Objetivo del Proyecto Genoma Humano
Anuncio
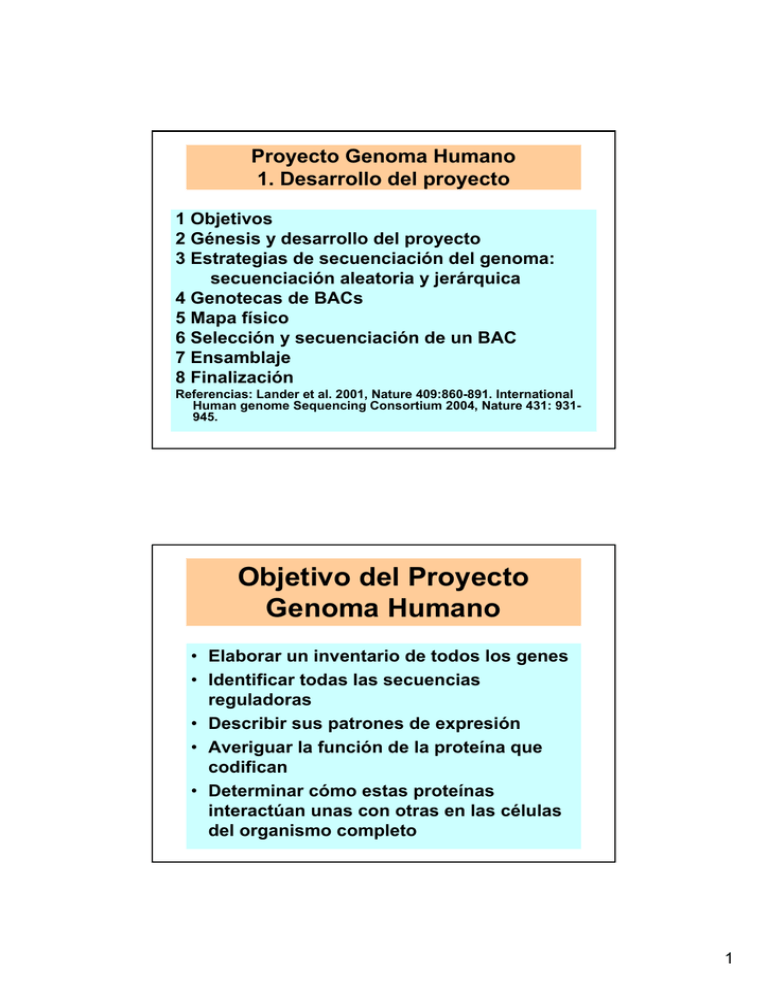
Proyecto Genoma Humano 1. Desarrollo del proyecto 1 Objetivos 2 Génesis y desarrollo del proyecto 3 Estrategias de secuenciación del genoma: secuenciación aleatoria y jerárquica 4 Genotecas de BACs 5 Mapa físico 6 Selección y secuenciación de un BAC 7 Ensamblaje 8 Finalización Referencias: Lander et al. 2001, Nature 409:860-891. International Human genome Sequencing Consortium 2004, Nature 431: 931945. Objetivo del Proyecto Genoma Humano • Elaborar un inventario de todos los genes • Identificar todas las secuencias reguladoras • Describir sus patrones de expresión • Averiguar la función de la proteína que codifican • Determinar cómo estas proteínas interactúan unas con otras en las células del organismo completo 1 El Proyecto Genoma Humano 1. Consorcio de 20 laboratorios públicos pertenecientes a 6 países. Liderado por F.S.Collins y E. Lander. 2. Discusión y debate en la comunidad científica 1984-1990. Iniciativa: Departament of Energy y National Institutes of Health (US). Comienzo del proyecto: 1990. Borrador: Oct 2000. Publicación: Feb 2001. Finalización: Octubre 2004. 3. Secuenciación aleatoria jerárquica (Hierarchical Shotgun Sequencing). 4. Material: DNA obtenido de donantes anónimos. La identidad de los donantes no es conocida (ni siquiera por ellos mismos). 5. Los datos se han hecho públicos a través de los bancos públicos de datos sin ninguna restricción a medida que se progresaba en el proyecto. 6. Publicación: Nature 409: 860-921 (15 febrero 2001); Nature 431: 931-945 (21 Octubre 2004). Celera Sequencing Project 1. Celera Genomics. Empresa privada de biotecnología que dirige J. Craig Venter y cotiza en bolsa. 2. Anuncio del proyecto: 1998. Comienzo de la secuenciación: 8 Sep 1999. Finalización de la secuenciación: 17 Jun 2000. Ensamblaje del borrador: 1 Oct 2000. 3. Estrategia: Secuenciación aleatoria del genoma (Wholegenome shotgun sequencing). 4. Material: Se reclutaron 21 donantes voluntarios. De ellos se seleccionaron 5 sujetos (dos hombres y tres mujeres): 2 caucásicos, un afroamericano, un asiático (chino) y un hispano (mejicano). 5. Condiciones para el acceso a los datos mediante acuerdo entre Science y Celera Genomics. Los datos está a disposición de los investigadores a partir de la fecha de publicación a través de la Web de Celera y con ciertas restricciones. 6. Publicación: Science 291: 1304-1351 (16 febrero 2001). 2 3 Características de una genoteca genómica • • • • Número de clones (N). Tamaño promedio (a) y varianza del inserto. Redundancia teórica (R = Na/b). Aleatoriedad (randomness): representación de secuencias genómicas diana. • Completa (completeness): si todas las secuencias diana en el genoma está representadas en la genoteca. • Fidelidad (fidelity): medida en que los insertos de los clones son copias fieles de las secuencias del genoma. P = 1 - (1 – a/b)N ≈ 1 – e-Na/b = 1 – e-R N = ln (1 – P)/ln (1 – a/b) R ≈ - ln (1 – P) donde N = número de clones; a = tamaño promedio del inserto; b = tamaño del genoma; R = redundancia teórica (Na/b); P = probabilidad de que una región dada esté incluida en la genoteca. 4 Shuttle vector pTARBAC2.1 (Zeng et al. 2001) Vector híbrido BAC/YAC que contiene: Centrómero y marcador His3 de levadura Replicón del plásmido F Gen de resistencia al cloranfenicol (CM-R) Gen sacB que codifica la levansucrasa (convierte la sucrosa en tóxico) Segmento de relleno pUClink eliminado durante la clonación Lugar de clonación flanqueado por primers universales T7 y SP6 RPCI = Roswell Park Cancer Institute, Buffalo (NY) Caltech= California Institute of Technology, Pasadena (California) 5 Genoteca ordenada: 8 x 12 = 96 pocillos 16 x 24 = 384 pocillos A B C D E F G H 1 2 3 4 5 6 7 8 9 10 11 12 Hibridación in situ de fluorescencia (FISH) de un paciente que presenta una translocación no-recíproca de un pequeño fragmento distal del cromosoma 10 en el cromosoma 9. (a) Sonda obtenida por microdisección del fragmento anormal del cromosoma 9. (b) Sonda procedente del brazo cromosómico 10p. 6 FISH en núcleos interfásicos 7 Análisis de la región del locus Rh mediante fiber-FISH. El locus Rh contiene 2 genes RHD y RHCE altamente homólogos. La hibridación con sondas de los intrones 3 (verde) y 7 (rojo) revela la presencia de deleciones y otras reordenaciones de la región. Figura 6.15. Dos métodos de cartografía física para detectar solapamiento entre clones genómicos 8 Ensamblaje de clones mediante fingerprinting Figure 6.2 Chain termination DNA sequencing (Sanger et al. 1977) 9 Secuenciación automática del DNA Secuenciación de un clon 100-200 kb • Fase 1. Secuenciación aleatoria – Construcción de una genoteca aleatoria en plásmido -> colección de clones 1-2 kb de tamaño promedio. – Secuenciación de uno o ambos extremos de un cierto número de clones -> colección de lecturas (“reads”). – Ensamblaje de las lecturas -> un cierto número de “contigs” con huecos (“gaps”) entre ellos. • Fase 2. Finalización (corrección de errores y rellenado de huecos mediante secuenciación dirigida) 10 Calidad de una secuencia (PHRAP score) • • • • P = Probabilidad de error de cada base Q = Calidad de una base Q = - 10 log P Al iniciar un proyecto de secuenciación es conveniente fijar cual es el objetivo: la calidad de la secuencia final a obtener. De ella depende la redundancia necesaria. • Q = 20, 30 y 40 corresponden a P = 1%, 0,1% y 0,01%. 10 11 Secuenciación de un clon 100-200 kb • Base-calling. PHRED permite obtener la probabilidad de error de cada base. • Ensamblaje. PHRAP permite ensamblar las lecturas en contigs. • Edición. CONSED permite visualizar el ensamblaje y la secuencia consenso así como calcular la probabilidad de error de cada base en la secuencia consenso. • Finalización. AUTOFINISH permite dirigir toda la operación de finalización basándose en la calidad de cada base. Borrador 7 Octubre 2000 • Cartografía. Ensamblaje de los clones BAC (por fingerprinting) en 1246 contigs. • Secuenciación y ensamblaje de 29.298 clones que representan un total de 4,26 Gb de secuencia. • Las secuencias brutas subyacentes suponen un total de 23 Gb (promedio 7.5x). 12 13 14 Fingerprint Sequenced- Gaps between clone contigs clone contigs draft clones El borrador del genoma humano (7-10-2000) representa el 88% de todo el genoma y un 93% de la eucromatina Genoma Humano (versión 21-Oct-2004) • Se han conseguido secuenciar 2.850 Mb (99% de la eucromatina). • La tasa de error es 1/100.000 bases (Q = 50). • Se ha reducido el número de “gaps” (huecos) de ~150.000 a sólo 341. • De ellos, 33 (total ~198 Mb) en la heterocromatina y 308 (total ~28 Mb) en la eucromatina. • Tamaño total: 2.850 + 198 + 28 = 3.080 Mb. • Número de genes: 20.000-25.000 (19.600 genes conocidos + 2.200 predicciones). • Pseudogenes: ~20.000. 15