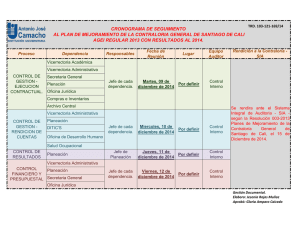
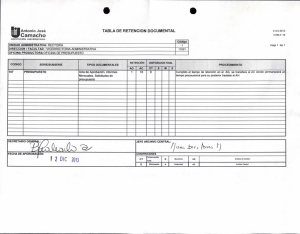
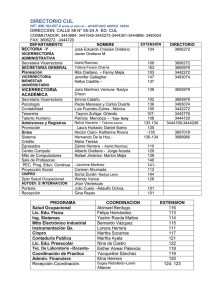
FORMULARIO DE PRESENTACIÓN Incentivo a la
Anuncio

FORMULARIO DE PRESENTACIÓN Incentivo a la consolidación de redes en la Universidad de Chile PROGRAMA U-REDES; Línea 2 PROYECTOS DOMEYKO II – UREDES 1. NOMBRE DE LA RED: BioMed-HPC: Red de Biología y Medicina Computacional de Alto Rendimiento 2. IDENTIFICACIÓN DEL JEFE DE PROYECTO O RESPONSABLE DE LA POSTULACIÓN Rut Nombre Dirección Facultad o instituto Centro, laboratorio o área Rango académico Teléfono E-mail Jerarquía y grado 21.323.545-1 Steffen Härtel Gründler Independencia 1027 Facultad de Medicina de la Universidad de Chile Laboratorio de Procesamientos de Imágenes (SCIAN-Lab) Centro de Espermiogramas Asistido por Internet (CEDAI SpA) Instituto de Neurociencias Biomédicas (BNI) Instituto de Ciencias Biomédicas (ICBM) Dr.rer.nat. 978 6366 [email protected] Profesor Asociado 3. a) IDENTIFICACIÓN DE ACADEMICOS PARTICIPANTES Nombre Universidad Facultad o Instituto Ricardo Verdugo (RV) [email protected] Ricardo Armisen (RA) [email protected] U-Chile ICBM, F-Med Centro Laboratorio o Área GENOMED Tipo de Jornada U-Chile ICBM, F-Med U-Cancer 44hr Conector U-Cancer Omar Orellana (OO) [email protected] U-Chile ICBM, F-Med 44 hr Conector ICBM Confirmará por email personal. Paulina Pino (PP) [email protected] Gonzalo Rojas Alcayaga (GR) [email protected] U-Chile 44hr U-Chile Salud Pública, F-Med FOUCH ICBM Microbiología Molecular Epidemiología Confirmará por email personal. Confirmará por email personal. Andrea Paula-Lima (AP) [email protected] U-Chile FOUCH Alejandro Maass (AM) [email protected] U-Chile FCFM, CMM LBMG 44hr Conector Salud Pública Conector Patología FOUCH Connector Microscopía FOUCH Conector Bioinf CMM Nancy Hitschfeld (NH) [email protected] U-Chile FCFM DCC 44hr Conectora DCC Confirmará por email personal. Eduardo Vera (EV) [email protected] U-Chile FCFM, CMM Director NLHPC 44hr Conector NLHPC Tomas Perez-Acle (TPA) [email protected] U-Chile FCFM, CMM Subdirector NLHPC 44hr Conector NLHPC Maria Luisa Cordero (MC) [email protected] U-Chile FCFM DFI 44hr Conectora Física / Microfluidos 44hr 44hr 44hr Función en la red BioMed-HPC Co-DIrector Firma Confirmará por email personal. Confirmará por email personal. Confirmará por email personal. 1 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Cristian Salgado (CS) U-Chile FCFM PMDC Lab 44hr Conector ICDB, PMDC Lab Connector FCQF U-Chile FCQF MMCM 44hr U-Chile FCQF Bioquímica 44hr Conector FCQF Romilio Espejo (RE) [email protected] U-Chile INTA OMICS 24hr Conector OMICS Sebastian Brauchi (SB) [email protected] UACh Instituto de Fisiologia Fisiologia Sensorial 44hr Conector UACh Bettina Müller (BM) [email protected] Convenio GOCCHI/ UChile GOCCHI Directora Ejecutiva GOCCHI 11hr Conector GOCCHI Confirmará por email personal. Luis Salinas (LS) [email protected] UTFSM CCTVal HPC 11hr Conector CCTVal Ver carta de apoyo. Juan Carlos Letelier (JCL) [email protected] U-Chile F-Ciencias Academico, Dpto de Biología 44hr Conector FCiencias Confirmará por email personal. Ricardo Cabrera (RC) [email protected] U-Chile F-Ciencias Bioquímica y Biología Molecular 44hr Conector FCiencias [email protected] Gerald Zapata (GZ) [email protected] Mauricio Báez (MB) [email protected] Confirmará por email personal. Confirmará por email personal. b) DESCRIPCIÓN DE INTEGRANTES EXTERNOS NACIONALES E INTERNACIONALES Integrantes Externos Nacionales (ver detalles de los integrantes en el Anexo I) REUNA La Corporación Red Universitaria Nacional (www.reuna.cl)... STI La Dirección de Servicios de Tecnologías de la Información y Comunicaciones de la U-Chile ... GOCCHI El Grupo Oncológico Cooperativo Chileno de Investigación (www.gocchi.org) agrupa ... CMM Centro de Modelamiento Matemático de la FCFM de la U-Chile (www.cmm.cl) ... OMICS El Centro Nacional de Genómica, Proteómica y Bioinformática 'OMICS Solutions' (www.omics-solutions.cl) ... ICBM Instituto de Ciencias Biomédicas de F-Med (www.icbm.cl) está constituido ... ESP La Escuela de Salud Públia de la F-Med reúne 53 académicos distribuidos en 4 divisiones ... CITC Centro de Investigación y Tratamiento del Cáncer, agrupa académicos de F-Med y del Hospital Clínico ... NLHPC El Laboratorio Nacional para Computación de Alto Rendimiento (www.nlhpc.cl) ... BNI El Instituto Milenio de Neurociencia Biomédica (www.bni.cl) formado en 2011 reúne ... FOUCH Facultad de Odontología (www.odontologia.uchile.cl) cobija al Instituto de Registro de Patología Oral ... CCTVal Centro Científico Tecnológico de Valparaíso (www.cctval.utfsm.cl) ... Integrantes Externos Internacionales (ver detalles de los integrantes en el Anexo I) Instituto Pasteur, Montevideo, Ury(www.pasteur.edu.uy) tiene el propósito de reforzar la integración ... Brown University, Providence, Rhode Island, USA (www.brown.edu). Tiene una exitosa experiencia ... GIGA, Grappe Interdisciplinaire de Génomique Appliquée, Liège, Bélgica (www.giga.ulg.ac.be) ... 4. ESTRATEGIA DE DESARROLLO COLABORATIVO PARA EL ESCALAMIENTO DE LA INVESTIGACIÓN Estrategia de BioMed-HPC hacia la referencia en I+D A nivel mundial, la Investigación de excelencia en el ámbito clínico, médico, biomédico y biológico depende en forma crucial de la capacidad de formar alianzas persistentes con disciplinas relacionadas a la matemática, computación e ingeniería. Instituciones emblemáticas en todo el mundo han respondido a está necesidad y desafío a través de la Creación de Centros o Institutos que persiguen misiones afines: (i) development of quantitative approaches for understanding the mechanisms and treatment of human disease through applications of mathematics, engineering and computational science (R. Winslow, 2 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Dir. of the Institute for Computational Medicine, J Hopkins University, www.icm.jhu.edu), (ii) creation of novel and impactful informatics and computationally-based methods, tools, algorithms, and resources to extend basic and clinical research capabilities and results (Department of Computational Medicine and Bioinformatics, U-Michigan), o (iii) usage of data and computational systems to make disease more preventable, illness more predictive and treatment more personalized (Computational Medicine Center, U-Cincinnati ), solo para nombrar algunos. Las disciplinas involucradas traspasan mundos, desde el ámbito de la Salud Pública (base de datos), Clínico/Hospitalario (sistemas de información), (Neuro)Ciencias Biomédicas (imágenes, bioinformática y biología computacional), Ciencias de Computación/Ingeniería (algoritmos), la Física hasta la Matemática (herramientas y modelos). Se reconoce que la creación del campo de la BioMedicina Computacional requiere un esfuerzo mayor a través de los años para generar una nueva cultura de trabajo desde las ciencias básicas hasta la investigación clínica, salud pública y la introducción de nuevos servicios en sistemas de salud. Hasta la fecha, Chile y la mayoría de los países Latinoamericanos no cuentan con respuestas adecuadas en este tema, principalmente por la falta inversión de fondos estratégicos con visión de mediano y largo plazo. Dentro de los pocos ejemplos positivos regionales está la reciente creación del Instituto de Investigación en Biomedicina de Buenos Aires CONICET - Instituto relacionado con la Sociedad Max Planck en Buenos Aires, dedicado a temas actuales de las biociencias, especialmente al ámbito de la investigación en biomedicina (www.mincyt.gov.ar/ministerio/polocientifico/ index.php). Si bien proyectos de está envergadura no son visibles aún en el escenario nacional, diferentes Facultades de la U-Chile (Facultad de Medicina, F-Med), la Facultad de Odontología (FOUCH), la Facultad de Ciencias y la Facultad de Ciencias Físicas y Matemáticas (FCFM) cuentan con investigadores(as) que requieren perspectivas similares para su desarrollo coherente y competitivo. Dentro de esta convicción, apuntamos en esta instancia a la formación de una Red de Biología y Medicina Computacional de Alto Rendimiento (BioMed-HPC) que persigue dos objetivos principales: A Crear conectividad de alta velocidad entre grupos de investigación que dependen en forma vital de la transferencia de grandes volúmenes de datos entre sus laboratorios y NLHPC/STI/ REUNA. B Incrementar la experticia en el manejo, storage and handling, de grandes volúmenes de datos biológicos y médicos y la implementación de algoritmos de simulación y análisis de datos en ambientes computacionales distribuidos de alto rendimiento. Antecedentes y fortalezas de investigación realizada por BioMed-HPC y sus miembros: Los integrantes de BioMed-HPC lideran (o participan en) proyectos nacionales e internacionales destacados de investigación (FONDECYT, Anillos, Núcleos e Institutos Milenio, FONDAP), Investigación y Desarrollo (FONDEF, Corfo), o formación de capital humano (MECESUP, DAAD, DFG) dentro de sus respectivas disciplinas. Representan líneas de investigación con alta productividad científica en los campos de la Bioinformática, Genómica, Genómica Funcional, Biología Computacional, Biología, Biofísica, entre otras. Sin embargo, la integración de las disciplinas que permite levantar proyectos asociativos de alta competitividad en el área de la biología y medicina computacional no existe hasta la fecha. BioMed-HPC desarrolla la excelencia en estas áreas sustentada en tres pilares disciplinarios: Análisis Computarizado de Imágenes, Genómica y Bioinformática, y Biología Computacional de Moléculas y Sistemas. Los laboratorios asociados son (ver detalle en Anexo II): Laboratorio Procesamiento de Imágenes Científicas SCIAN-Lab: Steffen Härtel SH, BNI/ICBM, F-Med, U-Chile Laboratorio de Genética de Sistemas y Genómica Biomédica (GENOMED): Ricardo Verdugo RV, F-Med, U-Chile Laboratorio de Microfluídica: María Cordero MC, Departamento de Física (DFI), FCFM, U-Chile Laboratorio de Biología Celular y Molecular (LBCM): Andrea Paula-Lima AP, FOUCH, U-Chile Laboratorio de Biología del Conocer: Juan Carlos Letelier, F-Ciencias, U-Chile Laboratorio de Bioquímica y Biología Molecular (LBBM): Ricardo Cabrera RC, F-Ciencias, U-Chile Laboratorio de Fisiología Sensorial: Sebastian Brauchi SB, F-Med, CAMI, UACh Valdivia Programa de Epidemiología del Instituto de Salud Pública: Paulina Pino PP, F-Med, U-Chile Modelación geométrica: Nancy Hitschfeld NH, Departamento de Ciencias de la Computación (DCC), FCFM , U-Chile Modelación y Mecánica Cuántica Molecular (MMCM): Gerald Zapata GZ, FCQF, U-Chile Laboratorio de plegamiento de proteínas: Mauricio Baez MB, FCQF, U-Chile 3 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Laboratorio de Modelación de Procesos y Computación Distribuida (PMDC Lab): Cristian Salgado CS, FCFM, ICDB, U-Chile Laboratorio de Bioinformática y Matemática del Genoma (LBMG): Alejandro Maass AM, CMM, FCFM, U-Chile Biología Computacional (DLab): Tomas Perez Acle TPA, NLHPC, CMM, FCFM, U-Chile Necesidad de conectividad de la red y oportunidad Un factor común al trabajo desarrollado por los miembros de la red es la utilización de datos cuantitativos y modelamiento matemático para entender/predecir procesos biológicos con relevancia en medicina. En la presente propuesta, agrupamos estas áreas temáticas dentro de la Biología y Medicina Computacional. En algunos casos, este proceso ya ha llevado al desarrollo de nuevas aplicaciones en el ámbito clínico, como el diagnóstico de infertilidad mediante procesamiento automatizado de imágenes y películas microscópicas a distancia (CEDAI Spa, SCIAN-Lab, SH) o el mejoramiento de procesos productivos e industriales tales como proyectos Genoma desarrollados en el LBMG (AM). Sin embargo, la consolidación y el futuro desarrollo de proyectos de investigación y desarrollo en biología y medicina computacional están en grave peligro por las barreras de infraestructura que impiden el desarrollo de colaboraciones necesarias. Este desafió no puede ser cubierto por proyectos individuales de los laboratorios o instituciones que forman y apoyan está red (ver tabla financiera) sino tiene que representar un esfuerzo común con el apoyo significativo de la iniciativa U-Redes. Mientras integrantes de F-Med, FOUCH, FCQF y F-Ciencias requieren experiencia matemática computacional, integrantes del FCFM buscan potenciar el modelamiento matemático asociado a problemas biomédicos. Se consideran campos con alto valor estratégico para I+D el modelamiento matemático del genoma y meta-genoma humano, descubrimiento de redes de interacción génica, modelamiento funcional de enfermedades, y descubrimiento de drogas. Está asociación permitirá a su vez generar sinergias con otras facultades e institutos con los cuales el CMM ya tiene lazos de larga data: como INTA, FCiencias, F-Agronomía, INRIA, entre otros. En segundo lugar, el NLHPC permitirá acceder a una capacidad de cómputos que no es factible alcanzar en las Facultades de la U-Chile. El NLHPC fue creado gracias al Programa de Equipamiento Mayor del PIA de CONICYT con el objetivo de estar al servicio de la comunidad académica y de la industria nacional para consolidarse como un referente de calidad de clase mundial con una oferta de servicios de excelencia para la investigación y desarrollo de HPC. Su capacidad de cómputo alcanzará sobre 3000 núcleos de procesamiento, lo cual excede del orden de 20 veces la capacidad HPC instalada. Es por tanto necesario lograr una buena conexión entre el NLHPC y las facultades de la U-Chile para potenciarse mutuamente en el campo de la biología computacional y matemática. Este proyecto U-Redes creará las condiciones necesarias. La propuesta BioMed-HPC busca fortalecer el desarrollo de investigación básica y aplicada en todas la áreas abarcadas por los miembros de la red, desde la física al cáncer o sistemas de información hospitalarios. Para alcanzar este objetivo, la estrategia de la red es: (i) Facilitar la gestión de proyectos colaborativos, sobre todo en aspectos financieros y organizacionales (ii) Asegurar el financiamiento necesario para mejorar la infraestructura de intranet en la U-Chile que permita el flujo de grandes cantidades de datos entre unidades académicas ubicadas en distintas facultades. El primer pilar se ejecutará a través de una Unidad de Operacional de la Red (financiada por U-Redes y contribuciones del ICBM, NLHPC, REUNA, STI, USLACRN). El segundo, mediante la organización de simposios y escuelas de verano (especialistas internacionales del amplio campo de la informática medica y HPC compartirán eventos con los integrantes de BioMed-HPC). En este contexto ya existe una tradición de eventos interdisciplinarios organizados: (i) ACCESS NOVA-meetings (EV) en el campo de redes avanzados, (ii) en el marco del Centro de Excelencia Heidelberg-Santiago en Informática Medica (coordinador académico SH) que generó summer schools, diplomados, simposio y cursos de postgrado en disciplinas en el interfase entre Informática, Biología y Medicina aún poco desarrollados en América Latina, o (iii) iniciativas nuevas de excelencia en el marco de la iniciativa NEUROSUR (BNI-Pastuer(Uy)), iniciada por científicos del BNI (U-Chile) que junta científicos de Argentina, Brasil, Chile y Uruguay por su asociación a centros o institutos de investigación, calidad científica y afinidad temática para fortalecer enlaces Latinoamericanos y definir estratégicas comunes en el diseño de plataformas científicas traslacionales. 5. OBJETIVO DEL PROYECTO DE CONSOLIDACIÓN DE RED a. OBJETIVO GENERAL DE LA CONSOLIDACIÓN DE LA RED BioMed-HPC El objetivo principal de BioMed-HPC es crear conectividad de alto nivel entre la F-Med, STI, REUNA, NLHPC/CMM/FCFM, GOCCHI/USLACRN y experticia en el manejo de grandes volúmenes de datos (storage and handling) provenientes de imágenes de origen biológico/médico y del campo de la genómica/proteómica. Está red (i) abre una perspectiva vital para proyectos vigentes y el desarrollo de futuras sinergias entre los académicos interconectados, (ii) genera un antecedente clave de competencias e infraestructura escalable para conectar otras Facultades de la U-Chile (FOUCH, FCQF, F-Ciencias) o el Hospital Clínico, y (iii) abre una perspectiva específica para desarrollar el campo de la Medicina Computacional dentro de la U-Chile en el interfase de jóvenes investigadores de los campos de las ciencias biomédicas, físicas, computación, bioinformática y neurociencias. 4 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Gracias a BioMed-HPC, se visualiza aumentar la productividad académica en todas sus facetas, basado en redes colaborativas y asociativas dentro de la Biomedicina Computacional con orientación en ciencia básica (FONDECYTS, Anillos, Núcleos Milenio) y investigación clínica aplicada (FONIS, FONDEF, CORFO). b. OBJETIVOS ESPECÍFICOS N° 1 2 3 4 Objetivo Específico Crear una estructura organizacional que facilite el funcionamiento de la red (corto plazo). Los académicos formarán el directorio de BioMed-HPC, quienes elegirán una directiva de 5 miembros. La directiva estará asesorada por un Consejo Asesor Nacional y Internacional y dispondrán de una Unidad Operacional, a cargo implementar las tareas de la red. Desplegar una infraestructura base y escalable que permita crear una conectividad con tecnología de punta, entre la FMed y el NLHPC (mediano plazo). En los primeros 6 meses, se procederá a ejecutar la instalación y puesta en marcha de una red fotónica de alta velocidad. Conectaremos a F-Med con el CMM por medio de una fibra dedicada a través del STI con la ayuda de equipos ópticos especializados que entregarán una capacidad inicial de 10Gbps. Está interconexión permitirá que otras facultades próximas (FOUCH, FCQF, GOCCHI y el Hospital Clínico) puedan conectarse a esta red. Mejorar la infraestructura de red de laboratorios computaciones en F-Med (mediano plazo). De forma paralela al objetivo 2, se adecuará parte de la red interna de la F-Med para soportar las altas velocidades de transferencia hacia y desde el CMM. Para ello se renovará equipamiento de la facultad para conectar, en un principio, los laboratorios SCIANLab y GENOMED al CMM a una alta velocidad. Está actualización de la red permitirá que en un futuro próximo, otros laboratorios y departamentos de la Facultad también puedan conectarse a la Red de alta velocidad en forma directa. Crear experticia en el manejo (storage and handling) de grandes volúmenes de datos en genómica e imágenes biomédicas a través de la organización de cursos de postgrado, simposios y escuelas de verano (mediano plazo) Organizaremos escuelas de verano en colaboración con el NLHPC, SCIAN, BNI y CCPVal para transferir conocimientos y experiencias sobre la implementación de análisis de datos en ambientes HPC distribuidos. Las clases estarán especialmente orientadas al análisis de datos genómicos, estructuras moleculares, e imágenes biomédicas. EL NLHPC ya tiene experiencia en la organización de eventos similares y SH (SCIAN, Dir. Académico del Centro de Excelencia en Informática Médica Santiago-Heidelberg. Este evento será apoyado/financiado con fondos de U-Redes (Ítem Capacitación, Difusión y Transferencia Tecnológica). La gestión de la organización de los eventos estará a cargo de la directiva del directorio y del Ing. en gestión de la Unidad Operacional. 5 Realizar transferencia y procesamiento de datos biológicos y biomédicos/clínicos a través de la red (mediano plazo) y elaborar protocolos de uso para futuros proyectos (mediano/largo plazo). Una vez que la conexión de alta velocidad entre la F-Med y el NLHPC sea establecida y mejoremos la conectividad interna en F-Med, realizaremos las primeras pruebas de transferencia de datos. La experiencia nos servirá para establecer protocolos de transferencia, manejo y almacenamiento de datos, uso de librerías de cómputos paralelos y utilización del software de administración de trabajos en el cluster. Una vez concluidos los estudios pilotos y los primeros protocolos, pasaremos a la etapa de producción que se extenderá a lo largo del proyecto y que nos permitirá mejorar nuestros protocolos y adquirir mayor experiencia que podrá ser compartida con todos los miembros de la red, colaboradores y participantes de las escuelas de verano. 6 Formulación de nuevos proyectos colaborativos en biomedicina computacional (mediano/largo plazo). La formulación de nuevos proyectos de investigación y desarrollo es una de las tareas más importantes del la red para asegurar su sustentabilidad en el tiempo. El recurso más importante de la red es su capital académico y profesional, quienes son expertos en sus respectivas áreas pero muchos de ellos nunca han desarrollado proyectos en conjuntos ya sea por limitantes técnicas o simplemente porque no se conocían. Este proyecto podrá dar solución a ambos problemas. Los objetivos 2-5 darán respuesta a las necesidades técnicas de conectividad y capacidad de realizar cómputos en ambientes HPC distribuidos. Para facilitar la interacción y colaboración académica, realizaremos al menos dos talleres por año para que todos los miembros de la red se conozcan, introduzcan su trabajo e intereses y trabajen en potenciales líneas de investigación colaborativa en grupos de interés común. 6. RESULTADOS ESPERADOS DIFERENCIAR LOS RESULTADOS DE CORTO, MEDIANO Y LARGO PLAZO Proyectos conjuntos, postulación a fondos concursables, generación de una nueva línea de investigación, productos servicios. 1. Consolidación de estructura de la red mediante organigramas propuestos (<6 meses, corto plazo). 2. Conexión intranet de 10Gbps entre F-Med y NLHPC (<6 meses, corto plazo). Esto crea un antecedente único en la región y facilitará el progreso en el desarrollo de algoritmos para analizar datos de alta relevancia biomédica en el campo de la visión computacional, análisis de estructuras 3D, imágenes biomédicos, servicios de teleanálisis, high throughput microscopy, bioinformática, salud pública, etc. 5 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE 3. Primeros ensayos y proyectos pilotos y creación de protocolos de uso de la red HPC (<1 año, corto y mediano plazo). El acceso al NLHPC abre un espectro amplio en el análisis de datos, la paralelisación de código, técnicas de análisis numérica de algoritmos para el uso optimizado del super-computo requiere protocolos que no existen hasta la fecha en el ámbito nacional. 4. Formación de capital humano a través de la organización de Cursos de Postgrado, Summer Schools y Symposia (siempre, corto-mediano-largo plazo). El eficiente uso de la infraestructura requiere ser acompañado por la generación de pericia en el manejo de software y programación para algoritmos de alta complejidad. A través de eventos que forman capital humano avanzado, Cursos de Postgrado, Summer Schools y Symposia, BioMed-HPC apunta a la generación de un futuro equipo capaz de afrontar los desafíos de I&D en el ámbito de la medicina computacional. 5. . Crear las bases académicas y tecnológicas para la formación de Anillos, Núcleos o Institutos Milenios en el campo de la Medicina Computacional (mediano-largo plazo). Este resultado destacaría la U-Chile, no solo en el ámbito nacional, sino también regional, más allá de la creación de resultados en investigación básica transferibles al sistema de salud Chileno en el mediano y largo plazo. a. CONCURSOS EXTERNOS A LOS QUE SE PRETENDE POSTULAR Siendo un proyecto de formación de investigación asociativa de excelencia de alto impacto, las capacidades de la investigación generada permite aplicar a todos los fondos accesibles dentro del espíritu de BioMed-HPC: FONDECYTS, FONDEF/IDEA, FONDEQUIP, Anillos de Investigación en Ciencia y Tecnología, Núcleos Milenio y Proyectos Internacionales. 7. ESTADO ACTUAL Y PERSPECTIVAS DE LA RED (PERTINENCIA Y POSICIONAMIENTO) a. PERTINENCIA DEL PROYECTO DOMEYKO II La propuesta de la red BioMed-HPC está en el corazón de los objetivos generales y específicos de U-Redes. BioMed-HPC se centra creando conectividad en si y en el manejo de la información científica y académica da alta demanda y complejidad. Los miembros de la red son académicos en diferente etapas de su desarrollo, aquellos que recién instalan sus laboratorios y conquistan sus primeros proyectos de investigación (RV, RA, AP, MC, CS, GZ, MB, RC), académicos que ya cuentan con laboratorios instalados y proyectos vigentes que generan datos y capacidades aisladas tanto en el área de la ciencia básica como ciencia aplicada o de interés estatal (SH, OO, PP, GR, AM, NH, EV, TPA, RE, SB, BM, LS, JCL), y entidades e institutos formados por académicos capaces de asumir el rol de plataformas tecnológicas en nivel nacional e internacional (CMM, BNI, Instituto Pasteur, CCTVal). La distancia física y administrativa entre las facultades de la U-Chile y por ende entre sus grupos de trabajo presentan un problema enorme para redes asociativos. Está brecha se cierre con BioMed-HPC en forma eficiente e elegante, creando una nueva cultura e estilo de trabajo académico que tanto se requiere para afrontar las necesidades vigentes en el área y la región. La oportunidad de BioMed-HPC consiste en crear por primera vez una conectividad de alta velocidad entre grupos de investigación que dependen en forma vital de la transferencia de grandes volúmenes de datos entre sus laboratorios y el NLHPC. Además se genera experiencia en el manejo (storage and handling) de grandes volúmenes de datos biológicos y médicos; y la implementación de algoritmos de simulación y análisis de datos en ambientes computacionales distribuidos de alto rendimiento. El impacto generado por los miembros y asociados se podrá medir en corto plazo (capacidad de almacenar por primera vez datos de proyectos vigentes en el ámbito de las imágenes digitales y la bioinformática), mediano plazo (creación de nuevos proyectos basados en procesamiento de datos masivos en ciencias básicas, aplicadas y de interés estatal) y largo plazo (solidificación del área de Medicina o Biomedicina Computacional hacia la creación de un nuevo campo de excelencia en este campo). Las participantes ya cuentan con trayectorias de excelencia en sus campos y forman partes de redes internacionales de primer nivel, en el caso de los grupos recién instalados, ellos tienen la mejor base académica, energía y visión para lograrla. No cabe ninguna duda que se realiza un impacto transcendente a través de BioMed-HPC. b. BREVE DESCRIPCIÓN DE HISTORIA Y ANTECEDENTES DE LA RED Y SUS MIEMBROS La red BioMed-HPC presenta una iniciativa sin antecedentes en el escenario nacional persiguiendo la visión de solidificar y posicionar el campo de la biomedicina computacional dentro de la escala internacional de investigación y desarrollo. En el pasado han habido iniciativas exitosas por iniciativa propia de los académicos para cerrar la brecha en forma paulatina y coherente. Entre SCIAN-Lab (F-Med, SH), DCC (NH) y LMBG (AM) se comparten alumnos de pre- y postgrado (uno de ellos premiado ´mejor tesis digital del año 2006´), y NH es co-investigadora por segunda vez en el Fondecyt regular de SH junto a jóvenes investigadores del CMM. En el marco del Centro de Excelencia Heidelberg-Santiago en Informática Médica (coordinador académico SH) colaboran intensamente AM (Matemática Aplicada, Bioinfomática), NH (Computación), PP (Salud Pública) generando escuelas de verano, diplomados, y un total de 20 cursos de postgrado novedosos en el ámbito de la informática médica. En este contexto AM y RV levantan dos cursos pioneros en el ámbito del Bioinfomática y PP en Epidemiología este semestre. Los primeros 7 alumnos del postulado Magíster en Informática Médica terminarán fines de 2013, un antecedente pionero en Chile y la mayoría de los países de América Latina. Cabe destacar que este esfuerzo se 6 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE levanta entre los participantes sin fondos de la U-Chile través del DAAD. Otro antecedente importante son los Simposios de AcessNova organizados por EV por más de una década. SH, AM, EV, JL, REUNA participan hace muchos años en este evento para el desarrollo de visiones dentro de conectividad, HPC y Massive Cientific Data Analysis en campos claves para la sociedad Chilena como minería y astronomía. Medicina y biología hasta la fecha no cuentan con fondos comparables, sin embargo el beneficio de sistemas de análisis remoto para servicios de salud en el campo de imágenes y los frutos de la investigación bioinformática con especial atención a iniciativas recientes en el ámbito de cáncer (ver U-Cancer, RA) o GOCCHI (BM) pueden, a corto y mediano plazo, crear servicios de excelencia para pacientes en el sistema de salud nacional. CEDAI SpA, un spin-off generado en consecuencia de un proyecto FONDEF de la F-Med, ha sido reconocido por la Secretaría General Iberoamericana con un Premio a la Innovación y el Emprendimiento que se entregará en Noviembre 2012 en el margen de la XXII Cumbre de Iberoamericana de Jefes de Estado y de Gobierno. Estos y otros esfuerzos pioneros de los miembros de BioMed-HPC en el pasado juntos a la perspectiva potente que se abre través de está iniciativa, no deja duda del impacto futuro en investigación y desarrollo de excelencia que tendrá el campo de la Biomedicina Computacional para la U-Chile y la región. c. SITUACION CON PROYECTO Y SIN PROYECTO La Red BioMed-HPC asegura la conectividad de alto nivel entre la F-Med (en cercanía a la FOUCH y la FCQF, el Hospital Clínico y GOCCHI) con el NLHPC del FCFM, REUNA y entidades nacionales e internacionales asociadas (ver arriba). Además generará expertise en el manejo (storage and handling) de grandes volúmenes de datos del campo de procesamiento de imágenes biológicas, biomédicas y el campo de la informática. Se realizarán transferencias y procesamientos de datos biológicos y biomédicos/clínicos a través de la red y se elaborarán protocolos de uso para futuros proyectos colaborativos de alta competitividad en el campo de la biomedicina computacional, imposible de levantar sin conectividad de primer nivel. Sin proyecto, el campo de la biomedicina computacional no se desarrollará, proyectos vigentes de I+D dentro del campo del teleanálisis médico no son factibles ya que no es posible replicar una plataforma HPC del NLHPC en cada unidad donde se generan datos de alto volumen que deben ser procesados. Investigadores jóvenes pierden un perspectiva vital para su futuro desarrollo y la competitividad de la U-Chile en este sector se sacrifica. A modo de ilustración, se presenta una ejercicio de cálculo de requerimientos de conectividad de un proyecto genoma. Un genoma humano secuenciado a una profundidad de 30x genera alrededor de 28.8 TB de datos en imágenes que deben ser almacenadas y procesadas para obtener entre 90 GB y 1.98 TB en secuencias cortas (reads), dependiendo de la cantidad de información que se guarde. La primera etapa suele hacerse en el lugar de secuenciación pero la segunda en un centro de cómputos que puede estar en la del CMM de Ingeniería o en la F-Med, dependiendo del proyecto. Suponiendo que se produjeran 100 GB por genoma, el traslado de datos por una conexión estándar de 100Mbps (equivalente a unos 7 Mega Bytes por segundo en la práctica) tomaría unas 4 hrs. Por lo tanto, un proyecto pequeño que secuencie 18 genomas tomaría 3 días solo en transferencia. Esto además tiene el problema que cualquier interrupción en la conexión puede arruinar los archivos, generando errores posteriores o necesitando repetir todo el proceso. Usando la tecnología de 1000 Gbps de red, el mismo proyecto de 18 genomas se transferiría en 4 min. En el escenario actual, los datos de secuenciación son producidos en OMICS y transportados mediante un disco duro externo hacia el CMM y luego de un análisis inicial, nuevamente en disco duro externo desde el CMM hasta GENOMED en F-Med. Este esquema de transporte de datos es obviamente inviable para proyecto de mayor envergadura. La situación es aún más dramática en aplicaciones que requieren procesamiento de datos en tiempo real. Por ejemplo, en el análisis de imágenes médicas para producir un diagnóstico y así guiar una decisión terapéutica. El traspaso de 10TB/semana de datos que están estimados para los proyectos SCIAN/BNI arriba descritos, requerirían 2.4 semanas solo para transferirse al clúster del NLHPC, sin contabilizar el tiempo que tomaría el procesamiento y el retorno de los resultados. En otras palabras, estos proyectos serían inviable con la situación actual. d. DESCRIPCIÓN Y DIAGRAMA DE SUSTENTABILIDAD DEL PROYECTO DOMEYKO II Modelos de asociación La red BioMed-HPC combina experticias complementarias en los campos biológico/médicos y computacionales. Esto queda evidenciado en el perfil de los integrantes, que incluyen miembros de varias facultades con diferentes competencias (ver Anexo 1). BioMed-HPC también estará asociado a organizaciones relevantes en el ámbito nacional e internacional que le permitirán orientar sus políticas estratégicas de desarrollo asegurando un posicionamiento de excelencia en el mediano plazo. Adicionalmente, BioMed-HPC contempla el asesoramiento técnico necesario, personal contratado y externo, para la implementación de una infraestructura sólida de tecnología de punta que sustente su funcionamiento y crecimiento en el largo plazo (ver diagrama). 7 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Sustentabilidad y Escalamiento de la Red El Directorio Académico estará a cargo de la planificación estratégica de la red, la identificación de áreas de desarrollo y la formulación de proyectos científicos y de I+D que sustentan la red. Los proyectos nuevos adjudicados gracias a la nueva infraestructura y soporte técnico creados a través de BioMed-HPC garantizarán la continuidad de la red en el futuro. La inversión inicial de BioMed-HPC permite reducir costos de inversión y mantención en el futuro para todos sus integrantes: (i) ahorro de infraestructura replicada, (ii) compartir personal calificado (iii) conectarse a la red en F-Med con protocolos establecidos. Dado el gran número de grupos participantes y los futuros miembros, los costos de mantención resultan fácilmente financiable a través de futuros proyectos (ver 6.2). A través de BioMed-HPC, tareas operacionales y administrativas serán asumidos por personal del Departamento de Informática (F-Med), STI, y NLHPC. De esta manera, los costos para el mantenimiento de la fibra óptica (500M$/mes) y el salario de personal técnico (1.500M$/mes) son financiables fácilmente por los proyectos de los miembros de la red. 8. PLAN DE TRABAJO DEL PROYECTO DOMEYKO II Año 1: Hito 1: Contratación de profesionales de la red (03.2013) Contrataremos 3 personas para integrar la Unidad Operacional. Las tareas financieras y secretariales estarán a cargo de un Ing. en gestión a ser contratado por media jornada en el ICBM. Además, se contratarán 22 hrs. de un Ing. en telecomunicaciones (STI/ICBM) y 44 hrs. de un administrador de sistemas /programador (NLHPC/ICBM). Estos interactuarán directamente con ingenieros del NLHPC para la implementación de rutinas de análisis de datos en el clúster de cómputos y la elaboración de protocolos, lo cual permitirá facilitar el acceso a los recursos computacionales a investigadores en laboratorios del ICBM sin experiencia en uso de equipamiento HPC. Este hito requerirá el aporte de 13.200M$ por parte del VID (U-Redes). Hito 2: Creación de un directorio académico y su directiva (06.2013). Se organizarán reuniones y talleres para identificar las problemáticas científicas que pueden beneficiarse de una aproximación computacional y de trabajos colaborativos entre los miembros de la red y colaboradores externos. También se desarrollarán las estrategias para facilitar al máximo la comunicación entre académicos con distintas formaciones para generar una cultura de red interdisciplinaria. Hito 3: Conexión de alto rendimiento entre F-Med y CMM (12.2013) REUNA instalará el switch óptico en F-Med e interfaces ópticas en nodo CMM y STI. Las actividades de instalación, ingeniería y gestión de los equipos serán financiadas con fondos de REUNA (10.000M$). Los equipos tendrán un costo total de 30.000M$ y serán financiados en partes iguales por VID (U-Redes ), STI, y F-Med. La instalación se hará durante los primeros 4 meses de proyecto, y las pruebas y puestas a 8 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE punto se realizarán a lo largo del primer año. Este hito requerirá el aporte de 12.000M$ por parte del VID (U-Redes) por concepto de mantención de la fibra óptica por 2 años (pagos mensuales de 20UF). Hito 4: Conexión de alto rendimiento entre laboratorios computacionales F-Med (12.2013) Los equipos del SCIAN y GENOMED lab en conjunto con la Unidad de Operacional realizarán un mejoramiento de la red interna F-Med para permitir que estos laboratorios puedan conectarse a 1Gbps entre si y con el switch fotónico de F-Med que permite el ingreso a la red de 10Gbps con el CMM. La red interna F-Med una vez mejorada, permitirá la formación de una primera grid de cómputos que luego podrá ser expandida a medida que nuevos laboratorios computacionales sean formados en F-Med u otras facultades en el campus norte (HCUCH, FOUCH, FCQF). Año 2: Hito 5: Primera escuela de verano de BioMed-HPC (12.2014). El Directorio Académico en colaboración con el NLHPC, SCIAN, BNI y CCPVa, y apoyados por el Ingeniero en Gestión, organizarán una escuela de verano para transferir conocimientos y experiencias sobre la implementación de análisis de datos en ambientes HPC distribuidos. Se dará prioridad a los miembros de la red para facilitar su acceso a los recursos computacionales de los que dispone. La organización de estos dos eventos tomará tiempo a lo largo de los años 1 y 2 para realizar contactos con expertos nacionales e internacionales. El trabajo administrativo será más intenso 2 meses antes de cada escuela, pero al estar acoplados a actividades ya andando (Cursos del NLHPC y Ms. Informática Médica U-Chile/U-Heidelberg) se facilitará la gestión. Este hito requerirá el aporte de 10.000M$ por parte del VID (U-Redes). Hito 6: Segunda escuela de verano de BioMed-HPC (12.2014). Al final del proyecto realizaremos la segunda escuela de verano. A diferencia de su primera versión, esta escuela tendrá énfasis en potenciales usuarios de cómputos HPC que no necesariamente tengan experiencia en este tipos de recursos y que puedan no haber sido miembros de BioMed-HPC. Este hito requerirá el aporte de 10.000M$ por parte del VID (U-Redes). Hito 7: Estudios pilotos y generación de protocolos BioMed-HPC (12.2014). La Unidad Operacional trabajará en la elaboración de protocolos de almacenamiento, transferencia y análisis de datos en el cluster del NLHPC y la grid de F-Med. Una primera versión estará disponible antes de la primera escuela de verano (diciembre 2013) y una segunda versión antes de la segunda escuela (diciembre 2014). Hito 8: Postulación a fondos concursables externos (12.2014). Será tarea constante de los miembros del Directorio Académico, la formulación de proyectos científicos y de innovación a fondos concursables externos. Ver sección CONCURSOS EXTERNOS A LOS QUE SE PRETENDE POSTULAR. a. CARTA GANTT Objetivos específicos (OE) OE 1 Estructura organizacional/contrataciones OE 2 Conectividad CMM / F-Med OE 3 Adecuación red F-Med 2013 E S G N L F S G N L M A S S G G N N L L T I R T I R M S G N L T I R T I R J S G N L T I R T I R 2014 J A S O N D T I R T I R T I R T I R T I R T I R T I R T I R E F M A M J J A S O N D OE 4 Transferencia tecnológica S G N L D D OE 5 S S S S S S S S S S S S S S S S S S Estudios pilotos / formulación G G G G G G G G G G G G G G G G G G de protocolos L L L L L L L L L L L L L L L L L L N N N N N N N N N N N N N N N N N N D D D D D D D D D D D D D D D D D D OE 6 S S S S S S S S S S S S S S S S S S S S S S S S Formulación de proyectos G G G G G G G G G G G G G G G G G G G G G G G G N N N N N N N N N N N N N N N N N N N N N N N N L L L L L L L L L L L L L L L L L L L L L L L L S: SCIAN, G: GENOMED, N: NLHPC, L: LBMG, R: REUNA, T: STI, I: Departamento Informática F-Med, R: Ingeniero en redes informáticas de GenoMed-HPC, D: Unidad Operacional 9 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE 9. DESCRIPCIÓN DE RECURSOS SOLICITADOS VID Valor M$Ch Recursos Humanos: Unidad Operacional: Ing. Redes Computacionales Programador/Administrador % % Total Valor M$Ch % 4.800 /5h 25 9.600 /11h,STI 4.800 /5h, ICBM 50 25 19.200/22h 100 8.400 /11h 25 16.800 /22h, NLHPC 8.400 /11h USLACRN 50 25 33.600/44h 100 9.600 /11h, CCTVal 100 9.600 /11h 100 16.800 /22h, ICBM 66.000 100 82 16.800 $Ch/22h 79.200 100 100 10.000, REUNA 100 10.000 , REUNA 100 10.000 100 10.000 100 Ing. Conexión UTFSM Géstor y Secretaria Subtotal SOCIOS/MANDANTES Valor M$Ch 13.200 18 Subcontratos Instalación red fotónica Subtotal Capacitación, Difusión y Transferencia Tecnológica Simposio / Escuela de verano 20.000 100 20.000 100 Subtotal 20.000 100 20.000 100 Gastos de Inversión: Cable Equipos, STI hasta F-Med 10.000 33 Equipos (F-Med interno) Equipos (BNI interno) Equipos (SCIAN-Lab diente A) Subtotal 10.000, F-Med 10.000, STI 33 33 30.000 100 15.000, F-Med 10.000, FOUCH 80 20 25.000 100 50.000, BNI 10.000, SCIAN 100 100 50.000 10.000 100 100 10.000 27 105.000 73 115.000 100 7.000 58 5.000, F-Med 42 12.000 100 Subtotal 12.000 100 12.000 100 TOTAL 50.200 21 236.600 100 Gastos de Operación: Mantención Fibra Óptica 186.400 79 Recursos Humanos Unidad Operacional Ing. Redes Computacionales: Encargados de la coordinación entre las partes para la creación de la red fotónica. Programador/Administrador: Encargado de proveer servicio de apoyo, mantención y operación para el uso de HPC por los miembros de la red. Ing. Conexión UTFSM: Encargada de la coordinación de la conexión de la UTFSM a la red fotónica de REUNA. Géstor / Secretaria: Encargado/a de la gestión, coordinación y comunicación de los eventos de la Red BioMed-HPC, incluyendo no sólo el simposio / escuela de verano, sino también la difusión y transferencia tecnológica a los miembros de la red. Subcontratos Instalación de equipos, ingeniería y gestion red optica: REUNA instalará el switch optico en F-Med e interfaces ópticas en nodo CMM y STI, así como la ingeniería de red respectiva para establecer la conexión F-Med/CMM y F-Med/STI. Realizará también por el periodo de duración del proyecto, en conjunto con STI, la administración de los equipos ópticos, así como la supervisión del enlace físico entre F-Med y STI de manera de gestionar alguna acción del proveedor de Telecom cuando sea necesario. Las actividades de instalación, ingeniería y gestión de los equipos serán financiadas con fondos de REUNA (10.000M$). Difusión y Transferencia Tecnológica Simposio / Escuela de verano: Organizaremos dos simposios o escuelas de verano para la transferencia de la experticia en el manejo y procesamiento (storage and handling) de grandes volúmenes de datos provenientes del campo de la bioinformática en un ambiente de computación de alto rendimiento (HPC). Estos eventos están destinados principalmente a miembros de la red a colaboradores que puedan beneficiarse de los recursos y competencias disponibles en BioMed-HPC. Gastos de Inversión Cable Equipos, STI hasta F-Med: La conectividad entre STI y F-Med(Unidad de Informática) se financiará con inversiones del STI y U-Redes (VID) (Anexo I) Equipos (F-Med interno): La conectividad F-Med(Unidad de Informática), SCIAN-Lab (Diente A) y GENOMED (Diente C) se financiará con inversiones de la F-Med ( Anexo II - Unidad de Informática) Equipos (BNI interno): La conectividad SCIAN-Lab (Diente A) y el nuevo edificio del BNI y sus 11 investigadores se financiará con inversiones del BNI ( Anexo III BNI) Equipos (SCIAN-Lab interno) La conectividad interna de 10 GIGA SCIAN-Lab (Diente A) se financiará con inversiones del SCIAN-Lab (Anexo II) Gastos de Operación Gasto Arriendo F.O. (por 2 años): Gasto de arriendo y mantención de la Fibra Obscura entre F-Med y STI ubicada en Torre 15, necesaria para mantener el enlace de alta velocidad fotónico en funcionamiento. 10 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Nombre del Postulante Steffen Härtel Gründler RUT: 21.323.545-1 Fecha de presentación: 10.09.2012 Firma 11 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE LISTA DE ANEXOS: INTEGRANTES Anexo I. DESCRIPCIÓN DE INTEGRANTES EXTERNOS NACIONALES E INTERNACIONALES Anexo II. DESCRIPCIÓN DE LABORATORIOS DE MIEMBROS PARTICIPANTES CARTAS DE COMPROMISO NACIONAL Anexo III. Carta_Compromiso_ICBM_Facultad_de_Medicina_BioMed-HPC Anexo IV. Carta_Compromiso_CITC_BioMed-HPC Anexo V. Carta_Compromiso_Luis_Salinas_Valparaiso_BioMed-HPC Anexo VI. Carta_Compromiso_CCTVal-UTFSM Anexo VII. Carta_Compromiso_BNI_BioMed-HPC Anexo VIII. Carta_Compromiso_F-Med_BioMed-HPC Anexo IX. Carta_Compromiso_FOUCH-HPC Anexo X. Carta_Compromiso_NLHPC-HPC Anexo XI. Carta_Compromiso_GOCCHI-HPC Anexo XII. Carta_Compromiso_STI-HPC Anexo XIII. Carta_Compromiso_SCIAN-HPC Anexo XIX. Carta_Compromiso_REUNA-HPC CARTAS DE COMPROMISO NACIONAL Anexo XX. Presupuesto red interna F-Med 10[Gbps] Anexo XXI. Presupuesto Red Óptica STI-F-Med CARTAS DE APOYO INTERNACIONAL Anexo XXII. Carta_Apoyo_GIGA-HPC Anexo XXIII. Carta_ Apoyo _Pasteur-HPC Anexo XXIV. Carta_ Apoyo _Brown-HPC 12 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Anexo I DESCRIPCIÓN DE INTEGRANTES EXTERNOS NACIONALES E INTERNACIONALES Integrantes Externos Nacionales REUNA gestiona la infraestructura digital de Red Académica de investigación, educación e innovación en Chile. La red interconecta a 17 Universidades, CONICYT, los principales observatorios en Chile (AURA, ESO, ALMA) y mediante alianzas a instituciones como Fundación de Ciencias para la Vida, INAPI, entre otras. La red tiene 11 puntos de presencia, entre Arica a Osorno, con una capacidad que va entre 0,155-2,5 Gbps, está última será prontamente ampliado a 1 Gbps. REUNA interconecta Redes Académicas Internacionales en América Latina (RedCLARA), América del Norte (Internet2 /Canarie), Europa (GÉANT), Asia (APAN) y Oceanía (AARNET). REUNA ha liderado proyectos de infraestructura digital desde sus orígenes, y recientemente (2010) establece una red óptica de alta velocidad entre Antofagasta y Santiago (7 nodos). En vinculación con el proyecto NLHPC - se encuentra implementando una ampliación de la red óptica en Santiago que integrara la U-Chile (CMM, STI), PUC, USACH, que permitirá la interconectividad de alta capacidad (hasta 32 canales de 10Gbps cada uno). STI La Dirección de Servicios de Tecnologías de la Información y Comunicaciones de la U-Chile busca introducir nuevos sistemas y prácticas para apoyar gestión y realización más eficiente y eficaz de diversos organismos de la Universidad. Actualmente tiene la responsabilidad de administrar la red de la Universidad. Una de sus prioridades estratégicas, busca impulsar una red experimental dedicada a la investigación entre las distintas facultades, siendo este proyecto un primer paso para conectar la FCFM con la F-Med y a futuro el resto de las facultades, conformando una red académica avanzada que cumpla con las crecientes necesidades de los académicos e investigadores de la Universidad. GOCCHI El Grupo Oncológico Cooperativo Chileno de Investigación (www.gocchi.org) agrupa investigadores a lo largo de Chile en especialidades médicas relacionadas con el tratamiento del cáncer. GOCCHI planifica y coordina estudios clínicos oncológicos nacionales e internacionales y busca promover y desarrollar la investigación oncológica en Chile, siguiendo rigurosamente la metodología científica y las Buenas Prácticas Clínicas en investigación. Tiene especial interés en promover y optimizar relaciones entre los centros de atención e investigación oncológica, privados y públicos y entre estos y el Ministerio de Salud y las universidades. A modo de ejemplo, GOCCHI coordina el estudio 'Perfil Molecular de Cáncer de Mama en Estadio Clínico II y III en Mujeres Latinoamericanas que Reciben Tratamiento Estándar de Referencia' del US Latinamerica Cancer Research Network (USLACRN) financiado por el National Cancer Institute de EE.UU y por aportes gubernamentales de Chile y que busca determinar los perfiles moleculares de los subtipos de cáncer de mama, detectando la prevalencia de cada uno en mujeres latinoamericanas. Este proyecto generará datos de genotipificación y expresión génica por microarreglos de 2000 pacientes enroladas en 5 países participantes (400 en Chile), los cuales requerirán de una gran capacidad de cómputos y competencias en modelamiento matemático e informáticos para ser analizados. Mediante la incorporación de GOCCHI en la red podremos cubrir está necesidad y sus miembros tendrán oportunidad de diseñar estudios en biología y medicina computacional basados en datos masivos de estudios clínicos. CMM Centro de Modelamiento Matemático de la FCFM de la U-Chile. La misión del CMM es crear nuevas matemáticas y usarlas para resolver problemas de otras ciencias, la industria y las políticas públicas. Su finalidad es desarrollar ciencia con los más altos estándares, los que guían asimismo sus actividades en investigación industrial y educación. Desde su inauguración, el CMM se a convertido en un centro de excelencia de clase mundial en investigación y formación avanzada en matemáticas aplicadas, reconocido internacionalmente como una plataforma para el modelamiento matemático industrial con alto impacto en innovación. En el ámbito de las instituciones, las principales contrapartes son el Centre National de la Recherche Scientifique (CNRS), la Universidad de Concepción (UDEC), y varios departamentos de la U-Chile. Las principales contrapartes industriales iniciales incluyen a la Corporación Nacional del Cobre (CODELCO) y a Dassault Aviation. Dentro del CMM, el Laboratorio de Bioinformática y Matemática del Genoma (LBMG) liderado por el Dr. Alejandro Maass, ha sido líder en el desarrollo de la genómica y bioinformática a nivel nacional y ha consolidado una red de investigación con múltiples organismos que incluyen el INTA, F-Med, F-Ciencias, F-Agronomía, INRI y FONDAP Center for Genome Regulation entre otros. OMICS El Centro Nacional de Genómica, Proteómica y Bioinformática 'OMICS Solutions' fue creado para promover el uso de la ciencias genómicas en Chile. El proyecto, financiado por CONICYT es patrocinado por la U-Chile, PUC, U-Andrés Bello, U-Talca y el Instituto Nacional de Investigaciones Agropecuarias. El Centro ha implementado un Laboratorio altamente especializado para prestar servicios y asesoría a la comunidad científica y profesional en las metodologías genómicas. Recientemente ha finalizado la instalación de cinco modernas plataformas de secuenciación masiva de ADN, que permiten conocer en detalle la composición genética de organismos y poblaciones, incluyendo humanos. Un Centro HPC beneficiaría grandemente a OMICS pues los datos obtenidos en cada corrida en cualquiera de estas plataformas pueden alcanzar volúmenes de terabytes que requieren capacidades especiales para trasmisión. 13 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE ICBM Instituto de Ciencias Biomédicas de F-Med está constituido por 240 académicos agrupados en nueve Programas Disciplinarios que orientan el desarrollo de las siguientes disciplinas: Biología Celular y Molecular, Farmacología Molecular y Clínica, Fisiología y Biofísica, Genética Humana, Inmunología, Microbiología y Micología, Anatomía y Biología del Desarrollo, Fisiopatología y Virología. El ICBM aspira a la excelencia en el cumplimiento de las funciones académicas de docencia, investigación y extensión o servicio a la comunidad. Las políticas del ICBM estimulan la colaboración de estos distintos grupos disciplinarios en todas las tareas académicas y para facilitar estas interacciones existen también otras instancias de asociación en torno a problemas relevantes de la biomedicina. Es en este contexto que el ICBM ha determinado que la Biología de Sistemas y la Bioinformática son áreas de gran relevancia para el futuro de la medicina que deben ser apoyadas y desarrolladas al interior de la institución. Está política se cristaliza mediante nuevas contrataciones de de académicos con perfiles afines y mediante el apoyo para la adquisición y renovación de infraestructura que esté de acuerdo a las necesidades de proyectos de este tipo. En particular, la reciente incorporación del Dr. Ricardo Verdugo y la contraparte financiera comprometida para la presente propuestá dan cuenta de la voluntad del ICBM para desarrollar su visión de excelencia en Biología y Medicina Computacional. ESP La Escuela de Salud Públia de la F-Med reúne 53 académicos distribuidos en 4 divisiones en las áreas de Epidemiología, Políticas, Promoción de Salud y Bioestadística. Actualmente se encuentra en etapa de transición a Instituto, lo que implica una reorganización en torno a programas temáticos que incluirán: Salud Ambiental, Salud Global y Modelación y Medición en salud entre otros. La ESP es el principal centro formador en esta área, a través de programas de postítulo (Especialidad Médica) y postgrado (Diplomas, Magíster y Doctorado). El Instituto aspira a constituirse en un motor de la investigación nacional mediante la realización de proyectos epidemiológicos y de estudios sociológicos presentados a los concursos de proyectos nacionales e internacionales. Sin embargo, un área crítica que puede dar gran impulso a la investigación en esta área, la constituyen los estudios realizados con datos secundarios, de estadísticas vitales, registros hospitalarios y de atención primaria, de la atención de enfermedades GES, de enfermedades de notificación obligatoria, imágenes, exámenes de laboratorio y muchas otros registros de interés en el país. El desarrollo de un área de informática biomédica en este ámbito permitirá contribuir a potenciar estos registros mejorando su calidad, integralidad y oportunidad- y sobre todo, a propiciar su conectividad de forma que se pueda estudiar a grades cohortes de nacimientos y su evolución a través de registros consecutivos en su curso de vida. El potencial de un escenario como ese para la investigación epidemiológica y para la salud pública es enorme. De hecho, una de las hipótesis más prolíficas en la investigación sobre el origen de las enfermedades crónicas surgió de la relativamente reciente capacidad de relacionar datos de nacimiento con los de mortalidad mediante aplicaciones informáticas. A lo menos tres académicos del Programa de Epidemiología del instituto estarán involucrados en el desarrollo de proyectos relacionados con esta área de la informática médica. CITC Centro de Investigación y Tratamiento del Cáncer, agrupa académicos de F-Med y del Hospital Clínico de la U-Chile. La misión del CITC es promover y potenciar el desarrollo de investigación en el área del cáncer, aplicando una estrategia integrada entre el ámbito clínico y básico, instalando un polo de Investigación Traslacional y Medicina Personalizada para el diagnóstico y tratamiento del Cáncer. Además se espera colaborar de manera significativa en la formación de recursos humanos para la investigación, diagnóstico, tratamiento y prevención del Cáncer. Dentro de su visión de desarrollo estratégico, el centro apoya investigación que apunte al 'Desarrollo de Epidemiología Molecular y Genética en Cáncer.' Mediante la incorporación del CITC en BioMed-HPC, se busca potenciar el alcance de ambos proyectos. Por un lado, los investigadores de BioMed-HPC tendrán oportunidad de diseñar proyectos que mediante la genómica y biología computacional apunten a mejorar el diagnóstico y tratamiento del cáncer usando muestras de pacientes nacionales. El CITC, a su vez, podrá desarrollar una área nueva de investigación dentro del paraguas multidisciplinario que conforman los miembros del Centro. El CITC estará representado por su Dir. Dr. R Armisen. Vale mencionar que este centro ha sido producto de una concurso U-Redes anterior que hizo posible la creación de 'U‐CANCER: Red de Medicina Trasnacional en Cáncer', cuyos miembros formaron recientemente el CITC. NLHPC El Laboratorio Nacional para Computación de Alto Rendimiento (www.nlhpc.cl) es un proyecto del CMM financiado por el Primer Concurso de Centros de Servicios de Equipamiento Mayor de uso Compartido, coordinado por el Programa de Investigación Asociativa de CONICYT. El NLHPC instalará en Chile una capacidad de HPC que permita satisfacer la demanda nacional por computación científica de alto rendimiento, ofreciendo servicios de alta calidad y promoviendo su uso en problemas tanto de investigación básica y aplicada, así como en aplicaciones industriales. Durante los últimos años, el desarrollo de las ciencias aplicadas y de la innovación industrial, han sido dirigidos por el uso sofisticado de las tecnologías de información y comunicaciones (TIC), proceso en el cual las tecnologías de HPC han jugado un rol preponderante. En Chile, algunas áreas de la ciencia así como ciertos sectores industriales, han alcanzado un nivel de madurez tal que, para mantener su competitividad a nivel global, se requiere del uso de tecnologías relacionadas con el HPC. Identificando las oportunidades que la disponibilidad de esta tecnología brindará al país, la mayoría de las universidades de investigación de Chile, lideradas por el CMM de la U-Chile, propusieron en 2010 a CONICYT la creación del NLHPC. El NLHPC está conformado por la U-Chile como Institución Responsable y como Instituciones Asociadas (AI) la PUC, UTFSM, USACH,UFRO, U-Talca y UCN, junto a REUNA. El NLHPC consolidará una 14 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE infraestructura de HPC en forma distribuida entre las AI, quedando instalado el nodo central de procesamiento en el CMM de la FCFM, un centro de excelencia con gran trayectoria en la gestión de proyectos colaborativos. El NLHPC contribuirá a este proyecto a través de la dedicación de media jornada de un ingeniero, destinado a atender los requerimientos que provengan de los laboratorios miembros de la red. El acceso a nuevos problemas provenientes de otras ciencias, así como el establecimiento de una comunidad en torno al HPC será uno de los principales beneficios para el NLHPC. BNI El Instituto Milenio de Neurociencia Biomédica (www.bni.cl) formado en 2011 reune a once investigadores principales en la F-Med en torno a equipamiento de punta para realizar investigación científica de excelencia y contribuir a la formación de capital humano avanzado. BNI propone una exploración integrada de la organización dinámica estructural y funcional del sistema nervioso en condiciones fisiológicas y patológicas, utilizando diversos modelos experimentales desde organismos completos hasta la célula. Dentro del BNI se generó la Plataforma BioMat, un grupo y equipamiento dedicado a la biomatemática-computacional que contribuye a una comprensión más profunda de los principios arquitectónicos y funcionales de los procesos neuronales, desde niveles sub- a supra-celulares. La biofísica y las matemáticas aplicadas en combinación con imágenes avanzadas y la informática fomentan una visión integrada para estudiar el diseño dinámico de las estructuras biológicas y sus patrones funcionales. Su objetivo central es descubrir nuevos procesos neuronales basados en modelos matemáticos que revelen principios morfo-funcionales en múltiples escalas. La plataforma incluye a tres especialistas, Ing. Electrónicos de la U-Conce, Ing. en Bioinformática de la U-Talca, y un post-doc en Computación de la U-Chile con doctorado en Francia. Actualmente, el desarrollo de modelos bi y tridimensionales está limitado por la rapidez de los algoritmos implementados. En los próximos 2 años, BNI realiza una inversión de 800.000M$Ch para crear un nuevo edificio I&D de excelencia que conectará dientes A y B de la F-Med, incluyendo salas de microscopía y de registros eléctricos de vanguardia. Incluye una unidad Science-to-Buisiness que albergará spin offs Biotecnológicos y proyectos I&D en vías de inserción al mercado. Con una inversión de 50.000M$Ch en redes y datos, el BNI aporta contrapartes para asegurar la conectividad de todas sus actividades y de sus 150 miembros a la red BioMed-HPC. FOUCH Facultad de Odontología cobija al Instituto de Registro de Patología Oral IREPO, sucesor del Plan Latinoamericano de Patología Oral (PLAPO), entidad creada a mediados de los 70 en un proyecto U-Chile/OPS. IREPO alberga un registro de 33.470 casos de patología oral, ordenados por nombres, clasificados por diagnósticos y compuestos por cortes histológicos y muestras. Este material es susceptible de ser reestudiado y sometido a nuevas pruebas diagnósticas, inmunohistoquímica o de DNA . Aprox 2.000 casos están documentados con diapositivas. Este material tiene valor patrimonial incalculable por la cantidad, calidad, estado de conservación del material y su capacidad de crecimiento y actualización. Aunque útil a la actividad clínica, su potencial mayor es docencia e investigación. Resulta fundamental la confirmación o actualización diagnóstica de los casos, de importancia para la taxonomias, la sistematización y comprensión de la patología. Una práctica imprescindible es el intercambio de opiniones asociado al intercambio de imágenes y la docencia que determina deterioro y pérdida por la manipulación del material, una amenaza a su integridad. El transporte del material prolonga y complica los procesos. Por ello se propone la digitalización bidimensional de las imágenes planas -historias clínicas y diapositivas- y digitalización tridimensional del material orgánico -placas histopatológica- para construir un banco de datos y generar y fortalecer las redes de interconexión con Unidades Académicas complementarias. Este desarrollo potenciará la investigación científica y el futuro Programa de Doctorado de nuestra Facultad. CCTVal Centro Científico Tecnológico de Valparaíso (www.cctval.utfsm.cl) de la Universidad Técnica Federico Santa María (UTFSM). El CCTVal es uno de los institutos chilenos de investigación avanzada fundados por CONICYT en el marco del Programa Basal de Financiamiento para Centros Científico-Tecnológicos de Excelencia (PFB). El CCTVal tiene tres componentes: Física de Altas Energías, Electrónica de Potencia, y HPC/Informática. Su objetivo principal es impulsar la investigación Física y el desarrollo de la ingeniería avanzada en el país implementando diversos proyectos de I&D. En este contexto, el CCTVal está entrando recientemente al área de salud mediante un proyecto de la patología digital, Procesamiento de Imágenes de Tejidos Mamarios Cancerígenos Tratados con Inmunohistoquímica (FB0821/14-RP), a cargo de la MS Ing. Informática Raquel Pezoa. Una colaboración recientemente establecida con SCIAN-Lab llevará la participación en el proyecto Microscopía Virtual - Centro de Patología Digital Asistida por Internet - (FONDEF D11I1096), reforzando mutuamente el desarrollo de la patología digital dentro de nuestro país. La incorporación del CCTVal a la red BioMed-HPC se considera esencial para el éxito de está colaboración y sus futuras perspectivas. La coordinación entre el CCTVal y BioMed-HPC estará a cargo del Dr. Luis Salinas y MS Raquel Pezoa (ver tabla de recursos). Integrantes Externos Internacionales Instituto Pasteur, Montevideo, Ury(www.pasteur.edu.uy). Con el propósito de reforzar la integración regional de iniciativas científicas Latinoamericanas, se realizó un primer encuentro entre Investigadores de la F-Med y científicos Uruguayos, Argentinos y de Brasil en el Instituto Pasteur de Montevideo. El encuentro potenció la interacción entre científicos en el ámbito de la investigación y desarrollo en temáticas de la neurociencia bajo estándares internacionales. 15 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE El impacto científico global, social y económico de la investigación desarrollada por estos grupos, la mayoría de las veces de forma aislada, está todavía por debajo de las expectativas y urgentes necesidades de los países de la región. La generación de plataformas comunes en el campo de la bioinformática y el procesamiento de imágenes biomédicas en combinación con HPC es una de las principales metas en esta alianza estratégica. Brown University, Providence, Rhode Island, USA (www.brown.edu). Tiene una exitosa experiencia de trabajo interdisciplinario y gran diversidad de esfuerzos en bioinformática y biología computacional, con participación de diversos departamentos (Biology, Neural Sciences, Computer Sciences, entre otros) y divisiones (Applied Mathematics, Engineering) académicas y los campos clínicos de su Escuela de Medicina en el estado de Rhode Island. Todos ellos están conectados por una red fotónica de alta velocidad (10 Gbps) al CCV (Center for Computation and Visualization), unidad que ofrece servicios de computación de alto rendimiento (HPC) y alta capacidad de almacenamiento (high density storage) en forma centralizada, creando un eco-sistema de colaboración a nivel local. El NLHPC/CMM-U-Chile y el CCVBrown han establecido una solida interacción conjunta en colaboración con IBM Research para enfrentar problemas de alta complejidad y datos masivos en biomedicina y medio ambiente, considerando que sus laboratorios pueden aportar grandes fortalezas para resolver problemas regionales que muestran varios grados de similitud (recursos naturales de la costa). Con apoyo de NSF-USA, el 2011, Brown estableció ICERM (Institute for Computational and Experimental Research in Mathematics) para dar amplio apoyo y profundizar la relación entre matemáticas y computación, para expandir los avances teóricos relacionados a la computación y enfrentar problemas reales con nuevas herramientas matemáticas. Ello complementa y refuerza la acción del CCV para un uso innovativo de HPC en el área biomédica. GIGA, Grappe Interdisciplinaire de Génomique Appliquée, Liège, Bélgica (www.giga.ulg.ac.be). GIGA es un centro de investigación en genómica y proteómica de excelencia de la U-Liège y de referencia mundial. Incluye siete plataformas tecnológicas, una oficina de transferencia de tecnología, un 'incubador' de compañías de Biotecnologías, y un centro de educación continua. Cada una de estas actividades está guiada por su actividad central que es la Genoproteómica. Ésta incluye estudios de genes que codifican información necesaria para el desarrollo y homeostasis de los organismos y la proteómica, que estudia la expresión, regulación e interacción de proteínas en los organismos. GIGA, representado por su Board Chairman Dr. Joseph Martial, consideran este proyecto de gran relevancia para el desarrollo de la investigación y docencia de postgrado en ambas instituciones y por ende ha comprometido su apoyo a BioMed-HPC en la carta adjunta a está postulación. 16 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Annex II DESCRIPCIÓN DE LABORATORIOS DE MIEMBROS PARTICIPANTES Laboratorio Procesamiento de Imágenes Científicas SCIAN-Lab: Steffen Härtel SH, BNI/ICBM, F-Med, U-Chile SCIAN-Lab fundado por Dr. SH en 2006 alberga diferentes proyectos que generan experticia básica en el manejo de grandes volúmenes de datos providentes del campo de la microscopía óptica con fines biológicos o biomédicos: (i) Fast Computational Schemes for the Analysis of Morpho-Topological Data from High Throughput Microscopy (FONDECYT 1120579, en colaboración con NH (DCC, FCFM) y Dr. T Asahi (CMM, FCFM)), (ii) Red de Microscopía de Super Resolución SOFI para Neurociencia (BNI/ICM, DFG), (iii) Centro de Espermiogramas Digitales Asistidos Por Internet (CEDAI) (FONDEF D11I1096, en colaboración con la Clínica las Condes y el IDIMI), (iv) Microscopía Virtual - Centro de Patología Digital Asistida por Internet - (FONDEF D11I1096, en colaboración con GR (FOUCH), patólogos del Hospital Clínico y R Pezoa, U-Santa María, Valpo). En el presente, ensayos in vivo realizados en investigación básica (neurociencia) a través de microscopía confocal de alta velocidad (spinning disk) generan hasta 100 GB de datos por experimento (en este momento la F-Med cuenta con tres microscopios spinning disk), experimentos a través del recién llegado Leica TCS LSI: Super Zoom Confocal con Matrix Software generan datos de un tamaño parecido por experimento (tamaño que va en incremento con el creciente uso de la automatización de escaneo de este equipo), secuencias SOFI están en el orden de 200 GB para una estructura biológica 3D en supra-resolución, y sistemas como el Hamamatsu NanoZoomer for Digital Pathology (NDP) que llegará a la F-Med al principio del 2013 generan imágenes de un tamaño de 30 GB por biopsia (www.microscopiavirtual.cl /wp/?page_id=5). Estos imágenes o películas en 2/3D requieren ser analizadas a través de HPC y distribuidas entre hospitales, clínicas y centros de investigación para un uso eficiente de los equipos (orden de 150.000 M$Ch). El tráfico de entre SCIAN-Lab/BNI y el NLHPC se estima a 1-10 TB por semana a partir del 2013, incrementando paulatinamente con el desarrollo de herramientas de análisis de estructuras celulares en el ámbito neuro-biológico, de la patología digital, espermiogramas digitales asistidos por internet, SOFI y aplicaciones dentro a través del LSI. Laboratorio de Genética de Sistemas y Genómica Biomédica (GENOMED): Ricardo Verdugo RV, F-Med, U-Chile Dentro de su marco de desarrollo estratégico, el ICBM ha apoyado la contratación del Dr. RV y montaje del laboratorio GENOMED em 2012, donde se utilizan herramientas de la genética, genómica y bioinformática para entender la relación entre genotipos y fenotipos complejos con el objetivo de desarrollar Medicina Genómica. LBMG, OMICS y GENOMED (ver abajo) participan del proyecto ChileGenomico donde se obtendrán perfiles genéticos de la población chilena mediante secuenciación y genotipificación por microarreglos de 500 individuos (chilegenomico.med.uchile.cl). Además GENOMED y participa del proyecto FONDEF Incorporación de la Secuenciación de Última Generación en el cuidado de los pacientes con cáncer del Dr. Ricardo Armisen (U-Cancer) para desarrollar un método de diagnóstico y prescripción de tratamiento del cáncer de mama basado en secuenciación de ADN tumoral. Estos proyectos demuestran la necesidad de la red de incluir académicos jóvenes y la capacidad de generar proyectos colaborativos que involucran experticias que van desde la matemática, bioinformática, genómica, hasta la medicina traslacional. El modelo de transferencia de datos genómicos usado hasta la fecha entre los punto de colección (OMICS) y de análisis de datos (LBMG y GENOMED) no es escalable (ver abajo sección Situación con y sin Proyecto). Por lo tanto, es indispensable contar una infraestructura de alta capacidad que permita el desarrollo de proyectos genómicos de mayor envergadura. Laboratorio de Microfluídica: María Cordero MC, Departamento de Física (DFI), FCFM, U-Chile En Junio 2012, se inauguró el Laboratorio de Microfluídica (DFI, FCFM) a cargo de la Dra. MC. Su principal objetivo es el estudio y descripción de flujos bifásicos al interior de redes de canales de tamaños micrométricos. En particular, se busca medir, comprender y modelar el comportamiento y transporte de gotas inmersas en un flujo externo y sujetas a diferentes fuerzas en ambientes confinados, como son los micro-canales. Gran parte del trabajo experimental se basa en el análisis de imágenes de microscopía de campo brillante y de fluorescencia, lo que genera desafíos en el desarrollo de técnicas apropiadas para medir a partir de imágenes, así como para transportar grandes volúmenes de datos. Uno de los principales empujes a tipo de investigación que se desarrolla en este laboratorio es su alto potencial aplicado, dada la posibilidad de utilizar los micro-canales como plataforma para realizar estudios biológicos o químicos. Para ello se utilizan las gotas y los flujos al interior de los microcanales para encapsular, transportar y manipular diferentes materiales, tales como reactivos, proteínas o células, y así realizar un estudio completo o un proceso de laboratorio de manera automatizada, con un bajo consumo de materiales y casi nula contaminación. En este sentido, buscamos potenciar la generación de proyectos colaborativos, multidisciplinarios y con fines aplicados, con la F-Med en particular y la U-Chile en general. Laboratorio de Biología Celular y Molecular (LBCM): Andrea Paula-Lima AP, FOUCH, U-Chile La FOUCH recientemente incorporó a cuatro académicos jovenes (Dres AP, Vicente Torres, Sonja Buvinic y Alejandro Escobar) formando el LBCM. Los investigadores participan en de Inserción de Capital Humano Avanzado en la Academia, U-Moderniza, Fondos Estratégicos BNI y Anillo, además de liderar Proyectos Fondecyt de Iniciación. En el LBCM se desarrollan líneas de investigación independientes y complementarias, en que se estudian distintos procesos a nivel celular, incluyendo las vías de transducción de señales involucradas en fenómenos con un alto impacto biomédico, tales como la enfermedad de Alzheimer, el cáncer, las distrofias musculares y las cardiopatías. Estos estudios utilizan aproximaciones experimentales 17 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE basadas en técnicas de microscopía y análisis avanzado de imágenes, entre ellas microscopía confocal de alta velocidad (spinning disk) para determinar la temporalidad de los procesos intracelulares, cuyos registros producen datos del orden de 10 GB. Además, la Dra. Paula-Lima participa en un proyecto básico-clínico que contempla registrar la actividad eléctrica cerebral mediante electroencefalografía continua de alta densidad en sujetos con Alzheimer y en ratas modelos. Estos experimentos producen datos del orden de 50-100 GB, cuyo procesamiento inicial (descomposición de las señales) puede aumentar 10 veces su tamaño original. Gracias a BioMed-HPC podemos realizar por primera vez modelamiento complejo de las señales electrofisiológicas, cuya necesidad de procesamiento y memoria RAM pueden requerir el trabajo conjunto de computadores durante días de análisis continuo en el escenario actual. Laboratorio de Biología del Conocer: Juan Carlos Letelier, F-Ciencias, U-Chile En la F-Ciencias existe una larga tradición de una visión realmente sistémica sobre la organización de los sistemas vivos. Está tradición está basada en la noción de autopoiesis (Maturana y Varela, 1972). Desde hace unos 10 años las ideas originales se han clarificado (un poco) y se ha comenzado a generar una teoría del metabolismo. Está teoría está basada en las nociones de clausura metabólica y aunque está todavía en pañales ya tiene interesantes conceptos a su haber. Obviamente que en el estado actual la teoría es analítica, es decir no usa computadores, ya se piensa que en los próximos años los nuevos resultados si requerirán de una interesante potencia de cálculo. El interés de nuestro grupo en este U-Redes es poder participar en una iniciativa con personas con interés en la teoría y capacidades no triviales de cálculo. Laboratorio de Bioquímica y Biología Molecular (LBBM): Ricardo Cabrera RC, F-Ciencias, U-Chile El LBBM desarrolla diferentes líneas de investigación en el contexto de un FONDECYT (1121170) y un Anillo (ACT1107): (i) entender y cuantificar procesos biológicos como la especificidad de las enzimas y (ii) la distribución de flujos de carbono, mediante el uso de herramientas de modelamiento. En el primer caso, llevamos a cabo estrategias de diseño racional para el incremento de la especificidad hacia nuevos ligandos en deshidrogenasas. Esto requiere la realización de simulaciones moleculares que permiten predecir cambios que se observarán in vitro, mediante análisis de cinética enzimática, para posteriormente evaluar in vivo el efecto celular de la modificación diseñada. Evaluamos decenas de mutantes simples y dobles, en presencia y ausencia de ligandos. Además colaboramos en la realización de este mismo tipo de cálculos con otros laboratorios, lo cuál hace que el trabajo en simulaciones moleculares sea permanente e intenso en nuestro grupo de investigación. Así, las dinámicas simuladas generan datos del orden de decenas de GB que posteriormente deben ser descargados desde el centro de cómputo para ser analizados. Lo que con un bajo ancho de banda se traduce en una limitación para el desarrollo de nuestros resultados. Laboratrio de Fisiologia Senasorial, Sebastian Brauchi SB, F-Med, CAMI, UACh Valdivia El laboratorio de fisiología sensorial y biofísica dirigido por el Dr SB, estudia principalmente proteínas de membrana, su organizacion estructural, su distribución sub-celular y el control celular a que están sujetos. La imagenología de TIRF y de super resolución constituyen algunas de las aproximaciones experimentales que se utilizan. Las técnicas de imagenología que utilizamos generan grandes cantidades de datos, y hoy no solo es costoso para nuestros colaboradores transportar los datos de vuelta a sus laboratorios sino que tambien es engorroso enviar nuestros propios datos para ser analizados utilizando mejores motores de analisis (ej. los disponibles en SCIAN-Lab, SH). Adicionalmente vemos que la actividad del Center for Advanced Microscopy and Imaging en UACh, una iniciativa que se decantara durante 2013 y 2014, se vera favorecida desde un inicio mediante la creación de una carretera de alta velocidad para el traspaso de datos entre investigadores. Desde ambos puntos de vista, el manejo inteligente de grandes cantidades de datos y la potenciación de una red de colaboración entre investigacdores, pertenecer a BioMed-HPC será un gran aporte a la comunidad nacional y sin duda tendrá un impacto que perdurara. Programa de Epidemiología del Instituto de Salud Pública: Paulina Pino (PP), F-Med, U-Chile En epidemiología se ha incursionado en el uso de grandes bases de datos poblacionales para el estudio de diversos eventos de salud. El Dr J Kaufman U-McGill, junto a PP y el joven investigador Dr. Faustino Alonso utilizan datos socioeconómicos del Censo 2002 y el registro de la edad gestacional y peso al nacer de los certificados de nacimiento, relacionándolos por geocodificación de la dirección de la madre a la fecha de nacimiento del hijo, para el estudio de las desigualdades sobre efectos reproductivos al nacer. Al menos dos proyectos PhD ya han utilizado datos nacionales de registros, de encuestas o de resultados de exámenes de laboratorio o de procedimientos, relacionándolos mediante el RUT, para el estudio de mortalidad por cáncer y de la evolución del sobrepeso y obesidad en escolares. Sin embargo, hay un largo camino en términos se calidad de los datos, conectividad y accesibilidad si se quiere al menos alcanzar los estándares logrados en países desarrollados. La asociatividad a través de BioMed-HPC constituye una gran oportunidad para impulsar el desarrollo y capacidad para el manejo de grandes volúmenes de datos. Se espera un gran impacto en el estudio de determinantes de la salud y su relación con eventos identificados en los registros de atención de salud, vigilancia epidemiológica y registros vitales entre otros. Modelación geométrica: Nancy Hitschfeld NH, Departamento de Ciencias de la Computación (DCC), FCFM , U-Chile El DCC tiene una gran experiencia en el desarrollo de discretizaciones espaciales en 2/3D y ing de software para generar triangulaciones y mallas mixtas (triángulos y cuadriláteros) en 2D, triangulaciones de superficie (2D1/2) y mallas de tetraedros y mallas mixtas (hexaedros, tetraedros, prismas o pirámides) tanto para trabajar con métodos numéricos de elementos finitos como de volúmenes finitos. Con SCIAN-Lab tenemos una colaboración científica (FONDECYT) para estudiar geométricamente la estructura de células o conjuntos de células de seres vivos con el fin de entender su proceso 18 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE de creación, evolución y comportamiento. En particular, a partir de la representación de una estructura con una triangulación de superficie que puede ser normalmente del orden de 10E6 de puntos hemos construido algoritmos para generar su skeleton y analizar su forma. Ahora estamos trabajando en la aceleración de algoritmos vía GPU o HPC dependiendo del tipo de problema a resolver. Para un colaboración más efectiva necesitamos intercambiar grandes volúmenes de datos y poder ejecutar y obtener datos de procesos que se corren a distancia. BioMed-HPC es esencial y beneficiaría enormemente futuras extensiones de la colaboración. Modelación y Mecánica Cuántica Molecular (MMCM): Gerald Zapata GZ, FCQF, U-Chile Dos investigadores jóvenes, Dr. GZ del FCQF y Dr. J. Cristian Salgado (CS) del FCFM han desarrollado una colaboración en una línea de investigación enfocada en aplicar métodos teóricos y computacionales para entender, predecir y cuantificar procesos biológicos involucrados en la interacción proteína-ligando. Buscan una descripción adecuada y detallada de la estructura electrónica y conformacional de moléculas de relevancia biológica, así como del análisis de los cambios conformacionales en proteínas con interés farmacológico, además de las propiedades químicas, biológicas asociadas a las interacciones moleculares. El enfoque es multidisciplinario incluyendo, herramientas de modelación molecular, dinámica molecular, docking, biología de sistemas y modelamiento matemático de sistemas biológicos. Su trabajo tiene aplicaciones terapéuticas e industriales en una amplia variedad de enfermedades incluyendo desordenes mentales, depresión, adicción. CS es un investigador joven asociado al Instituto Milenio de Dinámica Celular y Biotecnología (ICDB): Un centro de Biología de Sistemas y servirá como conector al Instituto de la red. Dos proyectos FONDECYT (2009 y 2012, GZ I-Resp) han permitido construir un cluster de cómputos en FCQF que cubren las necesidades de dichos proyectos. Además un proyecto FONDECYT de iniciación (2009) permitió implementar un clúster de cálculo científico similar en el laboratorio del CS (PMDC Lab, 80 cores). Esto ha permitido enriquecer la modelación molecular con problemáticas, conceptos, tecnología y metodologías usados en el dominio de la Biología de Sistemas y el modelamiento matemático de sistemas biológicos. Sin embargo el poco ancho de banda entre las facultades de que albergan ambos miembros de está pequeña red, ha sido una limitante para el desarrollo de proyectos de mayor envergadura. Con la red BioMed-HPC, la conexión óptima puede ser una realidad. Laboratorio de plegamiento de proteínas: Dr. Mauricio Baez, FCQF, U-Chile Nuestro grupo desea comprender los procesos de plegamiento que experimentan las proteínas luego de ser sintetizadas, vale decir los mecanismos mediante los cuales las cadenas polipeptídicas adquieren su estructura tridimensional. Unos de los objetivos principales es crear un puente entre las metodologías experimentales y computacionales que permiten describir estos mecanismos a nivel molecular. Para esto realizamos experimentos espectroscopia de fuerza en colaboración con el laboratorio de moléculas únicas de la U-Berkeley, liderado por Dr. C Bustamante y su laboratorio gemelo presente en la U-Cayetano Heredia en Lima. Usamos simulaciones moleculares basadas en potenciales Gō los cuales han mostrado ser exitosos para describir el mecanismo de plegamiento a nivel atómico. En nuestro grupo estas herramientas se han llevado al límite mediante la investigación proteínas con topologías anudadas que requieren un gran poder de computo debido a su complejidad topológica. La utilización de una red de alta velocidad para compartir y acceder al poder computo es claramente una ventaja para nuestro laboratorio. Laboratorio de Modelación de Procesos y Computación Distribuida (PMDC Lab): J. Cristian Salgado CS, FCFM, ICDB, UChile El PMDC Lab, albergado en el Dep Ing Química y Biotecnología, está liderado por Dr. CS. Su misión es 'Aplicar técnicas y metodologías matemáticas de frontera para desarrollar un profundo entendimiento de las reglas subyacentes que gobiernan complejos sistemas biológicos y productivos'. La investigación realizada tiene como eje central la aplicación del modelamiento matemático a complejos sistemas moleculares, biológicos y productivos, todos ellos de gran impacto social e industrial y con financiamiento de diversos proyectos FONDECYT. El trabajo es fuertemente interdisciplinario e incluye colaboraciones con investigadores de la FCFM, F-Ciencias, FCQF y F-Med de la U-Chile, y la Usach, PUC, y U-Andes. También se han establecido colaboraciones con investigadores internacionales destacándose Berkeley (Ca, USA) o la U-Pierre et Marie Curie (Francia). La fortaleza de estas colaboraciones permite la solución efectiva de problemas esencialmente interdisciplinarios como los asociados a la Biología de Sistemas y el modelamiento y simulación molecular, todos problemas complejos, de gran riqueza y diversidad fenomenológica y asociados al manejo y análisis de grandes volúmenes de información. El proyecto BioMed-HPC permitirá potenciar profundamente el trabajo colaborativo actualmente en desarrollo, y favorecer la gestación de nuevas colaboraciones. Laboratorio de Bioinformática y Matemática del Genoma (LBMG): Alejandro Maass AM, CMM, FCFM, U-Chile El LBMG liderado por el Dr. AM es un grupo multidisciplinario de investigadores e ingenieros en áreas de estadística, informática y biotecnología. Es el laboratorio de referencia en temas de bioinformática y modelamiento matemático para la compañía Chileno-Japonesa BioSigma S.A., cuya misión es ser líder mundial en el área de biotecnología para la minería. Participa además en proyectos para desarrollar genómica de uvas y de salmón. Con vasta experiencia en 'metagenómica'; secuenciamiento y análisis de genomas de bacterias; anotación funcional de genomas y modelamiento de redes biológicas; análisis estadístico de datos genómicos; además cuenta con bases de datos para la administración e integración de información genómica en el ambiente de un computador de alto rendimiento de clase mundial situado en el CMM. La red BioMed-HPC le permitirá ampliar el rango de aplicaciones, potenciando el modelamiento matemático asociado a problemas biomédicos: entre otros genética humana, analizando mutaciones, modelamiento funcional de enfermedades, simulaciones in silico de tratamientos y desarrollar estrategias para el descubrimiento de nuevas drogas. 19 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Biología Computacional (DLab): Tomas Perez Acle TPA, NLHPC, CMM, FCFM, U-Chile El objetivo principal del DLab liderado por Dr. TPA es la generación de modelos computacionales multi-escala de fenómenos biológicos complejos con el fin de obtener conocimientos sobre la estructura y la dinámica fundamental que subyace a los sistemas biológicos complejos. Para ello, utilizamos y producimos herramientas de simulación mediante la combinación de técnicas avanzadas de modelamiento matemático, termodinámica, física y química, junto con tecnologías de HPC. Si bien nuestro objetivo principal es producir una herramienta multi-escala para la simulación de sistemas biológicos, también estudiamos procesos a escalas individuales. En la micro-escala (nivel atómico), utilizamos herramientas de modelamiento y simulación molecular para entender las propiedades físico-químicas que rigen; la comunicación intercelular mediada por canales de Connexina, transporte de trans-membrana mediado por canales de iones activados por ligandos y por gradientes osmóticos, así como el reconocimiento molecular y los procesos de transducción de señales mediada por GPCRs. En la meso-escala (a nivel celular) utilizamos herramientas de topología de redes para inferir y caracterizar procesos de señalización celular, regulación génica, así como eventos de coordinación y codificación neuronal. En la macro-escala (nivel de población) usamos modelos de agentes y modelos basados en reglas para estudiar procesos de adaptación del comportamiento de poblaciones artificiales. 20 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Anexo III Carta_ Compromiso_ICBM_Facultad_de_Medicina_BioMed-HPC 21 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Anexo IV Carta_Compromiso_CITC_BioMed-HPC 22 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Anexo V Carta_Compromiso_Luis_Salinas_Valparaiso_BioMed-HPC 23 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Anexo VI Carta_Compromiso_CCTVal-UTFSM 24 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Anexo VII Carta_Compromiso_BNI_BioMed-HPC 25 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE 26 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Anexo VIII Carta_Compromiso_F-Med_BioMed-HPC 27 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Anexo IX Carta_Compromiso_FOUCH-HPC 28 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Anexo X. Carta_Compromiso_NLHPC-HPC 29 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Anexo XI. Carta_Compromiso_GOCCHI-HPC 30 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE 31 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Anexo XII Carta_Compromiso_STI-HPC 32 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Anexo XIII Carta_Compromiso_SCIAN-HPC 33 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Anexo XIX Carta_Compromiso_REUNA-HPC 34 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Anexo VII Presupuesto red interna F-Med 10[Gbps] 35 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Anexo VIII/ 1 de 3 Presupuesto Red óptica STI-F-Med 36 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Anexo VIII/ 2 de 3 Presupuesto Red óptica STI-F-Med 37 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Anexo VIII / 3 de 3 Presupuesto Red óptica STI-F-Med 38 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Anexo XXII. Carta_APOYO_GIGA-HPC 39 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE 40 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Anexo XXIII. Carta_APOYO_Pasteur-HPC 41 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE Anexo XXIV. Carta_ APOYO_Brown-HPC 42 PROGRAMA U-REDES VICERRECTORIA DE INVESTIGACION Y DESARROLLO UNIVERSIDAD DE CHILE