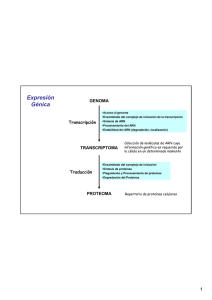
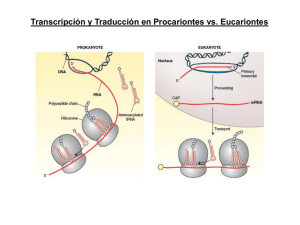
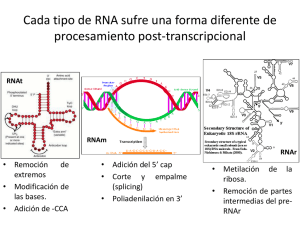
(Sintesis y Control de Traducción). - U
Anuncio

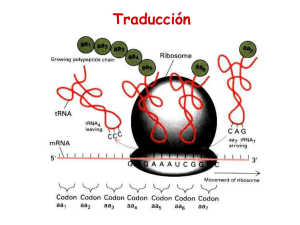
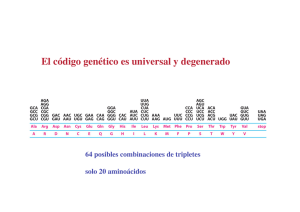
TRADUCCION -1950s Proteínas: arreglos de solo 20 aa en forma no azarosa -Ribosomas: lugar de síntesis de proteínas -1960s polisomas, tRNAs -1961 Jacob y Monod Teoría del mensajero - 1964s confirmación de mRNA intermediario informacional Componentes principales sistema traducción mRNA tRNA Aminoacil Aminoacil--tRNA sintetasa Ribosomas Factores proteicos mRNA : Secuencia codificante 5' leader No traducible (5´ (5´UTR) 3' trailer No traducible (3´ (3´UTR) 5' cap and 3' poly (A) tail (eucariotas) MECANISMOS DE SINTESIS PROTEICA El producto de traducción requiere de dos pasos de reconocimiento importantes: a) la elección del aminoácido correcto para la unión covalente con su tRNA b) la selección de los tRNAs cargados con sus aminoácidos específicos. El primero de estos pasos, es catalizado por enzimas específicas denominadas aminoacil tRNA sintasas (aaRSs), que unen un aminoácido al extremo 3´ de su ribosa para formar un aminoacil-tRNA tRNA Y T TyC arm D arm AminoacilAminoacil -tRNA sintetasas • Une los aa al correcto t RNA • Más de 20 enzimas diferentes • Requiere ATP Reconocimiento del tRNA tRNA:: puntos de contacto entre tRNA y sitio activo de proteína (1 –5 sitios, muchas veces incluye anticodon) Figura: representación de un aminoacil-tARN Este proceso necesita de la hidrólisis de ATP en dos reacciones secuenciales que se catalizan en la misma enzima: 1.- Primero el aminoácido es activado con ATP para formar aminoacil-adenilato. Este intermediario puede ser aislado con dificultad, pues permanece unido fuertemente a la enzima. Figura: activación de un aminoácido para la aminoacilación 2.- el anhídrido mixto formado en el paso 1, reacciona con el tARN para formar el aminoacil-tRNA de la siguiente forma: aminoacil-AMP + tRNA aminoacil-tRNA + AMP Algunas aaRSs unen exclusivamente aminoácidos en el grupo OH en la posición 2´ de su tRNA, otras de estas enzimas, también lo pueden hacer en el extremo 3´ (esto se supo al utilizar tRNAs carentes de alguno de los dos extremos). De manera natural, el grupo aminoacil se equilibra rápidamente entre las posiciones 2´ y 3´, por tanto la reacción general de aminoacilación es: Aminoácido + tRNA + ATP aminoacil-tRNA + AMP + PPi La reacción es reversible porque las energías de hidrólisis de los enlaces formados para el aminoacil-adenilato y en el aminoacil-tRNA son comparables a la hidrólisis del ATP. Aminoácido + tRNA + ATP aminoacil-tRNA + AMP + PPi La dirección de la reacción se mantiene por la acción de la pirofosfatasa inorgánica que actúa en el primer paso de la reacción. El tRNA es el aceptor de acilos en la activación de los aminoácidos. Existen dos clases de aminoacil-tRNA sintasas (2`y 3`) Las células deben tener al menos una aminoacil-tRNA sintasa para cada uno de los 20 aminoácidos. La similitud de las reacciones catalizadas por estas enzimas y el parecido estructural de todos los tRNAs sugiere que todas las aaRSs tienen un ancestro común y que estarían relacionadas estructuralmente. De las aproximadamente 100 aaRSs que se han caracterizado cada una de ellas tiene cuatro tipos de subunidades , 2 (la forma predominante), 4 y 22 que varían entre 334 y 112 residuos. A pesar de estas notables diferencias, existe una pequeña secuencia similar entre las enzimas específicas para cada aminoácido. La aminoacil-tRNA es muy específica En la síntesis de proteínas el tRNA adecuado es seleccionado sólo a través de interacciones codonanticodon, el grupo aminoacilo no participa en el proceso. El fenómeno fue demostrado utilizando la molécula de Cys-tRNACys en la cual el residuo de Cys esta marcado con 14C. Esta molécula fue desulfurizada reductivamente de tal forma que la Cys se transformó en Ala El híbrido resultante AlatRNACys marcado con 14C, se agregó a un sistema de síntesis de proteínas de reticulocitos de conejo libre de células. El experimento se siguió en la síntesis de la cadena de la hemoglobina, proteína que contiene varios puentes disulfuro. Se encontró que la hemoglobina sintetizada en estas condiciones no contenía marca radioactiva. De esta forma se comprobó que la única parte de la molécula del tRNA “cargado” que participa en el reconocimiento del codon, es el anticodon. La degeneración del código genético está mediada por la tercera posición de la interacción codón-anticodón que es variable Muchas células contienen tRNAs isoaceptores, lo que quiere decir que algunos tRNAs, son específicos para el mismo aminoácido. Por ejemplo el tRNAPhe de levadura que contiene el anticodón GAA, reconoce los codones UUC y UUU. Recordar que la interacción es antiparalela como se muestra a continuación: tRNA Une un aa específico en la terminación 3´ o 2`en algunos casos tRNAs isoaceptores con anticodones diferentes unen el mismo aa (varios codones para un aa) Hay varias especies de tRNA que pueden reaccionar con el mismo codon El anticodon hace apareamiento de bases con un codon de mRNA (o mas), la tercer base de codon con la primer base de anticodon puede aparear con otras bases además del apareamiento tradicional (Wobbling) Anticodon Codon 3´ - A - A - mG - · · · · · · 5´ - U - U - C - 3´ Anticodon Codon 5´ 5´ 3´ · · · · · 3´ -G-C-A- 5´ · · · · · · 5´ - U - U - U - 3´ -C-G-I- · - A - A - mG - 5´ 3´ Lo anterior significa que los pares de bases “flexibles” pueden aparecer en la tercera posición de la interacción codonanticodon La síntesis de proteínas procede desde el amino hacia el carboxilo terminal Esta dirección fue deducida por Howard Dintzis en 1961 a través de experimento de marcaje isotópico. Expuso reticulocitos que sintetizaban activamente hemoglobina en presencia de 3HLeucina en tiempos menores del que necesitan para producir al polipéptido entero (síntesis parcial) La cantidad de péptidos obtenidos por tripsinización se incrementaba con la proximidad al carboxilo terminal. La elongación ocurre por la unión de la cadena en crecimiento con el aminoácido cargado en el tRNA Durante la síntesis de polipéptidos, los aminoácidos son agregados secuencialmente en el extremo carboxilo de la cadena naciente unida al ribosoma. Si se obliga a salir a la cadena naciente del ribosoma mediante tratamiento con altas concentraciones de sal o detergentes, el residuo en el carboxilo terminal está siempre esterificado a un tRNA, formando un peptidil-tRNA (ingreso secuencial). El polipéptido naciente debe crecer entonces transfiriendo el aminoácido unido al tRNA hacia el peptidil-tRNA. El ribosoma posee dos sitios de unión, el sitio P que une al peptidil-tRNA y el sitio A que une al aminoacil-tRNA, estos sitios son “artificiales” Consecuentemente, después de la formación del enlace peptídico, el sitio P contiene un tRNA deacilado que es liberado y reemplazado por el nuevo peptidil-tRNA formado previamente en el sitio A y así sucesivamente. Además en el ribosoma existe un tercer sitio, el sitio E (Exit), que une momentáneamente a la cadena naciente. Ribosomas 4 sitios de binding mRNA--binding site mRNA A site (aminoacyl) P site (peptidyl) E site (exit) Procariotas : subunidades 30s y 50s 60S subunit 40S subunit DINAMICA DE TRADUCCION Traducción 3 fases •Iniciación •Elongación •Terminación INICIACION Procariotas -Tanscripción Tanscripción-- traducción acopladas Eucariotas -Iniciación citoplasmática Iniciación mRNAs que se Tienenenen común están transcribiendo -Disociación de ribosomas en subunidades -Reconocimiento de sitio de iniciación - Estrategias diferentes para por-interacción m-RNArpreiniciación reconocimiento msitio de FormaciónRNA complejo en subunidadde menor (Secuencia ShineShine-Dalgarno) iniciación -Unión de complejo preiniciación a mRNA Traducción CapCap-dependiente -Iniciación mediada por factores “scanning” Traducción CapCap-independiente (IREs) -3 Factores de iniciación (IF1,IF2,IF3) - 13 factores diferentes EUCARIOTAS INICIACION 3 Pasos 1) Unión del iniciador tRNAiMet a la subunidad menor 2) Unión de este complejo a mRNA y localización codón de iniciación 3) Unión de la subunidad mayor del ribosoma Cada uno de los pasaos de la iniciación de la traducción es facilitado por por proteínas denominadas Factores de iniciación. Iniciación RESUMEN: INICIACION La localización de un codón de inicio conservado entre las especies es una evidencia importante (>60% proteínas de E. Coli comienzan por Met). En la reacción de iniciación participan 3 factores importantes: IF-1, IF-2 e IF-3. IF-1 : localiza el codón de inicio (AUG) IF-1 + IF-3: estabilizan la subunidad mayor del ribosoma. IF-2 : se ordena junto al tRNA para formar el complejo de iniciación. Este complejo es dependiente de GTP. aa 50S ANTICODON IF-2 IF1 + IF-3 AUG 30S Ribosoma inactivo IF-1 RNAm IF-3 GDP GTP Ribosoma activo 50S ANTICODON aa 30S AUG REACCION DE ELONGACION La elongación de la cadena polipeptídica comienza con 3 etapas claves: La activación de los sitios A y P en el ribosoma. La liberación de los factores IF1 e IF-2. El ingreso de los precursores catalíticos EF-1, EF-2 y EF-3. En la reacción de elongación se producen 3 reacciones intermediarias. ELONGACION aa-tRNA:EF1A:GTP EF2:GDP EF1A:GDP EF2:GTP EF1B IF-3 50S aa1 ACTIVACION DE AMINOACIL-tRNA IF-1 + tRNA aa2 UNION ANTICODON ANTICODON P 30S A EF-1 AUG TRANSPEPTIDACION TRANSLOCACION aa ACTIVACION DE PEPTIDIL-tRNA P AUG EF-2 SE VUELVE A ACTIVAR LA ENZIMA AMINOACIL-tRNA EF-3 tRNA tRNA aa P AUG A ANTICODON aa3 TRANSLOCACION tRNA+ Durante esta etapa los sitios del Ribosoma vuelven a su “activación” inicial, es decir A en el sitio donde se activa la aminoacyl y P como el sitio donde se detecta actividad de la peptidil. REACCION DE TERMINACION La reacción de terminación de la cadena se inicia cuando llega un triplete o codón de término a la unidad ribosomal. La señal que da el codón de término es reconocida por los factores de liberación RF-1, RF-2 y RF-3. RF-1 localiza el codón de término. RF-2 + RF-3 neutralizan la cadena polipeptídica transfiriendo un H a la cadena. RF-3 es el encargado además de activar al GTP, energía que se gasta en esta reacción. RF-2 es el cofactor que separa las unidades ribosomales. TERMINACION RF3-GTP RF1 RF3-GDP RF1 La estructura de RF1 se asemeja a la del tRNA Complejo de terminación AAAAAAAAAAA Poli(A) RF1 RF3 PABP eIF4G mRNA Complejo de iniciación 5`-cap 70S POLIPEPTIDO LIBERADO 30S aa RIBOSOMA INACTIVO GTP tRNA RF-3 RF-3 P RF-2 A ANTICODON AUG STOP RF-1 Generalmente, varios ribosomas traducen de forma simultánea la misma molécula de RNAm, dando lugar a un polisoma o polirribosoma. De ese modo, se pueden sintetizar gran número de moléculas de proteínas en un breve período. Por ejemplo: el tiempo aproximado de síntesis de una proteína compuesta por 400 aminoácidos es de cerca de 20 segundos Control de la Traducción VELOCIDAD DE SINTESIS PROTEICA La proporción de proteínas que una célula posee, tiene una relación directa con sus requerimientos y con su estado ontogénico. A la fecha no existe evidencia de que los procariontes respondan a los cambios ambientales aumentando la velocidad de traducción; por lo tanto, su expresión genética es enteramente controlada en la trascripción. A partir de la observación de que los mRNAs procariontes tienen una vida media de pocos minutos, se supone no tienen necesidad de controles traduccionales. V (1/2) procariontes: 1,2 minutos V (1/2) eucariontes: horas a días. Estabilidad de mRNA EUCARIONTES El control eucariótico transcripcional está reservado para la diferenciación celular; existen además evidencias de que los eucariontes responden a sus necesidades, al menos en parte a través de controles trascripcionales. Lo anterior es al menos posible porque la vida media de sus mRNAs es de varias horas a días. Los sistemas mejor caracterizados son: la regulación de la síntesis de hemoglobina por hemo y los efectos de las proteínas inducidas por virus conocidas como interferón. REGULACION DE LA SINTESIS DE HEMOGLOBINA Los reticulocitos son células que sintetizan principalmente hemoglobina a una velocidad muy alta por lo cual son el modelo favorito para estudiar a la traducción eucarionte. En homogenizados frescos de estas células la síntesis de hemoglobina ocurre durante varios minutos y abruptamente se detiene por la inhibición de la iniciación de la traducción y la consecuente desagregación del polisoma. Este fenómeno se evita por la adición de hemo (que es un producto mitocondrial), que este sistema in vitro no puede sintetizar, lo que indica que la síntesis de la globina está regulada por la disponibilidad de hemo. Esta inhibición es revertida también por la adición del factor de iniciación eIF-2 y por altos niveles del GTP. Inhibido por hemo HCR= heme controlled repressor pro-HCR (inactivo) ATP HCR (activo) ADP eIF-2--P eIF-2—P eIF-2B eIF-2 GEF=complejo irreversible Pi H20 Fosfatasa de EIF-2 En ausencia de hemo, los lisados de reticulocitos acumulan una proteína, el represor controlado por hemo (HCR). Esta proteína fosforila específicamente la Ser 51 de la subunidad de eIF-2. Es un trímero que lleva GTP y Met-tRNA(MettRNA, es el tRNA iniciador en eucariontes que a diferencia de procariontes nunca está formilado), a la subunidad 40S del ribosoma. HCR es generado en ausencia de hemo, a partir de un proinhibidor en un proceso poco caracterizado que al parecer involucra al menos otra proteína. Tanto la forma fosforilada de eIF-2 como la no fosforilada pueden participar en la iniciación ribosomal: lo anterior se debe a que el GDP no se disocia espontáneamente de eIF-2 al finalizar la iniciación, como se hace en el proceso procarionte. Por el contrario eIF-2 intercambia este GDP por GTP en una reacción mediada por otro factor de iniciación, el eIF-2B La forma fosforilada de eIF-2 forma un compuesto más afín con eIF-2B que la forma desfosforilada. La cantidad de eIF-2B que existe en la célula es menos que la de eIF-2, lo que previene la formación del complejo eIF-2·GTP requerido para la inhibición de la iniciación de la traducción por HCR, la forma fosforilada de eIF-2 son reactivadas a través de la fosfatasa de eIF-2, que no es afectada por hemo Inhibido por hemo HCR= heme controlled repressor pro-HCR (inactivo) ATP eIF-2 Pi HCR (activo) ADP eIF-2--P eIF-2—P eIF-2B H 20 Fosfatasa de EIF-2 GTP eIF2B* GDP eIF2B GDP eIF2*GDP eIF-2*GTP La regulación del sistema vía grupo hemo, implica una adaptación del sistema generando una regulación : proteína-proteína SISTEMA INTERFERON Los interferones son glicoproteínas secretadas por células de vertebrados infectadas por virus. Después de unirse a receptores de superficie de otras células, los interferones se convierten en un estado antiviral, que impide la replicación de una amplia variedad de virus de RNAs y DNAs. El descubrimiento de estas moléculas en 1950, proviene de la observación de que los individuos infectados por un virus son resistentes a la infección por un segundo tipo de virus. Existen tres familias de interferones: tipo o interferón de leucocitos (células blancas), el tipo o interferón del fibrobalsto (tejido conectivo), esta familia está muy relacionada con la y el tipo o interferón del linfocito (células del sistema inmune). La síntesis del interferon es inducida por RNA de doble hebra (dsRNA). Estas moléculas son efectivas incluso a concentraciones tan bajas como 3 x 10-14 M, lo que los hace las moléculas biológicas más potentes, aunque su especificidad está lejos de la de anticuerpos contra virus. Los interferones previenen la proliferación viral principalmente al inhibir la síntesis de proteínas en las células infectadas, de hecho el interferón del linfocito incluso modula la respuesta inmune y lo hace a partir de dos formas diferentes Interferón que induce un inhibidor activado por dsRNA (Proteína Quinasa) ATP ADP EIF-2B eIF-2 Pi eIF-2--P eIF-2--P eIF-2B H20 Fosfatasa de EIF-2 El interferon que induce un inhibidor activado por dsRNA es una quinasa (DAI) que en presencia de dsRNA fosforila a la subunidad de eIF-2 exactamente igual que HCR (link a regulación por hemo) inhibiendo la iniciación ribosomal. Interferón induce 2,5A sintetasa dsRNA (2´,5´)-fosfodiesterasa ATP 2,5A ATP + AMP + PPi activación de 2`5`oligoadenilato sintetasa RNAsa L (inactiva) RNAsa L (activa) mARN nucleótidos degradación del mRNA Este compuesto 2´5´A activa una endonucleasa preexistente, la RNAasa L que degrada mRNA inhibiendo la síntesis de proteínas, 2´5´A es también rápidamente degradado por la (2´5´)-fosfodiesterasa, que es continuamente sintetizada para mantener el efecto. Inhibidor Cloranfenicol Estreptomicina Tetraciclina Comentarios Inhibe la petidil transferasa procariótica Inhibe la iniciación de la cadena peptídico procariótica, también induce a la lectura errónea de mRNA Inhibe la unión del aminoacil-tRNA procariótico a la subunidad pequeña del ribosoma Neomicina similar en actividad a la estreptomicina Eritromicina inhibe la translocación en procariotes a través de la subunidad grande del ribosom Ácido Fusidico similar a eritromicina solamente evitando que EFG se disocie de la subunidad grande Puromicina se asemeja a un aminoacil-tRNA, interfiere con la transferencia peptídica dando como resultado la terminación prematura en procariotas y eucariotas Toxina de Difteria cataliza la ribosilación-ADP de y la inactivación de eEF-2, el eEF-2 contiene un residuo His modificado conocido como diftamida, es este residuo el que es el blanco de la toxina de difteria . Residuo de diftamida ADP-ribosilada Ricina encontrado en habas de ricino, cataliza la ruptura de la subunidad grande de rRNA de los eucariotas Cicloheximida inhibe la peptidiltransferasa eucariótica La actinomicina D es un antineoplásico (anticancerigeno), producido por el hongo Streptomyces antibioticus que inhibe tanto a la RNA como a la DNA polimerasa, debido a que se une fuertemente al ADN de doble cadena. ESTABILIDAD DEL mRNA Regulación Proteína-Proteína : Operon Lac. Regulación por contenido iónico: Aconitasa aconitasa 5` CARENCIA Fe mRNA ferritina Receptor Transferrina EXCESO Fe Fe Fe Ferritina Receptor Transferrina