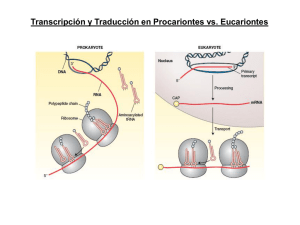
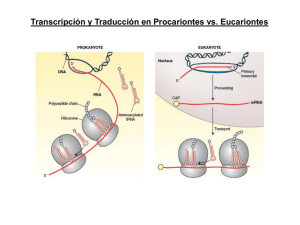
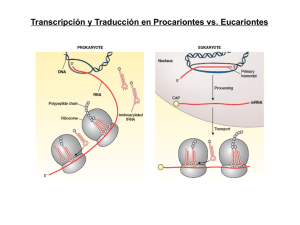
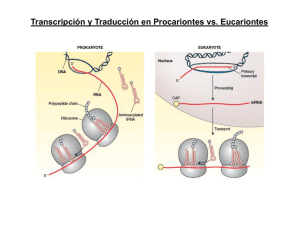
inicio de la traducción procarionte
Anuncio

Etapas de la traducción Inicio Alargamiento Terminación En procariontes: la traducción continúa a la transcripción La proteína se sintetiza empezando en el amino terminal Sitios en el ribosoma Centro peptidil transferasa (PTC) A: aceptor (aminoacil) P: peptidil E: salida (exit) Centro activador de GTPasa Centro decodificador Inicio subunidades ribosoma mRNA tRNAi-met factores de inicio GTP La interacción de la secuencia Shine-Dalgarno del mRNA con el rRNA 16S ubica el inicio de la traducción en procariotes Secuencia de reconocimiento del inicio de la traducción en procariontes: secuencia Shine-Dalgarno Presente en la región 5’ del mRNA, antes del codón de inicio de la traducción La secuencia Shine-Dalgarno interacciona con el rRNA16S (subunidad pequeña del ribosoma) En bacterias la selección del codón de inicio se realiza por interacción entre el mensaje y el ribosoma Shine-Dalgarno 5´AGGAGGU3´ INICIO Para el inicio de la traducción: Las subunidades ribosomales deben estar separadas La subunidad pequeña debe reconocer al mRNA El tRNA aminoacilado (fmet-tRNAmet o met-tRNA) debe colocarse en la posición P de la subunidad ribosomal pequeña Debe ocurrir el reconocimiento codón-anticodón de inicio Ocurre hidrólisis de al menos una molécula de GTP Factores de inicio de la traducción IFs (bacteria) o eIFs (eucariontes) Aminoácido con el que se inicia la traducción en procariotes En procariontes existen dos tRNAs de Metionina, uno iniciador y otro alargador El fMet-tRNA iniciador tiene características especiales En la mitad de las proteínas, la metionina es removida Reconoce los codones AUG ó GUG INICIO DE LA TRADUCCIÓN PROCARIONTE IF1 IF2 IF3 50S 30S [Mg2+] + 3 fMet 3 1 GDP 2 fMet 2 Shine-Dalgarno fMet 3 1 3 AUG + GTP fMet 2 2 1 AUG complejo de inicio de la traducción GTP 3 1 GTP FACTORES DE INICIO DE LA TRADUCCIÓN (PROCARIONTES) Factor Función IF1 Previene la unión de tRNAs en el sitio A de la subunidad 30S IF2 GTPasa que interacciona con 3 componentes claves durante iniciación: la subunidad 30S, IF1, y fMet-tRNAif-Met) IF3 Se une a 30S y evita re-asociación con 60S. Participa en el reconocimiento codon-anticodon f-met 5’ aa-tRNA IF3 IF1 E P A 3’ adición del 5’ 7mGpppG (cap) Procesamientos del mRNA eucarionte: terminación splicing - Capping - Splicing poliadenilación exportación - Poliadenilación nuclear - Exportación nuclear - Poliadenilación citoplasmática - Localización citoplasmática almacenaje Traducción Almacenaje traducción Degradación degradación En eucariotes se forma un complejo circularizado con los extremos 5’ y 3’ del mRNA Esto constituye el complejo 43S eIF2•GTP se une a tRNA-met para unirlo al complejo 43S eIF2B intercambia GDP x GTP en eIF2 eIF2•GDP Complejo mRNA-43S Complejo 48S (cuando ya se une el MettRNA) Ocurre un escaneo de la región 5’ no traducible hasta encontrar el primer AUG en contexto apropiado eIF4A: helicasa; utiliza ATP para deshacer estructura secundaria en el mRNA y permitir el paso del 40S Después de la formación del complejo ternario 48S, la subunidad grande del ribosoma se ensambla. Con este evento el ribosoma está listo para el alargamiento. Entorno del codón de inicio AUG En eucariontes: el primer AUG que permita pausar el ribosoma debe tener un entorno adecuado 5’CAP 40S C C A G C consenso C A U G G Resumen inicio de la traducción en eucariontes eIF3-eIF4G eIF4G: una proteína de anclaje PABP 2Apro eIF4E eIF4A eIF4A eIF3 132 1046 572 642 1201 1411 1560 Porción utilizada para la traducción independiente de Cap PABP eIF4A eIF4E eIF4A eIF3 132 Ya no funciona en traducción 572 642 1046 1201 1411 1560 X TRADUCCIÓN DEPENDIENTE DE 5’ CAP Apoptosis Infeccion viral Ciertos puntos del ciclo celular La iniciación de la traducción mediada por IRES (sitio de entrada interna del ribosoma) conlleva un inicio de la traducción independiente de CAP Los IRES presentan estructuras secundarias fundamentales para su función No existen motivos conservados dentro de estas secuencias, su actividad depende la integridad estructural La iniciación mediada por IRES no requiere al factor eIF4E, su dependencia de otros factores de inicio de la traducción es variable. • • • • • Rhinovirus Poliovirus Hepatitis A Virus Encephalomyocarditis Virus Foot and Mouth Disease Virus Alargamiento ribosoma mRNA tRNAs-aa factores de alargamiento (elongation factors) GTP GTP x aa Entrada del aminoacil tRNA Factores de alargamiento Bacteria EF-Tu EF-Ts EF-G Eucariontes eEF-1 eEF-1 eEF-2 1. Posicionamiento del aa-tRNAaa elongador correcto (EF-Tu/eEF-1) en el sitio A 2. Hidrólisis de GTP y cambio conformacional. Para regenerar EFTu•GTP se requiere EF-Ts Formación del enlace peptídico Bacteria Eucariontes La actividad peptidil transferasa, encargada de formar el enlace peptídico, no está en una proteína sino en el rRNA 23S y está conservada en todos los organismos 3. Ataque nucleofílico amino del aa2 al carboxilo del aa1 4. Posicionamiento del péptido sobre el tRNA del sitio A Translocación Bacteria Eucariontes EF-Tu EF-Ts EF-G eEF-1 eEF-1 eEF-2 5. Entrada de EF-G/eEF-2 6. Hidrólisis de GTP 7. Cambio conformacional y desplazamiento Ciclos de alargamiento Terminación ribosoma mRNA factores de terminación GTP proteína subunidades ribosoma mRNA tRNA libre Factores de terminación Bacteria Eucariontes RF1 eRF1 RF2 RF3 eRF3 RRF (ribosome release factor) IF3 EF-G eRF1 utiliza agua para hidrolizar el péptido Modelo de terminación propuesto para bacteria Moleculas que unen el mismo sitio en el ribosoma El gasto energético del proceso de traducción Se hidrolizan 2 GTP´s por cada aminoácido incorporado La hidrólisis promueve cambios conformacionales Cargado de tRNA con su aminoácido 1 ATP /aa TRADUCCIÓN Iniciación Alargamiento Terminación 1 GTP (1er aminoácido) 2 GTPs /aa 1 GTP Tarea: ¿Cuántos GTPs y ATPs se requieren para la síntesis de una proteína de 300 aa? Antibióticos inhibidores de la traducción Antibiótico/Toxina Organismo Función Tetraciclina Procarionte Sitio A subunidad 30S Cloramfenicol Procarionte Centro PTC subunidad 50S Puromicina Procarionte/Euca- Centro PTC subunidad 50S rionte Eritromicina Procarionte Tunel de salida del péptido naciente Acido fusídico Procarionte EF-G Ricina Procarionte Modifica el RNA en el centro activador de GTPasa Toxina de difteria Eucarionte Modifica EF-1A Cicloheximida Eucarionte Translocación del ribosoma durante elongación La puromicina inhibe la traducción porque se parece al aminoacil-tRNA La kirromicina bloquea a EF-Tu El ácido fusídico bloquea a EF-G Otros antibióticos... ¿Qué es lo que puede limitar la síntesis de proteínas en eucariontes? 1. Cantidad y eficiencia de mRNAs (específica) 2. Abundancia de ribosomas (global) 3. Actividad de la maquinaria traduccional (global o específica) 4. Velocidad de alargamiento (global o específica) Los dos principales puntos de regulación de la traducción en eucariontes eIF2 GTP eIF4E eIF4G eIF2B tRNAmet 2 Formación del complejo ternario PABP 1 Reconocimiento del mRNA por el complejo 43S