inicio de la traducción eucarionte
Anuncio

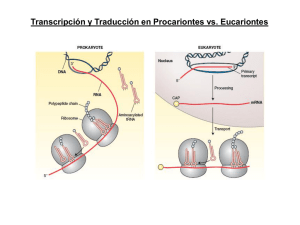
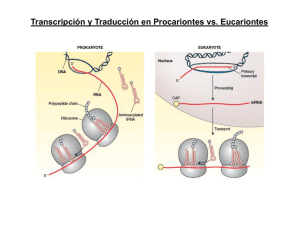
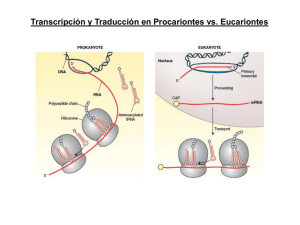
Transcripción y Traducción en Procariontes vs. Eucariontes Estructura del RNA mensajero: Procariontes vs. Eucariontes Alberts et al., 3rd ed., p.237 Etapas de la traducción v Iniciación v Elongación v Terminación Inicio subunidades separadas del ribosoma mRNA tRNAi-met factores de inicio GTP Para el inicio de la traducción: v Las subunidades ribosomales deben estar separadas v La subunidad pequeña debe reconocer al mRNA v El tRNA aminoacilado (fmet-tRNAmet o met-tRNA) debe colocarse en la posición P de la subunidad ribosomal pequeña v Debe ocurrir el reconocimiento codón-anticodón de inicio v Ocurre hidrólisis de al menos una molécula de GTP Factores de iniciación de la traducción IFs (bacteria) o eIFs (eucariontes) Existen dos tRNAs para Metionina, uno iniciador y otro elongador El fMet-tRNA iniciador tiene características especiales En la mitad de los casos, la metionina es removida de la proteína Reconoce los codones AUG ó GUG INICIO DE LA TRADUCCIÓN PROCARIONTE IF1 IF2 IF3 50S 30S [Mg2+] AUG 3 3 1 GDP 2 fMet + Shine-Dalgarno 3 1 fMet GTP 2 3 1 AUG complejo de inicio de la traducción fMet GTP + 2 fMet GTP 2 3 1 FACTORES DE INICIO DE LA TRADUCCIÓN (PROCARIONTES) Factor Función IF1 Previene la unión de tRNAs en el sitio A de la subunidad 30S IF2 GTPasa que interacciona con 3 componentes claves durante iniciación: la subunidad 30S, IF1, y fMet-tRNAif-Met) IF3 Se une a 30S y evita re-asociación con 50S. Participa en el reconocimiento codon-anticodon f-met 5’ aa-tRNA IF3 IF1 E P A 3’ INICIO DE LA TRADUCCIÓN EUCARIONTE 60S eIF1A eIF2 eIF3 eIF4 eIF5 40S [Mg2+] + Met 3 4 1A AUG 4 5 1A 2 3 4 Escaneo para buscar el AUG GDP Met GTP 5 2 3 1A AUG 3 Met GTP 5 2 4 3 1A AUG complejo de inicio de la traducción Met GTP + 2 Met GTP 2 3 1A Se forma un complejo circularizado con los extremos 5’ y 3’ del mRNA Los factores eIF4 NO existen en bacteria Interacciones en el complejo de inicio de la traducción Ocurre un escaneo de la región 5’ no traducible hasta encontrar el primer AUG en contexto apropiado eIF4A: helicasa; utiliza ATP para deshacer estructura secundaria en el mRNA y permitir paso de 40S Para regenerar eIF2•GTP que se une a tRNA-met para iniciar traducción, se requiere el factor eIF2B eIF2B intercambia GDP x GTP en eIF2 eIF2•GDP Resumen inicio de la traducción en eucariontes Complejo 43S eIF3-eIF4G Complejo 48S Traducción dependiente de CAP: Los mRNA eucariontes tienen CAP en el extremo 5’, por lo que su traducción es CAP dependiente y requiere a eIF4E Traducción independiente de CAP: Muchos virus que infectan células eucariontes tienen genoma de RNA que es traducido diréctamente. Estos virus NO portan CAP en su extremo 5’ y NO requieren a eIF4E. Su traducción inicia en un sitio interno río arriba del AUG llamado IRES (Internal Ribosome Entry Site). El IRES es reconocido por algunos de los eIFs celulares para reclutar la subunidad ribosomal 40S • • • • • Rhinovirus Poliovirus Hepatitis A Virus Encephalomyocarditis Virus Foot and Mouth Disease Virus eIF4G: una proteína de anclaje que es blanco de los virus IRES-dependientes PABP 2Apro eIF4E eIF4A eIF4A eIF3 132 1046 572 642 1201 1411 1560 Porción utilizada para la traducción independiente de Cap PABP eIF4A eIF4E eIF4A eIF3 132 Ya no funciona en traducción 572 642 1046 1201 1411 1560 X TRADUCCIÓN DEPENDIENTE DE 5’ CAP Apoptosis Infeccion viral Ciertos puntos del ciclo celular Elongación ribosoma mRNA tRNAs-aa factores de elongación GTP GTP x aa Factores de elongación Bacteria EF-Tu EF-Ts EF-G Eucariontes eEF-1α eEF-1β eEF-2 1. Posicionamiento del aa-tRNAaa elongador correcto (EF-Tu/eEF-1α) 2. Hidrólisis de GTP y cambio conformacional. Para regenerar EF-Tu•GTP se requiere EF-Ts Formación del enlace peptídico Bacteria Eucariontes Actividad peptidil transferasa del RNAr 23S/28S (Bases conservadas en todos los organismos) 3. Ataque nucleofílico amino del aa2 al carboxilo del aa1 4. Posicionamiento del péptido sobre el tRNA del sitio A Translocación Bacteria Eucariontes EF-Tu EF-Ts EF-G eEF-1α eEF-1β eEF-2 5. Entrada de EF-G/eEF-2 6. Hidrólisis de GTP 7. Cambio conformacional y desplazamiento ciclos de elongación Terminación ribosoma mRNA factores de terminación GTP proteína subunidades ribosoma mRNA tRNA libre Factores de terminación Bacteria Eucariontes RF1 RF2 RF3 RRF IF3 EF-G eRF1 eRF3 eRF1 utiliza agua para hidrolizar el péptido El gasto energético del proceso de traducción Se hidrolizan 2 GTP´s por cada aminoácido incorporado La hidrólisis promueve cambios conformacionales Cargado de tRNA con su aminoácido 1 ATP /aa TRADUCCION Iniciación 1 GTP (1er aminoácido) Elongación 2 GTPs /aa Terminación 1 GTP Resumen RIBOSOMA Centro peptidil transferasa (PTC) A: aceptor P: peptidil E: salida (exit) Centro activador de GTPasa Centro decodificador