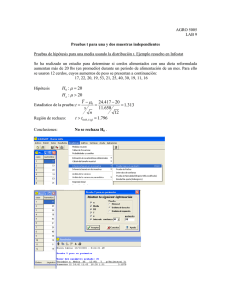
Modelos Mixtos en InfoStat
Anuncio

Modelos Mixtos en InfoStat Julio A. Di Rienzo Raúl Macchiavelli Fernando Casanoves Actualizado en Octubre de 2009 Modelos Mixtos en InfoStat Julio A. Di Rienzo es Profesor Asociado de Estadística y Biometría de la Facultad de Ciencias Agropecuarias de la Universidad Nacional de Córdoba, Argentina. Director del grupo de desarrollo de InfoStat y responsable de la implementación de la interfase con R que se presenta en esta obra ([email protected]). Raúl E. Macchiavelli es Catedrático de Biometría en el Facultad de Ciencias Agrícolas, Universidad de Puerto Rico - Mayagüez ([email protected]) Fernando Casanoves es el Jefe de la Unidad de Bioestadística del Centro Agronómico Tropical de Investigación y Enseñanza (CATIE). Anteriormente trabajó en la Facultad de Ciencias Agropecuarias de la Universidad Nacional de Córdoba, Argentina, donde participó del desarrollo de InfoStat ([email protected]). Modelos Mixtos en InfoStat AGRADECIMIENTOS Los autores agradecen a las Estadísticas Yuri Marcela García Saavedra y Jhenny Liliana Salgado Vásquez, de la Universidad del Tolima, Colombia, por la lectura crítica del manuscrito, la reproducción de la ejemplificación de este manual y los aportes sobre algunos detalles de la interfaz. Modelos Mixtos en InfoStat INDICE DE CONTENIDOS Introducción ................................................................................................................................1 Requerimientos (actualización 25/10/2009) .......................................................................1 Invocación del procedimiento de modelos lineales generales y mixtos ..................................2 Especificación de los efectos fijos...............................................................................................2 Especificación de los efectos aleatorios .....................................................................................5 Comparación de medias de tratamientos..................................................................................8 Especificación de la estructura de correlación y de varianza de los errores .........................9 Especificación de la estructura de correlación........................................................................ 10 Especificación de la parte fija .......................................................................................................... 12 Especificación de la parte aleatoria................................................................................................. 13 Especificación de la correlación de los errores ............................................................................... 14 Especificación de la estructura de varianzas de los errores .................................................... 18 Análisis de un modelo ajustado ...............................................................................................21 Ejemplos de Aplicación de Modelos Lineales Generales y Mixtos ......................................26 Estimación de componentes de varianza ................................................................................ 27 Aplicación de modelos mixtos para datos estratificados ........................................................ 49 Parcelas divididas ............................................................................................................................ 49 Parcelas divididas en un arreglo en bloques.................................................................................... 50 Parcelas divididas en un arreglo en diseño completamente aleatorizado........................................ 60 Parcelas subdivididas (split-split plot) ............................................................................................. 68 Aplicación de modelos mixtos para mediciones repetidas en el tiempo ................................ 77 Datos longitudinales......................................................................................................................... 77 Análisis de un ensayo de establecimiento de forrajeras ................................................................... 78 Análisis de un ensayo de drogas para asma..................................................................................... 96 Análisis de bolsas de descomposición ............................................................................................ 112 Uso de modelos mixtos para el control de la variabilidad espacial en ensayos agrícolas .... 125 Correlación espacial ...................................................................................................................... 125 Análisis de un ensayo comparativo de rendimientos en maní ........................................................ 126 Aplicaciones de modelos mixtos en otros diseños experimentales ...................................... 147 Diseño en franjas (strip-plot) ......................................................................................................... 147 Diseño experimental con dos factores y dependencia espacial ...................................................... 156 Diseños de testigos apareados........................................................................................................ 169 Referencias...............................................................................................................................181 Índice de cuadros ....................................................................................................................183 Índice de figuras......................................................................................................................183 ii Modelos Mixtos en InfoStat Introducción InfoStat implementa una interfase amigable de la plataforma R para la estimación de modelos lineales generales y mixtos a través de los procedimientos gls y lme de la librería nlme. La bibliografía de referencia de esta implementación, así como alguno de los ejemplos utilizados, corresponde a Pinheiro y Bates (2004). Las rutinas utilizadas para vincular la plataforma de desarrollo de InfoStat (Delphi) con el DCOM-R (una forma de correr R en el background) es un desarrollo de Dieter Menne ([email protected]). Requerimientos (actualización 25/10/2009) Para que InfoStat pueda tener acceso a R, debe estar instalado en su sistema el componente DCOM y R. Para ello se deben seguir los siguientes pasos (EN ESE ORDEN): a. Desinstalar R si estuviera previamente instalado en su computadora. b. Desinstalar DCOM si estuviera previamente instalado en su computadora c. Reiniciar su computadora d. Instalar DCOM DCOM 3.01B5 e. Reinstalar R R-2.9.2-win32 f. Correr R e instalar (desde los repositórios de R) la librería rscproxy g. Salir de R – Instalación concluida Nota: Si bien InfoStat se mantiene actualizado para las últimas versiones de DCOM y R, se recomienda utilizar las versiones que se pueden descargar de los vínculos (links) anteriores. En algunas configuraciones de Windows Vista hay inconvenientes para instalar la librería rscproxy. El síntoma es que cuando están instalando la librería, aparece un diálogo de opciones (dos opciones). No importa la opción que Ud. elija la instalación fallará. Solución: Entre al sitio del CRAN, busque y descargue manualmente el archivo rscproxy zipeado. Descomprímalo y cópielo (o muévalo) al directorio C:\Archio de Programas\R\R-2.9.0\library\. 1 Modelos Mixtos en InfoStat Invocación del procedimiento de modelos lineales generales y mixtos En el menú Estadísticas seleccionar el submenú Modelos lineales generales y mixtos, allí encontrará dos opciones. La primera, con el rótulo Estimación, invoca la ventana de diálogo que permite especificar la estructura del modelo. La segunda, rotulada Análisis –exploración de modelos estimados, se activa cuando algún modelo ha sido estimado previamente y contiene un conjunto de herramientas para el análisis diagnóstico. Especificación de los efectos fijos Comenzaremos indicando cómo ajustar un modelo de efectos fijos, utilizando el archivo Atriplex.IDB2 del conjunto de datos de prueba de InfoStat. Una vez abierto este archivo activar el menú Estadísticas, submenú Modelos lineales mixtos, opción Estimación. En la ventana de selección de variables, los factores de clasificación, covariables y variables dependientes pueden ser especificados como en un análisis de la varianza para efectos fijos. Para los datos en el archivo Atriplex.IDB2 especificar PG como variable respuesta y como criterios de clasificación a Tamaño y Episperma. Una vez que se acepta la selección realizada se mostrará la ventana principal de la interfase para modelos mixtos. Esta ventana contiene cinco solapas (Figura 1). Figura 1: Solapas con las opciones para especificación de un modelo lineal general y mixto. La primera permite especificar los efectos fijos del modelo y seleccionar opciones para la presentación de resultados y la generación de predicciones, obtener residuos del modelo y especificar el método de estimación. Por defecto el método de estimación es máxima verosimilitud restringida (REML). A la derecha de la ventana aparecerá una lista conteniendo las variables de clasificación y las covariables declaradas en la ventana de selección de variables. Para incluir un factor (variable de clasificación) o una covariable a la parte fija del modelo, basta hacer doble clic sobre el nombre del factor o covariable que se quiere incluir. Esta acción agregará una línea en la lista de efectos fijos. Doble clics adicionales sobre un factor o una covariable agregarán términos en líneas sucesivas, implícitamente separados por un signo “+” (modelo aditivo). Seleccionando con el ratón los factores principales y 2 Modelos Mixtos en InfoStat accionando el botón “*” se introduce un término que especifica la interacción entre los factores. Para el conjunto de datos en el archivo Atriplex.IDB2, incluir en el modelo de efectos fijos los factores Tamaño, Episperma y su interacción (Figura 2). Algunos de los textos en estas ventanas han sido aumentados de tamaño para mejorar su visualización (esto se logra moviendo el roller del ratón mientras la tecla Ctrl del teclado esta apretada). Si aceptamos esta especificación, en la ventana de resultados de InfoStat se obtendrá la salida que se muestra a continuación de la Figura 2. La salida que se obtiene es la más sencilla ya que no se han especificado características adicionales del modelo u otras opciones de análisis. La primera parte contiene la especificación de la forma en que se invocó la estimación del modelo en la sintaxis de R, e indica el nombre del objeto R que contiene al modelo y su estimación. En este caso modelo001_PG_REML. Esta especificación es sólo de interés para aquellos que están acostumbrados a ver las sentencias en R. La segunda parte muestra medidas de ajuste que son útiles para comparar distintos modelos ajustados a un conjunto de datos. AIC hace referencia al criterio de Akaike, BIC al Criterio Bayesiano de Información, logLik al logaritmo de la verosimilitud y Sigma a la desviación estándar residual. La tercera parte de esta salida presenta una tabla de análisis de la varianza mostrando las pruebas de hipótesis de tipo secuencial. 3 Modelos Mixtos en InfoStat Figura 2: Ventana con la solapa Efectos fijos desplegada para los datos del archivo Atriplex.IDB2. Modelos lineales generales y mixtos Especificación del modelo en R modelo001_PG_REML<-gls(PG~1+Tamano+Episperma+Tamano:Episperma ,method="REML" ,na.action=na.omit ,data=R.data01) Resultados para el modelo: modelo001_PG_REML Variable dependiente:PG Medidas de ajuste del modelo N 27 AIC 160.36 BIC 169.26 logLik -70.18 Sigma R2_0 9.07 0.92 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) Tamano Episperma Tamano:Episperma numDF F-value 1 1409.95 2 10.49 2 90.53 4 2.29 p-value <0.0001 0.0010 <0.0001 0.0994 4 Modelos Mixtos en InfoStat Especificación de los efectos aleatorios Los efectos aleatorios están asociados a grupos de observaciones. Ejemplos típicos son las medidas repetidas sobre un mismo individuo o las respuestas observadas en grupos de unidades experimentales homogéneas (bloques) o en los individuos de un mismo grupo familiar, etc. Estos efectos aleatorios son “agregados” a los efectos fijos de manera selectiva. Por lo tanto, en la especificación de los efectos aleatorios es necesario tener uno o más criterios de agrupamiento o estratificación, y elegir sobre qué efectos fijos se agregan los efectos aleatorios asociados. En el procedimiento lme de R, sobre el que se basa esta implementación, cuando hay más de un criterio de agrupamiento admisible, estos son anidados o encajados. En la segunda solapa del diálogo de especificación del modelo podemos elegir los criterios de estratificación o agrupamiento y la forma en que éstos incorporan efectos aleatorios a los componentes fijos. Para ejemplificar la especificación de los efectos aleatorios consideremos el archivo de prueba Bloque.IDB2. Este archivo contiene tres columnas: Bloque, Tratamiento y Rendimiento. En este ejemplo indicaremos que los bloques fueron seleccionados en forma aleatoria o producen un efecto aleatorio (por ejemplo, si los bloques son conjuntos de parcelas, el efecto de estos puede ser considerado aleatorio ya que su respuesta dependerá entre otras cosas de condiciones ambientales que no son predecibles), mientras que los tratamientos agregan efectos fijos. Para especificar este modelo, las dos primeras columnas del archivo de pruebas Bloque.IDB2 (Bloque y Tratamiento) se ingresarán como criterios de clasificación y la última (Rendimiento) como variable dependiente. El factor Tratamiento se incluirá en la solapa Efectos fijos como el único componente de esa parte del modelo. Para agregar el efecto aleatorio de los bloques, seleccionaremos la solapa Efectos aleatorios. Cuando se selecciona ésta solapa la lista Criterios de estratificación está vacía. Haciendo doble clic sobre Bloque en la lista de variables, se agrega éste factor de clasificación, como criterio de agrupamiento. La inclusión de un criterio de estratificación activa, en el panel inferior, un dispositivo que permite detallar la forma en que el efecto aleatorio entra en el modelo. En éste dispositivo hay una lista de componentes de la parte fija de modelo. El primer componente hace referencia a la Constante y el resto a los otros términos, en este caso Tratamiento (Figura 3). 5 Modelos Mixtos en InfoStat Figura 3: Ventana desplegada con la solapa Efectos aleatorios para los datos del archivo Bloque.IDB2. Dentro de la lista de términos fijos aparecen los criterios de estratificación previamente especificados. La combinación de ambas listas define los efectos aleatorios. Para ello, cada criterio de estratificación, dentro de cada efecto fijo, tiene asociado un check box. Cuando éste está tildado indica que hay un conjunto de efectos aleatorios asociados al efecto fijo correspondiente. El número de efectos aleatorios es igual al número de niveles que tiene el término fijo del modelo o a 1 en el caso de la constante o de las covariables. En el ejemplo que se ilustra se está incluyendo un efecto aleatorio inducido por los bloques sobre la constante. Esta especificación representa al siguiente modelo: yij i b j ij ; i 1,.., T ; j 1,..., B (1) donde yij es la respuesta al i-ésimo tratamiento en el j-ésimo bloque, la media general de rendimientos, i los efectos fijos de los tratamientos, b j el cambio del nivel medio de yij asociado al j-ésimo bloque y ij el término de error asociado a la 6 Modelos Mixtos en InfoStat observación yij . T y B son el número de niveles del factor de clasificación correspondiente al efecto fijo Tratamiento y al número de bloques respectivamente. Los b j se consideran, a diferencia de un efecto fijo, como variables aleatorias idénticamente distribuidas N 0, b2 y cuyas realizaciones se interpretan como los efectos de los distintos grupos o estratos (bloques, en este ejemplo). Luego, en estos modelos, los b j no se estiman, lo que se estima es el parámetro b2 que caracteriza a su distribución. Los ij también se interpretan como variables aleatorias idénticamente distribuidas N 0, 2 y describen a los errores aleatorios asociados a cada observación. Se supone, además, que las variables aleatorias b j y ij son independientes. La salida del ejemplo se muestra a continuación. La parte nueva de esta salida, respecto del ejemplo con el modelo lineal de efectos fijos, es que tiene una sección de parámetros para los efectos aleatorios. Especificación del modelo en R modelo002_Rendimiento_REML<-lme(Rendimiento~1+Tratamiento ,random=list(Bloque=pdIdent(~1)) ,method="REML" ,na.action=na.omit ,data=R.data03 ,keep.data=FALSE) Resultados para el modelo: modelo003_Rendimiento_REML Variable dependiente:Rendimiento Medidas de ajuste del modelo N 20 AIC 218.77 BIC 223.73 logLik -102.39 Sigma 160.65 R2_0 0.89 R2_1 0.93 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) Tratamiento numDF denDF F-value 1 12 2240.00 4 12 41.57 p-value <0.0001 <0.0001 Parámetros de los efectos aleatorios Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~1|Bloque Desvíos estándares relativos al residual y correlaciones (const) (const) 0.57 7 Modelos Mixtos en InfoStat En este caso se presenta la estimación de b (la desviación estándar de los b j relativa al residual) como 0.57. Al comienzo de la salida puede observarse la estimación de , la desviación estándar de los ij , como 160.65. Así, la varianza de los bloques puede calcularse como: b2 (0.57 160.65) 2 8385.15 Comparación de medias de tratamientos Siguiendo en la solapa Comparaciones (Figura 4), si en el panel que lista los términos fijos del modelo se tilda alguno de ellos, se obtiene una tabla de medias y errores estándares y una comparación múltiple entre medias del tipo LSD de Fisher (esta prueba está basada en una prueba de Wald) o la prueba de formación de grupos excluyentes DGC (Di Rienzo et ál. 2002). También se presentan varias opciones de corrección por comparaciones múltiples. Figura 4: Ventana desplegada con la solapa Comparaciones para los datos del archivo Bloque.IDB2. 8 Modelos Mixtos en InfoStat La salida correspondiente a la comparación de las medias de tratamientos se presenta a continuación. Medias ajustadas y errores estándares para Tratamiento LSD Fisher (alfa=0.05) Procedimiento de corrección de p-valores: No Tratamiento 300 225 150 75 0 Medias 3237.75 3093.50 2973.00 2498.50 1972.75 E.E. 92.47 A 92.47 A 92.47 92.47 92.47 B B C D Letras distintas indican diferencias significativas(p<= 0.05) La comparación de medias de tratamientos se muestra de la forma clásica como una lista ordenada en forma decreciente. Si el usuario desea controlar el error tipo I para la familia de todas las comparaciones de a pares, puede optar por alguno de los cuatro criterios implementados: Bonferroni (Hsu 199), Sidak (Hsu 1996), Benjamini-Hochberg (Benjamini y Hochberg 1995) o Benjamini-Yekutieli (Benjamini y Yekutieli 2001). Si para este mismo conjunto de datos se selecciona la opción Bonferroni, se obtiene el siguiente resultado: Medias ajustadas y errores estándares para Tratamiento LSD Fisher (alfa=0.05) Procedimiento de corrección de p-valores: Bonferroni Tratamiento 300 225 150 75 0 Medias 3237.75 3093.50 2973.00 2498.50 1972.75 E.E. 92.47 A 92.47 A 92.47 A 92.47 92.47 B B B B Letras distintas indican diferencias significativas(p<= 0.05) Especificación de la estructura de correlación y de varianza de los errores Las estructuras de varianzas y de covarianzas pueden modelarse separadamente. Para ello, InfoStat presenta dos solapas: en la solapa Correlación se encuentran las opciones para especificar la estructura de correlación de los errores y la solapa Heteroscedasticidad permite seleccionar distintos modelos para la función de varianza. A continuación se describen los contenidos de estas solapas. 9 Modelos Mixtos en InfoStat Especificación de la estructura de correlación Para ejemplificar la utilización de esta herramienta recurriremos a un ejemplo citado en Pinheiro y Bates (2004). Corresponde al archivo “Ovary” que contiene los datos de un estudio de Pierson y Ginther (1987) sobre el número de folículos mayores de 10 mm en ovarios de yeguas (mare). Estos números se registraron a los largo del tiempo desde 3 días antes de la ovulación y hasta 3 días después de la próxima ovulación. Los datos pueden cargarse desde la librería nlme utilizando el ítem de menú Aplicaciones>>Data set de R. Cuando se activa esta opción aparece la siguiente ventana de diálogo, que puede diferir en el número de librerías que estén instaladas en su configuración local de R (Figura 5). Figura 5: Ventana de diálogo para importar datos desde las librerías de R. En ella se muestra tildada la librería nlme y a la derecha la lista de archivos de datos en esa librería. Haciendo doble clic sobre “Ovary, nlme” se abrirá una tabla de datos de InfoStat conteniendo los datos correspondientes. El encabezamiento de la tabla abierta se muestra a continuación (Figura 6). 10 Modelos Mixtos en InfoStat Figura 6: Encabezamiento de la tabla de datos del archivo Ovary. Una gráfica de la relación entre número de folículos y el tiempo se muestra a continuación (Figura 7). 25 Follicles 20 15 10 5 0 -0,25 0,00 0,25 0,50 0,75 1,00 1,25 Time Figura 7: Relación entre el número de folículos (follicles) y el tiempo (Time). Pinheiro y Bates (2004) proponen ajustar un modelo donde el número de folículos depende linealmente del seno(2*pi*Time) y el coseno(2*pi*Time). Este modelo trata de reflejar las variaciones cíclicas del número de folículos mediante la inclusión de funciones trigonométricas. Además proponen la inclusión de un efecto aleatorio de yegua (Mare) sobre la constante del modelo y una auto-correlación de orden 1 de los errores dentro de cada hembra. El efecto aleatorio se incluyó para romper con la falta de independencia debida a efectos sujeto-dependientes que se expresan como perfiles 11 Modelos Mixtos en InfoStat paralelos del número de folículos a través del tiempo. El modelo propuesto tendría la siguiente forma general: yiTime 0 1sin 2* pi * Time 2 cos 2* pi * Time b0i it (2) donde los componentes aleatorios son b0i ~ N 0, bo2 y it ~ N 0, 2 . Por otra parte, la inclusión de una auto-correlación de orden 1 AR1 dentro de cada yegua tiene como propósito modelar una eventual correlación serial. Para especificar este modelo en InfoStat, indicaremos que follicles es la variable dependiente, que Mare es un criterio de clasificación y que Time es una covariable. Especificación de la parte fija La parte fija del modelo quedará indicada como se muestra en la Figura 8. InfoStat verifica que los elementos en esta ventana se corresponden con los factores y covariables listados en la parte derecha de la ventana. Figura 8: Ventana desplegada con la solapa Efectos fijos para los datos del archivo Ovary. 12 Modelos Mixtos en InfoStat Si no es así, porque no se han respetado minúsculas y mayúsculas (R es sensible a la tipografía), entonces InfoStat substituye eso términos por los apropiados. Pero si aún así, hay palabras que InfoStat no puede interpretar (como en este caso sin, cos y pi), entonces la línea queda marcada en rojo. Esto no quiere decir que esté incorrecta sino que puede estarlo y advierte al usuario para que la verifique. Especificación de la parte aleatoria La parte aleatoria se indica agregando a la lista de criterios de estratificación el factor Mare y especificando que el efecto yegua (Mare) es sobre la constante. Esto se indica tildando Mare dentro de Constante como se muestra en la Figura 9 (este tildado se agrega por defecto). Los términos sin(2*pi*Time) y cos(2*pi*Time) no presentan, en este caso, efectos aleatorios asociados. Figura 9: Ventana desplegada con la solapa Efectos aleatorios para los datos del archivo Ovary. 13 Modelos Mixtos en InfoStat Especificación de la correlación de los errores La especificación de la correlación autorregresiva de orden 1 para los errores dentro de cada hembra, se indica en la solapa Correlación 1 como se ilustra en la Figura 10. En R hay dos grupos de modelos de correlación. El primero corresponde a modelos de correlación serial, donde se supone que los datos están ordenados en una secuencia, y el segundo grupo modela correlaciones espaciales. En el primer grupo encontramos los modelos de simetría compuesta, sin estructura, autorregresivo de orden 1, autorregresivo continuo de orden 1 y el modelo ARMA(p,q), donde p indica el número de términos autorregresivos y q el número de términos de medias móviles (moving average). Todos estos modelos suponen que los datos están ordenados en una secuencia. Por defecto, InfoStat asume la secuencia en la que los datos están dispuestos en el archivo, pero si existe una variable que los ordena de manera diferente, ésta debe indicarse en el casillero Variable que indica el orden de las observaciones (para que este casillero se active hay que seleccionar alguna de las estructuras de correlación). Esta variable debe ser entera para la opción autorregresiva. Por este motivo, InfoStat agrega en la sentencia traducida al lenguaje R, una indicación para que la variable sea interpretada como entera. En el ejemplo que estamos ilustrando, la variable Time es un número real que codifica el tiempo relativo a un punto de referencia y está en una escala inapropiada para usarla como criterio de ordenamiento. Sin embargo, como los datos están ordenados por tiempo dentro de cada yegua (Mare), esta especificación puede omitirse (Figura 10). 1 Si los errores se suponen independientes (no correlacionados), entonces debe seleccionarse la primera opción de la lista de estructura de correlación (seleccionada por defecto). 14 Modelos Mixtos en InfoStat Figura 10: Ventana desplegada con la solapa Correlación para los datos del archivo Ovary. Si los datos no estuvieran ordenados en forma ascendente dentro del criterio de agrupamiento (Mare), habría que agregar una variable que identifique el orden. Para agregar una variable de ordenamiento su nombre puede escribirse o arrastrarse con el ratón desde la lista de variables, al casillero correspondiente. Es usual que la estructura de correlación esté asociada a un criterio de agrupamiento, en este caso Mare. Esto se indica en el panel rotulado Criterios de agrupamiento (para que este casillero se active hay que seleccionar alguna de las estructuras de correlación). Si se incluye más de un criterio, InfoStat construye tantos grupos como combinación de niveles en los factores de clasificación que se especifiquen. En la parte inferior de la ventana, rotulada Expresión resultante, se muestra la expresión R que se está especificando para la componente “corr=” de gls o lme. Esta expresión es sólo informativa y no puede editarse. 15 Modelos Mixtos en InfoStat A continuación se presenta la salida completa del modelo ajustado conteniendo la tabla de análisis de la varianza de los efectos fijos, que en este caso son pruebas sobre la pendiente asociada a las covariables sin(2*pi*Time) y cos(2*pi*Time). A continuación, se observa que la desviación estándar del componente aleatorio de la ordenada al origen es 0.77 veces la desviación estándar residual y que el parámetro phi del modelo autorregresivo es 0.61. Modelos lineales generales y mixtos Especificación del modelo en R Modelo000_follicles_REML<lme(follicles~1+sin(2*pi*Time)+cos(2*pi*Time) ,random=list(Mare= pdIdent(~1)) ,correlation=corAR1(form=~1|Mare) ,method="REML" ,na.action=na.omit ,data=R.data2 ,keep.data=FALSE) Variable dependiente:follicles Medidas de ajuste del modelo N 308 AIC 1562.45 BIC 1584.77 logLik -775.22 Sigma R2_0 3.67 0.21 R2_1 0.56 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) sin(2 * pi * Time) cos(2 * pi * Time) numDF denDF F-value 1 295 163,29 1 295 34,39 1 295 2,94 p-value <0,0001 <0,0001 0,0877 Parámetros de los efectos aleatorios Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~1|Mare Desvíos estándares relativos al residual y correlaciones (Const) (Const) 0.77 Estructura de correlación Modelo de correlación: AR(1) Formula: ~ 1 | Mare Parámetros del modelo Phi Estimación 0.61 16 Modelos Mixtos en InfoStat Los valores predichos por el modelo ajustado anteriormente versus el tiempo se presentan en la Figura 11. La línea de trazo negro representa la estimación del promedio poblacional y corresponde a la parte fija del modelo. Las curvas paralelas a esta son las predicciones para cada yegua derivadas de la inclusión de un efecto aleatorio (sujeto específico) sobre la constante. La inclusión de errores correlacionados según un modelo autorregresivo de orden 1 tuvo por objeto (según nuestra interpretación) contemplar la falta de independencia generada por el alejamiento de la curva de folículos de cada yegua respecto de las curvas de folículos que se generan permitiendo una variación sujeto-específica solo para la constante. Las discrepancias respecto del modelo que incluye solo desviaciones sujeto-específicas para la constante pueden visualizarse ajustando un modelo con efectos aleatorios sobre todos los parámetros de la parte fija (Figura 12). 22 Poblacional Mare 03 Mare 06 Mare 09 Mare 01 Mare 04 Mare 07 Mare 10 Mare 02 Mare 05 Mare 08 Mare 11 follicles 17 12 7 2 -0,30 0,10 0,50 0,90 1,30 Time Figura 11: Funciones ajustadas para el número poblacional de folículos (línea sólida negra) y para cada yegua originada por el efecto aleatorio sobre la constante (archivo Ovary). 17 Modelos Mixtos en InfoStat 22 18 follicles 14 10 6 2 -0,30 0,10 0,50 0,90 1,30 Time Figura 12: Funciones ajustadas para el número de folículos para cada yegua originada por la inclusión de efectos aleatorios sobre todos los parámetros de la parte fija del modelo (archivo Ovary). Especificación de la estructura de varianzas de los errores Este módulo permite contemplar modelos heteroscedásticos. La heteroscedasticidad sin embargo no tiene un origen único y así como se modela la correlación entre los errores, la heteroscedasticidad también puede modelarse. El modelo para las varianzas de los errores se puede especificar de la siguiente manera: var( i ) 2 g 2 ( i , z i , δ) donde g (.) se conoce como función de varianza. Esta función puede depender de la esperanza ( i ) de Yi (la variable de respuesta), de un conjunto de covariables z i y de un vector de parámetros δ . InfoStat, a través de R, estima los parámetros δ de acuerdo a la función de varianza seleccionada. La solapa Heteroscedasticidad se muestra en la Figura 13. Las funciones de varianza admitidas pueden ser identidad (varIdent), exponencial (varExp), potencia (varPower), potencia corrida por una constante (varConstPower), o fija (varFixed). R admite que varios modelos de varianza puedan superponerse, es decir, que para ciertos grupos de datos la varianza puede estar asociada con alguna covariable y para otros con otra. La especificación simultánea de varios 18 Modelos Mixtos en InfoStat modelos para la función de varianza se obtiene, simplemente, marcando y especificando cada uno de los componentes y agregándolos a la listas de funciones de varianza. InfoStat arma la sentencia apropiada para R. En la solapa Heteroscedasticidad para el ejemplo de los folículos, hemos indicado que la varianza de los errores es distinta para cada yegua, seleccionando varIdent como modelo de la función de varianza y escribiendo Mare en Criterios de agrupamiento. Figura 13: Ventana con la solapa Heteroscedasticidad desplegada para los datos del archivo Ovary. A continuación se presenta la salida del ajuste incluyendo estimaciones de la desviación estándar del error para cada yegua. También aquí las desviaciones estándar están expresadas en términos relativos a la desviación estándar residual. Además, el primer nivel del criterio de agrupamiento especificado para calcular estas desviaciones estándar diferenciales, es siempre inicializado en 1 porque de otra forma el modelo no es identificable. En la salida se observa que la hembra 5 tiene una variabilidad en el número de folículos comparativamente mayor que las otras hembras. 19 Modelos Mixtos en InfoStat El modelo para estos datos sería: yiTime 0 1sin 2* pi * Time 2 cos 2* pi * Time b0i it (3) donde los componentes aleatorios son b0i ~ N 0, bo2 y it ~ N 0, i2 . Obsérvese que la varianza residual está sub-indicada con el índice que identifica a las yeguas. Como es usual, los componentes aleatorios del modelo se suponen independientes. Luego si tomamos una yegua al azar la varianza de la respuesta sería la suma de las varianzas de la parte aleatoria, es decir var( yiTime ) b20 i2 , o sea (3.57*0.8)2 + (3.57*gi)2, donde gi es la función de varianza para una yegua elegida aleatoriamente. Ahora bien, cuando se condiciona a una yegua dada (por ejemplo la 5), el efecto individuo ( b0i ) está fijado, así que la varianza de la yegua 5 solo está asociada a la parte residual y además la función de varianza queda especificada, (es decir, hay que usar g5) y la varianza sería (3.57*1.34)2. Modelos lineales generales y mixtos Especificación del modelo en R Modelo001_follicles_REML<lme(follicles~1+sin(2*pi*Time)+cos(2*pi*Time) ,random=list(Mare= pdIdent(~1)) ,weight=varComb(varIdent(form=~1|Mare)) ,correlation=corAR1(form=~1|Mare) ,method="REML" ,na.action=na.omit ,data=R.data5 ,keep.data=FALSE) Variable dependiente:follicles Medidas de ajuste del modelo N 308 AIC 1569.02 BIC 1628.55 logLik -768.51 Sigma R2_0 3.57 0.21 R2_1 0.56 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) sin(2 * pi * Time) cos(2 * pi * Time) numDF denDF F-value 1 295 156.36 1 295 34.22 1 295 3.18 p-value <0.0001 <0.0001 0.0756 20 Modelos Mixtos en InfoStat Parámetros de los efectos aleatorios Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~1|Mare Desvíos estándares relativos al residual y correlaciones (Const) (Const) 0.80 Estructura de correlación Modelo de correlacion: AR(1) Formula: ~ 1 | Mare Parámetros del modelo Phi Estimación 0.61 Estructura de varianzas Modelo de varianzas: varIdent Formula: ~ 1 | Mare Parámetros del modelo Parámetro 1 2 3 4 5 6 7 8 9 10 11 Estim 1.00 1.01 1.20 0.82 1.34 1.05 0.92 1.06 0.93 0.99 0.77 Análisis de un modelo ajustado Cuando InfoStat ajusta un modelo lineal general o mixto con el menú Estimación, se activa el menú Análisis-exploración de modelos estimados. En este diálogo aparecen varias solapas como se muestra en la Figura 14. 21 Modelos Mixtos en InfoStat Figura 14: Ventana comparación de modelos generales y mixtos con la solapa Diagnóstico desplegada (archivo Atriplex.IDB2). El ejemplo usado en este caso es el del archivo Atriplex.IDB2, sobre el que se estimaron 2 modelos de efectos fijos, el modelo000_PG_REML que contiene los efectos Tamaño, Episperma y su interacción, y el modelo001_PG_REML que solo contiene los efectos principales de Tamaño y Episperma. La solapa Modelos sólo aparece en el caso que haya más de un modelo estimado y presenta una lista de los modelos evaluados en un “check-list”. Los modelos tildados, aparecen en una lista con sus estadísticos resumen y una prueba de hipótesis de igualdad de modelo cuya aplicabilidad debe tomarse con cautela ya que no todos los modelos son estrictamente comparables. De todas formas los criterios AIC y BIC son buenos indicadores para seleccionar el modelo más parsimonioso. La solapa Combinaciones lineales tiene como propósito probar hipótesis sobre combinaciones lineales. La hipótesis que se prueba es que la esperanza de la combinación lineal es cero. En esta ventana de diálogo aparecen listados los parámetros fijos del modelo que se haya seleccionado de la lista que aparece en la parte derecha de 22 Modelos Mixtos en InfoStat la pantalla (Importante: por defecto siempre está seleccionado el último de la lista). En la parte inferior de la pantalla hay un campo de edición donde pueden especificarse las constantes de la combinación lineal. A medida que los coeficientes se van agregando, los parámetros correspondientes se van coloreando para facilitar la especificación de las constantes, como se ilustra en la Figura 15. Figura 15: Ventana comparación de modelos generales y mixtos con la solapa Combinaciones lineales desplegada (archivo Atriplex.IDB2). Finalmente la solapa Diagnóstico tiene 3 subsolapas (Figura 14). La primera, identificada como “Residuos vs…” tiene dispositivos que sirven para generar de manera sencilla gráficos del tipo boxplot para los residuos estandarizados vs. cada uno de los factores fijos del modelo o diagramas de dispersión entre los residuos estandarizados y las covariables del modelo o los valores predichos. Asimismo, es posible obtener el gráfico QQ-plot normal. La segunda solapa, identificada como “ACF-SV”, permite generar un gráfico de la función de auto-correlación (útil para el diagnóstico de correlaciones seriales) y la tercera, identificada como LevelPlot, permite generar gráficos de residuos vs. coordenadas espaciales para generar un mapa del sentido e 23 Modelos Mixtos en InfoStat intensidad de los residuos. Esta herramienta es útil en el diagnóstico de estructuras de correlación espacial. Para ejemplificar el uso de la solapa ACF-FV consideremos el ejemplo de los folículos (archivo Ovary). En este ejemplo se argumentó que la inclusión del término autorregresivo de orden 1 tenía por objeto corregir una falta de independencia generada por las discrepancias entre los ciclos individuales de cada yegua respecto de los ciclos individuales que solo diferían del ciclo promedio poblacional por una constante (Figura 11). El gráfico de la autocorrelación serial de los residuos correspondiente a un modelo sin la inclusión de la autocorrelación de orden 1 muestra un claro patrón autorregresivo (Figura 16). Por otra parte, el gráfico de la autocorrelación de los residuos para el modelo que contempla la autocorrelación mediante un término autorregresivo de orden 1, corrige la falta de independencia (Figura 17). Autocorrelation 0.8 0.6 0.4 0.2 0.0 -0.2 0 5 10 Lag Figura 16: Función de autocorrelación de los residuos del modelo presentado en la Ecuación (2) excluyendo la modelación de la autocorrelación serial. 24 Modelos Mixtos en InfoStat Autocorrelation 0.8 0.6 0.4 0.2 0.0 -0.2 0 5 10 Lag Figura 17: Función de autocorrelación de los residuos del modelo presentado en la Ecuación (2) incluyendo la modelación de la autocorrelación serial. Las facilidades de la solapa Diagnóstico tienen por propósito permitir al investigador un rápido diagnóstico de los eventuales problemas de adecuación tanto de la parte fija como aleatoria del modelo ajustado. En la presentación de ejemplos se ilustrará más extensamente el uso de estas herramientas. 25 Modelos Mixtos en InfoStat Ejemplos de Aplicación de Modelos Lineales Generales y Mixtos Modelos Mixtos en InfoStat Estimación de componentes de varianza En áreas como el mejoramiento genético animal o vegetal es de particular interés el cálculo de componentes de varianza. Estos son usados para obtener heredabilidades, respuesta a la selección, coeficientes de variabilidad genética aditiva, coeficientes de diferenciación genética, etc. Los modelos lineales mixtos pueden usarse para estimar los componentes de varianza, por medio del estimador de máxima verosimilitud restringida (REML). En muchos estudios de genética de poblaciones se trabaja con varias poblaciones que a su vez están representadas por uno o más individuos de distintas familias. En este caso se cuenta con dos factores en el modelo, las poblaciones y las familias dentro de cada población. Para ejemplificar el uso de componentes de varianza se usan los datos que se presentan en el archivo Compvar.IDB2 (Navarro et ál. 2005). Estos datos provienen de un ensayo de siete poblaciones de cedro (Cedrela odorata L.) con un total de 115 familias. Para algunas familias se cuenta con repeticiones y para otras no. Además, el número de familias dentro de cada población no es el mismo. Las variables registradas son el largo promedio de las semillas (largo), el diámetro, el largo del tallo y número de hojas de plantines de cedro. Además de estimar los componentes de varianza, los investigadores están también interesados en comparar las medias de las poblaciones. Podemos considerar varios espacios de inferencia, de acuerdo al diseño y a los intereses de los investigadores. Si las poblaciones son una muestra aleatoria de un conjunto grande de poblaciones, entonces la inferencia estará orientada a este conjunto grande de poblaciones. El efecto de las poblaciones estudiadas es aleatorio, y el interés será la estimación de los componentes de varianza debida a poblaciones y a familias dentro de poblaciones. Otro aspecto de interés serán los predictores BLUP de los efectos aleatorios (en especial los de poblaciones). Si la inferencia se orienta solamente a las poblaciones estudiadas, el efecto de población es fijo, y el interés principal es estimar y comparar las medias de poblaciones. Si la media de una población se interpreta como un promedio a través de todas las posibles familias de dicha población (no solamente las estudiadas), entonces el efecto de familia Modelos Mixtos en InfoStat es aleatorio. En este caso interesará estimar el componente de varianza debido a familia dentro de poblaciones, y predecir los efectos de las familias estudiadas (BLUP). Un tercer espacio de inferencia es cuando el interés reside solamente en las poblaciones y las familias estudiadas. En este caso ambos efectos son fijos. Este tipo de modelo presenta severas limitaciones, tanto en su interpretación como en su implementación. Debido a esto, este modelo no se considerará en este tutorial. Para el análisis de los datos del archivo Compvar.IDB2 se ajustarán los dos primeros casos discutidos: Modelo 1: Poblaciones aleatorias y familias aleatorias Modelo 2: Poblaciones fijas y familias aleatorias Primero se selecciona el menú Estadísticas, submenú Modelos lineales generales y mixtos y escogemos Estimación. Al realizar esta selección aparecerá la ventana de selección de variables, donde especificamos como variables dependientes a Largo, Diametro, Largodetallo y Numerodehojas y como criterios de clasificación a Población y Familia (Figura 18). Figura 18: Ventana de selección de variables para Modelos lineales generales y mixtos con datos del archivo Compvar.IDB2. 28 Modelos Mixtos en InfoStat Modelo 1: Para el cálculo de los componentes de varianza se deben especificar las variables como en la Figura 18. Posteriormente, en la solapa Efectos aleatorios se debe declarar primero a Población y luego a Familia, ya que R asume que las distintas componentes aleatorias que se van agregando secuencialmente están anidadas en los factores declarados con anterioridad. En la subventana Mostrar se tildaron las opciones que se muestran en la Figura 19, y se sacó el tilde que tiene por defecto para presentar los Desvíos estándares relativos al desvío estándar residual. Figura 19: Ventana con la solapa Efectos aleatorios desplegada para los datos del archivo Compvar.IDB2 para la especificación del Modelo 1. En la solapa Efectos fijos no debe aparecer ningún efecto, y el método de estimación debe ser el de máxima verosimilitud restringida (REML), que es la opción por defecto. Observar que se desactivó la opción por defecto Desvíos estándares relativos al desvío estándar residual, por lo que las estimaciones que aparecen serán directamente los desvíos estándares absolutos. A continuación se presenta la salida obtenida con estas especificaciones solo para la variable Largo. 29 Modelos Mixtos en InfoStat Modelos lineales generales y mixtos Especificación del modelo en R modelo000_Largo_REML<-lme(Largo~1 ,random=list(Poblacion=pdIdent(~1) ,Familia=pdIdent(~1)) ,method="REML" ,na.action=na.omit ,data=R.data00 ,keep.data=FALSE) Resultados para el modelo: modelo000_Largo_REML Variable dependiente:Largo Medidas de ajuste del modelo N 214 AIC 2016.47 BIC 2029.91 logLik -1004.23 Sigma R2_0 21.53 R2_1 0.51 R2_2 0.76 AIC y BIC menores implica mejor Parámetros de los efectos aleatorios Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~1|Poblacion Desvíos estándares y correlaciones (const) (const) 27.16 Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~1|Familia dentro de Poblacion Desvíos estándares y correlaciones (const) (const) 14.80 Intervalos de confianza (95%) para los parámetros de los efectos aleatorios Formula: ~1|Poblacion sd(const) LI(95%) 15.09 Est. LS(95%) 27.16 48.89 Formula: ~1|Familia dentro de Poblacion sd(const) LI(95%) 10.72 Est. LS(95%) 14.80 20.43 Intervalo de confianza (95%) para sigma lower est. upper sigma 18.77 21.53 24.70 30 Modelos Mixtos en InfoStat A partir de las estimaciones de desvíos e intervalos de confianza para los desvíos, se obtienen las componentes de varianza y sus intervalos de confianza (Cuadro 1). Cuadro 1. Componentes de varianza estimados para los datos del archivo Compvar.IDB2 Componente Población Varianza estimada 2 pob 27.162 737.66 Familia dentro de 2fam ( pob ) 14.802 219.04 población Residual 2 res 21.532 463.54 IC para la varianza Variabilidad relativa al total (%) (15.092 , 48.882 ) 52.0 (10.722 , 20.432 ) 15.4 (18.77 2 , 24.702 ) 32.6 De acuerdo a los resultados presentados en la tabla anterior, es interesante resaltar que la variabilidad de la familias dentro de poblaciones es menor que la variabilidad residual con lo cual no hay una diferenciación de familias dentro de poblaciones. La mayor variación, en tanto, es atribuible a diferencias entre poblaciones. Ahora veremos cómo es el diagnóstico para el Modelo 1, es decir, tanto los efectos de familia como los de población aleatorios. Para esto vamos al submenú Análisisexploración de modelos estimados y se solicitan los gráficos de diagnóstico (Figura 20). El análisis diagnóstico de este modelo permite determinar una fuerte falta de homogeneidad de varianzas residual (Figura 21). 31 Modelos Mixtos en InfoStat 2 1 -2 -1 0 Cuantiles muestrales 1 0 -2 -1 Res.cond.estand.Pearson 2 Figura 20: Ventana Análisis-exploración de modelos estimados con la solapa Diagnóstico desplegada para el Modelo 1 con los datos del archivo Compvar.IDB2. 20 40 60 80 fitted(modeloLargocm_7_REML,level=2) -3 -2 -1 0 1 2 3 Cuantiles teóricos Figura 21: Gráficos de diagnóstico obtenidos para la variable largo y el modelo 1 para los datos del archivo Compvar.IDB2. 32 Modelos Mixtos en InfoStat En la figura anterior los residuos estandarizados de Pearsons son aproximaciones de errores y por lo tanto la heteroscedasticidad observada de modelarse a este nivel. Para corregir la falta de homogeneidad a este nivel se considera el Modelo 1 (Población y Familia como factores aleatorios) con varianzas residuales heterogéneas. Para incorporar las varianzas residuales eventualmente distintas para cada nivel de Población, en la solapa heterogeneidad se debe especificar el factor población como se muestra en la Figura 22. Figura 22: Ventana con la solapa Heteroscedasticidad desplegada para los datos del archivo Compvar.IDB2 para la especificación de varianzas heterogéneas para poblaciones. A continuación se presenta la salida para el Modelo 1 con varianzas residuales heterogéneas por Población y tildando en la solapa de Efectos aleatorios la opción Matriz de efectos aleatorios para obtener los estimadores BLUP. 33 Modelos Mixtos en InfoStat Modelos lineales generales y mixtos Especificación del modelo en R modelo004_Largo_REML<-lme(Largo~1 ,random=list(Poblacion=pdIdent(~1) ,Familia=pdIdent(~1)) ,weight=varComb(varIdent(form=~1|Poblacion)) ,method="REML" ,na.action=na.omit ,data=R.data00 ,keep.data=FALSE) Resultados para el modelo: modelo002_Largo_REML Variable dependiente:Largo Medidas de ajuste del modelo N 214 AIC 1872.14 BIC 1905.75 logLik -926.07 Sigma R2_0 2.32 R2_1 0.51 R2_2 0.51 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) numDF denDF F-value 1 108 21.59 p-value <0.0001 Parámetros de los efectos aleatorios Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~1|Poblacion Desvíos estándares y correlaciones (const) (const) 27.72 Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~1|Familia dentro de Poblacion Desvíos estándares y correlaciones (const) (const) 1.56 Intervalos de confianza (95%) para los parámetros de los efectos aleatorios Formula: ~1|Poblacion sd(const) LI(95%) 15.61 Est. LS(95%) 27.72 49.24 Formula: ~1|Familia dentro de Poblacion sd(const) LI(95%) 0.47 Est. 1.56 LS(95%) 5.14 34 Modelos Mixtos en InfoStat Estructura de varianzas Modelo de varianzas: varIdent Formula: ~ 1 | Poblacion Parámetros de la función de varianza Parámetro Charagre Escarcega Esclavos La Paz Pacífico Sur Xpujil Yucatán Estim 1.00 13.09 11.64 15.94 2.81 13.38 12.54 Coeficientes (BLUP) de los efectos aleatorios (~1|Poblacion) Charagre Escarcega Esclavos La Paz Pacífico Sur Xpujil Yucatán const -41.20 15.42 16.12 19.80 -36.51 23.29 3.08 Coeficientes (BLUP) de los efectos aleatorios (~1|Familia in Poblacion) Charagre/Ch_71 Charagre/Ch_710 Charagre/Ch_711 Charagre/Ch_712 Charagre/Ch_713 Charagre/Ch_714 Charagre/Ch_715 Charagre/Ch_72 Charagre/Ch_73 Charagre/Ch_74 Charagre/Ch_75 Charagre/Ch_76 Charagre/Ch_77 Charagre/Ch_78 Charagre/Ch_79 Escarcega/Es_1126 Escarcega/Es_1127 Escarcega/Es_1128 Escarcega/Es_1129 Escarcega/Es_1130 Escarcega/Es_1131 Escarcega/Es_1132 Escarcega/Es_1133 Escarcega/Es_1134 Escarcega/Es_1135 Escarcega/Es_1136 Escarcega/Es_1137 const -1.07 0.59 1.31 1.42 -0.95 -1.07 -0.70 0.70 -0.83 -0.35 -0.59 -0.08 -0.47 0.48 1.48 7.2E-04 0.18 0.14 0.07 3.6E-04 -0.06 0.21 0.01 -0.11 -0.09 -0.08 -0.17 35 Modelos Mixtos en InfoStat Escarcega/Es_1138 Escarcega/Es_1139 Escarcega/Es_1142 Escarcega/Es_1148 Esclavos/Ec_31 Esclavos/Ec_310 Esclavos/Ec_311 Esclavos/Ec_312 Esclavos/Ec_313 Esclavos/Ec_314 Esclavos/Ec_315 Esclavos/Ec_316 Esclavos/Ec_317 Esclavos/Ec_318 Esclavos/Ec_319 Esclavos/Ec_32 Esclavos/Ec_320 Esclavos/Ec_33 Esclavos/Ec_34 Esclavos/Ec_35 Esclavos/Ec_36 Esclavos/Ec_37 Esclavos/Ec_38 Esclavos/Ec_39 La Paz/LP_41 La Paz/LP_410 La Paz/LP_411 La Paz/LP_412 La Paz/LP_413 La Paz/LP_414 La Paz/LP_415 La Paz/LP_42 La Paz/LP_43 La Paz/LP_44 La Paz/LP_45 La Paz/LP_46 La Paz/LP_48 La Paz/LP_49 Pacífico Sur/PS_6204 Pacífico Sur/PS_6206 Pacífico Sur/PS_6207 Pacífico Sur/PS_6208 Pacífico Sur/PS_6209 Pacífico Sur/PS_6210 Pacífico Sur/PS_6211 Pacífico Sur/PS_6212 Pacífico Sur/PS_6213 Pacífico Sur/PS_6214 Pacífico Sur/PS_6215 Pacífico Sur/PS_6216 Pacífico Sur/PS_6217 Pacífico Sur/PS_6218 Pacífico Sur/PS_6219 Pacífico Sur/PS_6220 Pacífico Sur/PS_6221 Pacífico Sur/PS_6222 Pacífico Sur/PS_660 Xpujil/Xp_11 Xpujil/Xp_110 Xpujil/Xp_112 Xpujil/Xp_113 0.16 -0.08 0.08 -0.20 -0.08 0.08 -0.07 -0.03 -0.22 0.28 -0.34 0.15 0.04 -0.08 0.04 -0.07 0.18 -3.7E-03 -0.11 0.15 -0.17 0.18 0.08 0.05 -0.13 0.14 0.11 0.16 -0.08 -0.01 -0.13 0.01 -0.01 -0.01 0.02 -0.07 -0.01 0.07 -0.46 -0.58 0.52 -0.33 -0.15 0.31 -0.22 -0.43 0.03 -0.56 -0.07 1.80 -0.12 0.88 -0.35 -0.51 -0.12 -0.48 0.72 -0.12 0.02 3.8E-03 -0.07 36 Modelos Mixtos en InfoStat Xpujil/Xp_114 Xpujil/Xp_115 Xpujil/Xp_116 Xpujil/Xp_117 Xpujil/Xp_118 Xpujil/Xp_119 Xpujil/Xp_12 Xpujil/Xp_120 Xpujil/Xp_122 Xpujil/Xp_123 Xpujil/Xp_15 Xpujil/Xp_16 Xpujil/Xp_17 Xpujil/Xp_18 Xpujil/Xp_19 Yucatán/Yu_1111 Yucatán/Yu_1114 Yucatán/Yu_1115 Yucatán/Yu_1116 Yucatán/Yu_1117 Yucatán/Yu_1118 Yucatán/Yu_1119 Yucatán/Yu_1121 Yucatán/Yu_1122 Yucatán/Yu_1123 Yucatán/Yu_1124 Yucatán/Yu_1125 0.02 -0.12 0.17 0.11 0.08 0.18 -0.01 0.19 -0.21 -0.27 0.02 0.03 0.03 -0.05 0.07 -0.17 -0.19 -0.04 0.02 0.05 0.03 0.10 -0.06 0.20 -0.09 -0.05 0.20 Intervalo de confianza (95%) para sigma sigma lower est. 1.59 2.32 upper 3.38 Este modelo presenta valores más bajos de AIC y BIC que el modelo sin varianzas heterogéneas para Población y Familia dentro de Población. Observamos que las varianzas de las poblaciones son bien diferentes: La población La Paz tiene la mayor varianza estimada en (15.94*2.32)2 = 1367.57 mientras que la de menor varianza es (2.32*1)2 = 5.38. Al comparar los modelos con varianzas heterogéneas y homogéneas mediante una prueba de cociente de verosimilitud se corrobora que el modelo con varianzas heterogéneas es el mejor (p<0.0001) como se muestra en la siguiente salida. Comparación de modelos Modelo000_Largo_REML Modelo001_Largo_REML Call Model df AIC BIC 1 1 4 2016.47 2029.91 2 2 10 1872.14 1905.75 logLik Test L.Ratio -1004.23 -926.07 1 vs 2 156.33 p-value <0.0001 37 Modelos Mixtos en InfoStat Los residuos obtenidos para el Modelo 1 con varianzas distintas en cada población no muestran problemas de heteroscedasticidad y presentan una mejora en los supuestos 3 2 1 -2 -1 0 Cuantiles muestrales 1 0 -2 -1 Res.cond.estand.Pearson 2 3 distribucionales respecto al Modelo 1 con varianzas homogéneas (Figura 23). 10 20 30 40 50 60 70 -3 fitted(modeloLargocm_8_REML,level=2) -2 -1 0 1 2 3 Cuantiles teóricos Figura 23: Gráficos de diagnóstico obtenidos para la variable largo y el modelo 1 con varianzas residuales heterogéneas para poblaciones y los datos del archivo Compvar.IDB2. Modelo 2: Para este modelo se debe declarar Población en la solapa de Efectos fijos. Observar que en esta solapa se ha seleccionado además Coeficientes de los efectos fijos (Figura 24). En la solapa de Efectos aleatorios se ha declarado familias como aleatorio, se ha deseleccionado la opción por defecto de familia como efecto sobre la Constante (intercepto), y se ha seleccionado familia como afectando los parámetros del efecto población. La matriz de covarianzas de los efectos aleatorios asignados a poblaciones se suponen independientes (pdIdent). Se han seleccionado además las opciones Matriz de efectos aleatorios, Intervalo de confianza para los parámetros de la parte aleatoria e Intervalo de confianza para sigma (Figura 25). En la solapa Comparaciones se seleccionó la opción DGC para Población (Figura 26). 38 Modelos Mixtos en InfoStat Figura 24: Ventana con la solapa Efectos fijos desplegada para los datos del archivo Compvar.IDB2 para la especificación del Modelo 2. Figura 25: Ventana con la solapa Efectos aleatorios desplegada para los datos del archivo Compvar.IDB2 para la especificación del Modelo 2. 39 Modelos Mixtos en InfoStat Figura 26: Ventana con la solapa Comparaciones desplegada para los datos del archivo Compvar.IDB2 para la especificación del Modelo 2. A continuación de presenta la salida correspondiente a estas especificaciones: Especificación del modelo en R modelo001_Largo_REML<-lme(Largo~1+Poblacion ,random=list(Familia=pdIdent(~Poblacion-1)) ,method="REML" ,na.action=na.omit ,data=R.data00 ,keep.data=FALSE) Resultados para el modelo: modelo001_Largo_REML Variable dependiente:Largo Medidas de ajuste del modelo N 214 AIC 1967.65 BIC 1997.64 logLik -974.82 Sigma R2_0 21.54 0.51 R2_1 0.75 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) Poblacion numDF denDF F-value 1 108 601.79 6 108 27.23 p-value <0.0001 <0.0001 40 Modelos Mixtos en InfoStat Efectos fijos (Intercept) PoblacionEscarcega PoblacionEsclavos PoblacionLa Paz PoblacionPacífico Sur PoblacionXpujil PoblacionYucatán Value Std.Error 8.23 5.75 56.89 8.03 57.72 7.46 62.24 8.13 4.65 7.53 65.45 7.72 44.44 8.40 DF 108 108 108 108 108 108 108 t-value 1.43 7.08 7.74 7.66 0.62 8.48 5.29 p-value 0.1551 <0.0001 <0.0001 <0.0001 0.5382 <0.0001 <0.0001 Parámetros de los efectos aleatorios Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~Poblacion - 1|Familia Desvíos estándares y correlaciones Charagre Charagre 14.79 Escarcega 0.00 Esclavos 0.00 La Paz 0.00 Pacífico Sur 0.00 Xpujil 0.00 Yucatán 0.00 Escarcega 0.00 14.79 0.00 0.00 0.00 0.00 0.00 Esclavos 0.00 0.00 14.79 0.00 0.00 0.00 0.00 La Paz Pacífico Sur 0.00 0.00 0.00 0.00 0.00 0.00 14.79 0.00 0.00 14.79 0.00 0.00 0.00 0.00 XpujilYucatán 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 14.79 0.00 0.00 14.79 Intervalos de confianza (95%) para los parámetros de los efectos aleatorios Formula: ~Poblacion - 1|Familia sd( - 1) LI(95%) 10.71 est. 14.79 LS(95%) 20.42 Coeficientes (BLUP) de los efectos aleatorios (~Poblacion - 1|Familia) Ch_71 Ch_710 Ch_711 Ch_712 Ch_713 Ch_714 Ch_715 Ch_72 Ch_73 Ch_74 Ch_75 Ch_76 Ch_77 Ch_78 Ch_79 Ec_31 Ec_310 Ec_311 Ec_312 Ec_313 Ec_314 Ec_315 Ec_316 Ec_317 Ec_318 Ec_319 Ec_32 Ec_320 Ec_33 Charagre -1.08 0.62 1.34 1.47 -0.96 -1.08 -0.71 0.73 -0.84 -0.35 -0.60 -0.07 -0.48 0.50 1.53 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 Escarcega 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 Esclavos 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 -6.04 5.36 -5.56 -2.65 -16.00 20.66 -25.46 10.95 2.69 -5.80 2.94 -5.56 12.89 -0.46 La Paz Pacífico Sur 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 Xpujil Yucatán 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 41 Modelos Mixtos en InfoStat Ec_34 Ec_35 Ec_36 Ec_37 Ec_38 Ec_39 Es_1126 Es_1127 Es_1128 Es_1129 Es_1130 Es_1131 Es_1132 Es_1133 Es_1134 Es_1135 Es_1136 Es_1137 Es_1138 Es_1139 Es_1142 Es_1148 LP_41 LP_410 LP_411 LP_412 LP_413 LP_414 LP_415 LP_42 LP_43 LP_44 LP_45 LP_46 LP_48 LP_49 PS_6204 PS_6206 PS_6207 PS_6208 PS_6209 PS_6210 PS_6211 PS_6212 PS_6213 PS_6214 PS_6215 PS_6216 PS_6217 PS_6218 PS_6219 PS_6220 PS_6221 PS_6222 PS_660 Xp_11 Xp_110 Xp_112 Xp_113 Xp_114 Xp_115 Xp_116 Xp_117 Xp_118 Xp_119 Xp_12 Xp_120 Xp_122 Xp_123 Xp_15 Xp_16 Xp_17 Xp_18 Xp_19 Yu_1111 Yu_1114 Yu_1115 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 -0.06 16.20 16.63 6.49 -0.04 -7.09 19.12 1.15 -10.25 -10.94 -7.58 -16.32 14.26 -10.30 7.71 -18.99 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 -7.99 10.95 -12.84 12.89 5.36 3.67 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 -18.43 18.95 14.82 20.89 -12.12 -2.41 -18.67 1.23 -1.93 -1.68 1.96 -9.69 -2.39 9.48 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 -2.13 -2.73 2.48 -1.52 -0.67 1.51 -1.03 -2.01 0.18 -2.61 -0.31 8.55 -0.55 4.18 -1.64 -2.37 -0.55 -2.25 3.46 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 -14.96 2.35 -0.09 -7.12 1.61 -12.46 15.93 10.11 6.95 16.91 -2.14 18.36 -20.72 -27.03 1.86 2.99 3.95 -4.94 8.44 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 -14.89 -16.59 -3.24 42 Modelos Mixtos en InfoStat Yu_1116 Yu_1117 Yu_1118 Yu_1119 Yu_1121 Yu_1122 Yu_1123 Yu_1124 Yu_1125 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 1.86 4.53 2.83 8.66 -5.18 17.15 -7.85 -4.45 17.15 Intervalo de confianza (95%) para sigma sigma lower 18.77 est. 21.54 upper 24.71 Medias ajustadas y errores estándares para Poblacion DGC (alfa=0.05) Poblacion Xpujil La Paz Esclavos Escarcega Yucatán Pacífico Sur Charagre Medias 73.68 70.47 65.95 65.12 52.67 12.88 8.23 E.E. 5.16 5.74 4.75 5.61 6.13 4.87 5.75 A A A A A B B Letras distintas indican diferencias significativas(p<= 0.05) A continuación se muestra como ejemplo el cálculo de los BLUP para las familias de la población Charagre: Yˆcha ,71 ˆ ˆ cha ˆ71( cha ) 8.2296 0 (1.0823) 7.1473 Yˆcha ,72 ˆ ˆ cha ˆ72( cha ) 8.2296 0 0.7277 8.9573 Yˆcha ,73 ˆ ˆ cha ˆ73( cha ) 8.2296 0 (0.8396) 7.3900 Yˆcha ,74 ˆ ˆ cha ˆ74( cha ) 8.2296 0 (0.3542) 7.8754 Ahora realizaremos el análisis del ajuste del Modelo 2. En el submenú Análisisexploración de modelos estimados se pidieron los gráficos de diagnóstico (Figura 27). 43 Modelos Mixtos en InfoStat Figura 27: Ventana Análisis-exploración de modelos estimados con la solapa Diagnóstico desplegada para el Modelo 2 con los datos del archivo Compvar.IDB2. El gráfico de residuos condicionales estandarizados de Pearson vs. Valores ajustados (Figura 28) muestra varianzas residual heterogéneas para la variable Largo. 44 Charagre La Paz Xpujil 2 1 0 -1 -2 Res.cond.estand.Pearson 2 1 0 -1 -2 Res.cond.estand.Pearson Modelos Mixtos en InfoStat 20 60 80 Valores ajustados 1 0 -1 -2 Cuantiles muestrales 2 Poblacion 40 -3 -2 -1 0 1 2 3 Cuantiles teóricos Figura 28: Gráficos de diagnóstico obtenidos para la variable largo y el Modelo 2 para los datos del archivo Compvar.IDB2 Respecto a los supuestos distribucionales, es importante destacar que, existiendo heteroscedasticidad, el QQ-plot no debe ser interpretado hasta tanto no se corrija este problema. Para incorporar las varianzas heterogéneas del efecto Población, en la solapa heterogeneidad se debe especificar el factor Población como se mostró en la Figura 22. Este modelo presenta valores más bajos de AIC y BIC que el modelo sin varianzas heterogéneas para Población. Observamos que las varianzas de las poblaciones son bien diferentes: La población La Paz tiene la mayor varianza estimada en (15.94*2.32)2 = 1367.57, mientras que la de menor varianza es Charagre con (1*2.32)2 = 5.38. 45 Modelos Mixtos en InfoStat Modelos lineales generales y mixtos Especificación del modelo en R modelo002_Largo_REML<-lme(Largo~1+Poblacion ,random=list(Familia=pdIdent(~Poblacion-1)) ,weight=varComb(varIdent(form=~1|Poblacion)) ,method="REML" ,na.action=na.omit ,data=R.data01 ,keep.data=FALSE) Resultados para el modelo: modelo002_Largo_REML Variable dependiente:Largo Medidas de ajuste del modelo N 214 AIC 1823.20 BIC 1873.20 logLik -896.60 Sigma R2_0 2.32 0.51 R2_1 0.51 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) Poblacion numDF denDF F-value 1 108 509.60 6 108 86.55 p-value <0.0001 <0.0001 Efectos fijos (Intercept) PoblacionEscarcega PoblacionEsclavos PoblacionLa Paz PoblacionPacífico Sur PoblacionXpujil PoblacionYucatán Value Std.Error 8.23 0.61 57.32 5.90 57.72 4.33 62.33 7.16 4.65 1.28 65.43 5.54 44.44 6.00 DF 108 108 108 108 108 108 108 t-value 13.42 9.72 13.33 8.70 3.65 11.81 7.41 p-value <0.0001 <0.0001 <0.0001 <0.0001 0.0004 <0.0001 <0.0001 Parámetros de los efectos aleatorios Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~Poblacion - 1|Familia Desvíos estándares y correlaciones Charagre Escarcega Esclavos La Paz Pacífico Sur Xpujil Yucatán Charagre Escarcega 1.56 0.00 0.00 1.56 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 Esclavos La Paz 0.00 0.00 0.00 0.00 1.56 0.00 0.00 1.56 0.00 0.00 0.00 0.00 0.00 0.00 Pacífico Sur 0.00 0.00 0.00 0.00 1.56 0.00 0.00 Xpujil 0.00 0.00 0.00 0.00 0.00 1.56 0.00 Yucatán 0.00 0.00 0.00 0.00 0.00 0.00 1.56 Intervalos de confianza (95%) para los parámetros de los efectos aleatorios Formula: ~Poblacion - 1|Familia sd( - 1) LI(95%) 0.45 Est. 1.56 LS(95%) 5.38 46 Modelos Mixtos en InfoStat Estructura de varianzas Modelo de varianzas: varIdent Formula: ~ 1 | Poblacion Parámetros de la función de varianza Parámetro Charagre Esclavos Escarcega La Paz Pacífico Sur Xpujil Yucatán Estim 1.00 11.64 13.09 15.94 2.81 13.38 12.55 Para probar que este modelo menos parsimonioso es el de mejor ajuste se realizó una prueba del cociente de verosimilitud cuya salida se presenta a continuación. Comparación de modelos Model 001 df 9 AIC 1967.65 BIC 1997.64 logLik -974.82 Test 002 15 1823.20 1873.20 -896.60 1 vs 2 L.Ratio p-value 156.44 <0.0001 El modelo con varianzas heterogéneas para las distintas poblaciones es mejor que el de varianzas homogéneas (p<0.0001). Podemos observar que con la inclusión de varianzas heterogéneas para las distintas poblaciones el ajuste ha mejorado respecto a los ajustes anteriores (Figura 29). Tanto en los box-plot de los residuos condicionales estudentizados de Pearson como en el diagrama de dispersión de residuos condicionales estudentizados de Pearson versus predichos, ya no se evidencian problemas graves de falta de homogeneidad de varianzas. En el gráfico QQ-plot se observa una mejora en el supuesto distribucional. 47 3 2 1 0 -2 -1 Res.cond.estand.Pearson 2 1 0 -1 -2 Res.cond.estand.Pearson 3 Modelos Mixtos en InfoStat Charagre La Paz Xpujil 10 30 40 50 60 70 Valores ajustados 2 1 0 -1 -2 Cuantiles muestrales 3 Poblacion 20 -3 -2 -1 0 1 2 3 Cuantiles teóricos Figura 29: Gráficos de diagnóstico obtenidos para la variable largo y el modelo 2 para los datos del archivo Compvar.IDB2 una vez declaradas las varianzas residuales diferentes para cada población. 48 Modelos Mixtos en InfoStat Aplicación de modelos mixtos para datos estratificados Parcelas divididas Supongamos un experimento bifactorial en el que no es posible asignar al azar las combinaciones de ambos factores a las parcelas experimentales (PE). En algunos casos, grupos de PE reciben aleatoriamente los distintos niveles de uno de los factores de clasificación y dentro de estos grupos de parcelas, los niveles del segundo factor son asignados al azar. El experimento descripto anteriormente difiere de un experimento bifactorial convencional en que, si bien los niveles de los factores son asignados aleatoriamente a las PE, no son los tratamientos (i.e. las combinaciones de los niveles de los factores) los que están siendo asignados de esta forma. Esta manera particular de asignar las parcelas a los distintos niveles de los factores representa una restricción a la aleatorización, e induce estructuras de correlación que deben ser tenidas en cuenta en el momento del análisis. Este diseño se conoce como parcela dividida. El nombre surge de la idea de que PARCELAS principales reciben los niveles de un factor (también llamado a veces factor principal) y que estas parcelas son DIVIDIDAS en SUBPARCELAS que reciben los niveles del segundo factor de clasificación. Aunque en las parcelas divididas los niveles de un factor son asignados dentro de los niveles de otro factor, este NO ES un diseño anidado. Se trata de un experimento típicamente factorial donde los factores están cruzados. Es sólo la aleatorización la que se ha realizado en forma secuencial. De acuerdo a la forma en que están arregladas las parcelas principales, el diseño puede ser de: Parcelas divididas en un arreglo en bloques Parcelas divididas en un arreglo completamente aleatorizado Parcelas divididas en otros diseños 49 Modelos Mixtos en InfoStat Parcelas divididas en un arreglo en bloques El análisis clásico de un diseño en parcelas divididas con parcelas principales distribuidas en bloques completos incluye los siguientes términos en el modelo: Factor asociado a la parcela principal (FPP) Bloque Bloque*FPP (error de la parcela principal) Factor asociado a la subparcela (FSP) FPP*FSP Error (error para la subparcela) El punto clave para completar el análisis de este modelo es comprender que el error experimental para el FPP es diferente que para los términos del modelo que incluyen al FSP. El error experimental de las parcelas principales es mayor que el de las subparcelas. La varianza del error experimental de las parcelas principales en un diseño de parcelas divididas con parcelas principales repetidas en bloque completamente aleatorizados, se estima como el cuadrado medio (CM) de la interacción Bloque*FPP (se asume que no hay interacción Bloque*FPP y en consecuencia este CM estima el error entre parcelas principales tratadas de la misma forma). El CM de esta “interacción” es el que se usa como referencia para calcular el estadístico F de la prueba de hipótesis para el factor principal. El resto de las pruebas el CM residual es el apropiado para construir el estadístico F. El análisis de este diseño mediante un modelo lineal mixto se basa en la identificación de dos niveles de agrupamientos de las observaciones. El primer nivel esta dado por los bloques y el segundo nivel por las parcelas principales dentro de los bloques. Cada uno de estos niveles de agrupamiento genera una correlación, conocida como correlación intraclase, entre las observaciones que contiene. El modelo lineal mixto para este diseño es el siguiente: yijk i j ij bk pik ijk ; i 1,.., T ; j 1,..., G; k 1,..., B (4) 50 Modelos Mixtos en InfoStat donde yijk representa la respuesta observada en el k-ésimo bloque, i-ésimo nivel del factor principal y j-ésimo nivel de factor asociado a las subparcelas. representa la media general de la respuesta, i representa el efecto del i-ésimo nivel del factor asociado a las parcelas principales, j representa el efecto del j-ésimo nivel del factor asociado a las subparcelas y ij representa el efecto de la interacción del ij-ésimo tratamiento. Por otra parte bk , pik y ijk corresponden a efectos aleatorios de los bloques, de las parcelas dentro de los bloques y de los errores experimentales. Las suposiciones sobre estos componentes aleatorios es que bk ~ N 0, b2 , pik ~ N 0, p2 , ijk ~ N 0, 2 y que estos tres componentes aleatorios son independientes. A continuación ejemplificaremos el análisis de un diseño en parcelas divididas en bloques mediante la aplicación de un modelo lineal mixto. En este ejemplo (Di Rienzo 2007) se evalúan 4 variedades de trigo: BUCK-Charrua (BC), Las Rosas-INTA (LI), Pigué (Pe) y Pro-INTA Puntal (PP) bajo riego y secano con el diseño a campo presentado en la Figura 30. BC Pe PP LI Secano Pe BC LI PP Riego Figura 30: Esquema del diseño en parcelas divididas para el ejemplo de los datos en el archivo Trigo.IDB2. 51 Modelos Mixtos en InfoStat Los datos de este ejemplo se encuentran en el archivo Trigo.IDB2. El encabezamiento de la tabla de datos es la siguiente (Figura 31). Figura 31: Encabezamiento de la tabla de datos del archivo Trigo.IDB2. El factor en la parcela principal es Agua, el factor asociado a las subparcelas es Variedad y la variable de respuesta es el Rendimiento. Los bloques están claramente identificados, pero las parcelas principales no aparecen explícitamente. Esto es así porque en un diseño en parcelas divididas, las parcelas principales dentro de un bloque están confundidas con el factor principal. De esta forma las observaciones bajo “Riego” en el bloque 1, representan las observaciones de una de las parcelas principales de ese bloque. Para analizar este ejemplo invocaremos la estimación de un modelo lineal mixto. Esta invocación nos presentará, como es usual, la ventana de selección de variables. Su imagen, con la selección apropiada de variables de respuesta y factores se muestra en la Figura 32. Figura 32: Ventana de selección de variables para Modelos lineales generales y mixtos con datos del archivo Trigo.IDB2. 52 Modelos Mixtos en InfoStat Aceptando esta especificación, se mostrará el diálogo que permite especificar el modelo. La solapa de la parte fija, ya especificada, se muestra en la Figura 33. En ella aparecen los efectos principales Agua, Variedad y la interacción Agua*Variedad. Figura 33: Ventana con la solapa Efectos fijos desplegada para los datos del archivo Trigo.IDB2. Para la especificación de la parte aleatoria, en la solapa Efectos aleatorios se debe incorporar primero al factor Bloque y después al factor Agua. Esta es la forma de indicar que Agua está dentro de Bloque. La especificación de la parte aleatoria queda como se muestra en la Figura 34. 53 Modelos Mixtos en InfoStat Figura 34: Ventana con la solapa Efectos aleatorios desplegada para los datos del archivo Trigo.IDB2 con bloque y agua como criterios de estratificación. La salida correspondiente a esta estimación es la siguiente: Modelos lineales generales y mixtos Especificación del modelo en R modelo000_Rendimiento_REML<lme(Rendimiento~1+Agua+Variedad+Agua:Variedad ,random=list(Bloque=pdIdent(~1) ,Agua=pdIdent(~1)) ,method="REML" ,na.action=na.omit ,data=R.data00 ,keep.data=FALSE) Resultados para el modelo: modelo000_Rendimiento_REML Variable dependiente:Rendimiento Medidas de ajuste del modelo N 24 AIC 206.59 BIC 215.09 logLik -92.30 Sigma R2_0 51.65 0.84 R2_1 0.89 R2_2 0.91 AIC y BIC menores implica mejor 54 Modelos Mixtos en InfoStat Pruebas de hipótesis secuenciales (Intercept) Agua Variedad Agua:Variedad numDF denDF F-value 1 12 363.93 1 2 55.24 3 12 6.38 3 12 2.36 p-value <0.0001 0.0176 0.0078 0.1223 Parámetros de los efectos aleatorios Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~1|Bloque Desvíos estándares relativos al residual y correlaciones (const) (const) 0.55 Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~1|Agua dentro de Bloque Desvíos estándares relativos al residual y correlaciones (const) (const) 0.47 Las pruebas de hipótesis secuenciales son las correctas para este tipo de especificación del modelo. Esto es debido a que primero se aleatoriza el factor en la PP, luego se aleatoriza el factor en las SP y por último se especifica la interacción. Antes de continuar con nuestro análisis, haremos algunas validaciones simples de las suposiciones de estos modelos, revisando residuales estandarizados vs. predichos y otros criterios de clasificación, así como el QQ-plot normal de residuos estandarizados. Estos residuos son condicionales a los efectos aleatorios (es decir, aproximan los errores). Para ello invocaremos el submenú Análisis-exploración de modelos estimados. En el diálogo, seleccionaremos la solapa Diagnóstico y dentro de ella la subsolapa Residuos vs. (Figura 35). 55 Modelos Mixtos en InfoStat Figura 35: Ventana Comparación de modelos generales y mixtos con la solapa Diagnóstico desplegada para los datos del archivo Trigo.IDB2. Si se seleccionan los ítems dentro de la lista disponible, como se muestra en la Figura 35, se obtendrá el gráfico siguiente (Figura 36). Este aparece en una nueva ventana que genera R y su contenido puede copiarse oprimiendo el botón derecho del ratón, sobre la imagen. En el menú que se despliega se podrá optar por “Copy as metafile” o “Copy as bitmap”. 56 Riego 1.0 0.5 0.0 -1.0 Res.cond.estand.Pearson 1.0 0.5 0.0 -1.0 Res.cond.estand.Pearson Modelos Mixtos en InfoStat Secano BUCK-Charrua 300 400 500 Valores ajustados 600 1.0 0.5 0.0 -1.0 0.0 0.5 Cuantiles muestrales 1.0 Variedad -1.0 Res.cond.estand.Pearson Agua Pigue -2 -1 0 1 2 Cuantiles teóricos Figura 36: Herramientas gráficas para diagnóstico obtenidas para los datos del archivo Trigo.IDB2. Un examen rápido de la figura sugiere una posible heterogeneidad de varianzas entre variedades. Para poder probar si es necesario incluir la estimación de varianzas residuales diferentes para cada variedad hay que ajustar un modelo heteroscedástico y compararlo con el homoscedástico, utilizando algún criterio como el AIC o BIC (o una prueba del cociente de verosimilitud, ya que el modelo homoscedástico es un caso particular del heteroscedástico). Para ajustar el modelo heteroscedástico invocamos nuevamente al módulo de estimación de los modelos mixtos y en la solapa Heteroscedasticidad seleccionamos el modelo varIdent y una vez seleccionado hacemos doble clic sobre Variedad (en la lista a la derecha de la ventana) para especificar a esta variable como criterio de 57 Modelos Mixtos en InfoStat agrupamiento (Figura 37). Luego accionamos el botón Agregar para hacer efectiva la incorporación de esta especificación del modelo. Si por algún motivo la especificación ingresada no es deseada, haciendo doble clic sobre la misma, ésta se borra. Figura 37: Ventana con la solapa Heteroscedasticidad desplegada para los datos del archivo Trigo.IDB2 con selección de función varIdent con variedad como criterio de agrupamiento. Las medidas de ajuste del modelo especificado son las siguientes: Medidas de ajuste del modelo N 24 AIC 209.47 BIC 220.28 logLik -90.73 Sigma R2_0 24.49 0.84 R2_1 0.89 R2_2 0.90 AIC y BIC menores implica mejor 58 Modelos Mixtos en InfoStat Comparadas con las del modelo homoscedástico, no se observa una mejoría, por lo contrario tanto AIC como BIC aumentaron. Por este motivo se descarta el modelo heteroscedástico. Luego volviendo al modelo homoscedástico, realizaremos comparaciones múltiples del tipo LSD de Fisher para evaluar diferencias entre variedades. Para ello en la solapa Comparaciones subsolapa Medias, tildaremos la opción Variedad como se muestra la Figura 38. Figura 38: Ventana con la solapa Comparaciones desplegada para los datos del archivo Trigo.IDB2 y selección de la subsolapa Medias. Al final de la salida del programa se encontrará la comparación de medias. Se observa que solo BUCK-Charrua tuvo los rendimientos más bajos y esto ocurrió independientemente de si tenía riego o no. En tanto las otras variedades tuvieron rendimientos estadísticamente indistinguibles. 59 Modelos Mixtos en InfoStat Medias ajustadas y errores estándares para Variedad LSD Fisher (alfa=0.05) Procedimiento de correccion de p-valores: No Variedad Pro-INTA Puntal Pigue LasRosas-INTA BUCK-Charrua Medias 469.50 430.98 423.98 342.73 E.E. 28.48 A 28.48 A 28.48 A 28.48 B Letras distintas indican diferencias significativas(p<= 0.05) Parcelas divididas en un arreglo en diseño completamente aleatorizado A continuación ejemplificamos mediante un experimento cuyo objetivo fue evaluar el efecto de un coadyuvante sobre la cobertura de gotas y uniformidad de aplicación en distintas ubicaciones de las hojas dentro del canopeo de un cultivo de soja (Di Rienzo 2007). Para ello se seleccionaron 16 sitios en cada uno de los cuales se dispusieron 4 tarjetas hidro-sensibles, ubicadas a dos alturas del canopeo (inferior, superior) y apuntando, sus caras sensibles, en dos direcciones: hacia arriba y hacia abajo. Las tarjetas hidrosensibles muestran una mancha en el lugar donde cae una gota de agua. La superficie manchada en estas tarjetas es una medida de cuanto penetra y se dispersa el agua en una zona dada del canopeo. En 8 de los 16 sitios se agregó al agua de pulverización un coadyuvante (para disminuir la tensión superficial del agua y mejorar la dispersión de las gotas) y en los 8 restantes no. Por lo tanto en cada sitio de pulverización se obtienen 4 lecturas correspondientes a las combinaciones de las alturas (inferior y superior) y la ubicación de la cara sensible de la tarjeta (abajo y arriba). Luego en cada sitio hay una repetición completa de un experimento con 4 tratamientos SuAr, SuAb, InAr y InAb, y que se combinan con la utilización o no del coadyuvante en la solución de rociado. El experimento resultante es un trifactorial, con un factor principal (coadyuvante) asociado a parcelas principales (sitios donde se realiza el rociado) y dos factores (altura y ubicación de cara sensible de la tarjeta) asociados a las subparcelas (tarjetas dentro de 60 Modelos Mixtos en InfoStat sitio). El archivo conteniendo los datos se llama Cobertura de gotas.IDB2 y el encabezamiento de la tabla de datos se presenta en la Figura 39. Figura 39: Encabezamiento de la tabla de datos del archivo Cobertura de gotas.IDB2. En la tabla de datos hay una columna que identifica a la parcela y va numerada de 1 a 16. Este va a ser el único efecto aleatorio de nuestro modelo. El modelo lineal para las observaciones de este experimento es el siguiente: yijkl i j k ij ik jk ijk bl ijkl ; (5) i 1,.., 2; j 1,..., 2; k 1,..., 2; l 1,...,16 donde yijkl representa la respuesta observada en i-ésimo nivel del factor coadyuvante y j-ésimo nivel de factor altura, k-ésimo nivel del factor cara en la l-ésima parcela, representa la media general de la respuesta, i representa el efecto del i-ésimo nivel del factor asociado a las parcelas principales (coadyuvante), j representa el efecto del jésimo nivel del factor altura, k el k-ésimo nivel del factor cara, ambos asociados a las subparcelas y ij , ik , jk y ijk las interacciones de segundo y tercer orden correspondientes de los factores coadyuvante, altura y cara. Por otra parte bl y ijkl representan los efectos aleatorios de las parcelas y de los errores experimentales respectivamente. Las suposiciones sobre estos componentes aleatorios son que bl ~ N 0, b2 , que ijkl ~ N 0, 2 , y que estas dos componentes aleatorias son independientes. A continuación presentamos la forma en que se especifica el modelo anterior en InfoStat, su salida, interpretación y algunas acciones complementarias de validación del modelo. Para ello invocaremos el Menú: Modelos lineales generales y 61 Modelos Mixtos en InfoStat mixtos>>Estimación. El diálogo de selección de variables para este caso se presenta en la Figura 40. Figura 40: Ventana de selección de variables para Modelos lineales generales y mixtos con datos del archivo Cobertura de gotas.IDB2. La especificación de la parte fija del modelo para este ejemplo contiene los tres factores y sus interacciones dobles y triples (Figura 41). Figura 41: Ventana con la solapa Efectos fijos desplegada para los datos del archivo Cobertura de gotas.IDB2. 62 Modelos Mixtos en InfoStat El efecto aleatorio que consideramos en este ejemplo es el de Parcela (Figura 42). Figura 42: Ventana con la solapa Efectos aleatorios desplegada para los datos del archivo Cobertura de gotas.IDB2 con Parcela como criterio de estratificación. Luego de aceptar las especificaciones anteriores obtendremos la siguiente salida: Modelos lineales generales y mixtos Especificación del modelo en R modelo000_Cobertura_REML<lme(Cobertura~1+Coad+Altura+Cara+Coad:Altura+Coad:Cara+Altura:Cara+Coa d:Altura:Cara ,random=list(Parcela=pdIdent(~1)) ,method="REML" ,na.action=na.omit ,data=R.data00 ,keep.data=FALSE) Resultados para el modelo: modelo000_Cobertura_REML Variable dependiente:Cobertura Medidas de ajuste del modelo N 64 AIC 670.38 BIC 690.63 logLik -325.19 Sigma R2_0 65.17 0.76 R2_1 0.82 AIC y BIC menores implica mejor 63 Modelos Mixtos en InfoStat Pruebas de hipótesis secuenciales numDF denDF F-value (Intercept) 1 42 233.37 Coad 1 14 1.89 Altura 1 42 72.86 Cara 1 42 95.32 Coad:Altura 1 42 1.58 Coad:Cara 1 42 0.01 Altura:Cara 1 42 34.77 Coad:Altura:Cara 1 42 0.21 p-value <0.0001 0.1909 <0.0001 <0.0001 0.2152 0.9271 <0.0001 0.6476 Parámetros de los efectos aleatorios Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~1|Parcela Desvíos estándares relativos al residual y correlaciones (Intercept) (Intercept) 0.40 Una revisión de los residuos estandarizados de este modelo mediante las herramientas de diagnóstico en el Menú: Modelos lineales generales y mixtos>>Análisis-exploración de modelos estimados muestra una posible heterogeneidad de varianzas cuando se comparan las observaciones obtenidas cuando la cara sensible de la tarjeta hidrosensible 1 0 -1 -2 -3 Res.cond.estand.Pearson 2 3 se presenta hacia arriba (Figura 43). Ab Ar Cara Figura 43: Diagrama de cajas para los residuos estandarizados de Pearson para los niveles del factor Cara. 64 Modelos Mixtos en InfoStat Para ello invocaremos nuevamente el menú de estimación del modelo. Todas las especificaciones anteriores se han preservado por lo que sólo tenemos que concentrarnos en la especificación del modelo de la varianza. Para ello utilizaremos la solapa heteroscedasticidad como se muestra en la Figura 44. Figura 44: Ventana con la solapa Heteroscedasticidad desplegada para los datos del archivo Cobertura de gotas.IDB2 con Cara como criterio de agrupamiento. La salida resultante es la siguiente: Modelos lineales generales y mixtos Especificación del modelo en R modelo001_Cobertura_REML<lme(Cobertura~1+Coad+Altura+Cara+Coad:Altura+Coad:Cara+Altura:Cara+Coa d:Altura:Cara ,random=list(Parcela=pdIdent(~1)) ,weight=varComb(varIdent(form=~1|Cara)) ,method="REML" ,na.action=na.omit ,data=R.data00 ,keep.data=FALSE) 65 Modelos Mixtos en InfoStat Resultados para el modelo: modelo001_Cobertura_REML Variable dependiente:Cobertura Medidas de ajuste del modelo N 64 AIC 636.54 BIC 658.82 logLik -307.27 Sigma R2_0 21.26 0.76 R2_1 0.81 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) Coad Altura Cara Coad:Altura Coad:Cara Altura:Cara Coad:Altura:Cara numDF denDF F-value 1 42 176.66 1 14 4.19 1 42 53.72 1 42 98.43 1 42 13.83 1 42 0.01 1 42 35.90 1 42 0.22 p-value <0.0001 0.0599 <0.0001 <0.0001 0.0006 0.9259 <0.0001 0.6423 Parámetros de los efectos aleatorios Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~1|Parcela Desvíos estándares relativos al residual y correlaciones DS(Const) DS(Const) 1.06 Estructura de varianzas Modelo de varianzas: varIdent Formula: ~ 1 | Cara Parámetros del modelo Parámetro Ab Ar Estim 1.00 4.15 El modelo para estos datos sería yijk i j bk ijk , donde i j representa el efecto fijo de i-ésimo tratamiento en la j-ésima cara (cara puede ser Ab o Ar), bk es el efecto aleatorio de la k-esima parcela experimental que se supone N 0, b2 y ijk ~ N 0, 2j . Luego la varianza de una observación tomada en una parcela seleccionada aleatoriamente va a depender si se hace en la cara de abajo o arriba de la tarjeta. Así si tomamos una observación de la cara de abajo la varianza va a ser (21.26*(1.06+1))2 y si la tomamos en la cara de arriba: (21.26*(1.06+4.15))2. 66 Modelos Mixtos en InfoStat A continuación se presentan las medidas resumen del modelo homoscedástico y del heteroscedástico. Medidas de ajuste del modelo homoscedástico N 64 AIC 670.38 BIC 690.63 logLik -325.19 Sigma R2_0 65.17 0.76 R2_1 0.82 AIC y BIC menores implica mejor Medidas de ajuste del modelo heteroscedástico N AIC BIC logLik 64 636.54 658.82 -307.27 AIC y BIC menores implica mejor Sigma R2_0 21.26 0.76 R2_1 0.81 Si comparamos los AIC y BIC veremos que el último modelo ajustado es mejor y por lo tanto la interpretación de la pruebas de hipótesis debe basarse en este último. Obsérvese que en la estructura de varianzas, la desviación estándar residual de las observaciones en las tarjetas que apuntan hacia arriba es 4.15 veces mayor que la desviación estándar residual de las observaciones en las tarjetas que apuntan hacia abajo. Por otra parte, observando los resultados de las pruebas de hipótesis resulta que la interacción Coad:Altura:Cara no resultó significativa, por lo que se pueden observar las interacciones dobles (Figura 45). Entre estas, Coad:Altura y Altura:Cara son significativas. Estas interacciones se analizan utilizando la solapa Comparaciones de la ventana Modelos lineales generales y mixtos y tildando las correspondientes interacciones en la lista de términos del modelo que se presenta en esa ventana. Este procedimiento creará una tabla con las medias de todas las combinaciones resultantes de los niveles de los factores que intervienen en la interacción. El resultado, al final de la salida, presenta las siguientes tablas. Medidas ajustadas y errores estándares para Coad*Altura LSD Fisher (alfa=0,05) Procedimiento de corrección de p-valores: No Coad Si No Si No Altura Su Su In In Medias 253.94 204.69 94.38 86.13 E.E. 17.89 A 17.89 A 17.89 17.89 B B Letras distintas indican diferencias significativas(p<= 0.05) 67 Modelos Mixtos en InfoStat Medidas ajustadas y errores estándares para Altura*Cara LSD Fisher (alfa=0,05) Procedimiento de corrección de p-valores: No Altura Su In Su In Cara Ar Ar Ab Ab Medias 356.88 121.75 101.75 58.75 E.E. 22.74 A 22.74 7.73 7.73 B B C Letras distintas indican diferencias significativas(p<= 0.05) 400 300 250 300 Cobertura Cobertura 200 150 200 100 100 50 0 0 Superior Inferior Superior Sin coadyuvante Inferior Altura Altura Con coadyuvante Cara abajo Cara arriba Figura 45: Diagramas de puntos para estudiar la interacción entre Coad y Altura y entre Cara y Altura. Parcelas subdivididas (split-split plot) Este diseño utiliza el mismo principio que las parcelas divididas, excepto que lo extiende un paso más. El principio puede extenderse arbitrariamente a niveles más profundos de división. El modelo lineal para este diseño, suponiendo las parcelas principales agrupadas en bloques completos aleatorizados, es el siguiente: yijkl i j k ij ik jk ijk bl pil sp jil sspkjil ijkl (6) En la expresión anterior representa la media general, i el i-ésimo nivel del factor asociado a las parcelas principales, j el j-ésimo nivel del factor asociado a las subparcelas dentro de las parcelas principales, k el k-ésimo nivel del factor asociado a las sub-subparcelas (dentro de las subparcelas) y ij , ik , jk y ijk las correspondientes interacciones. Los términos aleatorios de éste modelo corresponden a los efectos de 68 Modelos Mixtos en InfoStat bloques, bl ~ N 0, b2 , los efectos de parcelas, pil ~ N 0, p2 , los efectos de 2 subparcelas, sp jil ~ N 0, sp2 , los efectos de sub-subparcelas, sspkjil ~ N 0, spp y el error experimental, ijkl ~ N 0, 2 . Todos ellos, como siempre, se suponen independientes. Consideremos ahora un ejemplo. Los datos están en el archivo Calidad del almidón.IDB2 (Di Rienzo 2007). En este experimento se evalúa el índice de absorción de agua (IAA) del almidón cocido y crudo obtenido de dos genotipos de Quínoa cultivada bajo 4 niveles de fertilización nitrogenada. Las variedades son Faro y UDEC10. Éstas se asignaron a grandes parcelas dispuestas en 3 bloques. Las parcelas en las que fueron sembradas las variedades fueron divididas en 4 subparcelas a las que se les asignaron 4 dosis de fertilización: 0, 75, 150 y 225 kg/ha. Las subparcelas fueron nuevamente divididas en 2 para asignar el tratamiento de cocción o sin cocción (crudo). El esquema para este diseño de experimento se presenta en la Figura 46. Sub-Parcela Parcela principal Bloque 1 Bloque 2 Bloque 3 Sub-sub Parcela Figura 46: Esquema del diseño en parcelas subdivididas para el ejemplo de los datos en el archivo Calidad del almidón.IDB2. Para el análisis de este diseño mediante un modelo mixto, además de la especificación de la parte fija, como en un clásico experimento tri-factorial, sólo debemos especificar la parte aleatoria para incluir el efecto aleatorio de los Bloques, de las Parcelas 69 Modelos Mixtos en InfoStat Principales dentro de Bloques y de las Subparcela dentro de Parcelas. El encabezamiento del archivo Calidad del Almidón.IDB2 se presenta en la Figura 47. Figura 47: Encabezamiento de la tabla de datos del archivo Calidad del Almidón.IDB2. La ventana de selección de variables para este ejemplo tendrá que contener la información que se presenta en la Figura 48. Figura 48: Ventana de selección de variables para Modelos lineales generales y mixtos con datos del archivo Calidad del Almidón.IDB2. La especificación de la parte fija deberá contener los factores e interacciones presentados en la Figura 49. 70 Modelos Mixtos en InfoStat Figura 49: Ventana con la solapa Efectos fijos desplegada para los datos del archivo Calidad del Almidón.IDB2. La parte aleatoria deberá tener declarados a los bloques (Bloque), a las parcelas principales dentro de Bloques (Genotipo) y a las subparcelas dentro de parcelas principales (Nitrógeno) (Figura 50). 71 Modelos Mixtos en InfoStat Figura 50: Ventana con la solapa Efectos aleatorios desplegada para los datos del archivo Calidad del Almidón.IDB2. La salida correspondiente es la siguiente: Modelos lineales generales y mixtos Especificación del modelo en R modelo000_IAA_REML<lme(IAA~1+Genotipo+Nitrogeno+Coccion+Genotipo:Nitrogeno+Genotipo:Cocci on+Nitrogeno:Coccion+Genotipo:Nitrogeno:Coccion ,random=list(Bloque=pdIdent(~1) ,Genotipo=pdIdent(~1) ,Nitrogeno=pdIdent(~1)) ,method="REML" ,na.action=na.omit ,data=R.data00 ,keep.data=FALSE) Resultados para el modelo: modelo000_IAA_REML Variable dependiente:IAA 72 Modelos Mixtos en InfoStat Medidas de ajuste del modelo N 48 AIC 116.45 BIC 145.76 logLik -38.22 Sigma R2_0 0.61 0.75 R2_1 0.75 R2_2 0.75 R2_3 0.75 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) Genotipo Nitrogeno Coccion Genotipo:Nitrogeno Genotipo:Coccion Nitrogeno:Coccion Genotipo:Nitrogeno:Coccion.. numDF denDF F-value 1 16 1389.20 1 2 14.49 3 12 0.78 1 16 32.90 3 12 0.88 1 16 37.67 3 16 1.74 3 16 0.46 p-value <0.0001 0.0626 0.5287 <0.0001 0.4769 <0.0001 0.1998 0.7108 Parámetros de los efectos aleatorios Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~1|Bloque Desvíos estándares relativos al residual y correlaciones (Const) (Const) 1.3E-05 Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~1|Genotipo dentro de Bloque Desvíos estándares relativos al residual y correlaciones (Const) (Const) 5.0E-06 Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~1|Nitrogeno dentro de Genotipo dentro de Bloque Desvíos estándares relativos al residual y correlaciones (Const) (Const) 1.8E-05 Podríamos seguir realizando pruebas de diagnóstico pero asumiremos que el modelo es correcto. La interpretación de las pruebas de hipótesis indica que sólo la interacción Genotipo:Cocción es significativa. Las comparaciones múltiples para las medias de tratamientos correspondientes a esta interacción se presentan a continuación. En estas pruebas se observa que sólo el almidón cocido del genotipo UDEC10 presenta un IAA significativamente mayor que el resto de combinaciones de Genotipo y Cocción. 73 Modelos Mixtos en InfoStat Medidas ajustadas y errores estándares para Genotipo*Coccion LSD Fisher (alfa=0,05) Genotipo UDEC10 Faro Faro UDEC10 Coccion Cocido Crudo Cocido Crudo Medias 4.64 2.97 2.90 2.56 E.E. 0.18 0.18 0.18 0.18 A B B B Letras distintas indican diferencias significativas(p<= 0.05) Una manera alternativa de formular el modelo anterior consiste en dejar los efectos fijos como en la Figura 49 y especificar los efectos aleatorios como se muestra en la Figura 51. Los resultados son exactamente los mismos que antes, excepto por el cálculo de los grados de libertad del denominador y por ende los valores de probabilidad. Esta es una aproximación también válida aunque la versión anterior es acorde con el análisis tradicional basado en efectos fijos. Además, las estimaciones de varianza están presentadas en forma diferente. Figura 51: Ventana con la solapa Efectos aleatorios desplegada para los datos del archivo Calidad del Almidón.IDB2 que contempla otra forma de especificar la parte aleatoria. 74 Modelos Mixtos en InfoStat Modelos lineales generales y mixtos Especificación del modelo en R modelo001_IAA_REML<lme(IAA~1+Genotipo+Nitrogeno+Coccion+Genotipo:Nitrogeno+Genotipo:Cocci on+Nitrogeno:Coccion+Genotipo:Nitrogeno:Coccion ,random=list(Bloque=pdIdent(~1) ,Bloque=pdIdent(~Genotipo-1) ,Bloque=pdIdent(~Genotipo:Nitrogeno-1)) ,method="REML" ,na.action=na.omit ,data=R.data00 ,keep.data=FALSE) Resultados para el modelo: modelo001_IAA_REML Variable dependiente:IAA Medidas de ajuste del modelo N 48 AIC 116.45 BIC 145.76 logLik -38.22 Sigma R2_0 0.61 0.75 R2_1 0.75 R2_2 0.75 R2_3 0.75 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) Genotipo Nitrogeno Coccion Genotipo:Nitrogeno Genotipo:Coccion Nitrogeno:Coccion Genotipo:Nitrogeno:Coccion.. numDF denDF F-value 1 30 1389.20 1 30 14.49 3 30 0.78 1 30 32.90 3 30 0.88 1 30 37.67 3 30 1.74 3 30 0.46 p-value <0.0001 0.0006 0.5157 <0.0001 0.4605 <0.0001 0.1807 0.7089 Parámetros de los efectos aleatorios Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~1|Bloque Desvíos estándares relativos al residual y correlaciones (Const) (Const) 1.3E-05 Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~Genotipo - 1|Bloque Desvíos estándares relativos al residual y correlaciones Faro UDEC10 Faro 5.0E-06 0.00 UDEC10 0.00 5.0E-06 75 Modelos Mixtos en InfoStat Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~Genotipo:Nitrogeno - 1|Bloque Desvíos estándares relativos al residual y correlaciones Faro:0 Faro:0 1.8E-05 UDEC10:0 0 Faro:75 0 UDEC10:75 0 Faro:150 0 UDEC10:150 0 Faro:225 0 UDEC10:225 0 UDEC10:0 0 1.8E-05 0 0 0 0 0 0 Faro:75 UDEC10:75 0 0 0 0 1.8E-05 0 0 1.8E-05 0 0 0 0 0 0 0 0 Faro:150 0 0 0 0 1.8E-05 0 0 0 UDEC10:150 0 0 0 0 0 1.8E-05 0 0 Faro:225 UDEC10:225 0 0 0 0 0 0 0 0 0 0 0 0 1.8E-05 0 0 1.8E-05 76 Modelos Mixtos en InfoStat Aplicación de modelos mixtos para mediciones repetidas en el tiempo Datos longitudinales Para la modelación de datos longitudinales el aspecto más importante a considerar es la estructura de la matriz de covarianza residual, que es posible modelar especificando la matriz de correlación. En algunos casos también las varianzas pueden ser distintas para algún criterio de agrupamiento y se debe modelar la heteroscedasticidad. Recordemos que existe correlación residual entre observaciones que comparten el mismo valor del criterio de estratificación, conocido también como sujeto, (por ejemplo, tomadas sobre la misma persona, la misma parcela, el mismo animal, el mismo árbol, etc.). Así, la matriz de covarianza residual para todas las observaciones será una matriz diagonal por bloques, y en cada bloque se reflejará la estructura deseada, i.e. simetría compuesta, autorregresiva de orden 1, etc. Para especificar esto, InfoStat presenta dos solapas. En la solapa Correlación se encuentran las opciones que permiten especificar la estructura de correlación de los errores y en la solapa Heteroscedasticidad se pueden seleccionar distintos modelos de varianza. Así, las distintas estructuras de la matriz de covarianza residual que se pueden ajustar resultan de combinar las distintas estructuras de correlación con la posible heteroscedasticidad en el tiempo. Si adicionalmente se desea especificar un efecto aleatorio también es posible hacerlo usando la solapa correspondiente. En este caso se debe tener mucha precaución de no combinar efectos aleatorios, estructuras de correlación y de heteroscedasticidad tales que el modelo final no sea identificable. Esto sucede cuando existe un conjunto infinito de valores de los parámetros para los cuales el modelo es indistinguible, y por lo tanto las soluciones a las ecuaciones de verosimilitud no son únicas. Ejemplos de estas situaciones ocurren cuando se especifica una estructura de correlación de simetría compuesta con un criterio de estratificación (por ejemplo, la parcela) y un efecto aleatorio de ese mismo criterio de estratificación sobre la constante. En este caso la estructura de covarianza de las observaciones será una matriz diagonal por bloques, y cada bloque tendrá una estructura de simetría compuesta. Por lo tanto, esta estructura tiene intrínsecamente dos parámetros. Pero de la manera que la hemos especificado aparecen tres parámetros (varianza del efecto aleatorio, correlación 77 Modelos Mixtos en InfoStat intraclase de la estructura de correlación residual y varianza residual). Esta sobreparametrización hace que existan infinitas soluciones, y por lo tanto los estimadores no se pueden interpretar (y en muchos casos el algoritmo numérico no converge). Otra situación común es la de una correlación sin estructura (corSymm) con un criterio de estratificación dado (por ejemplo la parcela) y un efecto aleatorio de ese mismo criterio de estratificación sobre la constante (intercept). Análisis de un ensayo de establecimiento de forrajeras A continuación se presenta un ejemplo de modelación de observaciones repetidas en el tiempo. Los datos provienen de un ensayo de establecimiento de forrajeras para comparar cinco métodos de labranza (labranza mínima, labranza mínima con herbicida, labranza mínima con herbicida y arado de disco a los 45 días, labranza cero, y labranza convencional) en la región central húmeda de Puerto Rico. La especie usada fue Brachiaria decumbens cv. Basilik. El experimento estaba diseñado en tres bloques completos aleatorizados, y se analizan aquí las medidas de cobertura (porcentaje de cobertura estimado en cada parcela). Hay 5 medidas repetidas, tomadas con intervalos de un mes entre agosto y diciembre de 2001 (Moser y Macchiavelli 2002). Los datos se encuentran en Cobertura forrajes.IDB2 en la carpeta de datos de prueba de InfoStat. Los perfiles promedio de cobertura observados en los cinco tiempos para cada uno de los tratamientos se presentan en la Figura 52. Cobertura de Brachiaria decumbens 50 Cobertura (%) 40 30 20 10 0 1 2 3 4 5 Tiempo Trat1 Trat2 Trat3 Trat4 Trat5 Figura 52: Relación entre cobertura y tiempo para cinco tratamientos del archivo Cobertura forrajes.IDB2. 78 Modelos Mixtos en InfoStat Como estrategia general para analizar estos datos primero se ajustarán modelos con distintas estructuras de covarianza, combinando apropiadamente estructuras de correlación residual, heteroscedasticidad residual y efectos aleatorios. Mediante criterios de verosimilitud penalizada (AIC y BIC) se elegirá el modelo que mejor describa los datos, y usando este modelo se realizarán inferencias acerca de las medias (comparar tratamientos, estudiar el efecto del tiempo, analizar si los perfiles promedio varían en el tiempo, si son paralelos, etc.). Para el elegir el mejor modelo comenzaremos proponiendo un modelo sencillo con pocos parámetros a estimar (i.e. parsimonioso), e iremos adicionando parámetros hasta llegar al modelo sin estructura, que es el menos parsimonioso. Se usarán las siguientes estructuras de covarianza para los datos (covarianza marginal): 1. Errores independientes y homoscedásticos. 2. Errores independientes y heteroscedásticos. 3. Correlación constante entre errores de la misma parcela y varianza residual constante en el tiempo. 4. Correlación constante entre errores de la misma parcela y varianza residual diferente en los distintos tiempos. 5. Estructura autorregresiva de orden 1 entre los errores de la misma parcela y varianza residual constante en el tiempo. 6. Estructura autorregresiva de orden 1 entre los errores de la misma parcela y varianza residual diferente en los distintos tiempos. 7. Estructura autorregresiva de orden 1 entre los errores de la misma parcela, varianza residual constante en el tiempo y efecto aleatorio de parcela. 8. Estructura autorregresiva de orden 1 entre los errores de la misma parcela, varianza residual diferente en los distintos tiempos y efecto aleatorio de parcela. 9. Sin estructura para las correlaciones entre errores provenientes de la misma parcela y varianzas residuales diferentes en el tiempo. Para ajustar estos modelos en primer lugar se deben declarar las variables como se indica en la Figura 53. 79 Modelos Mixtos en InfoStat Figura 53: Ventana de selección de variables para Modelos lineales generales y mixtos con datos del ejemplo Cobertura forrajes.IDB2. En todos los casos se usó el mismo modelo de medias, ya que la parte fija del modelo referido no cambió (imprescindible si se desea comparar estructuras de covarianza usando REML, y por ende los criterios de AIC y BIC) (Figura 54). A continuación se detallará la forma de declarar cada uno de los modelos a evaluar seguido por una salida de InfoStat con las medidas de ajuste correspondientes. 80 Modelos Mixtos en InfoStat Figura 54: Ventana con la solapa Efectos fijos desplegada para los datos del archivo Cobertura forrajes.IDB2. Modelo 1: Errores independientes y homoscedásticos En la solapa Correlación se debe declarar Errores independientes (Figura 55), que es la opción por defecto, y en la solapa Heteroscedasticidad no se declara nada. 81 Modelos Mixtos en InfoStat Figura 55: Ventana con la solapa Correlación desplegada para los datos del archivo Cobertura forrajes.IDB2 y selección de Errores independientes (Modelo 1). Medidas de ajuste del modelo N 75 AIC 465.05 BIC 517.45 logLik -204.53 Sigma R2_0 12.19 0.63 AIC y BIC menores implica mejor Modelo 2: Errores independientes y heteroscedásticos En la solapa Correlación se debe declarar Errores independientes (opción por defecto) y en la solapa Heteroscedasticidad se declara varIdent y en criterio de agrupamiento se declara Tiempo (Figura 56). 82 Modelos Mixtos en InfoStat Figura 56: Ventana con la solapa Heteroscedasticidad desplegada para los datos del archivo Cobertura forrajes.IDB2 con selección de función varIdent con tiempo como criterio de agrupamiento (Modelo 2). Medidas de ajuste del modelo N 75 AIC 465.83 BIC 525.71 logLik -200.91 Sigma R2_0 6.75 0.61 AIC y BIC menores implica mejor Modelo 3: Correlación constante entre datos de la misma parcela y varianza constante en el tiempo. En la solapa Correlación se eligió la opción Simetría Compuesta. Se debe declarar también el Criterio de agrupamiento, en este caso la Parcela, para indicar que se está modelando la correlación de datos provenientes de una misma parcela (Figura 57). En la solapa Heteroscedasticidad se dejó la opción por defecto, es decir no se eligió ningún criterio (para esto ir a la solapa Heteroscedasticidad y borrar la selección anterior desactivando todas las opciones y borrando varIdent(form=~1) con un doble clic). 83 Modelos Mixtos en InfoStat Figura 57: Ventana con la solapa Correlación desplegada para los datos del archivo Cobertura forrajes.IDB2 y selección de simetría compuesta para datos agrupados por parcela (Modelo 3). Medidas de ajuste del modelo N 75 AIC 458.67 BIC 512.94 logLik -200.34 Sigma R2_0 12.59 0.63 AIC y BIC menores implica mejor Modelo 4: Correlación constante entre datos de la misma parcela y varianza diferente en los distintos tiempos. En la solapa Correlación se eligió la opción Simetría compuesta y en la solapa Heteroscedasticidad eligió la opción varIdent y en Criterios de agrupamiento se declara Tiempo. 84 Modelos Mixtos en InfoStat Medidas de ajuste del modelo N 75 AIC 456.66 BIC 518.41 logLik -195.33 Sigma R2_0 8.05 0.48 AIC y BIC menores implica mejor Modelo 5: Estructura autorregresiva de orden 1 entre los errores de la misma parcela y varianza residual constante en el tiempo. En la solapa Correlación se eligió la opción Autorregresivo de orden 1 (Figura 58). Debido a que este modelo tiene en cuenta el orden en que fueron tomadas las observaciones, la variable que indica esto se debe declarar en la ventana correspondiente (en este caso la variable Tiempo). En la solapa Heteroscedasticidad se dejó la opción por defecto, es decir no se eligió ningún criterio (para esto ir a la solapa Heteroscedasticidad y borrar la selección anterior desactivando todas las opciones y borrando varIdent(form=~1) con un doble clic). Figura 58: Ventana con la solapa Correlación desplegada para los datos del archivo Cobertura forrajes.IDB2 y selección de modelo Autorregresivo de orden 1 para datos agrupados por parcela y orden de las observaciones indicado por la variable Tiempo (Modelo 5). 85 Modelos Mixtos en InfoStat Medidas de ajuste del modelo N 75 AIC 449.36 BIC 503.62 logLik -195.68 Sigma R2_0 12.40 0.63 AIC y BIC menores implica mejor Modelo 6: Estructura autorregresiva de orden 1 entre los errores de la misma parcela y varianza residual diferente en los distintos tiempos. En la solapa Correlación se eligió la opción Autorregresivo de orden 1. En la solapa Heteroscedasticidad se eligió la opción varIdent y se especificó el criterio de agrupamiento que en este caso es la variable tiempo. Medidas de ajuste del modelo N 75 AIC 452.40 BIC 514.15 logLik -193.20 Sigma R2_0 8.78 0.63 AIC y BIC menores implica mejor Modelo 7: Estructura autorregresiva de orden 1 entre los errores de la misma parcela, varianza residual constante en el tiempo y efecto aleatorio de parcela. En la solapa Correlación se eligió la opción Autorregresivo de orden 1 (igual que el Modelo 5, Figura 58) y en la solapa Heteroscedasticidad se dejó la opción por defecto, es decir no se eligió ningún criterio y se borraron las selecciones realizadas con anterioridad. En la solapa Efectos aleatorios se declaró a Parcela en Criterios de estratificación (Figura 59). 86 Modelos Mixtos en InfoStat Figura 59: Ventana con la solapa Efectos aleatorios desplegada para los datos del archivo Cobertura forrajes.IDB2 con parcela como criterio de estratificación (Modelo 7). Medidas de ajuste del modelo N 75 AIC 451.36 BIC 507.49 logLik -195.68 Sigma R2_0 12.40 0.63 R2_1 0.63 AIC y BIC menores implica mejor Modelo 8: Estructura autorregresiva de orden 1 entre los errores de la misma parcela, varianza residual diferente en los distintos tiempos y efecto aleatorio de parcela. 87 Modelos Mixtos en InfoStat En la solapa Correlación se eligió la opción Autorregresivo “continuo” de orden 1 (Figura 60) para mostrar que en este caso es igual al Autorregresivo de orden 1. Si los tiempos no fuesen equidistantes, la estructura corAR1 no es aplicable, y se debe usar su análoga continua (corCAR1). Para este ejemplo ambas estructuras son equivalentes debido a que los tiempos son equidistantes. En la solapa Heteroscedasticidad se dejó la opción varIdent (como en la Figura 56). En la solapa Efectos aleatorios se declaró a Parcela como Criterio de estratificación (como en el Modelo 7, Figura 59). Medidas de ajuste del modelo N 75 AIC 454.40 BIC 518.03 logLik -193.20 Sigma R2_0 8.78 0.63 R2_1 0.63 AIC y BIC menores implica mejor Figura 60: Ventana con la solapa Correlación desplegada para los datos del archivo Cobertura forrajes.IDB2 y selección de modelo Autorregresivo continuo de orden 1 para datos agrupados por parcela y orden de las observaciones indicado por la variable Tiempo (Modelo 8). 88 Modelos Mixtos en InfoStat Modelo 9: Sin estructura para los datos provenientes de la misma parcela. En la solapa Correlación se eligió la opción Sin estructura (Figura 61) y en la solapa Heteroscedasticidad se dejó tildada la opción varIdent (como en Figura 56). En la solapa Efectos aleatorios se eliminó Parcela en Criterios de estratificación. Figura 61: Ventana con la solapa Correlación desplegada para los datos del archivo Cobertura forrajes.IDB2 y selección de modelo Sin estructura para datos agrupados por parcela y orden de las observaciones indicado por la variable Tiempo (Modelo 9). Medidas de ajuste del modelo N 75 AIC 425.03 BIC 503.62 logLik -170.51 Sigma R2_0 6.82 0.60 AIC y BIC menores implica mejor 89 Modelos Mixtos en InfoStat Selección de la estructura de covarianza Si comparamos los valores de AIC (o los de BIC) para las estructuras que hemos ajustado, se puede observar que el menor valor se obtiene con el Modelo 9 (AIC = 425.03, BIC = 503.62). Por lo tanto elegimos la covarianza sin estructura. Observemos los parámetros estimados bajo este modelo: Medidas de ajuste del modelo N 75 AIC 425.03 BIC 503.62 logLik -170.51 Sigma R2_0 6.82 0.60 AIC y BIC menores implica mejor Estructura de correlación Modelo de correlacion: General correlation Formula: ~ as.integer(Tiempo) | Parcela Matriz de correlación común 1 2 3 2 0.3230 3 0.7160 0.2760 4 0.0650 0.0990 0.4600 5 0.0450 0.1240 0.4580 4 0.9950 Estructura de varianzas Modelo de varianzas: varIdent Formula: ~ 1 | Tiempo Parámetros del modelo Parámetro Estim 1 1.0000 2 1.1819 3 1.9653 4 2.3775 5 2.2697 Las varianzas estimadas para cada uno de los 5 tiempos se calculan de la siguiente manera: 90 Modelos Mixtos en InfoStat ˆ12 6.82112 46.53 ˆ 22 1.1819 6.8211 64.99 2 ˆ 32 1.9653 6.8211 179.71 2 ˆ 42 2.3775 6.8211 263.00 2 ˆ 52 2.2697 6.8211 239.69 2 Las 10 correlaciones estimadas aparecen directamente como una matriz en Estructura de Correlación. Inferencia sobre las medias Una vez elegida la estructura de covarianza de los datos (en este caso el modelo sin estructura) podemos proceder a realizar inferencias acerca de las medias. Los perfiles promedios observados para cada tratamiento se presentaron en la Figura 52. En un experimento factorial como este, donde se tiene el factor tratamiento y el factor tiempo, lo primero que se debe indagar es si existe interacción entre los tratamientos y el tiempo. Para ello podemos realizar una prueba de Wald, que aparece directamente en InfoStat como Trat:Tiempo en las pruebas secuenciales (recordemos que la interacción es el último término que colocamos en el modelo). Otra opción es realizar una prueba del cociente de verosimilitudes (LRT por sus siglas en inglés). Para esta última no podemos usar REML debido a que estamos probando modelos con efectos fijos diferentes, y por lo tanto los estimadores REML no son comparables. En su lugar se usa el estimador máximo verosímil (ML). Pruebas de hipótesis secuenciales numDF F-value p-value (Intercept) 1 127.93 <0.0001 Bloque 2 2.75 0.0739 Tratam 4 6.28 0.0004 Tiempo 4 16.77 <0.0001 16 1.49 0.1417 Tratam:Tiempo Para realizar una prueba de cociente de verosimilitudes podemos ajustar (con ML) dos modelos con la misma estructura de covarianza (en este ejemplo el modelo sin 91 Modelos Mixtos en InfoStat estructura) pero que difieren en su parte fija: uno contiene la interacción (modelo completo) y el otro no la contiene (modelo reducido): Modelo completo: Medidas de ajuste del modelo N 75 AIC 535.33 BIC 632.67 logLik -225.67 Sigma R2_0 5.29 0.60 logLik -237.51 Sigma R2_0 5.55 0.38 AIC y BIC menores implica mejor Modelo reducido: Medidas de ajuste del modelo N 75 AIC 527.01 BIC 587.27 AIC y BIC menores implica mejor Si bien la prueba LRT se puede obtener directamente desde el menú Análisisexploración de modelos estimados, solapa Modelos, a continuación se muestra otra forma de calcularla. En primer lugar se obtiene el estadístico de la prueba LRT, G 2 log lik completo 2 log lik reducido 2(225.67) 2(237.51) 23.68 . Este tiene 4226=16 grados de libertad, y arroja un valor p=0.0967, por lo que podemos decir que no existe interacción con un nivel de significancia del 5%. Este valor de probabilidad se obtiene a partir de una distribución chi-cuadrado con 16 grados de libertad, y puede ser calculado con la herramienta Calculador de probabilidades y cuantiles del menú Estadísticas de InfoStat (Figura 62). 92 Modelos Mixtos en InfoStat Figura 62: Ventana del Calculador de probabilidades y cuantiles de InfoStat. Ambas pruebas (Wald y LRT) indican una interacción no significativa (aunque los p-valores no son demasiado altos, p=0.1417 y p=0.0967 respectivamente), por lo que podemos (con precaución) realizar pruebas de efectos de tratamiento y tiempo por separado. Contrastando tiempos sucesivos Para comparar los tiempos sucesivos, es decir el tiempo 1 con el tiempo 2, el tiempo 2 con el tiempo 3 y así sucesivamente, se debe activar la solapa Comparaciones y dentro de esta la subsolapa Contrastes y seleccionar el efecto Tiempo (Figura 63). El resto de las ventanas debe quedar como en el Modelo 9, que fue elegido como el de mejor estructura de correlación para explicar el comportamiento de estos datos en el tiempo. 93 Modelos Mixtos en InfoStat Figura 63: Ventana con la solapa Comparaciones desplegada para los datos del archivo Cobertura forrajes.IDB2 y selección de la subsolapa Contrastes. Pruebas de hipótesis para contrastes F gl(num) gl(den) p-valor Cont.1 18.94 1 48 0.0001 Cont.2 2.70 1 48 0.1070 Cont.3 0.19 1 48 0.6640 Cont.4 0.34 1 48 0.5645 Total 16.77 4 48 <0.0001 Las salidas presentadas aquí corresponden a las estimaciones REML. Está claro a partir de estos resultados que, en promedio para los cuatro tratamientos, se ve un cambio significativo entre los tiempos 1 y 2, pero en tiempos posteriores la cobertura promedio no cambia significativamente. Las mismas conclusiones se obtienen realizando una comparación de medias de cada tiempo (LSD): 94 Modelos Mixtos en InfoStat Medias ajustadas y errores estándares para Tiempo LSD Fisher (alfa=0.05) Procedimiento de correccion de p-valores: No Tiempo 3 4 5 2 1 Medias 30.29 28.53 28.27 24.53 14.73 E.E. 3.46 4.19 4.00 2.08 1.76 A A A A B Letras distintas indican diferencias significativas(p<= 0.05) Comparación de tratamientos Medias ajustadas y errores estándares para Tratam LSD Fisher (alfa=0.05) Procedimiento de correccion de p-valores: No Tratam 5 1 4 3 2 Medias 39.60 31.27 24.96 17.33 13.19 E.E. 5.15 5.15 5.15 5.15 5.15 A A A B B B C C C Letras distintas indican diferencias significativas(p<= 0.05) A partir de esta comparación de medias ajustadas se puede concluir que los tratamientos 5, 1 y 4 son los que proveen mayor cobertura y no se diferencias estadísticamente entre sí. 95 Modelos Mixtos en InfoStat Análisis de un ensayo de drogas para asma Una compañía farmacéutica ha examinado los efectos de dos drogas (A y B) sobre la capacidad respiratoria de pacientes de asma (Littel et ál. 2002, 2006). Las dos drogas y un placebo fueron administradas a un grupo de pacientes en forma aleatoria. Se contó con 24 pacientes por cada uno de los tres tratamientos. A cada paciente se le midió la capacidad respiratoria basal (Cap_Rep_Bas) inmediatamente antes de aplicarle el tratamiento y la capacidad respiratoria cada hora durante las 8 horas siguientes (Cap_Respirat). Los datos están en el archivo CapacidadRespiratoria.IDB2. Usando nuevamente la estrategia definida en los ejemplos anteriores, primero se ajustarán modelos con distintas estructuras de covarianza, combinando apropiadamente estructuras de correlación residual, heteroscedasticidad residual y efectos aleatorios. Mediante criterios de verosimilitud penalizada (AIC y BIC) y pruebas de cociente de verosimilitud se elegirá el modelo que mejor describa los datos. Una vez seleccionado el modelo de estructura de covarianza adecuado se realizarán inferencias acerca de las medias (comparar medias de drogas, estudiar el efecto del tiempo, analizar si los perfiles promedio varían en el tiempo, si son paralelos, etc.). Es importante destacar que toda la inferencia sobre las medias estará basada en el modelo de estructura de covarianza seleccionado. Debido a que la variable que identifica al paciente (Paciente) en la base de datos toma valores iguales dentro de cada droga, para identificar a los 72 pacientes de este estudio se ha debido crear una nueva variable (paciente_droga) que identifica completamente al paciente. Para hacer esto se ha usado el menú Datos, submenú Cruzar categorías para formar una nueva variable (seleccionado Paciente y Droga en la ventana del selector de variables). Este es un experimento bifactorial y se usa un modelo que contempla los factores Droga, Hora y su interacción y la covariable Cap_Rep_Bas (todos de efectos fijos). Para realizar el análisis de este modelo, se deben declarar las variables de la siguiente forma (Figura 64). 96 Modelos Mixtos en InfoStat Figura 64: Ventana de selector de variables con los datos del archivo CapacidadRespiratoria.IDB2. En primer lugar se evaluará un conjunto de modelos para determinar cuál es el que mejor ajusta. Los modelos evaluados son: 1. Errores independientes y varianzas residuales homoscedásticas. 2. Simetría compuesta y varianzas residuales homoscedásticas. 3. Auto regresivo de orden 1 y varianzas residuales homoscedásticas. 4. Auto regresivo de orden 1 y varianzas residuales heteroscedásticas. 5. Auto regresivo de orden 1 y varianzas residuales homoscedásticas y efecto aleatorio de paciente. 6. Auto regresivo de orden 1 y varianzas residuales heteroscedásticas y efecto aleatorio de paciente. 7. Matriz de varianzas heteroscedásticas. y covarianzas sin estructura y varianzas residuales La especificación de la parte fija del modelo es la misma para los 7 modelos evaluados (Figura 65). Para obtener el ajuste del Modelo 1 solo se debe activar el botón Aceptar con el modelo de efectos fijos presentado a continuación: 97 Modelos Mixtos en InfoStat Figura 65: Ventana con la solapa Efectos fijos desplegada con los datos del archivo CapacidadRespiratoria.IDB2. Para ajustar el Modelo 2, se deben especificar las ventanas como en la Figura 65 y la Figura 66. El Modelo 3 se especifica como en la Figura 65 y la Figura 67. El Modelo 4 se especifica como el anterior más el agregado de varianzas residuales heterogéneas como en la Figura 68. El Modelo 5 se especifica con las ventanas presentadas en la Figura 65, la Figura 67 y la Figura 69. El Modelo 6 es como el Modelo 5 más la especificacion de varianzas residuales heterogéneas (Figura 68). El modelo 7 se especifica como se muestra en la Figura 65, Figura 68 y Figura 70). 98 Modelos Mixtos en InfoStat Figura 66: Ventana con la solapa Correlación desplegada, opción Simetría compuesta, con los datos del archivo CapacidadRespiratoria.IDB2. 99 Modelos Mixtos en InfoStat Figura 67: Ventana con la solapa Correlación desplegada, opción Autorregresivo de orden 1, con los datos del archivo CapacidadRespiratoria.IDB2. 100 Modelos Mixtos en InfoStat Figura 68: Ventana con la solapa Heteroscedasticidad desplegada con los datos del archivo CapacidadRespiratoria.IDB2. 101 Modelos Mixtos en InfoStat Figura 69: Ventana con la solapa Efectos aleatorios desplegada con los datos del archivo CapacidadRespiratoria.IDB2. 102 Modelos Mixtos en InfoStat Figura 70: Ventana con la solapa Correlación desplegada, opción Sin estructura, con los datos del archivo CapacidadRespiratoria.IDB2. Luego de ajustar los siguientes modelos estos son los resultados: Cuadro 2. Características y medidas de ajuste de los modelos evaluados para los datos del archivo CapacidadRespiratoria.IDB2. Efecto Modelo Aleatorio paciente Correlación Residual Varianzas residuales heterogéneas en el tiempo AIC BIC log lik 1 NO NO NO 968.94 1081.04 -458.47 2 NO Simetría Compuesta NO 401.29 517.71 -173.65 3 NO AR1 NO 329.04 445.45 -137.52 4 NO AR1 SÍ 324.57 471.17 -128.28 5 SÍ AR1 NO 303.03 423.76 -123.52 6 SÍ AR1 SÍ 287.80 438.71 -108.90 7 NO Sin estructura SÍ 270.27 533.29 -74.14 103 Modelos Mixtos en InfoStat A partir del Cuadro 2 podemos observar que los modelos 6 y 7 presentan los valores más bajos de AIC mientras que los modelos 5 y 6 presentan los valores más bajos para BIC. Una prueba formal de cociente de verosimilitud para comparar los modelos 5 y 6 puede obtenerse mediante: X 2 2(log lik modelo reducido - log lik modelo completo) 2(123.52 108.90) 29.24 Como ambos modelos difieren en 7 parámetros (el Modelo 5 tiene una única varianza residual y el Modelo 6 tiene 8 varianzas residuales), el estadístico de verosimilitud se compara con un valor crítico de una distribución chi-cuadrado con 7 grados de libertad. Al hacer esto con el calculador de probabilidades y cuantiles de InfoStat obtenemos un p-valor de 0.0001, lo que nos lleva a escoger el modelo completo (Modelo 6). La misma prueba se puede realizar con el menú Estadísticas>>Modelos lineales generales y mixtos>> Análisis-exploración de modelos estimados. Para comparar ambos modelos seleccionamos la solapa Modelos y obtenemos los siguientes resultados: Comparación de modelos Model 5 6 df 28 35 AIC 303.03 287.80 BIC 423.76 438.71 logLik -123.52 -108.90 Test 1 vs 2 L.Ratio p-value 29.23 0.0001 Los resultados de la prueba del cociente de verosimilitud indican que entre estos dos modelos el mejor es el Modelo 6. Luego, resta solo comparar el Modelo 6 con el 7. En este caso el modelo reducido es el 6 y el completo es el 7. Los resultados para esta comparación son: Comparación de modelos Model 7 6 df 61 35 AIC 270.27 287.80 BIC 533.29 438.71 logLik -74.14 -108.90 Test 1 vs 2 L.Ratio p-value 69.53 <0.0001 Los resultados indican que el Modelo 7 es el mejor. Por lo tanto el modelo seleccionado tiene una estructura de correlación residual sin estructura y varianzas residuales heterogéneas en el tiempo. La salida completa para este modelo se presenta a continuación: 104 Modelos Mixtos en InfoStat Modelos lineales generales y mixtos Especificación del modelo en R modelo009_Cap_Respirat_REML<gls(Cap_Respirat~1+Droga+Hora+Droga:Hora+Cap_Resp_base ,weight=varComb(varIdent(form=~1|Hora)) ,correlation=corSymm(form=~as.integer(as.character(Hora))|Paciente_Dro ga) ,method="REML" ,na.action=na.omit ,data=R.data09) Resultados para el modelo: modelo009_Cap_Respirat_REML Variable dependiente:Cap_Respirat Medidas de ajuste del modelo N 576 AIC 270.27 BIC 533.29 logLik -74.14 Sigma R2_0 0.48 0.55 AIC y BIC menores implica mejor Pruebas de hipótesis marginales(SC tipo III) (Intercept) Droga Hora Cap_Resp_base Droga:Hora numDF F-value 1 6.49 2 7.25 7 13.72 1 92.57 14 4.06 p-value 0.0111 0.0008 <0.0001 <0.0001 <0.0001 Pruebas de hipótesis secuenciales (Intercept) Droga Hora Cap_Resp_base Droga:Hora numDF F-value 1 3936.01 2 13.87 7 13.72 1 92.57 14 4.06 p-value <0.0001 <0.0001 <0.0001 <0.0001 <0.0001 Estructura de correlación Modelo de correlación: General correlation Formula: ~ as.integer(as.character(Hora)) | Paciente_Droga Matriz de correlación común 1 2 3 4 5 6 7 8 1 1.00 0.89 0.88 0.78 0.69 0.67 0.52 0.65 2 0.89 1.00 0.91 0.87 0.81 0.70 0.59 0.70 3 0.88 0.91 1.00 0.91 0.81 0.75 0.64 0.74 4 0.78 0.87 0.91 1.00 0.82 0.73 0.67 0.75 5 0.69 0.81 0.81 0.82 1.00 0.85 0.73 0.84 6 0.67 0.70 0.75 0.73 0.85 1.00 0.81 0.88 7 0.52 0.59 0.64 0.67 0.73 0.81 1.00 0.82 8 0.65 0.70 0.74 0.75 0.84 0.88 0.82 1.00 105 Modelos Mixtos en InfoStat Estructura de varianzas Modelo de varianzas: varIdent Formula: ~ 1 | Hora Parámetros de la función de varianza Parámetro 1 2 3 4 5 6 7 8 Estim 1.00 1.07 1.06 1.15 1.12 1.07 1.09 1.15 Medias ajustadas y errores estándares para Droga LSD Fisher (alfa=0.05) Procedimiento de correccion de p-valores: No Droga Medias B 3.33 A 3.11 P 2.82 E.E. 0.09 0.09 0.09 A A B Letras distintas indican diferencias significativas(p<= 0.05) Medias ajustadas y errores estándares para Hora LSD Fisher (alfa=0.05) Procedimiento de correccion de p-valores: No Hora 1 2 3 4 5 6 7 8 Medias 3.33 3.30 3.22 3.12 3.02 2.96 2.88 2.87 E.E. 0.06 0.06 0.06 0.06 0.06 0.06 0.06 0.06 A A B C D D E E Letras distintas indican diferencias significativas(p<= 0.05) Medias ajustadas y errores estándares para Droga*Hora LSD Fisher (alfa=0.05) Procedimiento de correccion de p-valores: No Droga B B B A B A B A B Hora 1 2 3 1 4 2 5 3 6 Medias 3.69 3.63 3.58 3.47 3.44 3.39 3.25 3.18 3.08 E.E. 0.10 0.10 0.10 0.10 0.11 0.10 0.11 0.10 0.10 A A A A B B B B B C C C C D D D D E E E F F G 106 Modelos Mixtos en InfoStat A A B A B P P P A A P P P P P 5 4 8 6 7 3 2 4 7 8 1 6 7 5 8 3.05 3.04 3.01 2.98 2.98 2.90 2.89 2.87 2.87 2.86 2.83 2.82 2.79 2.77 2.73 0.11 0.11 0.11 0.10 0.11 0.10 0.10 0.11 0.11 0.11 0.10 0.10 0.11 0.11 0.11 E E E E F F F F F F G G G G G G G G G G G G H H H H H H H H H H H H H H I I I I I I I I I I I I I Letras distintas indican diferencias significativas(p<= 0.05) Podemos ver que hay interacción significativa entre la droga y el tiempo (p<0.0001), por lo que procederemos a realizar un Gráfico de interacción. Para realizar este gráfico en primer lugar se copiaron las Medias ajustadas y errores estándares para Droga*Hora y se pegaron en una nueva tabla de InfoStat. Esta tabla fue guardada como MedCapRes.IDB2. Luego en el menú Gráficos>>Gráficos de puntos se declararon las variables como se muestra a continuación (Figura 71 y Figura 72): Figura 71: Ventana de selector de variables para los datos del archivo MedCapRes.IDB2. 107 Modelos Mixtos en InfoStat Figura 72: Ventana de selector de variables con solapa Particiones activada para los datos del archivo MedCapRes.IDB2. Es importante recalcar que debido a que los errores estándar de cada una de las combinaciones de tratamientos y horas son diferentes éstos deben ser tenidos en cuenta al momento de solicitar el gráfico. Esto se logra declarando la medida de error en la subventana Error. Con estas especificaciones se obtiene el gráfico para estudiar la interacción (Figura 73). 108 Modelos Mixtos en InfoStat 3.80 3.70 3.60 Droga A Droga B Placebo Capacidad respiratoria media 3.50 3.40 3.30 3.20 3.10 3.00 2.90 2.80 2.70 2.60 1 2 3 4 5 6 7 8 Hora Figura 73: Gráfico de puntos para estudiar la interacción entre tratamientos y hora con los datos del archivo CapacidadRespiratoria.IDB2. Podemos observar que mientras el placebo tiene una respuesta prácticamente constante, las drogas A y B aumentan la capacidad respiratoria después de su aplicación. Esta capacidad va disminuyendo con el tiempo, y siempre es superior el valor medio de la droga B respecto a la droga A. Para encontrar diferencias significativas entre los tratamientos en cada una de las horas se pueden realizar contrastes. En este caso, dentro de cada hora se pueden probar hipótesis sobre igualdad de medias entre drogas y placebo, y entre las dos drogas. Para la obtención de los contrastes (en este caso ortogonales) se deben declara en la solapa Comparaciones, subsolapa Contrastes como en la Figura 74. 109 Modelos Mixtos en InfoStat Figura 74: Ventana con la solapa Comparaciones, subsolapa Contrastes con los datos del archivo CapacidadRespiratoria.IDB2. A continuación se presentan lo valores de probabilidad para los contrastes solicitados: Pruebas de hipótesis para contrastes Droga*Hora Ct.1 Ct.2 Ct.3 Ct.4 Ct.5 Ct.6 Ct.7 Ct.8 Ct.9 Ct.10 Ct.11 Ct.12 Ct.13 Ct.14 Ct.15 Ct.16 Total F gl(num) 40.08 1 2.54 1 23.46 1 2.46 1 14.55 1 7.36 1 7.39 1 6.37 1 8.11 1 1.63 1 2.86 1 0.53 1 1.09 1 0.53 1 2.13 1 0.94 1 5.19 16 gl(den) 551 551 551 551 551 551 551 551 551 551 551 551 551 551 551 551 551 p-valor <0.0001 0.1119 <0.0001 0.1170 0.0002 0.0069 0.0068 0.0119 0.0046 0.2022 0.0914 0.4651 0.2965 0.4656 0.1446 0.3319 <0.0001 110 Modelos Mixtos en InfoStat Los Contrastes 1, 3, 5, 7 y 9 comparan el placebo con el promedio de las drogas para las horas 1, 2, 3 ,4 y 5 respectivamente. Debido a que todos estos son significativos (p<0.05) podemos decir que recién a la hora 6 de aplicadas las drogas estas pierden su efecto, ya que los contrastes 11, 13 y 15 no son significativos. Respecto a la comparación de las drogas entres sí, la superioridad de la B sobre la A se manifiesta (p<0.05) solo en las horas 3 y 4 (contrastes 6 y 8 respectivamente). 111 Modelos Mixtos en InfoStat Análisis de bolsas de descomposición En los ensayos de descomposición de hojarasca la materia seca remanente en cada tiempo es analizada, generalmente, mediante ANCOVA, usando el tiempo como covariable y transformación logarítmica de la respuesta, o ANOVA para un diseño en parcelas divididas, cuando los periodos de evaluación son equi-distantes. Las unidades de observación consisten en bolsas conteniendo el material vegetal. Usualmente estas bolsas son agrupadas para conformar una repetición y permitir su evaluación a lo largo del tiempo, evaluando el contenido de una bolsa en cada instancia de valoración. Aunque en cada tiempo las bolsas evaluadas son distintas, en muchas ocasiones la estructura de correlación que supone independencia o simetría compuesta (inducida por la agrupación de bolsas que representan una repetición) no es suficiente para explicar las correlaciones observadas. Las observaciones cercanas en el tiempo suelen estar más correlacionadas que las lejanas, o las correlaciones entre observaciones en los primeros tiempos son diferentes a las de los últimos. El uso de modelos mixtos permite no sólo manejar estructuras de correlación más complejas sino también la posibilidad de modelar varianzas heterogéneas. En estos modelos los tratamientos pueden ser incluidos como factores de clasificación y el tiempo puede modelarse tanto como una covariable o como un factor. Este último caso produce modelos menos parsimoniosos pero más flexibles para modelar diferentes tendencias en el tiempo. Por otra parte, la introducción de efectos aleatorios sobre los parámetros que involucran al tiempo puede ser usada para corregir falta de ajuste. En el ejemplo, que se presenta a continuación, se analizan un conjunto de datos proveniente de un ensayo de descomposición realizado en ambiente acuático tropical (Martinez 2006). Los tratamientos comparados consisten en: dos especies (Guadua sp. y Ficus sp.) de las cuales se obtiene el material vegetal y dos tamaño de la trama de la bolsa donde se coloca el material (tramado fino y grueso). Los cuatro tratamientos contaron con 5 repeticiones (conteniendo 7 bolsas cada una) y fueron evaluados en 7 tiempos. El propósito de este ensayo fue establecer el efecto de los factores y el tiempo sobre la tasa de descomposición. Los datos se encuentran en el archivo Descomposición.IDB2. Los datos originales (materia seca remanente) fueron transformados a logaritmos. El gráfico del logaritmo de la materia seca remanente (en adelante la respuesta) en función del tiempo y para cada tratamiento (Figura 75) muestra un decaimiento del peso seco 112 Modelos Mixtos en InfoStat remanente en función del tiempo. Se insinúa también una posible falta de homoscedasticidad en función del tiempo y dependiente de la especie y el tramado de la tela de la bolsa. Una primera aproximación a la modelación de estos datos podría ser el ajuste de un modelo de regresión con ordenadas al origen y pendientes diferentes. Para realizar este ajuste se invocó al módulo de modelos mixtos indicando como variable dependiente al LnPesoSeco, como factores de clasificación a Especie y Bolsa y como covariable al Tiempo. Luego en la solapa de la parte fija del modelo se indicaron los términos que se presentan en la Figura 76. El gráfico del modelo ajustado se presenta en la Figura 77. 1.5 1.0 0.5 0.0 LnPesoSeco -0.5 -1.0 -1.5 -2.0 -2.5 -3.0 -3.5 -4.0 0 10 20 30 40 50 60 70 80 90 Tiempo Ficus:Fino Ficus:Grueso Guadua:Fino Guadua:Grueso Figura 75: Gráfico de puntos para el logaritmo del peso seco remanente en función del tiempo, para los cuatro tratamientos (Especie-Bolsa). Archivo Descomposición.IDB2. 113 Modelos Mixtos en InfoStat Figura 76: Especificación del modelo de regresión lineal con ordenadas al origen y pendientes diferentes para el logaritmo de la materia seca remanente en función del tiempo para cuatro tratamientos dados por la especie de origen del material vegetal y el tramado de la bolsa que lo almacena. Archivo Descomposición.IDB2. 114 Modelos Mixtos en InfoStat 1.5 1.0 0.5 0.0 LnPesoSeco -0.5 -1.0 -1.5 -2.0 -2.5 -3.0 -3.5 -4.0 0 10 20 30 40 50 60 70 80 90 Tiempo Ficus:Fino Ficus:Grueso Guadua:Fino Guadua:Grueso Figura 77: Gráfico de puntos para el logaritmo del peso seco remanente en función del tiempo, para los cuatro tratamientos (Especie-Bolsa). Archivo Descomposición.IDB2. La Figura 77 muestra que el ajuste de rectas específicas por tratamiento, es una aproximación que, aunque plausible, no da cuenta de algunas particularidades de la pérdida de peso seco. Esto se refleja en la presencia de curvatura en los residuos (Figura 78). Una forma de resolver el problema de la presencia de curvatura es la imposición de un modelo que incluya términos cuadráticos para el tiempo. Para ello tendremos que extender el modelo propuesto en Figura 76, incluyendo todos los términos correspondientes al tiempo al cuadrado. Para simplificar la notación hemos creado tres variables T1 y T2 que representan el tiempo y el tiempo al cuadrado y Especie_Bolsa que identifica los cuatro tratamientos. T1 es el tiempo centrado respecto del valor 30 (días) y T2 el cuadrado de T1. La razón para centrar las covariables es romper la colinealidad que resulta de utilizar una regresora y su cuadrado y mejorar la condición de la matriz X’X. Las variables T1 y T2 así como Especie_Bolsa se incluyen en el archivo Descomposición.IDB2. En la invocación del módulo de modelos mixtos debería incluirse Especie_Bolsa como factor de clasificación y T1 y T2 como covariables. Luego en la solapa de los efectos fijos del modelo debería verse como se muestra en la Figura 79. 115 Modelos Mixtos en InfoStat 3.50 RE_0_LnPesoSeco 1.75 0.00 -1.75 -3.50 0 10 20 30 40 50 60 70 80 90 Tiempo Figura 78: Gráfico de residuos estudentizados (Pearson) vs. Tiempo, para un modelo de regresión de la materia seca residual en función del tiempo para cuatro tratamientos (Especia-Bolsa) con diferentes ordenadas y pendientes. Archivo Descomposición.IDB2. Figura 79: Especificación del modelo de regresión lineal con ordenadas y pendientes diferentes para el logaritmo de la materia seca remanente en función del tiempo para cuatro tratamientos dados por la especie de origen del material vegetal y el tramado de la bolsa que lo almacena. Archivo Descomposición.IDB2. 116 Modelos Mixtos en InfoStat 1.50 1.00 0.50 0.00 LnPesoSeco -0.50 -1.00 -1.50 -2.00 -2.50 -3.00 -3.50 -4.00 0 10 20 30 40 50 60 70 80 90 Tiempo Ficus:Fino Ficus:Grueso Guadua:Fino Guadua:Grueso Figura 80: Ajustes del modelo de regresión polinómica de orden 2 con ordenadas y pendientes diferentes para el logaritmo de la materia seca remanente en función del tiempo y el tiempo^2 (centrados) para cuatro tratamientos dados por la especie de origen del material vegetal y el tramado de la bolsa que lo almacena. Archivo Descomposición.IDB2. Los residuos del modelo ajustado según Figura 79, muestran dos problemas: heteroscedasticidad (que depende del tiempo y los tratamientos) y falta de ajuste, ya que para algunos tratamientos y tiempos, los residuos de Pearson aparecen por encima o por debajo de la línea del cero (Figura 81). En este punto optaremos por modelar primeramente, el problema de heteroscedasticidad utilizando una función de varianza identidad. Para ello, en la ventana de especificación del modelo dejaremos la parte fija tal cual se indicó en la Figura 79, pero en la solapa Heteroscedasticidad indicaremos que la varianza debe ser estimada de manera diferente para la combinación de tiempo y tratamiento según se muestra en la Figura 82. Los residuos estudentizados vs. tiempo para este modelo se presentan en la (Figura 83). Aún cuando se pudo subsanar, en gran medida, el problema de la heteroscedasticidad, persisten problemas de falta de ajuste que se visualizan en conjuntos de residuos de un único tratamiento en un tiempo dado que quedan ya sean todos positivos o negativos. 117 Modelos Mixtos en InfoStat 4.00 Residuos LnPesoSeco (Pearson) 3.00 2.00 1.00 0.00 -1.00 -2.00 -3.00 -4.00 -5.00 -6.00 -7.00 0 23 45 68 90 Tiempo Ficus:Fino Ficus:Grueso Guadua:Fino Guadua:Grueso Figura 81: Residuos estudentizados (Pearson) vs. Tiempo para el modelo de regresión polinómica de orden 2 con ordenadas y pendientes diferentes para el logaritmo de la materia seca remanente en función del tiempo y el tiempo^2 (centrados) para cuatro tratamientos dados por la especie de origen del material vegetal y el tramado de la bolsa que lo almacena. Archivo Descomposición.IDB2. Figura 82: Especificación de la parte heteroscedástica del modelo de regresión polinómica de orden 2 con ordenadas y pendientes diferentes para el logaritmo de la materia seca remanente en función del tiempo y el tiempo^2 (centrados) para cuatro tratamientos dados por la especie de origen del material vegetal y el tramado de la bolsa que lo almacena. Archivo Descomposición.IDB2. 118 Modelos Mixtos en InfoStat Una forma de resolver esta falta de ajuste, es agregar efectos aleatorios sobre el nivel medio para las combinaciones de tiempo y tratamientos. Si en la solapa Efectos aleatorios agregamos Tiempo_Especie_Bolsa y dejamos tildado el casillero correspondiente a la Constante estamos indicando que se trata de un corrimiento aleatorio que afecta la ordenada al origen para cada conjunto de observaciones correspondientes a la combinación Tiempo, Especie y Bolsa (Figura 84). Finalmente, el gráfico de residuos estudentizados de este modelo muestra una imagen donde no hay evidencia de falta de ajuste o presencia de heteroscedasticidad (Figura 85). Residuos LnPesoSeco (Pearson) 2.50 1.25 0.00 -1.25 -2.50 0 23 45 68 90 Tiempo Ficus:Fino Ficus:Grueso Guadua:Fino Guadua:Grueso Figura 83: Residuos estudentizados (Pearson) vs Tiempo para el modelo heteroscedástico de regresión con ordenadas y pendientes diferentes por tratamiento para el logaritmo de la materia seca remanente en función del tiempo y el tiempo^2 (centrados) para cuatro tratamientos dados por la especie de origen del material vegetal y el tramado de la bolsa que lo almacena. Archivo Descomposición.IDB2. 119 Modelos Mixtos en InfoStat Figura 84: Especificación de la parte aleatoria del modelo heteroscedástico de regresión polinómica de orden 2 con ordenadas y pendientes diferentes para el logaritmo de la materia seca remanente en función del tiempo y el tiempo^2 (centrados) para cuatro tratamientos dados por la especie de origen del material vegetal y el tramado de la bolsa que lo almacena. Archivo Descomposición.IDB2. Residuos LnPesoSeco (Pearson) 2.50 1.25 0.00 -1.25 -2.50 0 10 20 30 40 50 60 70 80 90 Tiempo Ficus:Fino Ficus:Grueso Guadua:Fino Guadua:Grueso Figura 85: Residuos estudentizados (Pearson) vs Tiempo para el modelo heteroscedástico de regresión con ordenadas y pendientes diferentes por tratamiento y el agregado de un efecto aleatorio sobre la constante que es particular para cada combinación de tiempo y tratamiento, para el logaritmo de la materia seca remanente en función del tiempo y el tiempo^2 (centrados) para cuatro tratamientos dados por la especie de origen del material vegetal y el tramado de la bolsa que lo almacena. Archivo Descomposición.IDB2. 120 Modelos Mixtos en InfoStat Finalmente, como el propósito de este ensayo fue calcular las tasas de descomposición y lo que hemos ajustado es un modelo lineal para el logaritmo del peso de materia seca remanente, podemos estimar la tasa de descomposición como la derivada de -exp(modelo ajustado). Utilizaremos la interfase con R para obtener estas derivadas. Apretando la tecla F9 se invoca la ventana del intérprete de R (Figura 86). A la derecha de la ventana aparecerán una lista de los objetos R que se hayan creado durante la sesión de trabajo. En esta lista debe aparecer el modelo ajustado utilizando el modulo de modelos mixtos, el nombre de estos objetos es “modelo”+ número correlativo_nombre de la variable dependiente_método de estimación. En nuestro ejemplo debería aparecer modelo#_LnPesoSeco_REML (en la posición # debe haber un número que depende del número de veces que se ajusto un modelo para la misma variable dependiente). En el ejemplo figura el modelo modeloOO1_LnPesoSeco_REML. Figura 86: Intérprete de R. Tiene 4 paneles. Script: contiene el o los programas R que se quieren ejecutar. Output: la salida de la ejecución de un script o de la visualización de un objeto, Objetos: la lista de los objetos residente en la memoria de R. Finalmente un panel inferior muestra los mensajes y reporte de errores que envía R a la consola. Para calcular las tasas de descomposición tenemos que comprender qué es lo que hemos ajustado con el modelo lineal estimado. La parte fija de modelo propuesto fue: 121 Modelos Mixtos en InfoStat Especie_Bolsa Especie_Bolsa*T1 Especie_Bolsa*T2 Este modelo especifica una regresión polinómica de segundo grado en el tiempo (centrado alrededor de 30 días) para cada una de las combinaciones de Especie y tramado de Bolsa. Así, lo que estimamos es una función de la forma: ln PesoSeco i 0 i1 T 30 i 2 T 30 2 (7) Donde el índice i indica el tratamiento (en este caso i identifica a las cuatro combinaciones de Especie y tramado de Bolsa). Es decir que vamos a tener una ecuación como (7) específica para cada condición. Los coeficientes estimados de la parte fija pueden obtenerse durante la estimación del modelo tildando, en la solapa Efectos fijos, la opción Mostrar coeficientes de la parte fija. Como vamos a utilizar R para calcular las derivadas de (7), revisaremos estos coeficientes desde R. Si en la ventana Script escribimos: Modelo004_LnPesoSeco_REML$coefficients$fixed y apretamos al final de la línea Shift Enter aparecerá en el output la siguiente salida: (Intercept) Especies_BolsaFicus_Grueso -0.7738921651 -0.5941956918 Especies_BolsaGuadua_Fino Especies_BolsaGuadua_Grueso 1.5901279280 1.5369627029 Especies_BolsaFicus_Fino:T1 Especies_BolsaFicus_Grueso:T1 -0.0326126598 -0.0508364778 Especies_BolsaGuadua_Fino:T1 Especies_BolsaGuadua_Grueso:T1 -0.0086055613 -0.0192635993 Especies_BolsaFicus_Fino:T2 Especies_BolsaFicus_Grueso:T2 0.0002938702 0.0004422140 Especies_BolsaGuadua_Fino:T2 Especies_BolsaGuadua_Grueso:T2 0.0000571603 -0.0002451274 Los primeros 4 coeficientes (leyendo de izquierda a derecha), corresponden a las ordenadas al origen i 0 de: Ficus_Fino, Ficus_Grueso, Guadua_Fino y 122 Modelos Mixtos en InfoStat Guadua_Grueso. La ordenada al origen de Ficus_Fino no aparece explícitamente porque está confundido con la ordenada al origen (Intercept) que se supone presente por defecto. Los segundos 4 coeficientes (-0.0326126598,…,,-0.0192635993) son los coeficientes i1 del término lineal de (7) y los últimos 4 (0.0002938702,…, -0.0002451274) son los coeficientes i 2 del término cuadrático en (7). Luego, el peso seco remanente para la Especie Ficus con Bolsa de tramado Fino la ecuación será: ln PesoSeco 0.7738921651 0.0326126598 (T 30) 0.0002938702(T 30) 2 Como la función (7) representa el peso seco remanente, el peso descompuesto debería calcularse como: (8) 2 i 2 T 30 (9) PesoSecoConsumido PesoInicial exp i 0 i1 T 30 i 2 T 30 2 En tanto la tasa de descomposición, sería la derivada de esta función, es decir: TasaDescomp exp i 0 i1 T 30 i 2 T 30 2 i1 El siguiente script genera una tabla cuya primera columna es el tiempo y las restantes las tasas de descomposición para cada uno de los tratamientos. Tener en cuenta que se debe especificar el modelo que mejor ajustó (en nuestro caso modelo004): a=modelo004_LnPesoSeco_REML$coefficients$fixed T=seq(0,90,1) dFF = -exp(a[1]+(T-30)*a[5]+(T-30)*(T-30)*a[9]) *(a[5]+2*(a[9] *(T-30))) dFG = -exp(a[2]+(T-30)*a[6]+(T-30)*(T-30)*a[10])*(a[6]+2*(a[10]*(T-30))) dGF = -exp(a[3]+(T-30)*a[7]+(T-30)*(T-30)*a[11])*(a[7]+2*(a[11]*(T-30))) dGG = -exp(a[4]+(T-30)*a[8]+(T-30)*(T-30)*a[12])*(a[8]+2*(a[12]*(T-30))) Tasas=as.data.frame=cbind(T,dFF,dFG,dGF,dGG) 123 Modelos Mixtos en InfoStat En la lista de objetos aparecerán los objetos a, T, dFF, dFG, dGF, dGG y Tasas. Haciendo clic sobre Tasas, con el botón derecho del ratón aparecerá un menú de acciones entre las que se encuentra Convertir matriz, data frame o vector a tabla InfoStat. Seleccionando esta opción obtendremos una nueva tabla InfoStat como la que se muestra a la derecha de este párrafo. Utilizando el submenú Diagrama de dispersión en el menú Gráficos podemos obtener la siguiente representación de las tasas de descomposición (Figura 87). Para ello se asignaron las variables dFF, dFG, dGF y dGG al Eje Y y la variable T al Eje X, en la ventana de diálogo emergente del submenú Diagrama de dispersión. 0.30 Tasa de descomposición 0.25 0.20 0.15 0.10 0.05 0.00 0 10 20 30 40 50 60 70 80 90 Tiempo Ficus Fino Ficus Grueso Guadua Fino Guadua Grueso Figura 87: Curvas de tasas de descomposición según especie y tramado de la bolsa de almacenamiento. 124 Modelos Mixtos en InfoStat Uso de modelos mixtos para el control de la variabilidad espacial en ensayos agrícolas Correlación espacial La estratificación o bloqueo de parcelas es una técnica usada para controlar los efectos de variación entre las unidades experimentales. Los bloques son grupos de unidades experimentales formados de manera tal que las parcelas dentro de los bloques sean lo más homogéneas posible. Los diseños con estratificación de parcelas tales como el diseño en bloques completos aleatorizados (DBCA), los diseños en bloques incompletos y los látices son más eficientes que el diseño completamente aleatorizado cuando las diferencias entre unidades experimentales que conforman un mismo estrato (bloque) son mínimas y las diferencias entre los estratos son máximas. Cuando esta condición no se cumple puede ocurrir una sobrestimación de la varianza del error y, si los datos son desbalanceados, también puede presentarse un sesgo en las estimaciones de los efectos de tratamientos. Cuando se evalúan muchos tratamientos en parcelas a campo, el tamaño de los bloques necesarios para lograr una repetición del ensayo es grande y por tanto resulta difícil asegurar la homogeneidad de las parcelas que conforman el bloque; las parcelas más próximas pueden ser más similares que las más distantes, generando variabilidad espacial (Casanoves et ál. 2005). La variabilidad espacial se refiere a la variación entre observaciones realizadas sobre parcelas con arreglos espaciales sobre el terreno. Debido a la existencia de variabilidad espacial dentro de bloques, el análisis de varianza estándar para los diseños que involucran el bloqueo de unidades experimentales no siempre elimina los sesgos en las comparaciones de efectos de tratamientos. La variación de parcela a parcela dentro de un mismo bloque puede deberse a competencia, heterogeneidad en la fertilidad del suelo, dispersión de insectos, malezas, enfermedades del cultivo o labores culturales, entre otros. Por este motivo se han propuesto procedimientos estadísticos que contemplan la variación espacial entre parcelas y que van desde el ajuste de medias de tratamientos en función de lo observado en las parcelas vecinas más cercanas (Papadakis 1937), hasta el uso de modelos que contemplan las correlaciones espaciales en términos del error y que también producen ajustes de medias de tratamientos (Mead 1971, Besag 1974, 1977, Ripley 1981). Gilmour et ál. (1997) particionan la variabilidad espacial entre parcelas de un ensayo en variabilidad espacial local y global. La variabilidad espacial local hace referencia a las 125 Modelos Mixtos en InfoStat diferencias entre parcelas a pequeña escala, donde se contemplan las variaciones intrabloque. La tendencia espacial local y la heterogeneidad residual se modelan mediante la matriz de varianza y covarianza residual. A través de un sistema de coordenadas bidimensionales es posible definir la ubicación de las parcelas en el campo. La modelación de la estructura espacial de parcelas a partir de funciones de distancia puede realizarse en el contexto de los modelos lineales mixtos (Zimmerman y Harville 1991, Gilmour et ál. 1997, Cullis et ál. 1998), donde además de contemplar la estructura de correlación entre observaciones provenientes de distintas parcelas es posible modelar heterogeneidad de varianza residual. Esto es muy útil en los ensayos comparativos de rendimiento ya que estos se llevan a cabo en distintos ambientes. Si la correlación solo depende de la distancia (magnitud y/o dirección de las distancias), los modelos que estiman las covarianzas entre observaciones se denominan estacionarios. Las funciones de correlación para modelos estacionarios pueden ser isotrópicas o anisotrópicas. Las primeras son idénticas en cualquier dirección (sólo dependen de la magnitud de las distancias) mientras que las segundas permiten diferentes valores de sus parámetros en diferentes direcciones (i.e. dependen también de la dirección sobre la cual se calculan las distancias). Análisis de un ensayo comparativo de rendimientos en maní Para ejemplificar las alternativas de análisis usaremos los datos que se encuentran en el archivo ECRmaní.IDB2 y provienen de un año agrícola de un ensayo comparativo de rendimientos (ECR) de líneas experimentales (genotipos) de maní (Arachis hypogaea L.) del Programa de Mejoramiento de Maní de la EEA-Manfredi, INTA, Argentina. En cada campaña los ECR se realizaron en tres localidades del área de cultivo en la provincia de Córdoba: Manfredi, General Cabrera y Río Tercero. El conjunto de líneas evaluadas fue el mismo para cada localidad. En cada una de las tres localidades los ensayos fueron conducidos según un DBCA con cuatro repeticiones, registrándose los valores de rendimiento en grano (kg/parcela). Los datos de rendimiento fueron analizados usando distintas modificaciones del siguiente modelo: yijk i j k jk ik ijkl ; i 1,..,16; j 1,..., 4; k 1,...,3 (10) 126 Modelos Mixtos en InfoStat donde yijk representa la respuesta observada en i-ésimo nivel del factor genotipo, j-ésimo nivel de factor bloque, y k-ésimo nivel del factor localidad, representa la media general de la respuesta, i representa el efecto del i-ésimo nivel del factor genotipo, j representa el efecto del j-ésimo nivel del factor bloque, k el k-ésimo nivel del factor localidad y ik la interacción entre los factores genotipo y localidad, ik el efecto de bloque dentro de localidad, y ijkl representa el error experimental. La suposición usual es que ijkl ~ N 0, 2 . Excepto por ijk y los efectos de bloque (cuando son considerados aleatorios) en la mayoría de los casos, todos los factores del modelo serán considerados como de efectos fijos. Esto tiene la finalidad de restringir la comparación de los modelos a su estructura de parcelas. Las distintas estructuras de parcela inducen una estructura de correlación entre las observaciones que puede ser contemplada en el marco de los modelos mixtos, incluyendo técnicas de análisis para el control de la variabilidad espacial. Se usarán las siguientes estructuras de covarianza para los datos (covarianza marginal): 1. Modelo BF: Efecto de Bloques fijos, errores independientes y varianza entre localidades constante. 2. Modelo BA: Efecto de Bloques aleatorios, errores independientes y varianza entre localidades constante. 3. Modelo BFH: Efecto de Bloques fijos, errores independientes y varianzas diferentes entre localidades. 4. Modelo BAH: Efecto de Bloques aleatorios, errores independientes y varianzas diferentes entre localidades. 5. Modelo Exp: Correlación espacial exponencial sin efecto de bloques y varianza entre localidades constante. 6. Modelo BFExp: Correlación espacial exponencial, efecto de bloques fijos, y varianza entre localidades constante. 7. Modelo ExpH: Correlación espacial exponencial sin efecto de bloques y varianzas diferentes entre localidades. 8. Modelo Gau: Correlación espacial Gaussiana sin efecto de bloques y varianza entre localidades constante. 9. Modelo Esf: Correlación espacial esférico sin efecto de bloques y varianza entre localidades constante. En los dos primeros modelos los ijk se asumirán como independientes con varianza constante 2, i.e. se supone que no existe variación espacial local (intrabloque) y además existe homogeneidad de varianzas residuales entre localidades. Los efectos de 127 Modelos Mixtos en InfoStat bloque serán considerados fijos y aleatorios, denotando los procedimientos como Modelo BF y Modelo BA respectivamente. Los procedimientos denotados como Modelo BFH y Modelo BAH se basarán también en un modelo para un DBCA pero contemplando la posibilidad de varianzas residuales heterogéneas según los distintos niveles del factor localidad. El quinto procedimiento consistirá en ajustar para cada localidad un modelo de correlación espacial isotrópico con función de correlación potencia (Modelo Exp) sin declarar el efecto de bloques. Este modelo supone que la función exponencial no solo contemplará la variación intrabloque sino también la variación entre bloques. El sexto procedimiento fue igual al anterior pero agregando un efecto fijo de bloque (Modelo BFExp). El séptimo modelo consistió en un modelo como el Exp pero permitiendo la posibilidad de varianzas (y correlaciones) diferentes para cada localidad. Los dos últimos procedimiento consistirán en ajustar para cada localidad un modelo de correlación espacial isotrópico con función de correlación Gaussiana (Modelo Gau) y con función de correlación Esférica, sin declarar el efecto de bloques. En todos los casos se utilizó estimación REML. En el selector de variables se indica al rendimiento (Rendim) como dependiente y bloque, local y geno como clasificatorias. Para ajustar el Modelo BF, en la solapa de efectos fijos se deben declarar los efectos como se muestra en la Figura 88. No se declara nada en el resto de las solapas. Para ajustar el Modelo BA, en la solapa efectos fijos y efectos aleatorios debe declararse los factores como se presenta en la Figura 89 y Figura 90 respectivamente. No se declara nada en el resto de las solapas. 128 Modelos Mixtos en InfoStat Figura 88: Ventana con la solapa Efectos fijos desplegada para los datos del archivo ECRmaní.IDB2 y el Modelo BF. Figura 89: Ventana con la solapa Efectos fijos desplegada para los datos del archivo ECRmaní.IDB2 y el Modelo BA. 129 Modelos Mixtos en InfoStat Figura 90: Ventana con la solapa Efectos aleatorios desplegada para los datos del archivo ECRmaní.IDB2 y el Modelo BA. Los modelos BFH y BAH contemplan errores independientes y varianzas entre localidades diferentes. Para especificar estos modelos, se procede igual que en los dos casos anteriores (i.e. BF y BA) pero agregando una función varIdent en la solapa Heteroscedasticidad, indicando como criterio de agrupamiento a la localidad (local). Una vez declarada la función y el criterio de agrupamiento hacer clic en Agregar (Figura 91). 130 Modelos Mixtos en InfoStat Figura 91: Ventana con la solapa Heteroscedasticidad usando local como criterio de agrupamiento para los datos del archivo ECRmaní.IDB2 y los Modelos BFH y BAH. El quinto modelo no incluye el efecto de bloque y modela la variabilidad entre bloques e intra-bloque por medio de una función exponencial isotrópica (modelo Exp) con varianzas constantes entre localidades. Par usar la función exponencial deberemos agregar al modelo las variables que denotan las coordenadas espaciales. Para esto en el selector de variables debemos colocar las variables la y lon en Covariables. En la solapa Efectos fijos dejamos geno, local y geno*local y en la solapa Efectos aleatorios no se declara ningún factor. En la solapa Heteroscedasticidad no debe quedar ninguna función declarada. Para declarar la correlación espacial tipo exponencial, en la solapa Correlación se debe seleccionar la función correspondiente y declarar las coordenadas en X y en Y, y el criterio de agrupamiento, en este caso local, ya que hay un sistema de coordenadas dentro de cada localidad (Figura 92). 131 Modelos Mixtos en InfoStat Figura 92: Ventana con la solapa Correlación usando las variables la y lon como coordenadas en X e Y respectivamente y local como criterio de agrupamiento para los datos del archivo ECRmaní.IDB2 y los Modelos Exp y BFExp. El sexto modelo, Modelo BFExp, es igual que el anterior pero declarando los efectos de bloque dentro de localidad como fijos (como en la Figura 88). La inclusión de los bloques fijos restringe la modelación de la variación espacial únicamente a la variación dentro de bloque. La variación entre bloques está siendo contemplada, en un sentido clásico, por la inclusión de los bloques en la parte fija. Así, declarar como coordenadas del modelo de correlación espacial a la y lon, parece redundante ya que bastaría con declarar sólo lon (coordenada que varia dentro de bloque). Sin embargo para omitir la coordenada la sería necesario declara un nuevo criterio de estratificación consistente en la combinación de los niveles de local y bloque. Esta forma alternativa produce idénticos resultados a los mostrados en el modelo BFExp. El séptimo modelo, modelo ExpH, es como el modelo Exp pero permitiendo varianzas heterogéneas entre las localidades (como en la Figura 91). Los modelos Gau y Esf se ajustan al igual que el Exp sin el efecto de bloque, y como se muestra en la Figura 92, 132 Modelos Mixtos en InfoStat pero eligiendo la función de correlación espacial Gaussiana y esférica respectivamente. En la solapa Heteroscedasticidad no debe quedar nada declarado. A continuación se presentan las salidas con las medidas de ajuste de los diferentes modelos. Modelo BF Modelos lineales generales y mixtos Especificación del modelo en R modelo000_rendim_REML<-gls(rendim~1+local+geno+local:geno+local/bloque ,method="REML" ,na.action=na.omit ,data=R.data00) Resultados para el modelo: modelo000_rendim_REML Variable dependiente:rendim Medidas de ajuste del modelo N 192 AIC 299.71 BIC 468.22 logLik -91.86 Sigma R2_0 0.35 0.86 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) local geno local:geno local:bloque numDF F-value 1 8372.75 2 280.56 15 6.02 30 4.32 9 4.77 p-value <0.0001 <0.0001 <0.0001 <0.0001 <0.0001 Modelo BA Modelos lineales generales y mixtos Especificación del modelo en R modelo001_rendim_REML<-lme(rendim~1+local+geno+local:geno ,random=list(bloque_local=pdIdent(~1)) ,method="REML" ,na.action=na.omit ,data=R.data00 ,keep.data=FALSE) Resultados para el modelo: modelo001_rendim_REML Variable dependiente:rendim 133 Modelos Mixtos en InfoStat Medidas de ajuste del modelo N 192 AIC 283.41 BIC 431.90 logLik -91.71 Sigma R2_0 0.35 0.81 R2_1 0.86 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) local geno local:geno numDF denDF F-value 1 135 1754.21 2 9 58.78 15 135 6.02 30 135 4.32 p-value <0.0001 <0.0001 <0.0001 <0.0001 Parámetros de los efectos aleatorios Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~1|bloque_local Desvíos estándares relativos al residual y correlaciones (const) (const) 0.49 Modelo BFH Modelos lineales generales y mixtos Especificación del modelo en R modelo002_rendim_REML<-gls(rendim~1+local+geno+local:geno+local/bloque ,weight=varComb(varIdent(form=~1|local)) ,method="REML" ,na.action=na.omit ,data=R.data00) Resultados para el modelo: modelo002_rendim_REML Variable dependiente:rendim Medidas de ajuste del modelo N 192 AIC 303.44 BIC 477.75 logLik -91.72 Sigma R2_0 0.36 0.86 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales numDF F-value (Intercept) 1 8547.37 local 2 292.67 geno 15 6.02 local:geno 30 4.36 local:bloque 9 4.76 Estructura de varianzas p-value <0.0001 <0.0001 <0.0001 <0.0001 <0.0001 134 Modelos Mixtos en InfoStat Modelo de varianzas: varIdent Formula: ~ 1 | local Parámetros de la función de varianza Parámetro gralcabr manf rio3 Estim 1.00 0.92 0.96 Modelo BAH Modelos lineales generales y mixtos Especificación del modelo en R modelo003_rendim_REML<-lme(rendim~1+local+geno+local:geno ,random=list(bloque_local=pdIdent(~1)) ,weight=varComb(varIdent(form=~1|local)) ,method="REML" ,na.action=na.omit ,data=R.data00 ,keep.data=FALSE) Resultados para el modelo: modelo003_rendim_REML Variable dependiente:rendim Medidas de ajuste del modelo N 192 AIC 287.12 BIC 441.55 logLik -91.56 Sigma R2_0 0.36 0.81 R2_1 0.86 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) local geno local:geno numDF denDF F-value 1 135 1765.74 2 9 59.53 15 135 6.01 30 135 4.36 p-value <0.0001 <0.0001 <0.0001 <0.0001 Parámetros de los efectos aleatorios Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~1|bloque_local Desvíos estándares relativos al residual y correlaciones (const) (const) 0.46 Estructura de varianzas 135 Modelos Mixtos en InfoStat Modelo de varianzas: varIdent Formula: ~ 1 | local Parámetros de la función de varianza Parámetro gralcabr manf rio3 Estim 1.00 0.92 0.95 Modelo Exp Modelos lineales generales y mixtos Especificación del modelo en R modelo004_rendim_REML<-gls(rendim~1+geno+local+local:geno ,correlation=corExp(form=~as.numeric(as.character(la))+as.numeric(as.c haracter(lon))|local ,metric="euclidean" ,nugget=FALSE) ,method="REML" ,na.action=na.omit ,data=R.data05) Resultados para el modelo: modelo004_rendim_REML Variable dependiente:rendim Medidas de ajuste del modelo N 192 AIC 273.43 BIC 421.92 logLik -86.72 Sigma R2_0 0.39 0.81 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) geno local geno:local numDF F-value 1 1687.54 15 7.27 2 56.18 30 5.33 p-value <0.0001 <0.0001 <0.0001 <0.0001 Estructura de correlación Modelo de correlación: Exponential spatial correlation Formula: ~ as.numeric(as.character(la)) + as.numeric(as.character(lon)) | local Metrica: euclidean Parámetros del modelo Parámetro range Estim 0.96 136 Modelos Mixtos en InfoStat Modelo BFExp Modelos lineales generales y mixtos Especificación del modelo en R modelo005_rendim_REML<-gls(rendim~1+geno+local+local:geno+local/bloque ,correlation=corExp(form=~as.numeric(as.character(la))+as.numeric(as.c haracter(lon))|local ,metric="euclidean" ,nugget=FALSE) ,method="REML" ,na.action=na.omit ,data=R.data05) Resultados para el modelo: modelo005_rendim_REML Variable dependiente:rendim Medidas de ajuste del modelo N 192 AIC 284.85 BIC 456.26 logLik -83.42 Sigma R2_0 0.35 0.86 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) geno local geno:local local:bloque numDF F-value 1 2785.57 15 7.86 2 92.79 30 5.74 9 3.46 p-value <0.0001 <0.0001 <0.0001 <0.0001 0.0007 Estructura de correlación Modelo de correlación: Exponential spatial correlation Formula: ~ as.numeric(as.character(la)) + as.numeric(as.character(lon)) | local Metrica: euclidean Parámetros del modelo Parámetro range Estim 0.78 137 Modelos Mixtos en InfoStat Modelo ExpH Modelos lineales generales y mixtos Especificación del modelo en R modelo006_rendim_REML<-gls(rendim~1+geno+local+local:geno ,weight=varComb(varIdent(form=~1|local)) ,correlation=corExp(form=~as.numeric(as.character(la))+as.numeric(as.c haracter(lon))|local ,metric="euclidean" ,nugget=FALSE) ,method="REML" ,na.action=na.omit ,data=R.data05) Resultados para el modelo: modelo006_rendim_REML Variable dependiente:rendim Medidas de ajuste del modelo N 192 AIC 275.01 BIC 429.44 logLik -85.50 Sigma R2_0 0.43 0.81 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) geno local geno:local numDF F-value 1 1633.46 15 7.15 2 61.51 30 5.53 p-value <0.0001 <0.0001 <0.0001 <0.0001 Estructura de correlación Modelo de correlación: Exponential spatial correlation Formula: ~ as.numeric(as.character(la)) + as.numeric(as.character(lon)) | local Metrica: euclidean Parámetros del modelo Parámetro range Estim 0.99 Estructura de varianzas Modelo de varianzas: varIdent Formula: ~ 1 | local Parámetros de la función de varianza Parámetro gralcabr manf rio3 Estim 1.00 0.85 0.81 138 Modelos Mixtos en InfoStat Modelo Gau Modelos lineales generales y mixtos Especificación del modelo en R modelo007_rendim_REML<-gls(rendim~1+geno+local+local:geno ,correlation=corGaus(form=~as.numeric(as.character(la))+as.numeric(as. character(lon))|local ,metric="euclidean" ,nugget=FALSE) ,method="REML" ,na.action=na.omit ,data=R.data05) Resultados para el modelo: modelo007_rendim_REML Variable dependiente:rendim Medidas de ajuste del modelo N 192 AIC 277.81 BIC 426.30 logLik -88.90 Sigma R2_0 0.37 0.81 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) geno local geno:local numDF F-value 1 3399.06 15 7.36 2 113.57 30 4.97 p-value <0.0001 <0.0001 <0.0001 <0.0001 Estructura de correlación Modelo de correlación: Gaussian spatial correlation Formula: ~ as.numeric(as.character(la)) + as.numeric(as.character(lon)) | local Metrica: euclidean Parámetros del modelo Parámetro range Estim 0.87 139 Modelos Mixtos en InfoStat Modelo Esf Modelos lineales generales y mixtos Especificación del modelo en R modelo008_rendim_REML<-gls(rendim~1+geno+local+local:geno ,correlation=corSpher(form=~as.numeric(as.character(la))+as.numeric(as .character(lon))|local ,metric="euclidean" ,nugget=FALSE) ,method="REML" ,na.action=na.omit ,data=R.data05) Resultados para el modelo: modelo008_rendim_REML Variable dependiente:rendim Medidas de ajuste del modelo N 192 AIC 277.72 BIC 426.21 logLik -88.86 Sigma R2_0 0.38 0.81 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) geno local geno:local numDF F-value 1 3170.04 15 7.61 2 105.96 30 5.15 p-value <0.0001 <0.0001 <0.0001 <0.0001 Estructura de correlación Modelo de correlación: Spherical spatial correlation Formula: ~ as.numeric(as.character(la)) + as.numeric(as.character(lon)) | local Metrica: euclidean Parámetros del modelo Parámetro range Estim 1.91 Comparación de los modelos ajustados Debido a que los modelos ajustados tienen distintas componentes en su parte fija, se compararán por medio de los criterios AIC y BIC aquellos que comparten los mismos efectos fijos. En primer lugar se comparan entonces el BF, BFH y BFExp (Cuadro 3). 140 Modelos Mixtos en InfoStat Cuadro 3. Criterios de bondad de ajuste para los modelos ajustados con efectos de bloque fijo en los datos del archivo ECRmani.IDB2 Modelo AIC BIC BF 299.72 468.22 BFH 303.44 477.75 BFExp 284.85 456.26 Para este grupo de modelos que contemplan efecto de bloques fijos se puede ver que el modelo con bloques fijos más una función de correlación exponencial provee el mejor ajuste. Esto implica la existencia de una correlación intra-bloque que es removida por la función de correlación exponencial. También se puede observar que no hay una mejora en estos modelos al permitir varianzas heterogéneas entre localidades (BF respecto a BFH). Si se calculan las varianzas a partir de los coeficientes para las distintas localidades se puede ver que estas son realmente similares: Varianza de gralcabr = (1*0.36)2 = 0.129 Varianza de manf = (0.92*0.36)2 = 0.109 Varianza de rio3 = (0.96*0.36)2 = 0.119 Los restantes 6 modelos se pueden comparar entre sí ya que todos comparten los mismos efectos fijos, i.e. geno, local y geno*local (Cuadro 4). Cuadro 4. Criterios de bondad de ajuste para los modelos ajustados sin efectos de bloque fijo en los datos del archivo ECRmani.IDB2 Modelo AIC BIC BA 283.41 431.90 BAH 287.12 441.55 Exp 273.43 421.92 ExpH 275.01 429.44 Gau 277.81 426.30 Esf 277.72 426.21 141 Modelos Mixtos en InfoStat Dentro de los modelos que contemplan efectos de bloque aleatorio se puede ver nuevamente que al permitir varianzas heterogéneas entre localidades el modelo no mejora, ya que AIC y BIC son más pequeños en BA comparados con BAH. Lo mismo ocurre cuando sólo se modela la variabilidad espacial por medio de una función de correlación exponencial, ya que al permitir varianzas heterogéneas (ExpH) no se logra una mejoría respecto a Exp. Comparando distintos modelos de correlación espacial, no se encontraron diferencias importantes para AIC y BIC entre los modelos Gau y Esf, pero estos criterios tuvieron valores inferiores para la función de correlación espacial exponencial. Este último modelo fue el de mejor ajuste dentro de los modelos sin efecto de bloque fijo. Si bien el primer grupo de modelos (BF, BFH y BFExp) no son comparables por medio de AIC y BIC con este último grupo, el investigador deberá poder discernir si sus bloques deben ser considerados fijos o aleatorios. La elección de uno u otro grupo de modelos tendrá efecto sobre las inferencias que se realicen. Esto se visualiza fácilmente al ver que los errores estándar usados para las comparaciones de medias cambian entre los modelos. Una discusión más detallada sobre la elección de bloques fijos o aleatorios puede encontrarse en Casanoves et ál. (2007). En este ejemplo los mejores modelos dentro de cada grupo (i.e. BFExp y Exp para el primero y segundo grupo de modelos respectivamente) tienen la misma estructura de covarianza pero difieren en su parte fija: unos contienen el efecto de bloque y otros no. Para decidir cuál de los dos modelos es el que conviene, podemos realizar una prueba de cociente de verosimilitudes, usando las estimaciones por ML para los modelos con y sin efectos de bloque (recordemos que para comparar modelos con distintos efectos fijos se debe usar ML): Modelo con bloque (completo BFExp): Medidas de ajuste del modelo N 192 AIC 163.82 BIC 356.01 logLik -22.91 Sigma R2_0 0.29 0.86 logLik -41.43 Sigma R2_0 0.34 0.81 AIC y BIC menores implica mejor Modelo sin bloque (reducido Exp): Medidas de ajuste del modelo N 192 AIC 182.85 BIC 345.73 AIC y BIC menores implica mejor 142 Modelos Mixtos en InfoStat Así, el estadístico G 2 log lik completo 2 log lik reducido 2 (22.91 41.43) 37.04 con 9 grados de libertad, y un valor p<0.0001, por lo que podemos decir, con una significancia del 5%, que conviene dejar el efecto de bloques fijos y la función de correlación exponencial. La comparación se puede hacer manualmente, o utilizando el módulo Análisis exploratorio de un modelo estimado. Seleccionando la solapa, Modelos y tildando los modelos estimados correspondientes a BFExp y ExP, se obtienen la salida mostrada a continuación. Comparación de modelos modelo009_rendim_ML modelo010_rendim_ML Model 1 2 df 59 50 logLik -22.91 -41.43 Test 1 vs 2 L.Ratio p-value 37.04 <0.0001 A continuación se presenta la salida completa correspondiente al modelo BFExp. Las pruebas de hipótesis para la interacción entre genotipo y localidad son significativas (p<0.0001) por lo que la recomendación de un genotipo puede cambiar dependiendo de la localidad. Puede observarse que debido al ajuste de la función de correlación espacial los EE de los genotipos no son únicos. Las comparaciones múltiples presentadas se realizaron mediante la aplicación del procedimiento DGC (Di Rienzo et ál. 2002). Esta procedimiento fue adaptado para contemplar las particularidades de la estructura de correlación entre estimaciones emergente de los modelos mixtos. La aplicación de este procedimiento es recomendada por el gran número de medias a comparar, ya que asegura una interpretación más sencilla que la que puede obtenerse de la aplicación de un test tipo LSD de Fisher. Para hacer recomendaciones se pueden usar las comparaciones de medias de las combinaciones de localidades y genotipos como así también el grafico de interacción (Figura 93). Modelos lineales generales y mixtos Especificación del modelo en R modelo010_rendim_REML<-gls(rendim~1+local+geno+local:geno+local/bloque ,correlation=corExp(form=~as.numeric(as.character(la))+as.numeric(as.c haracter(lon))|local ,metric="euclidean" ,nugget=FALSE) ,method="REML" ,na.action=na.omit ,data=R.data03) 143 Modelos Mixtos en InfoStat Resultados para el modelo: modelo010_rendim_REML Variable dependiente:rendim Medidas de ajuste del modelo N 192 AIC 284.85 BIC 456.26 logLik -83.42 Sigma R2_0 0.35 0.86 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) local geno local:geno local:bloque numDF F-value 1 2785.57 2 92.79 15 7.86 30 5.74 9 3.46 p-value <0.0001 <0.0001 <0.0001 <0.0001 0.0007 Estructura de correlación Modelo de correlación: Exponential spatial correlation Formula: ~ as.numeric(as.character(la)) + as.numeric(as.character(lon)) | local Metrica: euclidean Parámetros del modelo Parámetro range Estim 0.78 Medias ajustadas y errores estándares para local DGC (alfa=0.05) local manf gralcabr rio3 Medias 3.00 2.27 1.56 E.E. 0.08 0.08 0.08 A B C Letras distintas indican diferencias significativas(p<= 0.05) Medias ajustadas y errores estándares para geno DGC (alfa=0.05) geno mf435 mf407 mf429 mf415 mf420 mf421 mf431 mf405 manf68 mf408 manf393 colirrad mf404 mf433 mf432 mf410 Medias 2.73 2.59 2.51 2.49 2.38 2.36 2.34 2.31 2.24 2.22 2.22 2.21 2.14 1.96 1.96 1.78 E.E. 0.10 0.10 0.10 0.10 0.10 0.10 0.10 0.10 0.10 0.10 0.10 0.10 0.10 0.10 0.10 0.10 A A A A B B B B B B B B B C C C Letras distintas indican diferencias significativas(p<= 0.05) 144 Modelos Mixtos en InfoStat Medias ajustadas y errores estándares para local*geno DGC (alfa=0.05) local manf manf manf manf manf manf manf manf manf manf gralcabr manf manf gralcabr gralcabr manf gralcabr manf gralcabr manf gralcabr gralcabr gralcabr gralcabr gralcabr gralcabr gralcabr rio3 rio3 manf rio3 gralcabr rio3 rio3 gralcabr gralcabr rio3 rio3 rio3 gralcabr rio3 rio3 rio3 rio3 rio3 rio3 rio3 rio3 geno mf407 mf421 mf405 mf431 mf435 manf68 mf420 mf429 colirrad manf393 mf435 mf408 mf415 mf420 mf404 mf433 mf415 mf410 mf429 mf432 mf421 mf408 manf393 mf407 mf405 mf431 manf68 mf435 mf415 mf404 mf429 colirrad mf432 mf407 mf410 mf433 mf404 mf431 colirrad mf432 mf433 manf68 mf408 manf393 mf405 mf420 mf421 mf410 Medias 3.67 3.54 3.38 3.28 3.24 3.23 3.17 3.08 3.05 3.02 2.96 2.90 2.90 2.82 2.71 2.64 2.61 2.53 2.52 2.48 2.42 2.32 2.30 2.30 2.25 2.05 2.04 1.99 1.98 1.97 1.93 1.92 1.89 1.81 1.79 1.77 1.74 1.70 1.64 1.50 1.47 1.45 1.44 1.33 1.32 1.16 1.14 1.02 E.E. 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 0.17 A A B B B B B B B B B B B B C C C C C C C C C C C D D D D D D D D D D D D D D E E E E E E E E E Letras distintas indican diferencias significativas(p<= 0.05) 145 Modelos Mixtos en InfoStat Rendimiento (kg/parcela) 4 3 2 1 mf407 mf421 mf405 mf435 manf68 mf431 mf420 colirrad mf429 mf408 manf393 mf415 mf433 mf410 mf432 mf404 0 Genotipo Manfredi General Cabrera Río Tercero Figura 93: Diagrama de puntos para estudiar la interacción entre localidades y genotipos para la variable rendimiento. 146 Modelos Mixtos en InfoStat Aplicaciones de modelos mixtos en otros diseños experimentales Diseño en franjas (strip-plot) El diseño strip-plot es un resultado de las restricciones a la aleatorización. Al igual que en el diseño en parcelas divididas, el strip-plot es un resultado de cómo fue llevado a cabo un experimento que involucra dos o más factores. Estos factores (o sus combinaciones) se aplican en diferentes etapas, generalmente 2, y las restricciones a la aleatorización producen las unidades experimentales de diferentes tamaños y por ende diferentes términos de error para cada una de los factores o sus combinaciones (Milliken y Johnson 1984). Consideremos un ejemplo donde se desean evaluar tres niveles de fertilización con N (0, 50 y 100 kg N/ha) y dos niveles de riego (bajo y alto) sobre los rendimientos de maíz. El ensayo se condujo bajo un diseño en bloques completos al azar con cuatro bloques (datos: StripPlot.IDB2). Debido a restricciones de la aplicación de los tratamientos, en una primera etapa, en cada uno de los bloques, se aleatorizan los tres niveles de nitrógeno y en la segunda etapa, en cada bloque y en sentido trasversal al sentido de aplicación de los niveles de nitrógeno, se aleatorizan los niveles del factor riego. Si bien en el siguiente esquema (Figura 94).se presenta la aleatorización dentro de un bloque en particular, el experimento ha sido repetido en bloques, esquema necesario para poder obtener los distintos términos de error y que el modelo resultante tenga sentido. Si en cada etapa del diseño hubiera más de un factor, y estos no interactuaran entre sí, se podrían usar las interacciones de más alto orden como términos de error y así poder obtener las pruebas F sin necesidad de repeticiones. Modelos Mixtos en InfoStat Etapa 1 100 kg N/ha 0 kg N/ha 50 kg N/ha Etapa 2 Riego alto Riego bajo Figura 94: Esquema de un experimento conducido bajo un diseño strip-plot repetido en bloques completos al azar, con la aleatorización para un bloque particular de los factores cantidad de nitrógeno y cantidad de riego. Datos del archivo StripPlot.IDB2. Los datos de rendimiento se analizaron usando el siguiente modelo: yijk i j k ik jk ij ijk ; i 1,..,3; j 1, 2; k 1,..., 4 (11) donde yijk representa la respuesta observada en el i-ésimo nivel del factor nitrógeno, j-ésimo nivel de factor riego y k-ésimo nivel del factor bloque, representa la media general de la respuesta, i representa el efecto del i-ésimo nivel del factor nitrógeno, j representa el efecto del j-ésimo nivel del factor riego, k el k-ésimo nivel del factor 148 Modelos Mixtos en InfoStat bloque, ik el efecto del bloque k en el nivel i de nitrógeno (efecto aleatorio con el que se construye el término de error para nitrógeno), jk el efecto del bloque k en el nivel j de riego (término de error para riego), ij la interacción entre los factores nitrógeno y riego, y ijk representa el error residual (término de error para nitrógeno×riego). La suposición usual es que ik ~ N 0, 2 , jk ~ N 0, 2 y ijkl ~ N 0, 2 , siendo todos independientes. Este modelo puede ajustarse en InfoStat en el menú Análisis de varianza, de efectos fijos, declarando al rendimiento como variable dependiente y a riego, nitrógeno y bloque como variables clasificatorias. Luego, en la solapa Modelo se declaran los siguientes factores indicando su correspondiente término de error (Figura 95). Figura 95: Ventana del procedimiento de Análisis de varianza con la solapa Modelo desplegada para los datos del archivo StripPlot.IDB2. A continuación se presenta la salida correspondiente al análisis de la varianza tradicional. 149 Modelos Mixtos en InfoStat Análisis de la varianza Variable N Rendimiento 24 R² 0.99 R² Aj CV 0.97 1.65 Cuadro de Análisis de la Varianza (SC tipo III) F.V. SC gl CM F Modelo 1177.38 17 69.26 48.41 Bloque 123.46 3 41.15 28.77 Nitrogeno 339.08 2 169.54 60.13 Bloque*Nitrogeno 16.92 6 2.82 1.97 Riego 570.38 1 570.38 52.18 Bloque*Riego 32.79 3 10.93 7.64 Nitrogeno*Riego 94.75 2 47.38 33.12 Error 8.58 6 1.43 Total 1185.96 23 Test:LSD Fisher Alfa=0.05 DMS=2.05433 Error: 2.8194 gl: 6 Nitrogeno Medias n 0 68.25 8 A 50 71.75 8 B 100 77.38 8 p-valor (Error) 0.0001 0.0006 0.0001 (Bloque*Nitrogeno) 0.2147 0.0055 (Bloque*Riego) 0.0179 0.0006 C Letras distintas indican diferencias significativas(p<= 0.05) Test:LSD Fisher Alfa=0.05 DMS=4.29543 Error: 10.9306 gl: 3 Riego Medias n Bajo 67.58 12 A Alto 77.33 12 B Letras distintas indican diferencias significativas(p<= 0.05) Test:LSD Fisher Alfa=0.05 DMS=2.06945 Error: 1.4306 gl: 6 Nitrogeno Riego Medias n 0 Bajo 61.50 4 A 50 Bajo 66.00 4 0 Alto 75.00 4 100 Bajo 75.25 4 50 Alto 77.50 4 100 Alto 79.50 4 B C C D D Letras distintas indican diferencias significativas(p<= 0.05) Se encontró efecto de nitrógeno (p=0.0001), de riego (p=0.0055) y también interacción nitrogeno×riego (p=0.0006). Para estudiar la interacción se realizó un gráfico de puntos (Figura 96). 150 Modelos Mixtos en InfoStat 80 Rendimiento 75 70 Riego Alto Riego Bajo 65 60 55 0 50 100 Nitrogeno Figura 96: Diagramas de puntos para estudiar la interacción entre Riego y Nitrógeno y su efecto sobre el rendimiento. Datos archivo StripPlot.IDB2. A partir del gráfico de interacción y de las medias de los tratamientos se recomienda el riego alto con fertilización de nitrógeno de 50 kg, ya que esta combinación no difiere estadísticamente de 100 kg de nitrógeno con riego alto y ambas difieren del resto. Este ensayo pudo evaluarse bajo la óptica de los modelos lineales fijos debido a que está completamente balanceado. Estos datos también pueden analizarse como modelos mixtos como se detalla a continuación. Si hubiera existido desbalance o si los bloques hubiesen sido aleatorios, este enfoque es el único válido. Este modelo puede ajustarse en InfoStat en el menú Modelos lineales generales y mixtos, declarando a rendimiento como variable dependiente y a riego, nitrógeno y bloque como variables clasificatorias. Luego, en la solapa Efectos fijos se declaran los siguientes términos (Figura 97). 151 Modelos Mixtos en InfoStat Figura 97: Ventana con la solapa Efectos fijos desplegada para evaluar un modelo mixto con los datos del archivo StripPlot.IDB2 . En la solapa Efectos aleatorios se debe declarar el efecto de bloque tanto en la constante (k ) como en los factores fijos nitrógeno y riego ( ik y jk respectivamente) (Figura 98). 152 Modelos Mixtos en InfoStat Figura 98: Ventana con la solapa Efectos aleatorios desplegada para evaluar un modelo mixto con los datos del archivo StripPlot.IDB2 . Modelos lineales generales y mixtos Especificación del modelo en R modelo000_Rendimiento_REML<lme(Rendimiento~1+Nitrogeno+Riego+Nitrogeno:Riego ,random=list(Bloque=pdIdent(~1) ,Bloque=pdIdent(~Nitrogeno-1) ,Bloque=pdIdent(~Riego-1)) ,method="REML" ,na.action=na.omit ,data=R.data00 ,keep.data=FALSE) Resultados para el modelo: modelo000_Rendimiento_REML Variable dependiente:Rendimiento Medidas de ajuste del modelo N 24 AIC 106.09 BIC 115.00 logLik -43.05 Sigma R2_0 1.20 0.85 R2_1 0.94 R2_2 0.95 R2_3 0.99 AIC y BIC menores implica mejor 153 Modelos Mixtos en InfoStat Pruebas de hipótesis secuenciales (Intercept) Nitrogeno Riego Nitrogeno:Riego numDF denDF F-value 1 15 3061.88 2 15 60.13 1 15 52.18 2 15 33.12 p-value <0.0001 <0.0001 <0.0001 <0.0001 Parámetros de los efectos aleatorios Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~1|Bloque Desvíos estándares relativos al residual y correlaciones (const) (const) 1.83 Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~Nitrogeno - 1|Bloque Desvíos estándares relativos al residual y correlaciones 0 100 50 0 0.70 0.00 0.00 100 0.00 0.70 0.00 50 0.00 0.00 0.70 Modelo de covarianzas de los efectos aleatorios: pdIdent Formula: ~Riego - 1|Bloque Desvíos estándares relativos al residual y correlaciones Alto Bajo Alto 1.49 0.00 Bajo 0.00 1.49 Medias ajustadas y errores estándares para Nitrogeno LSD Fisher (alfa=0.05) Procedimiento de correccion de p-valores: No Nitrogeno 100 50 0 Medias 77.38 71.75 68.25 E.E. 1.40 1.40 1.40 A B C Letras distintas indican diferencias significativas(p<= 0.05) Medias ajustadas y errores estándares para Riego LSD Fisher (alfa=0.05) Procedimiento de correccion de p-valores: No Riego Medias Alto 77.33 Bajo 67.58 E.E. 1.47 1.47 A B Letras distintas indican diferencias significativas(p<= 0.05) 154 Modelos Mixtos en InfoStat Medias ajustadas y errores estándares para Nitrogeno*Riego LSD Fisher (alfa=0.05) Procedimiento de correccion de p-valores: No Nitrogeno 100 50 100 0 50 0 Riego Medias Alto 79.50 Alto 77.50 Bajo 75.25 Alto 75.00 Bajo 66.00 Bajo 61.50 E.E. 1.59 1.59 1.59 1.59 1.59 1.59 A A B B C C D E Letras distintas indican diferencias significativas(p<= 0.05) Podemos observar que bajo el enfoque de modelos mixtos en este experimento se llega a las mismas conclusiones que en el enfoque clásico y que los estadísticos F para las pruebas de efectos fijos son los mismos, pero los grados de libertad del denominador son diferentes. Debemos destacar que los modelos no son absolutamente equivalentes, ya que en el enfoque de modelos mixtos los bloques son aleatorios y en el clásico son fijos; y que las fórmulas para el cálculo de los grados de libertad del denominador en los estadísticos F son diferentes. 155 Modelos Mixtos en InfoStat Diseño experimental con dos factores y dependencia espacial En muchas situaciones se presentan niveles de un factor de interés que, por su naturaleza, no se pueden asignar en forma aleatoria. Este es el caso de las tomas de muestras de agua a lo largo de un río, cuando se evalúan efectos a distintas distancias en un bosque o cuando se toman muestras de suelo a distintas profundidades. El hecho de que no se puedan aleatorizar los niveles de un factor genera una dependencia espacial que debe ser contemplada. Aquí presentamos un ejemplo (datos Lombrices.IDB2) en donde se evalúan cuatro tipo de sombra en cultivos de café: testigo con sol (sol), leguminosa1 (SombraL1), leguminosa2 (SombraL2) y no leguminosa (SombraNL) en tres profundidades (1=0-10 cm, 2=10-20 cm y 3=20-30 cm). En cada una de las unidades experimentales (combinación de tratamientos y repeticiones) se tomaron muestras de 30×30 cm con 10 cm de profundidad en cada una de las tres profundidades. En cada muestra se recolectaron las lombrices y se obtuvo su peso vivo (biomasa). Las unidades experimentales estaban arregladas en un diseño completamente aleatorizado con tres repeticiones. La variable tratam_rep identifica a las unidades experimentales sobre las que se miden las distintas profundidades y fue generada desde el menú Datos, sub menú Cruzar categorias para formar una nueva variable (en la ventana de selección de variables se declaró a tratam y rep como variables). Para realizar el análisis de los datos del archivo Lombrices.IDB2, se deben declarar las variables como se muestra a continuación (Figura 99). 156 Modelos Mixtos en InfoStat Figura 99: Ventana se selector de variable para Modelos lineales generales y mixtos los datos del archivo Lombrices.IDB2. Luego, en la solapa Efectos fijos se deben declarar las variables como se muestra en la siguiente figura (Figura 100). 157 Modelos Mixtos en InfoStat Figura 100: Ventana con la solapa Efectos fijos desplegada para evaluar un modelo mixto con los datos del archivo Lombrices.IDB2. Por último, se declara en la solapa Correlación el modelo de Correlación espacial exponencial, identificando a profund como coordenada X y a tratam_rep como criterio de agrupamiento (Figura 101). 158 Modelos Mixtos en InfoStat Figura 101: Ventana con la solapa Correlación desplegada para evaluar un modelo mixto con correlación espacial exponencial en los datos del archivo Lombrices.IDB2. La salida correspondiente se presenta a continuación. Modelos lineales generales y mixtos Especificación del modelo en R modelo000_Biomasa_REML<-gls(Biomasa~1+Tratam+Profund+Tratam:Profund ,correlation=corExp(form=~as.numeric(as.character(Profund))|Tratam_Rep ,metric="euclidean" ,nugget=FALSE) ,method="REML" ,na.action=na.omit ,data=R.data00) Resultados para el modelo: modelo000_Biomasa_REML Variable dependiente:Biomasa 159 Modelos Mixtos en InfoStat Medidas de ajuste del modelo N 36 AIC 161.03 BIC 177.52 logLik -66.52 Sigma R2_0 3.46 0.97 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) Tratam Profund Tratam:Profund numDF F-value 1 3725.04 3 66.75 2 303.14 6 4.86 p-value <0.0001 <0.0001 <0.0001 0.0022 Estructura de correlación Modelo de correlación: Exponential spatial correlation Formula: ~ as.numeric(as.character(Profund)) | Tratam_Rep Metrica: euclidean Parámetros del modelo Parámetro range Estim 2.12 Todos los factores resultaron significativos, presentándose interacción entre tratamientos y profundidad (p=0.0022). El parámetro range tiene un valor estimado de 2.12. Este parámetro debe interpretarse con cuidado, dependiendo del modelo de correlación espacial usado. En la bibliografía geoestadística, el range se define, para procesos espaciales estacionales de segundo orden, como la distancia a partir de la cual las observaciones pueden considerarse independientes. El parámetro range que se muestra en la salida está relacionado a esta definición, pero no es la distancia a partir de la cual no hay más correlación (excepto en los modelos esférico y lineal). En los modelos de correlación espacial, en los que la covarianza alcanza cero solo asintóticamente (todos excepto el esférico y el lineal), no existe una distancia a la cual la correlación espacial se haga 0, por lo que se usa el concepto de practical range (distancia a partir de la cual la covarianza espacial se reduce al 5%, o equivalentemente, la distancia a la cual el semivariograma alcanza el 95% de su máximo). Esta distancia depende del modelo usado: para correlación espacial exponencial es 3 veces el range estimado, mientras que para correlación espacial Gaussiana es √3 veces el range estimado (Littel et ál. 2006). Para la correlación racional cuadrática este factor es aproximadamente 4.36. 160 Modelos Mixtos en InfoStat En este ejemplo se usó un modelo de correlación espacial exponencial. La profundidad 1 era entre 0 y 10 cm, la 2 entre 10 y 20 cm y la 3 entre 20 y 30 cm, es decir, la diferencia entre la profundidad 1 y 2 de la forma en que fueron declaradas, es de 1, sin embargo en la escala original esta diferencia es de 10. Por lo tanto, el practical range en la escala original es de 3×21.2 cm=63.6 cm. Esto implica que, para las profundidades estudiadas (0 a 30 cm), las observaciones de biomasa de lombrices nunca serán independientes (para que pudieran considerarse prácticamente independientes las observaciones deberían estar a más de 63.6 cm, lo que es imposible con estos datos). El modelo de correlación espacial exponencial isotrópico presentado aquí es equivalente a un modelo autorregresivo de orden 1 (Casanoves et ál. 2005). Si con este mismo conjunto de datos usamos ahora un modelo Autorregresivo de orden 1 (Figura 102) se obtiene la siguiente salida. Figura 102: Ventana con la solapa Correlación desplegada para evaluar un modelo mixto con correlación autorregresiva de orden 1 en los datos del archivo Lombrices.IDB2. 161 Modelos Mixtos en InfoStat Modelos lineales generales y mixtos Especificación del modelo en R modelo001_Biomasa_REML<-gls(Biomasa~1+Tratam+Profund+Tratam:Profund ,correlation=corAR1(form=~as.integer(as.character(Profund))|Tratam_Rep ) ,method="REML" ,na.action=na.omit ,data=R.data00) Resultados para el modelo: modelo001_Biomasa_REML Variable dependiente:Biomasa Medidas de ajuste del modelo N 36 AIC 161.03 BIC 177.52 logLik -66.52 Sigma R2_0 3.46 0.97 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) Tratam Profund Tratam:Profund numDF F-value 1 3725.05 3 66.75 2 303.14 6 4.86 p-value <0.0001 <0.0001 <0.0001 0.0022 Estructura de correlación Modelo de correlación: AR(1) Formula: ~ as.integer(as.character(Profund)) | Tratam_Rep Parámetros del modelo Parámetro Phi Estim 0.62 La única diferencia entre esta salida y la anterior es que en ésta se muestra el parámetro Phi de correlación (0.62) en vez del parámetro range. A continuación estudiaremos la validez de los supuestos de este modelo. Para esto, en el submenú Análisis-exploración de los modelos estimados se solicitaron los gráficos de diagnóstico que se presentan a continuación (Figura 103). 162 1.5 0.5 -1.5 -0.5 Res.cond.estand.Pearson 1.5 0.5 -0.5 -1.5 Res.cond.estand.Pearson Modelos Mixtos en InfoStat sol sombraL1 sombraNL 1 2 1.5 0.5 -0.5 -1.5 -0.5 0.5 Cuantiles muestrales 1.5 Profund -1.5 Res.cond.estand.Pearson Tratam 3 30 40 50 60 Valores ajustados 70 80 -2 -1 0 1 2 Cuantiles teóricos Figura 103: Herramientas gráficas para diagnóstico obtenidas para los datos del archivo Lombrices.IDB2. Como se puede observar la variabilidad de los residuos bajo los distintos tratamientos parece diferente. Para evaluar un modelo heteroscedástico por tratamientos, en la solapa Heteroscedasticidad se declararon las variables como en la (Figura 104) y se obtuvo la siguiente salida. 163 Modelos Mixtos en InfoStat Figura 104: Ventana con la solapa Heteroscedasticidad desplegada para evaluar un modelo mixto con en los datos del archivo Lombrices.IDB2. Modelos lineales generales y mixtos Especificación del modelo en R modelo002_Biomasa_REML<-gls(Biomasa~1+Tratam+Profund+Tratam:Profund ,weight=varComb(varIdent(form=~1|Tratam)) ,correlation=corAR1(form=~as.integer(as.character(Profund))|Tratam_Rep ) ,method="REML" ,na.action=na.omit ,data=R.data00) Resultados para el modelo: modelo002_Biomasa_REML Variable dependiente:Biomasa 164 Modelos Mixtos en InfoStat Medidas de ajuste del modelo N 36 AIC 164.03 BIC 184.06 logLik -65.02 Sigma R2_0 4.20 0.97 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) Tratam Profund Tratam:Profund numDF F-value 1 4300.37 3 54.19 2 511.72 6 6.32 p-value <0.0001 <0.0001 <0.0001 0.0004 Estructura de correlación Modelo de correlación: AR(1) Formula: ~ as.integer(as.character(Profund)) | Tratam_Rep Parámetros del modelo Parámetro Phi Estim 0.73 Estructura de varianzas Modelo de varianzas: varIdent Formula: ~ 1 | Tratam Parámetros de la función de varianza Parámetro sol sombraL1 sombraL2 sombraNL Estim 1.00 0.65 0.66 1.22 Los criterios AIC y BIC son mayores en el modelo heteroscedástico que en el homoscedástico, indicando que este último es el mejor. Similar conclusión se obtiene a partir de la prueba del cociente de verosimilitud (p=0.3916) al pedir la comparación de los modelos como se mostró en la sección Análisis de un modelo ajustado. Comparación de modelos df 14 AIC 161.03 BIC 177.52 logLik -66.52 Test modelo001_Biomasa_REML modelo002_Biomasa_REML 17 164.03 184.06 -65.02 1 vs 2 L.Ratio p-value 3.00 0.3916 Por este motivo, nos quedamos con el modelo homoscedástico y, debido a la presencia de interacción entre los dos factores, se realiza un diagrama de puntos para visualizar el comportamiento de las medias de biomasa de lombrices (Figura 105). 165 Modelos Mixtos en InfoStat 90 80 Biomasa 70 60 50 40 30 20 1 2 3 Profundidad Sol SombraL1 SombraL2 SombraNL Figura 105: Diagramas de puntos para estudiar la interacción entre tratamientos y profundidad y su efecto sobre la biomasa. Datos archivo Lombrices.IDB2. Como se puede observar, este gráfico sugiere la presencia de un comportamiento lineal para sol y uno cuadrático para los otros tratamientos. Para probar estas hipótesis se realizan contrastes ortogonales polinómicos a partir de la solapa Comparaciones, subsolapa Contrastes (Figura 106). 166 Modelos Mixtos en InfoStat Figura 106: Ventana con la solapa Comparaciones y la subsolapa Contrastes desplegada para evaluar un modelo mixto con los datos del archivo Lombrices.IDB2. A continuación se muestran los resultados de los contrastes. Se puede ver que el único tratamiento que presenta solo tendencia lineal y no cuadrática es el de sol (p<0.0001 y p=0.8147 respectivamente). El resto de los tratamientos, además de la tendencia lineal, presentan una tendencia cuadrática. Pruebas de hipótesis para contrastes Tratam*Profund Cont.1 Cont.2 Cont.3 Cont.4 Cont.5 Cont.6 Cont.7 Cont.8 Total F 111.81 0.06 222.11 26.66 164.40 10.52 92.62 7.26 79.43 gl(num) 1 1 1 1 1 1 1 1 8 gl(den) 24 24 24 24 24 24 24 24 24 p-valor <0.0001 0.8147 <0.0001 <0.0001 <0.0001 0.0035 <0.0001 0.0127 <0.0001 167 Modelos Mixtos en InfoStat Coeficientes de los contrastes Tratam sol sol sol sombraL1 sombraL1 sombraL1 sombraL2 sombraL2 sombraL2 sombraNL sombraNL sombraNL Profund 1 2 3 1 2 3 1 2 3 1 2 3 Cont.1 Cont.2 Cont.3 Cont.4 Cont.5 Cont.6 Cont.7 Cont.8 -1.00 1.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 -2.00 0.00 0.00 0.00 0.00 0.00 0.00 1.00 1.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 -1.00 1.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 -2.00 0.00 0.00 0.00 0.00 0.00 0.00 1.00 1.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 -1.00 1.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 -2.00 0.00 0.00 0.00 0.00 0.00 0.00 1.00 1.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 -1.00 1.00 0.00 0.00 0.00 0.00 0.00 0.00 0.00 -2.00 0.00 0.00 0.00 0.00 0.00 0.00 1.00 1.00 168 Modelos Mixtos en InfoStat Diseños de testigos apareados Este tipo de arreglo de tratamientos es común en la evaluación de nuevos cultivares (variedades, híbridos, etc.) en mejoramiento genético vegetal. Básicamente consisten en ubicar en forma aleatoria el conjunto de cultivares a evaluar intercalando siempre entre ellos un testigo común. La presencia de este testigo es la que permite de alguna forma modelar los efectos sistemáticos de la calidad del terreno donde se ubican las parcelas experimentales. Para ejemplificar su análisis se presenta un ejemplo con 16 híbridos (H1,…, H16) y un testigo, y así se tiene un total de 32 unidades experimentales. Los datos se encuentran en el archivo Testigos apareados.IDB2. Una alternativa básica y muy poco eficiente para analizar estos datos es realizar un ANOVA a una vía de clasificación, y comparar los tratamientos usando una estimación del término de error a partir de la varianza entre los testigos (únicos niveles del factor tratamiento que están repetidos). Este modelo es incapaz de contemplar los sesgos producidos por las diferencias sistemáticas entre unidades experimentales. Para obtener este modelo, se declara en el selector de variables a Rendimiento como variable dependiente y a Hibrido como variable de clasificación. En la solapa de Efectos fijos se declara al Hibrido como en la Figura 107. Luego, en la solapa Comparaciones se solicitó la prueba LSD de Fisher para Hibrido. 169 Modelos Mixtos en InfoStat Figura 107: Ventana con la solapa Efectos fijos desplegada para los datos del archivo Testigos_apareados.IDB2. La salida correspondiente se presenta a continuación. Modelos lineales generales y mixtos Especificación del modelo en R modelo000_Rendimiento_REML<-gls(Rendimiento~1+Hibrido ,method="REML" ,na.action=na.omit ,data=R.data21) Resultados para el modelo: modelo000_Rendimiento_REML Variable dependiente:Rendimiento Medidas de ajuste del modelo N 32 AIC 219.90 BIC 232.64 logLik -91.95 Sigma 101.35 R2_0 0.69 AIC y BIC menores implica mejor 170 Modelos Mixtos en InfoStat Pruebas de hipótesis secuenciales (Intercept) Hibrido numDF F-value 1 3580.56 16 2.12 p-value <0.0001 0.0763 Medias ajustadas y errores estándares para Hibrido LSD Fisher (alfa=0.05) Procedimiento de correccion de p-valores: No Hibrido H4 H3 H14 H10 H11 Testigo H5 H9 H2 H12 H8 H7 H16 H6 H1 H13 H15 Medias 1230.00 1222.00 1193.00 1168.00 1116.00 1115.81 1099.00 1063.00 1037.00 1033.00 975.00 966.00 928.00 907.00 886.00 876.00 756.00 E.E. 101.35 101.35 101.35 101.35 101.35 25.34 101.35 101.35 101.35 101.35 101.35 101.35 101.35 101.35 101.35 101.35 101.35 A A A A A A A A A A A A A B B B B B B B B B B B B C C C C C C C C C C C C C D D D D D D D D D Letras distintas indican diferencias significativas(p<= 0.05) La prueba F para Hibrido no resultó significativa (p = 0.0763) por lo cual no deben interpretarse las diferencias de medias presentadas en la prueba LSD de Fisher. La alternativa a este modelo es el uso de correlaciones espaciales para corregir las medias de cada híbrido por el “efecto del sitio” en donde fueron ubicadas por azar. Para esto, se procede a colocar la Posicion de la parcela como una covariable. En la solapa Efectos fijos se deja igual que en la Figura 107. En la solapa Correlación se especifican los diferentes modelos: Modelo 1: Correlación espacial exponencial ( Figura 108). Modelo 2: Correlación espacial Gaussiana (Figura 109). Modelo 3: Correlación espacial lineal (Figura 110). Modelo 4: Correlación espacial “rational quadratic” (Figura 111). Modelo 5: Correlación espacial esférica (Figura 112). 171 Modelos Mixtos en InfoStat A continuación se muestran las ventanas de selección de correlación espacial y las medidas de ajuste de cada uno de los modelos estimados. Figura 108: Ventana con la solapa Correlación desplegada para los datos del archivo Testigos_apareados.IDB2 y selección de Correlación espacial exponencial. Medidas de ajuste del modelo N 32 AIC 218.62 BIC 232.08 logLik -90.31 Sigma 112.79 R2_0 0.58 AIC y BIC menores implica mejor 172 Modelos Mixtos en InfoStat Figura 109: Ventana con la solapa Correlación desplegada para los datos del archivo Testigos_apareados.IDB2 y selección de Correlación espacial Gaussiana. Medidas de ajuste del modelo N 32 AIC 219.17 BIC 232.62 logLik -90.58 Sigma 106.78 R2_0 0.58 AIC y BIC menores implica mejor 173 Modelos Mixtos en InfoStat Figura 110: Ventana con la solapa Correlación desplegada para los datos del archivo Testigos_apareados.IDB2 y selección de Correlación espacial lineal. Medidas de ajuste del modelo N 32 AIC 219.13 BIC 232.58 logLik -90.56 Sigma 107.52 R2_0 0.56 AIC y BIC menores implica mejor 174 Modelos Mixtos en InfoStat Figura 111: Ventana con la solapa Correlación desplegada para los datos del archivo Testigos_apareados.IDB2 y selección de Correlación espacial “rational quadratic”. Medidas de ajuste del modelo N 32 AIC 218.81 BIC 232.26 logLik -90.40 Sigma 106.92 R2_0 0.59 AIC y BIC menores implica mejor 175 Modelos Mixtos en InfoStat Figura 112: Ventana con la solapa Correlación desplegada para los datos del archivo Testigos_apareados.IDB2 y selección de Correlación espacial esférica. Medidas de ajuste del modelo N 32 AIC 219.21 BIC 232.66 logLik -90.60 Sigma 137.39 R2_0 0.56 AIC y BIC menores implica mejor Todos los modelos ajustan bien, ya que sus valores de AIC y BIC son muy parecidos. El modelo con menores valores es el de Correlación espacial exponencial (AIC=218.62, BIC=232.08). La salida correspondiente a este modelo se presenta a continuación. 176 Modelos Mixtos en InfoStat Modelos lineales generales y mixtos Especificación del modelo en R modelo028_Rendimiento_REML<-gls(Rendimiento~1+Hibrido ,correlation=corExp(form=~as.numeric(as.character(Posicion)) ,metric="euclidean" ,nugget=FALSE) ,method="REML" ,na.action=na.omit ,data=R.data28) Resultados para el modelo: modelo028_Rendimiento_REML Variable dependiente:Rendimiento Medidas de ajuste del modelo N 32 AIC 218.62 BIC 232.08 logLik -90.31 Sigma 112.79 R2_0 0.58 AIC y BIC menores implica mejor Pruebas de hipótesis secuenciales (Intercept) Hibrido numDF F-value 1 582.79 16 5.27 p-value <0.0001 0.0012 Estructura de correlación Modelo de correlación: Exponential spatial correlation Formula: ~ as.numeric(as.character(Posicion)) Metrica: euclidean Parámetros del modelo Parámetro range Estim 2.74 Medias ajustadas y errores estándares para Hibrido LSD Fisher (alfa=0.05) Procedimiento de correccion de p-valores: No Hibrido H3 H4 H10 H5 Testigo H2 H11 H9 H14 H1 H12 H6 Medias 1248.31 1244.19 1145.64 1128.65 1096.98 1091.07 1078.43 1078.28 1070.07 1005.46 979.80 966.31 E.E. 85.33 85.33 85.33 85.33 45.09 85.33 85.33 85.33 85.33 85.33 85.33 85.33 A A A A A A A A A B B B B B B B B B B C C C C C C C C C 177 Modelos Mixtos en InfoStat H7 H8 H16 H13 H15 936.21 933.40 902.87 727.55 653.36 85.33 85.33 85.33 85.33 85.33 B B C C C D D D D E E Letras distintas indican diferencias significativas(p<= 0.05) Se encontraron diferencias entre híbridos (p = 0.0012). Mediante la prueba LSD de Fisher de comparación de medias se pudo determinar que los híbridos de mayor rendimiento fueron los H2, H3, H4, H5, H9, H10, H11, H14, y que éstos a su vez no difieren del testigo. Otra alternativa es pensar el problema como en los orígenes de la modelación espacial (Papadakis 1937), y utilizar un análisis de covarianza para ajustar las medias de los híbridos en las distintas posiciones. Para realizar una aproximación a este tipo de análisis se construyó una nueva variable llamada Tes, la cual contiene los rendimientos correspondientes a los testigos, luego se adicionó una nueva columna (Hib) en la que se copiaron los valores del rendimiento del hibrido más cercano a cada testigo. Se calculó luego la diferencia del rendimiento del testigo frente al hibrido (Dif). A continuación se realizó un análisis de regresión lineal considerando a Dif como variable dependiente y a Posicion como variable regresora. Se guardaron los predichos de este modelo con el fin de utilizarlos como una covariable en el análisis de las medias de híbridos. Luego, en la ventana del selector de variables de Modelos lineales generalizados y mixtos se declaran las variables como se muestra en la Figura 113. 178 Modelos Mixtos en InfoStat Figura 113: Ventana de selección de variables para Modelos lineales generales y mixtos con datos del archivo Testigos_apareados.IDB2. En la ventana de Efectos fijos se declara a Hibrido y a PRED_Dif. En la solapa Comparaciones se solicitó la prueba LSD de Fisher. La salida correspondiente se presenta a continuación. Modelos lineales generales y mixtos Especificación del modelo en R modelo029_Rendimiento_REML<-gls(Rendimiento~1+Hibrido+PRED_Dif ,method="REML" ,na.action=na.omit ,data=R.data29) Resultados para el modelo: modelo029_Rendimiento_REML Variable dependiente:Rendimiento Medidas de ajuste del modelo N 32 AIC 215.09 BIC 227.23 logLik -88.54 Sigma R2_0 79.89 0.82 AIC y BIC menores implica mejor 179 Modelos Mixtos en InfoStat Pruebas de hipótesis secuenciales (Intercept) Hibrido PRED_Dif numDF F-value 1 5763.58 16 3.42 1 10.15 p-value <0.0001 0.0129 0.0066 Medias ajustadas y errores estándares para Hibrido LSD Fisher (alfa=0.05) Procedimiento de correccion de p-valores: No Hibrido H4 H3 H10 H5 H2 H14 Testigo H11 H9 H12 H1 H8 H7 H6 H16 H13 H15 Medias 1295.07 1293.92 1150.88 1143.52 1129.47 1121.08 1115.81 1078.33 1052.73 988.48 985.32 985.27 983.12 944.67 828.68 810.93 663.53 E.E. 82.46 83.02 80.07 81.10 85.00 83.02 19.97 80.76 79.95 81.10 85.76 79.95 80.07 80.76 85.76 82.46 85.00 A A A A A A A A A B B B B B B B B B B B B C C C C C C C C D D D Letras distintas indican diferencias significativas(p<= 0.05) Si bien se llega a la misma conclusión con respecto a los cultivares que en el análisis usando correlación espacial exponencial, podemos observar que las medias ajustadas y los errores estándares son diferentes. También difiere el orden o ranking presente entre los híbridos que presentan los mayores rendimientos. Por último, el contemplar la correlación espacial es una alternativa mucho más sencilla para realizar este tipo de análisis. 180 Modelos Mixtos en InfoStat Referencias Benjamini Y., Hochberg Y.1995. Controlling the false discovery rate: a practical and powerful approach to multiple testing. J. R. Stat. Soc. Ser. B, 57:289-300. Benjamini Y., Yekutieli, D. 2001. The control of the false discovery rate in multiple hypothesis testing under dependency. The Annals of Statistics, 29(4):11651188. Besag J.E. 1974. Spatial interaction and the statistical analysis of lattice systems. J. R. Stat. Soc. Ser. B 36: 192-225. Besag J.E. 1977. Errors-in-variables estimation for Gaussian lattice schemes. J. R. Stat. Soc. Ser. B 39: 73-78. Casanoves F., Macchiavelli R., Balzarini, M. 2005. Error variation in multienvironment peanut trials: Within-trial spatial correlation and between-trial heterogeneity. Crop Science, 45: 1927-1933. Casanoves F., Macchiavelli R., Balzarini M. 2007. Models for multi-environment yield trials with fixed and random block effects and homogeneous and heterogeneous residual variances. Journal of Agriculture of the University of Puerto Rico, 91(3-4): 117-131. Cullis B.R., Gogel B.J., Verbyla A.P., Thompson R. 1998. Spatial analysis of multienvironment early generation trials. Biometrics 54: 1-18. Di Rienzo J.A., Guzman A.W., Casanoves F. (2002). A Multiple Comparisons Method based On the Distribution of the Root Node Distance of a Binary Tree Obtained by Average Linkage of the Matrix of Euclidean Distances between Treatment Means. JABES 7(2), 129-142. Di Rienzo J. 2007.Curso de Diseño de Experimentos 2007 de la Maestría en Estadística Aplicada – UNC. http://vaca.agro.uncor.edu/~estad/cursosposgrado.htm Gilmour A.R., Thompson R, Cullis B.R., Verbyla A.P. 1997. Accounting for natural and extraneous variation in the analysis of field experiments. J. Agric. Biol. Env. Stat. 2: 269:273. Hsu J.C. 1996. Multiple comparisons: Theory and methods. Firts edition. Chapman & Hall, London. 277 p. 181 Modelos Mixtos en InfoStat Littel R., Milliken G., Stroup W., Wolfinger R., Schabenberger O. 2006. SAS for Mixed Models. Second Ed., SAS Institute, Cary, NC. Littel R., Pendergast J., Natarajan R. 2002. Modelling Covariance Structure in the Analysis of Repeated Measures Data. Statistics in Medicine 19:1793-1819. Martínez N. 2006. Determinación de la tasa de degradación de hojarasca de guadua (Guadua angustifolia) e higuerón (Ficus glabrata) con y sin presencia de macroinvertebrados acuáticos en una quebrada del Valle del Cauca, Colombia. Disertación, Departamento de Biología, Universidad del Valle, Colombia. Mead R. 1971. Models for interplant competition in irregularly spaced population. In: Statistical Ecology, Patil G.P., Pielou E.C. and Waters W.E. (Eds.). Pensilvania State University Press, College Town, Pa, pp: 13-22. Milliken G.A., Johnson D.E. 1984. Analysis of Messy Data, Vol. 1. Van Nostrand Reinhold, NY. Moser E.B., Macchiavelli R. 2002. Model selection techniques for repeated measures covariance structures. Proceedings of the XIV Conference on Applied Statistics in Agriculture 14: 17-31. Navarro C., Cavers S., Pappinen A., Tigerstedt P., Lowe A., Merila J. 2005. Contrasting quantitative traits and neutral genetic markers for genetic resource assessment of Mesoamerican Cedrela odorata. Silvae Genetica. 54: 281–292. Papadakis J.S. 1937. Methode statistique pour des experiencessur champ. Institut d’Amelioration des Plantes a Thessaloniki. Pinheiro J.C., Bates D.M. 2004. Mixed-Effects Models in S and S-PLUS. Springer, New York. Pierson R.A., Ginther O.J. 1987. Follicular population dynamics during the estrus cycle of the mare. Animal Reproduction Science 14: 219–231. Ripley B.D. 1981. Spatial Statistics. Wiley, New York. Zimmerman D.L., Harville D.A. 1991. A random field approach to the analysis of field plot experiments and other spatial experiments. Biometrics 47: 223-239. 182 Modelos Mixtos en InfoStat Índice de cuadros Cuadro 1. Componentes de varianza estimados para los datos del archivo Compvar.IDB2.........................................31 Cuadro 2. Características y medidas de ajuste de los modelos evaluados para los datos del archivo CapacidadRespiratoria.IDB2. .....................................................................................................................................103 Cuadro 3. Criterios de bondad de ajuste para los modelos ajustados con efectos de bloque fijo en los datos del archivo ECRmani.IDB2 ..........................................................................................................................................................141 Cuadro 4. Criterios de bondad de ajuste para los modelos ajustados sin efectos de bloque fijo en los datos del archivo ECRmani.IDB2 ..........................................................................................................................................................141 Índice de figuras Figura 1: Solapas con las opciones para especificación de un modelo lineal general y mixto........................................2 Figura 2: Ventana con la solapa Efectos fijos desplegada para los datos del archivo Atriplex.IDB2. ............................4 Figura 3: Ventana desplegada con la solapa Efectos aleatorios para los datos del archivo Bloque.IDB2.......................6 Figura 4: Ventana desplegada con la solapa Comparaciones para los datos del archivo Bloque.IDB2. .........................8 Figura 5: Ventana de diálogo para importar datos desde las librerías de R...................................................................10 Figura 6: Encabezamiento de la tabla de datos del archivo Ovary................................................................................11 Figura 7: Relación entre el número de folículos (follicles) y el tiempo (Time). ...........................................................11 Figura 8: Ventana desplegada con la solapa Efectos fijos para los datos del archivo Ovary. .......................................12 Figura 9: Ventana desplegada con la solapa Efectos aleatorios para los datos del archivo Ovary................................13 Figura 10: Ventana desplegada con la solapa Correlación para los datos del archivo Ovary. ......................................15 Figura 11: Funciones ajustadas para el número poblacional de folículos (línea sólida negra) y para cada yegua originada por el efecto aleatorio sobre la constante (archivo Ovary)............................................................................17 Figura 12: Funciones ajustadas para el número de folículos para cada yegua originada por la inclusión de efectos aleatorios sobre todos los parámetros de la parte fija del modelo (archivo Ovary).......................................................18 Figura 13: Ventana con la solapa Heteroscedasticidad desplegada para los datos del archivo Ovary. .........................19 Figura 14: Ventana comparación de modelos generales y mixtos con la solapa Diagnóstico desplegada (archivo Atriplex.IDB2)..............................................................................................................................................................22 Figura 15: Ventana comparación de modelos generales y mixtos con la solapa Combinaciones lineales desplegada (archivo Atriplex.IDB2). ..............................................................................................................................................23 Figura 16: Función de autocorrelación de los residuos del modelo presentado en la Ecuación (2) excluyendo la modelación de la autocorrelación serial........................................................................................................................24 183 Modelos Mixtos en InfoStat Figura 17: Función de autocorrelación de los residuos del modelo presentado en la Ecuación (2) incluyendo la modelación de la autocorrelación serial........................................................................................................................25 Figura 18: Ventana de selección de variables para Modelos lineales generales y mixtos con datos del archivo Compvar.IDB2. ............................................................................................................................................................28 Figura 19: Ventana con la solapa Efectos aleatorios desplegada para los datos del archivo Compvar.IDB2 para la especificación del Modelo 1. ........................................................................................................................................29 Figura 20: Ventana Análisis-exploración de modelos estimados con la solapa Diagnóstico desplegada para el Modelo 1 con los datos del archivo Compvar.IDB2. .................................................................................................................32 Figura 21: Gráficos de diagnóstico obtenidos para la variable largo y el modelo 1 para los datos del archivo Compvar.IDB2. ............................................................................................................................................................32 Figura 22: Ventana con la solapa Heteroscedasticidad desplegada para los datos del archivo Compvar.IDB2 para la especificación de varianzas heterogéneas para poblaciones. ........................................................................................33 Figura 23: Gráficos de diagnóstico obtenidos para la variable largo y el modelo 1 con varianzas residuales heterogéneas para poblaciones y los datos del archivo Compvar.IDB2........................................................................38 Figura 24: Ventana con la solapa Efectos fijos desplegada para los datos del archivo Compvar.IDB2 para la especificación del Modelo 2. ........................................................................................................................................39 Figura 25: Ventana con la solapa Efectos aleatorios desplegada para los datos del archivo Compvar.IDB2 para la especificación del Modelo 2. ........................................................................................................................................39 Figura 26: Ventana con la solapa Comparaciones desplegada para los datos del archivo Compvar.IDB2 para la especificación del Modelo 2. ........................................................................................................................................40 Figura 27: Ventana Análisis-exploración de modelos estimados con la solapa Diagnóstico desplegada para el Modelo 2 con los datos del archivo Compvar.IDB2. .................................................................................................................44 Figura 28: Gráficos de diagnóstico obtenidos para la variable largo y el Modelo 2 para los datos del archivo Compvar.IDB2 .............................................................................................................................................................45 Figura 29: Gráficos de diagnóstico obtenidos para la variable largo y el modelo 2 para los datos del archivo Compvar.IDB2 una vez declaradas las varianzas residuales diferentes para cada población. ......................................48 Figura 30: Esquema del diseño en parcelas divididas para el ejemplo de los datos en el archivo Trigo.IDB2. ............51 Figura 31: Encabezamiento de la tabla de datos del archivo Trigo.IDB2. ....................................................................52 Figura 32: Ventana de selección de variables para Modelos lineales generales y mixtos con datos del archivo Trigo.IDB2. ..................................................................................................................................................................52 Figura 33: Ventana con la solapa Efectos fijos desplegada para los datos del archivo Trigo.IDB2..............................53 Figura 34: Ventana con la solapa Efectos aleatorios desplegada para los datos del archivo Trigo.IDB2 con bloque y agua como criterios de estratificación...........................................................................................................................54 Figura 35: Ventana Comparación de modelos generales y mixtos con la solapa Diagnóstico desplegada para los datos del archivo Trigo.IDB2.................................................................................................................................................56 Figura 36: Herramientas gráficas para diagnóstico obtenidas para los datos del archivo Trigo.IDB2..........................57 184 Modelos Mixtos en InfoStat Figura 37: Ventana con la solapa Heteroscedasticidad desplegada para los datos del archivo Trigo.IDB2 con selección de función varIdent con variedad como criterio de agrupamiento. ...............................................................58 Figura 38: Ventana con la solapa Comparaciones desplegada para los datos del archivo Trigo.IDB2 y selección de la subsolapa Medias..........................................................................................................................................................59 Figura 39: Encabezamiento de la tabla de datos del archivo Cobertura de gotas.IDB2. ...............................................61 Figura 40: Ventana de selección de variables para Modelos lineales generales y mixtos con datos del archivo Cobertura de gotas.IDB2. .............................................................................................................................................62 Figura 41: Ventana con la solapa Efectos fijos desplegada para los datos del archivo Cobertura de gotas.IDB2. .......62 Figura 42: Ventana con la solapa Efectos aleatorios desplegada para los datos del archivo Cobertura de gotas.IDB2 con Parcela como criterio de estratificación. ................................................................................................................63 Figura 43: Diagrama de cajas para los residuos estandarizados de Pearson para los niveles del factor Cara. ..............64 Figura 44: Ventana con la solapa Heteroscedasticidad desplegada para los datos del archivo Cobertura de gotas.IDB2 con Cara como criterio de agrupamiento. .....................................................................................................................65 Figura 45: Diagramas de puntos para estudiar la interacción entre Coad y Altura y entre Cara y Altura. ....................68 Figura 46: Esquema del diseño en parcelas subdivididas para el ejemplo de los datos en el archivo Calidad del almidón.IDB2. ..............................................................................................................................................................69 Figura 47: Encabezamiento de la tabla de datos del archivo Calidad del Almidón.IDB2.............................................70 Figura 48: Ventana de selección de variables para Modelos lineales generales y mixtos con datos del archivo Calidad del Almidón.IDB2. .......................................................................................................................................................70 Figura 49: Ventana con la solapa Efectos fijos desplegada para los datos del archivo Calidad del Almidón.IDB2. ....71 Figura 50: Ventana con la solapa Efectos aleatorios desplegada para los datos del archivo Calidad del Almidón.IDB2. ......................................................................................................................................................................................72 Figura 51: Ventana con la solapa Efectos aleatorios desplegada para los datos del archivo Calidad del Almidón.IDB2 que contempla otra forma de especificar la parte aleatoria. ..........................................................................................74 Figura 52: Relación entre cobertura y tiempo para cinco tratamientos del archivo Cobertura forrajes.IDB2..............78 Figura 53: Ventana de selección de variables para Modelos lineales generales y mixtos con datos del ejemplo Cobertura forrajes.IDB2. ..............................................................................................................................................80 Figura 54: Ventana con la solapa Efectos fijos desplegada para los datos del archivo Cobertura forrajes.IDB2. ........81 Figura 55: Ventana con la solapa Correlación desplegada para los datos del archivo Cobertura forrajes.IDB2 y selección de Errores independientes (Modelo 1). .........................................................................................................82 Figura 56: Ventana con la solapa Heteroscedasticidad desplegada para los datos del archivo Cobertura forrajes.IDB2 con selección de función varIdent con tiempo como criterio de agrupamiento (Modelo 2)..........................................83 Figura 57: Ventana con la solapa Correlación desplegada para los datos del archivo Cobertura forrajes.IDB2 y selección de simetría compuesta para datos agrupados por parcela (Modelo 3). .........................................................84 Figura 58: Ventana con la solapa Correlación desplegada para los datos del archivo Cobertura forrajes.IDB2 y selección de modelo Autorregresivo de orden 1 para datos agrupados por parcela y orden de las observaciones indicado por la variable Tiempo (Modelo 5). ...............................................................................................................85 185 Modelos Mixtos en InfoStat Figura 59: Ventana con la solapa Efectos aleatorios desplegada para los datos del archivo Cobertura forrajes.IDB2 con parcela como criterio de estratificación (Modelo 7)...............................................................................................87 Figura 60: Ventana con la solapa Correlación desplegada para los datos del archivo Cobertura forrajes.IDB2 y selección de modelo Autorregresivo continuo de orden 1 para datos agrupados por parcela y orden de las observaciones indicado por la variable Tiempo (Modelo 8). ........................................................................................88 Figura 61: Ventana con la solapa Correlación desplegada para los datos del archivo Cobertura forrajes.IDB2 y selección de modelo Sin estructura para datos agrupados por parcela y orden de las observaciones indicado por la variable Tiempo (Modelo 9). ........................................................................................................................................89 Figura 62: Ventana del Calculador de probabilidades y cuantiles de InfoStat..............................................................93 Figura 63: Ventana con la solapa Comparaciones desplegada para los datos del archivo Cobertura forrajes.IDB2 y selección de la subsolapa Contrastes. ...........................................................................................................................94 Figura 64: Ventana de selector de variables con los datos del archivo CapacidadRespiratoria.IDB2. .........................97 Figura 65: Ventana con la solapa Efectos fijos desplegada con los datos del archivo CapacidadRespiratoria.IDB2. ..98 Figura 66: Ventana con la solapa Correlación desplegada, opción Simetría compuesta, con los datos del archivo CapacidadRespiratoria.IDB2. .......................................................................................................................................99 Figura 67: Ventana con la solapa Correlación desplegada, opción Autorregresivo de orden 1, con los datos del archivo CapacidadRespiratoria.IDB2. ........................................................................................................................100 Figura 68: Ventana con la solapa Heteroscedasticidad desplegada con los datos del archivo CapacidadRespiratoria.IDB2. .....................................................................................................................................101 Figura 69: Ventana con la solapa Efectos aleatorios desplegada con los datos del archivo CapacidadRespiratoria.IDB2. .....................................................................................................................................102 Figura 70: Ventana con la solapa Correlación desplegada, opción Sin estructura, con los datos del archivo CapacidadRespiratoria.IDB2. .....................................................................................................................................103 Figura 71: Ventana de selector de variables para los datos del archivo MedCapRes.IDB2........................................107 Figura 72: Ventana de selector de variables con solapa Particiones activada para los datos del archivo MedCapRes.IDB2.......................................................................................................................................................108 Figura 73: Gráfico de puntos para estudiar la interacción entre tratamientos y hora con los datos del archivo CapacidadRespiratoria.IDB2. .....................................................................................................................................109 Figura 74: Ventana con la solapa Comparaciones, subsolapa Contrastes con los datos del archivo CapacidadRespiratoria.IDB2. .....................................................................................................................................110 Figura 75: Gráfico de puntos para el logaritmo del peso seco remanente en función del tiempo, para los cuatro tratamientos (Especie-Bolsa). Archivo Descomposición.IDB2. .................................................................................113 Figura 76: Especificación del modelo de regresión lineal con ordenadas al origen y pendientes diferentes para el logaritmo de la materia seca remanente en función del tiempo para cuatro tratamientos dados por la especie de origen del material vegetal y el tramado de la bolsa que lo almacena. Archivo Descomposición.IDB2. ..............................114 Figura 77: Gráfico de puntos para el logaritmo del peso seco remanente en función del tiempo, para los cuatro tratamientos (Especie-Bolsa). Archivo Descomposición.IDB2. .................................................................................115 186 Modelos Mixtos en InfoStat Figura 78: Gráfico de residuos estudentizados (Pearson) vs. Tiempo, para un modelo de regresión de la materia seca residual en función del tiempo para cuatro tratamientos (Especia-Bolsa) con diferentes ordenadas y pendientes. Archivo Descomposición.IDB2..................................................................................................................................116 Figura 79: Especificación del modelo de regresión lineal con ordenadas y pendientes diferentes para el logaritmo de la materia seca remanente en función del tiempo para cuatro tratamientos dados por la especie de origen del material vegetal y el tramado de la bolsa que lo almacena. Archivo Descomposición.IDB2. ..................................................116 Figura 80: Ajustes del modelo de regresión polinómica de orden 2 con ordenadas y pendientes diferentes para el logaritmo de la materia seca remanente en función del tiempo y el tiempo^2 (centrados) para cuatro tratamientos dados por la especie de origen del material vegetal y el tramado de la bolsa que lo almacena. Archivo Descomposición.IDB2................................................................................................................................................117 Figura 81: Residuos estudentizados (Pearson) vs. Tiempo para el modelo de regresión polinómica de orden 2 con ordenadas y pendientes diferentes para el logaritmo de la materia seca remanente en función del tiempo y el tiempo^2 (centrados) para cuatro tratamientos dados por la especie de origen del material vegetal y el tramado de la bolsa que lo almacena. Archivo Descomposición.IDB2.............................................................................................................118 Figura 82: Especificación de la parte heteroscedástica del modelo de regresión polinómica de orden 2 con ordenadas y pendientes diferentes para el logaritmo de la materia seca remanente en función del tiempo y el tiempo^2 (centrados) para cuatro tratamientos dados por la especie de origen del material vegetal y el tramado de la bolsa que lo almacena. Archivo Descomposición.IDB2.............................................................................................................118 Figura 83: Residuos estudentizados (Pearson) vs Tiempo para el modelo heteroscedástico de regresión con ordenadas y pendientes diferentes por tratamiento para el logaritmo de la materia seca remanente en función del tiempo y el tiempo^2 (centrados) para cuatro tratamientos dados por la especie de origen del material vegetal y el tramado de la bolsa que lo almacena. Archivo Descomposición.IDB2.............................................................................................119 Figura 84: Especificación de la parte aleatoria del modelo heteroscedástico de regresión polinómica de orden 2 con ordenadas y pendientes diferentes para el logaritmo de la materia seca remanente en función del tiempo y el tiempo^2 (centrados) para cuatro tratamientos dados por la especie de origen del material vegetal y el tramado de la bolsa que lo almacena. Archivo Descomposición.IDB2.............................................................................................................120 Figura 85: Residuos estudentizados (Pearson) vs Tiempo para el modelo heteroscedástico de regresión con ordenadas y pendientes diferentes por tratamiento y el agregado de un efecto aleatorio sobre la constante que es particular para cada combinación de tiempo y tratamiento, para el logaritmo de la materia seca remanente en función del tiempo y el tiempo^2 (centrados) para cuatro tratamientos dados por la especie de origen del material vegetal y el tramado de la bolsa que lo almacena. Archivo Descomposición.IDB2.............................................................................................120 Figura 86: Intérprete de R. Tiene 4 paneles. Script: contiene el o los programas R que se quieren ejecutar. Output: la salida de la ejecución de un script o de la visualización de un objeto, Objetos: la lista de los objetos residente en la memoria de R. Finalmente un panel inferior muestra los mensajes y reporte de errores que envía R a la consola.....121 Figura 87: Curvas de tasas de descomposición según especie y tramado de la bolsa de almacenamiento..................124 Figura 88: Ventana con la solapa Efectos fijos desplegada para los datos del archivo ECRmaní.IDB2 y el Modelo BF. ....................................................................................................................................................................................129 Figura 89: Ventana con la solapa Efectos fijos desplegada para los datos del archivo ECRmaní.IDB2 y el Modelo BA. .............................................................................................................................................................................129 187 Modelos Mixtos en InfoStat Figura 90: Ventana con la solapa Efectos aleatorios desplegada para los datos del archivo ECRmaní.IDB2 y el Modelo BA. ................................................................................................................................................................130 Figura 91: Ventana con la solapa Heteroscedasticidad usando local como criterio de agrupamiento para los datos del archivo ECRmaní.IDB2 y los Modelos BFH y BAH. ................................................................................................131 Figura 92: Ventana con la solapa Correlación usando las variables la y lon como coordenadas en X e Y respectivamente y local como criterio de agrupamiento para los datos del archivo ECRmaní.IDB2 y los Modelos Exp y BFExp......................................................................................................................................................................132 Figura 93: Diagrama de puntos para estudiar la interacción entre localidades y genotipos para la variable rendimiento. ....................................................................................................................................................................................146 Figura 94: Esquema de un experimento conducido bajo un diseño strip-plot repetido en bloques completos al azar, con la aleatorización para un bloque particular de los factores cantidad de nitrógeno y cantidad de riego. Datos del archivo StripPlot.IDB2. ..............................................................................................................................................148 Figura 95: Ventana del procedimiento de Análisis de varianza con la solapa Modelo desplegada para los datos del archivo StripPlot.IDB2. ..............................................................................................................................................149 Figura 96: Diagramas de puntos para estudiar la interacción entre Riego y Nitrógeno y su efecto sobre el rendimiento. Datos archivo StripPlot.IDB2. ....................................................................................................................................151 Figura 97: Ventana con la solapa Efectos fijos desplegada para evaluar un modelo mixto con los datos del archivo StripPlot.IDB2 . ..........................................................................................................................................................152 Figura 98: Ventana con la solapa Efectos aleatorios desplegada para evaluar un modelo mixto con los datos del archivo StripPlot.IDB2 . .............................................................................................................................................153 Figura 99: Ventana se selector de variable para Modelos lineales generales y mixtos los datos del archivo Lombrices.IDB2. ........................................................................................................................................................157 Figura 100: Ventana con la solapa Efectos fijos desplegada para evaluar un modelo mixto con los datos del archivo Lombrices.IDB2. ........................................................................................................................................................158 Figura 101: Ventana con la solapa Correlación desplegada para evaluar un modelo mixto con correlación espacial exponencial en los datos del archivo Lombrices.IDB2...............................................................................................159 Figura 102: Ventana con la solapa Correlación desplegada para evaluar un modelo mixto con correlación autorregresiva de orden 1 en los datos del archivo Lombrices.IDB2..........................................................................161 Figura 103: Herramientas gráficas para diagnóstico obtenidas para los datos del archivo Lombrices.IDB2..............163 Figura 104: Ventana con la solapa Heteroscedasticidad desplegada para evaluar un modelo mixto con en los datos del archivo Lombrices.IDB2. ...........................................................................................................................................164 Figura 105: Diagramas de puntos para estudiar la interacción entre tratamientos y profundidad y su efecto sobre la biomasa. Datos archivo Lombrices.IDB2. ..................................................................................................................166 Figura 106: Ventana con la solapa Comparaciones y la subsolapa Contrastes desplegada para evaluar un modelo mixto con los datos del archivo Lombrices.IDB2.......................................................................................................167 Figura 107: Ventana con la solapa Efectos fijos desplegada para los datos del archivo Testigos_apareados.IDB2. ..170 Figura 108: Ventana con la solapa Correlación desplegada para los datos del archivo Testigos_apareados.IDB2 y selección de Correlación espacial exponencial. ..........................................................................................................172 188 Modelos Mixtos en InfoStat Figura 109: Ventana con la solapa Correlación desplegada para los datos del archivo Testigos_apareados.IDB2 y selección de Correlación espacial Gaussiana..............................................................................................................173 Figura 110: Ventana con la solapa Correlación desplegada para los datos del archivo Testigos_apareados.IDB2 y selección de Correlación espacial lineal. ....................................................................................................................174 Figura 111: Ventana con la solapa Correlación desplegada para los datos del archivo Testigos_apareados.IDB2 y selección de Correlación espacial “rational quadratic”...............................................................................................175 Figura 112: Ventana con la solapa Correlación desplegada para los datos del archivo Testigos_apareados.IDB2 y selección de Correlación espacial esférica..................................................................................................................176 Figura 113: Ventana de selección de variables para Modelos lineales generales y mixtos con datos del archivo Testigos_apareados.IDB2...........................................................................................................................................179 189