Curso Herramientas Informáticas para el análisis estructural de
Anuncio

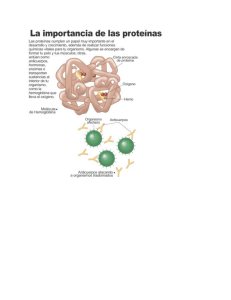
Análisis de proteínas Curso: Herramientas informáticas para el análisis estructural de ácidos nucleicos y proteínas 2004 ¿Qué determina su estructura? • Composición de las proteínas Las proteínas son polímeros de aminoácidos que se unen mediante una unión peptídica Todos los aminoácidos tienen un grupo amino (NH3), un grupo carboxilo (COOH) y un grupo lateral variable (R). Los aminoácidos se unen entre sí mediante unión peptídico entre el carboxilo y amino de aminoácidos adyacentes. El grupo lateral variable es el que determina las características fisico-químicas del aminoácido y el ángulo del enlace peptídico, y por lo tanto también el plegado de la proteína. Curso: Herramientas informáticas para el análisis estructural de ácidos nucleicos y proteínas 2004 Tipos de aminoácidos Dependiendo de su carga electrostática final, los aminoácidos pueden clasificarse en polares, no polares o cargados. Los polares y los cargados se encuentran normalmente en la superficie de las proteínas, e interaccionan con el medio. Los aminoácidos no polares se encuentran generalmente en el interior de las proteínas, donde pueden jugar un rol importante en su actividad. Hidrofóbicos <> Curso: Herramientas informáticas para el análisis estructural de ácidos nucleicos y proteínas 2004 La estructura primaria determina todos los niveles superiores de estructura La cadena polipeptídica se pliega según un determinado patrón para dar una estructura tridimensional particular. estructura primaria : esructura química, número y secuencia de aminoácidos estructura secundaria : plegado producido por las uniones puente de hidrógeno entre los grupos carboxilo e imida de la parte troncal de los aminoácidos. estructura terciaria: Una organización de estr. secundarias ligadas por fragmentos más “libres” de la cadena estabilizados principalmente por interacciones entre las cadenas laterales, por ejemplo por puentes disulfuro estructura cuaternaria : El agregado de varias cadenas polipeptídicas para formar una proteína funcional. Cuando se desnaturaliza una proteína in vitro, ésta puede volver a adquirir su forma original o nativa cuando las condiciones de la solución en la que se encuentra se llevan a las que tenía la proteína nativa originalmente. Esto indica que toda la información necesaria para el plegado de la proteína está contenida en su estructura primaria, su secuencia de aminoácidos. Curso: Herramientas informáticas para el análisis estructural de ácidos nucleicos y proteínas 2004 Homología Existe un número prácticamente infinito de posibles estructuras primarias. Por ejemplo: ¿Cuántos polipéptidos de 200 aminoácidos diferentes puede haber? : 20020 Este número tan grande (mucho mayor si consideramos que existen polipéptidos de más de 1000 residuos), nos permite arribar a 2 conclusiones 1. Sólo una fracción de los posibles polipéptidos existe(o ha existido) en la naturaleza. 2. Es altamente improbable que dos proteínas con una estructura primaria similar hayan aparecido independientemente. Dichas similitudes entonces, indican que lo más probable es que deriven de un antecsor común: son homólogas Curso: Herramientas informáticas para el análisis estructural de ácidos nucleicos y proteínas 2004 La estructura tridimensional de las proteínas está generalmente más conservada que la estructura primaria La estructura tridimensional es estabilizada por múltiples interacciones específicas entre los diferentes grupos químicos presentes Diferencias entre proteínas homólogas: generalmente se observa la aparición de aminoácidos similares en las mismas posiciones En consecuencia, las proteínas homologas tienen estructuras tridimensionales similares. De la misma manera, secuencias nucleotídicas diferentes pueden codificar para secuencias de aminoácidos similares. La escala relativa de conservación es la siguiente: genes < estr primaria de proteinas < estr tridimensional de proteinas Curso: Herramientas informáticas para el análisis estructural de ácidos nucleicos y proteínas 2004 Estructura secundaria Las proteínas no se pliegan al azar. Las estructuras básicas que forman son las láminas beta (beta sheets), y las hélices alfa. Las hélices alfa son muy flexibles y estables, y por lo tanto están generalmente asociadas a regiones de las proteínas que necesitan movilidad. Representación de tipos de hélices (de izq a der: modelo estilizado, esquema de hélice alfa, hélice 3.1 y hélice pi Curso: Herramientas informáticas para el análisis estructural de ácidos nucleicos y proteínas 2004 En las láminas beta están involucradas dos o más hebras de aminoácidos, que se apilan para formar una estructura plegada (estabilizada mediante puentes de hidrógeno). Esta estructura tiende a ser rígida, menos flexible que las hélices alfa. Thioredoxina Ejemplos de hebras que forman láminas paralelas (izquierda) y antiparalelas (derecha) Curso: Herramientas informáticas para el análisis estructural de ácidos nucleicos y proteínas 2004 Uno de los aminoácidos más importantes es la cisteína, pues es el único que contiene grupos sulfuro. Estos grupos interaccionan entre sí formando enlaces sulfhidrilo que unen cisteínas vecinas, acercando las cadenas entre sí. Los giros o vueltas (turns) están usualmente relacionados a los aminoácidos prolina y glicina, pequeños y muy frecuentes. Conociendo la geometría espacial que adoptan los aminoácidos de una región es posible determinar la posible estructura secundaria de una proteína. Curso: Herramientas informáticas para el análisis estructural de ácidos nucleicos y proteínas 2004 Predicción de la estructura y función de macromoléculas por comparación con otras previamente caracterizadas • por homología Cuando dos proteínas tienen una identidad mayor al 25-30%, existe una similitud estructural • Estadística Se basa en la “preferencia”de determinados aminoácidos por formar parte de una estructura determinada (método de Chou & Fasman (1978). • estereoquímica Se basa en las propiedades químicas de los aminoácidos. Se utiliza para predecir regiones hidrofóbicas, o regiones transmembrana (ej: método de Kyte & Doolittle (1982)) Curso: Herramientas informáticas para el análisis estructural de ácidos nucleicos y proteínas 2004 Programa Prosite Este programa permite realizar predicciones acerca de la función de proteínas desconocidas traducidas de secuencias de ADN.. Se basa en la identificación de residuos asociados a una determinada estructura o función, denominados motivos conservados (motifs), patrones (patterns), firmas (signature) o huellas (fingerprints). Contiene una base de datos de sitios y patrones de relevancia biológica ordenados de manera tal que es posible identificar su presencia en una secuencia incógnita El programa Prosite integra los datos de varias bases de datos relacionadas. Curso: Herramientas informáticas para el análisis estructural de ácidos nucleicos y proteínas 2004 Motivos y dominios Los motivos (o patrones) y los dominios surgen del alineamiento de secuencias de proteínas relacionadas, en donde se detectan las regiones más conservadas y se relacionan con una determinada característica. En general se denominan motivos cuando esas regiones son cortas, y dominios a las regiones más extensas. Existen muchas bases de datos que contienen secuencias relacionadas a determinadas funciones, a las que en general se denomina familias de proteínas. A partir de estas familias se deducen las regiones asociadas a cada función. Curso: Herramientas informáticas para el análisis estructural de ácidos nucleicos y proteínas 2004 El InterProScan hace una búsqueda integrada en muchas bases de datos incluyendo en Prosite y otras bases de datos de de motivos, dominios, fingerprints, etc Curso: Herramientas informáticas para el análisis estructural de ácidos nucleicos y proteínas 2004