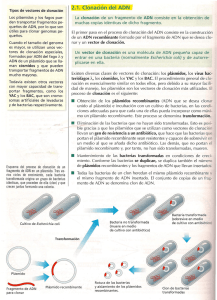
Introduccion Primeramente, tenemos la simulación de la obtención del plásmido recombinante donde En esta parte se diseñará en E. coli, una bacteria común que se encuentra en el intestino de animales de sangre caliente, para producir una proteína de anémona de mar llamada proteína fluorescente roja (RFP), que está dirigida por un gen llamado rfp. Una anémona de mar es un animal de cuerpo blando relacionado con el coral y la medusa. En el laboratorio, le dará a E. coli una nueva proteína que le dará un rasgo que antes no tenía: la capacidad de brillar. Está parte tiene dos simulaciones distintas. La primera parte de la obtención de plásmido recombinante es sobre la digestión de enzimas de restricción, aquí lo que haremos será En esta simulación, se realizará una digestión de restricción. Usarás enzimas de restricción para cortar las hebras de ADN de dos plásmidos y producir así fragmentos de ADN. Debido a que conocemos el tamaño de nuestros plásmidos y donde las enzimas cortarán el ADN, podemos predecir la longitud de los fragmentos que se producirán. También podemos confirmar la longitud de estos fragmentos, o determinar la longitud de los fragmentos producidos a partir de un constructo de plásmidos desconocido, a través de la electroforesis en gel. El descubrimiento, la caracterización y el aislamiento de los diferentes tipos de enzimas han facilitado en la investigación las tareas del estudio de los genomas, así como su expresión, su regulación y su posterior correlación con el desarrollo de diversas enfermedades. Hoy por hoy es común la aplicación de técnicas de biología molecular en diversas áreas, y en particular en las ciencias de la salud, ya que han revolucionado los métodos analíticos generando gran impacto en la investigación básica y la clínica, especialmente en el diagnóstico de enfermedades. Las enzimas de restricción son una herramienta primordial para la ejecución de diversos ensayos útiles en investigación clínica, desde la determinación de polimorfismos de longitud de fragmentos de restricción (restriction fragment length polymorphism, RFLP), así como en la construcción de vectores con ADN recombinante y clonación de secuencias de ADN provenientes de humano, virus, bacterias y demás microorganismos de interés para la generación de conocimiento o para su aplicación en el área de la medicina (Medina, A, 2019). De acuerdo a la segunda parte de la obtención de plásmido recombinante tenemos la ligación de los fragmentos de ADN, en está simulación aprenderemos a a unir dos fragmentos de ADN mediante una técnica llamada ligación. Usarás una enzima para unir los fragmentos creados a partir de una digestión de restricción previa de los plásmidos pKAN-R y pARA a fin de ensamblar todos los componentes necesarios para la expresión génica y la replicación de los plásmidos. Ligar los fragmentos de ADN de los plásmidos pKAN-R y pARA digeridos dará lugar a múltiples plásmidos recombinantes, pero solo uno de los productos es el plásmido recombinante que necesitas: el plásmido pARA-R. Lo siguiente es la electroforesis, en está simulación exploraremos el modo de utilización de una técnica denominada electroforesis en gel para verificar que todos los componentes deseados se hayan incluido en un plásmido recombinante. a electroforesis en gel es una poderosa técnica de laboratorio que permite la separación del material genético en bandas distintas según el tamaño molecular mediante la aplicación de una corriente eléctrica a un gel de agarosa. Como las moléculas genéticas no son visibles a simple vista, se añade un tinte de carga de ADN a cada muestra para visualizar la velocidad de funcionamiento del gel. Esto evita que las muestras migren accidentalmente a través del gel y se pierdan en la solución amortiguadora circundante que conduce la corriente (Dickey, J. L. 2012). La electroforesis en gel se utiliza para determinar si el gen de interés y todos los componentes necesarios se han incluido con éxito en el plásmido recombinante. Cuando sepas los tamaños de los fragmentos de ADN usados para crear el plásmido recombinante deseado, puedes confirmar visualmente que tienes el plásmido deseado comparando su tamaño con una escalera de ADN. Una escalera es una mezcla de fragmentos de ADN de tamaño conocido que se puede usar para estimar el tamaño en pares de bases de cualquier plásmido o fragmento de longitud desconocida. Por consiguiente tenemos el simulación de transformacion de las bacterias, lo que haremos será transformar la bacteria con el plásmido pARA-R recombinante que contiene el gen de interés, la proteína fluorescente roja (rfp). Una vez transformado, el gen rfp se puede expresar usando la propia maquinaria de la bacteria. El plásmido pARA-R también contiene un gen de resistencia a la ampicilina (ampR), que nos permite identificar las bacterias transformadas por su capacidad de crecer en presencia de ampicilina. El plásmido pARA-R también contiene un origen de replicación (ori) para asegurar que el plásmido se mantenga en la población bacteriana, así como el promotor pBAD y el gen araC, que regulan la expresión del gen. Por último tenemos la simulación de la siembra en placas de bacterias transformadas aprenderemos a sembrar en placas y cultivar bacterias en medios sólidos. Esta es una técnica común con una variedad de aplicaciones en el laboratorio. Aquí, usaremos la siembra para verificar que la bacteria E. coli ha adquirido plásmidos de ADN recombinante a través de la transformación. La transformación bacteriana es una técnica que permite que el ADN extraño, como los plásmidos, sea incorporado y expresado mediante los procesos propios de las bacterias. Objetivos Conocer un procedimiento de extracción de ADN plasmídico. Ejecutar experimentos de clonación de ADN. Comprender los procedimientos de obtención de células competentes. Realizar la transformación de células bacterianas con plásmidos recombinantes. Cultivar células transformadas en un medio sólido. Objetivos específicos Producir dos fragmentos de ADN: un fragmento contiene un gen de interés y el otro fragmento es una columna de plásmido que puede utilizarse para expresar proteínas en las células bacterianas. Crear el plásmido pARA-R recombinante para expresar la proteína fluorescente roja (RFP) en las bacterias. Crear experiencia con la electroforesis en gel, que se utiliza para separar e identificar una mezcla de biomoléculas, incluido el ADN; el tamañ o de los componentes de cada mezcla se puede identificar por su ubicació n en el gel Transformar la bacteria E. coli mediante la técnica de choque térmico. Preguntas: 1. Mencione dos factores que inciden en la eficiencia de transformación y posible soluciones La cepa de Producción: Un paso importante para alcanzar altas productividades consiste en la selección de la cepa de producción adecuada. Para ello deben considerarse aspectos cinéticos, el genotipo y el fenotipo de las cepas. En general se buscan cepas con baja tasa de mutación o de inserción de secuencias provenientes del plásmido. Los avances en el conocimiento de la fisiología de E. coli y en el desarrollo de herramientas moleculares han permitido la obtención de cepas diseñadas específicamente para la producción de proteína recombinante. Transferencia de oxígeno al biorreactor: Los cultivos de E. coli para la producción de PR son llevados a cabo bajo condiciones aerobias. De esta manera se obtienen mayores rendimientos de biomasa y producto, debido a que la generación de energía es mayor que bajo condiciones anaerobias. Además, bajo condiciones anaerobias la glucosa o fuente de carbono es oxidada sólo parcialmente por E. coli, lo que conlleva a la acumulación de subproductos de fermentación ácido–mixta, que resultan tóxicos. 2. ¿Cuáles métodos de transformación son más eficientes? Los dos métodos más populares de transformación bacteriana son (1) choque térmico de células competentes preparadas químicamente (transformación química) y (2) electroporación de células electrocompetentes. 3. Señalar la razón por la cual se recomienda utilizar un control negativo de cultivo. Un control negativo es un experimento en el que el microbiólogo sabe que habrá un resultado negativo. En el control negativo, el microbiólogo no espera ninguna respuesta. Implica probar el experimento con algo que sabes que no tendrá ningún efecto en él. Por otro lado, un control negativo es un experimento en el que el microbiólogo sabe que habrá un resultado negativo. En el control negativo, el microbiólogo no espera ninguna respuesta. Implica probar el experimento con algo que sabes que no tendrá ningún efecto en él. 4. ¿De qué diferente manera se trató el cultivo de bacterias P+ del cultivo de bacterias P-? 5. Durante las simulaciones se ha trabajado en condiciones asépticas. ¿Por qué esto es importante? Técnica aséptica es una habilidad fundamental ampliamente practicada en el campo de la Microbiología Ambiental que requiere un equilibrio de mindfulness y práctica en el laboratorio. Uso correcto de esta técnica reduce la probabilidad de contaminación bacteriana o micótica de reactivos, medios de cultivo y muestras ambientales. También es vital para garantizar la integridad de los datos y mantener la pureza de las bibliotecas de cultura que puede ser conformada por muy rara y difícil de cepas en condiciones asépticas. Fuentes de contaminación en el ambiente de laboratorio incluyen aire microorganismos (incluidos los adherirse al polvo y la pelusa partículas) microbios presentan en el espacio de trabajo de banco de laboratorio y cristalería no esterilizada o equipo y microbios transfieren desde el cuerpo y el cabello del investigador. El uso de la técnica aséptica es también una medida de seguridad que reduce el potencial para la transmisión de microorganismos a los investigadores, que es particularmente importante cuando se trabaja con agentes patógenos. Parte 3. Obtención de células competentes 1. 2. 3. Ingrese al simulador Lea el fundamento Observe el vídeo 3.1 Responda las siguientes preguntas: 4. ¿En qué fase de crecimiento deben estar las células? ¿Por qué? Fase logaritmica: Ya que es la fase en la cual los microorganismos se multiplican con rapidez. Durante cada intervalo de duplicación se producen tantas nuevas células como se habían producido anteriormente de manera acumulada. 5. ¿Por qué se deben mantener las células en hielo durante el proceso? La criopreservación tiene como objetivo el mantenimiento de la viabilidad y funcionabilidad celular a temperaturas bajas. La criobiología se refiere a entender los efectos de las temperaturas bajas sobre los sistemas celulares ya que el tiempo biológico es una consecuencia de determinadas reacciones bioquímicas y el frío prolonga el tiempo biológico puesto que enlentece estas reacciones. Sin embargo, este no es un proceso exento de problemas ya que puede inducir variaciones extremas en las propiedades químicas, térmicas y eléctricas las cuales pueden alterar las membranas celulares, los organelos y la delicada interacción célula-célula inherente en las células y tejidos a criopreservar (Gómez, Claudio, 2006). Conclusión En conclusión, tras haber desarrollado el simulador virtual Labxchange se estudió el proceso de digestión del ADN plasmídico, en el cual se emplearon dos tipos de plásmido pARA y pKAN-R que contienen los genes fragmentos de ADN por medio de enzimas fueron RE BamHI e HindIII que son las encargadas de cortar en sitios específicos o también se las conoce como tijeras moleculares, que está basado en el mecanismo de la unión nucleótidos a secuencias específicas que son los sitios de restricción presentando una secuencia palindrómica donde las enzimas ya nombradas realizan un corte de cohesivo en lugares determinados de la secuencia, con el fin de crear un plásmido recombinante. Para confirmar los resultados de la digestión de restricción, necesitarías usar una técnica llamada electroforesis en gel. Esta técnica utiliza un gel de agarosa poroso para separar las moléculas por tamaño y carga. Tener una buena comprensión de los tamaños de fragmentos previstos te permitirá interpretar de forma correcta si las enzimas cortan los plásmidos de manera correcta y eficiente en función del tamaño de las bandas observadas en el gel. Con el desarrollo de esta práctica, referente a la digestión de las enzimas de restricción, podemos concluir el alcance de los objetivos, el dominio del simulador con el paso a paso de la práctica y los cuidados al momento de realizar la clonación o corte para adquirir los fragmentos de plásmidos. En el siguiente simulador pudimos concluir que la ligadura es la unión de dos fragmentos de ácido nucleico mediante la acción de una enzima. Es un procedimiento de laboratorio esencial en la clonación molecular de ADN, mediante el cual se unen fragmentos de ADN para crear moléculas de ADN recombinante. Por otro parte los resultados de la electroforesis en gel proporcionarán evidencia de que está utilizando el plásmido recombinante pARA-R que contiene el gen rfp. El mismo procedimiento se usari ́a para verificar que un plásmido recombinante creado en el laboratorio conteni ́a él gen para una proteína. En conclusión, con el laboratorio de transformación de bacterias pudimos concluir que ara una transformación exitosa, el ADN del plásmido circular debe incorporarse a las células bacterianas. El control negativo contiene células competentes de E. coli, pero no plásmidos, por lo que las células bacterianas no se pueden transformar. Por último, tenemos el cultivo de bacteria transformada en el cual pudimos concluir que no todas las células son transformadas. Algunas de las células no transformadas pueden crecer alrededor de las colonias que si han sido transformadas. Referencias Guerrero, C., Pedras, C. M. G., Gambelas, C. De, Ciências, I. De, Mediterrânicas, A., É vora, U. De, Engenharia, C. De, Ajuda, T., Investigaçaõ , C. De, & Cima, A. (2012). 3. Resultados. 75(Figura 1), 8005. López, M. (2000). Digestión de DNA usando Enzimas de Restricción. Lunt, G. G. (1976). Fisiologiá vegetal. Biochemical Education, 4(3), 59– 60. https://doi.org/10.1016/0307-4412(76)90121-7 Medina, A. (2019). Las enzimas de restricción: las " tijeras moleculares " de los ingenieros genéticos. El Cuaderno de Porque Biotecnologiá . https://www.porquebiotecnologia.com.ar/Cuadernos/El_Cuaderno_34.pdf Toala Arias, F. J. (1390). ENZIMAS DE RESTRICCIÓN. 99–117. Universidad de Granada. (2020). Manual De Problemas, Prácticas De Laboratorio Y Simulación De Genética Ii. Reece, J. B., Taylor, M. R., Simon, E. J. y Dickey, J. L. (2012). Figura 12.13. Gel electrophoresis of DNA (Electroforesis en gel de ADN). En Campbell biology: Concepts & connections (7° ed., pág. 243). Ávila-Portillo, Luz Mabel, Madero, José I, López, Claudia, León, María Fernanda, Acosta, Lucía, Gómez, Claudia, Delgado, Lucy Gabriela, & Gómez, Claudio. (2006). Fundamentos de criopreservación. Revista Colombiana de Obstetricia y Ginecología, 57(4), 291-300. Retrieved June 22, 2022, from http://www.scielo.org.co/scielo.php?script=sci_arttext&pid=S003474342006000400008&lng=en&tlng=es. Durante está pracitica estaremos relizando varios simuladores para reforzar el tema de técnicas de ADN recombinante, estos laboratorios virtuales son impartidos por la plataforma de LabXchange en conjunto a la universidad de Harvar, especificamente estaremos viendo los laboratorios de Digestión con enzimas de restricción, Ligación de los fragmentos de ADN, Verificación de un plásmido recombinante mediante electroforesis en gel, transformacion de bacterias y cultivo de bacterias transformada. Por igual en cada laboratori estaremos realizando nuestras hipotesis acerca de los experimentos y realizadno diferentes pruebines dentro de estos laboratorios. En resumen lo que hicimos en la simulación del laboratorio de digestión con enzimas de restricción fue estudiar el proceso de digestión del ADN plasmídico, en el cual se emplearon dos tipos deplásmido pARA y pKAN-R que contienen los genes fragmentos de ADN por medio de enzimas fueron RE BamHI e HindIII que son las encargadas de cortar en sitios específicos o también se las conoce como tijeras moleculares, que está basado en el mecanismo de la unión nucleótidos a secuencias específicas que son los sitios de restricción presentando una secuencia palindrómica donde las enzimas ya nombradas realizan un corte de cohesivo en lugares determinados de la secuencia, con el fin de crear un plásmido recombinante.