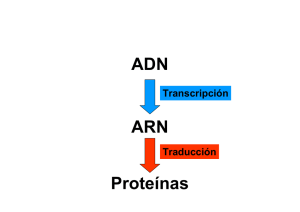
Código genético y traducción
Anuncio

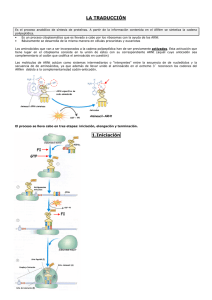
Código genético y traducción Nelson DL y Cox MM. (2000). Lehninger Nelson DL y Cox MM (2000) Lehninger Principios de Bioquímica, 3ª ed. Ed. Omega. Principios de Bioquímica 3ª ed Ed Omega Mathews C.K.; Van Holde K.E.; Ahern, K. G (2002) . Bioquímica, 3ª ed. Addison Wesley Experimentos en la síntesis de proteínas 1.‐ Paul Zamecnik 2.‐ Mahlon Hoagland y Zamecnik 3.‐ Francis Crick • Un Un aminoácido aminoácido determinado puede ser especificado por ser especificado por más de un codón, de manera que el manera que el código se designa como degenerado. como degenerado. De De LLa longitud de los ARNt l it d d l ARNt varía í entre 73 y 93 nucleótidos El inosinato puede formar puentes de hidrógeno ( , y ) con tres nucleótidos diferentes (U,C y A) Esto permite que algunos ARNt reconozcan más de un codón Síntesis de proteínas Síntesis de proteínas • Fase 1: Activación de los aminoácidos (aa) Fase 1: Activación de los aminoácidos (aa) 1)El grupo carboxilo de cada aa 1)El grupo carboxilo de cada aa de ser activado. de ser activado 2)Cada nuevo aa debe ser incorporado de acuerdo con la información contenida en el acuerdo con la información contenida en el ARNm (Se une el aa al ARNt específico covalentemente, consume energía y usa una enzima activadora dependiente de Mg2+). R Reacción realizada en el citosol. ió li d l i l • Fase 2: Inicio El ARNm (portador del código del polipéptido) se une a la menor de las dos subunidades ribosómicas y al aminoacil‐ARNt iniciador. La subunidad ribosómica mayor se une para formar el complejo de inicio. El aminoacil‐ ARNt iniciador se aparea con el codón AUG del ARN (principio ARNm ( i i i del d l polipéptido). li é tid ) Proceso P que requiere GTP 1) Subunidad ribosómica 30S une los factores de inicio impide la unión en 30S y 50 S. El ARNm se une a la 30S. AUG es guiado por la secuencia Shine‐Dalgarno. Shine Dalgarno. 2) El complejo formado por la subunidad ribosómica 30S el subunidad ribosómica 30S, el IF‐3, y el ARNm se une a IF‐2 ligado a GTP y al N‐ formilmetionil‐ARNt iniciador . 3) Este complejo se combina con la subunidad ribosómica 50S; el GTP se hidroliza a GDP y Pi que se desprenden del complejo. Los tres factores de inicio abandonan el ribosoma. Sitio A (aminoacilo) Sitio P (peptidilo) Sitio E (salida) se encuentra en la subunidad 50S subunidad 50S Las secuencias Shine‐Dalgarno se aparean con una secuencia cerca del extremo 3´del 3 del ARNr ARNr 16S Secuencias en el ARNm que sirven de señal para el inicio de la síntesis proteica en las bacterias. p • Fase Fase 3 3 La elongación requiere proteínas citosólicas denominados factores de iniciación. iniciación La fijación del aminoacil‐ARNt entrantes y el desplazamiento de los ribosomas a lo largo del ARNm están facilitados por la hidrólisis de GTP. GTP La elongación requiere: 1)el complejo de inicio 1)el complejo de inicio descrito anteriormente, 2) aminoacil‐ARNt 3)conjunto de tres proteínas (factores de elongación) 4)GTP • Fase Fase 4 4 Terminación y liberación: La finalización se da por un codón de terminación del ARNm por un codón de terminación del ARNm. La cadena polipéptidica se desprende del ribosoma con ayuda de proteínas ib d d í denominadas factores de liberación. Codones de terminación del terminación del ARNm (UAA, UAG, UGA • Fase 5: Plegamiento y modificación post g y p El polipéptido nuevo tiene que plegarse en su conformación tridimensional adecuada ¿Para conformación tridimensional adecuada ¿Para que?. Antes o después del plegamiento, el nuevo polipéptido puede experimentar nuevo polipéptido puede experimentar modificaciones enzimáticas. Eliminación de uno o más aa Eliminación de uno o más aa del extremo amino del extremo amino‐terminal terminal Adición de grupos: acetilo, fosforilo, metilo, carboxilo Figgure 5 5.20: Translatio on of an RN NA messagge intto a p protein. ESTRUCTURA PROTEICA Y FOLDING Plegamiento asistido Plegamiento asistido 1) Las chaperonas moleculares son proteínas 1) Las chaperonas moleculares son proteínas que interaccionan con polipéptidos parcial o incorrectamente plegados, facilitando rutas de plegamiento correctas. 2) Se localizan tanto en bacterias y humanos 3) Primera clase de chaperonas Hsp70 y Hsp40 (DnaK Y DnaJ) 4)La segunda clase de chaperonas se denominan Chaperoninas • Hsp70 Hsp70 estas proteínas son más abundantes en estas proteínas son más abundantes en células sometidas al estrés por temperaturas elevadas Se une a regiones ricas en residuos elevadas. Se une a regiones ricas en residuos hidrofóbicos de polipéptidos no plegados , protegen las proteínas que se han protegen las proteínas que se han desnaturalizado por calor y los péptidos que se están sintetizando (que aun no están se están sintetizando (que aun no están plegados) Las chaperoninas en el plegamiento de proteínas 1) La proteína desplegada se une a la bolsadel GroEL que no que no está bloqueada por GroES. 2) El ATP se une 2) El ATP se une a cada una de las subunidades del heptámero de GroEL. de GroEL. Proteína desplegada 7) Las proteínas que no se han plegado al l d l liberarse se vuelven a unir rápidamente 6) La proteína liberada está liberada está totalmente plegada o en una estado de plegamiento parcial comprometido para adoptar la conformación nativa 5) La proteínas se pliega en el pliega en el interior de la cavidad 3) La hidrólisis de ATP lleva a la liberación de 14 ADP y GroES. 4) 7 ATP y GroES se unen a GroEL con una bolsa llena. La ruta de plegamiento de muchas proteínas a uta de p ega e to de uc as p ote as necesita dos enzimas que catalizan reacciones de isomerización : 1)La proteína disulfuro isomerasa: Cataliza el intercambio o la mezcla de enlaces disulfuro h t hasta que se forman los enlaces de la f l l d l conformación nativa. 2)La péptido prolil cis‐trans 2)La péptido prolil cis trans isomerasa cataliza la cataliza la interconversión de los isómeros cis‐trans de los enlaces peptídicos p p de prolina p . Modificaciones postraduccionales Procesamiento proteolítico Procesamiento Químico *Unión de cadenas laterales de glúcidos g *Adición de grupos isoprenilo (sirve para anclar la proteína en una membrana ) anclar la proteína en una membrana ) *Adición de grupos prostéticos (biotina y el grupo hemo) grupo hemo) *Formación de puentes disulfuro (son comunes en proteínas eucarióticas í ió i d i d destinadas a la exportación) Destino de las proteínas en la célula La mayoría de las proteínas en (cloroplastos y mitocondrias) están codificadas por ADN nuclear. nuclear Los 10 a 15 residuos en el extremo amino‐terminal de algunas proteínas dirige la proteína a su destino final en la célula (estas secuencias son eliminadas por peptidasas p p específicas) p ) Proteínas de las mitocondrias Proteínas del cloroplasto Direccionamiento de proteínas nucleares Retículo endoplásmico Retículo endoplásmico