Tema2 comprimido
Anuncio

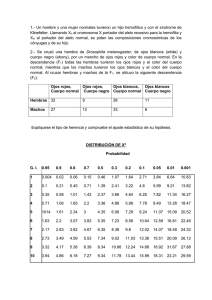
12/09/2013 Métodos para cuantificar la variación genética. Loci mendelianos 1.‐ Gen autosómico, indominante y bialélico 3 fenotipos que se corresponden con 3 genotipos Ejemplo: Locus sanguíneo LM – LN alelos M y N Fenotipos Genotipo Número observado Nº de alelos M 36 NM=NMM= MN 48 NNM= 16 NN = NNN= N NT individuos = M= N alelos = N= / = Frecuencias genotípicas: P = frecuencia de individuos M = H = frecuencia de individos MN = Q = frecuencia de individuos N = Para cada locus: N = sample size = tamaño muestral = nº de alelos Alelos H.O. y H. E. = heterocigosidad observada y esperada Heterocigosidad esperada (H.E.) = 2 . p . q = Frecuencias alélicas: Frecuencia alélica del alelo i = Nº de alelos i/nº total de alelos p = frecuencia del alelo M = Tabla de frecuencias alélicas Locus Alelos Muestra 1 Muestra 2 MN N LM LN H.O. H.E. Heterocigosidad Observada = Het obs (H.O.) = frecuencia de individuos heterocigóticos = Nº de heterocigóticos / nº total de individuos Relación entre alélicas y genotípicas: P = P + ½ H = NMM/NT + ½ NMN/NT = (NMM + ½ NMN)/NT = (2NMM + NMN)/2NT 2.‐ Gen autosómico, dominante y bialélico Fenotipo A = genotipo AA + genotipo Aa Fenotipo a = genotipo aa 3.‐ Gen autosómico, multialélico n alelos n(n+1)/2 genotipos (AiAj) ó (AiAi) Ej: Analizamos 100 individuos para la fosfatasa ácida y encontramos: 25 A, 9 B, 4 C, 30 AB, 20 AC y 12 BC Transformamos en genotipos 25 , 9 , 4 , 30 AB, 20 AC y 12 BC pA = (2xN + N + N )/2NT H.O. = (N + N + N )/NT H.E. : suma de lo que esperamos = 2pi.pj (j>i; i = 1 al nº de alelos) en todas las clases heterocigóticas = 1 ‐ pi2 4.‐ Gen ligado al sexo y bialélico Transformación de un gen multialélico en uno bialélico: AA = (AA) A‐ = (AB + AC) ‐ ‐ = (BB + BC + CC) Ej: G6pdh: detectamos 72 ♀♀ y 120 ♂♂ con la banda alta, 32♀♀ y 80♂♂ con la banda baja y 96 ♀♀ con ambas bandas Fenotipo Portador: estrictamente, aquellos individuos que en su genoma portan el alelo. Sin embargo, donde más se usa el término es en Medicina: usan el término portador (portador del alelo enfermo) para referirse a los individuos sanos portadores de un alelo recesivo, que pueden transmitirlo a su descendencia. Por lo que son individuos que no manifiestan la enfermedad pero si la transmiten Portador = P + H Portadores y sanos = H Genotipo Alelos Machos Hembras Total Machos Hembras Nm= 200 200 Nf= Ni= Nam= Naf= Total 120 ♂ y 72 ♀ A AB B Nº observado 96 ♀ 80 ♂ y 32 ♀ TOTAL Na= En este caso disponemos de tres muestras: machos, hembras y total. Podemos estimar: a) frecuencias alélicas para las 3 muestras: Hembras: pf= (2xNAA + NAB)/ (2xNf ) qf= (2xNBB + NAB)/ (2xNf ) pf + qf = 1 Machos : pm= NA/Nm qm= NB/Nm pm + qm = 1 Total: p = (2xNAA + NAB + NA)/ (2xNf + Nm) Como en este caso Nf = Nm también podemos calcular la p de la población como: p = (2.pf + pm)/3 b) al ser los genotipos distintos en machos que en hembras, sólo calculamos genotípicas, por separado, para machos y para hembras. No se puede calcular genotípicas para la muestra total. c) H.O. y H.E.: Sólo se puede calcular para las hembras, en la muestra de ♀♀ y en la total H.O. (♀) = NAB / Nf H.E. (♀) = 2pfi.pfj ó 2pi.pj Tabla de frecuencias alelicas: Locus Alelo Machos Hembras Total G6pdh A .. .. .. B .. .. .. Het. Obs. ♀ # (?) Het. Esp. ♀ # .. 5.‐ Autosómicos, multialélicos, con dominancia. Por ahora no podemos calcularlo 6.‐ Genes en organismos haplo‐diploides Llevan el mismo tratamiento que los ligados al sexo, igualando el diploide con las hembras y el haploide con los machos. 7.‐ Gen ligado al sexo, multialélico Por extensión de lo visto hasta ahora, para las hembras se calcula igual que los autosómicos y para los machos, las frecuencias alélicas coinciden con las fenotípicas. 1