PCR - Estación Fitopatolóxica do Areeiro
Anuncio

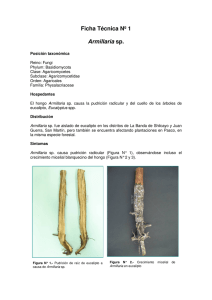
LA REACCIÓN EN CADENA DE LA POLIMERASA (PCR) COMO TÉCNICA DE DIAGNÓSTICO EN FITOPATOLOGÍA FORESTAL O. Aguín (1); M. Sabarís (1); A. Abelleira (1); C. Pintos (1) y J.P. Mansilla (1, 2) (1) Diputación Provincial de Pontevedra. Servicio Agrario. Estación Fitopatológica Do Areeiro. Subida a la Robleda, s/n. 36153 Pontevedra. Www.efa-dip.org (2) Departamento de Producción Vegetal, Universidad de Santiago de Compostela, Campus Universitario, 27002 Lugo INTRODUCCIÓN La identificación en el menor tiempo posible y de una forma fiable de organismos patógenos es fundamental para mininizar los daños que producen. Sin embargo, para algunos hongos, sobre todo los que atacan al sistema radicular, la determinación tradicional basada en la observación de estructuras morfológicas y en propiedades fisiológicas no es suficiente para obtener una identificación fiable. El desarrollo de técnicas moleculares, sobretodo con el descubrimiento en 1985 de la reacción en cadena de la polimerasa (PCR), ha permitido establecer la identidad de cada organismo y sus relaciones filogenéticas, lo que ha revolucionado la sistemática de hongos (EDEL, 1998). Las aplicaciones de la técnica PCR son muy amplias y en la actualidad se utiliza en la detección e identificación de virus, bacterias, hongos, nematodos, etc. (CENIS, 1993). El éxito de esta técnica consiste en su sencillez teórica y en la rapidez en su ejecución, ya que permite la identificación de un patógeno en un día. El poder aplicar con garantías técnicas moleculares supone una enorme ganancia en tiempo que beneficia el control de la enfermedad. Por este motivo el objetivo de este trabajo fue adaptar un protocolo de PCR, complementado con el análisis de la longitud de fragmentos de restricción (RFLP), para los géneros fúngicos Armillaria, Phytophthora y Fusarium. MATERIAL Y MÉTODOS Extracción del ADN Se utilizó el EZNA fungal DNA miniprep Kit AMPLIFICACIÓN Se utilizaron las Ready-To-Go-PCR beads ARMILLARIA: Micelio sobre planta Micelio en medio de cultivo Secuencias de cebadores y condiciones de amplificación para los géneros Armillaria, Phytophthora y Fusarium. HONGOS CEBADORES CONDICIONES DE AMPLIACIÓN (*) - 95 segundos a 951C. Armillaria 0-1: 5'-AGTCCTATGGCCGTGGTAT-3' Material fúngico utilizado: - 35 ciclos: 60 segundos a 601C PHYTOPHTHORA: Micelio en medio de cultivo 120 segundos a 721C 60 segundos a 951C -LR12R: 5' CTGAACGCCTCTAAGTCAGAA-3' - 10 minutos a 721C - 120 segundos a 961C Phytophthora ITS4: 5'-TCCTCCGCTTATTGATATGC-3' FUSARIUM: Micelio en medio de cultivo - 35 ciclos: 60 segundos a 961C 60 segundos a 551C 120 segundos a 721C ITS5: 5'-GGAAGTAAAAGTCGTAACAAGG-3' - 10 minutos a 721C - 182 segundos a 951C Fusarium ITS1: 5'-TCCGTAGGTGAACCTGCGG-3' ARMILLARIA: Alu I, Bsm I, Nde I - 35 ciclos: 60 segundos a 95 1C 60 segundos a 551C RFLP 60 segundos a 721C P3: 5'-CCTTGGTCCGTGTTTCAAGACGGG-3' - 3 minutos a 721C PHYTOPHTHORA: Rsa I, Hae III, Msp I * Condiciones propuestas para Armillaria (PÉREZ et al, 1999), Phytophthora (RISTAINO et al, 1998) y Fusarium (EDEL et al, 1996). RESULTADOS La amplificación del ADN de las muestras fúngicas permitió observar una banda situada aproximadamente a 900 pb en el caso de Armillaria y Phytophthora, mientras que para Fusarium los cebadores amplificaron una región de 1200 pares de bases. En las muestras con Armillaria, se produjo éxito en la amplificación en todos los casos, identificándose cuatro especies : A. mellea, A. ostoyae, A.cepistipes y A. gallica, en distintos hospedadores. En el caso de Phytophthora, mediante la aplicación de las técnicas PCR-RFLP se han detectado hasta el momento tres especies: P. cinnamomi, P. cactorum y P. capsici. En las muestras de Fusarium solo se ha llegado a determinar el género mediante PCR; estudios posteriores con las enzimas de restricción adecuadas permitirán la diferenciación de especies. 1500 Rsa I Hae III 1000 500 500 100 100 Gel de agarosa al 1%, mostrando el DNA amplificado de muestras de Fusarium Gel de agarosa al 2%, mostrando los fragmentos de restricción obtenidos al digerir el DNA amplificado de muestras de Phytophthora con Rsa I y Hae III Gel de agarosa al 3%, mostrando los fragmentos de restricción obtenidos al digerir el DNA amplificado de muestras de Armillaria con Alu I CONCLUSIONES El género Armillaria presentaba grandes dificultades en la identificación de especies y los métodos propuestos resultaban complicados, de mucha duración (entre cuatro o quinco semanas) y con resultados la mayor parte de las veces confusos. La aplicación de la técnica PCR junto con el análisis RFLP ha posibilitado la identificación de las especies en menos de un día de una forma fiable. Para Phytophthora y Fusarium, estas técnicas constituyen, por el momento, un buen complemento al diagnóstico por la metodología tradicional, pero estudios posteriores permitirán sin duda un mejor desarrollo de las mismas. III Congreso Forestal Español, Sierra Nevada (Granada) 25-28 septiembre 2001