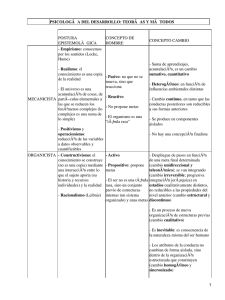
mutantes supresores bacterianos del acido lactico y su uso
Anuncio

k OFICINA ESPAÑOLA DE PATENTES Y MARCAS 19 k kInt. Cl. : C12N 15/74 11 Número de publicación: 2 185 669 7 51 ESPAÑA C12N 15/68 C12N 1/21 A23C 3/08 //(C12N 1/21 C12R 1:225) k TRADUCCION DE PATENTE EUROPEA 12 kNúmero de solicitud europea: 94929490.4 kFecha de presentación: 07.10.1994 kNúmero de publicación de la solicitud: 0 722 503 kFecha de publicación de la solicitud: 24.07.1996 T3 86 86 87 87 k 54 Tı́tulo: Mutantes supresores de las bacterias de ácido láctico y su utilización como marcadores k selectivos y como medios de contención en las bacterias de ácido láctico. 30 Prioridad: 08.10.1993 US 133390 13.05.1994 US 242098 P.O. Box 407 Bøge Allé 10-12 2970 Hørsholm, DK k 72 Inventor/es: Dickely, Françoise; k 74 Agente: Tomás Gil, Tesifonte-Enrique 45 Fecha de la publicación de la mención BOPI: 01.05.2003 ES 2 185 669 T3 k 73 Titular/es: CHR. HANSEN A/S 45 Fecha de la publicación del folleto de patente: 01.05.2003 Aviso: k Johansen, Eric; Nilsson, Dan; Hansen, Egon Bech y Stroeman, Per k En el plazo de nueve meses a contar desde la fecha de publicación en el Boletı́n europeo de patentes, de la mención de concesión de la patente europea, cualquier persona podrá oponerse ante la Oficina Europea de Patentes a la patente concedida. La oposición deberá formularse por escrito y estar motivada; sólo se considerará como formulada una vez que se haya realizado el pago de la tasa de oposición (art. 99.1 del Convenio sobre concesión de Patentes Europeas). Venta de fascı́culos: Oficina Española de Patentes y Marcas. C/Panamá, 1 – 28036 Madrid ES 2 185 669 T3 DESCRIPCION Mutantes supresores de las bacterias de ácido láctico y su utilización como marcadores selectivos y como medios de contención en las bacterias de ácido láctico. 5 10 Campo de la invención La presente invención proporciona mutantes útiles de bacterias del ácido láctico o plásmidos capaces de duplicarse en bacterias del ácido láctico, comprendiendo genes que codifican supresores de la mutación sin sentido, el uso de dichos genes supresores para confinar un replicón a una bacteria del ácido láctico especı́fica o a una bacteria del ácido láctico que crece en un ambiente particular y para controlar el número de células bacterianas del ácido láctico en un ambiente particular. Antecedentes de la técnica y técnica precedente 15 20 25 30 35 40 45 50 55 60 En la sı́ntesis in vivo de proteı́nas que ocurre en los ribosomas, el ARNm se traduce en cadenas de polipéptidos. No obstante, los codones de ARNm no reconocen directamente los aminoácidos que especifican de la manera en que una enzima reconoce un substrato. La traducción utiliza las moléculas “adaptadoras” que reconocen tanto un aminoácido como un grupo triple de bases nucleótidos (un codón). Estos adaptadores consisten en un grupo de pequeñas moléculas ARN conocidas como ARN de transferencia (o ARNt), cada una de las cuales tiene sólo de 70 a 90 nucleótidos de largo. Dichas moléculas de ARNt contienen los residuos impares de nucleótido que incluyen un triplete de CCA a un extremo de la molécula y, en una curva central, un triplete de secuencia variable que forma el denominado anticodón que puede unir las bases a un triplete complementario en la molécula ARNm, mientras el triplete de CCA en el extremo libre ’3 de la molécula está unido de manera covalente a un aminoácido especı́fico. Los tres tripletes del nucleótido UAG (codón ámbar), UGA (codón ópalo) y UAA (codón ocre) no se codifican en un aminoácido. Estas señales denominadas codones de terminación o codones sin sentido están implicadas en la terminación de cadenas de polipéptidos. Durante la traducción, dos factores proteı́nicos (R1 y R2) reconocen estos tripletes y efectúan la liberación de la cadena de polipéptidos del complejo ribosoma-ARNm-ARNt. Ocasionalmente tiene lugar una mutación en una célula resultante en un codón sin sentido que aparece en medio de un gen, causando la terminación prematura de la cadena y la producción de un fragmento proteı́nico. Dichos fragmentos raramente tienen actividad enzimática. El efecto de dicha mutación sin sentido puede ser invertido o suprimido por una segunda mutación en un gen que codifica un ARNt, la cual da lugar a la sı́ntesis de una molécula alterada de ARNt. Dicho ARNt alterado reconoce un codón sin sentido e inserta un aminoácido en ese momento en la cadena de polipéptidos. El gen que codifica el ARNt mutado se denomina gen supresor y el ARNt supresor de la mutación alterada sin sentido al que codifica se denomina generalmente supresor sin sentido o de terminación. Dichos supresores de terminación puede derivarse por substituciones de bases únicas, dobles o triples en la región del anticodón de ARNt. Los supresores de terminación fueron detectados en primer lugar en E. coli hace aproximadamente 25 años y desde entonces han sido estudiados en estas especies. Se considera que ya han sido identificados todos los supresores de terminación en E. coli. Recientemente, nuevos genes supresores de ARNt han sido sintetizados in vitro y posteriormente introducidos en E. Coli. Los supresores de terminación han sido también identificados en el bacteriófago T4 de E. coli y en la Salmonella typhimurium (Eggertson et al., 1988, Microbiological Reviews, 52,354-374). Además, se han identificado los supresores de terminación en los hongos eucarióticos que incluyen Neurospora spp., Saccharomyces cerevisiae y Schizosaccharomyces pombe. Hasta el momento, los supresores sin sentido o de terminación no han sido identificados en las especies bacterianas que pertenecen al importante grupo industrial de bacterias del ácido láctico, las cuales, i. a. son comúnmente utilizadas como fermentos lácticos en la producción de una variedad de productos alimenticios que incluyen productos lácteos, productos cárnicos, productos vegetales, productos de panaderı́a y vino, durante cuya producción estos fermentos lácticos producen el ácido láctico y otros ácidos orgánicos y, en muchos ejemplos, también los metabólitos que aumentan el sabor deseable. Además, los intentos de los presentes inventores de producir cepas supresoras de ámbar de bacterias del ácido láctico mediante la introducción de genes supresores clonados conocidos de E. coli, no han tenido éxito. De esta manera, se intentó introducir el gen E. coli supB (Thorbjarnadóttir et al., 1985), el gen E. 2 ES 2 185 669 T3 coli supE (Nakajima et al., 1981) y el gen E. coli supF (Ryan et al., 1980). Estos tres genes supresores fueron movidos a pFDi3 descrito en el ejemplo 3 posterior y analizados para la actividad supresora en Lactococcus lactis probando la expresión de la resistencia de la eritromicina. Ninguno de los tres genes supresores del E. coli mostró una actividad supresora en Lactococcus lactis. 5 10 15 20 En muchos ejemplos resulta de gran ventaja usar fermentos bacterianos del ácido láctico, los cuales están compuestos por dos o más especies diferentes puesto que la actividad metabólica de una especie puede realizar el crecimiento de otra especie o porque diferentes especies bacterianas del ácido láctico pueden tener efectos particularmente ventajosos en el desarrollo del sabor del producto de los alimentos a estadios especı́ficos de la producción de los alimentos. En consecuencia, existe una necesidad industrial de proporcionar fermentos bacterianos mixtos del ácido láctico en los que una caracterı́stica particular es recluida (o contenida) para una cepa particular. Comúnmente, los genes que codifican las caracterı́sticas deseadas de una bacteria del ácido láctico están localizados en replicones extracromosomales como los plásmidos. En consecuencia, puede resultar de gran ventaja tener dichos replicones contenidos en sus especies huéspedes originales. Tal como se utiliza en este caso, el término contenido indica el confinamiento de un replicón para una célula huésped especı́fica o para el mantenimiento estable de un replicón en una célula huésped bacteriana del ácido láctico cuando esta célula huésped está presente en un ambiente especı́fico. Este mantenimiento estable en una célula huésped particular de un replicón puede también denominarse estabilización de dicho replicón. El término “contención” puede también utilizarse aquı́ para englobar el fenómeno de que se controla el crecimiento y/o viabilidad de una cepa bacteriana de ácido láctico especı́fica en un ambiente particular. 25 30 35 40 45 Los métodos conocidos de los replicones que mantienen la estabilidad (estabilizantes) de una célula huésped incluyen la inserción de secuencias de ADN relativamente grandes como una función de partición. No obstante, como es bien conocido, la inserción de secuencias de ADN grandes implica el riesgo de eliminación de otras secuencias del replicón. Se ha descubierto ahora que pueden desarrollarse cepas bacterianas del ácido láctico que codifican los supresores sin sentido, lo cual proporciona los medios de un nuevo y ventajoso método de confinar replicones en cepas bacterianas de ácido láctico. En oposición a los métodos conocidos para estabilizar (confinar) replicones para células huésped, el método tal como se define en la presente hace posible el uso de genes que codifican el supresor ARNt, los cuales son pequeños y pueden insertarse sin eliminar genes deseados. En la producción de productos alimenticios donde se usan microorganismos vivos puede ser fundamental para la obtención de la calidad deseada de los productos el hecho de que se puedan controlar eficazmente los procesos microbianos. Esto es particularmente importante cuando se usan fermentos lácticos mixtos, tal como se han definidos anteriormente, los cuales comprenden una multitud de cepas. Dicho control ha sido hasta ahora difı́cil de conseguir ya que los mecanismos reguladores especı́ficos en el nivel de números de células y actividad y en el nivel de expresión genética, en particular cepas, tienen que seleccionarse individualmente para cada una de las cepas usadas en el cultivo mixto. No obstante, la presente invención ha hecho posible que el mismo gen supresor bajo el control del mismo mecanismo regulador pueda insertarse en todas las cepas de fermentos lácticos mixtos, por lo que la actividad de las especies de dicho cultivo puede regularse concomitantemente o, si diferentes secuencias reguladoras están insertadas en miembros de especies individuales de fermento láctico, la actividad de los miembros individuales puede regularse de manera independiente. Resumen de la invención 50 55 60 En consecuencia, la presente invención se refiere en un primer aspecto a un Método para aislar una bacteria del ácido láctico que codifica el supresor sin sentido, el cual incluye las fases de (i) mutación de un replicón capaz de duplicarse en una bacteria del ácido láctico, dicho replicón incluyendo un gen que codifica un marcador seleccionable que es expresable en la bacteria del ácido láctico, (ii) selección del replicón mutado de (i) un replicón que contiene una mutación sin sentido en el gen que codifica el marcador seleccionable, (iii) mutación de una bacteria del ácido láctico que no codifica un supresor sin sentido, (iv) introducción del replicón de la fase (ii) en dicha bacteria del ácido láctico mutada y (v) selección de la bacteria mutada de ácido láctico de (iv) una bacteria del ácido láctico transformada que codifica el supresor sin sentido en que se exprime el marcador seleccionable. En otro aspecto se proporciona un método para aislar una bacteria del ácido láctico que codifica el supresor sin sentido, el cual incluye las fases de (i) mutación de un replicón sin mutaciones sin sentido pero que contiene un marcador seleccionable, cuyo plásmido es intrı́nsecamente capaz de duplicarse en 3 ES 2 185 669 T3 5 10 una bacteria del ácido láctico, (ii) selección de un replicón de la fase (i) que contiene una mutación sin sentido que hace que el replicón sea incapaz de duplicarse, (iii) mutación de una bacteria del ácido láctico la cual no codifica un supresor sin sentido, (iv) introducción del replicón de la fase (ii) en dicha bacteria mutada de ácido láctico y (v) selección de una bacteria del ácido láctico transformada en lo cual el replicón introducido es capaz de duplicarse. En otros aspectos, la presente invención también se refiere a un cultivo puro aislado de una bacteria del ácido láctico que incluye un gen que codifica un supresor sin sentido, a una composición que incluye dicho cultivo puro aislado de una bacteria del ácido láctico tal como se define en la presente, un excipiente, y al uso de la composición como fermento láctico en la preparación de un producto alimenticio seleccionado de un producto lácteo, un producto vegetal, un producto cárnico y un producto de panaderı́a. La invención también se refiere a un plásmido que incluye ADN lactobacterial capaz de duplicarse en una bacteria del ácido láctico, el plásmido incluyendo un gen que codifica un supresor sin sentido. 15 20 25 30 35 En un aspecto interesante, la invención se refiere a un método para confinar un replicón extracromosómico capaz de duplicarse en bacterias del ácido láctico para un primer tipo de células bacterianas de ácido láctico, donde dicho replicón podrı́a transferirse naturalmente a un segundo tipo de células bacterianas de ácido láctico, cuyo método consta del suministro del primer tipo de células bacterianas de ácido láctico como células que contienen un gen que codifica un supresor sin sentido; las células siendo transformadas con el replicón en forma de un mutante sin sentido que ha perdido su capacidad de duplicarse en células bacterianas de ácido láctico, el producto genético del gen que codifica el ARNt supresor siendo capaz de restaurar la capacidad del replicón de duplicarse en células bacterianas de ácido láctico de manera que, si una célula del segundo tipo que no contiene un gen del ARNt supresor que codifica un producto de gen capaz de restaurar la capacidad del mutante sin sentido del replicón de duplicarse en bacterias del ácido láctico, recibe dicho replicón extracromosómico, el replicón no se duplicará en el segundo tipo de célula bacteriana de ácido láctico. En otro aspecto interesante, la presente invención se refiere a un método para mantener de manera estable un replicón extracromosómico en células huéspedes bacterianas de ácido láctico que crecen en un ambiente particular, el cual incluye el suministro de dichas células huéspedes como células mutantes sin sentido que han perdido la capacidad de crecimiento en dicho ambiente y que se han transformado con un replicón extracromosómico que contiene una gen del ARNt supresor que codifica un producto genético que restaura la capacidad de las células mutantes sin sentido para crecer en dicho ambiente de manera que, si se pierde el replicón de las células bacterianas de ácido láctico, las células no crecerán. 40 En un aspecto ulterior, la presente invención concierne un método para controlar el número de células bacterianas de ácido láctico en un ambiente particular que permite el crecimiento de bacterias del ácido láctico, el cual incluye el suministro de bacterias con una mutación sin sentido en un gen, la expresión del cual tiene un efecto sobre la viabilidad o el crecimiento de las bacterias y la inserción en las bacterias de un gen que codifica el ARNt supresor bajo el control de un promotor regulable, cuyo producto genético cuando se expresa a un nivel impide la expresión de la mutación sin sentido y el cual, cuando no se expresa o se expresa a un nivel diferente, hace que las células dejen de crecer o se mueran. 45 Descripción detallada de la invención 50 55 60 Como se ha mencionado anteriormente, el método de aislamiento de una bacteria del ácido láctico que codifica el ARNt supresor comprende como fase inicial el suministro de un replicón capaz de duplicarse en la bacteria del ácido láctico, dicho replicón contiene una mutación sin sentido en una codificación del gen para un marcador seleccionable (un gen marcador) y preferiblemente también una codificación del gen no mutado para otro producto genético seleccionable. El gen marcador seleccionable puede ser seleccionado de cualquier gen que codifica un fenotipo fácilmente detectable como un gen cuya expresión confiere resistencia a un antibiótico al que la bacteria del ácido láctico es sensible, incluyendo como ejemplos, la resistencia a la eritromicina, el cloramfenicol o la tetraciclina. Otros marcadores mutantes útiles incluyen fenotipos auxotróficos como mutantes cromosómicos puros o replicones como el plásmido o bacteriófagos mutantes que han perdido su capacidad inherente de replicación en la bacteria del ácido láctico. Según la presente invención, cualquier mutágeno convencional adecuado, incluyendo la radiación ultravioleta e ionizante y mutágenos quı́micos que incluyen mutágenos que afectan al ADN no replicante como HNO2 NH2 OH; agentes alquilantes que incluyen, por ejemplo, sulfonato del metano etı́lico (EMS) y N-metil-N’-nitro-Nnitrosoguanidina (NTG); y análogos de base o mutágenos que desplazan el marco de lectura del código genético, puede utilizarse para mutar el gen marcador. Además, la mutagénesis puede 4 ES 2 185 669 T3 ser mutagénesis dirigida, usando técnicas de ADN recombinante que incluyen el uso de cebadores en la reacción en cadena de la polimerasa, de elementos transposables o cepas mutágenas bacterianas, p. ej. la cepa mutágena LE30. 5 Aunque la mutación sin sentido, tal como se ha definido anteriormente, puede proporcionarse más cómodamente sometiendo el replicón al tratamiento de mutagenización anteriormente definido, no se excluye que un replicón que contiene una mutación sin sentido en la codificación genética para un marcador seleccionable pueda ser un mutante espontáneo aislado. 10 A continuación, se designa una mutación sin sentido por la designación convencional del gen en el que ha tenido lugar la mutación, seguido por una indicación del tipo de mutación sin sentido. Ası́, una mutación sin sentido reconocible bien por un supresor ámbar o por un supresor ocre, en los genes que codifican la resistencia a la eritromicina y al cloramfenicol, es designada erm-am y cat-am respectivamente. 15 En algunas formas de realización de la invención, el replicón mutado puede contener dos o más mutaciones sin sentido, p. ej. en genes que codifican la resistencia antibiótica como erm-am y cat-am. Dichos replicones que contienen mutaciones sin sentido múltiples pueden construirse convenientemente mediante la recombinación en un replicón de las secuencias de ADN que contienen las mutaciones. Ası́, como un Ejemplo que está descrito detalladamente a continuación, puede aislarse un plásmido inicial que contiene una mutación sin sentido en el gen erm. En una fase posterior, una secuencia de ADN que incluye este gen puede ser insertado en otro plásmido que lleve una mutación sin sentido en el gen cat. Un ejemplo tı́pico de un replicón que contiene dos mutaciones sin sentido es el plásmido pFDi10 como se describe posteriormente. 20 25 30 35 40 45 Cuando se obtiene un replicón mutante sin sentido aislado, se utiliza éste como medio para aislar una bacteria del ácido láctico en la que ha sido generada una mutación del ARNt supresor, sometiendo a una población madre de bacterias del ácido láctico que no codifica un supresor mutante a un tratamiento con un mutágeno tal como se ha definido anteriormente. El replicón mutante es introducido mediante técnicas de transformación convencionales en una población de células bacterianas de ácido láctico mutadas y una célula mutante de ARNt supresor es seleccionada entre estas células transformadas mediante el crecimiento de las células bajo condiciones que permiten al gen marcador del replicón expresarse y el aislamiento de las células huéspedes en las que se expresa la mutación sin sentido en el replicón. Cuando p. ej. la mutación en el replicón está en un gen que confiere resistencia a uno o más antibióticos, un mutante que codifica el ARNt supresor de las células bacterianas de ácido láctico transformadas puede cómodamente ser seleccionado de un medio que contenga este/estos antibiótico(s). El hecho de que una célula transformada capaz de crecer en dicho medio selectivo sea una célula que codifica el supresor real puede posteriormente verificarse, (i) transformando el mismo replicón que contiene mutaciones sin sentido en células huéspedes supresoras conocidas en las que el replicón puede duplicarse y en células huéspedes correspondientes sin el gen supresor y (ii) reintroduciendo el replicón en la supuesta célula que codifica el supresor para confirmar el fenotipo del supresor. Los transformadores ası́ obtenidos que suprimen sin sentidos pueden posteriormente ser polimerizados del replicón p. ej. a través del crecimiento de los transformadores en un medio no selectivo o mediante el tratamiento de éstos con un agente vulcanizante de plásmido convencional. El gen supresor puede localizarse en el cromosoma o en un replicón extracromosómico. 50 55 60 La mayorı́a de las mutaciones en un gen que codifica el ARNt que dan lugar a la formación de un supresor sin sentido están localizadas en el triplete anticodón y lo alteran a CUA, UUA o UCA. Dichos supresores pueden designarse como supresores ámbar, ocre y ópalo, respectivamente. Siguiendo las reglas de nomenclatura de Demerec et al. (Genetics, 1966, 54, 61-76) las cuales fueron sugeridas para los supresores de terminación (sin sentido) en E. coli el sı́mbolo “sup” y las mayúsculas asignadas como designaciones de gen, p. ej. supB, supC o supZ, se utilizan aquı́ también para designar los genes del supresor en las bacterias del ácido láctico. En este sistema, el término sup+ (p. ej. supB+) representa el alelo natural y sup-(e.g. supB-) representa el alelo mutante. Los supresores ámbar reconocen generalmente sólo codones ámbar mientras que todos los supresores ocre reconocen codones sin sentido tanto ámbar como ocre. El espectro de supresión de una mutación sin sentido depende no sólo del anticodón (ámbar o ocre), sino también del aminoácido insertado en el codón sin sentido. Cuando el ARNt supresor está causando la inserción de un aminoácido que es diferente a la proteı́na natural, la proteı́na resultante puede ser no funcional. En consecuencia, algunas mutaciones sin 5 ES 2 185 669 T3 sentido pueden sólo ser suprimidas por un tipo de supresores, mientras que otras pueden ser suprimidas por varios o todos los supresores. 5 Los supresores pueden mostrar una eficiencia de supresión pobre que significa que el término de traducción en el codón sin sentido no está completamente suprimido. De esta manera, un supresor con una eficiencia del 10 % permitirá que sólo un 10 % de la proteı́na codificada por un gen con una mutación sin sentido sea sintetizada por completo mientras que un 90 % de las moléculas proteı́nicas terminarán en el codón sin sentido. Muchos supresores ocre son sólo eficaces en un 5 % a 10 % mientras que los supresores ámbar normalmente tienen una eficiencia que varı́a del 25 % al 65 %. 10 15 20 25 Tal como se ha mencionado anteriormente, la presente invención se refiere en un aspecto a un método de aislamiento de una bacteria del ácido láctico que codifica el ARNt supresor en la que el replicón que está mutado para obtener la mutación sin sentido es uno que antes de la mutagénesis es capaz de duplicarse en la bacteria del ácido láctico y el cual ha adquirido una mutación sin sentido en uno o más genes, por lo que el replicón mutado aislado ya no es capaz de duplicarse en la bacteria del ácido láctico que no contenga un gen supresor, el producto del cual puede suprimir la mutación sin sentido de la replicación. Dichos replicones mutados sin sentido que han perdido su capacidad de replicación pueden derivarse de plásmidos o bacteriófagos que se duplican normalmente en bacterias del ácido láctico. Como ejemplo que también se describe detalladamente en el ejemplo 4A anteriormente mencionado, el plásmido del citrato de la cepa Lactococcus lactis subesp. lactis biovar diacetylactis DB1138 puede ser mutado por ej. mediante la reacción en cadena de la polimerasa para obtener una mutación sin sentido ámbar en el gen repB. El replicón del cual puede derivarse un mutante sin sentido no replicante puede ser también un bacteriófago que incluye, por Ejemplo, el Lactococcus fago ØMPC100 prolato, del cual pueden derivarse los mutantes con mutaciones sin sentido en genes esenciales para el desarrollo del fago. Dichos mutantes han perdido la capacidad del fago progenitor para formar placas en bacterias del ácido láctico sensibles. Los mutantes sin sentido de bacteriófagos pueden p. ej. generarse idóneamente por mutagenización con hidroxilamina (NH2 OH). 30 35 40 45 50 55 60 Tal como se ha mencionado anteriormente, en la presente se proporciona un cultivo puro aislado de una bacteria del ácido láctico que incluye un gen que codifica un ARNt supresor como un gen que codifica el ARNt supresor ámbar, ocre o ópalo. Dicho gen que codifica un supresor de terminación (sin sentido) puede localizarse en el cromosoma de la bacteria o puede, en otras formas de realización, localizarse extracromosómicamente p. ej. en un plásmido o estar incorporado en una célula como un profago. Cuando la bacteria del ácido láctico del cultivo anteriormente mencionada es una usada como fermento en la producción de alimentos, puede ser preferible que la bacteria sólo contenga ADN de ácido láctico de origen bacteriano, el cual incluye ADN aislado de plásmidos u otros replicones que tienen la bacteria del ácido láctico como su organismo huésped natural. En consecuencia, el ARNt supresor que contiene bacterias del ácido láctico puede, en formas de realización preferidas, contener un gen supresor que es un gen nativo o que se deriva de una bacteria heteróloga del ácido láctico. En la técnica, las bacterias del ácido láctico recombinantes que sólo contienen ADN de ácido láctico de origen bacteriano son también designadas como organismos “de grado alimenticio” ya que generalmente se considera que el uso de dichos organismos puede ser admisible por las autoridades gubernamentales competentes para el uso en la producción de alimentos. Como se entenderá a partir de lo anteriormente mencionado, el gen supresor contenido en el cultivo puro puede ser un gen insertado bien derivado de una cepa bacteriana de ácido láctico heterólogo o de un plásmido bacteriano de ácido láctico heterólogo o puede ser un gen supresor derivado de la mutagenización de un gen nativo homólogo que puede bien estar localizado cromosómica mente o extracromosómicamente. Cuando el gen es un gen insertado, puede estar insertado en el cromosoma o introducido en un plásmido o un bacteriófago. En formas de realización útiles de la invención, las células bacterianas de ácido láctico del cultivo puro pueden comprender, además del gen supresor, una mutación sin sentido que puede ser suprimida por el ARNt supresor. Cuando están presentes en las mismas células, el gen supresor y el gen con la mutación sin sentido pueden preferiblemente localizarse en replicones diferentes, p. ej. de modo que el gen supresor esté localizado en el cromosoma mientras que la mutación sin sentido tiene lugar en un gen contenido en un replicón extracromosómico. En otras formas de realización útiles, la ubicación de los dos genes es la inversa. 6 ES 2 185 669 T3 5 10 15 En algunas formas de realización preferidas, el supresor codificado por la bacteria del ácido láctico que constituye el cultivo puro aislado es un supresor ámbar. El supresor puede ser, p. ej., un supresor que suprime una mutación sin sentido que, en ausencia de un ARNt supresor capaz de suprimir la mutación, confiere auxotrofı́a como una mutación sin sentido en un gen involucrado en la sı́ntesis de nucleótidos de purina de sus precursores. Ası́ el cultivo puede comprender una bacteria del ácido láctico que es un mutante sin sentido puro. En consecuencia, en una forma de realización particularmente útil, la bacteria del ácido láctico en un cultivo es una cepa que contiene una mutación sin sentido que es suprimida por un gen supresor localizado en el plásmido de grado alimenticio en el modo descrito anteriormente. Como ejemplo, dicho plásmido puede construirse para contener una región de replicación de una bacteria del ácido láctico o un plásmido de origen natural en dicha bacteria, p. ej. la región de la replicación de un plásmido naturalmente albergado por un Lactococcus sp. que incluye la región de la replicación del plásmido de citrato en Lactococcus lactis subesp. lactis biovar diacetylactis y un gen supresor de una bacteria del ácido láctico tal como se define en la presente como p. ej. un gen de ARNt supresor de la cepa Lactococcus lactis FD100 como se describe a continuación. En dicho plásmido, el gen supresor funcionará como un marcador seleccionable si la mutación sin sentido es tal que en ausencia del gen supresor correspondiente hace que la bacteria sea incapaz de crecer en un ambiente particular, como la leche o cualquier otro producto alimenticio o producto agrı́cola en el que se utilizan bacterias del ácido láctico. 20 Un plásmido de construcción en el modo descrito anteriormente será útil como vector de clonación si está adicionalmente provisto de uno o más lugares de restricción únicos adecuados que incluyen el ADN aislado de una bacteria láctica o en una secuencia no codificante sintética ligadora/poliligadora. Dicho vector de clonación está incluido en la presente invención. 25 Preferiblemente dichos vectores de clonación de grado alimenticio tienen un tamaño que permite la inserción de genes deseables. En consecuencia, un vector de clonación de dimensiones ideales tal como se define en la presente tiene un tamaño que varı́a de entre 0,5 a 20 kb, aunque también pueden utilizarse vectores más grandes. En las formas de realización preferidas, el vector de clonación tiene un tamaño que varı́a de entre 1 a 10 kb o de entre 2 a 5 kb. Ejemplos de dichos vectores de clonación son los plásmidos pFG como se describen posteriormente. 30 35 40 45 50 55 60 Según la invención, dichos vectores de clonación pueden utilizarse para la inserción de genes que codifican productos genéticos deseables, en particular genes aislados de las bacterias del ácido láctico. Dichos genes útiles incluyen genes que codifican las enzimas que tienen un efecto ventajoso en la calidad de los productos alimenticios, cuya producción o preservación incluye la adición de cultivos bacterianos viables de ácido láctico tal como se ha descrito anteriormente. Ası́, los genes insertados en el vector de clonación anteriormente mencionado pueden codificar peptidasas, incluyendo dipeptidasas, ejemplos de dichas peptidasas son los productos genéticos de los genes pepN, pepC y pepR como se ejemplifica posteriormente. Otros productos genéticos interesantes incluyen lipasas, proteasas, nucleasas y enzimas que están implicadas en el metabolismo de los carbohidratos de la bacteria huésped. Los genes insertados pueden también ser genes procarióticos aislados de las especies bacterianas sin ácido láctico. Se contempla además que los genes útiles en el presente contexto sean genes eucarióticos p. ej. genes mamı́feros que codifican productos genéticos activos inmunológicamente, enzimáticamente o farmacológicamente, los cuales incluyen por Ejemplo enzimas proteolı́ticas como quimosina o plasminógeno. Se ha descubierto que una bacteria del ácido láctico que codifica normalmente una enzima particular puede tener la expresión de esta enzima aumentada por un factor de al menos 2 o 5 o incluso por un factor de al menos 10 siendo transformada con un vector de clonación tal como se ha definido anteriormente, en el que está insertado un gen que codifica una enzima que tiene una actividad similar. Ejemplos de dichos vectores son los derivados de pFG1 plásmido, pFG2, pFG3, pFG4, pFG5 y pFG6 como se describen en los ejemplos abajo mencionados. Se ha descubierto que el producto genético del gen supresor del vector de grado alimenticio anteriormente mencionado puede en algunos casos estar “sobreexpresado” hasta un cierto punto donde puede perjudicarse el crecimiento normal de la célula huésped. No obstante, también se ha descubierto que puede haber mutantes en los que el promotor natural para el gen supresor del vector está mutado, dando lugar a una expresión disminuida del gen supresor que permite que la célula huésped crezca de manera normal. En consecuencia, la presente invención presenta en una forma de realización un vector de clonación que incluye un gen supresor con un promotor conectado funcionalmente a éste en el que tiene lugar una mutación. También se ha descubierto que pueden tener lugar mutaciones en un cromosoma de la célula huésped bacteriana de ácido láctico que contiene mutación sin sentido, lo cual posibilita que la célula crezca de manera normal cuando alberga un plásmido vector a partir del cual un gen supresor está 7 ES 2 185 669 T3 sobreexpresado. Dichas células huéspedes, las cuales son claramente refractarias al efecto de la sobreproducción del gen supresor, pueden ser huéspedes muy útiles para clonar vectores como p. ej. el plásmido pFG1 anteriormente mencionado y sus derivados. 5 10 15 20 25 30 El cultivo bacteriano de ácido láctico puede comprender cualquier bacteria del ácido láctico. Como se utiliza en este caso, el término “bacterias del ácido láctico” designa a un grupo de bacterias que tienen como caracterı́stica común la capacidad de producir ácido láctico a partir de azúcares. La mayorı́a de las especies que pertenecen a este grupo pueden caracterizarse como gram-positivas, catalasas negativas, bacterias anaeróbicas o microaerofı́licas que pueden ser cocos o bastoncillos. El género anaeróbico Bifidobacterium está también generalmente incluido en el grupo de las bacterias del ácido láctico. En consecuencia, el cultivo puro de una bacteria del ácido láctico consta preferiblemente de bacterias seleccionadas de Lactococcus spp., Streptococcus spp., Lactobacillus spp., Leuconostoc spp., Pediococcus spp. y Bifidobacterium spp. En algunas formas de realización útiles, la bacteria del ácido láctico en el cultivo aislado es Lactococcus lactis. El cultivo puede, según la invención, comprender dos o más especies diferentes de bacterias del ácido láctico o dos o más cepas de las mismas especies. Como se ha mencionado anteriormente, esto es común en la producción de productos alimenticios, donde se utilizan fermentos bacterianos de ácido láctico para aplicarlos a cultivos mixtos, es decir cultivos que incluyen una multitud de cepas. Como ejemplo puede mencionarse que normalmente se utiliza un cultivo mixto de Lactobacillus bulgaricus y Estreptococcus thermophilus en la producción de yogur. En otros productos lácteos puede utilizarse un cultivo mixto de Bifidobacterium bifidum y Lactobacillus acidophilus. Además para su uso como fermentos alimenticios, las bacterias del ácido láctico según la presente invención pueden también aplicarse en la producción de pienso como ensilaje donde los fermentos son inoculados en la cosecha de alimento con el objetivo de obtener su preservación o en productos de desecho del animal ricos en proteı́nas como los desperdicios de la degollación o los desperdicios del pez, también con el objetivo de conservar estos desperdicios para la alimentación del animal. Aún otra aplicación significante de los cultivos bacterianos de ácido láctico, según la presente invención, es el uso de dichos cultivos como los denominados probióticos. Mediante el término “probiótico”, en el presente contexto, se entiende un cultivo microbiano el cual, cuando son ingeridos en forma de células viables por seres humanos o animales, confiere una condición de mejor salud, p. ej. suprimiendo los microorganismos nocivos en la trayectoria gastrointestinal, mejorando el sistema inmunológico o contribuyendo a la digestión de los nutrientes. 35 El cultivo tal como se ha definido anteriormente puede ventajosamente estar en una forma concentrada con p. ej. al menos 109 de unidades que forman la colonia por gramo de cultivo. Dichos concentrados pueden proporcionarse como una pasta de célula, p. ej. separada de una fermentadora o como un cultivo congelado o liofilizado. 40 En una forma de realización interesante, el cultivo según la invención consta de una bacteria del ácido láctico donde el gen que codifica un ARNt supresor está bajo el control de un estimulador regulable. Como se utiliza en este caso, el término “promotor regulable” se utiliza para describir una secuencia del promotor que incluye posiblemente secuencias reguladoras para el promotor cuyo promotor es regulable por uno o más factores seleccionados del pH y/o el contenido de arginina del medio, la temperatura de crecimiento, un cambio de la temperatura que suscita la expresión de genes de choque térmico, la composición del medio de crecimiento que incluye la fuerza iónica/contenido del NaCI y la fase de crecimiento/nivel de crecimiento de la bacteria del ácido láctico. Dicho promotor regulable puede ser el promotor nativo o un promotor insertado no relacionado naturalmente con el gen supresor, bien aislado desde la misma bacteria del ácido láctico o bien una secuencia del promotor heteróloga. 45 50 55 60 Una secuencia del promotor tal como se ha definido anteriormente puede comprender secuencias adicionales, por lo que el promotor será regulado por un evento estocástico. Dicha regulación puede p. ej. ser útil en cultivos bacterianos de ácido láctico para los cuales puede ser ventajoso tener una actividad gradualmente decreciente del gen supresor bajo control de la secuencia del promotor. Dichas secuencias adicionales pueden p. ej. ser secuencias la presencia de las cuales ocasiona una excisión recombinacional del promotor o de los genes que codifican las substancias que se necesitan positivamente para el funcionamiento del promotor. Como se ha mencionado anteriormente, la invención se refiere en otros aspectos a una composición que incluye un cultivo puro aislado de una bacteria del ácido láctico tal como se ha definido anteriormente y a un excipiente microbiológicamente aceptable y al uso de dicha composición como fermento en la Preparación de productos alimenticios. Es preferible que dicha composición contenga al menos 109 8 ES 2 185 669 T3 colonias formando unidades de bacterias. Preferiblemente, el soporte puede comprender nutrientes como carbohidratos asimilables o una fuente de nitrógeno que pueden utilizarse prontamente por la bacteria del ácido láctico. Normalmente, dicha composición se proporciona en forma de composición congelada o liofilizada. 5 10 15 20 Como se ha mencionado anteriormente, la invención se refiere, en otro aspecto, a un plásmido que incluye un gen que codifica un ARNt supresor. El gen puede, en algunas de las formas de realización preferidas, derivarse del cromosoma de una bacteria del ácido láctico. Según la presente invención, el plásmido es construido mediante la inserción de una secuencia de ADN aislada que incluye un gen supresor funcional en una bacteria del ácido láctico, en un plásmido inicial que puede duplicarse en una bacteria del ácido láctico. El plásmido inicial puede ser cualquiera que contenga una mutación sin sentido, p. ej. en un gen cuyo producto genético nativo sea requerido para la replicación del plásmido o en un gen que confiera resistencia antibiótica. La secuencia de ADN anteriormente mencionada, la cual incluye un gen supresor, es preferiblemente una secuencia pequeña como una secuencia que varı́a de entre 0,05 a 10 kb, más preferiblemente que varı́a de entre 0,1 a 5,1 kb, como p. ej. 3,2, 1,1 o 0,25 kb. El gen supresor puede preferiblemente codificar un ARNt con un anticodón que reconoce sólo codones ámbar, es decir un supresor ámbar. Como Ejemplo, la secuencia de ADN que codifica este tipo de ARNt puede ser la siguiente secuencia (SEC ID NO:1) 1 GGAGCCATGG CAGAGTGGTA ATGCAACGGA CTCTAAATCC GTCGAACCGT 51 GTAAAGCGGC GCAGGGGTTC AAATCCCCTT GACTCCTTA 25 El plásmido según la invención puede adicionalmente comprender un gen insertado que codifica un producto genético deseado. En este contexto, productos genéticos interesantes deseados incluyen enzimas hidrolı́ticas seleccionadas de proteasas como quimosina, peptidasas que incluyen endopeptidasas, lipasas, nucleasas y carbohidrasas; enzimas lı́ticas como lisozima o lisinas de fago; sustancias que mejoran el sabor; bacteriocinas que incluyen nisina, pediocina y bavaracina; aminoácidos; ácidos orgánicos y substancias activas farmacológicamente. 30 Según la invención, el gen que codifica el supresor soportado por el plásmido puede estar bajo el control de un promotor regulable tal como se ha definido anteriormente. 40 Tal como se ha descrito anteriormente, puede resultar de ventaja confinar un replicón extracromosómico a un tipo particular de bacterias del ácido láctico y, en consecuencia, la invención provee en un aspecto un método tal como se ha definido anteriormente para confinar un replicón extracromosómico a un primer tipo de células bacterianas de ácido láctico, donde el replicón podrı́a ser naturalmente transferido a un segundo tipo de células bacterianas de ácido láctico. El primer tipo de bacterias del ácido láctico es preferiblemente seleccionado de Lactococcus spp. incluyendo Lactococcus lactis,Streptococcus spp., Lactobacillus spp., Leuconostoc spp., Pediococcus spp. y Bifidobacterium spp. Como se utiliza en este caso, el término “confinar” indica que el replicón está estabilizado o contenido en el primer tipo de células. 45 Los replicones mutados sin sentido que han perdido su capacidad de replicación pueden ser derivados de plásmidos o bacteriófagos que se duplican naturalmente en bacterias del ácido láctico. Un ejemplo de dicho replicón no replicante es un mutante sin sentido del plásmido de citrato de la cepa Lactococcus lactis subesp. lactis biovar diacetylactis DB1138 en el modo descrito anteriormente. 35 Según el presente método, el gen que codifica el supresor puede localizarse en el cromosoma o puede ser un gen en un replicón como un plásmido o bacteriófago diferente del que será confinado en el primer tipo de células. 50 55 60 El replicón que será confinado puede, en formas de realización preferidas, ser un replicón recombinante que incluye un gen que codifica un producto de gen deseado tal como se ha definido anteriormente. Además de confinar un replicón tal como se ha definido anteriormente, también puede ser interesante confinar la expresión de un producto genético deseado a un tipo particular de bacterias del ácido láctico a través de un método donde el gen que codifica el producto genético deseado tiene una mutación sin sentido que hace que el producto genético no está expresado. Mientras dicho replicón está presente en una célula huésped bacteriana de ácido láctico que incluye un gen supresor que codifica un producto capaz de restaurar la capacidad del gen mutado sin sentido de expresarse normalmente, el gen está expresado. No obstante, si el replicón que incluye el gen que codifica el producto genético deseado escapa de la célula huésped primaria a un segundo tipo de bacterias del ácido láctico que no tiene un gen supresor como se ha definido anteriormente, entonces el producto genético no estará expresado en el segundo tipo de células. 9 ES 2 185 669 T3 5 10 15 20 25 30 35 40 45 50 55 60 El método de confinar un replicón, como se define en la presente, el cual es particularmente interesante cuando una composición de cultivo mixto de dos o más cepas de bacterias del ácido láctico entre la cual un replicón que codifica un producto genético deseado puede ser libremente transferido, se usa p. ej. en la producción de un producto alimenticio tal como se ha definido anteriormente. Ası́, la expresión de un producto genético determinado puede ser confinado a una cepa bacteriana de ácido láctico en el cultivo mixto que incluye una multitud de cepas. Según la presente invención, existe también en otra forma de realización útil proporcionada, un método como se ha definido anteriormente para mantener la estabilidad de un replicón extracromosómico, el cual incluye un plásmido y un bacteriófago, en células huéspedes bacterianas del ácido láctico en un ambiente particular. En formas de realización adecuadas de la invención, las células huéspedes bacterianas de ácido láctico que albergan el replicón que debe mantenerse estable, tienen una mutación sin sentido en uno o más genes que confieren auxotrofı́a a las células, por lo que las células pierden su capacidad de crecer en el ambiente en cuestión debido a una carencia de una sustancia nutritiva esencial, la cual no puede ser sintetizada por las células mutantes sin sentido. Como ejemplo, la mutación sin sentido puede hacer que las células huéspedes pierdan la capacidad de crecer en un medio que no contiene los precursores para la sı́ntesis en las células de nucleótidos de purina. Dichos mutantes auxotróficos se designan también mutantes Pur≡ . La leche es dicho medio que no contiene precursores nucleótidos en cantidades suficientes para el crecimiento de mutantes Pur≡ . En consecuencia, las células huéspedes bacterianas de ácido láctico del mutante Pur≡ sin sentido no serán capaces de crecer en la leche si el replicón que debe mantenerse se pierde de las células. En consecuencia, el gen supresor del replicón extracromosómico funciona como un marcador selectivo para las células huéspedes bacterianas de ácido láctico. En el presente contexto, el término “marcador selectivo” se utiliza para designar un genotipo que convierte a las células bacterianas de ácido láctico incapaces de crecer si el replicón que debe mantenerse se pierde de las células. Según la presente invención, se contempla que puedan aislarse mutantes sin sentido auxotróficos, lo cual permite mantener estable un replicón extracromosómico en una bacteria del ácido láctico que crece en otros ambientes especı́ficos que incluyen productos vegetales, productos cárnicos, productos de panaderı́a, vinos, zumos de fruta, tracto gastrointestinal, cultivos forrajeros o desperdicios que deben ser ensilados por una bacteria del ácido láctico. En otro aspecto interesante, la presente invención concierne a un método, tal como se ha definido anteriormente, para controlar el número de células bacterianas del ácido láctico en un ambiente particular que permite el crecimiento de bacterias del ácido láctico. La regulación del promotor regulable puede proporcionarse como se ha definido anteriormente. En una forma de realización especı́fica del método anteriormente mencionado, el gen que codifica el supresor es regulado de modo que la expresión del gen aumenta haciendo que las células bacterianas de ácido láctico crezcan más lentamente, cesen su crecimiento o mueran. Dicho efecto puede p. ej. observarse cuando la mutación sin sentido está localizada en un gen que codifica un producto genético que inhibe el crecimiento de la célula. Como ejemplo de un producto genético que tiene un efecto inhibitorio en el crecimiento de bacterias del ácido láctico puede mencionarse un antibiótico bacteriostático o bactericida. El producto genético puede también ser un producto como una enzima lı́tica que incluye lisozima y lisinas de fago, la cual puede expresarse en la bacteria que será controlada en cantidades que inhiben el crecimiento de la célula o causan la muerte de las células. En otra forma de realización especı́fica del presente método para controlar el número de bacterias del ácido láctico, el gen que codifica el ARNt supresor es regulado de modo que disminuye o termina la expresión del gen. Como ejemplo, las bacterias del ácido láctico que deben ser controladas son bacterias que contienen un fago lisogénico y que tienen la mutación sin sentido localizada en un gen seleccionado de un gen que codifica un producto genético que inhibe la introducción del fago en su ciclo litico por lo que, cuando disminuye o termina la expresión del gen que codifica el supresor sin sentido, el fago entra en el ciclo lı́tico, provocando que las bacterias del ácido láctico mueran. En otro ejemplo, las bacterias contienen una mutación sin sentido que está localizada en un gen que codifica un producto genético como un gen implicado en la sı́ntesis del nucleótido, la expresión del cual es necesaria para el crecimiento de las bacterias, por lo que, cuando disminuye o termina la expresión del gen que codifica el supresor sin sentido, el producto genético requerido para el crecimiento de las bacterias ya no se expresa, provocando el cese del crecimiento de las células bacterianas del ácido láctico. Leyendas de las figuras La figura 1 ilustra la construcción de plásmido pFDi10. Los segmentos rellenos indican ADN de 10 ES 2 185 669 T3 pCI372 o pCI3340, ADN de segmentos abiertos de pVA891 y ADN de segmentos rotos aislado de pCI160; Hi: Hı́ndlll, E: EcoRI, N: Ncol, C: Clal, X: Xbal, B: BamHlll, P: Pvull, Seg.: Stul. La figura 2 ilustra la construcción del plásmido pAK58. 5 10 15 20 La figura 3 muestra una secuencia de ADN de 300 bp (SEC ID NO:10) que incluye un gen supresor ocre aislado de la cepa del supresor mutante FD100. Los promotores potenciales están indicados con ∗, que marca las posibles regiones -35 y #, que marca las posibles regiones -10 . Las flechas indican una repetición invertida que forma parte de un terminador potencial de la transcripción. Se espera que las bases subrayadas sean transcritas y modificadas post-transcripción, p. ej. mediante la adición de CCA al extremo 3’ para formar un ARNt activo. La iniciación de ADN Lactococcus en los plásmidos pFDi17 (nucleótido 121), pFDi18 (nucleótido 121) y PFDi19 (nucleótido 132) está indicada. La figura 4 muestra una secuencia de ADN de 300 bp (SEC ID NO:11) que incluye un gen supresor ámbar aislado de la cepa del supresor mutante NJ 1. Los promotores potenciales están indicados con ∗, que marca las regiones posibles -35 y #, que marca las posibles regiones -10. Las flechas indican una repetición invertida que forma parte de un terminador potencial de la transcripción. Se espera que las bases subrayadas sean transcritas y modificadas post-transcripción, p. ej. mediante la adición de CCA al extremo 3’ para formar un ARNt activo. La figura 5 (A y B) muestra la alineación del gen supresor FD100 (SEC ID NO:32) (lı́nea de fondo) con 20 genes ARNt-gIn (SEC ID NO:12 a SEC ID NO:31). La figura 6 ilustra la construcción de pFG1. 25 La figura 7 ilustra la construcción de pAK117. Ejemplo 1 30 Construcción de un plásmido transportador (PFDi10) que lleva mutaciones sin sentido en dos genes de resistencia antibiótica El plásmido pFDi10 fue construido a través de diferentes fases de selecciones y clonaciones. Los plásmidos implicados en estas fases están catalogados en la tabla 1 y las cepas usadas están catalogadas en la tabla 2. Las fases individuales se ilustran en la figura 1 y se describen con detalle posteriormente. 35 A continuación, las mutaciones sin sentido en los genes erm y cat se designan erm-am y cat-am solamente para indicar que las mutaciones están suprimidas por supresores ámbar en E.coli. No se realizó ningún otro intento para analizar el tipo de mutaciones sin sentido. TABLA 1 Usos de los plásmidos para la construcción de pFDi10 40 Plásmido Huésped Genes de resistencia antibiótica Referencia pVA891 E.coli cat, erm Macrina et al. 1983 pCI160 E.coli Tet, bla Hill et al. 1988 pCI372 E.coli/L. Lactis cat Hayes et al. 1990 pCI3340 E.coli/L. Lactis cat Hayes et al. 1990 pVA89erm-am E.coli cat, erm-am Este trabajo pFDi6 E.coli/L. Lactis cat, erm Este trabajo pFDi6cat-am E.coli/L. Lactis cat-am, erm Este trabajo pFDi81 E.coli/L. Lactis cat, tet Este trabajo pFDi9 E.coli/L. Lactis cat-am, tet Este trabajo PFDi10 E.coli/L. Lactis cat-am, erm-am, tet Este trabajo 45 50 55 60 11 ES 2 185 669 T3 TABLA 2 Cepas usadas en la construcción de pFDi10 5 10 15 Cepa Especies Genotipo relevante Referencia MG1363 Lactococcus lactis LE3O E. coli K12 mutD Silhavy et al 1984 R594 E. coli K12 sup◦ Campbell 1965 BR2024 E. coli K12 sup◦ Austin et al. 1983 BR2025 E. coli K12 supD Austin et al. 1983 BR2026 E. coli K12 supE Austin et al. 1983 BR2027 E. coli K12 supF Austin et al. 1983 Gasson 1983 20 25 30 35 40 45 50 55 60 1. Construcción de PVA891 erm-am La cepa mutágena LE30 fue usada para mutagenizar pVA891. Las células competentes de LE30 fueron preparadas inoculando LE30 en medio mı́nimo AB (Clark & Maaloe 1967) y permitiendo el crecimiento a 37◦ C para continuar hasta que el OD600 fue de 0,4. Las células fueron recogidas por centrifugado, hechas competentes y posteriormente transformadas con pVA891 como describe Sambrook et al., 1989. Los transformantes fueron seleccionados mediante la colocación en placas de ágar LB completadas con 10 µg/ml cloramfenicol. Las células de placas que contienen un total superior a 1000 colonias fueron agrupadas añadiendo 2 ml de caldo LB por placa y resuspendiendo las células usando una varilla de vidrio inclinada. Las células de la suspensión fueron recogidas por centrifugado y los plásmidos extraı́dos como se describe en Sambrook et al., 1989. Esta preparación del plásmido fue el stock de plásmido mutado de pVA891. La cepa R594 fue transformada con el stock mutado de pVA891 y transformantes CmR seleccionados en LB completados con 10 µg/ml cloramfenicol. Más de 5x103 colonias fueron agrupadas en el modo descrito anteriormente y este cultivo mixto fue enriquecido para los mutantes de eritromicina sensibles mediante un procedimiento similar al enriquecimiento para los mutantes auxotróficos descritos por Miller 1972. El enriquecimiento se realizó mediante la inoculación del cultivo mixto en 250 ml de LB completado con 250 µg/ml de eritromicina a un OD600 de 0,02. Después de una incubación de 2 horas a 37◦ C el OD aumentó a 0,1. En este punto se añadió ampicilina a una concentración de 50 µg/ml. El cultivo fue incubado adicionalmente a 37◦C durante 2 horas. En ese momento el OD600 disminuyó a 0,04. Las células fueron recogidas por centrifugado y lavadas en un medio mı́nimo de AB y finalmente resuspendidas en un medio mı́nimo AB. Las diluciones de células resuspendidas fueron depositadas en placas en LB + 10 µg/ml de cloramfenicol. Se seleccionaron 300 colonias resistentes al cloramfenicol por la sensibilidad a la eritromicina mediante el estriado en LB + 250 µg/ml de eritromicina. Entre las 300 colonias evaluadas, 113 fueron consideradas sensibles a la eritromicina. De estos 113 mutantes individuales, se prepararon cultivos de 24 horas en LB + 10 µg/ml de cloramfenicol. Una agrupación con 50 µl de cada cultivo fue usada para la preparación de plásmidos. Esta agrupación de plásmido fue transformada en BR2024, BR2025 y BR2026, respectivamente. Las células transformadas fueron depositadas en placas en LB + 10 µg/ml de cloramfenicol y en LB + 250 µg/ml de eritromicina. Los transformantes resistentes al cloramfenicol fueron obtenidos con las tres cepas mientras que los transformantes resistentes a la eritromicina se obtuvieron sólo con la cepa BR2026. Un transformante resistente a la eritromicina de BR2026 fue purificado estriando colonias únicas dos veces. De este transformante se aisló el plásmido pVA891 erm-am. Para verificar la presencia de una mutación ámbar, el plásmido purificado pVA891 erm-am fue transformado en BR2024, BR2025 y BR2026, respectivamente y los transformantes seleccionados en LB + 250 µg/ml de eritromicina y en LB + 10 µg/ml de cloramfenicol. Los números de los transformantes 12 ES 2 185 669 T3 obtenidos están catalogados en la tabla 3. De hecho, el resultado muestra que pVA891 erm-am lleva una mutación en el gen erm que puede suprimirse eficientemente por el supresor ámbar supE y con una eficiencia inferior por el supresor ámbar supD. TABLA 3 Número de transformantes con pVA891 erm-am suprimido 5 Cepas 10 sup◦ BR2024 cmR eriR 15 20 25 30 35 supD BR2025 eriR /cmR No transformado con ADN 2 9 VA891 erm-am 600 4 cmR eriR 2 1,5x6−2 supE BR2026 eriR /cmR 4 255 70 cmR eriR 0 0,3 400 eriR /cmR 1 400 1 2. Construcción de pFDi6 El pFDi6 es un plásmido capaz de duplicarse tanto en E. coli como en Lactococcus lactis. El pFDi6 lleva dos marcadores de resistencia antibiótica (cat y erm). Estos dos marcadores se expresan bien en ambos huéspedes. El pFDi6 fue construido para insertar el gen erm de pVA891 en el plásmido transportador pCI3340. Esto se realizó mediante la digestión de 1 µg de ADN pVA891 simultáneamente con las enzima de restricción hindlll, Clal y Pvull (Pvull fue incluido para aumentar la frecuencia del evento deseado, ya que Pvull no disocia el fragmento que lleva el gen erm). 1 µg de pCI3340 fue digerido con las enzimas de restricción Hindlll y Clal. Los plásmidos digeridos fueron mezclados, ligados y transformados en R594 como se describe en Sambrook et al., 1989. Los transformantes fueron seleccionados en LB + 10 µg/ml de cioramfenicol. Se examinaron 222 colonias resistentes al cioramfenicol para comprobar su resistencia a la eritromicina y 24 fueron consideradas resistentes a ambos antibióticos. El ADN del plásmido fue extraı́do de 10 de éstos y se descubrió que los 10 tenı́an un tamaño y una estructura idéntica. Uno de éstos fue mantenido como pFDi6. 3. Construcción de pFDi6cat-am 40 45 50 55 60 Un derivado de pFDi6 que lleva una mutación sin sentido en el gen cat fue construido por un método análogo al usado en la construcción de pVA891 erm-am. El pFDi6 fue transformado en la cepa mutágena LE30 y transformantes seleccionados en LB + 250 µg/ml de eritromicina. Más de 103 colonias fueron agrupadas y el ADN del plásmido extraı́do. Este stock de ADN mutado de pFDi6 fue transformado en R594 y transformantes seleccionados en LB + 250 g/ml de eritromicina. Más de 103 transformantes fueron agrupados y usados para inocular 250 ml de LB + 10 µg/ml de cloramfenicol en un OD600 de 0,03. Después del crecimiento a 37◦C durante 2 1/2 horas el OD600 aumentó a 0,360. En ese momento se añadió la ampicilina a una concentración final de 50 µg/ml y ésta fue incubada continuamente durante 2 horas adicionales. En ese momento el OD600 disminuyó a 0,09 y las células fueron recogidas por centrifugado, lavadas en medio mı́nimo AB y resuspendidas en un medio mı́nimo AB, y las diluciones fueron depositadas en placa en LB + 250 µg/ml de eritromicina. Se examinaron 500 colonias resistentes a la eritromicina para comprobar su sensibilidad al cioramfenicol y 44 resultaron sensibles al cioramfenicol. Un cultivo de 24 horas de cada uno de estos 44 mutantes fue preparado en LB + 250 µg/ml de eritromicina. El ADN del plásmido fue extraı́do de una mezcla de los 44 cultivos. Esta mezcla de plásmidos mutados fue transformada en BR2024, BR2025, BR2026 y BR2027, respectivamente. Los transformantes resistentes a la eritromicina fueron obtenidos con las cuatro cepas mientras que los transformantes resistentes al cioramfenicol sólo pudieron ser obtenidos en BR2025, BR2026 y BR2027. Uno de los transformantes resistentes al cioramfenicol de BR2025 fue purificado y se demostró que el plásmido extraı́do de este transformante llevaba pFDi6cat-am mediante la repetición de la transformación en BR2024, BR2025, BR2026 y BR2027, respectivamente. La mutación ámbar en pFDi6cat-am es suprimida por supD, supE y supF. 13 ES 2 185 669 T3 4. Construcción de pFDi81 5 El gen de resistencia a la tetraciclina de pCI160 fue elegido para la construcción de pFDi81 y posteriormente de pFDi9 y pFDi10, ya que este gen de resistencia a la tetraciclina es bien expresado en Lactococcus spp (Hill et al. 1988). 10 1 µg de cada uno de los plásmidos pCI160 y pCI372 fue digerido con Hindll. Los digeridos fueron mezclados, ligados y transformados en células competentes de R594 como se describe en Sambrook et al. 1989. Los transformantes fueron seleccionados mediante su colocación en placa sobre ágar LB complementado con 10 µg/ml de cloramfenicol. Se examinaron 200 colonias para comprobar su resistencia a la tetraciclina estriando sobre ágar LB complementado con 10 µg/ml de tetraciclina. Se descubrió que 8 colonias llevaban los plásmidos con el gen de resistencia a la tetraciclina insertado en pCI372. Uno de éstos fue mantenido y el plásmido designado como pFDi81. 15 5. Construcción de pFDi9 20 El pFDi9 es un plásmido de la misma estructura que pFDi81, pero que lleva el gen cat-am en lugar del gen cat natural. El fragmento EcoRI-Stul de pFDi81 que lleva el gen cat fue sustituido con el fragmento EcoRI-Stul de pFDi6cat-am para construir pFDi9. 1 µg de ADN pFDi81 fue digerido con EcoRI, Stul y Ncol (Ncol fue incluido para reducir la frecuencia de clones que llevan el gen cat natural). 1 µg de pFDi6cat-am fue dividido con Stul + EcoRI. Los dos digeridos fueron ligados y transformados en BR2026 (supE). Los transformantes fueron seleccionados de ágar LB complementado con 10 µg/ml de cloramfenicol y 10 µg/ml de tetraciclina. 25 Se obtuvieron más de 500 transformantes. Tres colonias fueron purificadas y el ADN del plásmido fue extraı́do. La transformación en R594 y BR2026 respectivamente demostró que los tres plásmidos llevaban el gen cat-am ası́ como que podı́an obtenerse transformantes resistentes al cloramfenicol en la cepa supE BR2026 y no en la cepa de no supresión R594. Los transformantes resistentes a la tetraciclina fueron obtenidos con ambas cepas. Uno de los tres plásmidos fue mantenido como pFDi9. 30 6. Construcción de PFDi10 El gen erm-am de pVA891 erm-am fue insertado en pFDi9 para dar pFDi10. 35 40 45 50 1 µg de pVA891 erm-am fue digerido con Xbal + BamH1 + EcoRI (EcoRI fue incluido para aumentar la frecuencia del evento deseado). 1 µg de pFDi9 fue digerido con Xbal + BamHI. Los dos digeridos fueron ligados y transformados en BR2027. Los transformantes fueron seleccionados en ágar LB complementados con 10 µg/ml de tetraciclina (LB-tet). 50 colonias fueron estriadas sobre placas de LB complementadas con 250 µg/ml de eritromicina (LB-eri) y sobre placas LB complementadas con 10 µg/ml de cloramfenicol (LB-cam). Se descubrió que 15 colonias eran resistentes a los tres antibióticos. El ADN del plásmido fue extraı́do de cuatro de estos transformantes resistentes triples y transformado en R594 y BR2026, respectivamente. Las células transformadas fueron depositadas en placas con LB-eri, LB-tet y LB-cam, respectivamente. Estos cuatro plásmidos dieron transformantes de tetR de ambas cepas. Se obtuvieron transformantes resistentes al cloramfenicol para los cuatro plásmidos sólo en la cepa BR2026 que mostraba que los cuatro plásmidos llevaban un gen cat-am incapaz de dar resistencia al cloramfenicol en la cepa de no supresión R594. Tres de los cuatro plásmidos anteriormente mencionados dieron transformantes resistentes a la eritromicina de BR2026, pero ninguno de R594, y uno de los cuatro plásmidos dio transformantes resistentes a la eritromicina de ambas cepas. El último plásmido fue descartado por que tenı́a una reversión del gen erm-am. Uno de los tres plásmidos que tiene genes erm-arm, cat-am y genes de tet fue mantenido como pFDi1O. 55 Ejemplo 2 Selección de cepas que suprimen sin sentidos de Lactococcus lactis usando PFDi 10 60 1. Transformación de PFDi10 en Lactococcus lactis MG1363 Las células competentes de MG1363 fueron preparadas y transformadas mediante electroporación 14 ES 2 185 669 T3 5 10 como describen Holo y Nes 1989. 10 µg de pFDi10 fueron precipitados con etanol y el granulado seco fue resuspendido en 10 µl de agua estéril destilada. Se añadieron 40 µl de células competentes de MG1363 y la mezcla fue sometida a electroporación a través de un pulsador BioRad de genes usando la composición de 25µF, 2kv, 200Ω. El tiempo constante obtenido fue de 4,8. Se añadieron 960 µl de caldo SGM17 enfriado con hielo y la mezcla fue mantenida en hielo durante 5 minutos. La mezcla fue transferida a un tubo con 2 ml de caldo SGM17 e incubada a 30◦C durante 2 horas antes de depositar las diluciones hechas en el caldo SGM17 en placas de ágar SGM17 complementadas con 10 µg/ml de tetraciclina. Se obtuvo una eficiencia de transformación de 2 x 105 transformantes/µg. Una colonia fue purificada reestriándola dos veces en placas de ágar GM17 complementadas con 10 µg/ml de tetraciclina. Se verificó que la cepa resultante FD73 contenı́a pFDi10 mediante la extracción del ADN del plásmido como describen Israelsen y Hansen 1993. El plásmido extraı́do de FD73 tenı́a un tamaño idéntico a pFDi10 y un modelo de restricción idéntico cuando se digerı́a con Xbal + BamHI. Una muestra de FD73 fue depositada el 20 Septiembre 1993 con DSM-Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH, Mascheroder Weg 1 b, D-38124 Braunschweig Alemania, bajo el número de acceso DSM 8557. 15 20 Un cultivo de FD73 (MG1363/pFDi10) fue preparado para inocular una única colonia de FD73 en 10 ml de GM17 complementada con 10 µg/ml de tetraciclina seguida por una incubación a 30◦ C durante 18 horas. De este cultivo de 24 horas, 0,2 ml fue colocado en placa en cada uno de los medios siguientes: GM17 + 10 µg/ml de tetraciclina + 5 µg/ml de cioramfenicol + 1 µg/ml de eritromicina; GM17 + 5 µg/ml de cioramfenicol; GM17 + 1 µg/ml de eritromicina. De una dilución 10−5 del cultivo de 24 horas, 0,1 ml fue depositado en placas en GM17 + 10 µg/ml de tetraciclina. Las placas fueron incubadas a 30◦C durante 42 horas. Los resultados se resumen en la tabla 4. TABLA 4 25 30 35 40 45 Recuento de células en un cultivo de 24 horas de FD73 Medio CFU/ml tet 1x109 cam 7x101 eri 7x100 tet+cam+eri 0 Los resultados de este experimento muestran que ni el gen cat-arm ni del gen erm-am de pFDi10 confieren resistencia antibiótica a MG1363, lo cual indica que Lactococcus lactis MG1363 no es una cepa que inherentemente suprime sin sentidos. Los genes naturales correspondientes se expresan en MG1363 cuando el plásmido pFDi6 da resistencia al cioramfenicol y eritromicina cuando está transformado en MG 1363. 2. Aislamiento de mutantes que suprimen sin sentidos de Lactococcus lactis El Lactococcus lactis FD73 fue mutado con EMS mediante el siguiente procedimiento: 50 55 (i) 9 ml de GM17 + 10 µg/ml de tetraciclina fueron inoculados con una única colonia de FD73 seguida por una incubación durante 18 horas a 30◦ C; (ii) 270 µl de EMS fueron añadidos al cultivo anteriormente mencionado y la incubación continuó durante 100 minutos a 30◦ C; (iii) cada uno de los 10 tubos de 9 ml de GM17 fueron inoculados con 0,9 ml del cultivo tratado EMS y posteriormente incubados a 30◦ C durante 18 horas; 60 (iv) de cada uno de los tubos anteriormente mencionados 0,2 ml fue depositado en placas GM17 complementadas con 5 µg/ml de cloramfenicol + 1 µg/ml de eritromicina. Las placas fueron incubadas a 30◦ C durante 42 horas; 15 ES 2 185 669 T3 (v) una colonia de cada una de las placas anteriormente mencionadas que contenı́an de 13 a 30 colonias fue purificada mediante estriación en GM17 complementadas con 10 µg/ml de tetraciclina; 5 (vi) la resistencia al cloramfenicol y la eritromicina fue verificada estriando en placas GM17 con eritromicina y cloramfenicol, respectivamente; 10 (vii) cinco colonias que mostraban resistencia a cam, erm y tet fueron seleccionadas para un análisis posterior. Las cinco resultaron posteriormente ser cepas que suprimen sin sentidos. En la descripción siguiente se presentan sólo los datos para uno de los cinco mutantes independientes. El mutante descrito a continuación se designa FD87. 3. Identificación de la localización del gen supresor en FD87 15 20 25 30 El ADN del plásmido fue extraı́do del cultivo de 24 horas de FD87 en GM17 complementado con 10 µg/ml de tetraciclina. El plásmido extraı́do tenı́a, como se esperaba, el mismo tamaño que pFDi10. Los modelos de restricción obtenidos con BamHI y Hindlll fueron también idénticos a aquellos obtenidos con pFDi10. El plásmido extraı́do de F087 fue transformado en E. coli R594 (no supresor) y los transformantes resistentes a la tetraciclina seleccionados. Los transformantes fueron posteriormente examinados para la resistencia a la eritromicina y al cloramfenicol. Se descubrió que todos los transformantes seleccionados eran sensibles a la eritromicina y al cloramfenicol. Este resultado muestra que los genes cat y erm del plásmido todavı́a llevan la mutación ámbar y, de ese modo, que el evento mutacional en FDB7 no fue una reversión simultánea de ambas mutaciones ámbar en pFDi10. El FD87 fue polimerizado para el plásmido pFDi10 mediante la inoculación de 9 ml de GM17 con 0,1 ml de una dilución 10−6 de un cultivo fresco de 24 horas de FDB7. Este cultivo fue incubado durante 24 horas a 30◦C antes de depositar en placas 0,1 ml de una dilución 10−6 de este segundo cultivo en una placa GM17. La placa fue incubada a 30◦ C durante 18 horas. En la placa aparecieron 169 colonias y de éstas, 100 colonias fueron examinadas para la resistencia a tet, cam y eri mediante estriado sobre GM17, GM17 + tet, GM17 + cam y GM17 + eri, respectivamente. 97 colonias fueron resistentes a los tres antibióticos y tres colonias fueron sensibles a los tres antibióticos. Una de las colonias sensibles fue repurificada mediante estriado en GM17 y esta cepa fue designada FD100. El análisis de ADN del plásmido de FD100 no reveló ningún plásmido. 35 40 45 Como la variante de plásmido polimerizada FD100 es sensible a cat y eri, se podrı́a concluir que el mutante FD87 no adquirió genes de resistencia antibióticos alternativos. Esto indica fuertemente que la mutación dio lugar a un gen del ARNt supresor. Se demostró que éste era el caso mediante la transformación de FD100 con pFDi10 y la selección de transformantes resistentes a la tetraciclina en el modo descrito anteriormente para MG1363. Se descubrió que los transformantes de FD100 eran resistentes a la eritromicina y al cloramfenicol, mostrando que la cepa FD100 es un cepa supresora de sin sentidos capaz de suprimir ambas mutaciones ámbar de pFDi10 mientras que la cepa madre MG1363 es incapaz de suprimir cualquiera de estas dos mutaciones ámbar de pFDi10. Una muestra de FD100 fue depositada el 20 Septiembre 1993 con DSM-Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH, Mascheroder Weg 1 b, D-38124 Braunschweig, Alemania, bajo el número de acceso DSM 8561. Ejemplo 3 50 Clonación del gen supresor de FD100 1. Construcción del vector de clonación pFDi3 El pFDi3 es un vector transportador de E.coli-Lactococcus lactis que lleva un gen cat y un gen erm-am. 55 El pFDi3 fue construido mediante la clonación del gen erm-am de pVA891 erm-am en pCI372. Esta clonación fue realizada mediante la digestión de ADN pVAB91 erm-am con Xbal + BamHI + Pvull y la digestión de pCI372 con Xbal + BamHI como se describe en el ejemplo 1 para la construcción de pFDi10. 60 Cuando el pFDi3 es transformado en Lactococcus láctico MG1363, sólo se obtienen los transformantes resistentes al cloramfenicol ya que MG1363 es incapaz de suprimir la mutación del erm-am. 16 ES 2 185 669 T3 Este vector se utilizó para la clonación del gen del ARNt supresor de FD100 en MG1363, porque se esperaba que la clonación del gen supresor en pFDi3 producirı́a un plásmido que expresarı́a la resistencia a la eritromicina en MG 1363. 5 10 15 20 25 30 35 2. Clonación del gen supresor El ADN cromosómico de FD100 fue obtenido a partir de 100m1 de un cultivo fresco de 24 horas de la cepa crecida en GM17. Las células fueron recogidas mediante centrifugado a 7000 x g durante 10 minutos. Las células fueron resuspendidas en el tampón TE y recogidas mediante centrifugado. El granulado de la célula fue congelado y mantenido a -20◦ C durante 18 horas. El granulado fue descongelado y resuspendido en 3 ml de tampón STET [8 % p/v de Sucrosa, 5 % v/v de TritonX100, 50 mM de EDTA (pH 8,0), 50 mM de TrisCI (pH 8,0)]. Se añadieron 750 µl de 10 mg/ml de lisozima y la mezcla fue incubada a 37◦C durante 60 minutos. Se añadieron 750 µl de 10 % p/v de SDS y la incubación continuó a 37◦C durante 30 minutos seguida por una incubación a 65◦ C durante 30 minutos. La solución fue extraı́da con fenol: cloroformo (1:1) tres veces. El ADN fue precipitado añadiendo NaCI a una concentración final de 0,5M y añadiendo un volumen igual de isopropanol. El ADN precipitado fue enrollado alrededor de un asa de inoculación, lavado tres veces en un 70 % de etanol y resuspendido en 500 µl de TE-tampón. Se determinó que la concentración de ADN era de 1,8 µg/µl midiendo la absorción a 260 nm. 3,5 µg de ADN cromosómico FD100 fue digerido con Hindlll. 12 µg de ADN pFDi3 fue digerido con Hindlll y tratado con Fosfatasa alcalina de intestino vacuno (Boehringer Mannheim) como se describe en Sambrook et al. 1989. Las dos mezclas fueron extraı́das con fenol y precipitadas con etanol y ligadas como se describe en Sambrook et al. 1989. El ADN ligado fue precipitado con etanol y disuelto en 20 µl de agua estéril destilada y sometido a electroporación en Lactococcus lactis MG1363 como describen Holo y Nes 1989. Los transformantes fueron seleccionados en SGM17 y complementados con 1 µg/ml de eritromicina y en SGM17 complementados con 5 µg/ml de cloramfenicol. Las eficiencias obtenidas de la transformación fueron 20 transformantes resistentes a la eritromicina por µg de ADN y más de 105 transformantes resistentes al cloramfenicol por µg de ADN. Se obtuvieron un total de 241 transformantes resistentes a la eritromicina. El ADN del plásmido fue obtenido a partir de 11 transformantes resistentes a la eritromicina. Los plásmidos fueron digeridos con Hindlll y analizados por electroforesis en un 0,8 % de gel de agarosa. Estos 11 plásmidos adquirieron un fragmento de 3,2 kb. Dos de los plásmidos adquirieron también otros fragmentos. 40 45 El análisis de restricción usando Hindll reveló la presencia de un sitio Hindll en el fragmento Hindlll de 3,2 kb. Los digeridos de Hindll también revelaron que los nueve clones que llevaban un único fragmento nuevo tenı́an todos el fragmento en la misma orientación relativa al vector. Para aislar también los clones con la orientación opuesta, aislamos el ADN del plásmido de 20 transformantes adicionales y encontramos los plásmidos con el fragmento de 3,2 kb de Hindlll en la orientación opuesta. El pFDi11 es uno de los plásmidos con sólo un nuevo fragmento de Hindlll de 3,2 kb y el pFDi12 es un plásmido con el mismo fragmento de 3,2 kb insertado en la orientación opuesta. Ejemplo 4A 50 Construcción de un plásmido con una mutación sin sentido ámbar en la región de la replicación Cuatro cebadores fueron sintetizados para su uso en la sı́ntesis de la reacción en cadena de la polimerasa del fragmento de ADN deseado. Éstos tuvieron las secuencias siguientes: 55 Cebador 1 (SEC ID NO:2): 5’ TGAATTCAGAGGTTTGATGACTTTGACC 3’ Cebador 4 (SEC ID NO:3): 5 ’GGAATTCCTAACAAAAGACTATTAACGC 3’ Ámbar 1 (SEC ID NO:4): 5’ AAACTCTAGAGCAAGTATTCG 3’ Ámbar 2 (SEC ID NO:5): 5’ CTTGCTCTAGAGTTTTGTAG 3’ 60 17 ES 2 185 669 T3 5 El cebador 1 corresponde a los nucleótidos 610-621 y el cebador 4 es complementario a los nucleótidos 2340-2361 de la región de replicación del plásmido de citrato (Jahns et al, 1991). Ambos cebadores contienen sitios EcoRI en su extremo ’5. Ámbar 1 y Ámbar 2 contienen cada uno dos desequilibrios que introducen un codón ámbar en la secuencia de codificación (SEC ID NO:7) y crean un sitio Xbal como se muestra a continuación en la tabla 5: TABLA 5 Introducción de una mutación ámbar en repB del plásmido de citrato pAK49 10 15 20 25 30 Los cebadores, ámbar 1 (SEC ID NO:4) y ámbar 2 (SEC ID NO:5) están en la parte superior e inferior en la secuencia repB (SEC ID NO:7). Los pares de bases desequilibradas están indicados mediante ∗. El sitio Xbal introducido está subrayado y el codón ámbar (TAG) introducido en el codón 159 de repB indicado. Se realizaron reacciones en cadena de polimerasa con el cebador 1 y el ámbar 2 y con el cebador 4 y el ámbar 1 usando como molde el pAK49, el plásmido de citrato de la cepa Lactococcus lactis subesp. lactis biovar diacetylactis DB1138 clonado en pVA891 (Macrina et al, 1983). Se obtuvieron fragmentos de 0,8 y 0,9 kb, respectivamente. Éstos fueron digeridos con EcoRI y Xbal, mezclados y clonados en pIC19H digerido de EcoRI (Marsh et al, 1984). 35 Quince de los dieciocho clones analizados tenı́an ambos fragmentos, unidos en el sitio Xbal para dar un fragmento de 1,7 kb EcoRI. El insertado de dicho clon fue movido a pVA891 (Macrina et al, 1983) como un fragmento Hindlll que produce pAK58, el cual contiene una mutación ámbar en repB. Esta construcción se ilustra en la Fig. 2. 40 La electroporación de FD100 y MG1363 con pAK58 produjo unos transformantes resistentes a la eritromicina con FD100 pero no con MG1363 (no obstante, véase ejemplo 4B). En consecuencia: pAK58 sólo se duplica en una cepa que suprime sin sentidos, confirmando la presencia de una mutación ámbar en repB. 45 Ejemplo 4B Aislamiento de una cepa MG 1363 que suprime la mutación ámbar en pAK58 50 55 La electroporación de pAK58 en MG1363 fue realizada siguiendo los procedimientos estándar siguientes (Holo & Nes, 1989). Como se esperaba, el pAK58 fue incapaz de duplicarse en MG1363. Bajo condiciones en las que un plásmido de citrato replicante produjo aproximadamente 200.000 transformantes, pAK58 produjo dos. Estas dos colonias fueron analizadas adicionalmente y se observó que una contenı́a pAK58 intacto, mientras que la otra no contenı́a ningún plásmido. La cepa con pAK58 fue denominada NJ1/PAK58 y guardada para un análisis adicional. Una muestra de NJ1/pAK58 fue depositada el 20 de Septiembre de 1993 con DSM-Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH, Mascheroder Weg 1b, D-38124 Braunschweig, Alemania, bajo el número de acceso DSM 8559. 60 El plásmido pAK58 fue polimerizado de NJ1/pAK58 mediante el crecimiento de 30 generaciones sin antibióticos. Tras la colocación en placas y la prueba, se descubrió que el 16 % de las colonias evaluadas estaban libres de pAK58. Una de éstas fue denominada NJ1 y adicionalmente caracterizada. La 18 ES 2 185 669 T3 5 electroporación de NJ1 con pAK58 produce una alta frecuencia de transformantes, indicando que NJ1 contiene una mutación del supresor que permite la replicación de pAK58 a pesar de la mutación ámbar. Este supresor no suprimió ni las mutaciones sin sentido en pFDi10 ni las mutaciones sin sentido en los derivados del fago ØMPC100 (véase ejemplo 5). De esta manera, este supresor es diferente al supresor FD100 descrito anteriormente. Ejemplo 4C Clonación del gen supresor de la mutación sin sentido de NJ1 10 15 20 El ADN genómico total fue aislado de NJ1 siguiendo los procedimientos estándar siguientes (Johansen & Kibenich, 1992) y parcialmente digerido con Sau3AI. Estos fragmentos fueron ligados con pAK58 digerido con Bg/II y se realizaron transformaciones de MG1363. Un fragmento de ADN de NJ1 que contiene el gen supresor permitirá la replicación de pAK58 en MG1363. Dicho clon fue obtenido y designado pAK85. El 5,1 kb de ADN de pAK85 fue subclonado en pCI372 (Hayes et al, 1990) en una variedad de modos y evaluado para la supresión de la mutación ámbar en pAK58 mediante la electroporación de MG1363 con una mezcla del derivado pC1372 para ser evaluada y pAK58. Un clon, pAKB9.1, contiene un fragmento de 1,1 kb de EcoRI-Xbal y tiene actividad supresora. La secuenciación de pAKS9. 1 se realizó y reveló que el gen supresor es ARNt con un anticodón que reconoce sólo codones ámbar. Ası́, el supresor es un supresor ámbar. La secuencia de ADN codificante para este ARNt es la siguiente (SEC ID NO: 1): 1 GGAGCCATGG CAGAGTGGTA ATGCAACGGA CTCTAAATCC GTCGAACCGT 51 GTAAAGCGGC GCAGGGGTTC AAATCCCCTT GACTCCTTA 25 30 35 Se espera que los tres pares de bases finales sean reemplazados con CCA post-transcripción. Las investigaciones de homologı́a del banco de datos de la secuencia de ADN EMBL, versión 34.0, revelaron que este ARNt es probablemente un ARNt de serina. La secuencia de ADN del gen ARNt natural en MG1363 fue determinada por PCR ampliando una secuencia de 450 bp que contiene este gen, seguido por la secuenciación del fragmento amplificado. Se confirmó que el gen ARNt mutado en NJ1 era un gen ARNt de serina con el anticodón 5’ CGA 3’ natural y 5’ CTA 3’ mutante. Una muestra de MG1363/pAK85 fue depositada el 20 de Septiembre de 1993 con DSM-Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH, MascheroderWeg 1 b, D-38124 Braunschweig, Alemania, bajo el número de acceso DSM 8558. Ejemplo 5 Aislamiento de mutantes sin sentido del prolato llamado fago lactococcal ØMPC100 40 45 50 55 60 ØMPC100 es un fago Lactococcus de prolato de la colección de fago de Chr. Hansens Laboratorium A/S. Este fago proporciona grandes placas (diámetro 2-4 mm) cuando se deposita en placas sobre MG1363. Este fago fue usado para el aislamiento de mutantes con mutaciones sin sentido en genes esenciales para el desarrollo del fago. Dichos mutantes serán incapaces de formar placas en MG1363, pero podrán crecer en FD100. Un stock de fago ØMPC100 con una concentración de 1,8x1010 unidades que forman placas (PFU)/ml fue usado para la preparación de un stock de fago mutado. La mutagénesis fue realizada usando un tratamiento de hidroxilamina del stock de fago como se describe en Silhavy et al. 1984. El tratamiento de hidroxilamina de ØMPC100 durante 22 horas redujo la concentración con el mismo orden de magnitud como se describe para los fagos de E. coli. El nivel de supervivencia de ØMPC100 fue de 3x 10−3 . Las diluciones del stock de fago mutado fueron depositadas por placas sobre la cepa FD100 mediante la mezcla de 10 µl de dilución del fago, 200 µl de un cultivo fresco de 24 horas de FD100 en GM17 complementado con 10mM de CaCl2 y 10 mM de MgSO4 y 3 mlM17 de ágar blando complementado con 10 mM de CaCl2 y 10 MM de MgSO4 . (El ágar blando fue derretido mediante ebullición y enfriado a 42◦C.) La mezcla fue vertida sobre una placa de ágar GM17 e incubada a 30◦C durante 18 horas. En la placa con fagos mutados se observaron placas de tamaño normal ası́ como placas pequeñas. 81 placas fueron aisladas para la eliminación de las tapas de ágar que contienen una placa con una pipeta estéril Pasteur. Las tapas de ágar fueron transferidas a 200 µl M17 + 10 mM CaCl2 + 10 MM MgSO4 y se dejó que los fagos se difundiesen fuera del ágar durante 2 horas a 8◦C. 19 ES 2 185 669 T3 15 µl de cada uno de los 81 stocks de fago fueron esparcidos sobre dos placas con campos de FD100 y MG1363, respectivamente. Se observó que tres de los 81 fagos crecı́an sólo en FD100 mientras que el resto crecı́an igualmente bien en ambas cepas. 5 10 Los tres fagos mutantes que fueron designados ØMPC100a12, ØMPC100a16 y ØMPC199a77 fueron purificados mediante su colocación en placas únicas dos veces en la cepa FD100. En FD100 el mutante ØMPC100a77 proporciona placas pequeñas y los dos otros proporcionan placas de tamaño normal. Una muestra de ØMPC100a77 fue depositada el 20 de Septiembre de 1993 con DSM-Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH, Mascheroder Weg 1 b, D-38124 Braunschweig, Alemania, bajo el número de acceso DSM 8562. Las frecuencias de reversión de tres mutantes del fago fueron determinadas por valoración de los stocks de fago en el huésped permisivo FD100 y en el huésped no permisivo MG1363. TABLA 6 15 Frecuencias de reversión de los mutantes sin sentido ØMPC100 Concentración (PFU/ml) en 20 25 30 35 Frecuencia de reversión FD100 MG 1363 ØMPC100a12 1,5x106 0,6x102 4x10−5 ØMPC100a16 4,8x106 9,102 2x10−4 ØMPC100a77 8,7x106 7x102 8x10−5 Para verificar que las mutaciones del fago son suprimidas en FD100 por el mismo supresor que la mutación erm-am utilizada para la selección de FD100, se examinó si los mutantes de fago eran capaces de crecer en MG1363 con pFDi11 o pFDi12. Se descubrió que la introducción de pFDi11 o pFDi12 convertı́a a MG1363 en un huésped permisivo para los mutantes de fago anteriormente mencionados. Se puede concluir que el supresor clonado en pFDi11 y pFDi12 es capaz de suprimir la mutación erm-am, ası́ como las mutaciones del fago. Esto prueba que los mutantes de fago son mutantes sin sentido. Ejemplo 6 40 Determinación del tamaño del gen supresor de FD100 Puesto que un gen ARNt supresor es inferior a 100 bp se propuso determinar la ubicación del gen supresor en el fragmento 3,2 kb insertado en pFDi11 y pFDi12. 45 De pFDi11 fue eliminado un total de 3,7 kb, eliminando ası́ el gen erm-am y 2,2 kb del fragmento insertado. El plásmido resultante pFDi13 conserva 1,0 kb del insertado original. De pFDi12 un total de 2,5 kb fue eliminado, eliminando el gen erm-am y 1,0 kb del fragmento insertado. El plásmido resultante pFDi14 conserva 2,2 kb del inserción original. 50 pFDi13 y pFDi14 fueron construidos mediante la digestión de pFDi11 y pFDi12 respectivamente con Hindll. Los ADNs digeridos fueron extraı́dos con fenol, precipitados y ligados (separadamente). Las mezclas de la ligadura fueron transformadas en R594 como se describe en Sambrook et al. 1989. La selección de los transformantes fue realizada en LB complementados con 10 µg/ml de cloramfenicol. 55 Tres transformantes de cada experimento fueron analizados. Los tres transformantes del experimento pFDi11 resultaron idénticos y uno de ellos fue conservado como pFDi13. Los tres del experimento pFDi12 resultaron idénticos y uno de ellos fue mantenido como pFDi14. pFDi13 y pFDi14 fueron transformados en MG1363 mediante electroporación seleccionando los transformantes resistentes al cloramfenicol. 60 La presencia o ausencia del gen supresor en pFDi13 y pFDi14 fue determinada probando la supresión del mutante de fago ØMPC100a12. Los resultados se presentan en la tabla 7. 20 ES 2 185 669 T3 TABLA 7 Supresión de mutaciones sin sentido de ØMPC100a12 Cepa 5 10 15 Crecimiento de ØMPC100a12 MG1363 - FD100 + MG 1363 pFDi11 + MG 1363 pFDi12 + MG 1363 pFDi13 - MG1363 pFDi14 + 20 Estos resultados muestran que el supresor está en el fragmento 2,2 kb de PFDi14. 25 30 35 40 45 Con el objetivo de determinar la ubicación del gen supresor, incluso se hicieron eliminaciones más precisas en el plásmido pFDi14 usando el sistema Erase-a-Base (borra una base) de Promega. El sistema Erase-a-base genera eliminaciones unidireccionales en un plásmido usando Exonucleasa III. Las eliminaciones son forzadas a ser unidireccionales linealizando el plásmido con dos enzimas de restricción de las cuales una está generando preponderancias 3’ (Exonuoleasa III resistente). 5 µg de pFDi14 ADN fueron digeridos con Sacl (preponderancias 3’ de Exonucleasa III resistente) y BamHl (preponderancias 5’ de Exonucleasa III sensible). El tratamiento con Exonucleasa III a 30◦C, tomando muestras con intervalos de 1 minuto entre de 1 a 15 minutos después de la adición de Exonucleasa III, el tratamiento con nucleasa S1, el tratamiento con el fragmento Klenow de polimerasa ADN y la ligadura, se realizaron exactamente como se describe en el manual Erase-a-base de Promega. Cada una de las muestras ligadas fueron transformadas en Lactococcus lactis MG1363 por electroporación. Los transformantes fueron seleccionados en ágar SGM17 complementado con 10 µg/ml de cloramfenicol. Se obtuvieron transformantes de cada muestra. Se evaluaron 20 colonias de cada una de las últimas cinco muestras (las cuales supuestamente llevaban las eliminaciones más grandes) para la supresión de ØMPC100a12. De las 100 colonias evaluadas sólo cinco conservaron el supresor (2 de la muestra de 12 minutos, 2 de la muestra de 14 minutos y 1 de la muestra de 15 minutos). El ADN del plásmido fue extraı́do de diferentes transformadores supresores y no supresores. Los dos plásmidos más pequeños que todavı́a tenı́an el supresor fueron nombrados pFDi17 y pFDi18. El plásmido más grande (aunque más pequeño que pFDi17 y pFDi18) que no llevaba un supresor fue pFDi19. La longitud de la inserción cromosómica resultó ser 266 bp para pFDi17 y pFDi18. El fragmento conservado en pFDi19 fue 11 bp más corto (véase ejemplo 7). 50 Ejemplo 7 Secuencia nucleótida del gen del ARNt supresor de FD100 55 60 La secuencia de ADN de la inserción derivada de FD100 en cada uno de los tres plásmidos pFDi17, pFDi18 y pFDi19 fue determinada usando el equipo de secuencias de USB, Cleveland, Ohio, EEUU. Se siguió el protocolo provisto con el equipo para la secuenciación de plásmidos bicatenarios. La secuencia de ambas cadenas fue determinada para cada plásmido. La secuencia de una cadena fue determinada usando el cebador 5’ GCTAGAGTAAGTAGTT 3’ (cebador # 1206 de New England Biolabs, Beverly, Massachusetts, USA) (SEC ID NO:8), la secuencia de la otra cadena fue determinada usando el cebador 5’ CCTTTACCTTGTCTACAAACC 3’ (SEC ID NO:9). pFDi17 y pFDi18 contenı́an fragmentos con la misma longitud y secuencia. pFDi19, que no expresó 21 ES 2 185 669 T3 una actividad supresora, contenı́a un fragmento que era 11 bp más corto que el fragmento en pFDi17 y pFDi18 (cf. Figura 3). 5 La secuencia del fragmento que lleva el gen supresor fue comparada con todas las secuencias nucleótidas presentes en la base de datos EMBL, versión 34, usando el programa Fasta del paquete programa GCG (Devereux et al. 1984, Nucleic Acids Research, 12, 387-395). La secuencia que muestra la máxima homologı́a con la secuencia de la figura 3 fue la secuencia de ARNt-gln de Bacillus subtilis. Otros 19 genes del ARNt-gin de bacterias, cloroplastos y mitocondrias también mostraron homologı́as extremadamente altas. 10 15 La alineación del gen supresor FD100 con 20 genes del ARNt-gln se presenta en la figura 5. Esto muestra que el gen supresor es un gen del ARNt-gln. En la posición del anticodon, el supresor FDi100 ARNt-gLn tiene el triplete 3’ ATT 5 en lugar de los anticodones gln usuales 3’ GTT 5 o 3’ GTC 5’. Esto muestra que el supresor ARNt-gin es un supresor ocre que reconoce el codón de parada 5’ UAG 3’ como un codón gln. No obstante, este supresor ocre también reconoce el codón de parada ámbar 5 UAG 3’ (cf. ejemplo 5) probablemente debido al balanceo del “apareamiento de bases” en la tercera posición del codón. 20 La secuenciación del gen natural ha revelado posteriormente que este gen es un gen del ARNt-gln con el anticodón 3’ GTT 5’. La secuenciación de este alelo del ARNt-gln de los mutantes supresores descritos en el ejemplo 2 revelaron que no todos los mutantes supresores eran de este tipo. En consecuencia se espera que más análisis de los otros mutantes revele los supresores con otras especificidades. 25 Los 11 pares de bases extra en pFDi17 y pFDi18 comparados con pFDi19 contienen una secuencia que se parece a la parte -35 de un promotor de consenso. El gen supresor de pFDi19 no está expresado debido probablemente al defecto de un promotor. El plásmido pFDi19 puede supuestamente ser usado para construir los genes supresores con una expresión regulada o alterada. Ejemplo 8 30 Aislamiento de mutantes auxotróficos de purina y supresión de dos mutaciones en Lactococcus lactis localizadas en genes pur por un gen del ARNt supresor 35 40 Los mutantes auxotróficos de Lactococcus lactis de purina fueron aislados. La introducción de un gen del ARNt supresor en mutantes Lactococcus lactis pur dio como resultado prototrofı́a. En general, la sı́ntesis de novo de nucleótidos de purina de precursores pequeños requiere 10 reacciones enzimáticas, dando lugar a un monofosfato de inosina (IMP). El IMP se usa en la sı́ntesis de AMP y GMP. Las bases de purina, las cuales se originan intracelularmente o a partir de fuentes exógenas, y de nucleosidos son convertidas en nucleótidos por medio de vı́as de salvamento que han demostrado ser diferentes entre organismos diferentes (para revisiones véase: Nygaard 1983; Neuhard y Nygaard 1987). Apenas se conoce nada sobre el metabolismo de la purina en la bacteria gram-positivo anaeróbica Lactococcus lactis (Nilsson y Lauridsen, 1992). A continuación se describe el aislamiento de mutantes auxotróficos de purina y cómo éstos pueden utilizarse en combinación con genes supresores. 45 1. Materiales y métodos 1a. Cepas bacterianas y medios usados 50 55 La cepa sin plásmido Lactococcus lactis MG1363 (Gasson, 1983) fue crecida en el medio M17 (Oxoid) o en un medio definido con la misma composición como medio mı́nimo tamponado con fosfato de Clark y Maaløe (1967) excepto que el NaCI de este medio fue reemplazado por el mismo peso de acetato sódico (DN-medio). Como fuente de carbono en el medio M17 o medio DN se utilizó 0,5 % de glucosa. Para los objetivos de la selección, se utilizó 5 mg/L de medio de cloramfenicol. Se añadieron compuestos de purina como suplementos cuando resultaba apropiado en las siguientes concentraciones: adenina, hipoxantina, guanina y xantina, 15 mg/ml; adenosina, inosina y guanosina 30 mg/l. 1b. Aislamiento de cepas auxotróficas de purina Lactococcus lactis 60 El MG1363 fue cultivado durante 24 horas en el medio M17. Se añadió 1/33 vol de sulfonato de metano etı́lico (EMS, Merck) y el cultivo fue incubado adicionalmente durante 2 horas y media a 30◦ C. El cultivo fue dividido y diluido 50 veces en el medio DN fresco con hipoxantina para dar cinco cultivos que 22 ES 2 185 669 T3 5 posteriormente crecieron durante 24 horas. El medio DN con hipoxantina fue inoculado con un cultivo mutado a 2x107 bacteria/ml y crecido a 8x107 bacteria/ml. Las células fueron recogidas, lavadas dos veces en el medio DN sin hipoxantina y resuspendido a 4x106 bacteria/ml en el medio DN e incubado durante 2 horas a 30◦ C. Se añadió ampicilina a una concentración final de 100 µg/ml y el cultivo fue adicionalmente incubado durante 5 horas. Las células fueron recogidas por filtración (Millipore, tamaño 0,2 µm), lavadas 3 veces con el medio DN y suspendidas en 1/10 vol del medio DN con hipoxantina. 50 µl de cultivo (2x104 posible bacteria/ml) fueron extendidos sobre placas de ágar DN que contenı́an hipoxantina e incubados durante 24 horas. 582 colonias de cada cultivo fueron examinadas para el crecimiento con y sin hipoxantina en placas de ágar del medio DN. 10 1c. Manipulación del ADN 15 El ADN del plásmido Lactococcus lactis fue aislado según el método de Johansen y Kibenich (1992). EL Lactococcus lactis fue transformado por electroporación como es recomendado por Holo y Nes (1989). Los plásmidos de Lactococcus lactis pFDi17 y pFDi19 fueron usados en el modo descrito anteriormente . 2. Aislamiento de mutantes auxótrofos de purina 20 Cinco mutantes (DN207-211) de la cepa MG1363 con un fenotipo auxotrófico de purina fueron aislados en el modo descrito anteriormente en materiales y métodos. Exogenamente proporcionó adenina, hipoxantina, xantina, guanina o adenosina de ribonucleosidos, inosina y guanosina, crecimiento restaurado de DN207-211. 3. Supresión de mutaciones pur en DN207-211 por el gen del ARNt supresor 25 30 35 40 45 50 Los dos plásmidos pFDi17 (sup+), en los que se expresa un supresor ocre, y pFDi19 (sup-), el cual no expresa un supresor, fueron transformados en las cepas DN207-DN211, seguido por la selección por su resistencia al cloramfenicol en placas de ágar M17. Los transformantes fueron examinados para comprobar su crecimiento con y sin hipoxantina en las placas de ágar del medio DN . El plásmido pFDi17 (sup+) pudo transformar DN209 y DN210 en Pur+ , mientras que el pFDi19 (sup-) no pudo. En todas las otras cepas ninguno de los plásmidos pudieron transformar las células en Pur+ . Esto demuestra que las mutaciones que causan los fenotipos auxotróficos de purina en DN209 y DN21O fueron mutaciones sin sentido. Estos experimentos han demostrado que es posible aislar mutantes auxotróficos en Lactococcus lactis (aquı́ los mutantes auxotróficos de purina se utilizan como un sistema modelo) y examinar éstos para mutaciones sin sentido que pueden ser restauradas mediante la introducción de genes supresores en las células. La importancia de estas conclusiones es que dicho sistema puede utilizarse para el mantenimiento de plásmidos (como pFDi17) p. ej. durante la fermentación en medios que no soportan el crecimiento de mutantes auxotróficos a menos que la auxotrofı́a sea restaurada por un plásmido que contiene un gen supresor. La ventaja puede ser el usar sólo uno y el mismo gen supresor para la supresión de las mutaciones sin sentido ámbar/ocre localizadas en una variedad de genes diferentes de cepas de Lactococcus lactis. El único y mismo gen supresor puede también utilizarse para controlar la expresión de varios productos genéticos, como en el ejemplo con los productos genéticos pur en las cepas DN209 y DN210. En estas cepas, los productos genéticos pur sólo se expresan si el gen supresor está introducido en las células. Una muestra de DN209/pFDi17 fue depositada el 20 de Septiembre de 1993 con DSM-Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH, Mascheroder Weg 1 b, D-38124 Braunschweig, Alemania, bajo el número de acceso DSM 8560. 4. Estabilidad de pFDi17 en la cepa Lactococcus lactis DN209 55 Los cultivos de la cepa Lactococcus lactis DN209 con pFDi17 (sup+) o pFDi19 (sup-) fueron cultivados durante aproximadamente 100 generaciones en 9,5 % de leche LAB (9,5 % de leche desnatada en polvo en agua) con 0,5 % de ácidos casamino libres de purina como el medio basal. Un cultivo de DN209/pFDi19 y un cultivo de DN209/pFDi17 fueron cultivados en el medio basal completado con 15 mg/l de hipoxantina como fuente de purina, y un cultivo de DN209/pFDi17 fue cultivado solamente en el medio basal. 60 El DN209/pFDi19 no puede crecer en leche que no contenga una fuente de purina, pero añadiéndole una fuente de purina es posible observar pérdidas del plásmido durante el crecimiento. El DN209/pFDi17 23 ES 2 185 669 T3 fue capaz de crecer en el medio basal complementado con la fuente de purina ası́ como en el medio basal sin dicha complementa ción. Bajo estas condiciones experimentales, no se observó ninguna pérdida de plásmido en ninguno de los cultivos del DN209/pFDi17 en el cultivo DN209/pFDi19. 5 Ejemplo 9 Estrategia para la provisión de Leuconostoc spp que suprimen no sentidos 10 15 20 25 Se recomienda que se proporcione Leuconostoc spp que incluya un gen que codifica el ARNt supresor llevando a cabo los experimentos siguientes: En una primera fase se determina cuál de los replicones usados de forma habitual pueden utilizarse para transformar un Leuconostoc spp. que incluye Leuconostoc lactis y Leuconostoc cremoris p. ej. mediante electroporación de células competentes. Los replicones que deben evaluarse incluyen el replicón plásmido de citrato (pKR46), el replicón pCI3O5 (pCI3340) y el replicón pSH71 (pNZ18). Si el replicón plásmido de citrato funciona, puede utilizarse el pAK58 para aislar los supresores; si el replicón pCI305 funciona, puede utilizarse pFDi10 para seleccionar los supresores. Si ninguno de los replicones sugeridos funciona, debe construirse una nueva selección de plásmido p. ej. como se expone a continuación. El gen de resistencia al cloramfenicol de pNZ18 es eliminado y reemplazado con el gen de resistencia a la tetraciclina y los marcadores eri-am y cam-am de pFDi10. Se considera que el modo más fácil de conseguir esto es cionando 3,2 kb de fragmento Bg/ll de pNZ18, el cual contiene el origen de la replicación, en el sitio Bg/ll o BamHI de pFDi10. Esto requiere que los orı́genes de pC1305 y pSH71 puedan coexistir en el mismo plásmido. Si éste no es el caso, el replicón pCI305 puede desactivarse mediante digestión con Pacl, descarga de los extremos con polimerasa de ADN y ligadura. Esto introducirá un desplazamiento del marco de lectura de -2 en el gen repB. 30 En una fase posterior, un Leuconostoc spp supresor ámbar puede ser seleccionado introduciendo el plásmido nuevo en p. ej. Leuconostoc lactis DB1164 o Leuconostoc cremoris DB 1165 y seleccionando por la resistencia a la tetraciclina, seguido por la selección de mutantes que son simultáneamente resistentes a la eritromicina y al cloramfenicol (siguiendo la mutagénesis si es necesario), polimerizando cualquier cepa interesante de su plásmido y confirmando que la resistencia antibiótica se ha perdido y reintroduciendo el plásmido de la selección y confirmando que se ha recuperado la resistencia antibiótica. Esto sólo ocurre si se está produciendo la supresión. 35 Ejemplo 10 Construcción de pFG1, un vector de clonación de prado alimenticio 1. Introducción 40 45 50 55 60 Con el objetivo de usar microorganismos genéticamente manipulados en productos alimenticios se prefieren vectores derivados totalmente del organismo que se manipulará. Un vector útil contiene una región de replicación, un marcador seleccionable y un sitio de clonación múltiple que permite la inserción de genes deseables. Además, debe ser lo suficientemente pequeño como para permitir la inserción del ADN deseado sin dificultad. Un vector de clonación de grado alimenticio, pFG1 que se replica en bacterias del ácido láctico fue construido basado totalmente en secuencias de ADN de Lactococcus, y secuencias sintéticas. El pFG1 contiene la región de replicación del plásmido de citrato Lactococcus lactis subesp. lactis biovar diacetylactis, el gen del ARNt supresor ocre de la Cepa L. lactis FD100 que permite la selección de transformantes de Lactococcus lactis DN209 (ejemplo 8) y un polienlace sintético con 11 sitios de restricción únicos, idénticos a los encontrados en el vector pIC19R (Marsh et al., 1984). La utilidad de pFG1 fue demostrada mediante la clonación del gen pepN de Lactococcus lactis ssp. cremoris Wg2 (Strømann, 1992) para obtener el plásmido pFG2. Una cepa con pFG2 contiene 4-5 veces más actividad de aminopeptidasa de lisina que la misma cepa con sólo pFG1, y una cepa que lleva pFG2 es el primer elemento en una nueva lı́nea de cultivos de bacterias genéticamente manipulados para el control del sabor del ácido láctico. 2. Cepas bacterianas, plásmidos y medios El MS1363 es un derivado libre de plásmido de la cepa NCDO 712 Lactococcus lactis (Gasson, 1983). 24 ES 2 185 669 T3 El FD100 es un mutante de MS1363 que contiene un supresor ocre. El DN209 es un auxótrofo de purina de MG1363, el cual puede ser suprimido por el supresor ocre. Wg2 es una cepa natural de Lactococcus lactis ssp. cremoris. En algunas fases de la clonación se utilizó Escherichia coli DH5α. 5 10 15 20 El vector de clonación pICI9H [resistente a la ampicilina; AmpR ] (Marsh et al., 1984) fue usado en todo el proceso. A continuación, se describe la construcción del vector de grado alimenticio y la clonación del gen pepN. Las cepas Lactococcus fueron cultivadas a 30◦ C en un medio mı́nimo (receta 053 del departamento de genética, Laboratorio de Chr. Hansen) con un 1 % de M17. Las cepas de E. coli fueron cultivadas en un medio de LB a 37◦ C. Se usaron 50 µl/ml de ampicilina. La actividad de la aminopeptidasa de lisina fue evaluada usando el procedimiento estándar del Laboratorio de Chr. Hansen (procedimiento analı́tico P-019). 3. Preparaciones de plásmidos y transformaciones El ADN del plásmido para la secuenciación y electroporación fue preparado con el equipo de plásmido Qiagen (Diagen, Düsseldorf, Alemania). Las preparaciones plásmidas a pequeña escala de Lactococcus fueron realizadas según la receta 039 del departamento de genética, laboratorio de Chr. Hansen. Los plásmidos fueron introducidos en Lactococcus por electroporación de células competentes cultivadas en glicina (Holo y Nes, 1989, receta 018 del departamento de genética, laboratorio de Chr. Hansen). 4. Construcción del vector de clonación de grado alimenticio PFG1 4.1. Clonación de la región de replicación del plásmido de citrato 25 30 La reacción en cadena de la polimerasa (PCR) se realizó con dos cebadores diseñados para ampliar toda la región de replicación del plásmido de citrato como un fragmento de 1,7 kb . Los cebadores tenı́an sitios EcoRi en el extremo 5’ y el fragmento resultante fue clonado en pIC19H para dar pKR41 (Fig. 6). Este clon se ha descrito previamente (Pedersen et al., 1994). El replicón incluye el origen de la replicación y el gen repB del plásmido de citrato de L. lactis subesp. lactis biovar diacetilactis. Las secuencias flanqueantes de un total de 300 bp del mismo plásmido están también incluidas al ser dos enlaces sintéticos que contribuyen con un total de 6 bp a este fragmento. 4.2 Clonación del gen supresor ocre 35 El marcador seleccionable es el gen supB de la cepa L. lactis FD100. Este fragmento de ADN es de 208 bp y tiene una secuencia idéntica a la encontrada en FD100 excepto por la presencia de dos enlaces que contribuyen con un total de 6 bp. 40 45 50 55 El gen supresor ocre de la cepa FD100 ha sido clonado y está contenido en un plásmido llamado pFDi18 (ejemplo 6). El PCR que usa dos cebadores (Ocre-1 CGAATTCATAAATGCTTTCCCCTATTC (SEC ID NO:33); y Ocre2, CGAATTCTTGAAATTTATGAGGGTTTTTGG (SEC ID NO:34)) en pFDi18 dio como resultado un fragmento EcoRi de 208 bp que contiene el gen supresor ocre. Este fragmento fue clonado en pIC19H para proporcionar pAK95 (Fig. 6). 4.3 Combinación de la región de replicación con el gen supresor Los plásmidos pKR41 y pAK95 fueron digeridos con EcoRI, mezclados, ligados y usados para electroporar DN209, seleccionando el medio mı́nimo. Los transformantes con plásmidos que contienen al menos la región de replicación del plásmido de citrato y el gen supresor formarán colonias. Algunos plásmidos recombinantes también contienen pIC19H. Las colonias fueron raspadas de las placas de selección y los plásmidos extraı́dos. Los plásmidos en esta agrupación que contenı́an pIC19H fueron obtenidos mediante la transfomación de DH5α, seleccionando las colonias ampR. El análisis de los plásmidos de siete transformantes ampR reveló que todos tenı́an pIC19H, el fragmento EcoRi de 1,7 kb de pKR41 y el fragmento EcoRi de 208 bp de pAK95. Este hecho era previsible, ya que la doble selección usada sólo da plásmidos que contienen los tres fragmentos. Un clon fue conservado como pAK102 (Fig. 6). 4.4. Eliminación de los componentes sin grado alimenticio de pAK102 60 La fase final en la construcción del vector de clonación de grado alimenticio fue la eliminación de pAK102 de todos los pIC19H excepto para el sitio de clonación múltiple. Esto se realizó mediante la 25 ES 2 185 669 T3 digestión con Hindlll, auto ligando y electroporizando DN209 en el medio mı́nimo. Todas las colonias analizadas tenı́an el plásmido deseado. Una cepa fue conservada y depositada en el conjunto del cultivo como CHCC3061. El plásmido contenido en esta cepa fue denominado pFG1 y es el vector de grado alimenticio. El pFG1 tenı́a un tamaño total de 2003 bp. 5 El sitio de clonación múltiple (polienlace) es idéntico al que se encuentra en el vector llamado pIC19R (Marsh et al., 1984) y se muestra abajo (secuencia ID NO:35). El polienlace es de 69 bp y fue sintetizado totalmente in vitro por Marsh et al. (1984). Todos los sitios son únicos excepto para EcoRI y Clal. 10 15 20 25 30 El marcador seleccionable solamente puede, hasta ahora, utilizarse en DN209, el cual es un derivado de MG1363. El MG1363 es un derivado sin plásmido de NCD0712 y es, por consiguiente, Lac - y Prt-. Con el objetivo de usar pFG1 en otras cepas bacterianas de ácido láctico, se necesitarán mutaciones suprimibles por el supresor ocre. Las mutaciones preferidas estarán en los genes en la biosı́ntesis de purina o pirimidina, ya que la leche no contiene cantidades suficientes de estos compuestos para soportar el crecimiento de dichos mutantes, haciendo de esta manera de la leche un medio selectivo para los mutantes. Dichos mutantes pueden ser aislados por mutagénesis y técnicas de enriquecimiento habitualmente utilizadas en el departamento de genética del laboratorio de Chr. Hansens. Los plásmidos construidos usando DN209 pueden posteriormente transferirse de manera fácil a estos mutantes, dando lugar a nuevas cepas bacterianas de ácido láctico genéticamente manipuladas, las cuales incluyen cepas útiles de Lactococcus para una variedad de cultivos. A continuación se muestra la secuencia de ADN de pFG1 (secuencia ID NO:36): 35 40 45 50 55 60 26 ES 2 185 669 T3 5 10 15 20 25 30 35 Una cepa de DN\209/pFG1 fue depositada el 6 de mayo de 1994 bajo el No. DSM 9190 con DSMDeutsche SammIung von Mikroorganismen und Zelilkulturen GmbH, Mascheroder Wog 1b, D-38124 Braunschweig, Alemania. Ejemplo 11 Demostración de la utilidad del PFG1 como vector de cionación del grado alimenticio 40 45 50 Un fragmento de 3,5 kb BamHI/Sacl que contiene el gen pepN entero (también designado como el gen lap), el cual codifica una lisina-aminopeptidasa, fue extraı́do del plásmido pST03 (Strøman, 1992) en el sitio de cionación múltiple de pFG1 para producir un plásmido designado pFG2. La cepa transformada de DN209/pFG2 fue añadida al conjunto del cultivo del laboratorio de Chr. Hansen bajo el nombre CHCC3062 y depositada el 6 de mayo de 1994 bajo el No. DSM 9191 con DSM-Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH, Mascheroder Weg 1b, D-38124 Braunschweig,Alemania. Los extractos de las células libres fueron obtenidos a partir de DN209/pFG1 y DN209/pFG2 y analizados para la lisina-aminopeptidasa. La actividad enzı́rnática en DN209/pFG2 fue una proteı́na 228,3 nmoles/min/mg que era 4-5 veces más alta que para DN209/pFG1 (proteı́na 48,7 nmoles/min/mg). De esta manera, la introducción de copias extra del gen pepN en DN209 provoca el aumento de la expresión de dicho gen. Ejemplo 12 55 60 Estabilidad de plásmidos derivados de PFG en leche El gen de la β-galactosidasa de Leuconostoc mesenteroides subesp. cremoris fue insertado en pFG1 para producir pFG3. Este gen se obtuvo de un cion denominado pSB1 (Johansen y Kibenich, 1992a), el cual según el análisis de la secuencia de ADN contiene los genes lacL y lacM (datos no publicados). Las células que contienen este plásmido dan colonias azules en las placas que contienen X-gal mientras que las células sin plásmidos dan colonias blancas. Éste es un método simple para detectar la pérdida de plásmido y permite la filtración de muchas colonias para determinar la estabilidad de este plásmido. 27 ES 2 185 669 T3 DN209/pFG3 fue cultivado en GM17 o en leche y depositado varias veces en placas que contenı́an Xgal. La leche fue complementada con glucosa y ácidos de casamino porque DN209 está libre de plásmidos y, por lo tanto, de Lac− y Prt−. El porcentaje de colonias blancas se determinó en varios momentos y se presenta a continuación: 5 Colonias blancas Leche + glucosa + ácidos de casamino GM17 0 generaciones 0,1 % 0,1 % 10 generaciones 0,4 % 0,5 % 20 generaciones 0,1 % 13,1 % 30 generaciones 0,1 % 8,8 % 40 generaciones 0,2 % 3,4 % 10 15 20 25 Estos resultados demuestran claramente que la leche es un medio selectivo para las cepas que contienen derivados de pFG1, en este caso pFG3. También se obtuvo una estabilidad satisfactoria con GM17. Se cree que el descenso en porcentaje de las colonias blancas después de 20 generaciones es debido a la acumulación de variantes de DN209/pFG3 más rápidas en el crecimiento. Ejemplo 13 Aislamiento y caracterización de variantes de crecimiento más rápido de DN209/pFG1 30 35 40 Durante el curso de los estudios de estabilidad con DN209/pFG3 como se describe en el ejemplo 12 anteriormente mencionado, se constató que las variantes de crecimiento rápido de la cepa parecı́an acumularse. El análisis de estas variantes reveló una mutación en el gen estructural ARNt que podrı́a desestabilizar el ARNt resultante en la expresión reducida del gen supresor. La evaluación de DN209/pFG1 reveló la misma acumulación de variantes de crecimiento rápido. El DN209/pFG1 fue cultivado 45 generaciones en GM17 y posteriormente fue depositado en placas GM17. Las colonias grandes fueron introducidas en el medio mı́nimo para identificar aquellas que habı́an retenido pFG1. Doce mutantes fueron aislados de un único cultivo. El análisis de plásmido reveló que todos tenı́an pFG1 y que un mutante (#12) tenı́a un número de copia del plásmido reducido. El análisis de la secuencia de ADN del gen ARNt supresor reveló tres clases de mutantes. Una clase no tenı́a alteraciones claras. Una clase tenı́a una transversión de GC a CG en la región del promotor mientras que otra tenı́a una transición de TA a CG (SEC ID NO:37). Ambas se encuentran en o cerca de la región destinada a estar implicada en la respuesta rigurosa (Ogasawara et al., 1983, Ni1sson y Johansen, 1994) y se esperarı́a que disminuyesen la expresión del gen supresor. A continuación se ilustran: 45 50 55 Los plásmidos mutantes descritos aquı́ podrı́an utilizarse como el vector de grado alimenticio de segunda generación, ya que han superado un problema potencial con pFG1 (es decir una ligera inhibición del crecimiento). 60 La clase de mutantes sin alteraciones claras en el número de copia del plásmido o en el gen supresor son particularmente interesantes porque pueden contener mutaciones cromosómicas que superan la inhibición del crecimiento provocada por pFG1. Para confirmar que no contienen alteraciones plásmidas, 28 ES 2 185 669 T3 sometimos a electroporación el DN209 con pFG1, pFG1,1 y el plásmido sin alteraciones detectables (llamado pFG1,3). 5 10 El estriado de los transformantes resultantes reveló que las células que contenı́an pFG1,1 producı́an unas colonias de crecimiento más rápido que las células que contenı́an pFG1 y pFG1,3. De esta manera, una mutación plásmida es responsable del mejor crecimiento de DN209/PFG1,1, y pFG1,3 no contiene dicha mutación. Claramente entonces, la mutación que da lugar al crecimiento más rápido del original ’DN209’/pFG’1.3 debe estar en el cromosoma DN209. El plásmido fue polimerizado de ’DN209’/pFG1,3 dando lugar a una cepa denominada GH209 que esperamos sea un mejor huésped para pFG1 y los derivados varios, puesto que una mutación cromosómica en esta cepa supera la inhibición ligera del crecimiento provocada por pFG1. Ejemplo 14 15 Construcción de un derivado PFG que expresa una peptidasa lactococcal de histidilamino, alanil-amino y lisil-amino (PEPC) Un gen de 2,3 kb que codifica histidil-amino, alanil-amino y lisil-amino (pepC) fue aislado de la cepa Lactococcus lactis CHCC377 (colección de cultivos del laboratorio de Chr. Hansen). 20 1. Clonación y caracterización del gen pepC 25 30 35 40 45 El gen pepC fue clonado mediante la técnica PCR. La secuencia completa del nucleótido (nt) del gen pepC ha sido determinada (véase abajo) y está previsto que un marco de lectura abierto (ORF) de 1308 nt codifique un polipéptido de 436 aminoácidos (aa) (aproximadamente 52 kDa; pl 5,92). La región flanqueadora 5’ no contiene ninguna secuencia hidrófoba que codifique una secuencia guı́a potencial que sugiera una localización intracelular de PEPC como todas las aminopeptidasas hasta ahora conocidas en Lactococcus. Un promotor del consenso y elementos (-35, -10, y SD), los cuales están implicados en la transcripción y la iniciación de la traducción en Lactococcus, están presentes. El gen también contiene una corriente baja de repetición invertida del codón de parada TAA, el cual puede estar implicado en la terminación de la transcripción. Se descubrió una homologı́a secuencial con las enzimas proteolı́ticas de la familia de las proteinasas de cisteı́na (enzimas con un grupo de tiol activo), la cual incluye papaı́na, aleuraı́na y catepsinas B y H. No se descubrió ninguna homologı́a (significante) entre PEPC y las regiones exteriores de la papaı́na que codifican el sitio activo. El gen pepC no está (sobre)expresado en E. coli como p. ej. pepN, cuando un plásmido (pUC18) que alberga el gen pepC se transforma en la cepa DH5α de E. coli. Ninguna actividad enzimática pudo ser medida y ninguna banda extra o más pesada pudo ser detectada en la región de 50 kDa después de la electroforesis en gel de acrilamida, cuando se comparó con una cepa de control sin el fragmento del gen. Actualmente no se sabe si esto es debido al procedimiento de ensayo empleado o si está provocado por un promotor de pepC no funcional en E. coli. A continuación se muestra la secuencia que incluye pepC (SEC ID NO:38) 50 55 60 29 ES 2 185 669 T3 5 10 15 20 25 30 35 40 45 50 2. Construcción y caracterización de un derivado pFG que contiene el gen pepC CHCC377 55 60 El gen pepC CHCC377 fue insertado en el polienlace de pFG1 para obtener pFG4. El DN209 fue transformado con este plásmido y la actividad de la peptidasa de DN209/pFG4 fue comparada con aquella de DN209/pFG1 (control). La actividad de PEPC del control fue 3,1 nmoles/min/mg de proteı́na pero la de DN209/pFG4 fue 12,5 nmoles/min/mg de proteı́na, es decir aproximadamente un aumento 4 veces superior de actividad. Una cepa de DN209/pFG4 fue depositada el 6 de Mayo de 1994 bajo el número No. DSM 9192 con DSM-Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH, Mascheroder Weg 1b, D-38124 Braunschweig, Alemania. 30 ES 2 185 669 T3 Ejemplo 15 Construcción de derivados de PFG que contienen el gen de dipeptidasa (pepR) de la cepa Lactococcus NCDO712 5 10 Un fragmento Hindlll de 2,3 kb que se sobrepone al extremo 5’ del gen pepR fue clonado y caracterizado por secuenciación. El fragmento codifica un polipéptido (PEPR) de 471 aminoácidos de aproximadamente 52 kDa. El gen fue clonado en plásmido pUC18 y se descubrió que el producto genético no estaba sobrepuesto en E. coli y se confirmó que el producto genético es una dipeptidasa. En lo sucesivo el gen se designa como pepR y el producto genético como PEPR. A continuación se muestra una secuencia de 1419 bp que incluye la secuencia pepR (SEC ID NO:39): 15 20 25 30 35 40 45 50 55 60 31 ES 2 185 669 T3 Se construyeron nuevos plásmidos pFG5 y pFG6 para insertar el fragmento anteriormente mencionado de 2,3 kb en pFG1, en cualquier orientación. 5 Las cepas DN209/pFG5 y DN209/pFG6 fueron depositadas el 6 de Mayo de 1994 bajo los Nos. DSM 9193 y DSM 9194, respectivamente, con DSM-Deutsche Sammiung von Mikroorganismen und Zeilkulturen GmbH, Mascheroder Weg 1b, D-38124 Braunschweig, Alemania. Ejemplo 16 10 15 Control de la expresión genética y del crecimiento en bacterias del ácido láctico por la supresión regulada de una mutación rep-am Los experimentos descritos en este ejemplo ilustran cómo la regulación de la expresión de un gen supresor puede ser utilizada para controlar la expresión de otros genes. Este control de expresión genética es posteriormente extendido al control del crecimiento de bacterias del ácido láctico en ambientes diferentes. 1. Cepas bacterianas y medios de crecimiento 20 25 La cepa mutante Pur de Lactococcus lactis DN209 pur-am como se describe en el ejemplo 8 anteriormente mencionado fue utilizada como huésped para varios plásmidos. Los medios usados fueron el medio M17 complementado con 0,5 % de glucosa (GM17) o el medio DN libre de purina basado en el medio tamponado con fosfato de Clark y Maaløe (1967). Cuando fue requerido, se añadieron antibióticos a las concentraciones finales siguientes: clorarrı́fenicol 10 mg/l y eritromicina 2 mg/l. Se añadieron compuestos de purina, adenina, hipoxantina y guanosina a una concentración final de 15mg/l como se describe en los resultados. 2. Plásmidos 30 35 40 Los plásmidos pFDi17 (sup+) (ejemplo 6) que expresan un supresor ocre, pFDi19 (sup-) (ejemplo 6) que no expresa un supresor y pAK58 rep-am) (ejemplo 4) que contiene una mutación ámbar en el gen esencial de la replicación repB se han descrito anteriormente. Para los experimentos con expresión regulada del gen sup, el plásmido, pIAM4 fue construido de la siguiente manera: pFDi19 contiene un gen del ARNt supresor que es silencioso (sin expresión), porque al gen sup le falta un promotor para dirigir la expresión. La inserción de un promotor delante del gen sup restaura la expresión. No obstante, la expresión del promotor localizado delante del gen purD de Lactococcus lactis está regulada. La expresión de este promotor es dependiente de la accesibilidad de las purinas. Si las purinas están disponibles en el medio de crecimiento, la expresión de este promotor es reprimida. No obstante, si las purinas no están presentes en el medio, la expresión disminuye. En consecuencia, este promotor purD fue seleccionado para dirigir la expresión del gen sup de pFDi19. Un fragmento EcoRI de 850 bp que contiene el promotor Lactococcus lactis purD fue clonado en un sitio único EcoRI de pFDi19 que está localizado justo delante del gen sup de pFDi19. 3. Construcción de las cepas experimentales 45 La cepa DN209 fue transformada con los plásmidos pFDi17 (sup+), pFDi19 (sup-) y plAM4 (Ppur sup) y los transformantes fueron seleccionados para la resistencia al cioramfenicol. Las cepas DN209/pFDi17 y DN209/pFDi19 fueron construidas y usadas como cepas de control experimentales. 50 55 60 4. Resultados Las tres cepas DN209/pFDi17, DN209/pFDi19 y DN209/plAM4 representan cepas con genes supresores que se expresan de manera diferente. DN209/pFDi17 contiene el gen sup natural que es expresado más o menos constitutiva mente cuando se cultiva en el medio DN con y sin purinas. DN209/pFDi19 contiene un gen supresor que no está expresado porque al gen le falta un promotor. No obstante, DN209/plAM4 contiene un gen supresor que sólo se expresa en medios sin compuestos de purina, mientras que no se observa ninguna o sólo una expresión menor del gen supresor en medios que contienen purinas. Esto puede utilizarse para controlar la replicación del plásmido pAK58 que contiene una mutación ámbar en el gen esencial de la replicación repB. En consecuencia, la replicación de pAK58 es dependiente de la supresión de la mutación ámbar en el gen repB. Si pAK58 es capaz de duplicarse en una cepa huésped, esta cepa se convierte en resistente a la eritromicina. 32 ES 2 185 669 T3 5 10 15 En la tabla 8 mostrada abajo se exponen los resultados de intentos para introducir pAK58 en las cepas anteriormente mencionadas DN209/pFDi17, DN209/pFDi19 o DN209/plAM4(Ppur sup). Cuando fueron cultivados en placas de ágar del medio DN sin purinas pero con eritromicina, no se obtuvo ningún transformante con DN209/pFDi19(sup-), mientras que los transformantes podı́an obtenerse con ambos DN209/pFDi17(sup+) y DN209/plAM4(Ppur sup). Esto concuerda con el hecho de que pAK58 no puede existir en cepas huéspedes sin un gen supresor. No obstante, como puede observarse, sólo DN209/pFDi17/pAK58 y no DN209/plAM4/pAK58 fue capaz de crecer en medios que contenı́an eritromicina y purinas (DN complementada con adenina (ad), hipoxantina (hx) y guanosina (gu), o GM17). Esto es coherente con la suposición de que el gen supresor en DN209/plAM4 no está expresado en medios que contienen purinas y, en consecuencia, pAK58 no puede duplicarse y, por consiguiente, no se obtuvo ningún transformante resistente a la eritromicina. Este experimento demuestra que la supresión regulada, no sólo la regulación de la expresión del gen supresor sino también la expresión regulada de otros genes, dependen de la supresión y también de la regulación del crecimiento de las bacterias huéspedes. TABLA 8 Selección de cepas DN209 en las que se intenta la transformación con pAK58 20 Cepa 25 30 DN209/pFDi17 DN209/pFDi19 DN209/plAM4 Número de transformantes con 50 ng de pAK58 que seleccionan EriR y CamR en medios diferentes DN DN + ad, hx, gu GM17 > 103 0 > 103 > 103 0 0 500 0 0 Ejemplo 17 35 40 45 50 Construcción de una mutación ámbar localizada en un gen esencial El gen ftsH de Escherichia cofi es esencial para el crecimiento. Un gen que codifica una proteı́na de membrana con una actividad de ATPase putativa homóloga a esta proteı́na esencial de Escherichia coli, FtsH fue identificado como adyacente al gen hpt y al operón trnA en Lactococcus lactis (Nilsson et al., 1994). La secuencia de aminoácidos deducida de este producto de gen mostró una similitud de cuerpo entero con la proteı́na FtsH de Escherichia coli. Si se introduce una mutación ámbar en este gen esencial de una cepa de Lactococcus lactis dará como resultado una cepa cuya viabilidad depende de la expresión del gen supresor. En consecuencia, se introdujo una mutación ámbar por reacción en cadena de polimerasa (PCR) en el codón 325 del gen Lactococcus lactis ftsH y este gen mutante con sus regiones fianqueantes fue clonado en el vector de integración pV2 (Nilsson et al., 1994). La introducción de esta mutación ámbar en el cromosoma de Lactococcus lactis dará como resultado una cepa donde el mantenimiento de un gen supresor es esencial en todos los medios. Además, el crecimiento de dicha cepa puede controlarse con la supresión regulada tal como se explica en el ejemplo 16. Ejemplo 18 55 Procedimiento para seleccionar mutantes bacterianas de ácido láctico que no son capaces de crecer en leche La mutagénesis de la cepa de Lactococcus lactis CHCC2281 fue realizada mediante el tratamiento de un cultivo crecido de 10 mi realizado en el medio M17 con 300 µl EMS (Sigma #M0880) durante 2,5 h a 30◦ C. 60 El cultivo mutado fue distribuido en diez tubos de prueba, se añadieron 10 ml de medio M 17 a cada tubo y se dejaron que creciera a 30◦ C durante 18h. 33 ES 2 185 669 T3 De cada diez cultivos, 1,5 mi fueron usados para inocular 30 ml de medio M17 dejando que su crecimiento continuara a 30◦C hasta que OD en 600 nm (OD600 ) estuvo entre 0,2 y 0,3 cuando las células de 1,0 ml de cada cultivo fueron recogidas por centrifugado, lavadas dos veces en 0,9 % de solución estéril NaCl y resuspendidas en 20 ml de leche. 5 10 Los diez cultivos en leche fueron incubados a 30◦C durante 2h. Posteriormente se añadió ampicilina a una concentración de 100 µg/ml a cada uno y su incubación continuó durante 5h a 30◦C. Dos unidades de penicilinasa (Sigma #P0389) fueron añadidas a cada cultivo y la incubación continuó durante 30 minutos. Las diluciones fueron depositadas en placas de ágar M17 para obtener colonias únicas y las placas fueron incubadas a 30◦ C. Las colonias fueron seleccionadas por su capacidad de crecer en placas de ágar de leche. Se examinaron 200 colonias de cada uno de los cultivos enriquecidos y el número de mutantes incapaz de crecer en el ágar de leche era entre 10 y 40. Todos los enriquecimientos produjeron mutantes con el fenotipo deseado. 15 Posteriormente se descubrió que todos los mutantes excepto uno eran mutantes lac-, ya que fueron capaces de crecer en placas de ágar de leche complementadas con glucosa. Supuestamente, los mutantes lac- resultan de la pérdida del plásmido que lleva los genes lac. 20 25 30 35 La cepa mutante que no puede crecer en placas de ágar de leche + glucosa no fue suprimida al natural mediante la introducción del plásmido pFDi18 y, en consecuencia, la mutación no fue una mutación sin sentido. No obstante, los mutantes sin sentido deseados pueden aislarse si se evita el enriquecimiento de las cepas lac-. Esto puede conseguirse fácilmente mediante la adición de glucosa a la leche utilizada en los cultivos de enriquecimiento y en las placas de ágar de leche. Si la leche es sustituida por leche + glucosa en el medio de enriquecimiento, el procedimiento anteriormente mencionado producirá exclusivamente los mutantes de la misma clase, como aquel que no puede crecer en leche + glucosa. La filtración de una colección de este tipo de mutantes (como la filtración de mutantes pur) produce algunos mutantes que pueden ser suprimidos por el gen supresor clonado. En consecuencia, se contempla que el procedimiento de selección anteriormente mencionado, cuando se modifica como se ha indicado mediante el enriquecimiento del cultivo mutado en un medio de leche y glucosa, proporcionará los medios apropiados para seleccionar eficazmente las cepas mutantes bacterianas de ácido láctico, las cuales han perdido la capacidad de crecimiento en la leche como resultado de la mutación sin sentido. Dichas cepas serán útiles como base para construir, según la presente invención, las cepas bacterianas de ácido láctico que pueden contenerse en la leche. Ejemplo 19 40 Provisión de un derivado de PFDi10 capaz de duplicarse en una especie de Leuconostoc 45 Con el objetivo de demostrar la generalidad de las técnicas aquı́ descritas sobre las bacterias del ácido láctico, se construyó un derivado de pFDi10 (ejemplo 1) que puede utilizarse en miembros del género Leuconostoc, un género bacteriano de ácido láctico que está poco relacionado con el Lactococcus. Este procedimiento de construcción comprende la identificación de un replicón del plásmido de Leuconostoc, la adición de dicho replicón a pFDi10 y la introducción de este derivado en Leuconostoc. Se contempla que dicho enfoque es adecuado para cualquier bacteria del ácido láctico que contiene el plásmido. 50 1. Identificación de un replicón de Leuconostoc 55 60 El Leuconostoc mesenteroides subesp. cremoris DB1 165 contiene 4 plásmidos (Johansen y Kibenich, 1992). Dos de estos plásmidos contienen un sitio único Bg/ll que permite la cionación del plásmido entero como un fragmento de Bg/ll. La cionación fue en el sitio Bg/ll en el polienlace de pIC19H (Marsh et al., 1984). El pBL1 contiene el plásmido de 8,3 kb de DB1 165 y pBL2 contiene el plásmido de 3,6 kb clonado en pIC19H. Un fragmento BamHI-Clal de 2,2 kb de pVA891 (Macrina et al., 25 1983) que contiene el gen EriR fue insertado en pBL1 y pBL2 para dar pAK109 y pAK110, respectivamente. La cepa MG 1363 de Lactococcus lactis, la cepa DB 1164 de Leuconostoc lactis y la cepa DB1 165 de Leuconostoc cremoris fueron electroporadas con pAK1 09 y pAK110. Se obtuvieron colonias en dos electroporaciones y MG1363lpAK109 y DB1164/pAK1O9 fueron purificadas y la presencia de pAK1O9 fue confirmada mediante el análisis de plásmido. Ası́, pAK109 se replica en E.coli, Lactococcus lactis y 34 ES 2 185 669 T3 Leuconostoc lactis, y el sitio Bg/ll del plásmido 8,3 kb puede utilizarse para clonar sin interferencia con replicación. 2. Construcción de derivados PFDi10 que pueden duplicarse en Leuconostoc spr) 5 10 El polienlace de pFDi10 contiene un único sitio BamHI. El plásmido 8,3 kb, anteriormente mencionado, fue insertado en el sitio como un fragmento BamHI produciendo dos clones, pAK1 16 y pAK1 17, difiriendo sólo respecto a la orientación del fragmento de ADN insertado. Ası́, estos plásmidos contienen todos el pFDi10 (es decir, incluyen replicones para E. coli y Lactococcus, un gen de resistencia a la tetraciclina y mutaciones sin sentido en genes de resistencia al cloramfenicol y la eritromicina) y un replicón que es funcional en Leuconostoc spp. Estos plásmidos tienen un tamaño de aproximadamente 20,5 kb (Fig. 7). 3. Estrategia para la introducción de PAK 116 o PAK 117 en Leuconostoc spp 15 Los intentos para introducir pAK116 o pAK117 en Leuconostoc lactis DB1 164 o Leuconosfoc mesenteroı́des subesp. cremoris DB1 165 por electroporación fallaron, supuestamente debido al tamaño de este plásmido y a la baja eficiencia de la electroporación de Leuconostoc. 20 25 30 No obstante, se sabe (David et al., 1989; Dessart et al., 1991) que el Leuconostoc spp. puede ser transformado con éxito y, en consecuencia, se contempla que los transformantes de Leuconostoc spp., como los anteriormente mencionados, que contienen pAK116 o pAK117, pueden obtenerse mediante el acoplamiento de MG1 363/pAK116 o MG1363/pAK117 (que puede construirse por electroporación) con DB1 164 o DB1 165 y mediante la selección para la resistencia a la vancomicina (200 µg/ml) y a la tetraciclina (10 µg/ml). DB1164 y DB1165, como otras cepas de Leuconostoc son intrı́nsecamente resistentes a la vancomicina, mientras que MG1 363/pAK1 16 y MG1 363/pAK1 17 son sensibles a ese antibiótico. La resistencia a la tetraciclina es conferida por pAK1 16 o pAK1 17. Se contempla que los derivados anteriormente mencionados de pFDi10 pueden formar la base para seleccionar especies Leuconostoc que llevan genes supresores mediante el uso esencial de los mismos procedimientos que se detallan en el ejemplo 2, los cuales incluyen la guı́a de métodos microbiológicos estándares que son bien conocidos por los expertos en la técnica. Referencias 35 1. Andachi Y., F. Yamao, A. Muto and S. Osawa 1989: Codón recognition pattems as deduced from sequences of the complete set of transfer RNA species in Mycopiasma capricolum. J. Mol. Biol. 209, 37-54. 40 2. Austin and Wierzhicki 1983. Plasmid 10, 73-81. 3. Campbell, A. 1965: The steric effect in lysogenization by bacteriophage lambda. I Lysogenisation by a partially diploid strain of F. cofl K1 2. Virology 27, 329. 45 4. Ciark, D.J. and 0. Maaløe 1967: DNA replication and the division cycle in Escherichia coh. J. Mol. Biol. 23,99-112. 5. David, S., G. Simons and W.M. Vos 1989: Plasmid transformation by electroporation of Leuconostoc paramesenteroides and its use in molecular cloning. Appi. Environ. Microbiol. 55,1483-1489. 50 6. Demerec, M., E.A. Adelberg, A.J. Ciark and P.E. Harlman 1966: A proposal for a uniform nomenciature in bacterial genetics. Genetics 54, 61-76. 20 55 7. Dessart, S.R. and L.R. Steenson 1991: High frequency intergeneric and intrageneric conjugal transfer of drug resistance plasmids in Leuconostoc mesenteroides ssp. cremoris. J. Dairy Sci. 74,2912-2919. 8. Deno H. and M. Sugiura 1983: The nucleotide sequence of tRNA Ser (GCU) and tRNA (UUG) genes from tobacco chioroplasts. Nucleic Acids Res. 11, 8407-8414. 60 9. Eggertson, G. and D. Sall 1988: Transfer ribonucleic acid-mediated suppression of termination codons in Escherichia colL Microbiol Rev. 52, 354-374. 35 ES 2 185 669 T3 10. Gasson, M.J. 1983: Plasmid complements of Streptococcus lactis NCD0712 and other lactis streptococci after protoplast induced curing. J. Bacteriol. 154, 1-9. 5 11. Hanic-Joyce, P.J. and M.W. Gray 1990: Processing of transfer RNA precursors in a wheat mitochondrial extract. J. Biol. Chem. 265,13782-13791. 12. Hayes F., C. Daly and G.F. Fitzgerald 1990: ldentification of the ninimal replicón of Lactococcus lactissubsp. lactis UC317 plasmid pC1305. Appl. Environ. Microbiol 56, 202-209. 10 15 20 25 13. Hill, C., C. Venema, C. Daly and G.F. Fitzgerald 1988: Cloning and characterization of the tetracycline resistance determinant of and several promoters from within the conjugative transposon Tn919. Appl. Environ. Microbiol. 54, 1230-1236. 14. Hiratsuka, J., H. Shimada, R. Whittier, T. Ishibashi, M. Sakamoto, M. Mori, C. Kondo, Y. Honji, C.R. Sun, BY. Meng, Y.Q. Li, A. Kanno, Y. Nishizawa, A. Hirai, K. Shinozaki and M. Sugiura 1989: The complete sequence of the rice (Oryza sativa) chloroplast genome: intermolecular recombination between distinct tRNA genes accounts for a major plastid DNA inversion during the evolution of cereals. Mol. Gen. Genet. 217, 185.194. 15. Holo, H. and IF. Nes 1989: High-frequency transformation, by electro porati on, of Lactococcus lactis subsp cremoris grown with glycine in osmotically stabilized media. Appl. Environ. Microbiol 55, 3119-3123. 16. Israelsen, H. and E.B. Hansen 1993: Insertion of Transposon Tn917 Derivatives into the Lactococcus lactis subsp lactis Chromosome. Appl. Environ. MicrobioL 59, 21-26. 17. Jahns, A., A. Schafer, A. Geiss and M. Teuber 1991: Identification, cloning and sequencing of the replication region of Lactococcus lactis subsp. lactis biovar diacetylactis Bu2 citrate plasmid pSL2. FEMS Microbiol. Lett. 80, 253-258. 30 18. Johansen, E. and A. Kibonich 1992: Characterization of Leuconostoc isolates from commercial mixedstrain mesophilic starter cultures. J Dairy Sci. 75, 1186-1191 35 19. Johansen, E. and A. Kibenich 1992a: lsolation and characterization of IS1 165, an insertion sequence of Leuconostoc mesenteroides subsp. cremoris and other lactic acid bacteria,. Plasmid 27, 200-206. 20. Karabin, G.D. and R.B Hallick 1983: Euglena gracilis chIoroplast transfer RNA transcription units. Nucleotide sequence analysis of a tRNAThr-tRNAGly-tRNAMet-tRNASer-tRNAGIn gene cluster. J. Biol. Chem. 258, 5512-5518. 40 21. Ma, D.P., Y.W.Yang and S. Hasnain 1989: Nucleotide sequence of Chiarnydomonas reinhardfli mitochondrial genes coding for tRNA gln (UUG) and tRNA met (CAU). Nucleic Acids Res. 17,1256-1256. 45 22. Macrina, FL., R.P. Evans, J.A. Tobian and D.L. Hartley 1983: Novel shuffle plasmid vehicles for Escherichia Streptococcus transgenic cloning. Gene 25, 145-150. 23. Maid, U., R. Steinmueller and K. Zetsche 1992: Structure and expression of a plasmid-encoded groEl homologous heat-shock gene in athermophilic unicellular red alga. Curr. Genet. 21, 521-525. 50 24. Marsh, J.L., M. Erfle and E.J. Wykes. 1984: The pIC plasmid and phage vectors with versatile cloning sites for recombinant selection by insertional inactivation. Gene 32, 481-485. 25. Nagano, Y., R. Matsuno and Y. Sasaki 1991: Sequence and transcriptional analysis of the gene cluster trnQzfpA-psal-0RF231-petA in pea chioropiasts. Curr. Genet. 20, 431-436. 55 26. Nakajima, N., H. Ozeki and Y. Shimura 1981: Organisation and structure of an E. coli tRNA operon containing seven tRNA genes. Cell 23, 239-249. 60 27. Neuhard, J. and F Nygaard 1987: Purines and Pyrimidines. In Neidhardt FC (ed), Escherichia coli and Salmonella typhimurium. American Society for Microbiology, Washington, DC, pp 445-473 30 28. Neuhaus, H. 1989: Nucleotide sequence of the chloroplast genes fro tRNA Gin and the 4 kD K 36 ES 2 185 669 T3 polypeptide of photosystem 11 from mustard (S.a.). Nucleic Acids Res. 17, 444-444. 29. Nilsson, D. and B. Hove-Jensen 1987: P hosphoribosylpyrophosphate synthetase of Bacillus subtilis. Cloning, characterization and chromosomal mapping of the prs gene. Gene 53, 247-255 5 30. Nilsson, D. and E. Johansen 1994: A conserved sequence in tRNA ans rRNA promoters of Lactococcus lactis. Biochim. Biophys. Acta. Submitted for publication. 10 15 31. Nilsson, D., Lauridsen, A.A., Tomoyasu, T. & Ogura, T. 1994. A Lacfococcus lactis gene encodes a membrane protein with putative ATPase activity that is homologous to the essential Escherichia coli gene product. Microbiology, in press. 32. Nygaard, P. 1983: Utilization of preformed purine bases and nucleosides. In Munch-Petersen A (ed), Metabolism of nucleotides, nucleosides and nuoleobases in microorganisms. Academic Press, Inc., New York, pp 27-93 33. Oda, K., K. Yamato, E. Ohta, Y. Nakamura, N. Nozato, T. Kohohi T., Y. Ogura, T. Kanegae, K. Mashi and K. Chyarna 1992: Gene organization deduced from the complete sequence of liverwort Marchantia polymorpha mitochondrial DNA. J. Mol. Biol. 223, 1-7. 20 34. Oda, K., K. Yamato, E. Ohta, Y Nakarnura, M. Takernura, N. Nozato, K. Mashi and K. Ohyama 1992: Transfer RNA genes in the mitoohondrial genome from a liverwort, Marchantia polymorpha: the absence of chloropiast-like tRNAs. Nucleic Acids Res. 20, 3773-3777. 25 30 35. Ohyarna, K., H. Fukuzarna, T Kohohi, T Sano, H. Shirai, K. Umesono, Y. Shiki, M. Takeuchi, Z. Chang, SA. Aota, H. Inokuchi and H. Ozeki 1988: Structure and organization of Marchantia polymorpha Chloroplast genome. I. cloning and gene identification. J. Mol. Biol. 203, 281-298. 36. Ogasawara, N., S. Moriya and H. Yoshikawa 1983: Structure and organization of rRNA operons in the region of the replication region of the Bacillus subtilis chromosome. Nucleic Acids Res. 11, 6301-6316. 37. Pedersen, M.L., K.R. Arnved and F. Johansen 1994: Genetic analysis of the minimal replicón of the Lactococcus lactis subsp. lactis biovar diacetylactis citrate plasmid. Submitted for publication. 35 40 38. Pittet, A.C. and H. Hottinger 1989: Sequence of an hexameric tRNA gene cluster associated with rRNA genes in Lactobacillus bulgaricus. Nucl. Acid. Res. 17, 4873. 39. Ryan, M.J., E.L. Brown, R. Belangaje, H.G. Khorana and H.J. Fritz 1980: Cloning of two chemically synthesised genes for a precursor to the su+3 suppressor tRNAT Y r in D. Söll, J.N. Abelson and P.R. Schimmel (eds.) Transfer RNA: Biological Aspects. Cold Spring Harbor Laboratory, Cold Spring Harbor, NY, pp. 245-258. 40. Sambrook, J., E. F. Fritsch and T. Maniatis 1989: Molecular cloning: A Laboratory Manual. Cold Spring Harbor Laboratory, Cold Spring Harbor, NY. 45 41. Siemeister, G., C. Buchholz and W. Hachtel 1990: Genes for the plastid elongation factor Tu and ribosomal protein s7 and six tRNA genes on the 73 kb DNA from Astasia longa that resembles the cloroplast DNA of Euglena. Mol. Gen. Genet. 220, 425-432. 42. Silhavy, T.J., MI. Berman and L.W. Enquist 1984: Experiments with gene fusions. Cold Spring Harbor Laboratory, Cold Spring Harbor, NY. 50 43. Simeneau, P., R. Wenzel, R. Herrmann and P.C. Hu 1990: Nucleotide sequence of a tRNA cluster from Mycoplasma pneumoniae. Nucleic Acids Res. 18, 2814-2814. 55 44. Strøman, F 1992: Sequence of a gene (lap) encoding a 95.3-kDa aminopeptidase from Lactococcus lactis ssp. cremoris Wg2. Gene 113, 107-112. 45. Thorbjarnadóttir, S., H. Uemura, T. Dingermann, T. Rafnar, S. Thorsteindóttir, D. Söll and G. Eggertson 1985: Escerichia coli supH suppressor: Temperature-sensitive missense suppression caused by anticodón change in tRNAser 2. J. Bacteriol. 161, 207-211. 60 46. Tsudzuki, J., K. Nakashima, T.Tsudzuki, J. Hiratsuka, M. Shibata, T. Wakasugi and M. Sugiura 1992: Chioroplast DNA of black pine retains a residual inverted repeat lacking rRNA genes: nucleotide 37 ES 2 185 669 T3 sequences of tmQ, trnK, psbA, trn1 and trnH and the absence of rps16, Mol. Gen. Genet. 232, 206-214. 47. Wawrousek, FE, N. Narasimhan and J.N. Hansen 1984: Two large clusters with thirty-seven transfer RNA genes adjacent to ribosomal RNA gene sets in Bacillus subtills. J. Biol. Chem. 259, 3694-3702. 5 48. Wolfe, K.H., C.W. Morden, S.C. Ems and J.D. Palmer 1992: Rapid revolution of the plastid translational apparatus in a nonphotosynthetic plant: loss or accelerated sequence evolution of tRNA and ribosomal protein genes. J. Mol. EvoL 35, 304-317. 10 15 49. Yanase, H., H. Fukushi, N. Ueda, Y. Maeda, A. Toyoda and K. Tonomura 1991: Cloning, sequencing and characterization of the intracellular in envertase gene from Zymomonas mobilis. Agric. Biol. Chem. 55,1383-1390. 50. Yaniv M. and W.R. Folk 1975: The nucleotide sequences of the two glutamine transfer ribonucleic acids from Escherichia coli J. Biol. Chem. 250, 3243-3253. 20 25 30 35 40 45 50 55 60 38 ES 2 185 669 T3 REIVINDICACIONES 1. Método de aislamiento de una bacteria del ácido láctico que codifica un ARNt supresor, el cual incluye las fases de 5 10 (i) mutación de un replicón capaz de duplicarse en una bacteria del ácido láctico, dicho replicón incluyendo un gen que codifica un marcador seleccionable que puede expresarse en la bacteria del ácido láctico, (ii) selección, a partir del replicón mutado de (i), de un replicón que contiene una mutación sin sentido en el gen que codifica el marcador seleccionable, (iii) mutación de una bacteria del ácido láctico que no codifica un ARNt supresor, (iv) introducción del replicón de la fase (ii) en dicha bacteria del ácido láctico mutada, y 15 (v) selección, a partir de la bacteria del ácido láctico mutada de (iv), de una bacteria del ácido láctico transformada que codifica el ARNt supresor, en la cual se expresa el marcador seleccionable. 2. Método según la reivindicación 1 donde la bacteria del ácido láctico transformada en otra fase es polimerizada del replicón introducido en la fase (iv). 20 3. Método según la reivindicación 1 donde el ARNt supresor es seleccionado de un grupo que se compone de un supresor ámbar y un supresor ocre. 25 4. Método según la reivindicación 1 donde el gen que codifica el ARNt supresor es un gen que codifica ARNt. 5. Método según la reivindicación 1 donde el gen del ARNt supresor está localizado en el cromosoma. 30 6. Método según la reivindicación 1 donde el replicón que es mutado en la fase (i) es seleccionado de un plásmido y un bacteriófago. 7. Método según la reivindicación 1 donde el marcador seleccionable del replicón que es mutado en la fase (i) es seleccionado de la resistencia antibiótica y auxotropı́a. 35 8. Método de aislamiento de una bacteria del ácido láctico que codifica un ARNt supresor, el cual incluye las fases de (i) mutación de un replicón sin mutaciones sin sentido pero que contiene un marcador seleccionable, cuyo plásmido es intrı́nsecamente capaz de duplicarse en una bacteria del ácido láctico, 40 (ii) selección, a partir de la fase (i), de un replicón que contiene una mutación sin sentido que hace que el replicón sea incapaz de duplicarse, (iii) mutación de una bacteria del ácido láctico que no codifica un ARNt supresor, 45 (iv) introducción en dicha bacteria mutada de ácido láctico del replicón de la fase (ii), y (v) selección de una bacteria del ácido láctico transformada en la que el replicón introducido es capaz de duplicarse. 50 9. Método según la reivindicación 8 donde la bacteria del ácido láctico transformada en otra fase es polimerizada del replicón introducido en la fase (iv). 10. Método según la reivindicación 8 donde el replicón es seleccionado de un plásmido y un bacteriófago. 55 11. Método según la reivindicación 10 donde el replicón que es mutado en la fase (i) es un plásmido natural aislado a partir de una bacteria del ácido láctico. 12. Método según la reivindicación 10 donde el replicón es un plásmido de citrato. 60 13. Cultivo puro aislado de una bacteria del ácido láctico que incluye un gen que codifica un ARNt supresor. 39 ES 2 185 669 T3 14. Cultivo según la reivindicación 13 donde el gen que codifica un ARNt supresor es un gen que codifica ARNt. 5 15. Cultivo según la reivindicación 13 donde el gen que codifica el ARNt supresor está localizado en el cromosoma. 16. Cultivo según la reivindicación 13 donde el gen que codifica el ARNt supresor está localizado en un replicón no cromosómico. 10 17. Cultivo según la reivindicación 16 donde el gen es aislado de una bacteria del ácido láctico. 18. Cultivo según la reivindicación 13 donde el ARNt supresor es seleccionado del grupo que se compone de un supresor ámbar y un supresor ocre. 15 19. Cultivo según la reivindicación 13 que consta adicionalmente de una mutación sin sentido que puede ser suprimida por el ARNt supresor. 20. Cultivo según la reivindicación 19 donde la mutación sin sentido está localizada en un replicón diferente de aquel que contiene el gen que codifica el ARNt supresor. 20 21. Cultivo según la reivindicación 13 donde la bacteria del ácido láctico es seleccionada de Lactococcus spp., Streptococcus spp., Lactobacillus spp., Leuconostoc spp., Pediococcus spp. y Bifidobacterium spp. 25 22. Cultivo según la reivindicación 21 donde la bacteria del ácido láctico es Lactococcus lactis. 23. Cultivo según la reivindicación 13 donde el supresor es uno que suprime una mutación sin sentido la cual, en ausencia de un ARNt supresor capaz de suprimir la mutación, confiere auxotrofı́a. 30 24. Cultivo según la reivindicación 23 donde la mutación sin sentido es en un gen implicado en la sı́ntesis de nucleótidos de purina a partir de sus precursores. 25. Cultivo según la reivindicación 24 donde la bacteria del ácido láctico es un mutante pur. 35 26. Cultivo según la reivindicación 13 que contiene al menos 109 colonias que forman unidades de la bacteria del ácido láctico por g. 27. Cultivo según la reivindicación 26 el cual es seleccionado de un cultivo congelado y un cultivo liofilizado. 40 28. Cultivo según la reivindicación 13 donde el gen que codifica un ARNt supresor está bajo el control de un promotor regulable. 45 29. Cultivo según la reivindicación 28 donde el promotor regulable es un promotor que no está relacionado de manera natural con el gen. 50 30. Cultivo según la reivindicación 20 donde la bacteria del ácido láctico consta de un vector consistente en ADN proveniente de una bacteria M ácido láctico o de un plásmido que aparece naturalmente en una bacteria del ácido láctico, la cual incluye un marcador seleccionable, una región de replicación y al menos un sitio de restricción. 31. Cultivo según la reivindicación 30 donde el marcador seleccionable es el gen supresor. 32. Cultivo según la reivindicación 31 donde el vector es pFG1 (DSM 9190). 55 33. Cultivo según la reivindicación 31 donde el promotor para el gen supresor es mutado, por lo que la expresión del gen es alterada hasta un punto en el que el crecimiento de la bacteria del ácido láctico no disminuye en relación al de una bacteria que alberga un vector en el que el promotor para el gen supresor no es mutado. 60 34. Cultivo según la reivindicación 30 donde el vector comprende adicionalmente un gen insertado que codifica un producto de gen deseado. 40 ES 2 185 669 T3 35. Cultivo según la reivindicación 34 donde el producto de gen es una peptidasa. 5 36. Cultivo según la reivindicación 30 donde el vector es seleccionado de pFG2 (DSM 9191), pFG4 (DSM 9192), pFG5 (DSM 9193) y pFG6 (DSM 9194). 37. Composición que incluye un cultivo puro aislado de una bacteria del ácido láctico tal como se define en la reivindicación 20 y un soporte. 10 15 38. Composición según la reivindicación 37 que contiene al menos 109 colonias que forman unidades de la bacteria del ácido láctico por g. 39. Uso de una composición, como se define en la reivindicación 37, como cultivo de partida en la preparación de un producto alimenticio seleccionado de un producto lácteo, un producto vegetal, un producto cárnico y un producto de panaderı́a. 40. Plásmido que incluye ADN lactobacterial y que es capaz de replicarse en una bacteria del ácido láctico, el plásmido incluyendo un gen que codifica un ARNt supresor. 20 41. Plásmido según la reivindicación 40 donde el gen que codifica un ARNt supresor es derivado del cromosoma de una bacteria del ácido láctico. 42. Plásmido según la reivindicación 40 donde el gen que codifica un ARNt supresor es un gen que codifica ARNt. 25 43. Plásmido según la reivindicación 42 donde el supresor de la mutación sin sentido es seleccionado del grupo que se compone de un supresor ámbar y un supresor ocre. 30 35 40 44. Método de confinamiento de un replicón extracromosómico capaz de replicarse en bacterias del ácido láctico a un primer tipo de células bacterianas de ácido láctico, donde dicho replicón podrı́a ser naturalmente transferido a un segundo tipo de células bacterianas de ácido láctico, dicho método comprendiendo la provisión del primer tipo de células bacterianas de ácido láctico como células que contienen un gen que codifica el ARNt supresor, las células siendo transformadas con el replicón en la forma de un mutante sin sentido que ha perdido su capacidad de replicación en las células bacterianas de ácido láctico, el producto genético del gen que codifica el ARNt supresor siendo capaz de restaurar la capacidad del replicón para replicarse en células bacterianas de ácido láctico, por lo que, si una célula del segundo tipo, la cual no contiene un gen de ARNt supresor que codifica un producto de gen capaz de restaurar la capacidad del mutante sin sentido del replicón para replicarse en bacterias del ácido láctico, recibe dicho replicón extracromosómico, el replicón no se replicará en el segundo tipo de célula bacteriana de ácido láctico. 45. Método según la reivindicación 44 donde el primer tipo de células bacterianas de ácido láctico contiene el gen que codifica el ARNt supresor en el cromosoma. 45 50 55 60 46. Método según la reivindicación 44 donde el primer tipo de bacterias del ácido láctico es seleccionado de Lactococcus spp., Streptococcus spp., Lactobacillus spp., Leuconostoc spp., Pediococcus spp. y Bifidobacterium spp. 47. Método según la reivindicación 46 donde el primer tipo de bacterias del ácido láctico es Lactococcus lactı́s. 48. Método según la reivindicación 44 donde el primer tipo de células bacterianas de ácido láctico está presente en una composición que incluye una mezcla del primer tipo de células y, al menos, un segundo tipo de células bacterianas de ácido láctico que no contiene un gen del ARNt supresor que codifica un producto de gen capaz de restaurar la capacidad del mutante sin sentido del replicón para replicarse en las bacterias del ácido láctico. 49. Método según la reivindicación 48 donde el primer y segundo tipo de células bacterianas de ácido láctico son cultivadas en un producto alimenticio seleccionado de un producto lácteo, un producto vegetal, un producto cárnico y un producto de panaderı́a. 50. Método para mantener la estabilidad de un replicón extracromosómico en células huéspedes bac41 ES 2 185 669 T3 5 terianas de ácido láctico que crecen en un ambiente particular, el cual incluye la provisión de dichas células huéspedes como células mutantes sin sentido que han perdido la capacidad de crecimiento en dicho ambiente y transformadas con un replicón extracromosómico que contiene un gen ARNt supresor que codifica un producto de gen que restaura la capacidad de las células mutantes sin sentido para crecer en dicho ambiente, por lo que, si el replicón está perdido de las células bacterianas de ácido láctico, las células no crecerán. 51. Método según la reivindicación 50 donde las células mutantes sin sentido que han perdido la capacidad para crecer son células auxotróficas. 10 52. Método según la reivindicación 51 donde las células mutantes sin sentido tienen una mutación en un gen implicado en la sı́ntesis de nucleótidos de purina a partir de sus precursores. 15 53. Método según la reivindicación 52 donde las células huéspedes bacterianas de ácido láctico son mutantes pur. 54. Método según la reivindicación 50 donde el ambiente es un producto alimenticio seleccionado de un producto lácteo, un producto vegetal, un producto cárnico y un producto de panaderı́a. 20 25 55. Método para controlar el número de células bacterianas de ácido láctico en un ambiente particular que permite el crecimiento de bacterias del ácido láctico, el cual incluye la provisión de las bacterias con una mutación sin sentido en un gen, la expresión del cual tiene un efecto sobre la viabilidad o el crecimiento de las bacterias, y la inserción en las bacterias de un gen que codifica un ARNt supresor bajo el control de un promotor regulable, cuyo producto de gen, cuando se expresa a un nivel, impide la expresión de la mutación sin sentido y el cual, cuando no es expresado o es expresado a un nivel diferente provoca la interrupción del crecimiento de las células o su muerte. 56. Método según la reivindicación 55 donde el gen que codifica el ARNt supresor es regulado de modo que la expresión del gen es aumentada. 30 57. Método según la reivindicación 56, en el que se provoca la interrupción del crecimiento o la extinción de las células bacterianas de ácido láctico. 35 58. Método según la reivindicación 57 donde la mutación sin sentido está localizada en un gen seleccionado de un gen que codifica un producto de gen que inhibe el crecimiento de la célula y un gen que codifica un producto de gen, cuya expresión provoca la muerte de las células. 59. Método según la reivindicación 55 donde el gen que codifica el ARNt supresor es regulado de modo que la expresión del gen disminuye o termina. 40 60. Método según la reivindicación 59 donde las bacterias del ácido láctico contienen un profago y la mutación sin sentido está localizada en un gen que codifica un producto de gen que inhibe la entrada del profago en su ciclo lı́tico, por lo que, cuando la expresión del gen que codifica el ARNt supresor disminuye o termina, el fago entra en el ciclo lı́tico, causando la muerte de las bacterias del ácido láctico . 45 50 55 60 61. Método según la reivindicación 59 donde la mutación sin sentido está localizada en un gen que codifica un producto de gen, la expresión del cual es requerida para el crecimiento de las bacterias, por lo que, cuando la expresión del gen que codifica el ARNt supresor disminuye o termina, el producto de gen requerido para el crecimiento de las bacterias ya no continúa con su expresión, causando la interrupción del crecimiento de las células bacterianas de ácido láctico. NOTA INFORMATIVA: Conforme a la reserva del art. 167.2 del Convenio de Patentes Europeas (CPE) y a la Disposición Transitoria del RD 2424/1986, de 10 de octubre, relativo a la aplicación del Convenio de Patente Europea, las patentes europeas que designen a España y solicitadas antes del 7-10-1992, no producirán ningún efecto en España en la medida en que confieran protección a productos quı́micos y farmacéuticos como tales. Esta información no prejuzga que la patente esté o no incluı́da en la mencionada reserva. 42 ES 2 185 669 T3 43 ES 2 185 669 T3 44 ES 2 185 669 T3 45 ES 2 185 669 T3 46 ES 2 185 669 T3 47 ES 2 185 669 T3 48 ES 2 185 669 T3 49 ES 2 185 669 T3 50 ES 2 185 669 T3 LISTA DE SECUENCIAS (1) INFORMACIÓN GENERAL: 5 (i) SOLICITANTE (A) NOMBRE: Chr. Hansen’s Laboratorium A/S (B) CALLE: Boge Allé 10-12 10 (C) CIUDAD: Horsholm (D) PAÍS: Dinamarca 15 (E) CÓDIGO POSTAL (ZIP): 2970 (ii) TÍTULO DE LA INVENCIÓN: Mutantes supresores de las bacterias del ácido láctico y su utilización como marcadores selectivos y como medios de contención en las bacterias del ácido láctico. 20 (iii) NÚMERO DE SECUENCIAS: 39 (iv) FORMA DE LECTURA EN ORDENADOR: 25 (A) TIPO DE MEDIO: Floppy disk (B) ORDENADOR: PC compatible IBM (C) SISTEMA OPERATIVO: PC-DOS/MS-DOS 30 (D) SOFTWARE: Patent In Release #1.0, Versión #1.25 (2) INFORMACIÓN PARA LA SEC ID NO:1: 35 (i) CARACTERÍSTICAS DE LA SECUENCIA: (A) LONGITUD: 89 pares de bases (B) TIPO: ácido nucleico 40 (C) AISLAMIENTO: simple (D) TOPOLOGÍA: lineal 45 (ii) TIPO DE MOLÉCULA: ADN (genómico) (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:1: 50 55 (2) INFORMACIÓN PARA LA SEC ID NO:2: (i) CARACTERÍSTICAS DE LA SECUENCIA: (A) LONGITUD: 28 pares de bases 60 (B) TIPO: ácido nucleico 1 ES 2 185 669 T3 (C) AISLAMIENTO: simple (D) TOPOLOGÍA: lineal 5 (ii) TIPO DE MOLÉCULA: ADN (sintético) (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:2: 10 (2) INFORMACIÓN PARA LA SEC ID NO:3. 15 (i) CARACTERÍSTICAS DE LA SECUENCIA: (A) LONGITUD: 28 pares de bases (B) TIPO: ácido nucleico 20 (C) AISLAMIENTO: simple (D) TOPOLOGÍA: lineal 25 (ii) TIPO DE MOLÉCULA: ADN (sintético) (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:3: 30 (2) INFORMACIÓN PARA LA SEC ID NO:4: 35 (i) CARACTERÍSTICAS DE LA SECUENCIA: (A) LONGITUD: 21 pares de bases (B) TIPO: ácido nucleico 40 (C) AISLAMIENTO: simple (D) TOPOLOGÍA: lineal 45 (ii) TIPO DE MOLÉCULA: ADN (sintético) (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:4: 50 (2) INFORMACIÓN PARA LA SEC ID NO:5: 55 (i) CARACTERÍSTICAS DE LA SECUENCIA: (A) LONGITUD: 21 pares de bases 60 (B) TIPO: ácido nucleico (C) AISLAMIENTO: simple 2 ES 2 185 669 T3 (D) TOPOLOGÍA: lineal (ii) TIPO DE MOLÉCULA: ADN (sintético) 5 (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:5: 10 (2) INFORMACIÓN PARA LA SEC ID NO:6: (i) CARACTERÍSTICAS DE LA SECUENCIA: 15 (A) LONGITUD: 33 pares de bases (B) TIPO: ácido nucleico (C) AISLAMIENTO: doble 20 (D) TOPOLOGÍA: lineal (ii) TIPO DE MOLÉCULA: ADN (genómico) 25 (ix) CARACTERÍSTICAS: (A) NOMBRE/CLAVE: CDS (B) LOCALIZACIÓN: 1.33 30 (x) INFORMACIÓN SOBRE LAS PUBLICACIONES: (A) AUTORES: Macrina, F.L. 35 Evans, R.P. Tobian, J.A. Hartley, D.L. 40 (B) TÍTULO: Novel shuttle plasmid vehicles for Escherichia Streptococcus transgenic cloning (C) REVISTA: Gene 45 (D) VOLUMEN: 25 (F) PÁGINAS: 145-150 (G) FECHA: 1983 50 (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:6: 55 60 (2) INFORMACIÓN PARA LA SEC ID NO:7: (i) CARACTERÍSTICAS DE LA SECUENCIA: 3 ES 2 185 669 T3 (A) LONGITUD: 11 aminoácidos (B) TIPO: aminoácido 5 (D) TOPOLOGÍA: lineal (ii) TIPO DE MOLÉCULA: proteı́na (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:7: 10 15 (2) INFORMACIÓN PARA LA SEC ID NO:8: (i) CARACTERÍSTICAS DE LA SECUENCIA: 20 (A) LONGITUD: 16 pares de bases (B) TIPO: ácido nucleico 25 (C) AISLAMIENTO: simple (D) TOPOLOGÍA: lineal (ii) TIPO DE MOLÉCULA: ADN (sintético) 30 (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:8: 35 (2) INFORMACIÓN PARA LA SEC ID NO:9: (i) CARACTERÍSTICAS DE LA SECUENCIA: 40 (A) LONGITUD: 21 pares de bases (B) TIPO: ácido nucleico 45 (C) AISLAMIENTO: simple (D) TOPOLOGÍA: lineal (ii) TIPO DE MOLÉCULA: ADN (sintético) 50 (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:9: 55 (2) INFORMACIÓN PARA LA SEC ID NO:10: (i) CARACTERÍSTICAS DE LA SECUENCIA: 60 (A) LONGITUD: 300 pares de bases 4 ES 2 185 669 T3 (B) TIPO: ácido nucleico (C) AISLAMIENTO: doble 5 (D) TOPOLOGÍA: lineal (ii) TIPO DE MOLÉCULA: ADN (genómico) (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:10: 10 15 20 (2) INFORMACIÓN PARA LA SEC ID NO:11: 25 (i) CARACTERÍSTICAS DE LA SECUENCIA: (A) LONGITUD: 300 pares de bases 30 (B) TIPO: ácido nucleico (C) AISLAMIENTO: doble (D) TOPOLOGÍA: lineal 35 (ii) TIPO DE MOLÉCULA: ADN (genómico) (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:11: 40 45 50 (2) INFORMACIÓN PARA LA SEC ID NO:12: 55 (i) CARACTERÍSTICAS DE LA SECUENCIA: (A) LONGITUD: 72 pares de bases 60 (B) TIPO: ácido nucleico (C) AISLAMIENTO: simple 5 ES 2 185 669 T3 (D) TOPOLOGÍA: lineal (ii) TIPO DE MOLÉCULA: ADN (genómico) 5 (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:12: 10 (2) INFORMACIÓN PARA LA SEC ID NO:13: 15 (i) CARACTERÍSTICAS DE LA SECUENCIA: (A) LONGITUD: 72 pares de bases (B) TIPO: ácido nucleico 20 (C) AISLAMIENTO: simple (D) TOPOLOGÍA: lineal 25 (ii) TIPO DE MOLÉCULA: ADN (genómico) (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:13: 30 35 (2) INFORMACIÓN PARA LA SEC ID NO:14: (i) CARACTERÍSTICAS DE LA SECUENCIA: (A) LONGITUD: 72 pares de bases 40 (B) TIPO: ácido nucleico (C) AISLAMIENTO: simple 45 (D) TOPOLOGÍA: lineal (ii) TIPO DE MOLÉCULA: ADN (genómico) (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:14: 50 55 (2) INFORMACIÓN PARA LA SEC ID NO:15: 60 (i) CARACTERÍSTICAS DE LA SECUENCIA: (A) LONGITUD: 75 pares de bases 6 ES 2 185 669 T3 (B) TIPO: ácido nucleico (C) AISLAMIENTO: simple 5 (D) TOPOLOGÍA: lineal (ii) TIPO DE MOLÉCULA: ADN (genómico) (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:15: 10 15 (2) INFORMACIÓN PARA LA SEC ID NO:16: 20 (i) CARACTERÍSTICAS DE LA SECUENCIA: (A) LONGITUD: 72 pares de bases (B) TIPO: ácido nucleico 25 (C) AISLAMIENTO: simple (D) TOPOLOGÍA: lineal 30 (ii) TIPO DE MOLÉCULA: ADN (genómico) (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:16: 35 40 (2) INFORMACIÓN PARA LA SEC ID NO:17: (i) CARACTERÍSTICAS DE LA SECUENCIA: (A) LONGITUD: 72 pares de bases 45 (B) TIPO: ácido nucleico (C) AISLAMIENTO: simple 50 (D) TOPOLOGÍA: lineal (ii) TIPO DE MOLÉCULA: ADN (genómico) (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:17: 55 60 7 ES 2 185 669 T3 (2) INFORMACIÓN PARA LA SEC ID NO:18: (i) CARACTERÍSTICAS DE LA SECUENCIA: 5 (A) LONGITUD: 72 pares de bases (B) TIPO: ácido nucleico (C) AISLAMIENTO: simple 10 (D) TOPOLOGÍA: lineal (ii) TIPO DE MOLÉCULA: ADN (genómico) 15 (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:18: 20 (2) INFORMACIÓN PARA LA SEC ID NO:19: 25 (i) CARACTERÍSTICAS DE LA SECUENCIA: (A) LONGITUD: 72 pares de bases 30 (B) TIPO: ácido nucleico (C) AISLAMIENTO: simple (D) TOPOLOGÍA: lineal 35 (ii) TIPO DE MOLÉCULA: ADN (genómico) (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:19: 40 45 (2) INFORMACIÓN PARA LA SEC ID NO:20: (i) CARACTERÍSTICAS DE LA SECUENCIA: 50 (A) LONGITUD: 72 pares de bases (B) TIPO: ácido nucleico (C) AISLAMIENTO: simple 55 (D) TOPOLOGÍA: lineal (ii) TIPO DE MOLÉCULA: ADN (genómico) 60 (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:20: 8 ES 2 185 669 T3 5 (2) INFORMACIÓN PARA LA SEC ID NO:21: (i) CARACTERÍSTICAS DE LA SECUENCIA: 10 (A) LONGITUD: 72 pares de bases (B) TIPO: ácido nucleico 15 (C) AISLAMIENTO: simple (D) TOPOLOGÍA: lineal (ii) TIPO DE MOLÉCULA: ADN (genómico) 20 (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:21: 25 (2) INFORMACIÓN PARA LA SEC ID NO:22: 30 (i) CARACTERÍSTICAS DE LA SECUENCIA: (A) LONGITUD: 72 pares de bases 35 (B) TIPO: ácido nucleico (C) AISLAMIENTO: simple (D) TOPOLOGÍA: lineal 40 (ii) TIPO DE MOLÉCULA: ADN (genómico) (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:22: 45 50 (2) INFORMACIÓN PARA LA SEC ID NO:23: (i) CARACTERÍSTICAS DE LA SECUENCIA: 55 (A) LONGITUD: 72 pares de bases (B) TIPO: ácido nucleico 60 (C) AISLAMIENTO: simple (D) TOPOLOGÍA: lineal 9 ES 2 185 669 T3 (ii) TIPO DE MOLÉCULA: ADN (genómico) (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:23: 5 10 (2) INFORMACIÓN PARA LA SEC ID NO:24: (i) CARACTERÍSTICAS DE LA SECUENCIA: 15 (A) LONGITUD: 72 pares de bases (B) TIPO: ácido nucleico (C) AISLAMIENTO: simple 20 (D) TOPOLOGÍA: lineal (ii) TIPO DE MOLÉCULA: ADN (genómico) 25 (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:24: 30 (2) INFORMACIÓN PARA LA SEC ID NO:25: 35 (i) CARACTERÍSTICAS DE LA SECUENCIA: (A) LONGITUD: 72 pares de bases (B) TIPO: ácido nucleico 40 (C) AISLAMIENTO: simple (D) TOPOLOGÍA: lineal 45 (ii) TIPO DE MOLÉCULA: ADN (genómico) (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:25 50 55 (2) INFORMACIÓN PARA LA SEC ID NO:26: (i) CARACTERÍSTICAS DE LA SECUENCIA: 60 (A) LONGITUD: 72 pares de bases (B) TIPO: ácido nucleico 10 ES 2 185 669 T3 (C) AISLAMIENTO: simple (D) TOPOLOGÍA: lineal 5 (ii) TIPO DE MOLÉCULA: ADN (genómico) (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:26: 10 15 (2) INFORMACIÓN PARA LA SEC ID NO:27: (i) CARACTERÍSTICAS DE LA SECUENCIA: (A) LONGITUD: 75 pares de bases 20 (B) TIPO: ácido nucleico (C) AISLAMIENTO: simple 25 (D) TOPOLOGÍA: lineal (ii) TIPO DE MOLÉCULA: ADN (genómico) (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:27: 30 35 (2) INFORMACIÓN PARA LA SEC ID NO:28: (i) CARACTERÍSTICAS DE LA SECUENCIA: 40 (A) LONGITUD: 75 pares de bases (B) TIPO: ácido nucleico 45 (C) AISLAMIENTO: simple (D) TOPOLOGÍA: lineal (ii) TIPO DE MOLÉCULA: ADN (genómico) 50 (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:28: 55 60 (2) INFORMACIÓN PARA LA SEC ID NO:29: (i) CARACTERÍSTICAS DE LA SECUENCIA: 11 ES 2 185 669 T3 (A) LONGITUD: 75 pares de bases (B) TIPO: ácido nucleico 5 (C) AISLAMIENTO: simple (D) TOPOLOGÍA: lineal (ii) TIPO DE MOLÉCULA: ADN (genómico) 10 (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:29: 15 (2) INFORMACIÓN PARA LA SEC ID NO:30: 20 (i) CARACTERÍSTICAS DE LA SECUENCIA: (A) LONGITUD: 75 pares de bases 25 (B) TIPO: ácido nucleico (C) AISLAMIENTO: simple (D) TOPOLOGÍA: lineal 30 (ii) TIPO DE MOLÉCULA: ADN (genómico) (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:30: 35 40 (2) INFORMACIÓN PARA LA SEC ID NO:31: (i) CARACTERÍSTICAS DE LA SECUENCIA: 45 (A) LONGITUD: 72 pares de bases (B) TIPO: ácido nucleico (C) AISLAMIENTO: simple 50 (D) TOPOLOGÍA: lineal (ii) TIPO DE MOLÉCULA: ADN (genómico) 55 (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:31: 60 12 ES 2 185 669 T3 (2) INFORMACIÓN PARA LA SEC ID NO:32: (i) CARACTERÍSTICAS DE LA SECUENCIA: 5 (A) LONGITUD: 72 pares de bases (B) TIPO: ácido nucleico (C) AISLAMIENTO: simple 10 (D) TOPOLOGÍA: lineal (ii) TIPO DE MOLÉCULA: ADN (genómico) 15 (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:32: 20 (2) INFORMACIÓN PARA LA SEC ID NO:33: 25 (i) CARACTERÍSTICAS DE LA SECUENCIA: (A) LONGITUD: 27 pares de bases (B) TIPO: ácido nucleico 30 (C) AISLAMIENTO: simple (D) TOPOLOGÍA: lineal 35 (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:33: 40 (2) INFORMACIÓN PARA LA SEC ID NO:34: (i) CARACTERÍSTICAS DE LA SECUENCIA: 45 (A) LONGITUD: 30 pares de bases (B) TIPO: ácido nucleico (C) AISLAMIENTO: simple 50 (D) TOPOLOGÍA: lineal (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:34: 55 60 (2) INFORMACIÓN PARA LA SEC ID NO:35: (i) CARACTERÍSTICAS DE LA SECUENCIA: 13 ES 2 185 669 T3 (A) LONGITUD: 69 pares de bases (B) TIPO: ácido nucleico 5 (C) AISLAMIENTO: doble (D) TOPOLOGÍA: lineal (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:35: 10 15 (2) INFORMACIÓN PARA LA SEC ID NO:36: (i) CARACTERÍSTICAS DE LA SECUENCIA: 20 (A) LONGITUD: 2006 pares de bases (B) TIPO: ácido nucleico 25 (C) AISLAMIENTO: simple (D) TOPOLOGÍA: lineal (xi) DESCRIPCIÓN SECUENCIA: SEC ID NO:36: 30 35 40 45 50 55 60 14 ES 2 185 669 T3 5 10 15 20 25 30 35 40 45 (2) INFORMACIÓN PARA LA SEC ID NO:37: (i) CARACTERÍSTICAS DE LA SECUENCIA: 50 (A) LONGITUD: 46 pares de bases (B) TIPO: ácido nucleico (C) AISLAMIENTO: simple 55 (D) TOPOLOGÍA: lineal (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:37: 60 15 ES 2 185 669 T3 (2) INFORMACIÓN PARA LA SEC ID NO:38: (i) CARACTERÍSTICAS DE LA SECUENCIA: 5 (A) LONGITUD: 1311 pares de bases (B) TIPO: ácido nucleico (C) AISLAMIENTO: simple 10 (D) TOPOLOGÍA: lineal (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:38: 15 20 25 30 35 40 45 50 55 60 16 ES 2 185 669 T3 (2) INFORMACIÓN PARA LA SEC ID NO:39: (i) CARACTERÍSTICAS DE LA SECUENCIA: 5 (A) LONGITUD: 1419 pares de bases (B) TIPO: ácido nucleico (C) AISLAMIENTO: simple 10 (D) TOPOLOGÍA: lineal (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID NO:39: 15 20 25 30 35 40 45 50 55 60 17