Taxonomía de las bacterias acéticas
Anuncio

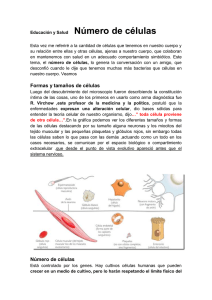
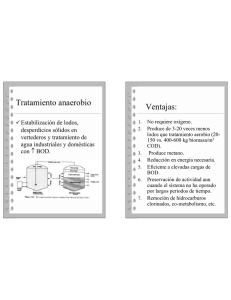
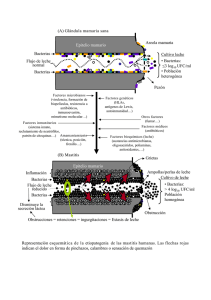
DESARROLLO DE TÉCNICAS MOLECULARES PARA LA IDENTIFICACIÓN DE BACTERIAS ACÉTICAS José M. Guillamón Departamento de Bioquimica i Biotecnologia Facultat d’Enologia de Tarragona Universitat Rovira i Virgili [email protected] Rovira i Virgili University BACTERIA ACÉTICAS. PRINCIPALES CARACTERÍSTICAS • Bacilos Gram negativos moviles • Catalasa Positivos • Metabolismo estrictamente aeróbio • Capacidad para crecer en medio ácido • Capacidad para oxidar una gran cantidad de sustratos • Aplicaciones industriales: vinagre, Ácido glucónico, sorbosa, celulosa, etc. 1 Taxonomía de las bacterias acéticas Taxonomía de las bacterias acéticas • Persoon (1822) Mycoderma • Pasteur (1868) Mycoderma aceti • Beijerinck (1900) Acetobacter 2 Taxonomía de las bacterias acéticas • Gluconobacter G. oxydans • Acetobacter: A. aceti A. pasteurianus A. hansenii A. liquefaciens Taxonomía de las bacterias acéticas Reordenación por pruebas moleculares (año 2003) 6 Géneros y 34 especies: • Acetobacter (14 especies) • Gluconobacter (3 especies) • Gluconoacetobacter (11 especies) • Acidomonas (1 especie) • Asaia (4 especies) • Kozakia (1 especie) 3 Taxonomía de las bacterias acéticas Especies aisladas en vinagres Acetobacter Gluconoacetobacter A. aceti Ga. hansenii A. pasteurianus Ga. europaeus Ga. xylinus Ga. oboediens Ga. intermedius Ga. entanii Aislamiento y Recuento 4 AISLAMIENTO Y RECUENTO DE ACÉTICAS Toma de muestra Uva, vino o vinagre Recuento en placa Dilución decimal o Centrifugación (10.000 rpm/10 min) Disolución del CaCO3 Medio de cultivo GYC • Glucosa 5% • Extracto de levadura 1% • Pimaricina (100mg/l) • Penicilina (3U/ml) • Carbonato Cálcico (0.5%) Incubar 48 horas/ 28ºC Tinción Gram y prueba de la catalasa Otros medios de aislamiento Medio RAE (Sokollek et al., 1998) Medio Manitol Glucosa D-Manitol 40 g 25 g Extracto levadura 10 g Extracto levadura 5g Peptona Peptona 3g 10 g Fosfato sódico 3,38 g Ácido cítrico 1,5 g Etanol 20 ml Ácido acético 10 ml Agar 10 g Agua Agar Agua 15 g 1 Litro 1 Litro 5 Técnicas de identificación y recuento independientes del cultivo Identificación de especies de bacterias acéticas mediante análisis de restricción del gen ribosomal 16S 1) Amplificación mediante PCR del gen ribosomal 16S 16S ITS 23S ITS 5S 2) Digestión del fragmento amplificado con diferentes enzimas de restricción 3) Separación de los fragmentos en base a su tamaño en un gel de agarosa 6 Técnicas moleculares de identificación La Reacción en Cadena de la Polimerasa (PCR) Permite amplificar una región del genoma bacteriano millones de veces Reacción de PCR: • DNA molde • Oligonucleótidos (20-24 nt) complementarios a los extremos del fragmento a amplificar • Nucleótidos • Polimerasa 7 Técnicas moleculares de identificación La Reacción en Cadena de la Polimerasa (PCR) Técnicas moleculares de identificación El análisis de restricción o RFLPs Enzimas de restricción EcoRI 8 Análisis de restricción del rDNA 16S 1. Amplificación del rDNA 16S mediante la PCR Se obtuvieron amplificados de todas las cepas de referencia de un tamaño aproximado de 1450 pb Productos de PCR ∼ 1450 bp Productos de PCR ∼ 1450 bp Análisis de restricción del rDNA 16S 2. Digestión del fragmento amplificado mediante diferentes enzimas de restricción (hasta 8 enzimas) Patrones de restricción obtenidos con TaqI Ga.liquefaciens A. pasteurianus Ga.G.oxydans hansenii A. aceti Ga. Xylinus Ga. europaeus 9 Tamaños de los fragmentos de restricción Cepa Enzima de restricción TaqI Enzima de restricción RsaI G. oxydans 1408 350+190+175+160+120+120+110 400+400+400+150+90 G. oxydans 360 350+190+175+160+120+120+110 400+400+400+150+90 G. oxydans 1484 350+190+175+160+120+120+110 400+400+400+150+90 G. oxydans 1414 350+190+175+160+120+120+110 400+400+400+150+90 A. aceti 1261 850+350+210 500+400+300+150+125 A. aceti 298 850+350+210 500+400+300+150+125 A. aceti 1505 850+350+210 500+400+300+150+125 A. aceti 1372 850+350+210 500+400+300+150+125 A. pasteurianus 1262 500+350+330+210 500+400+300+150+125 A. pasteurianus 1553 500+350+330+210 500+400+300+150+125 Ga. hansenii 1527 650+350+210+175 500+400+400+150 Ga. liquefaciens 1381 500+350+210+175+160 500+400+400+150 Ga. liquefaciens 1347 500+350+210+175+160 500+400+400+150 Ga. xylinus 1515 500+350+210+175+160 500+400+400+150 Ga. xylinus 1518 500+350+210+175+160 500+400+400+150 Ga. europaeus 6160 500+350+210+175+160 500+400+400+150 ESTUDIO ECOLÓGICO DE UNA FERMENTACIÓN VÍNICA Diversidad de especies de bacterias acéticas 100% 80% Ga. liquefaciens Ga. hansenii A. pasteurianus A. aceti G. oxydans 60% 40% 20% nt . F.F. Esp o F.F. Inoc . M.F. Esp ont. M.F. Ino c- Mosto 0% 10 ESTUDIO ECOLÓGICO DE UNA FERMENTACIÓN MALOLÁCTICA % especies de acéticas Diversidad de especies de acéticas durante la FML 100% 80% Ga. liquefaciens Ga. hansenii G. oxydans A. aceti 60% 40% 20% 0% 0 12 17 Día de Fermentación Malolactica Tipificación de cepas de bacterias acéticas mediante la técnica ERIC-PCR ⊇ En bacterias se han descrito secuencias repetitivas en el genoma: • Enterobacterial Repetitive Intergenic Consensus (ERIC) ⊇ Ya existen oligonucleotidos diseñados para la amplificación por PCR de las secuencias comprendidas entre estos elementos repetitivos ERICs LMG 1390 LMG 1484 LMG1282 LMG 1408 11 Recuento de poblaciones de bacterias acéticas mediante la PCR cuantitativa • No es necesario el cultivo previo • Cuantifica poblaciones de bacterias mediante cuantificación del ADN bacteriano de la muestra 12 Recuento de poblaciones de bacterias acéticas mediante la PCR cuantitativa Recuento de poblaciones de bacterias acéticas mediante la PCR cuantitativa 13 Perspectivas de futuro: • Cuantificar las distintas especies mediante la PCR cuantitativa en vinagre • Identificar mediante distintas técnicas independientes del cultivo la diversidad de especies y de cepas • Seleccionar cultivos iniciadores para acetificación (cepas seleccionadas) 14