Regulación de la Expresión en Procariotas
Anuncio

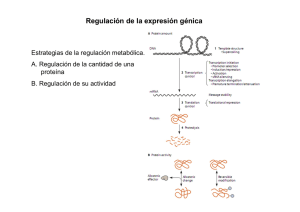
Regulación de la Expresión en Procariotas •Regulación a nivel de Transcripción (Iniciación o Terminación) ¾RNA polimerasa (factores V) ¾Proteínas regulatorias ¾Estructura del ARN transcripto. •Regulación a nivel de Traducción (Accesibilidad o no del RBS) 1 Regulación de la Iniciación de la Transcripción • Control constitutivo – Depende de la estructura del promotorÆ determina el nivel basal de transcripción • Secuencia -35 (unión RNA pol) • Secuencia -10 (complejo cerradoÆ abierto) • Iniciaciones abortivas (secuencia transcripto) • Control regulado – Depende de proteínas regulatorias – Cambio de subunidad VV70V32V54Vs) 2 RNA polimerasa + Factores V V70 : Factor general V32 : Heat shock Vs : estrés V54 : carencia de N •Secuencias conservadas en centros -35 y -10 •RNA pol + Vcon igual tamaño y ubicación respecto al sitio de iniciación •Regulación a nivel de modificaciones o actividad de la Proteína (Ej: V32 normalmente unida a DnaK (inestabilidad). Heat shock produce liberación de V32 ) 3 4 Regulación de la expresión en bacterias • OPERONES Æ un único promotor regula la expresión de genes relacionados (codifican proteínas involucradas en una misma vía metabólica o proceso) mRNA policistrónico •Regulación Positiva •Activación de la transcripción por proteína activadora (facilita la unión de la RNApol) Regulación Negativa: Inhibición de la transcripción por unión de proteína Represora (Inhibe la unión o iniciación de RNApol) 5 Regulación negativa Regulación positiva 6 Regulación de la expresión en bacterias 7 Operón Lactosa 8 •Proteína represora LacI Lac I : tetrámero – Monómero •DBD (HTH) N-terminal •Reg central unión al inductor •C-terminal: Oligomerización – Dímero Æ mayor afinidad por DNA Sitios de unión al DNA: Operador •O1 (región promotora) •O2 (+412) •O3 (-82) 9 10 11 Regulación Positiva CAP-AMPc (CRP) Dímero (2 subunidades idénticas) activado por AMPc Sitio de unión de CRP 22pb (repetición invertida) En operón lactosa: -61 Sistema de control de la captación de glucosa (PTS) regula la actividad de la adenilato ciclasa (IIA Gluc -IIA Gluc-P) 12 Operón Arabinosa AraC : Proteína activadora y represora CRP: unión a su sitio específico requerida para activación del operón AraC represora El promotor araBAD es un promotor débil AraC/arabinosa: Activadora CTGACG -- 18 -- TACTGT TTGACA -- 17 -- TATAAT 13 Operón Triptofano 14 Operón Triptofano Represión por producto final 15 UGG 16 17 Atenuación en trp • La [trp] determina la [trp-tRNA]. • En presencia de trp: – El ribosoma se mueve hasta el codón de terminación. – Se forma estructura secundaria en el ARNm que provoca la terminación de la transcripción (OFF). • En ausencia de trp: – El ribosoma se frena en codones Trp – Se forma una estructura secundaria alternativa en el RNA. – Continua la transcripción de los genes estructurales (ON). 18 •Regulación por atenuación en Operones de genes involucrados en biosíntesis de aminoácidos: his, phe, leu,etc En todos los casos los genes estructurales están precedidos por un RNA lider corto que codifica un polipéptido corto rico en el aminoácido producto final de la via Secuencia de aminoácidos de algunos péptidos lider 19 OPERON TRIPTOFANO Bacillus subtilis 20 Regulación de la asimilación de nitrógeno 21 Regulación a nivel traduccional Operones de Proteínas ribosomales Regulación de la traducción del ARNm 22 ARN reguladores de la expresión •Riboswitch: Regulación de la expresión a nivel de transcripción o traducción del ARN Ligandos •Parte de la región 5’ no traducida de un ARNm que funciona como sensora y regula la expresión. (aminoácidos, bases, azúcares, vitaminas, etc) •Comprende 2 dominios: 1) aptamero: unión a un ligando y 2) plataforma de expresión: acopla la unión del ligando a un cambio en la expresión. • Conformaciones mutuamente excluyentes de una larga región 5’ no traducida del ARNm (5’UTR) •Regulado por señal del entorno en forma de un metabolito (ligando) •No hay ninguna proteína involucrada •Unión del ligando directamente al ARNm 23 Tiamina (Tiamina (E.coli)) (FMN, SAM, guanina) GlcN6P mRNA glmS (Enz síntesis de GlcN6P) 24 Riboswitches sintéticos: búsqueda de nuevos aptámeros (ARN) que se unan a ligandos específicos y regulen la expresión. SELEX 25 ARN regulatorios Codificado en cis ARN antisentido Mecanismos regulatorios: Inhibición de maduración de primer, inhibición de la traducción o promoción de degradación o clivaje del ARN Codificado en trans Pequeños ARN regulatorios: - Apareamiento con sitio de iniciación de la traducción - Unión a una región del ARN puede cambiar la conformación de otra Æ expone o oculta región de SD - apareamiento con blancos ARNm generando ARNdc Æ clivaje por ARNasa E 26 •Regulación por RNA antisentido (sRNA) 27 SISTEMAS DE EXPRESIÓN PROCARIOTAS Vector de expresión procariota : set de elementos genéticos óptimos para transcripción y traducción •Promotor regulable •Terminadores de transcripción •Iniciación de traducción •Enhancer de traducción •Terminación de traducción •Estabilización de mRNA ATG R Promotor SD STOP Gen de interés TT 28 Promotores E.coli ¾Lac (lacI) ¾tac o trc (lacI) ¾Pho (fosfato) •Promotores fuertes (10-30% de proteína total) y regulables ¾Trp (triptofano) •Inducción rápida ¾araBAD (araC) •Nivel de expresión basal bajo (promotor altamente represible) ¾pL (O) (OcIts) 29 SISTEMAS DE EXPRESIÓN PROCARIOTA PROMOTOR trc o tac -35 promotor trp Promotor -10 promotor lac UV5 - Separación 17pb lac UV5 TTTACA -- 18 -- TATAAT lac wild-type TTTACA -- 18 -- TATGTT TTGACA -- 17 -- TATAAT Trp TTGACA -- 17 -- TTAACT TTGACA -- 17 -- TATAAT IPTG (isopropil-E-D-tiogalactósido) 30 31 SISTEMA DE EXPRESIÓN BASADO EN EL BACTERIÓFAGO T7 1- Fago OCE6 defectivo (no lítico) que expresa T7 RNA polimerasa 2- T7 polimerasa: transcribe genes tardíos del fago T7 (lisis) 3- + Plásmido que expresa T7 lisozima (inhibe T7 polimerasa) 32 +1 Promotor T7 TAATACGAACTCACTATAGGGAGA Terminador T7 RBS GAAGGAGA ATG 33