ESTAFILOCOCOS: procedimientos de aislamiento e identificación
Anuncio

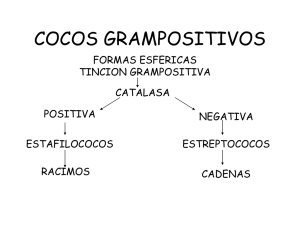
ESTAFILOCOCOS: procedimientos de aislamiento e identificación en el laboratorio ESTAFILOCOCOS: procedimientos de aislamiento e identificación en el laboratorio Loretta Durán Esther Damiani Christian Trigoso Resumen Los diferentes miembros del género estafilococos juegan un papel muy importante como causas de infecciones intrahospitalarias. Se observan elevadas tasas de resistencia a la meticilina tanto entre cepas de estafilocos coagulasa negativos como coagulasa positivos de origen intrahospitalario. Tradicionalmente, las infecciones estafilococcicas de origen comunitario son causadas por cepas de Staphylococcus aureus susceptibles a la meticilina. En la actualidad, sobre todo en los países industrializados, se observa el desarrollo de carácter epidémico de infecciones causadas por Staphylococcus aureus resistentes a la meticilina de origen comunitario. Dicho fenómeno cambia dramáticamente la selección de antimicrobianos requeridos para su tratamiento. El creciente número de infecciones causadas por los estafilococos coagulasa negativos y coagulasa positivos y el progresivo desarrollo de resistencia antimicrobiana hacen que su precisa identificación hasta el nivel de especie y la determinación de su perfil de susceptibilidad antimicrobiana sean de alta importancia para el laboratorio de bacteriología. El presente estudio resume los procedimientos recomendados para el aislamiento, identificación y determinación de la susceptibilidad antimicrobiana de estos patógenos en laboratorios clínicos de bacteriología. Correspondencia: Dra. “Loretta Ivana Duran Arias” <[email protected]> 277 Vigilancia, prevención y control de infecciones asociadas a servicios de salud INTRODUCCIÓN Los estafilococos son patógenos humanos muy importantes. Se encuentran ampliamente distribuidos en el medio ambiente. En los seres humanos son parte de la flora normal de la piel y de la faringe. Producen una gran variedad de infecciones cuya frecuencia e importancia continúan a acrecentarse. El género Staphylococcus pertenece a la familia Micrococcaceae. Se distinguen cerca de 31 especies diferentes. Clínicamente se dividen a los organismos en dos grupos: los estafilococos coagulasa positivas (S. aureus) y los estafilococos coagulasa negativas (S. epidermidis, S. saprophyticus, S. hominis, S. haemolyticus, S. warneri, S. capitis, etc.). Las especies más frecuentes y de mayor importancia clínica son: S. aureus, S. epidermidis, S. saprophyticus y S. hominis. TOMA DE MUESTRA Se recuperan principalmente en casos de infección de piel y tejidos blandos, heridas quirúrgicas, neumonía, bacteriemia, infecciones asociadas a catéter intravascular, endocarditis, e infecciones de articulaciones y huesos. Por lo tanto, en el laboratorio los aislamientos provienen principalmente de tres tipos de muestras: esputo, sangre y cultivos de abscesos y heridas quirúrgicas. Las características de estos procesos se resumen en la Guía Para la Toma de Muestras de Laboratorio descrita en otro lugar en esta monografía. (1) MICROBIOLOGÍA Morfología Su morfología microscópica muestra cocos Gram positivos agrupados en pares y en forma de racimos de uvas, lo que ayuda a diferenciarlos de los estreptococos que forman cadenas de cocos. Son inmóviles y no esporulados. La morfología macroscópica de S. aureus muestra colonias pigmentadas (que varían desde color amarillo claro hasta amarillo-naranja oscuro), lo que las distingue rápidamente de las colonias blancas brillantes formadas por los estafilococos coagulasa negativos. Los S. aureus crecen rápidamente bajo condiciones aeróbicas y anaeróbicas en agar sangre, y muchos otros medios especializados. Las colonias individuales 278 ESTAFILOCOCOS: procedimientos de aislamiento e identificación en el laboratorio tienden a ser bien definidas y lisas. Aunque su nombre (estafilococo dorado) refleja su tendencia de adquirir una pigmentación amarilla, tal fenómeno ocurre solo bajo determinadas condiciones de crecimiento. Por ejemplo, las colonias pequeñas y las colonias que crecen en ambiente anaeróbico no desarrollan dicha pigmentación. Son bastante resistentes a cambios ambientales, a la desecación y a incrementos de la temperatura de hasta 50ºC, lo que les permite sobrevivir por largo tiempo en el polvo, suelo, fomites y la ropa. Tales características facilitan su transmisión intrahospitalaria. Identificación En el laboratorio es crítico poder diferenciar entre las tres especies de estafilococos que causan el mayor número de infecciones humanas: S. aureus, estafilococos coagulasa negativas, y S. saprophyticus. La prueba más frecuentemente utilizada para dicho propósito es la prueba de la coagulasa. Las cepas de S. aureus son coagulasa positivas mientras que las otras especies son coagulasa negativas. El S. saprophyticus pertenece al grupo de estafilococos coagulasa negativas. Se diferencia de los otros miembros del grupo por su fermentación del manitol y su resistencia a la novobiocina. Resistencia a los antibióticos En este respecto se destacan dos procesos muy importantes: 1. La evolución de la resistencia a los betalactámicos puesto que dichos fármacos son los fármacos más eficaces contra los estafilococos. En situaciones donde la resistencia a la meticilina es elevada se requiere el uso de los glicopéptidos. 2. El desarrollo de cepas con resistencia intermedia o de alto nivel a la vancomicina; proceso que igualmente afecta a otros glicopéptidos. Producción de enzimas El S. aureus produce muchas enzimas, algunas de las cuales afectan la patogénesis del proceso de infección. Las enzimas más importantes son las siguientes: 1. Catalasa: Convierte al peroxido de hidrogeno en agua y oxigeno dentro 279 Vigilancia, prevención y control de infecciones asociadas a servicios de salud del S. aureus. Durante el proceso de fagocitosis puede reducir la capacidad de los leucocitos polimorfonucleares de matar al S. aureus. 2. Coagulasa: Su presencia permite distinguir entre el S. aureus y otros estafilococos. Juega un menor papel en la patogénesis de infección. 3. Hialuronidasa: Contribuye a la formación de abscesos y a la difusión de la infección a través de los tejidos. 4. Beta-lactamasa: Inactiva a los antibióticos betalactámicos. Toxinas El S. aureus produce varias toxinas. Aquellas de importancia clínica incluyen: 1. Toxina exfoliativa: Grupo constituido por lo menos por dos toxinas (eta y etb) que separan las células del estrato córneo y la capa granulosa de la epidermis. Los síntomas incluyen una eritrodermia generalizada y dolorosa que es acompañada por el desprendimiento de las capas superficiales de la epidermis. Este lugar de separación permite su distinción de la necrólisis epidérmica tóxica o síndrome de Lyell, causada por reacción a un fármaco, donde la separación ocurre en la capa basal. 2. Hemolisinas: La toxina hly (hemolisina) tiene un extenso campo de actividad lítica celular que incluye los fagocitos del hospedero. Es la mayor hemolisina del S. aureus. Su ausencia reduce dramáticamente la virulencia del S. aureus en modelos en animales. También produce hemolisina ß que contribuyen a su patogenicidad pero que son factores de virulencia de menor potencia. 3. Toxinas asociadas con el síndrome de choque toxico (SCT): Este síndrome fue descrito inicialmente en niños pero luego recibió mayor atención cuando ocurrió un brote epidémico en mujeres jóvenes durante sus reglas. Posteriormente se determinó la relación del SCT con el uso de tampones intravaginales hiper absorbentes. Subsiguientemente se ha observado el mismo síndrome en otros tipos de infecciones causadas por S. aureus, incluyendo infecciones de heridas quirúrgicas, operaciones del tabique nasal e inserción de prótesis mamarias. El SCT ocurre en personas que albergan cepas de S. aureus productoras de toxinas. La presentación clínica es aguda con fiebre, vómitos, diarrea y progresión al choque y desarrollo de disfunción de múltiples órganos. Se reconoce la 280 ESTAFILOCOCOS: procedimientos de aislamiento e identificación en el laboratorio presencia de eritrodermia generalizada, hiperemia conjuntival y faringea. La eritrodermia produce descamación extensa. El tratamiento principal se dirige al manejo del choque y a la administración de antimicrobianos contra S. aureus. 4. Enterotoxinas: El S. aureus produce por lo menos once enterotoxinas serologicamente distintas (Enterotoxinas estafilococcicas A a Q) que son la segunda causa más común de contaminación alimenticia en los EE.UU, particularmente de comidas basadas en productos lácteos. La falta de refrigeración adecuada o la presencia de comida no bien cocida permite la multiplicación del organismo y la producción de enterotoxinas. Dichas toxinas carecen de sabor y son relativamente estables frente al calor, lo que les permite producir enfermedad en comida parcialmente recalentada. Después de un corto periodo de incubación (1 a 6 horas) se manifiestan los síntomas de estímulo vagál, vómitos, retorcijones abdominales, cólicos y diarrea. La deshidratación, si severa, puede complicar la evolución clínica. No se observa fiebre. Por lo general, los síntomas duran menos de 24 horas. El diagnóstico requiere el aislamiento del S. aureus y/o su toxina del alimento contaminado. El tratamiento es sintomático, particularmente con rehidratación. Moléculas adherentes reconocedoras de componentes de la superficie Son proteínas en la superficie del organismo que le permiten adherirse a los tejidos del hospedero, por lo que se las ha denominado receptores. Aquellas cepas del S. aureus con elevados números de receptores para los huesos o para las proteínas que se encuentran en válvulas cardiacas dañadas tienen mayor chance de causar osteomielitis o endocarditis, respectivamente. Métodos especiales para distinguir cepas diferentes Durante un brote epidémico o una investigación epidemiológica nosocomial, es a veces importante poder diferenciar entre cepas de S. aureus. Los procedimientos de biotipificación y la comparación de los patrones de susceptibilidad antimicrobiana muchas veces proporcionan pautas o indicios acerca de la identidad de las cepas. Sin embargo, el S. aureus puede demostrar muy poca variabilidad en las reacciones bioquímicas de biotipificación o en sus patrones de susceptibilidad lo que complica los esfuerzos de diferenciación entre cepas. Por tal razón, se ha desarrollado otros métodos para poder distinguir una cepa de otra: 281 Vigilancia, prevención y control de infecciones asociadas a servicios de salud 1. Tipificación del bacteriófago. Es el método tradicional utilizado para distinguir cepas diferentes del S. aureus durante la investigación de brotes epidémicos. Sin embargo, su uso es limitado por los siguientes factores: elevado costo, necesidad de utilizar un laboratorio de referencia, pobre poder discriminativo, pobre reproducibilidad y elevada complejidad. Por encima, algunas cepas del S. aureus no son susceptibles a la fagolisis lo que hace que no se las pueda tipificar por este método (especialmente cepas SAMR). 2. Técnicas de tipificación epidemiológica por métodos de biología molecular. Cada grupo de organismos posee un patrón de ADN plasmídico o genómico que lo caracteriza. Cuando se identifica cepas con el mismo patrón de ADN en diferentes pacientes eso sugiere que los patógenos provienen de un mismo clono. El encontrar el mismo clono en diferentes pacientes que tienen una relación geográfica o de tiempo sugiere transmisión del patógeno de paciente a paciente a partir de una fuente común. Los métodos de biología molecular ayudan investigar brotes de infecciones nosocomiales, sobre todo por SAMR donde se ha acumulado bastante experiencia con esta técnica. Se recomienda utilizar el siguiente protocolo para identificar y separar a los diferentes miembros, clínicamente significativos, del género Staphylococcus y para establecer el antibiograma. 282 ESTAFILOCOCOS: procedimientos de aislamiento e identificación en el laboratorio Protocolo para Género: Staphylococcus (*)Depende del medio utilizado para esta prueba de acidificación de azucares, si se utiliza púrpura de bromocresol la interpretación será: • Violeta a Vino: Negativo • Amarillo: Positivo II.Pero si se utilizase el indicador azul de bromotimol la interpretación será como esta en EL cuadro. 283 284 �������� ��������� �������������� ����������� ���������� ����������� ���������� ���� ���� �������� �������� �������� ������� ������� ������ ��������� �������������� �������� �������� ���������� ������������� ��������������� ��������� �������� �������� �������� �������� Vigilancia, prevención y control de infecciones asociadas a servicios de salud Flujograma de interpretación de pruebas bioquímicas para SCN ESTAFILOCOCOS: procedimientos de aislamiento e identificación en el laboratorio ESQUEMA DE TRABAJO 1. Mètodo simlificado de identificación Tabla 1. Apertura para identificación de especies Staphylococcus novobiocina S. ���������� ���������� (+) 90 % o más de los aislamientos estudiados reacción positiva; (+) 90 % o más reacción devilmente positiva (-) 50 % o más de los atendidos reacción negativa,(V) 11-29 % de los atendidos reacción pacífica ND no determinada: () reacción tardía 285 Vigilancia, prevención y control de infecciones asociadas a servicios de salud GRUPO S. epidermis: S. epidermis, S. lugdunensis (TABLA 1.1) GRUPO 1.a: S. warneri, S. hominis subespecie hominis, S. lugdunensis, S. simulans (TABLA 1.2) GRUPO 1.b: S. capitis subespecie urealyticus, S. hominis subespecie hominis, S. simulans (tabla 1.3) GRUPO 1.c: S. haemolyticus, S. lugdunensis, S. auricularis (TABLA 1.4) GRUPO 1.d: S. achleiferi subespecie schleiferi, S. auricularis (TABLA 1.5) Tabla 1.1. Esquema de diferenciación de las especies del GRUPO S. epidermis ����������� (+) 90% o más de los aislamientos estudiados reacción positiva: (±) 90% o más reacción dévilmente positiva; (-) 90% o más de los aislamientos reacción negativa: (V) 11-89% de los aislamientos reacción positiva: ND no determinada; () reacción tardía 286 ESTAFILOCOCOS: procedimientos de aislamiento e identificación en el laboratorio Tabla 1.2. Esquema de diferenciación de las especies del GRUPO 1.a β (+) 90% o más de los aislamientos estudiados reacción positiva: (±) 90% o más reacción dévilmente positiva; (-) 90% o más de los aislamientos reacción negativa: (V) 11-89% de los aislamientos reacción positiva: ND no determinada; () reacción tardía Tabla 1.3. Esquema de diferenciación de las especies del GRUPO 1.b (+) 90% o más de los aislamientos estudiados reacción positiva: (±) 90% o más reacción dévilmente positiva; (-) 90% o más de los aislamientos reacción negativa: (V) 11-89% de los aislamientos reacción positiva: ND no determinada; () reacción tardía 287 Vigilancia, prevención y control de infecciones asociadas a servicios de salud Tabla 1.4. Esquema de diferenciación de las especies del GRUPO 1.c - + + + + + - - - (+) 90% o más de los aislamientos estudiados reacción positiva: (±) 90% o más reacción dévilmente positiva; (-) 90% o más de los aislamientos reacción negativa: (V) 11-89% de los aislamientos reacción positiva: ND no determinada; () reacción tardía Tabla 1.5. Esquema de diferenciación de las especies del GRUPO 1.d (+) 90% o más de los aislamientos estudiados reacción positiva; (±) 90% o más reacción débilmente positiva; (-) 90% o más de los aislamientos reacción negativa; (V) 11-89% de los aislamientos reacción positiva; ND no determinada; ( ) reacción tardía 288 ESTAFILOCOCOS: procedimientos de aislamiento e identificación en el laboratorio Tabla 2. Apertura para la identificación de las especies de Staphylococcus novobiocina resistente β (+) 90% o más de los aislamientos estudiados reacción positiva; (±) 90% o más reacción débilmente positiva; (-) 90% o más de los aislamientos reacción negativa; (V) 11-89% de los aislamientos reacción positiva; ND no determinada; ( ) reacción tardía GRUPO 2.a: S. kloosii, S. equorum, S. xylosus S. cohnii subespecie urealyticus, GRUPO 2.b: S. hominis subespecie novobiosepticus, S. saprophyticus subespecie saprophyticus, S. kloosii GRUPO 2.c: S. cohnii subespecie cohnii, S. kloosii, S. grupo sciuri 289 Vigilancia, prevención y control de infecciones asociadas a servicios de salud Tabla 2. 1. Esquema de diferenciación de las especies del GRUPO 2.a (+) 90% o más de los aislamientos estudiados reacción positiva; (±) 90% o más reacción débilmente positiva; (-) 90% o más de los aislamientos reacción negativa; (V) 11-89% de los aislamientos reacción positiva; ND no determinada; ( ) reacción tardía Tabla 2. 2. Esquema de diferenciación de las especies del GRUPO 2.b (+) 90% o más de los aislamientos estudiados reacción positiva; (±) 90% o más reacción débilmente positiva; (-) 90% o más de los aislamientos reacción negativa; (V) 11-89% de los aislamientos reacción positiva; ND no determinada; ( ) reacción tardía 290 ESTAFILOCOCOS: procedimientos de aislamiento e identificación en el laboratorio Tabla 2. 3. Esquema de diferenciación de las especies del GRUPO 2.c β β (+) 90% o más de los aislamientos estudiados reacción positiva; (±) 90% o más reacción débilmente positiva; (-) 90% o más de los aislamientos reacción negativa; (V) 11-89% de los aislamientos reacción positiva; ND no determinada; ( ) reacción tardía Bibliografía 1. Murray, BE.The life and times of the Enterococcus. Clin Microbiol Rev 1990; 3: 46. 2. Scheifler KH, Kilpper-Balz R. Transfer of Streptococcus faecalis and S. faecium to the genus Enterococcus nom. Rev. as Enterococcus faecalis comb. nov. and E. faecium, comb. nov. In J Syst Bacteriol 1984; 34:31. 3. Facklam RR, Sahm DA. Enterococcus. Manual of Clinical Microbiology, 6th ed, 1995.p.308. 4. Texeira LM, Faclam RR, Steigerwalt AG, et al. Correlation between phenotypic characteristics and DNA relatedness within Enterococcus faecium strains J Clin Microbiol 1995; 33:1520. 5. Singh KV, Coque TM, Weinstock GM, Murray BE. In vivo testing of an Enterococcus faecalis fea mutant and use of efeA homologs for species identification. FEMS Imnunol Med Microbiol 1998; 21: 323. 6. Coque TM, Murray BE. Identification of Enterococcus faecalis strains by DNA hybridoization and pulsed-field electrophoresis. J Vclin Microbiol 1995; 33:3368. 7. Tyrrell GJ, Bethune RN, et al. Species identification of enterococci via intergenic ribosomal PCR. J Clin Microbiol 1997; 35:1054. 8. Durán L, Damiani E, Trigoso C. Guía para la toma de muestras de laboratorio. En Infecciones por 291 Vigilancia, prevención y control de infecciones asociadas a servicios de salud enterococos y estafilococos: Manual de Aislamiento e Identificación y Guía de Tratamiento. Manuales Técnicos, Ministerio de Salud y Deporte, Bolivia, 2006 p4. 9. Leclercq R, Derlot E, Duval J, Courvalin P. Plasmid mediated resistance to vancomycin and teicoplanin in Enterococcus faecium. N Engl J Med 1988; 319:157-161. 10. Uttley AHC, Collins CH, Naidoo J, George RC. Vancomycin-resistant enterococci. Lancet 1988; 1:57-58. 11. Tenover FC, Tokars J, Swenson J, Paul S, Spitalny K, Jarvis W. Ability of clinical laboratories to detect antimicrobial agent-resistant enterococci. J Clin Microbiol 1993; 31:1695-1699. 12. Arthur M, Courvalin P. Genetics and mechanisms of glycopeptide resistance in enterococci. Antimicrob Agents Chemother 1993; 37:1563-1571. 13. Torres C, Reguera JA, Sanmartin MJ, Perez-Diaz JC, Baquero F. VanA-mediated vancomycin-resistant Enterococcus spp in sewage. J Antimicrob Chemother 1994; 33:553-561. 14. Cercenado E. Resistencia de los enterococos a los antibióticos glucopéptidos. Rev Clin Esp 1995; 195 (Supl 4):22-27. 15. Woodford N, Johnson AP, Morrison D, Speller DCE. Current perspectives on glycopeptide resistance. Clin Microbiol Rev 1995; 8:585-615. 16. Leclercq R, Courvalin P. Resistance to glycopeptides in enterococci. Clin Infect Dis 1997; 24:545-556. 17. Leclerq R. Enterococci acquire new kinds of resistance. Clin Infect Dis 1997; 24(Supl):S80-S84. 18. Perichon B, Reynolds O, Courvalin P. VanD-type glycopeptide resistant Enterococcus faecium BM4339. Antimicrob Agents Chemother 1997; 41:2016-2018. 19. Anónimo. National Committee for Clinical Laboratory Standards. Performance standards for antimicrobial susceptibility testing; eighth informational supplement. NCCLS 1998; 18(1): Documento M100-S8. 20. French GL. Enterococci and vancomycin resistance. Clin Infect Dis 1998; 27(Supl 1):S75-S83. 21. Linden PK. Clinical implications of nosocomial gram positive bacteremia and superimposed antimicrobial resistance. Am J Med 1998; 104(5A):24S-33S. 22. Liassine N, Frei R, Jan I, Auckenthaler R. Characterization of glycopeptide-resistant enterococci from a Swiss hospital. J Clin Microbiol 1998; 36:1853-1858. 23. Moellering RC. The specter of glycopeptide resistance: current trends and future considerations. Am J Med 1998; 104(5A):3S-5S. 24. Moellering RC. Vancomicyn-resistant enterococci. Clin Infec Dis 1998; 26:1196-1199. 25. Murray BE. Diversity among multidrug resistant enterococci. Emerging Infect Dis 1998; 4:37-47. 26. Walker M, Durán L, Flores A. Enterococos: Resistencia antimicrobiana y prevención de infección intrahospitalaria. En Infecciones por Enterococos y Estafilococos: Aislamiento e Identificación en Laboratorio y Guía de Tratamiento. Durán L, Damián E, Trigoso C, Eds. Instituto Nacional de Laboratorios en Salud- INLASA, Ministerio de Salud y Deporte, La Paz, Bolivia. 2006, p 44-57. 27. Durán L, Damiani E, Trigoso C. Guía para la toma de muestras de laboratorio. En Infecciones por enterococos y estafilococos: Manual de Aislamiento e Identificación y Guía de Tratamiento. Manuales Técnicos, Ministerio de Salud y Deporte, Bolivia, 2006 p4. 292 ESTAFILOCOCOS: procedimientos de aislamiento e identificación en el laboratorio 28. Woodford N, Johnson AP, Morrison D, Speller DCE. Current perspectives on glycopeptide resistance. Clin Microbiol Rev 1995; 8:585-615. 29. Anónimo. National Committee for Clinical Laboratory Standards. Performance standards for antimicrobial susceptibility testing; eighth informational supplement. NCCLS 1998; 18(1): Documento M100-S8. 30. Linden PK. Clinical implications of nosocomial Gram positive bacteremia and superimposed antimicrobial resistance. Am J Med 1998; 104(5A):24S-33S. 31. Moellering RC. The specter of glycopeptide resistance: Current trends and future considerations. Am J Med 1998; 104(5A):3S-5S. 32. Madigan MT, Martinko JM, Parker J. Biología de los microorganismos. Prentice Hall. 8 ª ed. Pág.: 986 33. Guerra Mercado J. Bacteriología clínica. 1º edición. La Paz - Bolivia. Pág:191-213 34. Herold BC, Immergluck LC, Maranan MC, et al. Community-acquired methicillin resistant S. aureus in Children with no identified predisposing risks. JAMA 1998; 279: 593 35. Gillet Y, Issartel B, Vanhems P, et al. Association beteween S. aureus strains carrying gene for PantonValentine leucocidin and highly letal necrotizing pneumonia in young immunocompetent patients. Lancet 2002; 359 (9308): 753 36. Proctor R. Staphylococcus. In APIC Text of Infection Control and Epidemiology. Association for Professionals in Infection Control and Epidemiology, 2nd Ed. Washington. 2005; 69-1 38. Rocca M.F., Manual de procedimientos, Identificación mediante caràcteres fenotípicos y normalización de la metodología para Staphylococcus coagulasa negativa, Servicio de Bacteriología especial, Depto. de Bacteriología; ANLIS - Dr. Carlos Malbran, Bs. As., 2006. 293 Vigilancia, prevención y control de infecciones asociadas a servicios de salud 294