Estudio de procesos dinamicos por RMN
Anuncio
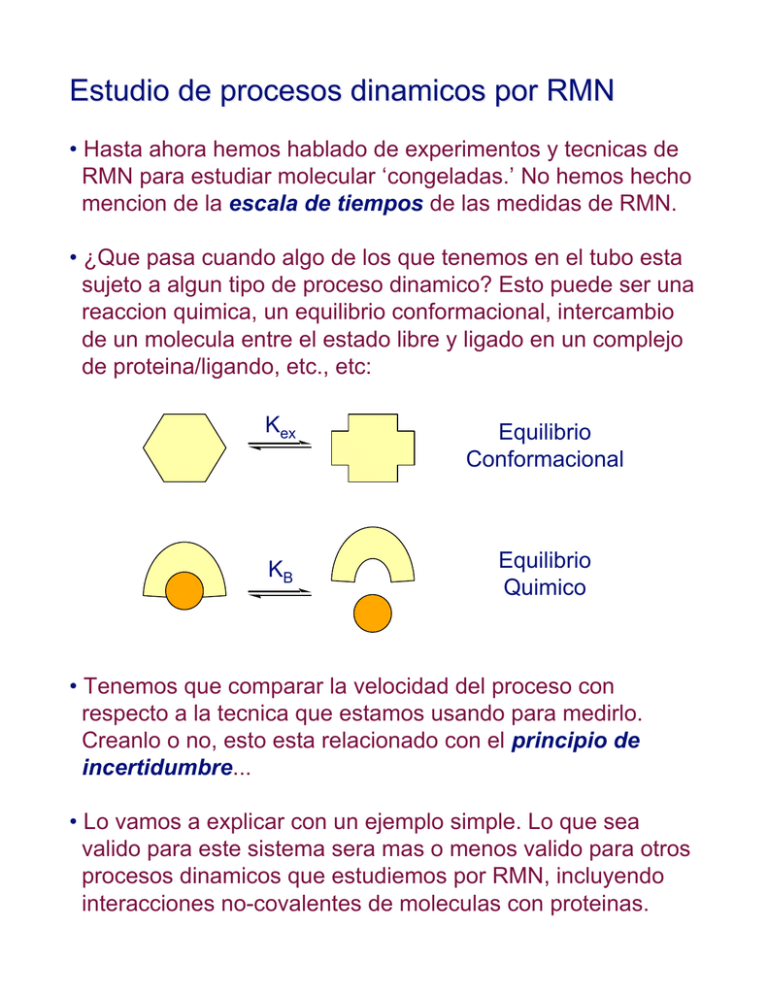
Estudio de procesos dinamicos por RMN • Hasta ahora hemos hablado de experimentos y tecnicas de RMN para estudiar molecular ‘congeladas.’ No hemos hecho mencion de la escala de tiempos de las medidas de RMN. • ¿Que pasa cuando algo de los que tenemos en el tubo esta sujeto a algun tipo de proceso dinamico? Esto puede ser una reaccion quimica, un equilibrio conformacional, intercambio de un molecula entre el estado libre y ligado en un complejo de proteina/ligando, etc., etc: Kex Equilibrio Conformacional KB Equilibrio Quimico • Tenemos que comparar la velocidad del proceso con respecto a la tecnica que estamos usando para medirlo. Creanlo o no, esto esta relacionado con el principio de incertidumbre... • Lo vamos a explicar con un ejemplo simple. Lo que sea valido para este sistema sera mas o menos valido para otros procesos dinamicos que estudiemos por RMN, incluyendo interacciones no-covalentes de moleculas con proteinas. Medida de constantes de velocidad • Digamos que el proceso que estamos analizando es la inversion de la N,N-dimetilformamida (DMF): O N O H N H • Sabemos que los metilos rojo y azul van a intercambiar sitios relativamente lento debido al caracter de doble enlace de la amida. Los dos metilos son quimica y magneticamente diferentes, y un espectro RMN de la DMF da una señal para cada metilo: • Esto significa que el intercambio entre los dos sitios es lo suficientemente lento comparado a la diferencia de frecuencia relativa entre las resonancias de las dos especies (roja y azul): 1 Intercambio (s) >> 1 o δr - δa Δδ Constantes de velocidad (continuado) • Ahora empezamos a calentar la muestra. Como la velocidad de la inversion depende de su ΔG y este esta afectado por T, temperaturas mas altas hacen que las cosas vayan mas rapido. Vemos lo siguiente: T TC • A cierta temepratura, llamada la temperatura de coalesenscia, la constante de intercambio entre las especies es comparable a la diferencia de corrimiento quimico entre los dos sitios: 1 Intercambio (s) ~ 1 o δr - δa Δδ • Despues de este punto, no podemos distinguir un sitio de otro con medidas de RMN porque el intercambio ocurre mas rapido que la diferencia de frecuencia relativa. Constantes de velocidad (mas...) • Vemos que hay dos regiones a medida que variamos la temperatura, llamdas de intercambio rapido y lento: Δδ * velocidad > 1 Intercambio lento Δδ * velocidad = 1 Transicion (TC) Δδ * velocidad < 1 Intercambio rapido • Como podemos estimar la temperatura de la transicion (la temperatura de coalecenscia), podemos sacar datos acerca de la termodinamica y la cinetica del proceso. • Si hiciecemos un estudio detallado, veriamos que tenemos que tomar en cuanta las poblaciones de los dos sitios (uno puede estar mas favorecido que el otro energeticamente), asi como tambien los anchos de linea de los picos. • Como hicimos con otras derivaciones matematicas, vamos a usar resultados aproximados que sirven para lo que queremos explicar. • Nos concentramos en el caso con sitios igualmente populados (energias iguales), osea que la diferencia de energia libre es debida solo al proceso de intercambio que estemos estudiando. Constantes de velocidad (...y mas) • A partir de Δδ (en Hz) en el limite de intercambio lento calculamos la constante de intercambio a la temperatura de coalecenscia: Kex = π * Δν / √ 2 = 2.22 * Δν • Usamos frecuencias en radianes y por eso es que aparece el factor de π. Esta ecuacion tiene muchas simplificaciones (nunca sabremos si la temperatura mas baja a la que hicimos el experimento es realmente intercambio lento, y no consideramos los anchos de linea). • De cualquier manera, funciona bastante bien. Con la temperatura de coalecenscia calculamos el ΔG‡ del proceso con otra relacion aproximada: ΔG‡ = R * TC* [ 22.96 + ln ( TC / Δν ) ] • Si no tomamos en cuenta las contribuciones entropicas al ΔG‡, podemos calcular las constantes de reaccion a cualquier otra temperatura solo con esta informacion. • Con RMN podemos medir constantes entre 10-2 y 108 s-1. Ejemplo de equilibrio conformacional • Como parte de mi trabajo con taxol, trate de preparar una analogo de la cadena lateral rigido para evaluar si el imponer rigidez mejoraba o empeoraba la actividad biologica. • Decidi hacer un sistema bifenilo, que a la larga demonstro ser una muy mala idea. Si hubiese leido un poco hubiese sabido que estas cosas se comportan medio raro.... • Considerado que tengo 10 pulgares, hacerlo fue todo un logro (me llevo muuuuucho tiempo). Al tomar un 1H, vi que al parecer tenia dos ‘cosas’ en solucion: O O NH O NH OMe OMe OH O OH • Algo estaba mal. Tenia solo una manchita por TLC, asi que las cuentas no me cerraban. Llegue a la conclusion de que teniamos un equilibio lento de dos conformeros en solucion, algo que los bifenilos hacen (haber leido...). Equilibrio conformacional (continuado) • Como me habia matado por 4 meses haciendo esto, no lo iba a dejar por eso. Tambien, tener dos conformeros no era bueno para probar nada. Si esto era un equilibrio, un cambio en temperatura tenia que afectar las velocidades de intercambio: • Vimos que a) habia coalesencia de los dos grupos de señales entre 80 y 100oC, y b) que el proceso era reversible. Equilibrio conformacional (mas…) • En este caso, la inversion de los anillos no ocurre sola, y hay otros cambios conformacionales asociados. Tambien puede haber puentes-H formandose/rompiendose, asi que es dificil elegir dos señales para hacer los calculos de la barrera de energia de inversion. • Si elejimos un par de picos aromaticos (los anillos son los se que invierten), medimos un δν de 0.04 ppm (o 20 Hz a 500 MHz): 20 Hz • Considerando que la coalecensia de estos dos picos ocurre a ~85oC (358 K), y usando la formula para ΔG‡: Kex ~ 44.4 s-1 ΔG‡ ~ 18.5 Kcal/mol • No esta tan mal despues de todo… Conformacion de ligandos - TRNOE • Una de las cosas mas importantes en el diseño de drogas es averiguar como se ligan a su receptor, Que generalmente es una proteina o ADN. • Con esta informacion e informacion de las REA podemos preparar analogos que no solo tengan los requerimientos quimicos para ser activos, pero tambien los requerimientos conformacionales impuestos por el sitio activo. • Una forma es estudiar la conformacion de la molecula aislada (por rayos-X o RMN), y asumir que va a tener la misma cononformacion en el sitio activo. • En moleculas flexibles (el 99.9% de los casos…), el cambio en el entorno (polaridad, grupos no-polares, puentes-H, etc.) al pasar del agua al sitio activo va a afectar la conformacion. + Libre Ligado • Osea, para estudiar la conformacion del ligando en el sitio activo hay que estudiarlo en el sitio activo o bajo el ‘efecto’ del sitio activo… Conformacion de ligandos (continuado) • Dependiendo del tamaño del receptor podemos determinar la estructura del complejo y usar eso (rayos-X, RMN). • Esto consume muchisimo tiempo, y vamos a estar determinando, principalmente, la estructura del receptor. Lo que queremos es saber la conformacion del ligando... • Ademas, la mayoria de los receptores son enormes (de 100 a 200 KDa), y los interesantes estan en membranas, osea que no les podemos (¿podiamos?) entrar con nada. • Lo que, en algunos casos, nos salva son las diferencias relativas entre la velocidad de crecimiento del NOE para protones del ligando (la velocidad de crecimiento de la relajacion cruzada) y las constantes de asociacion entre el ligando y el receptor. • Digamos que tenemos el siguiente complejo ligando/receptor: * HI * HS • Cuando este en el sitio activo los protones indicados van a tener NOE. Van a ser muy dificiles de ver en el complejo poque vamos a tener toneladas de NOE’s del receptor consigo mismo. Conformacion de ligandos (mas…) • Supongamos que el ligando se disocia y vuelve a la solucion Va a adoptar su conformacion en solucion rapidisimo: HI * HI * koff * H HS kon * H kunf * * HS • Generalmente, koff (o la constante de disociacion) es mucho mas lenta que kunf (la velocidad a la que el ligando sufre ‘unfolding’), osea que solo nos preocupamos por koff. Definimos todas las constantes de la siguiente forma: K= kon koff [ligando-receptor] = [receptor] [ ligando] • Independientemente de la constante koff, la interaccion NOE entre los dos protones que aparecio cuando el ligando estaba en el sitio activo va a persistir por un tiempo (que depende de la relajacion espin-red, T1). Conformacion de ligandos (y mas…) • Esto significa que si el proceso de asociacion/disociacion es rapido comparado con T1, el NOE que surgio entre los 1H’s cuando el ligando estaba en el sitio activo va a persistir luego de que el ligando se disocie y adopte su conformacion en solucion ¿Porque? • Tenemos que considerar todo el proceso: RIF IF Kon koff σISF RSF IB RIB σISB SF Kon koff SB RSB • El acople diploar entre I y S ocurre para el ligando en el sitio activo (‘bound,’ IB, SB) y en solucion (‘free,’ IF, SF). Cuando el ligando este en el sitio activo, su tiempo de correlacion va a ser enorme (tenemos a la proteina), y la relajacion cruzada entre I y S esta dominada por σISB. • Por eso los NOE’s que surgen en el sitio activo persisten en solucion. Ademas, en el complejo el ligando va a tener un ω * tc aprente mayor que 1, y siempre vamos a estar sobre el limite de difusion de espin (osea, NOE’s negativos...). Conformacion de ligandos (y mas...) • Es mas, si tenemos buen intercambio (‘turnover’) comparado con el tiempo de relajacion T1, vamos a tener varios ligandos interactuando con el mismo receptor antes de que el NOE del primero decaiga. • Osea, podemos hacer el experimento con un exceso de ligando (i.e., 10 veces mas), y las señales del ligando van a ser mas grandes que las del receptor (que van a ser anchas y van a aparecer todas superpuestas...). • Otra cosa buena de medir NOE’s de ligandos en el sitio activo por TRNOE es que como vamos a estar mirando al ligando en solucion, sus señales van a estar bien definidas (anchos de linea relativamente chicos) : L ligado L libre proteina • Las señales ‘finas’ son mas altas (tienen la misma area), se diferencian bien del ‘background’ de la proteina, y dan cross-peaks mucho mejor resueltos en un experimento 2D. Conformacion de ligandos (…y mas) • Si parece demasiado bueno para ser verdad, es demasiado bueno para ser verdad. Para poder usar TRNOE se tienen que dar un monton de cosas (todas juntas): • El ligando no puede interactuar mucho con el receptor, ya que necesitamos intercambio constante entre los ligandos libres y los ligandos en el sitio activo. • La constante Koff tiene que ser mucho mas chica que el tiempo de relajacion T1, sino el NOE desaparece antes de que lo podamos detectar. • En la practica, esto limita mucho el tipo de sistemas que podemos analizar usando esta tecnica... Gradientes de campo magnetico y difusion • Todo lo que hemos visto sucede en campos magneticos ‘perfectos’ (i.e., Bo homogeneos). Esto lo necesitamos para tener buena resolucion/sensibilidad. Sin embargo, crear un gradiente con caracteristicas conocidas en Bo es muy util. • Un gradiente de campo resulta en diferentes Bs. Si solo consideramos una variacion linear en el eje z (i.e., un gradiente-z, o Gz) y una muestra de agua, veremos que las moleculas de agua con distintas zs tendran diferentes δs (porque δ ∝ γ (Bo+ Gz)): Bo δ Bo+ Gz δ • La señal es proporcional a la ‘masa’ en la muestra… Gradientes de campo magnetico (continuado) • Y esto es util? Para empezar, podemos generar una imagen de la muestra si aplicamos un gradiente durante la adquisicion de datos. Como los espines en distintos lugares del tubo tienen distintos δs, obtenemos un espectro ‘continuo’ que refleja la forma del contenedor. ω (Hz) Bo+ Gz • Osea, un gradiente de campo linear deja a los espines codificados espacialmente. Esto significa que ‘sabemos’ a que parte del tubo (o ‘brazo’) pertenecia el espin basados en el gradiente que aplicamos. Si agregamos contraste basado en los tiempos de relajacion del agua en distintos tejidos obtenemos RMI (mas o menos…). • Ademas, si combinamos gradientes de distinto signo podemos codificar los espines, dejarlos evolucionar (pulsos, tiempos, etc.), y luego decodificarlos. • Espines que (no) se comportan de cierta manera durante el periodo de evolucion, (no) aparecen en el espectro… Gradientes y difusion • Y esto deriva en medidas de difusion. Medir el coeficiente de difusion (D) es muy importante en quimica/biologia. Reporta sobre mobilidad de moleculas, interacciones intramoleculares, etc., etc. • La espectroscopia de gradientes de campo pulsados es ideal para medir la difusion de particulas con nucleos que poseen RMN. La tecnica mas basica implica combinar un eco de espin con dos gradientes de signo opuesto y largo δ separados un tiempo Δ: 90y 180y Δ/2 1H: Δ/2 Δ Canal de gradientes: δ δ • Hay que analizar que hara el eco de espin bajo los efectos del gradiente codificador/decodificador sobre nucleos que difunden a distintas velocidades. Hay que recordar que un gradiente hace que las cosas se muevan mas rapido/lento en el sistema rotatorio (i.e., cambian el ‘corrimiento quimico’). Gradientes y difusion (continuado) • Para un espin que se mueve poco (luego del pulso π / 2): y y G(δ) x x y 180 Δ/2 y -G(δ) x Δ/2 x • Aca los puntos rojo/azul representan al mismo tipo de nucleo en distintas regiones del ‘tubo’… • Basicamente, como el espin no se mueve los gradientes tienen poco efecto. Osea, el espin no se movio del area original, y el gradiente decodificador tiene un efecto igual y opuesto al codificador (reenfoque). En otras palabras, la intensidad de la señal para ese nucleo no cambia… • Muy distinto para un espin que se mueve rapido… Gradientes y difusion (continuado) y y G(δ) x x y 180 Δ/2 y -G(δ) x Δ/2 x • En este caso, y como el nucleo se mueve lejos de donde estaba originalmente, el gradiente decodificador tiene un efecto completamente distinto (i.e., continua el desfasaje). En suma, la intensidad de la señal va a terminar muy atenuada. • El resultado final es que cuanto mas se muevan los espines, mas pequeñas seran las señales en el espectro. Elijiendo bien los valores de G, δ, y Δ podemos ajustar el experimento para estudiar difusividades de hasta 10-12 m2s-1 (en un Bruker como el de Malvin Norte). Gradientes y difusion (continuado) • Para calcular el valor de D, repetimos el experimento con muchos valores de G manteniendo δ y Δ constantes. • La variación de intensidad contra gradiente que se obiente guarda la siguiente relacion, y un regresion nos da el D. I(G) = Io x e [–γ2G2δ2D x (Δ – δ / 3)] • Datos reales para el [Emim][OAc] a 40 oC: y = m1*exp(-10000*m2*(2*pi*4... Value Error m1 1.0048 0.0017074 m2 1.5413e-14 6.2518e-17 Chisq 0.00016229 NA R 0.99995 NA 1.2 1 N N y = m1*exp(-10000*m2*(2*pi*4... Value Error m1 0.61144 0.00088819 m2 1.7099e-14 5.8874e-17 Chisq 4.1973e-5 NA R 0.99997 NA CH3 0.8 Intensity H3C O H3C O 0.6 0.4 0.2 0 0 5 10 15 20 Gz (G/cm) 25 30 35





