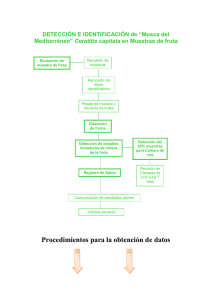
00 Hoja Universidad v2 F - Universidad Politécnica de Madrid
Anuncio

UNIVERSIDAD POLITÉCNICA DE MADRID E.T.S.I. AGRÓNOMOS BASES MOLECULARES DE LA RESISTENCIA A INSECTICIDAS EN LA MOSCA MEDITERRÁNEA DE LA FRUTA Ceratitis capitata (WIEDEMANN) TESIS DOCTORAL FRANCISCO COUSO FERRER Licenciado en Biología 2012 DEPARTAMENTO DE BIOTECNOLOGÍA E.T.S.I. AGRÓNOMOS BASES MOLECULARES DE LA RESISTENCIA A INSECTICIDAS EN LA MOSCA MEDITERRÁNEA DE LA FRUTA Ceratitis capitata (WIEDEMANN) FRANCISCO COUSO FERRER Licenciado en Biología PEDRO HERNÁNDEZ CRESPO Dr. en Ciencias Biológicas Universidad de Córdoba 1993 - ÍNDICE Pág. RESUMEN I ABSTRACT V Capítulo 1: INTRODUCCIÓN GENERAL 1 1.1. Ceratitis capitata 3 1.1.1. Descripción de la especie y distribución geográfica 3 1.1.2. Importancia como plaga 5 1.1.3. Métodos de lucha contra C. capitata 7 1.2. Resistencia a insecticidas 11 1.2.1. Definición de resistencia 11 1.2.2. La resistencia a insecticidas en números 12 1.2.3. Resistencia cruzada 14 1.2.4. Mecanismos de resistencia 15 1.2.4.1. Resistencia metabólica 1.2.4.1.1. Glutatión S-transferasas 16 1.2.4.1.2. Esterasas 16 1.2.4.1.3. Enzimas citocromo P450 17 1.2.4.1.3.1. Definición y nomenclatura 17 1.2.4.1.3.2. Identificación y diversidad 19 1.2.4.1.3.3. Estructura y función 21 1.2.4.1.3.4. Resistencia a insecticidas mediada por P450 21 1.2.4.2. Resistencia por modificación de la molécula diana 1.2.4.2.1. La acetilcolinesterasa 1.2.4.3. Otros mecanismos de resistencia 1.2.5. Resistencia en C. capitata y otras especies de la familia Tephritidae 1.3. Objetivos de la tesis 15 24 25 25 26 29 Pág. Capítulo 2: SELECCCIÓN DE RESISTENCIA EN LABORATORIO Y SUSCEPTIBILIDAD A INSECTICIDAS EN Ceratitis capitata 31 2.1. Introducción 33 2.2. Material y métodos 35 2.2.1. Ceratitis capitata en el laboratorio 35 2.2.1.1. Condiciones generales de cría 35 2.2.1.2. Mantenimiento de líneas de C. capitata bajo presión de selección 37 2.2.2. Líneas de laboratorio 38 2.2.3. Recogida de poblaciones de campo 40 2.2.3.1. Adaptación de las poblaciones de campo al laboratorio 2.2.4. Bioensayos de susceptibilidad a insecticidas 40 41 2.2.4.1. Condiciones generales 41 2.2.4.2. Bioensayos para estimar la CL50 43 2.2.4.3. Bioensayos de concentración discriminante 45 2.2.5. Bioensayos con el quimioesterilizante lufenurón 2.3. Resultados 2.3.1. Evaluación de la susceptibilidad a malatión y espinosad en poblaciones de campo 47 49 49 2.3.2. Selección de resistencia a malatión en el laboratorio: Establecimiento de las líneas W-4Km y W-10Km 52 2.3.3. Resistencia cruzada en la línea W-4Km 55 2.3.4. Selección de resistencia a espinosad en laboratorio: Establecimiento de la línea Xàbia-W-100s 2.4. Discusión 57 59 Capítulo 3: BASES PARA LA DETECCIÓN PRECOZ DE LA RESISTENCIA: DIVERSIDAD DE GENES CYP 65 3.1. Introducción 67 3.2. Material y métodos 71 Pág. 3.2.1. Insectos 71 3.2.2. Extracción de DNA 71 3.2.3. Extracción de RNAT y RNAm 72 3.2.4. Síntesis de cDNA 73 3.2.5. Amplificación por PCR de los genes CYP 74 3.2.6. Electroforesis y extracción de bandas 75 3.2.7. Clonación, RFLP y secuenciación 75 3.2.8. Análisis de las secuencias 76 3.2.9 Bioensayos de exposición a fenobarbital 77 3.2.10. PCR cuantitativa en tiempo real 77 3.3. Resultados 79 3.3.1. Diversidad de genes CYP 79 3.3.2. Expresión de los genes CYP 90 3.3.3. Inducción por fenobarbital 94 3.4. Discusión 97 Capítulo 4: DETERMINACIÓN DE LA ESTRUCTURA DE LOS GENES Ccace2 Y Ccae7 Y DESARROLLO DE SISTEMAS DE DETECCIÓN PRECOZ DE ALELOS DE RESISTENCIA 101 4.1. Introducción 103 4.2. Material y métodos 107 4.2.1. Obtención de intrones mediante GenomeWalkerTM 107 4.2.2. Amplificación de los exones de Ccace2 y Ccae7 113 4.2.3. Desarrollo de un método de detección de la mutación AChE G328A 115 4.3. Resultados 119 4.3.1. Estructura del gen Ccace2 119 4.3.2. Estructura del gen Ccae7 120 4.3.3. Amplificación de Ccae7 y Ccace2 en individuos de distintas regiones geográficas 121 4.3.4. Método de detección del alelo Ccace2R portador de la mutación AChE G328A 122 Pág. 4.3.5. Distribución geográfica del alelo Ccace2R 124 4.3.6. Frecuencia genotípica de Ccace2 en las líneas de laboratorio resistentes a malatión 130 4.4. Discusión 133 Capítulo 5: NUEVO MECANISMO DE RESISTENCIA A MALATIÓN EN Ceratitis capitata 137 5.1. Introducción 139 5.2. Material y métodos 143 5.2.1. Líneas de laboratorio y cruzamientos 143 5.2.2. Evaluación del número de copias del gen Ccace2 145 5.2.3. Cuantificación de la expresión de Ccace2 146 5.2.4. Análisis de la actividad AChE y su inhibición por malaoxón 147 5.2.5. Caracterización de los perfiles RFLP 148 5.2.6. Análisis de la mutación AChE G328A mediante secuenciación 149 5.2.7. Método PCR-RFLP para la detección de la duplicación Ccace2RS 149 5.2.8. Estimación del tiempo de desarrollo, porcentaje de emergencia y supervivencia de los adultos pertenecientes a las líneas C y W-10Km 5.3. Resultados 151 153 5.3.1 Cruzamientos y descendencia de las líneas C y W-10Km 153 5.3.2. Determinación del número de copias del gen Ccace2 155 5.3.3. Cuantificación de la expresión de Ccace2 156 5.3.5 Caracterización del perfil RFLP de los individuos con fenotipo heterocigoto para la mutación AChE G328A 158 5.3.6. Confirmación del genotipo asociado al perfil RFLP mediante cruzamientos y PCR cuantitativa 161 5.3.7. Análisis de los patrones de secuenciación de la mutación AChE G328A 163 5.3.8. Detección de la duplicación Ccace2RS mediante un sistema de doble PCR-RFLP 165 5.3.9. Detección de la duplicación Ccace2RS en poblaciones de campo 167 Pág. 5.3.10. Comparación del desarrollo, porcentaje de emergencia y supervivencia de adultos en las líneas C y W-10Km 169 5.4. Discusión 171 CONCLUSIONES 177 BIBLIOGRAFÍA 181 ANEXOS 215 I. Protocolo para la elaboración de la dieta para las larvas de C. capitata 217 II. Secuencias intrónicas de Ccace2 y Ccae7 219 ÍNDICE DE TABLAS 221 ÍNDICE DE FIGURAS 223 Resumen. La mosca mediterránea de la fruta Ceratitis capitata (Wiedemann, 1824) está considerada una de las plagas clave para la fruticultura. El malatión es un insecticida organofosforado que fue empleado mayoritariamente en España para el control de C. capitata hasta 2009, año en el que dejó de utilizarse por no estar incluido en el anexo I de la Directiva Europea 91/414/ECC. El incremento del uso del malatión, debido a las graves pérdidas económicas causadas por C. capitata, provocó la aparición de poblaciones de campo resistentes. El estudio de una población resistente a malatión, recogida en Castelló en 2004, permitió la identificación de dos mecanismos de resistencia: una mutación puntual (G328A) en la acetilcolinesterasa (AChE) y un mecanismo de resistencia metabólica, probablemente mediado por carboxilesterasas. Teniendo en cuenta estos antecedentes, nos propusimos estudiar los mecanismos implicados en la resistencia a malatión en C. capitata. Además, durante el desarrollo de esta Tesis, el malatión fue sustituido por otros insecticidas como el espinosad y la lambda-cialotrina para el control de la plaga. En este nuevo contexto, es extremadamente importante analizar la susceptibilidad de poblaciones de campo frente a espinosad y estudiar la posible existencia de resistencia cruzada a estos insecticidas, así como sentar las bases para el estudio de futuros mecanismos de resistencia. En primer lugar, analizamos mediante bioensayos con dosis discriminante la susceptibilidad a malatión y espinosad en doce poblaciones de C. capitata de Andalucía, Aragón, Cataluña, Comunidad Valenciana e Islas Baleares; y nuestros resultados sugirieron la presencia de individuos resistentes a malatión en la mayoría de las poblaciones analizadas. En el caso del espinosad, observamos que la susceptibilidad a este insecticida de origen biológico fue elevada en la mayoría de las poblaciones, sin embargo, la población recogida en Xàbia (Alicante) mostró un nivel de susceptibilidad unas dos veces menor al resto de poblaciones. Mediante la selección en laboratorio, obtuvimos dos líneas resistentes a malatión, W-4Km y W-10Km, con unos niveles de resistencia con respeto a la línea susceptible C de 178 y 400 veces, respectivamente. Además, se seleccionó por primera vez en C. capitata una línea altamente resistente a espinosad (Xàbia-W-100s), que actualmente es unas 500 veces más resistente que la línea de laboratorio C. I Con el objetivo de escoger la estrategia más adecuada para el manejo de la plaga, estudiamos la susceptibilidad a diferentes tipos de insecticidas en la línea resistente a malatión W4Km. En esta línea detectamos resistencia cruzada moderada a los organofosforados fentión, diazinón, fosmet, triclorfón y metil-clorpirifos (de 7 a 16 veces) y frente al carbamato carbaril, al piretroide lambda-cialotrina y al quimioesterilizante lufenurón (de 4 a 6 veces). Por otra parte, la resistencia cruzada frente a espinosad fue baja (1,5 veces). Es importante destacar que los niveles de resistencia estimados frente a todos los insecticidas fueron de uno o dos órdenes de magnitud inferiores al observado en la línea W-4Km frente a malatión (178 veces), hecho que podría deberse, al menos, a dos posibles hipótesis: que la mutación AChE G328A confiera mayor insensibilidad al malaoxón (forma activa del malatión) que a otros insecticidas que tienen como diana la AChE y/o, en segundo lugar, que el mecanismo de resistencia mediado por carboxilesterasas hidrolice el malatión de manera más eficiente que los otros insecticidas analizados. En el estudio de nuevos mecanismos de resistencia en C. capitata, por un lado, analizamos la diversidad de enzimas citocromo P450, asociadas con resistencia metabólica en otras especies, y por otro lado, desarrollamos un sistema para la detección de nuevas mutaciones puntuales que pudiesen aparecer en los genes que codifican la AChE (Ccace2) y la aliesterasa (Ccae7). Mediante el empleo de cebadores degenerados obtuvimos 37 genes CYP, que codifican enzimas P450, pertenecientes a cinco familias. Posteriormente, en un estudio de inducción con fenobarbital, observamos que la expresión de cuatro de los seis genes analizados era susceptible de ser inducida. Por otro lado, se puso a punto un sistema que permite amplificar y secuenciar, a partir de DNA genómico, los exones de los genes Ccace2 y Ccae7 en los que se han encontrado mutaciones relacionadas con resistencia a insecticidas en otras especies. Los resultados obtenidos facilitarán el estudio de nuevos mecanismos de resistencia mediados por estas enzimas en C. capitata. Se diseñó un método PCR-RFLP para identificar los individuos portadores de la mutación AChE G328A (alelo de resistencia Ccace2R) sin la necesidad de realizar bioensayos y que, además, permite detectar resistencia cuando ésta se encuentra a baja frecuencia. Según el análisis realizado, el alelo Ccace2R se observó en 25 de las 27 localidades españolas muestreadas en el territorio español, incluyendo las Islas Baleares y Canarias. Sin embargo, este alelo no se detectó en poblaciones procedentes de once países y de cinco continentes. El análisis de la presencia del alelo Ccace2R en las líneas resistentes a malatión durante el proceso de selección en el laboratorio mostró una rápida disminución de los homocigotos, tanto para el alelo susceptible como para el alelo de resistencia, en favor de los individuos heterocigotos. II Así, después de 52 generaciones de selección, se observó que la totalidad de los individuos analizados de la línea W-10Km presentaban un genotipo heterocigoto para la mutación AChE G328A. Este desequilibrio contradice la segregación mendeliana esperada para un gen con dos alelos pero podría ser explicado por la existencia de una duplicación del gen Ccace2. La demostración de la presencia de esta duplicación se realizó mediante: i) el cruzamiento de individuos heterocigotos de la línea W-10Km con homocigotos susceptibles de la línea C, que dio lugar a una descendencia en la que el 100% de los individuos eran heterocigotos; ii) la evaluación del número de copias del gen Ccace2 por PCR cuantitativa en tiempo real (qPCR), que resultó dos veces mayor en individuos de la línea W-10Km en comparación con los de la línea C; iii) el análisis del nivel de expresión de Ccace2, que fue el doble en la línea W-10Km con respecto a la línea C, y iv) el estudio de la actividad AChE, que resultó mayor en los individuos de la línea W-10Km. Según los resultados obtenidos, una duplicación del gen Ccace2 provoca la coexistencia en un mismo cromosoma del alelo silvestre y del alelo mutado y, además, las dos copias del gen Ccace2, al estar ligadas, producen una heterocigosis permanente (Ccace2RS). De esta manera se explica que el hecho de que 100% de los individuos de la línea W-10Km mostrasen un perfil de restricción correspondiente a un individuo heterocigoto ya que, en realidad, eran homocigotos estructurales para la duplicación (genotipo CCace2RS/RS). Se ha detectado un coste biológico asociado a la duplicación que consiste en un incremento en la mortalidad acumulada de los adultos a partir del séptimo día después de la emergencia. La descripción de la duplicación Ccace2RS supone la identificación de un nuevo mecanismo de resistencia a malatión en C. capitata. Finalmente, mediante el diseño de un método de doble PCR-RFLP se determinó la presencia de la duplicación Ccace2RS en la mayoría de las poblaciones españolas. La proporción de individuos portadores de la duplicación osciló entre el 5% y el 35%, observándose los mayores valores de frecuencia en las poblaciones de C. capitata recogidas en la cuenca mediterránea. Podemos por lo tanto concluir que la resistencia a malatión asociada a la mutación AChE G328A y a la duplicación Ccace2RS está ampliamente establecida en las poblaciones españolas de C. capitata. Nuestros resultados desaconsejan la utilización del malatión (si fuera de nuevo autorizado) o de otros organofosforados para el control de esta plaga. Además, una de las líneas resistentes a malatión mostró resistencia cruzada frente a insecticidas con diferentes modos de acción y que se utilizan actualmente para el control de C. capitata, tales como lambda-cialotrina y lufenurón. La alta susceptibilidad a espinosad observada en las poblaciones españolas, así como la reducida resistencia cruzada estimada para este insecticida, sugieren que su utilización es adecuada para el control de la plaga. Sin embargo, la utilización de un sólo insecticida puede entrañar riesgos por favorecer la selección de resistencia, de hecho, mediante selección en laboratorio se obtuvo una población altamente resistente a espinosad. Por tanto, es recomendable implementar programas de control integrado y de manejo de la resistencia en C. capitata utilizando III distintos sistemas de control e insecticidas con diferentes mecanismos de acción que permitan su sostenibilidad en el tiempo. Los sistemas de detección de alelos de resistencia desarrollados en este trabajo permitirán la detección precoz de resistencia en campo, facilitando la decisión sobre el sistema de control más adecuado. Además, los conocimientos generados podrán contribuir al desarrollo de nuevos sistemas de detección para otros mecanismos de resistencia. IV Abstract. The Mediterranean fruit fly, Ceratitis capitata (Wiedemann, 1824), is considered one of the most harmful pests in fruit crops. Until 2009, when malathion use was banned due to its not inclusion in the Annex I of Directive 91/414/EEC, the application of this organophosphate (OP) insecticide in Spain increased gradually due to the large economic losses caused by C. capitata. The increase in the frequency of treatments resulted in the development of resistant field populations. The study of a malathion-resistant population, collected in 2004 in Castelló (Comunidad Valenciana), allowed the identification of two resistance mechanisms: a single point mutation (G328A) in the target acetylcholinesterase (AChE), as well as a metabolic resistance mechanism, most likely carboxylesterase-mediated. Taking all the preceding into account, we studied the malathion resistance mechanisms in C. capitata. During the development of this PhD Thesis malathion use was banned by the European Union, being replaced by other insecticides, such as spinosad and lambda-cyhalotrin. Within this new working frame, the need to analyse the possible existence of cross-resistance to these insecticides and the susceptibility to spinosad in field populations was raised. This would define the baseline for future studies on resistance mechanisms. Firstly, through discriminant dose bioassays, we analysed malathion and spinosad susceptibility in twelve C. capitata populations from Andalucia, Aragon, Cataluña, C. Valenciana and the Baleares Islands. Our results suggest the presence of malathion-resistant individuals in most of the populations analysed. Regarding spinosad, we noticed a high susceptibility to this biologically derived insecticide in most of the populations, but in the one collected in Xabia (Alicante), which had a susceptibility level two times lower than the rest of populations. Through laboratory selection, we obtained two malathion-resistant strains, W-4Km and W-10Km, with resistance levels 178- and 400-fold, respectively, compared to the control susceptible C strain. Besides, a strain highly-resistant to spinosad (Xabia-W-100s), 500-times more resistant than control C strain, was selected. In order to decide the most appropriate management strategy for the pest, we studied the susceptibility to different insecticides in the malathion-resistant W-4Km strain. We detected a V moderated cross-resistance to the OPs fenthion, diazinon, phosmet, trichlorphon and methylchlorpyrifos (7- to 16-fold), and to the carbamate carbaryl, the pyretroid lambda-cyhalotrin and the chemosterilizer lufenuron (4- to 6-fold). On the other hand, cross-resistance to spinosad was low (1.5-fold). It is important to note that resistance levels to all insecticides were one or two orders of magnitude less than that observed against malathion in W-4Km strain (178-fold), a fact that might be due to, at least, two possible causes: mutation AChE G328A may provide a higher insensitivity to malaoxon (the active form of malathion) than to other insecticides having AChE as target, and/or, secondly, the carboxylesterase-mediated resistance mechanism hydrolyzes malathion more efficiently than all other analysed insecticides. To investigate new resistance mechanisms in C. capitata we analysed the diversity of the cytochrome P450 enzymes, which have been associated to metabolic resistance in insects, and we developed a new method to detect single point mutations in acetylcholinesterase (Ccace2) and aliesterase (Ccae7) genes that could appear. Using degenerate primers we obtained 37 CYP genes, coding P450 enzymes, included in five families. Afterwards, in a phenobarbital-induction study, we observed that the expression of 4 out of the 6 analysed genes could be induced. On the other hand, a system was set up to amplify and to sequence from genomic DNA the exons of genes Ccace2 and Ccae7 where mutations related to insecticide resistance have been found in other species. The results obtained could facilitate the study of new resistance mechanisms in C. capitata mediated by these enzymes. A PCR-RFLP method was designed to detect the presence of the mutation AChE G328A (resistance allele Ccace2R), with no need to perform bioassays and allowing detecting resistance at low frequency. According to the analysis, the resistance allele was found in 25 out of 27 sampled locations in Spain, including the Balearic and the Canary Islands. However, this allele was not detected in other populations collected in 11 countries from 5 continents. The follow-up of the presence of the allele Ccace2R in the malathion-resistant strains during the selection process in the laboratory showed a quick decrease in homozygous individuals, for both the susceptible and the resistant alleles, favouring heterozygous. Thus, after 52 generations of selection, all the individuals analysed from W-10Km strain showed a heterozygous genotype for mutation AChE G328A, contradicting mendelian segregation as expected for a gene with two alleles. Afterwards, we were able to demonstrate that this was caused by the presence of a duplication of the gene coding acetylcholinesterase by: i) crossing heterozygous individuals from W-10Km strain with susceptible homozygous from C strain, originating a F1 population in which 100% of individuals were heterozygous; ii) evaluating the number of copies of gen Ccace2 by quantitative PCR in real time (qPCR), that happened to be twice higher in individuals from W-10Km VI strain when compared with C strain; iii) analysing the level of expression of Ccace2, twice in W10Km strain when compared to C strain; iv) studying the acetylcholinesterase activity, that was higher in individuals from W-10Km strain. According to these results, duplication of gen Ccace2 originates the coexistence of the susceptible and the resistant allele in the same chromosome. The two linked copies of the gene Ccace2 provoke the existence of permanent heterozygosis (Ccace2RS). This explains why the 100% of individuals from W-10Km strain showed an heterozygous restriction pattern since, in fact, they were structural homozygotes for the duplication (genotype Ccace2RS/RS). A biological cost has been detected associated to this duplication, consisting in a rise in accumulated adult mortality from the seventh day after emergence. The Ccace2RS duplication described in this study represents a new resistance mechanism to malathion in C. capitata. Finally, by the design of a double PCR-RFLP method, the presence of Ccace2RS duplication was confirmed in most of the Spanish populations. We observed that the proportion of individuals carrying the duplication oscillated between 5 and 35%, the frequency being higher in those C. capitata populations collected in the area of the Mediterranean basin. Therefore, we can conclude that malathion resistance associated to mutation AChE G328A and to Ccace2RS duplication are widely distributed in Spanish populations of C. capitata. Our results advice against the use of malathion (if it came to be newly authorized for use) or other OPs for the control of this pest. Besides, one of the malathion-resistant strains showed cross-resistance against insecticides with diverse action modes that are currently used for pest control, such as lambdacyhalotrin and lufenuron. High susceptibility to spinosad in the Spanish populations, as well as the reduced cross-resistance estimated for this insecticide suggests its adequacy for Medfly control. However, the use of a single insecticide is a risky strategy since it favours the selection of resistance. In fact, a population highly resistant to spinosad was obtained through laboratory selection. Therefore, it is advisable to implement integrated pest management (IPM) and resistance management programs for C. capitata control. Using insecticides with different modes of action and diverse control systems would contribute to the sustainability of the pest control. The resistance allele detection systems developed through this work will allow the early detection of resistance in the field, making possible the selection of the most appropriate method for pest control. Besides, the generated knowledge may also contribute to the development of new detection systems for other resistance mechanisms. VII 1. Introducción general ______________________________________________________________________________________________________________________________________ Capítulo 1 INTRODUCCIÓN GENERAL 1 1. Introducción general ______________________________________________________________________________________________________________________________________ 1.1. Ceratitis capitata. 1.1.1. Descripción de la especie y distribución geográfica. Ceratitis capitata (Wiedemann, 1824), comúnmente conocida como la mosca mediterránea de la fruta, pertenece al orden Diptera, suborden Brachycera, familia Tephritidae. Dentro de esta familia, que engloba a más de 5.000 especies, se encuentran las “verdaderas moscas de la fruta”, que son un grupo de más de 1.400 especies que emplean frutas carnosas para su desarrollo. Los principales géneros de tefrítidos son Ceratitis, Bactrocera, Anastrepha y Rhagoletis, y todos ellos tienen especies que constituyen plagas (Aluja y Mangan, 2008). El género Ceratitis posee alrededor de 78 especies, de las cuales C. capitata es la más importante. Es una especie polífaga, que ataca a más de 300 especies frutícolas y hortícolas (Liquido et al., 1991; Copeland et al., 2002), y es una de las principales plagas de los cultivos frutícolas en el mundo. Es una especie multivoltina (Bateman, 1972), con tres o cuatro generaciones al año en zonas templadas con inviernos fríos, y hasta ocho en zonas en las que las temperaturas mínimas en invierno están por encima de los 0ºC (Ruiz-Castro, 1945), como es el caso de la vertiente mediterránea de la Península Ibérica. 3 1. Introducción general ______________________________________________________________________________________________________________________________________ La duración de su ciclo biológico, cuando las condiciones ambientales son favorables, es de aproximadamente un mes, aunque cuando la temperatura es baja puede durar entre dos y tres meses (Christenson y Foote, 1960). La hembra de C. capitata penetra la piel del fruto por medio del oviscapto (Fig. 1.1.a) y deposita en su interior de uno a diez huevos, que eclosionan transcurridos entre dos y cuatro días (McDonald y McInnis, 1985). Las larvas, que pasan por tres estadios, se alimentan de la pulpa de la fruta en la que se hospedan hasta que, una vez alcanzado el tercer estadio, saltan del fruto para pupar en el suelo. Los individuos adultos emergen tras un período de entre seis y once días, dependiendo de la temperatura, son de color pardo amarillento y pueden alcanzar los 5 mm de longitud. Existen rasgos anatómicos que permiten una clara diferenciación sexual a simple vista: las hembras presentan un ovipositor al final del abdomen (Fig.1.1.b) y los machos tienen un par de quetas negras en forma de rombo situadas en la región cefálica frontal. (Fig. 1.1.c/d). a b c d Figura 1.1. Detalle del ovipositor de las hembras y de las quetas de los machos de C. capitata. (a) Momento de la puesta de los huevos en el fruto por parte de la hembra. (b) Detalle del ovipositor. (c) Cabeza de un macho de C. capitata. (d) Detalle de la queta característica de los machos de la especie. Las micrografías, tomadas con microscopio electrónico de barrido, han sido realizadas en el Servicio de Microscopía de la UCM. C. capitata es una especie originaria de la zona subsahariana de África, desde donde se ha extendido al resto del mundo (Fig. 1.2), principalmente a la región mediterránea y a zonas del centro y sur de América, ocupando zonas templadas, tropicales y subtropicales (Fischer-Colibrie y 4 1. Introducción general ______________________________________________________________________________________________________________________________________ Busch-Petersen, 1989). Aunque con una distribución más reducida, también se pueden encontrar poblaciones de este tefrítido en EE.UU., México y zonas de Asia, Sudáfrica y Australia (Hagen et al., 1981; Fimiani, 1989; Fletcher, 1989; Davies et al., 1999, EPPO, 2011), siendo considerada actualmente como una importante plaga de cuarentena en numerosos países (Malacrida et al., 2007). En España su distribución es amplia, encontrándose poblaciones tanto en la Península Ibérica como en las Islas Baleares y Canarias (Fimiani, 1989; Beroiz et al., 2012). Ampliamente distribuida Distribución limitada No presente EPPO (European and Mediterranean Plant Protection Organization), 2011 Figura 1.2. Distribución de la mosca mediterránea de la fruta. Los tefrítidos han evolucionado hacia un estilo de vida de fitófagos oportunistas, por lo que las plantas que utilizan como hospedador tienen una gran influencia tanto en la supervivencia de la especies como en su dispersión geográfica. Además, debido a la combinación de factores naturales y humanos, como la globalización del comercio, esta familia de dípteros ha logrado extenderse rápidamente por todo el mundo (Malacrida et al., 2007). 1.1.2. Importancia como plaga. La picadura de la hembra de C. capitata durante la oviposición produce un orificio en la piel del fruto que favorece la entrada de microorganismos como hongos y bacterias en su interior. Esto, junto con el efecto producido por las galerías originadas por las larvas, favorece una prematura caída del fruto y su putrefacción, impidiendo que éste pueda ser empleado para el consumo humano. 5 1. Introducción general ______________________________________________________________________________________________________________________________________ Según la EPPO (European and Mediterranean Plant Protection Organization) en Europa y en la región mediterránea, algunos de los principales hospedadores de C. capitata son los cítricos (Citrus spp.), los aguacates (Persea americana), los higos (Ficus carica), los kiwis (Actinidia deliciosa), los mangos (Mangifera indica), los nísperos (Mespilus germanica) y los melocotones (Prunus spp.) (EPPO, 2011). En España, desde 1955, el Ministerio de Agricultura considera a la mosca mediterránea de la fruta como plaga de especial incidencia en la agricultura. El censo agrario realizado en 2009 por el Instituto Nacional de Estadística (consultados en INE, 2012) muestra que España dedica en torno a 300.000 ha al cultivo de cítricos, principalmente en la Comunidad Valenciana, Región de Murcia, Andalucía, Islas Baleares y Cataluña. Según los datos de la FAO del año 2010 (consultados en FAO, 2012), España es el primer productor europeo de cítricos con más de cinco millones de toneladas y uno de los principales productores a nivel mundial, sólo superado por China, Brasil, India, EE.UU. y México (Fig. 1.3). Mundo 25000 Europa 6000 2010 2010 Cítricos Mandarinas y naranjas Mandarinas y naranjas Producción (t x 1000) Producción (t x 1000) 20000 Cítricos 5000 15000 10000 4000 3000 2000 5000 1000 0 0 China Brasil India EE.UU. México España Turquía Egipto España Italia Grecia Portugal Croacia Figura 1.3. Principales países productores de cítricos. Datos del año 2010, pertenecientes a la FAO. Además, desde mediados de los años sesenta, España ha sido ininterrumpidamente el mayor exportador de naranjas y mandarinas a nivel mundial. Por ejemplo, en 2009 exportó alrededor de 3 millones de toneladas (Fig. 1.4), lo que representa un 56% de la producción total de los cítricos del país, y cuyo valor de mercado superó en 2009 los dos mil trescientos millones de euros (consultado en FAO, 2012). Las pérdidas económicas directas ocasionadas por C. capitata en los cultivos de cítricos españoles son muy elevadas debido al daño producido en la fruta tras el ataque de la plaga, 6 1. Introducción general ______________________________________________________________________________________________________________________________________ pérdidas que se han visto incrementadas en los últimos años tras la expansión de los cultivos de variedades extra-tempranas de cítricos, muy sensibles al ataque de la mosca. Por ejemplo, en la C. Valenciana, además de en los cítricos, C. capitata es capaz de desarrollarse en una gran cantidad de frutos a lo largo del año: desde las frutas de primavera (níspero, ciruela, melocotón…) hasta las de final de año (mandarinas, caqui…), pasando por una gran variedad de especies intermedias, lo que mantiene activas las poblaciones de C. capitata durante todo el año, produciendo graves pérdidas económicas en los cultivos citrícolas. Además de las pérdidas directas, se generan pérdidas indirectas, causadas por las restricciones de cuarentena exigidas en algunos países para la exportación y la comercialización. Actualmente, según la EPPO, C. capitata está considerada como una plaga de cuarentena tipo A2, categoría que posee unos estrictos protocolos de cuarentena. Concretamente el protocolo para C. capitata ha sido revisado recientemente, incluyendo para su diagnóstico caracteres morfológicos y métodos moleculares (EPPO, 2011). 2009 Argentina Mandarinas Marruecos Naranjas China Países Bajos Grecia Turquía EE.UU. Sudáfrica España 0 200 400 600 800 1000 1200 1400 1600 Cítricos exportados (t x 1000) Figura 1.4. Principales exportadores de cítricos a nivel mundial. Datos del año 2009, pertenecientes a la FAO. 1.1.3. Métodos de lucha contra C. capitata. Las principales estrategias de control de la mosca mediterránea de la fruta que se han realizado en España durante los últimos años son el monitoreo de las poblaciones y el control químico (Primo-Millo et al., 2003). El monitoreo de las poblaciones consiste en el empleo de 7 1. Introducción general ______________________________________________________________________________________________________________________________________ trampas que permiten, mediante la captura de individuos adultos, evaluar la abundancia de la plaga a lo largo de una campaña. El control químico es el método más utilizado, ya sea individualmente o combinado con otros métodos, y se basa en la aplicación de diferentes compuestos insecticidas. A continuación se describen brevemente algunos aspectos de los insecticidas que se han empleado en este trabajo y que, a excepción del carbaril, han sido o están siendo aplicados en España para combatir la mosca mediterránea de la fruta en los cultivos de cítricos: · Organofosforados. Los organofosforados son un grupo de insecticidas que, junto con los carbamatos, inhiben la actividad enzimática de la acetilcolinesterasa (Eto, 1974). En este trabajo hemos estudiado los efectos de seis organofosforados: fosmet, aplicado en trampas; diazinón, empleado para el tratamiento de la fruta caída al suelo; y malatión, fentión, triclorfón y metilclorpirifós, de aplicación aérea. · Espinosad. El espinosad es un insecticida de origen biológico constituido por productos (espinosinas) derivados de la fermentación realizada por una bacteria Gram + llamada Saccharopolyspora spinosa. El mecanismo de acción consiste principalmente en la activación de los receptores nicotínicos de la acetilcolina (Salgado et al., 1997), aunque también se han observado efectos negativos sobre la función del receptor GABA (Salgado, 1998). Este insecticida se aplica en una formulación que incluye un cebo de proteínas atrayentes y suele suministrarse a los cultivos en bandas o en parcheo. Su utilización, junto con el piretroide lambda-cialotrina, es la más extendida tras la prohibición del malatión a partir de 2009. · Lambda-cialotrina. Este insecticida es un piretroide sintético que actúa modulando los canales de sodio de las células nerviosas responsables de la transmisión del impulso nervioso (Narahashi, 1996). Su empleo frente a C. capitata se ha visto incrementado a partir de 2009 al no permitirse el uso de malatión. La aplicación de este insecticida se realiza en formulación cebo. · Carbaril. Es el único de los insecticidas ensayados en este trabajo que no ha sido autorizado para su uso contra C. capitata en España. Pertenece a la familia de los carbamatos y su mecanismo de acción es similar al de los organofosforados, inhibiendo la actividad acetilcolinesterasa (revisado por Oakeshott, 2005). El carbaril actúa por contacto, penetrando en el insecto a través de la cutícula. · Lufenurón. El lufenurón es una benzoilurea que actúa como regulador del crecimiento, inhibiendo la biosíntesis de quitina y produciendo alteraciones en el desarrollo de los insectos 8 1. Introducción general ______________________________________________________________________________________________________________________________________ durante sus primeros estadios (Navarro-Llopis et al., 2004). Además, en C. capitata, este insecticida posee una actividad quimioesterilizante (Casaña-Giner et al., 1999). El lufenurón se emplea en trampas en combinación con un cebo atrayente y su uso se ha incrementado durante las últimas campañas (Navarro-Llopis et al.; 2010). En España, el tipo de estrategia empleada para el control de la plaga dependerá de la especie de fruta afectada y de la comunidad autónoma en la que se lleve a cabo. En el plan de actuación integral para el control de la mosca mediterránea de la fruta en la C. Valenciana, donde el control de C. capitata es clave para la producción y exportación de cítricos se incluyen, además del control químico y del monitoreo, otros métodos de control como son el trampeo masivo, la técnica del insecto estéril (TIE) y la quimioesterilización (Castañera, 2003). Además, una práctica muy útil para disminuir los efectos de la plaga es la de eliminar toda la fruta caída en el suelo o que queda en el árbol tras la recolección para evitar la supervivencia de la mosca en su interior. El trampeo masivo consiste en capturar el mayor número de individuos de C. capitata que sea posible mediante trampas que contienen un cebo atrayente. Esta metodología se emplea normalmente a finales de la primavera y principios del verano, época en la que se produce un pico en la abundancia poblacional de la plaga. Su aplicación es especialmente útil en los terrenos próximos a las zonas urbanas, donde la aplicación de los tratamientos insecticidas por vía aérea es complicada (Alemany et al., 2004; Navarro-Llopis et al., 2008; Muñoz y García-Marí, 2009). La técnica del insecto estéril (TIE) o control autocida comenzó a aplicarse mediante un programa piloto en el municipio valenciano de Lliria durante los años 2003-2006. Desde 2006 hasta la actualidad el programa de actuación se ha extendido a toda la C. Valenciana. Este método de lucha consiste en la suelta de millones de machos estériles que se introducen en las poblaciones salvajes apareándose con las hembras y produciendo huevos infértiles (Gilmore, 1989). En estos momentos la capacidad de producción de la biofactoría, situada en Caudete de las Fuentes (Valencia), es de aproximadamente 500 millones de pupas a la semana. La principal ventaja de esta técnica es su elevada especificidad, ya que los machos estériles únicamente afectarían a la reducción de la descendencia de su misma especie. Sus principales desventajas son dos: sólo es eficaz cuando la abundancia de las poblaciones salvajes de la mosca es baja, por lo que su utilización ha de ser complementada con la de otros métodos de control (Argilés y Tejedo, 2007), y conlleva un elevado coste económico, al requerir la producción de millones de insectos a la semana. La reciente incorporación del quimioesterilizante lufenurón a las trampas con cebo atrayente también ha contribuido a una reducción significativa de las poblaciones de C. capitata 9 1. Introducción general ______________________________________________________________________________________________________________________________________ (Navarro-Llopis et al., 2004; 2010). La esterilidad provocada por este insecticida se ha observado tanto en hembras que han ingerido lufenurón, como en las que han copulado con machos que han ingerido el quimioesterilizante (Casaña-Giner et al., 1999). El control biológico, basado en la importación y el establecimiento de especies exóticas de depredadores y parasitoides, ha sido utilizado con éxito frente a varias plagas de cítricos (Wharton, 1989; Jacas et al., 2006). El control biológico de C. capitata se basa principalmente en la suelta masiva de parasitoides, método que tuvo mucho éxito en Hawai (Wong et al., 1991) y que actualmente, combinado con la liberación de machos estériles, se está aplicando en México, Guatemala y Costa Rica (Gurr y Kvedaras, 2009). En España, en el año 2002, se importaron dos especies de parasitoides bracónicos de los géneros Diachasmimorpha y Fopius y se realizaron ensayos para determinar su potencial como agentes de control de la mosca mediterránea de la fruta (Beitia et al., 2007). Además, se han realizado estudios para establecer el papel de los depredadores autóctonos en el control de las poblaciones de C. capitata (Urbaneja et al. 2006; Monzó et al., 2009; 2011). Incluso con la existencia de tan diversas técnicas, la aplicación de productos químicos sigue siendo el método más empleado contra C. capitata en España. El Ministerio de Agricultura, Alimentación y Medio Ambiente (MAGRAMA), elabora periódicamente una lista con los productos fitosanitarios aptos para su aplicación contra la mosca mediterránea de la fruta en España (http://www.magrama.gob.es/es/agricultura/temas/medios-de-produccion-/productos-fitosanitarios /fitos.asp). Durante la campaña 2007-2008 los insecticidas aprobados por el Ministerio frente a C. capitata fueron fentión, malatión, espinosad y lambda-cialotrina. De los insecticidas organofosforados fentión y malatión, fue este último el más utilizado y se aplicó en su formulación malatión-cebo en tratamientos aéreos y terrestres con una frecuencia que, en algunas zonas, alcanzó los diez tratamientos por año. A partir de 2009, debido a la no inclusión del malatión en el anexo I de la Directiva Europea 91/414/ECC, se prohibió el uso de malatión y se aplicaron fundamentalmente espinosad y lambda-cialotrina. Además, a partir de la campaña 2008-2009 se incrementó el uso del trampeo masivo con atrayentes y la aplicación del quimioesterilizante lufenurón (Navarro-Llopis et al., 2010). A mediados de 2010 (Directiva Europea 2010/17/UE) se aprobó de nuevo el malatión para su utilización en cultivos de fresas y de especies ornamentales, pero no para su aplicación contra C. capitata. Es importante destacar que las poblaciones de C. capitata analizadas en esta tesis fueron recogidas antes de 2009, época en la que el uso de malatión estaba autorizado. 10 1. Introducción general ______________________________________________________________________________________________________________________________________ 1.2. Resistencia a insecticidas. 1.2.1. Definición de resistencia. Según el IRAC (The Insecticide Resistance Action Committee) la resistencia se puede definir como una disminución heredable en la susceptibilidad de una plaga a un determinado insecticida que se refleja en un fallo continuado para alcanzar los niveles de control esperados aun cuando el insecticida se aplica en las concentraciones recomendadas (IRAC, 2012). La resistencia surge como una respuesta evolutiva natural al estrés ambiental y, aunque su aparición puede ser retrasada, difícilmente puede ser evitada (Hoy, 1998). Por ello, el control de plagas debe ir acompañado de un programa de manejo de la resistencia, siempre en el contexto del control integrado de plagas. A finales de los años cuarenta, pocos años después de su primera aplicación como insecticida, se describió el primer caso de resistencia a DDT en Musca domestica (Sacca, 1947). Este fenómeno se ha ido repitiendo con diferentes insecticidas hasta la actualidad, independientemente del mecanismo de acción y de la aparición de nuevas formulaciones (Whalon et al., 2012). Se han descrito casos de resistencia frente a los principales grupos de insecticidas: ciclodienos; carbamatos; DDT y análogos; organofosforados y piretroides; y más recientemente frente a los nuevos insecticidas neonicotinoides y espinosinas. El empleo de un determinado insecticida favorece la selección de aquellos individuos portadores de genes que confieren resistencia a dicho insecticida. La descendencia de estos individuos resistentes heredará esta característica, aumentando así el número de individuos resistentes en una determinada población, lo que provocará dificultades en el control de la plaga. Además, la pérdida de efectividad del insecticida, debida al aumento de la resistencia de la población, suele ir asociada a un incremento en la frecuencia de los tratamientos, lo que produce un coste ambiental y una disminución de la diversidad biológica del ecosistema. 11 1. Introducción general ______________________________________________________________________________________________________________________________________ 1.2.2. La resistencia a insecticidas en números. A partir de la base de datos de Arthropod Pesticide Resistance Database (Whalon et al., 2012) se puede realizar un análisis de la evolución de los casos de resistencia a lo largo del tiempo, así como de la distribución de los casos entre los diferentes taxones de insectos y ácaros. Desde los primeros descubrimientos de fallos en el control químico de los artrópodos plaga a principios del siglo XX, el número de especies en los que se han identificado casos de resistencia a insecticidas y/o acaricidas ha incrementado de manera exponencial hasta alcanzar las 573 especies en la actualidad (Fig. 1.5). 600 nº especies 500 400 300 200 100 0 1900 1920 1940 1960 1980 2000 2020 año Figura 1.5. Evolución de la resistencia a pesticidas. Se representa el número acumulado de especies de artrópodos en las que se ha detectado resistencia a insecticidas y/o acaricidas durante el último siglo (datos tomados de Whalon et al., 2012). Hasta el año 2012 se han descrito 8.795 casos de resistencia a insecticidas en 488 especies de insectos pertenecientes a 96 familias de 13 órdenes. El orden Diptera engloba el 40% de las especies descritas implicadas en procesos de resistencia, seguido de los órdenes Lepidoptera, Coleoptera y Homoptera (Fig. 1.6.A). Dentro del orden Diptera, la familia Culicidae contiene la mayoría de las especies relacionadas con resistencia (Fig. 1.6.B) y el 20% del total de los casos detectados. Esto se debe, problablemente, a que esta familia está muy estudiada, ya que dentro de ella se encuentran los géneros Anopheles y Aedes, vectores de enfermedades como la malaria, la fiebre amarilla o el dengue, además del género Culex, implicado en la transmisión de la enfermedad de la fiebre del Nilo. 12 1. Introducción general ______________________________________________________________________________________________________________________________________ Hymenoptera 3% Otros 7% Tephritidae 2% A B Chironomidae 3% Hemiptera 5% Calliphoridae 3% Diptera 40% Homoptera 12% Otras 14% Anthomyiidae 3% Agromyzidae 4% Simuliidae 7% Culicidae 64% Coleoptera 15% Lepidoptera 18% DÍPTEROS INSECTOS Figura 1.6. Distribución de las especies de insectos en los que se han identificado casos de resistencia. Se indica la proporción de las especies de insectos en los órdenes de la clase Insecta (A) y la distribución de las especies de dípteros en las diferentes familias (B). Datos tomados de Whalon et al. (2012). Es importante tener presente que dentro de las casi seiscientas especies de artrópodos en las que se han descrito casos de resistencia no todas presentan el mismo número de citas de poblaciones resistentes. Así, del total de casos de resistencia a pesticidas detectados hasta la fecha, únicamente nueve especies de insectos y un ácaro representan el 35% del total de los casos (Tabla 1.1). Tabla 1.1. Principales especies de artrópodos ordenadas según el número de casos de resistencia. (%) Representa el porcentaje de casos de cada especie con respecto al total de casos observados en los artrópodos. Datos tomados de Whalon et al. (2012). ______________________________________________________________________________________________________________________________________ Especie Familia Orden Nº casos % ______________________________________________________________________________________________________________________________________ Helicoverpa armigera Noctuidae Lepidoptera 639 6 Plutella xylostella Plutellidae Lepidoptera 453 5 Bemisia tabaci Aleyrodidae Homoptera 428 4 Myzus persicae Aphididae Homoptera 391 4 Tetranychus urticae Tetranychidae Acari 389 4 Aedes aegypti Culicidae Diptera 267 3 Culex quinquefasciatus Culicidae Diptera 256 3 Spodoptera litura Noctuidae Lepidoptera 238 2 Musca domestica Muscidae Diptera 227 2 Blattella germanica Blattellidae Dermaptera 213 2 ______________________________________ TOTAL 3501 35 ______________________________________________________________________________________________________________________________________ 13 1. Introducción general ______________________________________________________________________________________________________________________________________ 1.2.3. Resistencia cruzada. Una determinada población presenta resistencia cruzada cuando la resistencia que posee frente a un determinado insecticida favorece la resistencia frente a otros insecticidas, aunque la población no haya sido expuesta jamás a dichos insecticidas (IRAC, 2012). En el orden Diptera la mayoría de citas de resistencia cruzada a insecticidas pertenecen a estudios realizados en mosquitos (géneros Anopheles, Culex o Aedes), debido al gran interés en conocer la susceptibilidad a diferentes insecticidas de estos géneros que incluyen varios vectores de enfermedades infecciosas. Uno de los primeros trabajos de resistencia cruzada lo realizó Davidson en 1958 estudiando la susceptibilidad a diferentes tipos de insecticidas en tres especies del género Anopheles. En él, por ejemplo, describió que una población de A. gambiae altamente resistente al organoclorado dieldrina mostraba resistencia cruzada a otros ciclodienos, pero no frente a DDT. Además, en este género de mosquitos, se han descrito recientemente poblaciones resistentes a piretroides con resistencia cruzada a DDT (Fonseca-González et al., 2009). Por otro lado, en una población de C. pipiens resistente al organofosforados temefós se observó que mostraba resistencia cruzada a otros organofosforados y a DDT (Bisset et al., 1997). En el género Aedes se han descrito poblaciones resistentes a temefós de A. aegypti con resistencia cruzada a varios organofosforados (Rodríguez et al., 2002). En el suborden Brachycera del orden Diptera, que engloba a las moscas propiamente dichas, se ha detectado resistencia cruzada a varios organofosforados en Lucilia cuprina (Campbell et al., 1998a). Además, se ha descrito resistencia cruzada frente a otros piretroides en una población de M. domestica resistente a permetrina (Liu y Yue, 2000). Asimismo, se ha descrito resistencia cruzada al quimioesterilizante lufenurón en una línea de laboratorio de Drosophila melanogaster resistente a DDT (Daborn et al., 2002; Le Goff et al., 2003). Una de las escasas especies de importancia agrícola en la que se ha estudiado el fenómeno de resistencia cruzada ha sido en Bactrocera dorsalis, en la que una población resistente a espinosad no mostró resistencia cruzada frente a los diez insecticidas ensayados, entre los que había organofosforados, piretroides y un carbamato. En cambio, si que se observó cierta resistencia cruzada con respecto a espinosad (> 5 veces) en dos líneas de laboratorio de esta especie resistentes a los organofosforados malatión y naled (Hsu y Feng, 2006). 14 1. Introducción general ______________________________________________________________________________________________________________________________________ 1.2.4. Mecanismos de resistencia. Existen diferentes mecanismos mediante los cuales los insectos puedan desarrollar resistencia a los insecticidas. El conocimiento de los distintos mecanismos de resistencia es esencial para definir la estrategia más adecuada para su manejo. Sin embargo, hay que tener en cuenta que una determinada población puede presentar más de un mecanismo de resistencia, provocando mayores niveles de resistencia (Scott y Georghiou, 1986). Por ejemplo, frente al organofosforado malatión, además de haber sido descrita la resistencia debida a mutaciones puntuales en la molécula diana, también se ha observado que la resistencia puede estar provocada por un incremento en la actividad metabólica del insecto, ya sea mediada por las citocromo P450 (Welling et al., 1974; Morton y Holwerda, 1985; Maitra et al., 2000), por las glutatión Stransferasas (Wool et al., 1982; Taskin y Kence, 2004) o por las esterasas (Campbell et al., 1998b; Hemingway y Karunaratne, 1998). Los mecanismos de resistencia se clasifican en cuatro grandes grupos: resistencia metabólica, resistencia por modificación de la molécula diana, resistencia a la penetración y resistencia por comportamiento o etológica (Scott, 1991; Feyereisen, 1995; Hemingway et al., 2004; Feyereisen, 2005). 1.2.4.1. Resistencia metabólica. La resistencia metabólica es el tipo de resistencia más habitual en insectos y se produce cuando un insecticida, hidrolizado o transformado bioquímicamente, disminuye o pierde su capacidad de interaccionar con la molécula diana. Además, dentro de este tipo de resistencia se puede incluir el secuestro del insecticida por parte de una enzima. En una línea resistente, la detoxificación del insecticida se puede producir porque exista una enzima que metabolice más eficientemente dicho insecticida, o debido a que la cantidad de enzimas encargadas de metabolizar el xenobiótico sea mayor que la presente en una línea susceptible. La resistencia metabólica se debe principalmente a la implicación de tres grandes familias de enzimas: las glutatión Stransferasas (GSTs), las esterasas y las citocromo P450 (Hemingway, 2000; Ranson et al. 2002; Feyereisen, 2005). Además, es importante tener en cuenta que la resistencia metabólica no se produce exclusivamente por la acción de estas tres familias de enzimas, por ejemplo, recientemente se ha descrito un nuevo tipo de resistencia metabólica a insecticidas mediada por 15 1. Introducción general ______________________________________________________________________________________________________________________________________ una familia de glicoproteínas transportadoras de membrana que modulan el paso de sustancias a través de las membranas (Buss, 2008; Gahan et al., 2010; Labbé et al., 2011). Los sinergistas son compuestos que incrementan la efectividad de los insecticidas mediante el bloqueo de los sistemas de detoxificación que pudiesen inactivar al insecticida (Bernard y Philogene, 1993). El empleo de sinergistas ha sido útil para averiguar los mecanismos de resistencia en varias especies de artrópodos (Liu y Yue, 2000; Ahmad y Hollingworth, 2004; Espinosa et al., 2005). Los sinergistas más empleados, que inhiben a las tres principales familias de enzimas implicadas en detoxificación, son el DEM (maleato de dietilo) como inhibidor de las glutatión S-transferasas (Mulder y Ouwerkerk-Mahadevan, 1997); el DEF (SSS- tributilfosforotritioato) que inhibe a las esterasas (Oakeshott et al., 2005); y el PBO (butóxido de piperonilo) que inhibe la acción de las citocromo P450 (Feyereisen, 2005). 1.2.4.1.1. Glutatión S-transferasas. Las glutatión S-transferasas (GSTs) conforman una familia de enzimas implicadas en la detoxificación de multitud de xenobióticos. Estas enzimas catalizan la unión de una molécula de glutatión al insecticida, dando lugar a un compuesto hidrosoluble que se excreta más fácilmente. Además, las GSTs pueden utilizar el glutatión como cofactor para catalizar la deshidrocloración de algunos insecticidas (revisado por Enayati et al., 2005). En los dípteros, las GSTs se han relacionado con la resistencia a organofosforados, organoclorados, piretroides y DDT, tanto por sobreexpresión como por duplicación génica (Ranson et al., 2001; Ortelli et al., 2003; Enayati et al., 2005; Lumjuan et al., 2005). Hasta el momento se han descrito siete genes implicados en resistencia que codifican para GSTs, cinco de ellos se sobreexpresan en líneas resistentes y dos de ellos han sufrido una duplicación génica (Li et al., 2007). 1.2.4.1.2. Esterasas. Las carboxilesterasas o esterasas constituyen una gran familia de enzimas que son responsables el metabolismo lipídico y de la detoxificación de xenobióticos, entre otras funciones. En insectos se agrupan en clados filogenéticos ya que el origen de las diferentes esterasas se debe a la duplicación de un gen ancestral y a su posterior divergencia, organizándose finalmente en clusters (revisado por Oakeshott et al., 2005). Las esterasas están implicadas en resistencia a 16 1. Introducción general ______________________________________________________________________________________________________________________________________ organofosforados, carbamatos y piretroides debido a mutaciones puntuales, duplicaciones génicas, a regulación de la expresión, o a una combinación de los tres mecanismos (Li et al., 2007). La duplicación de determinados genes que codifican para esterasas produce un incremento en la degradación y el secuestro de los insecticidas, mecanismo que ha sido descrito en dípteros (Raymond et al., 1998; Karunaratne et al., 1998) y hemípteros (Field y Devonshire, 1998; Field et al., 1999; Vontas et al., 2000). Además, se han descrito casos de resistencia por sobreexpresión de las esterasas tanto en dípteros (Mouches et al., 1987; Hemingway y Karunamaratne, 1998; Qiao et al., 1998; Hemingway et al., 1998; Guerrero, 2000; Hawkes y Hemingway, 2002; Tao et al. 2006) como en pulgones (Field y Devonshire, 1998; Field et al., 1999; Pan et al., 2009). Finalmente, se han encontrado poblaciones resistentes a organofosforados de dípteros (Whyard et al., 1995; Campbell et al., 1998b; Claudianos et al., 1999) e himenópteros (Zhu et al., 1999), en los que una mutación puntual incrementa la capacidad hidrolítica de las esterasas disminuyendo al mismo tiempo su actividad carboxilesterasa. Una de las esterasas más importantes en resistencia es la aliesterasa, localizada dentro del cluster de las alfa-esterasas. Se han identificado dos mutaciones puntuales implicadas en procesos de resistencia en L. cuprina y M. domestica (Newcomb et al., 1997; Claudianos et al., 1999; Taskin y Kence, 2004; Hartley et al., 2006). Concretamente, la mutación Gly137Asp confiere resistencia a organofosforados en general, mientras que la mutación Trp251Ser/Leu es la responsable de resistencia específica a malatión (Campbell et al., 1998a, 1998b; Taskin y Kence, 2004). 1.2.4.1.3. Enzimas citocromo P450. El capítulo 3 de esta tesis se centra exclusivamente en el estudio de esta familia de enzimas de detoxificación, por lo que se ha realizado una descripción más exhaustiva de ellas en los siguientes apartados, basada en la revisión bibliográfica realizada por Feyereisen (2012). 1.2.4.1.3.1 Definición y nomenclatura. Las proteínas P450 son el producto de la traducción de los genes CYP que codifican para las enzimas monooxigenasas. Todas ellas presentan un pico de absorción a una longitud de onda de 450 nm, característica que da nombre a esta familia proteica (Omura y Sato, 1964). La principal 17 1. Introducción general ______________________________________________________________________________________________________________________________________ reacción que realizan estas enzimas es la transferencia de un átomo de oxígeno desde el oxígeno molecular hasta un determinado sustrato, tal y como generaliza la siguiente reacción: RH + O2 + NADPH + H+ → ROH + H2O + NADP+ Sin embargo, esta transferencia del átomo de oxígeno no es la única reacción catalítica que realizan estas enzimas. Las citocromo P450 poseen una gran variedad de actividades enzimáticas (oxidasa, reductasa, isomerasa…) y en conjunto pueden catalizar más de 60 reacciones químicas (Ortiz de Montellano, 1995; Mansuy, 1998; Guengerich, 2001). Las citocromo P450 conforman una de las familias proteicas más numerosas y están presentes en todos los organismos: bacterias, hongos, plantas y animales (Werck-Reichhart y Feyereisen, 2000). El genoma humano incluye 57 genes CYP, mientras que en insectos su número varía desde los 36 genes identificados en el piojo humano Pediculus humanus (Lee et al., 2010) hasta los 170 genes descritos en el mosquito Culex quinquefasciatus (Arensburgeret al., 2010). La gran diversidad de genes P450 es el resultado de sucesivas duplicaciones en el genoma seguidas de un proceso de divergencia evolutiva en las secuencias duplicadas. La primera nomenclatura de los genes y proteínas P450 de insectos se realizó cuando sólo se conocían 65 secuencias, y en ella se nombraban los genes en función del grado de homología que tenían entre sí las secuencias proteicas que codificaban para dichos genes (Nebert et al., 1987). Las reglas de nomenclatura han sido revisadas varias veces (Nebert et al., 1991; Nelson et al., 1993) hasta su formato actual (Fig. 1.7), donde cada gen, escrito en cursiva, se nombra con la raíz CYP seguida de un número arábigo que indica la familia, de una letra mayúscula que indica la subfamilia, y finalmente un número arábigo que identifica cada uno de los genes (Nelson et al., 1996). CYP: raíz de los genes P450 Familia: >40% identidad con otras proteínas CYP4 Subfamilia: >55% identidad con otras proteínas CYP4U CYP4U2 Identificador de Gen Figura 1.7. Nomenclatura de un gen CYP. Reglas consensuadas para la asignación de las diferentes categorías en las que se clasifican las proteínas P450 (Feyereisen, 2005). 18 1. Introducción general ______________________________________________________________________________________________________________________________________ Las proteínas codificadas por los genes CYP siguen la misma nomenclatura que los genes pero se escriben sin cursiva. De esta manera, la nomenclatura actualmente consensuada para los genes CYP no sigue la nomenclatura consensuada para otros genes (genes en minúscula y cursiva). Los genes P450 de D. melanogaster (Lindsley y Zimm, 1992) si mantienen la nomenclatura clásica, únicamente la primera letra del gen se escribe con mayúscula. Así por ejemplo, hablaríamos del gen CYP6A1 en M. domestica pero del gen Cyp6a2 en D. melanogaster (revisado por Feyereisen, 2011). Los diferentes alelos de un mismo gen se nombran con un sufijo “vn”, donde n es un número arábigo que identifica el alelo (Cohen et al., 1992). 1.2.4.1.3.2. Identificación y diversidad. En 1982 se clonó y secuenció el primer gen P450 (CYP2B1) en la rata común (FujiKuriyama et al., 1982). En insectos, los primeros genes identificados fueron CYP6A1, en M. domestica (Feyereisen et al., 1989), y Cyp4d1 (Gandhi et al., 1992) y Cyp6a2, en D. melanogaster (Waters et al., 1992). La amplificación por PCR empleando cebadores degenerados, basados en secuencias consenso de regiones conservadas, fue el primer paso para aislar cDNA de P450 en insectos y garrapatas (Feyereisen, 2005). Tomando como referencia la hélice I y la región conservada que flanquea a la cisteína del grupo hemo para el diseño de los cebadores degenerados, se pueden obtener productos de PCR de un tamaño aproximada de media kilobase, lo que representa la región central de los genes P450 y aproximadamente un tercio de la totalidad de su tamaño (Snyder et al., 1996). Siguiendo esta metodología se han descrito 17 nuevos genes CYP4 en Anopheles albimanus (Scott et al., 1994) y se ha ampliado la diversidad de genes P450 en varias especies de insectos (Feyereisen, 2005). El gran número de secuencias parciales obtenidas produjo dos problemas, en primer lugar, se infravaloró el número de genes encontrados ya que, debido al pequeño tamaño de las secuencias que se comparaban, genes diferentes fueron nombrados inicialmente como alelos de un mismo gen. En segundo lugar, el hecho de que las secuencias identificadas sólo representasen un tercio del total del gen, dificultó el cálculo del porcentaje de similitud, en el cual se basa la nomenclatura de los genes CYP. Gracias a la secuenciación del genoma completo de D. melanogaster (Adams et al., 2000) se pudo concretar el número de genes P450 para esta especie. Se identificaron un total de 90 genes de los cuales 83 codificaban proteínas potencialmente funcionales y los otros siete eran secuencias parciales o pseudogenes (Tijet et al., 2001). En 2002 se secuenció el genoma de A. gambiae (Holt et al., 2002) y se identificaron 111 genes CYP, entre los que se describieron cinco pseudogenes (Ranson et al., 2002). Existe una necesidad de estandarizar las reglas para la 19 1. Introducción general ______________________________________________________________________________________________________________________________________ identificación de los genes P450 (Claudianos et al., 2006; Strode et al., 2006; Lee et al., 2010) ya que normalmente se producen errores en la descripción de los genes a partir del banco de secuencias de un genoma debido, por ejemplo, a la fusión de la secuencia parcial de dos genes próximos (genes con secuencias similares) para dar lugar a un gen híbrido incorrecto. Es muy importante comparar la estructura intrón/exón de genes similares para facilitar su identificación. Actualmente, mediante nuevas técnicas de secuenciación, se ha estudiado la diversidad de genes P450 de varias especies sin necesitad de secuenciar el genoma completo. Así, en 2009 Zagrobelny et al. obtuvieron gran cantidad de transcritos P450 para larvas de Zygaena filipendulae y Pauchet et al. en 2010 identificaron 36 nuevos genes CYP en Manduca sexta. Aunque cada vez las técnicas de secuenciación y los ensamblajes son más precisos, el número de genes CYP identificados en cada especie puede variar con las revisiones de los ensamblajes de los bancos de secuencias, por lo que es necesario confirmar la secuencia completa de cada uno de los genes mediante su amplificación por PCR. El número de genes CYP identificados en insectos asciende a 1675, repartidos en 59 familias y 338 subfamilias (Nelson, 2009). El conjunto de todos los genes CYP de un determinado taxón, en analogía con los términos genoma, proteoma o transcriptoma, recibe el nombre de CYPoma (Lamb et al., 2002). La evolución de los CYPomas se basa en la duplicación génica seguida de la correspondiente divergencia o de la deleción del gen a lo largo del tiempo evolutivo (Feyereisen, 2011). Debido al elevado número de genes existentes dentro de un CYPoma, éstos se dividen en clados filogenéticos o clanes, formados por varias familias CYP. Concretamente en insectos, los genes se distribuyen en cuatro clanes: CYP-2, CYP-3, CYP-4 y mitocondrial (Feyereisen, 2006). El clan CYP-2 incluye genes que intervienen en funciones fisiológicas básicas para los insectos. El clan CYP-3 es el que engloba el mayor número de genes CYP de insectos, repartidos en unas 30 familias. Las principales familias del clan CYP-3 son CYP6, CYP9 y CYP28. Los genes de este clan tienen como característica común que intervienen en la respuesta a las condiciones ambientales externas (Berenbaum, 2002). Los genes incluidos en el clan CYP-4 son inducibles por xenobióticos y son el grupo de enzimas P450 menos estudiadas en insectos. Finalmente, el clan mitocondrial incluye P450 de insectos, de vertebrados y otros metazooos (Feyereisen, 2011), y de las familias englobadas en este clan, la CYP12 es la más importante. En cuanto a la diversidad observada dentro de la familia Tephritidae, se han identificado genes CYP en los géneros Ceratitis y Bactrocera. Así, han obtenido secuencias parciales de genes CYP en B. oleae (Tsoumani et al., 2011) y en B. papayae (ref. GB: AF532768). En B. dorsalis, recientemente, han clonado y caracterizado los genes CYP6A41 y CYP6EK1 (Huang et al., 2012) y se ha publicado el resultado de la secuenciación de su transcriptoma, donde se ha estimado que existen 90 genes CYP (Hsu et al., 2012a). En el único estudio realizado en C. capitata se 20 1. Introducción general ______________________________________________________________________________________________________________________________________ identificaron 14 genes CYP, entre los que se obtuvo la secuencia completa del gen CYP6A10 y la secuencia parcial del extremo 3’ de 5 genes de la familia CYP4, 8 de la familia CYP6 y un gen sin clasificar (Danielson et al., 1999). 1.2.4.1.3.3. Estructura y función. Las citocromo P450 presentan en su estructura génica intrones de pequeño tamaño con la organización típica GT/AG. La variación en la secuencia aminoacídica es de una magnitud tal que el único residuo absolutamente conservado en todas la proteínas P450 de distintos organismos es la cisteína del grupo hemo (Rupasinghe et al., 2006). Estas grandes diferencias observadas en la secuencia aminoacídica contrastan con el relativamente alto grado de conservación en la estructura terciaria presente en las proteínas P450 cristalizadas hasta el momento. En proteínas P450 de insectos se han descrito varias regiones conservadas que coinciden con los principales motivos que participan en la estabilización global de la estructura terciaria de la proteína: el motivo ExLR, localizado en la hélice K y el motivo PxxFxPE/DRF. Las P450 de los insectos se localizan tanto en las membranas de los microsomas como en las mitocondrias, pero su localización subcelular no está asociada con una determinada función catalítica. En general, las enzimas citocromo P450 de insectos son conocidas por su capacidad de metabolizar insecticidas, sin embargo, algunas de estas enzimas poseen además funciones fisiológicas de vital importancia para los insectos. Por ejemplo, algunas de ellas están implicadas en el catabolismo de ácidos grasos, mientras que otras participan en la biosíntesis de la hormona juvenil, de la ecdisona o de diferentes feromonas (Feyereisen, 2005). La regulación de la transcripción de los genes P450 puede producirse tanto por la influencia de un metabolito endógeno como de un xenobiótico. Las sustancias exógenas, como ocurre por ejemplo con algunas sustancias producidas por las plantas, pueden provocar la inducción de las enzimas P450. Algunos ejemplos de inductores de origen natural aplicados a los insectos son alcaloides, como la nicotina (Snyder et al., 1995) o la cafeína (Bhaskara et al., 2006) y terpenoides como el mentol, el α-pineno, el aceite de menta (Ranasinghe et al., 1997) o el limoneno (Giraudo et al., 2010). En cuanto a los xenobióticos de origen artificial, el inductor más empleado es el fenobarbital, capaz de activar la sobreexpresión de una treintena de genes CYP. El efecto del fenobarbital sobre la expresión de diferentes genes CYP en insectos ha sido evaluado tanto 21 1. Introducción general ______________________________________________________________________________________________________________________________________ mediante Northern blot (Snyder et al., 1995; Dunkov et al., 1997; Danielson et al., 1997, 1998; Li et al., 2000, 2002) como por microarrays (Le Goff et al., 2006, Willoughby et al., 2006; Sun et al., 2006) o mediante PCR cuantitativa en tiempo real (Le Goff et al., 2006, Sun et al., 2006; Zhou et al., 2010; Chung et al., 2011), variando su efecto inductor según sea el sexo del individuo y el gen analizado (Le Goff et al., 2006). Recientemente se ha demostrado que la aplicación de diferentes sustancias químicas sobre D. melanogaster provoca la inducción de aproximadamente una tercera parte de los genes de su CYPoma (Giraudo et al., 2010). Esto pone de manifiesto la gran importancia a nivel bioquímico, genético y fisiológico que poseen las P450 en la relación entre los insectos y el entorno en el que habitan (Feyereisen, 1999). La gran diversidad de las enzimas P450 y la inducción de su expresión en respuesta a diferentes xenobióticos contribuyen a incrementar la plasticidad adaptativa que poseen los insectos, por lo que las P450 pueden influir en el control de los insectos plaga. Está demostrado que un insecto tendrá diferentes respuestas ante un insecticida según el tipo de planta del que se esté alimentando (Ahmad, 1986), respuestas que responden a la inducción, entre otras, de enzimas P450. Por ejemplo, varios productos naturales suministrados a larvas de Helicoverpa zea produjeron la inducción de los genes CYP6B8 y CYP321A1, provocando una disminución de su susceptibilidad a diazinón y carbaril (Zeng et al., 2007; Wen et al., 2009). Además, se ha descrito que incluso los disolventes presentes en los herbicidas o insecticidas podrían actuar como inductores de genes CYP (Kao et al., 1995; Miota et al., 2000). 1.2.4.1.3.4. Resistencia a insecticidas mediada por P450. El primer estudio que demostró la inducción de las P450 en insectos en respuesta a la exposición a insecticidas se realizó en el chinche hematófago Triatoma infestans, en el que el DDT aplicado inducía la enzima NAD-quinasa (Agosin y Dinamarca, 1963), ahora incluida en la familia de enzimas P450. A principios de los años setenta se describió por primera vez la posible implicación de las P450 en procesos de resistencia a insecticidas (Perry y Buckner, 1970). Durante los siguientes años se realizó la caracterización y comparación de los espectros de absorción de los insectos susceptibles y resistentes para estudiar la resistencia mediada por P450 (Perry et al., 1971; Philpot y Hodgson, 1972; Tate et al., 1973). Posteriormente, se emplearon sinergistas como el PBO (butóxido de piperonilo) para relacionar las enzimas P450 con fenómenos de resistencia (Feyereisen, 2005). En los últimos años, mediante el empleo de nuevos métodos moleculares se ha incrementado el conocimiento sobre este grupo de enzimas y su implicación en la resistencia a insecticidas. 22 1. Introducción general ______________________________________________________________________________________________________________________________________ En insectos, el incremento en los niveles de expresión de uno o varios genes CYP ha sido descrito en líneas resistentes de varias especies. El orden Diptera es el que posee más casos de variación en la expresión de genes CYP en líneas resistentes, concretamente se ha detectado la sobreexpresión de más de cuarenta genes en las nueve especies del orden estudiadas (Feyereisen, 2005). El primer caso identificado de sobreexpresión de un gen CYP en insectos fue en la línea Rutgers de M. domestica, en la que se describió que el gen CYP6A1 era inducible por fenobarbital y se sobreexpresaba con respecto a una línea susceptible (Feyereisen et al., 1989). Sin embargo, no se confirmó su relación con la resistencia al organofosforado diazinón hasta que se expresó el gen en Escherichia coli y se observaron sus patrones redox (Sabourault et al., 2001). Posteriormente se han detectado otros genes CYP implicados en resistencia a insecticidas, como por ejemplo el gen CYP6D1, que está relacionado con la resistencia a piretroides en M. domestica (Liu y Scott, 1996), o el gen Cyp6a2, sobreexpresado en la línea 91-R de D. melanogaster resistente a DDT y malatión (Waters et al., 1992). Además, se ha descrito la sobreexpresión de genes CYP frente a propoxur (Daborn et al., 2002) y lufenurón (Daborn et al., 2002; Bogwitz et al., 2005) en D. melanogaster, y frente a imidacloprid en D. melanogaster (Daborn et al., 2002) y D. simulans (Le Goff et al., 2003). Por último, en el suborden Nematocera, se ha detectado sobreexpresión de determinados genes CYP en poblaciones resistentes a DDT y piretroides en seis especies de mosquitos (Feyereisen, 2012). Por otra parte, mediante el empleo de microarrays se ha analizado la expresión de múltiples genes de las diferentes familias génicas implicadas habitualmente en la detoxificación de insecticidas. Además, se ha identificado el nexo entre la resistencia a insecticidas y el incremento en los niveles de RNAm o de determinadas proteínas P450 (Bogwitz et al., 2005; Feyereisen, 2005; Wee et al., 2008; Hardstone et al., 2010). El uso de sistemas de expresión heteróloga, sobre todo en E. coli y nucleopoliedrovirus, se ha utilizado para establecer una relación entre la sobreexpresión de un determinado gen P450 y su contribución a la resistencia (Yang et al., 2008; Karunker et al., 2009). El reciente empleo de RNA de interferencia (RNAi) para silenciar un gen CYP concreto, permite mostrar claramente la función detoxificante de un determinado gen (Yang et al., 2007; Bautista et al., 2009; Zhu et al., 2010), pero es una técnica que no muestra la misma eficacia de interferencia en todas las especies de insectos. En la familia Tephritidae existen muy pocos estudios en los que se ponga de manifiesto la implicación de las P450 en la adquisición de resistencia. Únicamente se ha detectado, en una línea multiresistente de B. dorsalis, la implicación indirecta de las P450 en fenómenos de resistencia a insecticidas mediante el empleo de sinergistas (Hsu et al., 2004a), pero no se ha relacionado ningún gen CYP concreto con la resistencia. 23 1. Introducción general ______________________________________________________________________________________________________________________________________ 1.2.4.2. Resistencia por modificación de la molécula diana. Otro de los principales mecanismos de resistencia a insecticidas se debe a la modificación de la molécula diana o insensibilización del punto de acción del insecticida. La resistencia por modificación de la molécula diana conlleva modificaciones genéticas en el DNA que provocan cambios en la secuencia aminoacídica de la molécula que, a su vez, originan una variación estructural y/o funcional en la proteína, disminuyendo así la acción tóxica del insecticida. Las moléculas diana más estudiadas son el canal de sodio, el receptor del ácido gamma-aminobutírico, la acetilcolinesterasa y los receptores nicotínicos de la acetilcolina (ffrench-Constant, 1999; ffrenchConstant et al., 1998; 1999, 2004; Watson et al., 2010). 1.2.4.2.1. La acetilcolinesterasa. La acetilcolinesterasa (AChE, EC 3.1.1.7) es la principal enzima del sistema colinérgico y su función es regular los niveles de acetilcolina durante la transmisión del impulso nervioso (Fournier, 2005). La AChE hidroliza la acetilcolina en acetato y colina durante la transducción del impulso nervioso en la sinapsis colinérgica (Kozaki et al., 2002). En insectos, según los estudios realizados en D. melanogaster (Toutant, 1989; Adams et al., 2000) se asumía que sólo existía un gen ace que codificaba para la enzima acetilcolinesterasa, pero trabajos posteriores demostraron la existencia de dos genes ace en insectos no dípteros (Villatte y Bachmann, 2002) y en mosquitos (Bourguet et al., 1996a; Weill et al., 2002). Actualmente, se considera que existe un único gen ace en los dípteros del suborden Brachycera (moscas propiamente dichas), mientras que en el resto de dípteros y en la mayoría de los demás órdenes de insectos existen dos genes (Huchard et al., 2006). Se denominan ace1 a los genes ortólogos al gen Agace1 descrito en A. gambiae, y ace2 a los genes ortólogos al gen Dmace2 encontrado en D. melanogaster (Weill et al., 2002). Estudios de interferencia mediante RNA de doble cadena (RNAi) llevados a cabo en insectos que poseen ambos genes ace sugieren que ace1 es el gen responsable de la mayor parte de la actividad acetilcolinesterasa; aunque ambos genes pueden tener un papel esencial en la supervivencia y en la interacción con insecticidas (Revuelta et al., 2009; 2011). La AChE es la molécula diana de los organofosforados y los carbamatos, ya que estos compuestos tienen la capacidad de unirse al centro activo de la enzima y actúan como inhibidores funcionalmente irreversibles por fosforilación o carbamilación del grupo hidroxilo de una serina del centro activo, esencial para su mecanismo catalítico. Esta inhibición provoca la acumulación de 24 1. Introducción general ______________________________________________________________________________________________________________________________________ acetilcolina en el espacio sináptico, lo que produce la muerte del insecto (Aldridge, 1950). Desde la primera descripción de una disminución en la susceptibilidad de la acetilcolinesterasa frente a insecticidas (Smissaert, 1961) se han descrito multitud de casos de resistencia a organofosforados y carbamatos en los que las mutaciones puntuales en la AChE son el mecanismo de resistencia más habitual (Mutero et al., 1994; Walsh et al., 2001; Oakeshott et al., 2005). Concretamente en los dípteros, se han relacionado con resistencia varias mutaciones puntuales que alteran la actividad AChE en D. melanogaster (Fournier et al., 1993; Fournier y Mutero, 1994; Mutero et al., 1994; Villatte et al., 2000; Menozzi et al., 2004); en M. domestica (Feyereisen, 1995; Walsh et al., 2001; Kozaki et al., 2001); en B. oleae (Vontas et al., 2002), en B. dorsalis (Hsu et al., 2006), en C. capitata (Magaña et al., 2008) y en los mosquitos A. gambiae y C. pipiens (Weill et al., 2003). Estas mutaciones puntuales se han localizado principalmente en los exones E3, E4, E5, E6 y E7 del gen ace2, siguiendo la numeración de exones de este gen de D. melanogaster. En aquellas especies que presentan los dos genes ace, las mutaciones se han descrito fundamentalmente en el gen ace1 (Fournier, 2005; Kono y Tomita, 2006). 1.2.4.3. Otros mecanismos de resistencia. La resistencia a la penetración o la resistencia por comportamiento son dos mecanismos de resistencia a insecticidas poco conocidos debido a su dificultad de estudio. El primero de ellos se produce cuando la cutícula externa del insecto actúa de barrera frente al insecticida por haber desarrollado la capacidad de evitar su incorporación al cuerpo o disminuir la velocidad a la que ocurre su entrada. Este mecanismo de protección es no selectivo, por lo que los insectos que presenten este tipo de resistencia son menos susceptibles a la penetración de diferentes insecticidas. Por si sólo este mecanismo no aporta elevados niveles de resistencia pero la combinación con un segundo mecanismo puede provocar un incremento multiplicativo de la resistencia, como ocurre en Blatella germanica o en M. domestica (Scott y Georghiou, 1986; Wen y Scott, 1997; Wu et al., 1998). La resistencia por comportamiento se produce en aquellos insectos que son capaces de evitar la exposición al insecticida. Se ha descrito este mecanismo de resistencia frente a varios tipos de insecticidas como organofosforados, carbamatos o piretroides, en los que los insectos evitan ingerir alimentos que contengan el insecticida, o huyen de las zonas en las que ha habido fumigación (Sparks et al., 1989). Existen estudios en los que la resistencia se debe a la repulsión o irritación que provoca el insecticida, como ocurre en la mosca Haematobia irritans frente a 25 1. Introducción general ______________________________________________________________________________________________________________________________________ piretroides (Quisenberry et al., 1984). Este mecanismo de resistencia suele producirse en combinación con otros tipos de mecanismos, por lo que su identificación puede ser compleja. 1.2.5. Resistencia en C. capitata y otras especies de la familia Tephritidae. Hasta la fecha, dentro de la familia Tephritidae, se ha detectado resistencia a insecticidas en tres especies del género Bactrocera: B. cucurbitae, B. dorsalis y B. oleae, (Vontas et al., 2011); y en C. capitata (Magaña et al., 2007). En 1971 Krimbas y Tsakas describieron en B. oleae el primer indicio de resistencia frente a organofosforados en tefrítidos. Posteriormente varios autores han relacionado esta resistencia con diversas alteraciones en la acetilcolinesterasa, tanto en B. oleae (Vontas et al., 2001; 2002; Kakani et al., 2008) como en B. dorsalis (Hsu et al., 2006). En cuanto a la resistencia metabólica, se han descrito varios casos en poblaciones de B. oleae resistentes a organofosforados (Tsakas y Krimbas, 1970, Stasinakis et al., 2001). En B. dorsalis la implicación de las esterasas en la resistencia a organofosforados se ha confirmado mediante estudios con sinergistas (Hsu et al., 2004a). En la literatura científica existen muy pocos casos de resistencia frente a insecticidas piretroides en la familia Tephritidae. En B. dorsalis se han descrito dos casos de resistencia a lambda-cialotrina después de seleccionar en el laboratorio poblaciones recogidas en Taiwan (Hsu y Feng, 2002; Hsu et al., 2004b). Por el contrario, sí se detectó una elevada resistencia al piretroide alfa-cipermetrina en poblaciones de campo de B. oleae en Grecia (Margaritopoulos et al., 2008). En cuanto a los estudios de resistencia a espinosad, los datos son muy escasos. En la familia Tephritidae únicamente se ha detectado resistencia a espinosad en el género Bactrocera y en todos los casos descritos los niveles de resistencia son bajos. Así, se ha observado cierta resistencia en poblaciones de campo de B. oleae en California (EE.UU.) y en Grecia (Kakani et al., 2010). Además, recientemente se ha observado una baja susceptibilidad a espinosad en poblaciones de campo de B. cucurbitae en Hawai y en Taiwan (Hsu et al., 2012b). En el resto del orden Diptera sólo se ha descrito resistencia a espinosad en líneas de laboratorio de D. melanogaster, M. domestica y en una población de campo de Liriomyza trifolii (Sparks et al., 2012; Markussen y Kristensen, 2012). En 1982 Viñuela y Arroyo realizaron bioensayos con poblaciones de campo de C. capitata recogidas en diferentes regiones españolas y observaron que la mosca mediterránea de la fruta no 26 1. Introducción general ______________________________________________________________________________________________________________________________________ presentaba resistencia a tres organofosforados ampliamente usados para su control: malatión, diazinón y triclorfón. A finales de los años noventa se realizaron nuevos estudios en los que no se detectó ningún fallo en el control químico de la plaga (Viñuela, 1998). Durante las últimas décadas, el incremento de la frecuencia de los tratamientos de malatión como insecticida frente a C. capitata en España ha provocado la aparición de poblaciones resistentes (Magaña et al., 2007). Este hallazgo, supone el primer caso de resistencia a insecticidas en poblaciones de campo de la mosca mediterránea de la fruta. Esta resistencia ha sido relacionada con dos mecanismos de resistencia. El primero se debe a una mutación puntual en la acetilcolinesterasa, molécula diana del malatión. Concretamente la mutación AChE G328A, que ya había sido relacionada con resistencia a insecticidas en D. melanogaster (Menozzi et al., 2004) y M. domestica (Walsh et al., 2001), provoca una modificación en uno de los residuos próximo a la tríada catalítica, reduciendo la afinidad de la acetilcolinesterasa por el organofosforado malatión (Magaña et al., 2008). El segundo mecanismo de resistencia descrito en C. capitata está relacionado con la resistencia metabólica mediada por carboxilesterasas (Magaña et al., 2008). 27 1. Introducción general ______________________________________________________________________________________________________________________________________ 1.3. Objetivos de la tesis. La existencia de poblaciones españolas de Ceratitis capitata resistentes a malatión aconseja el desarrollo de métodos de detección que faciliten su seguimiento y que puedan ser utilizados para la elección de la estrategia más adecuada para el manejo de esta plaga. Para ello, es necesario profundizar en el conocimiento de los mecanismos asociados a la resistencia a insecticidas. Durante el desarrollo de esta Tesis el malatión dejó de ser utilizado en la Unión Europea contra la mosca mediterránea de la fruta, siendo sustituido por otros insecticidas como el espinosad y la lambda-cialotrina. En este nuevo contexto se planteó la necesidad de estudiar la posible existencia de resistencia cruzada a estos insecticidas, analizar la susceptibilidad de poblaciones de campo a espinosad y sentar las bases para el estudio de posibles nuevos mecanismos de resistencia Los objetivos de esta Tesis son: 1) Evaluar la susceptibilidad a malatión y espinosad en poblaciones españolas de C. capitata y obtener líneas resistentes mediante selección en el laboratorio. 2) Sentar las bases para el estudio de mecanismos de resistencia en C. capitata mediados por enzimas P450 a través de la identificación de genes CYP y el estudio de su inducción por xenobióticos. 3) Desarrollar un método molecular para determinar la presencia de alelos de resistencia a malatión en C. capitata y determinar la dispersión geográfica de dichos alelos en poblaciones de campo. 4) Caracterizar un nuevo mecanismo de resistencia a malatión identificado en una de las líneas de laboratorio altamente resistente a este insecticida. 29 Capítulo 2 SELECCIÓN DE RESISTENCIA EN LABORATORIO Y SUSCEPTIBILIDAD A INSECTICIDAS EN Ceratitis capitata Parte de los resultados mostrados en este capítulo forman parte de la siguiente publicación: Couso-Ferrer, F; Arouri, R; Beroiz, B; Perera, N; Cervera, A; Navarro-Llopis, V; Castañera, P; Hernández-Crespo, P; Ortego, F. (2011). Cross-resistance to insecticides in a malathion-resistant strain of Ceratitis capitata (Diptera: Tephritidae). J. Econ. Entomol.; 104(4): 1349-1356. 31 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ 2.1. Introducción. La mosca mediterránea de la fruta Ceratitis capitata (Wiedemann, 1824) está considerada como una de las plagas más importantes para los cultivos frutícolas a nivel mundial. En dos estudios realizados en España en los años ochenta y noventa se analizó la susceptibilidad de esta especie frente a varios insecticidas organofosforados y no se detectó ningún fallo en su control químico (Viñuela y Arroyo, 1982; Viñuela, 1998). En España, y más concretamente en la Comunidad Valenciana, se incrementó el uso del organofosforado malatión para combatir a C. capitata durante las dos últimas décadas debido a las grandes pérdidas económicas que ocasionaba esta plaga en los cultivos de cítricos. Este incremento, tanto en la concentración de malatión como en la frecuencia de los tratamientos, provocó la aparición de poblaciones de campo resistentes (Magaña et al., 2007), lo que supuso la primera descripción de un fenómeno de resistencia a insecticidas en poblaciones de campo de esta especie. Hasta 2008 el insecticida más empleado en los cultivos de cítricos frente a C. capitata fue el organofosforado malatión. Sin embargo, la Directiva Europea 91/414/ECC prohibió su uso frente a la mosca mediterránea de la fruta a partir del año 2009. Actualmente, los insecticidas más utilizados para el control de C. capitata en cítricos, en sustitución del malatión, están basados en dos sustancias activas: la lambda-cialotrina y el espinosad. Además, aunque en menor media, se 33 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ emplea el lufenurón, en el denominado control por quimioesterilización (Navarro-Llopis et al., 2010). El estudio de la línea de C. capitata resistente a malatión (línea W), que provenía de una población de campo recogida en 2004 en Castelló, permitió la identificación de dos mecanismos asociados a esta resistencia: una mutación puntual (G328A) en la acetilcolinesterasa (AChE), molécula diana de los organofosforados, y un mecanismo de resistencia metabólica mediado por carboxilesterasas (Magaña et al., 2008). No obstante, en los estudios de Magaña y colaboradores (2007 y 2008), no se determinó la importancia relativa de cada uno de los dos mecanismos sobre la resistencia a malatión observada en las poblaciones españolas. Los dos mecanismos de resistencia a malatión descritos en C. capitata podrían conferir resistencia cruzada a otros insecticidas. Por ello, es importante conocer si la efectividad de los insecticidas utilizados actualmente puede verse comprometida por la resistencia existente en campo. Por un lado, la resistencia mediada por una mutación en la AChE puede influir sobre la susceptibilidad a otros organofosforados y carbamatos, ya que comparten la misma enzima como diana (Oakeshott et al., 2005) y por otro lado, un mecanismo de resistencia metabólica mediada por carboxilesterasas podría conferir resistencia cruzada a distintos insecticidas, independientemente de la diana a la que van dirigidos, ya que actuaría favoreciendo la hidrólisis o el secuestro de la materia activa (Hemingway, 2000; Oakeshott et al., 2005). En los estudios de resistencia cruzada realizados por Magaña y colaboradores (2007) en la línea W, resistente a malatión, sólo se emplearon dos insecticidas: frente al organofosforado fentión se observó resistencia cruzada, mientras que no existieron diferencias significativas en la susceptibilidad a espinosad. Con el objetivo de estudiar, por una parte, la distribución en España de las poblaciones de C. capitata resistentes a malatión y, por otra, la posible existencia de diferentes mecanismos de resistencia frente a este organofosforado, se analizó la susceptibilidad a malatión y se emplearon sinergistas en poblaciones españolas de distintas áreas geográficas. Además, en la mayoría de estas poblaciones, se evaluó la susceptibilidad a espinosad. Al mismo tiempo, se seleccionaron dos líneas de laboratorio resistentes a malatión y a espinosad, con la finalidad de estudiar los mecanismos involucrados en la resistencia a estos insecticidas. Finalmente, en la línea seleccionada con malatión, se analizó la posible existencia de resistencia cruzada frente a diferentes tipos de insecticidas. 34 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ 2.2. Material y métodos. 2.2.1. Ceratitis capitata en el laboratorio. 2.2.1.1. Condiciones generales de cría. En una jaula de cría se colocaron pupas de C. capitata, en una placa Petri, de las que posteriormente emergerían los adultos. Los adultos de cada generación se mantuvieron en las jaulas durante un periodo de entre tres y cuatro semanas. En el interior de cada jaula se colocó un bebedero y un comedero (Fig. 2.2.1.a). El bebedero consistió en una botella de plástico de 250 ml llena de agua con una ranura en su tapa. A través de la ranura se colocó una tira de material absorbente que estaba siempre en contacto con el agua del recipiente y que sobresalía por la ranura de la tapa. El comedero era una placa Petri que contenía una mezcla de azúcar molido con extracto de levadura de cerveza (Laboratorios Conda S.A., Torrejón de Ardoz, Madrid, España, Cat. 1702) en una proporción 4:1 respectivamente. Se emplearon dos tipos de jaulas de cría de metacrilato transparente, las jaulas pequeñas, que eran unos cubos de 20 cm de arista, y las jaulas grandes con unas dimensiones de 30x30x50 cm. Una de las caras de las jaulas era una malla de nailon a través de la cual las hembras, adaptadas a la puesta en malla, introducían su ovipositor y depositaban los huevos, que caían en un recipiente con agua situado debajo de la 35 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ jaula (Fig. 2.2.1.b). Las jaulas de cría se mantuvieron en un insectario con un ciclo de luz/oscuridad de 16/8h y a una temperatura de 25±2ºC. Los primeros huevos, resultado de las cópulas producidas entre el tercer y quinto día después de la emergencia de los adultos, se desecharon. Las siguientes puestas de huevos se recogieron sucesivamente cada dos días con una jeringa y se depositaron en una dieta artificial para larvas a base de agua, azúcar, levadura de cerveza, salvado de trigo, agentes antimicrobianos y antifúngicos (Ver anexo I). Aproximadamente se sembraron 0,2 ml de huevos en 250 g de dieta que se colocaron en una caja de plástico de 12x5x2 cm cubierta con una lámina de papel de aluminio a modo de tapa (Fig. 2.2.2.a). Esta caja de plástico se colocó, apoyada en una placa Petri, dentro de otra caja mayor de plástico transparente de 25x15x4 cm que tenía en su tapa dos orificios tapados con papel de filtro para permitir la ventilación. Los huevos y las larvas se mantuvieron en cámaras climatizadas Sanyo MLR-350 (Sanyo Electric Biomedical Co., Japón) a una temperatura de 25±1ºC (o de 20±1ºC, para ralentizar el desarrollo) y con un ciclo de luz/oscuridad de 16/8h (Fig. 2.2.2.b). a b Figura 2.2.1. Jaula de cría para adultos. (a) Esquema de la jaula de cría donde se colocaron las pupas de C. capitata. Se observa el recipiente con agua y la placa Petri con la dieta para los adultos. (b) Fotografía de la cara de la jaula recubierta por una malla de nailon, a través de la cual las hembras depositaron los huevos, y del recipiente con agua donde se recogía la puesta. 36 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ a b c Figura 2.2.2. Cría de larvas. (a) Fotografía del sistema de cajas empleado para el desarrollo de C. capitata desde larva a pupa. (b) Cámara climatizada donde se mantuvieron las puestas de huevos hasta la aparición de las larvas. (c) Último estadio larvario en el que las larvas saltan de la dieta antes de pupar. El desarrollo larvario de C. capitata transcurre por tres estadios. Al finalizar el último estadio, las larvas realizan un salto desde el sustrato de alimentación hacia la caja externa, donde pupan (Fig. 2.2.2.c). Las pupas se retiraron cada dos días y se colocaron en cajas transparentes de plástico de 2,5 cm de alto por 9 cm de diámetro con un respiradero de 2 cm de diámetro en la tapa cubierto con papel de filtro. Dos o tres días antes de la emergencia de los adultos, las pupas se colocaron en una placa Petri en el interior de una jaula de cría en el insectario, donde emergían los adultos. 2.2.1.2. Mantenimiento de líneas de C. capitata bajo presión de selección. La selección mediante insecticidas se realizó sobre adultos de entre tres y cinco días de edad en el interior de las jaulas grandes en el insectario. Antes de comenzar el tratamiento con insecticida, se extrajeron de la jaula las pupas no emergidas para evitar la emergencia de nuevos individuos con posterioridad al inicio del tratamiento. El tratamiento de selección consistió en la sustitución del comedero con dieta normal por otra placa Petri que contenía nueve gramos de dieta mezclada con 1 ml de insecticida a la concentración deseada. Generalmente el tratamiento duró 48 h, pero en algunas ocasiones, si la mortalidad observada era baja, se renovaba el insecticida y se 37 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ prolongaba el tratamiento durante 24 o 48 h más. Finalizado el tiempo de exposición, se extrajo el insecticida de la jaula y se sustituyó por el comedero con dieta sin insecticida. La recogida de los huevos se inició 48 h después de haber finalizado el tratamiento insecticida para recoger únicamente la puesta de aquellos individuos más tolerantes al insecticida. El mantenimiento de las líneas después del tratamiento de selección siguió el procedimiento descrito en el apartado anterior. Para la selección con malatión se utilizó Malafin® 50 CE (Agrodan S.A.) y para la selección con espinosad se suministró Spintor-Cebo® (Dow AgroSciences), que contiene como componente insecticida una mezcla de espinosinas A y D. 2.2.2. Líneas de laboratorio. A continuación se describen las líneas de laboratorio de la mosca mediterránea de la fruta con las que se han realizado los experimentos que se describen en esta tesis. Para la nomenclatura de las líneas seleccionadas se empleó un término que define el origen de la población seguido de un valor numérico que muestra la concentración de insecticida aplicada durante la selección, y de una letra que indica el insecticida aplicado. En la figura 2.2.3 se puede observar, en forma de diagrama de flujo temporal, el origen y el desarrollo de las líneas de laboratorio estudiadas. · Línea C: Línea utilizada como control susceptible en los diferentes experimentos. Proviene de una población recogida con anterioridad al incremento intensivo del uso del malatión. Concretamente se recogió en los campos adyacentes al Instituto Valenciano de Investigaciones Agrarias (IVIA) de Moncada (Valencia, C. Valenciana) en 2001 (Magaña et al., 2007) y ha sido mantenida en el CIB desde 2004 sin haber recibido ningún tipo de tratamiento insecticida. · Línea W-4Km: Resulta de la selección de una población resistente a malatión recogida en el municipio de Castelló (C. Valenciana) en octubre de 2004 (Magaña et al., 2007). Las hembras de la línea W (primeras trece generaciones de selección) depositaban los huevos en manzanas, pero posteriormente fueron adaptadas a realizar la puesta a través de una malla de nailon. Esta línea fue mantenida bajo una presión creciente de selección frente a malatión según lo descrito en el apartado 2.2.1.2. Esta línea adquirió la denominación de W-4Km cuando en la generación F30 se fijó la presión de selección con malatión en un tratamiento de 4.000 ppm durante 48 horas. 38 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata Línea W-10Km Selección con malatión (10.000 ppm) a partir de la F40 Selección con malatión (4.000 a 10-000 ppm) hasta la generación F40 Línea Xàbia-W-100s Selección con espinosad (100 ppm) a partir de la F36 Selección con esiponad (2,5 a 100 ppm) hasta la generación F36 Línea Xàbia-W Cruce con F38 de W-4Km 39 (Magaña et al., 2007, 2008) Línea C desde 2004 en el CIB No tratada (Magaña et al., 2007) Selección con espinosad (1 a 2 ppm) durante 10 generaciones + Línea IVIA (2001) proviene de campos valencianos no tratados Población Xàbia (2007) Figura 2.2.3. Líneas de laboratorio. Origen y evolución de las líneas empleadas en esta tesis. Línea W-4Km Selección con malatión (4.000 ppm) a partir de la F31 Adaptación a la puesta en malla Selección con malatión (2.000 a 4.000 ppm) hasta la F31 (Magaña et al., 2008) Línea W Estudio de los mecanismos de resistencia Selección con malatión (1.000 a 3.000 ppm) durante 13 generaciones (Magaña et al., 2007) Población Castelló (2004) resistente a malatión _____________________________________________________________________________________________________________________________________________________________________________________________________________________ 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ · Línea W-10Km: Esta línea se obtuvo a partir de la línea W-4Km mediante una presión creciente de selección con malatión (ver apartado 2.2.1.2). A partir de la generación F40 se fijó la presión de selección en un tratamiento de 10.000 ppm de malatión durante 48 horas. · Línea Xàbia-W-100s: Procede de los adultos de C. capitata que emergieron a partir de la fruta infestada recogida en Xàbia (Alicante, C. Valenciana) en junio de 2007 (población JAV, ver tabla 2.2.1). Inicialmente la población JAV se adaptó a la puesta en manzana y en la generación F10 se cruzaron hembras vírgenes de la población JAV con machos vírgenes de la generación F38 de la línea W-4Km y viceversa. La descendencia de estos dos cruzamientos se juntó en la siguiente generación, estableciéndose la línea Xàbia-W. Esta línea se mantuvo bajo presión creciente de selección frente a espinosad según lo descrito en el apartado 2.2.1.2. A partir de la generación F36 la concentración aplicada para la selección se fijó en 100 ppm de espinosad durante 48 horas. 2.2.3. Recogida de poblaciones de campo. En diferentes parcelas de frutales y cítricos se recolectó fruta infestada por C. capitata. Se estudiaron un total de doce poblaciones localizadas en las comunidades autónomas de Andalucía, Aragón, Cataluña, C. Valenciana e Islas Baleares. En la tabla 2.2.1 se muestra su código de nomenclatura, la fecha de recogida, el tipo de fruto hospedador y los tratamientos aplicados en cada parcela en el caso de conocerse. La fruta se mantuvo entre una y dos semanas en el insectario en unas cajas de plástico con ventilación de 30x20x20 cm. En el fondo de las cajas se colocaron varias hojas de papel secante que evitaron la acumulación del líquido procedente de la descomposición de la fruta. Las cajas se revisaron diariamente, recogiendo y procesando las pupas según el protocolo de cría descrito en el apartado 2.2.1.1. Los adultos que emergieron de esas pupas se emplearon en bioensayos de susceptibilidad (apartado 2.2.4) o se utilizaron para establecer nuevas líneas de laboratorio (apartado siguiente). 2.2.3.1. Adaptación de las poblaciones de campo al laboratorio. Para establecer una línea de laboratorio a partir de individuos procedentes de la fruta recogida en el campo, los adultos se mantuvieron en condiciones similares a las descritas en el apartado 2.2.1.1, pero previamente las hembras fueron adaptadas a realizar la puesta a través de 40 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ la malla de nailon de las jaulas de cría. Durante las primeras generaciones en el laboratorio, en el interior de las jaulas se colocaron manzanas de la variedad Royal Gala donde las hembras realizaban la puesta. Las manzanas se recogieron cada dos días y en ellas se desarrollaron las larvas tal y como se describe en el apartado 2.2.3. Al cabo de varias generaciones, se forzó la puesta en malla de una parte de la población eliminando la manzana del interior de algunas jaulas de cría e impregnando la malla con zumo de manzana. Cuando una proporción alta de hembras puso sus huevos a través de la malla de nailon, se eliminaron las manzanas del interior del resto de jaulas y se prosiguió con el método de cría explicado en el apartado 2.2.1.1. 2.2.4. Bioensayos de susceptibilidad a insecticidas. 2.2.4.1. Condiciones generales. Para realizar los bioensayos de susceptibilidad a insecticidas, los adultos de C. capitata se introdujeron durante 10-15 minutos en una nevera a 4ºC para facilitar su manejo. Con unas pinzas entomológicas se depositaron entre 12 y 15 adultos, de entre 3 y 5 días de edad, en el interior de cajas transparentes de plástico de 2,5 cm de alto por 9 cm de diámetro, que contaban con un orificio de 2 cm de diámetro cubierto con papel de filtro a modo de respiradero (Fig. 2.2.4). En el interior de cada caja se colocaron dos recipientes de 1,5 cm de alto y 2,5 cm de diámetro, uno con una mecha de material absorbente con agua y otro con dieta (0,9 g de azúcar y extracto de levadura de cerveza en proporción 4:1), a la que se le habían añadido 100 µl del insecticida diluido en agua. El tiempo de duración del bioensayo de susceptibilidad fue de 48 h y se mantuvo en el interior de las cámaras climáticas con un fotoperiodo de 16/8h de luz/oscuridad y a una temperatura de 25ºC. En general, se realizaron cuatro réplicas experimentales por bioensayo, a no ser que se especifique lo contrario. Tras cumplirse el tiempo de duración del bioensayo, se anotaron los individuos muertos con respecto al número total de individuos de cada una de las cajas. Se consideraron como muertos, además de los inmóviles, aquellos individuos que, presentando indicios de estar afectados por el insecticida, no recuperaron la postura normal sobre sus patas al ser tocados con unas pinzas entomológicas. Todos los bioensayos para los diferentes insecticidas se realizaron por ingestión, con la excepción del bioensayo de susceptibilidad a carbaril, donde la aplicación del insecticida fue tópica. Esto fue debido a la imposibilidad de disolver el producto comercial a concentraciones suficientemente elevadas para obtener efecto tóxico 41 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata Valle de Río Verde (Granada) RIO VIY JAV Vila Joiosa (Alicante) Xàbia (Alicante) MAR MUR Marratxí (Mallorca) Muro (Mallorca) Islas Baleares W07 ALC Castelló’07 (Castellón) C. Valenciana Alcanar (Tarragona) Cataluña Villalengua (Zaragoza) VIG Jun, 2007 GIB Gibraleón (Huelva) Aragón Sep, 2008 AYA Ayamonte (Huelva) Nov, 2007 Nov, 2007 Jun, 2007 May, 2007 Dic, 2007 Oct, 2007 Sep, 2007 Sep, 2008 Jun, 2007 GOL Almuñecar (Granada) Dic, 2008 Fecha MAY Código Algarrobo Costa (Málaga) Andalucía Localidad (Provincia) Cítricos Naranja Naranja Naranja Naranja 42 Mandarina Melocotón Níspero Mandarina Mandarina Níspero Chirimoya Hospedador 2005 y 2006: Fentión. 2007: Espinosad y metil-clorpirifos 2005 y 2006: Fentión. 2007: Metil-clorpirifos y fentión No tratada No tratada 2005 y 2006: 5-10 T malatión. 2007: No tratada 2005 y 2006: 7 T Malatión. 2007: λ-cialotrina y 2 T malatión No tratada No tratada 2006 y 2007: 7-8 T malatión. 2008: 3 T malatión y 2 T espinosad 2003 a 2008: 7-8 T espinosad y 3 T λ-cialotrina No tratada No tratada Tratamientos en campo (T) recolección y el fruto recogido (hospedador). El código hace referencia a la nomenclatura abreviada asignada a cada población (adultos que emergen de la fruta recogida en un mismo campo de cultivo). En los tratamientos (T) se indica el historial de los insecticidas aplicados en cada año dirigidos contra C. capitata y la frecuencia de aplicación en el caso de conocerse. Tabla 2.2.1. Lugares de muestreo de C. capitata. Se indican las localidades en la que se recogió fruta infestada de C. capitata, la fecha de _____________________________________________________________________________________________________________________________________________________________________________________________________________ 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ por ingestión. El carbaril se disolvió en acetona (Merck KGaA, Alemania) y se aplicó tópicamente un volumen de 0,5 µl en el tórax de las moscas con un microaplicador semiautomático Burkard Precisor Miniaplicator 900X (Burkard Manufacturing Co., Inglaterra). La elaboración de un bioensayo se asumió correcta cuando la mortalidad de los controles no superaba el 10% (normalmente entre 0-5%). 2.2.4.2. Bioensayos para estimar la CL50 Se estudió la susceptibilidad de la línea de laboratorio W-4Km frente a los insecticidas enumerados en la tabla 2.2.2 (ver estructura química de los compuestos en la figura 2.2.5). Se analizaron seis insecticidas organofosforados (malatión, fentión, diazinón, fosmet, triclorfón y metil-clorpirifos), el piretroide lambda-cialotrina, el carbamato carbaril (único de los insecticidas analizados no aprobado para su uso contra C. capitata), y un insecticida de origen biológico (espinosad). En el diseño del bioensayo se aplicaron entre cinco y siete concentraciones crecientes de insecticida y un tratamiento control sin insecticida. En aquellos casos en los que se empleó por primera vez un insecticida, se realizó un preensayo en las mismas condiciones del propio bioensayo. El preensayo consistió en aplicar un rango amplio de concentraciones y, una vez observados los datos de mortalidad, se ajustaron las concentraciones para el bioensayo definitivo. Figura 2.2.4. Caja de bioensayo. Caja empleada en la realización de los bioensayos. Cada caja contenía un bebedero con agua y un comedero en el que se introducía la mezcla de dieta e insecticida. 43 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ MALATIÓN FENTIÓN FOSMET TRICLORFÓN DIAZINÓN METIL-CLORPIRIFOS LAMBDA-CIALOTRINA ORGANOFOSFORADOS PIRETROIDE Espinosina A: R = H Espinosina D: R = CH3 CARBARIL ESPINOSAD R LUFENURÓN CARBAMATO ESPINOSINA * BENZOILUREA Figura 2.2.5. Insecticidas. Estructura química de los compuestos insecticidas empleados en los bioensayos de susceptibilidad. (*) No se realizaron bioensayos de susceptibilidad para el lufenurón, si no que se evaluó su efecto quimioesterilizante en el apartado 2.2.5. Los porcentajes de mortalidad obtenidos en cada bioensayo de susceptibilidad fueron analizados con el programa PoloPlus v.1.0 (LeOra Software). Dicho programa calcula, corrigiendo la mortalidad según Abbott (1925), las concentraciones letales 50 (CL50) y su intervalo de confianza al 95% para cada población e insecticida. La CL50 indica la concentración de insecticida aplicada en dieta que provoca una mortalidad del 50% de los individuos en el tiempo que dura el bioensayo. Con el objetivo de comparar las CL50 de diferentes poblaciones o líneas de laboratorio se calculó la razón de las concentraciones letales 50 (RCL) y su intervalo de confianza al 95%. La RCL consiste en tomar como referencia una población, normalmente la línea más susceptible, y calcular la proporción de cada una de las poblaciones o líneas con respecto a ella. Si el intervalo de confianza generado para una determinada población no incluye el valor 1 quiere decir que existen diferencias significativas entre dicha población y la población susceptible tomada como referencia (Robertson y Preisler, 1992). 44 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ 2.2.4.3. Bioensayos de concentración discriminante. Teniendo en cuenta la susceptibilidad frente a malatión en las líneas susceptible C (CL50 = 18 ppm) y resistente W (CL50 = 1.460 ppm) (Magaña et al., 2007), se seleccionó una concentración de insecticida que pudiera ser utilizada para discriminar entre poblaciones de campo resistentes y susceptibles. Concretamente se emplearon 1.500 ppm de malatión, ya que experimentalmente se ha comprobado que concentraciones superiores a 300 ppm producen el 100% de mortalidad en la línea C. En el caso del espinosad se utilizó 1 ppm ya que la CL50 de la línea susceptible C fue igual a 0,4 ppm y la CL90 estimada fue de 1,3 ppm (Magaña et al., 2007). Además, en el caso del malatión y con el objeto de obtener información sobre el posible mecanismo de resistencia, se analizó el efecto de la administración de tres sinergistas: el maleato de dietilo (DEM), inhibidor de las glutatión S-transferasas (producto técnico al 97%, Aldrich Chemical Co); el SSS-tributilfosforotritioato (DEF), inhibidor de esterasas (producto técnico al 98%, Chem. Service Inc.); y el butóxido de piperonilo (PBO), inhibidor de las monooxigenasas (producto técnico al 90%, Aldrich Chemical Co). Estos bioensayos de insecticidas con sinergistas se realizaron siguiendo la metodología descrita en el apartado 2.2.4.1. Los sinergistas se aplicaron dos horas antes de la exposición al malatión, mediante un microaplicador semiautomático Burkard Precisor Miniaplicator 900X, para permitir la acción del sinergista antes de la aplicación del insecticida. La dosis correspondiente de sinergista disuelto en acetona se aplicó depositando 0,5 µl sobre el tórax de las moscas. Mediante ensayos previos se ajustaron las dosis aplicables por insecto, que fueron de 1 µg de DEM, 1 µg de DEF y 3 µg de PBO. Los resultados de mortalidad observados fueron corregidos según la fórmula propuesta por Abbott (1925). 45 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ® Lufenurón (QE) Adress GF-120* NF Espinosad (SP) ® Keyvin 80 pm ® Karate King ® Reldan E ® Dipterex Imidan WP ® Agrozinon ® Lebaycid 50 LE ® Malafin 50 ® Nombre comercial Carbaril (CA) λ-cialotrina (PI) Metil-clorpirifos (OP) Triclorfón (OP) Fosmet (OP) Diazinón (OP) Fentión (OP) Malatión (OP) Materia activa 3% (p/p) 0,02% (p/v) 80% (p/p) 2,5% (p/p) 22,4% (p/v) 50% (p/v) 50% (p/p) 60% (p/v) 50% (p/v) 50% (p/v) Concentración 46 Syngenta Agro S.A. Dow Agrosciences LLC Industrial Química Key, S.A. Syngenta Agro S.A. Dow Agrosciences Ibérica, S.A. Bayer Hispania, S.A. Kenogarg, S.A. Agrofit Sociedad Cooperativa Tecniagro, S.L. Agrodán, S.A. Fabricante 30.000 40 - 133 (2) 100 - 125 3.000 - 4.000 4.000 - 5.000 3.000 1.200 1.000 - 2.000 2.000 - 3.000 C.R.F. (1) Ingestión Ingestión Tópica Ingestión Ingestión Ingestión Ingestión Ingestión Ingestión Ingestión Aplicación Tabla 2.2.2. Listado de insecticidas empleados en los bioensayos de susceptibilidad. Se utilizaron insecticidas organofosforados (OP), un piretroide (PI), un carbamato (CA), una mezcla de espinosinas A y D (SP) y un quimioesterilizante (QE). La concentración indica el porcentaje de principio activo en el producto comercial en peso/peso (p/p) o en peso/volumen (p/v). (1) Muestra la concentración de insecticida recomendada por el fabricante (C.R.F.) para su uso contra C. capitata, en ppm. (2) El carbaril no se emplea específicamente contra C. capitata. Se indica además el método de aplicación empleado en los bioensayos de susceptibilidad. _____________________________________________________________________________________________________________________________________________________________________________________________________________________ 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ 2.2.5. Bioensayos con el quimioesterilizante lufenurón. El lufenurón (Fig. 2.2.4) es un regulador del crecimiento que interfiere en la síntesis de la quitina de los insectos, afectando tanto a la muda como al correcto desarrollo de los huevos, por tanto, uno de sus efectos insecticidas es la quimioesterilización (Wilson y Cryan, 1997). El bioensayo con lufenurón fue realizado por el grupo del Dr. Vicente Navarro Llopis de la Universidad Politécnica de Valencia. Se prepararon, para cada concentración de lufenurón, cuatro jaulas de 10x10x10cm de metacrilato con una cara cerrada con una tela de nailon. En cada jaula se introdujeron cinco hembras adultas de siete días de edad, que previamente habían copulado, y que habían estado sometidas a un periodo de ayuno de 24 horas. En total se ensayaron cinco concentraciones crecientes de lufenurón en la dieta, además de un control sin lufenurón. La dieta suministrada fue una mezcla en proporción 4:1 de azúcar y extracto de levadura de cerveza, respectivamente, y 0,5 ml de acetona por gramo de dieta sólida. En el caso de las jaulas tratadas con el quimioesterilizante, se disolvió el lufenurón en acetona y posteriormente, con la ayuda de un mortero, se homogeneizó con la dieta dejando evaporar la acetona. La duración del bioensayo fue de 24 horas, y se realizaron cuatro réplicas experimentales de cada tratamiento. Transcurrido el tiempo del bioensayo, se reemplazó la dieta con lufenurón por la dieta sin tratamiento. A partir de las 24 horas desde la retirada del lufenurón se recogió la puesta de las 24 horas posteriores. Los huevos se sembraron sobre agar y se mantuvieron a 26ºC durante 3 días. Al cuarto día se observaron los huevos eclosionados mediante una lupa binocular. Se observaron cincuenta huevos sembrados y se anotaron aquellos que habían eclosionado, calculando así el porcentaje de huevos no desarrollados con respecto al total de huevos. Los datos de esterilidad obtenidos fueron analizados con el programa PoloPlus v.1.0 (LeOra Software) de manera similar a la descrita en el apartado 2.2.4.2., calculándose la concentración esterilizante 50 (CE50) y su intervalo de confianza al 95%. La CE50 indica la concentración de quimoesterilizante aplicada en dieta que provoca una esterilidad del 50% de los huevos en las condiciones del bioensayo. La razón de las concentraciones esterilizantes 50 (RCE) y su intervalo de confianza al 95% se calcularon según lo descrito por Robertson y Preisler (1992). 47 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ 2.3. Resultados. 2.3.1. Evaluación de la susceptibilidad a malatión y espinosad en poblaciones de campo. Mediante un bioensayo de concentración discriminante (apartado 2.2.4.3), realizado sobre individuos adultos procedentes de fruta recogida en campos comerciales, se evaluó la susceptibilidad a malatión y espinosad. Además, con el objetivo de obtener información sobre la posible existencia de resistencia a malatión mediada por enzimas de detoxificación, se realizaron bioensayos con tres sinergistas. La mortalidad observada en los tratamientos control sin insecticidas osciló entre el 2% y 11% en la mayoría de poblaciones analizadas y sólo superó estos valores en las poblaciones recolectadas en Almuñecar (Granada) y Marratxí (Mallorca) (24 y 28% respectivamente) (Tabla 2.3.1). En ninguno de los bioensayos con 1.500 ppm de malatión se obtuvo una mortalidad del 100%, oscilando entre el 64 y el 97%. Las mortalidades más bajas (64% a 73%) se encontraron en las poblaciones de Xàbia (Alicante), Marratxí (Mallorca) y Alcanar (Tarragona). 49 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ Además, tampoco se alcanzaron mortalidades del 100% cuando se combinó la aplicación tópica de un sinergista (DEM, DEF o PBO) con el tratamiento de malatión en la dieta. En ninguna de las poblaciones estudiadas se observó una alteración en la susceptibilidad frente a malatión tras la aplicación del sinergista, a excepción de lo observado en la población mallorquina de Muro, donde el PBO redujo la susceptibilidad significativamente. En los tratamientos con 1 ppm de espinosad las mortalidades observadas estuvieron por encima del 60% en la mayoría de las poblaciones estudiadas, con dos excepciones: las poblaciones de Xàbia (Alicante) y Marratxí (Mallorca), donde los porcentajes de mortalidad fueron del 36% y 17%, respectivamente. La disponibilidad de un número suficiente de individuos pertenecientes a la población de Xàbia nos permitió realizar un bioensayo de susceptibilidad para estimar la CL50 frente a espinosad, así como establecer la población en el laboratorio y comenzar el proceso de selección de resistencia a espinosad, proceso que se describe posteriormente en el apartado 2.3.4. 50 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ALC ALM MAY AYA CAS GIB MAR MUR RIO VIY VIL JAV Alcanar (Tarragona) Almuñecar (Granada) Algarrobo Costa (Málaga) Ayamonte (Huelva) Castelló’07 (Castellón) Gibraleón (Huelva) Marratxí (Mallorca) Muro (Mallorca) Valle de Río Verde (Granada) Vila Joiosa (Alicante) Villalengua (Zaragoza) Xàbia (Alicante) 260 369 372 354 257 76 132 94 90 86 369 586 n 75 (40) 71 (39) 83 (56) 90 (54) 96 (60) 93 (60) 94 (57) 96 (59) 95 (40) 85 - - - - - (53) (26) 73 (44) 97 (47) 89 (30) 97 (42) 93 93 (55) 95 (58) 66 (101) 64 +DEM (98) M (40) 80 (58) 95 (60) 94 (52) 98 (40) 92 - - - - - (59) 98 (97) 70 +DEF (40) 77 (52) 94 (58) 93 (51) 98 (40) 67 * - - - - - (55) 98 (102) 57 +PBO (41) 36 (58) 71 (60) 89 (60) 86 (44) 77 (25) 17 (44) 74 - (30) 63 - (60) 80 (88) 77 Espinosad (1 ppm) 51 ________________________________________________________________________________________________________________________ CÓD. Población Malatión (1.500 ppm) % de Mortalidad Tabla 2.3.1. Análisis de la susceptibilidad a malatión y espinosad en poblaciones de campo de C. capitata. En la evaluación de la susceptibilidad a malatión, además de la aplicación de dicho insecticida en solitario (M), se realizó un tratamiento combinado con los sinergistas DEM (maleato de dietilo), DEF (SSS-tributilfosforotritioato) y PBO (butóxido de piperonilo). Se indica el número total de insectos del bioensayo (n) y los porcentajes de mortalidad, corregida según Abbott (1925), para los insecticidas malatión y espinosad. Entre paréntesis se indica el número de insectos utilizados en cada bioensayo. (*) Diferencias significativas con respecto a M ( χ 22g .l . = 25,4 > 3,841 , p<0,05). _____________________________________________________________________________________________________________________________________________________________________________________________________________ 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ 2.3.2. Selección de resistencia a malatión en el laboratorio: Establecimiento de las líneas W-4Km y W-10Km. A partir de la población Castelló, resistente a malatión (Magaña et al., 2007) se estableció en el laboratorio la línea W, mantenida bajo presión de selección con dicho organofosforado. Inicialmente su cría se realizaba sobre manzanas pero, a partir de la generación F13, las hembras fueron adaptadas a realizar la puesta a través de una malla de nailon, gracias a lo cual se facilitó el manejo de los insectos. La respuesta de esta línea a la presión de selección con malatión se puede observar en la tabla 2.3.2. En la generación F0, la CL50 estimada de la población Castelló recogida en campo fue de 1.874 ppm (Magaña et al., 2007). Durante las primeras doce generaciones la concentración de malatión empleada para la selección fue de 1.500-2.000 ppm, estimándose una CL50 de 3.962 ppm de malatión en la generación F13. A lo largo de las siguientes diez generaciones (entre la F13 y la F23) se suspendió el tratamiento insecticida y se observó que, en ausencia de presión de selección, la resistencia revertía a niveles muy bajos (163 ppm), disminuyendo más de diez veces respecto a la resistencia estimada en la F0 (RCLF0 = 0,09; intervalo de confianza [IC]: 0,02-0,30). En la generación F24 se aplicó de nuevo una concentración de selección de 2.000 ppm de malatión, y en cuatro o cinco generaciones la susceptibilidad de la línea W disminuyó rápidamente, alcanzando la CL50 un valor de 2.398 ppm. En la generación F30 se dividió la línea W en dos líneas sometidas a presiones de selección distintas. En la primera se fijó la concentración de selección en 4.000 ppm de malatión durante 48 horas, denominándose a partir de ese momento línea W-4Km. La resistencia a malatión estimada en la generación F32 de la línea W-4Km (CL50 = 6.170 ppm) respecto a la generación F0 se incrementó 3,3 veces (IC: 1,5-7,4), resultando ser 154 veces (IC: 78-304) más resistente a malatión que la línea susceptible de laboratorio. En la segunda línea de selección, la concentración de malatión empleada se incrementó paulatinamente hasta alcanzar las 10.000 ppm en la generación F40 (Tabla 2.3.3), denominándose a partir de este momento línea W-10Km. La presión de selección se mantuvo constante en 10.000 ppm de malatión a partir de esta generación, donde la CL50 se estimó en 16.163 ppm. El aumento en la presión de selección aplicado en la línea W-10Km respecto a W-4Km provocó que los niveles de resistencia se incrementasen en mayor medida en la línea W-10Km en relación a la F0 de Castelló (RCL 8,6, IC: 4,9-15,2), que fue 403 veces (IC: 288-564) más resistente a malatión que la línea susceptible de laboratorio. . 52 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata 0 0 0 2.000 2.000 2.000 2.000 2.000 F21 F22 F23 F24 F25 F26 F28 F30 4.000 0 F19 F32 0 F18 4.000 2.000 F13 F31 2.000 F9 Línea W-4Km 1.500 F2 (2) Línea W - CS F0 (1) Generación Castelló Población/Línea (3) 302 314 302 362 287 281 305 278 241 248 187 270 194 211 210 208 n 53 1,2 ± 0,3 1,1 ± 0,2 0,8 ± 0,1 0,8 ± 0,1 1,2 ± 0,1 1,1 ± 0,1 0,8 ± 0,1 1,1 ± 0,1 0,8 ± 0,1 0,9 ± 0,2 1,2 ± 0,3 0,7 ± 0,1 0,9 ± 0,2 0,7 ± 0,1 0,9 ± 0,1 0,9 ± 0,1 m ± E.E. (4) (5) (IC 95%) 6.170 (3.326 - 15.513) 5.456 (3.129 - 8.646) 2.398 (1.255 - 4.336) 6.146 (3.065 - 22.567) 627 (292 - 1.160) 671 (399 - 1.131) 1.769 (960 - 3.861) 896 (596 - 1.324) 613 (226 - 1.299) 223 (84 - 404) 163 (26 - 335) 310 (42 - 811) 3.962 (1.950 - 10.176) 1.237 (387 - 4.317) 1.406 (633 - 3.150) 1.874 (1.190 - 3.300) CL50 ◊ ◊ ◊ ◊ 18 18 18 14 22,5 ◊ 16,1 ◊ 17,6 ◊ 18 22 18 18 18 17 14 22 22 19 32 22 g.l. 31,6 38,5 30,8 ◊ 27,2 ◊ 12,3 ◊ 25,1 20,6 14,0 34,6 13,7 30,1 62,1 20,5 ◊ 2 χ (6) Tabla 2.3.2. Selección de resistencia a malatión en líneas de laboratorio de C. capitata: Establecimiento de W-4Km. (1) Indica el número de la generación durante el proceso de selección. (2) Concentración de malatión utilizada en la selección en ppm. (3) Número de individuos utilizados en el bioensayo. (4) Pendiente (m) y error estándar (E.E.) de la recta probit. (5) Concentración letal 50 (CL50) expresada en ppm de malatión con el intervalo de confianza (IC) al 95%. (6) Grados de libertad. (◊) Los datos se ajustan al modelo probit (p>0,05). _____________________________________________________________________________________________________________________________________________________________________________________________________________ 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata W-10Km Línea F40 F37 F30 (1) Generación (2) 10.000 7.000 4.000 CS (3) 309 261 310 n 54 2,1 ± 0,2 1,6 ± 0,3 0,9 ± 0,1 m ± E.E. (4) (5) (IC 95%) 16.163 (12.565 - 22.382) 8.427 (4.233 - 13.498) 1.201 (697 - 1.849) CL50 27,6 18 14 21,2 ◊ ◊ 18 g.l. 15,8 ◊ 2 χ (6) 10Km. (1) Indica el número de la generación durante el proceso de selección. (2) Concentración de malatión utilizada en la selección en ppm. (3) Número de individuos utilizados en el bioensayo. (4) Pendiente (m) y error estándar (E.E.) de la recta probit. (5) Concentración letal 50 (CL50) expresada en ppm de malatión con el intervalo de confianza (IC) al 95%. (6) Grados de libertad. (◊) Los datos se ajustan al modelo probit (p>0,05). Tabla 2.3.3. Selección de resistencia a malatión en líneas de laboratorio de C. capitata: Establecimiento de la línea W10Km. Partiendo de la línea W-4Km e incrementando la concentración de selección hasta 10.000 ppm de malatión, se estableció la población W- _____________________________________________________________________________________________________________________________________________________________________________________________________________ 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ 2.3.3. Resistencia cruzada en la línea W-4Km. En la línea W-4Km se analizó la posible existencia de resistencia cruzada a varios insecticidas. Se seleccionaron por su modo de acción y su estructura química un total de nueve insecticidas y se calculó para cada uno de ellos la razón de las concentraciones letales 50 (RCL) en relación a la línea susceptible C (Tabla 2.3.4). Además, se evaluó la capacidad esterilizante del lufenurón mediante el cálculo de la concentración esterilizante 50 (CE50) y de la razón de las concentraciones esterilizantes (RCE) con respecto a la línea susceptible C. La línea W-4Km fue 178 veces más resistente a malatión que la línea C, alcanzando unos niveles de resistencia (CL50 = 5.456 ppm) del rango de las concentraciones recomendadas por el fabricante del Malafin® 50 CE (2.000 - 3.000 ppm). Se detectaron diferencias de susceptibilidad entre ambas líneas a todos los insecticidas organofosforados estudiados (OP), aunque las RCL fueron menores que en el caso del malatión, ya que oscilaron entre 7 veces para el triclorfón y el metil-clorpirifos, y 16 veces para el diazinón, siendo todos los valores estadísticamente significativos. En el caso del carbamato carbaril no fue posible calcular la CL50 en la línea W-4Km pero, según los datos observados (17% de mortalidad con 2,56 ppm de carbaril), con la máxima dosis aplicada la resistencia sería mayor de 4 veces con respecto a la línea susceptible C. Además, se observó una RCL significativa 6 veces mayor con respecto a la línea control C cuando se analizó la susceptibilidad al piretroide λ-cialotrina y una RCE significativa de 6 veces más cuando se analizó la susceptibilidad al quimioesterilizante lufenurón. Cuando se comparó la susceptibilidad de ambas líneas frente al espinosad, aunque hubo diferencias significativas al evaluar la RCL, esta diferencia fue muy baja (1,5 veces). Con el objeto de determinar si las diferencias de susceptibilidad detectadas al comparar las CL50 de las líneas C y W-4Km eran debidas a la alta susceptibilidad de la línea C, se analizó la susceptibilidad frente al organofosforado malatión y el piretroide lambda-cialotrina de una población recogida en la finca de La Mayora (Algarrobo Costa, Málaga), finca experimental del CSIC en la que nunca se habían realizado tratamientos con insecticidas frente a C. capitata (Tabla 2.3.4, población MAY). En el caso del malatión, se observó que no existían diferencias significativas entre la CL50 de la población MAY y la CL50 de la línea susceptible de laboratorio C. Sin embargo, en el caso de la lambda-cialotrina, la RCL calculada para la población de MAY fue similar a la estimada para la línea W-4Km, siendo ambas poblaciones unas 6 veces más resistentes que la línea susceptible C. 55 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata C W-4Km MAY 4.000 - 5.000 Triclorfón (OP) Metil-clorpirifos (OP) 3.000 - 4.000 λ-cialotrina (PI) 40 - 133 3.000 Lufenurón (QE) (5) Espinosad (SP) Carbaril (CA) C W-4Km 3.000 Fosmet (OP) 1.000 - 1.250 C W-4Km 1.200 Diazinón (OP) C W-4Km C W-4Km C W-4Km C W-4Km C W-4Km C W-4Km 1.000 - 2.000 Fentión (OP) Línea C W-4Km MAY C.R.F. 2.000 - 3.000 Insecticida Malatión (OP) (1) 1.250 2.416 305 379 288 291 264 235 208 345 306 271 255 321 187 317 313 219 319 n 217 314 203 4,5 ± 0,5 1,9 ± 0,4 4,4 ± 1,0 4,0 ± 0,4 1,0 ± 0,2 - 1,0 ± 0,1 1,0 ± 0,2 1,8 ± 0,5 3,8 ± 0,4 1,9 ± 0,3 4,8 ± 1,2 3,1 ± 0,6 2,6 ± 0,4 - 2,7 ± 0,3 1,1 ± 0,2 1,0 ± 0,3 2,7 ± 0,4 m + E.E. 1,5 ± 0,4 1,2 ± 0,3 1,1 ± 0,2 56 (9 - 12) (110 - 229) (0 - 5) (8 - 14) (6 - 11) (40 - 70) 0,6 (0,5 - 0,7) 0,9 (0,8 - 1,0) (6) 9 52 - (36 - 110) (253 - 1.249) (280 - 959) (4 - 5) (23 - 41) (20 - 27) (127 - 195) 0,6 (0,3 - 1) 66 466 463 5 32 23 166 133 (108 - 160) (4) - 10 166 1 11 (2) CL50 (IC 95%) 31 (15 - 57) 5.456 (3.129 - 8.646) 76 (39 - 134) ◊ 90,1 221,8 12,2 18,9 ◊ ◊ 20,0 ◊ - 19,7 16,2 ◊ 10,7 ◊ ◊ 6,5 13,2 ◊ ◊ 12,3 22,1 ◊ 11,2 - ◊ 19,9 7,2 ◊ 18 25 18 22 18 18 18 14 9 13 17 13 18 18 14 18 18 17 14 18,5 ◊ 20,5 ◊ ◊ g.l. 18 18 9 2 χ 21,2 ◊ 16,1 ◊ 5,3 ◊ (3) (IC 95%) 6 (5 - 7) * 1,5 (1,3 - 1,9) * >4 6 (2 - 17) * 6 (2 - 17) * 7 (5 - 10) * 7 (5 - 10) * > 15 16 (10 - 25) * 8 (5 - 11) * 178 (82 - 272) * 2 (1 - 4) RCL laboratorio: C susceptible y W-4Km resistente a malatión. Además se estudió una población de campo no tratada recogida en Málaga (MAY). Se aplicaron insecticidas organofosforados (OP), un piretroide (PI), un carbamato (CA), una espinosina (SP) y un quimioesterilizante (QE). Excepto el bioensayo con carbaril que se realizó tópicamente, el resto de insecticidas se aplicaron por ingestión. (1) Concentración de insecticida recomendada por el fabricante contra C. capitata, en ppm. (2) La concentración letal 50 expresada en ppm, para el lufenurón se indica la CE50. (3) Razón de las concentraciones letales 50 respecto a la línea susceptible C. (4) Aplicando una concentración máxima de 2.000 ppm de fosmet, la mortalidad fue del 30%. (5) El carbaril no se emplea específicamente contra C. capitata. (6) Aplicando una dosis máxima de 2,56 ppm de carbaril, la mortalidad fue del 17%. (◊) Los datos se ajustan al modelo probit (p>0,05). (*) La concentración letal es significativamente diferente (p<0,05) si RCL no incluye el 1. Tabla 2.3.4. Susceptibilidad de C. capitata frente a diferentes insecticidas. Se analizaron individuos adultos de las líneas de _____________________________________________________________________________________________________________________________________________________________________________________________________________ 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ 2.3.4. Selección de resistencia a espinosad en laboratorio: Establecimiento de la línea Xàbia-W-100s. Los individuos provenientes de la fruta recogida en Xàbia (Alicante) mostraron, junto con los de Marratxí (Mallorca), los menores valores de susceptibilidad frente a espinosad (36% con 1 ppm, ver tabla 2.3.1). Para confirmar este resultado se realizó un bioensayo de susceptibilidad a espinosad con la población Xàbia (F0), estimando una CL50 de 1,6 ppm (Tabla 2.3.5). Los individuos supervivientes de los bioensayos de susceptibilidad a espinosad realizados en Xàbia (ver apartado 2.3.1) se mantuvieron en una jaula de cría con el objetivo de establecer una línea de laboratorio resistente a espinosad. Inicialmente fue mantenida sobre manzana y a partir de la cuarta generación se adaptó a la puesta en malla. Durante las primeras diez generaciones se aplicó una presión de selección de 1 ppm de espinosad durante 48 horas. Debido a un descenso en el número de individuos, posiblemente provocado por la presión de selección, se cruzó la generación F10 de la línea Xàbia con la generación F38 de la línea W-4Km, resistente a malatión. Hasta la generación F24 los niveles de susceptibilidad a espinosad de la línea Xàbia-W fueron similares a los de la población inicial, a pesar de que la selección se realizaba con concentraciones crecientes de espinosad, que llegaron hasta 4 ppm. Sin embargo, en la generación F25, cuando se seleccionaba con 6,5 ppm de espinosad, se observó una leve disminución en la susceptibilidad (CL50 =3,4 ppm). En la generación F29, la CL50 aumentó drásticamente hasta 187 ppm de espinosad, con una RCL de 499 (IC: 344-723) con respecto a la línea C, lo que supuso un incremento de 120 veces en la resistencia frente a espinosad con respecto a la población inicial. A partir de la generación F30 se fijó la concentración de selección en 100 ppm de espinosad, manteniéndose la susceptibilidad de la línea seleccionada, como se puede observar en la CL50 de la generación 36 (Tabla 2.3.5). 57 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata 6,5 10,0 F25 F29 100,0 4,0 F21 F36 3,0 F20 Xàbia-W-100s 2,5 F14 (2) Xàbia-W - CS F0 (1) Generación Xàbia Población/Línea (3) 366 329 189 302 363 262 244 n 58 1,9 ± 0.3 2,0 ± 0,4 2,5 ± 0,3 4,2 ± 0,6 3,5 ± 0,4 2,5 ± 0,4 2,4 ± 0,4 m ± E.E. (4) (5) (IC 95%) 156,6 (126,2 - 207,1) 187,0 (151,7 - 261,6) 3,4 (2,8 - 4,3) 1,4 (1,2 - 1,6) 1,5 (1,2 - 1,8) 0,6 (0,5 - 0,8) 1,6 (0,9 - 2,2) CL50 ◊ 28,0 ◊ 12,5 ◊ 20,4 15,1 ◊ 22 18 18 14 18 14 11,4 ◊ 35,6 18 g.l. 22,1 ◊ 2 χ (6) número de la generación durante el proceso de selección. (2) Concentración de espinosad utilizada en la selección en ppm. (3) Número de individuos utilizados en el bioensayo. (4) Pendiente (m) y error estándar (E.E.) de la recta probit. (5) Concentración letal 50 (CL50) expresada en ppm de malatión con el intervalo de confianza (IC) al 95%. (6) Grados de libertad. (◊) Los datos se ajustan al modelo probit (p>0,05). Tabla 2.3.5. Selección de resistencia a espinosad en laboratorio: Establecimiento de la línea Xàbia-W-100s. (1) Indica el _____________________________________________________________________________________________________________________________________________________________________________________________________________ 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ 2.4. Discusión. En un estudio previo, el análisis de nueve poblaciones de C. capitata recogidas durante los años 2004 y 2005 en la C. Valenciana, Cataluña y La Rioja, se mostró que todas las poblaciones de campo analizadas presentaban una susceptibilidad a malatión significativamente inferior a la de dos líneas de laboratorio, observándose que los niveles más bajos de susceptibilidad se encontraban en la C. Valenciana, niveles que estaban relacionados con la alta frecuencia de tratamientos con malatión (Magaña et al., 2007). En este trabajo, hemos analizado la susceptibilidad a malatión de poblaciones españolas recogidas durante los años 2007 y 2008, un periodo en el que el malatión estaba siendo utilizado de forma indiscriminada y única. En el estudio de susceptibilidad, en esta ocasión, hemos utilizado concentraciones discriminantes y hemos ampliado el área de muestro a las comunidades autónomas de Andalucía, Islas Baleares y Aragón. La concentración discriminante de malatión escogida (1.500 ppm) provocó el 100% de mortalidad en una población susceptible de laboratorio. En las poblaciones estudiadas la mortalidad osciló entre el 63 y el 97%. La menor mortalidad observada correspondió a las poblaciones de Alcanar (Tarragona) y Xàbia (Alicante) y sugiere la existencia de resistencia frente a malatión en estas localidades. La mortalidad estimada en Marratxí (Mallorca) fue también baja pero, debido al bajo número de individuos analizados, debe ser interpretada con cautela. En el resto de poblaciones la susceptibilidad fue mayor, pero la existencia de individuos supervivientes a la concentración discriminante aplicada podría ser compatible con la existencia de individuos resistentes a malatión 59 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ en la mayoría de las localidades. Se debe tener en cuenta que los bioensayos de susceptibilidad realizados en este capítulo únicamente permiten detectar poblaciones resistentes cuando la frecuencia del alelo que confiere resistencia es alta, por lo que los datos de mortalidad se deben interpretar con precaución. Por ejemplo, la CL50 de malatión estimada para una población no sometida a tratamientos insecticidas (población MAY) no fue significativamente distinta a la de la línea susceptible C, sin embargo, se detectaron supervivientes (7%) en el tratamiento con una dosis discriminante de malatión que provocaría el 100% de mortalidad en individuos susceptibles. En la actualidad se está poniendo mucho énfasis en otro tipo de aproximaciones como la detección de alelos de resistencia mediante métodos moleculares (Vontas et al., 2011), que permiten estimar la frecuencia de un determinado alelo de resistencia en una población y pueden detectar resistencia cuando ésta se encuentra en niveles no detectables por los bioensayos de susceptibilidad (por ejemplo Menozzi et al., 2004; Nardi et al., 2006: Alou et al., 2010; Kawada et al., 2011). Dos mecanismos distintos han sido asociados a la resistencia a malatión observada en una población recogida en Castelló en 2004: la mutación puntual G328A en la acetilcolinesterasa (AChE) y un mecanismo de detoxificación mediado por carboxilesterasas, sugerido por una disminución de la actividad aliesterasa y por el sinergismo del DEF, que provocaba una disminución de la CL50 de aproximadamente 8 veces (Magaña et al., 2008). La ausencia de sinergismo en el tratamiento con DEF sobre las poblaciones analizadas en este trabajo sugiere que el mecanismo de resistencia mediado por carboxilesterasas no es el responsable de los bajos niveles de susceptibilidad encontrados en las poblaciones de Xàbia o Alcanar, donde se debería haber observado un claro incremento en la mortalidad al aplicar el sinergista. Por otro lado, el tratamiento realizado con los sinergistas DEM y PBO en las poblaciones estudiadas tampoco produjo en ningún caso un aumento significativo de la mortalidad, lo que sugiere que la resistencia no está mediada por las enzimas de detoxificación GSTs y P450, respectivamente. Por el contrario, sí se observó una disminución significativa de la mortalidad en la población de Muro en el tratamiento con PBO. La disminución de la mortalidad provocada por el PBO, que podría ser debida a la inhibición de la transformación del malatión en su forma activa (malaoxón) por parte de las enzimas P450, ha sido observada anteriormente en C. capitata y no ha sido asociada a un mecanismo de resistencia (Magaña et al., 2007). El análisis del segundo mecanismo de resistencia descrito, es decir, la presencia de la mutación G328A en la acetilcolinesterasa, se abordará en el capítulo 4. Mediante la selección con malatión en el laboratorio se obtuvieron dos líneas resistentes: la línea W-4Km y la W-10Km. La línea W, que provenía de una población de campo recogida en Castelló en 2004 (Magaña et al., 2007), fue adaptada a la puesta en malla y fue sometida a un 60 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ proceso de selección con concentraciones crecientes de malatión. Este proceso derivó en el establecimiento de la línea resistente W-4Km, que triplicó los niveles de resistencia de la población original. A partir de la línea W-4Km, aumentando la presión de selección, se estableció una segunda línea resistente denominada W-10Km, cuya CL50 estimada (16.163 ppm) fue superior a la concentración de malatión recomendada por el fabricante (2.000 - 3.000 ppm) e incluso a la concentración (7.500 ppm) que se aplicó en campo (Primo-Millo et al., 2003). La estabilización de la línea W-4Km nos permitió realizar los estudios de resistencia cruzada presentados en este capítulo, así como otros estudios que se abordarán en los capítulos 4 y 5 de esta tesis. Además, el proceso de selección, que resultó en el establecimiento de la línea W-10Km, nos permitió estudiar los individuos que presentaban los mayores niveles de resistencia a malatión, dando lugar al descubrimiento de un nuevo mecanismo de resistencia que se detallará en el capítulo 5. Debido a la prohibición del uso del malatión contra C. capitata, desde 2009 se ha incrementado el uso de otros insecticidas como son el espinosad y la lambda-cialotrina. La aplicación de estos insecticidas alternativos, con mecanismos de acción diferentes al del malatión, debería favorecer el control de la plaga, pero esta mejora en el control sería necesario corroborarla realizando estudios que clarifiquen si existe o no resistencia cruzada a los nuevos insecticidas empleados. De esta manera se podrá seleccionar el insecticida o insecticidas más aptos así como el conjunto de estrategias de manejo para el correcto control de la plaga. En este trabajo hemos analizado la susceptibilidad de la línea W-4Km, resistente a malatión, frente a diez insecticidas: los organofosforados malatión, fentión, diazinón, fosmet, triclorfón y metil-clorpirifos; el piretroide lambda-cialotrina; el carbamato carbaril; la espinosina espinosad; y la benzoilurea quimioesterilizante lufenurón. La evaluación de estos insecticidas puede aportar información sobre los mecanismos asociados con la resistencia frente a malatión y sobre los insecticidas a emplear como alternativas a este organofosforado, evitando así el incremento de los alelos de resistencia. Actualmente en España, según el registro de productos fitosanitarios del Ministerio de Agricultura, Alimentación y Medio Ambiente, de los insecticidas empleados en este trabajo se aprueba contra C. capitata la utilización de metil-clorpirifos, triclorfón, fosmet, espinosad, lambda-cialotrina y lufenurón. No está aceptado el uso de diazinón y malatión, aunque éstos si se han utilizado frente a esta plaga en campañas anteriores (Beitia et al., 2011). El único insecticida analizado en este estudio no dirigido específicamente frente a la mosca mediterránea de la fruta es el carbaril. La línea W-4Km presentó resistencia cruzada moderada (7-15 veces) frente a los organofosforados analizados y frente al carbamato carbaril, sin embargo, estos niveles de resistencia son muy inferiores a los niveles detectados en la línea W-4Km frente a malatión (178 veces). Esta diferencia podría deberse a dos posibles hipótesis: en primer lugar, la mutación AChE G328A puede conferir mayor insensibilidad a malaoxón (forma activa del malatión) que frente a 61 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ otros insecticidas que tienen como diana la AChE, tal y como ha sido demostrado en Musca domestica (Walsh et al., 2001) y en Drosophila melanogaster (Menozzi et al., 2004); y en segundo lugar, que el mecanismo de resistencia mediado por carboxilesterasas (Magaña et al., 2008) hidrolice de un modo más eficiente el malatión (Campbell et al., 1998b) que el resto de insecticidas analizados. Un fenómeno similar ocurre en la mosca Lucilia cuprina en la que la resistencia a malatión y a otros organofosforados está asociada a una mutación puntual en la aliesterasa E3 (Campbell et al., 1998b). Sin embargo, en la secuenciación del gen de la aliesterasa de C. capitata realizada en individuos de la población W no se encontraron las mutaciones que habían sido asociadas con la resistencia a organofosforados en L. cuprina, aunque se encontraron otras mutaciones que no pudieron ser asociadas con la resistencia (Magaña et al., 2008). En cuanto a los insecticidas estudiados que no tienen a la acetilcolinesterasa como molécula diana, hemos detectado resistencia cruzada en la línea W-4Km tanto a lufenurón como a lambda-cialotrina. La resistencia a estos dos insecticidas difícilmente podría ser explicada por la presencia de la mutación G328A en la AChE, sin embargo, las carboxilesterasas, que intervienen en el segundo mecanismo previamente descrito, sí podrían estar implicadas en la resistencia al piretroide y al lufenurón, ya que se sabe que estas enzimas pueden detoxificar los insecticidas por hidrólisis y/o por secuestro (Hemingway 2000, Oakeshott et al., 2005). Los piretroides contienen un enlace éster en su estructura y, en algunos casos, se ha descrito resistencia cruzada entre malatión y piretroides asociada a un incremento en la actividad esterasa (Chen y Sun 1994, Bisset et al., 1997, Heidari et al., 2005). En el caso de las benzoilureas, no poseen enlaces tipo éster y, además, el principal mecanismo de resistencia a lufenurón identificado está mediado por la sobreexpresión de genes P450 (Daborn et al., 2002, Bogwitz et al., 2005). Sin embargo, en estudios in vivo realizados en insectos se ha descrito la ruptura del enlace entre los grupos amida y carboxilo de las benzoilureas (Ivie y Wright, 1978; El Saidy et al., 1989) y, según el resultado de bioensayos con sinergistas, se sugiere que la hidrólisis de dichos grupos está mediada por esterasas (El Saidy et al., 1989; Ishaaya, 1993). En el bioensayo de susceptibilidad en campo al piretroide lambda-cialotrina, realizado en individuos procedentes de la finca de La Mayora (MAY), se observó que el nivel de resistencia en esta población no tratada fue idéntico al estimado para la línea W-4Km, siendo en ambos casos seis veces superior al de la línea C. Por el contrario, la susceptibilidad a malatión fue similar en la población MAY y en la línea susceptible C. Aunque en la finca de La Mayora no se han realizado tratamientos insecticidas dirigidos contra C. capitata, la población MAY podría estar, en parte, formada por individuos que hubiesen migrado de alguna parcela próxima a la finca donde sí se hubiesen empleado piretroides. Este dato de susceptibilidad podría sugerir la existencia en España de poblaciones de campo de C. capitata resistentes a lambda-cialotrina. Para confirmar este 62 2. Selección de resistencia en laboratorio y susceptibilidad a insecticidas en Ceratitis capitata ______________________________________________________________________________________________________________________________________ resultado sería necesario recoger poblaciones de C. capitata en diferentes regiones españolas y realizar bioensayos de susceptibilidad al piretroide lambda-cialotrina. Hasta la fecha no se han descrito poblaciones de C. capitata resistentes a espinosad, aunque sí se ha encontrado resistencia a este insecticida, aunque moderada, en otras dos especies de tefrítidos: Bactrocera oleae y B. cucurbitae (Hsu y Feng, 2006; revisado por Vontas et al., 2011; Hsu et al., 2012b). El espinosad ha sido propuesto como una buena alternativa al malatión para el control de C. capitata en cítricos, en comparación con el fosmet y la lambda-cialotrina, tanto por su efectividad como por su bajo impacto sobre la fauna auxiliar (Urbaneja et al., 2009). Además, su uso contra C. capitata se ha incrementado desde la retirada del malatión. Con anterioridad a este trabajo, no existían datos sobre la susceptibilidad de las poblaciones de campo de C. capitata a este insecticida, con la excepción de la CL50 estimada para la población resistente a malatión analizada por Magaña et al. (2007), cuya CL50 fue similar a la de la línea susceptible de laboratorio. Nuestros resultados indican que, a pesar de que la susceptibilidad a espinosad en la población de campo es elevada y de que la resistencia a malatión no parece conferir resistencia cruzada a espinosad, el desarrollo de resistencia a este insecticida es posible a medio plazo si no se emplea racionalmente frente a C. capitata. De hecho, la pequeña diferencia de susceptibilidad frente a espinosad observada inicialmente en la población recogida en Xàbia (Alicante) ha resultado, después de haber sido seleccionada durante 30 generaciones en el laboratorio, en una línea resistente cuya CL50 (157–187 ppm) se encuentra en torno a la concentración de espinosad recomendada para el tratamiento con el insecticida GF-120 frente a C. capitata (133 ppm). Los resultados sugieren que existe un mecanismo de resistencia a espinosad en las poblaciones españolas, pero que posiblemente se encuentre en estos momentos en baja frecuencia. En conclusión, la resistencia a malatión parece estar ampliamente presente en las poblaciones españolas de C. capitata. Además, por selección en laboratorio de poblaciones de campo, se han podido obtener en este trabajo líneas altamente resistentes a malatión y espinosad. Estos resultados ponen de manifiesto la necesidad de establecer métodos de detección precoz de la resistencia en poblaciones de campo para elegir el insecticida más adecuado. En los siguientes capítulos se muestran diferentes estrategias para lograr esa detección precoz de la resistencia. 63 Capítulo 3 BASES PARA LA DETECCIÓN PRECOZ DE LA RESISTENCIA: DIVERSIDAD DE GENES CYP 65 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ 3.1. Introducción. Las citocromo P450 constituyen un grupo numeroso de enzimas que se han asociado a resistencia metabólica (Feyereisen, 2005). Las P450 poseen una gran variedad de actividades enzimáticas (oxidasa, reductasa, isomerasa…), bioquímicamente se definen como monooxigenasas y son el producto de la traducción de los genes CYP (revisado por Feyereisen, 2011). Este grupo de enzimas presenta un pico de absorción a una longitud de onda de 450 nm, característica que da nombre a esta familia proteica (Omura y Sato, 1964). Debido a su función de detoxificación, por oxidación de los xenobióticos, poseen un papel importante en el metabolismo de los insecticidas. En los años sesenta se describió por primera vez que las citocromo P450 estaban implicadas en el metabolismo de xenobióticos en insectos (Agosin, 1985) y a principios de los setenta se confirmó su implicación en procesos de resistencia a insecticidas (Perry y Buckner, 1970). Actualmente, se ha demostrado que las P450 están implicadas en la resistencia frente a los principales grupos de insecticidas, tales como organofosforados, piretroides, etc. (Feyereisen, 2012). En la familia Tephritidae, hasta el momento, no se ha encontrado una relación directa entre un gen CYP concreto y la resistencia a insecticidas, ya que sólo se ha detectado la implicación de las P450 de un modo indirecto en una línea multiresistente de Bactrocera dorsalis (Hsu et al., 2004a) y en varias poblaciones de campo de B. oleae recogidas en Grecia (Margaritopoulos et al., 2008). 67 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ La secuenciación completa de los genomas de varios insectos ha permitido estudiar tanto la diversidad como la evolución de los CYPomas, cuyo número total de genes varía desde los 36 del piojo humano (Lee et al., 2010) a los 170 genes descritos en el mosquito Culex pipiens (Arensburger et al., 2010). La diversidad de genes CYP existente en una determinada especie se debe a los fenómenos de divergencia, convergencia y/o desaparición de genes que hayan tenido lugar durante el proceso evolutivo de dicha familia génica. A partir de un ancestro común se pueden originar genes parálogos a través de varios mecanismos: duplicaciones génicas en tándem, entrecruzamientos desiguales, recombinaciones no homólogas, duplicaciones cromosómicas o retroposiciones (Feyereisen, 2011). Debido a su elevado número, las citocromo P450 se dividen jerárquicamente en clados, familias y subfamilias según la homología que presentan las proteínas codificadas por los genes CYP. Concretamente en insectos, existen cuatro grandes clados: CYP-2, CYP-3, CYP-4 y CYP mitocondrial (Feyereisen, 2006). Un estudio realizado en Drosophila melanogaster demostró que aproximadamente la tercera parte de su CYPoma está constituido por genes inducibles por xenobióticos (Giraudo et al., 2010), lo que pone de manifiesto la importancia de su regulación a nivel fisiológico. Uno de los compuestos más empleados en los estudios de inducción de las citocromo P450 es el fenobarbital, cuyo efecto ha mostrado un amplio rango de variación sobre la expresión de diferentes genes CYP en insectos, dependiendo tanto del sexo del individuo como del gen analizado (Le Goff et al., 2006). En la familia Tephritidae sólo se han aislado genes CYP en los géneros Bactrocera y Ceratitis. En el género Bactrocera se ha obtenido, mediante ESTs, una secuencia parcial de un gen CYP de B. oleae (Tsoumani et al., 2011) y un fragmento de un gen CYP en B. papayae (ref. GB: AF532768). En B. dorsalis, recientemente, se han clonado y caracterizado los genes CYP6A41 y CYP6EK1 (Huang et al., 2012) y, además, se ha publicado su transcriptoma, donde se han identificado un total de 90 genes CYP (Hsu et al., 2012a). En C. capitata se conoce la secuencia parcial de 14 genes pertenecientes a las familias CYP4 y CYP6, y la secuencia completa del gen CYP6A10 (Danielson et al., 1999). El conocimiento existente sobre la diversidad de los genes CYP en los insectos facilita la descripción de su expresión funcional y de los procesos de detoxificación de insecticidas en los que podrían participar. Por ello, el objetivo de este trabajo fue, en primer lugar, ampliar el conocimiento de la diversidad de genes CYP existentes en una especie plaga como es la mosca mediterránea de la fruta y, en segundo lugar, caracterizar los niveles basales de expresión y la posible inducción por fenobarbital de algunos de los genes CYP identificados, genes seleccionados 68 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ por su homología con otros genes CYP que previamente han sido asociados a resistencia a insecticidas en otros insectos. 69 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ 3.2. Material y métodos. 3.2.1. Insectos. Tanto en los ensayos realizados para estudiar la diversidad de genes CYP, como en el estudio de la inducción de la expresión por fenobarbital, se emplearon individuos adultos de entre tres y cinco días de edad de la línea susceptible de laboratorio C de C. capitata (descrita en el apartado 2.2.2). 3.2.2. Extracción de DNA. Previamente a la extracción de DNA, los insectos fueron congelados en nitrógeno líquido y almacenados a -80ºC. El DNA se extrajo de individuos completos siguiendo el método de extracción de fenol-cloroformo. Cada individuo se introdujo en un tubo eppendorf de 1,5 ml. A dicho tubo se le añadieron 500 µl de la solución de lisis (10 mM Tris pH 8, 25mM EDTA, SDS 0,5%) y se disgregó el tejido con un bastón homogeneizador de plástico. Una vez homogeneizado 71 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ se añadieron 30 µl de proteinasa K (10 mg/ml) y tras una ligera agitación manual de 5 s, se mantuvo a 37ºC durante toda la noche. Tras la incubación, al tubo de 1,5 ml se le añadieron 600 µl de fenol y se mezcló manualmente por volteo varias veces antes de centrifugarlo durante 5 min a 5.800 xg en una centrífuga Eppendorf 5424 (Eppendorf, Hamburgo). La fase superior se pasó a un nuevo tubo que contenía 600 µl de una solución de fenol:cloroformo (1:1); después de mezclarlo manualmente por volteo durante 20 o 30 s, tras una nueva centrifugación durante 5 min a 5.600 xg, se retiró la fase superior a un nuevo tubo eppendorf que contenía 550 µl de cloroformo. Después de mezclarlo y centrifugarlo 4 min a 5.800 xg, la fase superior se llevó a un nuevo tubo eppendorf de 1,5 ml al que se le añadió 1 volumen de isopropanol y se homogeneizó de nuevo por volteo. El tubo se centrifugó durante 5 min a 7.200 xg para sedimentar el DNA y se eliminó el isopropanol por decantación. Se añadió al tubo una disolución de lavado de etanol al 70% que se retiró después teniendo especial cuidado de no verter el precipitado de DNA o pellet. Posteriormente, se realizó un secado del pellet dejando el tubo abierto en la campana de flujo laminar Microflow H 1.20 (T.D.I., Alcobendas, Madrid) durante 10 min. Para resuspender el pellet de DNA se añadieron 100 µl de TE 0,1X y 2 µl de RNasa (1mg/ml). La concentración de DNA se midió con un NanoDrop (Thermo Scientific, Massachusetts, EE.UU.). El DNA se mantuvo almacenado a -20ºC hasta su utilización. 3.2.3. Extracción de RNAT y RNAm. La extracción de RNA total (RNAT) se realizó a partir de individuos completos, previamente conservados a -80ºC, utilizando TRIzol® Reagent (Life Technologies, Van Allen Way, Carlsbad, California, EE.UU.) y siguiendo las instrucciones del fabricante. La concentración del RNAT se midió con un NanoDrop (Thermo Scientific, Massachusetts, EE.UU.). A partir del RNAT se extrajo el RNA mensajero (RNAm) con el mRNA Purification Kit (Amersham Biosciences, Friburgo, Alemania). Así, haciendo pasar el RNAT a través de unas columnas de celulosa que contenían oligonucleótidos poli T, se separó el RNAm del resto de RNA. Las extracciones de RNAm se almacenaron a -80ºC para su uso posterior. 72 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ 3.2.4. Síntesis de cDNA. La obtención de DNA complementario (cDNA) a partir del RNAm se realizó mediante el MarathonTM cDNA Amplification Kit (BD Biosciences-Clontech, California, EE.UU.) con el fin de elaborar una librería de cDNA. El proceso de síntesis tuvo lugar en dos fases: en la primera se sintetizaron todas las cadenas simples de cDNA a partir del RNAm del transcriptoma y, en la segunda fase, se realizó la síntesis de la cadena complementaria. Así, en la primera fase se sintetizó la cadena simple de cDNA con la enzima transcriptasa inversa AMV RT, siguiendo las instrucciones del fabricante, partiendo de entre 1,5 y 2 µg de RNAT y utilizando 1,7 µg del cebador reverso poli-T Marathon cDNA synthesis primer (Tabla 3.2.1). En la segunda fase de la síntesis, para obtener cDNA de doble cadena mediante la DNA polimerasa T4 (Promega Co, Wisconsin, EE.UU.), se empleó como molde el cDNA de cadena simple sintetizado anteriormente. Finalmente, se realizó la ligación de unos adaptadores en los extremos de las cadenas de cDNA para facilitar la amplificación de los genes CYP por PCR RACE. Tabla 3.2.1. Cebadores empleados en el estudio de diversidad de genes CYP. Se indica los cebadores ordenados alfabéticamente por su nombre. La secuencia nucleotídica se muestra en sentido 5’ 3’ indicando el número de bases del oligonucleótido (b). El código de nomenclatura de los nucleótidos sigue los criterios de la IUPAC (Cornish-Bowden, 1985), la letra I indica inosina. (1) Cebador obtenido del estudio de Scott et al., 1994. Nombre Secuencia (5’ 3’) b dFCyp4a ACITTYATGTTYGARGGIYAYGAYACNAC 29 dFCyp6a CARGCITTYITITTYTWYIYIGCYGGNTT 29 dFCyp9a GCYCAGTGYYTIITNTTYTTYTTYGCNG 28 dFCyp12a MTGGAYWTSCTMWTIGCIGGWGYGGA 26 dFCyp18a GAYYTITTYWSIGCNGGIATGGARAC 29 dFCyp28a ACMTTYMTIYTIGAYGGIYTIGAIACNAC 29 dRHemo4 CCIATRCARTTICKIKGICCIGCISWRAAIGG 32 dRHemo6_9 ATGCARYTICKIKGICCIDCICCRAANGG 29 dRHemo12_18 CACATICKISKICCIAMICCRAANGG 26 CTGICCGATRCAGTTICGBGGICCIGCSIWGAABGG 36 M13F GTAAAACGACGGCCAGT 17 M13R GGAAACAGCTATGACCATG 19 Poli-T TTCTAGAATTCAGCGGCCGC(T)30N-1N 51 dRScott94 (1) ___________________________________________________________________________________________________________________________________ 73 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ 3.2.5. Amplificación por PCR de los genes CYP. Con el objetivo de obtener el mayor número de genes CYP se realizó la amplificación por PCR empleando una mezcla de cebadores, de aquí en adelante llamada cebadores degenerados, tanto a partir del DNA complementario (cDNA) como del DNA genómico. Los cebadores degenerados son una mezcla de cebadores similares entre sí que contienen mínimas variaciones en su secuencia para lograr amplificar varias secuencias nucleotídicas que codifican un mismo aminoácido. Como referencia para el diseño de los cebadores degenerados se emplearon las secuencias de aminoácidos de las familias CYP4, CYP6, CYP9, CYP12, CYP18 y CYP28 de D. melanogaster (Tabla 3.2.1), seis de ellos en sentido directo (cebadores dFCyp) a partir de la región hélice I, y cuatro cebadores en sentido reverso (cebadores dRHemo) basados en el grupo hemo. El tamaño esperado para el producto de PCR tras la amplificación, tomando cDNA como molde, tendría un tamaño aproximado de entre 400 y 500 pb (Fig. 3.2.1). En el caso de las amplificaciones realizadas sobre DNA genómico, el tamaño del amplicón aumentaría en función del tamaño del intrón existente en la región amplificada. Se ensayaron todas las posibles combinaciones entre los cebadores degenerados directos y los reversos con el objetivo de amplificar la mayor diversidad de genes CYP. La reacción de PCR se realizó en un termociclador Gene Amp® PCR System 2700 (Applied Biosystem, Foster City, EE.UU.) empleando la enzima Amplitaq Gold® (Roche Molecular Systems, Inc., New Jersey, EE.UU.) y con las siguientes condiciones de amplificación: inicialmente un periodo de desnaturalización a 95ºC durante 5 min, seguido de 35 ciclos de 94ºC 45 s, 46-52ºC 45 s, 72ºC 45 s y finalizando con un periodo de 72ºC durante 7 minutos. P450s (1700 pb) 5’-UTR Hélice I G. Hemo 3’-UTR 400 - 500 pb dFCyp dRHemo Figura 3.2.1. Estructura de un cDNA que codifica una P450. Se muestra la localización de las regiones conservadas hélice I y grupo hemo en el cDNA de un gen CYP. Situación de los cebadores en sentido directo (dFCyp) y reverso (dRHemo) empleados en la amplificación por PCR de DNA genómico y cDNA de C. capitata. 74 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ 3.2.6. Electroforesis y extracción de bandas. Los productos de PCR fueron migrados electroforéticamente para su separación por tamaño en un gel de agarosa D2 (Lab. Conda, Torrejón de Ardoz, Madrid, España) al 1,5% en TAE 1X modificado (Tris 10mM, EDTA 0,1mM pH 8.0), que contenía además 2 µl de bromuro de etidio (1 mg/ml) por cada 100 ml de gel. El marcador molecular referencia de 100 pb en escalera fue O’GeneRulerTM 100 bp Plus (Fermentas, Thermo Scientific, Massachusetts, EE.UU.). El DNA se migró en una cubeta electroforética Bio-Rad® Sub Cell (Bio-Rad Laboratories, California, EE.UU.). Los productos de PCR se visualizaron en un transiluminador UV/digitalizador GelDoc Universal Hood II (Bio-Rad Laboratories, Milán, Italia) y las bandas correspondientes en tamaño a los amplicones esperados se extrajeron del gel de agarosa y fueron purificadas usando las columnas Ultrafree-DA (Millipore Corporation, Massachusetts, EE.UU.) siguiendo las instrucciones del fabricante. Cada uno de los productos resultantes de la purificación de las bandas se almacenó a 20ºC para su posterior clonación. 3.2.7. Clonación, RFLP y secuenciación. Los productos de PCR purificados se clonaron empleando el sistema pGEM-T Easy Vector (Promega Corporation, Wisconsin, EE.UU.) siguiendo las instrucciones del fabricante. Las colonias positivas a la clonación, es decir, aquellas que podían contener insertos en el plásmido, fueron seleccionadas en base al color claro de las mismas, y fueron analizadas mediante PCR con los cebadores M13F y M13R (Tabla 3.2.1). El producto de PCR resultante se migró en un gel de agarosa al 1,5% para determinar el tamaño del inserto clonado (Fig. 3.2.2). a b Figura 3.2.2. Clonación y comprobación del inserto. (a) Placa Petri con colonias resultado de la clonación de uno de los productos de PCR obtenidos a partir de una determinada combinación de cebadores degenerados. Cada colonia positiva a la clonación fue analizada por PCR para confirmar la presencia de inserto y el tamaño del mismo (b). Los amplicones con un tamaño de 600-800 pb (ejemplo, en azul) fueron seleccionados para su posterior análisis por RFLP. 75 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ Los productos de PCR de aquellas colonias que contenían un inserto de 400-600 pb fueron analizados mediante RFLP (del inglés Restriction Fragment Length Polymorphism) con las enzimas de restricción Rsa I y Hpa II (Roche Applied Science, Penzberg, Alemania). La digestión se realizó en un volumen total de 25 µl en el buffer L, a 37ºC durante 2 horas, con una unidad de cada una de las enzimas. Finalmente, el producto de PCR digerido se migró electroforéticamente en un gel de agarosa D2 (Laboratorios Conda S.A., Torrejón de Ardoz, Madrid) al 2% en TAE 1X (Tris 40mM, EDTA 1mM pH 8.0). Los clones que presentaron distintos perfiles de restricción fueron enviados al servicio de secuenciación Secugen S.L. (Madrid) y secuenciados en un secuenciador automático ABI PRISM 377 (Applied Biosystems, California, EE.UU.). 3.2.8. Análisis de las secuencias. Los cromatogramas que contenían las secuencias nucleotídicas de los genes CYP de C. capitata fueron revisados mediante el programa Chromas V1.45 (Griffith University, Queensland, Australia), realizándose las correcciones manuales apropiadas. Una vez corregidas las secuencias nucleotídicas se realizó la identificación de los intrones presentes en el DNA genómico mediante la comparación de dichas secuencias con el genoma de D. melanogaster en un servidor web (http://www.fruitfly.org/seq_tools/splice.html). Después de eliminar las regiones intrónicas se realizó la traducción tanto de estas secuencias como de las obtenidas a partir del cDNA. Con el objeto de definir, por homología, la familia y subfamilia de cada uno de los genes obtenidos, se compararon las secuencias aminoacídicas obtenidas con las de D. melanogaster existentes en el servidor The Insect P450 Site del Institut Nationale de la Recherche Agronomique (INRA) (http://p450.sophia.inra.fr/blast/blast.html). Finalmente, se elaboró un árbol filogenético Neighbor Joining con el programa ClustalX V.1.85 (Thompson et al., 1997), donde se mostraros las relaciones existentes entre los genes CYP de D. melanogaster y los genes descritos para C. capitata en el presente trabajo. En análisis filogenético se realizaron 1000 réplicas de boostrap, indicando en los nodos del árbol sólo aquellos valores iguales o superiores a 500. 76 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ 3.2.9. Bioensayos de exposición a fenobarbital. Con el objetivo de estudiar la posible inducción de los genes CYP por xenobióticos se expusieron adultos de C. capitata a un agente inductor de la expresión. El inductor seleccionado fue el xenobiótico fenobarbital (Sigma P-5178, Chemical Co., Missouri, EE.UU.). La estructura química molecular de este xenobiótico se muestra en la figura 3.2.3. Figura 3.2.3. Estructura química del fenobarbital. Las condiciones del bioensayo de inducción con fenobarbital son similares a las condiciones generales de los bioensayos mostradas en el apartado 2.2.4.1. Se introdujeron 20 insectos en cada caja y se administró, mezclándolo con la dieta, fenobarbital al 1% y al 3%, realizando 4 réplicas experimentales y tomando como referencia un control al que no se le había aplicado fenobarbital. Después de 72 horas se anotó la mortalidad y se corrigió por Abbot con respecto a la mortalidad observada en el tratamiento control, que siempre fue inferior al 10%. Los individuos supervivientes fueron congelados en nitrógeno líquido y almacenados a -80ºC para su posterior análisis. 3.2.10. PCR cuantitativa en tiempo real. Para el estudio de inducción por fenobarbital se seleccionaron aquellos genes CYP de C. capitata con mayor homología a genes CYP que habían sido previamente relacionados con resistencia a insecticidas en otros dípteros. Se analizó la expresión en machos y hembras adultos de tres a cinco días de edad mediante PCR cuantitativa en tiempo real (qPCR). El gen empleado como control interno fue el gen que codifica la proteína ribosomal Ccp0 (ref. GenBank Y18444; Gagou et al., 2000). El RNAT se extrajo a partir de los insectos almacenados a -80ºC utilizando TRIzol® Reagent y siguiendo las instrucciones del fabricante. La extracción de RNAT se realizó en grupos de cinco insectos machos y cinco hembras. Se observó el efecto de los diferentes tratamientos con fenobarbital analizando la expresión de dichos genes en grupos de cinco insectos 77 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ para cada sexo y tratamiento, incluido el control sin fenobarbital. La primera cadena de cDNA se sintetizó a partir de 1 µg de RNAT utilizando iScript cDNA Syntesis Kit (Bio-Rad Laboratories, California, EE.UU.). La reacción de amplificación en tiempo real para cada gen CYP y para el gen control interno CCp0 se realizó en un detector continuo de fluorescencia DNA Engine Opticon2 (Bio-Rad Laboratories, California, EE.UU.). Se ensayaron varias parejas de cebadores para cada uno de los genes estudiados, seleccionando como óptimas aquellas parejas que tuviesen una eficiencia de amplificación entre el 80% y el 120% (ver tabla 3.3.3.). Además, para descartar la presencia de DNA genómico, el cual podría interferir en la cantidad de cDNA analizado en la qPCR, algunas de las parejas de cebadores se diseñaron de tal modo que flanqueasen una región intrónica, por lo que si existiese DNA genómico contaminando la extracción de RNA, observaríamos un amplicón doble en la qPCR. La reacción de PCR (15 µl) contenía 7,5 µl de qPCR MasterMix Plus for SYBR® Green (Eurogentec, Seraing, Bélgica), 1,25 µl de cada cebador 3,6 µM y 5 µl de cDNA diluido 100 veces en agua. El ciclo de amplificación estandarizado para todos los genes fue el siguiente: inicialmente 95ºC durante 10 min para desnaturalizar el cDNA y a continuación se realizó cuarenta veces el ciclo 95ºC 20s, 63ºC 20s y 72ºC 20s. La expresión de cada uno de los genes analizados se calculó siguiendo el método de ∆Ct (Pfaffl, 2001). Los resultados de expresión de los genes CYP se normalizaron con el valor de la expresión del gen Ccp0. El valor final de expresión para cada gen se calculó realizando la media aritmética de las expresiones obtenidas en cada una de las tres réplicas experimentales. La comparación estadística de los valores de expresión se realizó mediante la prueba t de Student o test de Dunnett, según corresponda, con el paquete estadístico SPSS v. 17.0, según corresponda. 78 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ 3.3. Resultados. 3.3.1. Diversidad de genes CYP. Empleando cebadores degenerados, diseñados a partir de las secuencias aminoacídicas de las familias CYP4, CYP6, CYP9, CYP12, CYP18 y CYP28 de D. melanogaster, y partiendo de cDNA y DNA genómico de C. capitata, se obtuvieron por PCR amplicones susceptibles de contener genes CYP de aproximadamente 400-600 pb (Fig. 3.3.1). 1 2 3 4 5 6 7 M 8 9 10 11 12 13 1 2 3 4 5 6 7 8 9 10 11 12 M 13 14 15 16 17 18 19 20 21 22 23 24 a b Figura 3.3.1. Amplificación por PCR de genes CYP. Migración electroforética en gel de agarosa al 1,5% de los productos de PCR resultado de la amplificación con cebadores degenerados de DNA genómico (a) y cDNA (b). Los amplicones obtenidos poseen un tamaño alrededor de los 500 pb, según el marcador de 100 pb empleado (M). En el gel (a) se muestran las combinaciones de cebadores que produjeron amplicones con DNA genómico como molde (calles 1 a 13). En el gel (b) se indica la totalidad de las 24 combinaciones, obsérvese que no todas las combinaciones de cebadores degenerados produjeron amplificación (calles 2 a 5). 79 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ De las 24 combinaciones de cebadores, 17 dieron lugar a amplicones del tamaño esperado y, de éstas, 12 produjeron la amplificación de genes CYP, por lo que la mitad de las combinaciones de cebadores degenerados empleadas amplificaron genes CYP (Tabla 3.3.1). La mayoría de las amplificaciones que contenían fragmentos de genes CYP (63%) fueron el resultado de las seis combinaciones correspondientes a los cebadores directos dFCyp4a y dFCyp6a con los reversos dRSCott94, dRHemo4a y dRHemo6_9a. En el caso del cebador reverso dRHemo12_18, aunque se obtuvieron por PCR fragmentos del tamaño esperado, no se obtuvieron amplicones de genes CYP en ninguna de sus combinaciones con los cebadores directos. Tabla 3.3.1. Combinaciones de cebadores degenerados para P450s. Se muestran las combinaciones de cebadores degenerados empleados en las PCR que, por su tamaño, dieron lugar a amplicones susceptibles de contener genes CYP (b b); y las combinaciones de cebadores con las que no se obtuvo producto de amplificación (-). Entre paréntesis se indica el porcentaje de clones con insertos de genes CYP obtenidos con cada combinación de cebadores. dFCyp4a dFCyp6a dFCyp9a dFCyp12a dFCyp18a dFCyp28a dRScott94 b(19%) b(10%) - - - - dRHemo4a b(11%) b(13%) - b(7%) b(16%) b(5%) dRHemo6_9 b (6%) b (4%) b(2%) b(3%) - b(4%) dRHemo12_18 b (0%) b (0%) b(0%) - b (0%) b(0%) __________________________________________________________________________________________________ Los productos de PCR con el tamaño esperado (400-600 pb) se clonaron y, del total de clones obtenido, se seleccionaron alrededor de 2.000 clones como susceptibles de poseer insertos de genes CYP según el tamaño del inserto que contenía cada clon (Fig. 3.2.2). Con el objetivo de evitar la secuenciación de clones con el mismo inserto, los 2000 clones se analizaron por RFLP con las enzimas Rsa I y Hpa II (Fig. 3.3.2) y se seleccionaron para su secuenciación un total de 500 clones, pertenecientes a doce combinaciones de cebadores degenerados. Aproximadamente el 70% de los clones secuenciados procedían de la amplificación de cDNA y el 30% restante se obtuvieron a partir de DNA genómico. 80 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ A B C D A E D C F D G H D A I E 500 pb Figura 3.3.2. Análisis RFLP de los clones. Migración electroforética, en un gel de agarosa al 2%, de la digestión con las enzimas de restricción Rsa I y Hpa II. Se muestra un ejemplo representativo del análisis realizado con los clones procedentes de la clonación de un determinado amplicón, originado a partir de una combinación de cebadores degenerados. Se realizó la secuenciación de aquellos clones con perfiles de restricción diferentes (letras diferentes). Como ejemplo, se resaltan tres clones con el perfil A, de los que sólo se secuenció uno de ellos. Las secuencias procedentes de la amplificación del DNA genómico contuvieron pequeños intrones con un tamaño que varió entre 58-80 pb. El tamaño de las secuencias nucleotídicas obtenidas, una vez eliminados los intrones, osciló entre 370 y 480 pb, codificando fragmentos proteicos de 123 a 160 aminoácidos. El alineamiento de las secuencias proteicas permitió identificar aquellas secuencias de nucleótidos que codificaban la misma proteína y descartarlas así para el estudio de diversidad de genes CYP en C. capitata. En la figura 3.3.3 se muestra, a modo de ejemplo, el alineamiento de 11 secuencias aminoacídicas, de las 500 analizadas, en el que se pueden observar los motivos ExLR y PERF, característicos de las citocromo P450. Además, a la derecha del motivo ExLR se observa una región muy variable que se corresponde con una zona de la enzima que está asociada con el reconocimiento del sustrato (Feyereisen, 2005). ExLR PERF 1 2 3 4 5 6 7 8 9 10 11 Figura 3.3.3. Alineamiento de las secuencias aminoacídicas obtenidas a partir de los genes CYP de C. capitata. Se muestra el alineamiento de 11 secuencias proteicas en el que se observan los motivos ExLR y PERF, característicos de las citocromo P450. Los haplotipos 1 a 4 pertenecen a la familia CYP4, del 5 al 9 a la CYP6 y el 10 y 11 a la CYP9. 81 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ Una vez realizado el análisis de las 500 secuencias aminoacídicas se obtuvieron un total de 72 haplotipos diferentes de genes CYP. Aquellas secuencias proteicas con un porcentaje de similitud igual o superior al 95% se consideraron como variantes alélicas de un mismo gen. Según este criterio, se identificaron un total de 35 posibles variantes alélicas, por lo que los restantes haplotipos se consideraron posibles genes CYP. A cada uno de los 37 genes se le puso un nombre según las normas de nomenclatura vigentes (Nelson et al., 1996) y, tomando como referencia el CYPoma de D. melanogaster, se le asignó una familia y, si se pudiese, una subfamilia y/o un gen. Los 37 genes se distribuyeron en cinco familias: doce se incluyeron en la familia CYP4, diecisiete en la familia CYP6, tres en la familia CYP9, cuatro en la familia CYP12 y un gen en la familia CYP18 (Fig. 3.3.4 y tabla 3.3.2). 7 1 1 22 6 4 Familias P450 CYP4 CYP6 CYP9 CYP12 CYP18 12 3 17 25 D. melanogaster C. capitata Figura 3.3.4. Distribución de los genes CYP en las cinco familias estudiadas. Se indica el número de genes CYP existentes en D. melanogaster y el número de genes encontrados en este trabajo en C. capitata en cinco de las principales familias CYP de insectos. Las relaciones entre los distintos genes CYP de C. capitata y D. melanogaster se muestran en un árbol filogenético Neighbor Joining generado a partir de las secuencias de aminoácidos (Fig. 3.3.5). Así, los 37 genes CYP identificados en C. capitata se dispusieron en los diferentes clados de la siguiente manera: un gen en el clado CYP-2, veinte genes en el clado CYP-3, doce genes en el clado CYP-4 y cuatro genes en el clado mitocondrial (nomenclatura de los clados según Feyereisen, 2006). 82 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP 02P2O 03QP7 02P2K 031OK 02T2A 021DJ 02P2C 02P24 03QP2 02P2D 031OJ 031NP 03YM8 02P28 03QOD 03QPU 021D3 02P2B 031OL 031P6 03YOD CYP4 (I) like CYP4 (II) like v1 v1 CYP4AC like CYP4AE like v1 v2 CYP4C3 like v1 v2 CYP4D (I) like v1 v1 CYP4D (II) like v1 v2 CYP4D (III) like v1 v2 CYP4G like v1 v2 CYP4G1 like v1 v2 CYP4G15 like CYP4P like CYP6 like JQ045589 JQ031144 JQ031143 JQ031141 JQ031142 JQ031139 JQ031140 JQ031138 JQ067922 JQ031137 JQ067921 JQ031136 JQ067920 JQ067918 JQ067919 JQ067916 JQ067917 JQ031135 JQ031134 JQ067915 JQ031133 Ref. GB 3 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 Clado CYP cDNA (379) cDNA (376) cDNA (379) cDNA (382) cDNA (382) cDNA (382) cDNA (382) cDNA (377) DNA (377) cDNA (385) DNA (385) cDNA (379) DNA (379) DNA (376) DNA (376) DNA (373) DNA (373) cDNA (364) cDNA (370) DNA (370) DNA (373) Origen (pb) 69 71 67 74 74 59 59 80 69 Intrón (pb) Cyp6a14 (Q9V4U7) Cyp4p1 (AAL49206) Cyp4g15 (AAF76522) Cyp4g1 (AAL13523) Cyp4g1 (AAL13523) Cyp4d2 (CAA80549) Cyp4d2 (CAA80549) Cyp4d1 (AAB71162) Cyp4c3 (Q9VA27) Cyp4ae1 (AAL13679) Cyp4ac1 (AAF52232) Cyp4e1 (NP_524771) Cyp4d2 (CAA80549) Gen CYP Dm 48 56 88 87 86 69 69 56 55 64 64 70 70 85 84 54 54 65 48 48 51 % Similitud 83 _________________________________________________________________________________________________________________________________________________________________________________________________________________________________________________ Haplotipo Gen CYP Cc por PCR procede de DNA genómico o cDNA, con el tamaño del amplicón en pares de bases (pb), entre paréntesis. Se indica el tamaño del intrón detectado en el caso de las amplificaciones de DNA genómico. Las secuencias aminoacídicas de Cc obtenidas en este trabajo se compararon con las de D. melanogaster (Dm); se muestra el nombre del gen y la referencia en Genbank de la secuencia más próxima de esta especie, así como el porcentaje de similitud entre ambas. Entre paréntesis se refleja el porcentaje de similitud con respecto a la secuencia de CYP6A10 de C. capitata de aquellos genes más próximos a este gen. Tabla 3.3.2. Diversidad de genes CYP en C. capitata. Se muestran, ordenados alfabéticamente, los genes CYP y las variantes alélicas detectadas en C. capitata (Cc), indicando su referencia en Genbank y el clado CYP al que pertenecen. La columna origen muestra si el gen amplificado _____________________________________________________________________________________________________________________________________________________________________________________________________________ 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP 02T1W 03QPH 031OT 021DS 02T11 02T21 03QPE 02P2Z 02P3F 02T12 031OY 02P3I 03QOT 02P33 03QOM 021DB F12.03VM7 03YLX 03YM1 03YMC D2.03VM7 02T22 03QPJ 02P3C 02T0N 02P3H CYP6A (I) like CYP6A (II) like v1 v2 v2 CYP6A (III) like v1 v2 CYP6A (IV) like v1 v1 CYP6A (V) like v1 v2 CYP6A (VI) like v1 v1 v2 v3 CYP6A (VII) like v1 v2 CYP6A (VIII) like v1 v2 CYP6A (IX) like v1 v2 CYP6A (X) like v1 v2 CYP6A (XI) like v1 v2 v3 v4 JQ045601 JQ067934 JQ067935 JQ067936 JQ045600 JQ067933 JQ045598 JQ045599 JQ045596 JQ045597 JQ045595 JQ067932 JQ045593 JQ067930 JQ045594 JQ067931 JQ067928 JQ067929 JQ045592 JQ067927 JQ067925 JQ067926 JQ045590 JQ045591 JQ067924 JQ067923 Ref. GB 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 Clado CYP cDNA (391) DNA (391) DNA (391) DNA (391) cDNA (390) DNA (391) cDNA (392) cDNA (394) cDNA (388) cDNA (382) cDNA (388) DNA (388) cDNA (382) DNA (388) cDNA (392) DNA (386) DNA (388) DNA (388) cDNA (388) DNA (388) DNA (391) DNA (391) cDNA (391) cDNA (391) DNA (391) DNA (391) Origen (pb) 72 72 72 61 78 67 67 58 58 66 59 61 58 59 Intrón (pb) Cyp6a2 (AAB36782) Cyp6a8 (Q27593) Cyp6a23 (Q9V771) Cyp6a23 (Q9V771) Cyp6a23 (Q9V771) Cyp6a14 (Q9V4U7) Cyp6a14 (Q9V4U7) Cyp6a14 (Q9V4U7) Cyp6a13 (AAK93147) Cyp6a13 (AAK93147) Cyp6a2 (AAB36782) Cyp6a2 (AAB36782) Cyp6a9 (Q27594) Cyp6a2 (AAB36782) Gen CYP Dm 53 54 54 53 53 56 59 59 58 56 60 58 56 56 55 55 51 51 59 59 53 53 63 65 65 58 % Similitud 84 _________________________________________________________________________________________________________________________________________________________________________________________________________________________________________________ Haplotipo Gen CYP Cc Tabla 3.3.2. Diversidad de genes CYP en C. capitata. (Continuación). _____________________________________________________________________________________________________________________________________________________________________________________________________________ 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP 03QOQ 03QPC 03QPD 03YOA 03QOE 021DD 03QPO 02T16 02T1N 031NF 02T1E 03YO7 03QOS 031P1 031OZ 03QOP 03YMF 03QOU 03QOV 03QOW 02T27 G3.03VMG A5.03VMG 00H90 CYP6A10 like v2 v3 v4 CYP6D (I) like CYP6D (II) like v1 v1 v2 v3 v4 v5 v6 CYP6D (III) like CYP6G like v1 v2 v3 CYP9 like CYP9B (I) like CYP9B (II) like v1 v2 CYP12B like v1 v1 CYP12C (I) like CYP12C (II) like CYP12E like JQ045619 JQ045618 JQ045617 JQ045616 JQ067942 JQ045614 JQ045615 JQ045613 JQ045612 JQ045609 JQ045610 JQ045611 JQ045608 JQ045606 JQ067937 JQ045607 JQ067938 JQ067939 JQ067940 JQ067941 JQ045605 JQ045602 JQ045603 JQ045604 Ref. GB Mitoc. Mitoc. Mitoc. Mitoc. Mitoc. 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 Clado CYP cDNA (392) cDNA (412) cDNA (411) cDNA (397) DNA (480) cDNA (408) cDNA (407) cDNA (407) cDNA (403) cDNA (413) cDNA (403) cDNA (403) cDNA (370) cDNA (373) DNA (373) cDNA (376) DNA (373) DNA (379) DNA (373) DNA (376) cDNA (388) cDNA (391) cDNA (388) cDNA (388) Origen (pb) 61 63 63 63 63 63 Intrón (pb) Cyp12e1 (NP_650003) Cyp12c1 (Q9VVR9) Cyp12c1 (Q9VVR9) Cyp12b2 (Q9V8M2) Cyp9b2 (Q9V4I1) Cyp9b2 (Q9V4I1) Cyp9b2 (Q9V4I1) Cyp6g2 (AAR88134) Cyp6d5 (NP_650327) Cyp6d4 (NP_651082) Cyp6d2 (AAQ22467) Cyp6a9 (Q27594) Gen CYP Dm 54 52 56 56 57 60 60 60 52 51 51 51 64 62 62 62 62 62 61 61 59 71 (97) 70 (96) 69 (95) % Similitud 85 CYP18A1 like 03YPC JQ045620 2 cDNA (376) Cyp18a1 (CAL69940) 88 ___________________________________________________________________________________________________________________________ Haplotipo Gen CYP Cc Tabla 3.3.2. Diversidad de genes CYP en C. capitata. (Continuación). _____________________________________________________________________________________________________________________________________________________________________________________________________________ 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ CYP- 4 CYP Mitoc. CYP- 2 CYP- 3 Figura 3.3.5. Disposición de los genes CYP de C. capitata. Representación esquemática del árbol filogenético Neighbor Joining de las secuencias aminoacídicas de los genes CYP de C. capitata, representados con trazos negros, y de D. melanogaster, en colores. Los genes CYP de C. capitata se agruparon en los cuatro clados existentes en insectos: CYP-4 (rojo), CYPmitocondrial (amarillo), CYP-2 (verde) y CYP-3 (azul). 86 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ En el clado CYP-4 (Fig. 3.3.6) se identificaron doce genes CYP de C. capitata, de los cuales tres se agruparon significativamente con Cyp4c3, Cyp4g1 y Cyp4g15 de D. melanogaster. Además, cuatro genes se incluyeron dentro de las subfamilias CYP4AC, CYP4AD, CYP4G y CYP4P, mientras que los genes CYP4 (I) like, CYP4 (II) like, CYP4AE like, CYP4D (I) like y CYP4D (III) like no se relacionaron significativamente con ninguna de las subfamilias conocidas de D. melanogaster. 0,01 Figura 3.3.6. Clado CYP-4. Árbol Neighbor Joining correspondiente al clado CYP-4 para los genes CYP de D. melanogaster (Dm) y C. capitata (Cc). En el análisis filogenético se empleó, para cada gen, un fragmento de 120-130 aminoácidos comprendido entre la región hélice Ι y el grupo hemo. Los valores de los nodos indican el valor de bootstrap para un total de 1000 réplicas. 87 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ En el clado mitocondrial (Fig. 3.3.7), según su homología con las secuencias aminoacídicas de D. melanogaster, se identificaron cuatro genes CYP de C. capitata: uno en la subfamilia CYP12B (CYP12B like) y tres genes que no se agruparon significativamente con subfamilias de D. melanogaster (CYP12C (I) like, CYP12C (II) like y CYP12E like). 0,01 Figura 3.3.7. Clado Mitocondrial. Árbol Neighbor Joining correspondiente al clado mitocondrial para los genes CYP de D. melanogaster (Dm) y C. capitata (Cc). En el análisis filogenético se empleó, para cada gen, un fragmento de 120-130 aminoácidos comprendido entre la región hélice Ι y el grupo hemo. Los valores de los nodos indican el valor de bootstrap para un total de 1000 réplicas realizadas. En el clado CYP-3 se identificaron un total de veinte genes de C. capitata pertenecientes a las familias CYP6 y CYP9 (Fig. 3.3.8). La distancia entre los genes de C. capitata y los de D. melanogaster en este clado fue, en términos generales, mayor que en los otros clados. Dentro de la familia CYP6, trece de los genes identificados se agruparon en la subfamilia CYP6A de D. melanogaster, entre los que se encuentran CYP6A10, descrito anteriormente en C. capitata (Danielson et al., 1999), y tres posibles variantes alélicas de CYP6A10 identificadas en este trabajo. Además, tres genes de C. capitata (CYP6D (I) like, CYP6D (II) like, CYP6D (III) like) se agruparon con genes de la subfamilia CYP6D. El gen CYP6G like fue el único que no se agrupó con genes de alguna de las subfamilias del clado CYP-3 de D. melanogaster. Finalmente, dentro de la familia CYP9 se identificaron tres genes próximos a la subfamilia CYP9B (CYP9B (I)-(II) like) y, CYP9 like. 88 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ Figura 3.3.8. Clado CYP-3. Árbol Neighbor Joining del clado CYP-3 para los genes CYP de D. melanogaster (Dm) y C. capitata (Cc). En el análisis filogenético se empleó, para cada gen, un fragmento de 120-130 aminoácidos comprendido entre la región hélice Ι y el grupo hemo. Los valores de los nodos indican el valor de bootstrap para un total de 1000 réplicas. 89 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ En el clado CYP-2 (Fig. 3.3.9) se identificó un único gen (CYP18A1 like) que se agrupó significativamente con el gen Cyp18a1 de D. melanogaster. 0,01 Figura 3.3.9. Clado CYP-2. Árbol Neighbor joining correspondiente al clado CYP-2 para los genes CYP de D. melanogaster (Dm) y C. capitata (Cc). En el análisis filogenético se empleó, para cada gen, un fragmento de 120-130 aminoácidos comprendido entre la región hélice Ι y el grupo hemo. Los valores de los nodos indican el valor de bootstrap para un total de 1000 réplicas. 3.3.2. Expresión de los genes CYP. Los niveles de expresión de 6 de los 37 genes CYP identificados en este trabajo se analizaron mediante qPCR en machos y hembras de C. capitata. Los genes se seleccionaron por su homología con otros genes CYP relacionados con resistencia a insecticidas en el orden Diptera. Los genes CYP6D (I) like y CYP6D (III) like fueron analizados por pertenecer a la subfamilia CYP6D asociada a resistencia a piretroides en Musca domestica (Liu y Scott, 1996; Kasai y Scott, 2000, 2001). El gen CYP6A (I) like fue seleccionado por su homología con CYP6A1 de M. domestica, relacionado con resistencia a organofosforados y carbamatos (Feyereisen et al., 1989; Sabourault et al., 2001), y con Cyp6a2 de D. melanogaster, implicado en resistencia a DDT y malatión (Waters et al., 1992; Maitra et al., 2000). CYP6G like se analizó por ser similar a Cyp6g1, implicado en resistencia a DDT en D. melanogaster (Daborn et al., 2001, 2002) y, por último, CYP12C (II) like por ser el más próximo a Cyp12d1/2, también asociado a resistencia a DDT en D. melanogaster (Le Goff et al., 2003). Además, aunque no ha sido relacionado con resistencia, se evaluó la expresión génica de CYP4G1 like. 90 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ R2 = 0,997 b dF/dT 10 5 0 a 60 65 70 75 80 85 90 95 deg. c Figura 3.3.10. Validación de los cebadores para PCR cuantitativa. Como ejemplo, se muestran los parámetros que definieron la aptitud de la pareja de cebadores para el gen Ccp0., empleado como control interno en la qPCR. (a) Curva de fusión (melting curve) a 78ºC para el gen Ccp0, representando la temperatura a la cual se produce la aparición del amplicón, independientemente de la concentración de cDNA molde. (b) Recta patrón que define las condiciones de linealidad de la amplificación a diferentes concentraciones de molde. (c) Curva de cuantificación del fluoróforo a diferentes concentraciones de molde. En la figura 3.3.10 se muestran, tomando como ejemplo el gen Ccp0, utilizado como control interno, los elementos evaluados para seleccionar los cebadores óptimos para la amplificación por qPCR de los diferentes genes. En el caso del gen CYP4G1 like se diseñaron dos parejas de cebadores en dos regiones diferentes del gen para confirmar los niveles de expresión. Los amplicones obtenidos para los seis genes CYP tenían entre 90-151 pb y los cebadores mostraron unas eficiencias de amplificación comprendidas entre el 81% y el 110%. Los coeficientes de determinación de las rectas patrón de amplificación de cada gen (R2) variaron entre 0,991 y 0,997 (Tabla 3.3.3). Las parejas de cebadores que amplificaban los genes CYP6D (I) like y CYP6D (III) like se diseñaron para que flanqueasen una región intrónica. Mediante el empleo de estas dos parejas de cebadores en una PCR sobre DNA genómico se corroboró la existencia de los correspondientes intrones y, dado que en la qPCR sobre cDNA no se observó un amplicón doble, se descartó que ocurriese una amplificación de DNA genómico en los ensayos de cuantificación de la expresión de los genes CYP. Los niveles de expresión del gen de referencia Ccp0 fueron similares a los niveles del gen CYP6D (III) like, con unos valores de ciclo de inicio de la amplificación (Ct) de 25,82±0,33 y 25,98±0,35 respectivamente. Estos niveles de expresión se sitúan en una zona intermedia si tenemos en cuenta la expresión estimada para el resto de genes CYP analizados (Fig. 3.3.11). 91 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ Machos 10000 Expresión relativa Hembras * 1000 * 100 * 10 * 1 CYP12C (II) like CYP6D (I) like CYP4G1 like CYP6A (I) like CYP6G like CYP6D (III) like Figura 3.3.11. Expresión de genes CYP en adultos de C. capitata. Análisis de los niveles de expresión de los genes CYP12C (II) like, CYP6D (I) like, CYP4G1 like, CYP6A (I) like, CYP6G like y CYP6D (III) like, analizados en machos y en hembras de C. capitata mediante qPCR. Los valores de expresión génica relativa se muestran en escala logarítmica; están normalizados con respecto a los valores de Ccp0 y relativizados con respecto a la expresión en hembras del gen CYP6A (I) like. Las barras de los histogramas representan la desviación estándar de la media. El asterisco indica que existen diferencias significativas en la expresión de un determinado gen, al comparar entre sexos (t Student, p< 0,05). El gen CYP6A (I) like, en hembras, fue el que menos se expresó, por lo que se empleó como referencia para estimar los valores de expresión del resto de genes. Los niveles basales de expresión de los adultos de la línea C de C. capitata para los seis genes CYP analizados mostraron diferentes rangos de expresión, variando hasta en tres órdenes de magnitud. Por ejemplo, en las hembras, el gen CYP6G like se expresó unas 5.000 veces más que CYP6A (I) like. La expresión en algunos de los genes analizados varió en función del sexo, observándose una expresión significativamente mayor en machos que en hembras en los genes CYP12C (II) like, CYP4G1 like y CYP6A (I), mientras que el gen CYP6D (III) like se expresó más en hembras que en machos. La mayor diferencia entre sexos se observó en CYP12C (II) like, gen en el que el nivel de expresión fue tres veces superior en machos que en hembras. En los genes CYP6D (I) like y CYP6G like no se observaron diferencias significativas en la expresión comparando ambos sexos (Fig. 3.3.11). 92 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP TCCAGGCTCTCTCCATACCA CGACTTTGTCACCAGGCTTC RP0 CGTGGACGTGGAAAGAAAAG R12C1 FP0_2 GTAATGTGCGCGAAACTGG CGAGAAATGGTAGTGGTGGA R6G2_2 F12C1 CGTTTGCGTGAGGAGGTG GGCAGACCGGGATATTTG R6D5_2 F6G2_2 GGCGCAACGTGATATTAGAG GTGTGCTCAGAATTGGGTATTT R6D2_2 F6D5_2 TGAAGCCCAGTGATCCTCTT F6D2_2 20 20 20 19 20 18 18 20 22 20 21 19 19 20 21 21 b 90 121 136 126 151 105 158 139 Amplicón (pb) 2 0,997 0,995 0,994 0,997 0,997 0,991 0,995 0,997 R 100 103 102 102 108 81 105 110 % Eficiencia 93 ________________________________________________________________________________________________________________________ Ccp0 CYP12C (II) like CYP6G like CYP6D (III) like * CYP6D (I) like * CGTCAGTTGATTGTCGTGTTG R6A1 GCGCAATCAAAGGTACTGG R4G1b_2 ACGCTCTCTTTTGCGCTCT TAAGGACGTGCAGGAAAAGG F4G1b_2 F6A1 GGTACATACGCAAGGTCTCCA R4G1 CYP6A (I) like CATAAGGACGTGCAGGAAAAG F4G1 3’) CYP4G1 like Secuencia (5’ Cebador Gen nombre del gen que amplifican. La secuencia nucleotídica se muestra en sentido 5’ 3’. Se muestra el número de bases (b) y el amplicón originado por la pareja de cebadores directo (F) y reverso (R) en pares de bases (pb). R2 indica el coeficiente de determinación de la recta patrón de amplificación. Se muestra el porcentaje de eficiencia de la pareja de cebadores. (*) Estos genes contienen un intrón en la región amplificada por la pareja de cebadores en el DNA genómico. Tabla 3.3.3. Genes seleccionados para el estudio de expresión. Se indican los cebadores empleados en la qPCR ordenados según el _____________________________________________________________________________________________________________________________________________________________________________________________________________ 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ 3.3.3. Inducción por fenobarbital. Después de exponer adultos de C. capitata durante 72 horas a una dieta que contenía 1% de fenobarbital, la mortalidad fue del 17%, y del 25% con 3% de fenobarbital. En los individuos supervivientes se analizaron, mediante qPCR, los niveles de expresión en los genes CYP6D (I) like, CYP6D (III) like, CYP6A (I) like, CYP6G like, CYP12C (II) like y CYP4G1 like, y se compararon con los niveles de expresión de los individuos no tratados con fenobarbital (Fig 3.3.12). La respuesta como inductor del xenobiótico fenobarbital fue desigual tanto al comparar su efecto en los distintos genes estudiados como entre machos y hembras (Fig. 3.3.12). En el análisis de la expresión de CYP6G like, el gen que presentó mayores tasas de expresión, no se detectó inducción en ninguno de los dos sexos (Fig. 3.3.12.E). En el análisis de los niveles de expresión de CYP4G1 like no se observaron diferencias significativas, tanto en machos como en hembras, al comparar los valores de los insectos tratados con fenobarbital y los del control sin xenobiótico (Fig. 3.3.12.C). Sin embargo, en CYP6D (I) like, otro gen que mostró elevados niveles de expresión respecto a CYP6A (I) like, sí se observó inducción significativa por la exposición a 1% y a 3% de fenobarbital tanto en machos (1,8 y 2,2 veces, respectivamente), como en hembras (2,5 y 2,4 veces, respectivamente) en comparación con los controles sin xenobiótico (Fig. 3.3.12.B). Unos valores similares de inducción (Fig. 3.3.12.F) se observaron en el otro gen de la subfamilia CYP6D, CYP6D (III) like, en las dos concentraciones de fenobarbital ensayadas y en ambos sexos (1,7 veces en machos y 1,9-2,1 veces en hembras). En los genes CYP12C (II) like y CYP6A (I) like, que mostraron los menores niveles de expresión basal, se observó inducción estadísticamente significativa, pero únicamente en uno de los dos sexos y frente a una de las dos concentraciones de fenobarbital (Fig. 3.3.12.A y D). Así, se observó inducción (1,8 veces) de CYP12C (II) like en hembras expuestas a 1% de fenobarbital, pero no se detectó inducción en la exposición a 3%, mientras que en CYP6A (I) like se observó un aumento significativo de la expresión en machos del tratamiento de 3% de xenobiótico (1,8 veces). 94 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP 7 1% FB 3% FB 6 5 4 * 3 2 1 8 7 6 3 Control 3% FB 5 4 3 8 7 1 0 Machos Control 6 5 4 3 8 7 Hembras Control CYP6D (III) like F 1% FB 3% FB 5 4 3 2 1 1 0 Hembras 3% FB 6 2 0 1% FB * 1,8 Hembras 3% FB Machos D 3 0 1% FB Control 4 2 CYP6G like 2,4 5 1 E 2,5 Hembras CYP6A (I) like 6 2 7 1,8 2,2 Machos 6 8 3% FB * * 4 0 1% FB Machos 1% FB * * 5 Hembras CYP4G1 like C 7 Control CYP6D (I) like B 1 Expresión relativa Expresión relativa Machos 8 2 1,8 0 Expresión relativa Control CYP12C (II) like A Expresión relativa 8 Expresión relativa Expresión relativa ______________________________________________________________________________________________________________________________________ * * 1,7 1,7 Machos * * 1,9 2,1 Hembras Figura 3.3.12. Efecto del fenobarbital en la expresión de los genes CYP en C. capitata. Análisis de la influencia del xenobiótico fenobarbital (FB) en los niveles de expresión en de CYP12C (II) like, CYP6D (I) like, CYP4G1 like, CYP6A (I) like, CYP6G like y CYP6D (III) like mediante qPCR. Se compararon los tratamientos de 1% y 3% de FB, sumnistrado en la dieta, con respecto a un control sin el xenobiótico. Los valores de expresión relativa están normalizados con la expresión de CCp0 y relativizados, para cada gen, con respecto a la réplica biológica del control sin FB que tenga el menor valor de expresión en cualquiera de los dos sexos. Las barras de los histogramas representan la desviación estándar de la media. (*) Diferencias significativas entre los niveles de expresión de un tratamiento de FB con respecto al control correspondiente de cada sexo y para cada gen (test de Dunnett, p< 0,05). Se indica el valor de inducción, con respecto al control de cada sexo, en aquellos tratamientos con diferencias significativas con respecto al control. 95 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ 3.4. Discusión. Las citocromo P450 engloban un elevado número de enzimas que participan en diversas funciones metabólicas. Una de sus funciones es la detoxificación de xenobióticos y han sido relacionadas con procesos de resistencia metabólica (Feyereisen, 2005). En este trabajo, partiendo de DNA genómico y de cDNA, se ha evaluado la diversidad de genes CYP presentes en la mosca mediterránea de la fruta C. capitata. El empleo de cebadores degenerados ha permitido la amplificación por PCR de un fragmento que representa un tercio del tamaño total del gen CYP y que está localizado en la región central, entre la hélice I y el grupo hemo. Tras el análisis de las secuencias y su comparación con los genes de D. melanogaster hemos obtenido un total de 72 haplotipos de genes CYP entre los que hemos identificado, basándonos en criterios de similitud, al menos, 37 genes diferentes. Al no existir solapamiento entre las secuencias que hemos obtenido y las 13 secuencias parciales descritas por Danielson y colaboradores (Danielson et al., 1999), no se han podido relacionar entre sí. Debido a que no se ha obtenido la secuencia completa de la región codificante de los genes, no ha sido posible la asignación de un nombre completo a cada gen. A los genes se les ha otorgado un nombre provisional en función de los porcentajes de similitud que definen las familias (>40%) y subfamilias (50%), tomando como referencia el CYPoma de D. melanogaster. La secuenciación del cDNA completo de todos los haplotipos que hemos descrito permitirá asignar a cada gen un nombre adecuado siguiendo la nomenclatura actual (Nelson et al., 1996). 97 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ El número de genes identificados en este trabajo mediante el proceso de amplificación por PCR, clonación y selección de los diferentes haplotipos por RFLP es elevado si se compara con la mayoría de trabajos en donde se ha evaluado la diversidad de genes CYP con metodologías similares, como por ejemplo los 17 genes hallados en Anopheles albimanus (Scott et al., 1994), los 10 genes en D. melanogaster (Dunkov et al., 1996), o los 14 genes descritos en C. capitata (Danielson et al., 1999). No obstante, recientemente se han obtenido resultados similares en cuanto a la diversidad de genes en Frankliniella occidentalis (Cifuentes et al., 2012). El número de secuencias parciales de genes CYP descritas en nuestro estudio es mayor al obtenido mediante nuevas técnicas de secuenciación en la mariposa Zygaena filipendulae (Zagrobelny et al., 2009) y similar al obtenido en la polilla Manduca sexta (Pauchet et al., 2010). Téngase en cuenta que, en base a la secuenciación masiva del genoma completo de Bombyx mori, se ha estimado que esta especie posee 85 genes CYP (Feyereisen, 2012), lo que sugiere que el número de genes CYP en lepidópteros no es necesariamente mucho menor que en dípteros. Por ejemplo, en D. melanogaster se han identificado 83 genes CYP que codifican proteínas P450 (Tijet et al., 2001). Los cebadores degenerados, diseñados a partir de las secuencias proteicas de las familias CYP4, CYP6, CYP9, CYP12, CYP18 y CYP28 de D. melanogaster, han amplificado genes pertenecientes a todas las familias esperadas excepto a la familia CYP28, obteniéndose la misma proporción de genes por familia que la existente en D. melanogaster (Tijet et al., 2001). Si ambas especies tuviesen una cantidad similar de genes en las cinco familias estudiadas, los 37 genes hallados con los cebadores degenerados en C. capitata representarían un 60% de la diversidad para dichas familias. Además, si asumimos que el número total de genes CYP de C. capitata es similar al de otros dípteros del suborden Brachycera (entre 76 y 91, según Feyereisen, 2011), los genes identificados en este trabajo representarían entre el 41% y el 49% del total del CYPoma de la mosca mediterránea de la fruta. Recientemente se ha analizado el transcriptoma de B. dorsalis (Hsu et al., 2012a) y se ha estimado en 90 el número de genes CYP existente en esta especie de tefrítido, muy próxima a C. capitata. La comparación del número de genes CYP de las familias comunes a ambos estudios nos indica que la diversidad que detectada en C. capitata representa un 55% con respecto a la de B. dorsalis, asumiendo que ambas especies poseen el mismo número de genes. El árbol filogenético, resultado de la comparación de las secuencias aminoacídicas de D. melanogaster con las secuencias de C. capitata, nos muestra que los nuevos genes encontrados se incluyen en los cuatro grandes clados de genes CYP de insectos: CYP-2, CYP-3, CYP-4 y CYP mitocondrial (Feyereisen, 2006). En el clado CYP-3 es donde hemos obtenido el mayor número de genes y de haplotipos, localizados principalmente en la familia CYP6. Se han identificado un total de 16 nuevos genes de la familia CYP6, lo que constituiría una amplia representación dentro de la 98 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ familia si comparamos esta diversidad con los 25 genes existentes en D. melanogaster (Tijet et al., 2001). La disposición en el árbol filogenético de algunas de las secuencias obtenidas en C. capitata en relación a las secuencias conocidas de D. melanogaster pone de manifiesto las limitaciones del sistema adoptado para otorgar un nombre provisional a los genes CYP de C. capitata. Como se ha dicho anteriormente, sólo las secuencias completas de las regiones codificantes permiten una nomenclatura adecuada (Feyereisen, 2005). Así, en este trabajo se han observado contradicciones entre las relaciones filogenéticas observadas en el árbol y el nombre asignado a cada gen de C. capitata según el porcentaje de homología con las secuencias de D. melanogaster. El porcentaje de similitud de CYP6G like no permitió su asignación a una subfamilia, pero en el árbol filogenético se agrupó con la subfamilia CYP6A. Además, algunas secuencias de C. capitata a las que se les había asignado una subfamilia por similitud, no se agruparon significativamente en el árbol con miembros de dicha subfamilia de D. melanogaster (por ejemplo, CYP4D3 like se agrupó con la familia CYP4AE). Finalmente, determinadas secuencias a las que se les asignó una subfamilia por los criterios de similitud, no se agruparon significativamente en el árbol con ninguna subfamilia de enzimas P450 de D. melanogaster (por ejemplo, CYP4D (I) like). Se debe tener en cuenta que estas dos especies de dípteros no incluirán necesariamente en su CYPoma genes representantes de las mismas subfamilias. En cualquier caso, las secuencias parciales de genes CYP identificadas en este estudio facilitarán la obtención de las secuencias completas de los genes de C. capitata. En la literatura científica no existen prácticamente datos sobre la expresión basal de los genes CYP en insectos, ya que la mayoría de estudios se centran en la comparación, ya sea a través de PCR cuantitativa o de microarrays, de los niveles de expresión de uno o varios genes ante un determinado tratamiento con respecto a un control, para evaluar así el efecto de dicho tratamiento. Los niveles transcripcionales basales de los seis genes analizados en C. capitata presentaron un rango de variación de hasta tres órdenes de magnitud al relativizar su expresión con respecto a la del gen CYP con menor valor de expresión (CYP6A (I) like), rango similar al detectado en la expresión de varios genes CYP en las larvas del mosquito Aedes aegypti (Poupardin et al., 2008; Riaz et al., 2009). En cuanto a la comparación de la expresión basal entre sexos, en cuatro de los seis genes se observó una expresión diferencial dependiente del sexo de hasta tres veces más, como se observa en el gen CYP12C (II) like, diferencias que podrían estar ligadas a los distintos requerimientos metabólicos de cada sexo y que han sido descritas en profundidad en D. melanogaster (Ranz et al., 2003; Arbeitman et al., 2004). Las diferencias de expresión de los genes CYP entre sexos deberían ser consideradas cuando se estudie la expresión de estos genes en relación a la resistencia a insecticidas, debiendo separar machos y hembras o realizando grupos equilibrados de ambos sexos. 99 3. Bases para la detección precoz de la resistencia: diversidad de genes CYP ______________________________________________________________________________________________________________________________________ Los trabajos realizados sobre la inducción de genes CYP por xenobióticos en insectos han sido recientemente revisados por Feyereisen (2012). Una gran variedad de moléculas pueden actuar como inductores de uno o más genes CYP en insectos pero, en base a los conocimientos ya adquiridos en los estudios realizados en vertebrados, el fenobarbital es el inductor más empleado. De hecho, el estudio sobre la inducción de genes CYP en D. melanogaster sugiere que una tercera parte del CYPoma de este insecto es susceptible de ser inducido por xenobióticos, que existen subconjuntos de inductores/inducidos, y que puede existir cierto grado de conservación sobre el tipo de inducción por fenobarbital observada en D. melanogaster y en vertebrados (Giraudo et al., 2010). En nuestro trabajo, se ha estudiado por primera vez la inducción de genes CYP por xenobióticos en C. capitata. Aunque sólo se ha analizado la expresión de seis genes, se ha podido observar que cuatro de ellos son inducidos a alguna de las concentraciones ensayadas o en alguno de los dos sexos La inducción por fenobarbital en diferentes genes CYP en insectos varía en un amplio rango según el gen analizado y dependiendo del sexo del individuo (Le Goff et al., 2006, Willoughby et al., 2006; Sun et al., 2006; Zhou et al., 2010; Chung et al., 2011). La regulación de los genes CYP que participan en los procesos fisiológicos es probablemente compleja y, en algunos casos, puede haber expresión coordinada de distintos genes CYP pertenecientes a un mismo cluster o que participan en una misma cascada metabólica (Feyereisen, 2012). Los valores de inducción con respecto al control que hemos obtenido en cuatro de los seis genes estudiados son bajos (1,7-2,5 veces), pero se encuentran en el intervalo de inducción encontrado en otras especies de insectos en experimentos similares realizados en tejidos específicos (Zhou et al., 2010; Chung et al., 2011), en estadios larvarios (Stevens et al., 2000; Li et al., 2002; Sun et al., 2006; Willoughby et al., 2006) o, como en nuestro caso, en individuos adultos (Scott et al., 1996; Brown et al., 2003; Sun et al., 2006; Willoughby et al., 2006; Le Goff et al., 2006). Según los casos descritos hasta el momento, la resistencia a insecticidas mediada por P450 se produce por un incremento de la actividad P450 como consecuencia de la duplicación de genes o de alteraciones en la regulación de su expresión. Se han estimado unos valores de inducción por fenobarbital en D. melanogaster de 15 veces en Cyp6a2 (Brun et al., 1996) y en M. domestica de 10 veces en su ortólogo CYP6A1 (Cariño et al., 1994), genes implicados en resistencia a organofosforados, carbamatos y DDT (revisado por Feyereisen, 2012). Recientemente, nuestro grupo ha seleccionado en el laboratorio una línea de C. capitata resistente al piretroide lambda-cialotrina cuyo mecanismo de resistencia, de acuerdo con los resultados obtenidos con sinergistas, podría estar mediado por P450 (resultados no publicados). El conocimiento de la diversidad de genes CYP será de utilidad para el estudio de este mecanismo de resistencia, que podrá facilitar el desarrollo de un sistema bioquímico o molecular de detección precoz de individuos resistentes. 100 Capítulo 4 DETERMINACIÓN DE LA ESTRUCTURA DE LOS GENES Ccace2 Y Ccae7 Y DESARROLLO DE SISTEMAS DE DETECCIÓN PRECOZ DE ALELOS DE RESISTENCIA 101 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ 4.1. Introducción. Los genes de Ceratitis capitata Ccace2 (ref. GenBank: FJ480223) y Ccae7 (ref. GenBank: FJ489674) codifican respectivamente para la acetilcolinesterasa (AChE, EC 3.1.1.7) y para una carboxilesterasa, la aliesterasa (AE7, EC 3.1.1.1). La función de la acetilcolinesterasa es la regulación de los niveles de acetilcolina durante la sinapsis colinérgica (Fournier, 2005). Esta enzima es la molécula diana de los insecticidas organofosforados y carbamatos, que actúan como inhibidores. El bloqueo del centro activo de la enzima provoca la acumulación de acetilcolina en el espacio sináptico, produciéndose la sobreexcitación nerviosa y la muerte del insecto (Aldridge, 1950). El mecanismo de resistencia en el que está involucrada habitualmente la AChE es la modificación estructural de la molécula diana debido a mutaciones puntuales (Mutero et al., 1994; Walsh et al., 2001). El cambio de un único aminoácido disminuye su susceptibilidad de ser inhibida por el insecticida y puede reducir su actividad enzimática. Así ocurre con la mutación AChE G328A que confiere resistencia al organofosforado malatión (Walsh et al., 2001; Menozzi et al., 2004; Baek et al., 2005; Lee et al., 2007; Khajehali et al., 2009). Actualmente se han descrito nueve mutaciones puntuales en la AChE (genes ace1 o ace2) asociadas a resistencia a insecticidas (Kono y Tomita, 2006). Las carboxilesterasas son una gran familia de enzimas que, entre otras funciones, se encargan de la detoxificación de xenobióticos (Oakeshott et al., 2005) y han sido implicadas en la resistencia a malatión (Campbell et al., 1998b; Hemingway y Karunaratne, 1998). La resistencia metabólica mediada por esterasas puede deberse a la sobreexpresión de las mismas, a 103 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ duplicaciones génicas, o a mutaciones puntuales que favorecen el incremento de su actividad hidrolítica (Oakeshott et al., 2005). En los dípteros Musca domestica, Lucilia cuprina y L. sericata se han identificado dos mutaciones asociadas a resistencia en el centro activo de la AE7: G137D, que confiere resistencia a varios organofosforados, y W251S/L ligada con una resistencia específica a malatión (Newcomb et al., 1997; Claudianos et al., 1999; Taskin y Kence, 2004; Hartley et al., 2006). Nuestro grupo ha determinado que en individuos de la línea W de C. capitata, resistente a malatión, la actividad acetilcolinesterasa es menos susceptible de ser inhibida por malaoxón, forma activa del malatión (Magaña et al., 2007). Además, hemos relacionado esta disminución en la susceptibilidad frente a malatión con la mutación puntual AChE G328A (numeración según Torpedo californica; Schumacher et al., 1986), situada en un aminoácido adyacente al glutámico de la tríada catalítica (Magaña et al., 2008). Esta mutación puntual en la acetilcolinesterasa se ha relacionado con la resistencia a insecticidas tanto en el gen ace2, en Drosophila melanogaster (Menozzi et al., 2004) y en M. domestica (Walsh et al., 2001), como en ace1 en la araña roja Tetranychus urticae (Khajehali et al., 2009). La resistencia a malatión en la línea W se debe además a un segundo mecanismo de resistencia en el que estarían implicadas las carboxilesterasas, ya que la aplicación de DEF, inhibidor de la actividad esterasa, reduce parcialmente la resistencia frente a malatión (Magaña et al., 2007). El análisis de la secuencia del gen Ccae7 de la población resistente no mostró las mutaciones asociadas a resistencia en otras especies de dípteros, sin embargo, se encontraron ocho sustituciones no sinónimas (exones 2, 4 y 5), no presentes en una línea susceptible de laboratorio, que no pudieron ser asociadas a resistencia (Magaña et al., 2008). La caracterización de un determinado mecanismo de resistencia favorece la elaboración de métodos de diagnóstico para la detección precoz de la resistencia en poblaciones de campo (Hemingway et al., 2002). Para este diagnóstico precoz se emplean técnicas bioquímicas y moleculares que permiten identificar los fenotipos y/o genotipos resistentes sin necesidad de realizar bioensayos de susceptibilidad. Se han desarrollado diversas técnicas para la detección de alelos de la acetilcolinesterasa que confieren resistencia a organofosforados en el pulgón Myzus persicae (Cassanelli et al., 2005), en el lepidóptero Cydia pomonella (Cassanelli et al., 2006), en la mosca Haematobia irritans (Foil et al., 2010), en cinco especies de mosquitos (Weill et al., 2004; Alout et al., 2007; Ben Cheikh et al., 2009), en Bactrocera dorsalis (Hsu et al., 2006) y en la mosca del olivo B. oleae (Hawkes et al., 2005; Margaritopoulos et al., 2008). En cuanto a las carboxilesterasas, se han elaborado métodos de detección de alelos que confieren resistencia a organofosforados en Culex pipiens (Berticat et al., 2000) y en H. irritans (Li et al., 2003). Uno de los métodos más empleados para la identificación de los genotipos implicados en resistencia es el sistema PCR-RFLP. En esta técnica se realiza una amplificación por PCR del gen implicado en 104 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ resistencia, seguida de una digestión del amplicón con una enzima de restricción, la cual distingue entre el alelo silvestre y el mutado para, posteriormente, visualizar el perfil de restricción en un gel de agarosa (Weill et al., 2004; Cassanelli et al., 2005, 2006; Hsu et al., 2006). Las secuencias nucleotídicas de los genes Ccace2 y Ccae7 han sido obtenidas a partir de la secuenciación del cDNA (Magaña et al., 2008). La amplificación por PCR de los exones completos a partir de DNA genómico resulta complicada debido a la existencia de intrones de gran tamaño. La estructura génica de la acetilcolinesterasa y de la aliesterasa están conservadas en dípteros, por lo que cabe esperar que los intrones existentes en C. capitata sean de gran tamaño, al igual que ocurre en D. melanogaster, donde existen varios intrones en ambos genes de entre dos y cinco kilobases (McQuilton et al., 2012). Además, la mutación AChE G328A que confiere resistencia a malatión en C. capitata se localiza en la primera base del exón seis de la acetilcolinesterasa (Magaña et al., 2008). Por lo tanto, para crear un método PCR-RFLP de detección de dicha mutación, tomando DNA genómico como molde de la PCR, es necesario conocer la secuencia del intrón 5, inmediatamente anterior al sitio puntual. Mediante el conocimiento de la estructura de los genes Ccace2 y Ccae7 nos planteamos amplificar y secuenciar aquellos exones susceptibles de estar implicados en resistencia a partir de DNA genómico, para así explorar la presencia de nuevas mutaciones relacionadas con resistencia a insecticidas en estas dos enzimas en poblaciones de C. capitata de diferentes regiones geográficas. El diseño de un sistema PCR-RFLP para la detección de la mutación AChE G328A en C. capitata, permitirá relacionar los niveles de susceptibilidad de las poblaciones de campo con la frecuencia alélica de dicha mutación puntual y además, se podrá determinar su dispersión geográfica. Además, el método de diagnóstico PCR-RFLP se podría implementar en los programas de monitoreo por trampeo masivo que se realizan de manera rutinaria en los campos comerciales para estimar los niveles poblacionales de la mosca mediterránea de la fruta, empleando para ello los insectos capturados en las trampas. 105 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ 4.2. Material y métodos 4.2.1. Obtención de intrones mediante GenomeWalkerTM. Para determinar la estructura génica de Ccae7 y Ccace2 hemos empleado el Kit GenomeWalkerTM (BD Biosciences-Clontech, California, EE.UU.), que permite identificar las secuencias de los intrones adyacentes a un determinado exón mediante la amplificación por PCR de librerías de DNA genómico digerido con diferentes enzimas de restricción (Fig. 4.2.1.). 107 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ DNA genómico División en cuatro librerías Digestión Unión del adaptador (AP) Amplificación del gen de interés en cada librería con los cebadores del adaptador (AP1 y AP2) y los específicos (CE1 y CE2) CE2 CE1 AP Intrón AP1 AP2 Exón Primera PCR: AP1-CE1 Segunda PCR: AP2-CE2 Visualización de los amplicones en gel de agarosa Figura 4.2.1. BD GenomeWalkerTM. Esquema del protocolo de elaboración de las librerías de DNA, amplificación por PCR y localización de los cebadores empleados en la identificación de la secuencia de los intrones de Ccae7 y Ccace2. La posición de los exones e intrones en los genes Ccae7 y Ccace2 de C. capitata se predijo en función de la estructura génica (Fig. 4.2.2.) de los genes homólogos de D. melanogaster Dmae7 y Dmace2 (ref. Genbank NP_524261 y NP_476953 respectivamente). 108 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ I5 I4 I3 I2 I1 Dmae7 I9 I7 I6 I5 I8 I2 I4 I1 I3 Dmace2 Figura 4.2.2. Estructura génica de Dmae7 y Dmace2. Esquema modificado de McQuilton et al. (2012) que representa la estructura de los genes ae7 y ace2 y de D. melanogaster, localizados en el cromosoma 3R. Se indica la posición y el número de intrones (I). Los cebadores específicos para C. capitata se diseñaron a partir de las secuencias de cDNA de Ccae7 y de Ccace2 (ref. FJ489674 y EU130781 respectivamente). En las tablas 4.2.1 y 4.2.2 se indican las secuencias de los cebadores AP1 y AP2, suministrados en el Kit GenomeWalkerTM, así como las de los cebadores específicos basados en las secuencias conocidas de cDNA de C. capitata. En este estudio se extrajo el DNA a partir de la cabeza de individuos adultos de la línea susceptible de laboratorio C de C. capitata (descrita en el apartado 2.2.2) mediante las columnas DNeasy® Blood & Tissue Kit (Qiagen, California, EE.UU.) y siguiendo las instrucciones del fabricante. Una vez extraído, se midió la concentración de DNA total con un NanoDrop (Thermo Scientific, Massachusetts, EE.UU.) y se mantuvo almacenado a -20ºC hasta su utilización. Se dividió en cuatro alícuotas un volumen de 100 µl de DNA total a una concentración de 0,1 µg/µl. Cada una de las alícuotas se digirió con una de las enzimas de restricción (Dra I, EcoR V, Pvu II y Stu I) recomendadas en el kit BD GenomeWalkerTM, las cuales reconocen dianas de restricción que coinciden con motivos típicos presentes en los intrones. Se obtuvieron así cuatro librerías de DNA total con diferentes cortes de restricción. 109 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia E5 Ccace2 TM RAChE_CcE5.1 (CE1) RAChE_CcE5.2 (CE2) FiAChE_CcE50 (CE1) FAChE_327N (CE2) RiAChE_CcE60 (CE1) RAChE_327 (CE2) FAChE_CcE6 (CE1) FAChE_CcE6.2 (CE2) RAChE_CcE5.1 (CE1) RAChE_CcE5.2 (CE2) FAChE_CcE7 (CE1) FAChE_CCE7.2 (CE2) I4 – 3’ I5 – 5’ I5 – 5’ I5 – 3’ I5 – 3’ I6 – 5’ I6 – 5’ I6 – 3’ I6 – 3’ I7 – 5’ I7 – 5’ AP2 GenomeWalker TM AP1 GenomeWalker Cebador I4 – 3’ Intrón (I) 3’) GTTAGCTGAACGAGGAGCTTCTGT GTTGGGTTGGTAACCCTGGCTT CCATTGTTGCACTGAGATCG TCATAGCCACGCATATCAGC ACAAGCGGAACGCGAGGCCATCA GGCAAAGTAACACAAGCGGAAC AATTTCCAAATATTTATCACGCGG CCGAAAAATGATGGCCTCGCGTTCCG GTGGCTATGACATCTTGATGGGCA CTCAAGCTGTAATGGCTTGCATGCGTG CCATTGTTGCACTGAGATCG TCATAGCCACGCATATCAGC ACTATAGGGCACGCGTGGT GTAATACGACTCACTATAGGGC Secuencia (5’ 24 22 20 20 23 22 24 26 24 27 20 20 19 22 b 110 ________________________________________________________________________________________________________________________ E7 E6 Exón (E) Gen 4.2.1). Se indica el exón tomado como referencia para el diseño, los intrones (I) y la región 5’ o 3’ del intrón que es amplificada por los cebadores. Se muestra el nombre del cebador, la secuencia nucleotídica y el número de bases (b). Tabla 4.2.1. Cebadores empleados en la amplificación mediante GenomeWalkerTM de los intrones de Ccace2. Listado de los cebadores específicos (CE1 y CE2) diseñados para amplificar por PCR en combinación con los cebadores AP1 y AP2 del Kit GenomeWalkerTM (Ver Fig. _____________________________________________________________________________________________________________________________________________________________________________________________________________ 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia E2 Ccae7 Cebador RAE7Ex2.1 (CE1) RAE7Ex2.2 (CE2) FAE7_CcEx4 FAE7_CcEx4.2 Intrón (I) I1 – 3’ (CE1) I1 – 3’ (CE2) I4 – 5’ (CE1) I4 – 5’ (CE2) 3’) ATGAGAACAGCGTGGGGTAAT GTAGAACCGTATGTGACACC AGGTGGTTGGGCATAAGGTA TTGAGGTGCTCTGAAGCGTA Secuencia (5’ 21 20 20 20 b 111 ________________________________________________________________________________________________________________________ E4 Exón (E) Gen tomado como referencia para el diseño, los intrones (I) y la región 5’ o 3’ del intrón que es amplificada por los cebadores. Se muestra el nombre del cebador, la secuencia nucleotídica y el número de bases (b). Tabla 4.2.2. Cebadores empleados en la amplificación mediante GenomeWalkerTM de los intrones de Ccae7. Listado de los cebadores específicos (CE1 y CE2) diseñados para amplificar con los cebadores AP1 y AP2 del Kit GenomeWalkerTM (Ver Fig. 4.2.1). Se indica el exón _____________________________________________________________________________________________________________________________________________________________________________________________________________ 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ Una vez realizadas las digestiones del DNA, se unió un adaptador (AP) a los extremos de los fragmentos obtenidos para, posteriormente, realizar una primera amplificación por PCR en un termociclador Gene Amp® PCR System 2700 utilizando un cebador AP1, complementario al adaptador, y un cebador específico (CE1) que se encuentra dentro de la secuencia conocida del exón. Las condiciones de la primera PCR fueron 95ºC 1min; un ciclo de 94ºC 25 s, 72ºC 3 min, repetido siete veces; seguido de otro ciclo de 94ºC 25 s, 67ºC 3min, repetido 32 veces; 67ºC 7 min y finalmente 4ºC 30 min. Tomando como molde el producto de la primera PCR diluida 1/50, se realizó una reamplificación por PCR con un cebador AP2, complementario del AP1, y con un segundo cebador específico (CE2), situado en la secuencia conocida de exón (Fig. 4.2.1). Las condiciones de amplificación de la segunda PCR fueron las siguientes: 95ºC 1min; un ciclo de 94ºC 25s, 72ºC 3 min, repetido cinco veces; seguido de otro ciclo de 94ºC 25 s, 67ºC 3 min, repetido 20 veces; 67ºC 7 min para finalizar con 4ºC 30 min. Los productos de PCR se migraron electroforéticamente para su separación por tamaño en un gel de agarosa al 1,5% en TAE 1X modificado (Tris 10mM, EDTA 0,1mM pH 8.0) que contenía 1µg/50mL de gel de bromuro de etidio (Sigma Chemical Co., Missouri, EE.UU.) y se visualizaron en un sistema de documentación de geles Gel Doc XR (Bio-Rad Laboratories, California, EE.UU.). Los amplicones obtenidos en las reamplificaciones por PCR de las cuatro librerías de DNA digerido, susceptibles de contener secuencias de intrones, se purificaron o con el PCR Purification Kit (QIAGEN CombH, Hilden, Alemania), o mediante la extracción de la banda correspondiente del gel de agarosa y su posterior purificación en columnas Ultrafree-DA (Millipore, Massachusetts, EE.UU.). Los productos de PCR purificados fueron clonados en el pGEM®-T Easy Vector System (Promega Corporation, Wisconsin, EE.UU.) siguiendo las instrucciones del fabricante. Finalmente, para confirmar que el amplicón clonado incluía la secuencia del exón y la secuencia desconocida del intrón adyacente, los productos de PCR fueron enviados a Secugen S.L. (Madrid) donde se secuenciaron en un secuenciador automático ABI PRISM 377 (Applied Biosystems, California, EE.UU.). Las secuencias nucleotídicas definitivas de cada intrón se obtuvieron después de la corrección manual de los cromatogramas con el programa Chromas V1.45 (Griffith University, Queensland, Australia). La posición inicial y final de los intrones adyacentes a las regiones de exón de los genes que codifican para la acetilcolinesterasa (AChE) y para la aliesterasa (AE7) de C. capitata se determinaron por el alineamiento con las secuencias de cDNA. 112 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ 4.2.2. Amplificación de los exones de Ccace2 y Ccae7. A partir de las secuencias obtenidas para las regiones intrónicas que flanqueaban los exones de los genes Ccace2 y Ccae7, se diseñaron cebadores específicos (Tabla 4.2.3) para amplificar por PCR a partir de DNA genómico aquellos exones en los que se habían descrito previamente mutaciones asociadas con la resistencia a insecticidas, tanto en genes ace como en genes ae7. Las condiciones de amplificación fueron las siguientes: 95ºC 10 min, 40 ciclos de 95ºC 30 s, 48-52ºC 30 s y 72ºC 45 s, seguidos de 72ºC 7 min y finalizando con un periodo de 30 min a 4ºC. Para comprobar el correcto funcionamiento de los cebadores diseñados, se realizó la amplificación de los exones de Ccace2 y Ccae7 de individuos de la línea de laboratorio W-4Km (descrita en el apartado 2.2.2), y de tres poblaciones de campo de Australia, Sudáfrica e Isla Reunión (Tabla 4.2.4). 113 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia 86 584 240 E4 (561) E5 (164) 161 E7 (150) E3 (151) 536 E6 (143) 364 234 E5 (204) E2 (313) 585 E4 (537) Ccae7 191 E3 (139) Ccace2 Amplicón Exón Gen 114 TTTCCCTGCCTTAGGCGCTAAGA GTTCCAGAAAATCGTGACAATGA Rin5Ex5_AE7 ATGCCAGTAGTTCTCACCTTGC Rin4Ex4_AE7 Fin4Ex5_AE7 AAACAGGCTTCCTTTCGCT TGCTATCATCCCACACATACCC RAE7_in3Ex3 FAE7_in3Ex4 CTTAAATCCTCACAGACTCTGCC GGTACATAAATTTATATATATACTCAC Rin2Ex2_AE7.1 FAE7_in2Ex3 CTCATACCTTTCTTCCGTTCTTAG ACTTACATGTGTAAAGTAATA Rin7Ex7_AChE Fin1Ex2_AE7 TTCAGCACACAAGTTGGGT TGCGCACAGTATGGCAACTCT Rin6AChE Fin6Ex7_AChE TCAACATGTTTTTCATTTTCGTTCCAG CACAGGAAGTACTCACCTTCATC Rin5Ex5_AChE FG328A_27 CATTTTCAGGAAAATCCTCAAGC TTGCAAAATATGCACACCCACTTACCGCC Rin4Ex4_AChE Fin4Ex5_AChE TCGTCTTCTAATATTCCCTACAGCACTCC Fin3Ex4_AChE TTAGCCTTTTTGTAGCTACTCACCTCACC Rin3Ex3_AChE 3’) GTATTCCCTTTTCGTGCGTTCACCTACAG Secuencia (5’ Fin2Ex3_AChE Cebador 23 23 22 19 22 23 27 24 21 19 21 27 23 23 29 29 29 29 b tamaño del amplicón obtenido en pares de bases, el nombre del cebador, la secuencia nucleotídica y el número de bases del cebador (b). Tabla 4.2.3. Cebadores empleados en la amplificación de los exones de Ccace2 y Ccae7. Una vez obtenida la secuencia de los intrones flanqueantes de los exones de Ccace2 y Ccae7 se diseñaron los siguientes cebadores para amplificar en C. capitata, partiendo de DNA genómico, los exones 3, 4, 5, 6 y 7 de Ccace2 y los exones 2, 3, 4 y 5 de Ccae7. Se indica el exón y su tamaño en pares de bases, entre paréntesis, el _____________________________________________________________________________________________________________________________________________________________________________________________________________ 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ 4.2.3. Desarrollo de un método de detección de la mutación AChE G328A. Las secuencias obtenidas mediante el método GenomeWalkerTM permitieron el desarrollo de un sistema de PCR-RFLP para la detección del alelo Ccace2R, que codifica para la mutación AChE G328A en C. capitata, previamente asociada con resistencia a malatión (Magaña et al., 2008). El sistema está basado en un método diseñado por Jacobson y Moskovits (1991) para la detección de SNPs (single nucleotide polymorphisms) en oncogenes. Una vez conocida la secuencia nucleotídica del extremo 3’ del intrón 5 del gen Ccace2, se diseñó un cebador basado en la secuencia de dicho intrón. Este cebador directo FG328A_27 (Tabla 4.2.3) contiene una modificación en la cuarta base de su extremo 3’ que crea un sitio de restricción en ausencia de la mutación puntual AChE G328A, es decir, cuando está presente el alelo silvestre susceptible a malatión (Ccace2S). La combinación de este cebador con el cebador reverso RiAChE_CcE60 (Tabla 4.2.1), situado en el exón 6 de Ccace2, permitió la amplificación por PCR de la región que contiene la mutación AChE G328A, partiendo de DNA genómico (Fig. 4.2.3). Se emplearon individuos adultos de las líneas de laboratorio C y W-4Km para la puesta a punto del método. A PCR G328A RiAChE_CcE60 FG328A_27 EXÓN 6 Ccace2 Intron 5 B PCR-RFLP 172 pb 27 pb 145 pb Bst NI Figura 4.2.3. Diseño del método de detección de la mutación AChE G328A en C. capitata. (A) Se muestra la localización de los dos cebadores empleados en la amplificación por PCR de la mutación AChE G328A, partiendo de DNA genómico. (B) Esquema del sitio de corte de la enzima Bst NI cuando el producto de PCR generado en la amplificación por PCR no posee la mutación puntual AChE G328A. 115 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ La reacción de PCR se realizó en un termociclador GeneAmp® PCR System 2700 (Applied Biosystem, Foster City, EE.UU.). El volumen final de cada reacción fue de 13 µl con los siguientes componentes: 2 unidades de la enzima Amplitaq Gold® (Roche Molecular Systems, Inc., New Jersey, EE.UU.), los cebadores a una concentración de 0,6 µM, MgCl2 2mM, dNTPs 0,2mM, buffer II 1X, y aproximadamente 100 ng de DNA total. Las condiciones de la PCR fueron las siguientes: 95ºC 10 min, seguidos de un ciclo de 95ºC 30s, 48ºC 30s, 72ºC 15s que se repitió cuarenta veces, finalizando la reacción de PCR con 72ºC 7 min y 4ºC 30min. Para la visualización del amplicón, se cargaron 3 µl del producto de PCR en un gel de agarosa al 2% (D-2 Agarose, Lab. Conda, Torrejon de Ardoz, Madrid, España) en TAE 1X (Tris 40mM, EDTA 1mM pH 8.0). Posteriormente, se realizó un RFLP con la enzima de restricción Bst NI (New England Biolabs, Massachusetts, EE.UU.). Así, a los 10 µl de producto de PCR se le añadieron 2 µl de una disolución que contenía 2 unidades de la enzima Bst NI, buffer NE-2 1X y BSA 100 µg/ml. El RFLP se realizó en el mismo termociclador mediante una incubación durante 2 horas a 60ºC. Los productos del RFLP se migraron electroforéticamente en un gel de agarosa al 3% donde se mezclaron en TAE 1X, en una proporción 1:1, dos tipos de agarosa (D-2 Agarose y NuSieve® GTG® Agarose, Lonza, Maine, EE.UU.). En la figura 4.2.4 se muestra el perfil de restricción teórico para este sistema de detección teniendo en cuenta la secuencia conocida de cDNA de C. capitata. PCR-RFLP PCR 172 pb 145 pb 27 pb (liberación del cebador) SS RS RR Genotipo Ccace2 Figura 4.2.4. Patrón PCR-RFLP esperado para Ccace2. Perfil de restricción teórico para los tres genotipos posibles después de digerir el producto de PCR con la enzima de restricción Bst NI, siendo S el alelo susceptible Ccace2S y R el resistente Ccace2R . El método de detección del aleo Ccace2R, que confiere resistencia frente a malatión en C. capitata, se empleó para analizar individuos provenientes de poblaciones extranjeras (Tabla 4.2.4) y españolas (Tabla 4.2.5), examinando un total de 20 individuos para cada una de las poblaciones. 116 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ Con el objeto de evaluar la complementariedad del método de detección del alelo Ccace2R con los muestreos que se realizan para cuantificar la abundancia en campo de las poblaciones de C. capitata, se analizaron individuos procedentes de trampas Moskisan® (Sansan Prodesing S.L., Burjassot, Valencia), situadas en una parcela de naranjos en la localidad de Sagunt (Valencia). Las moscas se recogieron en trampas que permanecieron en el campo una semana. Los individuos muertos encontrados en las trampas se conservaron en etanol al 95% hasta su posterior estudio en el laboratorio. Sólo se analizaron individuos hembras para evitar la inclusión en el estudio de los machos procedentes de la suelta de machos estériles que se pudieran haber efectuado en la misma zona. El protocolo de extracción de DNA y el análisis por PCR-RFLP realizado fue el descrito anteriormente en este apartado. Tabla 4.2.4. Poblaciones extranjeras de C. capitata utilizadas en los estudios moleculares de la resistencia a insecticidas. Se muestran los países y las poblaciones analizadas en cada país, indicando su código de nomenclatura y la fecha de recolección de la población. Además se indica si fueron empleadas en algún estudio previo. País Población Código Fecha Estudio previo Australia Australia occidental AUS Oct, 2005 - Brasil Sao Paulo BRA Oct, 2004 Beroiz et al., 2012 Francia Isla Reunión REU Nov, 2008 - Grecia Agrinio, Argos, Zacharo, GRE Dic, 2006 - Lefkada, Dominio, Patras Guatemala Antigua Guatemala GUA Ago, 2008 - Israel Ma'agan Michael ISR Jun, 2004 Beroiz et al., 2012 Marruecos Agadir AGA Dic, 2003 Beroiz et al., 2012 Portugal Isla Madeira MAD Mar, 2004 Beroiz et al., 2012 Sudáfrica Sudáfrica SUD Ene, 2006 - Túnez Béja, Tabarca TUN Dic, 2008 - Turquía Turquía TUR Dic, 2008 - _________________________________________________________________________________________ 117 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ Tabla 4.2.5. Poblaciones españolas de C. capitata analizadas en los estudios moleculares de resistencia a insecticidas. Se muestran, ordenadas alfabéticamente, las localidades que dan nombre a las poblaciones, indicando la provincia (según ISO 3166-2 para España), su código de nomenclatura, el fruto hospedador, la fecha de recolección de la fruta, y si fueron empleadas en algún estudio previo. CC.AA./Población Provincia Código Hospedador Fecha Estudio previo Algarrobo Costa (MA) MAY Chirimoya Dic, 2008 Capítulo 2 Almuñecar (GR) ALM Níspero Jun, 2007 Capítulo 2 Ayamonte (H) AYA Mandarina Sep, 2008 Capítulo 2 Andalucía Gibraleón (H) GIB Mandarina Sep, 2008 Capítulo 2 Torre de Benagalbón (MA) MAG Mango Oct, 2003 Beroiz et al., 2012 (Z) VIG Melocotón Sep, 2007 Capítulo 2 Alcanar (T) ALC Mandarina Oct, 2007 Capítulo 2 Amposta (T) AMP Mandarina Oct, 2003 Beroiz et al., 2012 Girona (GI) GER Manzana Oct, 2004 Beroiz et al., 2012 Tortosa (T) TOR Mandarina Oct, 2003 Beroiz et al., 2012 Alacant (A) ALI Higo Sep, 2004 Beroiz et al., 2012 Alcúdia (V) ALD Melocotón Jun, 2004 Magaña et al., 2007 Burriana (CS) BUR Níspero Jun, 2004 Magaña et al., 2007 Carlet (V) CAR Melocotón Jun, 2004 Magaña et al., 2007 Les Coves de Vinromà (CS) COV Pera Jul, 2003 Beroiz et al., 2012 Moncada (V) MON Melocotón Jun, 2003 Beroiz et al., 2012 Orihuela (A) ORI Cítricos Nov, 2005 Magaña et al., 2007 Sagunt (V) SAG Cítricos Sep, 2008 Este capítulo Aragón Villalengua Cataluña C. Valenciana Serra (V) SER Cítricos Oct, 2005 Magaña et al., 2007 Vila Joiosa (A) VIY Naranja May, 2007 Capítulo 2 Vila Real (CS) VIL Caqui Oct, 2005 Magaña et al., 2007 Xàbia (A) JAV Naranja Jun, 2007 Capítulo 2 Marratxí (PM) MAR Naranja Nov, 2007 Capítulo 2 Muro (PM) MUR Cítricos Nov, 2007 Capítulo 2 (TF) VAG Zapote May, 2004 Beroiz et al., 2012 (LO) ALB Manzana Oct, 2004 Magaña et al., 2007 I. Baleares I. Canarias Valle de Guerra La Rioja Albelda ___________________________________________________________________________________________________________________________________ 118 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ 4.3. Resultados. 4.3.1. Estructura del gen Ccace2. Mediante el método GenomeWalkerTM se obtuvieron las secuencias intrónicas que flanquean los exones 3, 4, 5, 6 y 7 del gen Ccace2 (ver anexo II), exones situados en su región central (Fig. 4.3.1). La estructura intrón-exón fue análoga a la observada en Dmace2, su ortólogo en D. melanogaster (Fig. 4.2.2) y tanto las posiciones físicas de los intrones dentro del gen como su estructura típica de inicio/fin (GT/AG) fueron similares. El tamaño de las secuencias intrónicas obtenidas varió entre los 133 pb y los 3.119 pb. Tanto la secuencia completa del intrón 3 (147 pb), obtenida a partir de una librería de cDNA (Kit Marathon®, Clontech), como las secuencias parciales del extremo 3’ del intrón 2 y del extremo 5’ del intrón 4 se obtuvieron previamente a éste (Tabla 4.3.1). Todos los fragmentos de intrón obtenidos tuvieron el tamaño suficiente como para diseñar los cebadores que permitiesen la amplificación por PCR del exón adyacente correspondiente. 119 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ 4.3.2. Estructura del gen Ccae7. Se estudió la estructura génica de la región central del gen Ccae7 que codifica para la aliesterasa (AE7) analizando, mediante GenomeWalkerTM, las secuencias intrónicas adyacentes a cuatro de los seis exones que conforman el gen (E2, E3, E4, E5). En la figura 4.3.1 se muestra un esquema de la estructura génica obtenida para Ccae7 de C. capitata, donde se observa la localización de los intrones y de los cebadores diseñados sobre ellos para posteriormente amplificar los exones adyacentes mediante PCR. La secuenciación GenomeWalker TM de los productos de PCR obtenidos mediante el protocolo (ver anexo II) mostró que el esquema intrón-exón, analizando la secuencia nucleotídica del gen Ccae7 (Fig. 4.3.1), coincidía con la descrita para Dmae7, su ortólogo en D. melanogaster (Fig. 4.2.2), así como la posición física de los intrones y su estructura típica de inicio/fin (GT/AG). El tamaño de las secuencias intrónicas de Ccae7 varió entre los 71 pb y los 347 pb (Tabla 4.3.1), lográndose la secuencia completa de los intrones 2, 3 y 4. La secuencia de los intrones 2 y 3 se obtuvo previamente a partir de una librería de cDNA (Kit Marathon®, Clontech). La secuencia del intrón 4 se obtuvo de manera indirecta, al ser un intrón de pequeño tamaño (71 pb) incluido en la amplificación del cebador directo FAE7_CcEx4.2 y el cebador AP2 del GenomeWalkerTM. Las secuencias de los intrones 1 y 5 fueron parciales, pero con un tamaño suficiente como para diseñar cebadores que permitiesen la amplificación por PCR del exón adyacente correspondiente. G328A Ccace2 (DNA genómico) I2 I3 E3 I4 I5 E4 E5 I6 E6 I7 E7 Ccae7 (DNA genómico) I1 I2 E2 I3 E3 I4 E4 I5 E5 Figura 4.3.1. Estructura génica de Ccae7 y Ccace2. Esquema de los exones amplificados por PCR a partir de la secuencias de los intrones adyacentes (trazo grueso del segmento del intrón) obtenidas mediante el GenomeWalkerTM. Las flechas indican la posición de los cebadores diseñados para amplificar por PCR la secuencia completa del exón correspondiente. En el gen Ccace2 se indica la posición de la mutación puntual AChE G328A, que confiere resistencia a malatión. 120 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ Tabla 4.3.1. Secuencias nucleotídicas de los intrones de Ccae7 y Ccace2. Se indica el tamaño en pares de bases (pb) de las secuencias parciales de cada uno de los intrones, entre paréntesis. Además se muestra el tamaño total de la secuencia de intrón conseguida. Gen Intrón Tipo de secuencia y tamaño (pb) Ccae7 I1 Parcial, desde el extremo 3’ (107) I2 Completa 347 I3 Completa 82 I4 Completa 71 I5 Parcial, desde el extremo 5’ (192) > 192 I2 Parcial, desde el extremo 3’ (133) > 133 I3 Completa I4 Parcial, desde el extremo 5’ (2.436) Ccace2 Total (pb) > 107 147 > 3.119 Parcial, desde el extremo 3’ (683) I5 Parcial, desde el extremo 5’ (874) > 1.178 Parcial, desde el extremo 3’ (304) I6 Parcial, desde el extremo 5’ (960) > 2.227 Parcial, desde el extremo 3’ (1.267) I7 Parcial, desde el extremo 5’ (453) > 453 ____________________________________________________________________________________________________ 4.3.3. Amplificación de Ccae7 y Ccace2 en individuos de distintas regiones geográficas. Las secuencias nucleotídicas de los intrones obtenidas para los genes Ccae7 y Ccace2 nos permitieron diseñar cebadores específicos para amplificar por PCR, a partir de DNA genómico, cada uno de los exones adyacentes correspondientes (Tabla 4.2.3). Se obtuvieron productos de PCR para todas las parejas de cebadores diseñadas, amplificando los exones 2, 3, 4 y 5 de Ccae7 y los exones 3, 4, 5, 6, 7 de Ccace2. En la figura 4.3.2 se muestra, como ejemplo del correcto funcionamiento del sistema de amplificación por PCR, el análisis realizado en insectos procedentes 121 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ de una línea de laboratorio (Línea W-4Km) y de tres diferentes regiones geográficas: Sudáfrica (SUD), Australia (AUS) e Isla Reunión (REU). Así, se confirmó experimentalmente que las parejas de cebadores diseñadas amplificaban correctamente los exones independientemente del origen de los individuos analizados. Ccae7 E2 E3 M E4 E5 584 pb 364 pb 240 pb 186 pb Ccace2 E3 E4 E5 E6 M E7 585 pb 536 pb 234 pb 161 pb 191 pb Figura 4.3.2. Amplificación de Ccae7 y Ccace2 en individuos de distintas regiones geográficas. Las fotografías muestran la amplificación por PCR de los exones de Ccae7 y Ccace2, a partir de DNA genómico, de cuatro individuos de distintas regiones geográficas, migrados en geles de agarosa 1,5% en TAE 1X. Para cada uno de los exones (E), de izquierda a derecha, se analizó un individuo de la población australiana (AUS), sudafricana (SUD), de Isla Reunión (REU) y de la línea de laboratorio W-4Km. Se empleó como referencia un marcador molecular en escalera de 100 pb (M). 4.3.4. Método de detección del alelo Ccace2R portador de la mutación AChE G328A. Al alelo del gen Ccace2, que codifica para la mutación AChE G328A, y confiere resistencia al malatión en C. capitata (Magaña et al., 2008), lo denominaremos Ccace2R, y al alelo silvestre, que muestra susceptibilidad frente a malatión lo llamaremos Ccace2S. A partir de la secuencia del extremo 3’ del intrón 5 de Ccace2, obtenido en el apartado 4.3.2, se comprobó que dicho intrón se situaba entre la primera y la segunda base del codón portador de la mutación AChE G328A, tal y como se predecía de la estructura génica de Dmace2 (McQuilton et al., 2012). El sitio puntual de la mutación AChE G328A se correspondió con la segunda posición del triplete, por lo que dicha base 122 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ ocuparía la primera posición del exón 6. Debido a que en la secuencia del intrón adyacente al sitio de la mutación puntual (últimas bases del extremo 3’ del intrón 5) no existía un sitio de restricción adecuado para detectar el alelo mutado mediante un sistema convencional de PCR-RFLP, fue necesario diseñar un cebador que crease dicho sitio de restricción. El cebador en sentido directo se diseñó con una modificación en su extremo 3’, dirigida a crear una diana de restricción para la enzima Bst NI dependiente de la ausencia de la mutación AChE G328A. Es decir, se cortaría aquel producto de PCR que correspondiese al alelo silvestre Ccace2S. Este cebador, denominado FG328A_27 (Tabla 4.2.2), en combinación con el cebador reverso RiAChE_CcE60 (Tabla 4.1.) amplificó, a partir de DNA genómico, un fragmento de 172 pb que, al ser expuesto a la enzima Bst NI, permitió detectar la presencia/ausencia del alelo Ccace2R al migrar el RFLP en un gel de agarosa. Así, se obtuvieron tres perfiles de restricción distintos que nos permitieron diferenciar, dependiendo de si existía la mutación AChE G328A y si ésta se encontraba en homocigosis o heterocigosis, los tres posibles genotipos del gen Ccace2: 1) individuos con genotipo silvestre, el producto de PCR de 172 pb fue digerido por Bst NI y se visualizó una sola banda de 145 pb, ya que el fragmento de 27 pb no se visualizó con nitidez en el gel de agarosa debido a su pequeño tamaño; 2) individuos homocigotos para la mutación AChE G328A, la enzima Bst NI no digirió el producto de PCR y se visualizó una sola banda de 172 bp; 3) individuos heterocigotos, se visualizaron las dos bandas anteriormente descritas de 172 pb y 145 pb correspondientes al alelo silvestre y al de resistencia (Fig. 4.3.3). Ccace2S Ccace2S Ccace2R Ccace2S M Ccace2R Ccace2R 172 pb 145 pb Figura 4.3.3. Método PCR-RFLP para la detección del alelo Ccace2R. Los productos del RFLP correspondientes a los tres posibles genotipos para Ccace2 se migraron en un gel de agarosa al 3% en TAE 1X. Se empleó un marcador molecular en escalera de 100 pb (M). 123 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ Este método de detección de la mutación puntual AChE G328A resultó ser rápido, ya que en un día se pudieron analizar hasta cuarenta insectos, desde la extracción de DNA genómico de la cabeza hasta la interpretación del perfil de restricción para identificar el genotipo del individuo para Ccace2. Además, el análisis PCR-RFLP se realizó a partir de DNA extraído tanto de insectos adultos completos, como de larvas y de pupas, obteniendo en todos los casos resultados satisfactorios. 4.3.5. Distribución geográfica del alelo Ccace2R. Mediante el método de detección PCR-RFLP explicado anteriormente se determinó la frecuencia del alelo Ccace2R en poblaciones de varias áreas geográficas. Se analizaron 27 poblaciones españolas y 11 poblaciones extranjeras de los cinco continentes. La figura 4.3.4.a muestra un ejemplo representativo de los análisis de PCR-RFLP realizados para determinar la distribución del alelo Ccace2R en las diferentes poblaciones, mostrando los perfiles de restricción de varios individuos de las poblaciones de Torre de Benagalbón (Málaga), Tortosa (Tarragona), Les Coves de Vinromà (Castellón) y Moncada (Valencia). Además, para evaluar la posible aplicación del método de detección de la mutación AChE G328A en insectos procedentes de trampas, se incluyó en este estudio la población Sagunt (Valencia), donde se analizaron moscas recogidas en una trampa Moskisan® que podían llevar muertas en la trampa hasta siete días (ver apartado 4.2.3). El análisis de la población de Sagunt (Valencia) se muestra en el figura 4.3.4.b. En ambas imágenes se identifica el genotipo de cada uno de los individuos analizados mediante el método PCR-RFLP para la detección del alelo Ccace2R, que codifica para la mutación AChE G328A y que confiere resistencia frente a malatión en la mosca mediterránea de la fruta C. capitata. 124 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ (a) MAG M TOR COV MON S-S S-S S-S S-S S-S S-S R-S R-S R-R R-S S-S R-R R-S R-S R-R S-S R-R R-S S-S R-S S-S S-S R-S M 200 pb 100 pb (b) R-S R-S R-R S-S R-R S-S S-S S-S M 172 pb 145 pb Figura 4.3.4. Análisis por PCR-RFLP del alelo Ccace2R en poblaciones de campo de C. capitata. Los productos de PCR-RFLP se migraron en un gel de agarosa al 3% en TAE 1X. Se empleó un marcador molecular en escalera de 100 pb (M). Para facilitar la compresión del esquema, Ccace2S se abrevia como “S” y Ccace2R como “R”. (a) Ejemplo de varios individuos de las poblaciones de Torre de Benagalbón, Málaga (MAG); Tortosa, Tarragona (TOR); Les Coves de Vinromà, Castellón (COV) y Moncada, Valencia (MON). (b) Individuos recolectados en trampas en la población de Sagunt, Valencia (SAG). 125 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ El análisis de las 27 poblaciones españolas mediante el método PCR-RFLP para la detección de la mutación AChE G328A mostró que el alelo Ccace2R, que confiere resistencia a malatión, se encontró en al menos un individuo de todas las poblaciones analizadas (Tabla 4.3.2 y Fig. 4.3.5), con las únicas excepciones de Torre de Benagalbón (Málaga) y Marratxí (Mallorca), lo que supone que el alelo Ccace2R estuvo presente en el 93% de las poblaciones analizadas. Albelda (04) Villalengua (07) Girona (04) Tortosa (03) Amposta (03) Alcanar (07) Les Coves de Vinromà (03) Vila Real (05) Burriana (04) Muro (07) Marratxí (07) Alcúdia (04) Sagunt (08) Gibraleón (08) Serra (05) Moncada (03) Ayamonte (08) Carlet (04) Xàbia (07) Vila Joiosa (07) Torre de Benagalbón (03) Algarrobo Costa (08) Almuñécar (07) Orihuela (05) Alacant (04) Valle de Guerra (04) Figura 4.3.5. Distribución geográfica del alelo Ccace2R en España. Localización de las poblaciones españolas analizadas. El sector negro de cada círculo representa el porcentaje de individuos que poseen el alelo Ccace2R. Entre paréntesis, a continuación del nombre de cada población, se indica el año de recogida. En las poblaciones de Gibraleón (Huelva), Almuñécar (Granada), Algarrobo Costa (Málaga) y Villalengua (Zaragoza) se registraron las menores frecuencias del alelo de resistencia, donde el número de individuos que portaban el alelo Ccace2R no superó el 10% (Tabla 4.3.2). Las poblaciones que exhibieron un mayor porcentaje de individuos con el alelo Ccace2R fueron Les Coves de Vinromà (90%) en Castellón y Amposta (85%) en Tarragona, seguidas de Valle de Guerra (80%) en Tenerife (Tabla 4.3.2). En estas cuatro poblaciones, la proporción del alelo Ccace2R con respecto a Ccace2S fue mayor o igual al 50%. Además, en 16 de las 25 poblaciones en las que se detectó el alelo Ccace2R el porcentaje de dicho alelo fue mayor o igual al 20% (Fig. 4.3.6). 126 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia C. Valenciana Cataluña Aragón Andalucía CC.AA. AYA GIB MAG Ayamonte Gibraleón Torre de Benagalbón ALD BUR CAR COV Burriana Carlet Les Coves de Vinromà TOR Tortosa Alcùdia GER Girona ALI AMP Amposta Alacant ALC Alcanar VIG ALM Villalengua MAY Almuñécar CÓD. Algarrobo Costa Población 10 80 55 58 74 25 35 15 60 92 0 95 90 95 95 Ccace2 S Ccace2 S 127 70 20 35 42 22 55 50 70 40 8 0 5 10 5 5 R Ccace2 S Ccace2 20 0 10 0 4 20 15 15 0 0 0 0 0 0 0 R Ccace2 R Ccace2 ___________________________________________________________________________________________________________ Genotipo (%) Tabla 4.3.2. Presencia del alelo Ccace2R en poblaciones españolas de C. capitata. 55 10 28 21 15 48 40 50 20 4 0 3 5 3 3 % alelo R Ccace2 90 20 45 42 26 75 65 85 40 8 0 5 10 5 5 % individuos con R alelo Ccace2 _____________________________________________________________________________________________________________________________________________________________________________________________________________ 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia VIL JAV Vila Real Xàbia Albelda Valle de Guerra ALB VAG MUR VIY Vila Joiosa Muro SER Serra MAR SAG Sagunt Marratxí MON ORI Orihuela CÓD. Moncada Población 56 20 80 0 40 35 90 40 45 55 30 Ccace2 S Ccace2 50 44 45 20 0 40 60 10 55 45 40 R Ccace2 S Ccace2 0 35 0 0 20 5 0 5 10 5 20 R Ccace2 R Ccace2 22 58 10 0 40 35 5 33 33 25 45 % alelo R Ccace2 44 80 20 0 60 65 10 60 55 45 70 % individuos con R alelo Ccace2 128 ______________________________________________________________________________________________________________________________________________________________________________________________________ La Rioja I. Canarias I. Baleares C. Valenciana CC.AA. S ___________________________________________________________________________________________________________ Genotipo (%) Tabla 4.3.2. Presencia del alelo Ccace2R en poblaciones españolas de C. capitata. (Continuación). _____________________________________________________________________________________________________________________________________________________________________________________________________________ 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ Cast elló Valle de Guerr a Les Coves de Vinromà Ampost a Tort osa Moncada Xàbia Gir ona Vila Real Serr a Sagunt Bur riana Ccace2 Ccace2 Orihuela Albelda S R Alcùdia Alcanar Alacant Mur o Carlet Vila Joiosa Ayamont e Villalengua Gibraleón Almuñecar ´ Algar r obo Cost a Torr e de Benagalbón Mar rat xí 0% 20% 40% 60% 80% 100% a le lo s Figura 4.3.6. Porcentaje de alelos Ccace2S y Ccace2R en las poblaciones españolas de C. capitata analizadas. En cuanto a las poblaciones extranjeras analizadas, no se detectó el alelo Ccace2R, que contiene la mutación AChE G328A que confiere resistencia a malatión en C. capitata, en ninguno de los individuos analizados (Fig. 4.3.7). 129 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ Túnez (08) Grecia (08) Turquía (08) Marruecos (03) Isla de Madeira (04) Israel (04) Isla Reunión (08) Guatemala (08) Brasil (04) Australia (05) Sudáfrica (06) Figura 4.3.7. Distribución geográfica del alelo Ccace2R en las poblaciones no españolas. Situación de las poblaciones recogidas en once países de cinco continentes. El color blanco del círculo indica que la totalidad de los individuos analizados no portaban el alelo Ccace2R. b) Entre paréntesis, a continuación del nombre de cada población, se muestra el año de recogida. 4.3.6. Frecuencia genotípica de Ccace2 en las líneas de laboratorio resistentes a malatión. Con el objetivo de estudiar la evolución temporal del alelo Ccace2R en una población sujeta a una presión de selección se analizaron con el método PCR-RFLP descrito previamente (apartado 4.3.4) individuos adultos, recogidos antes del tratamiento con insecticida, de las líneas de laboratorio resistentes a malatión: W (F2 y F10), W-4Km (F37) y W-10Km (F52). En la generación F2 de la línea W la frecuencia de individuos heterocigotos fue del 56%, un 6% eran homocigotos silvestres y un 38% homocigotos para la mutación AChE G328A. Estas frecuencias serían representativas de las existentes en la población Castelló de la que proceden (Magaña et al., 2007), ya que las moscas no fueron tratadas con malatión en la primera generación que se mantuvo en el laboratorio. En la generación F10, donde la concentración de selección fue de 2.000 ppm de malatión (Tabla 2.3.3), el porcentaje de heterocigotos se incrementó hasta el 70%, disminuyendo a la mitad el porcentaje de homocigotos para Ccace2R. Los análisis realizados en la generación F37 de la línea W-4Km, donde la selección se realizaba con 4.000 ppm de malatión, 130 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ mostraron que la mayoría de los individuos eran heterocigotos (96%) y no se detectaron individuos homocigotos silvestres. Finalmente, en el análisis de la generación F52 de la línea W10Km, tratada con 10.000 ppm de malatión, se observó que la totalidad de los individuos analizados eran heterocigotos para el gen Ccace2 (Fig. 4.3.8). Ccace2S-Ccace2S Ccace2R-Ccace2S 56% 70% F2 F10 _________________________ W 96% F37 100% Ccace2R-Ccace2R F52 ___________ ___________ W-4Km W-10Km Figura 4.3.8. Frecuencia genotípica de Ccace2 en las líneas de laboratorio resistentes a malatión seleccionadas a partir de la población W. Proporción de homocigotos para el alelo silvestre Ccace2S, heterocigotos y homocigotos para el alelo resistente Ccace2R para el gen Ccace2 en las generaciones F2 y F10 de la línea W, F37 de la línea de laboratorio W-4Km y F52 de la línea W-10Km. Se analizaron un mínimo de 20 individuos en cada una de las generaciones. 131 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ 4.4. Discusión. El análisis de la estructura génica de la acetilcolinesterasa y de la aliesterasa de C. capitata ha confirmado que la disposición de intrones y exones estudiados de los genes Ccace2 y Ccae7 es idéntica a la que presentan los genes ortólogos en D. melanogaster (McQuilton et al., 2012). En cuanto al tamaño de los intrones, se ha observado que en ambas especies de dípteros el gen ae7 presenta algunos intrones de pequeño tamaño. Sin embargo, los intrones del gen Ccace2 presentaron, en general, un tamaño mayor, en correspondencia con lo descrito en el gen Dmace2 de D. melanogaster (McQuilton et al., 2012). Debido al gran tamaño de algunos de estos intrones no se ha obtenido la secuencia completa de todos ellos, pero sí una secuencia con un tamaño adecuado para el diseño, en C. capitata, de cebadores específicos para amplificar por PCR los exones de ace2 y ae7 relacionados con resistencia en otros insectos, partiendo de DNA genómico. El análisis de las mutaciones en los genes Ccace2 y Ccae7, previo a este trabajo, se realizó mediante la amplificación por PCR del cDNA, por lo que era necesario purificar el RNA (Magaña et al., 2008). La extracción de RNA sólo se puede realizar a partir de individuos vivos y/o conservados a -80ºC, factor que limitaba el análisis a los individuos disponibles en el laboratorio o a los que eran recogidos en campo y criopreservados hasta su estudio. Los cebadores diseñados en este trabajo permiten la amplificación y secuenciación de los exones a partir de DNA genómico, que es menos sensible a la degradación que el RNA, no siendo necesario criopreservar los individuos antes 133 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ de su análisis. Esta técnica permite incluso analizar, con resultados satisfactorios, individuos procedentes de trampas de captura que podían llevar muertos en la trampa una semana antes de su recogida. Los cebadores empleados para amplificar los exones de los genes Ccace2 y Ccae7 fueron diseñados a partir de secuencias intrónicas adyacentes a los exones. Además, algunos de los cebadores incluyeron parte de la secuencia del exón. Los cebadores fueron diseñados con la intención de que fueran útiles para poder amplificar distintos alelos de estos genes, pudiendo así detectar mutaciones en las regiones codificantes del gen. La eficacia de los cebadores para amplificar los exones de los genes Ccace2 y Ccae7 en poblaciones de diferentes regiones geográficas (España, Sudáfrica, Australia o Isla Reunión) confirma el elevado grado de conservación existente en las regiones escogidas para el diseño de los cebadores. La mutación puntual AChE G328A ha sido asociada con la aparición de resistencia al organofosforado malatión en poblaciones españolas de la mosca mediterránea de la fruta (Magaña et al., 2007, 2008). El diseño de un método de diagnóstico para identificar aquellos individuos portadores de la mutación aumentaría el conocimiento sobre la distribución y dispersión de la resistencia y ayudaría a diseñar la estrategia adecuada para su manejo. Nuestro objetivo fue poner a punto un método sencillo para la detección de la mutación AChE G328A. El método de diagnóstico seleccionado fue mediante PCR-RFLP, método empleado para la detección de alelos de resistencia a insecticidas en otros insectos (Weill et al., 2004; Cassanelli et al., 2005, 2006; Hawkes et al., 2005; Hsu et al., 2006). Sin embargo, la mutación puntual no generaba o modificaba un sitio de restricción que diferenciase el alelo silvestre del mutado, por lo que no se pudo realizar una PCR-RFLP convencional. En la detección de marcadores tumorales definidos por SNPs (del inglés single-nucleotide polymorphism) se emplea asiduamente una técnica descrita en 1989 por Haliassos y colaboradores en la que se modifica deliberadamente la secuencia de uno de los cebadores empleados en la PCR para crear un sitio de restricción que permita la identificación de los diferentes alelos. Esta técnica, denominada PIRA-PCR (del inglés primer-introduced restriction analysis PCR) (Jacobson y Moskovits, 1991), ha sido empleada recientemente en la detección de alelos que confieren resistencia a insecticidas en B. oleae, Anopheles gambiae y A. culicifacies (Margaritopoulos et al., 2008; Janeira et al., 2008; Singh et al., 2009, 2010). Basándonos en la secuencia conocida del exón 6 de la AChE (Magaña et al., 2008) y en la secuencia del intrón 5 del gen que codifica para la acetilcolinesterasa que obtuvimos en este estudio, diseñamos una pareja de cebadores que nos permite amplificar por PCR la región del gen Ccace2 que contiene la mutación AChE G328A, realizando en uno de los cebadores la correspondiente modificación para crear artificialmente un sitio de restricción. Así, este sistema PCR-RFLP nos permite detectar la presencia del alelo mutado Ccace2R sin la necesidad de realizar bioensayos, tanto en insectos 134 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ adultos, como en larvas o pupas. Además, este método molecular nos permite detectar el alelo de resistencia aun cuando se encuentre a baja frecuencia en la población, hecho que no lograríamos mediante la realización de bioensayos de susceptibilidad. Se logró amplificar por PCR la región que contiene la mutación puntual AChE G328A en todas las poblaciones analizadas (españolas y de once países de cinco continentes), por lo que se valida el método de detección para la especie, independientemente de la posible variabilidad alélica dependiente de la región geográfica. Hasta la fecha sólo se han descrito poblaciones de C. capitata resistentes a malatión en España, posiblemente debido al elevado número de tratamientos con malatión aplicados frente a esta plaga en los campos españoles durante varios años (Magaña et al., 2007). Mediante el método PCR-RFLP desarrollado hemos confirmado la presencia del alelo Ccace2R en prácticamente la totalidad de las poblaciones españolas analizadas y su ausencia en las once poblaciones de C. capitata recogidas en otros países. La elevada frecuencia de individuos que portaron el alelo Ccace2R en determinadas poblaciones podría estar relacionada con el uso del malatión, ya que se observa que las mayores frecuencias del alelo mutado se encuentran en la Comunidad Valenciana y Cataluña, regiones que tradicionalmente han sufrido las mayores intensidades de tratamiento con malatión para intentar disminuir los daños ocasionados por C. capitata en los cítricos y otros frutales. El grado de expansión del alelo Ccace2R se muestra con la detección de individuos heterocigotos para la mutación AChE G328A en el análisis de la población recogida en la finca experimental del CSIC de La Mayora, situada en el municipio de Algarrobo-Costa (Málaga), a pesar de que en esta finca no se aplican insecticidas dirigidos frente a la mosca mediterránea de la fruta. Además, el alelo resistente se ha encontrado en poblaciones de las Islas Canarias y Baleares. El resultado positivo obtenido en el análisis de los individuos procedentes de las trampas del municipio de Sagunt (Valencia) demuestra que es posible la implementación del sistema de detección PCR-RFLP en el sistema de monitoreo para el seguimiento de la abundancia de la plaga. No obstante, a la hora de analizar con el método PCR-RFLP individuos recogidos en trampas situadas en zonas donde se realizan sueltas masivas de machos estériles, sería conveniente analizar exclusivamente hembras. La implementación del sistema de detección del alelo de resistencia en el monitoreo de poblaciones permitirá conocer la frecuencia genotípica del alelo Ccace2R en una determinada zona a lo largo de un periodo de tiempo y estudiar cómo varía dicha frecuencia después de la aplicación de un insecticida, observando si ha sido eficaz el método de control aplicado a esta plaga. Si se retomase la utilización del malatión o de otro organofosforado para el control de C. capitata en España, sería conveniente realizar un seguimiento de la frecuencia del alelo de resistencia Ccace2R en las poblaciones de campo. Es importante destacar que, en algunas de las poblaciones de C. capitata que hemos analizado, la frecuencia del alelo Ccace2R ha llegado a ser superior al 50%, valor que se considera 135 4. Determinación de la estructura de los genes ccace2 y ccae7 y desarrollo de sistemas de detección precoz de alelos de resistencia ______________________________________________________________________________________________________________________________________ elevado y que es habitual en las mutaciones relacionadas con la resistencia a piretroides en el canal de sodio en mosquitos (Reimer et al., 2005; Santolamazza et al., 2008; Sarkar et al., 2009; Kawada et al., 2011; Antonio-Nkondjio et al., 2011) y en la mutación A296S del receptor GABA en poblaciones de Anopheles funestus (Wondji et al., 2011). En relación a los alelos de la acetilcolinesterasa que confieren resistencia a insecticidas, únicamente se han descrito frecuencias superiores al 50% en poblaciones de A. gambiae en Burkina Faso (Dabiré et al., 2009) y de B. oleae en Grecia (Margaritopoulos et al., 2008). En otros estudios donde se ha estimado la frecuencia de los alelos de resistencia en genes ace se han encontrado frecuencias más bajas, como por ejemplo en H. irritans (Li et al., 2003), en varias especies del género Anopheles y Culex (Reimer et al., 2005; Djogbénou et al., 2008; Santolamazza et al., 2008; Sarkar et al., 2009; Alou et al., 2010; Verhaeghen et al., 2010; Kawada et al., 2011; Antonio-Nkondjio et al., 2011; Wondji et al., 2011) y en poblaciones de B. oleae (Nardi et al., 2006). Aunque el uso de malatión se prohibió desde el final de la campaña de 2008, actualmente se ha incorporado de nuevo al anexo I de la Directiva Europea 91/414/ECC, permitiendo su empleo en la Unión Europea pero, hasta el momento, no ha sido aprobado en España su uso específico frente a C. capitata. El hecho de que la frecuencia del alelo Ccace2R sea tan elevada y que la distribución geográfica de dicho alelo sea muy amplia, localizado en multitud de regiones de la Península Ibérica, en las Islas Baleares y Canarias, desaconseja el empleo de cualquier tipo de insecticida organofosforado o carbamato para el control químico de la mosca mediterránea de la fruta en España si no va acompañado de un manejo de la resistencia adecuado. De acuerdo con nuestro método de detección PCR-RFLP, la línea W-10Km está formada únicamente por individuos heterocigotos para la mutación AChE G328A. De igual modo, durante el proceso de selección de la línea W-4Km, se observó una disminución de los homocigotos para el alelo de resistencia y un incremento del porcentaje de individuos heterocigotos (96% en la generación F37). Lo esperable en una línea seleccionada es que los homocigotos susceptibles a la selección tiendan a desaparecer, incrementándose así la proporción del alelo de resistencia con respecto al susceptible. Además, según lo establecido por la herencia mendeliana clásica, la existencia de una población formada únicamente por individuos heterocigotos para un determinado gen con dos alelos es difícilmente explicable. Estas cuestiones, así como el estudio de la línea altamente resistente a malatión W-10Km se discutirán en el siguiente capítulo de esta tesis. 136 Capítulo 5 NUEVO MECANISMO DE RESISTENCIA A MALATIÓN EN Ceratitis capitata 137 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ 5.1. Introducción. La acetilcolinesterasa (AChE) es una enzima implicada en la sinapsis colinérgica durante la transmisión del impulso nervioso (Fournier, 2005). Los insecticidas organofosforados y carbamatos inhiben esta enzima mediante el bloqueo de su centro activo, por lo que cualquier modificación estructural en la AChE que impida o dificulte la unión de los insecticidas a dicho centro activo favorecerá la aparición de resistencia. Estas modificaciones en la AChE se producen debido a mutaciones puntuales en el gen ace que codifica la AChE. Las mutaciones puntuales, situadas en el centro activo de la enzima o en sus proximidades, crean impedimentos estéricos que dificultan la entrada del insecticida en el centro activo de la AChE (Mutero et al., 1994; Walsh et al., 2001; Oakeshott et al., 2005). En Ceratitis capitata se ha relacionado la mutación puntual AChE G328A con la resistencia al organofosforado malatión (Magaña et al., 2008). En el capítulo anterior hemos descrito un sistema PCR-RFLP para la detección del alelo de resistencia Ccace2R que porta esta mutación. Además, el análisis realizado en varias poblaciones de campo mediante un método PCR-RFLP mostró que la distribución geográfica del alelo Ccace2R es amplia en todo el territorio español, sin embargo, este alelo de resistencia no se detectó en otros países. 139 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ El análisis de la frecuencia del alelo Ccace2R en las líneas de laboratorio resistentes a malatión W-4Km y W-10Km (seleccionadas con 4.000 ppm y 10.000 ppm de malatión, respectivamente) mostró que se había producido un incremento en la frecuencia del alelo Ccace2R con respecto al alelo silvestre Ccace2s a lo largo del proceso de selección. Además, conforme se sucedieron las generaciones en las líneas resistentes a malatión, se observó un desequilibrio a favor de los individuos heterocigotos para estos dos alelos. Así, según el método PCR-RFLP, el análisis de los individuos de la línea W-10Km, realizado antes del tratamiento con malatión, mostró que la totalidad de los individuos a partir de la generación F52 eran heterocigotos. Estos resultados contradicen la segregación mendeliana esperada para un gen con dos alelos cuando se producen apareamientos al azar en ausencia de selección (Griffiths et al., 2008), ya que en la descendencia del cruzamiento de individuos heterocigotos sería esperable observar tanto individuos heterocigotos como individuos homocigotos para ambos alelos. A principios de los años noventa se describieron los primeros casos en los que se relacionaban las duplicaciones génicas con resistencia a insecticidas en varias especies de insectos (Devonshire y Field, 1991). En las dos últimas décadas se ha incrementado el número de casos de artrópodos en los que las duplicaciones producen un incremento en la síntesis de enzimas relacionadas con la resistencia metabólica y con la resistencia mediada por modificaciones en la molécula diana, sin embargo el número de estudios no supera la decena (Bass y Field, 2011). Recientemente, en mosquitos, se han descrito varios casos de duplicaciones del gen que codifica la acetilcolinesterasa (ace1, nomenclatura según Weill et al., 2002) relacionadas con resistencia a organofosforados en Culex pipiens (Bourguet et al., 1996a, 1996b; Labbé et al., 2007a; Alout et al., 2009; Ben Cheikh et al., 2009) y en Anopheles gambiae (Djogbénou et al., 2009). Concretamente, en C. pipiens (Labbé et al., 2007a, 2007b) se han descrito dos eventos de duplicación independientes en diferentes poblaciones del mosquito y que originan la presencia de dos alelos del gen ace1, el mutado y el silvestre, en un mismo cromosoma. Esta duplicación génica provocaría una heterocigosis permanente (Ccace2RS) (Labbé et al., 2007a). Hay que tener en cuenta que la adquisición de la resistencia provocada por una mutación puntual en la AChE conlleva un coste biológico asociado, ya que dicho gen codifica una AChE menos susceptible de ser inhibida por el insecticida pero, al mismo tiempo, es menos eficiente a la hora de hidrolizar la acetilcolina, sustrato indispensable para la transmisión del impulso nervioso. Se ha sugerido que en los individuos que portan la duplicación anteriormente descrita se reduciría el coste biológico, normalmente asociado a la presencia del alelo de resistencia, ya que el aumento del número de copias del gen compensaría la menor eficiencia de la enzima mutada (Labbé et al., 2007a). En este capítulo analizaremos la posible existencia de una duplicación del gen que codifica la acetilcolinesterasa en C. capitata, similar a la encontrada anteriormente en el mosquito C. 140 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ pipiens (Labbé et al., 2007a). Esta duplicación, que porta en el mismo cromosoma el alelo silvestre y el alelo de resistencia, representaría un nuevo mecanismo de resistencia a malatión en C. capitata. 141 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ 5.2. Material y métodos. 5.2.1. Líneas de laboratorio y cruzamientos. En este estudio se emplearon individuos de la línea de laboratorio resistente a malatión W10Km y de la línea susceptible C (descritas en el apartado 2.2.2). Además, en los experimentos en los que se indica, se utilizaron insectos de una isolínea (proveniente de una sola pareja), obtenida a partir de la línea W-4Km, constituida por individuos homocigotos para el alelo Ccace2R y denominada de aquí en adelante isolínea R/R. En el experimento realizado para demostrar la existencia de una duplicación del gen Ccace2 que provoca heterocigosis permanente (Labbé et al., 2007a), se analizó la descendencia del cruzamiento de la línea susceptible C con la línea resistente a malatión W-10Km. En el interior de unas cajas similares a las empleadas en los bioensayos de susceptibilidad a insecticidas (Fig. 2.2.3) se aislaron un macho y una hembra de ambas líneas. La puesta se recogió en un recipiente inferior que contenía agua (Fig. 5.2.1) y posteriormente se prosiguió con el protocolo de cría descrito en el apartado 2.2.1.1. Inicialmente se emplearon un total de veinte parejas, diez para cada dirección del cruzamiento. Sólo algunas hembras iniciaron la puesta y, de éstas, solo algunas pusieron huevos fértiles. Se seleccionaron para el experimento aquellas tres parejas en cada 143 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ dirección del cruzamiento con más de quince pupas en su descendencia, seis parejas en total. Los doce individuos parentales que formaban las parejas seleccionadas se genotiparon mediante el sistema de PCR-RFLP descrito en el capítulo anterior (apartado 4.2.3) para comprobar que los cruces se habían realizado conforme a lo esperado; es decir, que los individuos de la línea C presentaban un perfil de restricción homocigoto para el alelo silvestre y que los individuos de W10Km eran heterocigotos. Con el objeto de determinar si los dos alelos segregaban según la herencia mendeliana clásica se genotiparon, además de los parentales, un total de ochenta individuos de la descendencia. De cada una de las seis parejas se seleccionaron al azar entre diez y quince individuos, lo que representaba entre un 40% y un 100% de la descendencia de cada pareja. a b c Figura 5.2.1. Cajas para los cruzamientos. (a) Sistema de cajas empleado para la obtención de isolíneas y de cruzamientos dirigidos. (b) Los huevos se recogieron en el recipiente inferior, que contenía agua. (c) Detalle de los huevos depositados por la hembra a través de la rejilla Además, se realizaron otros cruzamientos entre las líneas de laboratorio W-10Km, C y la isolínea R/R pero, en estos casos, la mezcla de líneas se llevó a cabo introduciendo en dos jaulas de cría 25 machos de una línea con 25 hembras de la otra en ambas direcciones de cruzamiento. La puesta de las dos jaulas se recogió y se mantuvo según el protocolo de cría en laboratorio descrito en el apartado 2.2.1.1. 144 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ 5.2.2. Evaluación del número de copias del gen Ccace2. Con el objeto de identificar la presencia de una duplicación del gen que codifica la acetilcolinesterasa en la línea resistente W-10Km se evaluó el número de copias de Ccace2, mediante PCR cuantitativa en tiempo real (qPCR), tomando como molde DNA genómico. Se utilizaron como genes de referencia para la qPCR el gen que codifica la superóxidodismutasa (sod, ref. Genbank L35494) y el gen de la proteína ribosomal P0 (Ccpo, ref. GenBank Y18444). La qPCR se realizó en un termociclador Rotor Gene 6000 Corbett Life Science (Qiagen, Foster City, California, EE.UU.) tomando como molde 1 ng de DNA genómico. La reacción de PCR contenía 7,5 µl de Brilliant III Ultra-Fast SYBR® Green qPCR MasterMix (Agilent Technologies, California, EE.UU.), 1,25 µl de cada cebador 3,6 µM, para un volumen total de reacción de 15 µl. El ciclo de amplificación para todos los genes fue el siguiente: inicialmente 95ºC durante 10 min para desnaturalizar el DNA y a continuación se realizó cuarenta veces el ciclo 95ºC 20s, 58ºC 20s y 72ºC 20s. Las características de los cebadores empleados y su eficiencia se muestran en las tablas 5.2.1 y 5.2.2. Tabla 5.2.1. Cebadores empleados en la determinación del número de copias de Ccace2 y en la estimación de su expresión. Se muestra el nombre del cebador, su tamaño en bases (b) y su secuencia nucleotídica. El tamaño del amplicón originado en la qPCR por la pareja de cebadores directo (F) y reverso (R), se indica en pares de bases (pb). Gen Cebador b Secuencia (5’ Ccace2 FAChE4qPCR 20 GAAATCCGCAAAACACCACA RAChE4qPCR 18 CGGTCATAAAGCCACCAC FP0_2 20 TCCAGGCTCTCTCCATACCA RP0 20 CGACTTTGTCACCAGGCTTC FSOD 20 TCCCGTAGTGAAGGGTAATG RSOD 19 CGGTGTCAAGCCAGTCAAA Ccp0 Sod 3’) Amplicón (pb) 69 90 88 _______________________________________________________________________________________ Inicialmente se comparó el número de copias de Ccace2 presente en los individuos de la línea susceptible C con los de la línea resistente a malatión W-10km, analizando seis individuos de cada una de ellas. En experimentos posteriores, siguiendo el mismo procedimiento, se analizaron entre cuatro y seis individuos de los cuatro posibles genotipos restantes para Ccace2, obtenidos de los descendientes del cruce de las líneas de laboratorio W-10Km, C y de la isolínea R/R. Finalmente 145 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ se estudiaron varios individuos de dos poblaciones de campo analizadas mediante el método PCRRFLP en el capítulo anterior (Tabla 4.3.2): Orihuela (Alicante) y Alcanar (Tarragona). Tabla 5.2.2. Eficiencia de los cebadores empleados en la determinación del número de copias de Ccace2 y en la estimación de su expresión. Se indica el coeficiente de determinación de la recta patrón de amplificación (R2) y el porcentaje de eficiencia de la pareja de cebadores, según el molde empleado en la qPCR. 2 Gen Molde R Ccace2 DNA 0,999 113 cDNA 0,996 117 DNA 0,995 116 cDNA 0,999 101 DNA 0,998 113 Ccp0 Sod Eficiencia (%) El número de copias en el genoma de cada gen se calculó siguiendo el método de ∆Ct (Pfaffl, 2001), realizando la media aritmética de la cantidad de copias estimada para los individuos de cada línea. La cantidad de copias obtenida para el gen Ccace2 se normalizó con respecto a la cantidad de copias del gen Ccp0 y del gen sod. Con el programa SPSS v17.0 se realizó un test de la t de Student o un ANOVA, según corresponda, para comparar la cantidad de copias del gen Ccace2 presentes en los diferentes grupos de insectos. 5.2.3. Cuantificación de la expresión de Ccace2. Con el objetivo de cuantificar la expresión del gen Ccace2 en la línea resistente a malatión W-10Km y en la línea susceptible C, se evaluaron los niveles de expresión de Ccace2 mediante qPCR, tomando como referencia la expresión del gen Ccp0, que codifica la proteína ribosomal P0. En cada línea se analizaron un total de treinta individuos adultos, de entre tres y cinco días de edad, divididos en cinco grupos de seis insectos, conteniendo cada uno de los grupos tres machos y tres hembras de cada línea. El análisis de expresión se realizó a partir del tórax y del abdomen, mientras que las cabezas fueron almacenadas a -80ºC para su utilización en los ensayos de actividad enzimática (ver más adelante). El RNA total (RNAT) de cada grupo de individuos se 146 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ extrajo mediante TRIzol® Reagent (Life Technologies, Van Allen Way, Carlsbad, California, EE.UU.) siguiendo las instrucciones del fabricante. La concentración del RNAT se midió con un NanoDrop (Thermo Scientific, Massachusetts, EE.UU.). La primera cadena de cDNA se sintetizó a partir de 1 µg de RNAT utilizando iScript cDNA Syntesis Kit (Bio-Rad Laboratories, California, EE.UU.) siguiendo las instrucciones del fabricante. La reacción de amplificación a tiempo real se realizó en un termociclador con detector continuo de fluorescencia Rotor Gene 6000 Corbett Life Science (Qiagen, Foster City, California, EE.UU.) con los cebadores descritos en la tabla 5.2.1. La mezcla de la reacción de PCR (15 µl) contenía 7,5 µl de Brilliant III Ultra-Fast SYBR® Green qPCR MasterMix (Agilent Technologies, California, EE.UU.), 1,25 µl de cada cebador 3,6 µM y aproximadamente 8 ng de cDNA. El ciclo de amplificación fue el siguiente: inicialmente 95ºC durante 10 min para desnaturalizar el cDNA y a continuación se realizó cuarenta veces el ciclo 95ºC 20 s, 58ºC 20s y 72ºC 20s. La expresión de ambos genes se estimó siguiendo el método de ∆Ct (Pfaffl, 2001), normalizando la expresión de Ccace2 con la expresión del gen Ccp0. El valor final de la expresión de Ccace2 en cada línea se calculó realizando la media aritmética de las expresiones obtenidas en los cinco grupos de individuos. Finalmente se realizó un test t de Student con el programa SPSS v17.0 para comparar la expresión del gen Ccace2 en las líneas de laboratorio C y W-10Km. 5.2.4. Análisis de la actividad AChE y su inhibición por malaoxón. Se estimó la actividad AChE en la línea resistente a malatión W-10Km y en la línea susceptible C empleando para ello las cabezas de los individuos en los que previamente se había analizado la expresión del gen Ccace2. Cada uno de los cinco grupos de seis cabezas (tres de machos y tres de hembras) se disgregaron en 900 µl de una disolución de homogeneización (tampón fosfato Na 0,1M pH 7, 1% Tritón X-100 v/v). La suspensión proteica se centrifugó a 4ºC durante 10 min a 20.000 g en una centrífuga Universal 32 R (Hettich, Massachusetts, EE.UU.). El homogeneizado empleado en los ensayos enzimáticos se correspondió con la fase superior sobrenadante después de la centrifugación. La cantidad total de proteína en el homogeneizado se calculó según el método de Bradford (1976), utilizando como proteína estándar albúmina de suero bovino (Sigma Chemical Co., Missouri, EE.UU.). Las diluciones seriadas del estándar se prepararon en el mismo tipo de disolución en la que las muestras fueron homogeneizadas (Friednauer y Berlet, 1989). La actividad AChE se evaluó por el método espectrofotométrico descrito por Ellman et al. (1961). Se realizaron tres réplicas experimentales de cada medida de absorbancia de los 147 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ homogeneizados y se compararon con los pocillos sin muestra (blancos). Los 100 µl de mezcla de reacción en tampón fosfato pH 7 contenían 5 µl de homogeneizado, ATChI 2mM (acetiltiocolina yodada, Sigma Chemical Co., Missouri, EE.UU.) y DTNB 1 mM (5,5 ditio-bis-(ácido 2nitrobenzoico)), Sigma Chemical Co., Missouri, EE.UU.). El incremento de absorbancia se midió a 30ºC durante 5 minutos a una longitud de onda de 412 nm. La actividad AChE medida en mOD/min se convirtió a nanomoles de acetilcolina hidrolizada por minuto y por miligramo de proteína empleando el coeficiente de extinción de producto de Ellman (ε412= 1,36·104 M-1 cm-1) (Ellman, 1959). En los ensayos de inhibición de la actividad AChE se emplearon los mismos homogeneizados de cabeza de las líneas de laboratorio C y W-10Km que en la medición de la propia actividad enzimática. El agente inhibidor de la actividad AChE empleado fue el malaoxón Pestanal® al 94% (Ref. 36142, Riedel de Haën, Seelze, Alemania), diluido en etanol absoluto (Merck, Darmstadt, Alemania). Los homogeneizados de cabezas de ambas líneas se incubaron en las siguientes concentraciones de malaoxón: 3·10-5 M, 1·10-5 M, 3·10-6 M, 1·10-6 M, 3·10-7 M. Además, en el análisis del homogeneizado de la línea W-10Km se evaluó una concentración adicional de inhibidor de 1·10-4 M. Se analizó la variación de la actividad AChE en cada una de las concentraciones a los 10, 20, 30, 40 y 50 segundos, deteniendo la reacción mediante la adición de sustrato (ATChI) en exceso. Se realizaron tres réplicas experimentales de cada una de las concentraciones de malaoxón evaluadas y se midió la actividad AChE tal y como se describe en el párrafo anterior. La constante de inhibición (Ki) para los homogeneizados de las dos líneas de laboratorio analizadas se calculó siguiendo la metodología propuesta por Main (1964). 5.2.5. Caracterización de los perfiles RFLP. El método PCR-RFLP con la enzima Bst NI para la detección de la mutación AChE G328A (apartado 4.2.3) se empleó en el presente capítulo para genotipar tanto los individuos parentales como la descendencia de los diferentes cruzamientos realizados (ver apartado 5.2.1). Además, se realizó una caracterización de los patrones de restricción obtenidos mediante el sistema de documentación de geles Gel Doc XR (Bio-Rad Laboratories, California, EE.UU.) y con el programa Quantity One® v4.6.5 (Bio-Rad Laboratories, California, EE.UU.). Este programa analiza las imágenes de los geles y cuantifica la intensidad de las dos bandas, asignándole a cada una de ellas un porcentaje de intensidad en tanto por uno. Esta caracterización del perfil de restricción se realizó con el objetivo de, teniendo en cuenta la posible existencia de la duplicación, asociar un 148 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ determinado patrón RFLP con un determinado genotipo. Se analizaron individuos de las líneas de laboratorio C y W-10Km, e insectos de tres poblaciones de campo recogidas en los municipios de Les Coves de Vinromà (Castellón), Amposta y Tortosa (Tarragona) (Tabla 4.2.5). 5.2.6. Análisis de la mutación AChE G328A mediante secuenciación. Con la finalidad de asociar un patrón de secuenciación a cada uno de los genotipos posibles para Ccace2, asumiendo la existencia de la duplicación cromosómica, se analizaron los cromatogramas de aquellos individuos portadores de la duplicación tanto en homocigosis (Ccace2RS/RS) como en heterocigosis (Ccace2RS/R, Ccace2RS/S). Además, se estudiaron individuos de la línea susceptible C (Ccace2S/S) y la F1 del cruce de la línea C con la isolínea R/R, cuyo genotipo esperado sería Ccace2R/S. En primer lugar, se realizó la amplificación por PCR del fragmento de DNA que contiene el sitio puntual en el que se localiza la mutación AChE G328A. El amplicón se obtuvo empleando los cebadores FG328A_27 y RiAChE_CcE60, tal y como se describe en el apartado 4.2.3. En segundo lugar, los productos de PCR fueron enviados a Secugen S. L. (Madrid) donde se analizaron en un secuenciador automático ABI PRISM 377 (Applied Biosystems, California, EE.UU.), utilizando el cebador reverso RiAChE_CcE60 en la reacción de secuenciación. Los cromatogramas se visualizaron con el programa Chromas 1.45 (Griffith University, Queensland, Australia). 5.2.7. Método PCR-RFLP para la detección de la duplicación Ccace2RS. En el capítulo anterior se describió el diseño de un método PCR-RFLP para la detección de la mutación que confiere resistencia a malatión en C. capitata mediante el corte con la enzima Bst NI de la secuencia correspondiente al alelo silvestre (apartado 4.2.3). Además, en el análisis de los perfiles de restricción de determinados individuos heterocigotos para la mutación AChE G328A se observó que existían distintas intensidades en las bandas originas en el RFLP (ejemplo, calles 1 y 2 149 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ de la figura 4.3.4.B), hecho que podría estar relacionado con la presencia de la duplicación. Con el fin de diseñar un sistema de detección de la duplicación, se incorporó una segunda reacción al método PCR-RFLP (apartado 4.2.3) que detectaba, en este caso, el alelo que confiere resistencia a malatión. Para ello, se diseñó un nuevo cebador directo FG328A_27TseI (5’-TCA ACA TGT TTT TCA TTT TCG TTG CAG-3’), localizado en la misma posición que el cebador FG328A_27 descrito en el apartado 4.2.3. Este cebador posee una modificación en su extremo 3’ que produce una diana de restricción para la enzima Ape KI (New England Biolabs, Massachusetts, EE.UU.) en el producto amplificado cuando existe la mutación AChE G328A. El resultado teórico esperado del RFLP se muestra en el esquema de la figura 5.2.2. Ape KI Bst NI PCR-RFLP PCR PCR-RFLP PCR 172 pb 145 pb SS RS RR Fenotipo Ccace2 27 pb (liberación del SS RS RR cebador) Fenotipo Ccace2 Figura 5.2.2. Representación esquemática del método PCR-RFLP para la detección de la duplicación génica de Ccace2 en C. capitata. (Izquierda) Perfil de restricción esperado en la nueva reacción de restricción con la enzima Ape KI que produce la liberación del cebador en presencia del alelo R (Ccace2R). (Derecha) Recordatorio del corte producido por la enzima Bst NI (apartado 4.2.3). La reacción de PCR se realizó en un termociclador Gene Amp® PCR System 2700 (Applied Biosystem, Foster City, EE.UU.) con 2 unidades de la enzima polimerasa Amplitaq Gold® (Roche Molecular Systems, Inc., New Jersey, EE.UU.) a partir de un molde de 30 ng de DNA, empleando los cebadores FG328A_27TseI y RiAChEE_CcE60 en un volumen final de 10 µl de reacción. El ciclo de la reacción de PCR fue el siguiente: 95ºC 10 min, seguidos de un ciclo de 95ºC 30s, 48ºC 30s, 72ºC 15s que se repitió cuarenta veces, finalizando con un periodo de 72ºC durante 7 min. Una vez concluida la reacción, al producto de PCR se añadió una mezcla de restricción de 2 µl que contenía 0,05 unidades de la enzima de restricción Ape KI con el buffer NE3 recomendado por el 150 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ fabricante. La mezcla de producto de PCR y enzima de restricción se incubó durante 2 horas a 75ºC en el mismo termociclador empleado para la PCR. La migración electroforética del producto de restricción se realizó en un gel de agarosa al 3% en TAE 1X (Tris 40mM, EDTA 1mM pH 8.0). La composición del gel fue de 50% agarosa D-2 (Lab. Conda, Torrejón de Ardoz, Madrid, España) y 50% de NuSieve® GTG® Agarose (Lonza, Maine, EE.UU). La comparación de los perfiles de PCR-RFLP obtenidos utilizando, bien los cebadores FG328A_27 / RiAChEE_CcE60 en combinación con la enzima Ape KI (corta en ausencia de la mutación, descrito en apartado 4.2.3), bien los cebadores FG328A_27TseI / RiAChEE_CcE60 en combinación con la enzima Bst NI (corta en presencia de la mutación, descrito en este apartado), nos permitirá identificar la duplicación cuando se encuentre en uno de los dos cromosomas de un individuo. 5.2.8. Estimación del tiempo de desarrollo, porcentaje de emergencia y supervivencia de los adultos pertenecientes a las líneas C y W-10Km. En jaulas de cría independientes se depositaron pupas de la línea C (susceptible) y W10Km (resistente a malatión). Transcurridos 5-7 días desde la emergencia del primer adulto, previa eliminación de los huevos puestos con anterioridad, se recogieron de ambas jaulas los huevos depositados por las hembras durante 24 horas. Las puestas se sembraron en dieta y se mantuvieron en las condiciones de cría descritas en el apartado 2.2.1.1. Se consideró como tiempo cero experimental el momento en el que se realizó la siembra de los huevos. Se observó diariamente el estadio de desarrollo (larva, pupa o adulto) de los individuos de cada línea desde el momento de la puesta hasta la emergencia de los adultos y se calculó el día en el que el 100% de las larvas habían pupado. Para analizar el porcentaje de emergencia y el día de emergencia, del total de pupas obtenidas para cada línea, se seleccionaron al azar cien (entre los individuos que puparon en un intervalo de 4 días) y se anotó diariamente el número de adultos que emergieron en cada línea. Además, se analizó la supervivencia de los adultos emergidos a partir de esas cien pupas, anotando diariamente durante un periodo de quince días el número de insectos vivos de cada una de las líneas de laboratorio. Se realizaron tres réplicas experimentales no simultáneas de cada uno de los experimentos. 151 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ 5.3. Resultados. En el capítulo anterior se observó que, según el perfil de restricción obtenido mediante el método PCR-FLP (ver apartado 4.3.6), la línea de laboratorio resistente a malatión W-10Km estaba formada únicamente por individuos heterocigotos para la mutación AChE G328A. Una posible explicación para este resultado sería la existencia de una duplicación génica en homocigosis (Ccace2RS/RS) en los individuos de esta línea W-10Km, que provocaría la presencia del alelo silvestre (Ccace2S) y del mutado (Ccace2R) en las dos copias de un mismo cromosoma. A continuación se muestran los resultados de las diferentes aproximaciones realizadas en este capítulo para corroborar la existencia de esta duplicación tanto en líneas seleccionadas en el laboratorio como en poblaciones de campo de C. capitata. 5.3.1. Cruzamientos y descendencia de las líneas C y W-10Km. Para corroborar la hipótesis de la duplicación del gen Ccace2 se realizaron cruzamientos entre individuos de la línea resistente a malatión W-10Km con individuos de la línea susceptible C. Se genotiparon, mediante el sistema PCR-RFLP descrito en el capítulo anterior (apartado 4.2.3), 153 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ tanto los parentales como la descendencia de un total de 6 parejas, 3 parejas por dirección de cruzamiento. Según lo esperado, los seis parentales de la línea resistente W-10Km presentaron un perfil heterocigoto para la mutación AChE G328A (doble banda), mientras que los insectos parentales de la línea C mostraron un perfil asociado a la presencia del alelo silvestre en homocigosis. Los ochenta insectos analizados de la F1 de los cruzamientos de ambas líneas presentaron un perfil de restricción correspondiente a individuos heterocigotos para la mutación AChE G328A (Fig. 5.3.1). La ausencia de descendientes homocigotos para el alelo silvestre, tal como cabría esperar según la herencia mendeliana y en ausencia de selección, es compatible con la hipótesis de la duplicación génica. Así, los individuos de la F1 portarían en un cromosoma la duplicación génica (Ccace2RS) proveniente del parental W-10Km (homocigoto estructural para la duplicación) y, en el otro cromosoma, portarían el alelo silvestre proveniente de la línea C, con lo que su genotipo sería Ccace2RS/S. Resultado esperado (herencia mendeliana) Resultado observado línea C línea W-10Km línea C línea W-10Km SS RS SS RS X P: 50% SS X 50% RS 100% RS F1: Figura 5.3.1. Resultado esperado y observado en el cruzamiento de las líneas C y W-10Km. Perfiles de restricción esperados y observados en el análisis de la mutación AChE G328A mediante el método PCR-RFLP para la detección del alelo CCace2R en los individuos parentales (P) de la línea C y de la línea W-10Km, y en su descendencia (F1). 154 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ 5.3.2. Determinación del número de copias del gen Ccace2. Con el objetivo de determinar si existía una duplicación del gen Ccace2 en los individuos de la línea W-10Km se estimó el número de copias de este gen mediante qPCR sobre DNA genómico. El resultado se comparó con el análisis realizado en los individuos de la línea susceptible de laboratorio C, utilizando los genes Ccp0 y sod como genes de referencia. En la figura 5.3.2 se observa como los individuos de la línea W-10Km presentaron en su genoma doble número de copias del gen Ccace2 respecto a los individuos de la línea C. Este resultado apoya la hipótesis de la existencia de una duplicación del gen que codifica la acetilcolinesterasa en C. capitata. Nº relativo de copias de Ccace2 2,5 b Ccp0 b sod 2 1,5 a a 1 0,5 0 C W-10Km Figura 5.3.2. Cuantificación del número de copias del gen Ccace2 en dos líneas de laboratorio de C. capitata. Se muestra la cantidad relativa del gen Ccace2 en la línea susceptible C y la línea resistente al malatión W-10Km tomando DNA genómico como molde de la qPCR, con respecto al número de copias de los genes Ccp0 y sod. Las distintas letras muestran diferencias significativas según un ANOVA con test post hoc de Tukey (p<0,05). Las barras de error representan la desviación estándar de la media. No se observaron diferencias significativas en el análisis del número de copias de Ccace2 en las líneas C y W-10Km en función del gen de referencia empleado (Figura 5.3.2.), por lo que en las siguientes estimaciones del número de copias de Ccace2 únicamente se empleó como referencia el gen Ccp0. 155 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ 5.3.3. Cuantificación de la expresión de Ccace2. Para evaluar la funcionalidad de la duplicación del gen Ccace2 detectada en el genoma de los individuos de la línea W-10Km se estudió la expresión de Ccace2 mediante qPCR. Se analizaron cinco grupos de seis individuos de las líneas C y W-10Km, formado cada grupo por tres machos y tres hembras. La extracción de RNA se realizó a partir del tórax y el abdomen, empleando la cabeza para estimar la actividad AChE (ver apartado siguiente). Como se puede observar en la figura 5.3.3, la expresión relativa de Ccace2 fue el doble en la línea W-10Km, en comparación con la de la línea susceptible C. 3 Expresión relativa de Ccace2 b 2,5 2 1,5 a 1 0,5 0 C W-10Km Figura 5.3.3. Cuantificación de la expresión de Ccace2 en las líneas de laboratorio C y W-10Km. Las distintas letras muestran diferencias significativas según el test t de Student (p<0,05). Las barras de error representan el error estándar de la media. 5.3.4. Actividad AChE e inhibición por malaoxón. Una vez comprobado que la duplicación detectada en la línea W-10Km estaba acompañada de una mayor expresión de Ccace2 en esta línea resistente a malatión en comparación con la línea susceptible C, nos planteamos si este incremento en la expresión se vería reflejado en un aumento en la actividad enzimática. Con el objeto de comparar la actividad enzimática AChE y su inhibición por malaoxón en las líneas C y W-10Km, se analizaron los homogeneizados de las cabezas de los 156 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ grupos de insectos empleados en el estudio de la expresión de Ccace2. La actividad específica AChE en los grupos de insectos de la línea susceptible C varió entre 0,36 y 0,42 µmol·min-1·mg prot-1. Sin embargo, en los grupos de insectos de la línea W-10Km, la actividad específica AChE varió entre 0,45 y 0,55 µmol·min-1·mg prot.-1 (Tabla 5.3.1), siendo ésta un 30% mayor (p<0,001) que la actividad AChE estimada en los individuos de la línea susceptible C, línea que no portaban la mutación AChE G328A en el gen Ccace2. Tabla 5.3.1. Actividad AChE e inhibición por malaoxón en las líneas de laboratorio C y W-10Km. Se muestran los valores medios de actividad AChE y de la constante de inhibición (Ki) con su error estándar de la media (EE). Se indican las diferencias significativas con respecto a la línea susceptible C al realizar el test t de Student (* p<0,05, ** p<0,001). Actividad AChE ± EE Ki ± EE -1 (M-1·min-1) -1 (µmol·min ·mg prot ) Línea C (Ccace2S/S) 0,38 ± 0,01 2,09·106 ± 2,1·105 Línea W-10Km (Ccace2RS/RS) 0,49 ± 0,01 ** 1,36·106 ± 1,4·105 * La inhibición de la actividad enzimática AChE mediada por malaoxón se estimó mediante el cálculo de la constante de inhibición (Ki) en los mismos grupos de individuos. Cuanto mayor sea el valor de la Ki mayor será la afinidad del inhibidor por la enzima inhibida. Como se puede observar en la tabla 5.3.1, la Ki media calculada para los individuos de la línea susceptible C fue de 2,09·106 M-1·min-1, mientras que la Ki media de los individuos de la línea resistente W-10Km fue significativamente menor: 1,36·106 M-1·min-1 (p<0,05). Estos resultados demuestran que la línea W-10Km mantiene la insensibilidad frente al malaoxón sin que se vea reducida la actividad AChE, lo que indica que la duplicación observada al analizar el número de copias de Ccace2 no está originada por un pseudogen. 157 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ 5.3.5. Caracterización del perfil RFLP de los individuos con fenotipo heterocigoto para la mutación AChE G328A. En el capítulo anterior se diseñó un sistema PCR-RFLP que reconocía la presencia del alelo de resistencia Ccace2R, identificando aquellos individuos homocigotos y heterocigotos para la mutación AChE G328A. En el análisis de la F52 de la línea W-10Km mediante este sistema se observó que la totalidad de los individuos poseían el mismo perfil RFLP, consistente en una banda superior más intensa que la inferior, fenotipo RFLP asociado a los individuos heterocigotos para la mutación AChE G328A (Fig. 5.3.1). Además, en el análisis de las poblaciones de campo se observaron hasta tres perfiles RFLP heterocigotos distintos en función de la intensidad relativa de las dos bandas originadas (Fig. 5.3.4). A B B C C B 172 pb 145 pb Figura 5.3.4. Perfiles RFLP asociados a los individuos heterocigotos para AChE G328A. Se pueden observar las diferentes intensidades de las bandas (A, B, C) existentes en el patrón de restricción obtenido con la enzima Bst NI. En primer lugar, con el fin de asociar un perfil RFLP con la enzima Bst NI a cada uno de los cuatro genotipos que poseen el alelo de resistencia (Ccace2R/S, Ccace2RS/RS, Ccace2RS/S, Ccace2RS/R), elaboramos un modelo teórico de lo que ocurriría durante la PCR y el RFLP para cada uno de los cuatro genotipos. Así, en el amplicón obtenido tras la PCR habría tres posibles tipos de moléculas (Fig. 5.3.5): 1) moléculas de DNA con complementariedad perfecta y correspondientes al alelo Ccace2S, que serían cortadas por la enzima Bst NI. 2) moléculas de DNA con complementariedad perfecta y correspondientes al alelo Ccace2R, que no serían cortadas por la enzima Bst NI. 3) moléculas heterodúplex, que no serían cortadas por Bst NI debido a que en el sitio de corte existiría un fallo en la complementariedad de bases. 158 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ Bst NI DIANA: 5’-ttccagGCACTTA-3’ cadena S 3’-aaggtcCGTGAAT-5’ cadena S 5’-ttccagCCACTTA-3’ cadena R 3’-aaggtcGGTGAAT-5’ cadena R N O 5’-ttccagGCACTTA-3’ cadena S 3’-aaggtcGGTGAAT-5’ cadena R 5’-ttccagCCACTTA-3’ cadena R 3’-aaggtcCGTGAAT-5’ cadena S C O R T E Figura 5.3.5. Modelo teórico de PCR-RFLP para los individuos portadores del alelo de resistencia Ccace2R. Se muestran las cuatro combinaciones de DNA de doble cadena que se generarían tras la PCR en los individuos con genotipo Ccace2R/S, Ccace2RS/RS, Ccace2RS/R o Ccace2RS/S. Las cadenas S poseen el alelo silvestre (Ccace2S) y las R el de resistencia (Ccace2R). En amarillo se indica el sitio de la mutación puntual AChE G328A. Los triángulos negros muestran el punto de corte de la enzima Bst NI. Sólo serán cortadas aquellas moléculas de DNA con dos puntos de corte. Al realizar el RFLP del amplicón con la enzima Bst NI, sólo aquellas moléculas de DNA con la doble cadena S serían cortadas por la enzima, mientas que tanto los heterodúplex (cadena Rcadena S y viceversa), como las moléculas de DNA formadas por dos cadenas R permanecerían intactas. Por otro lado, la posible existencia de la duplicación génica, que provoca la presencia de los alelos Ccace2S y Ccace2R en un mismo cromosoma, daría lugar a un cambio en la proporción de los alelos, lo que repercutiría en la intensidad de las dos bandas originadas después del RFLP. En segundo lugar, teniendo en cuenta el razonamiento anterior y la relación existente entre los alelos silvestre y de resistencia, estimamos la proporción de las dos bandas del RFLP mediante un cuadro de Punnett (Fig. 5.3.6). Según nuestra estimación, el RFLP teórico de los individuos con genotipo Ccace2RS/R generaría una banda superior de 0,89 y una banda inferior de 0,11. Los individuos Ccace2RS/S mostrarían una banda superior de 0,55 y una inferior de 0,45. En el caso de los genotipos Ccace2R/S y Ccace2RS/RS la cantidad de alelos es proporcional, con lo que no se podrían diferenciar mediante este método los individuos con estos genotipos, mostrando ambos una banda superior de 0,75 y una inferior de 0,25 (Fig. 5.3.6). 159 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ RS/RS ó R/S RS/R Genotipo: Alelos Alelos RR(2/3) (2/3) SS(1/3) (1/3) PCR-RFLP teórico: RR(2/3) (2/3) 0,45 0,45 0,22 0,22 SS(1/3) (1/3) 0,22 0,22 0,11 0,11 Alelos Alelos RR(1/2) (1/2) SS (1/2) (1/2) RR(1/2) SS (1/2) (1/2) (1/2) 0,25 0,25 0,25 0,25 0,25 0,25 0,25 0,25 RS/S Alelos Alelos R R (1/3) (1/3) SS (2/3) (2/3) R (2/3) R (1/3) SS (2/3) (1/3) 0,11 0,22 0,11 0,22 0,22 0,22 0,45 0,45 Figura 5.3.6. Caracterización matemática de los perfiles de restricción heterocigotos para la mutación AChE G328A con Bst NI. En los genotipos, S representa el alelo Ccace2S, R es el alelo Ccace2R y RS representa la duplicación Ccace2RS. En el PCR-RFLP teórico se muestran, en tanto por uno, la proporción de cadenas que teóricamente se producirían durante la PCR en función de la cantidad inicial de los alelos, señalada entre paréntesis. En cada caso, sólo se cortarían con la enzima Bst NI las moléculas de doble cadena SS, resaltadas en gris en las tablas. Tomando como referencia los perfiles RFLP teóricos para los diferentes genotipos estimados en la figura 5.3.6, realizamos la cuantificación de las bandas de los perfiles RFLP de los individuos heterocigotos para la mutación AChE G328A con la enzima Bst NI en la línea de laboratorio resistente a malatión W-10Km (n=30) y en tres poblaciones recogidas en campo: Amposta (n=12), Les Coves de Vinromà (n=14) y Tortosa (n=11). Las intensidades de las bandas superior e inferior del perfil RFLP de cada individuo se representaron, en tanto por uno, en unos ejes de coordenadas en los que además se mostraron los valores teóricos estimados para cada 1,0 SUP SUP genotipo (Fig. 5.3.7). W-10Km 1,0 0,9 0,9 0,8 0,8 0,7 0,7 0,6 0,6 0,0 0,5 ῀ 0,0 0,0 0,5 0,1 0,2 0,3 0,4 0,5 AMP ῀ 0,0 0,1 0,2 0,3 Figura 5.3.7. Cuantificación de los perfiles RFLP heterocigotos para AChE G328A. Se representa en 0,4 1,0 COV 1,0 TOR 0,9 0,9 0,8 0,8 0,7 0,7 0,6 0,6 0,0 0,5 ῀ 0,0 0,0 0,5 0,1 0,2 0,3 0,4 0,5 0,5 INF SUP SUP INF ῀ 0,0 0,1 0,2 0,3 0,4 0,5 INF INF 160 ordenadas la intensidad de la banda superior (SUP) y en abscisas la de la banda inferior (INF), en tanto por uno. Cada punto negro representa un individuo. El punto azul indica el valor teórico de un genotipo Ccace2RS/R, el punto rojo de un genotipo Ccace2RS/RS o Ccace2R/S, y el punto verde de un genotipo Ccace2RS/S. Se analizó la línea W10Km y las poblaciones de Amposta (AMP), Les Coves de Vinromà (COV) y Tortosa (TOR). 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ En el análisis de las intensidades de las bandas del perfil RFLP observamos que todos los individuos de la línea W-10Km, homocigotos estructurales para la duplicación (genotipo Ccace2RS/RS), se situaron próximos al punto de color rojo que indica ese genotipo teórico. Por otro lado, en las poblaciones de Amposta (AMP) y Tortosa (TOR), los individuos se separaron en tres grupos: un grupo central próximo al punto rojo, de distribución similar a la observada en W-10Km y cuyo genotipo no se podría definir mediante el RFLP (podría ser Ccace2RS/RS o Ccace2R/S), un segundo grupo, próximo al punto azul, que tendría genotipo Ccace2RS/R y por último, un grupo próximo al punto verde que tendría genotipo Ccace2RS/S. Sin embargo, en la población recogida en Les Coves de Vinromà (COV) el agrupamiento no fue tan evidente, por lo que a algunos individuos no se les pudo asignar un genotipo (Tabla 5.3.2). Tabla 5.3.2. Asignación de genotipos para Ccace2 en función de la caracterización de los perfiles RFLP. Se indica el número de individuos de cada genotipo en las diferentes poblaciones. (nd) Indica que no se pudo determinar el genotipo. Genotipo W-10Km Amposta Les Coves de Vinromà Tortosa RS/R 0 2 4 2 R/S ó RS/RS 30 6 4 5 RS/S 0 4 3 4 nd 0 0 3 0 TOTAL 30 12 14 11 5.3.6. Confirmación del genotipo asociado al perfil RFLP mediante cruzamientos y PCR cuantitativa. Con el objetivo de comprobar que la asociación genotipo-perfil descrita anteriormente era correcta en todos los posibles genotipos de Ccace2, se estimó el número de copias del gen mediante qPCR y se realizó un genotipado mediante el sistema PCR-RFLP con la enzima Bst NI en las siguientes líneas y cruzamientos: · Línea susceptible C (genotipo Ccace2S/S). · Línea W-10Km, resistente a malatión y formada por individuos homocigotos estructurales para la duplicación (genotipo Ccace2RS/RS). · Isolínea R/R (genotipo Ccace2R/R). · F1 del cruce de la línea W-10Km con la línea C: genotipo Ccace2RS/S. · F1 del cruce de la línea W-10Km con la isolínea R/R: genotipo Ccace2RS/R. · F1 del cruce de la isolínea R/R con la línea C: genotipo Ccace2R/S. 161 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ En la figura 5.3.8 se muestra el perfil RFLP para la enzima Bst NI obtenido en los individuos parentales y en la F1 de los cruzamientos descritos previamente, y la correspondencia entre los patrones de restricción y el genotipo. En las líneas parentales se observó el perfil RFLP esperado según su genotipo conocido: i) la línea W-10Km mostró la doble banda asociada con la presencia de la mutación AChE G328A en heterocigosis, resultado de la duplicación; ii) en la línea C se observó el corte total de la banda superior, apareciendo únicamente la inferior, iii) en la isolínea R/R, al estar constituida por individuos homocigotos para el alelo de resistencia, no se produjo el corte del producto de PCR por la enzima Bst NI. Teniendo en cuenta la caracterización teórica de los perfiles RFLP realizada anteriormente (Fig. 5.3.6), en el análisis de la F1 del cruzamiento de la línea W-10Km con la isolínea R/R se observó un perfil RFLP en el que la banda superior era mucho más intensa que la inferior, relación que es compatible con la estimada para este genotipo Ccace2RS/R (89:11). En el caso de la F1 del cruce de la línea W-10Km con la línea C, se observó que la diferencia de intensidades entre ambas bandas era menor, siendo ligeramente más intensa la superior, siendo esto compatible con la intensidad estimada para los individuos con genotipo Ccace2RS/S (55:45). En el análisis de los individuos que poseen la misma proporción de alelo silvestre y de resistencia (genotipos Ccace2RS/RS y Ccace2R/S) se obtuvo un perfil RFLP en el que la banda superior era de mayor intensidad que la inferior y la relación entre sus intensidades era intermedia a las de los dos casos descritos anteriormente, por lo que coincidiría con una proporción 75:25 (Fig. 5.3.8). Línea W-10Km Isolínea R/R Línea W-10Km X RS/RS Isolínea R/R RS/RS Línea C RS/RS R/R RS/RS X S/S R/S Línea C Línea W-10Km X X R/R RS/R Línea W-10Km RS/RS S/S RS/S Figura 5.3.8. Correspondencia entre los patrones RFLP y los genotipos para la mutación AChE G328A y para la mutación estructural. En la asignación de los genotipos, S representa el alelo Ccace2S, R es el alelo Ccace2R y RS representa la duplicación génicaCcace2RS, resultado de la mutación cromosómica. 162 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ El número relativo de copias observado mediante qPCR se muestra en la figura 5.3.9. Los individuos de la isolínea R/R, de la línea C y los individuos resultado del cruce de éstas, con genotipo Ccace2R/S, mostraron el mismo número relativo de copias del gen Ccace2. Los individuos de la línea W-10Km, homocigotos estructurales para la duplicación génica (Ccace2RS/RS), presentaron el doble número de copias del gen Ccace2 con respecto a los individuos de la línea C. Por otro lado, los individuos de la F1 resultado del cruce de la línea W-10Km con la línea C o la isolínea R/R, heterocigotos estructurales para la duplicación (Ccace2RS/S o Ccace2RS/, respectivamente), presentaron 1,5 veces más copias del gen Ccace2 que los individuos silvestres. 2,5 a Nº relativo copias Ccace2 2,0 b b 1,5 c c c R/S R/R S/S 1,0 0,5 0,0 RS/RS RS/R RS/S Genotipos Figura 5.3.9. Cuantificación del número de copias del gen Ccace2 en diferentes genotipos. Se muestra la cantidad relativa del gen Ccace2 con respecto a Ccp0 en los seis genotipos posibles teniendo en cuenta la presencia de la duplicación Ccace2RS (RS). S representa el alelo Ccace2S y R es el alelo Ccace2R. Las distintas letras sobre las barras indican diferencias significativas según un ANOVA con test post hoc de Tukey (p<0,05). Las barras de error representan el error estándar de la media. 5.3.7. Análisis de los patrones de secuenciación de la mutación AChE G328A. Analizamos los patrones de secuenciación de aquellos genotipos que originaban un perfil RFLP heterocigoto para AChE G328A con la enzima Bst NI (Ccace2R/S, Ccace2RS/RS, Ccace2RS/S, Ccace2RS/R) y los comparamos con los patrones de los individuos con genotipo silvestre (Ccace2S/S) pertenecientes a la línea susceptible C. La secuenciación directa con el cebador RiAChE_CcE60 del 163 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ producto de PCR resultante de la amplificación del exón 6 de la acetilcolinesterasa mostró diferentes patrones de secuenciación en función del genotipo (Fig. 5.3.10). Fenotipos heterocigotos Fenotipo silvestre (línea C) Genotipo RFLP (Bst I) Cromatograma 0,00 Ccace2S/S 1,00 Ccace2R/S 0,77 Ccace2RS/RS 0,23 0,56 Ccace2RS/S 0,44 0,93 Ccace2RS/R 0,07 Figura 5.3.10. Patrones de secuenciación para la mutación AChE G328A. Se muestran los perfiles RFLP y los cromatogramas correspondientes a los cuatro genotipos heterocigotos posibles para la mutación AChE G328A y a un individuo de genotipo silvestre de la línea susceptible C, empleado como referencia. En los perfiles de restricción se indica el porcentaje, en tanto por uno, de cada una de las bandas generadas con la enzima Bst NI. El recuadro del cromatograma señala el sitio puntual de la mutación AChE G328A. 164 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ En los cromatogramas de los cuatro perfiles heterocigotos para la mutación AChE G328A se observó una doble lectura correspondiente a las bases citosina (C) y guanina (G) en la posición de la mutación puntual AChE G328A, mientras que los individuos con genotipo silvestre de la línea C mostraron un único pico correspondiente a la guanina. La guanina se corresponde con el alelo silvestre Ccace2S y la citosina con el alelo mutado Ccace2R que confiere resistencia a malatión. Tomando como referencia la proporción de ambos picos en el cromatograma de los individuos con genotipo Ccace2R/S y Ccace2RS/RS, que poseen idéntica proporción de alelos, observamos que la relación entre los picos varió en función del alelo predominante. Así, los individuos con genotipo Ccace2RS/R, que tienen mayor proporción de alelo Ccace2R, mostraron un pico de citosina mucho mayor que el de guanina. Por el contrario, los individuos con genotipo Ccace2RS/S mostraron mayor cantidad de pico de guanina que de citosina al comparar su cromatograma con el de los individuos Ccace2R/S y Ccace2RS/RS (Fig. 5.3.10). 5.3.8. Detección de la duplicación Ccace2RS mediante un sistema de doble PCR-RFLP. En el apartado 5.3.5 se explicó que el sistema de PCR-RFLP con la enzima Bst NI, descrito en el capítulo 4 y empleado para determinar la presencia del alelo de resistencia Ccace2R, también podía ser utilizado para la detección de la duplicación. Sin embargo, la caracterización de los perfiles RFLP de la enzima Bst NI puede ser confusa en ocasiones, debido a la dificultad para distinguir las proporciones relativas de las intensidades de las bandas del perfil de restricción en un gel de agarosa. Con el objetivo de detectar la duplicación Ccace2RS de una manera más precisa se desarrolló un método de doble PCR-RFLP que consistió en combinar el sistema de la enzima Bst NI con un segundo PCR-RFLP. En el nuevo sistema PCR-RFLP se utilizó un cebador (FG328A_27TseI) que creaba un sitio de restricción para la enzima Ape KI en presencia del alelo de resistencia Ccace2R. Del mismo modo que para la enzima Bst NI (Fig. 5.3.6), se estimaron las proporciones de las bandas de los perfiles de restricción para Ape KI (Tabla 5.3.3) y se observó que la proporción existente entre las dos bandas originadas en el RFLP difería en ambas enzimas únicamente en la estimación de los individuos heterocigotos estructurales para la duplicación (CCace2RS/S y CCace2RS/R). 165 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ Tabla 5.3.3. Proporción de las bandas estimada para el sistema de doble PCRRFLP. Se indica el porcentaje de intensidad de cada una de las dos bandas (superior SUP, inferior INF) que conforman el perfil de restricción para las enzimas Bst NI y Ape KI. En los genotipos, S representa el alelo Ccace2S, R es el alelo Ccace2R y RS representa la duplicación Ccace2RS. Bst NI Ape KI (% bandas) (% bandas) Genotipo SUP INF SUP INF RS/RS ó R/S 75 25 75 25 RS/R 89 11 55 45 RS/S 55 45 89 11 La relación entre el doble perfil PCR-RFLP y los posibles genotipos de Ccace2 (ver apartado 5.3.6) se muestra en la figura 5.3.11. Mediante el sistema de doble PCR-RFLP se obtuvieron dobles perfiles de restricción característicos para los genotipos homocigotos (Ccace2S/S y Ccace2R/R) y para los individuos heterocigotos estructurales para la duplicación (Ccace2RS/S y Ccace2RS/R). Los individuos con genotipos Ccace2R/S y Ccace2RS/RS, donde las proporciones de los alelos Ccace2R y Ccace2S son idénticas, no lograron ser diferenciados porque en ambos sistemas PCR-RFLP se produjo el mismo perfil, sin embargo, sí fueron identificados mediante qPCR (Fig. 5.3.9). RS/RS 1 RS/R 2 1 RS/S 2 1 R/S 2 1 R/R 2 1 S/S 2 1 2 172 pb 145 pb Figura 5.3.11. Sistema de doble PCR-RFLP para la detección de la mutación AChE G328A y de la duplicación Ccace2RS. En los genotipos, S representa el alelo Ccace2S, R es el alelo Ccace2R y RS representa la duplicación Ccace2RS. Se indican los patrones RFLP para las enzimas Bst NI (1) y Ape KI (2) en un gel de agarosa al 3%. 166 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ 5.3.9. Detección de la duplicación Ccace2RS en poblaciones de campo. Con el objetivo de estimar la frecuencia de individuos portadores de la duplicación Ccace2RS, mediante el sistema del doble PCR-RFLP, se analizaron de nuevo las 25 poblaciones españolas (recogidas entre los años 2003 y 2008) en la que se habían encontrado individuos heterocigotos para la mutación AChE G328A (apartado 4.3.5). Este nuevo sistema nos permitió confirmar los individuos homocigotos para los alelos Ccace2R y Ccace2S, e identificar aquellos heterocigotos estructurales para la duplicación (Ccace2RS/S, Ccace2RS/R). Los resultados del análisis de PCR-RFLP se muestran en la tabla 5.3.4. La duplicación RS Ccace2 se detectó mediante el método del doble PCR-RFLP en 18 de las 25 poblaciones españolas de C. capitata que habían presentado la mutación AChE G328A. La distribución geográfica de la duplicación, aunque se concentró en la cuenca mediterránea, fue amplia en todo el territorio español, ya que se observó tanto en las Islas Baleares como en Canarias (Tabla 5.3.4). El porcentaje de individuos portadores de la duplicación varió entre el 5%, detectado en Carlet y Burriana, y el 35% observado en Les Coves de Vinromà. No obstante, hay que tener en cuenta que estos valores representan la frecuencia genotípica mínima, ya que los individuos homocigotos estructurales para la duplicación (genotipo Ccace2RS/RS) no pueden ser distinguidos de los heterocigotos Ccace2R/S con el método del doble PCR-RFLP. Tabla 5.3.4. Frecuencias genotípicas de Ccace2 en poblaciones españolas de C. capitata. Se indica la frecuencia genotípica estimada mediante el sistema de doble PCR-RFLP. En los genotipos, S representa el alelo Ccace2S, R es el alelo Ccace2R y RS representa la duplicación Ccace2RS. __________________________________________________________________________________________________________________ Frecuencia genotípica para Ccace2 ________________________________________________________________ C. Autónoma Población RS/R RS/S R/R S/S RS/RS ó R/S ______________________________________________________________________________________________________________________________________ Andalucía Algarrobo-Costa Almuñecar Ayamonte Gibraleón - - - 0,95 0,95 0,90 0,95 0,05 0,05 0,10 0,05 Villalengua - 0,08 - 0,92 - 0,11 0,10 0,10 0,22 0,20 0,20 0,17 0,15 0,20 0,60 0,17 0,35 0,25 0,30 0,33 0,30 0,25 Aragón Cataluña Alcanar Amposta Girona Tortosa 167 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ Tabla 5.3.4. Frecuencias genotípicas de Ccace2 en poblaciones españolas de C. capitata. (Continuación). __________________________________________________________________________________________________________________ Frecuencia genotípica para Ccace2 ________________________________________________________________ C. Autónoma Población RS/R RS/S R/R S/S RS/RS ó R/S ______________________________________________________________________________________________________________________________________ C. Valenciana Alacant Alcùdia Burriana Carlet Les Coves de Vinromà Moncada Orihuela Sagunt Serra Vila Joiosa Vila Real Xàbia 0,20 0,05 0,05 - 0,17 0,05 0,05 0,15 0,21 0,11 0,25 0,10 0,11 0,30 0,04 0,10 0,20 0,21 0,05 0,11 0,05 0,05 0,20 0,74 0,58 0,55 0,80 0,10 0,32 0,55 0,50 0,40 0,90 0,37 0,40 0,22 0,25 0,30 0,15 0,35 0,21 0,40 0,28 0,25 0,47 0,10 Muro - 0,10 - 0,80 0,10 Valle de Guerra - 0,05 0,20 0,35 0,40 Albelda - - - 0,56 0,44 I. Baleares I. Canarias La Rioja ______________________________________________________________________________________________________________________________________ En aquellos individuos en los que no fue posible determinar el genotipo con el sistema de doble PCR-RFLP (Ccace2RS/RS y Ccace2R/S), se cuantificó el número de copias del gen Ccace2 mediante qPCR (ver apartado 5.3.2). Se analizaron un total de 13 individuos pertenecientes a dos poblaciones de campo recogidas en Alcanar (ALC, n=6) y Orihuela (ORI, n=7). Además, se analizaron 6 individuos de genotipo silvestre de la población ORI y, como control, se analizó uno Nº relativo copias Ccace2 de la línea C de laboratorio (Fig. 5.3.12). 2,5 Doble RFLP (Bst NI y Ape KI) 2 Ccace2RS/RS ó Ccace2R/S 1,5 1 0,5 Ccace2S/S Control ORI13 ORI12 ORI11 ORI9 ORI10 ORI8 ORI7 ORI6 ORI5 ORI4 ORI3 ORI2 ORI1 ALC6 ALC5 ALC4 ALC3 ALC2 ALC1 0 Figura 5.3.12. Cuantificación mediante qPCR del número de copias de Ccace2 en individuos de campo. Se muestra la cantidad relativa del gen Ccace2 con respecto a Ccp0 en las poblaciones de Alcanar (ALC) y Orihuela (ORI). Además, como control, se evaluó el número de copias en un individuo de la línea de laboratorio C. 168 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ De los trece individuos con genotipo indefinido (Ccace2R/S o Ccace2RS/RS), doce presentaron un número de copias de Ccace2 similar al del control, con lo que se les asignó el genotipo Ccace2R/S. Un individuo de la población de Alcanar presentó doble número de copias y se identificó como homocigoto estructural para la duplicación (genotipo Ccace2RS/RS). Con estos datos se calcularon las frecuencias alélicas de la población de Alcanar y se observó que los alelos Ccace2S, Ccace2R, y la duplicación Ccace2RS se encontraron en unos porcentajes de 77,5%, 12,5% y 10%, respectivamente. Del mismo modo, en la población de Orihuela, la frecuencia estimada para Ccace2S y Ccace2R fue del 75% y 25%, respectivamente, con lo que no se detectó la duplicación en esta población. 5.3.10. Comparación del desarrollo, porcentaje de emergencia y supervivencia de adultos en las líneas C y W-10Km. Se estimaron algunos parámetros indicadores de la posible existencia de un coste biológico asociado a la duplicación Ccace2RS comparando en las líneas de laboratorio C y W-10Km: i) los tiempos de desarrollo, ii) el porcentaje de emergencia y iii) la supervivencia de los adultos. Tomando como tiempo cero del experimento el momento en el que las puestas de huevos fueron recogidas, no se detectaron diferencias entre ambas líneas en cuanto al desarrollo larvario, ya que el día en el que el 100% de las larvas habían pupado fue de 19,6±0,3 días en W-10Km y de 20,0±0,0 días en la línea C. Tampoco se encontraron diferencias en el día de emergencia de los adultos de la línea W-10Km (28,7±0,8 días) con respecto a la línea susceptible C (29,1±0,2 días). En cuanto al porcentaje de emergencia, aunque el valor fue mayor en la línea C (94±0,8%) que en W-10Km (91±1,2%), las diferencias entre las líneas no fueron significativas. El análisis de la supervivencia, realizado en ambas líneas durante un periodo de quince días después de la emergencia de los adultos, nos indicó que a partir del séptimo día existieron diferencias significativas en la viabilidad de los adultos (Fig. 5.3.13), siendo mayor la mortalidad en los individuos de la línea W-10Km. Estas diferencias se incrementaron en el tiempo. Así, en el día decimoquinto, último día del experimento, la mortalidad acumulada fue 3,4 veces mayor en la línea resistente a malatión W-10Km que en la línea susceptible C. 169 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ 100% Supervivencia * 90% * W-10Km * * C * * * W-10Km 80% * * 70% 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 días Figura 5.3.13. Supervivencia de los adultos de las líneas C y W-10Km después de la emergencia. Se muestra el porcentaje de individuos vivos a lo largo de quince días. Las barras de error representan el error estándar de la media. Los asteriscos indican diferencias significativas con respecto a la línea C (t de Student, p<0,05). 170 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ 5.4. Discusión. El nuevo mecanismo de resistencia a malatión descrito en C. capitata en este capítulo consiste en una mutación cromosómica que produce una duplicación del gen Ccace2 que codifica la acetilcolinesterasa (AChE). Esta duplicación génica provoca la coexistencia de los alelos silvestre Ccace2S y resistente Ccace2R en un mismo cromosoma, y es similar a la encontrada anteriormente en C. pipiens (Bourguet et al., 1996b). La primera evidencia de la existencia de esta duplicación se observó al genotipar la línea W-10Km mediante el sistema PCR-RFLP con la enzima Bst NI, observándose que la totalidad de los individuos presentaban un perfil de restricción correspondiente a individuos heterocigotos para la mutación AChE G328A (apartado 4.3.6). Según la segregación mendeliana de un gen con dos alelos en ausencia de selección, el cruce entre individuos heterocigotos para dicho gen debe dar lugar tanto a individuos heterocigotos como a individuos homocigotos para los dos alelos del gen (Griffiths et al., 2008). En el análisis, con el mismo método, de la generación filial (F1) de las dos direcciones del cruzamiento entre la línea W10Km y la línea susceptible C (formada por homocigotos para Ccace2S) se demostró que toda la descendencia presentaba un perfil de restricción asociado a individuos heterocigotos (apartado 5.3.1). Este hecho es compatible con que en la línea W-10Km se haya producido una duplicación en tándem del gen Ccace2 que favorezca que dos copias del gen (una mutada y otra no mutada) se transmitan ligadas, generándose así una heterocigosis permanente (Labbé et al., 2007a). Si las dos copias del gen no estuvieran ligadas habríamos identificado en la F1, al menos, individuos 171 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ homocigotos para el alelo Ccace2S, en una proporción aproximada del 50%. La ausencia de descendientes homocigotos para el alelo silvestre indica que estos individuos de la F1 serían heterocigotos estructurales. Así, estos individuos portarían en un cromosoma una duplicación génica (Ccace2RS) y, en el otro cromosoma, portarían el alelo silvestre proveniente de la línea C, con lo que su genotipo sería Ccace2RS/S. La cuantificación mediante qPCR del número de copias del gen Ccace2 en ambas líneas parentales (C y W-10Km) y en la F1 corroboró la existencia de la duplicación (apartado 5.3.2). Además, la existencia de la duplicación y su segregación se comprobó analizando, mediante qPCR, las líneas W-10Km y C, y la isolínea R/R, y la F1 resultado del cruzamiento entre ellas, obteniendo así los seis genotipos posibles para CCace2: Ccace2RS/RS, Ccace2S/S, Ccace2R/R, Ccace2RS/R, Ccace2RS/S y Ccace2R/S. En los seis casos, el número de copias del gen Ccace2 esperado para cada genotipo coincidió con el estimado mediante qPCR (ver figura 5.3.9). Finalmente, se ha demostrado que la duplicación detectada en el RFLP y en la qPCR no es un pseudogen y es funcional, ya que tanto los niveles de expresión de Ccace2 (apartado 5.3.3) como la actividad AChE fueron superiores en la línea W-10Km que en la línea C. (apartado 5.3.4). En ácaros e insectos se han relacionado con resistencia varias amplificaciones y duplicaciones de genes que codifican enzimas de detoxificación (Oakeshott et al., 2005), sin embargo, el número de casos de duplicaciones génicas relacionadas con la resistencia a insecticidas mediada por modificación de la molécula diana es escaso (revisado por Bass y Field, 2011). En el pulgón Myzus persicae se ha relacionado con resistencia frente a ciclodienos una duplicación del gen que codifica la subunidad gamma del receptor del ácido aminobutírico (GABA) (Anthony et al., 1998); y tanto en el ácaro Tetranychus urticae como en el pulgón Aphis gossypii se ha asociado a resistencia a organofosforados la amplificación del gen ace1 (Kwon et al., 2010; Shang et al., 2012). De hecho, se ha sugerido que los eventos de duplicación o amplificación en artrópodos pueden ser más comunes de lo que los datos reflejan, pudiendo existir zonas del genoma más propensas a albergar duplicaciones (Bass y Field, 2011). No obstante, la duplicación encontrada en C. capitata, que da lugar a una heterocigosis permanente, se ha encontrado únicamente en el gen ace1 de mosquitos (Bourguet et al., 1996b; Djogbénou et al., 2009), habiéndose además demostrado la existencia de duplicaciones independientes en distintas zonas geográficas (Labbé et al., 2007a). Los distintos escenarios que pueden provocar la duplicación que genera heterocigosis permanente y que permiten la permanencia de ambos genes ligados en la población han sido discutidos anteriormente (Haldane, 1954; Spofford 1969; Otto y Yong, 2002; Labbé et al., 2007a). En los análisis realizados en las poblaciones de campo de C. capitata no hemos detectado 172 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ individuos con genotipo silvestre que tuviesen doble número de copias del gen Ccace2, hecho que indicaría que el evento de la duplicación génica es previo a la mutación AChE G328A, no obstante, habría que comprobar esta hipótesis analizando un mayor número de individuos. Así, en el contexto de nuestros resultados, no es posible determinar si ha podido ocurrir en primer lugar la duplicación y a continuación la mutación en una de las dos copias del gen, o si una mutación ha estado seguida de una recombinación no homóloga que originaría la duplicación en tándem. Se ha demostrado que la mutación puntual encontrada en el alelo Ccace2R provoca una disminución de la inhibición por malaoxón, la molécula activa del malatión, pero también una disminución de la actividad AChE (Magaña et al., 2008) que podría estar asociada a un coste biológico. En C. pipiens (Bourguet et al., 1996b) se ha sugerido que la disminución de actividad en la AChE alterada puede provocar un exceso de acetilcolina en el espacio sináptico que podría acarrear un coste biológico asociado al alelo de resistencia. En este trabajo hemos comprobado que los individuos de C. capitata portadores de la duplicación poseen al mismo tiempo una afinidad baja por el insecticida y una mayor actividad AChE, con toda probabilidad producida por el aumento de la expresión que acompaña al incremento en el número de copias del gen. Además, el hecho de que la duplicación se haya fijado en la línea W-10Km parece indicar que la duplicación Ccace2RS confiere a los individuos que la portan una doble ventaja con respecto a los individuos que poseen el alelo de resistencia Ccace2R: por un lado, la presencia del alelo Ccace2R conferiría resistencia al malatión y, por otro lado, el aumento del número de copias del alelo silvestre reduciría el coste biológico producido por la menor eficiencia catalítica de la enzima mutada, hecho que ha sido observado en C. pipiens (Raymond et al., 2001). No obstante, la compensación no resulta suficiente para eliminar totalmente el coste biológico en C. capitata en ausencia de selección. Así, hemos observado que en individuos portadores de la duplicación en homocigosis se produce una disminución en la supervivencia de los adultos a partir del séptimo día tras la emergencia (apartado 5.3.10). Es esperable que el coste biológico observado en individuos portadores de la duplicación sea menor que el observado en individuos portadores del alelo resistente sin duplicación, pero esta hipótesis deberá ser verificada en posteriores estudios mediante el análisis del coste biológico asociado a la mutación AChE G328A en los individuos homocigotos para el alelo mutado (Ccace2R/R). El conocimiento de los mecanismos asociados a resistencia y de los genes implicados permite desarrollar métodos de detección precoz, basados en técnicas de bioquímica o de biología molecular, que pueden ser útiles para el seguimiento y el manejo de la resistencia (Vontas et al. 2011). En el capítulo anterior describimos un método PCR-RFLP con la enzima de restricción Bst NI que identificaba la presencia de la mutación AChE G328A en los individuos en los que no se había producido el corte del amplicón, ya que la enzima reconocía el alelo silvestre Ccace2S (apartado 173 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ 4.2.3). Este método tiene la ventaja de poder ser aplicado sobre individuos muertos provenientes de trampas de monitoreo convencionales (ver figura 4.3.4.B), siendo posible la identificación de resistencia aún cuando el porcentaje de individuos resistentes sea bajo. Un objetivo que nos planteamos en este capítulo fue la detección de dicha duplicación de un modo simple y rápido. Para ello, inicialmente, estudiamos los patrones de secuenciación de los individuos heterocigotos para la mutación. El análisis de los cromatogramas no sólo reveló una doble lectura en el sitio de la mutación puntual AChE G328A, fenómeno similar al observado para una mutación en el receptor GABA de Anopheles funestus (Wondji et al., 2011), sino que la intensidad de los dos picos que mostraban la heterocigosis estaba relacionada con la cantidad de alelos (apartado 5.3.7). Así, se observaron patrones característicos para los genotipos Ccace2R/S/Ccace2RS/RS, Ccace2RS/R y Ccace2RS/S y, aunque detectamos la duplicación cuando estaba presente en uno de los dos cromosomas, las diferencias no eran tan evidentes como para emplear este método para el diagnóstico de la presencia de la duplicación. Posteriormente, analizamos los perfiles de restricción, obtenidos con la enzima Bst NI, de los individuos heterocigotos para la mutación AChE G328A y comprobamos que, si se atendía a la intensidad de las dos bandas generadas por la enzima de restricción, se podían observar distintos patrones de PCR-RFLP. Sin embargo, la visualización de las diferencias de intensidad entre bandas puede no ser clara en algunos casos. Finalmente, diseñamos un sistema de doble PCR-RFLP (apartado 5.3.8) que nos permitió identificar con mayor precisión los genotipos para el gen CCace2, a excepción de aquellos individuos en los que la proporción entre los dos alelos (mutado y no mutado) no está descompensada (genotipos Ccace2R/S y Ccace2RS/RS), en el primer caso por la ausencia de la duplicación y en el segundo por la presencia de la duplicación en las dos copias del cromosomas Hemos demostrado que la distinción entre estos dos genotipos es posible mediante el análisis del número relativo de copias del gen Ccace2 por qPCR. Además, mediante el sistema de doble PCR-RFLP, hemos asociado doble perfil de restricción a los individuos heterocigotos estructurales para la duplicación (Ccace2RS/S y Ccace2RS/R), hecho que nos permite detectar la duplicación sin emplear qPCR. El análisis de poblaciones españolas con el método del doble RFLP demostró que la duplicación Ccace2RS, fijada en la línea resistente de laboratorio W-10Km, se encuentra ampliamente distribuida por todo el territorio español, encontrándose en 18 de las 25 poblaciones analizadas, incluyendo las Islas Canarias y Baleares (apartado 5.3.9). Esta elevada dispersión de la duplicación concuerda con lo observado en C. pipiens (Ben Cheikh et al., 2009; Alout et al., 2009, 2011) y en A. gambiae (Djogbénou et al., 2009), ya que cuando se detecta la duplicación de ace1 en una región, se observa en la mayoría de las poblaciones y de los individuos. Además, en determinadas poblaciones de la cuenca mediterránea, la frecuencia de la duplicación era moderadamente alta entre los años 2003 y 2008, siendo igual o superior al 20% en Amposta, Girona y Tortosa (Cataluña); y en Les Coves de Vinromà, Moncada, Serra y Xàbia (C. Valenciana). 174 5. Nuevo mecanismo de resistencia a malatión en Ceratitis capitata ______________________________________________________________________________________________________________________________________ Como el método del doble PCR-RFLP no permite la distinción entre los genotipos Ccace2R/S y Ccace2RS/RS, solo ha sido posible realizar las estimaciones absolutas de frecuencia de cada uno de los alelos y de la duplicación en aquellas poblaciones analizadas mediante qPCR: Alcanar (Cataluña) y Orihuela (C. Valenciana). El malatión ha estado excluido temporalmente del registro de productos fitosanitarios de la Unión Europea durante los años 2009 y 2010 pero, recientemente, ha sido incorporado al registro y se permite su aplicación en los cultivos de especies ornamentales y de fresas, por lo que no se puede descartar que en las próximas campañas se apruebe de nuevo su uso en España para el tratamiento de las poblaciones de C. capitata, quizás mediante el empleo de una nueva formulación. En cualquier caso, el malatión se sigue utilizando para el control de C. capitata en multitud de países fuera de la Unión Europea. La amplia dispersión del alelo Ccace2R que hemos encontrado en las poblaciones españolas y la presencia de la duplicación, que provoca la heterocigosis permanente, podría suponer un grave problema si se utilizase de nuevo el malatión u otro organofosforado para el control de esta plaga. Además, la resistencia a malatión está asociada a resistencia cruzada frente a otros insecticidas utilizados actualmente para el control de C. capitata, como el caso de lambda-cialotrina, por lo que los tratamientos con malatión en campo podría favorecer la resistencia frente a otros insecticidas. Es por tanto muy importante obtener estimaciones precisas de las frecuencias de cada uno de los alelos y de la duplicación en distintas zonas geográficas, que permitan realizar un seguimiento de su progreso en el tiempo. Actualmente, se están recogiendo individuos en algunas de las localidades analizadas durante el periodo 2003-2008 con el objetivo de estimar la evolución en el tiempo de la frecuencia de los alelos de Ccace2 así como de la duplicación descrita en este capítulo. La detección precoz de la resistencia, unida al uso racional de los insecticidas y a la integración de distintos sistemas de control, permitirá la sostenibilidad del uso de insecticidas para el control de C. capitata y contribuirá a reducir los graves daños económicos que esta especie causa en la agricultura. 175 CONCLUSIONES 177 Conclusiones ______________________________________________________________________________________________________________________________________ Conclusiones. A partir de los resultados obtenidos en la presente tesis doctoral se extraen las siguientes conclusiones: 1. Los bioensayos realizados con una dosis discriminante sugieren la existencia de individuos resistentes a malatión en poblaciones de campo de C. capitata recogidas en Cataluña, Comunidad Valenciana, Andalucía, Islas Baleares y Aragón. 2. La susceptibilidad de las poblaciones españolas de C. capitata frente a espinosad fue elevada en general y similar a la de la línea susceptible C, aunque en una población recogida en Xàbia (Alicante) se observó una susceptibilidad aproximadamente dos veces menor. 3. Mediante selección en laboratorio se han obtenido las líneas W-4Km y W-10Km, 178 y 400 veces más resistentes a malatión que la línea susceptible C, respectivamente. Además, por primera vez en C. capitata, se ha seleccionado una línea 500 veces más resistente a espinosad (Xàbia-W-100s) que la línea susceptible C. 4. En la línea W-4Km se ha detectado resistencia cruzada moderada (entre 4 y 16 veces) a varios organofosforados, a carbaril, a lambda-cialotrina y a lufenurón, sin embargo, la resistencia cruzada a espinosad fue baja (1,5 veces). 5. El análisis de la diversidad de genes CYP en C. capitata ha permitido la identificación de, al menos, 37 genes que codifican enzimas P450 pertenecientes a cinco familias. 6. La expresión de 6 genes CYP, con homología a genes asociados con resistencia a insecticidas en otras especies, fue dependiente del sexo y presentó variaciones basales de hasta tres órdenes de magnitud entre diferentes genes. Hemos observado inducción de la expresión por fenobarbital en cuatro de ellos. 179 Conclusiones ______________________________________________________________________________________________________________________________________ 7. Se ha puesto a punto un sistema que permite amplificar por PCR y secuenciar, partiendo de DNA genómico, los exones de los genes Ccace2 y Ccae7 donde se han encontrado mutaciones relacionadas con resistencia a insecticidas en otras especies. 8. Se ha diseñado un sistema PCR-RFLP que permite la detección del alelo Ccace2R que confiere resistencia a malatión en C. capitata. Este alelo está ampliamente distribuido por todo el territorio español (en 25 de las 27 poblaciones analizadas), aunque no ha sido detectado en poblaciones procedentes de once países y de cinco continentes. 9. Se ha identificado un nuevo mecanismo de resistencia a malatión en C. capitata consistente en una duplicación del gen Ccace2 que da lugar a la existencia de los alelos Ccace2S y Ccace2R en un mismo cromosoma. Las dos copias del gen, al estar ligadas, originan una heterocigosis permanente. 10. Mediante la combinación de un sistema de doble PCR-RFLP y PCR cuantitativa, se ha detectado la duplicación Ccace2RS en la mayoría de poblaciones españolas de C. capitata analizadas, oscilando su frecuencia entre el 5% y el 35%. 11. Los dos genes que conforman la duplicación son funcionales, ya que en los individuos que portan la duplicación aumenta la expresión del gen Ccace2 y la actividad acetilcolinesterasa, manteniéndose al mismo tiempo la baja sensibilidad al malaoxón, forma activa del malatión. 12. Se ha observado un coste biológico asociado a la duplicación génica que se manifiesta en una disminución de la supervivencia de los adultos de la línea W-10Km. 13. Los conocimientos adquiridos en esta Tesis serán de utilidad para el seguimiento y manejo de la resistencia y, por tanto, podrán contribuir a la sostenibilidad del uso de insecticidas para el control de C. capitata. 180 BIBLIOGRAFÍA 181 Bibliografía ______________________________________________________________________________________________________________________________________ Abbott, W. S. (1925). A method of computing the effectiveness of an insecticide. J. Econ. Entomol. 18: 265-267. Adams, M. D., Celniker, S. E., Holt, R. A., Evans, C. A., Gocayne, J. D., et al. (2000). The genome sequence of Drosophila melanogaster. Science. 287: 2185-2195. Agosin, M. & Dinamarca, M. L. (1963). The effect of DDT on the level of di- and triphosphopyridine nucleotides in Triatoma infestans. Exp. Parasitol. 13: 199-203. Agosin, M. (1985). Role of microsomal oxidations in insecticide degradation. In: Kerkut, G.A., Gilbert, L.I. (Eds.), Comprehensive Insect Physiology, Biochemistry and Pharmacology, vol. 12. Pergamon, Oxford. 647-712. Ahmad, S. (1986). Enzymatic adaptations of herbivorous insects and mites to phytochemicals. J. Chem. Ecol. 12: 533-560. Ahmad, M. & Hollingworth, R. M. (2004). Synergism of insecticides provides evidence of metabolic mechanisms of resistance in the obliquebanded leafroller Choristoneura rosaceana (Lepidoptera: Tortricidae). Pest Manag. Sci. 60: 465-473. Alemany A., Miranda, M. A., Alonso, R & Martín Escorza, C. (2004). Efectividad del trampeo masivo de hembras de Ceratitis capitata (Diptera: Tephritidae) a base de atrayentes alimentarios. "Efecto-borde" y papel de los frutales abandonados como potenciadores de la plaga. Bol. San. Veg. Plagas. 30 (1-2): 255-264. Aldridge, W. N. (1950) Some properties of specific cholinesterase with particular reference to the mechanism of inhibition by diethyl p-nitrophenyl thiophosphate (E 605) and analogues. Biochem J. 46: 451-460. Alou, L. P. A., Koffi, P. A., Adja, M.A., Tia, E., Kouassi, P. K., et al. (2010) Distribution of ace-1R and resistance to carbamates and organophosphates in Anopheles gambiae s.s populations from Cote d'Ivoire. Malar. J. 9: 167. Alout, H., Berthomieu, A., Cui, F., Tan, Y., Berticat, C., et al. (2007) Different amino-acid substitutions confer insecticide resistance through acetyleholinesterase 1 insensitivity in Culex vishnui and Culex tritaeniorhynchus (Diptera : Culicidae) from China. J. Med. Entomol. 44: 463-469. 183 Bibliografía ______________________________________________________________________________________________________________________________________ Alout, H., Labbe, P., Berthomieu, A., Pasteur, N. & Weill, M. (2009). Multiple duplications of the rare ace-1 mutation F290V in Culex pipiens natural populations. Insect Biochem. Mol. Biol. 39: 884-891. Alout, H., Labbé, P., Pasteur, N. & Weill, M. (2011). High incidence of ace-1 duplicated haplotypes in resistant Culex pipiens mosquitoes from Algeria. Insect Biochem. Mol. Biol. 41: 29-35. Aluja, M. & Mangan, R. L. (2008). Fruit fly (Diptera: Tephritidae) host status determination: critical conceptual, methodological, and regulatory considerations. Annu. Rev. Entomol. 53: 473502. Anthony, N., Unruh, T., Ganser, D. & ffrench-Constant, R. (1998). Duplication of the Rdl GABA receptor subunit gene in an insecticide-resistant aphid, Myzus persicae. Mol. Gen. Genet. 260: 165-175. Antonio-Nkondjio, C., Tene Fossog, B., Ndo, C., Menze Djantio, B., Zebaze Togouet, S., AwonoAmbene, P., Costantini, C., Wondji, C. & Ranson, H. (2011). Anopheles gambiae distribution and insecticide resistance in the cities of Douala and Yaoundé (Cameroon): influence of urban agriculture and pollution. Malar. J. 10: 154. Arbeitman, M. N., Fleming, A. A., Siegal, M. L., Null, B. H. & Baker, B.S. (2004). A genomic analysis of Drosophila somatic sexual differentiation and its regulation. Development. 131: 20072021. Arensburger, P., Megy, K., Waterhouse, R. M., Abrudan, J., Amedeo, P., et al. (2010). Sequencing of Culex quinquefasciatus establishes a platform for mosquito comparative genomics. Science. 330: 86-88. Argilés, R. & Tejedo, V. (2007). La lucha contra la mosca de la fruta mediante la técnica del insecto estéril en la Comunidad Valenciana. Monografía Ceratitis capitata. Levante agrícola. 385: 157-162. Baek J. H., Kim, J. I., Lee, D. W., Chung, B. K., Miyata, T., et al. (2005). Identification and characterization of ace1-type acetylcholinesterase likely associated with organophosphate resistance in Plutella xylostella. Pestic. Biochem. Physiol. 81:164-175. 184 Bibliografía ______________________________________________________________________________________________________________________________________ Bass, C. & Field, L. M. (2011). Gene amplification and insecticide resistance. Pest. Manag. Sci., 67: 886-890. Bateman, M. A. (1972). The ecology of fruit flies. Ann. Rev. Entomol. 17: 493-518. Bautista, M. A., Miyata, T., Miura, K., & Tanaka, T. (2009). RNA interference-mediated knockdown of a cytochrome P450, CYP6BG1, from the diamondback moth, Plutella xylostella, reduces larval resistance to permethrin. Insect Biochem. Mol. Biol. 39: 38-46. Beitia, F.; Sabater-Muñoz, B. & Malagón, J. (2011). Estrategias de manejo integrado de la mosca mediterránea de la fruta en la Comunidad Valenciana. Vida Rural. 323: 52-58. Beitia, F., Pérez-Hinarejos, M., Santiago, S., Garzón, E., Tarazona, I. & Falcó, J. V. (2007). Control biológico con parasitoides. Levante Agrícola. 385: 145-150. Ben Cheikh, R., Berticat, C., Berthomieu, A.,et al. (2009). Genes conferring resistance to organophosphorus insecticides in Culex pipiens (Diptera: Culicidae) from Tunisia. J. Med. Entomol. 46(3): 523-530. Bernard, C. B., & Philogene, B. J. (1993). Insecticide synergists: role, importance, and perspectives. J. Toxicol. Environ. Health. 38: 199-223. Berenbaum, M. R. (2002). Postgenomic chemical ecology: From genetic code to ecological interactions. J. Chem. Ecol. 28: 873-896. Beroiz, B., Ortego, F., Callejas, C., Hernandez-Crespo, P., Castañera P. & Ochando, M. D. (2012). Genetic structure of Spanish populations of Ceratitis capitata revealed by RAPD and ISSR markers: implications for resistance management. Span. J. Agric. Res. 10(3): 815-825. Berticat, C., Dubois, M. P., Marquine, M., Chevillon, C. & Raymond, M. (2000). A molecular test to identify resistance alleles at the ampliÞed esterase locus in the mosquito Culex pipiens. Pest. Manag. Sci. 56: 727-731. Bhaskara, S., Dean, E. D., Lam, V., & Ganguly, R. (2006). Induction of two cytochrome P450 genes, Cyp6a2 and Cyp6a8, of Drosophila melanogaster by caffeine in adult flies and in cell culture. Gene. 377: 56-64. 185 Bibliografía ______________________________________________________________________________________________________________________________________ Bisset, J., Rodrıíguez, M., Soca, A., Pasteur, N. & Raymond, M. (1997) Cross-resistance to pyrethroid and organophosphorus insecticides in the southern house mosquito (Diptera: Culicidae) from Cuba. J. Med. Entomol. 34: 244-246. Bogwitz, M. R., Chung, H., Magoc, L., Rigby, S., Wong, W., et al. (2005). Cyp12a4 confers lufenuron resistance in a natural population of Drosophila melanogaster. Proc. Natl. Acad. Sci. USA. 102: 12807-12812. Bourguet, D., Raymond, M., Fournier, D., Malcolm, C. A., Toutant, J. P. & Arpagaus, M. (1996a). Existence of two acetylcholinesterases in the mosquito Culex pipiens (Diptera: Culicidae) J. Neurochem. 67: 2115-2123. Bourguet, D., Raymond, M., Bisset, J., Pasteur, N. & Arpagaus, M. (1996b). Duplication of the Ace.1 locus in Culex pipiens mosquitoes from the Caribbean. Biochem. Genet. 34: 351-362. Bradford, M. M. (1976). A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein dye binding. Anal. Biochem. 72: 248-254. Brown, D., Zhang, L., Wen, Z. & Scott, J. G. (2003). Induction of P450 monooxygenases in the German cockroach, Blattella germanica. L. Arch. Insect Biochem. Physiol. 53: 119-124. Brun, A., Cuany, A., Le Mouel, T., Berge, J. & Amichot, M. (1996). Inducibility of the Drosophila melanogaster cytochrome P450 gene, CYP6A2, by phenobarbital in insecticide susceptible or resistant strains. Insect Biochem. Mol. Biol. 26(7): 697-703. Buss, D. (2008) Interaction of pesticides with p-glycoprotein and other ABC proteins: A survey of the possible importance to insecticide, herbicide and fungicide resistance. Pest. Biochem. Physiol. 90(3): 141-153. Campbell, P. M., J. L. Yen, A. Masoumi, R. J. Russell, P. Batterham, J. A. McKenzie & J. G. Oakeshott. (1998a). Cross-resistance patterns among Lucilia cuprina (Diptera: Calliphoridae) resistant to organophosphorus insecticides. J. Econ. Entomol. 91: 367-375. Campbell, P. M., Newcomb, R. D., Russell, R. J. & Oakeshott, J. G. (1998b). Two different amino acid substitutions in the ali-esterase, E3, confer alternative types of organophosphorous insecticide resistance in the sheep blow fly, Lucilia cuprina. Insect Biochem. Mol. Biol. 28: 139-150. 186 Bibliografía ______________________________________________________________________________________________________________________________________ Cariño, F. A., Koener, J.F., Plapp, F. W. & Feyereisen, R. (1994). Constitutive overexpression of the cytochrome P450 gene CYP6A1 in a house fly strain with metabolic resistance to insecticides. Insect Biochem. Mol. Biol. 24(4): 411-418. Casaña-Giner, V., Gandia-Balaguer, A., Mengod-Puerta, C., Primo-Millo, J. & Primo-Yufera, E.. (1999). Insect growth regulators as chemosterilants for Ceratitis capitata (Diptera: Tephritidae). J. Econ. Entomol. 92: 303-308. Cassanelli, S., Cerchiari, B., Giannini, S., Bizzaro, D., Mazzoni, E. & Manicardi, G. C. (2005). Use of the RFLP-PCR diagnostic test for characterizing MACE and kdr insecticide resistance in the peach potato aphid, Myzus persicae. Pest Manag. Sci. 61: 91-96. Cassanelli, S., Reyes, M., Rault, M., Manicardi, G. C. & Sauphanor, B. (2006). Acetylcholinesterase mutation in an insecticide-resistant population of codling moth Cydia pomonella (L.). Insect Biochem. Mol. Biol. 36: 642-653. Castañera, P. (2003). Control integrado de la mosca mediterránea de la fruta, Ceratitis capitata (Wiedemann) (Diptera: Tephritidae) en cítricos. Phytoma España. 153: 131-133. Chen, W. L., & C. N. Sun, C. N. (1994). Purification and characterization of carboxylesterases of a rice brown planthopper, Nilaparvata lugens Stal. Insect. Biochem. Mol. Biol. 24: 347-355. Christenson, L. D. & Foote, R. H. (1960). Biology of fruit flies. Ann. Rev. Entomol. 5: 171-192. Chung, H., Boey, A., Lumb, C., Willoughby, L., Batterham, P. & Daborn, P. J. (2011). Induction of a detoxification gene in Drosophila melanogaster requires an interaction between tissue specific enhancers and a novel cis-regulatory element, Insect Biochem. Mol. Biol. 41: 863871. Cifuentes, D., Chynoweth, R., Guillén, J., De La Rúa, P. & Bielza, P. (2012). Novel Cytochrome P450 Genes, CYP6EB1 and CYP6EC1, are Over-Expressed in Acrinathrin-Resistant Frankliniella occidentalis (Thysanoptera: Thripidae). J. Econ. Entomol. 105(3): 1006-1018. Claudianos, C., Russell, R. J. & Oakeshott, J. G. (1999). The same amino acid substitution in orthologous esterases confers organophosphate resistance on the house fly and a blowfly. Insect Biochem. Mol. Biol. 29: 675-686. 187 Bibliografía ______________________________________________________________________________________________________________________________________ Claudianos, C., Ranson, H., Johnson, R. M., Biswas, S., Schuler, M. A., et al. (2006). A deficit of detoxification enzymes: Pesticide sensitivity and environmental response in the honeybee. Insect Mol. Biol. 15: 615-636. Cohen, M. B., Schuler, M. A., & Berenbaum, M. R. (1992). A host-inducible cytochrome P-450 from a host-specific caterpillar: Molecular cloning and evolution. Proc. Natl. Acad. Sci. USA. 89: 10920-10924. Copeland, R. S., Wharton, R., Luke, Q. & De Meyer, M. (2002). Indigenous host of Ceratitis capitata (Diptera: Tephritidae) in Kenya. Ann. Entomol. Soc. Am. 95: 672-694. Cornish-Bowden, A. (1985). Nomenclature for incompletely specified bases in nucleic acid sequences: recommendations. Nucl. Acids Res. 13(9): 3021-3030. Dabiré, K. R., Diabaté, A., Namontougou, M., Djogbenou, L., Kengne, P., Simard, F., Bass, C. & Baldet, T. (2009). Distribution of insensitive acetylcholinesterase (ace-1R) in Anopheles gambiae s.l. populations from Burkina Faso (West Africa). Trop. Med. Int. Health. 14: 396403. Daborn, P., Boundy, S., Yen J., Pittendrigh, B. & ffrench-Constant, R. (2001). DDT resistance in Drosophila correlates with Cyp6g1 over-expression and confers cross-resistance to the neonicotinoid imidacloprid. Mol. Genet. Genomics. 266: 556-563. Daborn, P. J., Yen, J. L., Bogwitz, M. R., Le Goff, G., Feil, E., et al. (2002). A single P450 allele associated with insecticide resistance in Drosophila. Science. 297: 2253-2256. Danielson, P. B., MacIntyre, R. J., & Fogleman, J. C. (1997). Molecular cloning of a family of xenobiotic-inducible drosophilid cytochrome p450s: Evidence for involvement in hostplant allelochemical resistance. Proc. Natl. Acad. Sci. USA. 94: 10797-10802. Danielson, P. B., Foster, J. L., McMahill, M. M., Smith, M. K., Fogleman, J. C. (1998). Induction by alkaloids and phenobarbital of Family 4 Cytochrome P450s in Drosophila: evidence for involvement in host plant utilization. Mol. Gen. Genet. 259: 54-59. Danielson, P. B., Foster, J. L., Cooper, S. K., & Fogleman, J. C. (1999). Diversity of expressed cytochrome P450 genes in the adult Mediterranean Fruit Fly, Ceratitis capitata. Insect Mol. Biol. 8: 149-159. 188 Bibliografía ______________________________________________________________________________________________________________________________________ Davidson G. (1958). Studies on insecticide resistance in anopheline mosquitos. Bull. World Health Organ. 18: 579-621. Davies, N., Villablanca, F. X. & Roderick, G. K. (1999). Bioinvasions of the medfly Ceratitis capitata: source estimation using DNA sequences at multiple intron loci. Genetics. 153: 351-360. Devonshire, A. L. & Field, L. M. (1991). Gene amplification and insecticide resistance. Annu. Rev. Entomol. 36:1-23. Djogbénou, L., Dabiré, R., Diabaté, A., Kengne, P., Akogbeto, M., Hougard, J. M. & Chandre, F. (2008) Identification and geographic distribution of the ACE-1R mutation in the malaria vector Anopheles gambiae in south-western Burkina Faso, West Africa. Am. J. Trop. Med. Hyg. 78: 298-302. Djogbénou, L., Labbé, P., Chandre, F., Pasteur, N. & Weill, M. (2009). Ace-1 duplication in Anopheles gambiae: a challenge for malaria control. Malar. J. 8: 70. Dunkov, B. C., Rodriguez-Arnaiz, R., Pittendrigh, B., ffrench-Constant, R. H. & Feyereisen, R. (1996). Cytochrome P450 gene clusters in Drosophila melanogaster. Mol. Gen. Genet. 251: 290-297. Dunkov, B. C., Guzov, V. M., Mocelin, G., Shotkoski, F., Brun, A., et al. (1997). The Drosophila cytochrome P450 gene Cyp6a2: Structure, localization, heterologous expression, and induction by phenobarbital. DNA Cell. Biol. 16: 1345-1356. El Saidy, M. F., Auda, M. & Degheele, D. (1989). Detoxification mechanisms of diflubenzuron and teflubenzuron in the larvae of Spodoptera littoralis (Boisd.). Pest Biochem. Physiol. 35: 211-222. Ellman, G. L. (1959). Tissue sulfhydryl groups. Arch. Biochem. Biophys. 82: 70-77. Ellman, G. L., Courtney, K. D., Andrews, V. & Featherstone, R. M. (1961). A new and rapid colorimetric determination of acetylcholinesterase activity. Biochem. Pharmacol. 7: 88-95. Enayati, A. A., Ranson, H. & Hemingway, J. (2005). Insect glutathione transferases and insecticide resistance. Insect Mol. Biol. 14: 3-8. 189 Bibliografía ______________________________________________________________________________________________________________________________________ EPPO (2011). Ceratitis capitata. EPPO Bulletin. 41: 340-346. Espinosa, P. J., Contreras, J., Quinto, V., Gravalos, C., Fernandez, E. & Bielza, P. (2005). Metabolic mechanisms of insecticide resistance in the western flower thrips, Frankliniella occidentalis (Pergande). Pest Manag. Sci. 61: 1009-1015. Eto, M. (1974). Organophosphorus Pesticides: Organic and Biological Chemestry. CRC Press, Cleveland. FAO, Faostat (consultado en 2012). Food and Agriculture Organization of the United Nations http://faostat.fao.org/site/291/default.aspx Feyereisen, R., Koener, J. F., Farnsworth, D. E., & Nebert, D. W. (1989). Isolation and sequence of cDNA encoding a cytochrome P-450 from an insecticide-resistant strain of the house fly, Musca domestica. Proc. Natl. Acad. Sci. USA. 86: 1465-1469. Feyereisen, R. (1995). Molecular biology of insecticide resistance. Toxicol. Lett. 82/83: 83-90. Feyereisen, R. (1999). Insect P450 enzymes. Annu. Rev. Entomol. 44: 507-533. Feyereisen, R. (2005). Insect cytochrome P450. In L. I. Gilbert, K. Iatrou, & S. S. Gill (Eds.), Comprehensive Molecular Insect Science. 4: 1-77. Oxford, UK: Elsevier. Feyereisen, R. (2006). Evolution of insect P450. Biochem. Soc. Trans. 34: 1252-1255. Feyereisen, R. (2011). Arthropod CYPomes illustrate the tempo and mode in P450 evolution. Biochim. Biophys. Acta. 1814: 19-28. Feyereisen, R. (2012). Insect CYP genes and P450 enzymes. In Gilbert, L. I. (Eds.), Comprehensive Molecular Insect Science. 7: 236-316. Oxford, UK: Elsevier. ffrench-Constant, R. H., Pittendrigh, B., Vaughan, A. & Anthony, N. (1998). Why are thereso few resistance-associated mutations in insecticide target genes? Phil. Trans. R. Soc. Lond. B. 353: 1685-1693. ffrench-Constant, R. H. (1999). Target site mediated insecticide resistance: what questions remain? Insect Biochem. Mol. Biol. 29: 397-403. 190 Bibliografía ______________________________________________________________________________________________________________________________________ ffrench-Constant, R. H., Park, Y., & Feyereisen, R. (1999). Molecular biology of insecticide resistance. In A. Puga, & K. B. Wallace (Eds.), Molecular Biology of the Toxic Response (533-551). London, UK: Taylor & Francis. ffrench-Constant, R. H., Daborn, P. J. & Le Goff, G. (2004). The genetics and genomics of insecticide resistance. Trends in Genet. 20: 163-170. Field, L. M., & Devonshire, A. L. (1998). Evidence that the E4 and FE4 esterase genes responsible for insecticide resistance in the aphid Myzus persicae (Sulzer) are part of a gene family. Biochem. J. 330: 169-173. Field, L. M., Blackman, R. L., Tyler-Smith, C. & Devonshire, A. L. (1999). Relationship between amount of esterase and gene copy number in insecticide-resistant Myzus persicae (Sulzer). Biochem. J. 339: 737-742. Fimiani, P. (1989). Mediterranean region, pp. 39-50. In A. S. Robinson and G. Hoorper [eds.], World Crop Pests. Vol. 3A. Fruit flies. Their Biology, Natural Enemies and Control. Elsevier, Amsterdam, The Netherlands. Fisher-Colbrie, P. & Busch-Petersen, E. (1989). Temperate Europe and west Asia, pp. 91-99. In A. S. Robinson and G. Hooper [eds.], World Crop Pests. Vol. 3A. Fruit flies. Their Biology, Natural Enemies and Control. Elsevier, Amsterdam, The Netherlands. Fletcher, B. S. (1989). Movements of tephritid fruit flies, pp. 209-217. In A. S. Robinson and G. Hooper [eds.], World Crop Pests. Vol. 3B. Fruit flies. Their Biology, Natural Enemies and Control. Elsevier, Amsterdam, The Netherlands. Foil, L. D., Guerrero, F. D. & Bendele, K. G. (2010). Detection of target site resistance to pyrethroids and organophosphates in the horn fly using multiplex polymerase chain reaction. J. Med. Entomol. 47(5): 855-861. Fonseca-González I., Cárdenas R., Quiñones M. L., McAllister J. & Brogdon W.G. (2009). Pyrethroid and organophosphates resistance in Anopheles (N.) nuneztovari Gabaldón populations from malaria endemic areas. Colombia Parasitology Research. 105(5): 1399-1409. Fournier, D., Mutero, A., Pralavorio, M., Bride, J. M. (1993). Drosophila acetylcholinesterase: Mechanisms of resistance to organophosphates. Chem. Biol. Interact. 87(1-3), 233-238. 191 Bibliografía ______________________________________________________________________________________________________________________________________ Fournier, D. & Mutero, A. (1994). Modification of acetylcholinesterase as a mechanism of resistance to insecticides. Comp. Biochem. Physiol. 108C: 19-31. Fournier, D. (2005). Mutations of acetylcholinesterase which confer insecticide resistance in insect populations, Chem. Biol. Interact. 157: 257-261. Friedenauer, S. & Berlet, H. H. (1989). Sensitivity and variability of the Bradford protein assay in the presence of detergents. Anal. Biochem. 178(2): 263-268. Fuji-Kuriyama, Y., Mizukami, Y., Kawajiri, K., Sogawa, K., & Muramatsu, M. (1982). Primary structure of a cytochrome P-450: Coding nucleotide sequence of phenobarbital-inducible cytochrome P-450 cDNA from rat liver. Proc. Natl. Acad. Sci. USA. 79: 2793-2797. Gahan, L. J., Pauchet, Y., Vogel, H. & Heckel, D. G. (2010). An ABC transporter mutation is correlated with insect resistance to Bacillus thuringiensis Cry1Ac toxin. PLoS Genet. 6(12): e1001248. Gandhi, R., Varak, E. & Goldberg, M. L. (1992). Molecular analysis of a cytochrome P450 gene of family 4 on the Drosophila X chromosome. DNA Cell. Biol. 11: 397-404. Gagou, M., Ballesta, J. P. & Kouyanou, S. (2000). Cloning and characterization of the ribosomal protein Ccp0 of the medfly Ceratitis capitata. Insect Mol. Biol. 9(1): 47-55. Gilmore, J. E. (1989). Sterile insect technique (SIT), pp. 353-363. In A. S. Robinson and G. Hoorper [eds.], World Crop Pests. Vol. 3B. Fruit flies. Their Biology, Natural Enemies and Control. Elsevier, Amsterdam, The Netherlands. Giraudo, M., Unnithan, G. C., Le Goff, G. & Feyereisen, R. (2010). Regulation of cytochrome P450 expression in Drosophila: Genomic insights. Pestic. Biochem. Physiol. 97: 115-122. Griffiths, A. J. F., Wessler, S. R., Lewontin, R. C. & Carroll, S. B. (2008). Genética. 9ª Edición en español. McGraw-Hill. Guengerich, F. P. (2001). Common and uncommon cytochrome P450 reactions related to metabolism and chemical toxicity. Chem. Res. Toxicol., 14: 611-650. 192 Bibliografía ______________________________________________________________________________________________________________________________________ Guerrero, F. D. (2000). Cloning of a horn fly cDNA, HialphaE7, encoding an esterase whose transcript concentration is elevated in diazinon-resistant flies. Insect Biochem. Mol. Biol. 30: 1107-1115. Gurr, G. M. & Kvedaras, O. L. (2010). Synergizing biological control: Scope for sterile insect technique, induced plant defences and cultural techniques to enhance natural enemy impact. Biol. Control. 52(3): 198-207. Hagen, K. S., Allen, W. W. & Tassan, R. L. (1981). Mediterranean fruit fly the worst may be yet to come. Calif. Agric. 35: 5-7. Haldane, J. B. S. (1954). The biochemistry of genetics. London: George Allen and Unwin. Haliassos, A., Chomel, J. C., Kruh, J., Kaplan, J. C. & Kitzis, A. (1989). Detection of minority point mutations by modified PCR technique: A new approach for a sensitive diagnosis of tumorprogression markers. Nucleic Acids Res. 17: 8093-8099. Hardstone, M. C., Komagata, O., Kasai, S., Tomita, T. & Scott, J. G. (2010). Use of isogenic strains indicates CYP9M10 is linked to permethrin resistance in Culex pipiens quinquefasciatus. Insect Mol. Biol. 19: 717-726. Hartley, C. J., Newcomb, R. D., Russell, R. J., Yong, C. G., Stevens, J. T., Yeates, D. K., La Salle, J. & Oakeshott, J. G. (2006). Amplification of DNA from preserved specimens shows blowflies were preadapted for the rapid evolution of insecticide resistance Proc. Natl. Acad. Sci. USA. 103: 8757-8762. Hawkes, N. J. & Hemingway, J. (2002). Analysis of the promoters for the β-esterase genes associated with insecticide resistance in the mosquito Culex quinquefasciatus. Biochim. Biophys. Acta 1574: 51-62. Hawkes, N. J., Janes, R. W., Hemingway, J. & Vontas, J. (2005) Detection of resistance-associated point mutations of organophosphate-insensitive acetylcholinesterase in the olive fruit fly, Bactrocera oleae (Gmelin). Pest. Biochem. Phys. 81: 154-163. 193 Bibliografía ______________________________________________________________________________________________________________________________________ Heidari, R., Devonshire, A. L., Campbell, B. E., Dorrian, S. J., Oakeshott, J. G. & Russell, R. J. (2005). Hydrolysis of pyrethroids by carboxylesterases from Lucilia cuprina and Drosophila melanogaster with active sites modified by in vitro mutagenesis. Insect Biochem. Mol. Biol. 35: 597-609. Hemingway, J. & Karunaratne, S. H. (1998). Mosquito carboxylesterases: a review of the molecular biology and biochemistry of a major insecticide resistance mechanism. Med. Vet. Entomol. 12: 1-12. Hemingway, J., Hawkes, N., Prapanthadara, L., Jayawardenal, K. G. I. & Ranson, H. (1998). The role of gene splicing, gene amplification and regulation in mosquito insecticide resistance. Philos. Trans. R. Soc. London B. 353: 1695-1699. Hemingway, J. (2000). The molecular basis of two contrasting metabolic mechanisms of insecticide resistance. Insect Biochem. Mol. Biol. 30: 1009-1015. Hemingway, J., Field, L. & Vontas, J. (2002). An overview of insecticide resistance. Science. 298: 96-97. Hemingway, J., N. J. Hawkes, L. McCarroll & H. Ranson. (2004). The molecular basis of insecticide resistance in mosquitoes. Insect Biochem. Mol. Biol. 34: 653-665. Holt, R. A., Subramanian, G. M., Halpern, A., Sutton, G. G., Charlab, R., et al. (2002). The genome sequence of the malaria mosquito Anopheles gambiae. Science. 298: 129-149. Hoy, M. A. (1998). Myths, models and mitigation of resistance to pesticides. Philos. Trans. R. Soc. Lond. B. Biol. Sci. 353: 1787-1795. Hsu, J. C. & Feng, H. T. (2002). Susceptibility of melon fly (Bactrocera cucurbitae) oriental fruit fly (B. dorsalis) to insecticides in Taiwan. Plant Prot. Bull. 44: 303-314. Hsu, J. C., Feng, H. T. & Wu, W. J. (2004a) Resistance and synergistic effects of insecticides in Bactrocera dorsalis (Diptera: Tephritidae) in Taiwan. J. Econ. Entomol. 97: 1682-1688. Hsu, J. C., Wu, W. J. & Feng, H. T. (2004b). Biochemical mechanisms of malathion resistance in oriental fruit fly (Bactrocera dorsalis), Plant Prot. Bull. 46: 55-266. 194 Bibliografía ______________________________________________________________________________________________________________________________________ Hsu, J. C. & Feng, H. T. (2006) Development of resistance to spinosad in oriental fruit fly (Diptera: Tephritidae) in laboratory selection and cross-resistance. J. Econ. Entomol. 99: 931-936. Hsu, J. C., Haymer, D. S., Wu, W. J. & Feng, H. T. (2006). Mutations in the acetylcholinesterase gene of Bactrocera dorsalis associated with resistance to organophosphorus insecticides, Insect Biochem. Mol. Biol. 36: 396-402. Hsu, J. C., Chien, T. Y., Hu, C. C., Chen, M. J. M., Wu, W. J., Feng, H. T., Haymer, D. S. & Chen, C. Y. (2012a). Discovery of genes related to insecticide resistance in Bactrocera dorsalis by functional genomic analysis of a de novo assembled transcriptome. PLoS One 7(8): e40950. Hsu, J. C., Haymer, D. S., Chou, M. Y., Feng, H. T., Chen, H. H., Huang, Y. B. & Mau, F. L. R. (2012b). Monitoring resistance to spinosad in the melon fly (Bactrocera cucurbitae) in Hawaii and Taiwan. The Scientific World Journal, vol. 2012. Article ID 750576. Huang, Y., Jiang, H.-B., Shen, G. M., Dou, W. & Wang, J. J. (2012), Molecular characterizations of two cytochrome P450 genes encoding CYP6A41 and CYP6EK1 from the oriental fruit fly, Bactrocera dorsalis (Diptera: Tephritidae). Arch. Insect. Biochem. Physiol. 79: 31-46. Huchard, E., Martinez, M., Alout, H., Douzery, E.J.P., Lutfalla, G., Berthomieu, A., Berticat, C., Raymond, M. & Weill, M. (2006). Acetylcholinesterase genes within the Diptera: takeover and loss in true flies. Proc. Roy. Soc. B. Biol. Sci. 273: 2595-2604. INE (consultado en 2012). Censo Agrario de 2009 del Insitituto Nacional de Estadística. http://www.ine.es/jaxi/menu.do?type=pcaxis&path=%2Ft01%2Fp042/E01&file=inebase&L =0 IRAC (consultado en 2012). The Insecticide Resistance Action Committee: http://www.iraconline.org/ Ishaaya, I. (1993). Insect detoxifying enzymes: their importance in pesticide synergism and resistance. Arch. Insect. Biochem. Physiol. 22: 263-276. Ivie, G. W. & Wright, J. E. (1978). Fate of diflubenzuron in the stable fly and house fly. J. Agric. Food Chem. 26: 90-94. 195 Bibliografía ______________________________________________________________________________________________________________________________________ Jacas, J. A., Urbaneja, A. & Viñuela, E. (2006). History and future of introduction of exotic arthropod biological control agents in Spain: a dilemma? BioControl. 51: 1-30. Janeira, F., Vicente, J. L., Kanganje, Y., Moreno, M., do Rosário, V. E., Cravo, P. & Pinto, J. (2008). A Primer-introduced restriction analysis-polymerase chain reaction method to detect knockdown resistance mutations in Anopheles gambiae. J. Med. Entomol. 45: 237-241. Jacobson, D. R. & Moskovits, T. (1991). Rapid, nonradioactive screening for activating ras oncogene mutations using PCR-primer introduced restriction analysis (PCR-PIRA). PCR Methods Appl. 1: 146-148. Kakani. E. G., Ioannides, I. M., Margaritopoulos, J. T., Seraphides, N. A., Skouras, P. J., Tsitsipis, J. A. & Mathiopoulos, K. D. (2008). A small deletion in the olive fly acetylcholinesterase gene associated with high levels of organophosphate resistance, Insect Biochem. Mol. Biol. 38: 781-787. Kakani, E. G., Zygouridis, N. E., Tsoumani, K. T., Seraphides, N., Zalom, F. G. & Mathiopoulos, K. D. (2010). Spinosad resistance development in wild olive fruit fly Bactrocera oleae (Diptera: Tephritidae) populations in California, Pest Manag. Sci. 66: 447-453. Kao, L. M., Wilkinson, C. F., & Brattsten, L. B. (1995). In vivo effects of 2,4-D and atrazine on cytochrome P-450 and insecticide toxicity in southern armyworm (Spodoptera eridania) larvae. Pestic. Sci. 45: 331-334. Karunaratne, S. H. P. P., Vaughan, A., Paton, M. G. & Hemingway, J. (1998). Amplification of a serine esterase gene is involved in insecticide resistance in Sri Lankan Culex tritaeniorhynchus. Insect Mol. Biol. 7: 307-15. Karunker, I., Morou, E., Nikou, D., Nauen, R., Sertchook, R., et al. (2009). Structural model and functional characterization of the Bemisia tabaci CYP6CM1vQ, a cytochrome P450 associated with high levels of imidacloprid resistance. Insect Biochem. Mol. Biol. 39: 697706. Kasai, S., & Scott, J. G. (2000). Overexpression of cytochrome P450 CYP6D1 is associated with monoxygenase-mediated pyrethroid resistance in house flies from Georgia. Pestic. Biochem. Physiol. 68: 34-41. 196 Bibliografía ______________________________________________________________________________________________________________________________________ Kasai, S., & Scott, J. G. (2001). Expression and regulation of CYP6D3 in the house fly, Musca domestica (L.). Insect Biochem. Mol. Biol. 32: 1-8. Kawada, H., Futami, K., Komagata, O., Kasai, S., Tomita, T., Sonye, G., Mwatele, C., Njenga, S. M., Mwandawiro, C., Minakawa, N. & Takagi, M. (2011). Distribution of a Knockdown Resistance Mutation (L1014S) in Anopheles gambiae s.s. and Anopheles arabiensis in Western and Southern Kenya. PLoS One. 6: 24323-24323. Khajehali, J., Van Leeuwen, T., Grispou, M., Morou, E., Alout, H., Weill, M., Tirry, L., Vontas, J. & Tsagkarakou, A. (2010). Acetylcholinesterase point mutations in European strains of Tetranychus urticae (Acari: Tetranychidae) resistant to organophosphates. Pest Manag. Sci. 66: 220-228. Kono, Y. & Tomita, T. (2006). Amino acid substitutions conferring insecticide insensitivity in Aceparalogous acetylcholinesterase. Pestic. Biochem. Physiol. 85: 123-132. Kozaki, T., Shono, T., Tomita, T. & Kono, Y. (2001). Fenitroxon insensitive acetylcholinesterases of the housefly, Musca domestica associated with point mutations. Insect Biochem. Mol. Biol. 31: 991-997. Kozaki, T., Shono, T., Tomita, T., Taylor, D. & Kono, Y. (2002). Linkage analysis of an acetylcholinesterase gene in the house fly Musca domestica (Diptera: Muscidae). J. Econ. Entomol. 95: 129-133. Krimbas, C. B. & Tsakas, S. (1971). The genetics of Dacus oleae. IV. Changes of esterase polymorphism in a natural population following insecticide control-Selection or drift? Evolution 25: 454-460. Kwon, D. H., Clark, J. M. & Lee, S. H. (2010). Extensive gene duplication of acetylcholinesterase associated with organophosphate resistance in the two-spotted spider mite. Insect Mol. Biol. 19: 195-204. Labbé, P., Berthomieu, A., Berticat, C., Alout, H., Raymond, M., et al. (2007a). Independent duplications of the acetylcholinesterase gene conferring insecticide resistance in the mosquito Culex pipiens. Mol. Biol. Evol. 24: 1056-1067. 197 Bibliografía ______________________________________________________________________________________________________________________________________ Labbé, P., Berticat, C., Berthomieu, A., Unal, S., Bernard, C., Weil,l M. & Lenormand, T. (2007b). Forty years of erratic insecticide resistance evolution in the mosquito Culex pipiens . PLoS Genet. 3: 2190-2199. Labbé, R., Caveney, S. & Donly, C. (2011), Genetic analysis of the xenobiotic resistance-associated ABC gene subfamilies of the Lepidoptera. Insect Mol. Biol. 20: 243-256. Lamb, D. C., Skaug, T., Song, H.L., Jackson, C.J., Podust, L.M., Waterman, M.R., Kell, D.B., Kelly, D.E. & Kelly, S.L. (2002). The cytochrome P450 complement (CYPome) of Streptomyces coelicolor A3(2). J. Biol. Chem. 277: 24000-24005. Le Goff, G., Boundy, S., Daborn, P. J., Yen, J. L., Sofer, L., et al. (2003). Microarray analysis of cytochrome P450 mediated insecticide resistance in Drosophila. Insect Biochem. Mol. Biol. 33: 701-708. Le Goff, G., Hilliou, F., Siegfried, B. D., Boundy, S., Wajnberg, Soferb, L., Pascaline, ffrenchConstant, R. H. & Feyereisen, R. (2006). Xenobiotic response in Drosophila melanogaster: Sex dependence of P450 and GST gene induction. Insect Biochem. Mol. Biol. 36: 674-682. Lee, D. W., Choi, J. Y., Kim, W. T., Je, Y. H., Song, J. T., et al. (2007) Mutations of acetylcholinesterase 1 contribute to prothiofos-resistance in Plutella xylostella (L.). Biochem. Biophys. Res. Commun. 353: 591-597. Lee, S. H., Kang, J. S., Min, J. S., Yoon, K. S., Strycharz, J. P., et al. (2010). Decreased detoxification genes and genome size make the human body louse an efficient model to study xenobiotic metabolism. Insect Mol. Biol. 19: 599-615. Li, X., Berenbaum, M. R., & Schuler, M. A. (2000). Molecular cloning and expression of CYP6B8: A xanthotoxininducible cytochrome P450 cDNA from Helicoverpa zea. Insect Biochem. Mol. Biol. 30: 75-84. Li, X., Berenbaum, M. R., & Schuler, M. A. (2002). Plant allelochemicals differentially regulate Helicoverpa zea cytochrome P450 genes. Insect Mol. Biol. 11: 343-351. 198 Bibliografía ______________________________________________________________________________________________________________________________________ Li, A. Y., Guerrero, F. D., Garcia, C. A. & George, J. E. (2003). Survey of resistance to permethrin and diazinon and the use of a multiplex polymerase chain reaction assay to detect resistance alleles in the horn fly, Haematobia irritans irritans (L.). J. Med. Entomol. 40: 942-949. Li, X., Shuler, M. A. & Berenbaum, M. R. (2007). Molecular mechanisms of metabolic resistance to synthetic and natural xenobiotics. Annu. Rev. Entomol. 52: 231-253. Lindsley, D. L., & Zimm, G. G. (1992). The Genome of Drosophila melanogaster. San Diego, CA: Academic Press, Inc. Liquido, N. J., Shinoda, L. A. & Cunningham, R. T. (1991). Host plants of the Mediterranean fruit fly, Ceratitis capitata (Wiedemann) (Diptera: Tephritidae): an annotated world review. Misc. Publ. Entomol. Soc. Am. 77: 1-52. Liu, N., & Scott, J. G. (1996). Genetic analysis of factors controlling high-level expression of cytochrome P450, CYP6D1, cytochrome b5, P450 reductase, and monooxygenase activities in LPR house flies, Musca domestica. Biochem. Genet. 34: 133-148. Liu, N. & Yue, X. (2000). Insecticide resistance and cross-resistance in the house fly (Diptera: Muscidae). J. Econ. Entomol. 93: 1269-1275. Lumjuan, N., McCarroll, L., Prapanthadara, L., Hemingway, J. & Ranson, H. (2005). Elevated activity of an Epsilon class glutathione transferase confers DDT resistance in the dengue vector, Aedes aegypti. Insect Biochem. Mol. Biol. 35: 861-71. Magaña, C., Hernández-Crespo, P., Ortego, F. & Castañera, P. (2007). Resistance to malathion in field populations of Ceratitis capitata. J. Econ. Entomol. 100(6): 1836-1843. Magaña, C., Hernández-Crespo, P., Brun-Barale, A., Couso-Ferrer, F., Bride, J. M., Castañera, P., Feyereisen, R. & Ortego, F. (2008). Mechanisms of resistance to malathion in the medfly Ceratitis capitata. Insect Biochem. Mol. Biol. 38: 756-762. Main, I. H. M. (1964). The inhibitory actions of prostaglandins on respiratory smooth muscle. Br. J. Pharmacol. Chemother. 22: 511-519. 199 Bibliografía ______________________________________________________________________________________________________________________________________ Maitra, S., Dombrowski, S. M., Basu, M., Raustol, O., Waters, L. C. & Ganguly, R. (2000). Factors on the third chromosome affect the level of cyp6a2 and cyp6a8 expression in Drosophila melanogaster. Gene. 248: 147-156. Malacrida, A. R., Gomulski, L. M., Bonizzoni, M., Bertin, S, Gasperi, G. & Guglielmino, C. R: (2007). Globalization and fruitfly invasion and expansion: the medfly paradigm. Genetica. 131: 1-9 Mansuy, D. (1998). The great diversity of reactions catalyzed by cytochrome P450. Comp. Biochem. Physiol. 121C: 5-14. Margaritopoulos, J. T., Skavdis, G., Kalogiannis, N., Nikou D., Morou, E., Skouras, P. J., Tsitsipis, J. A. & Vontas, J. (2008). Efficacy of the pyrethroid alpha-cypermethrin against Bactrocera oleae populations from Greece and improved diagnostic for an iAChE mutation, Pest Manag. Sci. 64: 900-908. Markussen, M. D. & Kristensen, M. (2012), Spinosad resistance in female Musca domestica L. from a field-derived population. Pest. Manag. Sci. 68: 75-82. McDonald, P. T. & McInnis, D. O. (1985). Ceratitis capitata: Effect of host fruit size on the number of eggs per clutch. Entomol. Exp. Appl. 37: 207-211. McQuilton, P., St. Pierre, S. E., Thurmond, J. & the FlyBase Consortium (2012). FlyBase 101 - the basics of navigating FlyBase. Nucleic Acids Res. 40 (Database issue): D706-14 Menozzi, P., Shi, M. A., Lougarre, A., Tang, Z. H. & Fournier, D. (2004). Mutations of acetylcholinesterase which confer insecticide resistance in Drosophila melanogaster populations. BMC Evol. Biol. 4: 4-10. Miota, F., Siegfried, B. D., Scharf, M. E., & Lydy, M. J. (2000). Atrazine induction of cytochrome P450 in Chironomus tentans larvae. Chemosphere. 40: 285-291. Monzó, C., Mollà, O., Castañera, P. & Urbaneja, A. (2009). Activity-density of Pardosa cribata in Spanish citrus orchards and its predatory capacity on Ceratitis capitata and Myzus persicae. BioControl. 54: 393-402. 200 Bibliografía ______________________________________________________________________________________________________________________________________ Monzó, C., Sabater-Muñoz, B., Urbaneja, A. & Castañera, P. (2011). The ground beetle Pseudophonus rufipes reveals as predator of Ceratitis capitata in citrus orchards. Biol. Control. 56: 17-21. Morton, R.A. & Holwerda, B.C. (1985). The oxidative metabolism of malathion and malaoxon in resistant and susceptible strains of Drosophila melanogaster. Pestic. Biochem. Physiol. 24: 19-31. Mouches, C., Magnin, M. Berge, J. B., de Silvestri, M., Beyssat, V., Pasteur, N. & Georghiou, G. P. (1987). Overproduction of detoxifying esterases in organophosphate-resistant Culex mosquitoes and their presence in other insects. Proc. Natl. Acad. Sci. USA. 84: 2113-2116. Mulder, G. J. & Ouwerkerk-Mahadevan, S. (1997). Modulation of glutathione conjugation in vivo: how to decrease glutathione conjugation in vivo or in intact cellular systems in vitro. Chem. Biol. Interact. 105: 17-34. Mutero, A., Pralavorio, M., Bride, J. M. & Fournier, D. (1994), Resistance-associated point mutations in insecticide-insensitive acetylcholinesterase. Proc. Natl. Acad. Sci. USA. 91: 5922-5926. Muñoz, A. & García-Marí (2009). Factores que influyen en la eficacia del trampeo masivo para el control de la mosca de la fruta Ceratitis capitata (Diptera: Tephritidae). Bol. San. Veg. Plagas. 35: 401-418. Narahashi, T. (1996) Neuronal ion channels as the target sites of insecticides. Pharmacol. Toxicol. 79: 1-14. Nardi, F., Carapelli, A., Vontas, J. G., Dallai, R., Roderick, G. K. & Frati, F. (2006). Geographical distribution and evolutionary history of organophosphate-resistant Ace alleles in the olive fly (Bactrocera oleae). Insect Biochem. Mol. Biol. 36: 593-602. Navarro-Llopis, V. N., Sanchis-Cabanes, J., Ayala, I., Casana-Giner, V. & Primo-Yufera, E. (2004). Efficacy of lufenuron as chemosterilant against Ceratitis capitata in field trials. Pest. Manag. Sci. 60: 914-920. 201 Bibliografía ______________________________________________________________________________________________________________________________________ Navarro-Llopis, V., Alfaro, F., Domiguez, J. Sanchis, J. & Primo, J. (2008). Evaluation of traps and lures for mass trapping of Mediterranean fruit fly in citrus groves. J. Econ. Entomol. 101(1): 126-131. Navarro-Llopis, V., Domínguez-Ruiz, J., Zarzo, M., Alfaro, C. & Primo, J. (2010). Mediterranean fruit suppression using chemosterilants for area-wide integrated pest management. Pest Manag. Sci. 66: 511-519. Nebert, D. W., Adesnik, M., Coon, M. J., Estabrook, R. W., Gonzalez, F. J., et al. (1987). The P450 gene superfamily: Recommended nomenclature. DNA. 6: 1-11. Nebert, D. W., Nelson, D. R., Coon, M. J., Estabrook, R. W., Feyereisen, R., et al. (1991). The P450 superfamily: Update on new sequences, gene mapping, recommended nomenclature. DNA Cell. Biol., 10(1): 1-14. Nelson, D. R., Kamataki, T., Waxman, D. J., Guengerich, F. P., Estabrook, R. W., et al. (1993). The P450 superfamily: Update on new sequences, gene mapping, accession numbers, early trivial names of enzymes, nomenclature. DNA Cell Biol. 12: 1-51. Nelson, D. R., Koymans, L., Kamataki, T., Stegeman, J. J., Feyereisen, R., et al. (1996). P450 superfamily: Update on new sequences, gene mapping, accession numbers and nomenclature. Pharmacogenetics, 6: 1-42. Nelson, D.R. (2009) The Cytochrome P450 Homepage. Human Genomics. 4: 59-65. Newcomb, R. D., Campbell, P. M., Ollis, D. L., Cheah, E., Russell, R. J. & Oakeshott, J. G. (1997). A single amino acid substitution converts a carboxylesterase to an organophosphorus hydrolase and confers insecticide resistance on a blowfly. Proc. Natl. Acad. Sci. USA. 94: 7464-7468. Oakeshott, J. G., C. Claudianos, P. M. Campbell, R. D. Newcomb, & R. J. Russell. (2005), Biochemical genetics and genomics of insect esterases, pp. 309-381. In L. I. Gilbert, K. Iatro, and S. Gill (eds.), Comprehensive molecular insect science. Elsevier, Oxford, United Kingdom. Omura, T., & Sato, R. (1964). The carbon monoxide-binding pigment of liver microsomes I. Evidence for its hemoprotein nature. J. Biol. Chem. 239: 2370-2378. 202 Bibliografía ______________________________________________________________________________________________________________________________________ Ortelli, F., Rossiter, L. C., Vontas, J., Ranson, H. & Hemingway, J. (2003). Heterologous expression of four glutathione transferase genes genetically linked to a major insecticide-resistance locus from the malaria vector Anopheles gambiae. Biochem. J. 373: 957-63. Ortiz de Montellano, P. R. (1995). Oxygen activation and reactivity. In T. Omura, Y. Ishimura, & Y. Fujii-Kuriyama (Eds.). Cytochrome P-450 (2nd ed.). (pp. 245-303). New York, NY: Plenum Press. Otto, S. P. & Yong, P. (2002). The evolution of gene duplicates. Adv. Genet. 46: 451-483. Pan, Y., Guo, H. & Gao, X. (2009). Carboxylesterase activity, cDNA sequence, and gene expression in malathion susceptible and resistant strains of the cotton aphid, Aphis gossypii, Comp. Biochem. Physiol. B: 152(3): 266-270. Pauchet, Y., Wilkinson, P., Vogel, H., Nelson, D. R., Reynolds, S. E., et al. (2010). Pyrosequencing the Manduca sexta larval midgut transcriptome: Messages for digestion, detoxification and defence. Insect Mol. Biol. 19: 61-75. Perry, A. S. & Buckner, A. J. (1970). Studies on microsomal cytochrome P-450 resistant and susceptible houseflies. Life Sci. 9: 335-350. Perry, A. S., Dale, W. E. & Buckner, A. J. (1971). Induction and repression of microsomal mixedfunction oxidases and cytochrome P-450 in resistant and susceptible houseflies. Pest. Biochem. Physiol. 1: 131-142. Pfaffl, M. W. (2001). A new mathematical model for relative quantification in realtime. RT-PCR. Nucleic Acids Res. 29: 2002-2007. Philpot, R. M. & Hodgson, E. (1972). Differences in the cytochrome P-450s from resistant and susceptible houseflies, Chem. Biol. Interact. 4: 399-408. Poupardin, R., Reynaud, S., Strode, C., Ranson, H., Vontas, J., & David, J. P. (2008). Crossinduction of detoxification genes by environmental xenobiotics and insecticides in the mosquito Aedes aegypti: Impact on larval tolerance to chemical insecticides. Insect Biochem. Mol. Biol. 38: 540-551. 203 Bibliografía ______________________________________________________________________________________________________________________________________ Primo-Millo, E., Alfaro-Lassala, F. & Argilés-Herrero, R. (2003). Plan de actuación contra la mosca de la fruta (Ceratitis capitata) en la Comunidad Valenciana. Phytoma España. 153: 127130. Qiao, C. L., Marquine, M., Pasteur, N. & Raymond, M. (1998). A new esterase amplification involved in OP resistance in Culex pipiens mosquitoes from China. Biochem. Genet. 36: 417-426. Quisenberry, S. S., Lockwood, J. A., Byford, R. L., Wilson, H. K. & Sparks, T. C. (1984). Pyrethroid resistance in the horn fly, Haematobia irritans (L.) (Diptera: Muscidae). J. Econ. Entomol. 77: 1095-1098. Ranasinghe, C., Headlam, M. & Hobbs, A. A. (1997). Induction of the mRNA for CYP6B2, a pyrethroid inducible cytochrome P450, in Helicoverpa armigera (Hubner) by dietary monoterpenes. Arch. Insect. Biochem. Physiol. 34: 99-109. Ranson, H., Rossiter, L., Ortelli, F., Jesen, B., Wang, X., Roth, C. W., Collins, F. H. & Hemingway, J. (2001). Identification of a novel class of insect glutathione S-transferases involved in resistance to DDT in the malaria vector Anopheles gambiae. Biochem. J. 359: 295-304. Ranson, H., Claudianos, C., Ortelli, F., Abgrall, C., Hemingway, J., et al. (2002). Evolution of supergene families associated with insecticide resistance. Science. 298: 179-181. Ranz, J. M., Castillo-Davis, C. I., Meiklejohn, C. D. & Hartl, D. L. (2003). Sex-dependent gene expression and evolution of the Drosophila transcriptome. Science. 300: 1742-1745. Raymond, M., Chevillon, C., Guillemaud, T., Lenormand, T. & Pasteur, N. (1998). An overview of the evolution of overproduced esterases in the mosquito Culex pipiens. Philos. Trans. R. Soc. London B. 353: 1-5. Raymond, M., Berticat, C., Weill, M., Pasteur, N. & Chevillon, C. (2001). Insecticide resistance in the mosquito Culex pipiens: What have we learned about adaptation? Genetica. 112/113: 287-296. Reese, M. G., Eeckman, F. H., Kulp, D. & Haussler, D. (1997). “Improved splice site detection in genie”. J. Comp. Biol. 4(3): 311-323. 204 Bibliografía ______________________________________________________________________________________________________________________________________ Reimer, L. J., Tripet, F., Slotman, M., Spielman, A., Fonjo, E. & Lanzaro, G. C. (2005). An unusual distribution of the kdr gene among populations of Anopheles gambiae on the island of Bioko, Equatorial Guinea. Insect Mol. Biol. 14: 683-688. Revuelta, L., Piulachs, M. D., Bellés, X., Castañera, P., Ortego, F., Díaz-Ruíz, J. R., HernándezCrespo, P. & Tenllado, F. (2009). RNAi of ace1 and ace2 in Blattella germanica reveals their differential contribution to acetylcholinesterase activity and sensitivity to insecticides. Insect Biochem. Mol. Biol. 39: 913-919. Revuelta, L., Ortego, F., Díaz-Ruíz, J. R., Castañera, P., Tenllado, F. & Hernández-Crespo, P. (2011). Contribution of Ldace1 gene to acetylcholinesterase activity in Colorado potato beetle, Insect Biochem. Mol. Biol. 41: 795-803. Riaz, M. A., Poupardin, R., Reynaud, S., Strode, C., Ranson, H. & David, J. P. (2009). Impact of glyphosate and benzo[a]pyrene on the tolerance of mosquito larvae to chemical insecticides. Role of detoxification genes in response to xenobiotics. Aquat. Toxicol. 93(1): 61-69. Robertson, J. L. & H. K. Preisler. (1992). Pesticide Bioassays with Arthropods. CRC Press, Boca Raton, FL. Rodríguez, M. M., Bisset, J. A., Ruiz, M. & Soca, A. (2002). Cross-resistance to pyrethroid and organophosphorus insecticides induced by selection with temephos in Aedes aegypti (Diptera: Culicidae) from Cuba. J. Med. Entomol. 39: 882-888. Ruiz-Castro, A. (1945). Fauna entomológica de la vid en España. Estudiosistemático y biológico de las especies de mayor importancia económica. III Diptera. Inst. Esp. de Entomología., Madrid Rupasinghe, S., Schuler, M. A., Kagawa, N., Yuan, H., Lei, L., et al. (2006). The cytochrome P450 gene family CYP157 does not contain EXXR in the K-helix reducing the absolute conserved P450 residues to a single cysteine. FEBS Lett.. 580: 6338-6342. Sabourault, C., Guzov, V. M., Koener, J. F., Claudianos, C., Plapp, F. W., Jr., & Feyereisen, R. (2001). Overproduction of a P450 that metabolizes diazinon is linked to a loss-of-function in the chromosome 2 ali-esterase (MdalphaE7) gene in resistant house flies. Insect Mol. Biol. 10: 609-618. 205 Bibliografía ______________________________________________________________________________________________________________________________________ Sacca, G. (1947). Sull'esistenza di mosche domestiche resistenti al DDT. Riv. Parassitol. 8: 127128. Salgado, V. L., Watson, G. B. & Sheets, J. J. (1997). Studies on the mode of action of spinosad, the active ingredient in Tracer insect control. Proc Beltwide Cotton Conf, National Cotton Council, Memphis, TN, 1082-1086. Salgado, V. L. (1998). Studies on the mode of action of spinosad: insect symptoms and physiological correlates. Pestic. Biochem. Physiol. 60: 91-102. Santolamazza, F., Calzetta, M., Etang, J., Barrese, E., Dia, I., Caccone, A., Donnelly, M. J., Petrarca, V., Simard, F., Pinto, J. & Della Torre, A. (2008). Distribution of knock-down resistance mutations in Anopheles gambiae molecular forms in west and west-central Africa Malar. J. 7: 74. Sarkar, M., Borkotoki, A., Baruah, I., Bhattacharyya, I. K. & Srivastava, R. B. (2009) Molecular analysis of knock down resistance (kdr) mutation and distribution of kdr genotypes in a wild population of Culex quinquefasciatus from India. Tropical Medicine & International Health 14(9): 1097-1104. Schumacher, M., Camp, S., Maulet, Y., Newton, M., MacPhee-Quigley, K., Taylor, S. S., Friedmann, T. & Taylor, P. (1986). Primary structure of Torpedo californica acetylcholinesterase deduced from its cDNA sequence. Nature. 319: 407-409. Scott, J. G. & Georghiou, G. P. (1986). Mechanisms responsible for high levels of permethrin resistance in the house fly. Pestic. Sci. 17: 195-206. Scott, J. G. (1991). Insecticide resistance in insects. In: Pimental, D. (Ed.), Hanbook of Pest Management. CRC Press, Boca Raton. 663-677. Scott, J. A., Collins, F. H., & Feyereisen, R. (1994). Diversity of cytochrome P450 genes in the mosquito, Anopheles albimanus. Biochem. Biophys. Res. Commun. 205: 1452-1459. Scott, J. G., Sridhar, P. & Liu, N. (1996). Adult specific expression and induction of cytochrome P450tpr in house flies. Arch. Insect Biochem. Physiol. 31: 313-323. 206 Bibliografía ______________________________________________________________________________________________________________________________________ Shang, Q., Pan, Y., Fang, K., Xi, J., Wong, A., Brennan, J. A. & Cao, C. (2012). Extensive Ace2 duplication and multiple mutations on Ace1 and Ace2 are related with high level of organophosphates resistance in Aphis gossypii. Environ. Toxicol. In press. doi: 10.1002/tox.21778 Singh, O. P., Bali, P., Hemingway, J., Subbarao, S. K., Das, A. P. & Adak, T. (2009). PCR-based methods for the detection of L1014 kdr mutation in Anopheles culicifacies sensu lato. Malar. J. 8: 154. Singh, O. P., Dykes, C. L., Das, M. K., Pradhan, S., Bhatt, R.M., Agrawal, O.P. & Adak, T. (2010) Presence of two alternative kdr-like mutations, L1014F and L1014S, and a novel mutation, V1010L, in the voltage gated Na+ channel of Anopheles culicifacies from Orissa, India. Malar. J. 9: 146. Smissaert, H. R. (1964). Cholinesterase inhibition in spider mites susceptible and resistant to organophosphate. Science. 143: 129-131. Snyder, M. J., Stevens, J. L., Andersen, J. F. & Feyereisen, R. (1995). Expression of cytochrome P450 genes of the CYP4 family in midgut and fat body of the tobacco hornworm, Manduca sexta. Arch. Biochem. Biophys. 321: 13-20. Snyder, M. J., Scott, J. A., Andersen, J. F. & Feyereisen, R. (1996). Sampling P450 diversity by cloning polymerase chain reaction products obtained with degenerate primers. Methods Enzymol. 272: 304-312. Sparks, T. C., Lockwood, J. A., Byford, R. L., Graves, J. B. & Leonard, R. B. (1989). The role of behavior in insecticide resistance. Pestic. Sci. 26: 383-399. Sparks, T. C., Dripps, J. E., Watson, G. B. & Paroonagian, D. (2012). Resistance and crossresistance to the spinosyns - A review and analysis, Pestic. Biochem. Physiol. 102(1): 1-10. Spofford, J. B. (1969). Heterosis and the evolution of duplications. Am. Nat. 103: 407-432. Stasinakis, P., Katsares, V. & Mavragani-Tsipidou, P. (2001). Organophosphate resistanceand allelic frequencies of esterases in the olive fruit fly Bactrocera oleae (Diptera: Tephritidae), J. Ag. Urban Entomol. 18: 157-168. 207 Bibliografía ______________________________________________________________________________________________________________________________________ Stevens, J. L., Snyder, M. J., Koener, J. F. & Feyereisen, R. (2000). Inducible P450s of the CYP9 family from larval Manduca sexta midgut. Insect Biochem. Mol. Biol. 30: 559-568. Strode, C., Steen, K., Ortelli, F. & Ranson, H. (2006). Differential expression of the detoxification genes in the different life stages of the malaria vector Anopheles gambiae. Insect Mol. Biol. 15: 523-530. Sun, W., Margam, V. M., Sun, L., Buczkowski, G., Bennett, G. W., Schemerhorn, B., Muir, W. M. & Pittendrigh, B. R. (2006). Genome-wide analysis of phenobarbital-inducible genes in Drosophila melanogaster. Insect Mol. Biol. 15: 455-464. Tao, L. M., Yang, J. Z., Zhuang, P. J. & Tang, Z. H. (2006). Effect of a mixture of iprobenfos and malathion on the development of malathion resistance in the mosquito Culex pipiens pallens Coq. Pest Manag. Sci. 62: 86-90. Taskin, V. & Kence, M. (2004). The genetic basis of malathion resistance in housefly (Musca domestica L.) strains from Turkey. Russ. J. Genet. 40: 1215-1222. Tate, L. G., Plapp, F. W. JR. & Hodgson, E. (1973). Cytochrome P-450 difference spectra of rnicrosomes from several different insecticide-resistant and susceptible strains of housefly, Musca domestica. Chem. Biol. Interact. 6: 237-247. Thompson, J. D., Gibson, T. J., Plewniak, F., Jeanmougin, F. & Higgins, D. G. (1997) The ClustalX windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Res. 25: 4876-4882. Tijet, N., Helvig, C. & Feyereisen, R. (2001). The cytochrome P450 gene superfamily in Drosophila melanogaster: Annotation, intron-exon organization and phylogeny. Gene. 262: 189-198. Toutant, J. P. (1989). Insect acetylcholinesterase: catalytic properties, tissue distribution and molecular forms. Prog. Neurobiol. 32: 423-446. Tsakas, S. & Krimbas, C. B. (1970). The genetics of Dacus oleae. IV. Relation between adult esterase genotypes survival to organophosphate insecticides. Evolution. 24: 807-815. 208 Bibliografía ______________________________________________________________________________________________________________________________________ Tsoumani, K., Augustinos, A. A., Kakani, E. G. & Mathiopoulos, K. D. (2011). Isolation annotation applications of expressed sequence tags from the olive fly Bactrocera oleae, Mol. Genet. Genomics. 285: 33-45. Urbaneja, A., Mari, F. G., Tortosa, D., Navarro, C., Vanaclocha, P., Bargues, L. & Castanera, P. (2006). Influence of ground predators on the survival of the mediterranean fruit fly pupae, Ceratitis capitata, in Spanish citrus orchards. BioControl 51: 611-626. Urbaneja, A., Chueca, P., Montó, H., Pascual-Ruiz, S., Dembilio, O., Vanaclocha, P., Abad-Moyano, R., Pina, T. & Castañera, P.. 2009. Chemical alternatives to malathion for controlling Ceratitis capitata (Diptera: Tephritidae), and their side effects on natural enemies in Spanish citrus orchards. J. Econ. Entomol. 102: 144-151. Verhaeghen, K., Van Bortel, W., Roelants, P., Okello, P. E., Talisuna, A. & Coosemans, M. (2010). Spatio-temporal patterns in kdr frequency in permethrin and DDT resistant Anopheles gambiae s.s. from Uganda. Am. J. Trop. Med. Hyg. 82: 566-573. Villatte, F., Ziliani, P., Marcel, V., Menozzi, P. & Fournier, D. (2000). A high number of mutations in insect acetylcholinesterase may provide insecticide resistance. Pest Biochem. Physiol. 67: 95-102. Villatte, F. & Bachmann, T. T. (2002). How many genes encode cholinesterase in arthropods? Pest. Biochem. Physiol. 73: 122-129. Viñuela, E. & Arroyo, M. (1982). Resultados del ensayo de la susceptibilidad de 6 poblaciones españolas de Ceratitis capitata Wied. para detección de su posible resistencia a los insecticidas. An. INIA/ Ser. Agríc. 21: 183-192. Viñuela, E. (1998). La resistencia a insecticidas en España. Bol. San. Veg. Plagas 24: 487-496. Vontas J. D., Small, G. J. & Hemingway, J. (2000). Comparison of esterase gene amplification, gene expression and esterase activity in insecticide susceptible and resistant strains of the brown planthopper, Nilaparvata lugens (Stal). Insect Mol. Biol. 9: 655-60. Vontas J. G., Cosmidis, N., Loukas, M., Tsakas, S., Hejazi, M. J., Ayoutanti, A. & Hemingway, J. (2001), Altered acetylcholinesterase confers organophosphate resistance in the olive fruit fly Bactrocera oleae, Pestic. Biochem. Physiol. 71: 124- 132. 209 Bibliografía ______________________________________________________________________________________________________________________________________ Vontas, J. G., Hejazi, M. J., Hawkes, N. J., Cosmidis, N., Loukas, M. & Hemingway, J. (2002), Resistance-associated point mutations of organophosphate insensitive acetylcholinesterase in the olive fruit fly Bactrocera oleae, Insect Mol. Biol. 11: 329-336. Vontas, J., Hernández-Crespo, P., Margaritopoulos, J. T, Ortego, F., Feng, H.-T, Mathiopoulos, K. D. & Hsu, J. C. (2011). Insecticide resistance in Tephritid flies, Pestic. Biochem. Physiol. 100(3): 199-205. Walsh, S. B., Dolden, T. A., Moores, G. D., Kristensen, M., Lewis, T., Devonshire, A. L. & Williamson, M. S. (2001). Identification and characterization of mutations in housefly (Musca domestica) acetylcholinesterase involved in insecticide resistance. Biochem. J. 359: 175-181. Waters, L. C., Zelhof, A. C., Shaw, B. J. & Chang, L. Y. (1992). Possible involvement of the long terminal repeat of transposable element 17.6 in regulating expression of an insecticide resistance-associated P450 gene in Drosophila. [published erratum appears in Proc. Natl. Acad. Sci. USA 1992 Dec 15;89(24):12209] Proc. Natl. Acad. Sci. USA. 89: 4855-4859. Watson, J. D. & Crick, F. H. (1953). Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid. Nature. 171(4356): 737-738. Watson, G. B., Chouinard, S. W., Cook, K. R., Geng, C., Gifford, J. M., Gustafson, G. D., Hasler, J. M., Larrinua, I. M., Letherer, T. J., Mitchell, J. C., Pak, W. L., Salgado, V. L., Sparks, T. C. & Stilwell, G. E. (2010). A spinosyn-sensitive Drosophila melanogaster nicotinic acetylcholine receptor identified through chemically induced target site resistance, resistance gene identification, and heterologous expresión. Insect Biochem. Mol. Biol. 40: 376-384. Wee, C. W., Lee, S. F., Robin, C., & Heckel, D. G. (2008). Identification of candidate genes for fenvalerate resistance in Helicoverpa armigera using cDNA-AFLP. Insect Mol. Biol. 17: 351360. Weill, M., Fort, P., Berthomieu, A., Dubois, M. P., Pasteur, N. & Raymond, M. (2002). A novel acetylcholinesterase gene in mosquitoes codes for the insecticide target and is nonhomologous to the ace gene in Drosophila. Proc. Roy. Soc. London B Biol. Sci. 269: 20072016. 210 Bibliografía ______________________________________________________________________________________________________________________________________ Weill, M., Lutfalla, G., Mogensen, K., Chandre, F., Berthomieu, A., Berticat, C., Pasteur, N., Philips, A., Fort, P. & Raymond, M. (2003). Comparative genomics: insecticide resistance in mosquito vectors. Nature. 423: 136-137. Weill, M., Malcolm, C., Chandre, F., Mogensen, K., Berthomieu, A., Marquine, M. & Raymond, M. (2004). The unique mutation in ace-1 giving high insecticide resistance is easily detectable in mosquito vectors. Insect Mol. Biol. 13: 1-7. Welling, A. W., DeVries, A. W. & Voerman, S., (1974). Oxidative cleavage of a carboxyester bond as a mechanism of resistance to malaoxon in house-flies. Pestic. Biochem. Physiol. 4: 3143. Wen, Z., & Scott, J. G. (1997) Cross-resistance to imidacloprid in strains of German cockroach (Blattella germanica) and house fly (Musca domestica). Pestic Sci. 49: 367-371. Wen, Z., Zeng, R. S., Niu, G., Berenbaum, M. R. & Schuler, M. A. (2009). Ecological significance of induction of broadsubstrate cytochrome P450s by natural and synthetic inducers in Helicoverpa zea. J. Chem. Ecol. 35: 183-189. Werck-Reichhart, D. & Feyereisen, R. (2000). Cytochromes P450: A success story. Genome Biology. 1: 3003.1-3003.9. Whalon, M. E., Mota-Sanchez, D., Hollingworth, R. M. & Duynslager, L. (2012). http://www.pesticideresistance.org/ Wharton, R. A. (1989). Classical biological control of fruit-infesting tephritidae., pp. 303-313. In A. S. Robinson and G. Harper [eds.], World Crop Pests. Vol. 3B. Fruit flies. Their Biology, Natural Enemies and Control. Elsevier, Amsterdam, The Netherlands. Wilson, T. G. & Cryan, J. R. (1997). Lufenuron, a chitin-synthesis inhibitor, interrupts development of Drosophila melanogaster. J. Exp. Zool. 278: 37-44. Whyard, S., Downe, A. E. R. & Walker, V. K. (1995). Characterization of a novel esterase conferring insecticide resistance in the mosquito Culex tarsalis. Arch. Insect Biochem. Physiol. 29: 329-42. 211 Bibliografía ______________________________________________________________________________________________________________________________________ Willoughby, L., Chung, H., Lumb, C., Robin, C., Batterham, P., & Daborn, P. J. (2006). A comparison of Drosophila melanogaster detoxification gene induction responses for six insecticides, caffeine and phenobarbital. Insect Biochem. Mol. Biol. 36: 934-942. Wondji, C. S., Dabiré, R. K., Tukur, Z., Irving, H., Djouaka, R., et al. (2011) Identification and distribution of a GABA receptor mutation conferring dieldrin resistance in the malaria vector Anopheles funestus in Africa. Insect Biochem. Mol. Biol. 41: 484-491. Wong, T. T. Y., Ramadan, M. M., McInnis, D. O., Mochizuki, N., Nishimoto, J. I. & Herre, J. C. (1991). Augmentative releases of Diachasmimorpha tryoni (Hymenoptera: Braconidae) to suppress a Mediterranean fruit fly (Diptera: Tephritidae) population in Kula, Maui, Hawaii. Biol. Control. 1: 2-7. Wool, D., Noiman, S., Manheim, O. & Cohen, E. (1982). Malathion resistance in Tribolium strains and their hybrids: inheritance patterns and possible enzymatic mechanisms (Coleoptera: Tenebrionidae). Biochem. Genet. 20: 621-636. Wu, D., Scharf, M. E., Neal, J. J., Suiter, D. R. & Bennett, G. W. (1998). Mechanisms of fenvalerate resistance in the German cockroach, Blattella germanica (L.). Pestic. Biochem. Physiol. 61: 53-62. Yang, J., McCart, C., Woods, D. J., Terhzaz, S., Greenwood, K. G., et al. (2007). A Drosophila systems approach to xenobiotic metabolism. Physiol. Genomics. 30: 223-231. Yang, Y., Yue, L., Chen, S. & Wu, Y. (2008). Functional expression of Helicoverpa armigera CYP9A12 and CYP9A14 in Saccharomyces cerevisiae. Pestic. Biochem. Physiol. 92: 101105. Zagrobelny, M., Scheibye-Alsing, K., Jensen, N. B., Moller, B. L., Gorodkin, J., & Bak, S. (2009). 454 pyrosequencing based transcriptome analysis of Zygaena filipendulae with focus on genes involved in biosynthesis of cyanogenic glucosides. BMC Genomics. 10: 574. Zeng, R. S., Wen, Z., Niu, G., Schuler, M. A. & Berenbaum, M. R. (2007). Allelochemical induction of cytochrome P450 monooxygenases and amelioration of xenobiotic toxicity in Helicoverpa zea. J. Chem. Ecol. 33: 449-461. 212 Bibliografía ______________________________________________________________________________________________________________________________________ Zhou, X., Ma, C., Li, M., Sheng, C., Liu, H. & Qiu, X. (2010), CYP9A12 and CYP9A17 in the cotton bollworm, Helicoverpa armigera: sequence similarity, expression profile and xenobiotic response. Pest Manag. Sci. 66: 65-73. Zhu, Y. C., Dowdy, A. K. & Baker, J. E. (1999). Detection of single-base substitution in an esterase gene and its linkage to malathion resistance in the parasitoid Anisopteromalus calandrae (Hymenoptera: Pteromalidae). Pestic. Sci. 55: 398-404. Zhu, F., Parthasarathy, R., Bai, H., Woithe, K., Kaussmann, M., et al. (2010). A brain-specific cytochrome P450 responsible for the majority of deltamethrin resistance in the QTC279 strain of Tribolium castaneum. Proc. Natl. Acad. Sci. USA. 107: 8557-8562. 213 ANEXOS 215 ANEXO I Protocolo para la elaboración de la dieta para las larvas de C. capitata. Componentes: ® · 100 g salvado de trigo Santiveri · 300 g azúcar molido ® · 145,2 g levadura de cerveza Santiveri ® · 11,2 g Nipagin (metil p-hidroxibenzoato) Sigma ® · 11,2 g Nipasol (N-propil-p-hidroxibenzoato) Sigma · 2.400 ml Agua destilada · 10 g ácido benzoico (Ref: 12349 Sigma-Aldrich Inc.) Elaboración: 1. Se disuelven los 10 g de ácido benzoico en 1.500 ml de agua destilada calentando la mezcla en agitación a 75-80ºC en un matraz durante aproximadamente media hora, tapando el matraz con papel de aluminio. 2. En una bandeja se mezclan el salvado de trigo, el azúcar, la levadura y los productos ® ® antibacterianos Nipagin y Nipasol . 3. Antes de añadir la disolución de ácido benzoico a la bandeja, se añaden 900 ml de agua destilada al matraz y se deja enfriar unos 15 minutos. 4. Finalmente se añaden los 2400 ml de la disolución de ácido benzoico a la mezcla existente en la bandeja y se homogeniza. 5. La dieta se repartirá en varios recipientes, anotando la fecha, para su congelación a -20ºC y posterior uso. 217 ANEXO II. SECUENCIAS INTRÓNICAS DE Ccace2 y Ccae7 Secuencias nucleotídicas de los intrones de los genes Ccae7 y Ccace2. Todas las secuencias nucleotídicas se muestran en sentido 5’ 3’ indicando su tamaño en pares de bases (pb). Para el mejor entendimiento de la nomenclatura de los intrones y su localización en la estructura génica, véase la Figura IV.3.1. El símbolo […] indica que la secuencia intrónica está incompleta en ese extremo. Subrayadas las bases típicas que identifican el principio y el fin de los intrones (GT/AG). >Intrón 2 Ccace2. Secuencia parcial que incluye el extremo 3’ (133 pb) […]ATTGAAAAAAGCTTTTAATTTCCAATAAGCGAAATAGTTATACAACACATAGCTTTTGTACTTGCATACATACGTATACATACATATTA TATAACTAAATTAATGTATTCCCTTTTCGTGCGTTCACCTACAG >Intrón 3 Ccace2. Secuencia completa (147 pb) GTGAGTAGCTACAAAAAGGCTAAAGTATCTAATGTCTGCATTCAAACATACAAACGCAAATACACCCAAAATTCTATACTCGTACAAAGCA ACTCATTCATTGCTCTTACTCTCATATTTTCTTTCGTCTTCTAATATTCCCTACAG >Intrón 4 Ccace2. Secuencia parcial que incluye el extremo 5’ (2.436 pb) GTAAGTGGGTGTGCATATTTTGCAAAAAATTTAAATTGAATTCAGCTTTTTTTTGGAACAACTAGCTTTTATACGTTTTGGAGATGTGTTG TGTTCATGTGTATTTGAATACAAAAACAAGAATTGGTCAGATTTTAATGAAATGACTCACAAAGGATTTTTCTGCGCAGGAAATGAGAAAA TGGGGATTGTTACTTATTCGAGTTTGAGTCATTGGTATTCTGAAACGTTCCGTTCAGTAGACTTGTACGAAGTTTCAGCTAAATTCTGTTC CATTATTATATCTTAAATGCATTCAGAGAGCCCTCTTGGAGTTCGGGGAGTCAACTCCACAGTAAAGAAATTGAGGTGGTGCGTGAAGTCT ACTCCTCCTTTTATCTTATCAATTTGCTAGGTGGAGCACCTATTGAAGATGCGGGGTATTACAAGAGGAACTATTAAGTCTAATAACTCTT GAAAAAGTGTTAGTGTGACGGAGAAAGCTTCGTGGAATTCTTATCACCAGCATTCCTTGTGATGGCAAATATCTAAGCCTGGCATAGTATG GATTCAAAAAAAAAAAAAAAAAAAAAAAATGGATGGGAACTTTCTAACTTATGTACGTAATTCCCCCAAGAAGTTGTTAAAAATTTCACAA CTATTCTTGTCTAGTGATTTTCCGGAAGTTTTTTATTTTTTTTATTAACTCGGTTTTCTCGCTTTTTTTTTAAGGGAAAGAAAATTCAGAA TTCATTCGAAAGAGAATTCAAGGCCTAAATTTAAGTTGGATTGCTCTCATGGAATTTGGACTTTACCATTCACCACAGGAAAAATTCTATC AGTGTGATATCCCACGATCCTGGTAGACGATCGGCCAGTCGAAAAACGAAAACGTGAAATGTTCTCTCAATAAACTCATTGATCGATGTGT GTGAAGTGGCGATTGTTGAAGCCAAATTTTGCCGAGATCACGAGCTTCTATTTCAGGCATCAATTAGTCGTTCATCCTGCCGCGATAACGA TCGCCATGACGGTTACGTACTCATATCTTAACAAAATTTGGCTCGAGAAGGTCGAACCTTCTTAGAACTTCTCAAGTGTGCATAGAACAAG GCGATTTCACATGGAAAGATCGAGCGACCTCAATTCTCGCATAAACTATATTTTGTATACTTTTAATGAAAGATCTCGAAGTAAAATGCGC CAAGAAGATCCATATGTTCGTGCAAGTTGATGCGAACGGCGCCAAATCGTTTTCGTGTACATACTGAGCTACGGCCGCTATCTGAGCCGTT TGATACCAAGCGACGTTTTAGACATTGTGATTCCGTGTGATTTCTTCAGTTTAATGCAGGAGAAATAATGAGAGCTGCAGAGCTAAATTGA GGAGATACAGTCTTGATATGACAATATTGGCCTCAACAACCGCGCCCTGAGTTCTGCCTCCCCCAGTCTTGACAAAGCAACGAAGTGAATA GGTCTGCCAGTGAACGAGGAGAAGTATTTCCTGTCATCAAACCAACAATATTCAAATGTTTCTTCTTTGCTTACGTCGAAAGGAAATTCTC TTATATTGAATGACATTATGTAAAAGAACACAAATAAATCTTTGTTATCCGTGCGAAAGGAAAAATATTTGTTTGCAGTTGAGAAAATTAT AACAAATCTTGCCATCTTGACCGATAGAAAATTACTTTCGCACCGGACTCCTGCAAGTTTTTTTTTCCATAAATCTAGAGATCTGCAAATA CTTAAGCCAGTTTGGGATCACTTATAAAATCGTTTCATTTTACTTGGAGTATAAGGTTTCAGCTTTCTGTTTAAAAAGACTTAAGAAATGG AATAAGTACGTCTTCCAGTCGTTTGTTAGTCTGGATTATTCGTTAATAATTAACACAATGCAAACCAACATTGAAGAAATAATAACATTCC TCAATCAGATTTTTTTTTAACTCCCTATTTAATAGACACTTCCTCACATGGCTTCTTTTAAGGTTGATCCACCATCTCTAGTGTTCATGGT TGAGGTATCAACGGCTTGTTTTGCCCACAAATTGAAGATTTAATTACAGAAAAGTATTTTTATAAAATCCCCTAAAGATTTCTCCACAAAC GTAATGACACACATTTCGATCACTGAAACAATTTCCCCGGGACGAAGACCTGATAATCAATCTCTATTTAGTTATAAAAAGACACTAATAG CATATTTCCACCGTACGTTAAATGCATTTAAATATTGCATTTCAATGCGTGATTTCCCATGTGTTTGTTGTTGTGAATTTTAGTGAATTAA CTCATTTTCGGTATTTTAGCAGAAAATGTTATGTTAACAGTCGTTGCAGTCACTACTAATGTAATCATTTCGGAAGTTTCGGCATATTTTT TGCCAGAGTCGTGTATTTTCGGGTTTTATGAGGGGAGAAACTTTTATTTTTCATGGTGAAATTTTTTCAG[…] >Intrón 4 Ccace2. Secuencia parcial que incluye el extremo 3’ (683 pb) […]TTTTTTCAGCTGATTTCACTAAACAAATAATTTGAGAGTATTTGTTCCCAATGTGAAAATAGAAACATGCATAAAAGCATTTCGTTTCA ATAGSGAAGGCCCGTGTAGAAAAAATGTGCACTTGCAAATCATAAGATTGCATAAATAGCCAAATAAAATTAATGCGGTTCCTTGAATTTA TGATTTCAAAAGTTGTGACAAATATTGCAAACTGCTTGACATTTCATTCTCTTAACAGAGACATTAAATGCATTTAAATGCTCGGTGGAAA TAGGCTATAGGACTTAAAATTGTGCTCTTAAATGTGCTAACTACAGCAAGTTTGAAAAACATATGGTAGCTACAAAACTTCACATTAGAAA AAAAGTAAAAAATATTTTTCTTATTGCCAATGCATATTTAATACAAAACCCAAAATAATTTTTTTTAGTTGTGTTGGGGTGGAATTCACAA AGTTATTTAGTCTTTTATTTGTATCATTATTTTACAATAATATTGAGAATAATATTTAGGGTACCTGACTTTTATGAGCACCGATTTTAAA ATCAATACGATATTGATTTGCTTCAGTTTTGATCATCAGGGCCTTGGTTTAAGTATTTTTTTACACTAAATTTTTTTTTTTATACTTAAAA ACGTATTTTGGTTTGTTTTCACTTACCTTTTTCCCTTTCCATTTTCAG >Intrón 5 Ccace2. Secuencia parcial que incluye el extremo 5’ (874 pb) GTGAGTACTTCCTGTGATGAAATATAAAATTTATAATTTTTTGAACAAAGTTAAGGAATGAGACAAAATTTGTGCTCAATGAGAAGTGTCA GTAAAAATTAGTCAGTAAAAATTAGTCAGTAAAAATTAGTCAGTAAAAATTAGTCACTAAACAAATTGAGAAAGTATAACGTCTTGCTCTT TTAAAAAGGAAACTAAATACTGTTTCACACAAAAATAAAAAAATAAATAAAAATTGTGTTACATTACATAATCTTTAATAATATGGAAAAT ATACAGCGTAAAAGCGAACTACCTTTTAATCCAAAATAAGTTTCCAACATTAACGAAAAAAATGCACCTAATAAAGTCATCAACTTCTGGC TCACATTTCCGCGCCATAAGCGCGCGCTCAGCACACAAAAATAATCACAATGAAGTTGGTGTTGTTGAAAATTGTTGTCGCTACCACTAAG TGGACTCACAAACTCTCTGGCACATAAACATTTTTATATATCAAAGAAATGTGTGTGAACAAATATAGAAGCGTGCAAACGCCTTTCTAAA GTGGCAACAGTCAACCATCTGCCGCATGCACTTCATACTTCTGCATCTTTCCaCTACAGCACGACGCGACACAGTTGCCAATTGCAACTGT GCTAAGTTGCAACAAACAATACAAATTTTTCTTCTTTTTTTTttGTTGATGTCCCATTTTCCAAAATTACTTTGCCTCGGCGTTTCATTGG 219 CAACGAAATGATGTTACAAAGGGTGTGTAGCGCGTATGTATGTAGGCGACAAAGTGAGACTGGCTGTAAAGTAGTGAAGAAAAGTAGGATG CTCTGTAACGAGTTTTACTCTGACGTCAGAGTtGAACCAGAAAAATGCGGCTCAC[…] >Intrón 5 Ccace2. Secuencia parcial que incluye el extremo 3’ (304 pb) […]TTTTTTTTAAATCACTTTTTATATGAAGGAAGTCCCCAGTTTCCTATTTAATGGTTGGGCAACGAAAATTTTTCTTTTAAAACTTGCTG TAACAACAAGAACAATAGmCATTTCTTTCCCTGGATGGTTGGCACAATAACACGAATAGACGAGAAGCAATATTGCCGGCTGAAGACTCAT CATTTTCGCCGTTAGTTTTCACAACAGATTTGAACACATTTATTTATGTTGGTATTTTTTTCTATTTTAGAAATATTTTTAAGTCAATGAA TTGATTTCAACATGTTTTTCATTTTCGTTTCAG >Intrón 6 Ccace2. Secuencia parcial que incluye el extremo 5’ (960 pb) GTAAGTAAATTGCTTTAATTTATGTATTTAAGTATGTATTGCAAAAAAAGTCATAACATAAAGACAAAAGTTATTCGCGAATGGTGTATAC ATATATGTTTGTATTAAATGTTTGGTTTTCAATAAGGCAATCCTTTTTTCGGAGTTGTTTACTTTAACAGCTCTTCATTAATTTATGTCCA GTTAAGTTAGGCATTTCCAGAGTGAACAGACTTATACCGGAACAACGTTTGAAAATCGTCCAAATTTATTGCCAAAATTATTGTTCGGTTC GCGCGACGCATCGCAGTGCAACCATTTATTCATTTCAAATCACGTTTGCTCTAGTGGATAATTTGCATCGTCAGAGTTGCCATACTGTGCG CACCGAAGACATTGCTGCTGTGGAGCAGACAATAGGAGCTGGCCCAGCTCCAAAAATTTCATGGAAAATTTTACGGAAGGATAGACTCATG CATTAATTTTTCGTACCTGAGTTGGAAGATGTTGATGTGGACGACCTTTCGTTACAACAAGAGACCACTATAAGCTTACAGACAATGAATC AATCAATTTATTGAAGGAAACTTTTTATGCGCTCCTTATATTGCGTCGTGCTCCCATGGCGTGGCTTTAAAGTTCTTACGATTTATTACTA CTGCGGGGTTTTTACGGGGTTATGCGAAGTCGTTTTTCTACGCGCGGAAAGCCTACTTTTGTAATAAAGTTGTTTTTCTGGCAACAACAAT AAGTTACATACATTTTGTTTTCCTTGAAAACCACATGTATACGATATTTGTTATCACCTATGTAAtATAATATGAAAAGATTTGTCCTGAA TGACTGACACATCAATCCATAGCCTAAATATTTGCTCGTAGAAAAATTAAACTTGGAAATTGACCTGTGACCGAATTGATGGCTTGGTTTT GTTTATTATGTGAAACATATGAGTATATGAGCCaAGAGTAGTTTCTCTCT[…] >Intrón 6 Ccace2. Secuencia parcial que incluye el extremo 3’ (1.267 pb) […]AAATTATTCTACAAAAATTTTTTTTAATTATAACTTATTAGATATTTTTTGATCAACATTCAAAGAAACTATAATTAATGCCAAAAATT AAAAAAAAAATTCAAATTTCGCATATTGCTCAAGTAAATGGTAATCCATTATTTTTGCAAAAGTTGGCTTTAGTGATATGTATCCCGCGTT CTAATTGGGTTTTCCAATAAGAGATGTTATTTTGAATGAAAAATGGAACGATTCACACTTCAACAACGCAATGAAATAATTAAAATTCAAT AAAAAAATTTCATCGAAAAATTATATTGACTAACGAGGCCCAGTTACACCTTGATAGCTACATCTATAAGCGCAACTGTCTGACTAACCTT TCAAATTTTTACGTAGTTTATGGTCCAGCAGGGTCATTTGACCTTTATTTTTCGGAAATGAATGGATTGCGCTATCGATAGATGATTAATG ATTTTTTTACGAGCGAAGCAAAGTTTACTTTCAACATGATAATGCTACGTGTTTATTTGAATATTAAAATAACACATTTATTGGAAAACCC TGTACGAGTATAAGTTCGATTGGACTACGCTGGGTGGCATTAAAAAGTTGAGATTTGGTTCTAAAAAAGACAAAGGGAGAAGTGTTGGTCG CATATCTAAGGAGGTGGGGAGGCAAATGGTCGAAAATTCAATAAAATGATGAAAATCGTCGAATCATTGCACAAAAAGCTTGATCTGTGGA TGCCACATAAATTAACGAAAATATTCTGATGTACCGAATCATTTCGGAAGCAAATTTTGGACGGAAGTGCAGCAAGAAGCATTGCAAGAGC TACCAATGCATCCAGAAAAAGTAACAGGTTGGTGCGGACTGATGGCATCTTTGGACCGTACTTCTTCAAAGACGACGCGAATCGTAACGTA ACTGTGAATGGTGAGTGCCACCTTGAGATGATATCATACTTTTTCGATTGGCCCCTAGATCGTGTGACGCAACGTATTTAGACTATTTTTG TGGGGCTCTGTTAAGTCTAATCTTTCTAATCTCCAACATAATGTTTTTTACTTGAATTAAATTAAAATTAGTAGTACAAGATTTACTAACT TGATACAAGTTTTTTTTGCAGGGCTTAAATTCTCCCAGTACATTAAAATTTCTTAAGATTTTGGTCAACCTTTTACGTTATAACATTATTT ATAACATATGTATATATTATAATATAAACTATCGGACCTTCATCTTAAGCCATAATTAATAAAAATAATATGATTTATTATTTCAG >Intrón 7 Ccace2. Secuencia parcial que incluye el extremo 5’ (453 pb) GTAAGTATTGGTAATAAAAAAAACAATAACAATGATAAACTAAAAAAAGTACTCTGCCACGCATCAGCGAGTTTCAAAATATTTTTAAATT AAAAATATGATGATTTTTTTAATTTTTATAAACCCACGACTTGTTACGACTTGCAAAATACAAAAGTCCGTGAACATAAAGAAGAAATACA ATATCGCAAGTCTAAGCAAAGAAGGCTCGAACAATAACAAATTTAGTTAAAAGGGTAATCGCTGAATCTAAGGAGTTCAATGGCTCATTTT GAAATATGGCTCAGTTGTATAGTGTCCACACCATCAATGGTTTAGGGCTAAATTGGAATCTATGCAGAAACAATTCCTACTACTCGCGTAA GAAATTTAAACTGGGATACGAATGAGAGTCTCCGTCCTCACAAAAGTTTTTTGGCGCTCATTAAAATGCCAACAATACAATACTAAATT[…] _________ >Intrón 1 Ccae7. Secuencia parcial que incluye el extremo 3’ (107 pb) […]AAATAAACGCTTCATAGCAACAATTGTTAGGCAATGGAAAAAAATTTATTTTTTTTAAGGACAAGCACAATCTAAATATCTAACTCATA CCTTTCTTCCGTTCTTAG >Intrón 2 Ccae7. Secuencia completa (347 pb) GTGAGTATATATATAAATTTATGTACCAAAACAGTGAAACGTTGCATCGTTAAACGAAGGAGAAAAATCTCAAATTTCTACTATTAGTTCT CATAAAAAGAATAAAGAATATATCGGCTTATGAATTTTTAAAGAATAATTGTTTAGATTATAAAAATTACCTATAGATCCTGTTCTAAAAT TTCGCTTTGCAGTTTATAGTATAACATACCTTTGTATGTGTTCATTACTTATTTCTTAATATAATAATACTTTGCTCAATCAGTCCGAAAC TTGCCCGCAGCCTTGTTACTTTATATCTTTTTCTTTTACTGTTTGATATTCAAAAATAACTTAAATCCTCACAG >Intrón 3 Ccae7. Secuencia completa (82 pb) GTATGTGTGGGATGATAGCATATTTTACAGGCAATAATATTTCGTACGAGTATATTATTATTTTACATACAAATATAAACAG >Intrón 4 Ccae7. Secuencia completa (71 pb) GTGAGAACTACTGGCATTGCCACATACATTTTTGAATCATTATAATAAATTTATAATTTTCCCTGCCTTAG >Intrón 5 Ccae7. Secuencia parcial que incluye el extremo 5’ (192 pb) GTGAGTATCGCAAAAATAATTATAAAAATGAAAATTAATTCATTGTCACGATTTTCTGGAACTTAATTAAATAAATAATAATAATTGAAAA TCATATTTTTATATGTATTTGCGTATGAATAATATCAAATATCACGCTTCTAGATGAGCTTTTGGCGGCTTCTTTACCAAAAACCTCTTAA AGGGGGATTT[…] 220 ÍNDICE DE TABLAS Pág. Tabla 1.1. Principales especies de artrópodos ordenadas según el número de casos de resistencia. 13 Tabla 2.2.1. Lugares de muestreo de C. capitata. 42 Tabla 2.2.2. Listado de insecticidas empleados en los bioensayos de susceptibilidad. 46 Tabla 2.3.1. Análisis de la susceptibilidad a malatión y espinosad en poblaciones de campo de C. capitata. 51 Tabla 2.3.2. Selección de resistencia a malatión en líneas de laboratorio de C. capitata: Establecimiento de W-4Km. 53 Tabla 2.3.3. Selección de resistencia a malatión en líneas de laboratorio de C. capitata: Establecimiento de la línea W-10Km. 54 Tabla 2.3.4. Susceptibilidad de C. capitata frente a diferentes insecticidas. 56 Tabla 2.3.5. Selección de resistencia a espinosad en laboratorio: Establecimiento de la línea Xàbia-W-100s. 58 Tabla 3.2.1. Cebadores empleados en el estudio de diversidad de genes CYP. 73 Tabla 3.3.1. Combinaciones de cebadores degenerados para P450s. 80 Tabla 3.3.2. Diversidad de genes CYP en C. capitata. 83 Tabla 3.3.3. Genes seleccionados para el estudio de expresión. 90 Tabla 4.2.1. Cebadores empleados en la amplificación mediante GenomeWalkerTM de los intrones de Ccace2. 110 Tabla 4.2.2. Cebadores empleados en la amplificación mediante GenomeWalkerTM de los intrones de Ccae7. 111 Tabla 4.2.3. Cebadores empleados en la amplificación de los exones de Ccace2 y Ccae7. 114 Tabla 4.2.4. Poblaciones extranjeras de C. capitata utilizadas en los estudios moleculares de la resistencia a insecticidas. 117 Tabla 4.2.5. Poblaciones españolas de C. capitata analizadas en los estudios moleculares de resistencia a insecticidas. 118 Tabla 4.3.1. Secuencias nucleotídicas de los intrones de Ccae7 y Ccace2. R Tabla 4.3.2. Presencia del alelo Ccace2 en poblaciones españolas de C. capitata. 121 127 Tabla 5.2.1. Cebadores empleados en la determinación del número de copias de Ccace2 y en la estimación de su expresión. 145 Tabla 5.2.2. Eficiencia de los cebadores empleados en la determinación del número de copias de Ccace2 y en la estimación de su expresión. 146 221 Pág. Tabla 5.3.1. Actividad AChE e inhibición por malaoxón en las líneas de laboratorio C y W-10Km. 157 Tabla 5.3.2. Asignación de genotipos para Ccace2 en función de la caracterización de los perfiles RFLP. 161 Tabla 5.3.3. Proporción de las bandas estimada para el sistema de doble PCR-RFLP. 166 Tabla 5.3.4. Frecuencias genotípicas de Ccace2 en poblaciones españolas de C. capitata. 167 222 ÍNDICE DE FIGURAS Pág. Figura 1.1. Detalle del ovipositor de las hembras y de las quetas de los machos de C. capitata. 4 Figura 1.2. Distribución de la mosca mediterránea de la fruta. 5 Figura 1.3. Principales países productores de cítricos. 6 Figura 1.4. Principales exportadores de cítricos a nivel mundial. 7 Figura 1.5. Evolución de la resistencia a pesticidas. 12 Figura 1.6. Distribución de las especies de insectos en los que se han identificado casos de resistencia. 13 Figura 1.7. Nomenclatura de un gen CYP. 18 Figura 2.2.1. Jaula de cría para adultos. 36 Figura 2.2.2. Cría de larvas. 37 Figura 2.2.3. Líneas de laboratorio. 39 Figura 2.2.4. Caja de bioensayo. 43 Figura 2.2.5. Insecticidas. 44 Figura 3.2.1. Estructura de un cDNA que codifica una P450. 74 Figura 3.2.2. Clonación y comprobación del inserto. 75 Figura 3.2.3. Estructura química del fenobarbital. 77 Figura 3.3.1. Amplificación por PCR de genes CYP. 79 Figura 3.3.2. Análisis RFLP de los clones. 81 Figura 3.3.3. Alineamiento de las secuencias aminoacídicas obtenidas a partir de los genes CYP de C. capitata. 81 Figura 3.3.4. Distribución de los genes CYP en las cinco familias estudiadas. 82 Figura 3.3.5. Disposición de los genes CYP de C. capitata. 86 Figura 3.3.6. Clado CYP-4. 87 Figura 3.3.7. Clado Mitocondrial. 88 Figura 3.3.8. Clado CYP-3. 89 Figura 3.3.9. Clado CYP-2. 90 Figura 3.3.10. Validación de los cebadores para PCR cuantitativa. 91 Figura 3.3.11. Expresión de genes CYP en adultos de C. capitata. 92 Figura 3.3.12. Efecto del fenobarbital en la expresión de los genes CYP en C. capitata. 95 Figura 4.2.1. BD GenomeWalkerTM. 108 Figura 4.2.2. Estructura génica de Dmae7 y Dmace2. 109 Figura 4.2.3. Diseño del método de detección de la mutación AChE G328A en C. capitata. 115 Figura 4.2.4. Patrón PCR-RFLP esperado para Ccace2. 116 223 Pág. Figura 4.3.1. Estructura génica de Ccae7 y Ccace2. 120 Figura 4.3.2. Amplificación de Ccae7 y Ccace2 en individuos de distintas regiones geográficas. R Figura 4.3.3. Método PCR-RFLP para la detección del alelo Ccace2 . R Figura 4.3.4. Análisis por PCR-RFLP del alelo Ccace2 en poblaciones de campo de C. capitata. R Figura 4.3.5. Distribución geográfica del alelo Ccace2 en España. 122 123 125 126 Figura 4.3.6. Porcentaje de alelos Ccace2S y Ccace2R en las poblaciones españolas de C. capitata analizadas. 129 R Figura 4.3.7. Distribución geográfica del alelo Ccace2 en las poblaciones no españolas. 130 Figura 4.3.8. Frecuencia genotípica de Ccace2 en las líneas de laboratorio resistentes a malatión seleccionadas a partir de la población W. 131 Figura 5.2.1. Cajas para los cruzamientos. 144 Figura 5.2.2. Representación esquemática del método PCR-RFLP para la detección de la duplicación génica de Ccace2 en C. capitata. 150 Figura 5.3.1. Resultado esperado y observado en el cruzamiento de las líneas C y W-10Km. 154 Figura 5.3.2. Cuantificación del número de copias del gen Ccace2 en dos líneas de laboratorio de C. capitata. 155 Figura 5.3.3. Cuantificación de la expresión de Ccace2 en las líneas de laboratorio C y W-10Km. 156 Figura 5.3.4. Perfiles RFLP asociados a los individuos heterocigotos para AChE G328A. 158 Figura 5.3.5. Modelo teórico de PCR-RFLP para los individuos portadores del alelo de resistencia Ccace2R. 159 Figura 5.3.6. Caracterización matemática de los perfiles de restricción heterocigotos para la mutación AChE G328A con Bst NI. 160 Figura 5.3.7. Cuantificación de los perfiles RFLP heterocigotos para AChE G328A. 160 Figura 5.3.8. Correspondencia entre los patrones RFLP y los genotipos para la mutación AChE G328A y para la mutación estructural. 162 Figura 5.3.9. Cuantificación del número de copias del gen Ccace2 en diferentes genotipos. 163 Figura 5.3.10. Patrones de secuenciación para la mutación AChE G328A. 164 Figura 5.3.11. Sistema de doble PCR-RFLP para la detección de la mutación AChE G328A y de la duplicación Ccace2RS. 166 Figura 5.3.12. Cuantificación mediante qPCR del número de copias de Ccace2 en individuos de campo. 168 Figura 5.3.13. Supervivencia de los adultos de las líneas C y W-10Km después de la emergencia. 170 224