Teórica 17 Regulación génica
Anuncio
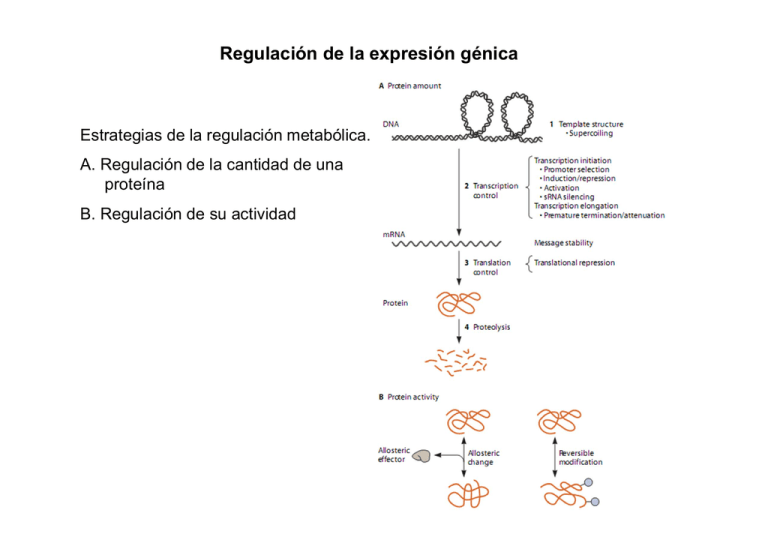
Regulación de la expresión génica Estrategias de la regulación metabólica. A. Regulación de la cantidad de una proteína B. Regulación de su actividad Procesos regulatorios que afectan los niveles de una proteína en las bacterias Genes de expresión constitutiva: presentan una expresión relativamente constante. Genes guardianes (house-keeping genes). Ej. enzimas del metabolismo central. Genes de expresión regulable : su expresión varía en respuesta a diferentes estímulos Inducción: aumento del nivel de una proteína en respuesta a un estímulo Represión: disminución del nivel de una proteína en respuesta a un estímulo Las bacterias y arqueas poseen genes organizados en operones. Presentan regulación coordinada. ARNm policistrónicos Secuencias consenso de los promotores bacterianos (E. coli) Promotores arqueanos . Inicio de la transcripción Interacción entre la ARN polimerasa bacteriana y los elementos del promotor La actividad de los promotores está regulada por proteínas activadoras y represoras Proteínas de unión al ADN Funcionan como dímeros o multímeros Interaccionan con el promotor y la ARN polimerasa Activadores : aumentan la velocidad de inicio de la transcprición Represores : Inhiben el inicio de la transcripción Sujetos a control alostérico o modificación química (fosforilación) Activadores - Los activadores (A) generalmente se unen upstream de caja -35 - Interaccionan con ARN pol o generan cambio conformacional en el Promotor (P) haciéndolo accesible a la ARN pol. (a) Activador (A) unido a distancia del promotor (P), genera torción en el ADN, estabiliza interacción de ARN pol. con P. (b) La distancia entre las cajas -35 y -10 es mayor a la usual (19 pb en lugar de 17pb). El A reposiciona las cajas haciendo más eficiente la transcripción. Represores (a- b) Inhiben la transcripción uniéndose en posiciones que interfieren con la ARN pol. (c ) Bloquean actividad de un activador : Anti-activación Regulación de la transcripción por la estructura del promotor y la actividad de factores σ Las bacterias presentan varios factores σ que reconocen promotores específicos. El σ70 controla la expresión de los genes durante el crecimiento exponencial. Otros funcionan en situaciones particulares. La regulación por factores σ es un tipo de sistema de regulación global. Las proteínas anti-σ se unen a los factores σ e inhiben su actividad Circuitos de control del inicio de la transcripción Control positivo: mediado por activador Control negativo: mediado por represor Se puede lograr inducción o represión por control + o dependiendo de la interacción de la molécula señal con la proteína reguladora. Represión catabólica de Carbono mediada por la proteína activadora CAP (CRP) en E. coli Crecimiento diauxico de E. coli en presencia de glucosa y lactosa. Es el resultado de un mecanismo regulatorio llamado represión catabólica (RCC, represión catabólica de C). Regulador: proteína CRP o CAP (cAMP receptor protein; catabolite gene-activator protein) A. Crecimiento usando glucosa B. Fase lag C. Crecimiento usando lactosa Mecanismos moleculares para la inducción y represión del operón lac en E. coli. RCC en E. coli. El sistema PTS cumple un rol importante en la regulación genica. En bajos niveles de glu, EIIA-P se une y activa la adenilato ciclasa (AC), aumenta cAMP y favorece la formación de cAMP-CRP. Este complejo se une y activa los promotores de genes catabólicos. En presencia de glu, EIIA inhibe el transporte de fuentes de C alternativas mediante exclusión del inductor. Las diferencias en las secuencias consenso del promotor lac hace que la ARN polimerasa se pegue débilmente en ausencia de CRP Niveles de organización de los genes en las bacterias Regulón: conjunto de genes / operones controlados por un regulador en común. Modulón o red de regulación global: conjunto de operones /regulones con su regulación individual a su vez controlados por un regulador en común (regulador maestro, master regulator) Acoplamiento entre la expresión génica y la morfogénesis. Síntesis del flagelo en Enterobacterias. Flagelo formado por: cuerpo basal, gancho y filamento (flagelina). Regulón flagelar: más de 50 genes organizados en ~17 operones regulados coordinadamente. Se expresan en cascada. Un sigma específico (σ28) regula la síntesis de la flagelina. El σ28 es inhibido por un factor antisigma (FlgM) hasta que se forman el gancho y cuerpo basal del flagelo. Luego FlgM es secretado facilitando la unión del σ28 al promotor de la flagelina (activacion sintesis flagelina)). Genes regulados por recombinación génica. Variación de fase. Síntesis de flagelinas en Enterobacterias Salmonella typhimurium (patógeno humano) posee un mecanismo para evadir el sistema inmune cambiando el tipo de proteína flagelar (flagelina tipo FljB y FliC ), 1 vez cada ~ 1000 generaciones mediante un proceso llamado variación de fase. Control de la traducción: Regulación por ARN Control de la estabilidad y la traducción de los ARNm mediante: - Proteínas con afinidad por ARN - ARNs pequeños no codificantes (ncRNA) - Estructuras en 5’-UTR (riboswitch) El 5’-UTR sirve como elemento de control a. Factores que afectan la estabilidad de los ARNm b. Inhibición de la traducción y posterior degradación del ARNm Regulación por formación de estructuras alternativas en 5’-URT a. Estructuras alternativas que dependen de la temperatura. Adaptación al shock por frío o calor. Ej. Síntesis de RpoH, σ alternativo inducido por shock de calor. TIR: región de inicio de la traducción b. Riboswitch. Estructuras alternativas inducidas por unión con ligandos pequeños (AA, nt, vitaminas). Pueden activar o inhibir la traducción Control antisense por ARNs no codificantes pequeños (ncARN) Control antisense por ARNs no codificantes pequeños (ncARN), codificados en cis o en trans Cis: codificados en el mismo locus que el target. Ej. Reg. Replicación de plásmidos; sist. Toxinaantitoxina. Trans: codificados en distinto locus que el target. Presentan complementaridad parcial con el target. Se inducen por cambios ambientales.









