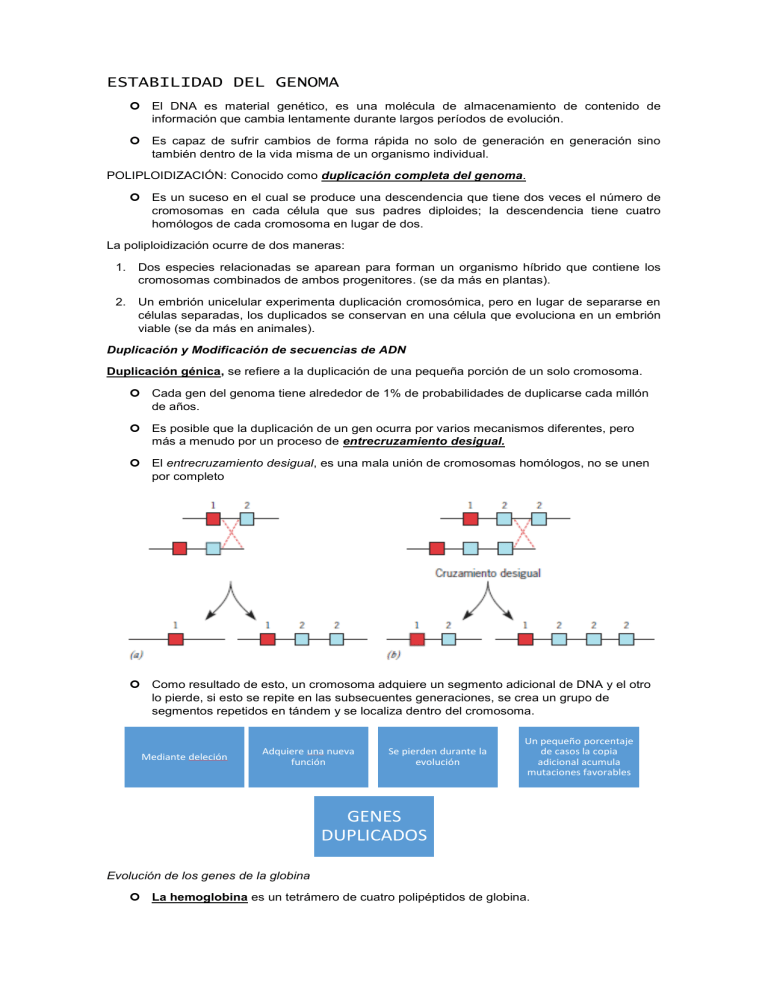
ESTABILIDAD DEL GENOMA El DNA es material genético, es una molécula de almacenamiento de contenido de información que cambia lentamente durante largos períodos de evolución. Es capaz de sufrir cambios de forma rápida no solo de generación en generación sino también dentro de la vida misma de un organismo individual. POLIPLOIDIZACIÓN: Conocido como duplicación completa del genoma. Es un suceso en el cual se produce una descendencia que tiene dos veces el número de cromosomas en cada célula que sus padres diploides; la descendencia tiene cuatro homólogos de cada cromosoma en lugar de dos. La poliploidización ocurre de dos maneras: 1. Dos especies relacionadas se aparean para forman un organismo híbrido que contiene los cromosomas combinados de ambos progenitores. (se da más en plantas). 2. Un embrión unicelular experimenta duplicación cromosómica, pero en lugar de separarse en células separadas, los duplicados se conservan en una célula que evoluciona en un embrión viable (se da más en animales). Duplicación y Modificación de secuencias de ADN Duplicación génica, se refiere a la duplicación de una pequeña porción de un solo cromosoma. Cada gen del genoma tiene alrededor de 1% de probabilidades de duplicarse cada millón de años. Es posible que la duplicación de un gen ocurra por varios mecanismos diferentes, pero más a menudo por un proceso de entrecruzamiento desigual. El entrecruzamiento desigual, es una mala unión de cromosomas homólogos, no se unen por completo Como resultado de esto, un cromosoma adquiere un segmento adicional de DNA y el otro lo pierde, si esto se repite en las subsecuentes generaciones, se crea un grupo de segmentos repetidos en tándem y se localiza dentro del cromosoma. Mediante deleción Adquiere una nueva función Se pierden durante la evolución Un pequeño porcentaje de casos la copia adicional acumula mutaciones favorables GENES DUPLICADOS Evolución de los genes de la globina La hemoglobina es un tetrámero de cuatro polipéptidos de globina. Cada gen de globina está formado por tres exones y dos intrones. Al estudiar genes que codifican a ciertos polipéptidos semejantes a la globina se revela la presencia de cuatro exones y tres intrones, y se ha propuesto que representan la forma ancestral del gen de la globina Al analizar las secuencias de DNA de los grupos del gen de la globina, se encontraron “genes” cuyas secuencias son homólogos respeto de los genes funcionales de la globina, pero han acumulado mutaciones profundas que los han hecho no funcionales, los genes de este tipo se conocen como seudogenes. Genes Saltarines… La naturaleza dinámica del gen • Transposiciones: Ciertos elementos genéticos experimentan movimiento de un lugar en un cromosoma a un sitio por entero diferente • Elementos Transponibles: Entidades genéticas móviles • Transposones: Elementos transferibles que se mueven de un lugar al otro en el genoma, descubiertos en 1960 en bacterias, la mayoría codifica una proteína, transposasa. Transposones 3% Retrotransposones 40% Transposones: Mecanismo de corte y empalme Mariner, reinos de plantas y animales Retrotransposones Mecanismo de copia y pegado Interviene un RNA intermediario Se transcribe el DNA, se crea un RNA, lo retrotranscribe, por la transcriptasa inversa Se elabora una copia de DNA bicatenario y se integra al sitio blanco Elementos genéticos Móviles en la evolución Secuencias moderadamente repetidas Elementos Diseminados SINE LINE L1 completa 6000 pares de bases GH, 500,000 Familias L1 Familias Alu En mas de un millon de sitios en el GH. 2% seres hmanos poseen un nuevo inserto genómio L1 Cuál es su función???? Considerados de manera primaria como “basura” Un elemento transponible es un tipo de parásito genético que puede invadir el genoma de un huésped diseminarse dentro de él y transmitirse a su descendencia. Sin embargo pueden hacer contribuciones positivas, recordando que la evolución es un proceso oportunista. Entonces como participaron en la evolución…. Dos segmentos no unidos del genoma pueden conectarse y crear un nuevo segmento compuesto En algunos casos parecen haber dado lugar a ciertos genes, la enzima telomerasa, con función clave en la replicación del DNA pudo originarse en una transcriptasa inversa codificada por un retrotransposón antiguo. SECUENCIACION DE GENOMAS La primera secuencia completa de un organismo procariota se notificó en 1996 y el siguiente año se presento la de un eucariota, S. Cerevisiae. Genoma Humano, alrededor de 3200 Millones de pares de bases. Primer informe de la secuencia nucleotídica de todo el GH 2001 Fue una aproximación, cada segmento se secuenció 4 veces y se excluyeron algunas regiones Concluyeron que el GH tal vez tenia unos 30,000 genes codificantes No se determino su secuencia completa, se supuso que contenia unos 50,000 y quizá hasta 150,000 genes La versión terminada de la secuencia del genoma humano se informó en 2004 luego de: Cada sitio se secuenció de siete a diez veces para asegurar un alto grado de exactitud La secuencia contenía un número mínimo de huecos, los que persistían contenían regiones de los cromosomas Actualmente las estimaciones colocan la cifra alrededor de 20,000 genes. Esto significa que los seres humanos tiene casi el mismo número de genes que un gusano microscópico, una planta de mostaza, así como la mayoría de vertebrados Como se explica entonces la complejidad de los seres vivos?? Un solo gen puede codificar varias proteínas relacionadas como resultado de un proceso llamado corte y empalme alternativo. Más del 90% de genes humanos. Tienen más exones. Puede haber mucho más pero el punto general: La diferencia aparente en la complejidad entre distintos grupos de organismo multicelulares depende menos dela información genética en el genoma de un organismo que de la forma en que se aplica esta. “si se conserva debe ser importante”… La mayor parte del genoma consiste en DNA que reside entre los genes La mayor parte de las secuencias intergénicas e intrónicas tienden a cambiar con rapidez a medida que evoluciona el organismo Cada uno de los cerca de 20,000 genes humanos codificadores de proteína consiste sobre todo en porciones no codificadoras (intones) Los segmentos que codifican proteínas o regulas la expresión génica tienden a conservarse La porción del genoma que codifica proteínas representa 1.5 % del DNA total Se acepta el principio de la evolución molecular si se conserva debe ser importante, las secuencias no codificantes ni reguladoras consideradas inútiles tiene una función no definida aún Codifican RNA pequeños descubiertos hace poco y que aún no se ha determinado su función. El proyecto ENCODE, busca identificar todos los elementos funcionales del genoma. No se cuenta con el conocimiento necesario para reconocer muchos de estos pero con los estudios quedo un punto claro: Un porcentaje significativo de las secuencias de DNA funcionales está en evolución constante, no se mantiene. La base genética del ser humano En 2005 se publicó un borrador del genoma del chimpancé. En general difiere del humano en casi 4%. Esto significa decena de millones de diferencias. Se supone que las diferencias entre factores de trasncripción del chimpancé y el ser humano son la causa de las diferencias de expresión de las proteínas cerebrales. Uno de los genes, llamado HAR1, se extiende sólo 118 pares de bases pero contiene 18 sustituciones de bases que los distinguen de la región correspondiente en el genoma del chimpancé. El gen que ha causado mayor interés es el que codifica un factor de transcripción llamado FOXP2 (Gen del habla), las mutaciones en este gen, hacen que la persona sufra graves trastornos del habla y el lenguaje, son incapaces de movimientos musculares finos de los labios y la lengua. Variación genética dentro de la población humana VARIACIÓN DE SECUENCIA Los polimorfismos genéticos son sitios en el genoma que varían de un individuo a otro. El tipo más común de variabilidad genética en seres humanos se presenta en nucleótidos individuales, llamados polimorfismos de nucleótidos individuales. VARIACION ESTRUCTURAL: Se refiere a cambios de algunos segmentos de genoma. Casi siempre afectan segmentos de DNA que varía de miles a millones de pares de bases, se denominan variantes estructurales. MUTACIONES Cambios al azar o provocados por agentes mutagénicos en el material genético celular no dirigidos y de efectos imprevistos. Tiene menor probabilidad en organismos superiores y mayor en los inferiores como las bacterias y virus. Tipo de Mutaciones por su Efecto NOCIVAS: La mayoría pertenecen a este grupo, pues un cambio al azar de cualquier información siempre lleva a una información “sin Sentido” o defectuosa para el individuo NEUTRAS: No tienen efecto alguno BENEFICIOSAS: Para el individuo y para la especie si se trasmite a su descendencia, aparecen con menor frecuencia Tipos de Mutaciones por su Origen ESPONTÁNEAS • Las menos numerosas, se producen por un error en la replicación del DNA, • Por errores en el movimiento y comportamiento de los cromosomas durante la división celular INDUCIDAS • La mayoría de las mutaciones existentes • Provocadas por agentes mutagénicos Tipo de Mutaciones según localización Génicas o puntuales • • • Sustitución de bases Deleción de bases Adición de bases Cromosómicas, estructurales • • • • Deleción cromosómica Duplicación cromosómica Inversión cromosómica Translocación cromosómica Genómicas, o cromosómicas numéricas • • EUPLOIDIA • Triploidia • Tetraploidia • Poliploidia ANEUPLOIDIA • Nulisomica • Monosomica • Trisómico MUTACIONES GENICAS: Son aquellas que producen alteraciones en la secuencia de nucleótidos de un gen. SUSTITUCIONES DE PARES DE BASES, que pueden ser: 1. Transiciones, que es el cambio en un nucleótido de una base purica por otra purica, o de una pirimidina por otra pirimidina. 2. Transversiones, que es el cambio en un nucleótido de una pase purica por una pirimidina o viceversa. PÉRDIDA O INSERCIÓN, de bases resultando en: 1. Adiciones génicas: Es la inserción de nucleótidos en la secuencia del gen. 2. Deleciones génicas: Es la pérdida de nucleótidos MUTACIONES CROMOSÓMICAS ESTRUCTURALES: Son los cambios en la estructura interna de los cromosomas, se pueden agrupar en dos tipos: Las que suponen pérdida o duplicación de segmetos: 1. Deleción cromosómica: es la pérdida de un segmento del cromosoma. 2. Duplicación cromosómica: es la repetición en un segmento del cromosoma Las que suponen variaciones en la distribución de los segmentos de los cromosomas 1. Inversiones: un segmento del cromosoma se encuentra colocado en una posición invertida 2. Translocaciones: un segmento del cromosoma se encuentra situado en otro cromosoma AGENTES MUTAGÉNICOS AGENTES QUÍMICOS • Acido nitroso, peroxido de hidrógeno, gas mostaza, dioxido de carbono en altas concentraciones. • La mayoría provoca emparejamientos erróneos de las bases, y otros intercalándose entre las bases. RADIACIONES • Rayos X, beta, gama, entre otros. De efectos ionizantes con cabios de bases o los de mayor energía que provocan ruptura de ADN o de los cromosomas. Origen de Algunas Mutaciones Cromosómicas Estructurales Todos los cambios estructurales de los cromosomas se pueden explicar por rotura y reunión de sus fragmentos, se consideran tres casos. 1. Roturas que afectan a un cromosoma: Si la rotura ocurre dentro del brazo del cromosoma, los fragmentos pueden reunirse y dar lugar a una deleción o una inversión más un fragmento sin centrómero 2. Roturas que afectan a cromosoma distinto: Si la rotura ocurre en dos cromosomas homólogos simultáneamente. Después de la rotura la reunión de los fragmentos puede producir una duplicación más una deleción. 3. Roturas que afectan a cromosoma distinto: Si la rotura ocurre en dos cromosomas no homólogos simultáneamente. Después de la rotura se produce un intercambio de fragmentos dando lugar a una translocación entre cromosomas no homólogos: translocación recíproca Efecto Fenotípico de las Mutaciones Cromosómicas Estructurales Las deleciones y duplicaciones producen un cambio en la cantidad de genes y por tanto tienen efectos fenotípicos, por lo general deletéreos. Mutaciones Cromosómicas Numéricas: Son alteraciones en el número de cromosomas de la especie y pueden ser; EUPLOIDIAS Origen Si durante la meiosis se produce en algunas células la no disyunción de todos los cromosomas homólogos se originarán dos gametos con 2n cromosomas y dos gametos sin cromosomas (0). La unión de estos gametos entre sí o con gametos n, puede producir zigotos haploides, triploides o tetraploides (n+0, n+2n, 2n+2n) Las anomalías en los euploides son menores que en los aneuploides, en los que los efectos fenotípicos son mayores al no mantenerse equilibradas las dosis relativas de genes. ANEUPLOIDIAS Aneuploidias más importantes en la especie humana AGENTES MUTÁGENOS: es todo factor capaz de aumentar la frecuencia de mutación natural. actuarán como agentes mutágenos todos aquellos agentes capaces de alterar el material genético y en particular, aquellos que alteren la secuencia del ADN. Los principales agentes mutágenos son: Agentes Físicos 1. Las radiaciones electromagnéticas como los rayos X y los rayos gamma. 2. Las radiaciones corpusculares como los rayos α, los rayos ß y los flujos de protones o neutrones que generan los reactores nucleares u otras fuentes de radiactividad natural o artificial. Agentes Químicos 1. 2. 3. 4. Los análogos de las bases nitrogenadas. El ácido nitroso (HNO2), porque desamina ciertas bases nitrogenadas. Los alcaloides como la cafeína, la nicotina, etc. El gas mostaza, el agua oxigenada (H2O2), el ciclamato, etc. NUCLEO CELULAR • Estructura común para toda la importancia en almacenamiento y utilización de la información genética • Definición: Masa amorfa viscosa encerrada en una envoltura nuclear compleja, que forma una transición entre núcleo y citoplasma. Dentro del núcleo la célula tiene: • Cromosomas que se observan como fibras de nucleoproteínas, denominada cromatina. • Uno o más nucléolos: estructuras electrodensas forma irregular que funcionan en la síntesis de RNA ribosómico y en el ensamble de ribosomas • Nucleoplasma: sustancia liquida en la que los solutos del núcleo se disuelven • Matriz Nuclear: red fibrilar que contiene proteínas Envoltura Nuclear: Permite la separación del material genético del citoplasma, y esto puede ser la distinción más importante entre células eucariotas y procariotas • Se considera como punto de la evolución biológica a la aparición de la envoltura nuclear La envoltura nuclear consta de: • Dos membranas celulares organizadas en paralelo una con la otra y separadas por un espacio de 10 a 50 nanómetros. • Sirve como barrera que protege los iones, solutos y macromoléculas que pasan del núcleo al citoplasma… • Membrana externa esta tapizada por Ribosomas, y se continúa con el retículo endoplásmico rugoso La superficie interna se une mediante proteínas de tipo integral a una delgada red de filamentos llamada LAMINA NUCLEAR Brinda apoyo mecánico a la envoltura nuclear y sirve como sitio de unión para las fibras de cromatina de la periferia nuclear Participa en la duplicación y transcripción del DNA Al igual que en el citoplasma la integridad de los filamentos intermedios se regula por fosforilación y desfosforilación. • Las mutaciones en uno de los genes de la lámina son causa de diversas enfermedades en los humanos ▫ Forma rara de distrofia muscular llamada EDMD2 ▫ Síndrome de progeria de Hutchinson-Gilford La estructura del complejo del poro nuclear y su función: Son compuertas para cruzar la barrera que forma la envoltura nuclear. Membrana Plasmática: Previene el paso de macromoléculas entre citoplasma y espacio extracelular Poro nuclear: Punto de actividad para el movimiento de RNA y proteínas en ambas direcciones Los poros nucleares contienen un aparato complejo en forma de canastilla denominado complejo de poro nuclear Complejo poro nuclear: • Similar a un tapón • Complejo supramolecular característico (15 a 30 veces la masa del ribosoma) • Simetría octagonal (repetición octuple de varias estructuras) A pesar de su complejidad y tamaño contiene alrededor de 30 proteínas denominadas nucleoporinas Nucleoporinas: • Entre estas hay un subgrupo que tiene una gran cantidad de repeticiones fenilalaninaglicina (FG)en su secuencia de aminoacidos. • Las repeticiones FG se aglomeran en una región particular conocida como dominio FG. • Las nucleoporinas con FG recubren el conducto del NPC, formando una red que impide la difusión de macromoléculas más grandes • Algunas proteínas contienen un grupo de aminoácidos cercanos a su grupo carboxilo terminal que funcionan como una señal de localización nuclear. Existe otro grupo de proteínas que funcionan como receptores de transporte Pasos principales durante la importación nuclear de una proteína nuclear clásica: Inicia como una proteína que contiene una señal de localización nuclear unida a un receptor soluble heterodimérico, conocido como importina α/β que reside en el citoplasma El receptor de transporte escolta a la proteína a la superficie externa del núcleo donde se ancla a los filamentos citoplásmicos El complejo receptor-cargamento se muevo por el poro nuclear Cuando ya paso por el poro nuclear, aparece una proteína de unión con GTP llamada RAN, puede encontrarse de dos formas: 1. Activa unida con GTP 2. Inactiva unida con GDP ▫ La función de está en la regulación del transporte nucleocitoplásmico se basa en mecanismos en los que la célula mantiene una alta concentración de proteína Ran-GTP en el núcleo y una concentración muy baja de la misma en el citoplasma. ▫ Las proteínas exportadas del núcleo contienen secuencias aminoacídicas denominadas señales de exportación nuclear, NES, que son reconocidas por receptores de transporte que las acarrean a través de la envoltura nuclear hasta el citoplasma. Empaquetamiento del genoma: Una célula humana contiene cerca de 6.4 mil millones de pares de bases de DNA repartidos en 46 cromosomas Cada cromosoma replicado contiene una molécula continua y única de DNA, entre más largo el cromosoma más largo el DNA que contiene. Cada par de bases mide 0.34 nm de longitud, todas medirian 2 metros Tanto de guisantes como de vacas, H4 difiere solo en dos residuos de aminoácidos: Interactúan con el esqueleto de DNA que es igual en todos los organismos Casi todos los aminoácidos de una histona interactúan con otras moléculas ya sea de DNA o con otras histonas. Roger Kornberg en 1974 propuso una estructura nueva para la cromatina, postuló que el DNA y las histonas se organizan en subunidades repetidas, denominadas nucleosomas. • cada nucleosoma contiene una partícula nuclear de nucleosoma, que consiste en 146 pares de bases de DNA superenrollado envuelto por lo menos dos veces alrededor de un complejo en forma de disco de ocho moléculas de histona. El núcleo de histona de cada nucleosoma consta de dos copias de cada histona, ensambladas en un octágono (H2A, H2B, H3 Y H4) H1 se refiere como histona de unión, porque enlaza parte del DNA de unión que conecta una partícula con la siguiente. Las ocho moléculas de histona que comprenden una proteína nuclear del nucleosoma se organizan en cuatro heterodímeros: dos dímeros H2A H2B y dos dímeros H3-H4. Funciones de las histonas La cromatina compactada se conoce como heterocromatina, que difiere de la eucromatina, que retorna al estado disperso. Heterocromatina Se divide en dos clases 1. Heterocromatina constitutiva, permanece en estado compactado en todas las células durante todo el tiempo y por tanto representa el DNA que en forma permanente no realiza transcripción. 2. Heterocromatina facultativa, es una cromatina que se inactiva de manera específica durante ciertas fases de la vida de un organismo en cierto tipo de células. Telómeros, se evidencian en las puntas de cada molécula de DNA compuestas por un tramo inusual de secuencias repetidas, forma una cubierta en cada extremo del cromosoma. PROTEÍNAS 1. Como se llaman las macromoléculas que realizan todas las actividades celulares, son las herramientas y maquinas moleculares que hacen que las cosas sucedan? Proteínas 1. Describa en que forma pueden actuar las proteínas? Como enzimas, estructurales, hormonas, factores de crecimiento y activadores génicos, funciones reguladoras como receptores de membrana transportadores, entre funciones adicionales actúan como anticuerpos, toxinas, absorben o refractan la luz. 2. Como están formadas las proteínas? Polímeros formados por monómeros de aminoácidos 3. Como están formadas las proteínas? Polímeros formados por monómeros de aminoácidos 4. Cuantos son los aminoácidos usados habitualmente en la construcción de proteínas, ya sea de un virus o un humano? 20 5. Todos los aminoácidos tienen un grupo carboxilo y un grupo amino separados entre si por un solo átomo de carbono llamado? CARBONO ALFA 6. Durante el proceso de síntesis de proteínas, cada aminoácido se une con dos aminoácidos más y forman un polímero largo, continuo, no ramificado llamado Cadena Polipeptídica 7. Que tipo de enlace une a los aminoácidos que forman la cadena polipeptídica al sintetizar proteínas Enlace peptídico 8. Como se denominan los aminoácidos incorporados en la cadena polipeptidica Residuos 9. Muchas proteinas tienen otros tipos de componentes que se agregan después de la síntesis del polipétido, cuales son: Carbohidrastos glucoproteinas, grupos que contiene meta metaloproteinas y grupos orgánicos, (flavoproteinas) 10. Los aminoácidos se clasifican según una de sus estructuras cual es: Cadenas laterales 11. Cuáles son las 4 categorías en que se clasifican los aminoácidos Polares con carga Aquellos con propiedades únicas polares sin carga no polares 12. Cuáles son los aminoácidos del grupo polares con carga Acido aspártico acido glutámico 13. Cuáles son los aminoácidos polares sin carga? o Aparagina o glutamina o treonina 14. Cuales son los aminoácidos no polares? Alanina valina leucina isoleucina o o lisina y arginina serina tirosina triptófano fenilalanina metionina 15. Cuales son los aminoacidos con propiedades unicas Glicina, prolina y cisteína 16. Como se llaman las modificaciones que se dan después de la incorporación de aminoácidos no básicos a la cadena polipeptidica: Modificaciones postraduccionales 17. Cuál es la modificación postraduccional pas difundida e importante: Adición de un grupo fosfato a un residuo de serina, treonjina o tirosina 18. Por su organización las proteinas se obserban en cuatro niveles que son: Primario, secundario, terciario y cuaternario 19. Cuál es el nivel que se refiere a la secuencia de aminoácidos en una proteína: Primario 20. Como se llama el tipo de anemia grave y hereditaria que se debe a un solo cambio en la secuencia de aminoácidos, donde una vaina esta donde debería estar un ácido glutámico: Anemia depranocitica 21. A que se refiere el termino conformación: Disposición tridimensional de los átomos de una molécula 22. A que se refiere la conformación de hoja plegada beta: Varios segmentos de un polipeptido dispuestos de lado a lado 23. Cuál es la diferencia entre proteínas fibrosas y globulares: Las primeras tienen forma alargada y las otras son compactas 24. Mencione tres proteínas fibrosas: Colagenas, elastinas y queratinas 25. Cuál fue la primera proteína globular cuya estructura terciaria fue descubierta: Mioglobina 26. Los cambios predecibles dentro de una proteína que se activan por la unión de una molécula específica se describen como: Cambios en la conformación 27. Como se llama la proteína formada por dos subunidades idénticas: Homodímero 28. Como se llama la proteína formada por dos subunidades no idénticas: Heterodímero 29. Como se llama el establecimiento de una relación física entre distintas proteínas con una función específica: Complejo multiproteínico 30. Las proteínas que tienen múltiples parejas de unión como las17 se denominan: Proteínas ejes 31. Como se denomina el desdoblamiento o desorganización de una proteína: Desnaturalización 32. Que es una proteína nativa: Es el nombre que recibe al tener una conformación plegada 33. Como se llaman las familias de proteínas que evolucionaron hasta una función que es ayudar a las proteínas no plegadas o plegadas a alcanzar su conformación tridimensional: Chaperonas moleculares 34. Donde se sintetizan las cadenas polipeptídicas: Ribosomas 35. Todo el inventario de proteínas que produce un organismo se conoce como Proteoma 36. Campo creciente de la bioquímica de las proteínas: Proteomica REPLICACION DEL ADN REPLICACION DEL DNA La estructura que propusieron Watson y Crick en 1953 para el DNA incluía un mecanismo que sugería su “autoduplicación”. Las dos cadenas de la doble hélice se mantienen juntas por medio de puentes de hidrógeno entre las bases. Supusieron que la replicación ocurría por separación gradual de las cadenas de la doble hélice algo muy semejante a la separación de las dos mitades de una cremallera. REPLICACION SEMICONSERVA La replicación de este tipo se dice que es semiconservadora, puesto que cada dúplex descendiente contiene una cadena de la estructura parental. Las dos cadenas originales debían permanecer juntas (después de servir como plantillas), así como también las dos cadenas sintetizadas de nuevo. Como resultado, una de las cadenas dúplex hijas debía contener sólo el DNA parental, mientras que las otras dúplex hijas, sólo el DNA resintetizado. REPLICACIÓN DISPERSIVA Las cadenas parentales deben cortarse en fragmentos y las nuevas cadenas sintetizarse en segmentos cortos. Los fragmentos previos y los nuevos deben unirse para formar cadenas completas. Como resultado, los dúplex hijos contendrían cadenas constituidas por DNA nuevo y antiguo. Demostración experimental de que la replicación del DNA en bacterias es semiconservadora. El DNA se extrajo de la bacteria en diferentes fases del experimento, se mezcló con una solución concentrada de cloruro de cesio (CsCl) y se centrifugó hasta el equilibrio a alta velocidad en una ultracentrífuga. Replicación en bacterias Disponibilidad de mutantes que no pueden sintetizar una u otra proteína requerida para el proceso de replicación. El aislamiento de mutantes incapaces de replicar su cromosoma puede parecer paradójico: ¿cómo pueden las células con un defecto en este proceso vital cultivarse? La paradoja se ha resuelto al obtener mutantes sensibles a la temperatura (TS), en los que la deficiencia sólo es evidente a una temperatura elevada, denominada temperatura no permisiva (o restrictiva). Horquillas de replicación y replicación bidireccional La replicación comienza en un sitio específico en el cromosoma bacteriano conocido como el origen. El origen de replicación en el cromosoma de E. coli es una secuencia específica llamada oriC en la que diferentes proteínas se unen para iniciar el proceso de replicación. Tras comenzar, la replicación se aleja del origen en ambas direcciones, es decir, en forma bidireccional. Los puntos donde el par de segmentos replicados se unen con los segmentos no replicados se denominan horquillas de replicación Cada horquilla de replicación corresponde al sitio donde: 1) la doble hélice parental se separa y 2) los nucleótidos se incorporan en las cadenas complementarias resintetizadas. Desenrollamiento del dúplex y separación de las cadenas Entran en juego las topoisomerasas responsables del desenrollamiento del DNA Una enzima la girasa de DNA, una topoisomerasa II libera la tensión mecánica que se crea durante la replicación en E. coli. Las DNA Polimerasas Kornberg et al. purificaron una enzima de extractos bacterianos a la que incorporaron precursores de DNA marcados con radiactividad en un polímero ácido insoluble de DNA. La enzima se denominó DNA polimerasa Para que tuviera lugar la reacción, la enzima requería la presencia de DNA y los cuatro trifosfatos de desoxirribonucleósido (dTTP, dATP, dCTP y dGTP). El DNA recién sintetizado y marcado con radiactividad tenía la misma composición de bases que el DNA original no marcado, lo cual sugería que las cadenas de DNA original habían servido como plantillas para la reacción de polimerización. La cadena que provee el OH 3' terminal se denomina iniciador. Todas las DNA polimerasas tanto de procariotas como de eucariotas tienen estos dos requerimientos básicos: Una cadena de DNA plantilla para copiar y Una cadena iniciadora en la cual pueden agregarse los nucleótidos. Por otra parte, una cadena sencilla circular provee un DNA plantilla pero carece del iniciador. La molécula de doble cadena parcial satisface ambos requerimientos y promueve la incorporación de nucleótido. El diagrama de Watson y Crick sugería que una de las cadenas nuevamente sintetizadas se polimerizaba en la dirección 5' → 3', mientras que la otra cadena lo hacía en la dirección 3' → 5'. ¿Acaso había otras enzimas encargadas de la construcción de la cadena en la dirección 3' → 5'?, Estudios posteriores revelaron que la enzima de Kornberg, o DNA polimerasa I, era sólo una de las distintas DNA polimerasas presentes en las células bacterianas. La principal enzima responsable de la replicación del DNA es la DNA polimerasa III. Una célula bacteriana típica contiene 300 a 400 moléculas de DNA polimerasa I, pero sólo unas 10 copias de la DNA polimerasa III. Las cantidades mucho mayores de la DNA polimerasa I en la célula han minimizado la presencia de la DNA polimerasa III. En conclusión no se pueden sintetizar cadenas de DNA en dicha dirección. Ambas cadenas recién sintetizadas se ensamblan en la dirección 5' → 3'. Las moléculas de la polimerasa encargadas de la construcción de las dos cadenas de DNA se mueven en una dirección 3' a 5' a lo largo de la plantilla y ambas construyen una cadena que crece desde su extremo 5’ fosfato terminal. Por consecuencia, una de las cadenas resintetizadas crece en dirección del asa de replicación donde las cadenas de DNA parental se separan, mientras la otra cadena crece y se aleja de la horquilla. Pronto se obtuvo evidencia de que la cadena que crece y se aleja de la horquilla de replicación se sintetizaba de manera discontinua, esto es, en fragmentos Las dos cadenas hijas se sintetizan por procesos muy diferentes. La cadena que se sintetiza de modo continuo se conoce como cadena adelantada debido a que lo hace conforme avanza la horquilla de replicación. La cadena que se sintetiza de forma discontinua se llama cadena retrasada dado que el inicio de cada fragmento debe esperar a que las cadenas progenitoras se separen y expongan un fragmento Términos no apropiados, se sintetizan de forma simultánea, una de forma continua y otra discontinua, replicación semidiscontinua Reiji Okazaki descubrió que las cadenas primero se forman por pequeños segmentos, al cultivar y cosechar inmediatamente después encontró muchos fragmentos de DNA a diferencia si cosechaba uno o dos minutos después donde ya encontraba una molécula de DNA más grande. A estos fragmentos se les conoció como Fragmentos de Okazaki, ligados a través de la DNA Ligasa. Como se inicia la síntesis de estos fragmentos, sabemos que las polimerasas no inician la cadena, es un tipo de RNA polimerasa denominado primasa. La Maquinaria que opera en la horquilla El desenrollamiento del dúplex y la separación de las cadenas requieren la ayuda de dos tipos de proteínas que se unen al DNA, Una helicasa (o enzima que desenrolla al DNA) Y proteínas que se unen al DNA monocatenario (SSB). En las bacterias la helicasa y la primasa se relacionan de forma transitoria formando el primosoma. Estructura y función de las ADN polimerasas: La enzima que sintetiza las cadenas de DNA durante la replicación en E. coli es la DNA polimerasa III, que es parte de una “maquinaria de replicación” llamada holoenzima DNA polimerasa III Uno de los componentes no catalíticos de la holoenzima denominada pinza 𝛃 (abrazadera), mantiene la polimerasa relacionada con la plantilla de DNA. Las DNA polimerasas (como las polimerasas de RNA) poseen dos propiedades contrastantes: Tienen que permanecer vinculadas con la plantilla sobre largos tramos para sintetizar una cadena complementaria continua y No se pueden fijar con tanta fuerza que les impida desplazarse de un nucleótido al siguiente. El término replisoma se usa a menudo para referirse al complejo completo de proteínas que están activas en la horquilla de replicación, incluidas la holoenzima DNA polimerasa III, la helicasa, SSB y primasa. LA DNA POLIMERASA I Consiste sólo en una subunidad, interviene de manera primaria en la reparación del DNA, proceso mediante el cual se corrigen las secciones dañadas del DNA Actividades de la exonucleasa de las ADNpolimerasas La DNA polimerasa I siempre contiene actividades de exonucleasa, es decir, son capaces de degradar polímeros de DNA al eliminar uno o más nucleótidos del extremo de la molécula Las exonucleasas pueden dividirse en actividades de exonucleasa 5' → 3' y 3' → 5‘, según sea la dirección en la cual se degrada la cadena. Polimerasa I posee ambas actividades además de la polimerización Alta Fidelidad durante la replicación del ADN La actividad de exonucleasa 3' → 5' remueve alrededor de 99 de cada 100 bases mal pareadas, lo que incrementa la fidelidad a casi 10−7 a 10−8. Otra característica importante de la replicación en bacterias es la velocidad. Inicio de la Replicación Inicio de la Replicación Las células de los organismos superiores pueden tener mucho más de mil veces el DNA que las bacterias tienen, si bien sus polimerasas incorporan nucleótidos en el DNA a velocidades mucho más lentas. Para adecuar estas diferencias, las células eucariotas replican su genoma en pequeñas porciones, denominadas replicones. Existen secuencias que promueven la replicación del DNA, y se conocen como secuencias de replicacion autonoma (ARS). Las ARS que se han aislado y analizado comparten distintos elementos. El elemento central de una ARS consiste en una secuencia conservada de 11 pares de bases, que funciona como un sitio de unión específico para un complejo multiproteínico esencial llamado complejo de reconocimiento de inicio (ORC). Si la ARS muta de modo que sea incapaz de unirse al ORC, no es posible el inicio de la replicación. Como en la DNA polimerasa III de E. coli, la polimerasa que replica el DNA eucariota está presente como un dímero, lo que sugiere que las cadenas adelantada y retrasada se sintetizan de manera coordinada por un solo complejo de replicación o replisoma En la actualidad se han aislado las cinco DNA polimerasas “típicas” de las células eucariotas y se conocen como α, β, γ, δ, ε. De estas enzimas, la polimerasa γ replica el DNA mitocondrial y la β funciona en la reparación del DNA. Las otras tres polimerasas tienen funciones de replicación. La polimerasa α está muy relacionada con la primasa y juntas inician la síntesis de cada fragmento de Okazaki. De la misma manera que la principal enzima de replicación de E. coli, las polimerasas δ y ε requieren una “pinza deslizante” que mantiene unida la enzima al DNA y ello hace posible a ésta moverse de manera continua a lo largo de la plantilla. PCNA tiene una participación importante en la orquestación de los fenómenos que ocurren durante la replicación, reparación y recombinación del DNA. Por esta capacidad para unirse con un conjunto diverso de proteínas, PCNA se ha denominado “cinturón de herramientas moleculares”. Replicación y estructura nuclear: El aparato de replicación consiste en un complejo de proteínas que opera dentro de los confines de un núcleo estructurado La horquilla de replicación activa en un momento particular no se distribuye de forma aleatoria a través del núcleo celular; en realidad, se localiza en 50 a 250 sitios, conocidos como lugares de replicación Estructura y replicación de la cromatina Los cromosomas de las células eucariotas consisten en DNA unido en complejos con conjuntos regulares de proteínas histonas que se presentan en forma de nucleosomas. La mejor estudiada de estas chaperonas de histona, CAF-1, se atrae a la horquilla de replicación en movimiento mediante una interacción con la pinza deslizante PCNA. REPARACION DEL ADN La falla para reparar estas lesiones produce anomalías permanentes, o mutaciones, en el DNA. Si la mutación tiene lugar en una célula destinada a ser un gameto, la alteración genética puede pasar de una generación a la próxima. Las células tienen una amplia variedad de sistemas de reparación que corrigen casi cualquier tipo de daño al cual una molécula de DNA es vulnerable Tanto las células procariotas como las eucariotas poseen una variedad de proteínas que recorren extensos tramos de DNA y buscan alteraciones químicas o distorsiones sutiles del DNA dúplex. La reparación del daño del DNA en células eucariotas es complicada por la inaccesibilidad relativa del DNA dentro de las fibras de cromatina plegadas del núcleo. Escisión de Nucleótidos y Reparación La reparación de la escisión nucleotidica (NER) opera por un mecanismo de corte y pegado que elimina diversas lesiones voluminosas, incluidos los dímeros de pirimidina y los nucleótidos en los cuales distintos grupos químicos se unen. Se conocen dos vías de la reparación de la escisión nucleotidica: Vía 1: Vía acoplada a la transcripción: En esta las cadenas de DNA plantilla de los genes que se transcriben de forma activa se reparan de manera preferencial. La reparación de una cadena plantilla ocurre al parecer a medida que el DNA se transcribe y la presencia de la lesión puede señalarla una RNA polimerasa atorada. Esta vía de reparación preferencial garantiza que estos genes de gran importancia para la célula, que son los genes que la célula transcribe de manera activa, reciban la prioridad más alta en la “lista de reparación”. Vía 2: Vía genómica global Mecanismo más lento y menos eficiente Corrige las cadenas de DNA en el resto del genoma Componente Clave Un componente clave de la maquinaria de reparación NER es el factor TFIIH, una gran proteína que también participa en el inicio de la transcripción. Escisión de nucleótidos Incluidas dentro de las diferentes subunidades de TFIIH están dos subunidades (XPB y XPD) que poseen actividad de helicasa; estas enzimas separan las dos cadenas del dúplex (paso 2) en la preparación para la eliminación de las lesiones. Un par de endonucleasas (paso 3) corta entonces la cadena dañada en ambos lados de la lesión y el segmento de DNA se elimina (paso 4). Una vez suprimido, el espacio se reocupa por medio de una DNA polimerasa (paso 5) y la cadena se liga por medio de una DNA ligasa (paso 6). Reparación por escisión de bases Un sistema de reparación por escisión elimina los nucleótidos alterados generados por los reactivos químicos presentes en la dieta o por el metabolismo. Se han identificado diferentes glucosilasas de DNA, cada una de ellas más o menos específica para un tipo en particular de base alterada, incluidos el uracilo (formado por la remoción hidrolítica del grupo amino de la citosina), la 8-oxoguanina (secundaria al daño de radicales libres del oxígeno) y la 3-metiladenina (generada por la transferencia de un grupo metilo de un donador de metilo). Reparación de la unión deficiente Las células pueden eliminar bases mal unidas incorporadas por la DNA polimerasa y las que se escapan de la lectura y corrección por medio de la enzima exonucleasa. Este proceso se conoce como reparación de la unión deficiente. Reparación de la unión deficiente. Reparación de la rotura de doble cadena Los rayos X, los rayos gamma y las partículas liberadas por los átomos radiactivos se describen como radiación ionizante porque generan iones que atraviesan la materia. Las roturas de la doble cadena (DSB) también pueden deberse a ciertos químicos, incluidos los utilizados en la quimioterapia del cáncer (p. ej., bleomicina) y radicales libres generados por el metabolismo normal de la célula. Una sola rotura de la doble cadena puede causar anomalías cromosómicas serias y, al final, letales para las células. La vía principal en las células de mamíferos se llama unión de extremos no homólogos, en la cual un complejo de proteínas se une a los extremos rotos del DNA dúplex y cataliza una serie de reacciones que de nueva cuenta unen las cadenas rotas.









