Diversidad genética de los aislados colombianos de
Anuncio

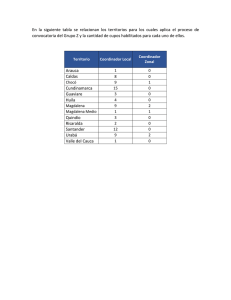
Genética de poblaciones Diversidad genética de los aislados colombianos de Mycosphaerella fijiensis Morelet basada en marcadores de microsatélites I. Perea, E. Rodríguez Arango, E. Márquez y R. Arango L a Sigatoka negra o raya negra de la hoja, causada por el hongo Mycosphaerella fijiensis Morelet (Stover 1980), fue reconocida por primera vez en la costa sudoriental de Viti Levu en Fiji en 1963 (Rhodes 1964). Posteriormente, la enfermedad fue registrada en las islas del Pacífico, Asia, África y finalmente, en América Latina en 1972 en La Lima, Honduras (Stover y Dickson, 1976). El hongo es haploide durante casi todo su ciclo de vida y se reproduce tanto asexual como sexualmente, vía conidias y ascósporas, respectivamente. Varios estudios han descrito la variabilidad genética global de M. fijiensis (Carlier et al. 1994, 1996), basados principalmente en el análisis de los aislados de los hongos de diferentes continentes utilizando RFLP y marcadores PCR-RFLP (Rivas et al. 2004). La realización de estudios a escala local podría ayudar a mejorar el manejo local de la enfermedad y programas para el mejoramiento de la resistencia a la Sigatoka negra. En Colombia, la Sigatoka negra fue observada por primera vez en 1981 (Mourichon y Fullerton 1990) en la región productora de banano de Urabá, de donde probablemente se propagó al resto del país. Las principales áreas productoras de banano en Colombia están localizadas en Urabá y Magdalena, donde los bananos se cultivan a alta densidad en grandes plantaciones y son controlados utilizando fungicidas químicos (Figura 1). Las áreas de producción platanera se localizan principalmente en Tolima y Arauca donde los plátanos se cultivan en pequeñas y medianas fincas, en asociación con café, cacao u otros, con poco o ningún control químico. En el presente estudio se utilizaron los marcadores de microsatélites para realizar una encuesta preliminar de la variabilidad genética de 40 aislados de M. fijiensis de las principales regiones locales bananeras y plataneras de Colombia. Materiales y métodos 240 Km 240 Km Magdalena Venezuela Panamá Urabá Arauca Tolíma Ecuador Se tomaron muestras de las hojas de banano y plátano infectadas con M. fijiensis en cuatro localidades en Colombia. De los 40 aislados recolectados, 11 provenían de Urabá, 10 de Tolima, 9 de Arauca, y 10 de Magdalena. Los cultivares de los cuales se tomaron las 18 muestras incluían ‘Grand naine’ (AAA) en Magdalena y Urabá, y ‘Dominico hartón’ (AAB) en Tolima y Arauca. Cada aislado fue obtenido de hojas de plantas diferentes recolectadas al azar. Todos los aislados fueron cultivados a partir de una sola ascóspora y mantenidos en el PDA (agar de dextrosa de papa) en tubos de ensayo a 28 ± 2°C e incubados en oscuridad por 30 días. De cada aislado se extrajo el ADN utilizando un método basado en CTAB (Weising et al. 1991) con menores modificaciones descritas en Romero et al. 1999. El análisis de los microsatélites se realizó utilizando siete pares de cebadores para los loci Mf SSR 005, Mf SSR 025, Mf SSR 061, Mf SSR 137, Mf SSR 203, Mf SSR 194 y Mf SSR244 (Neu et al. 1999). El análisis PCR fue realizado en un búfer con volumen de reacción de 25 ml que contenía 2 mM de MgCl2, 0.2 mM de dNTPs, 12.5 pmol de cada cebador, 0.625 unidades de polimerasa de ADN Taq DNA (Promega, California), 1X de búfer de PCR (50 mM de KCl , 10 mM de Tris HCl, pH 8.3), Perú Brasil Figura 1. Principales regiones bananeras y plataneras de Colombia. Figure 1. Main banana and plantain growing regions in Colombia. InfoMusa - Vol. 14 N°2 Diciembre 2005 y 10-50 ng de patrón de ADN. Después de la desnaturalización inicial se realizaron amplificaciones en un thermocycler (MJ Research PT-200, Mass.) programado para 2 min. a 94°C; 30 ciclos de 30 segundos a 94°C, 45 segundos a 55°C y 45 segundos a 72°C; con una extensión final de 7 min. a 72°C. Para determinar el tamaño de los alelos, un volumen de 2 ml del producto de PCR, combinado con 3 ml de formamido de búfer de carga (loading buffer) (80% (w/v), 10 mM de EDTA pH (8.0), 1 mg/ml de bromofenol azul y 1mg/ml de xileno cianol FF), fue desnaturalizado a 98°C por 5 min. Esta mezcla fue cargada en el gel desnaturalizador de poliarilamida al 6%, que contenía 7 mol/L de urea. La electroforesis fue llevada a cabo con el búfer 1X TBE en un sistema Sequi-Gen ® (Bio-Rad). Los tamaños de alelos fueron estimados utilizando una escalera de ADN de 10bp (Gibco BRL). Los datos se consideraron como haploides, ya que el ADN fue aislado del cultivo de una sola ascóspora. Para cada locus se calculó la cantidad de alelos. La diversidad génica fue estimada utilizando el índice de Nei (h) (Nei 1978) y la diversidad genotípica, utilizando la estadística G de Stoddart y Taylor, considerando cada genotipo como un haplotipo con loci múltiples (Hayden et al. 2003). Para comparar las muestras de diferentes tamaños, el valor de G fue dividido por el tamaño de la muestra. Los cálculos de la diversidad génica se realizaron utilizando el programa de computadora GDA (Lewis y Zaykin 2001). La diferenciación genética se estimó utilizando las estadísticas Fst de Wright como se describe en Weir y Cockerham (1984), utilizando el programa de computadora Arlequín (Schneider et al. 2000). El significado de los valores Fst fue examinado con el método descrito en Excoffier et al. (1992) utilizando 3024 permutaciones. Resultados y discusión La diversidad génica estimada en la muestra de Arauca (0.42) fue más alta que los valores obtenidos para Urabá (0.33) y Magdalena (0.36), aunque en ausencia de la prueba apropiada es imposible distinguir las muestras (Tabla 1). Utilizando nueve marcadores de microsatélites, incluyendo siete utilizados en nuestro estudio, Molina y Kahl (2004) obtuvieron una diversidad génica de 0.13 para la región de Magdalena. La diferencia podría deberse a diferencias en el muestreo o al hecho de que los últimos autores utilizan más marcadores. La diversidad global génica (h) de nuestras muestras fue 0.46 (Tabla 1), un valor similar al 0.40 registrado por Neu et al. (1999) para las poblaciones de M. fijiensis en México y al 0.42 registrado por Molina y Kahl (2004) para un conjunto de aislados de Costa Rica, Colombia y Venezuela. Los productos de PCR revelaron dos alelos en cada locus, exceptuando Mf SSR 194 y Mf SSR 244, los cuales tenían tres alelos por locus. En promedio, la población tenía 2.03 alelos por locus (Tabla 2). Este valor es aparentemente más bajo que los valores registrados para México (2.6) y Nigeria (2.7) utilizando los mismos marcadores de microsatélites (Neu et al. 1999), pero no es posible decir si las diferencias son significativas estadísticamente. Entre los 40 aislados estudiados se descubrió un total de 36 distintos haplotipos de loci múltiples. Los valores G calculados para cada localidad muestreada mostraron altos niveles de diversidad genotípica (Tabla 2). La diversidad genotípica se encuentra cerca del máximo teórico, ya que la mayoría de los haplotipos fueron observados solo una vez. Esto es muy similar a lo que fue descrito por Carlier et al. (1996) quienes observaron que cada aislado de M. fijiensis correspondía a un solo haplotipo de loci múltiple, en 19 loci de RFLP evaluados. Algunos de los aislados similares provenían de localidades distantes, por ejemplo, el aislado 990920 de Tolima tenía el mismo haplotipo que el aislado 981111 de Arauca. La colección de aislados con haplotipos similares puede representar el resultado de Tabla 1. Estimados de la diversidad génica de Nei en siete loci de los aislados de Mycosphaerella fijiensis provenientes de cuatro poblaciones en Colombia. Locus MfSSR 005 MfSSR 025 MfSSR 061 MfSSR 137 MfSSR 194 MfSSR 244 MfSSR 203 Media Desviación estándar Arauca 0.34 0.34 0.34 0.49 0.56 0.44 0.44 0.42 0.08 Magdalena 0.18 0.32 0.18 0.42 0.48 0.58 0.42 0.36 0.15 InfoMusa - Vol. 14 N°2 Diciembre 2005 Tolima 0.32 0.50 0.50 0.48 0.42 0.18 0.42 0.40 0.11 Urabá 0.49 0.29 0.46 0.29 0.00 0.39 0.39 0.33 0.16 Total 0.39 0.39 0.42 0.45 0.62 0.48 0.48 0.46 0.07 19 Tabla 2. Resultados de los análisis de cuatro poblaciones de Mycosphaerella fijiensis en Colombia utilizando siete marcadores de microsatélites. Número de aislados Número de genotipos Número de alelos/locus Diversidad génica (h) Diversidad genotípica (G) Tamaño de G/muestra Arauca 9 9 2.1 0.42 9 1 Magdalena 10 10 2.0 0.36 9.36 0.85 Tolima 10 10 2.0 0.40 10 1 Urabá 11 9 1.8 0.33 10 1 Total 40 36 2.03 0.46 38.1 0.95 Tabla 3. Niveles de diferenciación genética (Fst) entre las cuatro poblaciones de Mycosphaerella fijiensis en Colombia. Tolima Urabá Arauca Magdalena Tolima 0.00000 0.20158* 0.07566 0.10979* Urabá Arauca 0.00000 0.13086* 0.26992* 0.00000 0.03130 Magdalena 0.00000 * Denota un valor p igual o menor que 0.05 (Excoffier et al. 1992). un muestreo independiente de la combinación más frecuente de alelos. El estimado global de Fst fue 0.145. Tal como se ve en la tabla 3, los valores de Fst variaron de 0.07566 hasta 0.26992 indicando algún grado de diferenciación, aunque los niveles logrados no fueron tan altos como los niveles obtenidos por Rivas et al. (2004) entre las poblaciones de varios países de América Latina, que variaron entre 0.03 y 0.58. Esto no es sorprendente, ya que nuestros datos fueron recolectados solo en un país. Aunque la cantidad de aislados en nuestro estudio es pequeña, se debe realizar más estudios, ya que nuestros datos ofrecen un primer estimado de la variabilidad genética de M. fijiensis en algunas regiones de Colombia. Agradecimiento I. Perea y E. Rodríguez Arango trabajan en la Unidad de Biotecnología Vegetal, Corporación para Investigaciones Biológicas, Cra. 72 A No. 78B- 141 Medellín, Colombia. E. Márquez trabaja en la Escuela de Biociencias, Facultad de Ciencias, Universidad Nacional sede Medellín, Calle 64 Cra. 65 Autopista Norte, Medellín, Colombia, y Rafael Arango trabaja en ambas instituciones. Autor para correspondencia: [email protected] Este trabajo fue apoyado en parte por el Instituto Colombiano para el Desarrollo de la Ciencia y la Tecnología “Francisco José de Caldas” (Colciencias), subvención de Colombia No. 2213-05-157-97, la Corporación para Investigaciones Biológicas, CIB y el Programa de Postrado en Biotecnología de la Universidad Nacional de Colombia, sede Medellín. Agradecemos al Dr. Dieter Kammer y al Dr. Gunter Kahl por suministrar cebadores de microsatélites asi como a la Dra. Alba Estella Riveros, Sra. Alegría Saldarriaga y Sr. Alberto Martínez por su ayuda en el muestreo y aislamiento de Mycosphaerella spp.; también agradecemos al Dr. Nicolás Jaramillo, Dr. Mario Lobo, Dra. Ángela Restrepo y Dra. Helga VonPlaten, por revisar y mejorar el manuscrito. Referencias Carlier J., M.H. Lebrun, M.F. Zapater, C. Dubois & X.Mourichon. 1996. Genetic structure of the global population of banana black leaf streak fungus Mycosphaerella fijiensis. Molecular Ecology 5:499510. 20 Carlier J., X. Mourichom, D. Gonzales de León, M.F. Zapater & M.H. Lebrun. 1994. DNA restriction fragment length polymorphisms in Mycosphaerella species that cause banana leaf spot diseases. Phytopatology 84:751-756 Excoffier L., P. Smouse & J. Quatro. 1992. Analysis of molecular variance inferred from metric distances among DNA haplotypes: Application to human mitochondrial DNA restriction data. Genetics 131: 479-491. Hayden H.L., J. Carlier & E.A.B. Aitken. 2003 Genetic structure of Mycosphaerella fijiensis populations from Australia, Papua New Guinea and the Pacific Islands. Plant Pathology 52:703-712 Lewis P. & D. Zaykin. 2001. Genetic Data Analysis ( GDA) User’s manual. Molina C.M. & G. Kahl. 2004. Genomics of two banana pathogens: genetic diversity, diagnostics, and phylogeny of Mycosphaerella fijiensis and M. musicola. Pp. 127-146 in Banana Improvement Cellular, Molecular Biology, and Induced Mutations (S. Mohan Jain and R. Swennen, eds). Science Publishers Inc., Enfield, USA. Mourichon X. & R.A. Fullerton. 1990. Geographical distribution of the two species, Mycosphaerella musicola Leach (Cercosporamusae) and Mycosphaerella fijiensis (Cercospora fijiensis), respectively agents of Sigatoka disease and black leaf streak disease in bananas and plantains. Fruits 45(3):213-218 Nei M. 1978. Estimation of average heterozygozity and genetic distance from small number of individuals. Genetics 89:583-590 Neu C., D. Kaemmer, G. Kahl, D. Fisher & K. Weising. 1999. Polymorphic microsatellite markers for the banana pathogen Mycosphaerella fijiensis. Molecular Ecology 8:523-525 Rivas G.G., M.F. Zapater, C. Abadie & J. Carlier. 2004. Founder effects and stochastic dispersal at the continental scale of the fungal pathogen of bananas Mycosphaerella fijiensis. Molecular Ecology 13: 471-482. Rhodes P.L. 1964. A new banana disease in Fiji. Commonwealth Phytopathology News 10:38-41 Romero M., T. Diaz, D. Castañeda & R. Arango. 1999. Diagnostico para PCR del complejo Sigatoka en Colombia. Revista Facultad de Agronomía. 52:425-434 InfoMusa - Vol. 14 N°2 Diciembre 2005 Schneider S., D. Roessli & L. Excoffier. 2000. Arlequin Stover R.H. & J. Dickson. 1976. Banana leaf spot caused ver 2000: A software for population genetics data by Mycosphaerella musicola and Mycosphaerella analysis. Laboratory, fijiensis var difformis: a comparison of the first central Stover R. 1980. Sigatoka leaf spot of banana and Weir B. & C. Cockerham. 1984. Estimation of F-statistic for the Genetics and Biometry University of Geneva, Switzerland. plantains. Plant Disease 64:750-755 American epidemics. FAO Plant Prot. Bull. 24:36-42. analysis of population structure. Evolution 38:1358-1370. Estimación del tamaño del sistema radical utilizando muestras testigo H. H. Mukasa, D. Ocan, P. R. Rubaihayo y G. Blomme L a excavación del sistema radical de una planta de banano que crece en el campo es un proceso destructivo y consumidor de tiempo. Por ejemplo, se necesitan seis hombreshora para excavar y evaluar el sistema radical de una planta de banano maduro (Blomme 2000). La estimación del tamaño del sistema radical de una mata tomando muestras de las raíces podría ser mucho más rápida. Para la fresa (Fragana xananassa Duch.), Fort y Shaw (1998) demostraron una correspondencia sustancial en la variabilidad de las muestras testigo del suelo y la masa radical de toda la planta, indicando que los cambios en el crecimiento del sistema radical podrían ser estimados eficazmente a partir de las muestras testigo del suelo. Las muestras testigo del suelo requieren no más del 10% del tiempo empleado para recolectar y procesar el sistema radical de la planta entera. Blomme (2000) evaluó plantas de banano cultivadas en el campo en la estación de pluviometría alta del Instituto Internacional de Agricultura Tropical en Nigeria y reportó que las características del sistema radical de la mata podrían ser evaluadas adecuadamente a partir de las muestras testigo. En adición, la obtención de las raíces de las muestras testigo del suelo requirió sólo una fracción del tiempo (por ejemplo, el 5% para dos muestras testigo del suelo) necesitado para excavar y evaluar el sistema radical completo de una planta madura. El objetivo de este estudio consistió en evaluar si el enfoque de muestras testigo proveería suficiente información para estimar el tamaño del sistema radical de la mata en un amplio rango de genotipos de bananos de Africa Oriental, que crecen en las fincas. Materiales y métodos Este estudio fue realizado en las fincas en los distritos de Masaka y Bushenyi en el sudoeste de Uganda, dos áreas productoras de banano importantes. El sitio Masaka-Lwengo se clasifica bajo el sistema banano-café, mientras que el sitio Bushenyi, bajo el sistema de montaña (INIBAP 2003). La altitud en el sitio Masaka-Lwengo varía entre 1080-1330 metros, con una precipitación anual promedio de 1200 mm y suelos clasificados como Luvisoles (FAO 1998). La altitud en el sitio Bushenyi varía entre 1600-1800 metros con una precipitación anual promedio de 1588 mm y suelos clasificados como Acrisoles (FAO 1998). En cada sitio se evaluaron ocho genotipos de Musa: los genotipos de banano de altiplanos de Africa Oriental (AAA-EAHB) ‘Mpologoma’, ‘Lwadungu’, ‘Nakitembe’, ‘Mbwazirume’ y ‘Kibuzi’, el banano de postre ‘Sukali Ndizi’ (AAB), el plátano ‘Gonja’ (AAB) y el banano cervecero ‘Kayinja’ (ABB). En cada sitio fueron evaluadas veinte plantas por genotipo con dos a cinco matas por finca. Las matas seleccionadas estaban en la cuarta etapa de desarrollo y consistían de una planta madre lista para cosechar con dos o tres retoños. La selección de los genotipos AAAEAHB se basó en las series de clones con el fin de incluir un genotipo representativo de cada serie de cuatro clones (Karamura y Pickersgill 1999). El sistema radical fue evaluado utilizando el método de muestra testigo (Blomme 2000). Se tomaron tres muestras testigo del suelo en cada mata: de la posición contigua al retoño más alto y a 90° y 180° según las manecillas del reloj desde el retoño más alto. Los testigos tenían un diámetro de 25 cm y una altura de 80 cm. Los testigos fueron marcados utilizando un anillo de metal y se removieron con un pequeño azadón. Las muestras fueron lavadas para descartar las partículas de suelo y para cada muestra testigo se recolectaron los datos del peso seco y el largo de la raíz adventicia (Tennant 1975). Posteriormente, las plantas fueron enteramente excavadas y algunas de las características fueron medidas en la planta completa. El análisis estadístico se realizó utilizando el paquete estadístico Genstat (Genstat 1999). Se ejecutó una prueba ANOVA para determinar los efectos de la localización de la planta y de la InfoMusa - Vol. 14 N°2 Diciembre 2005 21 Sistema radical