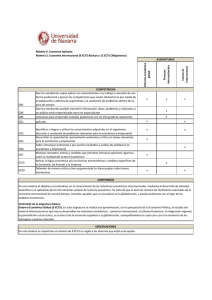
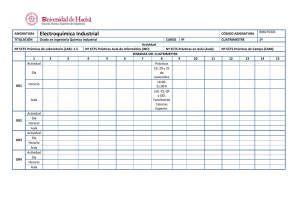
master en biotecnología molecular, celular y genética
Anuncio

Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 MASTER EN BIOTECNOLOGÍA MOLECULAR, CELULAR Y GENÉTICA PROGRAMA DE POSTGRADO: BIOCIENCIAS Y CIENCIAS AGROALIMENTARIAS Curso 2009-10 1 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 ÍNDICE PRESENTACIÓN……………………………………………………...…4 1. GUIA DOCENTE ASIGNATURAS COMUNES Transferencia de Tecnología, Protección de Resultados de Investigación y Creación de Empresas de Base Tecnológica…………………………………………………………....7 Introducción a la Bioinformática………………………………………...9 2. GUÍA DOCENTE ASIGNATURAS METODOLÓGICAS Bioquímica de Proteínas y Proteómica………………...………..…...17 Técnicas Básicas del DNA Recombinante ……………………...…..21 Metodología de la Experimentación en Biología Celular………...…27 3. ITINERARIO 1: BIOTECNOLOGÍA VEGETAL Y AMBIENTAL Técnicas Avanzadas en Genómica Funcional………………...…….32 Biotecnología Ambiental……………………………………………….39 Biotecnología Vegetal……………………………………………….…43 Biotecnología de Eucariotas Unicelulares…………………………...47 Transporte de Solutos en Membranas Vegetales…………………..51 Metabolómica…………………………………………………………...52 4. ITINERARIO 2: BIOTECNOLOGÍA SANITARIA Técnicas Avanzadas en Genómica Funcional ………….………..…32 Técnicas Avanzadas de Imagen Celular……………….…………....59 Inmunobiología Molecular………...………………….………………..62 Avances en Neuroendocrinología………………….…………………65 Cultivos Celulares…………………………………….………………...71 Metabolómica ……………………………………….………………….52 5. ITINERARIO 3: ANÁLISIS GENÉTICO EN BIOTECNOLOGÍA Transformación Aplicada a la Mejora Vegetal. ……………..………76 Mecanismos de Patogénesis y Resistencia en las Enfermedades de las Plantas ………………………………………………………………78 Recursos Fitogenéticos y Evolución de Plantas Cultivadas……….81 2 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Manipulación Cromosómica en Plantas……………………………...82 Epigenética……………………………………………………………...84 Genética del Comportamiento………………………………………...88 Marcadores Moleculares y su Uso en Mejora Genética…………....92 Análisis e Interpretación de Genomas………………………………..95 Filogenias y Análisis de Datos Genéticos……………………………98 6. HORARIO DEL MASTER…………………………………...…….100 7. LÍNEAS DE INVESTIGACIÓN…………………………………....102 3 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 GUíA DEL MÁSTER EN BIOTECNOLOGíA MOLECULAR CELULAR Y GENÉTICA PROGRAMA DE POSTGRADO: BIOCIENCIAS Y CIENCIAS AGROALIMENTARIAS Curso 2009-10 Esta guía tiene como objetivo orientar a cerca del contenido y metodologías de docencia y los métodos de evaluación de las asignaturas del Master. Asimismo se incluye el horario para facilitar la elección de las asignaturas a cursar. Se recuerda al alumno que, de partida, debe elegir su tutor, quien le orientará sobre las asignaturas optativas a seleccionar y la línea de investigación sobre la que desarrollará el trabajo final del Máster. Contenido: 1. Guía docente de las asignaturas comunes (8 ECTS) 2. Guía docente de las asignaturas metodológicas (8 ECTS) 3. Itinerario 1: Biotecnología Vegetal y Ambiental. 4. Itinerario 2: Biotecnología Sanitaria. 5. Itinerario 3: Análisis Genético en Biotecnología. 6. Horario del Master. 7. Líneas de investigación, de donde se elegirá el tutor y el tema para el trabajo experimental del Master (16 ECTS) 4 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 ESQUEMA ACADÉMICO DEL MÁSTER MASTER en BIOTECNOLOGIA MOLECULAR, CELULAR Y GENETICA FORMACIÓN PROFESIONAL 16 créditos de Asignaturas comunes (2 X 4ECTS) 1. Transferencia de Tecnología, protección de resultados de investigación y creación de empresas de base tecnológica 2. Introducción a la Bioinformática Asignaturas metodológicas: (2 X 4ECTS) a elegir 2 de: 1. Bioquímica de proteínas y Proteómica. 2. Técnicas básicas del DNA recombinante 3. Metodología de la experimentación en Biología Celular FORMACIÓN INVESTIGADORA 16 créditos de A elegir 2 de las asignaturas de Investigación Transversales de la UCO (2 x 4 ECTS) Asignaturas metodológicas: (2 X 4ECTS) a elegir 2 de: 1. Bioquímica de proteínas y Proteómica. 2. Técnicas básicas del DNA recombinante 3. Metodología de la experimentación en Biología Celular + 28 créditos (7 x 4) (especialización en un itinerario con al menos 5 asignaturas) I tinerario 1: Biotecnología Vegetal y Ambiental Itinerario 2: Biotecnol. Sanitaria Itinerario 3: Análisis Genético en Biotecnología (7 a elegir) - Técnicas avanzadas en Genómica Funcional - Biotecnología ambiental - Biotecnología vegetal - Biotecnología de eucariotas unicelulares -Transporte de solutos en membranas vegetales - Metabolómica - Técnicas avanzadas en Genómica Funcional - Técnicas avanzadas de imagen celular - Inmunobiología molecular - Avances en Neuroendocrinología - Cultivos Celulares - Metabolómica - Transformación aplicada a la mejora vegetal - Mecanismos de patogénesis y resistencia en las enfermedades de las plantas - Recursos fitogenéticos y evolución de plantas cultivadas - Manipulación cromosómica en plantas - Epigenética - Genética del comportamiento - Marcadores moleculares y su uso en mejora genética - Análisis e interpretación de genomas - Filogenias y análisis de datos genéticos 16 ECTS Trabajo Fin de Master 16 ECTS TFM investigación 16 ECTS TFM investigación 5 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 1. GUÍA DOCENTE ASIGNATURAS COMUNES 6 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Asignatura: TRANSFERENCIA DE TECNOLOGÍA, PROTECCIÓN DE RESULTADOS DE INVESTIGACIÓN Y CREACIÓN DE EMPRESAS DE BASE TECNOLÓGICA Total de créditos ECTS: 4 (100 horas) Tipo de asignatura: Obligatoria. Coordinador: Manuel Pineda Priego. La OTRI Discusión del Artículo 83 de la LOU El proceso de transferencia Funciones de la OTRI Los contratos con otras entidades El intercambio de personal universidad-empresa El programa Torres Quevedo Otros programas nacionales Programas internacionales Protección de resultados Propiedad intelectual e industrial: conceptos nacionales e internacionales El Copyright y los derechos de autor La propiedad industrial y sus modalidades Invenciones (patentes y modelos de utilidad) Variedades vegetales Marcas Signos distintivos Funciones de la patentes Gestión de la propiedad II en la Universidad: explotación y licencia Vías de protección: nacional, europea y PCT Las patentes en Biotecnología Las empresas de base tecnológica El concepto y su regulación El proceso de creación y su forma jurídica Participación del profesorado: ley de incompatibilidades La participación de las universidades: reglamentación y procedimientos 7 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 TABLA 1: La estructura curricular a MATERIA/ ASIGNATURA (Unidad de matrícula) Transferencia de tecnología, protección de resultados de investigación y creación de empresas de base tecnológica b OBJETIVOS DE APRENDIZAJE 1. Conocer el proceso de transferencia de conocimiento a la sociedad 2. Identificar invenciones patentables 3. Conocer los mecanismos de búsqueda de recursos 4. Conocer el procedimiento de creación de empresas de base tecnológica c d e f g h i Nº CRS ECTS TIPO SECUEN CIA CTER. DESARROLLO ACTIVIDADES DE APRENDIZAJE EVALUACIÓN (d) Obligatoria (e) 1º semestre (f) Teórica/ Aplicada/Metodológica/Práctica (PFC, Prácticum, Problemas/casos) (g) Presencial/Semipresencial/No presencial) (h) Grandes tipologías: 1. Clases, seminarios 2. Prácticas "regladas": laboratorio, campo, seminarios, externas 3. Trabajos en grupo 4. Trabajos individuales (i) Ejecuciones del estudiante sobre las que se tendrá que evaluar: 1. Asistencia y participación en clase 2. Exámenes (papel y lápiz) 3. Ensayo, trabajo individual o en grupo 4. Exposiciones o demostraciones 5. Informes de prácticas 8 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Asignatura: INTRODUCCIÓN A LA BIOINFORMÁTICA Total de créditos ECTS: 4 Tipo de asignatura: Elegible. Nombre del Profesor responsable: Prof. Dr. Antonio Rodríguez Franco, Profesor Titular de Bioquímica y Biología Molecular, Departamento de Bioquímica y Biología Molecular, UCO. 1 ECTS. Profesores participantes[1]: Dr. Manuel Tejada Jiménez, Contratado Postdoctoral, Departamento de Bioquímica y Biología Molecular, UCO. 1 ECTS. Dr. Juan Ruano Ruiz, Unidad de Lípidos y Arteriosclerosis. Facultativo Especialista de Área de la Unidad de Gestión Clínica de Medicina Interna. Hospital Universitario Reina Sofía. 1 ECTS. Dr. Juan Ramón Peinado Mena, Contratado Postdoctoral, Unidad de Endocrinología, Departamento de Biología Celular, Fisiología e Inmunología, UCO. 1 ECTS. Competencias y objetivos del aprendizaje. Al final del curso, el alumno deberá conocer: 1. Diferentes sistemas operativos e informáticos. La manipulación de diferentes formatos de archivos (texto, binario) y de archivos de bases de datos de secuencias. Acceso WEB. Manipulación de gráficos 2. Las principales bases de datos primarias y secundarias de ácidos nucleicos y de proteínas y la forma de acceder a las mismas y de manejarlas. 3. Los principales conceptos informáticos y de Bioinformática/Biología Computacional y las estrategias que se utilizan para al estudio de la estructura y función de las proteínas y de los ácidos nucleicos. 4. Las principales aplicaciones bioinformáticas para la visualización, modelado y análisis estructural de las proteínas. 5. Algunas de las herramientas de biología computacional para la predicción de genes y de la estructura de las proteínas a partir de la secuencia. 6. Los métodos para la predicción de función de la proteínas: colocalización cromosómica, duplicaciones y fusiones génicas, coexpresión y co-evolución. 7. El sistema de clasificación funcional jerárquico basado en términos GO. 8. La generación de redes semánticas de genes/proteínas a través de métodos de text mining en la literatura científica. 9 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Metodología. La Asignatura se dará presencialmente por los diferentes profesores y está implementada en la plataforma digital Moodle. Desde la misma, los alumnos tendrán acceso a los recursos más importantes (foros, chat, wiki, etc) así como a problemas y cuestiones bien definidos en cada tema que tendrán que resolver como parte del trabajo que deben realizar. Antes de cada “sesión” o bloque temático se tratará de proporcionar cuando sea posible a los estudiantes un “screencast” conteniendo información sobre la lección preparado ad hoc. Los estudiantes visualizarán este video, que les servirá de guión para luego ponerlo en práctica por sí mismos con el ejercicio práctico que les haya correspondido. Evaluación La calificación final se obtendrá de la suma de varias calificaciones: 1. Examen final: 30%. 2. Elaboración de ejercicios prácticos: 15%. 3. Cantidad y calidad de la participación en los foros de Moodle: 10%. 4. Resolución del problema del curso, que debe ser redactado en formato de artículo científico: 30%. 5. Lectura crítica de artículos científicos: 15%. La asistencia a las clases presenciales es obligatoria. PROGRAMA ABREVIADO TEMA 1. Conceptos básicos de Informática e Internet. TEMA 2. Bases de datos biológicas. TEMA 3. Análisis de secuencias biológicas. TEMA 4. Analisis de los genes a partir de la secuencia. TEMA 5. Análisis de las proteínas a partir de la secuencia. TEMA 6. Visualización y manipulación de la estructura 3D de las proteínas. TEMA 7. Análisis de datos de experimentos con chips de arrays. TEMA 8. Objetivos futuros de la Bioinformática. 10 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 PROGRAMA DESARROLLADO TEMA 1. Conceptos básicos de Informática e Internet. (Manuel Tejada Jiménez) OBJETIVOS: Toma de contacto, comprensión conceptual y familiarización con las herramientas de hardware y software. Conocer las características esenciales de las aplicaciones y servicios web. Conocer las definiciones de Bioinformática y Biología Computacional y los diferentes formatos de archivos. CONTENIDO: Conocer la estructura básica de una computadora y el funcionamiento básico de sus componentes (CPU, memoria, dispositivos E/S) al utilizar una aplicación (software, archivos, GUI, sistema operativo). Conocer la existencia de diferentes sistemas operativos (UNIX/Linux, Mac OS, Microsoft Windows). Conocer las características esenciales en relación a las aplicaciones de escritorio: ·Modelo matemático/estadístico, algoritmo, script, programa o aplicación, plugins, , suite informática, entorno de desarrollo informático; ·Editores de texto vs. procesadores de texto; ·Tipos y Manipulación de Imágenes ·Lenguaje máquina y lenguaje de alto nivel, compilador, debugs; ·Licencias GNU vs. software comercial; ·Órdenes en línea de comandos (terminal, prompt, intérprete de programa) vs GUI. Conceptos útiles sobre la Internet: dominios/servidores web, IP/DNS, protocolos HTTP/FTP, navegadores web (Firefox/Mozilla, Camino, Safari, Opera, Internet Explorer), web 2.0, documentos y editores html/CSS, scripts PHP/CGI/Ruby/Java, sesiones web y activación de cookies. Publicación en páginas WEB Modos de entradas de los datos (formulario/archivo/bacthing/workflows) y de salida de los resultados (pantalla/download/e-mail). Consideraciones prácticas sobre las herramientas/servicios web creados por instituciones educativas/públicas. Manejo de los diferentes tipos de archivos frecuentes en Bioinformática (.txt, .doc, .xls, .cvs, .xml, pdb, etc) TEMA 2. Bases de datos biológicas (Antonio R. Franco) OBJETIVOS: Conocer las características y manejar las diferentes bases datos en Ciencias de la Vida. 11 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 CONTENIDO: - Principales bases de datos de ácidos nucleicos y proteinas: EMBL, GenBank, UniProt, SwissProt, etc - Acceso a otras bases de datos, como la de publicaciones cientificas (PubMed, Mediline), del genoma humano (OMIM, MapView), de protocolos, de genomas completos, arrays, proteómica, etc. - Servicios de búsqueda de bases de datos: SRS, Entrez. Identificación de los diferentes campos de cada ficha, de los métodos de filtrado y recuperación de datos. - Propiedades y Anotaciones en las secuencias TEMA 3. Análisis de secuencias biológicas. (Antonio R. Franco) OBJETIVOS: Comparación de secuencias de ácidos nucleicos y de proteínas. CONTENIDO: - Alineamiento y comparación de pares de secuencias. Programa Dotlet. Algoritmos globales (FASTA) y locales (BLAST). Matrices de comparación Penalización de creación de huecos y de extensión en los alineamientos. - Alineamiento y comparación de 3 o más secuencias. - Aplicaciones TEMA 4. Analisis de los genes a partir de la secuencia. (Manuel Tejada Jiménez) OBJETIVOS: Análisis funcional y estructural de las secuencias de núcleotidos. CONTENIDOS: - Predicción de ORFs, TFBS en regiones promotoras, intrones/exones, snARN, microRNA - Identificación de dianas para enzimas de restricción. Digestión ‘virtual’ de secuencias. TEMA 5. Análisis de las proteínas a partir de la secuencia. (Juan R Peinado) OBJETIVOS: Conocer las estrategias bioinformáticas para la predicción de la función de una proteína a partir de datos estructurales y genómicos. 12 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 CONTENIDO: - Paquetes bioinformáticos para la predicción de propiedades físico químicas y estructurales de las proteínas a partir de la secuencia. Predicción de péptidos señal, residuos/regiones potencialmente sometidas a regulación enzimática (fosforilación, mutilación, glicosilación, etc). - Identificar las principales estrategias y métodos computacionales para la predicción de regiones estructuralmente ordenadas y desordenadas en la proteína - Otros métodos para la predicción de función: localización cromosómica, duplicaciones y fusiones génicas, co-expresión y co-evolución. - Conocer la estructura jerárquica que rige las clasificaciones de ontologías (‘ramas’, ‘hijos’ y ‘madres’), así como las principales bases de datos sobre términos GO, las herramientas de búsqueda y los métodos de análisis (enriquecimiento de términos en grupos de genes/proteínas, análisis comparativos). TEMA 6. Visualización y manipulación de la estructura 3D de las proteínas. (Juan R Peinado) OBJETIVOS: Visualizar y manipular las estructuras tridimensionales de las proteínas y sus interacciones con ligandos específicos. CONTENIDO: - Formas de representar las proteínas (cintas, cartoon, cordón). - Conceptos de plegamiento/flexibilidad, estructuras secundaria, terciaria y cuaternaria. - Acceso a estructuras macromoleculares en la web. - Destreza en los principales programas para el manejo de datos macromoleculares y coordenadas tridimensionales. - Bases de datos especializadas y clasificación de estructuras resueltas. - Predicción de estructuras por modelado, “threading” y ”ab initio”. - Aplicaciones para la predicción funcional del efecto de sustituciones aminoacídicas TEMA 7. Análisis de datos de experimentos con chips de arrays. (Juan Ruano Ruiz) OBJETIVOS: Conocimiento de la validez y el alcance de la información obtenida mediante tecnologías de arrays de genes y de proteínas. 13 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 CONTENIDO: - Identificación de los tipos de chips de arrays y utilidad para el análisis de muestras biológicas a gran escala (expresión génica (tags, exones), variaciones génicas (SNP, CNV), tilling arrays (metilación de islas CpG, ChIP on chip, MeDIP-chip, CGH), proteínas). Chips comerciales y chips ‘a la carta’ de baja densidad. - Identificación de los factores comunes más importantes a tener en cuenta: a) diseño del experimento (hipótesis/objetivos planteados, controles externos e internos –spikes-, réplicas biológicas y técnicas, cáculo de la ‘n’) b) marcaje e hibridación del chip (fluoróforos, dye-swap, problemas de saturación) c) preprocesamiento de los datos de lectura (ruido de fondo, cuantificación de la señal, normalización, identifación de las flags) d) análisis de los resultados (expression diferencial, métodos de clustering, clasificación, métodos automatizados de anotación funcional) - Principales nociones sobre lo algoritmos matemáticos, técnicas estadísticas y aplicaciones para el análisis de los resultados. El problema del false discovery rate y posibles soluciones que se ofrecen. - Repositorios públicos para alojar/consultar los resultados. TEMA 8. Objetivos futuros de la Bioinformática. (Juan Ruano Ruiz) OBJETIVOS: Conocer el estado actual y futuro de la Bioinformática CONTENIDO: Creación de algoritmos/modelos matemáticos más eficientes; Herramientas estadísticas cada vez más complejas (métodos de clasificación basados en maching learning, análisis multivariante, false discovery rate, significación estadística). Lenguajes de programación potentes, orientados a objetos y con menos nivel de abstracción (Ruby, Python). Flujos automatizados de trabajo (BIOMOBY, Taverna). Redes neuronales. Métodos de integración de los procesos de análisis (Biología de Sistemas) y búsqueda (Redes Semánticas) de datos biológicos de diferente naturaleza (genómica, proteómica, transcriptómica, metabolómica, etc). Potencia y velocidad de computación: grid computing, paralelismo en computación y supercomputadoras. 14 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Text mining para la extracción de nuevos conocimientos y generación de hipótesis a partir de la información contenida en las publicaciones científicas (abstract, full text). BIBLIOGRAFÍA 1. http://www.uco.es/~bb1rofra/analisis/ 2. Orengo C.A., Jones D.T. y Thorton J.M. (2003) Bioinformatics. Genes, Proteins & Computers. BIOS Scientific Publishers. ISBN 1-85996-054-5 3. Current Protocols in Bioinformatics.(2005) John Wiley & Sons, Inc 4. http://www.ebi.ac.uk 5. http://www.ncbi.nlm.nih.gov 6. http://www.ddbj.nig.ac.jp/ 7. http://www.expasy.org 15 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 2. GUÍA DOCENTE ASIGNATURAS METODOLÓGICAS 16 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Asignatura: BIOQUÍMICA DE PROTEÍNAS Y PROTEÓMICA Total de créditos ECTS: 4 (100 horas) Tipo de asignatura: Elegible. PROFESORES: Prof. Dr. José Antonio Bárcena Ruiz. 1,0 ECTS. Dr. Manuel José Rodríguez Ortega, 2,0 ECTS. Dr. Víctor Manuel Luque Almagro, 2,0 ECTS. Colaboradores invitados: - Dr. Samuel Ogueta Villarreal, Servicio de Proteómica del SCAI, UCO. - Dra. Consuelo Gómez Díaz, Servicio de Proteómica del SCAI, UCO. - Dr. Fernando Corrales, CIMA, Universidad de Navarra. - Dr. Antonio Romero Ruiz, Hospital Virgen del Rocío, Sevilla OBJETIVOS ESPECÍFICOS DEL CURSO: Objetivos concretos -Aprender a manipular muestras biológicas para estudios Proteómicos. -Comprender los fundamentos de las técnicas preparativas de separación de proteínas empleadas en Proteómica. -Comprender los aspectos prácticos de la electroforesis bidimensional de proteínas para estudios de Proteómica de expresión diferencial. -Manejo práctico de programas de análisis de imagen para estudio de geles bidimensionales. -Conocer los fundamentos y variantes de la espectrometría de masas aplicada al estudio de las proteínas. -Preparar y analizar muestras mediante espectrometría de masas para identificación de proteínas. - Aprender a interpretar datos de espectrometría de masas de péptidos y utilizar motores de búsqueda conociendo las posibilidades y las limitaciones. -Comprender las estrategias experimentales más adecuadas para cada tipo de estudio proteómico. Competencias a adquirir -Capacidad de diseñar experimentos en cada campo de las Biociencias con el fin de aprovechar las posibilidades que ofrece la Proteómica para dar respuestas a los problemas biológicos. - Conocimiento de todos aspectos experimentales que conducen a la identificación del proteoma: Fraccionamientos electroforéticos y cromatográficos; espectrometría de masas de mezclas peptídicas; herramientas bioinformáticas. -Conocimiento de las limitaciones de la información proporcionada por un análisis proteómico. 17 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 -Capacidad para interpretar y ampliar la información estándar de un análisis de espectrometría de masas de péptidos por huella peptídica y por fragmentación. CONTENIDOS DEL CURSO: TEORÍA (1,5 ECTS): J. Antonio Bárcena y Manuel J. Rodríguez Ortega: T1: Introducción a las terminologías y ciencias “ómicas”. Proteoma y Proteómica. Separación y fraccionamiento de proteínas y péptidos mediante electroforesis bidimensional y cromatografía líquida. T2: Espectrometría de masas. Identificación de proteínas y péptidos mediante PMF y MS/MS. T3: Interpretación de espectros MS y MS/MS. Secuenciación “de novo”. Ejercicios prácticos. T4: Modificaciones postraduccionales y Proteómica cuantitativa. T5: Proteómica aplicada: data mining, perfiles de expresión proteica. Proteómica cuantitativa. Interactómica. Descubrimiento de biomarcadores. Biología de Sistemas. Ontologías. PRÁCTICAS (2,5 ECTS): Manuel J. Rodríguez Ortega y Víctor Luque. Realización de una electroforesis bidimensional de dos muestras biológicas comparables (Laboratorio Severo Ochoa): P1: (individual) Isoelectroenfoque (IEF). P2: (individual) 2ª dimensión; fijación y tinción. Análisis de imágenes de los geles bidimensionales mediante aplicaciones informáticas apropiadas (SCAI: laboratorio de proteómica y sala de informática): P3: (en grupo de 12) escaneo del gel e introducción al análisis de imagen. P4: (individual) utilización del software Melanie para el análisis de los geles y selección de manchas diferenciales. Extracción y digestión con tripsina de manchas proteicas diferenciales seleccionadas del gel de electroforesis 2D (Laboratorio Severo Ochoa): P5: (grupos de 6) Picado y digestión. Obtención de las “huellas peptídicas” por MS mediante MALDI-TOF (SCAI: laboratorio de proteómica): P6: (en grupos de 6) colocación en placa y disparo en el espectrómetro. Identificación de proteínas a partir de su huella peptídica mediante consulta en bases de datos proteicas, con el empleo de algoritmos específicos. Confirmación de la identificación por fragmentación de 3 péptidos por MALDITOF/TOF (SCAI: laboratorio de proteómica): Análisis y discusión de los resultados individuales (en el aula): P7: (en un único grupo) METODOLOGÍA DE ENSEÑANZA-APRENDIZAJE: Aproximaciones metodológicas. El contenido de la parte teórica se impartirá en el aula mediante clases magistrales de 120 minutos de duración, en las que se delinearán los diferentes temas haciendo hincapié en los conceptos esenciales ilustrados mediante la descripción de casos modelo. Los estudiantes completarán estas clases con la consulta en la bibliografía recomendada 18 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 para cada tema en particular. Los alumnos dispondrán de antemano de copias del material audiovisual de cada clase o tema. Las clases prácticas se impartirán en tres sitios: 1) Individualmente en el laboratorio de prácticas del segundo ciclo del Departamento de Bioquímica y Biología Molecular en el edificio Severo Ochoa. 2) En grupos de un máximo de 6 alumnos, en el laboratorio de proteómica del SCAI usando la infraestructura informática y de espectrometría de masas. 3) Una sesión inicial en grupos de 12 alumnos y luego individualmente, en sala de informática para manejo de software específico y simulaciones. Competencias a adquirir en actividades de carácter presencial. Al final del curso los estudiantes deben ser capaces de: Realizar una separación de proteínas por electroforesis 2D e interpretar los resultados. Evaluar la idoneidad de cada tipo de método de preparación de proteínas para análisis protéomico. Interpretar un análisis de huella peptídica y de fragmentación de péptidos para la identificación de proteínas y evaluar su validez. Decisión del tipo de aproximación proteómica adecuado para cada objetivo científico. Utilizar las bases de datos de acceso libre en Internet para recabar información estructural y funcional sobre proteínas. Otras actividades académicas dirigidas. Asistencia a seminarios o conferencias relacionados con la temática del curso. Elaboración de un informe de valoración personal sobre cada seminario. CRITERIOS DE EVALUACIÓN: Resolución de ejercicios prácticos sobre interpretación de espectros PMF y MS/MS: 30%. Presentación y en su caso, exposición oral, del informe práctico: 50% El progreso, implicación y aprovechamiento de cada estudiante se seguirá en cada sesión teórica y práctica mediante preguntas durante el desarrollo de las mismas: Informes sobre los seminarios y conferencias a los que asista: 20%. BIBLIOGRAFÍA BÁSICA: Libros o revisiones generales: PENNINGTON AND DUNN (2000). Proteomics. From protein sequence to function. BIOS Scientific. O’CONNOR, DC (2008) Proteomics. Scion Publishers. WESTERMEIER R, NAVEN T (2002) Proteomics in Practice. A Laboratory manual of Proteome Analysis. Wiley-VCH, Darmstadt, Germany. LINK & LE BAER (2009) Proteomics. Cold Spring Harbor Laboratory Protocols. CSHL Publ. 19 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 VARIOS AUTORES (2003) Proteomics. Nature, 422 (6928):191-237. STEEN, H & MANN, M (2004) The ABC’s (and XYZ’s) of peptide sequencing. Nature Reviews, 5:699-711. JENSEN, ON (2006) Interpreting the protein language using proteomics. Nat Rev Mol Cell Biol vol. 7 pp. 391-403. Bibliografía específica: Se proporcionará con el material didáctico de cada tema. Revistas periódicas (por orden de de interés): Molecular and Cellular Proteomics. Proteomics. Journal of Proteome Research Journal of Proteomics Practical Proteomics Nature Protocols Nature Methods Nature Biotechnology J. Chromatogr. J. Chromatogr. B Biomed. Sci. Appl. Anal. Biochem. Electrophoresis. Curr Opin. Biotechnol. Anal. Chem. J. Protein Chem. 20 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Distribución, temporalización y planificación. Distribución ECTS de la Asignatura BIOQUÍMICA DE PROTEÍNAS Y PROTEÓMICA (a) 1 ECTS = 25 horas trabajo. (b) Estimación de estudio personal del alumno durante el curso 12 (trimestral) o 36 (anual) semanas: 1,5 horas de estudio por cada hora de teoría y 0,75 horas de estudio por cada hora de prácticas. (c)Las tutorías se encuentran incluidas en el total de Actividades Académicamente Dirigidas. Actividad Actividad Docente Clases en aula Actividades dirigidasc Clases en laboratorio Exámenes Evaluación Alumno Procedimiento Peso en la nota final Horas presenciales Horas no presenciales Horas Profesor Exposición de la Teoría. Apoyo con audiovisuales Tomar apuntes, copiar el material audiovisual Varias preguntas cortas y una larga. Se valorará razonamiento y capacidad de síntesis 40 % 10 h 20 h b 30 h Resultados de prácticas Interpretación de espectros y criterios de búsqueda en bases de datos Exposición y discusión en grupo de los resultados individuales. Identificar sin ambigüedad las proteínas diferenciales No evaluable 2h 2h 4h Prácticas de laboratorio Preparación del material para cada grupo y alumno. Explicación y supervisión continua. Cuaderno de laboratorio, anotaciones, experimentos, informe, etc. Evaluación continuada, cuaderno, resultados, informes, actitud 18 h 20 h 38 h Teoría Poner, atender y corregir el examen. Calificar globalmente al alumno Preparación de examen (35 horas) Realización de examen (2 horas) 2h 26 h 28 h 32 h 68 h 100 h Materia Teoría TOTAL CARGA DOCENTE DEL ALUMNO 50% 90 % ECTS a Asignatura: TÉCNICAS BÁSICAS DEL DNA RECOMBINANTE Total de créditos ECTS: 4 Tipo de asignatura: Elegible. Profesorado: Concepción de la Hera y/o Carmen Ruiz Roldán (Módulo 1: Clonación,1 ECTS) Enriqueta Moyano Cañete y/o Carmen Michán Doña (Módulo 2: Hibridación de ácidos nucleicos, 1 ECTS) 21 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Conrado Moreno Vivián y Mª Dolores Roldán Ruiz (Módulo 3: PCR y mutagénesis dirigida, 1 ECTS y Módulo 4: Expresión de proteínas, 1 ECTS) La necesidad de impartir un Máster de calidad en una asignatura de alta experimentalidad como es Técnicas básicas del DNA Recombinante, exige que el número máximo de alumnos por grupo sea 10. En los cursos académicos en los que el número de matriculados sea superior a diez, será necesaria la distribución de alumnos en varios grupos y consecuentemente, la participación de más profesores. Por esta razón, se han incluido más de cuatro nombres de profesores, cuya participación estará vinculada a las necesidades docentes de cada año académico. Profesor invitado: se contará con la colaboración del Dr. José Gadea Vacas (Universidad Politécnica de Valencia-CSIC), experto en genómica funcional y técnicas de matrices de DNA que impartirá un seminario Objetivos específicos del curso - Adquirir los conocimientos básicos, tanto a nivel teórico como práctico, para su iniciación en la utilización de las principales técnicas de genómica y de ingeniería genética. - Conocer las técnicas de clonación, hibridación de ácidos nucleicos, PCR y mutagénesis dirigida, expresión heteróloga de genes para proteínas recombinantes. - Adquirir la capacidad de aplicación de técnicas básicas en diferentes muestras biológicas. - Introducir al alumno en el análisis funcional de genomas mediante matrices de DNA. Contenidos del curso Módulo 1: Clonación Programa teórico: 1. Qué es la clonación 2. Estrategias de clonación de ADN 2.1. Obtención del ADN Amplificación de fragmentos ADN genómico (PCR) Obtención de fragmentos mediante enzimas de restricción 2.2. Vectores de clonación 2.3. Organismos hospedadores 2.4. Métodos de transformación 2.5. Selección de las células transformadas 3. Aplicaciones de la clonación Programa práctico: - Clonación de un fragmento de ADN genómico de Fusarium oxysporum en Escherichia coli. 1. Diseño de oligonucleótidos 2. Amplificación mediante PCR del fragmento de interés 3. Ligación del fragmento en el vector pGEM-T 4. Transformación de Escherichia coli 22 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 5. Selección y análisis de los transformantes a) Amplificación del fragmento mediante PCR de colonia b) Análisis de los tamaños de los plásmidos recombinantes mediante electroforesis en gel de agarosa. Módulo 2: Hibridación de ácidos nucleicos Programa teórico: 1. Fundamentos de la hibridación de ácidos nucleicos 2. Tipos de técnicas de hibridación 3. Etapas y factores que afectan a la hibridación 4. Ventajas e inconvenientes de los diferentes tipos de membranas y de marcaje de la sonda 5. Kits comerciales Programa práctico: - Realización de un Southern blot 1. Fraccionamiento de una muestra de DNA mediante electroforesis en gel de agarosa. 2. Transferencia de la muestra de DNA a un filtro de nailon 3. Preparación de una sonda de DNA marcada con digoxigenina 4. Proceso de hibridación, lavados y detección de la señal Módulo 3: PCR y mutagénesis dirigida Programa teórico: 1. Fundamentos de la PCR 1.1. Introducción y consideraciones generales 1.2. Características de las DNA polimerasas termoestables 1.3. Características de los oligonucleótidos cebadores 1.4. Tipos de PCR: PCR convencional, PCR inversa, RACE-PCR, PCR en tiempo real, PCR in situ, otros tipos de PCR 1.5. Aplicaciones de la PCR 2. Mutagénesis dirigida 2.1. Introducción y consideraciones generales 2.2. Tipos de mutagénesis 2.3. Descripción de algunos sistemas comerciales de mutagénesis dirigida 2.3.1. QuikChange™ (Stratagene) 2.3.2. ExSite™ (Stratagene) 2.3.3. Chamaleon™ (Stratagene) 2.3.4. Code20™ (Biolabs) 2.3.5. Sculptor™ (Amersham) 2.3.6.Altered Sites® (Promega) 2.3.7.Otros sistemas Programa práctico: - Utilización de la PCR y el DNA total de la bacteria alcalófila degradadora de cianuro y de cianato Pseudomonas pseudoalgaligenes CECT5344 junto con los cebadores cynLF9 y cynLR5 para introducir los sitios de corte para las enzimas de 23 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 restricción PstI y NotI en el gen cynB que codifica el componente transmembrana de un transportador de tipo ABC posiblemente de cianato. Módulo 4: Expresión de proteínas Programa teórico: 1. Introducción: Conceptos de expresión homóloga y heteróloga 2. Técnicas de análisis transcripcional 2.1. Identificación de promotores y de secuencias reguladoras de unión DNAproteína. Experimentos de huella y de retardo de la movilidad en gel 2.2. Identificación del extremo 5’ del transcrito. Experimentos de extensión de cebadores y de digestión con nucleasa S1 2.3. Técnicas de identificación y cuantificación de transcritos 2.4. Fusiones transcripcionales y traduccionales de genes informadores 3. Expresión heteróloga de proteínas 3.1. Objetivos y problemática de la expresión de genes clonados 3.2. Principales sistemas de expresión 3.3. Tipos de expresión (proteínas de fusión, de secreción, etc.) 3.4. Vectores de expresión y vectores lanzaderas 3.5. Factores que afectan a la expresión y estrategias para su optimización 3.6. Expresión y purificación de proteínas marcadas (6xHis, etc.) 3.7. Descripción de algunos vectores y sistemas comerciales de expresión (pET, pQE, pMAL, pGEX, etc.) 3.8. Cuantificación de la expresión 4. Análisis funcional de los genomas: genómica y proteómica 4.1. Técnicas de análisis genómico de la expresión génica 4.2. Identificación de proteínas y modificaciones post-transcripcionales 4.3. Interacciones proteína-proteína 4.4. Bioensayos Programa práctico: - Análisis de la expresión en E. coli de una proteína recombinante. 1. Utilización de una estirpe de E. coli previamente transformada con una construcción que permite sobreexpresar la nitrorreductasa NprA de la bacteria Rhodobacter capsulatus fusionada a una secuencia marcadora de seis histidinas en el extremo N-terminal. 2. Inducción de la expresión con IPTG y detección de la proteína recombínate mediante Western-blot con anticuerpos frente a las oligohistidinas. Metodología de enseñanza-aprendizaje Al comenzar la asignatura se entregará a cada alumno en CD y/o en forma de libro todo el material necesario (explicación y planificación del curso, bibliografía, contenido de explicaciones teóricas, protocolos prácticos, etc). Esta información estará disponible en el aula virtual de la UCO (http://www3.uco.es/moodle). Clases Teóricas: se impartirá en el aula mediante clases magistrales en las que se desarrollarán los diferentes temas, profundizando en los conceptos básicos para la realización posterior de las prácticas correspondientes. 24 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Los alumnos completarán estas clases con la consulta en la bibliografía recomendada para cada tema en particular. Clases prácticas: se impartirán en grupos reducidos de no más de 10 alumnos /grupo en los laboratorios de prácticas de tercer ciclo del Departamento de Bioquímica y Biología Molecular y del Departamento de Genética de la UCO. Las prácticas de uno de los grupos del módulo 2 se impartirán preferentemente en inglés. Criterios de evaluación La participación y el interés en el curso se valorarán de 0 a 2 puntos. La realización de cuestiones tanto teóricas como prácticas relacionadas con cada uno de los módulos del curso se valorará de 0 a 2 puntos. El examen se valorará de 0 a 6 puntos. Si no se supera la asignatura en la convocatoria de junio se realizará un examen en septiembre con preguntas de teoría (de 0 a 6 puntos) y de prácticas (de 0 a 4 puntos). Bibliografía básica Al comenzar la docencia de la asignatura se entregará la bibliografía específica para cada módulo. A continuación se suministra una relación bibliográfica de carácter general para toda la asignatura. 1. Sambrook J y Russell DW (2001). Molecular Cloning: A Laboratory Manual, 3rd ed, Vols 1-3. Cold Spring Harbor Laboratory Press. 2. Glick BR y Pasternak JJ (2003). Molecular Biotechnology. Principles and Applications of Recombinant DNA. 3rd ed, ASM Press. 3. Ausubel FM, Brent R, Kingston RE, Moore DD, Seidman JG, Smith JA y Struhl K (2000). Current Protocols in Molecular Biology, Vols 1-4. John Wiley & Sons. 4. Newton CR y Graham A (1997). PCR. 2nd ed. Bios. Sci. Publ., SpringerVerlag. 5. Tait RC (1997). An Introduction to Molecular Biology. Horizon Scientific Press. 6. Glover DM y Hames BD (1995). DNA Cloning: A Practical Approach, Vols. 1-4. IRL Press. 7. Brown TA (1992). Gene Cloning: An Introduction, 2nd ed. Chapman & Hall. 8. Old RW y Primrose SB (1989). Principles of Gene Manipulation. An Introduction to Genetic Engineering, 4th ed. Blackwell Scientific Publications. 9. Winnacker, E-L (1987). From Genes to Clones. Introduction to Gene Technology. VCH Publishers. 10. Wu R, Grossman L y Moldave K, eds. (1989). Recombinant DNA Methodology. Academic Press. 25 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 ANEXO I: Distribución ECTS de la Asignatura TÉCNICAS BÁSICAS DEL DNA RECOMBINANTE a b ( ) 1 ECTS = 25 horas trabajo. ( ) Estimación de estudio personal del alumno durante el curso 12 (trimestral) o 36 (anual) semanas: 1,5 horas de estudio por cada hora de teoría y 0,75 horas de estudio por cada hora de prácticas. (c)Las tutorías se encuentran incluidas en el total de Actividades Académicamente Dirigidas. Actividad Actividad Docente Clases en aula Clases en laboratorio o en aula de informática Actividades dirigidas tutoríasc Materia Alumno Presentación de normas, exposición de la teoría, apoyo con audiovisuales, entregar cuestiones para su realización Tomar notas y apuntes, completar copias del material audiovisual, formular preguntas y dudas Prácticas Presentación de normas, explicación de las prácticas, aclaración del protocolo experimental Realizar la práctica, hacer anotaciones, discutir resultados, etc. Preguntas sobre los protocolos, cuestiones prácticas, problemas o ejercicios Cuestiones y tutorías Preparación de cuestiones teóricas y prácticas y discusión de las mismas y otros temas de interés a alumnos Responder las cuestiones. Presentar dudas y participar en la pagina web de la asignatura Se valorará los conocmientos y la respuesta razonada de las cuestiones 20% Es una actividad no evaluable - La asistencia y la participación es fundamental y por ello será evaluada 20% Teoría Actividades dirigidas Seminario Exposición del seminario Asistencia a clases y actividades Teoría, prácticas y seminario Controlar la asistencia a las actividades presenciales del curso Asistir y participar en las clases de teoría y prácticas y al seminario Teoría y prácticas Poner, vigilar y corregir el examen, calificar globalmente al alumno Preparación y realización del examen Procedimiento Peso en la nota final Profesor Realizar cuestiones sobre el seminario en el aula Exámenes Evaluación Preguntas en las que se valorará el conocimiento de la materia, el razonamiento y la capacidad de síntesis. TOTAL CARGA DOCENTE DEL ALUMNO Horas presenciales Horas no presenciales Horas 8h 10,5 hb 18,5 h 22 h 16,5 h 38,5 h 26 h 26 h - 1h ECTS a 60% 1h Las indicadas anteriormente en las clases de teoría, prácticas y actividades dirigidas 1h 15 h 16 h 32 h 68 h 100 h 26 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Curso: METODOLOGÍA DE LA EXPERIMENTACIÓN EN BIOLOGÍA CELULAR Departamento de Biología Celular, Fisiología e Inmunología. CRÉDITOS: 4 ECTS PROFESORES (Indicando el nº de créditos que imparten): 1.- José Antonio González Reyes (1 crédito ECTS). 2.- Mª Isabel Burón Romero (1 crédito ECTS). 3.- Mª del Mar Malagón Poyato (1 crédito ECTS). 4.- José Manuel Villalba Montoro (1 crédito ECTS). OBJETIVOS ESPECÍFICOS DEL CURSO: Aprendizaje teórico y práctico de las metodologías de estudio más utilizadas en los distintos campos de estudio de la Biología, a nivel celular. Se pretende dotar al alumno de conocimientos básicos y recursos para el abordaje técnico en líneas de investigación biológica que utilicen modelos de estudio celulares y/o métodos de análisis del nivel celular y tisular. Se pretende acercar al alumno al manejo de distintos organismos y materiales de trabajo, al planteamiento experimental de problemas concretos y al análisis, discusión y presentación de resultados CONTENIDOS DEL CURSO: Fraccionamiento celular.- Conceptos básicos sobre toma de muestras para fraccionamiento celular. Homogenización de células, órganos y tejidos animales y vegetales. Centrifugación y ultracentrifugación. Centrifugación diferencial y en gradientes. Tipos de gradientes. Estimación de pureza de fracciones: métodos espectrofotométricos, inmunológicos y morfométricos. Solubilización de fracciones de membrana para la obtención de proteínas. Introducción a los detergentes biológicos. Identificación y localización de moléculas y estructuras en células y tejidos animales y vegetales. Técnicas histoquímicas e inmunohistoquímicas. Determinación de la expresión celular de genes por hibridación in situ. Concepto y objetivos. Fijación y preparación de las muestras biológicas. Tipos de sonda y marcado. Condiciones de hibridación. Especificidad y sensibilidad de la hibridación Identificación y localización de moléculas y estructuras a nivel subcelular en muestras animales, vegetales y células en cultivo. Técnicas de microscopía electrónica aplicadas a citoquímica e inmunocitoquímica. 27 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 METODOLOGÍA DE ENSEÑANZA-APRENDIZAJE: Clases teóricas: en aula, con presentaciones virtuales y apoyo de medios audiovisuales y potenciando la participación activa del alumno. estas clase tiene carácter obligatorio. Clases prácticas: en laboratorio, grupos reducidos (no mas de16 alumnos) y con carácter obligatorio. Actividades dirigidas: realización de memorias a partir de la actividad desarrollada en cada sesión práctica. Posibilidad de hacer ejercicios y problemas planteados por el profesor. Aunque esta actividad tiene un carácter optativo, la presentación de las correspondientes memorias es obligatoria. Tutorías: orientación del profesor al alumno, personalizada. Presencial o en espacio virtual de aprendizaje CRITERIOS DE EVALUACIÓN: Para la calificación final se contempla la siguiente distribución de puntuación y método de seguimiento: - 50% Conocimientos adquiridos: examen final. - 25% Asistencia con aprovechamiento: control de presencia. - 25% Memorias de prácticas y actividades relacionadas (espacio virtual). Evaluación del profesor, seguimiento en tutoría y/o espacio web BIBLIOGRAFÍA BÁSICA: Cell Biology: A Laboratory Handbook. JE Celis, N Carter (Editor), K Simons, JV Small, T Hunter, D Shotton. 2ª Edición. Academic Press (New York, USA). 2005. Biological Centrifugation (The Basics). J Graham. BIOS Scientific Publishers Ltd (Oxford, UK). 2001. Centrifugation: A Practical Approach. IRL Press, Oxford. 1984. Isolation of Membranes and Organelles from Plant Cells. Academic Press (San Diego, USA). 1983. Aqueous Two-Phase Systems (Methods in Enzymology). JN Abelson, MI Simon, H Walter and G Johansson. Academic Press Inc. (San Diego, CA, USA). 1994. Subcellular Fractionation: A Practical Approach (Practical Approach Series) J Graham and D Rickwood. Academic Press (New York, USA). 1997. Methods in Enzimology. Vol 182. Guide to protein purification. Academic Press, San Diego, 1990. Electron Microscopy: Methods and Protocols (Methods in Molecular Biology). 2ª Edición. J Kuo. Humana Press Inc (New York, USA). 2007. 28 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Microscopy, Immunohistochemistry, and Antigen Retrieval Methods: For Light and Electron Microscopy. MA Hayat. Kluwer Academic/Plenum Publishers (New York, USA). 2002. Introduction to Immunocytochemistry. JM Polak. BIOS Scientific Publishers Ltd (Oxford, UK). 2003. Immunocytochemistry and In Situ Hybridization in the Biomedical Sciences. JE Beesley. Birkhäuser Boston (New York, USA). 2001. Practical in Situ Hybridization. T Schwarchzacher and P Heslop-Harrison. BIOS Scientific Publishers Ltd (Oxford, UK). 2000. In Situ Hybridization Protocols (Methods in Molecular Biology). IA Darby and TD Hewitson. Humana Press Inc. (Totowa, New Jersey, USA). 2006. In situ Hybridization. Principles and Practice. J.M. Polak y J.O.D. McGee, Oxford Science Publications. 1990. In situ hybridization Histochemistry. M.F. Cheselet. CRC Press. 1990. ALGUNOS RECURSOS “ON-LINE” http://homepages.gac.edu/~cellab/chpts/chpt3/intro3.html http://fig.cox.miami.edu/~cmallery/255/255tech/255techniques.htm http://csm.colostate-pueblo.edu/biology/dcaprio/412L/CFbreak.html http://www.sumanasinc.com/webcontent/animations/content/cellfractionation.html http://swehsc.pharmacy.arizona.edu/exppath/micro/em.html http://www.udel.edu/biology/Wags/b617/b617.htm http://www.protocol-online.org/prot/Image_Techniques/Microscopy/Electron_Microscopy/ http://www.mwrn.com/microscopy/electron/microscope.aspx http://www.ub.edu/biocel/wbc/tecnicas/me.htm http://www.cytochemistry.net/Cytochem.htm http://www.roche-applied-science.com/PROD_INF/BIOCHEMI/no1_98/p10.pdf http://www.ifom-ieo-campus.it/RESEARCH/Mol_path/Final_Protocols/ISH_protocol.pdf http://www.genedetect.com/insitu.htm 29 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Distribución de horas presenciales y no presenciales. Criterios de evaluación: Distribución ECTS de la Asignatura METODOLOGÍA DE LA EXPERIMENTACIÓN EN BIOLOGÍA CELULAR a b ( ) 1 ECTS = 25 horas trabajo. ( ) Estimación de estudio personal del alumno durante el curso 12 (trimestral) o 36 (anual) semanas: 1,5 horas de estudio por cada hora de teoría y 0,75 horas de estudio por cada hora de prácticas. Actividad Actividad Docente Clases en aula Evaluación Alumno Procedimiento Peso en la nota final Horas presenciales Horas no b presenciales Horas Profesor Exposición de la teoría con apoyo de medios audiovisuales Planteamiento de cuestiones. Toma de apuntes, planteamiento de cuestiones; respuesta a cuestiones planteadas por el profesor Tipo de cuestiones planteadas por el alumno. Respuesta a cuestiones planteadas por el profesor. Se valorará razonamiento y capacidad de síntesis 50 % 6h 9h 15 h Presentación de normas. Explicación de las prácticas Realización de experimentos, recogida de datos Evaluación continuada, cuaderno de datos y memorias de prácticas. Se valorará la actitud así como la capacidad de análisis y síntesis 25 % 24 h (6 sesiones de 4 h) 10 h 34 h Análisis de resultados Explicación de métodos para el análisis de datos experimentales Análisis de resultados; de conclusiones; elaboración de una memoria para cada práctica Evaluación continuada; memorias de prácticas. Se valorará el razonamiento y las capacidades de análisis y comunicación. 25 % 32 h 32 h Teoría y prácticas Orienta y resuelve dudas Preparar cuestiones y solicitar orientación personalizada Es una actividad no evaluable, sólo de comunicación - 3h 3h Confeccionar, vigilar y corregir el exámen. Evaluación Preparación y realización del examen Materia Teoría Prácticas de laboratorio Clases en laboratorio Tutorías Examen TOTAL CARGA DOCENTE DEL ALUMNO ECTS a - 2h 14 h 16 h 100% 32 h 68 h 100 h 30 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 3. ITINERARIO 1: BIOTECNOLOGÍA VEGETAL Y AMBIENTAL 31 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Asignatura: TÉCNICAS AVANZADAS EN GENÓMICA FUNCIONAL Total de créditos ECTS: 4 Tipo de asignatura: Elegible. Profesorado: José Luis Caballero Repullo y/o Enriqueta Moyano Cañete (Modulo1: Genotecas substractivas, 2 ECTS). Mª José Prieto Álamo (Coordinadora) y/o Juan Jurado Carpio (Modulo 2: Microarrays de DNA, 1 ECTS) Carmen Pueyo de la Cuesta y/o Nieves Abril Díaz (Módulo 3: Perfiles transcripcionales por RT-PCR en tiempo real, 1ECTS) La impartición de una docencia de calidad en una asignatura de alta experimentalidad como es Técnicas Avanzadas en Genómica Funcional, exige que el número máximo de alumnos por grupo sea 10. Si el número de matriculados es superior a diez, será necesaria la distribución de alumnos en varios grupos y consecuentemente, la participación de más profesores. Por esta razón, se han incluido más de cuatro profesores, cuya participación estará vinculada a las necesidades docentes en función del número de alumnos. Profesores invitados: Dra. Mercedes Cousinou Rodríguez, Servicio de Genómica del SCAI, Universidad de Córdoba. Dr. José Gadea Vacas, Laboratorio de Genómica, Instituto de Biología Molecular y Celular de Plantas, Universidad Politécnica de Valencia-CSIC. Objetivos del curso Generales - Conocer los fundamentos teóricos y prácticos de la Genómica funcional. - Entender las distintas aproximaciones de la Genómica funcional al estudio de los procesos biológicos. - Manejo de técnicas y equipos en estudios genómicos de muestras biológicas. - Familiarizarse con el procesamiento y análisis de los resultados obtenidos en los diversos experimentos. - Percibir el alcance de las aplicaciones biotecnológicas de las metodologías aprendidas. Específicos Módulo 1. Genotecas substractivas - Conocer los procedimientos más empleados y novedosos en la construcción de genotecas representativas de cDNA - Comprender la importancia del enriquecimiento de éstas en determinados genes mediante hibridación substractiva (clonación substractiva). 32 Master en Biotecnología, Molecular Celular y Genética - - Curso 2009-2010 Diferenciar entre el escrutinio convencional de una genoteca mediante sondas conocidas y los escrutinios diferenciales (sondas no conocidas), con el fin de aislar genes específicos relacionados con un determinado proceso. Conocer cómo estos procedimientos hacen uso de otras técnicas, como la PCR, que mejoran e incrementan notablemente los resultados. Conocer las etapas relevantes de estas técnicas, con especial énfasis en sus ventajas e inconvenientes Conocer qué número mínimo de clones debe poseer una genoteca y comprender las diferencias entre título primario de una genoteca y título de una genoteca amplificada. Módulo 2. Microarrays de DNA - Conocer los principales aspectos teóricos y prácticos de los experimentos que conllevan el uso de microarrays de DNA. - Conocer diferentes técnicas de validación de los resultados obtenidos con microarrays. - Conocer nuevas aplicaciones de los microarrays: hibridación genómica comparativa (CGH), metilación del DNA (islas CpG), interacciones DNAproteína, procesamientos alternativos de transcritos primarios, análisis de miRNA. - Comprender las ventajas e inconvenientes de estos análisis y su potencialidad. Módulo 3. Perfiles transcripcionales por RT-PCR en tiempo real - Comprender los fundamentos de la RT-PCR cuantitativa. - Comprender la importancia del diseño de cebadores para obtener la máxima eficiencia de amplificación de las secuencias diana. - Conocer la importancia de contrastar la bondad de los estándares elegidos en la cuantificación relativa por RT-PCR. - Diferenciar entre cuantificación relativa y cuantificación absoluta por RTPCR en tiempo real. Conocer las ventajas e inconvenientes de la cuantificación relativa y de la cuantificación absoluta, y en sus posibles aplicaciones según el tipo de estudio. Competencias a adquirir: Capacidad de diseñar experimentos aplicando cada una de las metodologías descritas. Capacidad de valorar las diferentes estrategias para el aislamiento de genes mediante construcción, enriquecimiento y escrutinio de genotecas con objeto de economizar tiempo y costes de ejecución en proyectos relacionados con el tema. Desarrollo de la capacidad de manipulación práctica de fagos recombinantes, siembra y titulación de las genotecas. Conocimiento de todos los aspectos experimentales que conllevan la metodología de microarrays de DNA (marcaje, hibridación, escaneado, extracción y tratamiento de los datos, análisis estadístico, herramientas 33 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 bioinformáticas, etc.) así como de las ventajas y limitaciones de la tecnología de microarrays. Capacidad para llevar a cabo experimentos de análisis masivo de expresión génica diferencial en un conjunto de muestras, utilizando la metodología de microarrays de DNA. Conocimiento de todos los aspectos experimentales que conlleva la cuantificación de transcritos (aislamiento de RNA de calidad, síntesis de cDNA, diseño de cebadores que amplifiquen las secuencias diana con máxima eficiencia, elección de estándares, etc.) Capacidad para utilizar la RT-PCR para cuantificación absoluta o relativa de la expresión génica, aplicando una u otra según convenga al tipo de estudio concreto que se quiera realizar. Desarrollo de las habilidades bioinformáticas necesarias para conocer y utilizar las múltiples bases de datos de acceso libre en internet de secuencias de DNA, Gene Ontology, etc. Programa de la asignatura: Módulo 1. Genotecas substractivas (2 ECTS; José Luis Caballero Repullo y/o Enriqueta Moyano Cañete). 1. Introducción a la obtención y uso de genotecas substractivas: 1.1. Procedimientos más empleados y novedosos en la construcción de genotecas representativas de cDNA. 1.2. Enriquecimiento en determinados genes mediante hibridación substractiva (clonación substractiva) para su escrutinio convencional mediante sondas conocidas o mediante escrutinios diferenciales (sondas no conocidas), con el fin de aislar genes específicos relacionados con un determinado proceso. 2. Escrutinio de una genoteca construida en fago lambda: 2.1. Titulación de la genoteca mediante siembra en placas. 2.2. Obtención de membranas réplica por duplicado de los clones/halos de lisis sembrados/as. 2.3. Hibridación con sondas de ADNc a partir de ARNm de muestras procedentes de condiciones biológicas diferentes. Módulo 2. Microarrays de DNA (1 ECTS; Mª José Prieto Álamo y/o Juan Jurado Carpio). 1. Introducción a la tecnología de microarrays de DNA y sus aplicaciones: 1.1. Conceptos básicos y recomendaciones generales en el manejo de microarrays. 1.2. Microarrays de expresión. 1.3. Otras aplicaciones de la tecnología de microarrays: hibridación genómica comparativa, metilación del DNA, interacciones DNAproteína, procesamientos alternativos de transcritos primarios, análisis de miRNA. 34 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 2. La tecnología de microarrays aplicada al estudio de la expresión génica: 2.1. Preparación de las muestras: marcaje, purificación y control espectrofotométrico de la calidad del marcaje. 2.2. Hibridación y escaneado de microarrays. Extracción de datos: GenePix. 2.3. Visión general del análisis de datos de un experimento con microarrays. Obtención de genes diferencialmente expresados. 2.4. Análisis funcional e interpretación biológica de los resultados. Módulo 3. Perfiles transcripcionales por RT-PCR en tiempo real. (1 ECTS; Carmen Pueyo de la Cuesta y/o Nieves Abril Díaz). 1. Introducción a la cuantificación por RT-PCR: 1.1. Ventajas e inconvenientes de distintos métodos para cuantificar transcritos. 1.2. Métodos para el aislamiento de RNA y síntesis de cDNA. 1.3. Etapas en la amplificación por PCR. 1.4. Parámetros y estrategias para el diseño de cebadores. 1.5. Naturaleza exponencial de la amplificación por PCR y eficiencia de amplificación. 1.6. Ventajas e inconvenientes de los estándares internos vs externos, y de las cuantificaciones relativas vs absolutas. 1.7. Medidas por RT-PCR a tiempo final vs medidas por RT-PCR en tiempo real. 1.8. Concepto de Ct y recta de calibración. 1.9. Método de Pfaffl. 2. Cuantificación absoluta y relativa de transcritos por RT-PCR en tiempo real: 2.1. Determinación de la eficiencia de amplificación de la/s pareja/s de cebadores utilizados en la cuantificación de transcrito/s de interés. 2.2. Determinación de los valores de Ct en las muestras objeto de análisis. 2.3. Cuantificaciones relativas mediante el método de Pfaffl. 2.4. Obtención de la recta de calibración. 2.5. Cuantificaciones absolutas por extrapolación en la recta de calibración. 2.6. Comparación de las medidas expresadas en diferencias en número de moléculas de transcritos vs número de veces de variación. Metodología de enseñanza-aprendizaje Al comenzar la asignatura cada alumno dispondrá de todo el material necesario (planificación del curso, bibliografía, contenido de las explicaciones de los fundamentos teóricos, protocolos prácticos, material necesario para la preparación de los seminarios, etc.) en el aula virtual de la UCO (http://www3.uco.es/moodle/), así como en formato CD y/o libro. Así mismo, se suministra una bibliografía general básica y material docente para que los alumnos que lo deseen puedan mejorar sus conocimientos previos sobre los principales métodos y técnicas utilizados en Genómica funcional, tanto de aquellas objeto de estudio en este curso como de otras que no pueden ser abordadas por la limitada duración del mismo. 35 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 La metodología docente incluirá los siguientes aspectos fundamentales: Clases prácticas, que se impartirán en grupos reducidos (no más de 10 alumnos/grupo) en: 1. Aula, para la explicación inicial de los fundamentos teóricos que se impartirán haciendo hincapié en los conceptos esenciales ilustrados mediante la descripción de casos modelo. También se llevará a cabo en el aula la exposición y discusión en grupo de los resultados. Los estudiantes completarán sus conocimientos con la consulta de la bibliografía recomendada para cada módulo. 2. Laboratorio de prácticas de tercer ciclo del Departamento de Bioquímica y Biología Molecular, en el edificio Severo Ochoa. 3. Sala de informática para manejo de software específico y simulaciones. 4. Servicio de Genómica del SCAI, UCO. Actividades dirigidas, que consistirán en la realización de un seminario bajo la supervisión de un profesor (en grupos de 2 alumnos o individualmente). Las exposiciones de estos seminarios durarán unos 20-30 min y se realizarán al finalizar la docencia de la asignatura. Los temas de los seminarios (trabajos científicos o resolución de problemas) se propondrán al comenzar el curso. Tutorías, donde el profesor orientará al alumno de forma personalizada. Podrán ser presenciales o a través del espacio virtual de aprendizaje. Estudio y trabajo personal de cada alumno para la asimilación de los conceptos aprendidos. Se evaluará mediante la realización de un examen final donde se demostrarán los conocimientos adquiridos. Criterios de evaluación Debido al carácter práctico de este curso, la asistencia a las actividades presenciales se considera un requisito básico para la consecución de los objetivos propuestos. De este modo, la asistencia y participación en el curso se valorará de 0 a 2 puntos (0, si se asiste a menos del 80% de las horas presenciales; 1, si la asistencia está entre el 80 y el 90%; 2, si se asiste a más del 90%). El examen final se valorará de 0 a 6 puntos. La realización y exposición del seminario se valorará de 0 a 2 puntos. Para superar la asignatura será necesario obtener al menos 3 puntos en el examen final, 1 punto en los seminarios y una calificación global de 5. Si no se supera la asignatura en la convocatoria de junio se realizará un examen en Septiembre que se valorará de 0 a 10 puntos, donde será necesario obtener al menos 5 puntos para aprobar. Bibliografía básica A continuación se suministra una relación bibliográfica de carácter general para toda la asignatura. 1. Ausubel FM, Brent R, Kingston RE, Moore DD, Seidman JG, Smith JA y Struhl K (2000). Current Protocols in Molecular Biology, Vols 1-4. John Wiley & Sons. 36 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 2. Botwell D and Sambrook J (Ed). (2003) DNA Microarrays (A Molecular Cloning Manual), Cold Spring Harbor Laboratory Press. 3. Caballero, J.L., Valpuesta Fernández, V. y Muñoz Blanco, J. (2001) Introducción a la Biotecnología Vegetal: Técnicas y Aplicaciones. Capítulo: Construcción de sondas y genotecas sustractivas. Escrutinios diferenciales. ISBN. 8479593806. Editorial: CajaSur Córdoba (SPAIN) 4. Churchill GA (2002). Fundamentals of experimental design for cDNA microarrays. Nature Genetics, 32:490-495. 5. Cui X, Churchill GA (2003). Statistical tests for differential expression in cDNA microarray experiments. Genome Biology, 4:210.1-210.10. 6. Duggan DJ, Bittner M, Chen Y, Meltzer P, Trent JM (1999). Expression profiling using cDNA microarrays. Nature Genetics, 21:10-14. 7. Freeman WM, Robertson DJ, Vrana KE (2000). Fundamentals of DNA hybridization arrays for gene expression analysis. Biotechniques, 29:1042.1055. 8. Glick BR y Pasternak JJ (2003). Molecular Biotechnology. Principles and Applications of Recombinant DNA. 3rd ed, ASM Press. 9. Houghton SG, Cockerill FR (2006). Real-time PCR: overview and applications. Surgery, 139:1-5. 10. Kaltenboeck, B, Wang Ch (2005). Advances in real-time PCR: application to clinical laboratory diagnostics. Adv Clin Chem, 40:219-59. 11. Kubista M, Andrade JM, Bengtssona M, Forootand A, Jonáke J, Linda K, Sindelkae R, Sjöbacka R, Sjögreend B, Strömboma L, Ståhlberga A, Zorica N (2006). The real-time polymerase chain reaction. Molecular Aspects of Medicine, 27:95-125 12. Pfaffl, M W (2001). A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Res, 29:2002-2007 13. Sambrook J y Russell DW (2001). Molecular Cloning: A Laboratory Manual, 3rd ed, Vols 1-3. Cold Spring Harbor Laboratory Press. Al comenzar la docencia de la asignatura se proporcionará la bibliografía específica con el material didáctico de cada módulo. 37 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 ANEXO I: Distribución ECTS de la Asignatura TÉCNICAS AVANZADAS EN GENÓMICA FUNCIONAL (a) 1 ECTS = 25 horas trabajo. (b) Estimación de estudio personal del alumno durante el curso 12 (trimestral) o 36 (anual) semanas: 1,5 horas de estudio por cada hora de teoría y 0,75 horas de estudio por cada hora de prácticas. (c)Las tutorías se encuentran incluidas en el total de Actividades Académicamente Dirigidas. Actividad Actividad Docente Clases en laboratorio o en aula de informática Materia Todo el contenido del programa Actividades dirigidas Seminarios Tutorias Todo el contenido del programa Asistencia a clases y actividades dirigidas Exámenes Profesor Alumno Preparación del material. Explicación de los fundamentos Toma teóricos de la apuntes. práctica y de Formula los protocolos preguntas y experimentales. dudas. Explicación del Realiza los uso de los experimentos. programas de Recoge los ordenador datos, realiza, adecuados a cálculos, y cada técnica análiza los impartida. resultados. Planteamiento de cuestiones y aclaración de dudas. Supervisión continua. Distribuye los Consulta de trabajos, dudas, recomienda exposición bibliografía, del seminario orienta, en el aula, resuelve y etc. aclara dudas Trabajo colaborativo alumnoprofesor. Discusión de los contenidos del programa de forma global Resolución de dudas y participación en el espacio virtual de aprendizaje Evaluación Procedimiento Peso en la nota final Evaluación continuada (asistencia, participación, actitud). Resultados experimentales. Cuestiones prácticas, problemas o ejercicios. Examen donde se valorará el conocimiento de la materia, el razonamiento y la capacidad de síntesis. Se valorará contenido, exposición, presentación y preparación del tema No evaluable La asistencia participativa no se considera obligatoria pero es fundamental y por ello será evaluada Horas presenciales Horas no presenciales Horas 60% 26 h 30 h 56 h 20% 4h 15 h 19 h ECTS a - Prácticas y seminarios Controla la asistencia y la actitud en las actividades presenciales del curso Asiste con actitud positiva a las clases y seminarios Prácticas Pone, atiende y corrige el examen. Califica globalmente al alumno Preparación y realización del examen 2h 23 h 25 h TOTAL CARGA DOCENTE DEL ALUMNO 32 68h 100 h 20% Las indicadas anteriormente en las clases y actividades dirigidas 38 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Asignatura: BIOTECNOLOGÍA AMBIENTAL Total de créditos ECTS: 4 Tipo de asignatura: Elegible. Profesores participantes: Módulos 1 (Introducción), 3 (Biomarcadores convencionales de contaminación) y 4 (Nuevos biomarcadores) - Prof. Dr. Juan López Barea, Catedrático de Bioquímica y Biología Molecular, Departamento de Bioquímica y Biología Molecular, UCO. 1 ECTS. Módulo 2. Biotecnología microbiana. - Prof. Dr. Francisco Castillo Rodríguez, Catedrático de Bioquímica y Biología Molecular, Departamento de Bioquímica y Biología Molecular, UCO. 1 ECTS. Módulo 5. Integración de tecnologías ómicas en la monitorización de ecosistemas terrestres: el ratón moruno (Mus spretus) como bioindicador. - Profa. Dra. Carmen Pueyo de la Cuesta, Catedrática de Bioquímica y Biología Molecular, Departamento de Bioquímica y Biología Molecular, UCO. 1 ECTS. Parte experimental. - Dr. José Alhama Carmona, Profesor Contratado Doctor, Departamento de Bioquímica y Biología Molecular, UCO. 1 ECTS. En esta parte colaboran dos expertos con las siguientes dedicaciones: - Dr. Victor Luque Almagro, Doctor contratado por el Programa Juan de la Cierva, Departamento de Bioquímica y Biología Molecular, UCO. 0,25 ECTS. - Dra. Julia Ruiz Laguna, Doctora contratada con cargo a proyecto, Departamento de Bioquímica y Biología Molecular, UCO. 0,25 ECTS. Profesores colaboradores invitados: - - - Dr. Rafael Blasco Pla, Profesor Titular de Bioquímica y Biología Molecular, Universidad de Extremadura, UNEX. 0,25 ECTS Prof. Dr. José Luis Gómez Ariza, Catedrático de Química Analítica, Universidad de Huelva, UHU. 0,25 ECTS Objetivos: Conocer los principales contaminantes ambientales, su absorción y distribución y sus efectos sobre los seres vivos de distintos niveles, desde microorganismos a animales superiores. Comprender el uso de microorganismos para la bioeliminación de contaminantes, centrándose en algunos ejemplos en los que tienen experiencia los profesores participantes. 39 Master en Biotecnología, Molecular Celular y Genética - - - Curso 2009-2010 Familiarizarse con los principales biomarcadores moleculares usados de forma convencional para la monitorización de la contaminación. Análisis masivo de datos en organismos expuestos a contaminantes y seguimiento integral de los ecosistemas mediante metodologías derivadas de la nueva Biología de Sistemas Descripción global de ecosistemas, búsqueda de nuevos biomarcadores no sesgados y comprensión de los mecanismos moleculares de toxicidad por técnicas “ómicas”: metagenómica, metaproteómica, proteómica, transcriptómica y metalómica en estudios ambientales Integración de proteómica, transcriptómica y metalómica en el estudio de ecosistemas de la Ría de Huelva y el Entorno de Doñana con distintos niveles de contaminación. Programa teórico (3 ECTS): Módulo 1. Introducción (aprox. 1,5 horas) 1. Contaminantes (hidrocarburos lineales, PAHs, PCBs, plaguicidas, metales). 2. Disposición de xenobióticos. 3. Biotransformación en animales (reacciones de Fases I y II). 4. Modificación de la biotransformación (factores biológicos y ambientales, regulación génica). 5. Estrés oxidativo (especies reactivas de oxígeno, daños en biomoléculas). 6. Defensas antioxidativas y su regulación. Módulo 2. Biotecnología microbiana (duración aprox. 7 horas) 1. El equilibrio de los elementos biogenésicos en la biosfera: alteraciones de los ciclos del carbono, nitrógeno y azufre-hierro. 2. Aplicaciones de la metagenómica al estudio de los microorganismos en su medio ambiente. 3. Bioeliminación de contaminantes. Rutas metabólicas biodegradativas. Evolución natural y artificial de rutas metabólicas: ingeniería metabólica. 4. Aplicaciones prácticas de la biodegradación: eliminación de cianuros de origen industrial por bacterias. Módulo 3. Biomarcadores convencionales de contaminación (aprox. 3,5 horas) 1. Enzimas biotransformadoras (cit P450, GSH transferasas) y antioxidativas. 2. Daños oxidativos en biomoléculas (oxidación y rotura del DNA; estado redox del glutatión; peroxidación lipídica; oxidación de proteínas; proteólisis). 3. Biomarcadores específicos (metalotioneínas, esterasas). 4. Aplicación al estudio del litoral andaluz, el accidente de Aznalcóllar y el Entorno de Doñana. 5. Limitaciones de los biomarcadores convencionales. Módulo 4. Nuevos biomarcadores de contaminación (aprox. 4 horas) 1. Análisis masivo de los efectos biológicos de los contaminantes. 40 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 2. Estudios iniciales de proteómica ambiental: señales de expresión protéica: identificación de proteínas en organismos modelo expuestos a contaminantes. 3. Las especies no modelo exigen la secuenciación de novo: exposición de bivalvos, crustáceos y peces a contaminantes; estudios en Doñana con crustáceos, bivalvos y ratones silvestres. 4. Causas de alteración protéica (síntesis/proteólisis, carbonilos, GSH, NO2). 5. Presente y futuro de la metaproteómica: aplicación a la Ría de Huelva. Módulo 5. Integración de tecnologías “ómicas” en la monitorización de ecosistemas terrestres: el ratón moruno (Mus spretus) como bioindicador (aprox. 8 horas) 1. Interés de M. spretus en la monitorización de ecosistemas: especie de vida libre próxima al organismo modelo mejor conocido (Mus musculus). 2. El Estero de Domingo Rubio (EDR) como laboratorio “al aire libre”. 3. Transcriptómica: perfiles de expresión transcripcional por qRT-PCR y micrpochips hete-rólogos de DNA. Integración de resultados proteómicos y transcriptómicos en el EDR. 4. Contaminantes metálicos y su especiación; metalobiomoléculas; metaloproteínas; metalómica y sus aplicaciones ambientales. Integración de la proteómica, transcriptómica y metalómica en EDR y Entorno de Doñana. Programa Práctico (1 ECTS): Módulo 1. Estudio del regulador CynF del operón cyn que codifica la cianasa de Pseudomonas pseudoalcaligenes (aprox. 2,5 horas) 1. Actividad cianasa en la estirpe silvestre y en un mutante cynF cultivadas en distintas fuentes de N. Módulo 2. Cuantificación por PCR en tiempo real del nº de transcritos de los genes GstM1, GstA2 y cyp2E1 en M. spretus (aprox. 2,5 horas) 2. Efecto de la zona, del sexo y del tejido en la expresión génica. Módulo 3 Herramientas usadas en el análisis de resultados de Proteómica Ambiental (aprox. 6 horas) 3. Digitalización de los geles bidimensionales. Análisis de imagen de los geles 2-DE(software PDQuest, DeCyder, MelanieSIB, Proteomweaver). 4. Algoritmos empleados en la secuenciación de novo (Lutefisk, Peaks, etc.). 5. Identificación de proteínas mediante BLAST en distintas bases de datos y búsqueda de estructura y función (UNIPROT, INTERPRO, EXPASSY, SWISS MODEL, INGENUITY PATHWAY ANALYSIS) 6. Análisis de clusters (GENESIS) 41 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Bibliografía Libros de texto: Burdon RH (1999). Genes and the Environment. Taylor & Francis, London, 243 pp. Castillo Rodríguez F, et al (2005). Biotecnología Ambiental. Editorial Tébar, Madrid, 614 pp. Simpson RJ (2003). Proteins and Proteomics. Cold Spring Harbour Laboratory Press, New York, 926 pp. Timbrell J (2002). Introduction to Toxicology (3rd ed). Taylor & Francis, London, 394 pp. Walker CH, et al (2001). Principles of Ecotoxicology (2nd ed). Taylor & Francis, London, 309 pp. Artículos: Alhama J, et al (2006). J Chromatogr A, 1107: 52-58. Bonilla-Valverde D, et al (2004). Toxicology, 197: 123-138. Dowling V, Sheehan D (2006). Proteomics, 6: 5597-5604. Fraser-Liggett CM (2005). Genome Research, 15: 1603-1610. Funes V, et al (2006). Environ Pollut, 139: 214-223. Gómez-Ariza JL, et al (2007). Anal Bioanal Chem, 388: 1295-1302. Gonzalez-Fernandez M, et al (2008). Anal Bioanal Chem, 390: 17-28. Huertas MJ, et al (2006). Biochem Soc Transact, 34: 152-155. Karl DM (2007). Nature Reviews Microbiology, 5: 759-769. López-Barea J, Gómez-Ariza JL (2006). Proteomics, 6 (Suppl 1): S51-S62 (2006). Luque-Almagro V, et al (2005). Appl Environ Microbiol, 71: 940-947. Luque-Almagro V, et al (2005). Biochem Soc Transact, 33: 168-169. Luque-Almagro, V.M., et al (2007) Environ. Microbiol. 9, 1541-1549 Monsinjon T, Knigge T (2007). Proteomics, 7: 2297-3009. Montes-Nieto R, et al (2007). Proteomics, 7: 4376-4387. Pérez-Reinado et al (2008) Environ. Microbiol. (en prensa) Roldán MD et al (2008) FEMS Microbiol Rev (en prensa) Rodríguez-Ortega MJ, et al (2003). Proteomics, 3: 1535-1543. Romero-Ruíz A, et al (2003). Environ Toxicol Chem, 22: 92-100. Romero-Ruiz A, et al (2006). Proteomics, 6 (Suppl 1): S245-S255 (2006). Ruíz-Laguna J, et al (2001). Biomarkers, 6: 146-160. Ruiz-Laguna J, et al (2005). Gene Express, 12: 165-176. Ruiz-Laguna J, et al (2006). Environ Sci Technol, 40: 3646-3652. Vioque-Fernández A, et al (2007). Toxicol Lett, 168: 260-268. Wilmes P, Bond PL (2006). Trends Microbiol, 14: 92-97. 42 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Distribución ECTS de la Asignatura: BIOTECNOLOGÍA AMBIENTAL (a) 1 ECTS = 25 horas trabajo. (b) Estimación de estudio personal del alumno durante el curso 12 (trimestral) o 36 (anual) semanas: 1,5 horas de estudio por cada hora de teoría y 0,75 horas de estudio por cada hora de prácticas. (c)Las tutorías se encuentran incluidas en el total de Actividades Académicamente Dirigidas. Actividad Actividad Docente Clases en aula Clases en laboratorio Actividades dirigidas Exámenes Evaluación Profesor Alumno Procedimiento Peso en la nota final Teoría Exposición de la Teoría. Apoyo con audiovisuales Toma apuntes. Formula preguntas y dudas Eval contin asist, particip., actitud). Examen preg. cortas (razonam, cap. síntesis) 60% 24 46 70 Prácticas de laboratorio Explicación superv. cont. Manejo softare para las técnicas enseñadas Cuaderno de laboratorio, cálculo y análisis de datos Eval continuada (asistencia, participación, actitud), 30% 8 4 12 Seminarios Orientación, de trabajos a presentar Exposición del seminario. Formulación de preguntas Contenido, exposición, respuesta a preguntas 10% 3 5 8 Teoría Redactar y corregir. Calificar globalmente Preparación y realización 1 9 10 36(36%) 64 (64%) 100(100%) Materia TOTAL CARGA DOCENTE DEL ALUMNO 100 % Horas presenciales Horas no presenciales Horas ECTS a Asignatura: BIOTECNOLOGÍA VEGETAL Total de créditos ECTS: 4 Tipo de asignatura: Elegible. Profesorado: Juan Muñoz Blanco Jesús V. Jorrín Novo Ana Mª Maldonado Alconada Rosario Blanco Portales Introducción: La revolución verde, liderada por Norman Borlaug, Monkombu Swaminathan and Gurdev Khush, permitió triplicar la producción de alimentos a lo largo de las tres últimas décadas del siglo XX. Este extraordinario aumento de la producividad fue debida a la adopción de de variedades de plantas mejoradas genéticamente conjuntamente con una mejora del manejo de los sistemas de cultivo. A pesar del desarrollo de esa Agricultura intensiva, en muchos paises la la velocidad de demanda de alimentos supera la velocidad de producción de los mismos. Por ello, se necesita incrementar cada vez mas la proporción de terreno paras fines agrícolas y la utilización de Agroquímicos, ambas 43 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 situaciones producen efectos medio ambientales indeseados. La población mundial se ha incrementado en 2.5 billones de personas en los últimos 50 años, estimándose que en la actualidad es de 6.1 billones de personas. También se ha indicado que el aumento de la población no se estabilice hasta el año 2100. Por todo ello, se necesita que además de los recursos actuales disponibles en Agricultura se desarrollen e implementen nuevas tecnologías enfocadas a: a. Aumento de la producción de alimentos de alta calidad y bajo coste b. Que dichos aumentos de producción no motiven un impacto medio-ambiental insostenible. c. Mejora y explotación de las metodologías moleculares tanto aplicadas a la mejora genética asistida por marcadores (molecular breeding), como al desarrollo de plantas modificadas genéticamente y que presenten características agronómicas mejoradas o alimentos de mayor calidad nutricional y mas saludables (healthy foods). La Biotecnología Vegetal moderna, entendida como el desarrollo y aplicación de tecnologías basadas en los seres vivios o en sus procesos para obtener bienes y servicios, forma parte de la última revolución tecnológica que ha irrumpido en muchas áreas del quehacer humano y, especialmente, en su actividad productiva. En los inicios del siglo XXI presenta un desarrollo imparable y prometedor. La diversidad de Biotecnología, o la existencia de posibles Biotecnologías, puede ser tan amplia como son los organismos vivos protagonistas de la misma o las áreas de la producción a las que se puede aplicar. Esta gran diversidad, así como la amplitud de conocimientos y el rigor exigido a los mismos, obligan a centrar los objetivos del Programa Docente en la Biotecnología de las Plantas Superiores. Sin embargo el ámbito de aplicación se puede extender desde la sanidad humana y animal hasta la alimentación y la protección del medio ambiente. La variedad de disciplinas científicas que participan en el desarrollo de la Biotecnología exige la contribución de expertos de áreas que desde la mejora genética vegetal hasta la innovación y creación de empresas biotecnológicas, pasando por genéticos, biólogos celulares y moleculares, bioquímicos, microbiólogos y bio-informáticos. El impacto social de esta tecnología exige una reflexión sobre éticos y legales que plantea su desarrollo. Objetivos: Entre los objetivos docentes de la asignatura se presentan los siguientes: 1. Presentar los conocimientos básicos sobre el funcionamiento molecular de las especies vegetales y su relación con el entorno y que son esenciales para el desarrollo de proyectos de Biotecnología Vegetal. 44 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 2. Estudiar todas las técnicas moleculares utilizadas para el aislamiento de genes, el conocimiento de sus funciones y su aplicación en el desarrollo de la Biotecnología Vegetal en los diferentes campos de la actividad productiva. 3. Conocimiento en profundidad de las herramientas biotécnológicas modernas aplicables en Biotecnología Vegetal y de como deben de ser utilizadas para obtener cultivares mejorados genéticamente. 4. Revisar los aspectos sociales del desarrollo de la Biotecnología, incluidos los aspectos legales y el análisis de la empresa biotecnológica, y reflexionar sobre las implicaciones éticas del desarrollo de la misma. Estructura de la Asignatura: La asignatura se impartirá integrando las clases teóricas con las clases prácticas. Inicialmente se diseñará un objetivo Biotecnológico práctico que se desarrollará en el laboratorio. A lo largo del desarrollo del citado proyecto se irán impartiendo las clases téóricas correspondientes al temario y relacionadas los mas posible con el trabajo experimental que se esté haciendo en el laboratorio. En paralelo se les dará al alumno un tema monográfico de interés en biotecnología vegetal para que planteen un proyecto de aplicación biotecnológica que tendran que desarrollar (con la supervisión y tutoría de un profesor de la asignatura) y posteriormente exponer. Temario: Los temas se compondrán de una apartado teórico (exposición en aula) y de un apartado práctico (proyecto de desarrollo práctico tutelado por el profesor en el laboratorio). Tema 1.- Aplicaciones de las metodologías de “alto rendimiento” (micro-arrays, proteómica y metabolómicas) en la identificación de genes implicados en procesos de interés biotecnológico en plantas. Horas teórico-prácticas : 5 Tema 2.- Herramientas moleculares de interés en Biotecnología Vegetal: Promotores, vectores binarios y sistemas de transformación. Horas teórico-prácticas: 4 Tema3.- Genética Directa: Generación y utilización de mutantes en Biotecnología Vegetal. RILs y NILs. Tilling. Horas teórico-prácticas: 3 Tema 4.- Genética inversa: Silenciamiento y sobre-expresión génica y sus aplicaciones en Biotecnología Vegetal. Metodologías de ARN antisentido, RNAi y sistema Gateway. Horas teórico-prácticas: 4 Tema 5.- Aplicaciones relacionadas con la resistencia de las plantas frente a insectos y microorganismos patógenos (Estrés biótico). Horas teórico-prácticas: 2 45 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Tema 6.- Aplicaciones relacionadas con la resistencia de las plantas frente a estrés abiótico (especialmente sequía, salinidad y heladas). Horas teórico-prácticas: 2 Tema 7.- Abordajes biotecnológicos para la obtención de nutraceúticos en plantas (alimentos funcionales). Horas teórico-prácticas: 2 Tema 8.- Aproximaciones experimentales para la obtención de alimentos vegetales de mayor valor nutricional (calidad alimentaria). Horas teórico-prácticas: 2 Tema 9.- Química genómica aplicada a plantas. Horas teórico-prácticas: 1 Tema 10.- Las plantas como biofactorías y como organismos utilizables en fitorremediación Horas teórico-prácticas: 2 Métodos de evaluación: a) Evaluación continua b) Desarrollo del tema monográfico (aplicación biotecnológica) y exposición del mismo. Bibliografía básica: Libros - Introducción a la Biotecnología Vegetal: Métodos y aplicaciones. Ed. J.L Caballero, V. Valpuesta y J. Muñoz Blanco. CAJASUR, 2001. ISBN 84-7959-380-6. - Smith AM, Coupland G, Dolan L, Harberd N, Jones J, Martin C, Sablowski R, Amey A (2010) Plant Biology. Garland Science. ISBN 978-0-8153-4025-6 - Neal Stewart C (2008). Plant Biotechnology and Genetics. Wiley & Sons. ISBN 978-0-470-04381-3. - Slater A, Scott NW, Fowler MR (2008). Oxford University Press. ISBN 978-0-19-928261-6. Revistas - Trends in Plant Science - Trends in Biotechnology - Plant Biotechnology Journal - Current Opinion in Plant Biology - Cellular and Molecular Life Sciences 46 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Distribución ECTS de la Asignatura: BIOTECNOLOGÍA VEGETAL (a) 1 ECTS = 25 horas trabajo. (b) Estimación de estudio personal del alumno durante el curso 12 (trimestral) o 36 (anual) semanas: 1,5 horas de estudio por cada hora de teoría y 0,75 horas de estudio por cada hora de prácticas. (c)Las tutorías se encuentran incluidas en el total de Actividades Académicamente Dirigidas. Actividad Actividad Docente Materia Evaluación Profesor Alumno Procedimiento Peso en la nota final Tomar apuntes, discutir el material audiovisual Tipo de preguntas. Se valorará razonamiento y capacidad de síntesis 35 8h 8h 16h Horas presenciales Horas no presenciales Horas ECTS a Clases en aula Teoría Exposición de la Teoría. Apoyo con audiovisuales Prácticas en laboratorio Prácticas de aspectos metodológicos de la asignatura Preparación de la parte práctica de la asignatura Realización de actividades, ejercicios y demostraciones prácticas en el laboratorio Evaluación continuada, ejercicios, informes, actitud 35 19 h 29 h 58 h Proyecto Distribución de los trabajos, recomendar bibliografía, orientar Exposición y discusión del proyecto en el aula Se valorará contenido, preparación, presentación, interés, etc.. 30 4h 16 h 20 h Teoría y prácticas Discusión de las materias teóricopráctica de modo global y otros temas de interés a alumnos Preparar cuestiones y participar en la pagina web de la asignatuta Es una actividad no evaluable - 5h 5h Teoría y/o problemas Realizar y corregir el examen. Calificar globalmente al alumno 1h 10h 21h 32 h 68 h 100h Actividades dirigidasc Tutorias Exámenes Preparación de examen Realización de examen TOTAL CARGA DOCENTE DEL ALUMNO 80 % Asignatura: BIOTECNOLOGÍA DE EUCARIOTAS UNICELULARES Total de créditos ECTS: 4 Tipo de asignatura: Elegible. Profesorado: Juan Carlos García Mauricio Isidoro García García Aurora Galván Cejudo Emilio Fernández Reyes 47 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 OBJETIVOS ESPECÍFICOS DEL CURSO: - Familiarizarse con las principales propiedades de las levaduras y algas que las capacitan para desarrollar aplicaciones biotecnológicas. Conocer las estrategias moleculares, genómicas y proteómicas en aplicaciones industriales de levaduras y algas. Conocer aplicaciones biotecnológicas actuales de las levaduras y algas, así como sus perspectivas futuras. Familiarizarse con los principales tipos de biorreactores y fotorreactores, y comprender las claves para su diseño y funcionamiento. PROGRAMA TEÓRICO Programa de la parte de levaduras: 1.- Las levaduras. Características morfológicas y fisiológicas. 2.- Genómica y proteómica de levaduras industriales. Aplicación de los chips de DNA a las levaduras industriales. Proteómica en levaduras vínicas. 3.- Levaduras de flor. Base molecular de la formación del velo de flor. Aplicaciones biotecnológicas. 4.- Mejora de levaduras industriales mediante técnicas de ingeniería genética. Sistemas de transformación genética en levaduras. Regulación de la expresión génica: promotores de interés biotecnológico. Levaduras transgénicas. Legislación en torno al empleo de ingeniería genética en alimentación. Perspectivas de futuro. 5.- Inmovilización de levaduras. Cultivos de levaduras inmovilizadas. Ventajas y dificultades de usar levaduras inmovilizadas. Métodos de inmovilización. Aplicaciones de la inmovilización celular en vinificación. Bioinmovilización. Programa de la parte de biorreactores: 1.- Aspectos generales de los biorreactores. Tipos y modos de funcionamiento. 2.- Cinética bioquímica. Análisis y diseño. 3.- Biorreactores para levaduras. Relación entre tipo de producto y elección del biorreactor. Biorreactores con células libres e inmovilizadas. 4.- Biorreactores para algas. Fotobiorreactores. Aspectos generales de su diseño y funcionamiento. Programa de la parte de algas: 1.- Las algas eucariotas. Características y ciclos reproductivos. Características anatómicas interesantes. Fotosíntesis e interacciones con la luz. 2.- Papel biogeoquímico de las algas. Limitación de nutrientes en el crecimiento de algas. Las algas y los ciclos del carbono, nitrógeno, fósforo, y azufre. 3.- Utilización de algas en alimentación humana y animal. Polisacáridos derivados de las algas. Aplicaciones de las algas como fertilizantes, suplementos terapeúticos, y en cosmética. Las toxinas derivadas de las microalgas. 48 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 4.- Transformación nuclear de microalgas eucarióticas. Métodos y características. Construcciones utilizadas. Dificultades para la expresión estable de los transgenes. 5.- Transformación de cianobacterias. Métodos y aplicaciones. 6.- Diatomeas. Biología. Manipulación genética. Bioquímica y aplicaciones tecnológicas. Síntesis de ácidos grasos. Biomineralización. 7.- Transformación de los cloroplastos de Chlamydomonas. Métodos. Poliploidía y los problemas de la heteroplasmia. 8.- El sesgo en el uso de codones en la expresión de genes heterólogos. Expresión en el núcleo frente al cloroplasto. 9.-Secuenciación de los genomas de algas. Decisiones sobre los organismos a secuenciar. Secuencias completas, parciales y de cDNA. Manejo de las bases de datos de los genomas. Los genomas de virus. 10.-Mutagénesis insercional en los estudios de genómica funcional. 11.- Optimización de la expresión de proteínas recombinante en los cloroplastos. 12.- Ficorremediación de metales pesados usando microalgas transgénicas. Biosensores de algas. 13.- La producción de hidrógeno a partir de algas transgénicas. Estrategias de ingeniería de microalgas eucariotas. 14.- Vacunas de microalgas. PROGRAMA PRÁCTICO Práctica 1: Bioinmovilización de levaduras. Aplicación de biocápsulas de levaduras a distintas fermentaciones. Práctica 2: Transformación de algas con marcadores moleculares. Análisis de transformantes para detectar la presencia de un marcador en la inactivación insercional de un gen blanco. Práctica 3: Determinación de la ecuación cinética para el crecimiento celular. Análisis del funcionamiento de un biorreactor. CRITERIOS DE EVALUACIÓN: 1.- Criterios cualitativos de evaluación: Nivel de aprendizaje alcanzado. Capacidad para integrar sus conocimientos y expresarlos de forma oral y escrita. Sentido crítico y capacidad de generar ideas de modo individual y en grupo. Capacidad para mantenerse informado de modo personal. 2.- Métodos de evaluación: Evaluación continua. Trabajos realizados. Participación en las tutorías. Examen de preguntas cortas sobre aspectos teóricos de la materia (1 hora). 3.- Criterios cuantitativos de evaluación: Cumplimiento de las horas presenciales (Obligatorio 80%). Examen y evaluación continua: 4 puntos. Evaluación continua y parte práctica: 2 puntos. 49 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Trabajo y presentación de proyecto: 2 puntos. Participación y comentarios de seminarios: 1puntos. Participación en tutorías: 1 puntos. BIBLIOGRAFÍA BÁSICA: - Barbosa, M. Microalgal photobioreactors. Scale-up and optimisation. 2003. Ph.D. Thesis, Wageningen University, The Netherlands. ISBN: 90-5808-898-7. -Barsanti, L., Gualtieri, P. Algae. Anatomy, Biochemistry and Biotechnology. 2006. CRC Press-Taylor and Francis. Boca Raton. - Carrascosa, A.V., Muñoz, R., González, R. Microbiología del vino. AMV Ediciones, Madrid. ISBN: 84-87440-06-1. - Dutta, R. Fundamentals of Biochemical Engineering. 2008. Springer. ISBN: 978-3-540-77900-1. -Glick, B.R., Pasternak, J.J. Molecular Biotechnology. Principles & Applications of Recombinant DNA. 1998. ASM Press, Washington. -León, R., Galván, A., Fernández, E. Transgenic microalgae as green cell factories. 2007. Springer Science-Landes Bioscience.New York, Austin. - Nedovic, V., Willaert, R. Applications of cell Immobilisation Biotechnology. 2005. Springer. The Netherlands. ISBN: 1-4020-3229-3. - Xiao, W. Yeast Protocols. Methods in Molecular Biology. 2006. Humana Press Inc. Totawa, New Jersey. ISBN: 1-58829-437-4. Distribución ECTS de la Asignatura: BIOTECNOLOGÍA DE EUCARIOTAS UNICELULARES (a) 1 ECTS = 25 horas trabajo. (b) Estimación de estudio personal del alumno durante el curso 12 (trimestral) o 36 (anual) semanas: 1,5 horas de estudio por cada hora de teoría y 0,75 horas de estudio por cada hora de prácticas. (c)Las tutorías se encuentran incluidas en el total de Actividades Académicamente Dirigidas. Actividad Actividad Docente Materia Evaluación Profesor Alumno Procedimiento Peso en la nota final Tomar apuntes, discutir el material audiovisual Tipo de preguntas. Se valorará razonamiento y capacidad de síntesis 40 % 20 h 20h b 40h Horas presenciales Horas no presenciales Horas ECTS a Clases en aula Teoría Exposición de la Teoría. Apoyo con audiovisuales Prácticas en laboratorio Prácticas de aspectos metodológicos de la asignatura Preparación de la parte práctica de la asignatura Realización de actividades, ejercicios y demostraciones prácticas en el laboratorio Evaluación continuada, ejercicios, informes, actitud 20% 7h 7h 14 h Proyecto Distribución de los trabajos, recomendar bibliografía, orientar Exposición y discusión del proyecto en el aula Se valorará contenido, preparación, presentación, interés, etc.. 20% 4h 16 20 h Teoría y prácticas Discusión de las materias teóricopráctica de modo global y otros temas de interés a Preparar cuestiones y participar en la pagina web de la asignatuta Es una actividad no evaluable - 5 5h Actividades dirigidasc Tutorias 50 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 alumnos Exámenes Teoría y/o problemas Realizar y corregir el examen. Calificar globalmente al alumno Preparación de examen Realización de examen TOTAL CARGA DOCENTE DEL ALUMNO 80 % 1h 20h 21h 32 h 68 h 100h Asignatura: TRANSPORTE DE SOLUTOS EN MEMBRANAS VEGETALES Total de créditos ECTS: 4 Tipo de asignatura: Elegible. Profesorado: Manuel Benlloch Marín José María Fournier Andray José Ramos Ruiz (todos intervienen a partes iguales) Objetivos: Se pretende que el alumno adquiera conocimientos específicos, tanto teóricos como prácticos, sobre los procesos de transporte de solutos a nivel celular y a nivel de la planta, que se consideran necesarios para conocer las relaciones hídricas y la nutrición mineral de las plantas Programa Teórico 1. Estructura y función de la membrana 2. Bioenergética del transporte a través de la membrana 3. Transporte iónico a través de la planta 4. Regulación del flujo hídrico en la planta. Acuaporinas. 5. Bases moleculares de la tolerancia al estrés salino 6. Transporte y compartimentación de asimilados en la planta. Carga y descarga del floema. Programa Práctico. Actividades complementarias Se realizaran prácticas de laboratorio sobre transporte de solutos y agua, utilizando como material biológico células de levadura y plantas de girasol. Los alumnos elegirán, a ser posible, el tipo de práctica a realizar. Al comienzo de las mismas recibirán bibliografía relacionada con la práctica seleccionada, que servirá de apoyo para la realización del trabajo de laboratorio y para la redacción del manuscrito final, en el que se recogerán los resultados obtenidos. Cada alumno expondrá de forma oral un resumen de su trabajo de investigación relacionándolo con la bibliografía utilizada. En el caso de que en un mismo tema participaran más de un alumno, se deja a su criterio el grado de participación que cada uno de ellos tenga en la exposición oral. En todos los casos se pretenderá ajustar el horario de practicas con las actividades de las 51 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 mismas, tanto en lo referente al trabajo de laboratorio como a la preparación del manuscrito. Bibliografía Marschner, H. (1995). Mineral Nutrition of Higher Plants. Academic Press, London. Harold, F.M. (1986). The Vital Force: A Study of Bioenergetics. W. H. Freeman and Company, New York. Wilkinson, R.E. (1994). Plant-Environment Interactions. Marcel Dekker, Inc., New York Hohmann, S and Willem H. Mager (2005). Yeast Stress Responses (Topics in Current Genetics) Springer Yeo, A. R. and Flowers, T. J. (2007). Plant Solute Transport. Blackwell Publishing, Oxford. Evaluación La evaluación será contínua. Se tendrá en cuenta el grado de participación del alumno en las diferentes actividades de la asignatura: clases teóricas, trabajo en el laboratorio y exposición de los resultados. También se valorará la calidad de la exposición final del trabajo de laboratorio: originalidad, claridad y nivel de comprensión del proceso estudiado. Asignatura: METABOLÓMICA Total de créditos ECTS: 4 Tipo de asignatura: Elegible. Profesorado: María Dolores Luque de Castro Feliciano Priego capote Objetivos del curso: (1) Introducir a los alumnos en la materia objeto del curso. (2) Poner de manifiesto la estrecha relación entre ésta y otras ómicas como la proteómica y la genómica, así como las ventajas de su uso conjunto. (3) Mostrar la vertiente analítica de la metabolómica, extrapolable a las demás ómicas. (4) Formar a los alumnos en los aspectos prácticos de la metabolómica mediante las adecuadas clases prácticas. Programa teórico: LECCIÓN 1. INTRODUCCIÓN A LA METABOLÓMICA Introducción. Sub-disciplinas de la Metabolómica: Clasificación. Estrategias usadas en Metabolómica. La Metabolómica en el contexto de las disciplinas ómicas: La biología de sistemas. 52 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 LECCIÓN 2. LAS HERRAMIENTAS ANALÍTICAS DE LA METABOLÓMICA Introducción. Selección de la muestra. Preparación de la muestra. Técnicas de análisis sin separación previa —espectrometría de resonancia magnética nuclear -(NMR), espectrometría de masas (MS), espectrometría de infrarrojo (IR). Técnicas de análisis con separación previa — cromatografía de gases (GC), cromatografía de líquidos (LC), electroforesis capilar (CE). LECCIÓN 3. LA QUIMIOMETRÍA Y LA BIOINFORMÁTICA EN METABOLÓMICA Introducción. Modelado y análisis de datos: La combinación del diseño de experimentos y el análisis multivariante. Etapas y aproximaciones quimiométricas de los estudios en metabolómica. Herramientas quimiométricas características de la metabolómica. Utilización de bases de datos. LECCIÓN 4. ÁREAS GENÉRICAS DE APLICACIÓN DE LA METABOLÓMICA Introducción. La metabolómica como herramienta en biomedicina: Investigación y desarrollo farmacéuticos y estudios clínicos. Aplicación de la metabolómica en botánica. Biomarcadores. LECCIÓN 5. NUTRIMETABOLÓMICA Introducción: Relación dieta-salud. Logros genéricos en nutrición y salud. Logros recientes en nutrición y salud. Las ómicas y el binomio nutrición-salud. La nutrimetabolómica para entender los efectos de la alimentación en la salud. El papel del microbioma en nutrimetabolómica. Alimentos transgénicos y nutrimetabolómica. Contribuciones de interés en el campo de la nutrimetabolómica. Contribuciones del Grupo a la nutrimetabolómica LECCIÓN 6. XENOMETABOLÓMICA Concepto y extensión de la disciplina. Metabolómica de fármacos, drogas y tóxicos en individuos: Métodos analíticos. Xenometabolómica ambiental: Estrategias y métodos analíticos. Contribuciones realizadas en la UCO. LECCIÓN 7. LIPIDÓMICA Introducción. La determinación de lípidos: Etapas de un método analítico. Papel de las lipasas en la transformación de lípidos. Los lípidos como biomarcadores de enfermedades. Investigaciones sobre lipidómica realizadas por el grupo PAI FQM-227. PROGRAMA PRÁCTICO Programa práctico: Práctica 1: Biomarcadores del metabolismo óseo. Preparación de la muestra de suero. Análisis automatizado de vitamina D y sus metabolitos mediante extracción en fase sólida (Prospekt)–separación individual mediante HPLC– identificación/cuantificación mediante espectrometría de masas de triple cuadruplo. Tratamiento de los datos e interpretación de los resultados. Práctica 2: Metaboloma del olivo. Selección de la muestra (hojas o frutos, según la época) y su preparación. (A) Perfil de lípidos y antioxidantes mediante 53 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 lixiviación asistida por ultrasonidos, microondas o extractante sobrecalentado– separación individual mediante cromatografía de gases y electroforesis capilar, respectivamente– identificación/determinación mediante espectrometría de masas en tándem y mediante espectrometría de masas de triple cuadrupolo, respectivamente. (B) Huella dactilar metabólica, utilizando la clasificación de variedades por su perfil fenólico mediante el método de determinación de antioxidantes. Tratamiento de los datos obtenidos en (A) y (B) e interpretación de los resultados. BIBLIOGRAFÍA BÁSICA: Affolter M, Bergonzelli GE, Blaser K, Blum-Sperisen S, Corthésy B, Fay L, GarcíaRódenas C, Lopes L, Marvin-Guy L, Mercenier A, Mutch D, Panchaud A, Raymond F, Schmidt-Weber C, Schumann A, Spertini F, Williamson G, Kussmann M (2006): Omics for prevention: gene, protein and metabolite profiling to better understand individual disposition to disease. 57th Nestle Nutrition Workshop. Primary Prevention by Nutrition Intervention in Infancy and Childhood. (24–28 May 2005). Allen J, Davey HM, Broadhurst D, Rowland JJ, Oliver SG, Kell DB (2004): Discrimination of modes of action of antifungal substances by use of metabolic footprinting. Appl Environm Microbiol 70: 6157–6165. Bang JW. Crockford DJ. Holmes E. Pazos F. Sternberg MJE, Muggleton HS. Nicholson JK (2008) Integrataaive top-down system metabolic modelling in experimental disease states via data-driven Bayesian methods. J Proteome Res 7: 497–503. Colquhoun IJ. (2007): Use of NMR for metabolic profiling in plant systems. J Pestic Sci 32(3): 200–212. Davies KM (2007): Genetic modification of plant metabolism for human health benefits. Mut Res, 622(1-2): 122–37. Dobrovolsky VN, Bowyer JF, Pabarcus MK, Heflich RH, Williams LD, Doerge DR, Arvidsson B, Bergquist J and Casida JE (2005): Effect of arylformamidase (kynurenine formamidase) gene inactivation in mice on enzymatic activity, kynurenine pathway metabolites and phenotype. Biochim Biophys Acta 1724 (1-2): 163–172. Evans DA, Hirsch JB, Dushenkov S (2006): Phenolics, nutrigenomics. J Sci Food Agriculture 86: 2503–2509. inflammation and Fiehn O, Kristal B, van Ommen B, Sumner LW, Sansone SA, Taylor C, Hardy N, Kaddurah-Daouk R (2006): Establishing reporting standards for metabolomic and metabonomic studies: a call for participation. Omics J Integrative Biol 10(2): 158–63. Fogg-Johnson N, Kaput J (2007): Providing valid personalized nutritional advice to consumers on the basis of their genetic makeup will require scientific collaboration, data sharing, and joint funding strategies. Moving forward with nutrigenomics. Food Technol 61: 50–56. Griffiths WJ (Editor) (2008): Metabolomics, Metabonomics and Metabolite Profiling. Royal Society of Chemistry, Cambridge, UK. 54 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Gu H, Chen H, Pan Z, Jackson AU, Talaty N, Xi B, Kissinger C, Duda C, Mann D, Raftery D, Graham R (2007): Monitoring diet effects via Biofluids and their implications for metabolomics studies. Anal Chem 79: 89–97. Hoefgen R. Nikiforova VJ. (2008): Metabolomics integrated with transcriptomics: assessing systems response to sulphur-deficiency stress. Physiologia Plantarum 132: 190–198. Huang X, Regnier FE (2008): Differential metabolomics using stable isotope labelling and two-dimensional gas chromatography with time-of-flight mass spectrometry. Anal Chem 80: 107–114. Ito H, Gonthier MP, Manach C, Morand C, Mennen L, Rémésy C, Scalbert A, (2005): Polyphenol levels in human urine after intake of six different polyphenol-rich beverages. Br J Nutr 94: 500–509. Japón-Luján R, Luque-Rodríguez JM, Luque de Castro MD (2006): Multiariate optimisation of microwave-assisted extraction of oleuropein and related biophenols from olive leaves. Anal Bioanal Chem 385: 753–759. Japón-Lujan R, Priego-Capote F, Marinas A, Luque de Castro MD (2008): Liquid chromatography/triple quadrupole tandem mass spectrometry with multiple reaction monitoring for optimal selection of transitions to evaluate nutraceuticals from olive-tree materials. Rapid Commun Mass Spectrom 22: 855–864. Kaput J, Perlina A, Hatipoglu B, Bartholomew A, Nikolsky Y (2007): Nutrigenomics: concepts and applications to pharmacogenomics and clinical medicine. Pharmacogenomics 8: 369–390. Kaderbhai NN, Broadhurst DI Ellis DI, Goodacre R, Kell DB (2003): Monitoring metabolite secretion by Escherichia coli tryptophan metabolism mutants using FT-IR and direct injection electrospray mass spectrometry. Compar Funct Genomics 4: 376– 391. Kell DB. (2006): System biology, metabolic modelling and metabolomics in drug discovery and development. Drug Discovery 11 (23/24): 1085–1098. Kinloch A, Tatzer V, Wait R, Peston D, Lundberg K, Donatien P, Moyes D, Taylor PC, Venables PJ (2005). Identification of citrullinated α-enolase as a candidate autoantigen in rheumatoid arthritis. Arthritis Research & Therapy, 7:R1421–R1429doi Kussmann M, Raymond F, Affolter M (2006): OMICS-driven biomarker discovery in nutrition and health. J Biotechnol 124: 758-787. Lawrence GR (2005): Pathogenesis of osteoporosis: concepts, conflicts, and prospects. J Clin Invest 115: 3318–3325). Lindon JC. Nicholson JK. (2008): Analytical technologies for metabonomics and metabolomics, and multi-omic information recovery. Trends Anal Chem. DOI: 10.1016/j.trac.2007.08.009. Lindon JC, Nicholson JK, Holmes E (2007): The Handbook or Metabonomics and Metabolomics. Elsevier, Amterdam. 55 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Lindon JC, Nicholson JK, Holmes E (2005): NMR spectroscopy of biofluids. Metabonomics Toxicity Assessment 105–172. Lindon JC et al (2005): Summary recommendations for standardization and reporting of metabolic analyses. Nature Biotechnol 23(7): 833–838. Maher AD, Zirah SFM, Holmes E, Nicholson JK (2007): Experimental and analytaical variation in human urine in 1H NMR spectroscopy-based metabolic pehnotyping studies. Anal Chem 79: 5204–5211. Maiani G, Serafini M, Salucci M, Azzini E, Ferro-Luzzi A (2007); Application of a new high-performance liquid chromatographic method for measuring selected polyphenols in human plasma, J Chromatogr B 692: 311–317. Malayappan B, Garrett TJ, Segal M, Leeuwenburgh C (2007): Urinary analysis of 8oxoguanine, 8-oxoguanosine, fapy-guanine and 8-oxo-2'-deoxyguanosine by highperformance liquid chromatography-electrospray tandem mass spectrometry as a measure of oxidative stress. J Chromatogr 1167(1): 54–62. Mariman ECM (2006): Nutrigenomics and nutrigenetics: the 'omics' revolution in nutritional science. Biotech Applied Biochem 44: 119–128. Martin FPJ, Dumas ME, Wang Y, Legido-Quigley C, Yap Ivan KS; Tang H, Zirah S, Murphy GM, Cloarec O, Lindon JC, Sprenger N, Fay LB, Kochhar S, van Bladeren P, Holmes E, Nicholson JK (2007) A top-down systems biology view of microbiomemammalian metabolic interactions in a mouse model. Mol Syst Biol 3: 112–120. Montañez R. Sánchez-Jiménez F, Aldana-Montes JF. Medina MA. (2007): Polyamines: metabolism to systems biology and beyond. Amino Acids 33: 283–289. Mullinix KP (2007): The future of personalized nutrition. Food Technol 61: 96–105. Rezzi G, Ramadan Z, Martin FPJ, Fay LB, van Bladeren P, Lindon JC, Nicholson JK, Kochhar S (2007): Human Metabolic Phenotypes Link Directly to Specific Dietary Preferences in Healthy Individuals.J Proteome Res 6(11): 4469–4477. Romero A, Bastida S, Sánchez-Muñiz FJ, (2006): Cyclic fatty acid monomer formation in domestic frying of frozen foods in sunflower oil and high oleic acid sunflower oil without oil replenishment. Food Chem Toxicol 44(10): 1674–1681. Romero R. et al. (2008): The use of high-dimensional biology (genomics, transcriptomics, proteomics, and metabolomics) to understand the preterm parturition syndrome. Intern J Obst Gynaecol DOI: 10.1111/j.1471-0528.2006.01150.x. Ryan D. Robards K. (2006) Metabolomics: the greatest omics of them all? Anal Chem 78: 7954–7958. Ruiz-Jiménez J, Priego-Capote F, Mata-Granados JM, Quesada-Gómez JM, Luque de Castro MD. (2007): Determination of the ubiquinol-10 and ubiquinone-10 (coenzyme Q10) in human serum by liquid chromatography tandem mass spectrometry to evaluate the oxidative stress. J Chromatogr 1175(2): 242–248. Sánchez-Ávila N, Priego-Capote F, Luque de Castro MD (2007): Ultrasound-assisted extraction and silylation prior to gas chromatography–mass spectrometry for the characterization of the triterpenic fraction in olive leaves. J Chromatogr 1165(1–2): 158–165. 56 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Sing OV. (2006): Proteomics and metabolomics: The molecular make-up of toxic aromatic pollutant bioremediation. Proteomics 6: 5481–5492. Subbiah MTR (2007): Nutrigenetics and nutraceuticals: the next wave riding on personalized medicine. Transl Res 149: 55–61. Trauger SA. et al. (2008): Correlatinf the transcriptome, proteome, and metabolome in the environmental adaptation of a hyperthermophile. J Proteome Res 7: 1027–1035. Trygg J, Holmes E, Lundstedt T (2007): Chemometrics in metabonomics. J Rev Proteome Res 6: 469–479. Wang Y, Lawler D, Larson B, Ramadan Z, Kochhar S, Holmes E, Nicholson JK (2007): Metabonomic investigations of aging and caloric restriction in a life-long dog study. J Proteome Res 6(5): 1846–1854. Wang X, Liu R, Sun J, Guan S, Yang M, Bi K, Guo D (2007): HPLC method for the determination and pharmacokinetic studies of four triterpenoids in rat plasma after oral administration of Ganoderma lucidum extract. Biomed Chromatogr 21: 389–396. Watson AD (2006): Lipidomics: a global approach to lipid analysis in biological systems. J. Lipid Res 47: 2101–2111. Weckwerth W. (2008): Integration of metabolomics and proteomics in molecular plant physiology – coping with the complexity by data-dimensionality reduction. Physiologia Plantarum 132: 176–189. Wenk MR (2005): The emerging field of lipidomics. Nat Rev Drug Discovery 4: 594610. Werf MJ. Overkamp KM. Muilwijk B. Coulier L. Hankemeier T. (2007): Microbial metabolomics: towards a platform with full metabolome coverage. Anal Biochem 370: 17–25. Criterios de evaluación: Se exigirá el cumplimiento de un mínimo de horas presenciales del 80% del total. La evaluación será un compendio de los siguientes aspectos: -Un examen de preguntas cortas sobre los aspectos teóricos de la materia (4 puntos) -La evaluación continuada del interés demostrado en las clases teóricas y en las tutorías (2 puntos). -Trabajo de revisión crítico (1.5 puntos). -Interés y capacidad demostradas en las clases prácticas de laboratorio (2.5 puntos). 57 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 4. ITINERARIO 2: BIOTECNOLOGÍA SANITARIA 58 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Asignatura: TÉCNICAS AVANZADAS DE IMAGEN CELULAR Total de créditos ECTS: 4 Tipo de asignatura: Elegible. Profesorado: Justo P. Castaño Fuentes Rafael Vázquez Martínez Antonio J. Martínez Fuentes Raul M. Luque Huertas Profesores colaboradores invitados: Dr. Mario Durán Prado Instituto de Parasitología López Neira. CSIC. Granada. Dr. Spencer L. Shorte. Institut Pasteur. Paris, Francia. (1 ECTS) Competencias y objetivos del aprendizaje: COMPETENCIAS TRANSVERSALES/GENÉRICAS: Competencias básicas de la profesión investigador en biomedicina Capacidad de aplicar la teoría a la práctica COMPETENCIAS ESPECÍFICAS: • Cognitivas (Saber): Conocer los fundamentos teórico-prácticos de las principales técnicas avanzadas de adquisición y análisis de imagen de células y moléculas. • Procedimentales/Instrumentales (Saber hacer): Destreza en técnicas de adquisición, procesamiento y análisis de imágenes de células y estructuras subcelulares en distintas condiciones experimentales concretas, aplicando criterios metodológicos para la resolución de problemas de estudio. Habilidad en la redacción, exposición y discusión de memorias de trabajos experimentales. • Actitudinales (Ser): Espíritu crítico. Capacidad de elección metodológica, análisis de resultados y obtención de conclusiones. Objetivos: Se persigue que el alumno alcance un aprendizaje teórico y práctico de las principales técnicas y metodologías avanzadas que se emplean en la adquisición, procesamiento y análisis cuantitativo de imágenes de células, tanto vivas como fijadas, así como de orgánulos subcelulares y de moléculas. El alumno podrá 59 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 adquirir los conocimientos y recursos necesarios para abordar líneas de investigación biomédica que requieran modelos de estudio celulares vivos y/o fijados sobre los que se aplican métodos de análisis a través de la imagen cuantitativa. De esta forma, el alumno podrá aplicar este conjunto de técnicas al estudio de distintos organismos y modelos experimentales, seleccionando y aplicando la metodología más conveniente al planteamiento experimental que requiera la resolución de problemas concretos y aprendiendo asimismo a realizar el análisis, discusión y presentación de resultados. Programa Teórico: 1.-Principios básicos del Análisis de Imagen. Técnicas de adquisición, digitalización y procesamiento de imágenes. Filtros electrónicos para la mejora de imagen. Principios de Morfometría, Estereología y Densitometría. Análisis de imagen aplicado a la Citología cuantitativa en Biomedicina. 2.- Microfluorimetría. Fundamentos de microfluorimetría cuantitativa. Técnicas de microfluorimetría para el estudio de iones intracelulares y otros segundos mensajeros (AMPc): fundamentos, métodos de excitación doble-emisión simple y excitación simple-emisión doble. Cuantificación por microfluorimetría de la incorporación de membrana para análisis de secreción celular. Evaluación de interacciones moleculares mediante técnicas microfluorimétricas: aplicaciones del FRET (Fluorescence Resonance Energy Transfer). 4.- Microscopía confocal. Técnicas de microscopía confocal: fundamentos, tipos y métodos. Microscopía confocal para la visualización y adquisición de imágenes biológicas. Aplicaciones cuantitativas de la microscopía confocal: FRET, FRAP, FLIP. Métodos de análisis de células vivas mediante microscopía confocal. 5.- Bioluminiscencia. Principios, tipos y aplicaciones de la bioluminiscencia en el estudio del funcionamiento de las células. Sistemas de adquisición, cuantificación y análisis de bioluminiscencia. Estudios de la regulación de la expresión génica en células individuales vivas. Prácticas: 1. Adquisición, digitalización y procesamiento de imágenes para la evaluación de parámetros morfométricos y densitométricos mediante un sistema semiauto-mático de análisis de imagen. 2. Evaluación de la concentración de calcio libre citosólico en células adenohipofisarias mediante microfluorimetría. 3. Bases del manejo del microscopio confocal para el examen de muestras biológicas. 60 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 4. Cuantificación de la expresión génica en células individuales vivas mediante bioluminiscencia. Metodología: Presenciales: 35 • Clases Teóricas: 7 • Clases Prácticas: 26 • Examen final: 2 No presenciales: 65 Actividades en colaboración con el profesor: 3 • Tutorías individualizadas (presenciales / virtuales) Actividades autónomas del alumnado: 62 Se incluyen la realización de Actividades Académicas Dirigidas y el trabajo colaborativo en el espacio virtual. • Horas de estudio de teoría: 8 • Estudio y elaboración de datos experimentales 10 • Preparación de Trabajos (memorias) : 32 • Preparación de Exámenes: 12 Técnicas Docentes Clases teóricas: En aula, con presentaciones virtuales. Carácter obligatorio. Tutorías: Orientación del profesor al alumno, personalizada. Presencial o en espacio virtual de aprendizaje Actividades dirigidas: Realización de memorias a partir de la actividad desarrollada en cada sesión práctica. Posibilidad de hacer ejercicios y problemas planteados por el profesor. Carácter optativo Clases prácticas: En laboratorio, grupos reducidos (no más de 15) alumnos. Carácter obligatorio Estudio y trabajo personal: Asimilación de conceptos aprendidos. Actividad en el espacio virtual de aprendizaje. Examen final: Realización de un test para evaluar los conocimientos básicos adquiridos. Carácter obligatorio Criterios de evaluación y seguimiento Para la calificación final se contempla la siguiente distribución de puntuación y método de seguimiento: • 50% Conocimientos adquiridos: EXAMEN FINAL • 25% Asistencia con aprovechamiento: CONTROL DE PRESENCIA • 25%. Memorias de prácticas y actividades relacionadas (espacio virtual). EVALUACION DEL PROFESOR, SEGUIMIENTO EN TUTORIA Y/O ESPACIO WEB. 61 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Asignatura: INMUNOBIOLOGÍA MOLECULAR Total de créditos ECTS: 4 (100 horas) Tipo de asignatura: Elegible Profesorado: José Peña Martínez. CR: 2 Rafael Solana Lara. CR: 1 Manuel Santamaría Osorio. CR: 1 Objetivos: Introducir al alumno en la función de las moléculas de histocompatibilidad (MHC) en la salud y la enfermedad. Ofrecer conocimientos actuales de las moléculas del complejo mayor de histocompatibilidad y de los receptores que las reconocen y actúan como ligando de las mismas. Resaltar la gran importancia de estas moléculas en la regulación de la respuesta inmune a través de su capacidad de de presentación de antígenos a los linfocitos T y su contribución a definir de lo propio y no propio a cada organismo. También se estudiaran los receptores que reconocen a estas moléculas MHC descritos recientemente y que tienen una gran importancia en los fenómenos de defensa del organismo y en el reconocimiento inmune de los componentes propios. Programa teórico: 1. Moléculas del complejo mayor de histocompatibilidad (MHC). Estructura, tipos, distribución y relevancia biológica. 2. Genética de las moléculas MHC y regulación de su síntesis. 3. Polimorfismo de las moléculas MHC: su origen y su importancia en la evolución de las poblaciones. 4. Función de las moléculas MHC en la presentación de antígenos y como marcadores de lo propio y lo extraño. 5. Biología de los receptores inhibidores y activadores que reconocen las moléculas MHC. Estudio de las distintas familias de receptores y sus implicaciones funcionales. 6. Importancia de las moléculas MHC y sus receptores en los fenómenos de tolerancia inmunológica y en inmunopatología de las enfermedades autoinmunes. 7. Importancia de las moléculas MHC en los trasplantes de órganos sólidos y de médula ósea. Procesos de alorreconocimiento y rechazo. 8. Moléculas MHC y en especial HLA-G y sus receptores en la aceptación inmune del feto por la madre. 62 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 9. Implicaciones del MHC en enfermedades virales con especial referencia a los fenómenos de “escape viral”. 10. MHC en inmunología tumoral con especial referencia a los fenómenos de escape y crecimiento de células tumorales. 11. Utilidad de las moléculas MHC en el estudio de migraciones de poblaciones, diagnóstico de enfermedades y estudios de paternidad. Programa de prácticas1 1. Laboratorio de Inmmunología con especial referencia al laboratorio de histocompatibilidad. Características y requerimientos del mismo. Sistemas de acreditación internacional de sus resultados. 2. Laboratorio de citometría. Fundamentos y equipos. Estudio de los sistemas de separación de células (sorting) y sus aplicaciones en biología y en Medicina. 3. Estudio por técnicas serológicas de las moléculas HLA. Uso de los anticuerpos y técnicas de citotoxicidad en placas de Teresaki. 4. Estudio de niveles y especificidades de anticuerpos citotóxicos anti-HLA. Ventajas e inconvenientes de las distintas técnicas existentes. 5. Análisis génico de las moléculas HLA clase I y II por baja y alta resolución. Uso de las técnicas de PCR y de PCR a tiempo real en al estudio del genotipo HLA. 6. Estudio de las moléculas de histocompatibilidad solubles y sus receptores 7. Análisis geonómico de los receptores de las moléculas de histocompatibilidad. Moléculas de la superfamilia de las inmunoglobulinas como receptores tipo lectina. 8. Citometría aplicada al estudio de las moléculas de histocompatibilidad en linfocitos y las distintas familias de receptores de las moléculas HLA. 9. Estudio inmunohistológico de moléculas HLA y sus receptores en muestras de tejido sano y en diversas patologías, especialmente en trasplantes y SIDA. Bibliografía/referencias/enlaces: Se recomendarán diversos libros de texto, libros-online y trabajos monográficos sobre inmunología, esencialmente en el área de las moléculas de histocompatibilidad. Entre estos libros se consideraran, entre otros: 1. Peña, J. (Coordinador): Inmunología. Bases moleculares y celulares. Editorial Pirámide. (Grupo Anaya). 2001. ISBN: 84-368-0828-2 (segunda edición). Madrid. 2. Solana, R. and Peña, J.: MHC antigens and NK cells. Editorial R.G. Landes Company. ID: 2693 USA.1995. 1 Las practicas se realizarán el laboratorio de Inmunología ubicado en la Facultad de Medicina y en el laboratorio del Servicio de Inmunología del Hospital Reina Sofía al ser el profesor responsable de este curso, el director de dicho servicio al poseer una plaza hospitalaria vinculada a la Universidad. 63 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 3. Peña, J: Inmunopatología. Bases moleculares y celulares. Editorial ARAN. Madrid. 2001. ISBN: 84-86725-91-7. Madrid. Así como: 1. Abbas, A 2007. Inmunología Cel. y Mol. Interamericana. 2. Janeway, C. 2008. Inmunobiología. Churchil Levistone 3. Klein, J. 2004. Immunology. Blackwell Scientific Public. 4. Kubi, J. Inmunología.2005. Freemen and Company 5. Paul, W.E. 2005. Fundamental Immunology. Raven Press. 6. Regueiro, J. R. 2008. Inmunología. Panamericana. 7. Roitt, I.M. 2008. Inmunología. Panamericana Así como las paginas webs especialmente diseñadas para este objetivo: Libro inmunología enlinea: http://www.inmunologiaenlinea.com Libro de trasplante online: http://www.inmunologiatrasplante.com Criterios de evaluación: Se realizará tanto del contenido teórico como las habilidades adquiridas en el desarrollo de las distintas clases prácticas. Para ello se realizará una prueba escrita y otra consistente en el desarrollo de un caso practico al final del periodo formativo. Se considerará de manera muy prioritaria la asistencia a las clases tanto teóricas como prácticas. Advertencias al alumno. Se hace una indicación de que las clases se impartirán en la Facultad de Medicina. ANEXO I: Distribución ECTS de la Asignatura AVANCES EN INMUNOLOGÍA (a) 1 ECTS = 25 horas trabajo. (b) Estimación de estudio personal del alumno durante el curso 12 (trimestral) o 36 (anual) semanas: 1,5 horas de estudio por cada hora de teoría y 0,75 horas de estudio por cada hora de prácticas. (c)Las tutorías se encuentran incluidas en el total de Actividades Académicamente Dirigidas. Actividad Actividad Docente Materia Teoría Evaluación Profesor Alumno Procedimiento Exposición de la Teoría. Apoyo con audiovisuales Tomar apuntes, copiar el material audiovisual Tipo de preguntas. Se valorará razonamiento y capacidad de síntesis Clases en aula Clases en laboratorio Ejercicios y problemas Respuestas y soluciones Apuntes. Formulación de preguntas y dudas Problemas numéricos. Se valorarán razonamientos, unidades y convenios, resultados, lenguaje, etc. Prácticas de laboratorio Presentación de normas. Explicación de las prácticas Cuaderno de laboratorio, anotaciones, experimentos, ejercicios, informe, etc. Evaluación continuada, cuaderno, ejercicios, informes, actitud Peso en la nota final Horas presenciales Horas no presenciales Horas 24 h 36 h b 60 h 4 8 12h 12 h 9 21 h ECTS a 40 % 25% 64 Master en Biotecnología, Molecular Celular y Genética Actividades dirigidasc Distribución de los trabajos, recomendar bibliografía, orientar Seminarios. Exposición del seminario en el aula Se valorará contenido, exposición, presentación, y preparación, etc.. Curso 2009-2010 20% 4h 17 21 h Asignatura: AVANCES EN NEUROENDOCRINOLOGÍA Total de créditos ECTS: 4 (100 horas) Tipo de asignatura: Elegible Manuel Tena Sempere Leonor Pinilla Jurado (Responsable de la asignatura) Profesorado: Objetivos: - Conocer los principios básicos del control neuroendocrino de diversas funciones corporales - Conocer los sistemas neuronales (circuitos hipotalámicos y neuropéptidos) y señales periféricas involucrados en el control de la función reproductora - Conocer los principales factores hormonales implicados en el control de la ingesta y el metabolismo, con especial atención a señales recientemente identificadas - Saber interpretar elementos las interacciones periféricos fisiológicas (hormonas) y entre centrales (neuropéptidos) en el control neuroendocrino de diversas funciones corporales - Ser capaz de aplicar diversas técnicas analíticas y diseños experimentales al estudio de los mecanismos neuroendocrinos de control de la reproducción y el balance energético Programa PROGRAMA TEÓRICO (12 horas presenciales) - Tema 1: Aspectos generales del desarrollo y la función del eje reproductor - Duración: 3 horas - Fecha de impartición 4 de Mayo de 2009 - Horario: 16.00-19.00 horas 65 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 - Tema 2: Control integral de la ingesta - Duración: 3 horas - Fecha de impartición: 5 de Mayo de 2009 - Horario: 16.00-19.00 horas - Tema 3: Interacciones entre señales que informan del estado energético y funcionamiento del eje reproductor - Duración: 3 horas - Fecha de impartición: 6 de Mayo de 2009 - Horario: 16.00-19.00 horas - Tema 4: Control neuroendocrino de la reproducción: Sistema Kiss/GPR54 - Duración: 3 horas - Fecha de impartición: 7 de Mayo 2009 - Horario: 16.00-19.00 PROGRAMA PRÁCTICO (12 horas presenciales) - Tema 1: Introducción: Experimentación con animales y legislación al respecto. Tipos de animalarios. Importancia y necesidad de los Comités de Ética en experimentación - Tema 2: Experimentación animal y humana: Técnicas experimentales “in vivo” e “in vitro”. Recogida de datos - Tema 3: Manejo de la rata desde el nacimiento y condiciones de estabulación. Diferenciación sexual en el momento del nacimiento. Destete. Signos externos de pubertad en machos y hembras. Ciclo vaginal. Fertilidad, preñez y pseudopreñez - Tema 4: Administración de drogas: Administración sistémica (inhalatoria, oral, subcutánea, intraperitoneal, intravenosa), central (intracerebroventricular, en núcleos) o directa en órganos. Elección de vías de administración según el tipo de estudio - Tema 5: Disección de órganos endocrinos en la rata. Obtención de muestras biológicas. Almacenamiento de muestras según el tipo de señal a analizar - Tema 6: Cirugía endocrina en la rata: Ovariectomía, orquidectomía, adrenalectomía, pancreatectomía, pinealectomía, tiroidectomía e hipofisectomía. Cuidados postoperatorios 66 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 - Tema 7: Obtención de muestras de sangre en los animales de experimentación. Técnicas de obtención de sangre en roedores: Decapitación, punción yugular e intracardiaca y canulación yugular. Elección de la técnica según tipo de estudio a realizar. Tomas seriadas de sangre y estudios de pulsatilidad hormonal. Anestésicos: Tipos. Indicaciones y contraindicaciones según el tipo de estudio y las hormonas a analizar - Tema 8: Secreción hormonal “in vitro”: incubaciones estáticas de órganos y células dispersas y cultivos celulares. Medios de incubación. - Tema 9: Medida de hormonas en sangre y tejidos. Radioinmunoensayo de hormonas esteroideas y proteicas: Fundamentos teóricos. Marcaje de hormonas y rendimiento del mismo. Cálculo de la actividad específica. Titulación de anticuerpos. Curvas patrón y sensibilidad del ensayo. Variaciones intra e interensayo. Cálculo de los resultados. - Duración: 12 horas - Fechas de impartición: 11, 12 y 13 de Mayo de 2009 - Horario: 16.00-20.00 horas Bibliografía/referencias/enlaces - Interacting appetite-regulating pathways in the hypothalamic regulation of body weight. Kalra SP, Dube MG, Pu S, Xu B, Horvath TL, Kalra PS. Endocrine Reviews 20:68-100 (1999) - The timing of normal puberty and the age of sexual precocity: variations around the world, secular trends and changes after migration. Parent AS, Teilmann G, Juul A, Skakkebaek NE, Toppari J, Bourguignon JP. Endocrine Reviews 24:668-693 (2003) - Biological, physiological, pathological and pharmacological aspect of ghrelin. van der Lely AJ, Tschöp M, Heiman ML, Ghigo E. Endocrine Reviews 25:426-457 (2004) - Ghrelin and reproduction: a novel signal linking energy status and fertility. Barreiro ML, Tena-Sempere M. Molecular and Cellular Endocrinology 226:1-9 (2004) - Ghrelin in growth and development. Chanoine JP. Hormona research 63:119 (2005) 67 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 - GPR54 and kisspeptin in reproduction. Tena-Sempere M. Hum. Reprod. Uptake 12:631-639 (2006) - Role of ghrelin in reproduction. García MC, López M, Álvarez CV, Casanueva FF, Tena-Sempere M, Diéguez C. Reproduction 133:531-540 (2007) - New frontiers in kisspeptin/GPR54 physiology as fundamental gatekeepers of reproductive function. Roa J, Aguilar E, Diéguez C, Pinilla L, TenaSempere M. Frontiers in Neuroendocrinology 29:48-69 (2008) Distribución de horas presenciales: Ver cuadro adjunto - Indicar el carácter obligatorio o no de cada actividad presencial - Clases en el aula: Se valorará la asistencia, sin considerarse obligatoria - Clases en el laboratorio: Se considera obligatoria la asistencia a, al menos, un 65% de las actividades Criterios de evaluación: Ver cuadro adjunto Advertencias al alumnado: - Dado que en el curso se manipularán ratas, se desaconseja la matriculación en el mismo de personas que, por distintas causas (alergias, miedo, ideología….etc.) no puedan manipular las mismas. - Dado que del total de 24 horas presenciales, la asistencia a 8 horas de laboratorio asegura la posibilidad del examen, no se contemplan de inicio medidas alternativas. Los requerimientos para el examen de Septiembre serán similares a los de la convocatoria de Junio - Lugar de impartición: Facultad de Medicina. Campus Menéndez Pidal - En caso de ausencia de la Dra. Pinilla, ésta será sustituida por el Dr. TenaSempere y viceversa 68 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 69 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Asignatura: CULTIVOS CELULARES Total de créditos ECTS: 4 Tipo de asignatura: Elegible. Profesorado: Fco. Javier Alcaín Tejada Socorro García Navarro Rafael Manuel Vázquez Martínez Rafael Villalba Montoro* Objetivos: Conocer las técnicas de cultivos celulares y adquirir experiencia en la manipulación de células en cultivo. Se pretende que el alumno adquiera destreza en el laboratorio de cultivos celulares, particularmente en la esterilización del material, preparación de medios, manipulación estéril de líneas celulares y cultivos primarios. Que el alumno aprenda como se establecen, mantienen y conservan las líneas celulares; como se obtienen los hibridomas y los anticuerpos monoclonales. Que conozca y aprenda las técnicas de manipulación celular (transfección y silenciamiento génico) y las técnicas básicas de ingeniería tisular. Número de horas de trabajo del alumno: Nº de Horas en créditos ECTS: ...100..... Presenciales: 41 • Clases Teóricas: ...12....... • Clases Prácticas:.27...... • Examen final:……2 No presenciales: 59 Actividades en colaboración con el profesor: ..2........ • Tutorías individualizadas(presenciales / virtuales): 2 • Trabajo colaborativo en espacio virtual Actividades autónomas del alumnado: 57 • Realización de Actividades Académicas Dirigidas (memorias de prácticas): 10 • Horas de estudio de teoría: 18 • Estudio y elaboración de datos experimentales 20 • Preparación de Trabajos (memorias) : • Preparación de Exámenes: 9 Técnicas docentes Este curso se desarrollará a través de las actividades lectivas, prácticas y de evaluación, operando ya sea en grupos plenarios, en subgrupos o individualmente. Para ello se utilizarán el aula de teoría, el laboratorio y el aula virtual. En estos escenarios, se utilizarán como herramientas educativas: 70 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 a.- Unidades de estudio independiente, es decir, formas de estrategia y organización del estudio autónomas, propias de cada alumno o grupo de alumnos, para el tratamiento de un tema encomendado. b.- La participación en las actividades comunes, como espacio de reflexión, producción y puesta a prueba colectiva e individual de resultados parciales o finales de los ejercicios propuestos. Estas actividades incluirán asimismo prácticas de laboratorio y visitas a Servicios de la Universidad de Córdoba y al Banco de Tejidos y Unidad de Terapia Celular del Centro de Transfusión Sanguínea de Córdoba. c.- Lecturas dirigidas, bajo la forma de documentos propios y artículos científicos, orientados sobre la base del contenido del curso, proporcionados por el docente. d.- Sesiones de tutoría y orientación individual. e.- Docencia virtual mediante la utilización de recursos on-line y de la información contenida en un CD con la totalidad de los contenidos del curso Programa Teórico 1.- El laboratorio de cultivos celulares: Diseño y equipamiento. Diseño del laboratorio. Cabinas de seguridad microbiológica. Centrífugas. Incubadores. Superficies de trabajo y suelo. Material de plástico y desechable. Cuidado y mantenimiento del laboratorio. Requerimientos y medidas de seguridad del laboratorio de cultivos celulares. Salas blancas, estructura y diseño. Evaluación del riesgo. Desinfección. Eliminación de residuos. 2. El entorno del cultivo celular. Preparación, conservación y esterilización del material y reactivos comúnmente utilizado en el cultivo de células animales. Selección del sistema de cultivo (escala). Sistemas de cultivo alternativos. Cultivos celulares a gran escala: soluciones. Incubadores y botellas “roller”. Otros cultivos a escala. Constituyentes básicos del medio. Sales inorgánicas. Sistemas de tamponado. Carbohidratos. Vitaminas. Proteínas y péptidos. Ácidos grasos y lípidos. Oligoelementos. Suero. Normas para el uso del suero. Origen del suero. 3. Características y naturaleza del sustrato. .Factores de adhesión celular (colágeno, gelatina, fibronectina, laminina) y polímeros. Interacciones células– substrato:. Medios semisólidos: matrices. Biomateriales en Ingeniería Tisular: Principios básicos en el diseño de biomateriales y substratos. Biocompatibilidad y respuesta a biomateriales 4.- Cultivos primarios. Métodos de aislamiento y dispersión de tejidos y órganos. Separación celular. Cultivo en monocapa y cultivo en suspensión. Obtención de fibroblastos embrionarios de ratón (MEFs) para el cultivo de células madre. 5.- Líneas celulares. Subcultivo de las monocapas celulares. Cinética del crecimiento y mantenimiento de las células en cultivo. Establecimiento y mantenimiento de líneas celulares. Autentificación de líneas celulares. Criopreservacion y almacenamiento de líneas celulares. 71 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 6. Contaminaciones de los cultivos celulares. Principales contaminantes de los cultivos celulares. Origen de los contaminantes biológicos. Control de la contaminación. Protocolos de actuación ante la contaminación 7. Preparación y uso de hibridomas. Clonación de células por diluciones seriadas. Clonación de células en medio semisólido 8. Manipulación celular. Transfección de genes en células de mamíferos. Transfección estable. Protocolos básicos. Marcadores y medios selectivos: selección de células transfectadas resistentes a antibióticos. Silenciamiento génico: siRNA basado en vectores. Clonación del siRNA en vectores GenScript pRNA. 9. Ingeniería tisular. Generalidades: Introducción al estudio de la Ingeniería tisular. Fundamentos de la terapia basada en células. Normas de buenas prácticas de fabricación (GMPs). La Ingeniería Tisular en el marco normativo europeo. La legislación española del medicamento. Ensayos clínicos y biobancos. Donación de células y tejidos. Selección de donantes. Establecimientos de Tejidos (Conservación y criopreservación). Gestión de riesgos. Normas de seguridad en instalaciones criogénicas. 10. Células madre: concepto y tipos Investigación con células madres. Aplicaciones de la ingeniería tisular celular de los distintos sistemas orgánicos (sistema cardiovascular, nervioso, aparato locomotor, génitourinario, digestivo, piel) PROGRAMA PRÁCTICO 1. Preparación, conservación y esterilización del material y reactivos comúnmente utilizado en el cultivo de células animales. Manipulación en esterilidad. Recuento celular. Viabilidad celular. (4 horas) 2. Cultivo primario: Métodos de aislamiento y dispersión de tejidos y órganos. Obtención de dispersiones celulares a partir de hipófisis de rata. Separación de células hipofisarias de rata por centrifugación en gradiente de densidad de percoll. (6 horas) o Dispersión celular de corazones de embriones de pollo y cultivo en monocapa de cardiomiocitos (4 horas). 3. Líneas celulares. Cinética del crecimiento y mantenimiento de las células en cultivo. Subcultivo de las líneas celulares. Congelación y descongelación de las líneas. (3 horas) 4. Sincronización celular. Estimulación por factores de crecimiento del paso quiescencia-proliferación: estudio del ciclo celular. (3 horas) 5. Transfección celular. (4 horas) 6. Criopreservación y Bancos de Tejidos: Crioprotectores y curvas de congelación: (3 horas). 7. Ingeniería tisular: Manejo en salas blancas. Monitorización de condiciones ambientales. Cultivo sobre matriz para trasplante (4 horas) 72 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 ACTIVIDADES DIRIGIDAS (10 HORAS) 1. Diseño de un laboratorio de cultivos: equipamiento básico, distribución etc.. y medidas de seguridad del laboratorio de cultivos celulares. 2. Diseño de salas blancas y monitorización de condiciones ambientales para Ingeniería Tisular del trasplante en humanos. 3. Conocimiento de instalaciones criogénicas. Normas de seguridad Bibliografía GENERAL - Adams, R.L.P. Cell Culture for Biochemists. Elsevier. (1990). - Barnes, D.W., D.A. Sirbasku, G.H. Sato. Cell Culture Methods for Molecular and Cell Biology. Vol. 1: Methods for Preparation of Media, Supplements, and Substrata for Serum-Free Animal Culture. Alan R. Liss, Inc. (1984). - Barnes, D.W.; D.A. Sirbasku y G.H. Sato. Cell Culture Methods for Molecular and Cell Biology. Vol. 2: Methods for Serum Free Culture of Cells of the Endocrine System. Alan R. Liss, Inc. (1984). - Conn, P.M. Methods in Neurosciences. Vol. 2: Cell Culture. Academic Press, Inc. (1990). - Freshney, R.I. Culture of Animal Cell: A Manual of Basic Technique. IRL Press. (1983). - Freshney, R.I. Animal Cell Culture: A Practical Approach. IRL Press. (1986). - Jakoby, W.B. y I.H. Pastan. Methods in Enzymology. Vol. LVIII: Cell culture. Academic Press Inc. (1979). - Lydersen, B.K. Large Scale Cell Culture Technology. Hanser Publishers, (1987). - Pollard, J.W. y J.M. Walker. Methods in Molecular Biology. Vol. 6. Plant Cell and Tissue Culture. Human Press, (1990) - Vasil, I.K. Cell Culture and Somatic Cells Genetics of Plants. Vol 3: Plant Regeneration and Genetic Variability. Academic Press, Inc. (1986). Criterios de evaluación y seguimiento Los alumnos realizarán un examen teórico-práctico final. Además, presentarán una memoria del las actividades realizadas en las sesiones prácticas. Para la calificación final se contempla la siguiente distribución de puntuación y método de seguimiento: • 60% Conocimientos adquiridos: EXAMEN FINAL • 40%. Memorias de prácticas y actividades relacionadas (espacio virtual).EVALUACION DEL PROFESOR, SEGUIMIENTO EN TUTORIA Y/O ESPACIO WEB Advertencias: Dado el carácter fundamentalmente práctico del contenido de la asignatura, es obligatoria la asistencia a las clases teóricas y prácticas y la presentación al examen final como medida del aprovechamiento y consecución de los objetivos. Al inicio del curso, el alumno que no pueda asistir a alguna(s) de las sesiones presenciales, por los motivos que sean, debe anunciarlo y justificarlo 73 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 debidamente. De esta forma se consensuará previamente con el alumno la forma de recuperación de las actividades obligatorias. En casos de enfermedad o imprevistos sin posibilidad de programación previa, será el profesor(es) responsable(s) quien establecerá el criterio de evaluación, y la posibilidad de recuperación de las faltas e asistencia. Distribución ECTS de la Asignatura CULTIVOS CELULARES (a) 1 ECTS = 25 horas trabajo. (b) Estimación de estudio personal del alumno durante el curso 12 (trimestral) o 36 (anual) semanas: 1,5 horas de estudio por cada hora de teoría y 0,75 horas de estudio por cada hora de prácticas. Actividad Actividad Docente Clases en aula Clases en laboratorio Actividades dirigidas Tutorias Exámenes Evaluación Profesor Alumno Procedimiento Peso en la nota final Teoría Exposición de la Teoría. Apoyo con audiovisuales Tomar apuntes, comprender los conceptos apoyados en el material audiovisual.Participar. Examen final tipo test. 60 % 12h 18h 30 h Prácticas de laboratorio Presentación/ Explicación de métodos y técnicas. Entrega de documentación y guias para el desarrollo experimental Anotaciones detalladas en , cuaderno de laboratorio.Realización de experimentos, Obtención de resultados. Evaluación continuada de asistencia, destreza, actitud, resolucion de problemas. 25% 27h 20 47h Realización y Entrega de documentos. Se valorará: la forma y contenido de la presentación, el razonamiento y la capacidad de síntesis. 15% 10h 10h Teoría y prácticas Se trata de Trabajo Colaborativo (todo el grupo, profesores y alumnos) Discusión de las materias teórico-práctica de modo global y otros temas de interés a alumnos Resolver cuestiones y participar en la pagina web de la asignatuta Es una actividad no evaluable, sólo de comunicación - 2 2h Teoría y/o problemas Poner, vigilar y corregir el examen. Evaluar el aprendizaje y rendimiento del alumno 2h 9h 11 h 41h 59 h 100h Materia Tareas autónomas. Memorias de Prácticas y Actividades relacionadas. Establecimiento de criterios, modelos, formato para la presentación , Orientar, aclarar dudas en tutoria. Preparación y Realización de examen TOTAL CARGA DOCENTE DEL ALUMNO 100% Horas presenciales Horas no presenciales Horas 74 ECTS a Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 4. ITINERARIO 3: ANÁLISIS GENÉTICO EN BIOTECNOLOGÍA 75 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Asignatura: TRANSFORMACIÓN APLICADA A LA MEJORA VEGETAL Total de créditos ECTS: 4 Tipo de asignatura: Elegible. Profesorado: Francisco Barro Losada Juan Gil Ligero Objetivos: Proporcionar los conocimientos básicos de ingeniería genética (IG). Aplicación de la IG a la Mejora de cereales y leguminosas. Conocer los sistemas de transformación genética de plantas. Cómo desarrollar e implementar un protocolo de transformación genética. Programa Teórico 1. Introducción. 2. Sistemas de transformación 2.1. Agrobacterium 2.2. Transferencia Directa de Genes 2.2.1. Electroporación de tejidos 2.2.2. Transformación de protoplastos 2.2.3. Microinyección/microfibrillas 2.2.4. Bombardeo con micropartículas 3. Vectores para la transferencia de genes 3.1. Promotores 4. Sistemas de selección 4.1. Selección positiva 4.2. Selección negativa 5. Genes marcadores 6. Aplicaciones agrícolas 6.1. Mejora para la resistencia a herbicidas 6.2. Resistencia a enfermedades y plagas 6.3. Mejora de la calidad Práctico 1. 2. 3. 4. Aislamiento de explantes y cultivo in vitr. Práctica transformación mediante bombardeo de partículas. Cuantificación de la expresión de transgenes. Análisis estadístico. 76 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Bibliografía/referencias/enlaces Aldemita RR, Hodges TK (1996) Agrobacterium tumefaciens-mediated transformation of japonica and indica rice varieties. Planta 199:612-617 Altpeter F, Baisakh N, Beachy R, Bock R, Capell T, Christou P, Daniell H, Datta K, Datta S, Dix PJ, Fauquet C, Huang N, Kohli A, Mooibroek H, Nicholson L, Nguyen TT, Nugent G, Raemakers K, Romano A, Somers DA, Stoger E, Taylor N, Visser R (2005) Particle bombardment and the genetic enhancement of crops: myths and realities. Mol Breed 15:305-327 Altpeter F, Vasil V, Srivastava V, Vasil IK (1996) Integration and expression of the high-molecular-weight glutenin subunit 1Ax1 gene into wheat. Nat Biotechnol 14:1155-1159 Barro F, Barcelo P, Lazzeri PA, Shewry PR, Martín A, Ballesteros J (2002) Field evaluation and agronomic performance of transgenic wheat. Theor Appl Genet 105:980-984 Barro F, Cannell ME, Lazzeri PA, Barcelo P (1998) The influence of auxins on transformation of wheat and tritordeum and analysis of transgene integration patterns in transformants. Theor Appl Genet 97:684-695 Barro F, Rooke L, Bekes F, Gras P, Tatham AS, Fido R, Lazzeri PA, Shewry PR, Barcelo P (1997) Transformation of wheat with high-molecular-weight subunit genes results in improved functional-properties. Nat Biotechnol 15:1295-1299 Dai SH, Zheng P, Marmey P, Zhang SP, Tian WZ, Chen SY, Beachy RN, Fauquet C (2001) Comparative analysis of transgenic rice plants obtained by Agrobacterium-mediated transformation and particle bombardment. Mol Breed 7:25-33 Travella S, Ross SM, Harden J, Everett C, Snape JW, Harwood WA (2005) A comparison of transgenic barley lines produced by particle bombardment and Agrobacterium-mediated techniques. Plant Cell Rep 23:780 Wu H, Sparks C, Amoah B, Jones HD (2003) Factors influencing successful Agrobacterium-mediated genetic transformation of wheat. Plant Cell Rep 21:659-668 Criterios de evaluación Se valorará la asistencia y participación en las clases teóricas, la asistencia y resultados obtenidos en los trabajos prácticos, así como el trabajo de revisión presentado. Los alumnos que no cumplan con la asistencia a las actividades obligatorias del Curso no podrán superar la asignatura en la convocatoria de Junio. Para estos alumnos el examen de septiembre consistirá en una prueba escrita sobre cuestiones teóricas y prácticas del programa de la asignatura. 77 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Distribución ECTS de la Asignatura TRANSFORMACION APLICADA A LA MEJORA VEGETAL (a) 1 ECTS = 25 horas trabajo. (b) Estimación aproximada de estudio personal del alumno durante el curso 12 (trimestral) o 36 (anual) semanas: 1,5 horas de estudio por cada hora de teoría y 0,75 horas de estudio por cada hora de prácticas. (c)Las tutorías se encuentran incluidas en el total de Actividades Académicamente Dirigidas. Actividad Actividad Docente Materia Evaluación Alumno Procedimiento Peso en la nota final Horas presenciales Horas no presenciales Horas Profesor Prestar atención, tomar notas, enterarse, preguntar si no se entera Se valorará razonamiento y capacidad de síntesis 35 % 15 h 25 h 40 h ECTS a Clases en aula Teoría Exposición de la Teoría. Apoyo con audiovisuales Clases en laboratorio Prácticas de laboratorio Explicación de las prácticas Anotaciones, experimentos, ejercicios, estadística. Evaluación continuada, ejercicios, actitud 40% 20 h 20 h 40 h Trabajos tutorizados. Seminarios Distribución de los trabajos, recomendar bibliografía, orientar Recoger información, analizarla, obtener conclusiones, redactar y exponer el trabajo Se valorará bibliografía utilizada, contenidos y presentación 25% 5h 15 h b 20 h 40 h 60 h 100 h Actividades dirigidasc TOTAL CARGA DOCENTE DEL ALUMNO Asignatura: MECANISMOS DE PATOGENÉSIS Y RESISTENCIA EN LAS ENFERMEDADES DE LAS PLANTAS Total de créditos ECTS: 4 Tipo de asignatura: Elegible. Profesorado: Antonio Di Pietro David Turrà Alejandro Pérez Sara Fondevilla Objetivos: En el curso se dan a conocer a los alumnos los principios básicos que determinan las enfermedades en plantas. Las clases teóricas y prácticas se centran tanto en los mecanismos de infección de los patógenos, como en la respuesta de defensa y en las bases de la resistencia en plantas. Se explica la aplicación de estos conocimientos a la mejora genética de plantas. 78 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Programa: Primera parte (Genética-UCO) Programa teórico - Mecanismos de patogénesis - Señalización y regulacion de los mecanismos de infección - Comunicación patógeno-planta Programa práctico - Inoculación de plantas con cepas de Fusarium oxysporum - Observación microscópica de F. oxysporum expresando GFP - Detección de mutantes de F. oxysporum obtenidos por mutación dirigida. Segunda parte (IAS-CSIC) Programa teórico - Señalización y regulación de mecanismos de defensa - Principios de la mejora genética por resistencia a enfermedades Programa práctico - Estudio de macro- y microscópico de mecanismos de resistencia a royas y oidios en cereales y leguminosas. - Evaluación de resistencia a hongos necrotrofos en cereales y leguminosas. - Estudio de componentes de resistencia a plantas parásitas en leguminosas Bibliografía/referencias/enlaces Di Pietro, A., Madrid, M.P., Caracuel, Z., Delgado-Jarana, J., Roncero, M.I.G. (2003) Fusarium oxysporum: exploring the molecular arsenal of a vascular wilt fungus. Mol. Plant Pathol. 4:315-326. Dita, M.A., N. Rispail, E. Prats, D. Rubiales & K.B. Singh, 2006. Biotechnology approaches to overcome biotic and abiotic stress constraints in legumes. Euphytica 147(1-2): 1-24. Gold, S.E., García-Pedrajas, M.D., Martínez-Espinoza, A.D. (2001) New (and used) approaches to the study of fungal pathogenicity. Annu. Rev. Phytopathol. 39: 337-365 Jones, J.D.G., Dangl, J.L. (2006) The plant immune system. Nature 444:323329. Lengeler, K.B. et al. (2000) Signal transduction cascades regulating fungal development and virulence. Microbiol. Mol. Biol. Rev. 64:746-785. Sillero, J.C., S. Fondevilla, J. Davidson, M.C. Vaz Patto, T.D. Warkentin, J. Thomas & D. Rubiales, 2006. Screening techniques and sources of resistance to rusts and mildews in grain legumes. Euphytica 147(1-2): 255-272. 79 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Rispail N., M.A. Dita, C. González-Verdejo, A. Pérez-de-Luque, M.A. Castillejo, E. Prats, B. Román, J. Jorrín & D. Rubiales, 2007. Plant Resistance to Parasitic Plants: Current Approaches for an old foe. New Phytologist 173: 703-712. Distribución de horas presenciales: VER TABLA ADJUNTA Criterios de evaluación: Evaluación continua (100% presenciales) En caso de más de 15 alumnos se desdoblará el grupo. En caso mayor de ausencia, las Dras Mª Angeles Castillejo y Mónica Fernández-Aparicio sustituirán a los profesores del CSIC. - En caso de fuerza mayor y por ausencia excepcional y justificada de algún alumno se compensarían parte de las clases perdidas con un trabajo de revisión. En caso de no asistencia a ninguna clase se exigirá un periodo de colaboración práctica en el laboratorio o un trabajo de revisión, además de la elaboración de un informe y/o la realización de un examen. - Los lugares de impartición de las clases teóricas y prácticas serán los reservados en el Edificio Gregor Mendel (Campus de Rabanales) y en el IAS-CSIC. - fechas horarios 20/04 , de 16:00 a 22/04, 20:00 23/04 27/04-30/04 Lugar clases Laboratorio de Práctica, Edificio Gregor Mendel Rabanales IAS, CSIC Profesores Antonio Di Pietro X1583221Q créditos 2 Alejandro Pérez de Luque 1 30805160H Sara Fondevilla Aparicio 1 44222867P Distribución ECTS de la Asignatura MECANISMOS DE PATOGENÉSIS Y RESISTENCIA EN LAS ENFERMEDADES DE LAS PLANTAS (a) 1 ECTS = 25 horas trabajo. (b) Estimación de estudio personal del alumno durante el curso 12 (trimestral) o 36 (anual) semanas: 1,5 horas de estudio por cada hora de teoría y 0,75 horas de estudio por cada hora de prácticas. (c)Las tutorías se encuentran incluidas en el total de Actividades Académicamente Dirigidas. Actividad Actividad Docente Materia Evaluación Alumno Procedimiento Peso en la nota final Horas presenciales Horas no presenciales Horas Profesor Tomar apuntes, copiar el material audiovisual Tipo de preguntas. Se valorará razonamiento y capacidad de síntesis 50 % 12 h 30 h b 42 ECTS a Clases en aula Teoría Exposición de la Teoría. Apoyo con audiovisuales Clases en laboratorio Prácticas de laboratorio Presentación de normas. Explicación de las prácticas Cuaderno de laboratorio, anotaciones, experimentos, ejercicios, informe, etc. Evaluación continuada, cuaderno, ejercicios, informes, actitud 50% 20 h 26 h 46 Tutorias Teoría y prácticas Discusión de las materias teórico- Preparar cuestiones y participar en Es una actividad no evaluable - 8 4 12 80 Con formato: Numeración y viñetas Master en Biotecnología, Molecular Celular y Genética práctica de modo global y otros temas de interés a alumnos Curso 2009-2010 la pagina web de la asignatuta TOTAL CARGA DOCENTE DEL ALUMNO 40 h 60 h 100 Asignatura: RECURSOS FITOGENÉTICOS Y EVOLUCIÓN DE PLANTAS CULTIVADAS Total de créditos ECTS: 4 Tipo de asignatura: Elegible. Profesorado: Luís Miguel Martín Martín Juan Bautista Álvarez Cabello Objetivos: Comprender cómo se han generado los recursos genéticos a partir de la evolución que han tenido las plantas cultivadas, y las repercusiones económicas y sociales de todo el proceso. Metodología: NÚMERO DE HORAS DE TRABAJO DEL ALUMNO: Nº de Horas/ Créditos ECTS: ........100,0 horas/ 4.0 ECTS Clases Teóricas: ......... 20.0 h Clases Prácticas: ..........8.0 h Actividades en colaboración con el profesor: Exposiciones y Seminarios…8.0 h Tutorías especializadas colectivas (presenciales o virtuales): 4.0 h Actividades autónomas del alumnado: Horas de estudio y preparación de trabajos… 50.0 h Tutorías especializadas individuales (presenciales o virtuales). 4.0 h Realización de Exámenes:… 4.0 h PROGRAMA: Tema 1. Sociedades preagrícolas. Tema 2. El origen de la Agricultura. Tema 3. Genética de poblaciones vegetales. Tema 4. El concepto de planta cultivada. Tema 5. El concepto de mala hierba. Tema 6. La taxonomía de las plantas cultivadas. Tema 7. La dinámica de la domesticación. Tema 8. La variación en el espacio y en el tiempo. Tema 9. El Centro del Oriente Medio. Tema 10. El Centro Africano. Tema 11. El Centro Chino. Tema 12. El Centro del Sur de Asia y Oceanía. 81 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Tema 13. Los Centros Americanos. Tema 14. La domesticación en la actualidad. Tema 15. Evolución de plantas cultivadas y recursos genéticos. Bibliografía general: Cubero, J.I. 2002. Introducción a la Mejora Genética Vegetal. Mundi-Prensa. 2ª edición. Harlan, J. R. 1992. Crop and Man. American Society of Agronomy, Inc. Crop Science Society of America. Inc Madison. Winscosin. USA. Con cada tema se entregará un guión detallado y una relación bibliográfica específica. TÉCNICAS DOCENTES: Sesiones académicas teóricas X Sesiones académicas prácticas X Exposición y debate: X Visitas y excursiones: Tutorías especializadas: X Otros (especificar): Conferencias de profesionales de la Mejora de Plantas. Asignatura: MANIPULACIÓN CROMOSÓMICA EN PLANTAS Total de créditos ECTS: 4 Tipo de asignatura: Elegible. Profesorado: Adoración Cabrera Caballero, Dpto. Genética, UCO Antonio Martín Muñoz, IAS, CSIC Pilar Prieto Aranda, IAS, CSIC Programa de la asignatura 1. Introducción 1.1. La paradoja del valor C. 1.2. Estructura del cromosoma eucarióntico. Sub y microscópica. 1.3. El cromosoma en mitosis y meiosis. 1.4. Bloques cromosómicos 1.5. Evolución genómica. NOR en algodón. El impulso molecular. 1.6. Importancia de la poliploidía en la Naturaleza y en la Mejora Genética Vegetal. 2. Cambios numéricos y estructurales. Poliploidía y aneuploidía 2.1. Cambios numéricos. Nomenclatura, producción e identificación, comportamiento meiótico, fertilidad y uso. 2.1.1. Diploide.Euploide 2.1.2. Aneuploides 2.1.3. Poliploides 2.1.3.1. Autoploides 82 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 2.1.3.2. Aloploides. 2.1.4. Haploides. 2.2. Cambios estructurales. Efectos genéticos. 2.2.1. Inversiones. Para y pericéntricas 2.2.2. Traslocaciones 3. Híbridos interespecíficos y anfiploides. 3.1. Ideas generales 3.2. Relaciones evolutivas en las gramíneas 3.2.1. Homología y homeología 3.2.2. Control de apareamiento homeólogo 3.2.2.1.Caracterización de mutantes de apareamiento (mutantes Ph) 3.2.2.2.Aplicación en mejora genética vegetal como herramienta para la transferencia de genes de interés entre especies relacionada 3.2.3. Manipulación cromosómica. 3.2.3.1. Heterosis y nuevas especies en las gramíneas 3.2.3.2. Triticale y tritórdeo 3.4. Androesterilidad e híbridos interespecíficos. 3.4.1. Utilización del híbrido- anfiploide para la obtención de líneas aloplásmicas. 3.4.2. Uso de la hibridación somática para la manipulación de los genomas citoplásmicos. Tema 4. Hibridación In situ en plantas: Métodos 4.1. Hibridación in situ Fluorescente (FISH) 4.1.1. Sondas de ADN. Aislamiento, tipos y preparación de sondas. 4.1.1.1. ADN genómico 4.1.1.2. Secuencias de ADN repetido de función conocida Secuencias teloméricas Secuencias centroméricas Secuencias de genes ribosómicos (NORs) 4.1.1.3. Secuencias de ADN repetido de función desconocida 4.1.1.4. BACs y YACs 4.1.1.5. Sondas obtenidas por microdisección 4.1.1.6. Sondas obtenidas por citometría de flujo 4.1.2. Marcaje de sondas. Métodos de marcaje. 4.1.3. Preparaciones cromosómicas: mitóticas y meióticas. 4.1.4. Hibridación in situ DNA:DNA 4.1.5. Detección de la hibridación. Análisis de señales fluorescentes. 4.2. PRINS 4.3. FISH Multicolor 4.4. Fibra de cromatina extendida 4.5. Q-FISH: Análisis de telómeros Tema 5. Hibridación In situ en plantas: Aplicaciones 5.1. Identificación genómica 5.2. Identificación cromosómica. 5.3. Estudio de la composición genómica de híbridos interespecíficos y especies aloploides. 5.4. Estudio del apareamiento cromosómico meiótico 5.5. Análisis de cambios evolutivos cromosómicos: numéricos y estructurales 83 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 5.6. Detección de introgresiones. 5.7. Estudio de la arquitectura molecular de los cromosomas 5.8. Organización cromosómica y función de la cromatina. Compartimentación nuclear. Dominios cromosómicos 5.9. Mapeo físico mediante FISH. Secuencias repetidas y de copia única. 5.10. Estudios de homeología 5.11. Localización de transgenes 5.12. Estudios de expresión génica. Hibridación in situ con sondas de ARN. Marcaje y localización de proteínas. Uso de la microscopía confocal para experimentos de expresión in vivo (FLIP y FRAP). Tema 6. Utilización de los cambios cromosómicos númericos y estructurales en la localización y mapeo de genes 6.1. Utilización de aneuploides: 6.1.1. Trisómicos, monosómicos y nulisómicos 6.2. Líneas de adición, sustitución y líneas ditelosómicas 6.3. Mapeo físico mediante líneas de translocación y deleción. 6.4. Correlación entre distancias genéticas y físicas PRÁCTICAS 1. 2. 3. 4. 5. Visualización de cromosomas mitóticos Visualización de cromosomas meióticos Cruzamientos Rescate de embriones inmaduros Visualización de preparaciones hibridadas con sondas fluorescentes. Asignatura: EPIGENÉTICA Total de créditos ECTS: 4 Tipo de asignatura: Elegible. Profesorado: Rafael Rodríguez Ariza María Teresa Roldán Arjona María Teresa Morales Ruiz Dolores Córdoba Cañero Objetivos: - - Entender el concepto de epigenética y su campo de estudio Conocer los principales tipos de marcas epigenéticas y los mecanismos moleculares responsables de su establecimiento, mantenimiento y modificación. Familiarizarse con los métodos experimentales empleados para analizar modificaciones epigenéticas Entender el papel que desempeñan los procesos epigenéticos en distintos aspectos de los ciclos vitales de los organismos. Programa: 84 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Programa de clases teóricas: 1. Introducción a la Epigenética Genética y epigenética. Definición de epigenética. Fenómenos epigenéticos. La cromatina. Tipos de marcas epigenéticas. PARTE 1. MECANISMOS MOLECULARES 2. Cromatina: estructura y función La estructura básica de la cromatina: el nucleosoma. La fibra de 30 nm. Estructura de la cromatina y actividad génica. Cambios en el empaquetamiento del ADN durante el ciclo celular 3. Metilación de ADN Metiltransferasas. Patrones de metilación en animales y plantas. Funciones biológicas de la metilación. Metilación y silenciamiento génico transcripcional. Efectos de la metilación sobre los genomas 4. Modificaciones en la estructura y composición de la cromatina Modificaciones covalentes de las histonas: el código de histonas. Incorporación de variantes histónicas. Remodelado de la cromatina. 5. El papel de del ARN en el control de las modificaciones epigenéticas La interferencia mediada por ARN (RNAi) y silenciamiento a nivel postranscripcional. El papel del RNAi en la formación de heterocromatina. Metilación de ADN dirigida por ARN. PARTE 2. PROCESOS EPIGENÉTICOS 6. Silenciamiento de transposones, genes y cromosomas Silenciamiento de elementos transponibles. Impronta parental. Inactivación del cromosoma X. 7. Cambios epigenéticos durante el ciclo vital de animales y plantas Estabilidad y flexibilidad de la regulación epigenética durante el desarrollo en mamíferos. Reprogramación epigenética. 8. Epigenética y enfermedades humanas Enfermedades causadas por defectos en impronta. Enfermedades causadas por defectos en la maquinaria epigenética. Cáncer y epigenética. Medio ambiente y epigenética. Programa de clases prácticas: Práctica 1. Análisis del estado de metilación de una secuencia de ADN mediante tratamiento con McrBC. Práctica 2. Análisis del estado de metilación de una secuencia de ADN mediante inmunoprecipitación de ADN metilado (MeDIP). Práctica 3. Utilización de ADN glicosilasas para detectar la presencia de 5-metilcitosina en el ADN Bibliografía: 85 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Allis, C. D., Jenuwein, T., and Reinberg, D. (2007). Epigenetics (Cold Spring Harbor, N.Y., Cold Spring Harbor Laboratory Press). Turner, B. M. (2001). Chromatin and gene regulation: mechanisms in epigenetics (Oxford ; Malden, MA, Blackwell Science). Schones, D. E., and Zhao, K. (2008). Genome-wide approaches to studying chromatin modifications. Nat Rev Genet 9, 179-191. Reik, W. (2007). Stability and flexibility of epigenetic gene regulation in mammalian development. Nature 447, 425-432. Kouzarides, T. (2007). Chromatin modifications and their function. Cell 128, 693-705. Jiang, Y. H., Bressler, J., and Beaudet, A. L. (2004). Epigenetics and human disease. Annu Rev Genomics Hum Genet 5, 479-510. Jones, P. A., and Baylin, S. B. (2007). The epigenomics of cancer. Cell 128, 683-692. Distribución de horas presenciales y no presenciales: Ver cuadro adjunto Todas las actividades presenciales son obligatorias. Será requisito indispensable la asistencia a un mínimo de un 80 % de las clases teóricas y prácticas. Criterios de evaluación: Ver en al cuadro adjunto el peso que se da a cada una de las actividades presenciales y no presenciales para la calificación final. Advertencias al alumno: - En caso de que, por causas de fuerza mayor y por ausencia excepcional y debidamente justificada, algún alumno no cumpla con la asistencia a las actividades obligatorias del curso, se dará la opción de recuperar el 80% de las actividades/clases perdidas en horario y lugar previamente acordado con los profesores de la asignatura. - En caso de no superar la asignatura en la convocatoria de junio se exigirá la repetición de las memorias correspondientes a todas las actividades prácticas y su entrega en septiembre para repetir la evaluación. 86 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Otras consideraciones: 1. En caso de ausencia forzada de alguno de los tres profesores, será sustituido por alguno de los dos restantes. 2. Si el número de alumnos es superior a 20, se establecerán dos grupos para la realización de las clases prácticas. 3. Los lugares de impartición de las clases teóricas y prácticas serán los reservados en el campus de Rabanales, oportunamente anunciados en la página web de posgrado (http://www.uco.es/estudios/postgrado/). Distribución de horas presenciales y no presenciales. Criterios de evaluación: Distribución ECTS de la Asignatura EPIGENÉTICA (a) 1 ECTS = 25 horas trabajo. (b) Estimación de estudio personal del alumno durante el curso 12 (trimestral) o 36 (anual) semanas: 1,5 horas de estudio por cada hora de teoría y 0,75 horas de estudio por cada hora de prácticas. Actividad Actividad Docente Clases en aula Alumno Procedimiento Peso en la nota final Horas presenciales Horas no presencialesb Horas Profesor Teoría Exposición de la Teoría con apoyo de medios audiovisuales Planteamiento de cuestiones. Toma de apuntes, planteamiento de cuestiones; respuesta a cuestiones planteadas por el profesor Tipo de cuestiones planteadas por el alumno. Respuesta a cuestiones planteadas por el profesor. Se valorará razonamiento y capacidad de síntesis 20 % 8h 12 h 20 h Prácticas de laboratorio Presentación de normas. Explicación de las prácticas Realización de experimentos, recogida de datos, Evaluación continuada, cuaderno de datos experimentales, actitud 20% 24 h 18 h 42 h Análisis de resultados Explicación de métodos para el análisis de datos experimentales Análisis de resultados; extracción de conclusiones; elaboración de una memoria para cada práctica Evaluación continuada; memoria de prácticas. Se valorará razonamiento y capacidad de análisis 60 % 6 30 36 h Teoría y prácticas Orienta y resuelve dudas Preparar cuestiones y solicitar orientación personalizada Es una actividad no evaluable - 2h 0h 2h 100 % 40 h 60 h 100 h Materia Clases en laboratorio Tutorías Evaluación TOTAL CARGA DOCENTE DEL ALUMNO ECTS a 87 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Asignatura: GENÉTICA DEL COMPORTAMIENTO Total de créditos ECTS: 4 Tipo de asignatura: Elegible. Profesorado: Encarna Alejandre Durán (2 créditos) Manuel Ruiz Rubio (2 créditos). Objetivos: Dar a conocer a los alumnos los principios básicos de la función de los genes en el comportamiento. Profundizar en las bases moleculares de las enfermedades del comportamiento y aplicar dichos conocimientos para entender el comportamiento humano. Programa Teórico: Introducción a la genética del comportamiento. Enfermedades humanas que afectan al comportamiento: Genética del autismo Genética del trastorno de hiperactividad (TDAH) Genética del síndrome X-frágil Variación alélica en las poblaciones humanas y su relación con el comportamiento: Polimorfismo en el gen MAOA y su relación con el maltrato. Sistemas modelos para el estudio del comportamiento: Base molecular del comportamiento social en Caenorhabditis elegans El comportamiento y su relación con los genes, el ambiente y epigenética Bases moleculares del comportamiento emocional. Seminarios: ANOREXIA ALZHEIMER ESQUIZOFRENIA SINDROME DE RETT DEPRESION TRASTORNO BIPOLAR ADICCION AL TABACO ALCOHOLISMO Síndrome de ANGELMAN y PRADER-WILI (Levinson 2006; Haile, Kosten et al. 2007; Hunt 2007; Kas, Fernandes et al. 2007; Levitt and Manson 2007; Schnoll, Johnson et al. 2007; Tsankova, Renthal et al. 2007; Voracek and Loibl 2007; Mineur and Picciotto 2008; Moy and Nadler 2008) 88 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Programa Práctico: - Extracción de ADN a partir de muestras de sangre y estudio de polimorfismo en los genes DRD4 (receptor de la dopamina) y 5HTT (transportador de la serotonina) en humanos. - Observación y análisis del comportamiento social de C. elegans. Bibliografía: Libros Reilly P R (2004) Is it in your genes? Cold Spring Harbor Laboratoty Press. New York Novo Villaverde F J (2007) Genética Humana. Perrazo Prentice Hall. Khoury M J, Little J, Burke W (2004) Human Genome Epidemiology. Oxfor University Press Jimenez Escrig A (2003) Manual de Neurogenética. Díaz de Santos, Madrid Plomin R., DeFries J.C., McClearn G.E. y McGuffin P (2000) Behavioral Genetics. Worth Publishers, New York Faraone S.V., Tsuang M.T. y Tsuang D. W. (1999) Genetics of mental disorders. Guilford Press, New York Revisiones Haile, C. N., T. R. Kosten, et al. (2007). "Genetics of dopamine and its contribution to cocaine addiction." Behav Genet 37(1): 119-45. Hunt, G. J. (2007). "Flight and fight: a comparative view of the neurophysiology and genetics of honey bee defensive behavior." J Insect Physiol 53(5): 399-410. Kas, M. J., C. Fernandes, et al. (2007). "Genetics of behavioural domains across the neuropsychiatric spectrum; of mice and men." Mol Psychiatry 12(4): 324-30. Levinson, D. F. (2006). "The genetics of depression: a review." Biol Psychiatry 60(2): 84-92. Levitt, M. and N. Manson (2007). "My genes made me do it? The implications of behavioural genetics for responsibility and blame." Health Care Anal 15(1): 33-40. Mineur, Y. S. and M. R. Picciotto (2008). "Genetics of nicotinic acetylcholine receptors: Relevance to nicotine addiction." Biochem Pharmacol 75(1): 323-33. Moy, S. S. and J. J. Nadler (2008). "Advances in behavioral genetics: mouse models of autism." Mol Psychiatry 13(1): 4-26. Schnoll, R. A., T. A. Johnson, et al. (2007). "Genetics and smoking behavior." Curr Psychiatry Rep 9(5): 349-57. Tsankova, N., W. Renthal, et al. (2007). "Epigenetic regulation in psychiatric disorders." Nat Rev Neurosci 8(5): 355-67. Voracek, M. and L. M. Loibl (2007). "Genetics of suicide: a systematic review of twin studies." Wien Klin Wochenschr 119(15-16): 463-75. 89 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Enlaces http://www.ncbi.nlm.nih.gov/projects/gensat/ http://neuroscienceupdate.cumc.columbia.edu/index.html http://www.bbc.co.uk/radio4/reith2003/lecturer.shtml http://www.sciammind.com/ Distribución de horas presenciales: VER CUADRO ADJUNTO Todas las actividades son obligatorias. Criterios de evaluación Asistencia 30% Exposición del trabajo 30% Resolución de cuestiones individualmente 30% Participación 10% Advertencias al alumno EN CASO DE NO ASISTENCIA JUSTIFICADA, EL ALUMNO TENDRÍA QUE REALIZAR LOS CUESTIONARIOS DE EVALUACIÓN DEL CURSO, YA SEA EN LA CONVOCATORIA DE JUNIO COMO EN LA DE SEPTIEMBRE. Otras consideraciones: - En caso de fuerza mayor cualquiera de los profesores se sustituirían mutuamente. - El curso se impartirá en el edificio C-5 90 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Distribución ECTS de la Asignatura: GENÉTICA DEL COMPORTAMIENTO (a) 1 ECTS = 25 horas trabajo. (b) Estimación de estudio personal del alumno durante el curso 12 (trimestral) o 36 (anual) semanas: 1,5 horas de estudio por cada hora de teoría y 0,75 horas de estudio por cada hora de prácticas. (c)Las tutorías se encuentran incluidas en el total de Actividades Académicamente Dirigidas. Actividad Actividad Docente Materia Teoría Profesor Alumno Procedimiento Exposición de la Teoría. Apoyo con audiovisuales Tomar apuntes, copiar el material audiovisual Tipo de preguntas. Se valorará razonamiento y capacidad de síntesis Clases en aula Práctica en laboratorio Actividades dirigidasc Tutorias Exámenes Evaluación Peso en la nota final Horas presenciales Horas no presenciales Horas 12 h 18 h 30 h 8h 12 h 20 h ECTS a Ejercicios y problemas Respuestas y soluciones Apuntes. Formulación de preguntas y dudas Problemas numéricos. Se valorarán razonamientos, unidades y convenios, resultados, lenguaje, etc. Prácticas de laboratorio Presentación de normas. Explicación de las prácticas Cuaderno de laboratorio, anotaciones, experimentos, ejercicios, informe, etc. Evaluación continuada, cuaderno, ejercicios, informes, actitud 20 % 10 h 7 h 17 h Seminarios. Distribución de los trabajos, recomendar bibliografía, orientar Exposición del seminario en el aula Se valorará contenido, exposición, presentación, y preparación, etc.. 21 % 3h 15 h 18 h Teoría y prácticas Discusión de las materias teóricopráctica de modo global y otros temas de interés a alumnos Preparar cuestiones y participar en la pagina web de la asignatuta Es una actividad no evaluable - 1h 1h 2h Teoría y/o problemas Poner, vigilar y corregir el examen. Calificar globalmente al alumno Preparación de examen (25 horas) Realización de examen (2 horas) 3h 10 h 13 h 37 h 63 h 100 h TOTAL CARGA DOCENTE DEL ALUMNO 59 % 85 % 91 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Asignatura: MARCADORES MOLECULARES Y SU USO EN MEJORA GENÉTICA Total de créditos ECTS: 4 Tipo de asignatura: Elegible. Profesorado: Luis Morera Sanz Juan José Garrido Pavón Manuel Barbancho Medina Teresa Millán Valenzuela Objetivos: Formación de los alumnos en aspectos teóricos y prácticos relacionados con los marcadores moleculares y sus aplicaciones en la mejora genética animal y vegetal. Programa: BLOQUE DE GENÉTICA ANIMAL ¾ Contenidos teóricos: - 1. Conceptos y técnicas básicas relacionadas con la detección de microsatélites. Métodos de identificación de microsatélites: Identificación a partir del estudio de genotecas. - 2. Aplicaciones de los microsatélites: Mapas genéticos. Localización de QTLs. Pruebas de paternidad. Análisis de variabilidad genética en las poblaciones. - 3. Otros marcadores. SNPs. Concepto y distribución en el genoma. Técnicas de detección. Detección por electroforesis: SSCP. Aplicaciones en genética animal. RAPDs. Concepto, variación en poblaciones animales y técnica de detección. Aplicaciones en genética animal. Prácticos: 1. Identificación de microsatélites en muestras biológicas. 2. Detección de SNPs por electroforesis: polimorfismo SSCP. 3. Detección de SNPs por PCR-RFLP. BLOQUE DE GENÉTICA VEGETAL ¾ Contenidos teóricos: 1. Marcadores moleculares en la selección del material vegetal de partida de un programa de Mejora. 2. Introducción al análisis de ligamiento y elaboración de mapas genéticos. 3. Fundamentos teóricos sobre la detección de QTL (Quantitative Trait Loci), concepto de “selección asistida por marcadores” (MAS). 4. Desarrollo de marcadores diagnóstico. Estrategia de genes candidatos 92 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 5. Uso de marcadores moleculares en la identificación varietal. Prácticos: 1. - Extracción rápida de ADN en material vegetal. 2. - Análisis de marcadores RAPDs (Random Amplified Polymorphic DNA), STS (Sequence Tagged Sites) y AFLPs (Amplified Fragment Length Polymorphisms). 3. - Interpretación y elaboración de bases de datos a partir de lectura de geles y análisis de fragmentos con electroforesis capilar. 4. Análisis de ligamiento y elaboración de mapas genéticos (programas Joinmap y Mapmaker) 5. Análisis de QTL (Quantitative Trait Loci) - Bibliografía/referencias/enlaces BLOQUE DE GENÉTICA ANIMAL DeNise S et al., Power of exclusion for parentage verification and probability of match for identity in American kennel club breeds using 17 canine microsatellite markers. Animal Genetics, 2004, 35: 14 – 17. Forman et al., Characterization of the COMMD1 mutation causing copper toxicosis in Bedlington terrier. Animal Genetics, 2005, 36: 497 - 501 Morera L et al., A polymorphic microsatellite located on pig chromosome band 12p11-2/3p13, within the 3’UTR of the ITGB3 gene. Animal Genetics 2002, 33: 239-240. Morera L et al., Genetic variation detected by microsatellites in five Spanish dog breeds. Journal of Heredity 1999, 90(6): 654-666. Pompanon F et al., Genotyping errors: causes, consequences and solutions. Nature Reviews: Genetics, 2005, 6: 847 – 859. Ruane J y Zimmerman M., Biotecnología agrícola para países en desarrollo. Resultados de un foro electrónico. Organización de las Naciones Unidas para la Agricultura y la Alimentación (FAO). Roma, 2003. Yuzbasiyan-Gurkan V et al., Linkage of a microsatellite marker to the canine copper toxicosis locus for Bedlington terriers. American Journal of Veterinary Research 1997, 58: 23-27. BLOQUE DE GENÉTICA VEGETAL Agarwal M., Shrivastava N, Padh H. (2008) Advances in marker technique and their applications in plant sciences. Plant Cell Rep 27: 617-631. Collard BCY, MZZ Jahufer, JB Brouwer, ECK Pang (2005). An introduction to markers, quantitative trait loci (QTL) mapping and marker-assisted selection for crop improvement: The basic concepts Euphytica 142: 169–196 93 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Gale, K.R. (2005). Diagnostic DNA markers for quality traits in wheat. Journal of Cereal Science 41, 181-192. Guimaraes E., Ruane J, Scherf BD, Sonnino A, Dargie JD. (2007). MarkerAssisted selection. Current status and future perspectives in crops, livestock, forestry and fish. Guimaraes E., Ruane J, Scherf BD, Sonnino A, Dargie JD (Eds). FAO, Rome. Martín A. (2002). Los Marcadores genéticos en Mejora Vegetal. En: Genómica y Mejora Vegetal. Nuez F, Carrillo JM, Lozano R (Eds) Mundi –Prensa Nuez F y Carrillo JM. (2000). Los Marcadores Genéticos en la Mejora Vegetal. Nuez F y Carrillo JM (Eds) Univ Politécnica de Valencia. Criterios de evaluación. Se valorará la asistencia y participación en las clases teóricas, la asistencia y resultados obtenidos en los trabajos prácticos, así como el trabajo de revisión presentado. Los alumnos que no cumplan con la asistencia a las actividades obligatorias del curso no podrán superar la asignatura en la convocatoria de Junio. Para estos alumnos el examen de septiembre consistirá en una prueba escrita sobre cuestiones teóricas y prácticas del programa de la asignatura. Distribución ECTS de la Asignatura MARCADORES MOLECULARES Y SU USO EN MEJORA GENÉTICA (a) 1 ECTS = 25 horas trabajo. (b) Estimación aproximada de estudio personal del alumno durante el curso 12 (trimestral) o 36 (anual) semanas: 1,5 horas de estudio por cada hora de teoría y 0,75 horas de estudio por cada hora de prácticas. (c)Las tutorías se encuentran incluidas en el total de Actividades Académicamente Dirigidas. Actividad Actividad Docente Materia Evaluación Alumno Procedimiento Peso en la nota final Horas presenciales Horas no presenciales Horas Profesor Prestar atención, intentar enterarse, preguntar si no se entera Tipo de preguntas. Se valorará razonamiento y capacidad de síntesis 35 % 10 h 28 h b 38 h ECTS a Clases en aula Teoría Exposición de la Teoría. Apoyo con audiovisuales Clases en laboratorio Prácticas de laboratorio Explicación de las prácticas Anotaciones, experimentos, ejercicios, informe, etc. Evaluación continuada, ejercicios, informes, actitud 40% 18 h 24 h 42 h Trabajos tutorizados. Distribución de los trabajos, recomendar bibliografía, orientar Recoger información, analizarla, obtener conclusiones y redactar el trabajo Se valorará bibliografía utilizada, contenidos y presentación 25% 4h 16 h 20 h 32 h 68 h 100h Actividades dirigidasc TOTAL CARGA DOCENTE DEL ALUMNO 94 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Asignatura: ANÁLISIS E INTERPRETACIÓN DE GENOMAS Total de créditos ECTS: 4 (100) Tipo de asignatura: Optativa. Profesores: Concha de la Hera Mª Carmen Ruiz Roldán Objetivos: Aprendizaje teórico y práctico de la estructura y organización de los genomas eucarióticos, a la luz de los resultados de los Proyectos Genoma y las informaciones que se derivan de la Genómica Comparativa. Se pretende acercar al alumno al manejo de bases de datos, al planteamiento experimental de problemas concretos y al análisis, discusión y presentación de los resultados. Programa de clases teóricas: 1. La Organización y Estructura de los Genomios. 2. De los Mapas Genéticos a los Proyectos Genoma 3. Disección, interpretación e impacto de la información obtenida tras la secuenciación de genomios 4. Genómica comparativa: Conservación de secuencias. Expansión de familias génicas. Sintenia. Programa de clases prácticas: 1. Análisis de secuencias repetidas en Fusarium 1.1. Identificación “in silico” de secuencias repetidas en el genomio de especies del género Fusarium: análisis de familias de transposones. 1.2. Comparación de la inestabilidad genómica entre estirpes silvestres y mutantes de Fusarium oxysporum mediante un ensayo fenotípico que detecta el movimiento de los transposones. 2. Análisis de familias génicas en Fusarium 1.1. Identificación “in sílico” de familias génicas en distintas especies del género Fusarium. 1.2. Detección de la variabilidad existente en una familia génica entre distintas formae speciales de F.oxysporum, mediante análisis por PCR. 1.2. Determinación del patrón de expresión de los miembros de una de las familias génicas mediante análisis transcripcional. Bibliografía: Libros: 1. Watson, JD; Caudy AA; Myers, RM y Witkowski JA. (2007) Recombinant DNA: Genes and Genomes, a short course. W.H. Freeman and Company. Cold Spring Harbor Laboratory Press. 95 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 2. Primrose SB y Twyman RM (2006) Principles of Gene Manipulation and Genomics. Blackwell Publishing. 3. Brown TA (2002) Genomes. Biocientific Publishers Ltd. Revisiones y Artículos Científicos: 1. Scannell DR, Butler G and Wolfe KH (2007) Yeast genome evolution-the origin of the species. Yeast 24:929-942 2. Tirosh I, Bilu Y and Barkai N (2007) Comparative biology: beyond sequence analysis. Current Opinion in Biotechnology 18: 371-377 Enlaces: http://www.broad.mit.edu/annotation/genome/fusarium_group/MultiHo me.html Distribución de horas presenciales y no presenciales: Todas las actividades presenciales son obligatorias. Será requisito indispensable la asistencia a un mínimo de un 80 % de las clases teóricas y prácticas. Criterios de evaluación: Ver en al cuadro adjunto el peso que se da a cada una de las actividades presenciales y no presenciales para la calificación final. 96 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 ANEXO I: Distribución ECTS de la Asignatura ANÁLISIS E INTERPRETACIÓN DE GENOMAS (a) 1 ECTS = 25 horas trabajo. (b) Estimación de estudio personal del alumno durante el curso 12 (trimestral) o 36 (anual) semanas: 1,5 horas de estudio por cada hora de teoría y 0,75 horas de estudio por cada hora de prácticas. Actividad Actividad Docente Clases en aula Alumno Procedimiento Peso en la nota final Horas presenciales Horas no presenciales Horas ECTS Profesor Teoría Exposición de la Teoría. Apoyo con audiovisuales Tomar apuntes, comprender los conceptos apoyados en el material audiovisual. Participar. Examen al finalizar el curso 50 % 10h 15h 25 h Prácticas de laboratorio Presentación/ Explicación de métodos y técnicas. Entrega de documentación y guías para el desarrollo experimental Anotaciones detalladas en cuaderno de laboratorio. Realización de experimentos, Obtención de resultados. Evaluación continuada de asistencia, destreza, actitud, resolución de problemas. 30% 20 15h 35h Presentación/ Explicación de métodos y técnicas. Realización de actividades y análisis de resultados. Evaluación continuada de asistencia, destreza, actitud, resolución de problemas 20% 10 15h 25h Es una actividad no evaluable, sólo de comunicación - 5h (individuales en despacho) 5h 2h 8h 10 h 42h 58 h 100h Materia Clases en laboratorio Análisis y comparación de secuencias en bases de datos. Tutorías Exámenes Evaluación Teoría y prácticas Teoría y/o problemas Se trata de Trabajo Colaborativo (todo el grupo, profesores y alumnos) Discusión de las materias teóricoprácticas de modo global y otros temas de interés a alumnos Poner, vigilar y corregir el examen. Evaluar el aprendizaje y rendimiento del alumno Preparación y Realización de examen TOTAL CARGA DOCENTE DEL ALUMNO 100% 97 a Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Asignatura: FILOGENIAS Y ANÁLISIS DE DATOS GENÉTICOS Total de créditos ECTS: 4 Tipo de asignatura: Elegible. Profesorado: Mª Ángeles Alonso Moraga Andrés Muñoz Serrano OBJETIVOS: Conocer la estructura filogenética de los grandes taxos. Conocer qué datos genéticos se utilizan para generar filogenias. Trabajar las metodologías utilizadas para inferir filogenias y sus limitaciones. Conocer los modelos de evolución molecular. Manejar programas generalistas de reconstrucción filogenética. Estudiar el origen de la variabilidad de los datos científicos. Analizar las causas de esta variabilidad. Estudiar los diferentes métodos de estima de los parámetros que determinan esta variabilidad. Y realizar la resolución en ordenador mediante paquetes estadísticos. NÚMERO DE HORAS DE TRABAJO DEL ALUMNO: Nº de Horas/ Créditos ECTS: ........100 horas/ 4.0 ECTS • Clases Teóricas: .........30 h • Clases Prácticas: ........ 10 h Actividades en colaboración con el profesor: • Exposiciones y Seminarios…10 h • Tutorías especializadas colectivas (presenciales o virtuales): 4 h Actividades autónomas del alumnado: • Horas de estudio y preparación de trabajos… 39 h • Tutorías especializadas individuales (presenciales o virtuales). 4 h • Realización de Exámenes:… 3 h Criterios de evaluación: Evaluación continua de los trabajos dirigidos. Programa: DATOS GENÉTICOS DE LOCI SIMPLES Tema 1: Base molecular de la evolución. Arqueología del genoma. Mecanismos de la evolución. Estructura y función de los genes. Tema 2: Árboles filogenéticos. Terminología. Reconstrucción de la historia de un carácter. Tema 3: Medida del cambio evolutivo. Alineamiento de secuencias y homología. Distancias genéticas. Tema 4: Reconstrucción de filogenias: métodos de distancias, de parsimonia, máxima verosimilitud y bayesiano. Error de muestreo. Tema 5: Modelos de evolución molecular. Composición de bases y uso de codones. Reloj Molecular. Selección natural a nivel molecular. Tema 6: Uso y discusión de lo programas de filogenias. 98 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Bibliografía Muñoz Serrano, A. 2002. Estadística Aplicada Uni y Multivariante. E.; Consejería de Agricultura y Pesca. Sevilla (España). Cuadras, C.M. 1981. Métodos de Análisis Multivariante. Ed:EUNIBAR. Barcelona (España). Nei M. and Kumar S. 2000. Molecular Evolution and Phylogenetics. Oxford University Press Page R.D.N. and Holmes E.C. 1998. Molecular Evolution: A Phylogenetic Approach. Ed. Blackewll Science. Sokal, R. R. and F. J. Rohlf. 1995. Biometry: the principles and practice of statistics in biological research. E.: W. H. Freeman, New York (USA). Weir B.S. 1996. Genetic Data Análisis II. Ed. Sinauer. 99 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 Horario del Master Semana 2-6N 9-13N 16-20N 23-27N 30N-4D L Transversales y comunes Transversales y comunes Transversales y comunes Transversales y comunes Bioq Prot:Proteo 9:00-13:00 (B) Met Exp Biol Cel 4:00-8:00 7-11D M X J V Bioq Prot:Proteo 9:00-13:00 (B) Met Exp Biol Cel 4:00-8:00 Bioq Prot:Proteo 9:00-13:00 (B) Met Exp Biol Cel 4:00-8:00 Bioq Prot:Proteo 9:00-13:00 (B) Met Exp Biol Cel 4:00-8:00 Immaculada Bioq Prot:Proteo 9:00-13:00 (A) Met Exp Biol Cel 4:00-8:00 Bioq Prot:Proteo 9:00-13: (A) Met Exp Biol Cel 4:00-8:00 Bioq Prot:Proteo 9:00-13:00 (B) GenetComport 13:00-14:00 Met Exp Biol Cel 4:00-8:00 Bioq Prot:Proteo 9:00-13:00 (A) Met Exp Biol Cel 4:00-6:00 14-18D Bioq Prot:Proteo 9:00-13:00 Bioq Prot:Proteo 9:00-13:00 Bioq Prot:Proteo 9:00-13:00 Bioq Prot:Proteo 9:00-13:00 4E-8E Navidad Navidad Navidad 11-15 E TecBasDNARec 9.30-10-30 (A y B) Presentación del curso. TecBasDNARec 9.30-12.30 (A) Módulo 1 TecBasDNARec 9.30-12.30 (A) Módulo1 TranspSolMemVeg 17:30-19:30 TecBasDNARec 9.30-13.30 (A) Módulo 3 Bioq Prot:Proteo 9:00-13:00 GenetComport 13:00-14:00 TranspSolMemVeg 17:30-19:30 TecBasDNARec 9.30-12.30 (A) Módulo 3 15.30-18.30 (B) Módulo 1 15.30-18.30 (B) 15.30-19.30 (B) 15.30-18.30 (B) TranspSolMemVg 17:30-19:30 TranspSolMemVg 17:30-19:30 TranspSolMemVg 17:30-19:30 TranspSolMemVg 17:30-19:30 TecBasDNARec 9.30-10.30 (A) Módulo 2 TecBasDNARec 9.30-12.30 (A) Módulo 2 TecBasDNARec 9.30-13.30 (A) Módulo 4 10.30-13.30 (A) Módulo 4 15.30-18.30 (B) Módulo 2 15.30-19.30 (B) Módulo 4 TranspSolMemVg 17:30-19:30 TecAvazGenFun 15:30-19:00 GenetComport 9:00-13:30 TecAvazGenFun 15:30-19:00 AnalInterpGenom 9:00-14:00 TranGenetMejVeg 16:00-20:30 TecAvazGenFun 15:30-19:00 GenetComport 9:00-13:30 TecAvazGenFun 15:30-19:00 AnalInterpGenom 9:00-14:00 TranGenetMejVeg 16:00-20:30 Santo TOMAS AnalInterpGenom 9:00-14:00 TranGenetMejVeg 16:00-20:30 Biotec Amb 16:00-20:00 CultCel 16:00-18:00 AnalInterpGenom 9:00-14:00 TranGenetMejVeg 16:00-20:30 Biotec Amb 16:00-20:00 CultCel 16:00-18:00 TranGenetMejVeg 16:00-20:00 10.30-12.30 (A) Módulo 1 18-22 E 15.30-17.30 (B) Módulo 1 TranspSolMemVg 17:30-19:30 TecBasDNARec 9.30-12.30 (A) Módulo 2 15.30-18.30 (B) Módulo 2 15.30-16.30 (B) Módulo 2 25-29E 1-5 F 8-12 F 15-19 F TranspSolMemVg 17:30-19:30 TecAvazGenFun 15:30-19:00 GenetComport 9:00-13:30 TecAvazGenFun 15:30-19:00 GenetComport 9:00-13:30 16.30-19.30 (B) Módulo 4 TranspSolMemVg 17:30-19:30 TecAvazGenFun 15:30-19:00 GenetComport 9:00-13:30 TecAvazGenFun 15:30-19:00 GenetComport 9:00-13:30 TecAvazGenFun 16:00-18:00 AnalInterpGenom 9:00-14:00 TranGenetMejVeg 16:00-20:30 Biotec Amb 16:00-20:00 CultCel 16:00-18:00 AnalInterpGenom 9:00-14:00 TranGenetMejVeg 16:00-20:30 Biotec Amb 16:00-20:00 CultCel 16:00-18:00 Santo TOMAS TecAvazGenFun 15:30-19:30 AnalInterpGenom 9:00-14:00 TranGenetMejVeg 16:00-20:30 Biotec Amb 16:00-20:00 CultCel 16:00-18:00 100 Master en Biotecnología, Molecular Celular y Genética 22-26 F 1-5 M 8-12 M 15-19M 22-26 M 29M-2A 5-9A 12-16A 19-23 A 26-30A 3-7M 10-14 M 17-21 M 24-28M MarcMolMejGen 9:00-14:00 RecFitoEvolPl 16:00-19:00 Biotec Amb 16:00-20:00 Cult Cel 16:00-18:00 RecFitoEvolPl 16:00-19:00 Biotec Veg 16:00-18:00 TecnAvazImagCel 16:00-18:00 RecFitoEvolPl 16:00-19:00 Biotec Veg 16:00-18:00 TecnAvazImagCel 16:00-18:00 ManCromosPlant 16:00-20:30 Metabol 9:30-13:00 15:30-19:30 SEMANA SANTA Epigenetica 16:00-20:300 ImmunolMolec 9:00-13:00 MecPatogen 16:00-20:30 ImmunolMolec 9:00-13:00 MecPatogen 16:00-20:30 BiotecEucUnic 16:00-18:00 Avanz Neuroend 16:00-19:00 BiotecEucUnic 16:00-18:00 Avanz Neuroend 16:00-20:00 BiotecEucUnic 16:00-18:00 FilogenAnalDGen 16:00-20:30 BiotecEucUnic 16:00-18:00 Curso 2009-2010 MarcMolMejGen 9:00-14:00 RecFitoEvolPl 16:00-19:00 Biotec Amb 16:00-20:00 Cult Cel 16:00-18:00 RecFitoEvolPl 16:00-19:00 Biotec Veg 16:00-18:00 TecnAvazImagCel 16:00-18:00 RecFitoEvolPl 16:00-19:00 Biotec Veg 16:00-18:00 TecnAvazImagCel 16:00-18:00 ManCromosPlant 16:00-20:30 Metabol 9:30-13:00 15:30-19:30 MarcMolMejGen 9:00-14:00 RecFitoEvolPl 16:00-19:00 Biotec Amb 16:00-20:00 Cult Cel 16:00-18:00 RecFitoEvolPl 16:00-19:00 Biotec Veg 16:00-18:00 TecnAvazImagCel 16:00-18:00 MarcMolMejGen 9:00-14:00 RecFitoEvolPl 16:00-19:00 Biotec Amb 16:00-20:00 CultCel 16:00-18:00 RecFitoEvolPl 16:00-19:00 Biotec Veg 16:00-18:00 TecnAvazImagCel 16:00-18:00 MarcMolMejGen 9:00-14:00 RecFitoEvolPl 16:00-19:00 Biotec Amb 16:00-20:00 CultCel 16:00-18:00 RecFitoEvolPl 16:00-19:00 Biotec Veg 16:00-18:00 TecnAvazImagCel 16:00-18:00 Biotec Veg 16:00-18:00 TecnAvazImagCel 16:00-18:00 ManCromosPlant 16:00-20:30 Metabol 9:30-13:00 15:30-19:30 Biotec Veg 16:00-18:00 TecnAvazImagCel 16:00-18:00 ManCromosPlant 16:00-20:30 Metabol 9:30-13:00 15:30-19:30 Biotec Veg 16:00-18:00 TecnAvazImagCel 16:00-18:00 ManCromosPlant 16:00-20:30 Metabol 9:30-13:00 15:30-19:30 Epigenetica 16:00-20:30 Epigenetica 16:00-20:30 Epigenetica 16:00-20:30 Epigenetica 16:00-20:30 ImmunolMolec 9:00-13:00 MecPatogen 16:00-20:30 ImmunolMolec 9:00-13:00 Epigenetica 16:00-20:30 ImmunolMolec 9:00-13:00 Epigenetica 16:00-20:30 ImmunolMolec 9:00-13:00 MecPatogen 16:00-20:30 BiotecEucUnic 16:00-18:00 Avanz Neuroend 16:00-19:00 BiotecEucUnic 16:00-18:00 Avanz Neuroend 16:00-20:00 BiotecEucUnic 16:00-18:00 FilogenAnalDGen 16:00-20:30 ImmunolMolec 9:00-13:00 1 MAYO MecPatogen 16:00-20:30 BiotecEucUnic 16:00-18:00 Avanz Neuroend 16:00-19:00 BiotecEucUnic 16:00-18:00 Avanz Neuroend 16:00-20:00 BiotecEucUnic 16:00-18:00 FilogenAnalDGen 16:00-20:30 MecPatogen 16:00-20:30 BiotecEucUnic 16:00-18:00 Avanz Neuroend 16:00-19:00 BiotecEucUnic 16:00-18:00 1 MAYO BiotecEucUnic 16:00-18:00 FilogenAnalDGen 16:00-20:30 FERIA BiotecEucUnic 16:00-18:00 FilogenAnalDGen 16:00-20:30 FERIA BiotecEucUnic 16:00-18:00 BiotecEucUnic 16:00-18:00 31M-4J ASIGNATURAS TRANSVERSALES CIENTÍFICO-TÉCNICAS Y COMUNES HORARIO ASIGNATURA 9.30 – 11.30 Búsqueda bibliográfica 11.30 – 13.30 Experimentación animal en investigación y sus alternativas 16.30 – 18.30 18.30 – 20.30 11:30-13:30 Teoría, metodología y evaluación de la Comunicación y divulgación de la ciencia investigación científica Transferencia de Ética aplicada a la ciencia Diseños de experimentos y tecnología y empresas de y a la vida profesional fundamentos de análisis de base tecnológica datos Bioinformática 101 Master en Biotecnología, Molecular Celular y Genética Curso 2009-2010 4. Líneas de investigación Línea ESTUDIO DEL APAREAMIENTO CROMOSÓMICO DURANTE LA MEIOSIS EN CEREALES Y SU APLICACIÓN PARA INTRODUCIR GENES DE INTERÉS AGRONÓMICO EN TRIGO FUNCION DE LINFOCITOS T EN INMUNOPATOLOGIA HUMANA Tutor Adoración Cabrera Caballero Pilar Prieto Aranda Manuel Santamaría Ossorio TÍTULO: METABOLOMA DEL OLIVO: RUTAS METABÓLICAS DE COMPUESTOS ANTIOXIDANTES DEL OLIVO M.D. Luque de Castro F. Priego Capote DESARROLLO DEL MAPA GENÉTICO DE LA ROSA (Rosa SP) Teresa Millán Valenzuela TRANSPORTE Y REGULACIÓN DE LA ASIMILACIÓN DE NITRATO Y METABOLISMO DEL MOLIBDENO EN ALGAS EUCARIOTAS Emilio Fernández Reyes Aurora Galván Cejudo Ángel Llamas Azúa CARACTERIZACIÓN FUNCIONAL DE UNA PROTEINA FAWRKY DE FRESA Y SU HOMÓLOGA EN Arabidopsis, ATWRKY75: DOS REGULADORES POSITIVOS DE RESISTENCIA BIORREACTORES PARA ACETIFICACIÓN OBTENCIÓN DE UNA VACUNA CURATIVA FRENTE AL HIV-1 (SIDA) PROTEÓMICA Y METAPROTEÓMICA AMBIENTALES PROTEÓMICA VEGETAL: PROTEÓMICA DE EXPRESIÓN DIFERENCIAL Y MODIFICACIONES POSTRADUCCIONALES (PROTEOMA REDOX) PROTEÓMICA DE LEVADURAS ENOLÓGICAS ADAPTACIONES Y MECANISMOS DE REGULACIÓN EN EL METABOLISMO DEL NITRÓGENO Y CARBONO DE LA CIANOBACTERIA Phroclocococcus DESMETILACIÓN DE ADN Y REGULACIÓN EPIGENÉTICA UTILIZACIÓN DE Caenorhabditis Elegans COMO MODELO EXPERIMENTAL PARA EL ESTUDIO DE LA BASE MOLECULAR DEL AUTISMO EVALUACIÓN DE LA ACTIVIDAD GENOTÓXICA Y ANTITUMORAL DE ALIMENTOS SISTEMA KISS-1/GPR54: CARACTERIZACIÓN FISIOLÓGICA E IMPLICACIONES FISIOPATOLÓGICAS Y TERAPÉUTICAS SELECCIÓN DE CANDIDATOS PARA VACUNAS MEDIANTE NUEVAS ESTRATEGIAS PROTEÓMICAS INGENIERÍA GENÉTICA EN HONGOS PATÓGENOS INMUNOSENESCENCIA E INFECCIONES VÍRICAS MEJORA ASISTIDA POR MARCADORES EN Juan Muñoz Blanco José Luis Caballero Repullo Isidoro García García José Peña Martínez Juan López Barea José Alhama Carmona Jesús V. Jorrín Novo Ana Mª Maldonado Alconada Juan Carlos García Mauricio Jesús Díez Dapena José Manuel García Fernández Mª Teresa Roldán Arjona Rafael Rodríguez Ariza Teresa Morales Ruiz Mª Dolores Córdoba Cañero Manuel Ruiz Rubio Encarnación Alejandre Durán Andrés Muñoz Serrano Ángeles Alonso Moraga Leonor Pinilla Jurado Manuel J. Rodríguez Ortega Concepción de la Hera Díaz de Liaño Carmen Ruiz Roldán Rafael Solana Lara Teresa Millán Valenzuela 102 Master en Biotecnología, Molecular Celular y Genética GARBANZO (Cicer arietinum L.) SEÑALES ENDOCRINAS QUE ADECUAN EL FUNCIONAMIENTO DEL EJE REPRODUCTOR A LAS RESERVAS ENERGÉTICAS DEL ORGANISMO Curso 2009-2010 Juan Gil Ligero Manuel Tena Sempere DEGRADACIÓN BACTERIANA DE CIANURO. APLICACIONES BIOTECNOLÓGICAS Francisco Castillo Rguez. Conrado Moreno Vivián Mª Dolores Roldán Ruiz ACTIVIDAD ESFINGOMIELINASA Y NIVELES DE CERAMIDA EN LOS FIBROBLASTOS EMBRIONARIOS DE RATÓN KNOCKOUTS NQO1-/DURANTE LA SENESCENCIA REPLICATIVA Francisco J. Alcaín Tejada CONTRIBUCIÓN DE NUEVAS SEÑALES ENDOCRINAS A LA REGULACIÓN FISIOLÓGICA DE LOS ADIPOCITOS Socorro García Navarro PAPEL DE LA UBIQUINONA EN LA DEFENSA CELULAR FRENTE AL ESTRÉS OXIDATIVO José M. Villalba Montoro José A. González Reyes Mª Isabel Burón Romero REGULACIÓN CELULAR Y MOLECULAR DE LA SECRECIÓN HORMONAL Justo P. Castaño Fuentes Rafael Vázquez Antonio J. Mtez. Fuentes Mª del Mar Malagón Poyato RUTAS DE SEÑALIZACIÓN IMPLICADAS EN LA PATOGÉNESIS FÚNGICA Antonio Di Pietro SIMULACIÓN Y DISEÑOS DE EXPERIMENTOS APLICADOS A LOS ANÁLISIS BIOMÉTRICOS Andrés Muñoz Serrano MODIFICACIONES REDOX DE LAS PROTEÍNAS: “REDOXINAS” Y PROTEÓMICA REGULACIÓN DE LOS FLUJOS DE CATIONES EN LEVADURAS Y PLANTAS José A. Bárcena Ruiz Carmen Alicia Padilla Peña Colab. Brian McDonagh José Ramos Ruiz Manuel Benlloch Marín José Mª Fournier Andray 103