BIR VOL IV BM 2015
Anuncio

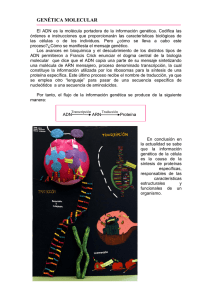
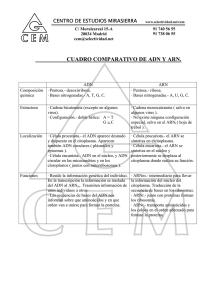
Manual bIR VOLUMEN IV: MANUAL DE BIOLOGÍA MOLECULAR Autor: Dr. Tomás E. Verdura González Editora: Dra. Iliana Perdomo López Manual BIR. VOLUMEN IV: MANUAL DE BIOLOGÍA MOLECULAR Autor: Dr. Tomás E. Verdura González © Iliana Perdomo López (editora). Depósito legal: DLC 614-2012 Reservado todos los derechos. Está prohibido, bajo las sanciones penales y el resarcimiento civil previsto en las leyes, reproducir, registrar o transmitir esta publicación, íntegra o parcialmente por cualquier sistema de recuperación y por cualquier medio, sea mecánico, electrónico, magnético, por fotocopia o por cualquier otro, sin la autorización previa por escrito de la editora. ÍNDICE BIOLOGÍA MOLECULAR BIR 1.- COMPONENTES DE LOS ÁCIDOS NUCLEICOS 2.- ESTRUCTURA DE LOS ÁCIDOS NUCLEICOS 1 13 2.1- ESTRUCTURA PRIMARIA 14 2.2- PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS. 14 3.- ESTRUCTURA SECUNDARIA 17 3.1- REGLAS DE CHARGAFF 17 3.2.- MODELO DE WATSON Y CRICK: FORMA B DEL ADN 17 3.3.- VARIACIONES EN LA ESTRUCTURA DEL ADN 20 3.4.- PROTEÍNAS DE UNIÓN AL ADN 26 3.5.- PROTEÍNAS DE UNIÓN AL ARN 29 4.- ESTRUCTURAS DE ORDEN SUPERIOR DEL ADN Y DEL ARN 31 4.1.- SUPERENROLLAMIENTO DEL ADN (ESTRUCTURA TERCIARIA) 31 4.2.- ESTRUCTURA DE LOS ARNs 35 4.3.- LOS RIBOSOMAS 41 5.- CONDENSACIÓN DEL ADN Y CROMOSOMAS 43 5.1.- PROTEÍNAS COMPONENTES DE LA CROMATINA 43 5.2.- NIVELES DE CONDENSACIÓN DEL ADN NUCLEAR EUCARIÓTICO 45 5.3.- EL CROMOSOMA METAFÁSICO 51 5.4.- DOTACIÓN GENÉTICA EN EUCARIOTAS 57 6.- ORGANIZACIÓN DEL GENOMA DE EUCARIOTAS 59 6.1.- CARACTERÍSTICAS GENERALES DEL GENOMA. DIFERENCIAS ENTRE PROCARIOTAS Y EUCARIOTAS 59 6.2.- COMPLEJIDAD DEL GENOMA EUCARIÓTICO 60 6.3.- COMPLEJIDAD DEL GENOMA HUMANO 61 6.4.- ADN DE COPIA ÚNICA, SIMPLE O NO REPETITIVO 62 6.5.- ADN REPETITIVO 62 6.6.- ESTUDIO EXPERIMENTAL DE LA COMPLEJIDAD 69 7.- EL CICLO CELULAR 73 7.1.- CONTROL DEL CICLO CELULAR 74 7.2.- DIVISIÓN CELULAR 87 7.3.- DIFERENCIAS ENTRE MITOSIS Y MEIOSIS 96 7.4.- LA GAMETOGÉNESIS 97 8.- GENÉTICA MENDELIANA Y AMPLIACIÓN DEL MENDELISMO 99 8.1.- LAS LEYES DE MENDEL 99 8.2.- AMPLIACIÓN DEL MENDELISMO 106 8.3.- HERENCIA EN RELACIÓN CON EL SEXO 114 8.4.- COMPROBACIÓN DE LAS PROPORCIONES MENDELIANAS. PRUEBA CHI-CUADRADO 118 8.5.- POLIGENIA. HERENCIA MULTIFACTORIAL 119 8.6.- LA HERENCIA NO NUCLEAR 120 9.- LIGAMIENTO Y RECOMBINACIÓN 123 9.1.- GENES LIGADOS 123 9.2.- LA RECOMBINACIÓN 126 9.3.- CARTOGRAFÍA EN ORGANISMOS DIPLOIDES 128 9.4.- SEGREGACIÓN Y RECOMBINACIÓN MITÓTICA 134 9.5.- INTERCAMBIOS ENTRE CROMÁTIDAS HERMANAS 134 9.6.- CONFECCIÓN DE MAPAS DE CROMOSOMAS EN HUMANOS 134 10.- MECANISMOS DE RECOMBINACIÓN 137 11.- LA REPLICACIÓN DEL ADN 147 11.1.- CARACTERÍSTICAS 147 11.2.- ENZIMOLOGÍA DE LA REPLICACIÓN 150 11.3.- ETAPAS DE LA REPLICACIÓN 154 11.4.- REPLICACIÓN MITOCONDRIAL 161 11.5.- REPLICACIÓN POR CÍRCULOS RODANTES 162 12.- LA TRANSCRIPCIÓN 163 12.1.- CONCEPTO DE GEN 163 12.2.- ENZIMOLOGÍA DE LA TRANSCRIPCIÓN 165 12.3.- TRANSCRIPCIÓN DEL GENOMA MITOCONDRIAL 175 12.4.- INHIBIDORES DE LA TRANSCRIPCIÓN 175 13.- REGULACIÓN DE LA EXPRESIÓN GÉNICA EN EUCARIOTAS: CONTROL PRETRANSCRIPCIONAL Y TRANSCRIPCIONAL 177 13.1.- CONTROL PRETRANSCRIPCIONAL 177 13.2.- CONTROL TRANSCRIPCIONAL 184 14.- CONTROL POSTRANSCRIPCIONAL O PROCESAMIENTO DEL ARN 191 14.1.- PROCESAMIENTO DEL ARN MENSAJERO 193 14.2.- PROCESAMIENTO DE LOS ARNs RIBOSÓMICOS Y TRANSFERENTES 200 14.3.- MADURACIÓN DEL ARN MITOCONDRIAL 200 14.4.- REGULACIÓN POSTRANSCRIPCIONAL Y PRETRADUCCIONAL 201 15.- EL CÓDIGO GENÉTICO 203 15.1.- CARACTERÍSTICAS DEL CÓDIGO GENÉTICO 203 15.2.- DESCIFRAMIENTO DEL CÓDIGO GENÉTICO 205 16.- TRADUCCIÓN O SÍNTESIS DE PROTEÍNAS 207 16.1.- FASE PREVIA: ACTIVACIÓN DE LOS AMINOÁCIDOS 208 16.2.- INICIACIÓN 209 16.3.- ELONGACIÓN 213 16.4.- TERMINACIÓN 215 16.5.- ENÉRGETICA 216 16.6.- INHIBIDORES DE LA TRADUCCIÓN 217 16.7.- REGULACIÓN TRADUCCIONAL 219 17.- MODIFICACIONES POSTRADUCCIONALES 221 17.1.- TRÁFICO O DESTINO DE LAS PROTEÍNAS 221 17.2.- MADURACIÓN DEL POLIPÉPTIDO NACIENTE 223 17.3.- DEGRADACIÓN DE LAS PROTEÍNAS 232 18.- MUTACIÓN 235 18.1.- MUTACIONES GÉNICAS 235 18.2.- MECANISMOS DE ORIGEN DE LAS MUTACIONES GÉNICAS 238 18.3.- MUTACIONES CROMOSÓMICAS ESTRUCTURALES 242 18.4.- MUTACIONES CROMOSÓMICAS NUMÉRICAS 242 19.- REPARACIÓN DEL ADN 243 19.1.- SISTEMAS PREVENTIVOS 243 19.2.- REVERSIÓN DIRECTA DE LA LESIÓN 243 19.3.- REPARACIÓN POR ESCISIÓN 244 19.4.- REPARACIÓN POSREPLICACIÓN 247 19.5.- REPARACIÓN GO 250 20.- ENFERMEDADES GENÉTICAS. ENFERMEDADES MONOGÉNICAS Y MULTIFACTORIALES 251 20.1.- CLASIFICACIÓN DE LAS ENFERMEDADES GENÉTICAS 251 20.2.- ENFERMEDADES MONOGÉNICAS NUCLEARES 253 20.3.- ENFERMEDADES MITOCONDRIALES 263 20.4.- ENFERMEDADES POLIGÉNICAS, MULTIFACTORIALES, COMPLEJAS O MIXTAS 20.5.- ENFERMEDADES COMPLEJAS: ENFERMEDAD DE ALZHEIMER 264 266 21.- CROMOSOMOPATÍAS O ENFERMEDADES CITOGENÉTICAS 267 21.1.- CARIOTIPO HUMANO Y SISTEMA INTERNACIONAL DE NOMENCLATURA 267 21.2.- CROMOSOMOPATÍAS ESTRUCTURALES 269 21.3.- CROMOSOMOPATÍAS NUMÉRICAS 280 22.- BASES MOLECULARES DEL CÁNCER 287 22.1.- GENES RESPONSABLES DEL CÁNCER 287 22.2.- RUTAS REGULADORAS DE LA PROLIFERACIÓN CELULAR 290 23.- MÉTODOS DE HIBRIDACIÓN 295 23.1.- HIBRIDACIÓN EN FASE LÍQUIDA 296 23.2.- HIBRIDACIÓN EN SOPORTE SÓLIDO 296 23.3.- HIBRIDACIÓN IN SITU 298 23.4.- MICROARRAY O MICROCHIP DE ADN 302 23.5.- SINTESIS DE ADNc 305 23.6.- CGH (HIBRIDACIÓN GENÓMICA COMPARADA) 305 23.7.- TRANSFERENCIA WESTERN 306 24.- CLONACIÓN ACELULAR: REACCIÓN EN CADENA DE LA POLIMERASA (PCR) 307 24.1.- OBJETIVO Y APLICACIONES DE LA PCR 307 24.2.- REQUERIMIENTOS 308 24.3.- ETAPAS DEL PROCESO 308 24.4.- CARACTERÍSTICAS DEL PROCESO 310 24.5.- VARIANTES DEL MÉTODO 311 25.- TECNOLOGÍA DEL ADN RECOMBINANTE 315 25.1.- NUCLEASAS 315 25.2.- ENZIMAS DE RESTRICCIÓN 315 25.3.- CLONACIÓN CELULAR DE MOLÉCULAS DE ADN 319 25.4.- GENOTECAS 328 26.- MAPA GENÉTICO Y FÍSICO DEL GENOMA 331 26.1.- MAPA GENÉTICO O DE LIGAMIENTO 331 26.2.- MAPA FÍSICO 331 26.3.- SECUENCIACIÓN 336 27.- LOS GENES EN LAS POBLACIONES 343 27.1.- EQUILIBRIO DE HARDY-WEINBERG 343 27.2.- MODIFICACIÓN DE LAS FRECUENCIAS GÉNICAS Y GENOTÍPICAS 345 27.3.- CÁLCULO DEL COEFICIENTE DE CONSANGUINIDAD INDIVIDUAL 346 BIBLIOGRAFÍA 347 Biología Molecular 1. InspiracleBIR/2015 COMPONENTES DE LOS ÁCIDOS NUCLEICOS. (2011) 212; (2010) 167, 219; (2009) 260; (2008) 174, 215, 216, 237; (2007) 185, 186; (2005) 241; (2003) 188. Los ácidos nucleicos son macromoléculas catenarias compuestas exclusivamente por C, H, O, N y P y constituidas por monómeros denominados nucleótidos. Existen dos tipos de ácidos nucleicos, ADN y ARN, formados cada uno por un tipo de nucleótido: - ADN (ácido desoxirribonucleico) por desoxirribonucleótidos. - ARN (ácido ribonucleico) por ribonucleótidos. Independientemente de su tipo, cada nucleótido consta de tres componentes: 1. Una pentosa. Aparece en su forma más estable, la configuración β: la β-D-ribofuranosa en el ARN y la β-D-2-desoxirribofuranosa en el ADN. Fuente: Figura apartado 2.2 (pag 11) del libro “Texto ilustrado de Biología Molecular e Ingeniería Genética., Barcelona, 1ª ed., 2008”. 2. Una base nitrogenada heterocíclica. Moléculas con más de un átomo de nitrógeno. Pertenecen a dos familias distintas: - pirimidínicas: derivadas de la pirimidina, formadas sólo por un anillo. Son tres: citosina (C), timina (T) y uracilo (U). - púricas: formadas por dos anillos condensados. Son dos, adenina (A) y guanina (G). 1 InspiracleBIR/2015 Biología Molecular Fuente: Figura apartado 2.31 (pag 12) del libro “Texto ilustrado de Biología Molecular e Ingeniería Genética., Barcelona, 1ª ed., 2008”. Las bases nitrogenadas poseen las siguientes propiedades: a) Hidrofobicidad. La naturaleza aromática de los anillos hace que las bases tengan un marcado carácter apolar, sean hidrofóbicas y poco solubles en agua a pH celular. A pH ácido o alcalino adquieren carga y se hacen más solubles en agua. b) Existencia de dipolos. Salvo la adenina, todas las bases poseen átomos de nitrógeno y oxígeno, lo que determina que se formen entre ellas enlaces polares como los puentes de hidrógeno. c) Disposición coplanar de los enlaces de cada anillo. d) Tautomería. Consiste en el desplazamiento de un H desde un átomo de N o de O nuclear hacia otro extranuclear y viceversa. Hay dos tipos de tautomerías: d.1. Tautomería ceto-enol. El grupo ceto forma parte de un enlace amida cíclica o lactama. Al migrar el átomo de H desde el N nuclear al O del grupo ceto, se convierte en el tautómero lactima (enol). Esta tautomería afecta a T, G, C y U. 2 Biología Molecular InspiracleBIR/2015 d.2. Tautomería imina-amina. Aquí el grupo exocíclico es una imina, que al recibir el H desde el N nuclear se convierte en un grupo amina primaria. Afecta a A, G y C. En condiciones fisiológicas normales, los equilibrios de tautomerización están desplazados hacia las formas ceto y amino, que son más estables. Por lo tanto, en las bases libres, estas formas son unas 10000 veces más abundantes que las enol e imina. Fuente: Figura (pag 15) del libro “Texto ilustrado de Biología Molecular e Ingeniería Genética., Barcelona, 1ª ed., 2008”. e) Carácter básico. Son bases débiles porque con valores de pka entre 9 y 10 pueden captar protones. 3 Biología Molecular 2. InspiracleBIR/2015 ESTRUCTURA DE LOS ÁCIDOS NUCLEICOS. (2011) 213, 246; (2010) 202, 231; (2009) 243; (2008) 158, 243; (2006) 193; (2005) 242. Las funciones de los dos tipos de ácidos nucleicos vienen determinadas por su localización subcelular y su conformación tridimensional. Diferencias entre el ADN y el ARN ADN Función ARN Depositario y transmisor de Interviene en la transmisión la información genética, de la información desde el organizada en genes que ADN hasta los productos Composición Localización codifican proteínas o ARNs. génicos. Azúcar: 2´-desoxirribosa. Azúcar: Ribosa. Bases: A, T, G, C. Bases: A, U, G, C. Eucariotas: en el núcleo Eucariotas: en el como cromosomas, matriz temporalmente, mitocondrial y estroma de matriz cloroplastos. Procariotas: núcleo citosol, mitocondrial y estroma. en la zona Procariotas: citosol. nucleoide del citosol, una Virus: en algunos en la molécula de ADN circular cápside. “cromosoma” y varios plásmidos. Virus: en algunos en la cápside. En los ácidos nucleicos se pueden considerar varios niveles estructurales, al igual que en las proteínas: - Estructura primaria: es la unión de los nucleótidos en un polímero lineal. El orden de las bases define la secuencia. - Estructura secundaria: corresponde a la conformación local, a la disposición relativa de los nucleótidos próximos. Está definida por la asociación de dos cadenas polinucleotídicas a través de las bases nitrogenadas. - Estructuras de orden superior: para el ADN se incluyen las estructuras resultantes del superenrollamiento y de la asociación con proteínas básicas para formar la cromatina. No están determinadas por las estructuras de los niveles inferiores. 13 Biología Molecular InspiracleBIR/2015 Hebras equilibradas: regiones del ADN en las que una de las cadenas contiene igual proporción de purinas y pirimidinas. Como consecuencia la hebra complementaria también es equilibrada. e) Helicidad y carácter dextrógiro. El apareamiento completo de las dos hebras sólo se puede establecer si los pares de bases sucesivos van girando unos con respecto a los otros (34,6º en el B-ADN). Como consecuencia la cadena es una doble hélice dextrógira. Fuente: Parámetro apartado 5.2.6 (pag 46) del libro “Texto ilustrado de Biología Molecular e Ingeniería Genética., Barcelona, 1ª ed., 2008”. f) Carácter anfipático y estabilidad. Los esqueletos azúcar-fosfato se disponen hacia el exterior, lo cual muestra un marcado carácter hidrofílico y minimiza la repulsión electrostática entre los fosfatos. Las bases se dirigen hacia el interior de la hélice, perpendiculares a su eje y superpuestas como monedas. Como consecuencia de la proximidad y paralelismo de los anillos aromáticos se forman fuerzas de Van der Waals (hidrofóbicas), llamadas interacciones de apilamiento, que son inespecíficas y en su conjunto la contribuyen a la estabilidad de la molécula de una forma muy elevada. 19 Biología Molecular InspiracleBIR/2015 Fuente: Figura apartado 6.1.1 (pag 53) del libro “Texto ilustrado de Biología Molecular e Ingeniería Genética., Barcelona, 1ª ed., 2008”. Fuente: Figura apartado 6.1.1 (pag 53) del libro “Texto ilustrado de Biología Molecular e Ingeniería Genética., Barcelona, 1ª ed., 2008”. 21 Biología Molecular InspiracleBIR/2015 Tipos de ARN interferente Los ARN interferentes son moléculas pequeñas (de 20 a 25 nucléotidos) que se generan por fragmentación de precursores más largos. Se pueden clasificar en tres grandes grupos: 1) siRNA El acrónimo siRNA proviene el inglés small interfering RNA: en español, ARN interferente pequeño. Son moléculas de ARN bicatenario perfectamente complementarias de aproximadamente 20 o 21 nucleótidos (nt) con 2 nucleótidos desemparejados en cada extremo 3'. Cada hebra de ARN tiene un grupo fosfato 5' y un grupo hidroxilo (-OH) 3'. Esta estructura proviene del procesamiento llevado a cabo por Dicer, una enzima que corta moléculas largas de ARN bicatenario (dsRNA, double stranded RNA) en varios siRNA.3 Una de las hebras del siRNA (la hebra 'antisentido') se ensambla en un complejo proteico denominado RISC (RNA-induced silencing complex), que utiliza la hebra de siRNA como guía para identificar el ARN mensajero complementario. El complejo RISC cataliza el corte del ARNm complementario en dos mitades, que son degradadas por la maquinaria celular, bloqueando así la expresión del gen. Los siRNA pueden ser también introducidos de forma exógena en las células utilizando métodos de transfección basándose en la secuencia complementaria de un gen en particular, con la finalidad de reducir significativamente su expresión. Mecanismo de RNAi / ribointerferencia mediado por siRNA. 39 Biología Molecular InspiracleBIR/2015 5.3 EL CROMOSOMA METAFÁSICO Los cromosomas metafásicos son corpúsculos observables a microscopio óptico con aspecto de bastoncillos con dos cromátidas (cada una de las dos moléculas de ADN que forma el cromosoma) más o menos separadas entre sí. Las proteínas SMC mantienen la estructura de los cromosomas Las proteínas SMC (mantenimiento estructural de los cromosomas) constan de 5 dominios, los dominios globulares amino y C-terminal se unen para formar un sitio de hidrólisis del ATP y están conectados por dos regiones en hélice α unidas por un domino bisagra. Generalmente son proteínas diméricas y forman un complejo en forma de V. Las proteínas pertenecientes a la familia SMC se encuentran en todos los organismos, de las bacterias a los humanos. Los eucariotas tienen dos tipos principales, las cohesinas y las condensinas. Las cohesinas juegan un importante papel en la unión de las cromátidas hermanas inmediatamente después de la replicación y las mantienen juntas cuando los cromosomas se condensan en la metafase . Esta asociación es esencial para la correcta segregación de los cromosomas en la metafase. Las condensinas son esenciales para la condensación de los cromosomas en la entrada de las células en la mitosis. En el laboratorio, las condensinas se unen al DNA y forman vueltas superhelicoidales positivas; provocando un enrollamiento adicional del ADN, al contrario de los nucleosomas, que lo subenrollan. 5.3.1. Morfología La región más estrecha del cromosoma por la que se hayan unidas las dos cromátidas hermanas, se llama centrómero o constricción primaria. En los centrómeros se hallan los puntos de anclaje a los microtúbulos, los cinetocoros, compuestos principalmente por ARN y proteína. El centrómero delimita los brazos cromosómicos, cuatro antes de la mitosis y dos después. El brazo corto se nombra con la letra p (petit) y el largo con q (queue) y así 9q designa el brazo largo del cromosoma 9. Los cromosomas se clasifican según la posición del centrómero en: - metacéntricos: en la mitad, los brazos son de igual longitud. - telocéntrico: en el extremo del cromosoma. - submetacéntricos o subtelocéntricos: brazos de tamaño diferente. - acrocéntricos: un brazo grande y otro muy pequeño. En los brazos cortos de estos cromosomas, excepto en el Y, existen otros estrechamientos denominados constricciones secundarias, que delimitan una pequeña zona esférica terminal que se denomina satélite cromosómico. En los satélites se encuentran los genes del ARNr 18S y 28S. 51 InspiracleBIR/2015 - Biología Molecular genes truncados: son copias incompletas del gen por pérdida de una región situada en uno de los extremos, el 3´ o el 5´. - fragmentos de genes: si han perdido ambos extremos y sólo se conserva el interior del gen. En este grupo hay tres tipos de familias: - Con homología sólo en parte de la secuencia de ADN, lo que da lugar a proteínas con grandes regiones de secuencia y estructuras comunes (dominios). - Con homología escasa, lo que genera proteínas con pequeños motivos comunes, cada uno de pocos aminoácidos. - Grandes “superfamilias” con muy débil homología en la secuencia y por tanto en la de las proteínas. El ejemplo más representativo es el de los genes de la globina. También los de los genes HLA-I. 64 InspiracleBIR/2015 Biología Molecular Tercer punto de control Se encuentra en la fase M, entre la metafase y la anafase. Se encarga de revisar que todos los cromosomas se hayan unido al huso mitótico. Si detecta que uno de los cinetocoros no se encuentra unido, manda una señal negativa al sistema de control bloqueando la activación de proteínas implicadas en la separación de las cromátidas hermanas. Específicamente inactiva al conjunto APC- cdc20, lo que inhibe la liberación de la separasa, impidiendo que las cromátides hermanas se separen hasta que la señal desaparezca. Control extracelular del ciclo celular La mayoría de los mitógenos controlan la tasa de división celular actuando en la fase G1; liberan el control negativo del ciclo celular permitiendo la entrada a la fase S. Actúan uniéndose a receptores de membrana con actividad de tirosina-cinasas los cuales activan a la proteína G monomérica Ras cambiándola de su estado unido a GDP por GTP; esta activación desencadena una cascada de fosforilaciones a través de las proteínas MAPK (cinasas activadas por mitógenos). A su vez estas proteínas MAPK transmiten el estimulo a diversas moléculas efectoras (cinasas de proteínas o factores de trascripción). Esta cascada de fosforilaciones ocasiona la trascripción de genes tempranos (entre los que destacan los que codifican a las ciclinas de G1), algunos de estos genes a su vez activan la trascripción de otros genes denominados genes tardíos. De esta manera la vía de señalización Ras-MAPK transmite señales extracelulares al núcleo activando la maquinaria del ciclo celular. 84 InspiracleBIR/2015 Biología Molecular Comienza cuando los cromosomas anafásicos llegan a sus respectivos polos. SEGUNDA DIVISIÓN MEIÓTICA Después de la telofase, existe un corto período de interfase que algunas veces es de larga duración y en el que no hay duplicación del ADN. Las dos células hijas llegan con una dotación haploide de cromosomas, aunque cada uno está constituido por dos cromátidas (n cromosomas y 2C de ADN). La segunda división tiene como objeto separar esas cromátidas hermanas dejando dos células haploides, n, con un contenido en ADN igual a C. La profase II es corta y similar a la de una mitosis. En la metafase II, los cromosomas se disponen en el plano ecuatorial. El centrómero se rompe separándose los cinetocoros y las dos cromátidas hijas se dirigen a los polos opuestos durante la anafase II. p. 75 (2010). Cada uno de los cuatro núcleos de la telofase II tendrá una cromátida de lo que inicialmente fue una tétrada, que será diferente genéticamente de cualquiera de las presentes en los cromosomas materno y paterno. Además, cada núcleo haploide tiene una composición genética diferente. 7.3. DIFERENCIAS ENTRE MITOSIS Y MEIOSIS 96 Biología Molecular InspiracleBIR/2015 10. MECANISMOS DE RECOMBINACIÓN. (2013) 95. La fuente primaria de variabilidad es la mutación, sin embargo, en los organismos con reproducción sexual la producción de individuos con nuevas combinaciones de alelos a partir de los ya existentes en la población, es el mecanismo más importante de producción de variabilidad genética. Existen diferentes tipos de recombinación: - recombinación homóloga general. - recombinación específica de sitio. - transposición. En eucariotas la recombinación genética se produce por la distribución independiente de los cromosomas durante la meiosis y por la existencia de un intercambio físico de segmentos cromosómicos entre dos cromátidas no hermanas de dos cromosomas homólogos, a través del entrecruzamiento que tiene lugar en la profase I de la meiosis. En procariotas la recombinación genética se lleva a cabo mediante fenómenos parasexuales: conjugación, transformación, transducción y sexducción. MECANISMOS MOLECULARES DE LA RECOMBINACIÓN GENERAL HOMÓLOGA 1. MODELO DE HOLLIDAY El primer modelo aceptado fue propuesto por Holliday en 1964 para procariotas. Consiste en un proceso de ruptura de dos moléculas de ADN homólogas en el mismo lugar de una sola cadena en cada doble hélice, seguido de un intercambio de fragmentos entre ellas y una reconstitución de dos moléculas que contienen partes intercambiadas. Los pasos básicos son los siguientes: - Apareamiento de los cromosomas homólogos mediado por el complejo sinaptonémico. Las dobles hélices implicadas en el entrecruzamiento rotan de manera que queden enfrentadas con la misma polaridad. - Producción de cortes en las dos cadenas de la misma polaridad y desenrollamiento de la doble hélice. Este paso lo lleva a cabo el complejo RecBCD con actividad nucleasa y helicasa, que genera cadenas sencillas. La actividad nucleasa reconoce el sitio chi, una secuencia constituida por 8 pb (5´-GCTGGTGG-3´) y que se encuentra en multitud de sitios del genoma. La cadena sencilla originada, una vez estabilizada por la proteína Ssb desencadena el proceso. - Los extremos rotos invaden la doble hélice contraria: una hélice sencilla desplaza a la cadena de su misma polaridad y la otra hace lo mismo. Las cadenas desplazadas se unen a RecA, que reconoce el ADN de cadena sencilla y promueve la invasión de esta cadena hacia una doble hélice que esté en su proximidad. Si encuentra homología desplaza a la cadena de igual polaridad. El que se mueve es siempre el extremo 3´-OH. La cadena 137 Biología Molecular InspiracleBIR/2015 La hebra crece por su extremo 3´ y se dice que la síntesis tiene lugar en dirección 5´→3´. 11.2.3. Tipos de ADN polimerasas La síntesis de ADN ocurre de forma idéntica en todos los organismos y en todas las ubicaciones subcelulares, pero es efectuada por enzimas distintas en distintos casos. Se denominan polimerasas de ADN dirigidas por ADN (o dependientes de ADN), o comúnmente ADN polimerasas (ADNpol). Las principales polimerasas son: Molécula sintetizada Molécula molde Nombre completo ADN ADN ADN Nombre común polimerasa ADN polimerasa dependiente de ADN ADN ARN ADN polimerasa Transcriptasa dependiente de ARN ARN ADN ARN ARN ARN ARN Telomerasa polimerasa ARN dependiente de ADN inversa polimerasa Primasa polimerasa ARN replicasa dependiente de ARN 11.2.3.1. ADN polimerasas de procariotas En procariotas se han descrito 3 ADN polimerasas: ADN polimerasa I, ADN polimerasa II y ADN polimerasa III. La I y la II son monoméricas. La primera descubierta, por Kornberg en 1958, y la mejor estudiada es la ADNpol I de E. coli. Su hidrólisis con subtilisina o tripsina origina dos fragmentos: - Fragmento grande o de “Klenow”: es el N-terminal, de 68kDa. Contiene los centros activos responsables de las actividades polimerasa y 3´-exonucleasa. Se emplea para la síntesis in vitro de ADN. - Fragmento pequeño: el C-terminal, de 35kDa y estructura poco conocida. Sólo muestra la actividad 5´-exonucleasa. Es única entre las polimerasas porque la actividad exonucleasa 5´→3´ le permite hidrolizar secuencialmente ADN o ARN a partir del extremo 5´ de la hebra. Así es capaz de eliminar el cebador y sustituirlo por ADN durante la síntesis discontinua de la hebra retardada. También interviene en la escisión de los dímeros de pirimidina (actividad de reparación) y experimentalmente se emplea para preparar sondas marcadas. 151 Biología Molecular InspiracleBIR/2015 Formación y composición de la holoenzima ADNpol III. La holoenzima contiene dos copias del núcleo catalítico de la enzima lo que permite que la hebra guía y retardada se sinteticen de forma coordinada. 153 InspiracleBIR/2015 Biología Molecular 12.2.2.4. Polimerasas En procariotas y mitocondrias y cloroplastos de eucariotas, una sola ARN polimerasa sintetiza los tres tipos de ARNs: ARNm, ARNt y ARNr. En el núcleo de eucariotas existen 3 ARN polimerasas diferentes: ARNpol-I, ARNpol-II y ARNpol-III, que sintetizan cada una distintos tipos de ARN. Sus características son las siguientes: ARNpol-I Localización Nucleolo ARNpol-II ARNpol-III ARNpol Nucleoplasma Nucleoplasma Mitocondrias Cloroplastos Tránscrito Pre- primario ARNr Pre-ARNm Pre-ARNt ARNr, ARNm grande Pre-ARNr y ARNt 45S precursor pequeño mitocondriales de: o Producto ARNr 28S ARNm ARNt génico final ARNr 18S Mayoría de ARNsn (ARN ARNr 5S ARNr 5,8S U1-U5) snRNA U6 miRNA snoRNA 50 -70% 20 – 40% ≈ 10% Ninguno Fuerte inhibición Inhibición débil Actividad cloroplásticos enzimática Efecto de la α-amanitina Ninguno Sí afectada por la rifampicina No se sabe si se sintetizan directamente en forma activa o como pre-ARNs que necesitan maduración. Las ARN polimerasas no requieren cebador y carecen de actividad correctora exonucleasa 3´→5´. Estructura de la ARN polimerasa de E. coli La ARN polimerasa de procariotas, la mejor estudiada, está formada por 4 (puede que 5) polipéptidos asociados en la llamada enzima “mínima” o “núcleo” . Para iniciar la transcripción, la enzima necesita de la unión de otro polipéptido independiente, el factor σ. Enzima núcleo: Subunidad α: ensambla la enzima núcleo y se une a proteínas reguladoras. Subunidad β: sitio catalítico de la polimerasa. Subunidad β´: se une al ADN molde. Subunidad ω: 168 InspiracleBIR/2015 Biología Molecular Fuente: Tabla apartado 21.3.3.4 (pag 52) del libro “Texto ilustrado de Biología Molecular e Ingeniería Genética., Barcelona, 1ª ed., 2008”. 14.1.3.1 Otros tipos de intrones Grupo I: Pertenecen los intrones de algunos genes que codifican ARNr nucleares, mitocondriales y de cloroplastos. Su procesamiento requiere como cofactor externo un nucleósido o un nucleótido de guanina. El splicing no utiliza ATP como fuente de energía. Grupo II: Aparecen en genes de mitocondrias o cloroplastos que codifican preARNm. No necesitan cofactor externo, sino que usan el 2´-OH de una A del propio intrón. No necesitan spliceosoma, sino que experimentan un autosplicing catalizado por el propio ARN. Estos ARNs con función autocatalítica constituyen uno de los casos de ribozimas. Grupo III: Es el más numeroso y el ya visto para los intrones de los genes nucleares que codifican proteínas. Grupo IV: comprende ciertos genes de ARNts. El splicing requiere ATP. 198 InspiracleBIR/2015 Biología Molecular b) ARNm policistrónicos: sólo aparecen en procariotas y virus y codifican varios polipéptidos. La traducción se inicia, simultáneamente o no, en varios codones de inicio y finaliza en sus respectivos codones de terminación. En la traducción se distinguen varias etapas: una fase previa de activación, iniciación, elongación y terminación. 16.1. FASE PREVIA: ACTIVACIÓN DE LOS AMINOÁCIDOS La unión del aminoácido correcto a cada ARNt es el paso más crítico de la expresión génica y debe realizarse de forma correcta. La unión del aminoácido con el ARNt se realiza en dos reacciones catalizadas por la misma enzima, una aminoacil-ARNt sintetasa (aaRS): R-CH(NH2)-COOH + AMP-P-P + enzima → Enzima-AMP-CO-CH(NH2)-R + PPi (aminoácido) (ATP) La hidrólisis del PPi impulsa la reacción: PPi (aminoacil-AMP-enzima) → 2Pi Enzima-AMP-CO-CH(NH2)-R + ARNt-OH → ARNt-O-CO-CH(NH2)-R + AMP + enzima (aminoacil-AMP-enzima) (aminoacil-ARNt) Reacción global Aminoácido + ATP + ARNt → Aminoacil-ARNt + AMP +2Pi El producto final de la doble reacción es un aminoacil-ARNt, con un enlace éster entre el grupo 3´-OH terminal del ARNt y el grupo carboxilo del aminoácido. 16.1.1. Las aminoacil-ARNt sintetasas Tipos Existen dos tipos que se diferencian en la segunda reacción: a) Las de clase I forman el éster inicialmente sobre el grupo 2´-OH del ARNt, con lo que se necesita una transesterificación posterior. La mayoría son monoméricas. Reconocen los 10 aminoácidos más grandes y polares: Arg, Cys, Gln, Glu, Ile, Leu, Met, Trp, Tyr y Val. b) Las de clase II unen el aminoácido directamente al grupo 3´-OH del ARNt. Son diméricas y reconocen los otros 10 aminoácidos, más pequeños e hidrófobos. Especificidad Cada aminoacil-ARNt sintetasa reconoce un único aminoácido, (del que toma el nombre) y todos los ARNt cuyos anticodones codifican ese mismo aminoácido (ARNt sinónimos o isoaceptores). De acuerdo con esto, en la célula existen 20 aaRS distintas. El papel de estas enzimas es esencial en la fidelidad de la síntesis proteica. 208 Biología Molecular InspiracleBIR/2015 Mecanismo de reconocimiento de los aminoácidos por las sintetasas Es similar al reconocimiento de cualquier otro sustrato por la enzima correspondiente. Cada aminoácido encaja en un determinado lugar del centro activo de la enzima gracias a diversos tipos de interacciones. Mecanismo de reconocimiento de los ARNt por las sintetasas Los principales puntos de reconocimiento están en el brazo aceptor (extremo 3´) y en el brazo anticodón, a veces incluyendo al propio anticodón. Muchas de las sintetasas de la clase I reconocen el brazo del anticodón; muchas de las aaRS de la clase II reconocen sólo el brazo aceptor, algunas reconocen ambos brazos y las hay que requieren además otras secuencias y aspectos estructurales del ARNt. El mecanismo general de reconocimiento es diferente del de las proteínas de unión al ADN (que formaban puentes de hidrógeno con grupos de los surcos mayor y menor). El ARNt no es tan adecuado para interaccionar con la proteína como lo hace el ADN. 16.1.2. Corrección de errores cometidos por las aaRS La elevada especificidad de las aminoacil-ARNt sintetasas hace que los errores sean infrecuentes. Aún así, existen mecanismos de corrección que actúan sucesivamente en el centro activo de la enzima antes y después de cada etapa de la reacción de síntesis. A pesar de todos estos mecanismos de corrección, la tasa de error de la traducción es mucho mayor que la de la replicación, de uno por cada 10.000 aminoácidos (frente a uno por cada 106-108 nucleótidos en la replicación). 16.1.3 Balance energético de la activación En cada reacción de aminoacilación se hidrolizan dos enlaces fosfato de alta energía para formar el enlace entre el aminoácido y el ARNt. 16.2. INICIACIÓN. Es la etapa previa a la formación del primer enlace peptídico, determina la velocidad global de la síntesis y transcurre de forma semejante en procariotas y eucariotas salvo por las proteínas implicadas. 16.2.1. El punto de inicio. La traducción nunca comienza en el primer nucleótido del extremo 5´del ARNm, por eso siempre se representa una región líder 5´ no traducida. Sólo un codón AUG, situado internamente en la molécula, actúa como punto de inicio. Este AUG debe ser reconocido por el ribosoma y el ARNt iniciador. 209 Biología Molecular 16.6. InspiracleBIR/2015 INHIBIDORES DE LA TRADUCCIÓN Muchas sustancias impiden o dificultan la traducción, son importantes porque pueden emplearse como antibióticos, especialmente los que son selectivos sobre procariotas. La selectividad de los antibióticos y toxinas sobre organismos procariotas o eucariotas se debe a las diferencias en sus ribosomas y en algunos casos, como en las tetraciclinas, a la incapacidad de la molécula de atravesar la membrana de las células eucariotas. El efecto inhibidor puede ejercerse en distintas etapas del proceso de traducción: Etapa afectada Ejemplos Activación de lós Mupirocina aminoácidos (o ácido pseudomónico) Iniciación Estreptomicina (paso 2, complejo (un aminoglucósido) de preiniciación) Pactamicina Showdomicina Interferón Iniciación Eritromicina (paso 3, complejo de iniciación) Elongación (paso 4, ubicación) Elongación (paso 5, transpeptidación) Tetraciclinas Kirromicina Ricina (glicoproteína de origen vegetal; con 2 cadenas A y B; la primera es la toxina, 66 kDa) Cloranfenicol Cicloheximida Puromicina Efecto sobre Detalles de la acción Procariotas o Eucariotas P Inhibe competitivamente la Ile-tRNA sintetasa, evitando la incorporación de Ile y dteniendo la síntesis proteica P Se fija de modo irreversible a la subunidad menor 30S, por interacción con varias de sus proteínas y con el Rrna 16S. Distorsiona la entrada de fMet-tRNA iniciador y también produce errores de lectura de mRNA durante la elongación, al interferir con el apareamiento codón/anticodón E Impide la ubicación de Met-tRNA iniciador en la subunidad menor 40S E Impide la formación del complejo MettRNAi : elF-2 : GTP E Induce la expresión de una proteína quinasa que fosforila a elF-2, inactivándolo (de forma similar al HCl) P Se une a un sitio específico en el rRNA 23S de la subunidad mayor 50S. Impide la asociación con la subunidad menor; otra acción es interfiriendo la translocación. P>E Se unen a la subunidad menor, interfiriendo con la fijación del aa-tRNA al sitio A P Bloquea la disociación de GDP del factor EF-Tu (equivalente al eEF-1α eucariótico), lo que evita su salida del ribosoma. E Se une a proteínas de la subunidad mayor 60S, bloqueando la unión de aa-tRNA: Eef1α : GTP, y posiblemente también de Eef2(transpeptidación) Py Se une selectivamente a una proteína de la mitocondrial subunidad mayor 50S, interfiriendo en la interacción del aa-tRNA con el centro activo de la peptidiltransferasa, como inhibidor competitivo E Similarmente al cloranfenicol, inhibi la actividad peptidiltransferasa pero sólo en el ribosoma eucariótico (subunidad mayor 60S) PyE Análogo estructural del aa-tRNA, forma enlace con el péptido provocando su terminación prematura 217 InspiracleBIR/2015 18.2. Biología Molecular MECANISMOS DE ORIGEN DE LAS MUTACIONES GÉNICAS Existen dos grandes categorías de mutaciones: espontáneas e inducidas. Las espontáneas son las que se producen de manera natural, sin que se utilice ningún agente inductor específico, mientras que las inducidas son las que surgen artificialmente por la acción de un agente inductor. 18.2.1 MECANISMOS DE ORIGEN DE LAS MUTACIONES ESPONTÁNEAS Se producen principalmente como consecuencia de errores en la replicación o por la acción de procesos celulares que alteran las bases nitrogenadas, lo que se denominan lesiones espontáneas. A. Los errores replicativos de emparejamientos erróneos de bases, si no son reparados, originan transiciones. A.1. Uno de los mecanismos de producción de estos errores es la tautomería. En el ADN las formas tautoméricas predominantes de las bases son las formas ceto de la guanina y la timina y las amino de la citosina y de la adenina. Las formas tautoméricas raras son las enol de la G y la T y las imino de la C y la A. En ocasiones alguna base puede adquirir una forma tautomérica rara y durante la replicación se emparejará incorrectamente originando una transición. A.2. Otro tipo de fallo replicativo son los deslizamientos de las hélices en regiones con repetición de nucleótidos, lo que origina inserciones y deleciones de bases (según la cadena que sufra el deslizamiento) que finalmente darán lugar a cambios en la pauta de lectura del gen. En humanos estos deslizamientos de hélices, denominados bucles de inserción/deleción (IDL) deben ser frecuentes y existen mecanismos especiales de reparación de los mismos. Se ha demostrado en diversas enfermedades hereditarias que la patología se origina por el incremento en el número de tripletes en los genes responsables que muestran en su secuencia regiones con repeticiones de nucleótidos (p. ej., un determinado triplete repetido diez veces). Enfermedades causadas por expansión de tripletes repetidos - Corea de Huntington. - Distrofia miotónica. - Síndrome del X frágil. - Atrofia muscular espinobulbar. - Algunos tipos de ataxia. 238 Biología Molecular InspiracleBIR/2015 BIBLIOGRAFÍA - FERNÁNDEZ PIQUERAS, J. et al: Genética. Ariel Ciencia, 1ª ed., 2002. - GRIFFITHS, A., SUZUKI, D., LEWONTIN, R., MILLER, J.: Genética. McGrawHill Interamericana. 7ª ed., 2002. - KLUG, W.S., CUMMINIGS, M.R. y SPENCER, C.A.: Conceptos de Genética. Pearson Prentice Hall. Madrid, 8ª ed. 2006. - LUQUE, J. y HERRÁEZ, A.: Texto ilustrado de Biología Molecular e ingeniería genética. Barcelona, 1ª ed. 2008. - MENSUA, J. L.: Genética: Problemas y ejercicios resueltos. Pearson Prentice Hall. 2002. - NELSON, D.L. y COX, M.M.: Lehninger. Principios de Bioquímica. Ediciones Omega, Barcelona, 4ª ed., 2006. - PANIAGUA GÓMEZ ÁLVAREZ, R.: Citología e histología vegetal y animal. McGraw-Hill Interamericana.4ª ed., 2007. - SOLARI, A. J.: Genética humana: fundamentos y aplicaciones en medicina. Editorial Médica Panamericana. Buenos Aires, 3ª ed., 2004. - TAMARIN, R. H.: Genética. Editorial Reverté, S. A. España, 4ª ed., 1996. - BROWN, T.A. “Genomas” Editorial médica Panamericana. 3ª edición. 2008. Páginas web - http://www.ucm.es/info/genetica/grupod - http://www.drscope.com/pac/mg/b1/index.htm - http://www.es.embnet.org/-Imc/ - http://genmolecular.wordpress.com/ligamiento 347