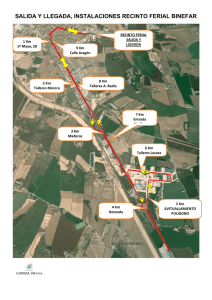
lacasas myceliophthora purificadas y acidos nucleicos que las
Anuncio

k OFICINA ESPAÑOLA DE PATENTES Y MARCAS 19 k 2 165 420 kInt. Cl. : C12N 15/53, C12N 1/15 11 Número de publicación: 7 51 ESPAÑA A61K 7/13, A61K 7/06 D21C 5/00, //(C12N 1/15 C12R 1:66) k TRADUCCION DE PATENTE EUROPEA 12 kNúmero de solicitud europea: 95921503.9 kFecha de presentación: 31.05.1995 kNúmero de publicación de la solicitud: 0 765 394 kFecha de publicación de la solicitud: 02.04.1997 T3 86 86 87 87 k 54 Tı́tulo: Lacasas myceliophthora purificadas y ácidos nucléicos que las codifican. k 30 Prioridad: 03.06.1994 US 253781 15.05.1995 US 441146 1445 Drew Avenue Davis, CA 95616, US Novozymes A/S k 72 Inventor/es: Berka, Randy M.; k 74 Agente: Tomás Gil, Tesifonte-Enrique 45 Fecha de la publicación de la mención BOPI: 16.03.2002 45 Fecha de la publicación del folleto de patente: 16.03.2002 ES 2 165 420 T3 k 73 Titular/es: Novozymes Biotech, Inc. Aviso: k Brown, Stephen H.; Xu, Feng; Schneider, Palle; Aaslyng, Dorrit Anita y Oxenböll, Karen M. k En el plazo de nueve meses a contar desde la fecha de publicación en el Boletı́n europeo de patentes, de la mención de concesión de la patente europea, cualquier persona podrá oponerse ante la Oficina Europea de Patentes a la patente concedida. La oposición deberá formularse por escrito y estar motivada; sólo se considerará como formulada una vez que se haya realizado el pago de la tasa de oposición (art. 99.1 del Convenio sobre concesión de Patentes Europeas). Venta de fascı́culos: Oficina Española de Patentes y Marcas. C/Panamá, 1 – 28036 Madrid ES 2 165 420 T3 DESCRIPCION Lacasas Myceliophthora purificadas y ácidos nucléicos que las codifican. 5 10 Campo de la invención La presente invención se refiere a fragmentos aislados de ácido nucleico que codifican una enzima oxidorreductasa micótica y las enzimas purificadas producidas de ese modo. Más particularmente, la invención se refiere a fragmentos de ácido nucleico que codifican una fenol-oxidasa, especı́ficamente una lacasa, de un ascomiceto termófilo, la Myceliophthora. Estado de la técnica 15 20 25 30 35 40 45 50 Las lacasas (oxidorreductasas de benzenodiol:oxı́geno) son enzimas que contienen multi-cobre que catalizan la oxidación de los fenoles. Las oxidaciones mediante lacasas dan como resultado la producción de intermediarios ariloxi-radicales a partir de un substrato fenólico adecuado; el acoplamiento definitivo de los intermediarios producidos de este modo provee una combinación entre los productos de la reacción dimérica, oligomérica y polimérica. Estas reacciones resultan importantes en cuanto a su naturaleza en las vı́as biosintéticas que conducen a la formación de melanina, alcaloides, toxinas, ligninas y ácidos húmicos. Las lacasas son producidas por una amplia variedad de hongos, incluyendo los ascomicetos como el Aspergillus, el Neurospora y el Podospora, el deuteromiceto Botrytis y los basidiomicetos como la Collybia, Fomes, Lentinus, Pleurotus, Trametes y las formas perfectas de Rizoctonia. La lacasa muestra un amplio margen de especificidad en el substrato y cada lacasa micótica diferente normalmente difiere sólo cuantitativamente de otras en cuanto a su capacidad para oxidar substratos fenólicos. Debido a la diversidad de substratos, por lo general las lacasas cuentan con una amplia variedad de aplicaciones industriales potenciales. Entre éstas, se pueden encontrar la modificación de ligninas, el refuerzo del papel, la inhibición de la transferencia de tintos en detergentes, la polimerización de fenoles, la producción de zumos, la producción de resinas fenólicas y el tratamiento de aguas residuales. Aunque las capacidades catalı́ticas son similares, las lacasas hechas a partir de especies micóticas diferentes presentan unos puntos óptimos de temperatura y pH diferentes, y estos a su vez también pueden ser diferentes dependiendo del substrato especı́fico. Se han aislado varias de estas lacasas micóticas y se han clonado los genes de varias de ellas. Por ejemplo, Choi et al. (Mol. Plant-Microbe Interactions 5: 119-128, 1992) describe la caracterización molecular y la clonación del gen que codifica la lacasa del hongo del chancro del castaño, la Cryphonectria parasitica. Kojima et al. (J. Biol. Chem. 265: 15224-15230, 1990; JP 2-238885) proporciona una descripción de dos formas alelas de la lacasa del basidiomiceto de pudrición blanca Coriolus hirsutus. Germann y Lerch (Experientia 41: 801,1985; PNAS EEUU 83: 88548858, 1986) han hecho mención de la clonación y la secuenciación parcial del gen de la lacasa Neurospora crassa. Saloheimo et al. (J. Gen. MicroBiol. 137: 1537-1544, 1985; WO 92/01046) han descrito un análisis estructural del gen de la lacasa del hongo Phlebia radiata. Los intentos de expresar los genes de la lacasa en sistemas micóticos heterólogos frecuentemente dan como resultado unos rendimientos muy bajos (Kojima et al., arriba; Saloheimo et al., Bio/Technol. 9: 987-990, 1991). Por ejemplo, la expresión heteróloga de la lacasa Phlebia radiata en Trichoderma reesei sólo dio como resultado 20 mg por litro de enzima activa (Saloheimo, 1991, arriba). Aunque las lacasas presentan un gran potencial comercial, su capacidad de expresar la enzima en cantidades significantes resulta fundamental para su utilidad comercial. En la actualidad no existe ninguna lacasa que se exprese a altos niveles en huéspedes empleados comercialmente como el Aspergillus. De este modo, existe la necesidad de obtener una lacasa que se pueda producir en cantidades comercialmente útiles (es decir, a gramo por litro o más). La presente invención satisface dicha necesidad. Resumen de la Invención 55 60 La presente invención se refiere a un constructo de ADN que contiene una secuencia de ácido nucleico que codifica una lacasa Myceliophthora. La invención también se refiere a una lacasa aislada codificada por la secuencia del ácido nucleico. Preferentemente, la lacasa es substancialmente pura. Por “substancialmente pura” se entiende una lacasa que está esencialmente libre (es decir ≥ 90 %) de otras proteı́nas que no sean lacasas. Para facilitar la producción de la nueva lacasa, la invención también comprende vectores y células huéspedes que incluyen la secuencia del ácido nucleico reivindicada, cuyos vectores y células huéspedes resultan útiles en la producción recombinante de la lacasa. La secuencia es enlazada operablemente a las señales de transcripción y traducción capaces de dirigir la expresión de la proteı́na lacasa en la célula 2 ES 2 165 420 T3 huésped elegida. Una célula huésped preferida es una célula micótica, aunque se prefiere aún más que sea del género Aspergillus. La producción recombinante de la lacasa según la invención se consigue cultivando una célula huésped transformada o modificada con el constructo según la invención, o sus derivados, bajo condiciones adecuadas para expresar la proteı́na lacasa y recuperar la proteı́na lacasa del cultivo. 5 Las lacasas según la presente invención resultan útiles en varios procesos industriales en los que se requiere la oxidación de fenoles. Estos procesos incluyen la manipulación de la lignina, la producción de zumos, la polimerización de fenoles y la producción de resinas fenólicas. 10 Breve Descripción de las Figuras La figura 1 muestra un mapa de restricción de un fragmento 7.5 EcoRI en pRaMB1. La región que hibrida la sonda del gen de la lacasa N. crassa aparece sombreada. 15 La figura 2 muestra las secuencia del nucleótido (ID SEC N◦ : 1) y del aminoácido (ID SEC N◦ : 2) de la lacasa Myceliophthora thermophila. Las letras escritas en minúsculas en la secuencia del nucleótido indican la posición de los intrones. Las secuencias TATA y CAAT putativas en la región del promotor aparecen en negrita y subrayadas. Las estructuras del enlace consensuado (PuCTPuAC) dentro de los intrones aparecen subrayadas. 20 La figura 3 ilustra el constructo del plásmido pRaMB5. Descripción detallada de la Invención 25 30 35 40 45 50 55 60 La Myceliophthora thermophila es un ascomiceto termófila descrita originariamente por Apinis (Nova Hedwigia 5: 57-78, 1963) y a la que se le da el nombre de Sporotrichum thermophile. Las subsecuentes revisiones taxonómicas han situado este organismo en el género de los Chrysosporium (Von Klopotek, A. Arch. Microbiol. 98: 365-369, 1974) y, posteriormente, en el género Myceliophthora (Van Oorschot, Persoonia 9: 401-408, 1977). Varios organismos conocidos con otros nombres también parecen pertenecer a esta especie. Entre estos se incluye el Sporotrichum cellulophilum (Patente U.S. N◦ 4,106,989); la Thielavia thermophila (Fergus y Sinden, Can. J. Botany 47: 1635-1637, 1968); el Chrysosporium fergussi y el Corynascus thermophilus (Von Klopotek, arriba). A esta especie se la conoce como fuente de varias enzimas diferentes industrialmente útiles, como las celulasas, la B-glucosidasa y la xilanasa (ver, p. ej., Oberson et al., Enzyme Microb. Technol. 14: 303-312, 1992; Merchant et al., Biotechnol. Lett. 10: 513-516, 1988; Breuil et al. Biotechnol. Lett. 8: 673-676, 1986; Gilbert et al., Bioresource Technol. 39: 147-154, 1992). Actualmente se ha determinado que la Myceliophthora produce una lacasa con un pH neutro y que el gen que codifica esta lacasa puede ser utilizado para producir grandes rendimientos de enzimas en sistemas de huéspedes convenientes como el Aspergillus. Para identificar la presencia de un gen de lacasa en la Myceliophthora, se emplea una porción 5’ del gen de la lacasa Neurospora crassa (Icc1) a modo de sonda, bajo condiciones de rigor leve, en una hibridación de Southern del ADN genómico total de las diferentes especies micóticas. Se detecta una secuencia especı́fica de lacasas de aproximadamente 12 kb en el ADN de la Myceliophthora. A continuación, se emplea el fragmento de N. crassa para filtrar aproximadamente 20.000 placas de una biblioteca de ADN genómico de M. thermophila en un vector de clonación bacteriófago λ EMBL4. Se hidridan ocho placas enérgicamente con la sonda; de entre estas ocho, se aı́sla el ADN de tres. Cada uno de estos clones contiene un fragmento 7.5 EcoRI que también se hibrida con la sonda (Figura 1). Se subclona uno de los fragmentos hasta obtener pBR322 para generar el plásmido pRaMB1. Al emplear la sonda Icc1 se determina la posición de la región de codificación del clon. La región de codificación de la M. thermophila completa parece estar contenida en un segmento de 3,2 kb de NheI-BgllI, que entonces se clona para obtener pUC119 y se secuencia a través del método “primer walking”. Una vez se ha determinado la secuencia, se asignan las posiciones de los intrones y exones dentro del gen basándose en la alineación de la secuencia de aminoácidos deducida del producto del gen de lacasa N. crassa correspondiente. Resulta que, según esta comparación, el gen (IccM) de M. thermophila está compuesto de siete exones (nucleótidos 246, 79, 12, 70, 973, 69 y 411) interrumpidos por seis intrones (nucleótidos 85, 84, 102, 72, 147, y 93). La región de codificación, excluyendo las secuencias que intervienen, es muy rica en GC (65,5 % de G+C) y codifica una preproenzima de 620 aminoácidos: un péptido con una señal de 22 aminoácidos, un propéptido de 25 aminoácidos y una lacasa madura que incluye 573 aminoácidos. La secuencia del gen de la M. thermophila y la secuencia predicha de aminoácidos se muestran en la figura 2 (ID SEC N◦ s: 1 y 2). 3 ES 2 165 420 T3 5 10 15 20 25 30 35 A continuación, se emplea el gen de la lacasa para crear un vector de expresión que transforme las células huéspedes de Aspergillus. El vector, pRaMB5 contiene el promotor de la TAKA-amilasa del A. oryzae y las regiones del terminador. El constructo de pRaMB5 aparece descrito en la figura 3. Las células Aspergillus se cotransforman con la ayuda del vector de expresión y un plásmido que contiene el marcador seleccionable pyrG o amdS. Los transformantes se seleccionan basándose en el medio selectivo apropiado que contiene el ABTS. Las colonias que producen lacasas presentan un halo verde y se pueden aislar inmediatamente. Los transformantes seleccionados son cultivados en frascos de agitación y en caldos de cultivo evaluados para detectar actividad lacasa a través del método de la siringaldazina. Los cultivos del frasco de agitación son capaces de producir 0,2 o más g/litro de lacasa y, en fermentación, se observan unos rendimientos de aproximadamente 1-2 g/litro. Según la invención, se puede obtener un gen de Myceliophthora que codifique una lacasa mediante los métodos descritos anteriormente o mediante cualquiera de los métodos alternativos conocidos en la materia, usando la información provista aquı́. El gen se puede expresar de forma activa mediante un vector de expresión. Un vector de expresión útil contiene un elemento que permite la integración estable del vector en el genoma huésped celular o la replicación autónoma del vector en una célula huésped independientemente del genoma celular huésped y, preferentemente, uno o más marcadores fenotı́picos que admitan una fácil selección entre las células huésped transformadas. El vector de expresión también puede incluir secuencias de control que codifiquen un promotor, el sitio de enlace del ribosoma, la señal de iniciación de la traducción y, opcionalmente, un gen represor o diferentes genes activadores. Para permitir la secreción de la proteı́na expresada, se pueden insertar los nucleótidos que codifican una señal de secuencia antes de codificar la secuencia del gen. Para efectuar la expresión bajo la dirección de las secuencias de control, se enlaza un gen de lacasa a emplear según la invención de forma que sea operable con las secuencias de control en el marco de lectura apropiado. Entre las secuencias del promotor que pueden ser incorporadas en los vectores plásmidos y que pueden dirigir la transcripción del gen de lacasa, se incluyen las siguientes (aunque no se limitan a ellas): el promotor de β-lactamasa procariótico (Villa -Kamaroff, et al., 1978, Proc. Natl. Acad. Sci. EE.UU. 75:3727-3731) y el promotor tac (DeBoer, et al., 1983, Proc. Natl. Acad. Sci. EE.UU. 80:21-25). También se pueden encontrar otras referencias en “Useful proteins from recombinant bacteria” en Scientific American, 1980, 242:74-94; y en Sambrook et al., Molecular Cloning, 1989. El vector de expresión que transporta el constructo de ADN según la invención puede ser cualquier vector que pueda ser sometido convenientemente a procedimientos de ADN recombinante y la elección del vector depende generalmente de la célula huésped en la que se va a introducir. De este modo, el vector puede ser un vector de replicación autónoma, es decir, un vector que existe como entidad extracromosómica, cuya réplica es independiente de la réplica cromosómica, p. ej., un plásmido, o un elemento extracromosómico, un minicromosoma o un cromosoma artificial. Alternativamente, el vector puede ser un vector que, cuando es introducido en una célula huésped, se integra en el genoma huésped celular y se replica junto con el(los) cromosoma(s) en el(los) que ha sido integrado. 40 45 50 55 60 En el vector, la secuencia de ADN de la lacasa se conectarı́a de manera operable a una secuencia adecuada del promotor. El promotor puede ser cualquier secuencia de ADN que muestre una actividad transcripcional en la célula huésped elegida y puede provenir de genes que codifiquen proteı́nas ya sean homólogas o heterólogas de la célula huésped. Algunos ejemplos de promotores adecuados para desarrollar la transcripción del constructo de ADN según la invención, especialmente en un huésped bacteriano, son el promotor del operón lac de E. coli, los promotores dagA del gen de agarasa de Streptomyces coelicolor, los promotores del gen de la α-amilasa del Bacillus ficheniformis (amyL), los promotores del gen de amilasa maltogénico del Bacillus stearothermophilus (amyM), los promotores de la α-amilasa del Bacillus amyloliquefaciens (amyQ) o los promotores de los genes xy1A y xy1B del Bacillus subtilis. En un huésped de una levadura, un promotor útil es el promotor eno-1. Para lograr la transcripción en un huésped micótico, algunos ejemplos de promotores útiles son los derivados del gen que codifica la TAKA-amilasa A. oryzae, la proteinasa aspártica Rhizomucor miehei, la α-amilasa neutral A. Niger, la α-amilasa ácida estable A. Niger, la glucoamilasa (glaA) A. Niger o A. awamori, la lipasa Rhizomucor miehei, la proteasa alcalina A. oryzae, la isomerasa de fosfato de triosa A. oryzae o la acetamidasa A. nidulans. Se prefieren la TAKA-amilasa y los promotores glaA. El vector de expresión según la invención también puede comprender un terminador de transcripción adecuado y, en las eucariotas, las secuencias de poliadenilación se pueden conectar operablemente a la secuencia de ADN que codifica la lacasa según la invención. Las secuencias de terminación y poliadenilación pueden provenir idóneamente de las mismas fuentes que el promotor. El vector puede comprender, además, una secuencia de ADN que permita que el vector se replique en la célula huésped en cuestión. Algunos ejemplos de este tipo de secuencias son los origenes de la replicación de los plásmidos pUC19, 4 ES 2 165 420 T3 pACYC177, pUB110, pE194, pAMB1 y plJ702. 5 10 El vector también puede comprender un marcador seleccionabe, p. ej., un gen, el producto del cual implementa un defecto en la célula huésped, como los genes dal de B. subtilis o B. licheniformis o uno que confiera resistencia antibiótica como la resistencia a la ampicilina, a la canamicina, al cloranfenicol o a la tetraciclina. Algunos ejemplos de marcadores de selección del Aspergillus incluyen el amdS, pyrG, argB, niaD, sC y hygB, dicho marcador incrementando la resistencia a la higromicina. Para su uso en una célula huésped de Aspergillus se prefieren los marcadores amdS y pyrG de A. nidulans o A. oryzae. Un marcador mamı́fero usado frecuentemente es el gen de la reductasa de dihidrofolato (DHFR). Además, la selección se puede llevar a cabo mediante cotransformación, p. ej., en el modo descrito en la WO 91/17243. 25 En general se prefiere que la expresión genere un producto que sea extracelular. De este modo, las lacasas según la presente invención pueden comprender una prerregión que permita la secreción de la proteı́na expresada en el medio de cultivo. En caso de que ası́ se desee, esta prerregión puede ser nativa en relación a la lacasa según la invención o sustituida por una prerregión o señal de secuencia diferente, llevándose a cabo de manera conveniente al sustituir las secuencias de ADN que codifican las prerregiones correspondientes. Por ejemplo, la prerregión puede derivarse de una glucoamilasa o un gen de amilasa de una especie de Aspergillus, un gen de amilasa de una especie de Bacillus, una gen de lipasa o de proteinasa de Rhizomucor miehei, el gen para el α-factor de Sacaromices cerevisiae o el gen de la preproquimosina de ternero. En aquellos casos en los que el huésped es una célula micótica, se prefiere particularmente la prerregión de la TAKA-amilasa A. oryzae, la amilasa neutra A. Niger, la forma Bacillus de la amilasa maltogénica NCIB 11837, la α-amilasa B. stearothermophilus o la subtilisina Bacillus licheniformis. Una señal de secuencia eficaz es la señal de la TAKA-amilasa A. oryzae, la señal de la proteinasa aspártica Rhizomucor miehei y la señal de la lipasa Rhizomucor miehei. 30 Los procedimientos empleados para enlazar el constructo de ADN según la invención con el promotor, el terminador y otros elementos, respectivamente, y para insertarlos en los vectores adecuados que contengan la información necesaria para efectuar la replicación son bien conocidos por los expertos en la materia (cf., por ejemplo, Sambrook et al. Molecular Cloning, 1989). 15 20 35 40 45 La célula según la invención que comprende bien un constructo de ADN bien un vector de expresión según la invención en el modo en que se ha definido anteriormente es empleada de forma ventajosa como célula huésped en la producción recombinante de una enzima según la invención. La célula puede ser transformada gracias al constructo de ADN en el cromosoma huésped. En general se considera que esta integración constituye una ventaja ya que es más probable que la secuencia de ADN se mantenga estable en la célula. La integración de constructos de ADN en el cromosoma huésped se puede llevar a cabo según los métodos convencionales, p. ej., mediante recombinación homóloga o heteróloga. Alternativamente, la célula puede ser transformada gracias a un vector de expresión como el descrito arriba en conexión con los diferentes tipos de células huésped. La célula huésped puede ser seleccionada de entre las células procarióticas como, por ejemplo, las células bacterianas. Algunos ejemplos de bacterias adecuadas son las bacterias gram positivas como el Bacillus subtilis, el Bacillus licheniformis, el Bacillus lentus, el Bacillus brevis, el Bacillus stearothermophilus, el Bacillus alkalophilus, el Bacillus amyloliquefaciens, el Bacillus coagulans, el Bacillus circulans, el Bacillus lautus, el Bacillus megaterium, el Bacillus thuringiensis, o el Streptomyces lividans o el Streptomyces murinus, o las bacterias gram negativas como la E. coli. La transformación de las bacterias puede, por ejemplo, llevarse a cabo transformando el protoplasto o empleando células competentes conocidas en cierto modo per se. 50 55 60 La célula huésped también puede ser un eucariota, como las células mamiferas, las células de insectos, las células vegetales o las células preferentemente micóticas, incluyendo la levadura y los hongos filamentosos. Por ejemplo, entre las células mamiferas útiles se incluyen las células CHO o las células COS. Una célula huésped de levadura puede ser seleccionada de entre una especie de Sacaromices o Schizosacaromices, p. ej., la Sacaromices cerevisiae. Los hongos filamentosos útiles pueden ser seleccionados a partir de una especie de Aspergillus, p. ej., el Aspergillus oryzae o el Aspergillus Niger. Alternativamente, se puede emplear una cepa de una especie de Fusarium, p. ej., el F. oxysporum, como célula huésped. Las células micóticas pueden ser transformadas mediante un proceso que implica la formación del protoplasto y la transformación de los protoplastos seguido de la regeneración de la membrana celular conocido en cierto modo per se. En la EP 238 023 se describe un procedimiento adecuado para transformar las células huésped de Aspergillus. Malardyer et al., 1989, describe un método adecuado para transformar una especie de Fusarium. 5 ES 2 165 420 T3 5 En consecuencia, la presente invención provee un método para producir una lacasa recombinante según la invención, cuyo método comprende el cultivo de una célula huésped en el modo descrito anteriormente bajo condiciones propicias para producir dicha enzima y la recuperación de la enzima de las células y/o el medio de cultivo. El medio que se emplea para cultivar las células puede ser cualquier medio convencional adecuado para el crecimiento de la célula huésped en cuestión y para la obtención de la expresión de la lacasa según la invención. Los medios adecuados pueden ser obtenidos de los proveedores comerciales o preparados según las fórmulas publicadas (p. ej., en los catálogos de la American Typer Culture Collection). 10 15 20 25 En una forma de realización preferida, la producción recombinante de lacasas en cultivo se consigue en presencia de una cantidad excesiva de cobre. A pesar de que los metales traza que se añaden al medio de cultivo contienen tı́picamente una pequeña cantidad de cobre, los experimentos llevados a cabo en conexión con la presente invención muestran que la adición de un suplemento de cobre al medio puede incrementar en gran medida el rendimiento de la enzima activa. Preferentemente, se añade cobre al medio en forma soluble, preferentemente en forma de sal cúprica soluble como, por ejemplo, el cloruro cúprico, el sulfato cúprico o el acetato cúprico. La concentración final de cobre en el medio se encontrarı́a en el margen de 0,2-2 mM, aunque se prefiere que se encuentre en el margen de 0,05-0,5 mM. Este método puede ser empleado para intensificar el rendimiento de cualquier lacasa micótica producida por recombinación, ası́ como otras enzimas que contengan cobre, particularmente las oxidoreductasas. La enzima resultante puede ser recuperada del medio a través de procedimientos convencionales entre los que se incluyen la separación de las células del medio centrifugándolas o filtrándolas, la precipitación de los componentes proteı́nicos del sobrenadante o filtrado mediante una sal, p. ej., sulfato amónico, seguido de la purificación mediante una variedad de procedimientos cromatográficos, p. ej., la cromatografı́a de intercambio iónico, la cromatografı́a de permeación sobre gel, la cromatografı́a de afinidad o similares. Preferentemente, la proteı́na aislada es aproximadamente pura al 90 % como se determinada a través de un sistema de dilución simple por electroforesis en gel de poliacrimida, siendo la pureza más importante en el caso de los alimentos, los zumos o las aplicaciones detergentes. 30 35 40 45 50 55 60 En una forma de realización particularmente preferida, se consigue la expresión de lacasa en una célula huésped micótica, como el Aspergillus. Como se describe con detalle en los siguientes ejemplos, el gen de la lacasa es ligado a un plásmido que contiene el promotor de la α-amilasa TAKA Aspergillus oryzae, ası́ como el marcador seleccionable amdS del Aspergillus nidulans. Alternativamente, el amdS se puede situar sobre un plásmido separado y se puede emplear en la cotransformación. El plásmido (o plásmidos) se emplea(n) para transformar una célula huésped de la especie Aspergillus, como el A. oryzae o el A. Niger, según los métodos descritos en Yelton et al. (PNAS EEUU 81: 1470-1474,1984). Resultará evidente a los expertos en la materia que la invención no se limita al uso de los fragmentos de ácido nucleico descritos aquı́ especı́ficamente, por ejemplo, en la figura 1. También les resultará evidente que la invención comprende aquellas secuencias de nucleótidos que codifican las mismas secuencias de aminoácidos representadas en la figura 1, aunque difieran de las secuencias de nucleótidos representadas especı́ficamente debido a la degeneración del código genético. Además, se entenderá que la referencia que se hace a la figura 1 tanto en la descripción como en las reivindicaciones engloba tanto la secuencia genómica representada en ella como las secuencias de ADNc y ARN correspondientes, al mismo tiempo, se entenderá que las frases “constructo de ADN” y “secuencias de ácidos nucleicos” en el modo en que se emplea aquı́ engloban todas las variaciones anteriormente mencionadas. En general, se entiende que el término “constructo de ADN” hace referencia a una molécula de ADN, bien mono o bicatenaria, que puede ser parcialmente aislada de un gen de origen natural o que ha sido modificada para que contenga los segmentos de ADN combinados y yuxtapuestos de modo que, de lo contrario, no podrı́an existir en la naturaleza. La lacasa Myceliophthora descrita aquı́ tiene una actividad especı́fica particularmente alta sobre un substrato de siringaldazina en relación con otras lacasas extracelulares de ascomiceto o deuteromiceto conocidas en las se ha descrito dicha actividad especı́fica. La presente secuencia provee un medio a través del cual se pueden aislar otras lacasas de ascomiceto y/o deuteromiceto. La identificación y el aislamiento de los genes de lacasa derivados de fuentes diferentes a las que aparecen en los ejemplos de la presente se pueden conseguir empleando la metodologı́a descrita en los presentes ejemplos, con las cepas disponibles públicamente de ascomiceto y deuteromiceto. En particular, la secuencia especı́fica descrita aquı́ se puede emplear para diseñar cebadores y/o sondas que resulten útiles para aislar genes de lacasa similares mediante una RCP (reacción en cadena de la polimerasa) estándar o mediante técnicas de hibridación de Southern. En consecuencia, la presente invención engloba dichas lacasas de ascomiceto y deuteromiceto 6 ES 2 165 420 T3 que presentan una actividad especı́fica de al menos unos 30 SOU/mg, preferentemente de al menos unos 40 SOU/mg, “SOU” estando definido como µmol del substrato oxidado por minuto medido mediante siringaldazina como substrato, con un pH óptimo. 5 10 15 20 25 30 Además, la invención también engloba otras lacasas Myceliophthora, incluyendo las formas alternativas de lacasa que se pueden encontrar en la M. thermophila, ası́ como las lacasas que se pueden encontrar en otros hongos incluidos dentro de la definición de Myceliophthora tal y como se define en Van Oorschot, 1977 arriba. La identificación y el aislamiento de los genes de lacasa de fuentes diferentes a las que se especifican en los ejemplos de la presente se pueden conseguir empleando la metodologı́a descrita en los presentes ejemplos, con las cepas disponibles al público de Myceliophthora. Alternativamente se puede emplear la secuencia descrita aquı́ para diseñar cebadores y/o sondas útiles para aislar genes de lacasa con la ayuda de la RCP estándar o mediante técnicas de hibridación de Southern. Otras especies nombradas de Myceliophthora incluyen la Myceliphthora hinnulea (Awao et al., Mycotaxon. 16: 436-440, 1983), la Myceliophthora vellerea (Guarro et al, Mycotaxon. 23: 419-427, 1985) y la Myceliophthora lutea Costatin. También se engloban las lacasas que son sinonı́micas, p. ej., los estados anamorfos o perfectos de especies o variedades del género Myceliophthora. Las variedades de Myceliophthora están disponibles al público en varias colecciones de cultivos, como ATCC 48102, 48103, 48104 et al. CBS 117.65, 131.65, 379.65 et al., DSM. 1799 (M. thermophila), ATCC 52474, CBS 539.82, 540.82 et al. (M. hinnulea), DSM 62114, CBS 146.50, 147.50, 157.51 et al (M. lutea) y CBS 478.76, 479.76 y 715.84 (M. vellerea). La invención también engloba cualquier secuencia de nucleótidos variante y la proteı́na codificada por ella, proteı́na que retiene, como mı́nimo, aproximadamente una homologı́a del 80 %, aunque se prefiere que sea al menos del 85 %, y más preferentemente como mı́nimo del 90-95 %, con la secuencia de aminoácidos representada en la figura 1, y que retiene cualitativamente la actividad lacasa de la secuencia descrita aquı́. Las variantes útiles dentro de las categorı́as definidas anteriormente incluyen, por ejemplo, aquellas en las que las se han llevado a cabo las sustituciones conservadoras del aminoácido, sustituciones que no influyen significativamente en la actividad de la proteı́na. Por substitución conservadora se entiende que los aminoácidos de la misma clase pueden ser sustituidos por cualquier otro de dicha clase. Por ejemplo, los residuos alifáticos no polares Ala, Val, Leu e Ile se pueden intercambiar, como pueden serlo los residuos básicos Lys y Arg o los residuos acı́dicos Asp y Glu. De forma similar, Ser y Thr son mutuamente sustituciones conservadoras, como lo son Asn y Gln. A los expertos en la materia les resultará evidente que dichas sustituciones pueden efectuarse en la parte externa de las regiones fundamentales para la función de la molécula y, además, dar como resultado una enzima activa. La retención de la actividad deseada puede determinarse fácilmente conduciendo un método de oxidación de ABTS estándar, como se describe en los presentes ejemplos. 35 40 45 50 55 La proteı́na se puede emplear en un número de procesos industriales diferentes. Estos procesos incluyen la polimerización de lignina, el papel kraft y los lignosulfatos, en solución, con el objetivo de producir una lignina con un peso molecular más elevado. Una lacasa neutra/alcalina constituye una ventaja particular en la que la lignina kraft es más soluble a pHs más altos. Estos métodos aparecen descritos en, por ejemplo, Jin et al., Holzforschung 45(69): 467-468, 1991; en la Patente USA N◦ 4,432,921; EP 0 275 544; PCT/DK93/00217,1992. La lacasa según la presente invención también se puede emplear para llevar a cabo la depolimerización in situ de la lignina en la pasta de papel kraft, produciendo de ese modo una pasta de papel con un contenido inferior en lignina. Este uso de la lacasa constituye una mejora en relación al uso actual del cloro para llevar a cabo la depolimerización de la lignina, que conduce a la producción de compuestos aromáticos clorurados, que son subproductos anti-ecológicos de las fábricas de papel. Dichos usos aparecen descritos en, por ejemplo, Current Opinion in Biotechnology 3: 261-266, 1992; J. Biotechnol. 25: 333-339, 1992; Hiroi et al., Svensk papperstidning 5: 162 -166, 1976. Puesto que el ambiente en una fábrica de papel es tı́picamente alcalino, la presente lacasa resulta más útil que otras lacasas conocidas, que funcionan mejor bajo condiciones acı́dicas. La oxidación de los tintes o de los precursores del tinte, ası́ como de otros compuestos cromóforos conlleva la decoloración de los compuestos. La lacasa se puede emplear con este objetivo, pudiendo resultar particularmente ventajosa en una situación en la que no se desee que se produzca una transferencia de tintes entre los tejidos, p. ej., en la industria textil y en la industria de los detergentes. Se pueden hallar métodos para inhibir la transferencia de tintes y la oxidación de tintes en WO 92/01406; WO 92/18683; EP 0495836; Calvo, Mededelingen van de Faculteit Landbouwwetenschappen/Rijiksuniversitet Gent. 56: 1565-1567, 1991 Tsujino et al., J. Soc. Chem. 42: 273-282, 1991. 60 La lacasa resulta particularmente adecuada para su uso en tintes para el pelo. En este tipo de aplicación, la lacasa entra en contacto con un precursor del tinte, preferentemente en el pelo, con lo que se 7 ES 2 165 420 T3 5 10 15 20 25 30 35 40 consigue una oxidación controlada del precursor del tinte para convertir el precursor en un tinte o un pigmento que produce un compuesto, como un compuesto quinoide. El precursor del tinte es preferentemente un compuesto aromático que pertenece a una de las tres familias quı́micas mayores: las diaminas, los aminofenoles (o aminonaftoles) y los fenoles. Los precursores del tinte pueden ser empleados por sı́ solos o combinados. Al menos uno de los intermediarios en la copolimerización debe ser una ortoo para-diamina o un aminofenol (intermediario primario). Se pueden encontrar algunos ejemplos en la sección IV, abajo, entre los que se incluyen la p-fenileno-diamina (pPD), la p-toluileno-diamina, la clorop-fenilenodiamina, el p-aminofenol, el o-aminofenol o el 3,4-diaminotolueno, los compuestos adicionales también aparecen descritos en la patente US N◦ 3,251,742, cuyo contenido se incorpora aquı́ como referencia. En una forma de realización, los materiales de partida incluyen no sólo la enzima y un intermediario primario, sino también un modificador (acoplador) (o combinación de modificadores), modificador que es tı́picamente una meta-diamina, un meta-aminofenol o un polifenol. Algunos ejemplos de compuestos para modificadores incluyen la m-fenileno-diamina, el 2,4-diaminoanisol, el α-naftol, la hidroquinona, el pirocatecol, el resorcinol y el 4-clororesorcinol. El modificador reacciona ante el intermediario primario en presencia de la lacasa, convirtiéndola en un compuesto coloreado. En otra forma de realización se puede emplear la lacasa con el intermediario primario directamente para oxidarlo hasta conseguir un compuesto coloreado. En cualquier caso, el proceso de coloración se puede llevar a cabo con la ayuda de uno o más intermediarios primarios, bien solos o combinados con uno o más modificadores. Las cantidades de componentes están en consonancia con las cantidades comerciales usuales para componentes similares y las proporciones de componentes pueden variar en consecuencia. El uso de esta lacasa constituye una mejora en relación al uso más tradicional del H2 O2 , en el que éste último puede dañar el cabello y su uso normalmente requiere un pH elevado, que también resulta nocivo para el cabello. Por el contrario, la reacción con lacasas se puede llevar a cabo con un pH alcalino, neutro o incluso ácido, y el oxı́geno necesario para que se desarrolle la oxidación provienen del aire, más que a través de una oxidación quı́mica fuerte. El resultado proporcionado por el uso de la lacasa Myceliophthora es comparable al que se consigue con el uso de H2 O2 , no sólo en cuanto al desarrollo del color, sino también en lo que concierne a la estabilidad ante los lavados y a la fotoestabilidad. Una ventaja comercial adicional radica en que se puede fabricar un único paquete recipiente que contenga tanto la lacasa como el precursor, en una atmósfera libre de oxı́geno, cuya disposición no es posible con el uso de H2 O2 . También se puede emplear la presente lacasa para polimerizar compuestos fenólicos presentes en lı́quidos. Un ejemplo de este uso concierne al tratamiento de zumos, como el zumo de manzana, de modo que la lacasa acelerará una precipitación de los compuestos fenólicos presentes en dicho zumo, produciendo de ese modo un zumo más estable. Dichas aplicaciones aparecen descritas en Stutz, Fruit processing 7/93, 248-252, 1993; Maierer et al., Dt. Lebensmittel-rindschau 86(5): 137-142, 1990, Dietrich et al., Fluss. Obst 57(2): 67-73, 1990. Las lacasas como la lacasa Myceliophthora también resultan útiles en la detoxificación del suelo (Nannipieri et al., J. Environ. Qual. 20: 510 -517,1991; Dec y Bollag, Arch. Environ. Contam. Toxicol. 19: 543-550, 1990). La invención es ilustrada, además, a través de los siguientes ejemplos no limitativos. 45 Ejemplos I. Aislamiento del gen de lacasa Myceliophthora thermophila 50 A. Materiales y métodos 1. Extracción de ADN y análisis de hibridación 55 60 El ADN celular total se extrae de las células micóticas de la cepa E421 de Myceliophthora thermophila desarrolladas durante 24 horas en 25 ml de medio YEG (0,5 % de extracto de levadura, 2 % de glucosa) usando el siguiente protocolo: se recogen micelios mediante filtración a través de un filtro Miracloth (Calbiochem) y se lavan una vez con 25 ml de tampón con TE. Se drena el tampón sobrante de los micelios, que posteriormente se congelan en nitrógeno lı́quido. Los micelios congelados se reducen a un polvo fino en un molinillo de café eléctrico y se añade dicho polvo a 20 ml de tampón con TE y 5 ml de SDS al 20 % (p/v) en un tubo centrifugador plástico desechable. Se invierte la mezcla poco a poco varias veces para asegurar dicha mezcla y se extrae dos veces con un volumen idéntico de alcohol de fenol:cloroformo:isoamil (25:24:1). Se añade acetato sódico (3M de solución) para dar como resultado 8 ES 2 165 420 T3 5 10 15 20 25 30 una concentración final de 0,3 M y se precipitan los ácidos nucleicos con 2,5 volúmenes de etanol helado. Se centrifugan los tubos a 15.000 x g durante 30 minutos y se deja que el granulado se seque al aire durante 30 minutos antes resuspenderlo en 0,5 ml de tampón con TE. Se añade la ribonucleasa libre de DNase A a una concentración de 100 µg/ml y se incuba la mezcla a 37◦C durante 30 minutos. Se añade la proteinasa K (200 µg/ml) y se incuba cada tubo durante una hora más a 37◦C. Finalmente, se extrae cada muestra dos veces con alcohol de fenol :cloroformo:isoamil antes de precipitar el ADN con acetato sódico y etanol. Se deja que los granulados de ADN se sequen al vacı́o, se resuspenden en un tampón con TE y se almacenan a 4◦ C. Se analizan las muestras de ADN celular total derivadas de los transformantes y una cepa de control no transformada mediante hibridación southern. Aproximadamente 5 µg de ADN son digeridos con EcoRI y se fraccionan por tamaño sobre un gel de agarosa al 1 %. Se fotografı́a el gel bajo UV de longitud de onda corta y se sumerge durante 15 minutos en 0,5 M de NaOH, 1,5 M de NaCl seguido de 15 minutos en 1 M de Tris-HCl, pH 8, 1,5 M de NaCl. El ADN en el gel se transfiere a una membrana de hibridación Zeta-ProbeTM (Laboratorios BioRad) mediante transferencia capilar en 20 X SSPE (R. W. Davis et al., Advanced Bacterial Genetics, A Manual for Genetic Engineering. Cold Spring Harbor Press. 1980). Se cuecen las membranas durante 2 horas a 80◦C al vacı́o y se sumergen durante 2 horas en el siguiente tampón de hibridación a 45◦ C removiéndolo suavemente: 5 X SSPE, formamida al 35 % (v/v), SCS al 0,3 %, 200 µg/ml de ADN de testı́culos de salmón desnaturalizados y cortados. El fragmento de la sonda especı́fica con lacasas (aprox. 1,5 kb) que codifica la porción 5’ del gen lcc1 de N. crassa se amplifica a partir del ADN genómico de la N. crassa usando unas condiciones de RCP estándar (Perkin-Elmer Cetus, Emeryville, CA) con el siguiente par de cebadores: el cebador delantero, 5’CGAGACTGATAACTGGCTTGG 3’; el cebador inverso, 5’ACGGCGCATTGTCAGGGAAGT 3’. El segmento de ADN amplificado se clona en primer lugar en un vector que clona el TA (Invitrogen, Inc., San Diego, CA); a continuación, se purifica mediante electroforesis en gel de agarosa que sigue la digestión con EcoRI. Se radiomarca el fragmento purificado de la sonda mediante traslación por muescas con α[32P]dCTP (Amersham) y se añade al tampón de hibridación bajo una actividad de aproximadamente 1 x 106 cpm por ml de tampón. Se incubó la mezcla durante toda la noche a 45◦ C en un baño marı́a removido. Tras la incubación, se lavan las membranas una vez en 0,2 X SSPE con SDS al 0,1 % a 45◦ C seguido de dos lavados en 0,2 X SSPE (ningún SDS) a la misma temperatura. Se deja que las membranas se sequen en toallas del papel durante 15 minutos, a continuación se envuelven con Saran WrapT M y se exponen a una a pelı́cula de rayos X durante toda la noche a -70◦C mediante filtros intensificadores (Kodak). 2. Bibliotecas de ADN e identificación de clones de lacasas 35 40 45 50 55 Las bibliotecas de ADN genómico se construyen en el vector bacteriófago de clonación λ-EMBL4 (J.A.Sorge, en Vectors, A Survey of molecular Cloning Vectors and Their Uses, Rodriguez et al., eds, págs. 43-60, Butterworths, Boston, 1988). Resumidamente, el ADN celular total se dirigiere parcialmente con Sau3A y se divide por tamaño en geles de agarosa con un bajo punto de fusión. Se cortan los fragmentos de ADN que emigran entre 9kb y 23 kb y se eluyen del gel usando β-agarasa (New England Biolabs, Beverly MA). Los fragmentos de ADN eluido se ligan con los brazos del vector λ-EMBL4 dividido con BamHI y defosforilado, y se envuelven las mezclas de ligación mediante extractos de embalaje comerciales (Stratagene, La Jolla, CA). Las bibliotecas de ADN envueltas se sitúan en placas y se amplifican sobre células K802 de Escherichia coli. Aproximadamente 10.000-20.000 placas de cada biblioteca se filtran a través de una hibridación por placas con el fragmento de ADN Icc1 radiomarcado usando las condiciones descritas anteriormente. Las placas que transmiten las señales de hibridación con la sonda se purifican dos veces en células K802 de E. coli y se purifica el ADN del fago correspondiente a partir de lisatos de alta concentración usando un kit Qiagen Lambda (Qiagen, Inc., Chatsworth, CA). 3. Análisis de los genes de la lacasa El mapeo de restricción de los clones de la lacasa se crea al emplear métodos estándar (Lewin, Genes. 2a¯ ed., Wiley & Sons, 1985, Nueva York). La secuenciación de ADN se lleva a cabo mediante un secuenciador de ADN automatizado modelo 373A de Applied Biosystems (Applied Biosystems, Inc., Foster City, CA) usando la técnica primer walking con la quı́mica del terminador para tintes (H. Giesecke et al., J. Virol. Methods 38: 47-60, 1992). Los cebadores de la secuenciación de los oligonucleótido se sintetizan en un sintetizador de DNA/RNA Modelo 394 de Applied Biosystems. 60 9 ES 2 165 420 T3 B. Resultados y discusión 1. Identificación de la Secuencia del gen de la lacasa 5 10 Las muestras del ADN celular total se obtienen a partir de las especies Neurospora crassa, Botritis cinerea y Myceliophthora. Las partes alı́cuotas de estas preparaciones de ADN se digieren con BamHI y se destilan mediante electroforesis en gel de agarosa. El ADN en el gel se filtra en un filtro de membrana Zeta-ProbeT M (Laboratorios BioRad, Hercules, CA) y se somete a examen bajo condiciones de rigor leves con un fragmento radiomarcado que codifica una porción del gen Icc1 de la N. crassa, como se describe arriba. Las secuencias especı́ficas con las lacasas se detectan en los genomas de las M. thermophila y N. crassa de control, aunque no en el ADN genómico de B. cinerea con esta sonda. 2. Clonación y caracterización del gen de la lacasa Myceliophthora thermophila (MtL) 15 20 25 30 35 40 45 50 55 60 Se filtran aproximadamente 20.000 placas de una biblioteca de ADN genómica de M. thermophila construida en un vector de clonación λ-EMBL4. La biblioteca está compuesta de aproximadamente 10.000 clones independientes con insertos que varı́an en tamaño de 9kb a 23kb. Asumiendo un tamaño medio de inserto de 10 kb y un tamaño de genoma total de 4 x 107 bp para la M. thermophila, esta cifra equivale aproximadamente a 2,5 veces el número de clones requeridos para representar el genoma entero. Se identifican ocho placas que hibridaron fuertemente la sonda del gen de la lacasa N. crassa. Se aı́sla el ADN de tres de ellas, se divide con EcoRI y se analiza mediante electroforesis en gel de agarosa e hibridación southern. Estos tres clones contienen un fragmento EcoRI de 7,5 kb que hibrida la sonda especı́fica de la lacasa. Se subclona uno de estos fragmentos EcoRI en pBR322 (Bolivar et al., Gene 2: 95-113, 1977) para generar el plásmido pRaMB1. En la figura 1 se muestra un mapa de restricción de este segmento de ADN. La posición de la región de codificación de la lacasa en este clon se determina mediante hibridación con el fragmento del gen Icc1 descrito anteriormente. Basándose en los datos obtenidos del mapeo y para un tamaño estimado de proteı́na lacasa de aproximadamente 80 kdal, se estima que la región de codificación de la lacasa M. thermophila entera está contenida en un segmento NheI-Bg1II de 3,2 kb que, a continuación, se subclona en pUC119 (Viera y Messing, Methods Enzymol. 153: 3-11, 1987). La secuencia de nucleótidos de este segmento se determina usando el método primer walking (Giesecke et al., arriba). La secuencia del ácido nucleico se muestra en la figura 2 y en la ID de SEC N◦ : 1. La secuencia de aminoácidos deducida de MtL se obtiene a partir de una homologı́a de secuencias de aminoácidos con la lacasa N. crassa. En el nivel aminoácido, estas dos lacasas comparten aproximadamente el 60 % de la identidad de la secuencia. Esta semejanza es mucho mayor en aquellas regiones que corresponden a las cuatro histidinas y a una cisteı́na implicadas en la formación de grupos de cobre trinucleares (Perry et al., J. Gen. Microbiol. 139: 1209-1218, 1993; Coll et al. Appl. Environ. Microbiol. 59: 4129-4135, 1993; Messerschmidt et al. J. Mol. Biol. 206: 513-530, 1989). Existen 11 sitios potenciales para la glicosilación N-vinculada en la secuencia de aminoácidos deducida de MtL. Los primeros 22 aminoácidos de MtL parecen comprender un péptido con una señal canónica con una disociación predicha detrás de un residuo Ala (vonHeijne, J. Mol. Biol. 173:243-251, 1984). Aunque se desconoce la secuencia amino terminal de la MtL nativa, el terminal amino de la MtL recombinante producida en A. oryzae se ve bloqueada por un residuo del piro-glutamato. La extracción enzimática de este residuo seguida de la secuenciación del aminoácido sugiere que la MtL madura inicia con un residuo Gln (posición 1 en la figura 2; ID de SEC N◦ : 2). De este modo, la MtL se sintetiza claramente como preproenzima de 620 aminoácidos que presenta un péptido con la señal de 22 aminoácidos y un propéptido de 25 residuos. La lacasa Neurospora crassa (NcL) se procesa de forma similar a su amino terminal final. Además, la NcL también se procesa proteolı́ticamente en su C-terminal, cosa que se deriva en la extracción de 13 aminoácidos (Germann et al. J. Biol. Chem. 263: 885-896, 1988). El sitio del procesamiento se engloba dentro de la secuencia Asp-Ser-Gly-Leu∗Arg558 (donde ∗ designa el sitio de disociación). Existe una secuencia similar cerca el C-terminal final de MtL (Asp-Ser -Gly-Leu-LYS560), sugiriendo que la enzima Myceliophthora también se puede ver sometida al procesamiento C-terminal (Asp-Ser-Gly-Leu∗Lys560 ) que eliminarı́a 12 aminoácidos. Las posiciones de seis intrones (nucleótidos 85, 84, 102, 72, 147 y 93) dentro de la región que codifica el Icc1 se determinan comparando la secuencia de aminoácidos deducida de MtL a la de NcL y aplicando las reglas consensuadas sobre las caracterı́sticas de los intrones en hongos filamentosos (Gurr et al., en Gene Structure in Eukaryotic Microbes, J.R. Kinghorn, ed., págs. 93-139, IRL Press, Oxford, 1987). Los 1860 nucleótidos de la secuencia de codificación, excluyendo los intrones, son ricos en guanosina y citosina (65,5 % de G+C). Las pautas de uso de codones en este gen refleja la composición básica del ADN en una fuerte desviación (89,7) para aquellos codones que finalizan en G o C. 10 ES 2 165 420 T3 II. Expresión de la lacasa Myceliophthora en Aspergillus A. Materiales y métodos 5 10 1. Variedades del huésped bacteriano y variedades micóticas La Escherichia coli JM101 (Messing et al., Nucl. Acids Res. 9:309-321, 1981) se emplea como huésped para construir y propagar la rutina de los vectores de expresión de una lacasa en este estudio. Los huéspedes micóticos para la expresión de la lacasa incluı́an las variedades de Aspergillus Niger Bo-1, AB4.1 y AB1.13 (Mattern et al., Mol. Gen. Genet. 234: 332-336), ası́ como un mutante que requiere uridina (pyrG) de la cepa HowB104 del Aspergillus oryzae α-amilasa deficiente. 2. Plásmidos 15 20 25 El plásmido pRaMB2 es un derivado de pUC119 que contiene un fragmento de Bg1II-NheI de 3,2 kb de ADN genómico de M. thermophila que codifica MtL. El vector pMWR se construye insertando el promotor de la TAKA-amilasa A. oryzae y los elementos del terminador pTAKA17 (Christensen et al., Bio/Technol. 6: 1419-1422, 1988; EP 238 023) dentro de pUC18 (Yanisch-Perron et al., Gene 33: 103-119, 1985). En este vector, hay un único sitio SwaI en el extremo del elemento del promotor y un único sitio NsiI al comienzo del terminador para lograr la clonación direccional de las secuencias de codificación. El vehı́culo de clonación pUC518 se deriva insertando un pequeño enlazador que contiene sitios de restricción NsiI, ClaI, XhoI y Bg1II entre los sitios adyacentes BamHI y XbaI de pUC118 (Vieira y Messing, arriba). El plásmido pToC68 (WO 91/17243) contiene el promotor de la TAKA-amilasa de A. oryzae y el terminador glaA de A. Niger y el pToC90 (WO 91/17243) transporta el gen amdS de A. nidulans. 3. Construcción de Vectores de expresión de la lacasa 30 35 40 La estrategia de construcción para el vector de expresión de la lacasa pRaMB5 aparece representado en la figura 3. El promotor que dirige la transcripción del gen de la lacasa se obtiene a partir del gen de la α-amilasa (TAKA-amilasa) de A. oryzae (Christensen et al., arriba), ası́ como la región del terminador de la TAKA-amilasa. El plásmido se construye en primer lugar al modificar pMWR3 insertando un pequeño enlazador que contiene un sitio ApaI entre los sitios SwaI y NsiI, creando un plásmido llamado pMWR3SAN. El RCP dirigido por la polimerasa PfuI (Stratagene, La Jolla, CA) se emplea para amplificar un segmento de ADN corto que codifique la porción 5’ de MtL, desde el codón de iniciación hasta un sitio PstI interno (aproximadamente 0,5 kb). El cebador delantero de esta reacción RCP está diseñado para crear un sito EcoRI justo en la parte superior del codón de iniciación. A continuación, el fragmento amplificado se digiere con EcoRI y PstI [durante este paso, se debilita el sito EcoRI mediante un tratamiento con dNTPs y la ADN polimerasa 1 (fragmento Klenow)] y se purificó mediante electroforesis en gel de agarosa. La porción 3’ de la región de codificación de la M. thermophila se escinde de pRaMB2 como un fragmento PstI-ApaI de 2kb (este segmento también contiene aproximadamente 110 bp de la región 3’ no trasladada). Estos dos fragmentos se combinan con pMWR3-SAN divididos con SwaI y ApaI en una ligación de reacción en tres partes para generar el vector de expresión de la lacasa pRaMB5. 4. Transformación de las células huésped del Aspergillus 45 50 55 60 Los métodos para llevar a cabo la cotransformación de las cepas de Aspergillus son tal y como se han descrito en Christensen et al., arriba. Para introducir los vectores de expresión HowB 104 pyrG de la lacasa en A. oryzae, se emplean cantidades iguales (aproximadamente de 5 µg cada uno) de vector de expresión de la lacasa y uno de los siguientes plásmidos: pPYRG (Fungal Genetics Stock Center, Kansas City, KS) que contiene el gen pyrG de A. nidulans (Oakley et al, Gene 61385-399, 1987)- pSO2 que alberga los clones del gen pyrG de A. oryzae; pPRYG24 que contiene el gen pyrG de A. ficuum (=A. niger). Los transformantes protróficos (Pyr+ ) se seleccionan a partir de un medio mı́nimo de Aspergillus (Rowlands y Girador, Mol. Gen. Genet. 126: 201-216, 1973) y los transformantes son transformantes en los que se busca la capacidad de producir lacasas en un medio mı́nimo que contenga 1 mM de 2,2’azinobis(3-ácido etilbenztiazolinesulfónico) [ABTS]. Las células que segregan la lacasa activa oxidan el ABTS, produciendo un halo verde que rodea la colonia. En último lugar, los protoplastos Bo-1 de A. Niger se cotransforman al emplear cantidades idénticas (aproximadamente, de 5 µg cada una) de vector de expresión de la lacasa y pToC90 que contiene el gen amdS de A. nidulans (acetamidasa) (Hynes et al., Mol. Cell Biol. 3: 1430-1439, 1983). Los transformantes AmdS+ se seleccionan a partir de un medio mı́nimo Cove (Cove, Biochim. Biophys. Acta 113: 51-56, 1966) con glucosa al 1 % a modo de fuente de carbono y acetamida a modo de fuente de nitrógeno único y en ellos se busca que expresen la lacasa en un medio cove con 1 mM de ABTS. 11 ES 2 165 420 T3 5. Análisis de transformantes productores de lacasas 5 10 15 Se purifican dos veces los transformantes que producen una actividad de lacasas sobre las placas de agar a través de conidiosporas y se crean suspensiones de esporas en Tween-80 estéril al 0,01% a partir de cada una. La densidad de esporas en cada suspensión se calcula espectrofotométricamente (A595 nm). Se emplean aproximadamente 0,5 unidades de absorbencia de esporas para inocular 25 ml de medio con ASPO4 o MY50 en frascos plásticos de 125 ml. Se incuban los cultivos a 37◦ C con aireación vigorosa (aproximadamente 200 rpm) durante cuatro o cinco dı́as. Se recogen los caldos de cultivo al centrifugarlos y se determina la cantidad de actividad de lacasas en el sobrenadante usando siringaldazina a modo de substrato. En resumen, se mezclan 800 µl de tampón de ensayo (25 mM de acetato sódico, pH 5.5, 40 µM de CuSo4 ) con 20 µl de sobrenadante de cultivo y 60 µl de 0,28 mM de siringaldazina (Sigma Chemical Co., St. Louis, MO) en EtOH al 50 %. La absorbencia a 530 nm se mide en el tiempo en un espectrofotómetro UV-vis Genesys 5 (Milton-Roy). Una unidad de lacasa (LACU) se define como la cantidad de enzima que oxida un µmol de substrato por minuto a temperatura ambiente. La electroforesis en gel de SDS-poliacrilamida (PAGE) se lleva a cabo usando geles de gradiente prefundidos al 10-27 % de Novex (San Diego, CA). Las bandas proteı́nicas se desarrollan usando azul brillante Coomassie (Sigma). B. Resultados y discusión 20 25 30 35 1. Expresión de la lacasa Myceliophthora Los transformantes productores de lacasas se detectan mediante la incorporación de ABTS en los medios selectivos. Al usar pyrG o amdS como marcador seleccionable, las frecuencias de cotransformación varı́an de entre aproximadamente un 30 % y un 70 %. La expresión heteróloga de MtL parece ser mucho mayor con los transformantes de A. oryzae. Además, la producción parece ser mejor en un medio ASPO4 comparado con MY50, aunque las razones por las que ocurre son desconocidas. El análisis del sistema de dilución simple por electroforesis en gel de poliacrimida de las muestras del caldo de cultivo muestra una banda de lacasas prominente a aproximadamente 80 kdal, que es similar al tamaño de enzima nativa purificada de M. thermophila. Unos análisis similares de los filtrados del cultivo de los Bo-transformantes de A. Niger indican que la banda de lacasas aparece oscurecida por una glucoamilasa muy intensa y unas bandas proteı́nicas de amilasa estable mediante ácido. Los resultados se muestran en la Tabla 1. TABLA 1 Expresión MtL entre los transformantes seleccionados A. oryzae y A. Niger Cepa Huésped Transformante ADNs Transformantes 40 MTLACU/ML ASPO4 MY50 HowB104 pyrG de A. oryzae no transformado RaMB5.15 RaMB5.30 RaMB5.33 RaMB5.108 RaMB5.111 RaMB5.121 RaMB5.142 ninguno pRaMB5+pPYRG pRaMB5+pPYRG pRaMB5+pPYRG pRaMB5+PSO2 pRaMB5+PSO2 pRaMB5+PSO2 pRaMB5+PSO2 0,00 0,85 0,71 0,60 0,68 0,70 0,49 0,54 0,00 0,29 0,87 0,26 0,19 0,17 0,20 0,04 Bo-1 de A. Niger no transformado ninguno 0,00 0,00 RaMB5.1 RaMB5.25 RaMB5.49 RaMB5.51 RaMB5.53 RaMB5.62 pRaMB5+pToC90 pRaMB5+pToC90 pRaMB5+pToC90 pRaMB5+pToC90 pRaMB5+pToC90 pRaMB5+pToC90 n.d. n.d. n.d. n.d. n.d. n.d. 0,20 0,09 0,06 0,12 0,21 0,16 45 50 55 60 n.d.= no determinado 12 ES 2 165 420 T3 2. Expresión en presencia o en ausencia de cobre en exceso 5 10 15 20 25 30 35 Se añadió asépticamente una parte alı́cuota de 1 ml de una suspensión de esporas con el transformante HowB104-pRaMB5.30 de Aspergillus oryzae (aproximadamente 109 esporas/ml) a un frasco de agitación de 500 ml que contenı́a 100 ml de medio en dicho frasco de agitación estéril (maltosa, 50 g/l; MgSO4 .7H2 O, 2 g/l; KH2 PO4 , 10 g/l; K2 SO4 , 2 g/l; CaCl2 .2H2 O 0,5 g/l; ácido cı́trico, 2 g/l; extracto de levadura, 10 g/l; metales traza [ZnSO4 .7H2 O, 14,3 g/l; CUSO4 .5H2 O, 2,5 g/l; NiCl2 .6H2 O, 0,5 g/l; FeSO4 .7H2 O, 13,8 g/l, MnSO4 .H2 O, 8,5 g/l; ácido cı́trico, 3,0 g/l], 0,5 ml/L; urea, 2 g/l, hecho con agua corriente y ajustado a un pH de 6.0 antes de autocodificarlo) y se incubó a 37◦ C en un agitador giratorio a 200 rpm durante 18 horas. Se transfirieron asépticamente 50 ml de este cultivo a un fermento de 3 litros que contenı́a 1,8 litros de medios de fermentación (MgSO4 .7H2 O, 2 g/l; KH2 PO4 , 2 g/l; ácido cı́trico 4 g/l; K2 SO4 , 3 g/l; CaCl2.2H2 O, 2 g/l; metales traza, 0,5 ml/L; antiespuma plurónica, 1 ml/l). Se mantiene la temperatura del fermento a 34◦ C al hacer circular agua enfriada a través de la cubierta de fermentación. Se introduce aire estéril a chorro a través del fermento a un ritmo de 1,8 litros/min (1v/v/m). Se mantiene el ritmo de agitación entre 600 y 1300 rpm a aproximadamente el nivel mı́nimo requerido para mantener el nivel de oxı́geno disuelto en el cultivo por encima del 20 %. Se añaden nutrientes estériles (Nutriose 725 [jarabe de maltosa], 225 g/l; urea, 30 g/l; extracto de levadura, 15 g/l; antiespuma plurónica, 1,5 ml/L, hecha con agua destilada y sometida a autoclave) al fermento usando un bomba peristáltica. El perfil del ritmo de alimentación durante la fermentación es el siguiente: inicialmente, se añaden 30 g de alimento antes de la inoculación; 0-24 h, 2 g/l h; 24-48 h, 4 g/l h, 48h-final, 6 g/l. Se crea cobre a modo de stock de 400x en agua o a modo de tampón adecuado, se esteriliza el filtro y se añade asépticamente al tanque hasta alcanzar un nivel final de 0,5 mM. La fermentación descrita anteriormente también se lleva a cabo sin añadir el suplemento de cobre al medio del tanque. Se retiran las muestras empleadas para determinar la actividad enzimática y se filtran a través de un Miracloth para eliminar los micelios. Se somete a examen estas muestras para calcular la actividad de la lacasa mediante el ensayo LACU descrito anteriormente. Se ha comprobado que la actividad de la lacasa aumenta continuamente durante el transcurso de la fermentación, con un valor de aproximadamente 45 LACU/ml conseguido tras 180 horas de fermentación que contiene cobre en exceso. En el caso de una actividad especı́fica de 22 LACU/mg, le corresponde 2g/l de lacasa recombinante expresada. Por otra parte, la actividad máxima de lacasas conseguida en la fermentación sin suplemento de cobre es de aproximadamente 10 LACU/ml tras 170 horas o aproximadamente de un 25 % de la hallada en presencia de cobre adicional. III. Purificación y caracterización de la lacasa Myceliopthora A. Materiales y Métodos 1. Materiales 40 45 Los productos quı́micos empleados como tampones y substratos son productos comerciales con calidad de, como mı́nimo, reactivo. La endo/N -glicosidasa F y la amino-peptidasa de piroglutamato se compran de Boehringer Mannheim. La cromatografı́a se lleva a cabo bien en una FPLC Pharmacia o en un sistema de baja presión convencional, Las pruebas espectroscópicas se llevan a cabo bien en un espectrofotómetro (Shimadzu PC160) o un lector de microplacas (Molecular Devices). Los tampones de Britton & Robinson (B&R) se preparan según el protocolo descrito en Quelle, Biochemisches Taschenbuch, H.M. Raven, II. Teil, S. 93 u. 102, 1964. 2. Ensayo enzimático 50 55 60 La actividad de la lacasa se determina con la ayuda de siringaldazina oxidante a 30◦ C en una cubeta de cuarzo de 1 cm. Se mezclan 60 µl de solución controlada con siringaldazina (0,28 mM en etanol al 50 %) y 20 µl de muestra con 0,8 ml de solución tamponada precalentada. La oxidación se controla a 530 nm durante 5 minutos. La actividad se expresa como µmol de substrato oxidado por minuto. Se emplean tampones B&R con pHs diferentes. En la presente se hace referencia a la unidad de actividad con el nombre de “SOU”. También se emplea un tampón de 25 mM de acetato sódico, 40 µM de CuSO4 , pH 5.5, para determinar la actividad, a la que se hace referencia con el nombre de LACU, tal como se ha definido anteriormente. Los ensayos oxidantes con 2,2’-azinobis(3-etilbenzo ácido tiazolina-6-sulfónico) (ABTS) se llevan a cabo empleando 0,4 mM de ABTS, tampón B&R, pH 4.1, a temperatura ambiente al controlar ∆A405. Se lleva a cabo un ensayo de superposición de actividad de oxidasa al verter ABTSagarosa enfriada (0,05 g de ABTS, 1 g de agarosa, 50 ml de H2 O, calentada para disolver la agarosa) sobre un gel de IEF nativo y al incubarlo a temperatura ambiente. El análisis sobre la termoestabilidad 13 ES 2 165 420 T3 de la lacasa (r-MtL) se lleva a cabo usando muestras que presentan una actividad de 3 SOU pre-incubada en un tampón B&R, pH 6, a temperaturas diferentes. Se someten a examen las muestras tras diluirlas 400 veces en el mismo tampón a temperatura ambiente. 5 10 15 20 3. Purificación de un caldo de fermentación Se filtran 3,7 litros de caldo filtrado con tela para fabricar queso (pH 7.6, 16 mS) a través de papel de filtrado Whatman #2. El caldo se concentra en un concentrador en espiral (Amicon) con una membrana S1Y100 (con corte de exclusión de peso molecular:100) de 3700 ml a 200 ml. Se ajusta el concentrado a 0,75 mS al diluirlo en agua y se reconcentra en S1Y100 a 170 ml. El caldo lavado y concentrado presenta un color verdoso denso. Se congela el caldo durante toda la noche a -20◦C, se descongela al dı́a siguiente y se carga en una columna de Q-sefarosa XK26 (120 ml), preequilibrada con 10 mM de Tris, pH 7.5, 0.7 mS (tampón A). La banda de la lacasa azul migra ralentizando la columna durante la carga. Un grupo de fracciones azules pasa a través de la columna una vez se ha cargado y lavado mediante un Tampón A. Un segundo grupo se eluye durante el gradiente lineal con el Tampón B (Tampón A más 2 M de NaCl). Después, se eluye algún material marrón sin actividad de lacasas con 1 M de NaOH. El análisis del sistema de dilución simple por electroforesis en gel de poliacrimida muestra que esta preparación da como resultado una lacasa pura. 4. Análisis de contenido del aminoácido, extensión de la glicosilación y secuencia N-terminal 25 30 35 La secuenciación del N-terminal se lleva a cabo en un secuenciador ABI 476A. El análisis de aminoácidos totales a partir de los que se determina el coeficiente de extinción de r-MtL, se lleva a cabo en un instrumento HP AminoQuant. La desglicosilación se lleva a cabo a través de una endo/Nglucosidasa F según las instrucciones del fabricante y el contenido en carbohidratos se calcula a través de la diferencia de movilidad en el modo en que se determina en el sistema de dilución simple por electroforesis en gel de poliacrimida. El desbloqueo del N-terminal con la amino-peptidasa del piroglutamato se lleva a cabo según las instrucciones del fabricante. Se tratan aproximadamente 80 µg r-MtL con 4 µg de peptidasa con o sin la presencia de 1 M de urea o 0,1 M de guanidina HCl antes de introducirlo en una membrana de PVDF para la secuenciación. Se obtuvieron y secuenciaron cerca de 20 pmol de proteı́na. El sistema de dilución simple por electroforesis en gel de poliacrimida y el análisis con el IEF nativo se llevan a cabo bien en una célula Novex o en un Mini Protean II y en células Mini IEF modelo 111 (Bio-Rad). Los análisis de filtración por gel se llevan a cabo en un Sephacryl S-300 (Pharmacia), a partir del cual se calcula el peso molecular nativo usando un azul dextrano (2000 kdal), IgG bovino (158 kdal), albúmina de suero bovino (66 kdal), ovalbumina (45 kdal) y mioglobina de corazón de caballo (17 kdal) para calibrar la columna. 40 B. Resultados y discusión 1. Purificación y caracterización de r-MtL a partir de un caldo de fermentación 45 50 55 60 Se aislaron cerca de 2-3 g de r-MtL de 3,7 l de caldo de fermentación. La concentración inicial usando una membrana con corte de exclusión de peso molecular de 100 kdal eliminó cantidades significantes de materia marrón y proteı́nas contaminantes pequeñas. La baja afinidad de r-MtL en comparación con la matriz de Q-sefarosa equilibrada con 10 mM de Tris, pH 7.5, facilita su separación de otras impurezas más acı́dicas y más fuertemente enlazadas. Como se muestra con la ayuda del sistema de dilución simple por electroforesis en gel de poliacrimida, esta preparación tuvo como resultado la lacasa esencialmente pura en las fracciones más activas localizadas en torno al valor máximo. Se puede seguir purificando otras fracciones menos activas bien en un Mono-Q con un gradiente más bajo o en una columna de filtración en gel, como S-300, a partir de la que se separan los contaminantes debido a su peso molecular más pequeño. Se consigue una purificación global de 18 veces mayor y una recuperación del 67 %. Como se discutirá a continuación, la existencia de dos bandas de elución de r-MtL en un cromatograma de Q-sefarosa se debe probablemente a una glicosilación diferencial. El r-MtL purificado muestra un peso molecular de 100-1 40 kdal en una filtración en gel S-300 y un peso molecular de 85 kdal en un sistema de dilución simple por electroforesis en gel de poliacrimida. El aumento de la movilidad r-MtL en el sistema de dilución simple por electroforesis en gel de poliacrimida tras la desglicosilación sugiere que los carbohidratos constituyen el 14 % de su masa total. El IEF nativo muestra una banda mayor con un pl a -4,2 que se muestra activo en el ensayo de recubrimiento ABTS. 14 ES 2 165 420 T3 5 10 La secuenciación directa del N-terminal del r-MtL purificado de las muestras bien en solución desalada o en una membrana de PVDF carece de éxito. Sin embargo, el tratamiento de r-MtL con la amino-peptidasa de piroglutamato dio como resultado una proteı́na con el N-terminal desbloqueado. Esto sugiere el procesamiento de un propéptido durante la maduración de r-MtL, un caso postranslacional similar al de la lacasa N. crassa pero que no se encuentra en otras lacasas como la Rizoctonia solani. El esquema propuesto aparece representado abajo. MKSFISAATLWIVGILTPSVAAAPPSTEPQRDLLVPITEREEAAVKARQQSCNTPS | <-péptido con la señal putativa-> | <-propéptidoputativo–> | <-N -terminal El espectro del r-MtL azul presenta una absorción máxima a 276 y 589 nm. 15 20 25 30 La actividad de la lacasa se evalúa usando tanto siringaldazina como ABTS a modo de substratos. Expresada por AbS276 o por mg, la lacasa presenta un valor de 20 ó 45 unidades para un SOU con un pH de 6.5, respectivamente. Los ensayos de LACU dan como resultado un valor de 10 ó 22 unidades por AbS276 o por mg. El perfil del pH de la actividad r-MtL se encuentra bastante cerca del tipo salvaje, con un pH óptimo de 6.5. El lı́mite superior de temperatura para retener la actividad completa tras una preincubación de 20 minutos observada para r-MtL es de aproximadamente 60◦C. El r-MtL purificado no muestra ninguna pérdida de actividad tras un almacenamiento de 5 semanas congelado en un tampón de elución con Qsefarosa a -20◦ C. Cuando se comparan las dos formas de r-MtL obtenidas a partir del caldo de fermentación aislado en Q-sefarosa, no existe ninguna diferencia significante en cuanto al sistema de dilución simple por electroforesis en gel de poliacrimida, PAGE nativo, IEF nativo, filtración en gel S-300, espectro con UV visibles, actividad especı́fica hacia la siringaldazina y ABTS, y en lo que concierne a las mediciones de la secuenciación del N-terminal desbloqueado. Posiblemente, la diferente pauta de elución de la Q-sefarosa surge a partir de algún tipo de glicosilación diferencial. IV. Uso de la lacasa Myceliophthora para teñir cabellos 35 El efecto de la coloración de la lacasa Myceliophthora se calcula a partir de diferentes precursores del tinte y por encima de un 0,1 % de p-fenilenodiamina comparado con varios modificadores. Materiales Precursores del tinte 40 0,1 % de p-fenileno-diamina en 0,1 M de tampón con k-fosfato, pH=7.0) 0,1 % de o-aminofenol en 0, 1 M de tampón con k-fosfato, pH=7.0) 45 Enzimas Lacasa Myceliophthora thermophila recombinante, 16 LACU/ml (en la solución de coloración final). Equipamiento 50 Datacolor Textflash 2000 (CIE-lab) Valoración del color del pelo 55 El color cuantitativo de los mechones de pelo se determina en un Datacolor Textflash 2000 mediante el uso de los parámetros de CIE-lab L∗ (“0” = negro y “100” = blanco) combinados con a∗ (“-” = verde y “+” rojo). 60 15 ES 2 165 420 T3 Resultados Efecto de la coloración 5 Se emplean mechones de cabello rubio europeo (1 gramo) para someter a examen la lacasa Myceliophthora thermophila en el contexto de tintes oxidantes de pelo. Se utilizan la p-fenileno diamina y el o-aminofenol a modo de precursores del tinte. Tinte de pelo 10 Se mezclan 4 ml de solución con el precursor de tinte con 1 ml de lacasa en un mezclador Whirley, se aplica a los mechones de pelo y se tiñe a 30◦C durante 60 minutos. A continuación, se enjuagan dichos mechones con agua corriente durante aproximadamente 3 minutos, se presionan entre dos dedos, se peinan y se deja que se sequen al aire. 15 Los resultados del ensayo sobre el efecto de la coloración se muestran abajo en las Tablas 1 y 2. TABLA 1 20 o-aminofenol 25 ∗ = 0 = negro, 100 = blanco enzima L∗ a∗ Pelo rubio sin tratar - 70,3 2,3 Lacasa + 57,7 15,3 enzima L∗ a∗ Pelo rubio sin tratar - 70,3 2,3 Lacasa + 29,1 4,1 a∗:- = verde, + = rojo TABLA 2 30 p-fenilenodiamina 35 40 ∗ = 0 = negro, 100 = blanco a∗: - = verde, + = rojo Resultado del ensayo En las tablas 1 y 2 se puede observar que la lacasa Myceliophthora thermophila se puede emplear en la coloración oxidante del cabello. 45 Depósito de Materiales Biológicos 50 Se han depositado los siguientes materiales biológicos bajo los términos del Tratado de Budapest en la Colección de Cultivos del Servicio de Patentes sobre Investigaciones Agrı́colas, Northern Regional Research Center, 1815 University Street, Peoria, Ilinois, 61604 el 25 de mayo de 1994 y a los que se le ha dado el siguiente número de acceso. Depósito Número de acceso E. coli JM 1 01 que contiene pRaMB5 NRRL B-21261 55 60 16 ES 2 165 420 T3 Listado de secuencias (1) INFORMACION GENERAL: 5 (i) SOLICITANTE: (A) NOMBRE: Novo Nordisk Biochem, Inc. (B) CALLE: 1445 Drew Avenue 10 (C) CIUDAD: Davis, California (D) PAIS: Estados Unidos de América (E) CODIGO POSTAL (CP): 95616-4880 15 (F) TELEFONO: (916) 757-8100 (G) TELEFAX: (916) 758-0317 20 (i) SOLICITANTE: (A) NOMBRE: Novo Nordisk A/S (B) CALLE: Novo Alle 25 (C) CIUDAD: Bagsvaerd (D) PAIS: Dinamarca 30 (E) CODIGO POSTAL (CP): DK-2880 (F) TELEFONO: +45 4444 8888 (G) TELEFAX: +45 4449 3256 35 (ii) TITULO DE LA INVENCION: LACASAS MYCELIOPHTHORA PURIFICADAS Y ACIDOS NUCLEICOS QUE LAS CODIFICAN (iii) NUMERO DE SECUENCIAS: 2 40 (iv) DIRECCION POSTAL: (A) DESTINATARIO: Novo Nordisk of North America, Inc. (B) CALLE: 405 Lexington Avenue, Suite 6400 45 (C) CIUDAD y ESTADO: Nueva York, Nueva York (D) PAIS: EEUU 50 (E) CP: 10174-6401 (v) FORMATO LEGIBLE POR ORDENADOR: (A) TIPO MEDIO: Disquete 55 (B) ORDENADOR: IBM PC compatible (C) SISTEMA OPERATIVO: PC-DOS/MS-DOS (D) SOFTWARE: PatentIn Release #1.0, Versión #1.25 (EPO) 60 (vi) DATOS SOBRE LA SOLICITUD ACTUAL: 17 ES 2 165 420 T3 (A) NUMERO DE SOLICITUD: no se le ha asignado todavı́a (B) FECHA DE SOLICITUD: 31 de mayo de 1995 5 (C) CLASIFICACION: (vii) DATOS SOBRE LA SOLICITUD ANTERIOR: (A) NUMERO DE SOLICITUD: US 08/253,781 10 (B) FECHA DE SOLICITUD: 03 de junio de 1994 (viii) INFORMACION SOBRE EL AGENTE/REPRESENTANTE: (A) NOMBRE: Lowney, Karen A. 15 (B) NUMERO DE REGISTRO: 31,274 (C) NUMERO DE REFERENCIA/IDENTIFICACION: 4184.204-WO 20 (ix) INFORMACIAN SOBRE LAS TELECOMUNICACIONES: (A) TELEFONO: 212 867 0123 (B) TELEFAX: 212 867 0298 25 (2) INFORMACION SOBRE LA ID DE SEC N◦ : 1: (i) CARACTERISTICAS DE LA SECUENCIA: 30 (A) LONGITUD: 3187 pares base (B) TIPO: ácido nucleico (C) TIPO DE ENLACE: doble 35 (D) TOPOLOGIA: lineal (ii) TIPO DE MOLÉCULA: (ADN) genómico (vi) FUENTE ORIGINAL: 40 (A) ORGANISMO: Myceliophthora thermophila (ix) CARACTERISTICA: 45 (A) NOMBRE/CLAVE: intrón (B) LOCALIZACION: 833... 917 (ix) CARATERISTICA: 50 (A) NOMBRE/CLAVE: intrón (B) LOCALIZACION: 996... 1077 (ix) CARACTERISTICA: 55 (A) NOMBRE/CLAVE: intrón (B) LOCALIZACION: 1090... 1188 60 (ix) CARACTERISTICA: (A) NOMBRE/CLAVE: intrón 18 ES 2 165 420 T3 (B) LOCALIZACION: 1261... 1332 (ix) CARACTERISTICA: 5 (A) NOMBRE/CLAVE: intrón (B) LOCALIZACION: 2305... 2451 10 (ix) CARACTERISTICA: (A) NOMBRE/CLAVE: intrón (B) LOCALIZACION: 2521... 2613 15 (ix) CARACTERISTICA: (A) NOMBRE/CLAVE: CDS 20 (B) LOCALIZACIAN: unión (587..832, 918..995, 1078..1089, 1189..1260, 1333..2304, 2452..2520, 2614..3024) (xi) DESCRIPCION DE LA SECUENCIA: 25 30 35 40 45 50 55 60 19 ES 2 165 420 T3 5 10 15 20 25 30 35 40 45 50 55 60 20 ES 2 165 420 T3 5 10 15 20 25 30 35 40 45 50 55 60 21 ES 2 165 420 T3 5 10 15 20 25 30 35 40 45 50 55 60 22 ES 2 165 420 T3 5 10 (2) INFORMACION SOBRE LA ID DE SEC N◦ : 2: (i) CARACTERISTICAS DE LA SECUENCIA: (A) LONGITUD: 620 aminoácidos 15 (B) TIPO: aminoácido (C) TIPO DE ENLACE: simple (D) TOPOLOGIA: lineal 20 (ii) TIPO DE MOLECULA: proteı́na (vi) FUENTE ORIGINAL: 25 (A) ORGANISMO: Myceliophthora thermophila (xi) DESCRIPCION DE LA SECUENCIA: ID DE SEC N◦ : 2: 30 35 40 45 50 55 60 23 ES 2 165 420 T3 5 10 15 20 25 30 35 40 45 50 55 60 24 ES 2 165 420 T3 5 10 15 20 25 30 35 40 45 50 55 60 25 ES 2 165 420 T3 REIVINDICACIONES 1. Lacasa Myceliophthora substancialmente pura que presenta una secuencia de aminoácidos que es homóloga en al menos un 80 % a la secuencia de aminoácidos establecida en la ID de SEC N◦ 2. 5 10 2. Lacasa Myceliophthora según la reivindicación 1 que presenta una secuencia de aminoácidos que es homóloga en al menos un 85 % a la secuencia de aminoácidos establecida en la ID de SEC N◦ 2. 3. Lacasa Myceliophthora según la reivindicación 1 que presenta una secuencia de aminoácidos que es homóloga en al menos un 90 % a la secuencia de aminoácidos establecida en la ID de SEC N◦ 2. 4. Lacasa Myceliophthora según la reivindicación 1 que presenta una secuencia de aminoácidos que es homóloga en al menos un 95 % a la secuencia de aminoácidos establecida en la ID de SEC N◦ 2. 15 5. Lacasa según la reivindicación 1 que es una lacasa Myceliophthora thermophila. 6. Lacasa Myceliophthora según la reivindicación 1 que presenta una secuencia de aminoácidos establecida en la ID de SEC N◦ 2. 20 7. Lacasa según la reivindicación 1 que presenta una actividad especı́fica de, como mı́nimo, 30 SOU/mg en el caso de la siringaldazina con un pH óptimo. 8. Constructo de ADN que incluye una secuencia de ácido nucleico que codifica una lacasa Myceliophthora según la reivindicación 1. 25 30 9. Constructo de ADN que incluye una secuencia de ácido nucleico que codifica una lacasa Myceliophthora según la reivindicación 2. 10. Constructo de ADN que incluye una secuencia de ácido nucleico que codifica una lacasa Myceliophthora según la reivindicación 3. 11. Constructo de ADN que incluye una secuencia de ácido nucleico que codifica una lacasa Myceliophthora según la reivindicación 4. 35 12. Constructo de ADN que incluye una secuencia de ácido nucleico que codifica una lacasa Myceliophthora según la reivindicación 5. 13. Constructo de ADN que incluye una secuencia de ácido nucleico que codifica una lacasa Myceliophthora según la reivindicación 6. 40 14. Constructo de ADN que incluye una secuencia de ácido nucleico que codifica una lacasa Myceliophthora según la reivindicación 7. 15. Vector recombinante que incluye el constructo de ADN según la reivindicación 8. 45 50 16. Vector según la reivindicación 15 en el que el constructo el enlazado de manera operable con una secuencia del promotor. 17. Vector según la reivindicación 16 en el que el promotor es un promotor micótico o un promotor derivado de una levadura. 18. Vector según la reivindicación 17 en el que el promotor es el promotor de la TAKA-amilasa del Aspergillus oryzae. 55 19. Vector de reivindicación 17 en el que el promotor es el promotor de la glucoamilasa (gluA) de Aspergillus Niger o Aspergillus awamori. 20. Vector según la reivindicación 15 que también comprende un marcador seleccionable. 60 21. Vector según la reivindicación 20 en el que el marcador seleccionable es seleccionado del grupo consistente en amdS, pyrG, argB, niaD, sC y hygB. 22. Vector según la reivindicación 20 en el que el marcador seleccionable es el marcador amdS de 26 ES 2 165 420 T3 Aspergillus nidulans o Aspergillus oryzae o el marcador pyrG de Aspergillus nidulans, Aspergillus Niger, Aspergillus awamori o Aspergillus oryzae. 5 23. Vector según la reivindicación 20 que comprende tanto el promotor de la TAKA-amilasa de Aspergillus oryzae como el marcador amdS o pyrG del Aspergillus nidulans o Aspergillus oryzae. 24. Célula huésped recombinante que incluye un constructo de ADN heterólogo según la reivindicación 8. 10 25. Célula según la reivindicación 24 que es una célula micótica. 26. Célula según la reivindicación 24 que es una célula de Aspergillus. 27. Célula según la reivindicación 24 en la que el constructo es integrado en el genoma huésped celular. 15 28. Célula según la reivindicación 24 en la que el constructo está contenido en un vector. 29. Célula según la reivindicación 24 en la que la secuencia de ácido nucleico codifica una lacasa que presenta la secuencia de aminoácidos representada en la ID de SEC N◦ 2. 20 30. Método para obtener una lacasa según la reivindicación 1, que comprende el cultivo de una célula huésped recombinante que incluye un constructo de ADN que contiene una secuencia de ácido nucleico que codifica una [la] lacasa según la reivindicación 1 bajo condiciones propicias para expresar la lacasa y el recuperar la enzima del cultivo. 25 31. Método para aumentar el rendimiento de la lacasa recombinante activa según la reivindicación 1, que comprende el cultivo de una célula huésped recombinante que incluye un constructo de ADN que contiene una secuencia que codifica una enzima que contiene cobre, bajo condiciones propicias para la expresión de la enzima, en presencia de, como mı́nimo, aproximadamente 0,02 mM de cobre. 30 32. Método para polimerizar un substrato de lignina o lignosulfato en solución que comprende la puesta en contacto del substrato con una lacasa según la reivindicación 1. 35 33. Método para llevar a cabo una depolimerización in situ en pasta de papel kraft que comprende la puesta en contacto de la pasta de papel con una lacasa según la reivindicación 1. 34. Método para oxidar tintes o precursores del tinte que comprende la puesta en contacto de dicho tinte con una lacasa según la reivindicación 1. 40 35. Método para tintar el pelo que comprende la puesta en contacto de una lacasa Myceliophthora según la reivindicación 1, en presencia o en ausencia de por lo menos un modificador, con al menos un precursor del tinte, durante un perı́odo de tiempo y bajo unas condiciones suficientes como para permitir la oxidación del precursor del tinte hasta convertirse en el tinte en sı́. 45 36. Método según la reivindicación 35 en el que el precursor del tinte es seleccionado del grupo consistente en una diamina, un aminofenol y un fenol. 37. Método según la reivindicación 35, en el que el modificador, cuando se usa, es una meta-diamina, un meta-amino-fenol o un polifenol. 50 38. Método según la reivindicación 36 en el que el precursor del tinte es un intermediario primario seleccionado del grupo consistente en una orto- o una para-diamina o en un aminofenol. 39. Método según la reivindicación 35 en el que se emplea más de un modificador. 55 40. Método según la reivindicación 35 en el que se emplea un sólo modificador. 41. Método según la reivindicación 35 en el que se emplean tanto el intermediario primario como el modificador. 60 42. Composición para tintes que incluye una lacasa Myceliophthora según la reivindicación 1 combinada con al menos un precursor del tinte. 27 ES 2 165 420 T3 43. Composición para tintes según la reivindicación 42, que comprende, además, por lo menos un intermediario primario y por lo menos un modificador. 5 44. Recipiente que contiene una composición para tintes según la reivindicación 42 en una atmósfera libre de oxı́geno. 45. Recipiente según la reivindicación 44 que comprende, además, por lo menos un precursor de tinte derivado de un intermediario primario combinado con, como mı́nimo, un modificador. 10 46. Método para polimerizar u oxidar un compuesto fenólico o anilı́nico que comprende que el compuesto fenólico o anilı́nico entre en contacto con una lacasa Myceliophthora según la reivindicación 1. 15 47. Constructo de ADN que incluye la secuencia de ácido nucleico que codifica una lacasa Myceliophthora contenida en NRRL B-21261. 20 25 30 35 40 45 50 55 60 NOTA INFORMATIVA: Conforme a la reserva del art. 167.2 del Convenio de Patentes Europeas (CPE) y a la Disposición Transitoria del RD 2424/1986, de 10 de octubre, relativo a la aplicación del Convenio de Patente Europea, las patentes europeas que designen a España y solicitadas antes del 7-10-1992, no producirán ningún efecto en España en la medida en que confieran protección a productos quı́micos y farmacéuticos como tales. Esta información no prejuzga que la patente esté o no incluı́da en la mencionada reserva. 28 ES 2 165 420 T3 29 ES 2 165 420 T3 30 ES 2 165 420 T3 31 ES 2 165 420 T3 32 ES 2 165 420 T3 33