Descargar - Ingeniería Genética A
Anuncio
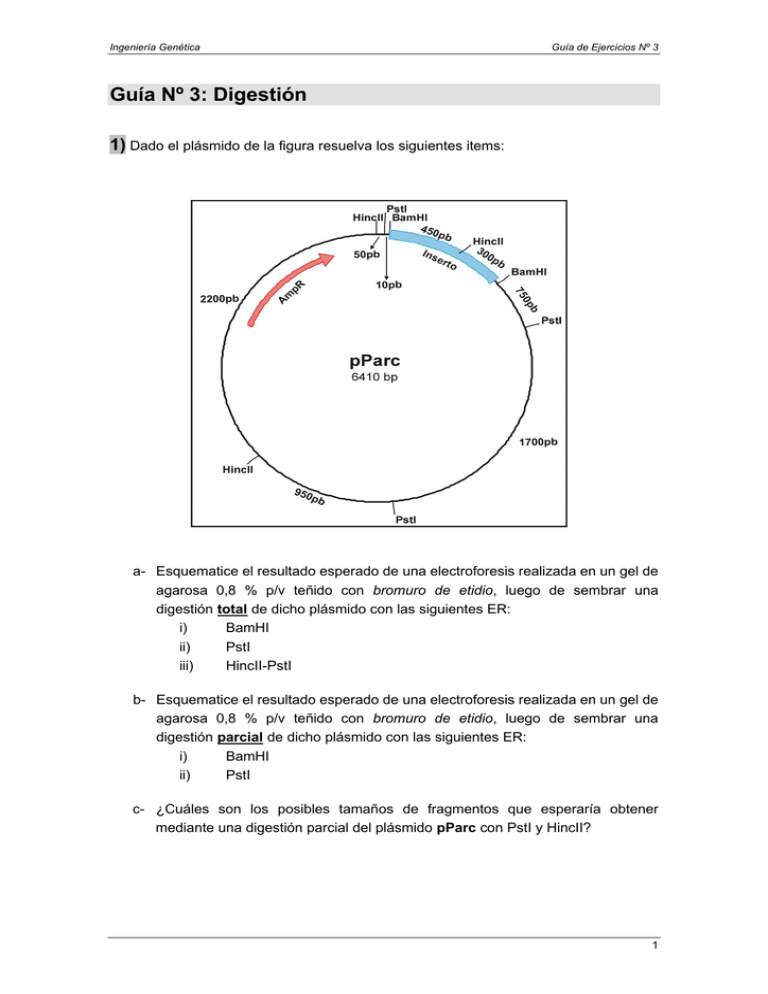
Ingeniería Genética Guía de Ejercicios Nº 3 Guía Nº 3: Digestión 1) Dado el plásmido de la figura resuelva los siguientes items: a- Esquematice el resultado esperado de una electroforesis realizada en un gel de agarosa 0,8 % p/v teñido con bromuro de etidio, luego de sembrar una digestión total de dicho plásmido con las siguientes ER: i) BamHI ii) PstI iii) HincII-PstI b- Esquematice el resultado esperado de una electroforesis realizada en un gel de agarosa 0,8 % p/v teñido con bromuro de etidio, luego de sembrar una digestión parcial de dicho plásmido con las siguientes ER: i) BamHI ii) PstI c- ¿Cuáles son los posibles tamaños de fragmentos que esperaría obtener mediante una digestión parcial del plásmido pParc con PstI y HincII? 1 Ingeniería Genética Guía de Ejercicios Nº 3 d- Usted digiere el plásmido pParc con EcoRI para comprobar si tiene sitio de reconocimiento para dicha enzima. Como resultado, usted encuentra que luego de una digestión total con EcoRI se generan dos fragmentos: uno de 3150 pb y otro de 3260 pb. Para determinar la ubicación física de dichos sitios en pParc usted realiza digestiones dobles totales teniendo los siguientes resultados: EcoRI-HincII: 1950 pb, 1750 pb, 1200 pb, 1000 pb, 510 pb. EcoRI-PstI: 2150 pb, 1510 pb, 1050 pb, 1000 pb, 700 pb. EcoRI-BamHI: 3150 pb, 1450 pb, 1060 pb, 750 pb. Ubique en pParc los sitios de reconocimiento para EcoRI. e- Obtenga el plásmido pParcII, mediante la elimininación del inserto contenido en pParc. Detalle todos los pasos que debería hacer para lograrlo. 2) Usted posee un fragmento que fue liberado de un vector mediante una digestión con la enzima EcoRI. Para realizar un mapa físico de dicho fragmento usted realiza simple y doble digestiones con las enzimas PstI, BamHI y XhoI y las resuelve en un gel de agarosa 0.8 % p/v. También siembra en el mismo gel un ladder de peso molecular. Luego de teñirlo con bromuro de etidio y revelarlo con luz UV usted encuentra los siguientes resultados al analizar las distancias migradas de cada banda: pb 100 200 300 400 500 600 700 800 900 1000 1400 Ladder distancia (cm) 3,96 3,25 2,7 2,45 2,1 1,8 1,5 1,3 1,1 0,8 0,2 ER PstI BamHI XhoI PstI-XhoI XhoI-BamHI PstI-BamHI Distancia migrada (cm) 2,93 2,25 1,55 3,67 0,72 4,26 1,93 1,44 4,26 3,67 2,25 1,79 5,26 4,26 1,93 1,55 3,67 2,93 2,25 1,93 En base a los resultados obtenidos: i) Grafique el resultado de la electroforesis resuelta (incluya el ladder, los controles y las digestiones). ii) Construya el mapa físico del fragmento. 2 Ingeniería Genética 3) Guía de Ejercicios Nº 3 El DNA del plásmido pHUB1 es circular, de cadena doble y tiene 5,7 kpb. Este plásmido lleva un gen cuyo producto proteico confiere resistencia a la bacteria huésped al antibiótico tetraciclina (tetR). Además, este cccdsDNA tiene un sitio único de corte para las enzimas de restricción, EcoRI, HpaI, BamHI, PstI, SalI y BglII. Por otro lado, el clonado en los sitios BamHI y SalI anula el fenotipo resistencia a tetraciclina, mientras que esto no sucede cuando se utilizan estrategias basadas en la apertura con las otras endonucleasas mencionadas. Con el objetivo de confeccionar el mapa físico del plásmido, se realizaron digestiones con distintas combinaciones de enzimas de restricción. Los productos de dicha reacción fueron fragmentos cuyos tamaños se muestran en la tabla. Con toda esta información, dibuje el mapa correspondiente al plásmido pHUB1. Enzimas de Restricción EcoRI EcoRI, BamHI EcoRI, HpaI EcoRI, SalI EcoRI, BglII EcoRI, PstI PstI, BglII BglII, HpaI Tamaño del fragmento (kpb) 5,7 0,4-5,3 0,5-5,2 0,7-5,0 1,1-4,6 2,4-3,3 1,3-4,4 0,6-5,1 3









