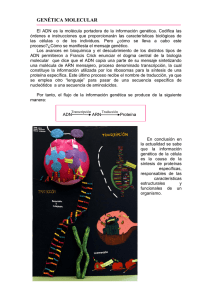
IDENTIFICACIÓN DE MARCADORES GENÉTICOS
Anuncio

UNIVERSIDAD NACIONAL DE HUANCAVELICA (Creada por Ley Nro. 25265) PROYECTO DE INVESTIGACIÓN IDENTIFICACIÓN DE MARCADORES GENETICOS MOLECULARES SNPs PARA CARACTERISTICAS DE IMPORTANCIA ECONOMICA EN LA ALPACA HUACAYA LINEA DE INVESTIGACIÓN Preservación de la biodiversidad y el ecosistema de la zona de influencia del proyecto Camisea INVESTIGADORES: RESPONSABLE : M.Sc. Ing. WILLIAM SALAS CONTRERAS - UNH ASESOR : Ph D. Blgo. JOSE ESPINOZA BABILÓN - UPCH FECHA DE REGISTRO: INICIO : ............................................................. CULMINACIÓN : ............................................................. HUANCAVELICA, MAYO DEL 2014 ÍNDICE Pág. 1. PROBLEMA 1.1 Planteamiento del problema 1.2 Formulación del problema 1.3 Objetivos 1.3.1 Objetivo general 1.3.2 Objetivos específicos 1.6 Justificación CAPITULO I 3 3 4 4 4 4 5 CAPITULO II 2. MARCO TEÓRICO 2.1 Antecedentes 2.2 Bases teóricas 2.3 Hipótesis 2.4 Definición de términos 2.5 Identificación de variables 2.6 Definición operativa de variables e indicadores 7 7 8 10 11 15 15 CAPITULO III 3. METODOLOGÍA DE LA INVESTIGACIÓN 3.1 Tipo de investigación 3.2 Diseño de investigación 3.3 Método de investigación 3.4 Población, muestra, muestreo 3.5 Fundamento y técnica a utilizar 3.6 Procedimiento para el análisis 3.7 Toma de muestras 3.8 Análisis de laboratorio 3.9 Ámbito de estudio 16 16 16 16 16 16 18 20 21 27 CAPITULO IV 4. ASPECTO ADMINISTRATIVO 4.1 Recursos humanos 4.2 Recursos materiales 4.3 Presupuesto (cadena de gasto mensualizado) 4.4 Financiamiento 4.5 Cronogramas de actividades 28 28 29 31 37 37 BIBLIOGRAFIA ANEXO 38 41 CAPITULO I 1. PROBLEMA 1.1 PLANTEAMIENTO DEL PROBLEMA El Perú cuenta con la mayor población de alpacas (Vicugna pacos) en el mundo llegando alrededor de 3 millones de ejemplares. En la alpaca, el principal producto económico constituye la fibra, sin embargo, a lo largo del tiempo esta fibra ha ido disminuyendo su calidad con lo cual el precio en el mercado ha disminuido considerablemente. El gobierno peruano con el fin de contrarrestar esta situación ha propiciado la creación de programas de mejora genética encaminados a elevar la productividad y calidad de la alpaca, programas que a la fecha no han obtenido resultados satisfactorios. Un adecuado programa de mejoramiento genético en alpacas requiere de dos herramientas básicas para su desarrollo y aplicación: i) Selección y ii) Sistemas de reproducción. Una selección adecuada de alpacas reproductoras depende de la información sobre animales para la toma de decisiones, donde los registros productivos y de filiación son fuentes de información indispensables. Lamentablemente en el Perú, los registros de los criadores de alpacas son deficientes o inexistentes, debido a un inadecuado manejo y baja confiabilidad en la identificación de parentesco. Estudios previos han reportado al menos un 25% de errores de filiación (Rodríguez y col., 2006) en registros en centros reproductores de alpacas. Los errores de filiación en alpacas producen una merma considerable en la ganancia genética esperada para esta especie; merma que hasta la fecha no ha sido cuantificada, provocando el fracaso o retraso de cualquier programa de mejoramiento genético dirigido a elevar la calidad de la fibra. Además, la ausencia de un sistema de validación de pedigrí se convierte en una limitante para la apertura del mercado nacional e internacional de venta de crías certificadas de alto valor genético. El impacto generado por errores de filiación en registros productivos en ganado doméstico, sugieren que tasas de solo el 10% producen una disminución del orden del 11 al 15% de la ganancia genética total (Banos y col., 2001), siendo además un factor importante a considerar en la estimación del mérito genético de un animal reproductor. En la alpaca, esta situación se agrava debido a la carencia de productos y subproductos alternativos a la fibra con capacidad de aprovechamiento y demanda en el mercado nacional e internacional. Adicionalmente, la ausencia de métodos adecuados y acelerados de selección genética disminuye el valor del ganado lo cual se manifiesta en una baja calidad de fibra. El intervalo generacional en la alpaca alrededor de 4 años también constituye un problema para métodos adecuados de selección, siendo la selección genética asistida por marcadores una alternativa tecnológica a ser explotada. Los marcadores genéticos SNPs en genes codominantes relacionados con características de importancia económica como diámetro de fibra principal característica económica en los sistemas de producción de alpacas. Las consecuencias de una ausencia de alternativas tecnológicas en base a genética molecular y su incipiente aplicación en sistemas de producción de alpacas vienen generando que los pobladores alto andinos que subsisten con los productos generados por la alpaca no dispongan de suficientes recursos económicos para solventar su alicaída economía familiar. Se estima que alrededor de 200,000 familias equivalentes a un millón de habitantes distribuidos en 14 de los 24 departamentos del Perú subsisten gracias a este recurso pecuario. De otro lado, cabe mencionar que en la actualidad se están realizando trabajos de investigación científica aplicando las ventajas de la genética molecular y la disponibilidad de una batería de microsatélites y SNPs por si sola ya permite emprender inmediatamente una serie de estudios importantes para los programas de mejoramiento genético. Así, tenemos que las aplicaciones a corto plazo incluyen, básicamente, la identificación y discriminación de individuos, pruebas de filiación, estudios de consanguinidad y variabilidad, control y verificación de empadres, aplicados en forma efectiva los programas de mejora genética podrían ayudar a elevar la calidad genética. 1.2 FORMULACIÓN DEL PROBLEMA La genética de los rasgos económicamente importantes de la alpaca se desconoce debido a una insuficiente caracterización de marcadores genéticos moleculares en la alpaca. Los marcadores microsatélites y los polimorfismos de nucleótidos únicos (SNPs) son marcadores de elección para el mapeo de los rasgos productivos y son una herramienta útil para solucionar el problema. El insuficiente número de marcadores microsatélites y SNPs identificados y validados son un problema en la alpaca, los cuales constituyen una causa de los pobres resultados obtenidos en los programas de mejora genética. Por lo expuesto, el problema que busca abordar el presente proyecto constituye: La insuficiente identificación y desarrollo de marcadores SNPs y microsatélites como herramientas de mejora genética en alpacas Huacaya. El contar con un panel de marcadores moleculares de la alpaca también reforzara los estudios de diversidad genética del recurso y ayudaran a los programas de mejoramiento genético de la alpaca. ¿Existen marcadores moleculares SNPs para características de importancia económica en la alpaca Huacaya? 1.3 1.4 OBJETIVOS 1.3.1 OBJETIVO GENERAL Generar un panel de marcadores genéticos moleculares de la alpaca Huacaya 1.3.2 OBJETIVOS ESPECIFICOS Desarrollar libros de registros y pedigrí validados mediante ADN Identificar marcadores genéticos de ADN tipo SNPs para el mejoramiento genético y evaluación de la diversidad genética JUSTIFICACIÓN Siendo el Perú un país poseedor de una población de más 3 millones de alpacas distribuidas principalmente en zonas alto andinas sobre los 3500 metros de altitud con sistemas de producción constituidos principalmente por pequeños y medianos productores (cerca del 90%) la venta de fibra de alpaca constituye un importante recurso económico para la manutención de sus familias. (FAO, 2005) Además el Perú es considerado como el principal productor de fibra de alpaca en el mundo, con un 85% de la producción nacional orientada al mercado internacional .La venta de fibra de alpaca al mercado internacional representa en promedio el 1.35% de las exportaciones .Por otro lado cabe mencionar que actualmente, esta cifra ha disminuido debido a la pérdida de la calidad de la fibra, lo cual ha ocasionado la disminución de su valor comercial (FAO, 2005). El conocimiento del estatus genético del animal es un elemento clave para una correcta toma de decisiones en los programas de conservación y uso sostenible de cualquier recurso genético, así como para el desarrollo de pruebas de identidad y parentesco. La capacidad de las especies domésticas para mejorar su productividad y adaptarse a diferentes condiciones medioambientales, requiere de tener acceso a todo su potencial adaptativo, lo cual está estrechamente relacionado con la diversidad genética existente (Tapia y col., 2006). Durante los últimos años la conservación de razas y/o poblaciones ha tomado un gran auge debido principalmente a la concientización del hombre en la necesidad de preservar dichos recursos genéticos. La importancia de la biodiversidad y la conservación quedó patente a partir de la cumbre de las Naciones Unidas sobre el medio ambiente y el desarrollo celebrada en Río de Janeiro en 1992 estableciéndose entonces la necesidad de estudiar los diferentes componentes de la diversidad genética. El recurso genético animal constituye un patrimonio de inestimable valor, la pérdida de diversidad genética merma nuestra capacidad de mantener y mejorar la producción y productividad pecuaria y reduce la aptitud para hacer frente a nuevas condiciones ambientales (FAO, 2005). Es menester anotar que la importancia de la conservación de razas se resume en cuatro aspectos: De orden genético-productivo: la diversidad es necesaria para mantener la variabilidad de las poblaciones la cual permite la adaptación a diferentes ambientes algunas veces adversos. De orden científico: el estudio de cada raza en particular es de interés para detectar posibles genes únicos y valiosos en el momento actual o en el futuro de estas poblaciones. De orden histórico cultural: la conservación de determinadas razas y especies representan un patrimonio genético de un país y como historia viva y paralela al desarrollo de la población humana. De orden ecológico-ambiental: los ecosistemas son el resultado del equilibrio entre clima, flora y fauna y cualquier factor que afectará a alguno de estos componentes estaría atentando contra ese equilibrio deteriorando el medio y la simbiosis ecológica de la zona (Simons 1984). -Antiguamente los registros genealógicos se hacían por examen aparente o simplemente no existían. -En la actualidad muchos de estos estudios sobre conservación de razas se basan en los análisis genéticos a través del uso de marcadores moleculares de ADN (uno de estos marcadores son los microsatélites o short sequence repeat tandem o SSRT). -Para Huancavelica y otros criadores de alpacas la mejora genética asistida con marcadores moleculares permitirá mejorar la fibra la que redunda en una mayor economía del poblador andino. Además también repercute en la industria con mayores volúmenes y homogeneidad en calidad, y la industria textil contará con mejor materia prima para la producción de telas. -Afortunadamente la aparición de los marcadores moleculares está ayudando a eliminar tanto los inconvenientes de una selección basada en el análisis exclusivo del fenotipo, como la identificación de especies y variedades de una forma más rigurosa y repetitiva. Finalmente, es importante identificar marcadores moleculares porque con ellos se podrá realizar mejoramiento genético logrando mejores animales lo cual a su vez permitirá mejorar los precios en beneficio de los criadores y los usuarios del producto final CAPITULO II 2. MARCO TEÓRICO 2.1 ANTECEDENTES Tradicionalmente se consideraba al guanaco, el ancestro de estas dos especies, mientras que se pensaba que la vicuña nunca había sido domesticada. Recientes investigaciones, vinculan a la alpaca con la vicuña, y datan su domesticación una antigüedad de 6000 a 7000 años, en los Andes Peruanos. En nuestro territorio, en la Puna Norte y Puna Sur hay evidencias arqueológicas del probable comienzo de la domesticación de los camélidos, entre 3500 a 5000 años atrás iniciado por cazadores. Los análisis genéticos, como el ADN mitocondrial, confirmaron la similitud genética entre la llama y el guanaco y entre la vicuña y la alpaca, revelando hibridación bidireccional. Por análisis de microsatélite ADN se sugiere que la alpaca desciende de la vicuña y que debiera ser reclasificada como Vicugna pacos (Wheeler, 1995) Tapia et al (2006), estudió comparativamente el material genético de los camélidos sudamericanos, un aspecto biológico que no había sido explorado; analizó, entre otros temas, la organización de secuencias repetidas de ADN, que suelen encontrarse en proporción alta en los mamíferos, y cuyo papel en la estructura, evolución y regulación de los genes no es aún conocido. Estas secuencias pueden aparecer más de un millón de veces y, desde un punto de vista evolutivo, podrían establecer nuevas relaciones entre los genes estructurales y regulatorios, ya que las secuencias repetidas podrían variar algunas de sus características a lo largo del tiempo sin perder su identidad. Ello convierte a este tipo de ADN en valiosa herramienta para inferir relaciones evolutivas entre grupos de organismos. Hasta la fecha se han identificado y caracterizado 23 marcadores de microsatélites polimórficos aislados de una genoteca genómica de camélidos sudamericanos, utilizando una sonda de poli (TG) 24. Estos SSRT presentan una gran diversidad en cuanto al número de alelos en los distintos taxa de los camélidos sudamericanos, así como también en los índices de heterocigocidad para cada locus. Finalmente ya se tiene algunos marcadores apropiados para determinar relaciones de filiación. Esto permitirá validar de una forma objetiva los registros genealógicos de camélidos sudamericanos actualmente existentes en el país, tema en el que una vez más han tomado la iniciativa países del hemisferio norte donde estas especies son exóticas (wheeler, 1965). Los rasgos productivos en alpacas incluyen características de la fibra, resistencia a las infecciones, así como rasgos reproductivos y metabólicos. Algunos loci asociados a la variación de la lana han sido identificados por estudios de mapeo genético en ovejas. Estudios en oveja Merino han demostrado la existencia de variantes genéticas para genes de queratina (KRT33) y proteínas asociadas a queratina (Tricohialina y KAPs). En alpacas, a la fecha, se cuenta con 6,611 ESTs no anotados que constituyen un recurso a ser explotado para la generación de marcadores genéticos mediante el tamizaje dirigido a la identificación de microsatélites y SNPs, los cuales podrían ser evaluados en poblaciones fenotípicamente diferenciadas en rasgos productivos como diámetro de fibra y densidad del vellón. Esta etapa está en sus inicios ya que se necesitan decenas de marcadores para realizar los trabajos de identificación de genes relevantes. Todos los marcadores existentes en camélidos fueron obtenidos aleatoriamente por lo que poco contribuyen en el mejoramiento de características deseadas. En Uruguay, trabajando con 494 ovinos Texel se analizaron los genes CAPN1, GH, CAST y FABP9 los cuales sirvieron para implementar la selección asistida con marcadores moleculares para el incremento del progreso genético en esta especie (Eileen, 2011). De otro lado utilizando SNPs en Perú se trató de identificar polimorfismos del gen tlr4 en crías de alpacas con neumonía provocada por Pasteurella multocida no encontrándose polimorfismos tipo SNP en esta especie (Guzman, 2011). 2.2 BASES TEÓRICAS MICROSATÉLITES Los microsatélites, también llamados “secuencias simples repetidas” (SSRs), “Secuencias cortas repetidas en tandem” (STRs), son secuencias cortas de ADN que consisten en repeticiones de 1 a 6 pares de bases de largo (Figura 1) (Hancock, 1991; (Freeland, 2005). Por otro lado los microsatélites presentan elevado polimorfismo genético (presencia de múltiples alelos Estas características han permitido ser considerados como los marcadores de elección para estudios de genética poblacional, identificación, parentesco y mapeo genético (Bruford y Wayne, 1996; Schlötterer y Harr, 2001; Freeland, 2005). Figura 1. Diagrama mostrando la ubicación de tres loci microsatélites (TA)n, (TAA)n y (GC)n en un segmento cromosomal. Se aprecia el grado de homocigosidad para el locus 1 (TA)4, y heterocigosidad para el locus 2 (TAA)7, (TAA)8 y el locus 3 (GC)7, (GC)8. Modificado por Freeland (2005) DIVERSIDAD GENÉTICA La diversidad genética se define como la cantidad de variación genética medible que está contenida dentro de una población o especie (Freeland, 2005). La diversidad genética es una de las características más importantes en cualquier población, siendo esencial para su adaptación a cambios en el medio ambiente, sobrevivencia a largo plazo y evolución (Nei y Kumar, 2000). Niveles bajos de diversidad genética aumentan la probabilidad de incrementar los niveles de consanguinidad provocando efectos sobre la viabilidad de los individuos y de las poblaciones. La estimación de la diversidad genética de una población constituye un punto central en genética poblacional y tiene una implicancia extremadamente importante en biología de la conservación (Freeland, 2005). La rama de la genética que estudia la generación y el mantenimiento de la diversidad genética y/o polimorfismo genético, así como los mecanismos de la evolución a nivel poblacional se denomina “Genética Poblacional” (Nei y Kumar, 2000). PARENTESCO E IDENTIDAD La determinación de parentesco (paternidad y maternidad) se basa en la búsqueda de incompatibilidades genéticas entre los alelos presentes en un hijo y un alegado pariente (Figura 2) para un grupo de loci analizados asumiendo la ausencia de mutación y un modelo de herencia mendeliana simple (Butler, 2005). Las técnicas para determinación de parentesco se basan en la exclusión genética, la cual utiliza incompatibilidades genéticas entre los supuestos padres y las crías, para rechazar una particular hipótesis de asignación de parentesco. El número mínimo de inconsistencias genéticas entre un supuesto padre y una cría para determinar la exclusión genética es tres (Cifuentes y col., 2006). Existen 5 indicadores para medir la robustez de una técnica de determinación de parentesco: la probabilidad de exclusión, el contenido de información polimórfica, el índice de paternidad, el valor de LOD y el estadístico Delta. Individuo al azar Alegado padre E,F Madre (pariente conocido) C,D A,B C,B Cría (a) C,D Crías A,B D,B C,B D,A D,B C,A D C D D C (b) Alelo paterno Figura 2. Representación esquemática de una prueba de parentesco y su fundamento. (a) Asignación de paternidad. (b) La determinación de parentesco se basa en una regla de herencia mendeliana: una cría tiene dos alelos para cada marcador autosomal, uno heredado del padre y otro de la madre. Modificado por Butler (2005). POLIMORFISMO GENÉTICO Polimorfismo genético es un parámetro de la variabilidad genética poblacional que se define como la existencia de dos o más alelos asociados a un locus en una población con una frecuencia relativa sustancial (usualmente mayor del 1%). La generación de polimorfismo genético en un locus se debe principalmente a mutaciones puntuales, inserciones, deleciones, conversión génica y recombinación intraalélica (Nei, 1973), los cuales se han estabilizado en una población y región geográfica determinada. POLIMORFISMO DE NUCLEOTIDOS UNICO (SNPs) Los SNPs (single nucleotid polimorfism) son considerados solamente como un cambio de una simple base en una secuencia de ADN. Los SNPs presentan diferentes alternativas de secuencia (alelos) en individuos normales en una o varias poblaciones dadas (figura 1), sin embargo para ser considerado un SNP este deben presentar frecuencias alélicas mayores del 1%. Las variantes debido a inserciones /deleciones de bases (indels) nopueden ser consideradas como SNPs, asi mismo, como las variantes de nucleotidos únicos en cDNAs se denomina cSNPs y son clasificadas como SNPs bajo el criterio que también son variantes de ADN genómico Los SNPs se consideran marcadores principalmente, sin embargo, en humanos se ha demostrado que la existencia de SNPs tri y tetra alelicos pero con una frecuencia muy rara al punto de casi no existir. Los SNPs se heredan como marcadores bialelicos codominantes en forma mendeliana simple. El grado de polimorfismo medido como contenido de información polimórfica (PIC) no es alto como en los microsatélites multialelicos, pero su gran abundancia en el genoma balancea esta deficiencia. Los SNPs son el polimorfismo más abundante en cualquier tipo de organismo, constituyen el último nivel de variación genética entre individuos. Del total de variación genética en humanos, un 90% corresponde a SNPs. En humanos se han reportado en bases de datos 5 millones de SNPs de los cuales 4 millones han sido validados (Sobrino y col., 2005). Los SNPs se pueden encontrar tanto en regiones codantes y no codantes del genoma, sin embargo existe una mayor densidad de SNPs en regiones no codantes. 2.3 HIPÓTESIS El origen común de los mamíferos se refleja en la secuencia de sus genes. Los camélidos como cualquier mamífero tiene un genoma de alrededor de 3 mil millones de bases y cerca de 25,000 genes. Las secuencias traducidas a proteínas (exones) constituyen menos del 1.5 % del genoma, el resto son secuencias que interrumpen a los genes (intrones) y secuencias entre genes. La secuencia del genoma humano y otras especies han revelado que los individuos no son idénticos, sino que hay variaciones prácticamente en todos los genes. Los microsatélites son unas secuencias que se encuentran cada 10,000-40,000 pares de bases y que son muy variables (polimórficas, y ofrecen hasta 90 % de informatividad). Los marcadores microsatélites son ubicuos, y (cada 40,000 bases), habrá mucha probabilidad de encontrarlos dentro de cualquier gen. Los microsatélites son altamente polimórficos, y van a ser útiles para seguir su segregación (y del gen asociado) a través de las generaciones, permitiendo ser utilizados en pruebas de identidad, parentesco, consanguinidad, variabilidad genética y selección asistida. En consecuencia la hipótesis que planteamos es la siguiente: En la alpaca Huacaya las características de importancia económica están determinados por marcadores moleculares SNPs 2.4 DEFINICIÓN DE TÉRMINOS MARCADORES MOLECULARES.- Los marcadores moleculares son biomoléculas que se pueden relacionar con un rasgo genético. Las biomoléculas que pueden ser marcadores moleculares son las proteínas (antígenos e isoenzimas) y el ADN (genes conocidos o fragmentos de secuencia y función desconocida) esta cadena de ADN son fragmentos específicos que pueden ser identificados en todo el genoma. Los marcadores moleculares están situados en lugares específicos del genoma y se usan para “marcar” la posición de un gen en concreto o la herencia de una característica particular. En un cruzamiento genético, las características de interés seguirán generalmente unidas a los marcadores moleculares. Por lo tanto, se pueden seleccionar individuos en los que el marcador molecular esté presente, ya que el marcador indica la presencia de la característica deseada. Son todas aquellas moléculas (proteínas o ADN) que se pueden identificar y caracterizar para definir un genotipo determinado. Muchos de ellos permiten la asociación de genotipos con fenotipos específicos. ALPACA.- Según algunos investigadores, la alpaca proviene de la domesticación de la vicuña, y habita en la zona alta andina por encima de los 3,800 msnm en Perú, Bolivia, Chile, Argentina y, en menor medida, en los Estados Unidos, Canadá, Nueva Zelanda y Australia. La alpaca es un animal de fina estampa, armoniosa en su caminar, de cuerpo esbelto cubierto de fibra que en su conjunto se denomina vellón. Presenta almohadillas plantares, característica que le otorga la condición de animal ecológico al no dañar el pasto, ni provocar erosión. La alpaca como especie doméstica es criada en rebaños; su producción principal es la fibra que presenta un número variado de colores, pasando del blanco al café, hasta el negro; también el color roano y el gris. De otro lado, la carne fresca de alpaca es materia prima de alta calidad para la elaboración de gran variedad de embutidos y conservas, además, se pueden preparar gran variedad de platos. Dicha carne es reconocida como uno de los alimentos más nutritivos, pues posee 22% de proteínas, 56 miligramos de colesterol por cada 100 gramos de carne y un contenido graso de 3% por lo que es considerado un producto light. HUELLA GENETICA.- Fragmentos característicos del material genético que diferencian a un individuo de otro. Técnica por la que se analiza el ADN de un individuo para observar el patrón de repetición de secuencias. Este patrón es único para cada individuo. MEJORAMIENTO GENETICO.- Consiste en aplicar principios biológicos económicos y matemáticos, con el fin de encontrar estrategias óptimas para aprovechar la variación genética existente en una especie animal en particular para maximizar su mérito. Esto involucra tanto la variación genética como entre los individuos de una raza como la variación entre razas y cruces. GENÉTICA MOLECULAR.- La genética molecular es el campo de la biología que estudia la estructura y la función de los genes a nivel molecular. La genética molecular emplea los métodos de la genética mendeliana, cuantitativa, poblacional y la biología molecular. SECUENCIACION DE ADN.- La secuenciación de ADN es el proceso por el cual se establece el orden preciso de bases a lo largo de una cadena de ADN. Esto se hace comúnmente al someter los fragmentos de ADN a electroforesis en gel, lo cual lleva a la separación del ADN en fragmentos que difieren en tamaño sólo por una base. Cada fragmento de ADN se detecta debido a la presencia de un marcador radioactivo o fluorescente. Los instrumentos más usados para establecer las secuencias del ADN emplean un rayo láser para detectar el ADN marcado con fluorescencia. A partir del patrón de los fragmentos de ADN resultantes se puede deducir la secuencia subyacente del ADN. GENOMA.- Conjunto completo de los genes de un organismo. El genoma humano, por ejemplo, contiene entre 30000 y 40000 genes. MAPEAR GENES.- Conocido también como mapa de ligamiento, es un mapa de marcadores polimórficos que define las distancias por los eventos de recombinación ocurridos en la región cromosómica que los contiene. Se refiere a los cromosomas de una especie, que muestra la posición de sus genes conocidos o de los marcadores correspondientes. FINGERPRINTING.- Conocido también como “fingerprinting del ADN” o “trazado del perfil del ADN”, el fingerprinting en las plantas se basa en el supuesto de que cada variedad o población individual tiene un perfil genético, revelado mediante su ADN, que es exclusivo de esa variedad o población. Los investigadores obtienen muestras de ADN a partir del tejido vegetal y usan varias técnicas para producir un “fingerprint” (una huella característica y singular), que se ve como una serie de bandas de tamaño variable, muy similar a un código de barras. Las bandas correspondientes a una variedad pueden ser comparadas con las de otras variedades para detectar similitudes y diferencias. Cuanto más similitudes hay, más emparentadas están las dos variedades y se pueden determinar los progenitores de la variedad en cuestión o de las variedades hermanas que comparten los mismos progenitores. ADN.-Moléculas de las células que contienen la información genética y que son responsables de la transferencia de ésta, de generación en generación. Cada molécula de ADN está constituida por dos cadenas formadas por un elevado número de compuestos químicos llamados nucleótidos. Estas cadenas forman una especie de escalera retorcida de doble hélice. Cada nucleótido está formado por tres unidades: una molécula de azúcar llamada desoxirribosa, un grupo fosfato y uno de cuatro posibles compuestos nitrogenados llamados bases: adenina, guanina, timina o citosina. El ADN incorpora las instrucciones de producción de proteínas. GEN.- La unidad funcional y física de herencia que pasa de padres a sus descendientes. Los genes son piezas de ADN, y la mayoría de los genes contienen la información necesaria para la formación de una proteína específica. MICROSATELITES o SSR (Repetición de secuencias discretas).- En los genomas existe un ADN ubicuo y abundante denominado "microsatélite" que consiste en mono, di, tri y tetranucleótidos repetidos en tándem. Este ADN, que es muy polimórfico, se ha utilizado como marcador molecular cuando la secuencia del motivo repetido se clona y secuencia para usarla en el análisis de poblaciones. De esta manera se han estudiado con éxito numerosos árboles, aunque en algunos se ha visto que los microsatélites son menos variables de lo que se esperaba. Otras siglas para designar esta técnica son: ISSR, IMA, ISA, IRA, y RAMP. HIBRIDACION.- En biología molecular, hibridación es el proceso de unir dos hebras complementarias de ADN. TAXA.- En biología, un taxón (del griego ordenamiento) es un grupo de organismos emparentados, que en una clasificación dada han sido agrupados, asignándole al grupo un nombre en latín, una descripción, y un tipo, que si el taxón fuera una especie este sería un espécimen o ejemplar concreto. Cada descripción formal de un taxón asociada al nombre del autor o autores que la realizan, los cuales se hacen figurar detrás del nombre. En latín el plural de taxón es taxa, y es como suele usarse en inglés, pero en español el plural adecuado es taxones. La ciencia que define a los taxones se llama taxonomía. POLIMORFISMO.- Un polimorfismo es un sitio a lo largo de la secuencia del ADN donde distintas personas pueden tener secuencias diferentes. Puede tratarse de la sustitución de una simple letra (digamos que donde yo tenga una g usted tiene una t. O puede ser más complicado y donde hay una secuencia repetida, yo tenga veinte copias y usted 18). En resumen, es un sitio en el ADN donde hay una variación común entre individuos. Los cambios poco frecuentes en general no se llaman polimorfismos. Tienen que presentarse en una frecuencia de al menos uno por ciento para llenar el requisito de esta definición. LOCUS.- En biología, el locus es el lugar físico donde está un gen en un cromosoma. Todos los alelos de un gen están en el mismo locus, o sea, que si en un ser vivo hay un solo gen que, por ejemplo codifica una proteína, éste, estará en el mismo lugar en toda su especie. FILIACIÓN.- la filiación es la relación ascendente entre un grupo de parientes. Por ejemplo, entre los hijos y sus padres, o entre los abuelos y sus nietos. Se opone a la relación de descendencia. REGISTROS GENEALÓGICOS.- Esta unidad tiene como objeto certificar la información proveniente de las descendencias de animales registrados (Pedigrí), a fin de garantizar la pureza racial de los mismos. Archiva registros genealógicos de varias especies y razas desde mediados del siglo pasado. Estos datos son la base de las evaluaciones genéticas, a la cual debe adicionarse medidas objetivas de productividad. EXONES.- Es la región de un gen que contiene la información para producir la proteína del gen. Cada exón codifica una porción específica de la proteína completa. En eucariotas los exones de un gen se separan por regiones largas de ADN (llamadas intrones) que no codifican. INTRONES.- Es una secuencia no codificadora de ADN que separa a dos exones. El intrón inicialmente se transcribe en la molécula de ARN mensajero pero después es eliminado durante el proceso de maduración del ARN. PCR.- (reacción en cadena de la polimerasa), descrita en 1988. La PCR es una de las técnicas esenciales para la preparación de huellas dactilares, si bien no vale para elaborar marcadores por sí sola, la reacción básica de la PCR comienza con la desnaturalización del ADN molde para separar las cadenas, continúa con el alineamiento de un par de oligonucleótidos con su ADN molde, y termina con la polimerización para sintetizar un nuevo ADN entre los dos oligonucleótidos. 2.5 IDENTIFICACIÓN DE VARIABLES Variable independiente: Marcadores genéticos moleculares SNPs Variable dependiente: Características de importancia económica 2.6 DEFINICIÓN OPERATIVA DE VARIABLES E INDICADORES OBJETIVO Generar un panel de marcadores genéticos moleculares de la alpaca Huacaya. VARIABLES INDEP. Marcadores genéticos SNPs DEP. Características de importancia económica INDICADORES ESCALA Secuencia de ADN Número de repeticiones Alelos presentes medidos con repeticiones Mayor demanda de la alpaca. Mayor peso vivo Registros del control productivo CAPITULO III 3. METODOLOGÍA DE LA INVESTIGACIÓN 3.1 TIPO DE INVESTIGACIÓN Aplicada 3.2 NIVEL DE INVESTIGACION Tecnológica. 3.3 DISEÑO DE INVESTIGACIÓN Experimental. 3.4 MÉTODO DE INVESTIGACIÓN Matemático estadístico 3.5 POBLACIÓN, MUESTRA, MUESTREO Población Muestra Muestreo : 700 alpacas del centro experimental de Lachocc - UNH : 300 alpacas. : Dirigido. Localización del estudio: El ámbito de estudio está comprendido en el departamento, provincia y distrito de Huancavelica, en el Centro de Investigación de Camélidos Sudamericanos-Lachocc perteneciente a la Universidad Nacional de Huancavelica. Duración: El tiempo que demandara el trabajo de investigación propuesto es de 20 meses sin considerar el tiempo que demore la aprobación. 3.6 FUNDAMENTO Y TÉCNICA A UTILIZAR FUNDAMENTO Todo ser vivo, tanto plantas como animales están constituidos y controlados por un factor llamado “factor genético” el cual determina el comportamiento y manifestación del animal. Este factor genético está representado por una sustancia llamada ADN la cual a su vez es una secuencia de bases nitrogenadas distribuidas al azar y que en conjunto de dos, tres o más bases determinan secuencias llamadas intrones exones o genes los cuales son los responsables de la diferenciación entre animales desde el punto de vista genético. Un marcador genético es un segmento de ADN con una ubicación física identificable en un cromosoma y cuya herencia se puede rastrear en una familia. Un marcador puede ser un gen o puede ser un fragmento de ADN sin función conocida. Los marcadores se usan a menudo como una forma indirecta de rastrear el patrón hereditario de un gen que no ha sido identificado todavía y cuya ubicación aproximada se desconoce. Los marcadores se usan para el mapeo genético como el primer paso para encontrar la posición e identidad de un gen. La razón principal del incremento del uso de los SSRs como una herramienta molecular es que proveen la más alta incidencia de polimorfismo o PIC (Polymorphic Information Content) en comparación con otras técnicas como RFLPs y RAPDs. Poseen, además, atributos muy valorables porque tienen la característica de: a.- Ser altamente informativos: presentan herencia codominante y muchos alelos son encontrados entre individuos estrechamente relacionados. b.- Ser técnicamente simples: la tecnología del PCR puede fácilmente y rápidamente ser utilizada para la automatización en el uso de estos marcadores. c.- Ser una técnica sensible: sólo pequeñas cantidades de ADN son requeridas. d.- Ser analíticamente simples: los datos son producidos de forma confiable y altamente reproducibles. e.- Ser muy abundantes: los microsatélites están uniformemente dispersos a través del genoma, aproximadamente cada 10 Kbp. f.- Ser ampliamente aplicables: los loci son altamente conservados entre especies relacionadas y algunas veces entre géneros. Técnica de Sambrook (1989) Sambrook, reporta una técnica para extracción de ADN genómico basada en el uso de soluciones detergentes para lisis de membranas celulares y nucleares, seguido de un proceso de eliminación de proteínas mediante desnaturalización con fenol y un paso de eliminación de la fase orgánica con solventes como cloroformo. Finalmente, una etapa de precipitación del ADN genómico utilizando sales y alcoholes. Para luego responder el ADN genómico en una solución tamponada. Probabilidad de Exclusión (PE) La probabilidad de exclusión (PE) constituye la capacidad de un marcador genético para excluir a un falsamente asignado padre en términos de probabilidad (Weir, 1994). La probabilidad de exclusión depende del número de alelos y de las frecuencias alélicas del marcador y no de las frecuencias genotípicas observadas (Butler, 2005). a) PE1: Probabilidad de exclusión para un candidato a pariente. Dónde: pi es la frecuencia del alelo I y n es el número de alelos en un locus. b) PE2: Probabilidad de exclusión para candidato a pariente dado el genotipo de un pariente conocido del sexo opuesto. Dónde: pi es la frecuencia del alelo I y n es el número de alelos en un locus. c) PE3: Probabilidad de exclusión para un par de candidatos a parientes. Dónde: pi es la frecuencia del alelo I y n es el número de alelos en un locus. d) PEA = Probabilidad de exclusión acumulada o total. PE (acumulado o total) = 1- (1 - P1) (1 - P2) (1 - P3)…….. (1 - Pk) Dónde: Pk es la probabilidad de exclusión del locus K. 2.2.3. Contenido de información polimórfica (PIC) El contenido de información polimórfica (PIC) constituye una medida de la informatividad y polimorfismo de un locus genético, en estudios de parentesco refleja la probabilidad que una cría lleve un alelo raro, el cual permitiría la deducción del genotipo parental para un locus dado (Botstein y col., 1980). El valor de PIC es determinado mediante la siguiente formula: n n-1 PIC = 1 - ( ∑ pi 2 ) - ∑ i=1 n ∑ 2 pi 2 pj 2 i=1 j=i+1 Donde: pi es la frecuencia del alelo I, pj es la frecuencia del alelo J y n es el número de alelos en un locus (Botstein y col., 1980). 2.2.7. Identidad y Probabilidad de identidad (PID) La determinación de identidad se basa en la búsqueda de genotipos idénticos asumiendo la asociación de alelos dentro y entre loci al azar (Waits y col., 2001). Un estimador de identidad es la probabilidad de identidad (PID), la cual refleja la probabilidad que dos individuos seleccionados poseen un genotipo idéntico al analizar un mismo locus. La probabilidad de identidad es calculada mediante la siguiente formula: P(ID) = ∑ pi 4 + ∑ ∑ (2 pi pj)2 Dónde: pi es la frecuencia del alelo I, pj es la frecuencia del alelo J e i j ; Butler, 2005). Cuantificación de la Diversidad Genética Existen estimadores de la diversidad genética basados principalmente en frecuencias alélicas o frecuencias genotípicas. Los principales estimadores de la diversidad genética son: la diversidad alélica (A), proporción de loci polimórficos (P), heterocigosidad esperada (H E) y heterocigosidad observada (HO) (Freeland, 2005) . Diversidad Alélica La frecuencia relativa de un alelo en particular en una población es denominada frecuencia alélica. La frecuencia alélica es considerado un indicador fundamental en estudios evolutivos, porque un cambio genético en una población es usualmente descrito por un cambio en la frecuencias de sus alelos (Nei y Kumar, 2000). La frecuencia alélica en especies diploides consiste en número de alelos de un tipo dado dividido entre dos veces el tamaño poblacional para un locus autosomal (Templeton, 2006) y la diversidad alélica (A) constituye simplemente el numero promedio de alelos por locus (Nei y Kumar, 2000). Heterocigosidad La heterocigosidad (H) constituye la proporción de individuos heterocigotos en la población. Un valor de heterocigosidad elevado significa que existe una elevada diversidad alélica presente en la muestra analizada (Butler, 2005). La heterocigosidad puede ser dividida en dos indicadores: la heterocigosidad esperada (HE) y la heterocigosidad observada (HO). La heterocigosidad esperada (HE) también llamada diversidad génica (Nei, 1973), es definida como la probabilidad que dos alelos escogidos al azar en una población sean diferentes (Weir, 1996). n HE = 1 - ∑ pi 2 i=1 Donde: pi es la frecuencia del alelo I y n es el número de alelos en un locus (Nei, 1973). La HE es usada comúnmente como una medida de diversidad genética que refleja el nivel de heterocigosidad que podría esperarse en una población que se encuentre en equilibrio de HardyWeinberg (Freeland, 2005). La heterocigosidad observada (HO) es definida como la proporción de individuos heterocigotos observados para un locus en particular (Weir, 1996). Coeficiente de Consanguinidad (F o FIS) Consanguinidad, se define al resultado del cruzamiento entre individuos estrechamente relacionados. La forma más intensa de consanguinidad es originada por autofecundación y es dada en individuos hermafroditas. La consanguinidad puede ser medida mediante dos importantes coeficientes: el coeficiente de parentesco o coancestria (FJK) y el coeficiente de consanguinidad (FI, F o FIS) (Maynard, 1999). El coeficiente de parentesco o coeficiente de coancestria (FJK) constituye la probabilidad que dos genes homólogos de dos individuos J y K seleccionados al azar sean idénticos por descendencia (Maynard, 1999). El concepto de identidad por descendencia puede definirse como la propiedad que dos alelos dados sean copias de un solo alelo que existió en un ancestro reciente (Freeland, 2005). El coeficiente de consanguinidad (FI, F o FIS) constituye la probabilidad que dos alelos en un locus en un individuo sean idénticos por descendencia (Maynard, 1999; Freeland, 2005). El coeficiente de consanguinidad describe la proporción por la cual la heterocigosidad disminuye en relación con una población que presenta cruzamientos al azar asumiendo que ambas presentan la misma frecuencia génica inicial (Maynard, 1999). DESCRIPCIÓN DE LA TÉCNICA 1.- Selección fenotípica. La selección fenotípica consiste en la elección de n grupo de animales mediante la evaluación de un set de caracteres fenotípicos con capacidad de medición. 2.- Identificación. a) Identificación de marcadores microsatélites in silico. La identificación in silico consiste en la identificación y elección de marcadores microsatélites en base a información reportada sin la necesidad de efectuar una técnica de laboratorio. La identificación in silico consiste en un método de tamizado y constituye el primer paso en cualquier estudio de identificación y caracterización de un set de marcadores genéticos. b) Caracterización de marcadores microsatélites fluorescentes mediante electroforesis en capilar. La caracterización de microsatélites fluorescentes consiste en el marcado con un fluorocromo que permite la detección de la fluorescencia emitida al ser excitado por un rayo láser y capturada la fluorescencia por un sensor automático. La electroforesis en capilar se basa en el uso de un capilar y una matriz o polímero líquido que permite la separación del ADN en base a su tamaño. Previamente se solicitara la adquisición del secuenciamiento del primer a trabajar en un laboratorio de Estados Unidos, lo cual posteriormente se procesara en el secuenciador automático propiedad del Instituto Peruano de Energía Nuclear (IPEN). 3.- Análisis de identidad El análisis de identidad se basa en el índice de paternidad y los valores de LOD que son razones de probabilidad de ocurrencia entre una hipótesis de asignación de filiación versus una hipótesis de no filiación. 4.- Análisis de consanguinidad Se basa en estadística F o estadística de Wright que determina niveles de relación entre los individuos mediante la capacidad de compartir ciertos alelos. 5.- Análisis de variabilidad genética. Se basa en estadística F o estadística de Wright, en los niveles de heterocigosidad (Numero de heterocigotos en un población). 6.- Análisis de asociación genética entre SNPs y características fenotípicas de la fibra Se basa en la determinación de relaciones estadísticas entre las variables genéticas (alelos de los diferentes marcadores genéticos) y las variables fenotípicas a estudiar. 3.7 PROCEDIMIENTOS PARA EL ANALISIS COMPONENTE 1 DESARROLLO E IMPLEMENTACION DE LIBROS DE REGISTRO Y DE PEDIGRI VALIDADOS MEDIANTE ADN Actividad 1. Generación de libros de registro fenotípico en alpacas reproductoras de alta calidad. Evaluación fenotípica y selección de alpacas reproductoras de alta calidad. Se evaluaran fenotípicamente todas las alpacas Huacaya adultas entre 2 y 6 años de edad y las crías de alpaca, de ambos sexos (machos y hembras) sin discriminar el color ni la edad proveniente del Centro de Investigación de la Universidad Nacional de Huancavelica para los principales descriptores raciales y de producción (Tabla 1). Los datos fenotípicos colectados serán anotados en una ficha de evaluación para luego ser almacenados electrónicamente en una base de datos (archivo Excel-Microsoft). Trescientas alpacas serán seleccionadas mediante el cumplimiento del estándar racial, en estricto orden de mérito fenotípico. Las alpacas seleccionadas serán identificadas mediante microchips insertados debajo de la piel a nivel del cartílago auricular. Descriptores e Indicadores Fenotípicos: Sexo Se determinara el sexo de los animales por medio de inspección visual al momento de la evaluación. Color Se determinara el color del vellón mediante inspección visual y comparación con un catálogo de colores de vellón que servirá como patrón de color. Peso vivo Se determinara el peso vivo de los animales mediante una balanza digital para ganado. Tamaño Se determinara el tamaño de las alpacas mediante el uso de una cinta métrica. La medición de la alzada a la cruz tomara como puntos de referencia: la punta de la pezuña y la parte superior de la cruz (unión de los dos miembros anteriores) y la longitud horizontal tomando como puntos de referencia el punto medio del pecho y la base de la cola. Como medidas anexas se tomaran la longitud del cuello y el volumen torácico. Peso de Vellón El peso de vellón se determinara mediante una balanza común tipo reloj. Esta medición se realizara por duplicado. Densidad de Vellón Se determinaran la densidad del vellón mediante conteo del número de folículos primarios y secundarios por centímetro cuadrado a través de microscopia de una biopsia de piel de aproximadamente 2 centímetros cuadrados. Rizos Se determinara el número de rizos mediante inspección visual y medición con una regla graduada en centímetros. Finura Se determinara el valor de finura de fibra mediante colección de una muestra de 100 gramos de vellón del costillar medio. El promedio, desvió estándar, coeficiente de variación y % de pelos mayores de 30 micras será determinado utilizando el equipo de análisis de fibra OFDA 2000. Generación de informe sobre características fenotípicas en alpacas reproductoras de alta calidad Las fichas fenotípicas serán introducidas en formato electrónico Microsoft Excel y serán analizadas en los programas estadísticos de SAS Y SPSS para la determinación de media, desviación estándar y coeficiente de variación para las alpacas procedentes del Centro de Investigación, para la emisión de una línea de base fenotípica. Además se realizara un análisis de correlación y regresión entre los principales indicadores fenotípicos como peso vivo, peso de vellón, uniformidad, densidad, finura de la fibra, entre otros. Actividad 2. Generación de libros de pedigrí Las 300 alpacas seleccionadas serán introducidas en un registro de reproductores como medio de identificación y control genealógico. Se realizaran cruzamientos entre las alpacas seleccionadas mediante empadre controlado y será detallado en un registro de empadre como medio de verificación en la elaboración de un libro de pedigrí de alpacas de alta calidad. Las relaciones de filiación serán validadas mediante una prueba de ADN utilizando marcadores microsatélites previamente reportados (Agapito y col., 2008). COMPONENTE 2 IDENTIFICACIÓN Y DESARROLLO DE MARCADORES GENÉTICOS SNPs PARA EL MEJORAMIENTO GENÉTICO DE ALPACA Actividad 1 Elaboración de un banco de ADN de alpacas reproductoras Se establecerá un banco de ADN de los reproductores almacenados a -20°C que sirva como referencia para futuros estudios de genética y genómica de la alpaca Colección de muestras de sangre Se colectarán 3ml de sangre mediante punción en la vena yugular utilizando tubos vacutainer con anticoagulante (EDTA) aproximadamente a 700 alpacas Huacaya (Vicugna pacos) seleccionadas fenotípicamente previamente e identificadas con microchips. Extracción de ADN ADN genómico será extraído a partir de 200 microlitros de sangre con anticoagulante mediante el método de extracciones sucesivas con solventes orgánicos (Fenol-Cloroformo) (Sambrook et. al., 1989). El ADN extraído se colocará en tubos de microcentrífuga de 1.5 ml de capacidad y se almacenará en una congeladora a -20°C. Se procederá a su cuantificación mediante comparación con diluciones seriadas de un marcador de peso molecular λ ADN Hind III® (FERMENTAS) en un gel de agarosa al 1.0% Determinación de la calidad y concentración del ADN genómico La calidad del ADN genómico extraído será determinada mediante electroforesis en gel de agarosa al 1% -TBE 0.5X (Tris-Borato-EDTA). El ADN genómico será cuantificado mediante espectrofotometría utilizando un equipo Nanodrop. Finalmente, el ADN genómico obtenido será diluido con una solución TE (Tris-EDTA) hasta una concentración final de 5 ng/µL. Actividad 2 Identificación y caracterización de marcadores SNPs en alpacas reproductoras de alta calidad Se identificara y validara un panel de 20 marcadores SNPs de alpaca y se explorara su aplicación en las actividades de mejoramiento genético para lo cual se utilizaran 2 bibliotecas de cDNA de piel de alpaca: i) una reportada y anotada en la base de datos del Genebank y ii) otra generada recientemente en la Unidad de Biotecnología Molecular (UBM) de la Universidad Peruana Cayetano Heredia. Un listado inicial de ESTs para los 3 genes candidatos será elaborado en base a las secuencias disponibles en Genebank y las obtenidas mediante la biblioteca de cDNA de la UBM. Se identificaran SNPs manualmente mediante la observación y análisis de los cromatogramas obtenidos por secuenciamiento directo de dos bibliotecas de cDNA de piel de alpaca. Secuencias de los genes de keratinas y proteínas asociadas a keratinas disponibles en Genebank y en la biblioteca de cDNA de piel de alpaca seran alineadas utilizando CLUSTAL W program para obtener una secuencia consenso a partir de la cual se diseñaran cebadores específicos para amplificar fragmentos únicos entre 300 y 500 bp utilizando el programa Primer Designer 3 software (Scientific Educational Software, Durham, USA). La especialidad de los cebadores (presencia de un solo producto de amplificación) será evaluada mediante amplificación por PCR a partir de DNA genómico utilizando condiciones y ciclos termales estandar. Fragmentos de 300 a 500 pares de bases de los 4 genes candidatos serán amplificados mediante PCR utilizando los cebadores diseñados previamente utilizando Quiagen Multiplex PCR Kit (QUIAGEN) en un termociclador modelo 9700 GeneAmp® (Applied Biosystem). Los productos de PCR seran secuenciados usando ABI PRISM Big Dye Terminador Cycle Sequencing Ready Reaction Kit y un secuenciador automático ABI PRISM 3130 Genetic analyser®. La edición de las secuencias se realizara mediante el Clustal W y Mega v 4.0. Un alineamiento múltiple de las secuencias generadas se realizara con el programa Mega v4.0, para identificar la presencia de SNPs en los pooles de ADN genómico para cada uno de los 4 genes secuenciados. Actividad 3 Elaboración de pedigríes e identificación de familias de referencia Información disponible en libros de registro, libros de nacimientos, libros de empadre, libros de compra, venta de alpacas del centro de reproductores serán analizados para la construcción de libros de pedigrí. Se cotejará el pedigrí mediante la validación del parentesco de los animales inscritos, mediante genotipificación con un panel de 10 marcadores microsatélites y análisis con el programa Cervus v3.0 (Kalinowski et al., 2007). Adicionalmente se cotejara el pedigree con los genotipos obtenidos a partir del análisis de 12 marcadores SNPs previamente determinados. La determinación de las genealogías se realizara mediante el programa PyPedal. Se determinara el nivel de heterocigocidad de la población mediante la medición del coeficiente de consanguinidad individual (F), coeficiente de relación promedio (AR) mediante el programa FSTAT v4.02 (Weir & Cockerham, 1984) y Pypedal. Actividad 4 Base de datos de genotipos de ADN alpacas reproductoras Se elaborara un catálogo de ADN genómico y genotipos de 12 marcadores microsatélites de alpacas, tanto en formato electrónico (Microsoft – Excel) y en forma física (Impresiones). COMPONENTE 3 APLICACIÓN DE MARCADORES MICROSATELITES Y SNPs EN LA GENERACION DE HERRAMIENTAS PARA MEJORA GENETICA EN UN PLANTEL DE REPRODUCTORES Actividad 1 Identificación de los niveles de consanguinidad y variabilidad genética en el plantel reproductor Amplificación y análisis de ADN microsatélite Doce loci microsatélites descritos para alpacas y llamas (Lang et al.1996, Penedo et al., 1998; Penedo et al., 1999) serán amplificados en dos reacciones de PCR múltiple utilizando Qiagen Multiplex PCR Kit (QIAGEN) en un termociclador modelo 9700 GeneAmp® (Applied Biosystem). Los productos de PCR serán separados mediante un Analizador Genético ABI 3130 ADN sequencers® (Applied Biosystems). El tamaño de los productos amplificados se determinaran con el programa Genemapper® v.4.0 (Applied Biosystems). Análisis de variabilidad genética y consanguinidad en alpacas Se determinara la variabilidad genética para la población de alpacas del Centro de Investigación mediante el análisis de las frecuencias alélicas, número de alelos por locus, heterocigosidad observada y esperada (Nei, 1973) e índice de Garza-Williamson (Garza y Williamson, 2001), contenido de información polimórfica (PIC), probabilidad de exclusión (Jamieson & Taylor, 1997) para cada locus utilizando el programa Arlequín v3.1 (Excoffier, Laval y Schneider, 2005). Se determinara las relaciones de filiación entre los animales utilizando una prueba de determinación de parentesco (Agapito et al., 2008) utilizando el programa Cervus 3.0 (Kalinowski et al., 2007). Los niveles de consanguinidad serán determinados mediante el coeficiente de consanguinidad (FIS) e índice de fijación (FST) para cada población y para la totalidad de individuos analizados utilizando el programa Genepop v4.0 (Raymond y Rousset, 1995) y Arlequín v3.1 (Excoffier, Laval y Schneider, 2005). Actividad 2 Análisis de asociación genética entre 3 genes vinculados a la producción de fibra de alpaca y características de fibra Se utilizaran 50 alpacas emparentadas (medios hermanos) para un ensayo de asociación con medios hermanos. Los haplotipos para los 3 genes analizados serán determinados mediante el programa DNA baser y Arlequin v3.1 para las 20 alpacas analizadas. Un análisis de asociación para cada uno de los alelos y haplotipos de los genes THH, KRTAP1-1 y KRT33 con las principales características de fibra (diámetro, lustre, confort, densidad) será realizado mediante un análisis de varianza (ANOVA) utilizando el programa SPSS v13.0 COMPONENTE 4 FORTALECIMIENTO DE LAS CAPACIDADES DE INVESTIGACION EN GENOMICA DE ALPACAS Actividad 1 Desarrollo e implementación de cursos de capacitación en genética molecular y genómica Se implementara un curso teórico sobre Genética y Genómica Molecular con una duración total de 20 horas dirigidas a estudiantes y profesionales. Se implementara un curso práctico sobre Técnicas y Aplicaciones en Genética Molecular con una duración total de 20 horas dirigidas a estudiantes y profesionales. Actividad 2 Difusión de resultados Se publicara un artículo científico en una revista nacional y/o internacional, además se realizara una presentación de los resultados en una conferencia científica nacional. 3.7 DE LA TOMA DE MUESTRA La muestra utilizada para el análisis genético será sangre de alpaca, las cuales se tomarán de la vena ubicada en la parte interna de la pierna, para lo cual estos animales serán derribados luego del cual se procederá a tomar la muestra de sangre en una cantidad aproximada de 4 ml con una aguja de dos puntas las cuales se depositarán en un tubo al vacío con anticoagulante incluido (vacutainer) previamente se anotarán el número de arete de la alpaca para su identificación posterior. La muestra previamente homogenizada con el anticoagulante será transportada en hielo hasta el laboratorio de Biotecnología de la Universidad Nacional de Huancavelica. 3.8. DEL ANALISIS EN LABORATORIO (GENETICO) EXTRACCION DE ADN: ELIMINACIÓN DE GLÓBULOS ROJOS (HEMOLISIS) La extracción de los glóbulos rojos se hará en el laboratorio de Biotecnología de la Universidad Nacional de Huancavelica: PROTOCOLO PARA ELIMINAR GLÓBULOS ROJOS HEMOLISIS 1.- Colocar la muestra de sangre en un tubo falcón de 15 ml 2.- Completar hasta 15 ml con la solución de hemólisis 3.- Homogenizar con movimiento suave 4.- Llevar al refrigerador a 4ºC por dos horas para lisar los glóbulos rojos (en la parte inferior del refrigerador) 5.- Centrifugar a 3000 rpm por 15 a 30 minutos aproximadamente 6.- Se observa el pellet 7.- Sacar toda la sangre de encima 8.- Nuevamente enrazar con la solución de hemólisis 9.- Centrifugar nuevamente a 3000 rpm por espacio de 15 a 30 minutos 10.- Sacar nuevamente el sobrenadante LISIS DE GLÓBULOS BLANCOS Luego de eliminar los glóbulos rojos es necesario eliminar los glóbulos blancos, significa que la purificación es por partes para quedarse finalmente con el ADN limpio, para lo cual se usara el siguiente protocolo: PROTOCOLO PARA LISAR GLÓBULOS BLANCOS 1.- Colocar en cada tubo con el contenido de la muestra sin glóbulo rojo 2.5 ml de la solución de lisis 2.- Agregar a cada tubo 250 ml de SDS (al 10%) 3.- Homogenizar suavemente 4.- Aplicar la proteinaza 20 ml (de una proteinaza que está a una concentración de 20 miligramos /ml) 5.- Luego incubar a 53 °C por 18 a 22 horas 6.- Aplicar 675 L de Cl Na (6 molar ) en cada tubo 7.- Homogenizar hasta que salga espuma 8.- Centrifugar a 3,000 rpm por tiempo de 30 minutos 9.- El sobrenadante (ADN ) transferir a otro tubo falcon 10.- Echar 2 ml de isopropanol 11.- Poner a un ependorff de 1.5 ml el ADN que está en forma de moco 12.- Centrifugar a máxima velocidad por 2 minutos 13.14.15.16.17.18.19.- Se desecha el líquido sobrenadante Luego echar 0.5 ml de alcohol al 70% Centrifugar 2 minutos a máxima velocidad Nuevamente echar 0.5 ml de alcohol al 70% Centrifugar nuevamente 2 minutos a máxima velocidad El sedimento es ADN limpio Dejar abierto el tubo pero cubierto con papel absorbente para que evapore el alcohol por 24 horas 20.- Echar buffer T-E (TRIS –EDTA) en cantidad conveniente (0.25 ml) COMPOSICIÓN DE LA SOLUCION DE LISIS (para 16 tubos) Tris HCl (2molar) ClNa (6 molar) EDTA (0.25 molar) Agua destilada En cada tubo: SDS (al 10%) Proteinaza (20mlgr/ml) 200 L 320 L 320 L 40 ml 250 L 20 L LECTURA DE CALIDAD DE ADN La lectura de la calidad de ADN se hará en el espectrofotómetro a 260 y 280 nanómetros lo cual revela la calidad de ADN. 3.9 ÁMBITO DE ESTUDIO El ámbito de estudio está comprendido en el departamento y provincia de Huancavelica, en el Centro de Investigación de Camélidos Sudamericanos-Lachocc perteneciente a la Universidad Nacional de Huancavelica. CAPITULO IV 4. ASPECTO ADMINISTRATIVO 4.1 RECURSOS HUMANOS La ejecución del presente proyecto cuenta con el siguiente personal que describimos a continuación: INVESTIGADORES Ing. M. Sc. WILLIAM SALAS CONTRERAS Docente Principal de la Universidad Nacional de Huancavelica Especialidad: Genética Animal. Ph. D. JOSE ESPINOZA BABILÓN Docente Principal de la Universidad Peruana Cayetano Heredia Especialidad: Genética Molecular Instituto al que pertenece: Instituto de Genética y Biología Molecular Institución a la que pertenece: Facultad de Ciencias y Filosofía COLABORADORES 4.2 Alumnos de la Escuela Académico Profesional de Zootecnia de la Universidad Nacional de Huancavelica. Personal técnico del Centro Experimental Lachocc-Huancavelica. RECURSOS MATERIALES DE LA LOCALIZACION DEL ESTUDIO El trabajo se llevará cabo en el Centro de Investigación de Camélidos Sudamericanos de Lachocc (CIDCS) de la Universidad Nacional de Huancavelica, la cual abarca una extensión de 6,200 hectáreas de praderas, incluidas 40 hectáreas de bofedales y se encuentra ubicada en la Sierra Centro del Perú, departamento y provincia de Huancavelica a una altitud de 4300 metros sobre el nivel del mar. Las características agroecológicas de Lachocc corresponden a puna seca que incluye rocas, bofedales y riachuelos en la que prevalece una temperatura promedio anual de 6 ºC (3.8 ºC en julio y 8.2 ºC en noviembre), una precipitación pluvial de 688.3 mm/año (con meses húmedos de diciembre a marzo y meses secos de mayo a octubre) y una predominancia de Festuca dolicophylla, Stipa spp, y el Calamagrostis spp; pasturas características de puna seca (Huancavelica). DE LOS ANIMALES Los animales que se utilizaran serán 300 alpacas de color blanco de la variedad Huacaya tomados del total existente. DE LOS MATERIALES: DE CAMPO 1.- Máquina fotográfica 2.- Lapiceros 3.- Cuaderno de campo 4.- Aguja de dos puntas para muestrear sangre 5.- Tubos vaccutainer, con anticoagulante k3 EDTA 6.- Alcohol 7.- Lápiz DE LABORATORIO REACTIVOS 1.- Alcohol isoamílico 2.- Cloroformo 3.- Cloruro de sodio 4.- Etanol 70% y 90% 5.- Isopropanol 6.- RNAasa (10mg/mL) 7.- SDS (dodecil sulfato de sódio) 8.- EDTA 9.- Tubos falcon de 15 ml 10.- Tubos eppendorf de 1.5L y de reacción de 0.2 L EQUIPOS 1.- Baño maría 2.- Balanza analítica 3.- Bandejas para tinción 4.- Cámara de electroforesis horizontal 5.- Cámara de electroforesis vertical 6.- Cámara extractora 7.- Centrífuga refrigerada de 1300 rpm 8.- Congeladora 9.- Destilador de agua 10.- Espectrofotómetro 11.- Fuente de poder de 140 V 12.- Fuente de poder 13.- Horno microondas 14.15.16.17.18.19.20.21.22.23.24.- Microcentrifuga de tubos ependorff 1000 rpm Micropipetores Potenciometro Purificador de agua ultrapura Shaker Termociclador Transiluminador de luz ultravioleta UV Transiluminador de luz visible Vortex Poliacrilamida Vidrios para la poliacrilamida OTROS MATERIALES 1.- Erlenmeyer 125, 250 y 500 ml 2.- Guantes de goma 3.- Papel aluminio 4.- Papel toalla 5.- Pipetas pasteur 6.- Placas PCR 7.- Probetas de 50, 100 500 y 1000 ml 8.- Puntas para micropipetas: 0.5, 10, 100, 1000 L 9.- Tubos de centrífuga de 15 ml 10.- Tubos ependorff de 1.5 ml y de reacción de 0.2 ml 4.3 FINANCIAMIENTO El presente proyecto será financiado por la Universidad Nacional de Huancavelica, con el Fondo del Gas de Camisea (FOCAM) 4.4 CRONOGRAMA DE ACTIVIDADES (MENSUAL) Actividad (meses) C1.DESARROLLO E IMPLEMENTACIÓN DE LIBROS DE REGISTRO Y PEDIGRÍ VALIDADOS MEDIANTE ADN C1.A1.Generación de libros de registro fenotípico en alpacas reproductoras de alta calidad C1.A2.Generación de libros de pedigrí C2.IDENTIFICACIÓN Y DESARROLLO DE MARCADORES GENÉTICOS SNPs 1 2 3 4 5 6 7 8 9 10 11 12 X X X X X X X X X X X X X X X 13 14 15 16 17 18 19 20 PARA EL MEJORAMIENTO GENÉTICO DE ALPACA C2.A1.Elaboración de un banco de ADN de alpacas reproductoras C2.A2. Identificación y caracterización de marcadores microsatélites en alpacas reproductoras de alta calidad C2.A3.Elaboración de pedigríes e identificación de familias de referencia C2.A4.Base de datos de genotipos de ADN alpacas reproductoras C3. APLICACIÓN DE MARCADORES SNPs PARA LA GENERACIÓN DE HERRAMIENTAS PARA MEJORA GENÉTICA EN UN PLANTEL DE REPRODUCTORES C3.A1.Identificación de los niveles de consanguinidad y variabilidad genética en el plantel reproductor C3.A2.Identificación de valores de heredabilidad de caracteres cuantitativos para uso en selección C4.FORTALECIMIENTO DE LAS CAPACIDADES DE INVESTIGACIÓN EN GENÓMICA DE ALPACAS C4.A.1.Desarrollo e implementación de cursos de capacitación en genética molecular y genómica C4.A2.Capacitación de profesionales C4.A3. Difusión de resultados e informe final X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X 4.5 PRESUPUESTO PRESUPUESTO DEL PROYECTO DE INVESTIGACION "IDENTIFICACIÓN DE MARCADORES GENETICOS MOLECULARES SNPs EN LA ALPACA HUACAYA QUE INFLUYEN EN CARACTERISTICAS DE IMPORTANCIA ECONOMICA" DESCRIPCION DEL BIEN CANT UNID COSTO UNIT. PRECIO TOTAL 1 unidad 155000 155000 155000 1 unidad 200000 200000 200000 1 unidad 200000 200000 200000 Unidad 8500 8500 8500 1 2 EQUIPAMIENTO 2.6.32.12 2.6.32.9 99 2.6.32.9 99 Equipo automatizado para extracción de ADN Sistema multicapilar para electroforesis (sistema quiaxel) Dispensador automatizado para reacciones de PCR en tiempo real (quiagility) Computadora core i7 3.1 de 3 teras de disco duro 1 termociclador de gradiente 1 unidad 30670 30670 30670 2.6.32.9 99 Congeladora de -20 °C 1 unidad 2000 2000 2000 2.6.32.12 Escritorio tipo gerente 1 unidad 600 600 600 2.6.32.12 Escritorio tipo secretaria 1 unidad 400 400 400 Impresora HP LaserJet Pro M425dn, función, imprime/copia/scanea 1 Equipo 2000 2000 2000 1 Unidad 2500 2500 2500 2.6.32.9 99 2.6.32.31 2.6.32.9 99 2.6.32.12 Lectora de microchip IDE FX pet. Mueble para computadora 1 unidad 300 300 300 2.6.32.9 99 Refrigeradora 1 unidad 2000 2000 2000 2.6.32.12 Sillón giratorio tipo secretaria 1 unidad 400 400 400 Vortex 1 unidad 4000 4000 4000 2.6.32.9 99 608370 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 INSUMOS 2.3.1.8.2.1 Tubos de 1.5 ml(caja x 1000 tubos) 1 bolsa 100 100 100 2.3.1.8.2.1 tips sin filtro de 0.5 a 10 µL (bolsa x 1000) 1 bolsa 100 100 100 2.3.1.8.2.1 Tips sin filtro de 200µL (bolsa x 1000) 1 bolsa 100 100 100 2.3.1.8.2.1 Tips sin filtro de 1000 µL(bolsa x 1000) 1 bolsa 100 100 100 tips con filtro de 0.5 a 10 µL(caja x 960 tips) 1 caja 380 380 380 2.3.1.8.2.1 tips con filtro de 200 µL(caja x 960 tips) 1 caja 380 380 380 2.3.1.8.2.1 tips con filtro de 1000 µL(caja x 1000 tips) 1 caja 380 380 380 gradilla para tubos de 0.2 ml PCR 2 unidad 70 140 140 3 unidad 15 45 45 5 unidad 15 75 75 1 unidad 1200 1200 1200 1 unidad 1200 1200 1200 1 unidad 1200 1200 1200 1 unidad 1200 1200 1200 2.3.1.8.2.1 2.3.1.8.2.1 2.3.1.8.2.1 2.3.1.8.2.1 2.3.1.8.2.1 2.3.1.8.2.1 2.3.1.8.2.1 2.3.1.8.2.1 2.3.199.12 2.3.199.12 2.3.199.12 2.3.199.12 gradilla para tubos de 1.5 ml PCR gradilla para tubos eppendorf Micropipeta de rango variable de 0.5 a 10 µl Micropipeta de rango variable de 10 a 100 µl Micropipeta de rango variable de 20 a 200 µl Micropipeta de rango variable de 100 a 1000 µl taq polimerasa hot start (250 U) 1 unidad 1000 1000 1000 agua de grado molecular de 500 ml 1 unidad 800 800 800 kit de extracción de DNA sangre, tejido animal (kit x 50) 1 unidad 1100 1100 2200 Hot Start Taq Plus master mix (250 Det) 1 unidad 2400 2400 2400 2.3.199.12 2.3.199.12 2.3.199.12 2.3.199.12 kit para PCR en tiempo real con Sybrgreen (400 det) 1 unidad 2500 2500 2500 0.2 mL Thin Wall PCR tubes domed cap (1000 tubes) 1 Frasco 150 150 150 1 Frasco 100 100 1000 1.5 mL Boil Proof microtubes(500) Axygen 2µL Universal fit filter tips, racked&presterilized (10Rack x 96 tips) 1 Frasco 600 600 600 2.3.199.12 3130POP-7TM 1 Frasco 1000 1000 1000 2.3.199.12 Agarosa 100 gramos 1 Frasco 200 200 200 2.3.199.12 Agujas 21 1/2 para tubo vacutainer (caja x 100) 2 Caja 60 120 120 2.3.16.199 Arete metálico Cajas Criobox 1 ciento 300 300 300 1 Caja 50 50 50 2.3.199.12 dNTP set, PCR grade 4x100µL 1 Frasco 400 400 400 2.3.199.12 EDTA Disodium Salt (500 gr) 1 Frasco 500 500 500 2.3.199.12 Etanol 2.5 litros 1 Frasco 180 180 180 2.3.16.199 frasco transparente tapa rosca (1000 ml) 2 unidad 30 60 60 2.3.16.199 Gradilla y soporte para tubos PCR 2 unidad 70 140 140 2.3.16.199 Gradillas para tubos ependorff 2 unidad 15 30 30 2.3.16.199 Holders para colección 3 Caja 20 60 60 2.3.199.12 Loading buffer gel 6X Microchip 1 Frasco 200 200 400 2 Caja 600 1200 1200 Papel toalla Proteinasa K de 20 mg/ml 30 Rollo 5 150 150 200 400 400 2.3.16.199 2.3.199.12 2.3.16.199 2.3.199.12 frasco 2 Puntas para micropipetas de 0.5 µL con filtro x 10 cajas 1 bolsa 130 130 130 SDS 50 Gramo 6 300 300 Tubo Vacutainer de 10 mL (caja x 100)con anticoagulante K3 EDTA 3 Caja 100 300 300 2.3.16.199 Tubos PCR (1000 tubos) 1 bolsa 220 220 220 2.3.16.199 Tubos ependorff (1000 tubos de 1.5ml) 1 bolsa 200 200 200 2.3.16.1 99 Aretador para ganado 1 Unidad 150 150 150 2.3.16.199 2.3.199.12 2.3.16.199 21540 2.3.2.1.22 2.3.2.1.21 2.3.2.1.12 2.3.2.1.11 PASANTIA Viáticos para viajes diversos al interior del país Pasajes para viajes diversos al interior del país 100 Día 200 20000 2000 2000 2000 2000 2000 2000 2000 2000 2000 2000 2000 2000 2000 70 Pasaje 50 3500 350 350 350 350 350 350 350 350 350 350 350 350 350 Viáticos al exterior del país 1 viaje 20000 20000 10000 10000 Pasajes al exterior 1 Viaje 4000 4000 2000 2000 47500 2.3.12 VESTUARIO 2.3.1.2.11 Guardapolvo blanco 2.3.13 COMBUSTIBLE 2.3.1.3.11 Gasolina (84 octanos) 680 Galon 16 10880 10880 2.3.1.3.11 Petróleo (diesel B5) 677 Galon 16 10832 10832 10 Unidad 60 600 600 600 21712 MATERIALES DE ESCRITORIO 2.3.15.12 Engrapador tipo tijera 1 unidad 50 50 50 2.3.15.12 Papel bond A4 de 80 gramos 30 Millar 40 1200 1200 2.3.15.12 Tóner para impresora laser 2 Unidad 300 600 600 1850 2.3.27.1.2 CAPACITACION Servicio de asesoría para la ejecución del proyecto de investigación ; capacitación a productores y profesionales. Entrenamiento en técnicas de biología y genética molecular avanzado en las instalaciones de la unidad de Biotecnología Molecular UPCH 1 Consultor 30000 30000 15000 2.3.27.1.2 Servicio de consultoría y entrenamiento en técnicas de biología y genética molecular avanzado en las instalaciones de la unidad de Biotecnología Molecular UPCH 1 Especialista 20000 20000 10000 10000 2.3.27.32 Capacitación a productores y profesionales, alumnos y tesistas 1 Especialista 10000 10000 2500 2500 2.3.27.32 Capacitación en uso de software exclusivo para genómica y biología molecular 2 Mes 3000 6000 3000 3000 3500 3500 15000 2500 66000 ASISTENTES DE INVESTIGACION 2.3.27.2 99 Asistente de investigación experto en preparación de muestras 3 Mes 3500 10500 3500 2500 2.3.27.2 99 Asistente experto en procesamiento de material genético (ADN) 3 Mes 3500 10500 2.3.27.2 99 Asistente de laboratorio experto en manejo de software de ADN 3 Mes 3500 10500 2.3.27.2 99 Asistente de laboratorio para compilación de información genética 3 Mes 3500 10500 2.3.27.2 99 Contador publico colegiado 2 Mes 3300 6600 2.3.27.2 99 Asistente de investig. de campo para genética y selec.(Bach. ing. Zoot.) 3 Mes 3200 9600 3 Mes 2350 7050 2350 2350 2350 2.3.27.2 99 Técnico de campo (Bach. Zootecnista o afín ) Asistente Administrativo 20 Mes 1400 28000 1400 1400 2.3.27.2 99 Experto en calificación fenotípica de alpacas 1 evento 8000 8000 8000 2.3.27.2 99 Experto en calificación de caracteres reproductivos de alpacas 1 evento 8000 8000 Apoyo secretarial 14 Mes 1000 14000 2.3.27.2 99 servicio de limpieza 4 Mes 1000 4000 2.3.27.2 99 Apoyo secretarial 8 Mes 1000 2.3.27.2 99 8000 3500 3500 3500 3500 3300 3500 3500 3500 3500 3500 1400 1400 1400 3300 3200 3200 3200 1400 1400 1400 1400 1000 1000 1000 1000 1000 1000 1400 1400 1400 1000 1000 1400 1400 8000 1000 1000 1000 1000 135250 PUBLICACIÓN, COMUNICACIÓN Y DIFUSIÓN 2.3.27.2 99 Publicación de trabajo en revista científica Ponencia de presentación de resultados 2.3.22.44 Impresión de manuales didácticos 50 Doc. 20 1000 2.3.22.41 Gigantografia 1 Unidad 618 618 2.3.22.41 4000 1 Doc 4000 4000 1 Ponen. 5000 5000 5000 1000 618 2.3.27.2 99 Ponentes para el curso de genómica en Huancavelica 2 Espec 5000 10000 5000 5000 20618 CONSULTORIA 2.3.27.21 Evaluación de proy. Monitoreo y eval. final del proyecto 3 Jurado 3500 10500 10500 10500 OTROS SERVICIOS 2.3.25.12 Alquiler de movilidad 180 Dia 200 36000 36000 IMPREVISTOS 20000 APOYO A LA DUI GRAN TOTAL 999940 3600 3600 3600 3600 3600 3600 3600 3600 3600 3600 BIBLIOGRAFIA 1. Agapito, J., Rodríguez, J., Herrera-Velit, P., Timoteo, O., Rojas, P., Boettcher, P., Garcia, F., Espinoza, J. (2008) Parentage testing in alpacas (Vicugna pacos) by using semi-automated fluorescent multiplex PCRs with 10 microsatellite markers. Animal genetics, 39:201-203. 2. Banos, G., Wiggans, G. R., Powell. R. L. (2001) Impact of paternity errors in cow identification on genetic evaluations and international comparisons. J. Dairy Sci., 84:2523–2529. 3. Botstein D., White R.L., Skolnick M., Davis R.W. (1980) Construction of a genetic linkage map in man using restriction fragment length polymorphisms . American journal of human genetics, 32:314–31. 4. Butler, J.M. (2005) Forensic DNA typing. Biology, technology and genetics of STR markers. 2da. Ed. Elseiver Academic Press. USA: 455-539. 5. Cifuentes, L., Martinez, E., Acuña, M., Jonquera, H. (2006) Probability of Exclusion in Paternity Testing: Time to Reassess. J. Forensic Sci., 51(2):349-350. 6. Eileen A. (2011). Mejoramiento genético ovino, Facultad de veterinaria de la Universidad del Uruguay 7. Excoffier, L., Laval, G., Schneider, S. (2005) Arlequin ver. 3.0: An integrated software package for population genetics data analysis. Evolutionary Bioinformatics Online, 1:47-50. 8. FAO - Organización de las Naciones Unidas para la Agricultura y la Alimentación. (2005) Situación actual de los camélidos sudamericanos en Perú. Proyecto de cooperación técnica en apoyo a la crianza y aprovechamiento de los camélidos sudamericanos en la región andina (TCP/RLA/2914):1-62. 9. Freeland, J.R. (2005) Molecular Ecology. John Wiley y Sons Inc. USA: 110-111. 10. Garza, J.C., Williamson, E.G. (2001) Detection of reduction in population size using data from microsatellite loci. Molecular ecology, 10:305-318. 11. Guzman, K. (2011) Identificación de polimorfismos del gen tlr4 en crías de alpacas con cuadros de neumonías por Pasteurella multocida, UNMSM -Peru 12. Hancock, J. (1991) Microsatellites and other simple sequences: genomic context and mutational mechanisms. In: Microsatellites. Evolution and applications. Ed. Goldstein, D., Schlötterer, C. Oxford University Press. Oxford: 1-9. 13. Jamieson A., Taylor S.C. (1997) Comparisons of three probability formulae for parentage exclusion. Animal genetics, 28:397–400. 14. Kalinowski S, Taper M, Marshall T. 2007. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment. Molecular ecology 16: 1099-1106. 15. Lang K.D., Wang Y., Plante Y. (1996) Fifteen polymorphic dinucleotide microsatellites in llamas and alpacas. Animal genetics, 27:293. 16. Maynard, J. (1999) Evolutionary Genetics. 2º ed. Oxford University Press: 139-160. 17. Nei, M. (1973) Analysis of gene diversity in subdivided populations. Proceedings of the national academy of sciences USA, 70:3321--3323. 18. Nei, M., Kumar, S. (2000) Molecular Evolution and Phylogenetics. Oxford University Press. USA: 231-275. 19. Penedo M.C., Caetano A.R., Cordova K.I. (1998) Microsatellite markers for South American camelids. Animal genetics, 29:411–412. 20. Penedo M.C., Caetano A.R., Cordova K.I. (1999) Eight microsatellite markers for South American camelids. Animal genetics, 30:166–7. 21. Raymond, M., Rousset, F. (1995) GENEPOP (version 1.2): population genetics software for exact tests and ecumenicism. Journal of heredity, 86:248-249. 22. Rodríguez, J., Dodd, C., Rosadio, R., Wheeler, J., Bruford, M. (2006) Paternity testing using microsatellite DNA in alpacas (Vicugna pacos). In: South American Camelids Research - Volume I. Proceedings of the 4th European Symposium on South American Camelids and DECAMA European Seminar. Eds. Gerken M y Renieri C. Wageningen Academic Publishers. Netherlands: 273-278 pag. 23. Sambrook, J., Fritsch, E.F., Maniatis, T. (1989) Molecular Cloning – A Laboratory Manual, 2nd Edition. Cold Spring Habour Laboratory Press, New York. 24. Tapia, I., Varv, S., Bennewitz, J., Maleviciute, J., Fimland, E., Grislis, Z., Meuwissen., T.H.E., Miceikiene, I., Olsaker, I., Viinalass, H., Vilkki, J., Kantaneno, J. (2006) Prioritization for Conservation of Northern European Cattle Breeds Based on Analysis of Microsatellite Data. Conservation Biology, 20(6):1768-1779. 25. Templeton, A. (2006) Population Genetics and Microevolutionary Theory. John Wiley y Sons, New Jersey: 82-246 26. Weir B. & Cockerham C. 1984. Estimating F-statistics for the analysis of population structure. Evolution 38:1358-1370. 27. Weir, B. (1996) Genetic data análisis II. Methods for discrete population genetic data. Sinauer Associates Inc Publishers. Massachusetts: 150-156; 209-211. 28. Wheeler, J.C. (1995) Evolution and present situation of the South American Camelidae. Biol. J. Linn. Soc., 54:271-295. ANEXOS ANEXO 01: MARCO LÓGICO MEDIOS DE VERIFICACION SUPUESTOS FIN Elevar la producción y calidad de características de importancia económica en alpacas Huacaya Incremento de las características de importancia económica Adecuado y eficiente programa de mejoramiento genético asistido por marcadores genéticos. PROPÓSITO Identificar y desarrollar marcadores SNPs como herramientas de mejora genética en alpacas huacaya. Marcadores genéticos SNPs reportados. Existencia de marcadores genéticos SNPs polimorficos en el genoma de la alpaca. COMPONENTES - - Desarrollo e implementación de libros de registro y de pedigrí validados mediante ADN Identificación y desarrollo de marcadores genéticos SNPs para el mejoramiento genético de alpaca. Informe del estatus fenotípico y productivo de las alpacas del Centro Experimental de Lachocc -Huancavelica. Libro de pedigrí Cronograma de empadre de alpacas reproductoras Banco de ADN genómico. Banco de genotipos. Informe de la caracterización genética Libro de pedigrí con ADN Los investigadores se encuentran comprometidos con el proyecto. La universidad cumple con el normal funcionamiento administrativo Universidad ejecuta el normalmente el reglamento. Universidad cumple con ejecutar el presupuesto del proyecto en las fechas indicadas. - Aplicación de marcadores SNPs en la generación de herramientas para mejora genética en un plantel de reproductores. - Fortalecimiento de las capacidades de investigación en genómica de alpacas. ACCIONES 1. Generar un libro de registro fenotípico en alpacas reproductoras. Nivel de consanguinidad Niveles de heterosis Heredabilidad El plantel de reproductores de alpaca huacaya de la Universidad Nacional de Huancavelica no se comercializan. Número de cursos y seminarios a los productores de alpacas e interesados. Número de profesionales capacitados. Publicación en revistas indexadas. La Universidad Nacional de Huancavelica cumple con ejecutar el presupuesto del proyecto en las fechas indicadas. Evaluación fenotípica Los investigadores se encuentran comprometidos con el proyecto. 2. Generar libros de pedigrí en alpacas reproductoras. Libros de inscripción de alpacas de alta productividad 3. Generar bancos de ADN genómico de alpacas de alta productividad. Catálogo de ADN 4. Adecuar técnicas para identificación y desarrollo de marcadores SNPs en alpacas. Protocolos de genotipificación y análisis de SNPs. 5. Identificar familias de referencia y pedigrís validados en alpacas reproductoras. 6. Generar bancos de genotipos de alpacas de alta productividad. Identificación de pedigríes 7. Generar información sobre los niveles de consanguinidad y variabilidad genética en alpacas reproductoras Informes sobre consanguinidad y variabilidad genética 8. Generar información sobre parámetros de selección de características cuantitativas en alpacas. Informes sobre parámetros de selección 9. Mejorar la capacitación en genética y genómica molecular. Cursos de capacitación 10. Profesionales capacitados Promover la formación de profesionales e investigadores jóvenes. 11. Promover la difusión del conocimiento en el área de genética y genómica de alpacas. Catálogo de genotipos Artículo científico La universidad cumple con el normal funcionamiento administrativo Universidad ejecuta él normalmente la reglamentación del proyecto Universidad cumple con ejecutar el presupuesto del proyecto en las fechas indicadas ANEXO 02: MATRIZ DE CONSISTENCIA PROBLEMA OBJETIVO HIPOTESIS VARIABLES ¿Existen marcadores moleculares SNPs para características de importancia económica en la alpaca Huacaya? Generar un panel de marcadores genéticos moleculares SNPs de la alpaca En la alpaca Huacaya las características de importancia económica están determinados por marcadores moleculares SNPs Independiente -Marcadores moleculares SNPs INDICADORES METODOS Y TECNICAS Secuencia de ADN. METODO: -Experimental Número de repeticiones. TECNICA: -Análisis por microssatélite SNPs -Hibridación de ADN Dependiente Características de -Secuenciación importancia Características económica de importancia económica