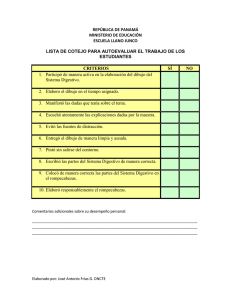
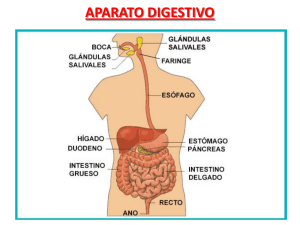
proyecto: riqueza de especies bacterianas y diversidad de
Anuncio

PROYECTO: RIQUEZA DE ESPECIES BACTERIANAS Y DIVERSIDAD DE GENES RELEVANTES EN EL TRACTO DIGESTIVO DE INSECTOS DESCORTEZADORES DEL GENERO DENDROCTONUS SIP 20070651 INFORME FINAL I. INTRODUCCION I.1. MICROORGANISMOS DEL INTESTINO En el tracto digestivo de los insectos se albergan una gran variedad de microorganismos; algunos de cuales habitan cavidades concretas, están rodeados por las células de tejido epitelial o se mantienen como simbiontes intracelulares (endosimbiontes). Los microorganismos del intestino están compuestos por una amplia variedad de especies, que incluyen bacterias, arqueas y eucariotes. En particular, la región del proctodeo es la más densamente poblada y con mayor diversidad de especies de microorganismos asociados (Anklin-Mϋhlemann et al., 1995; Bignell et al., 1980; Cazemier et al., 1997; Cruden y Markovetz, 1987; Werner, 1926; Wiedenmann, 1930). El proctodeo de termitas, grillos y cucarachas; que sobreviven a base de dietas de difícil asimilación y limitadas en nutrientes, tienen poblaciones de bacterias firmemente adheridas al integumento (Breznak y Pankrast, 1977; Cruden y Markovetz, 1979; Fogelsong et al., 1975; Ulrich et al., 1981). Sin embargo, otros microorganismos del intestino de insectos, como los protozoarios y las espiroquetas, establecen grandes y persistentes poblaciones sin estar adheridos a las superficies del canal alimentario (Breznak, 2000). La dieta influye claramente en la densidad y proporción de los diferentes microorganismos que podemos encontrar en el canal alimentario de mamíferos y artrópodos (Hungate, 1966; Kauffman et al., 2000). Mientras que algunas bacterias de los tractos digestivos son capaces de utilizar una amplia variedad de nutrientes, otras sólo sobreviven empleando nutrientes específicos. El crecimiento y supervivencia de diferentes especies microbianas está en función de las condiciones ambientales que se pueden encontrar en el tracto digestivo de los huéspedes. Estas condiciones pueden variar temporal y espacialmente dentro del mismo individuo o entre diferentes individuos (Nardi et al., 2002). La naturaleza facultativa u obligatoria de las interacciones insectomicroorganismo es dinámica y compleja. La interrelación entre las funciones microbianas y la fisiología de los insectos es una área aún pobremente estudiada de la biología de los insectos. De los microorganismos del intestino de los insectos, sólo los de las termitas se han estudiado ampliamente y la interdependencia obligada de esta asociación se ha reconocido (Breznak, 1982). Otros insectos en los cuales también se ha estudiado con cierta profundidad a los microorganismos del intestino son las cucarachas y los áfidos (Cruden y Markovetz, 1979; Douglas, 1998). Así por ejemplo, el tiempo de vida de las termitas superiores se puede reducir de 250 a 13 días si son eliminadas las bacterias y espiroquetas de su intestino empleando algunos antibióticos (Eutick et al., 1978). Aunque las células del mesenterón de las termitas superiores son capaces de digerir la lignocelulosa sin necesitar la cooperación directa de los microorganismos de su intestino se ha propuesto que la producción de estas celulasas endógenas no es suficiente para abastecer a las termitas de la energía que ellas necesitan para su desarrollo y reproducción. (Bignel, 2000; Tokuda y Watanabe, 2007) Las células de ningún insecto son capaces de fijar nitrógeno ni de convertir el dióxido de carbono en acetato; todos estos procesos anaeróbicos son exclusivos de los microorganismos del intestino. En particular, la acetogénesis microbiana provee, al menos, la tercera parte de la energía requerida por las termitas (Breznak, 2000). Asimismo, la mayoría de las especies de cucarachas se alimentan de cualquier material orgánico disponible, incluso de los residuos celulósicos de las plantas, los cuales son pobres en nitrógeno. Si las poblaciones microbianas asociadas al tracto alimentario de las cucarachas se eliminan, se observa un efecto negativo sobre el peso del adulto, el volumen del proctodeo, la producción de metano y el tiempo de generación (Gijzen y Barugahare, 1992). Finalmente, las bacterias del intestino de los áfidos mantienen un sutil balance entre ellas y el insecto huésped. Erwinia aphidicola puede mostrarse como un patógeno en su relación con los insectos, sin embargo generalmente mantiene un equilibrio con otras poblaciones de enterobacterias que suprimen su crecimiento excesivo. Si esto sucede, se inhibe el cambio del exoesqueleto (ecdisis) y causa una mayor mortalidad en los insectos adultos (Harada y Ishikawa, 1997). I.2. ESCARABAJOS DESCORTEZADORES Los escarabajos descortezadores se encuentran en la subfamilia Scolytinae, éstos típicamente se alimentan de sustratos pobres. Ellos colonizan y se alimentan de una gran variedad de tejidos de las plantas que incluyen los tejidos de la madera (corteza y floema), frutos y la médula de las ramas. Solamente los escarabajos de la ambrosia no se alimentan de los tejidos de las plantas, sino de sus hongos asociados. Los escolítidos se pueden agrupar en tres categorías con base en sus estrategias de alimentación: saprófagos, los cuales se alimentan exclusivamente de tejidos de árboles muertos o decaídos; fitófagos, que se alimentan de de tejidos de plantas vivas o recientemente muertas y los micetófagos, que viven en la madera ó en otros tejidos de las plantas donde cultivan hongos, de los que se alimentan (Six, 2003). 1.3 SIMBIOSIS EN Dendroctonus Los escarabajos descortezadores comprenden un grupo económicamente y ecológicamente importante que habita en los tejidos subcorticales de los árboles, especialmente en el floema y la corteza externa (Wood, 1982). Algunas especies de descortezadores se alimentan de árboles enfermos ó muertos y juegan un papel ecológico benéfico, debido a que participan en la descomposición y el reciclaje de nutrientes, además de proveer de alimento a algunas aves y otros animales silvestres (Schowalter, 1981; Raffa et al., 1993), como es el caso de Dendroctonus valens. Sin embargo, otras especies de escarabajos descortezadores son importantes plagas forestales que se pueden extender a millones de hectáreas contiguas de bosque y que causan pérdidas a gran escala (Coulson, 1979; Wallin y Raffa, 2004). Dada la pobre composición de nutrientes que tienen la albura (parte más externa del xilema secundario) y el floema de los cuales se alimentan los escarabajos del género Dendroctonus, parece probable que los microorganismos asociados provean de algunos nutrientes que suplementan su dieta o les facilitan su asimilación. La mayoría del conocimiento de los microorganismos asociados al género Dendroctonus involucra a los hongos, particularmente a aquellos que son transportados externamente sobre el exoesqueleto o en estructuras especializadas conocidas como micangios (Hsiau y Harrington, 2003; Krokene y Solhein, 1998; Lim et al., 2004, 2005; Paine et al., 1997). El micangio es una invaginación del integumento que posee glándulas o células secretoras que alberga hongos simbióticos, entre los que se encuentran Entomocorticium spp., Ceratocystiopsis ranaculosus, etc. (Batra, 1963; Levieux et al., 1991; Six, 2003). Se ha sugerido que los hongos micangiales proveen de compuestos nitrogenados, vitaminas, esteroles y otros factores de crecimiento a escarabajos del género Hypothenemus (Beaver, 1986). Algunos hongos micangiales de D. frontalis pertenecientes al género Entomocorticium concentran y transportan compuestos nitrogenados de la savia (proteínas y aminoácidos) a la cámara de alimentación de las larvas del insecto, lo cual favorece el crecimiento de las poblaciones de los insectos (Ayres, 2000). Las poblaciones de D. frontalis que se desarrollan en presencia de Entomocorticium poseen un alto contenido lipídico, lo que disminuye el tiempo de desarrollo mínimo de las larvas de ~40 a ~18 días (Barras y Hodges, 1973; Wagner et al., 1984). Los hongos micangiales parecen beneficiarse de la simbiosis al recibir nutrientes producidos por las glándulas asociadas al micangio, al ser transportados e inoculados a otros árboles durante los periodos de dispersión y colonización del insecto (Paine y Birch, 1983) y posiblemente también reciben una protección física contra la luz UV y la desecación (Six, 2003). Otros hongos como Pichia pinus y Kuraishia capsulata asociados a D. ponderosae convierten el trans-verbenol, una feromona de agregación, a verbenona, una feromona de antiagregación. Esta conversión posiblemente juega un papel muy importante en la terminación del ataque masivo al árbol por este insecto (Hunt y Borden, 1990). En D. terebrans, D. frontalis e Ips avulsus se han aislado constantemente cepas de Pantoea agglomeras (anteriormente clasificada como Enterobacter agglomerans) y Enterobacter spp. las cuales son bacterias fijadoras de nitrógeno potenciales y posiblemente suplementan la dieta de estos insectos con compuestos nitrogenados (Bridges, 1981). Por otra parte, Penicillium chrysogenum que tiene la capacidad de degradar la carboximetil-celulosa se aisló en una ocasión del tracto digestivo de D. frontalis, aunque no se lograron aislar bacterias celulolíticas, por lo que se mantuvo la duda acerca del impacto que tiene la microbiota asociada al intestino de este insecto sobre la degradación de la celulosa (Delalibera et al., 2005). A pesar de la gran importancia económica y ecológica de los escarabajos en general y en particular del género Dendroctonus, se conoce poco sobre la diversidad de la microbiota de su intestino. II. DIAGRAMA GENERAL DE TRABAJO III. MATERIAL Y METODOS III.1. COLECTA DE INSECTOS Los insectos se colectaron en bosques de pinos de diferentes localidades de la República Mexicana, se extrajeron de árboles infestados y se colocaron en recipientes de plástico, con un papel húmedo, debidamente etiquetados indicando fecha y localidad. Los insectos se mantuvieron a 4ºC y se transportaron al laboratorio donde se procedió a su disección. En el cuadro 1 se muestran los lugares donde se realizaron las colectas de insectos. Todos los ejemplares se mantuvieron a 4ºC y se transportaron al laboratorio. Los insectos se disectaron para obtener su tracto digestivo, el cual se seccionó en mesenterón anterior (MP), mesenterón posterior (MP) y proctodeo (P), estas partes se colocaron en tubos de 1.5 ml que contenían 200 μl de medio de cultivo líquido cuando la muestra se procesó para el cultivo de microorganismos ó 200 μl de agua para la muestras a las cuales se les extrajo DNA metagenómico Cuadro 1. Escarabajos descortezadores colectados en este estudio. Coordenadas Fecha de Especie Estado Localidad geográficas colecta San Juanito Chihuahua Casas Grandes Dendroctonus valens Jalisco Los Pozos, Gómez Farias Distrito Federal CICS, Milpa Alta 27o55’22’’ N 107º55’47’’ O 30º09’76’’ N 108 º 57’03’’ O 19º88’20’’ N 103º40’40’’ O 19º 04’ N 98º 58’ O 25 de Octubre 2005 28 de Octubre de 2005 25 de Junio de 2006 25 de Marzo de 2006 III.2 DISECCIÓN Y EXTRACCIÓN DEL TUBO DIGESTIVO DE LOS INSECTOS Los escarabajos se limpiaron superficialmente con etanol al 96%, se expusieron durante 10 minutos a luz UV y se lavaron en una solución reguladora de fosfatos (50 mM, pH 7.2), en esta solución se sacrificaron por ahogamiento. Una vez sacrificado el insecto, se colocó en una caja Petri con regulador de fosfatos y se inició la disección quitando los élitros y las alas, posteriormente se cortó la parte abdominal y se separó el cuerpo graso del tubo digestivo. Una vez obtenido el tubo digestivo se pasó a otra caja Petri con PBS, se separó en las distintas secciones: mesenterón anterior, mesenterón posterior y proctodeo y cada sección se colocó en tubos Eppendorf estériles con 100 μL de PBS 120 mM pH 8. Se almacenaron a -20ºC hasta su posterior uso. III.3 ANÁLISIS BASADOS EN MÉTODOS MOLECULARES III.3.1 EXTRACCIÓN DEL DNA TOTAL Se descongelaron los tubos conteniendo las partes del tubo digestivo y se agregaron 200 μL de DNAzol® (Invitrogen, CA) y 100 mg de perlas de vidrio de 0.5 mm previamente esterilizadas. Se agitaron con Vortex hasta el rompimiento total del tejido y se centrifugaron 10 min a 14,000 rpm. Se hicieron extracciones del sobrenadante con 200 μL de fenol-cloroformo 25:25, el DNA de la fase acuosa se precipitó con un volumen de isopropanol e incubación a -20ºC por 30 min. Finalmente, el DNA se lavó con 0.5 mL de etanol 70% y se resuspendió en 50 μL de agua desionizada estéril y se almacenó a -20ºC hasta su uso. III.4 CUANTIFICACIÓN Y VISUALIZACIÓN DEL DNA Para cuantificar y determinar la pureza el DNA, se empleó un espectrofotómetro de UV y se tomaron lecturas de absorbencia a 260 y 280 nm del DNA en la muestra (Sambrook et al., 2002). También se visualizó el DNA extraído por electroforesis en gel de agarosa al 0.7% en amortiguador TAE 1X. III.5 CONSTRUCCIÓN DE LA LIBRERÍA DE GENES RIBOSOMALES III.5.1 AMPLIFICACIÓN DE LOS GENES 16S rRNA Se amplificó un fragmento de aproximadamente 1,500 pb del gen 16S rRNA con un protocolo e iniciadores descritos previamente en la literatura (Relman, 1993). Los iniciadores utilizados fueron: Derecho 8 (5’GCG GAT CCG CGG CCG CTG CAG AGT TTG ATC CTG GCT CAG3’) y Reverso 1492 (5’GGC TCG AGC GGC CGC CCG GGT TAC CTT GTT ACG ACT T3’) III.5.2 CLONACIÓN DEL PRODUCTO DE PCR Se purificaron los productos de PCR para la clonación con el kit de purificación “PCR Purification Kit” (Qiagen, Valencia, CA). El amplificado purificado se clonó en el vector pCR2.1-TOPO de 3.9 Kb del kit TOPO TA Cloning (Invitrogen, Carlsbad, CA) siguiendo el protocolo del fabricante. La tranformación se realizó en células quimiocompetentes de la cepa DH10b de Escherichia coli. El crecimiento de las clonas se efectuó en medio LB con ampicilina (50 μg/mL), X-gal (40 μg/ mL) e IPTG (0.5 mM). Las clonas con los plásmidos recombinantes se reconocieron por la coloración blanca de las colonias y por su resistencia a la ampicilina. III.5.3 CULTIVO DE LAS CLONAS SELECCIONADAS Y OBTENCIÓN DEL DNA PLASMÍDICO Las clonas seleccionadas se picaron con palillos de madera estériles y se sembraron en tubos con tapón de rosca con 5 mL de medio LB con ampicilina (50 μg/mL) durante 24 h a 37°C. El DNA plasmídico se obtuvo por medio de la técnica de lisis alcalina (Sambrook et al., 2002). III.5.4 BÚSQUEDA DE CLONAS POSITIVAS Se identificaron las clonas que contenían el inserto por liberación del mismo con la endonucleasa de restricción EcoRI (Invitrogen, Carlsbad, CA) cuyas secuencias de corte flanquean el sitio de inserción en el plásmido. III.6 SECUENCIACIÓN Para priorizar los esfuerzos de secuenciación se realizó un análisis RFLP con la enzima HpaII y se secuenció una clona de cada perfil representativo. La secuenciación de los plásmidos purificados se realizó de manera automatizada con un secuenciador ABI PRISM 310 Genetic Analyzer PE (Applied Biosystems). Se realizaron reacciones de secuenciación por clona representativa a partir del extremo 5’ y 3’ con los iniciadores M13-F (5’-GTAAAACGACGGCCAG-3’) y M13-R (5’-CAGGAAACAGCTATGAC-3’). III.7 ANÁLISIS DE LAS SECUENCIAS OBTENIDAS Las secuencias obtenidas de los diferentes microorganismos fueron analizadas con el programa CHIMERA-CHECK del Ribosomal Database Project (http://rdp8.cme.msu.edu/htm/analyses.html) para descartar las posibles secuencias quiméricas. Posteriormente las secuencias obtenidas se sometieron a una búsqueda de secuencias similares en GenBank por medio del programa BLASTN versión 2.2.3 (Altschul et al., 1997) en la página de la NCBI (http://www.ncbi.nlm.nih.gov/). Una vez determinada la identidad, las secuencias relacionadas se seleccionaron considerando los niveles jerárquicos y linaje taxonómico descrito en la base de datos TaxBrowser del NCBI (http://www.ncbi.nlm.nih.gov/sites/entrez?db=taxonomy). Para obtener el mejor alineamiento, múltiples alineamientos de las secuencias se hicieron con el programa CLUSTAL X versión 2.0 (Thompson, 1997); los parámetros usados fueron los de lento/seguro, 15.5 para apertura de gaps, 6.6 para extensión de los mismos, eliminación de secuencias divergentes en 30% o más, peso de 0.5 para las transiciones y la matriz IUB para peso del DNA, no se utilizó matriz negativa. Los alineamientos se terminaron de editar manualmente con el programa “Seaview” (Galtier et al., 1996). Las relaciones filogenéticas entre las secuencias se establecieron por métodos de distancia. El modelo de sustitución nucleotídica para estimar las distancias evolutivas (ED) se eligió tomando en cuenta las frecuencias nucleotídicas determinadas con el programa DAMBE (Xia y Xie, 2001), que por medio de una tabla de contingencia de Χ2 indica si las proporciones nucleotídicas corresponden a lo esperado y si esto es significativo estadísticamente; el índice de transiciones y transversiones se calculó con el programa MEGA versión 2.1. Para obtener los árboles filogenéticos se empleó el paquete de programas computacionales MEGA versión 4.0, se empleó el método de agrupamiento de Neighbor Joining y se realizó un análisis de remuestreo (“bootstrap”) con 1000 aleatorizaciones. III.8 ANÁLISIS DE RAREFACCIÓN E ÍNDICES DE DIVERSIDAD Para efectuar los analisis de rarefacción y calcular el estimador de la riqueza Chao 1, las secuencias de las clonas obtenidas a partir de la librería de genes ribosomales del tracto digestivo de 3-5 hembras de D. valens se alineron, con el programa Clustal X 2.0 (Thompson et al., 1997). Una matriz de distancia fue calculada empleando la paquetería PHYLIP 3.67 apartir del modelo de sustitucion de Kimura-2-Parámetros (Felsestein, 1993). La determinacion de la riqueza y unidades taxonómicas operativas se llevo a cabo con en el programa DOTUR. Las curvas de rarefación se construyeron a niveles de distancia genética de 20, 10, 5, 3 y 1%, las cuales corresponden a los niveles taxonómicos de phylum, clase, género, especie y cepa respectivamente (Scholoss y Handelsman, 2005). En nuestro caso, los análisis de rarefacción fueron útiles para estimar el grado en que el muestreo cubrió la diversidad presente en el intestino de D. valens a partir de la librería de genes. Las curvas de rarefacción son construidas mediante el trazado del número total de OTUs encontradas versus el número total de secuencias analizadas. Generalmente, la inclinación de una curva de rarefacción es alta al inicio y gradualmente disminuye, provocando que la curva alcance un punto máximo y se vuelva asintótica; en este punto, el muestreo se considera adecuado. En este trabajo, solo se presentan las curvas de rarefacción para las OTUs con una distancia genética no mayor de 20, 5 y 3%, la cuales indicativas de las diferencias nivel de phylum, género y especie respectivamente. A pesar de que los análisis de rarefacción son útiles para estimar si el muestreo fue adecuado, éstos no son adecuados para estimar la riqueza de una comunidad. Por lo tanto el estimador de la riqueza Chao 1 fue también calculado con la paquetería DOTUR (Scholoss y Handelsman, 2005). Chao 1 es en estimador no paramétrico que determina la diversidad de una comunidad basándose en el numero de “singletons” (OTUs representadas por sólo una secuencia) y “doubletons” (OTUs representadas por dos secuencias) encontrados en una muestra (Bohannan y Hughes, 2003). La riqueza terminal de Chao 1 se reporta para secuencias con diferencias no mayores al 3% entre ellas. El estimador de la riqueza terminal Chao 1 se calcula empleando la siguiente relación: SChao1 = Sobs +[n2(n1-1)/2(n2+1) ], cuando n1>0 y n2≥0 y cuando n1=0 y n2=0 SChao1 = Sobs + (n1²/ 2n2), cuando n1=0 y n2≥0 Donde, S Chao1 = Riqueza estimada Sobs= Número de especies observadas n1 = Número de OTUs con sólo una secuencia n2 = Número de OTUs con sólo dos secuencias III.9 ANÁLISIS MEDIANTE DGGE DE LA REGIÓN V3 DEL GEN 16S rRNA III.9.1 AMPLIFICACIÓN DE LA REGIÓN V3 DEL GEN 16S rRNA Para amplificar la región variable 3 del gen 16S rRNA se usaron los iniciadores Derecho 2 (5’GCG CCG CCG CGC GCG GGC GGG GCG GGG GCA CGG GGG GCC TAC GGG AGG CAG CAG3’) (en negritas se destaca el broche de GC) y Reverso 3 (5’ATT ACC GCG GCT GCT GG3') (Muyzer et al., 1993). La mezcla de reacción fue la siguiente: Reactivo DNA Regulador Oligonucleótidos DNTPs Taq polimerasa MgCl2 Agua Concentración final 4 ng 1X 0.4 pM 0.2 mM 1 unidad 3 mM Hasta completar 25 μL Las condiciones para la PCR fueron las siguientes: Ciclos Desnaturalización inicial Desnaturalización Hibridación Polimerización Extensión final Temperatura Tiempo (°C) (seg) 92 300 92 45 55 30 72 45 72 420 No. de ciclos 1 35 1 III.9.2 ELECTROFORESIS EN GEL CON GRADIENTE DESNATURALIZANTE (DGGE) Para la DGGE se prepararon las siguientes soluciones madre: Capacidad relativa de desnaturalización Reactivo Acrilamida-Bis acrilamida (37.5:1) 40% TAE 50X Glicerol Urea Formamida Solución A 100 % 50 mL 2.5 mL 5.0 mL 105.4 g 100 mL Solución B 0% 50 mL 2.5 mL 5.0 mL 0 0 Agua desionizada hasta completar 250 mL Las soluciones se guardaron en frascos ámbar a 4°C. La solución A se diluyó con la solución B para obtener los porcentajes de desnaturalización relativa del 60% y 30%. Se prepararon dos tubos con 12 mL de las soluciones de alto y bajo porcentaje, se agregaron 13 μL de TEMED y 40 μL de persulfato de amonio al 10% (APS). El gradiente de desnaturalización se formó con un gradientador de vasos comunicantes al tiempo que se introdujo la mezcla para llenar el espacio entre los vidrios que contienen el gel, a los que previamente se les colocó un cuadro de “gel bond”. Finalmente se preparó un tubo con 7 mL de solución B, 7 μL de TEMED y 20 μL de APS al 10% con el cual se formó el gel de carga. Se dejó polimerizar la poliacrilamida durante hora y media. La cámara de electroforesis se llenó con amortiguador TAE 1X, se montó el gel y se calentó a 60°C. Se colocaron las muestras en el gel (aproximadamente 20 μL con 4 μL del amortiguador de carga). Se corrió el gel a 200 V durante 5 min para introducir las muestras al gel de carga. Para separar las muestras, el gel se corrió a 85 V durante 16 h. III.9.3 TINCIÓN Y OBSERVACIÓN DE PERFILES La tinción con sales de plata se realizó en cuatro etapas: 1. Fijación. El gel se colocó en 200 mL de solución de fijación y se mantuvo en agitación durante 3 min a 26 rpm. 2. Tinción. Posteriormente, el gel fijado se colocó en 200 mL de solución de tinción y se mantuvo en agitación durante 10 min a 26 rpm. Después se lavó con agua destilada durante 2 min. 3. Revelado. El gel se colocó en otra charola, que previamente se lavó con aproximadamente 10 mL de la solución de revelado para eliminar las sales contaminantes que pudieran precipitar la plata. Los geles se mantuvieron en 200 mL de la solución de revelado y en agitación hasta la aparición de las bandas. La reacción de revelado se detuvo añadiendo con 200 mL de solución de fijación durante 5 min. Después se lavó con 200 mL de agua destilada durante 5 min. 4. Preservación: En seguida el gel se colocó en 200 mL de solución de preservación en agitación durante 10 min. Finalmente, el gel se colocó en un transiluminador de luz blanca y se digitalizó en un Eagle Eye o con ayuda de una cámara forográfica. El análisis de los perfiles obtenidos se realizó manualmente. El análisis de DGGE se llevó a cabo para comparar la fracción bacteriana cultivable con los amplificados de genes ribosomales provenientes del DNA metagenómico, que teóricamente se aproxima a la comunidad bacteriana total. El DNA metagenómico se aisló de las diferentes partes del tracto digestivo de 3-5 individuos (hembras y machos) de D. valens colectados en el campo. Las cepas aisladas a partir de los tractos digestivos se crecieron en medio LB durante toda la noche y posteriormente se les extrajo el DNA. Los productos de PCR se obtuvieron a partir de la amplificación de la región 3 variable del gen 16S rRNA y se separaron por electroforesis en gel con gradiente desnaturalizante. La población mínima de una especie detectable por PCR-DGGE usualmente debe constituir al menos el 1% de la total de poblaciones que componen la comunidad en estudio. Asimismo, las especies con poblaciones más abundantes forman las bandas más intensas (Muyzer y Smalla, 1998). III.9.4 DETECCIÓN POR PCR DE LOS GENES REPRESENTATIVOS DEL CICLO DEL NITRÓGENO. Los genes que codifican para la amonio monooxigenasa (amoA), nitrito reductasa dependiente de cobre (nirK) y citocromo cd1 (nirS), óxido nítrico reductasa (nosZ) y 16S rRNA del género Nitrobacter se amplificaron por procedimientos previamente descritos, cuyos oligonucleótidos iniciadores se describen en el Cuadro 1. Debido a que existen varias familias del gen que codifica para la dinitrogenasa (nifH) y éstas mantienen una conservación a nivel de nucleótidos relativamente baja, fue necesario diseñar 4 juegos de iniciadores para PCRs específicas que nos permitieran abarcar la mayoría de los miembros de cada una de las familias de este gen. En la medida de lo posible se evitó incluir nucleótidos degenerados para mantener una especificidad relativamente alta. Los oligonucleótidos se diseñaron a partir de alineamientos múltiples de las secuencias completas de los genes nifH disponibles en el banco de genes (GeneBank). Cada par de iniciadores amplifica uno de los cuatro grupos o familias de genes nifH reconocidos en el alineamiento (Cuadro 2). Cuadro 2. Oligonucleótidos utilizados para amplificar los genes importantes en el ciclo del nitrógeno. Gen Nombre del iniciador Secuencia (5’ →3’) Posición amoA1F GGGGTTTCTACTGGTGGT N 332-N349 amoA2R FGPS1269 CCCCTCKGSAAAGCCTTCTTC TTTTTTGAGATTTGCTAG N802-N822 FGPS872 nirK1 CTAAAACTCAAAGGAATTGA GGMATGGTKCCSTGGCA N526-N542 nirK5 nirS1 GCCTCGATCAGRARCCAGGTTTCC CCTAYTGGCCGCCRCART N1023-N1040 N763-N780 nirS6 CAMPNos661 CGTTGAACTTRCCGGT CTACTACTACTACGGCTGGGGGCTG ACCAA N1638-N1653 CAMPNos1773 nifHEnter-F CATCATCATCATATRTCGATCARCTG BTCG TT ACACCATTATGGAGATGG N1773-N1803 nifHEnter-R CTGATGGAGTTCGGCATC N805-N822 nifHAzo-F GACTCCACCCGCCTGATCCT N130-N152 nifHAzo-R ATGACCGTGATCGAATACGATC N676-N697 nifHClo-F GCTATTTAYGGAAARGTTG N13-N26 nifHClo-R ATGATGGCAHTRTATGC N454-N470 AmoA 16S Rrna Nitrobacte r NirK NirS Organismo (número de acceso del gen en GenBank) Referencia Nitrosomonas europea Rotthauwe et al., 1997 Nitrobacter Degrande y Bardin, 1995 Alcaligenes faecalis Braker et al., 1998 Pseudomonas stutzeri Braker et al., 1998 Pseudomonas stutzeri Scala et al., 1999 N661-N691 NosZ N167-N184 Klebsiella pneumonie (43874) Azotobacter vinelandii (M11579) Este trabajo NifH nifHRFZ-F TYGGCAAGTCCACCACC N41-N57 nifHRFZ-R nifD-F TCCGGYGAGATGATGGCGC CGCGGCTGCGCCTAYGCMGG N454-N472 nifD-R GARTGCATCTGRCGGAAMGG Clostridium pasteurianum (AY603957) Azospirillum brasilense (M64344) Este trabajo NifD La mezcla de reacción para las amplificaciones fue la siguiente. Reactivo Concentración final DNA Regulador Oligonucleótidos 10-100 ng 1X 1 pM dNTPs Polimerasa MgCl2 Agua 0.2 mM 1 unidad 3 mM Hasta completar 25 μL Nota: Las cantidades de los reactivos pudieron variar dependiendo del protocolo descrito para cada gen en particular. Las condiciones de la PCR para los genes nifH fueron las siguientes: Ciclos Desnaturalización inicial Desnaturalización Hibridación Polimerización Extensión final Temperatura Tiempo (°C) seg 94 600 94 60 TMºC 60 72 60 72 600 No. de ciclos 1 35 1 Las temperaturas, los tiempos y el número de ciclos pueden variar dependiendo del protocolo descrito para la amplificación de cada gen. III.10 MÉTODOS BASADOS EN EL CULTIVO III.10.1 CULTIVO Y AISLAMIENTO DE BACTERIAS HETERÓTROFAS De 5 a 10 partes del tubo digestivo de D. valens se colocaron en tubos con PBS y se maceraron suavemente con perlas de vidrio. Posteriormente, se hicieron diluciones decimales hasta 10-5 en regulador de fosfatos y se sembraron y extendieron por duplicado 100 μL de cada dilución en cajas de Petri con medio de gelosa nutritiva. Las cajas se incubaron a 28ºC durante 72 h. Los microorganismos heterótrofos aerobios, cultivables y viables se reportaron en UFC/tracto digestivo. III.10.2 OBTENCIÓN DE DNA POR EL MÉTODO DE CULLEN Y HIRSCH Se cosechó la biomasa de un cultivo de 72 h, el DNA genómico de cada cepa se obtuvo con una técnica previamente descrita (Cullen y Hirsch, 1988). III.10.3 ANÁLISIS RAPD COMO PRUEBA TAMIZ PARA SECUENCIACIÓN Se desarrollaron PCR-RAPD’s (amplificación aleatoria del DNA polimórfico) para distinguir entre los diferentes aislados de las cepas de bacterias aisladas de los insectos adultos colectados en el estado de Chihuahua y para dar prioridad a los esfuerzos de identificación basada en el análisis de la secuencia del gen 16S rRNA. De este modo se partió del supuesto que las cepas que desplegaron perfiles de RAPD iguales pertenecen a la misma especie, mientras que perfiles diferentes pertenecen es altamente improbable que pertenezcan a la misma especie. Se empleó el iniciador OPB-01 (Williams et al., 1990) y la siguiente mezcla de reacción. Reactivo DNA Regulador Iniciador dNTPs Polimerasa MgCl2 Agua Concentración final 5 ng 1X 0.4 pM 0.2 mM 1U 3 mM Hasta los 25 μL Las condiciones generales para efectuar el RAPD fueron las siguientes: 1) Un ciclo de desnaturalización inicial a 94oC durante 5 minutos. 2) 40 ciclos con las siguientes condiciones: i) Desnaturalización a 94oC durante 1 minuto. ii) Hibridación a 38oC durante 1 minuto. iii) Polimerización a 72oC durante 1 minuto. 3) Un ciclo final de i) Polimerización a 72oC durante 1 minuto. III.10.4 IDENTIFICACIÓN DE LAS CEPAS AISLADAS. Se realizaron infiriendo las relaciones filogenéticas utilizando la secuencia del gen 16S rRNA. Se consideraron los criterios de Roselló-Mora que definen los límites de especie (similitud >97%) y género (similitud entre 95% y <97%) respectivamente (Rosello-Mora y Amann, 2002). III.10.5 ENSAYO DE LA ACTIVIDAD CELULOLÍTICA La capacidad de degradar celulosa fue probada en los microorganismos aislados pertenecientes al phylum Actinobacteria. Se emplearon cajas de agar Rojo Congo celulosa y la degradación de la celulosa se observó como un halo amarillo o anaranjado alrededor de la colonia. La actividad de la enzima se definió como el diámetro del halo claro dividido entre el diámetro de la colonia. Dos mediciones de cada colonia fueron tomadas y al menos se les efectuó la medición a dos colonias de cada microorganismo probado. III.10.6 REDUCCION DE ACETILENO Para estimar la fijación de nitrógeno, se llevaron a cabo ensayos de reducción del acetileno. El acetileno fue producido mediante la disolución de carburo de calcio en agua empleando una trampa de agua para capturar el acetileno formado en la reacción. El acetileno fue inyectado en viales (10 mL) a una concentración del 1% por reemplazo de un volumen idéntico de aire. El ensayo se realizó en frascos viales que contenían un individuo (larva o adulto), insectos esterilizados por calor húmedo fueron utilizados como testigos. Los insectos se mantuvieron en este sistema cerrado durante de 68 h a 25°C. La reducción de acetileno se midió por cromatografía de gases utilizando un detector de ionización de flama (Varian 3300 gas chromatograph). Se realizó una ANOVA de un factor para comprobar estadísticamente si existían diferencias significativas entre todos los tratamiento formados y posteriormente si hubo diferencias significativas se realizó la prueba secuencial de Tukey HSD con el programa estadístico OpenStat4 ver 7 (Miller, 2005). IV. RESULTADOS. (NO SE INCORPORARON LAS IMÁGENES POR MOTIVOS DE ESPACIO) IV.1 ANÁLISIS DE LA DIVERSIDAD BACTERIANA ASOCIADA AL TRACTO DIGESTIVO DE Dendroctonus valens IV.1.1 DIVERSIDAD DETECTADA POR MÉTODOS BASADOS EN EL CULTIVO IV.1.1.1 CULTIVO DE MICROORGANISMOS A PARTIR DE LAS DIFERENTES REGIONES DEL TRACTO DIGESTIVO DE Dendroctonus valens La población total de bacterias heterótrofas aerobias cultivables en medio gelosa nutritiva del tracto digestivo de D. valens fue de 2.11 x 106 UFC/intestino aproximadamente. El proctodeo es la región que posee el mayor porcentaje (63%) de la población total de bacterias heterótrofas aerobias, seguido del mesenterón anterior y posterior (Cuadro 3.). Cuadro 3. Número de bacterias por sección de tracto digestivo. Población de Bacterias heterótrofas aerobias (106 UFC/sección)a Especie Mesenterón Mesenterón Proctodeo anterior posterior Dendroctonus valens a 0.41 ± 0.0621 0.34 ± 0.056 1.35 ± 0.067 Los valores son la media de dos cuentas viables en medio gelosa nutritiva ± D. E. Se aislaron 98 cepas provenientes de las diferentes secciones del tracto digestivo de individuos de D. valens de las diferentes localidades geográficas. La mayoría de las cepas fueron bacilos Gram negativos anaerobios facultativos, también se aislaron algunos microorganismos Gram positivos. Tal es el caso de las cepas MA1B, MA3A, MA2, PL2 y PL5 cuya identificación basada en el gen 16S rRNA arrojó la presencia de las especies Janibacter melonis, Cellulosimicrobium celulans, Leifsonia sp., Cellulomonas xylanilytica y Cellulosimicrobium celulans. IV.1.1.2 Análisis RAPD La PCR-RAPD con el oligonucleótido OPB-01 produjo 4 patrones de bandas diferentes en las 22 cepas de bacterias aisladas del tracto digestivo de insectos adultos de D. valens obtenidos en la localidad de San Juanito Chihuahua (Fig. 4). La cepa 1.10E resultó ser una levadura por lo que su patrón desplegado no fue considerado. Figura 4. Patrones obtenidos por la técnica de RAPD con el oligonucleótido OPB-01 del DNA genómico de las cepas aisladas del tracto digestivo de insectos adultos de Dendroctonus valens, de la localidad de San Juanito Chihuahua (M. Marcador de talla molecular). Este mismo oligonucleótido produjo 8 patrones diferentes en las 17 cepas de bacterias aisladas del tracto digestivo de larvas de D. valens de la localidad del CICS, Milpa Alta, Distrito Federal (Fig. 5). Figura 5. Patrones obtenidos por la técnica de RAPD con el oligonucleótido OPB-01 del DNA genómico de las cepas aisladas del tracto digestivo de insectos adultos de Dendroctonus valens, del CICS, Milpa Alta, Distrito Federal (M. Marcador de talla molecular y TN Testigo negativo) Tomando como molde el DNA de las 33 cepas de la localidad de El Wicle, Casas Grandes, Chihuahua el oligo desplegó 4 patrones diferentes (Fig. 6). La cepa P6W no fue considerada para secuenciación por tratarse de un posible contaminante. Figura 6. Patrones obtenidos por la técnica de RAPD con el oligonucleótido OPB-01 del DNA genómico de las cepas aisladas del tracto digestivo de insectos adultos de Dendroctonus valens, de El Wicle, Casas Grandes, Chihuahua (M. Marcador de talla molecular) Para los insectos provenientes de la localidad de Los Pozos, Gómez Farias, Jalisco se procedió a aislar microorganismos en gelosa nutritiva y en el medio propuesto por Bridges para el aislamiento de bacterias fijadoras de nitrógeno, así se obtuvieron un total de 26 cepas las cuales desplegaron 8 patrones diferentes (Fig. 7). Figura 7. Patrones obtenidos por la técnica de RAPD con el oligonucleótido OPB-01 del DNA genómico de las cepas aisladas del tracto digestivo de insectos adultos de Dendroctonus valens, de los Pozos, Gómez Farías, Jalisco (M. Marcador de talla molecular) En resumen, todas las cepas consideradas se agruparon de acuerdo al perfil RAPD desplegado y de ahí se seleccionaron entre 1 y 3 cepas de cada perfil para su identificación molecular mediante el análisis de la secuencia del gen 16S rRNA (Cuadro 4). C San Juanito, I C Chihuahua LOCALIDAD Cuadro 4. Clasificación de las cepas aisladas del tracto digestivo de Dendroctonus valens de acuerdo a los patrones que desplegaron con la técnica de PCR-RAPD empleando el oligonucleótido OPB-01. PATRÓN CEPAS 1 3J, 12EF, 8EF, 11J, 8J, 10EF, 1.7E, 6EF, 7EF, 13EF, 1.4E y 17EF 2 5EF y 1EF 3 15EF y 5EFB 4 2EF I 1L El Wicle, Casas Grandes, Chihuahua. Los Pozos, Gómez Farías, Jalisco. II III IV V VI VII VIII A B C D 2LA, 6L, 7L, 17LA, 17LB y 3L 2LB 3LA, 12L y 19LB 8L 4L y 9L 16L y 5L 19LA 1WB 2WA, 2WC, 4WD, 4WB, 6WD, 8WB y 8WA 3WB, 3WD, 9WA, 9WB, 10WA y 10WC 5WA, 5WB, 7WB, 7WD, 11WA, 11WD, 12WA, 12WB, 13WD, 13WC, 14WC, 14WD, P1W, P2W, P3W, P5W, P7W y P8W a b c d e f MA1B, MA1A, PL3B Y PL3A MA2, MA4, PL1 MA3A MA3B, MA1Bfx, PL4, MP3A Y MP3B MP1 MP3AB, PL5, MA1Afx, MPABfx, P1Afx, P1Bfx, P2fx, P3Afx y P3Bfx PL2 MP1Afx y MP2fx g h 5.1.1.3 AMPLIFICACIÓN DEL GEN 16S rRNA. El gen 16S rRNA se amplificó a partir del DNA genómico de las cepas aisladas del tracto digestivo de insectos adultos de D. valens que desplegaron patrones de RAPD diferentes, los fragmentos del tamaño esperado (1,500 pb aproximadamente) se muestran en la Fig. 8. Figura 8. Amplificación del 16S rRNA de las cepas aisladas del tracto digestivo de Dendroctonus valens. (M. Marcador de talla molecular) IV.1.1.4 ANÁLISIS FILOGENÉTICO El cultivo de bacterias heterótrofas aerobias hemos mostró 11 géneros bacterianos asociados al tracto digestivo de larvas y adultos de D. valens. El análisis de las secuencias del gen 16S rRNA de estos microorganismos indicó que el 54.5% correspondieron a géneros del phylum γ-Proteobacteria y el 45.5% al phylum Actinobacteria (Cuadro 5). El tamaño de las secuencias de cepas representativas de los diferentes patrones de RAPD varió entre 1268 y 1449 nucleótidos y el alineamiento final consto de 1521 nucleótidos, los gaps no fueron considerados en el análisis. La topología de estas secuencias y las obtenidas del GenBank se muestra en la figura 9. Cuadro 5. Aislados bacterianos obtenidos del tracto digestivo de Dendroctonus valens identificados mediante el análisis de la secuencia del 16S rRNA. Porcentaje Organismo conocido más cercano de Aislado Phylum (No. de acceso) similitud (%) 13EF 17EF 3J 6J 7J 9J 2LB 2WA 3WB 1EF 2EF 15EF 19LA 2LA 3LA 1WB 5WA 11WA P7W 16L 1L MP1Afx 97.9 98 98 97.8 98 97.8 98.1 98 97.7 99.2 99.7 99.7 99.2 99.8 99.7 98.8 98.6 98.9 98.8 99.4 96 97 γ-Proteobacteria Rahnella aquatilis (U90757) Serratia liquefaciens (AJ306725) Serratia proteamaculans (AY040208) Enterobacter aerogenes (AJ251468) Pantoea cedenensis (AF130971) Erwinia mallotivora (AJ233414) MP1 8L MA3B 4L MA2 MA1B PL5 MA3A PL2 96.9 98.8 97.8 99.4 98.8 99.6 98.5 98.6 99.2 Acinetobacter lwoffii (DQ328322) Acinetobacter haemolyticus (AM184255) Stenotrophomonas maltophilia (DQ424872) Leifsonia shinshuensis (DQ232614) Janibacter melonis (AY522568) Cellulosimicrobium cellulans (AY665978) Actinobacteria Cellulomonas xylanilytica (AY303668) Figura 9. Relaciones filogenéticas de las secuencias del gen 16S rRNA de las cepas aisladas del tracto digestivo de Dendroctonus valens. El árbol se construyó a partir de un alineamiento múltiple de secuencias de 1521 nucleótidos de γ-proteobacterias y actinobacterias empleando el modelo de Jukes-Cantor y el método de agrupamiento de Neighbor-joining. En el Cuadro 5 se muestra la asignación de las clonas a nivel de especie y la ubicación en los correspondientes Phyla bacterianos. De este modo se identificaron nueve especies de γ-proteobacterias y 4 de actinobacterias. Por otra parte, en el Cuadro 6 se correlacionan los patrones desplegados por las cepas mediante la técnica de RAPD-PCR y su afiliación taxonómica de las cepas representativas de cada patrón de RAPD. En la topología las cepas 2WA, 3WB, 2LB, 3J, 7J, 6J, 9J, 13EF, y 17EF se asociaron con secuencias de microorganismos asociados a cepas de Rahnella aquatilis aisladas del tracto digestivo de larvas y adultos de Dendroctonus frontalis (Vasanthakumar et al., 2006). Las cepas 1EF, 2EF, 15EF, 2LA, 3LA, y 19LA se asociaron con especies del género Serratia; las cepas 1WB, 5WA, 11WA y P7W se agruparon con Enterobacter aerogenes, la cepa 16L se agrupa con Pantoea cedenensis y las cepas MP1Afx, MP1 y 1L se agruparon con secuencias de cepas obtenidas de D. frontalis (Vasanthakumar et al., 2006), todas ellas se agruparon con la secuencia de una Erwinia mallotivora. La cepa 4L se agrupó con Stenotrophomonas maltophilia, la cual ha sido reportada como un organismos capaz de fijar nitrógeno (Park et al., 2005), la cepa MA3B se agrupó con Acinetobacter haemolyticus y la cepa 8L con Acinetobacter lwoffii. Las cepas MA1B y MA2 se asociaron con Janibacter melonis y Leifsonia shinshuensis respectivamente; las cepas MA3A y PL5 se asociaron con Cellulomicrobium cellulans y la cepa PL2 con Cellulomonas xylanilytica. Los porcentajes de similitud de las secuencias obtenidas con aquellas de microorganismos reportadas en el banco de genes se muestran en el cuadro 5. Las cepas 2LB, 3J, 9J, 13EF, 7J, 6J y 17EF tienen una similitud del 97.7-98.1% con la secuencia de Rahnella aquatilis (U90757), las cepas 1EF, 2EF, 15EF y 19LA tuvieron una similitud de 99.2-99.7% con la secuencia de Serratia liquefaciens (AJ306725). Mientras que las secuencias 2LA y 3LA tuvieron una similitud del 99.8 y 99.7% con la secuencia de Serratia proteamaculans (AY040208); no obstante todas ellas también tuvieron porcentajes de similitud muy altos con otras cepas del genero Serratia (Datos no mostrados). Las cepas1WB, 5WA, 11WA y P7W presentaron una similitud del 98.6-98.9% con la secuencia de Enterobacter aerogenes (AJ251468), la cepa 16L del 99.4% con la secuencia de Pantoea cedenensis (AF130971) y las cepas 1L, MP1Afx y MP1 presentaron una similitud del 96-97% con la secuencia de Erwinia mallotivora (AJ233414). La cepa 8L se asoció con un 98.8 con Acinetobacter lwoffii (DQ328322), la cepa MA3B presentó una similitud del 97.8% con Acinetobacter haemolyticus (AM184255) y la cepa 4L posee un similitud del 99.4% con Stenotrophomonas maltophilia (DQ424872). Todos estos organismos pertenecen al phylum γ-Proteobacteria. En éste mismo cuadro podemos observar que la cepa MA2 tuvo una similitud del 98.8% con Leifsonia shinshuensis (DQ232614), la cepa MA1B del 99.6% con Janibacter melonis (AY522568), la cepa PL5 tuvo un 98.5% de similitud con Cellulosimicrobium cellulans (AY665978) y la cepa MA3A y PL2 del 98.699.2% con Cellulomonas xylanilytica (AY303668). Todos estos microorganismos pertenecen al phylum Actinobacteria. Cuadro 6. Afiliación filogenética de las secuencias de los genes 16S rRNA de las cepas aisladas del tracto digestivo de D. valens. Localidad San Juanito, Chihuahua o 27 55’22’’ N 107º55’47’’ O CICS, Milpa Alta, Distrito Federal 19º 04’ N 98º 58’ O El Wicle, Casas Grandes, Chihuahua 30º09’76’’ N 108 º 57’03’’ O Los Pozos, Gómez Farías, Jalisco 19º88’20’’ N 103º40’40’’ O Estadio del Insecto colectado Patrón desplegado de PCRRAPD Aislado representativo Adultos 1 3J, 13EF, 17EF 2 1EF 3 15EF 4 2EF I 1L II 2LA III 2LB IV 3LA V 8L VI 4L VII 16L VIII 19LA A 1WB B 2WA C 3WB D a 5WA, 11WA, P7W MA1B b MA2 c MA3A d MA3B e MP1 f PL5 g PL2 h MP1AFx Larvas Adultos Adultos Relación filogenética Especie más cercana reportada en GenBank (No. de acceso) Rahnella aquatilis (U90757) Serratia liquefaciens (AJ306725) Serratia liquefaciens (AJ306725) Serratia liquefaciens (AJ306725) Erwinia mallotivora (AJ233414) Serratia proteamaculans (AY040208) Rahnella aquatilis (U90757) Serratia proteamaculans (AY040208) Acinetobacter lwoffii (DQ328322) Stenotrophomonas maltophilia (DQ424872) Pantoea cedenensis (AF130971) Serratia liquefaciens (AJ306725) Enterobacter aerogenes (AJ251468) Rahnella aquatilis (U90757) Rahnella aquatilis (U90757) Enterobacter aerogenes (AJ251468) Janibacter melonis (AY522568) Leifsonia shinshuensis (DQ232614) Cellulomonas xylanilytica (AY303668) Acinetobacter haemolyticus (AM184255) Erwinia mallotivora (AJ233414) Cellulosimicrobium cellulans (AY665978) Cellulomonas xylanilytica (AY303668) Erwinia mallotivora (AJ233414) Similitud (%) Grupo microbiano de afiliación 98, 97.9, 98 99.2 Rahnella aquatilis Serratia liquefaciens 99.7 Serratia liquefaciens 99.7 Serratia liquefaciens 96 Erwinia sp. o Pantoea sp. Serratia proteamaculans Rahnella aquatilis 99.8 98.1 99.7 99.8 Serratia proteamaculans Acinetobacter lwoffii 99.4 Stenotrophomonas maltophilia 99.4 Pantoea cedenensis 99.2 Serratia liquefaciens 98.8 98 Enterobacter aerogenes Rahnella aquatilis 97.7 Rahnella aquatilis 98.6, 98.9, 98.8 99.6 Enterobacter aerogenes Janibacter melonis 98.8 Leifsonia shinshuensis 98.6 Cellulomonas xylanilytica Acinetobacter haemolyticus Erwinia sp. o Pantoea sp. Cellulosimicrobium cellulans 97.8 96.9 98.5 99.2 97 Cellulomonas xylanilytica Erwinia mallotivora IV.1.1.5 ACTIVIDAD CELULOLÍTICA Al observar que algunos de las cepas aisladas se identificaron como Cellulomonas xylanilytica y Cellulosimicrobium cellulans, especies en las que se ha reportado la capacidad de degradar la celulosa (Schumann, 2001; Rivas 2004), se decidió hacer un ensayo para evaluar la actividad celulolítica de las cepas del phylum Actinobacteria empleando agar rojo congo celulosa (Cuadro 7 y Fig. 10). La población de bacterias celulolíticas aerobias asociadas al tracto digestivo de D. valens fue de 9 x 102 ± 2.1 x 102 UFC/tracto digestivo. Cuadro 7. Actividad celulolítica de las cepas pertenecientes al phylum Actinobacteria asociadas al canal alimentario de Dendroctonus valens. Insecto de Clave del Grupo microbiano de Actividad procedencia aislado afiliación enzimáticaa PL2 Cellulomonas xylanilytica 2.7 Cellulosimicrobium PL5 2.9 Dendroctonus cellulans valens MA1B Janibacter melonis 3 (Adulto) MA2 Leifsonia shinshuensis 3.1 MA3A a Cellulomonas xylanilytica 2.4 La actividad enzimática se definió como el diámetro del halo claro dividido entre el diámetro de la colonia. Figura 10 Actividad celulolítica de algunas de las cepas aisladas en agar rojo congo. A. MA1B Janibacter melonis, B. PL2 Cellulomonas xylanilytica, C. PL5 Cellulosimicrobium cellulans. La capacidad de algunas de las cepas de degradar la celulosa es una evidencia indirecta de la posible participación de los microorganismos asociados al canal alimentario de D. valens en la digestión de la celulosa. IV.1.1.6 REDUCCIÓN DE ACETILENO EN LOS INSECTOS La reducción de acetileno fue detectada en larvas e insectos adultos vivos (15.96 ± 2.22; 12.59 ± 3.39 nmol de etileno/h/insecto; promedio ± ES), mientras que en larvas e insectos adultos muertos y esterilizados, y en el testigo sin insectos no se encontró actividad (1.12 ± 0.47; 0.34 ± 0.22; 0 ± 0 nmol de etileno/h/insecto; promedio ± ES) (ANOVA de un factor: F= 15.09; P < 0.05). Estos resultados indican que la fijación de nitrógeno ocurre in vivo en D. valens (Fig. 11) y nos permite deducir que algún miembro de la comunidad procariótica asociada al insecto realizó esta actividad, dado que es conocido que ninguna célula u organismo eucariote tiene la capacidad para fijar nitrógeno molecular. Figura. 11 Actividad de la nitrogenasa in vivo en larvas y adultos de D. valens. (LV, Larvas vivas; AV, Adultos vivos; LM, Larvas muertas esterilizadas; AM, Adultos muertos esterilizados; T, Testigos con papel.) (Las barras representan el error estándar). Al efectuar la prueba de Tukey HSD encontramos que no existen diferencias significativas entre la cantidad de etileno producido por las larvas y los adultos (q= 1.645; P > 0.05) y si se encuentra una diferencia significativa entre los experimentos efectuados con individuos vivos y los realizados con individuos muertos y esterilizados, como ya se mencionó anteriormente (L.V.-L.M.: q=7.251; A.V.-A.M.: q=5.99; P < 0.05; nomenclatura como en la figura 11). IV.1.2 DIVERSIDAD DETECTADA POR MÉTODOS MOLECULARES IV.1.2.1 ANÁLISIS RFLP DE LAS CLONAS POSITIVAS Para evitar la secuenciación excesiva de las clonas de la librería de genes ribosomales de las diferentes partes del tracto digestivo de hembras de D. valens se efectuó una prueba de polimorfismo de la longitud de fragmentos de restricción (RFLP) empleando la enzima HpaII (Moyer et al.,1996). Un total de 12 patrones diferentes fueron observados. El mesenterón anterior y el proctodeo desplegaron 10 y 6 patrones diferentes respectivamente (Fig. 12 y Fig. 13). Ambas regiones anatómicas del canal alimentario compartieron 4 patrones RFLP. Figura 12. Perfiles de restricción de la enzima HpaII de las clonas de la librería de genes ribosomales del mesenterón anterior de hembras de D. valens. Cada color representa un patrón diferente. Los patrones que son iguales en este despliegue, pero que muestran un color diferente, fueron distinguidos a partir de su patrón desplegado con EcoRI. Figura 13. Perfiles de restricción de la enzima HpaII de las clonas de la librería de genes ribosomales del proctodeo de hembras de D. valens. Cada color representa un patrón diferente. Los patrones que son iguales en este despliegue, pero que muestran un color diferente, fueron distinguidos a partir de su patrón desplegado con EcoRI. IV.1.2.2 ANÁLISIS DE LAS SECUENCIAS DE LAS CLONAS DE LAS LIBRERIAS DE GENES RIBOSOMALES. Mediante la construcción de librerías de genes ribosomales logramos asociar cuatro géneros bacterianos al mesenterón anterior y proctodeo de hembras adultas de D. valens y ambas librerías se analizaron conjuntamente. El análisis de las secuencias con el programa CHIMERA-CHECK permitió reconocer que varias clonas correspondían a quimeras, posiblemente formadas durante la amplificación de los genes ribosomales a partir del DNA metagenómico. En el resto de las secuencias (n=9) se identificaron hasta especie miembros de los géneros Rahnella, Erwinia o Pantoea, Lactococcus y Acinetobacter (Fig. 14). Figura 14. Relaciones filogenéticas de las secuencias del gen 16S rRNA de las clonas obtenidas de las librerías de genes ribosomales del DNA metagenómico del tracto digestivo de Dendroctonus valens. El árbol se construyó a partir de un alineamiento de 1593 bases de secuencias de γproteobacterias y firmicutes empleando el modelo de sustitución de Kimura 2 Parámetros y el método de agrupamiento de Neighbor-joining. En el Cuadro 8 se correlacionan los patrones desplegados por las clonas mediante la técnica de RFLP y su afiliación taxonómica obtenida a partir de la secuenciación del gen 16S rRNA. Cuadro 8. Afiliación filogenética de las secuencias de los genes 16S rRNA de las clonas obtenidas a partir de las librerías de genes ribosomales del DNA metagenómico del tracto digestivo de D. valens. Localidad Estadio del Insecto colectado Patrón desplegado por RFLP (número de clonas) Relación filogenética Clona representativa Especie más cercana reportada en GenBank (No. de acceso) Similitud (%) Grupo microbiano de afiliación El Wicle, Casas Grandes, Chihuahua 30º09’76’’ N 108 º 57’03’’ O Adultos (Hembras) A (8) B (7) C (1) D (1) E (8) F (4) MAF16 PF1 PF4 99.5 99.5 99.5 Lactococcus lactis 99.1 ---- Acinetobacter schindleri ---- Rahnella aquatilis (U90757) Rahnella aquatilis (U90757) Pantoea dispersa (AB273743) Quimera 96.8 Rahnella sp. 97.7 Rahnella aquatilis ---- Pantoea dispersa ---- 97.5 Rahnella aquatilis 97.8 Rahnella aquatilis MAF26 Rahnella aquatilis (U90757) Rahnella aquatilis (U90757) Quimera ---- ---- MAF34 Quimera ---- ---- MAF35 Quimera ---- ---- PF23 MAF20 MAF2 MAF7 PF20 G (9) H (3) I (1) J (1) K (1) L (1) PF22 MAF4 MAF15 Lactococcus lactis (EU104368) Lactococcus lactis (EU104368) Acinetobacter schindleri (AJ275041) Quimera Lactococcus lactis 97.6 IV.1.2.3 ANÁLISIS DE RAREFACCIÓN E ÍNDICES DE DIVERSIDAD Las curvas de rarefacción de las librerias construidas a partir de DNA metagenómico del canal alimentario de hembras adultas de D. indican que existe una comunidad bacteriana relativamente simple, es decir con una riqueza de especies baja (Fig. 15, 16 y 17). Aunque no se obtuvo una forma asintótica en la curva de rarefracción de la librería de genes ribosomales a nivel de especie, es decir con un máximo de 3% de disimilitud, la gráfica adquiere una tendencia asintótica cuando analizamos las secuencias a nivel de género y phylum, lo que implica considerar las secuencias con una diferencia no mayor al 5% y 20% respectivamente (Fig. 16 y 17). Esto sugiere que logramos reconocer la mayoría de los géneros y phyla asociados al tracto digestivo de este insecto. Figura 15. Análisis de rarefacción de la librería del gen 16S rRNA construida a partir del DNA del tracto digestivo de 3 individuos hembra adultos de D. valens. La curva de rarefacción se construyó con base a análisis hecho en DOTUR empleando el algoritmo de asignación del vecino más lejano. La curva de rarefacción representa a las secuencias con una similitud del 97% entre ellas, lo cual corresponde al nivel taxonómico de especie. Figura 16. Análisis de rarefacción de la librería del gen 16S rRNA construida a partir del DNA del tracto digestivo de 3 individuos hembra adultos de Dendroctonus valens. La curva de rarefacción se construyó con base en un análisis hecho en DOTUR empleando el algoritmo de asignación del vecino más lejano. La curva de rarefacción representa a las secuencias con una similitud del 95% entre ellas, lo cual corresponde al nivel taxonómico de género. Figura 17. Análisis de rarefacción de la librería del gen 16S rRNA construida a partir del DNA del tracto digestivo de 3 individuos hembra adultos de D. valens. La curva de rarefacción se construyó con base a análisis hecho en DOTUR empleando el algoritmo de asignación del vecino más lejano. La curva de rarefacción representa a las secuencias con una similitud del 80% entre ellas, lo cual corresponde al nivel taxonómico de phylum. Para determinar si se había analizado un número suficiente de secuencias de la librería de genes ribosomales construida se calculó el estimador no paramétrico de la riqueza de especies Chao 1. La curva de colector para el estimador Chao 1 con una diferencia del 3% entre las secuencias muestra una línea asintótica, lo cual indica que la mayoría de las especies ya han sido analizadas (Datos no mostrados). La riqueza de Chao1 terminal estimada para la librería ribosomal analizada fue de 5 OTUs, en la cual una OTU fue definida como un grupo de secuencias que no difieren más del 3% entre ellas, es decir a nivel de especie. IV.1.2.4 PCR-DGGE DE LA REGIÓN V3 DE GENES RIBOSOMALES El número mayor de bandas (n=12) fue desplegado en el mesenterón anterior y el proctodeo de hembras adultas de D. valens. En el caso de los machos las regiones que desarrollaron el mayor número de bandas fue el mesenterón anterior y el proctodeo con 8 bandas cada uno. Si consideramos a cada una de las bandas desplegadas en el DGGE como una especie diferente de microorganismo podemos asumir que la comunidad bacteriana asociada a las diferentes partes del tracto digestivo es relativamente simple (12 especies) y que mantiene cierta homogeneidad a lo largo del canal alimentario (6 especies en común), presentándose la mayor diversidad en el proctodeo (Fig. 18). En total se comparten 6 bandas en todas las regiones del canal alimentario tanto de hembras como de machos y solamente la banda No. 9 es exclusiva del tracto digestivo de los machos (Fig. 18). Durante las repeticiones del DGGE se observó que en la mayoría de los geles la región que constantemente mostraba el mayor número de bandas era el proctodeo tanto en las hembras como en los machos (Datos no mostrados). También se observó que algunas bandas aparecían esporádicamente y existían pequeñas variaciones en el patrón desplegado por las diferentes secciones del canal alimentario tanto de hembras como de machos, lo que posiblemente indica que la estructura poblacional que albergan estos insectos puede cambiar durante el transcurso del tiempo o que hay un cierto grado de variación en la comunidad de especies bacterianas en este hábitat (Datos no mostrados). Al comparar los patrones desplegados por los amplificados de la región 3V del 16S rDNA de las diferentes secciones del tracto digestivo y algunas de las cepas aisladas podemos observar que la banda No. 5 puede corresponder a Rahnella aquatilis y la banda No. 6 a Enterobacter aerogenes, dado que estas bandas comigraron a la misma altura en ambos casos (Fig. 19). Las banda No. 5 es muy evidente en el proctodeo de machos, lo cual nos indica que la población de E. aerogenes es una de la más abundantes en esta sección. Por otro lado, la banda No. 6 se presenta con gran intensidad en todas las secciones del canal alimentario de hembras y machos. Lo anterior nos indica que R. aquatilis es una bacteria que se encuentra ampliamente distribuida en el tracto digestivo de este insecto. Figura 18. PCR-DGGE de la región 3 variable de los genes 16S rRNA obtenidos a partir del DNA metagenómico de las diferentes partes del tracto digestivo de individuos colectados en el campo de D. valens. Los productos fueron separados por electroforesis en un gradiente de urea formamida del 30 al 60%. Figura 19. PCR-DGGE de la región 3 variable de los genes 16S rRNA obtenidos a partir del DNA metagenómico de las diferentes partes del tracto digestivo de individuos de D. valens y algunas cepas aisladas. Los productos fueron corridos en un gradiente urea formamida (30-60%). La flecha No. 5 representa a una de las bandas que despliega Rahnella aquatilis y la flecha No. 6 representa a una de las bandas que despliega Enterobacter aerogenes, estos dos organismos fueron aislados e identificados mediante la secuenciación del gen 16S rRNA. IV.1.2.5 DISEÑO DE OLIGONUCLEÓTIDOS PARA AMPLIFICAR EL GEN nifH. Debido a la falta de resultados positivos al tratar de amplificar el gen nifH con los oligonucleótidos originalmente propuestos para efectuar éste trabajo, se diseñaron nuevos. Los iniciadores se diseñaron a partir del alineamiento de secuencias completas del gen nifH obtenidas del banco de genes de la NCBI, la secuencia de los oligonucleótidos propuestos, la temperatura de alineamiento, el tamaño del amplificado esperado y al grupo de microorganismos a los cuales van dirigidos se resumen en el cuadro No. 9. Ahora que se tienen identificadas las bacterias que posiblemente fijen nitrógeno se diseñaran oligos específicos para amplificar el gen nif H con base a las secuencias de este gen ya reportadas en la literatura. Cuadro 9. Oligonucleótidos diseñados para la amplificación del gen nifH Grupo microbiano Oligonucleótidos Secuencia (5´- 3´) Tm (oC) Enterobacterias Fentero Rentero Fazoto Razoto Fclos Rclos FFRA RFRA ACACCATTATGGAGATGG GATGCCGAACTCCATCAG GACTCCACCCGCCTGATCCT GATCGTATTCGATCACGGTCAT GCTATTTAYGGAAARGTTG GCATAYADTGCCATCATT TYGGCAAGTCCACCACC GCGCCATCATCTCRCCGGA 54 Tamaño esperado (pb) 650 65 550 51 450 55 400 Azotobacter spp. Clostridium spp. FrankiaRhizobiumAzospirillum 5.1.2.6 AMPLIFICACIÓN DE LOS GENES IMPORTANTE EN EL CICLO DEL NITRÓGENO Hasta el momento sólo hemos logrado amplificar el gen nifH de enterobacterias en el mesenterón anterior del macho (Fig 20.) y el gen nifD en todas las partes del tubo digestivo de hembras (Fig. 21). La presencia de estos genes, el aislamiento de bacterias capaces de reducir el acetileno y la reducción de acetileno que llevaron a cabo larvas y adultos de D. valens in vivo son evidencias de que existen poblaciones de microorganismos asociados al tracto digestivo del descortezador que son capaces de fijar nitrógeno y estarían participando activamente en el suplemento de la dieta de este insecto con compuestos nitrogenados asimilables. Figura 20. Amplificación del gen nifH de enterobacterias a partir del DNA metagenómico de las diferentes partes del canal alimentario de D. valens. Figura 21. Amplificación del gen nifD a partir del DNA metagenómico de las diferentes partes del canal alimentario de D. valens. Otros genes bacterianos asociados al ciclo del nitrógeno (amonio monooxigenasa (amoA), nitrito reductasa dependiente de cobre (nirK) y citocromo cd1 (nirS), óxido nítrico reductasa (nosZ) y los genes 16S rRNA del género Nitrobacter) no han sido amplificado, a pesar de que se han ensayado diferentes condiciones de amplificación por PCR (Datos no mostrados). DISCUSION Los insectos son unas de las formas de vida más exitosas sobre el planeta. Uno de los factores más importantes del éxito de los insectos es que se han adaptado a una amplia variedad de dietas. La flexibilidad de los hábitos alimentarios de los insectos está asociada, de manera muy significativa, a la contribución que llevan a cabo los microorganismos del intestino. Por otro lado, poco se sabe acerca de la diversidad, fisiología y ecología de los microorganismos asociados al tracto digestivo de los escarabajos que se desarrollan dentro de la madera y la corteza de los árboles (Brand et al., 1975; Bridges, 1981; Bridges et al., 1984; Delalibera et al., 2005; Moore 1972a, b; Vasanthakumar et al., 2006). Por lo que el presente trabajo pretende contribuir al conocimiento de la diversidad de bacterias y genes bacterianos relevantes asociados al tracto digestivo de D. valens. Para lograr entender las posibles contribuciones que tienen los microorganismos del intestino en la digestión y nutrición de su huésped es necesario identificarlos y asociarlos a un nicho ecológico. Este es el primer trabajo que estudia la microbiota asociada al canal alimentario de D. valens empleando métodos basados en el cultivo y métodos independientes del cultivo. El tracto digestivo de D. valens está constituido por las regiones estomodeo, mesenterón y proctodeo que poseen estructuras tisulares bien diferenciadas y las cuales están separadas por dos estrechamientos parecidos a una válvula (Díaz et al., 1998). En este trabajo, inicialmente se encontró que el número de bacterias cultivables asociadas al proctodeo es un orden de magnitud mayor al observado en las otras dos secciones del mesenterón (Cuadro 3). Este resultado coincide con otros trabajos realizados con otros artrópodos como termitas, cucarachas y grillos, en los cuales se ha determinado que el proctodeo alberga un mayor número de bacterias cultivables que otras partes del tracto digestivo (Breznak, 1982; Cruden y Markovetz, 1984; Ulrich et al., 1981). La misma conclusión fue alcanzada empleando técnicas basadas en la microscopía de fluorescencia donde se determinó que el número de bacterias que residen en el proctodeo de las cucarachas Periplaneta americana, Periplaneta australasia, Pynoscelus surinamensis, Blaberus fuscus y Gromphadorhina portentosa era diez veces mayor al número encontrado en el estomodeo y mesenterón. En este mismo trabajo se reportó que los proctodeos del miliápodo (Chicobulus sp), del grillo (Acheata domestica) y del escarabajo (Pachnoda marginata) presentan un mayor número de bacterias (Cazemier et al., 1997). El elevado número de bacterias asociadas al proctodeo de D. valens sugiere, que esta parte del tubo alimentario juega un papel muy importante en el proceso digestivo de este insecto. Los resultados reportados en este trabajo demuestran que el mesenterón y proctodeo de D. valens albergan una comunidad bacteriana poco diversa. Los métodos basados en el cultivo y los independientes de éste permitieron reconocer e identificar tan sólo 17 especies bacterianas (Cuadros 6 y 8) pertenecientes a los grupos de γ-Proteobacteria, Actinobacteria y Firmicutes (Fig. 9 y 14). Esta aparente baja diversidad observada se hace evidente cuando la comparamos con la diversidad bacteriana observada en los intestinos de las termitas, los cuales albergan 50 filotipos (Okhuma et al., 1996). En trabajos realizados en otros coleópteros (Pachnoda ephippiata y Melolontha melolonta) se reportó que la comunidad microbiana asociada a sus tractos digestivos pertenece a los Firmicutes (Lactobacillales, Clostridiales y Bacillales), Actinobacteria, γ−, β− y δ−Proteobacteria, Bacteroidetes, y Actinobacteria (Egert et al., 2003 y 2005). Los trabajos antes mencionados poseen un número amplio de microorganismos identificados, lo cual contrasta con nuestros resultados y pone en evidencia la escasa diversidad de microorganismos que se encuentran alojados en el canal alimentario de D. valens. El único trabajo previo que explora la diversidad microbiana en el tracto digestivo de una especie del género Dendroctonus, es el de D. frontalis, en donde los microorganismos se afiliaron a los grupos α, γ−Proteobacteria y Firmicutes (Vasanthakumar et al., 2006), lo cual concuerda con los resultados que se obtuvieron en este trabajo. Particularmente, no hemos encontrado microorganismos anaerobios estrictos (Clostridiales y Bacteroidetes) que comúnmente se encuentran asociados al canal alimentario de otros insectos (Egert et al. 2005; Moran et al., 2005; Ohkuma et al., 1996), esto posiblemente es debido a que en el tracto digestivo de D. valens no se alcanzan niveles de potencial de óxido-reducción suficientemente bajos como los que se observan en los intestinos de termitas y otros insectos. Entre las bacterias cultivables que se identificaron en este trabajo se encuentran Rahnella aquatilis, Enterobacter aerogenes, Stenotrophomonas maltophilia, Pantoea sp., Acinetobacter sp. y Serratia sp. Estos microorganismos también se han detectado en D. frontalis. Sin embargo, en D. valens no se logró documentar la presencia de bacterias como Pseudomonas fluorescens, Bacillus sp., Microccocus sp., Mycoplasma sp., Rhodobacter sp, Leuconostoc mesenteroides, Enterococcus sp. y Lactobacillus sp. (Bridges et al., 1984; Moore, 1972a, b; Vasanthakumar et al., 2006). Algunas de estas variaciones pueden ser debidas a la diferente ecología y hábitos que tienen ambas especies ó a las estrategias para el aislamiento de bacterias practicadas en cada uno de los trabajos. Muchos de los géneros bacterianos asociados al canal alimentario de D. valens se han aislado de los intestinos de otros invertebrados. El género Rahnella ha sido frecuentemente aislado del tracto digestivo de larvas y adultos de D. valens y D. frontalis (Vasanthakumar et al., 2006). En particular, Rahnella aquatilis o alguna de sus genomoespecies se han aislado del tracto digestivo del escarabajo xilófago, Saperda vestita y del intestino de caracoles, babosas y lombrices de tierra (Delalibera et al., 2005; Muller et al.; 1995a, b). Asimismo, este género se ha identificado como un endófito de coníferas y se le ha reportado como una endobacteria de ectomicorrízas (Cankar et al., 2005; Izumi et al., 2006), lo cual sugiere que esta bacteria puede estar siendo ingerida constantemente por el insecto al alimentarse del floema de su huésped. Investigaciones futuras podrán revelar si las cepas de Rahnella en el tracto digestivo de Dendroctonus spp, son las mismas que las que se encuentran en el floema de las coníferas o si se trata de poblaciones o ecotipos independientes. Asimismo, será muy interesante averiguar si estas bacterias, que tienen la capacidad potencial de fijar nitrógeno, están activas reproductiva y metabólicamente y si llevan a cabo la fijación de nitrógeno en el tracto digestivo del insecto. Para estos propósitos convendría averiguar si los miembros de este género aparecen en una librería de cDNA de genes ribosomales construida a partir de rRNA y estimar la expresión de alguno de los genes de la nitrogenasa a partir del contenido del tracto digestivo de miembros de Dendroctonus. Otro género aislado frecuentemente en este trabajo fue Serratia. Las secuencias generadas a partir de nuestros aislados se asociaron principalmente con Serratia liquefaciens y Serratia proteomaculans, pero mantuvieron una alta similitud con otras especies de Serratia. Esto obliga a realizar una caracterización fenotípica que complemente la identificación molecular ensayada hasta este momento (Holt et al., 1994). Miembros del género Serratia son comúnmente aislados del canal alimentario de otros insectos (Brand et al., 1975; Broderick et al., 2004; Lundgren et al., 2007). Serratia marcescens, Serratia entomophila, Serratia proteamaculans se han descrito como entomopatógenos facultativos (Hurts et al., 2007; Steinhaus, 1956). Asimismo, Serratia se reportó como un patógeno de D. frontalis (Moore, 1972a). El constante aislamiento de estas bacterias a partir de individuos aparentemente sanos y colectados en el campo de D. valens, sugiere que estos microorganismos forman parte de la microbiota normal del insecto y mantienen un balance con otras poblaciones bacterianas que impiden el desarrollo de su potencial patogénico, como sucede con otras bacterias entomopatógenas (Harada e Ishikawa, 1997). En adultos y larvas del modelo de estudio de este trabajo se aislaron microorganismos que se asociaron al grupo Erwinia/Pantoea. El género Erwinia se ha asociado a diversos insectos como arañuelas, áfidos, escarabajos pepino y la mosca del olivo (Capuzzo et al., 2005; de Vries et al., 2004; Ferrari et al., 2007; Harada e Ishikawa, 1997). En algunos casos, las especies de este grupo actúan como entomopatógenos (Grenier et al., 2006; Harada e Ishikawa, 1997) y en otros aceleran el proceso de desarrollo y aumentan el número de huevos que ponen los insectos (de Vries et al., 2004). El género Pantoea también se ha encontrado en varios insectos como las arañuelas, mosquitos, áfidos, moscas y escarabajos (Delalibera et al., 2005; Mramba et al., 2006; Straif, et al., 1998; Vorwerk et al., 2007; Wells et al., 2002). En particular en el género Dendroctonus se ha reportado la presencia de la bacteria fijadora de nitrógeno Pantoea agglomerans (Bridges, 1981; Bridges et al., 1984; Vasanthakumar et al., 2006). La baja similitud que presentan las secuencias de genes ribosomales de los aislados obtenidos con los diferentes especies del grupo Erwinia/Pantoea impide asociarlos a una especie en particular, debido a ello, es complicado sugerir una función dentro de la comunidad bacteriana o en la simbiosis con D. valens. A partir de los tractos digestivos de insectos colectados en Casas Grandes, Chihuahua se aislaron constantemente cepas de Enterobacter aerogenes. Esta especie y otras del mismos género se han aislado también del canal alimentario de las crisopas pardas, de termitas, de la polilla gitana y de escarabajos barrenadores de la madera, entre otros insectos (Broderick et al., 2004; Eutick, et al., 1978; Schloss et al., 2006; Woolfolk y Inglis, 2003). Previamente, Enterobacter aerogenes también se aisló en D. frontalis. Las especies del género Enterobacter son una de las principales poblaciones bacterianas que se albergan en el tracto digestivo de varias termitas, entre ellas Coptotermes acinaciformis (Adams y Boopathy, 2005; Eutick, et al., 1978). Enterobacter es una bacteria anaerobia facultativa que se ha descrito como un “pepenador de oxigeno” y se ha sugerido que elimina el oxígeno que se permeabiliza a través del exoesqueleto y que puede afectar los procesos anaeróbicos (acetogénesis, hidrogénesis, metanogénesis, reducción del sulfato, y fijación de nitrógeno) que son fundamentales para la supervivencia de las termitas. La glucosa, sacarosa y xilosa son carbohidratos fermentables que se forman durante la degradación de la celulosa y que pueden ser metabolizados por varias especies del género Enterobacter (Adams et al., 2005). La xilosa no se encuentra comúnmente en la naturaleza en un estado libre, sino como un polímero denominado xilano, el cual se puede encontrar en asociación con la celulosa de las plantas (Jeffries y Jin, 2000). Por lo que canal alimentario del insecto ofrece un lugar en el cual estos polímeros del floema y la albura de las plantas pueden ser degradados para su asimilación. E. aerogenes dentro del canal alimentario de D. valens podría estar generando microambientes anóxicos, que favorezcan la fijación de nitrógeno y participando en los pasos finales de la degradación de la celulosa y el xilano. Acinetobacter lwoffi y A. haemolyticus forman parte de los microorganismos que se detectaron dentro del canal alimentario de D. valens. Especies del género Acinetobacter se han encontrado en los tractos digestivos de diversos artrópodos (Dunn y Stabb, 2005; Schäfer, 1996; Schloss et al., 2006; Sezen et al., 2007; Xiang et al.; 2006). En D. frontalis se reportó la presencia de bacterias del género Acinetobacter lo que coincide con los resultados para D. valens. Miembros de Acinetobacter tienen la capacidad potencial de degradar celulosa y han sido aislados del canal alimentario de las termitas y de otros ambientes (Ekperigin, 2006; Schfäer et al., 1996; Vimala et al., 2004). Con base en lo antes mencionado, se puede inferir que las cepas de Acinetobacter podrían estar participando en la degradación de la celulosa en el tubo digestivo de D. valens. Stenotrophomonas maltophilia es otra bacteria que hemos identificado como miembro de la comunidad bacteriana asociada a D. valens. El género Stenotrophomonas también ha sido encontrado en otros insectos y en D. frontalis (Campbell et al., 2004; Monttlor Curley et al., 2007; Schloss et al., 2006; Vasanthakumar et al., 2006). Particularmente, S. maltophilia se ha reportado como una bacteria fijadora de nitrógeno (Park et al., 2005) que podría estar suministrando compuestos nitrógenados asimilables a D. valens. El phylum Actinobacteria está representado por cuatro especies dentro de la comunidad bacteriana asociada al tracto digestivo de D. valens, las cuales fueron identificadas como Cellulosimicrobium cellulans, Cellulomonas xylanilytica, Leifsonia shinshuensis y Janibacter melonis. Bacterias del phylum Actinobacteria se han encontrado frecuentemente dentro de los canales alimentarios de diversos insectos incluyendo a insectos que se alimentan de la madera y del floema de las plantas (Bridges, 1981; Bridges et al., 1984; Broderick et al., 2004; Egert et al., 2003 y 2005; Schäfer et al., 1996; Schloss et al., 2006; Vasanthakumar et al., 2006). En particular, Cellulosimicrobium cellulans secreta un complejo de enzimas líticas capaces de degradar la pared celular de levaduras (varias preparaciones comerciales de glucanasas se obtienen a partir de este microorganismo) (Ferrer, 2006). Se ha identificado y aislado a este microorganismo y creemos que puede estar jugando un papel muy importante en el control de las poblaciones de levaduras asociadas al canal alimentario de D. valens. Todas las actinobacterias que se identificaron en este trabajo fueron capaces de degradar la celulosa (Fig. 10 y Cuadro 7). Los resultados anteriores son una evidencia indirecta de la posible participación de los microorganismos asociados al canal alimentario de D. valens en la digestión de la celulosa contenida en el floema de la plantas. Esta asociación ha sido ampliamente estudiada en insectos como las termitas (Saxena et al., 1993; Schäfer et al., 1996; Varma et al., 1994; Wenzel et al., 2002) y las cucarachas comedoras de madera (Cruden y Markovetz, 1979) y se ha visto el impacto negativo que tiene la carencia de estos microorganismos sobre el desarrollo y reproducción de los insectos. La capacidad de degradar la carboximetilcelulosa (CMC) de Cellulomonas xylanilytica y C. cellulans se ha reportado previamente (Rivas et al., 2004; Schumann et al., 2001). Sin embargo, hasta nuestro conocimiento en el caso de Janibacter melonis y Leifsonia shinshuensis no existen reportes previos en los cuales se les asocié con dicha capacidad. La densidad de bacterias celulolíticas aerobias asociadas a D. valens determinada en este trabajo fue de 900 ± 210 UFC/tracto digestivo, lo que equivale al 0.04% de la población total de bacterias cultivables en medio Gelosa Nutritiva. En la cucaracha Eublaberus posticus se reportó que las bacterias capaces de degradar la CMC constituyen alrededor del 1% de las bacterias cultivables asociadas a su tracto digestivo (Cruden y Markovetz 1979). La aparente baja proporción de bacterias celulolíticas asociadas al trato digestivo de D. valens podría deberse, al menos parcialmente, a que nosotros empleamos como sustrato celulosa en polvo y no CMC. La CMC es un derivado de la celulosa que frecuentemente se usa en los ensayos de actividad de endoglucanasas (Karlsson et al., 2002). La mayoría de los trabajos que caracterizan a los microorganismos celulolíticos emplean la sal disódica de la CMC debido a que posee una alta solubilidad en agua. Sin embargo, existen evidencias de que la capacidad de degradar la celulosa se encuentra distribuida en un menor número de microorganismos que la capacidad de degradar alguno de sus derivados como la CMC (Reese et al., 1950). Los nichos ecológicos en los cuales se encuentran involucradas las bacterias asociadas a los intestinos de los insectos se ha estudiado en muy pocas especies (Dillon y Dillon, 2004). Particularmente, la capacidad de degradar la celulosa sólo se ha descrito en las termitas, cucarachas y algunos barrenadores de la madera (Cruden y Markovetz, 1979; Delalibera et al., 2005; Saxena et al., 1993; Schäfer et al., 1996; Varma et al., 1994; Wenzel et al., 2002). En este trabajo identificamos y cuantificamos a las bacterias celulolíticas aerobias asociadas al trato digestivo de D. valens e inferimos que conjuntamente con otras poblaciones bacterianas (Serratia, Enterobacter y Acinetobacter) y de levaduras pueden estar contribuyendo en la digestión completa de la celulosa y al aprovechamiento de este polisacárido como fuente de carbono. Esta interdependencia cometabólica también se ha documentado ampliamente en las termitas (Schäfer et al., 1996). Los métodos independientes del cultivo revelan comúnmente una mayor diversidad de especies bacterianas en comparación con los métodos basados en el cultivo (Broderick et al., 2004; Eckburg et al., 2005; Paster et al., 2001). En nuestro caso, reconocimos 13 especies de bacterias de 11 géneros y dos phyla; mientras que en las librerías de genes ribosomales solamente logramos caracterizar 4 especies incluidas en 4 géneros y dos phyla (Fig. 9 y 14) (Cuadros 6 y 8). Este resultado no es inusual y se ha observado en otros ambientes (Dumbar et al., 1999). Particularmente, los aislados bacterianos del phylum Actinobacteria no se encontraron en las librerías de genes ribosomales. Sin embargo, el género Lactobacillus solamente fue encontrado mediante el uso de métodos moleculares. Una posible explicación para las diferencias observadas es el sesgo que pueden introducir por una parte las técnicas de extracción de DNA y la amplificación por PCR, y por otra parte los métodos de cultivo utilizados (medios y condiciones de incubación). En particular los métodos basados en el cultivo sobre representan las especies que crecen fácil y rápidamente en los medios sintéticos y no necesariamente son los mas abundantes de una comunidad. En este caso, se podrían aislar especies presentes en muy bajos números, lo cual los hace indetectables por métodos moleculares. Por estos motivos, los estudios que intentan describir de manera amplia la diversidad de especies en un ambiente deben incluir métodos basados en el cultivo e independientes del cultivo; dado que ambas estrategias se complementan y confirman. Los análisis de rarefacción de nuestros datos sugieren que la riqueza de especies presente en el tracto digestivo de D. valens es relativamente baja, sin embargo, todavía falta por analizarse parte de la comunidad presente en este ambiente ( Fig. 15, 16 y 17). Para poder llegar a una conclusión fiable de la riqueza de especies de una comunidad los análisis de rarefacción deben ser combinados con estimadores de la riqueza de especies (Hughes et al., 2001). Por lo tanto, también calculamos el estimador de la riqueza de especies no paramétrico Chao1, el cual es concordante con las curvas de rarefacción (Chao 1= 5 OTUs y Fig. 15). Estos datos coinciden con los obtenidos para D. frontalis en los cuales el estimador Chao 1 tuvo un valor de 1-3 OTUs en las librerías de larvas y de 3 y 6 OTUs en las librerías de adultos (Vasanthakumar et al., 2006) La PCR-DGGE de la región 3 variable del 16S rRNA demostró que la comunidad bacteriana presente en las diferentes partes del tracto digestivo de las hembras y de los machos de D. valens fue homogénea; sin embargo, se observan bandas comunes entre ellos y bandas exclusivas entre machos y hembras. La región con mayor diversidad microbiana fue el proctodeo de las hembras (Fig. 18), lo que sugiere que la comunidad bacteriana presente en el insecto no sobrepasa 12 especies bacterianas, lo cual de algún modo corrobora los resultados obtenidos mediante el cultivo y la librería de genes ribosomales. Asimismo, estos datos son consistentes con lo reportado en D. frontalis (Vasanthakumar et al., 2006). La comparación del patrón e intensidad de las bandas observadas en el DGGE de la comunidad total de las diferentes partes del canal alimentario y las cepas aisladas (Fig. 19), permite inferir que E. aerogenes es una población abundante en el proctodeo de las hembras y que R. aquatilis es una bacteria que se encuentra ampliamente distribuida en el tracto digestivo de D. valens. Durante las repeticiones del DGGE se observó que en la mayoría de los geles la región que constantemente mostraba el mayor número de bandas era el proctodeo tanto en hembras como en machos. También se observó que algunas bandas aparecían esporádicamente y existían pequeñas variaciones en el patrón desplegado por las diferentes secciones del canal alimentario tanto de hembras como de machos, lo que posiblemente indica que la estructura poblacional que albergan estos insectos puede cambiar durante el transcurso del tiempo o que hay un cierto grado de variación en la comunidad de especies bacterianas en este hábitat (Datos no mostrados). Sin embargo, en general no se observaron diferencias substanciales en la comunidad bacteriana total de las diferentes partes del canal alimentario, sexos y localidades de muestreo. En la librería de genes ribosomales identificamos las siguientes especies bacterianas: R. aquatilis, Pantoea dispersa, Acinetobacter schindleri y Lactococcus lactis (Fig. 14 y Cuadro 8). Solamente las bacterias del género Lactococcus no se encontraron mediante técnicas basadas en el cultivo. La presencia de bacterias del género Lactococcus se ha documentado en diversos artrópodos (Bauer et al., 2000; Peloquin y Greenberg, 2003). La presencia de bacterias lácticas, entre ellas las del género Lactococcus, se ha demostrado extensamente en el proctodeo de las termitas (Eutick et al., 1978; Tholen et al., 1997; Schultz y Breznak, 1978). En el canal alimentario de los mamíferos las bacterias lácticas son importantes colonizadores del epitelio intestinal (Tannock, 1997) y gracias a su actividad antagonista contra otras bacterias se ha descrito que participan en la defensa del tejido epitelial contra la invasión de patógenos (Rolfe, 1997). En las termitas las bacterias lácticas reducen el oxígeno y desplazan los procesos de fermentación hacia lactato y potencialmente pueden aumentar la obtención de energía (Condon, 1987). Otra ventaja selectiva de las bacterias lácticas asociadas a las termitas es su capacidad de metabolizar el ácido úrico (Potrikus y Breznak; 1980, 1981). Con las evidencias anteriormente mencionadas, se puede proponer que posiblemente los microorganismos del género Lactococcus participan en el mantenimiento del equilibrio entre las poblaciones bacterianas del tracto digestivo y en la mejora de la ganancia energética producto de la fermentación de los azúcares liberados de la degradación de la celulosa. Asimismo, esta bacteria puede contribuir en el reciclaje del ácido úrico, lo cual aumenta la eficiencia del uso de los compuestos nitrógenados, los cuales constituyen uno de los factores limitantes para el desarrollo de los insectos que se alimentan de la albura y el floema de las plantas. El contenido de nitrógeno en el tejido de las plantas es muy bajo si lo comparamos con el de los tejidos de los herbívoros. Como consecuencia, el nitrógeno de la dieta puede limitar el crecimiento y reproducción de los herbívoros y seleccionar los atributos que incrementen su adquisición (Mattson, 1980). La supervivencia y crecimiento de muchos artrópodos con dietas que presentan una relación C:N muy alta sugiere que estos insectos no obtienen suficiente nitrógeno de su alimento y que deben obtener compuestos nitrogenados de otras fuentes. Particularmente, los insectos del género Dendroctonus se alimentan del floema de las plantas, un sustrato que posee un bajo contenido de compuestos nitrogenados. Debido a lo anterior los escarabajos de éste género requieren de 16 a 26 veces más nitrógeno que el presente en los tejidos vegetales de los cuales se alimentan. Uno de los posibles mecanismos mediante el cual los insectos pueden subsanar esta deficiencia es la capacidad de fijar nitrógeno asociada a las bacterias de su canal alimentario. Lo anterior se ha demostrado en una amplia variedad de insectos como termitas, cucarachas, escarabajos descortezadores, escarabeidos, la mosca mediterránea de la fruta y el escarabajo venado (Behar et al., 2005; Benemann, 1973; Breznak et al., 1973; Breznak et al., 1974; Bridges, 1981; Citernesi et al., 1977; Kuranouchi et al., 2006). La presencia de las bacterias que potencialmente tienen capacidad para fijar nitrógeno dentro del canal alimentario del insecto, como R. aquatilis, Stenotrophomonas maltophilia y Pantoea spp.; la reducción de acetileno in vivo de larvas e insectos adultos y de las bacterias aisladas del tracto digestivo (Datos no mostrados); y la presencia de los genes nifH y nifD en el DNA metagenómico del canal alimentario de D. valens apunta a que la fijación biológica de nitrógeno se lleva acabo en el tracto digestivo de D. valens. En la literatura se reporta que R. aquatilis, Serratia, Pantoea agglomerans, Enterobacter spp. y S. maltophilia poseen la capacidad de fijar nitrógeno (Asis et al., 2004; Berge et al., 1991; Gyaneshwar 2001; Heulin et al., 1994; Park et al., 2005; Sandhiya et al., 2005; Wright et al., 1981), lo que sugiere de manera indirecta que posiblemente estos microorganismos contribuyen al aporte de nitrógeno a los insectos. La capacidad de reducir el acetileno se demostró in vivo en larvas e insectos adultos de D. valens (16.0 ± 2.2; 12.6 ± 3.4 nmol de etileno/h/insecto respectivamente), si se compara este valor con lo obtenido para otros coleópteros como el escarabajo venado, Dorcus rectus, en el cual se reporta 1.76 ± 0.52 nmol de etileno/h/larva (Kuranouchi, 2006), se tiene que la capacidad de reducir el acetileno de las larvas de D. valens es 9 veces mayor con respecto a la capacidad de reducir el acetileno de las larvas de Dorcus rectus. Ambos insectos se alimentan de sustratos pobres en nitrógeno. También estimamos que el nitrógeno que pueden fijar las larvas y los adultos de nuestro modelo de estudio equivale a 5.6 y 4.4 μg de proteína/día/insecto respectivamente. Los datos anterior son muy cercanos a lo reportado para la mosca mediterránea de la fruta, Ceratitis capitata, a la cual se le estimó que puede fijar el nitrógeno equivalente a 6 μg de proteína/día/insecto. El resultado anterior debe tomarse con cautela debido a que en repeticiones posteriores se obtuvieron magnitudes variables (Datos no mostrados). La fijación de nitrógeno se ha demostrado después de la ingesta masiva de bacterias fijadoras de nitrógeno como Klebsiella y Enterobacter spp. en insectos mantenidos por un periodo prolongado de tiempo en condiciones del laboratorio (Murphy et al., 1994). La alta variación en la magnitud de la reducción del acetileno podría deberse a la composición de la comunidad bacteriana asociada al canal alimentario del insecto; a la edad y actividad fisiológica de los adultos colectados; al lugar y temporada de muestro; a la etapa particular del ciclo de vida del insecto; al alimento tomado; y al propio árbol huésped del que provienen los insectos, entre otras variables. También hemos demostrado la presencia de los genes nifH y nifD en el DNA metagenómico de las diferentes regiones anatómicas del canal alimentario de D. valens (Ueda et al., 1995; Zerh y McReynolds, 1989). Ambos genes son ampliamente utilizados como marcadores que demuestran la presencia y permiten estimar la presencia, diversidad y filogenia de estos genes, y por ende de bacterias fijadoras de nitrógeno, en diferentes entornos. Los datos obtenidos son evidencias directas e indirectas de que la fijación biológica de nitrógeno se está llevando a acabo in vivo en algunos momentos del ciclo de vida de D. valens. Para elucidar totalmente la capacidad de fijación de nitrógeno in vivo asociada a D. valens son necesarios estudios diseñados para específicamente resolver las preguntas que se mantienen vigentes. CONCLUSIONES Se identificaron 14 bacterias heterótrofas aerobias cultivables Cuyas especies son: Rahnella aquatilis, Serratia liquefaciens, S. proteamaculans, Acinetobacter lwoffii, A. haemolyticus, Stenotrophomonas maltophilia, Pantoea cedenensis, Erwinia mallotivora, Erwinia/Pantoea sp., Enterobacter aerogenes, Janibacter melonis, Leifsonia Cellulosimicrobium cellulans. shinshuensis, Cellulomonas xylanilytica y En las librerías de genes ribosomales identificamos 4 especies bacterianas cuya identificación correspondió a: Lactococcus lactis, Acinetobacter schindleri, Rahnella aquatilis y Pantoea dispersa. Los genes nifH y nifD fueron detectados en el DNA metagenómico de las diferentes regiones anatómicas del canal alimentario de D. valens Los genes amoA, 16S rRNA de Nitrobacter, nirK, nirS y nosZ no fueron detectados en el DNA metagenómico de las diferentes regiones anatómicas del canal alimentario de D. valens Las larvas e insectos adultos de D. valens llevan a cabo fijación de nitrógeno. Cellulosimicrobium cellulans, Cellulomonas xylanilytica, Leifsonia shinshuensis y Janibacter melonis tienen capacidad de degradar la celulosa como única fuente de carbono. IMPACTO Este trabajo es estrictamente básico, su trascendencia principal será en la aportación de información científica y formación de estudiantes de licenciatura y posgrado. Sin embarbo, el entender el papel de la simbiosis de Dendroctonus con sus simbiontes microorganismos puede contribuir al entendimiento del ciclo de vida, adaptación y sobrevivencia de este insecto descortezados que se constituye en una plaga de los bosques de pinos en México, con el consecuente impacto económico y ecológico. No se derivarán directamente de los resultados informados aquí estrategias claras de control de la plaga, pero si revelamos la importancia que tienen, en la biología del insecto, las bacterias simbiontes. Posiblemente estos microorganismos contribuyen a que el insecto sobreviva en un ambiente químicamente hostiles, a que su comunicación química ocurra de manera eficiente y a que complemente su dieta que sabemos es muy pobre. Cualquier factor o estrategia que limite o afecte la comunidad bacteriana del insecto tendrá graves consecuencias sobre la sobrevivencia de este insecto y se constituirá en una herramienta para de control de este insecto plaga. BIBLIOGRAFIA 1. Adams, L. y R. Boopathy 2005 Isolation and characterization of enteric bacteria from the hindgut of Formosan termite. Bioresour. Technol. 96: 1592-1598. 2. Ahmadjian, V., y S. Paracer. 1986. Symbiosis: An introduction to biological associations. University Press of New England. 3. Altschul, S.F., T.L. Madden, A.A. Schäffer, J. Zhang, Z. Zhang, W. Miller, y D.J. Lipman. 1997. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs, Nucleic Acids Res. 25:3389-3402. 4. Anklin-Mϋhlemann, R., D.E. Bignell, P.C. Veivers, R.H Leuthold y. M. Slaytor. 1995. Morphological, microbiological and biochemical studies of the gut flora in the fungus-growing termite Macrotermes subhyalinus. J. Insect Physiol. 41:929–940. 5. Asis C.A. Jr y K. Adachi. 2004. Isolation of endophytic diazotroph Pantoea agglomerans and nondiazotroph Enterobacter asburiae from sweetpotato stem in Japan. Lett. Appl. Microbiol. 38:19-23. 6. Ayres, M.P. y S.F. MacLean, Jr. 1987. Development of birch leaves and the growth energetics of Epirrita autumnata (Geometridae). Ecology 68:558-568. 7. Ayres, M.P., R.T. Wilkens, C.J. Ruel, M.J. Lombardero y E. Vallery. 2000. Nitrogen budges of phloem-feeding bark beetles with and without symbiotic fungi. Ecology. 81:2198-2210. 8. Batra, L.R. 1963. Ecology of ambrosia fungi and their dissemination by beetles. Trans. Kansas. Acad. Sci. 66:213-236. 9. Barras, S.J. 1973. Reduction of progeny and development in the southern pine beetle following removal of symbiotic fungi. Can. Entomol. 105:1295-1299. 10. Barras, S.J. y T.J. Perry. 1975. Interrelationships among microorganisms, bark or ambrosia beetles, and woody plant tissue: an annotated bibliography, 1965-1974. United States Department of Agriculture Forest Service, Southern Forest Experiment Station, General Technical Report SO-10. 11. Bauer, S., A. Tholen, J. Overmann y A. Brune. 2000. Characterization of abundance and diversity of lactic acid bacteria in the hindgut of wood- and soil-feeding termites by molecular and culture- dependent techniques. Arch Microbiol 173:126-137. 12. Beaver, R.A. 1986. The taxonomy, mycangia, and biology of Hypothenemus curtipennis (Schedl), the first know cryphaline ambrosia beetle (Coleoptera: Scolytinae). Entomol. Scand. 17: 131-135. 13. Becker, G. 1971. Physiological influences on wood-destroying insects of wood compounds and substances produced by microorganisms. Wood Sci. Tech. 5:236-246. 14. Behar, A., B. Yuval, y E. Jurkevitch. 2005. Enterobacteria-mediated nitrogen fixation in natural populations of the fruit fly Ceratitis capitata. Mol. Ecol. 14:2637-2643. 15. Benemann J.R. 1973. Nitrogen fixation in termites. Science 181: 164-165. 16. Berge, O., T. Heulin, W. Achouak, C. Richard, R. Bally y J. Balandreau. 1991. Rahnella aquatilis, a nitrogen-fixing enteric bacterium associated with the rhizosphere of wheat and maize. Can. J. Microbiol. 37:195-203. 17. Bignell, D.E., H. Oskarsson y J.M Anderson.1980. Distribution and abundance of bacteria in the gut of a soil-feeding termite Procubitermes aburiensis (Termitidae, Termitinae). J. Gen. Microbiol. 117:393–403. 18. Bignell, D.E. 2000 Introduction to symbiosis. In: Abe, T., D.E. Bignell, M. Higashi (Eds), Termites: Evolution, Sociality, Symbioses, Ecology. Kluwer Academic. Dordrect, Netherlands. 19. Bishop, P.E. y R.D. Joerger. 1990. Genetics and molecular-biology of alternative nitrogenfixation systems. Annu. Rev. Plant Physiol. Plant Mol. Biol. 41:109–125. 20. Bohannan, B.J.M. y J. Hughes. 2003. New approaches to analyzing microbial biodiversity data. Curr. Opin. Microbiol. 6:282-287. 21. Braker, G., A. Fesefeldt y K.P. Witzel. 1998. Development of PCR primer systems for amplification of nitrite reductase genes (nir K and nirS) to detect denitrifying bacteria in environmental samples. Appl. Environ. Microbiol. 64:3769-3775. 22. Brand, J.M., J.W. Bracke, A.J. Markovetz, D.L. Wood y L.E. Browne. 1975. Production of verbenol pheromone by a bacterium isolated from bark beetles. Nature 254:136–137. 23. Breznak, J.A., W.J. Brill, J.W. Mertins y H.C. Coppel. 1973. Nitrogen fixation in termites. Nature 244:577-580. 24. Breznak J.A., J.W Mertins y H.C. Coppel. 1974. Nitrogen fixation and methane production in wood-eating cockroach, Cryptocercus punctulatus Scudder (Orthoptera: Blattidae). Univ. Wis. For. Res. Notes 184, Madison, WI. 25. Breznak, J.A. y H.S: Pankratz. 1977. In situ morphology of the gut microbiota of wood-eating termites [Reticulitermes flavipes (Kollar) and Coptotermes formosamus Shiraki]. Microbiol. 33: 406-426. 26. Breznak, J.A. 1982 Intestinal microbiota of termites and other xylophagous insects. Ann. Rev. Microbiol. 36: 323-343. 27. Breznak, J.A. 2000 Ecology of prokaryotic microbes in the guts of wood- and litter-feeding termites. In: Abe, T., D.E. Bignell, M. Higashi (Eds), Termites: Evolution, Sociality, Symbioses, Ecology. Kluwer Academic. Dordrect, Netherlands. 209-231. 28. Bridges, J.R. 1981. Nitrogen-Fixing Bacteria Associated with Bark Beetles. Microb. Ecol. 7:131-137. 29. Bridges, J.R., J.E. Marler y B.H. McSparrin. 1984. A quantitative study of the yeasts and bacteria associated with laboratory-reared Dendroctonus frontalis Zimm. (Coleopt., Scolytidae). Sonderdruck aus Bd. 97:261-267. 30. Broderick N.A., K.F. Raffa, R.M. Goodman y J. Handelsman. 2004. Census of the bacterial community of the gypsy moth larval midgut by using culturing and culture-independent methods. Appl. Environ. Microbiol. 70: 293–300. 31. Boucher, D.H., S. James y K.H. Keeler. 1982. The ecology of mutualism. A. Rev. Ecol. Syst. 13:315-347. 32. Buchner, P. 1965. Endosymbionts of animals with plant microorganisms. John Wiley y Sons, Inc., New York, N.Y. 33. Bürgmann, H., F. Widmer, W.V. Sigler, y J. Séller. 2004 New molecular screening tools for analysis of free-living diazotrophs in soil. Appl. Environ. Microbiol. 70:240-247. 34. Campbell, B.C. 1989. On the role of microbial symbiotes in herbivorous insects. En E. Bernays (ed.), Insect-plant interactions. CRC Press, Boca Raton, Fla. 35. Campbell, C.L., D.L. Mummey, E.T. Schmidtmann y W.C. Wilson. 2004. Culture-independent analysis of midgut microbiota in the arbovirus vector Culicoides sonorensis (Diptera: Ceratopogonidae). J. Med. Entomol. 41:340–348. 36. Cankar, K., H. Kraigher, M. Ravnikar y M. Rupnik. 2005. Bacterial endophytes from seeds of Norway spruce (Picea abies L. Karst). FEMS Microbiol. Lett. 244: 341-345. 37. Capuzzo, C., G. Firrao, L. Mazzon, A. Squartini y V. Girolami. 2005. ‘Candidatus Erwinia dacicola’, a coevolved symbiotic bacterium of the olive fly Bactrocera oleae (Gmelin). Int J. Syst Evol Microbiol 55: 1641–1647. 38. Cazemier, A.E., J.H.P. Hackstein, H.J.M. OpdenCamp, J. Rosenberg y C. van der Drift. 1997. Bacteria in the intestinal tract of different species of arthropods. Microb. Ecol. 33:189-197. 39. Cibrián, D.T., J.T. Mendez, R. Campos, H.O. Yates, y J.E. Flores. 2000. Insectos forestales de México. Universidad Autónoma Chapingo. 40. Citernesi U, R. Neglia, A. Seritti, A.A. Lepidi, C. Gilippi, G. Bagnoli, M.P. Nuti y R. Galluzzi. 1977. Nitrogen fixation in the gastro-enteric cavity of soil animals. Soil Biol. Biochem. 9:71–72. 41. Clancy, K.M. 1992. Response of western spruce budworm (Lepidoptera: Tortricidae) to increased nitrogen in artificial diets. Environ. Entomol. 21:331-344. 42. Clancy, K.M., M.R. Wagner y R.W. Tinus. 1988. Variation in host foliage nutrient concentrations in relation to western spruce budworm herbivory. Can. J. For. Res. 18:530-544. 43. Condon, S. 1987. Responses of lactic acid bacteria to oxygen. FEMS Microbiol Rev 46:269– 280. 44. Coulson, R.N. 1979. Population dynamics of bark beetles. Annu. Rev. Entomol. 24:417-477. 45. Cruden, D.L. y A.J. Markovetz. 1979. CarboxymethylCellulose decomposition by intestinal bacteria of cockroaches. Appl. Eviron. Microbiol. 38:369-372. 46. Cruden, D.L. y A.J. Markovetz. 1984. Microbial aspects of the cockroach hindgut. Arch Microbiol. 138:131-139. 47. Cruden, D.L. y A.J. Markovetz. 1987. Microbial ecology of the cockroach gut. Ann. Rev. Microbiol. 41:617–643. 48. Cullen D.W. y P.R. Hirsch. 1988. Simple and rapid method for direct extraction of microbial DNA from soil to PCR. Soil Biol Biochem 30:983-993. 49. de Bary, A.H. 1878 Vortrag: Über Symbiose. En Versammlung deutscher naturforscher und arzte En Cassel Kassel: Baier & Lewalter. 50. de Vries, E.J., G. Jacobs, M.W. Sabelis, S.B.J. Menken y J.A.J. Breeuwer. 2004 Diet- dependent effects of gut bacteria on their insect host: the symbiosis of Erwinia sp. and western flower thrips. Proc. R. Soc. Lond. 271:2171-2178. 51. Delalibera, I., J. Handelsman y K.F. Raffa. 2005. Contrasts in cellulolytic activities of gut microorganisms between the wood borer, Saperda vestita (Coleoptera : Cerambycidae), and the bark beetles, Ips pini and Dendroctonus frontalis (Coleoptera : Curculionidae). Environ. Entomol. 34:541-547. 52. Díaz, E., R. Cisneros, G. Zuñiga y E. Uria-Galicia. 1998 Comparative anatomical and histological study of the alimentary canal of Dendroctonus parallelocollis, D. rhizophagus, and D. valens (Coleoptera: Scolytidae). Ann. Entomol. Soc. Am. 91:479-487. 53. Díaz, E., O. Arciniega, L. Sánchez, R. Cisneros y G. Zúniga. 2003 Anatomical and histological comparison of the alimentary canal of Dendroctonus micans, D. ponderosae, D. pseudotsugae pseudotsugae, D. rufipennis, and D. terebrans (Coleoptera: Scolytidae). Ann. Entomol. Soc. Am. 96:144-152. 54. Dillon, R.J. y V.M. Dillon. 2004. The gut bacteria of insects: nonpathogenic interactions. Annu. Rev. Entomol. 49:71-92. 55. Douglas, A.E. 1989. Mycetocyte symbiosis in insects. Biol. Rev. 64:409-434. 56. Douglas, A.E. 1998. Nutritional interactions in insect-microbial symbioses: Aphids and their symbiotic bacteria Buchnera. Annu. Rev. Entomol 42:17-37. 57. Douglas A. E. 2006. Phloem-sap feeding by animals: problems and solutions. J. Exp. Bot. 57:747–754. 58. Dunbar, J., S. Takala, S.M. Barns, J.A. Davis y C.R. Kuske. 1999. Levels of bacterial community diversity in four arid soils compared by cultivation and 16S rRNA gene cloning. Appl. Environ. Microbiol. 65:1662-1669. 59. Dunn, A.K. y E.V. Stabb. 2005. Culture-independent characterization of the microbiota of the ant lion Myrmeleon mobilis (Neuroptera: Myrmeleontidae). Appl. Environ. Microbiol. 71:87848794. 60. Eckburg, P.B., E.M. Bik, C.N. Bernstein, E. Purdom, L. Dethlefsen, M. Sargent, S.R. Gill, K.E. Nelson y D.A. Relman. 2005. Diversity of the human intestinal microbial flora. Science 308:1635-1638. 61. Egert, M., B. Wagner, T. Lemke, A. Brune y M.W. Friedrich. 2003 Microbial community structure in midgut and hindgut of the humus-feeding larva of Pachnoda ephippiata (Coleoptera: Scarabaeidae). Appl. Environ. Microbiol. 69:6659–6668. 62. Egert, M, U. Stingl, L.D. Bruun, B. Pommerenke, A. Brune y M.W. Friedrich. 2005 Structure and topology of microbial communities in the major gut compartments of Melolontha melolontha larvae (Coleoptera: Scarabaeidae). Appl. Environ. Microbiol. 71:4556-4566. 63. Ekperigin, M.M. 2007 Preliminary studies of cellulase production by Acinetobacter anitratus and Branhamella sp. Afr. J. Biotechnol. 1: 028-033. 64. Eutick, M.L., P. Veivers, R.W. O’Brien y M. Slaytor. 1978. Dependence of the higher termite, Nasutitermes exitiosus and the lower termite, Coptotermes lacteus on their gut flora. J. Insect Physiol. 24:363-368. 65. Falcon, L.I., F. Cipriano, A.Y. Chistoserdov y E.J. Carpenter. 2002. Diversity of diazotrophic unicellular cyanobacteria in the tropical North Atlantic Ocean. Appl. Environ. Microbiol. 68: 5760–5764. 66. Felsestein J. 1989. PHYLIP-Phylogeny inference package version 3.5c. Cladistic 5:164-166. 67. Ferrari, M.J., D. Du, J.A. Winsor y A.G. Stephenson. 2007. Inbreeding depression of plant quality reduces incidence of an insect-borne pathogen in a wild gourd. Int. J. Plant Sci. 168:603–610. 68. Ferrer, P., T. Halkier, L. Hedegaard, D. Savva, I. Diers y J.A. Asenjo. 2006. Nucleotide sequence of a 13-1,3-glucanase Isoenzyme II(A) gene of Oerskovia xanthineolytica LL G109 (Cellulomonas cellulans) and initial characterization of the recombinant enzyme expressed in Bacillus subtilis. J. Bacteriol. 178:4751-4757. 69. Fogelsong, M.A., D.H. Walker, F.S. Puffer y A.J. Markovetz. 1975. Ultrastructural morphology of some prokaryotic microorganisms associated with the hindgut of cockroaches. J. Bacteriol. 123:336-345. 70. Forcella, F. 1981. Twig nitrogen content and larval survival of twig girdling beetles Oncideres cingulata (Coleoptera: Cerambycidae). Coleopterists Bulletin 35:211-212. 71. Francis C.A., J.M. Beman y M.M.M. Kuypers. 2007. New processes and players in the nitrogen cycle: the microbial ecology of anaerobic and archaeal ammonia oxidation. ISME J. 1:19-27. 72. Galtier, N., M. Gouy, y C. Gautier. 1996. SEAVIEW and PHILO_WIN: two graphic tools for sequence alignment and molecular phylogeny. Comput. Appl. Biosci. 12:543-548. 73. Gill, S.R., M. Pop, R.T. DeBoy, P.B. Eckburg, P.J. Turnbaugh, B.S. Samuel, J.I. Gordon, D.A. Relman, C.M. Fraser-Liggett y K. E. Nelson. 2006. Metagenomic analysis of the human distal gut microbiome. Science. 312:1355-1359. 74. Gil, R., A. Latorre y A. Moya 2004. Bacterial endosymbionts of insects: insights form comparative genomics. Environ. Microbiol. 6:1109-1122. 75. Gijzen, H.J. y M. Barugahare. 1992. Contribution of anaerobic protozoa and methanogens to hindgut metabolic activities of the American cockroach, Periplaneta Americana. Appl. Environ. Microbiol. 58:2565-2570. 76. Grenier, A.M., G. Duport, S. Pagès, G. Condemine y Y. Rahbé. 2006 The phytopathogen Dickeya dadantii (Erwinia chrysanthemi 3937) is a pathogen of the pea aphid. Appl. Environ. Microbiol. 72:1956-1965. 77. Gyaneshwar P, E.K. James, N. Mathan, P.M. Reddy, B. Reinhold-Hurek y J.K. Ladha. 2001 Endophytic colonization of rice by a diazotrophic strain of Serratia marcescens. J. Bacteriol.183:2634-45. 78. Harada, H. y H. Ishikawa. 1997. Experimental pathogenicity of Erwinia aphidicola to pea aphid, Acyrthosiphon pisum. J. Gen. Appl. Microbiol. 43:349-354. 79. Henry, S.M. 1962. The significance of microorganisms in the nutrition of insects. Transactions of the New York Academy of Sciences, Series II 24:676-683. 80. Heulin, T., O. Berge, P. Mavingui, L. Gouzou, K.P. Hebbar y J. Balandreau. 1994. Bacillus polymyxa and Rahnella aquatilis, the dominant N2-fixing bacteria associated with wheat rhizosphere in French soils. Eur. J. Soil. Biol. 30:35-42. 81. Hodges, J.E. y P.L. Lorio, Jr. 1969. Carbohydrate and nitrogen fractions of the inner bark of loblolly pines under moisture stress. Can. J. Bot. 47:1651-1657. 82. Holt, J.G., N.R. Krieg, P.H.A. Sneath, J.T. Staley y S.T. Williams. 1994. Bergey’s manual of determinative. Bacteriology. 9th ed. Editors.Williams and Wilkins. Baltimore, Maryland USA. 83. Hsiau, P.T-W. y T.C. Harrington. 2003. Phylogenetics and adaptations of basidiomycetous fungi fed upon by bark beetles (Coleoptera:Scolytidae). Symbiosis. 34:111-131. 84. Hughes, J.B., J.J. Hellmann, T.H. Ricketts y B.J.M. Bohannan. 2001. Counting the uncountable: statistical approaches to estimating microbial diversity. Appl. Environ. Microbiol. 67:4399-4406. 85. Hungate, R.E. 1966. The rumen and its Microbes. Academic Press, New York. 86. Hunt, D.W.A. y J.H. Borden. 1990. Conversion of verbenols to verbenone by yeasts isolated from Dendroctonus ponderosae (Coleoptera: Scolytidae). J. Chem. Ecol. 16:1386-1397. 87. Hurst, M.R.H., Jones S.M., B. Tan y T.A. Jackson. 2007 Induced expression of the Serratia entomophila Sep proteins shows activity towards the larvae of the New Zealand grass grub Costelytra zealandica. FEMS Microbiol. Lett. 275:160-167. 88. Ishikawa, H. 2003. Insect simbiosis: An introduction. En Insect symbiosis. Bourtzis, K., and T.A. Miller. CRS Press. 89. Izumi, H., I.C. Anderson, I.J. Alexander, K. Killham y E.R.B. Moore. 2006 Endobacteria in some ectomycorrhiza of Scots pine (Pinus sylvestris) FEMS Microbiol. Ecol. 56:34-43. 90. Jeffries, T.W. y Y.-S. Jin. 2000. Ethanol and thermotolerance in the bioconversion of xylose by yeasts Adv. Appl. Microbiol. 47:221-268. 91. Kadavy D.R., B. Plantz, C.A. Shaw, J. Myatt, T.A. Kokjonh y K.W. Nickerson. 1999. Microbiology of the oil fly, Helaeomyia petrolei. Appl. Environ. Microbiol. 65: 477-1482. 92. Karlsson, J., D. Momcilovic, B. Wittgren, M. Schϋlein. F. Tjerned y G. Brinkmalm. 2002. Enzymatic degration of carboxymethyl cellulose hydrolyzed by the endoglunases Cel5A, Cel7B, and Cel45Acore from Trichoderma reesei. Biopolymers. 63:32-40. 93. Kaufman, M.G., E.D. Walker, D.A. Odelson y M.J. Klug. 2000. Microbial community ecology and insect nutrition. Am. Entomol. 46:173-184. 94. Kneip C., P. Lockhart, C. Voss y U.G. Maier. 2007 Nitrogen fixation in eukaryotes – New models for symbiosis BMC Evol. Biol. 7:55-66. 95. Krokene, P. y H. Solheim. 1998. Pathogenicity of four blue stain fungi associated with aggressive and nonaggressive bark beetles. Phytopathology. 88:39-44. 96. Kuranouchi, T., T. Nakamura, S. Shimamura, H. Kolima, K. Goka, K. Okabe y A. Mochizuki. 2006 Nitrogen fixation in the stag beetle, Dorcus (Macrodorcus) rectus (Motschulsky) (Col., Lucanidae). J. Appl. Entomol. 130:471-472. 97. Lammers, P.J. y R. Haselkorn. 1984. Sequence of the nifD geneogy coding for the alpha subunit of dinitrogenase from the cyanobacterium Anabaena. Cell 44:905–911. 98. Levieux, J., P. Cassier, L. Guillaumin y A. Roques. Structures implicated in the transportation of pathogenic fungi by the European bark beetle, Ips sexdentatus Boerner: ultrastructure of a mycangium. Can. Entomol. 123:245-254. 99. Lim, Y.W., S.M. Alamounti, J.J. Kim y C. Breuil. 2004. Multigene phylogenies of Ophiostoma clavigerum and closely related species from bark beetle-attacked Pinus in North America. FEMS Microbiol. Lett. 237:89-96. 100. Lim, Y.W., J.J. Kim, M. Lu y C. Breuil. 2005. Determining fungal diversity on Dendroctonus ponderosae and Ips pini affecting lodgepole pine using cultural and molecular methods. Fungal Divers. 19:79-94. 101. Liu, X., S.M. Tiquia, G. Holguin, L. Wu, S.C. Nold, A.H. Devol, K. Luo, A.V. Palumbo, J.M. Tiedje, y J. Zhou. 2003. Molecular diversity of denitrifying genes in continental margin sediments within the oxygen-deficient zone off the Pacific coast of Mexico. Appl. Environ. Microbiol. 69:3549-3560. 102. Lundgren, J.G., R.M. Lehman y J. Chee-Sanford. 2007 Physiology, biochemistry, and topology: Bacterial communities within digestive tracts of ground beetles (Coleoptera: Carabidae). Ann. Entomol. Soc. Am. 100:275-282. 103. Maier, R.M., I.L. Pepper y C.P. Gerba. 1999 Environmental microbiology. Academic Press. 104. Martin, M.M. 1979. Biochemical implications of insect mycophagy. Biol. Rev. 54:1-21. 105. Mazur, B.J. y C.F. Chui, 1982. Sequence of the gene coding for the beta-subunit of dinitrogenase from the blue-green alga Anabaena. Proc. Natl. Acad. Sci. U. S. A. 79:6782-6786. 106. Mattson, W.J. 1980. Herbivore in relation to plant nitrogen content. Ann. Rev. Ecol. Syst. 11:119-161. 107. Mevarech, M., D. Rice y R. Haselkorn. 1980. Nucleotide sequence of a cyanobacterial nifH gene coding for nitrogenase reductase. Proc. Natl. Acad. Sci. U. S. A. 77:6476–6480. 108. Miller, B. 2005. OpenStat user's manual, portable data file (OS2MANUAL.PDF), http://statpages.org/miller/openstat/ , 2003. 109. Mittler, T.E. 1988. Applications of artificial feeding techniques for aphids. En A.K. Minks and P. Harrewijn (ed.), Aphids: their biology, natural enemies, and control. Elsevier Biomedical Press, Amsterdam, TheNetherlands. 110. Monttlor Curley, C., E.L Brodie, M.G. Lechner y A.H. Purcell. 2007 Exploration for facultative endosymbionts of glassy-winged sharpshooter (Hemiptera: Cicadellidae). Ann. Entomol. Soc. Am. 100:345-349. 111. Moore, G.E. 1972 (a). Microflora from the alimentary tract of healthy southern pine beetles, Dendroctonus frontalis (Scolytidae), and their possible relationship to pathogenicity. J. Invertebr. Pathol. 19:72-75. 112. Moore, G.E. 1972 (b). Pathogenicity of ten strains of bacteria to larvae of the southern pine beetle [Dendroctonus frontalis]. J. Invertebr. Pathol. 20:41-45. 113. Moran, N.A., P. Tran y N.M. Gerardo. 2005 Symbiosis and insect diversification: an ancient symbiont of sap-feeding insects from the bacterial phylum Bacteroidetes. Appl. Environ. Microbiol. 12:8802-8810. 114. Mramba, F., A. Broce y L Zurek 2006. Isolation of Enterobacter sakazakii from stable flies, Stomoxys calcitrans L. (Diptera: Muscidae). J. Food. Prot. 69:671-673. 115. Mulder A, A.A. Graaf, L.A. Robertson y J.G. Kuenen. 1995. Anaerobic ammonium oxidation discovered in a denitrifying fluized bed reactor. FEMS Microbiol. Ecol. 16:177-184. 116. Muller, H.E., G.R. Fanning y D.J. Brenner. 1995a. Isolation of Ewingella americana from mollusks. Curr. Microbiol. 31:287-290. 117. Muller, H.E., G.R. Fanning, y D.J. Brenner. 1995b. Isolation of Serratia fonticola from mollusks. Syst. Appl. Microbiol. 18:279-284. 118. Murphy K.M., D.S. Teakle y I.C. Macrae. 1994. Kinetics of colonization of adult Queensland fruit-flies Bactrocera tryoni by dinitrogenfixing alimentary-tract bacteria. Appl. Environ. Microbiol. 60:2508–2517. 119. Muyzer G., E.C. De Waall y A.G. Vitterlinden. 1993 Profiling of complete microbial populations by denaturing gradient gel electrophoresis analysis of poliymerase chain reactionamplified genes coding for 16S rDNA. Appl Environ Microbiol 59:695-700. 120. Muyzer, G. y K. Smalla 1998 Application of denaturing gradient gel electrophoresis (DGGE) and temperature gradient gel electrophoresis (TGGE) in microbial ecology. Antoine van Leeuwenhoek. 73:127-141. 121. Nardi, J.B., R.I. Mackie y J.O. Dawson. 2002. Could microbial symbionts of arthropod guts contribute significantly to nitrogen fixation in terrestrial ecosystems? J. Insect Physiol. 48:751763. 122. Noda, S., M. Ohkuma, R. Usami, K. Horikoshi, y T. Kudo. 1999. Culture-independent characterization of a gene responsible for nitrogen fixation in the symbiotic microbial community in the gut of the termite Neotermes koshunensis. Appl. Environ. Microbiol. 65:4935-4942. 123. Ohkuma, M., S. Noda y T. Kudo. 1999. Phylogenetic diversity of nitrogen fixation genes in the symbiotic microbial community in the gut of diverse termites. Appl. Environ. Microbiol 65:4926-4934. 124. Ohkuma, M. y T. Kudo. 1996. Phylogenetic diversity of the intestinal bacterial community in the termite Reticulitermes speratus. Appl. Environ. Microbiol. 62:461-468. 125. Paine, T.D. y M.C. Birch. 1983. Acquisition and main tenance of mycangial fungi by Dendroctonus brevicomis (Coleoptera Scolytidae). Environ. Entomol. 12:1384-1386. 126. Paine, T.D., K.F. Raffa, y T.C. Harrington. 1997. Interactions among scolytid bark beetles, their associated fungi, and live host conifers. Annu. Rev. Entomol. 42:179-206. 127. Park M., C. Kim, J.Yang, H. Lee, W. Shin., S. Kim y T. Sa. 2005. Isolation oand characterization of diazotrophic growth promoting bacteria from rhizosphere of agricultural crops of Korea. Microbiol. Res. 160:127-33. 128. Peloquin, J.J. y L. Greenberg. 2003. Identification of midgut bacteria from fourth instar red imported fire ant larvae, Solenopsis invicta buren (Hymenoptera: Formicidae) J. Agric. Urb. Entomol. 20:157-164. 129. Potrikus, C.J. y J.A. Breznak. 1980. Uric acid-degrading bacteria in guts of termites [Reticulitermes flavipes (Kollar)]. Appl Environ Microbiol 40:117-124. 130. Potrikus C.J. y J.A. Breznak. 1981. Gut bacteria recycle uric acid nitrogen in termites: a strategy for nutrient conservation. Proc. Natl. Acad. Sci. USA 78:4601-4605. 131. Purkhold, U., A. Pommerening-Röser, S. Juretschko, M.C. Schmid, H.P. Koops y M. Wagner. 2000. Phylogeny of all recognized species of ammonia oxidizers based on comparative 16S rRNA and amoA sequence analysis: Implications for molecular diversity surveys. Appl. Environ. Microbiol. 66:5368-5382. 132. Paster, B.J., S.K. Boches, J.L. Galvin, R.E. Ericson, C.N. Lau, V.A. Levanos, A. Sahasrabudhe y F.E. Dewhirst. 2001. Bacterial diversity in human subgingival plaque. J. Bact. 183:3370-3383. 133. Raffa, K.F., T.W. Phillips y S.M. Salom. 1993. Strategies and mechanisms of host colonization by bark beetles. EN T. Schowalter and G. Filip (eds.), Beetle pathogen interactions in conifer forests. Academic, San Diego, CA. 134. Raymond, J., J.L. Siefert, C.R. Staples y R.E. Blankenship. 2004. The natural history of nitrogen fixation. Mol. Biol. Evol. 21:541–554. 135. Reese, E.T., R.G.H. Siu y H.S. Levison. 1950. The biological degradation of soluble cellulose derivatives and its relationship to the mechanism of cellulose hydrolysis. J. Bacteriol. 59:485-497. 136. Relman, D.A. 1993. Universal bacterial 16S rRNA amplification and sequencing. En American Society of Microbiology (ed) Diagnostic molecular microbiology: principles and applications. Nueva York. 137. Rivas, R., M.E. Trujillo, P.F. Mateos, E. Martínez-Molina y E. Velázquez. Cellulomonas xylanilytica sp. nov., a cellulolytic and xylanolytic bacterium isolated from a decayed elm tree. Int. J. Syst. Evol. Microbiol. 54:533-536. 138. Robbins, C.T. 1983. Wildlife feeding and nutrition. Academic Press, New York, New York, USA. 139. Rolfe, R.D. 1997. Colonization resistance. En: Mackie R.I. y White B.A. (eds) Gastrointestinal microbiology, vol 2. Chapman and Hall, New York, USA. 140. Rosello-Mora R. y R. Amann. 2001. The species concept for prokaryotes. FEMS-Microbiol. Rev. 25:39-67. 141. Rotthauwe, J.H, K.P. Witzel y W. Liesack. The ammonia monooxigenase structural gene amoA as a functional marker: molecular fine-scale analysis of natural ammonia-oxidizing populations. Appl. Environ. Microbiol. 63:4704-4712. 142. Sandhiya G.S., T.C. Sugitha, D. Balachandar y K. Kumar. 2005 Endophytic colonization and in planta nitrogen fixation by a diazotrophic Serratia sp. in rice. Indian J Exp Biol. 43:802-807. 143. Sambrook J. y D.W. Russell. 2001. Molecular Cloning: a laboratory manual ·3rd Edition Cold Spring Harbor Laboratory Press. Cold Spring Harbor, New York. 144. Saxena, S., J. Bahadur y A. Varma. 1993. Cellulose and hemicelluloses degrading bacteria from termite gut and mound soils of India. Indian J. Microbiol. 33:55-60. 145. Scala, D., y L.J. Kerkhof. 1999. Diversity of nitrous oxide reductase (nosZ) genes in continental shelf sediments Appl. Environ. Microbiol. 65:1681-1687. 146. Schäfer, A. 1996. Hemicellulose-degrading bacteria and yeasts from the termite gut. J. Appl. Bacteriol. 80:471–478. 147. Schloss, P.D. y J. Handelsman. 2005. Introducing DOTUR, a computer program for defining operational taxonomic units and estimating species richness. Appl. Environ. Microbiol. 71:15011506. 148. Schloss, P.D., I. Delalibera, Jr., J. Handelsman y K.F. Raffa. 2006. Bacteria associated with the guts of two wood boring beetles: Anoplophora glabripennis and Saperda vestita (Cerambycidae). Environ. Entomol. 35:625-629. 149. Schowalter, T.D. 1981. Insect herbivore relationship to the state of the host plant: biotic regulation of ecosystem nutrient cycling through ecological succession. Oikos 37:126-130. 150. Schultz, J.E. y J.A. Breznak. 1978. Heterotrophic bacteria present in hindguts of wood eating termites [Reticulitermes flavipes (Kollar)]. Appl. Environ. Microbiol. 35:930-936. 151. Schumann, P., N. Weiss y E. Stackebrandt. 2001. Reclassification of Cellulomonas cellulans (Stackebrandt and Keddie 1986) as Cellulosimicrobium cellulans gen. nov., comb. nov. Int. J. Syst. Evol. Microbiol. 51: 1007-1010. 152. Scriber, J.M. y F. Slansky, Jr. 1981. The nutritional ecology of immature insects. Annu. Rev. Entomol. 26:183-211. 153. Sezen, K., I. Demir y Z. Demirbaǧ. 2006. Identification and pathogenicity of entomopathogenic bacteria from common cockchafer, Melolontha melolontha (Coleoptera: Scarabaeidae). New Zealand Journal of Crop and Horticultural Science 35:79-85. 154. Six, D.L. 2003. Bark beetle-fungus symbiosis. En Insect symbiosis. Bourtzis, K., and T.A. Miller. CRS Press. 155. Slansky, E 1993. Nutritional ecology - the fundamental quest for nutrients. En Caterpillars. N.E. Stamp y T.M. Casey, editors. Chapman & Hall, New York, New York, USA. 156. Slansky, F., Jr. y J.M. Scriber. 1985. Food consumption and utilization. En Comprehensive insect physiology. G.A. Kerkut y L.I. Gilbert, editors, biochemistry, and pharmocology. Pergamon, Oxford, UK. 157. Slansky, F. y G.S. Wheeler. 1992. Caterpillars compensatory feeding response to diluted nutrients leads to toxic allelochemical dose. Entomol. Exp. Appl. 65:171-186. 158. Steinhaus, E.A. 1959. Serratia marcescens Bizio as an insect pathogen. Hilgardia 28:351380. 159. Straif, S.C., C.N.M. Mbogo, A.M. Toure, E.D. Walker, M. Kaufman, Y.T. Toure y J.C. Beier. 1998. Midgut bacteria in Anopheles gambiae and An. funestus (Diptera: Culicidae) from Kenya and Mali. J. Med. Entomol. 35:222-6. 160. Tabashnik, B.E. 1982. Responses of pest and nonpest Colias butterfly larvae to intraspecific variation in leaf nitrogen and water content. Oecologia 55:389-394. 161. Tannock, G.W. 1997. Probiotic properties of lactic-acid bacteria: Plenty of scope for fundamental R and D. Trends. Biotechnol. 15:270–74. 162. Tholen, A., B. Schink y A. Brune. 1997. The gut microflora of Reticulitermes flavipes, its relation to oxygen, and evidence for oxygen-dependent acetogenesis by the most abundant Enterococcus sp. FEMS Microbiol. Ecol. 24:137-149. 163. Thompson, J.D., T.J. Gibson, F. Plewniak, F. Jeanmougin y D.G. Higgins. 1997. The ClustalX windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Res. 24:4876-4882. 164. Treusch A.H., S. Leininger, A. Kletzin, S.C. Schuster, H-P. Klenk y C Scheleper 2005 Novel genes for nitrite reductase and Amo-related proteins indicate a role of uncultivated mesophilic crenarchaeota in nitrogen cycling. Environ. Microbiol. 7:1985-1995. 165. Trier, T.M. y W.J. Mattson. 1997. Needle mining by the spruce budworm provides sustenance in the midst o f privation. Oikos 79:241-246. 166. Tokuda, G. y H. Watanabe 2007 Hidden cellulases in termites: revision of an old hypothesis. Biol. Lett. 3:336-339. 167. Ueda, T., Y. Suga, N. Yahiro y T. Matsuguchi. 1995. Remarkable N2-fixing bacterial diversity detected in rice roots by molecular evolutionary analysis of nifH gene sequences. J. Bacteriol. 177:1414-1417. 168. Ulrich, R., D. Buthula y M. Klug. 1981. Microbiota associated with the gastrointestinal tract of the common house cricket, Anacheta domestica. Appl. Environ. Microbiol. 41:246-254. 169. Varma, A., B.K. Kolli, J. Paul, S. Saxena y H. König. 1994. Lignocellulose degradation by microorganisms from termite’s hills and termite guts: a survey the present state of art. FEMS Microbiol. Rev. 15:9-28. 170. Vasanthakumar A., I. Delalibera, Jr, J. Handelsman, K.D. Klepzig, P.D. Schloss y K.F. Raffa. 2006 Characterization of gut-associated bacteria in larvae and adults of the southern pine beetle, Dendroctonus frontalis Zimmermann. Eviron. Entomol. 35: 1710-1717. 171. Venter J.C., K. Remington, J.F. Heidelberg, A.L. Halpern, D. Rusch y J.A. Eisen. 2004. Environmental genome shotgun sequencing of the Sargasso Sea. Science 304:66-74. 172. Vimala, A., A. Sree y M. Bapuji. 2004. Isolation and characterisation of Acinetobacter sp. from a marine sponge Axinella agariciformis. Asian J Microbiol. Biotechnol. Environ. Sci. 6:297300. 173. Vorwerk, S., D. Martinez-Torres y A. Forneck. 2007. Pantoea agglomerans-associated bacteria in grape phylloxera (Daktulosphaira vitifoliae, Fitch). Agric. For. Entomol. 9: 57-64. 174. Wagner, T.L., J.A. Gagne, P.J.H. Sharpe, y R.N. Coulson. 1984. A biophysical model of southern pine beetle, Dendroctonus frontalis Zimmermann (Coleoptera: Scolytidae), development. Ecol. Model. 21: 125-147. 175. Wallin, K.F. y K.F. Raffa. 2004. Feedback between individual host selection behavior and population dynamics in an eruptive insect herbivore. Ecol. Monogr. 74:101-116. 176. Wells, M.L., R.D. Gitaitis y F.H.Sanders. 2002 Association of tobacco thrips, Frankliniella fusca (Thysanoptera: Thripidae) with two species of bacteria of the genus Pantoea Ann. Entomol. Soc. Am. 95: 719-723. 177. Wenzel, M., I. Schönig, M. Berchtold, P. Kämpfer y H. König. 2002. Aerobic and facultatively anaerobic cellulolytic bacteria from the gut of the termite Zootermopsis angusticollis. J. Appl. Microbiol. 92: 32-40. 178. Werner, E., 1926. Die ernährung der larvae von Potosia cuprea Fbr. (Cetonia floricola Hbst.). En Beitrag zum problem der cellulosverdauung bei insectenlarven. Zeitschrift für Morphologie und Ökologie der Tiere 6: 150–206. 179. White, T.C.R. 1993. The inadequate environment: nitrogen and the abundance of animals. Springer-Verlag, New York, New York, USA. 180. Widmer, F., B.T. Shaffer, L.A. Porteuous y R.J. Seidler. 1999. Analysis of nifH gene pool complexity in soil and litter at a Douglas Fir forest site in the Oregon cascade mountain range. Appl. Environ. Microbiol. 65:374-380. 181. Wiedenmann, J.F., 1930. Die Zelluloseverdauung bei Lamellicornierlarven. Zeitschrift für Morphologie und Ökologie der Tiere 19: 228-258. 182. Williams, J.G.K., A.R. Kubelik, K.J. Livak, J.A. Rafalsky y S.V. Tingey. 1990. DNA polymorphism amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Res.18:6531-6535. 183. Wood, S.L. 1982. The bark and ambrosia beetle of North and Central America (Coleoptera: Scolytidae). A taxonomic monograph. Great Basin Nat. Mem. 184. Woolfolk, S.W. y G.D. Inglis. 2004. Microorganisms associated with field-collected Chrysoperla rufilabris (Neuroptera: Chrysopidae) adults with emphasis on yeast symbionts. Biol. Control. 29:155-168. 185. Wright S.F. y R.W. Weaver 1981. Enumeration and identification of nitrogen-fixing bacteria from forage grass roots Appl Environ Microbiol. 42:97–101. 186. Wu, L., D.K. Thompson, G. Li, R.A. Hurt, J.M. Tiedje y J. Zhou. 2001. Development and evaluation of functional gene arrays for detection of selected genes in the environment. Appl. Environ. Microbiol. 67:5780-5790. 187. Xia X. y Z. Xie 2001 DAMBE: Data analysis in molecular biology and evolution. J. Heredity 92:371-373. 188. Xiang, H., G.F. Wei, S. Jia, J. Huang, X.X. Miao, Z. Zhou, L.P. Zhao y Y.P. Huang. 2006. Microbial communities in the larval midgut of laboratory and field populations of cotton bollworm (Helicoverpa armigera). Can. J. Microbiol. 52:1085-1092. 189. Yang, Y. y A. Joern. 1994. Compensatory feeding in response to food quality by Melanopus differentiallis. Physiol. Entomol. 19:75-82. 190. Zehr, J.P., M. Mellon, S. Braun, W. Litaker, T. Steppe y H.W. Paerl. 1995. Diversity of heterotrophic nitrogen fixation genes in a marine cyanobacterial mat. Appl. Environ. Microbiol. 61:2527-2532.