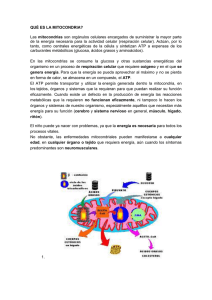
“Estudios sobre la participación de AtFH en la biogénesis de
Anuncio

Universidad Nacional de General San Martín UNSAM Instituto de Investigaciones Biotecnológicas Instituto Tecnológico de Chascomús IIB-INTECH-CONICET “Estudios sobre la participación de AtFH en la biogénesis de proteínas Fe-S y hemoproteínas en mitocondrias de plantas” Tesis para optar al título de Doctor en Biología Molecular y Biotecnología Bioquímica María Victoria Maliandi Director: Dr. Diego F. Gómez-Casati Co-Directora: Dra. MaríaVictoria Busi 2009 Indice Indice Indice I Agradecimientos VI Publicaciones VIII Abreviaturas IX 1. Introducción 1.1. Estructura y función de la mitocondria 2 1.2. Fosforilación oxidativa 5 1.3. Comunicación núcleo-mitocondria 11 1.4. Biogénesis de los grupos Fe-S 13 1.4.1. Biogénesis de grupos Fe-S en plantas 17 1.5. Producción de especies reactivas de oxígeno 23 1.6. Hierro y daño oxidativo 26 1.7. Frataxina: estructura y función 30 1.8. Biogénesis de hemoproteínas 36 2. Hipótesis y Objetivos Hipótesis y Objetivos 42 3. Materiales y Métodos 3.1. Material vegetal y condiciones de crecimiento 45 3.2. Cepas bacterianas 45 3.3. Vectores plasmídicos 46 3.4. Medios de cultivo, antibióticos y otros reactivos 3.4.1. Medios de cultivo bacteriano 46 3.4.2. Medios líquidos y soluciones buffer 46 3.4.3. Antibióticos y otros compuestos relacionados 47 3.5. Oligonucleótidos 47 I Indice 3.6. Preparación y análisis de ADN 3.6.1. Propagación de ADN plasmídico 3.6.1.1. Preparación de bacterias Escherichia coli competentes 50 3.6.1.2. Transformación de bacterias competentes de E. coli 50 3.6.2. Aislamiento de ADN plasmídico 3.6.2.1. Aislamiento de ADN plasmídico (kit comercial) 51 3.6.2.2. Aislamiento de ADN plasmídico mediante lisis alcalina 51 3.6.3. Electroforésis de ADN en geles de agarosa 52 3.6.4. Purificación de ADN a partir de geles de agarosa 52 3.7. Manipulación enzimática de ADN 3.7.1. Reacción de ligación 52 3.7.2. Reacciones de digestión con enzimas de restricción 52 3.8. Reacción en cadena de la polimerasa (PCR) 53 3.9. Extracción de ARN total 53 3.10. Cuantificación de ARN total 54 3.11. RT-PCR semicuantitativa 54 3.12. PCR en tiempo real (QPCR) 55 3.13. Extracción de ADN genómico 56 3.14. Cuantificación de ADN 56 3.15. Hibridación de ácidos nucleicos (Southern-blot) 56 3.16. Preparación y análisis de proteínas 3.16.1. Extracción de proteínas 58 3.16.2. Determinación del contenido proteico 58 3.16.3. Electroforesis de proteínas 58 3.17. Obtención de anticuerpos policlonales 59 3.18. Análisis por western blot 59 3.19. Aislamiento de plantas mutantes homocigotas 60 3.20. Construcción de vectores binarios para transformación de plantas de A.thaliana 60 II Indice 3.21. Preparación de células competentes de Agrobacterium tumefaciens y su transformación 3.21.1. Preparación de células competentes 61 3.21.2. Transformación de las células de A. tumefaciens 61 3.21.3. Obtención de ADN plasmídico de A. tumefaciens 62 3.22. Transformación de plantas de A. thaliana utilizando el método de infiltración por floral dip 63 3.23. Esterilización de semillas de Arabidopsis 63 3.24. Selección de plantas transformadas 64 3.25. Clonado, expresión y purificación de proteínas 3.25.1. Clonado molecular de AtFH de A. thaliana 64 3.25.2. Expresión y purificación de las proteínas recombinantes AtFH-His6 y AtFH- STOP de A. thaliana 64 3.26. Ensayo de degradación oxidativa de 2-deoxiribosa 66 3.27. Determinación de Fe 66 3.28. Determinación de la concentración de ATP 67 3.29. Medidas de respiración 67 3. 29.1. Tratamiento con Rotenona 68 3.30. Aislamiento de mitocondrias y determinaciones enzimáticas 3.30.1. Aislamiento de mitocondrias 68 3.30.2 Medida de la actividad enzimática de aconitasa, succinato deshidrogenasa y malato deshidrogenasa 69 3.31. Infiltración de hojas con H2O2 69 3.32. Detección histoquímica de superóxido en hojas de Arabidopsis 70 3.33. Detección histoquímica de especies reactivas de oxígeno en hojas y raíces de plantas de Arabidopsis thaliana 3.34. Determinación del contenido de hemo y actividad catalasa 3.34.1. Determinación de la concentración de hemina 70 70 70 3.34.2. Medida de la actividad catalasa en hojas, flores y mitocondrias aisladas y recuperación de la actividad catalasa por el agregado de hemina exógena 71 III Indice 3.35. Métodos bioinformáticos 72 4. Resultados y discusión 74 Capítulo I 4.1. Caracterización de plantas de Arabidopsis thaliana deficientes en la expresión de AtFH 4.1.1. Estructura del gen AtFH de Arabidopsis thaliana 75 4.1.2. Selección de plantas mutantes homocigotas de A. thaliana con inserción de T-ADN en el gen AtFH 76 4.1.3. Construcción de líneas transgénicas de Arabidopsis thaliana que expresan AtFH en orientación antisentido y sentido 80 4.1.4. Efecto de la deficiencia de AtFH en plantas de Arabidopsis 4.1.4.1. Análisis fenotípico de plantas de Arabidopsis deficientes en la expresión de AtFH 4.1.5. Análisis fenotípico de las líneas mutantes atfh-2 y atfh-3 82 86 4.1.6. Caracterización de las plantas de Arabidopsis thaliana que expresan el gen AtFH en orientación sentido (s-AtFH) 4.1.6.1. Fenotipo de plantas transgénicas s-AtFH 87 Capítulo II 92 5.1. Clonado, expresión y purificación de AtFH recombinante 92 5.2. Atenuación de la reacción de Fenton por AtFH 95 Capítulo III 99 6.1. Efecto de la deficiencia de AtFH sobre la función mitocondrial en Arabidopsis thaliana 101 6.2. Efecto de AtFH sobre la transcripción y actividad del CI mitocondrial 6.2.1. Determinación de los niveles de expresión de genes del CI en plantas con deficiencia en AtFH 6.2.2. Efecto de la deficiencia de AtFH sobre la actividad del CI 104 108 IV Indice 115 Capítulo IV 7.1. Efecto de AtFH sobre la transcripción de genes que codifican proteínas Fe-S mitocondriales 117 7.2. AtFH y stress oxidativo 7.2.1. Plantas deficientes en frataxina muestran un aumento en el contenido de Fe y en la producción de especies reactivas de oxígeno (ROS) 121 7.2.2. Análisis molecular de marcadores de estrés oxidativo 125 7.2.3. Respuesta de AtFH frente a agentes oxidantes 128 Capítulo V 8.1. Participación de AtFH en la biogénesis de hemoproteínas 131 8.1.1. La deficiencia de AtFH provoca una disminución en el contenido de hemo 8.1. 2. Efecto de la deficiencia de AtFH sobre la actividad catalasa 131 134 8.1.3. Efecto de la deficiencia de AtFH en enzimas claves de la síntesis del hemo 139 9. Conclusiones 145 10. Resumen 148 11. Referencias 151 V Agradecimientos Agradecimientos: A mi director de tesis, Dr. Diego Gómez Casati, por estimularme día a día, por ensañarme a pensar y por haberme dado las herramientas necesarias para desarrollarme en el área de la investigación y haber podido llevar a cabo este trabajo de tesis. A mi codirectora, Dra. María Victoria Busi, por haberme abierto las puertas en el laboratorio aquel febrero de 2004. Por haberme guiado y enseñado desde mis comienzos y por su constante predisposición tanto en lo laboral como en lo personal. Al Consejo Directivo y al Director del Instituto de Investigaciones Biotecnológicas- Instituto Tecnológico de Chascomús (IIB-INTECH) Dr. Alberto Carlos Frasch, por permitirme realizar este trabajo de tesis en este Instituto. A la Universidad Nacional de General San Martín por haberme dado la oportunidad de cursar la carrera de Doctorado en esta importante casa de estudios. A la Agencia Nacional de Promoción Científica y Tecnológica (ANPCyT) y al Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET), por otorgarme la beca de iniciación y finalización respectivamente, que posibilitaron la realización de este trabajo de tesis. Al Dr. Eduardo Zabaleta, que me dio la posibilidad de realizar una pasantía en el laboratorio y así poder comenzar a trabajar en investigación. A José Luis Burgos por el apoyo técnico prestado durante el desarrollo de toda esta tesis. Al instituto INFIVE, UNLP y al instituto IFISE, UNR, por haberme permitido realizar las medidas de respiración y de niveles de ATP respectivamente, informadas en este trabajo de tesis. VI Agradecimientos Al Dr. Alejandro Araya, por su asesoramiento en la discusión de los resultados de la PCR en tiempo real. A todos los que componen la “UB6” por crear un muy lindo ambiente de trabajo y compañerismo. A Vale, por su excelente calidad humana y por siempre estar predispuesta a ayudar. A mis amigos, Colo, Huguito, y José Luis, por habernos permitido distraer minutos de trabajo para realizar nuestras típicas charlas, por apoyarnos en momentos en los que lo hemos necesitado y por la buena relación que siempre nos ha acompañado. A todos mis amigos del INTECH que he ido conociendo durante todos estos años y con los cuales he pasado momentos que siempre recordaré. A Diego y Vicky, porque siempre me han brindado tanto en lo personal como en lo laboral, un gran apoyo y no tengo más que agradecimientos hacia ellos. A mis amigos de la vida, del cole y de la facu, con los que cuento incondicionalmente. A mi familia, que siempre me acompañan y apoyan en todas las decisiones que tomo en mi vida, y que sin ellos no hubiese podido lograr nada de lo que he realizado hasta ahora. VII Publicaciones Parte de estos resultados fueron presentados en: Publicaciones: 1- Busi, M.V.; Maliandi, M.V.; Valdez, H.; Clemente, M.; Zabaleta, E.J.; Araya, A. and Gomez-Casati, D.F. (2006) Deficiency of Arabidopsis thaliana frataxin alters mitochondrial Fe-S proteins activity and induces oxidative stress. The Plant Journal 48, 873-882. 2- Maliandi, M.V.; Busi, M.V.; Clemente, M.; Zabaleta, E.J.; Araya, A. and GomezCasati, D.F. (2007) Expression and one-step purification of recombinant Arabidopsis thaliana frataxin homolog (AtFH). Protein Expression and Purification 51, 157-161. 3- Maliandi, M.V.; Busi, M.V.; Araya, A. and Gómez Casati, D.F. Role of frataxin in the biogenesis of hemoproteins in Arabidopsis. Enviado para su publicación. Presentaciones a Congresos 1- Busi MV, Clemente M, Maliandi MV, Rius S, Burgos JL, Valdez H, Zabaleta E, Araya A and GómezCasati D. Oxidative stress and A. thaliana Frataxin homolog (AtFH). XL Reunión SAIB, Posadas, Misiones, Argentina, 2004. 2- Busi, MV; Valdez, H; Clemente, M; Maliandi, MV; Zabaleta, E; Araya, A and Gómez-Casati, D. Arabidopsis thaliana frataxin homolog (AtFH) deficiency increases oxidative stress and modulates Fe-S protein activity in mitochondria. International Congress on Plant Mitochondrial Biology, Obernai, France, jun 2005. 3- Martín, M.; Maliandi, M.V.; Busi, M.V.; Lamattina, L.; Gómez-Casati, D.; Zabaleta, E. Arabidopsis thaliana frataxin (AtFH) is involved in oxidative stress and iron homeostasis. XLI Reunión SAIB, Pinamar, Argentina, 2005. 4- Maliandi, M.V.; Valdez, H.;, Clemente, M.; Busi, M.V.; Zabaleta, E.; Araya, A. and Gomez-Casati, D. Molecular Cloning, Expression and Purification of Arabidopsis thaliana Frataxin homolog (AtFH). XLI Reunión SAIB, Pinamar, Argentina, 2005. 5- Maliandi, M.V.; Busi, M.V.; Zabaleta, E.J.; Araya, A. y Gomez-Casati, D.F. Caracterización funcional de frataxina de Arabidopsis thaliana. XXVI Reunión Argentina de Fisiología Vegetal (SAFV) , Chascomús, Argentina, 2006. 6- Rodriguez Colman, M.; Maliandi, M.V.; Busi, M.V.; Graziano, M.; Gomez-Casati, D.; Lamattina, L.; Zabaleta, E.; Martin, M. A. thaliana frataxin is involved in iron homeostasis and oxidative stress. XLII Reunión SAIB, Rosario, Argentina, 2006. 7- Maliandi, M.V.; Rius, S.P.; Busi, M.V. y Gómez-Casati, D. A simply method for the addition of rotenone in Arabidopsis leaves. XLII Reunión SAIB, Rosario, Argentina, 2006. 8- Maliandi, M.V.; Busi, M.V.; Valdez, H.A.; Clemente, M.; Zabaleta, E.; Araya, A. and Gomez-Casati, D.F. Deficiency of Arabidopsis thaliana frataxin alters activity of mitochondrial Fe–S proteins and induces oxidative stress. XXXVII Annual Meeting of SBBq (Brazilian Biochemistry and Molecular Biology Society) and XI PABMB Congress (Pan- American Association for Biochemistry and Molecular Biology), San Pablo, Brasil, 2008. 9- Maliandi, M.V.; Busi, M.V y Gómez-Casati, D. Participation of AtFH in the biogenesis of hemecontaining proteins. XLIII Reunión SAIB, Carlos Paz, Argentina, 2008 VIII Abreviaturas Abreviaturas: ácido desoxiribonucleico ADN copia ADN genómico ADN mitocondrial ácido ribonucleico ácido ribonucleico mensajero adenosina trisfosfato albumina sérica bovina bromuro de cetil trimetil amonio 5-bromo-4-cloro-indolil-fosfato buffer (tomada del inglés) buffer fosfato salino cantidad suficiente para 2ƍ,7ƍ-diclorodihidrofluoresceina diacetato densidad óptica ADN ADNc ADNg ADNmt ARN ARNm ATP BSA CTAB BCIP sc. amortiguadora de pH PBS c.s.p H2DCFDA D.O diclorofenol indofenol DCPIP dietil pirocarbonato docecil sulfato sódico electroforesis en gel de poliacrilamida especies reactivas de oxigeno flavina adenina dinucleótido fluoruro de fenilmetilsulfonil frataxina homólogo de Arabidopsis thaliana frataxina homólogo de levaduras gentamicina higromicina isopropil ȕ-D-tiogalactopiranosido kanamicina kilodalton levaduras mutantes deficientes en frataxina milimolar nicotinadamida adenina dinucleótido reducido nitroblue tetrazolium pares de bases peróxido de hidrógeno peso fresco peso seco peso/peso peso/volumen Reacción en cadena de la polimerasa DEPC SDS PAGE ROS FAD PMSF AtFH YFH gem hig IPTG kan kDa ǻyfh mM NADH NBT pb H 2 O2 PF PS P/P P/V PCR IX Abreviaturas porción del plásmido Ti retro-transcripción rifanpicina volumen/volumen wild type (tomado del inglés), tipo salvaje T-ADN RT rif V/V wt X Introducción Introducción 1.1. Estructura y función de la mitocondria: Las mitocondrias son organelas dinámicas y pleomórficas de las células eucariotas involucradas en distintos procesos como por ejemplo la fosforilación oxidativa, el metabolismo del carbono y del nitrógeno, la termorregulación, la homeostasis del calcio y hierro (Mackenzie and McIntosh, 1999; Orrenius et al., 2003), la regulación de la apoptosis (Youle and Karbowski, 2005) y la síntesis de grupos Fe-S y hemo (Muhlenhoff et al., 2002; Lange et al., 1999). Estas organelas poseen una doble membrana y difieren en tamaño, forma y cantidad, dependiendo del tipo celular, función tisular y la actividad metabólica del tejido donde se encuentran. Se ha informado que en los tejidos metabólicamente más activos, las mitocondrias ocupan una gran proporción del volumen celular (Lehninger et al., 1993). La membrana externa mitocondrial no forma pliegues y rodea completamente la organela. Esta membrana es permeable a iones y pequeñas moléculas. La membrana interna presenta invaginaciones denominadas crestas mitocondriales que proporcionan una gran área superficial y es impermeable a la mayoría de las moléculas pequeñas e iones, incluido los protones (H+). En general, las únicas especies que pueden atravesar la membrana interna son aquellas para las que existen proteínas transportadoras específicas. Es en las crestas de la membrana interna, donde se encuentran los complejos multiproteicos de la cadena de transporte de electrones y el complejo ATP sintasa (Lehninger et al., 1993; Taiz and Zeiger, 1998). El espacio interno situado entre la membrana externa y la interna se denomina espacio intermembrana. En este compartimento se acumulan altas concentraciones de protones que provienen de la matriz mitocondrial. El mecanismo detallado que acopla la transferencia de electrones a la translocación de protones es poco conocido. El flujo de protones a través de canales proteicos y a favor de su gradiente electroquímico provee la energía libre necesaria para la síntesis de ATP, catalizado por el complejo ATP sintasa. El compartimento interno de la mitocondria, denominado matriz mitocondrial, contiene diferentes complejos proteicos y enzimas como el complejo 2 Introducción piruvato deshidrogenasa, las enzimas del ciclo del ácido cítrico, enzimas relacionadas con la oxidación de ácidos grasos y metabolismo de aminoácidos, ribosomas y ADN mitocondrial (Lehninger et al., 1993; Logan, 2006) (Figura 1). Figura 1: Estructura esquemática de la mitocondria. Tomada de Proyecto biosfera, www. recursos.cnice.mec.es. A diferencia de otras estructuras membranosas como los lisosomas, el complejo de Golgi o la envoltura celular, las mitocondrias tienen naturaleza semiautónoma: deben dividirse para pasar a la siguiente generación a partir de mitocondrias previamente existentes. Esta división se logra por fisión como ocurre en la mayoría de los procariotas (Lenhinger., 2005). Debido su origen endosimbiótico y por encontrarse en algunas algas inferiores, podría esperarse que la mitocondria utilice la maquinaria de fisión heredada de bacterias progenitoras. FtsZ es una GTPasa, estructuralmente homóloga a la tubulina que media la división celular en bacterias al ensamblarse a un anillo en el sitio medio de la división celular (McFadden and Ralph, 2003). En cloroplastos de plantas superiores, se conocen dos genes FtsZ codificados a nivel nuclear, FtsZ1 y FtsZ2 (Vitha et al., 2001). Dichos genes se han encontrado a su vez en procariotas relacionados con las mitocondrias y presumiblemente participen en la división mitocondrial de algas y otros 3 Introducción protistas (Beech and Gilson, 2000; Beech et al., 2000; Takahara et al., 2000). Sin embargo, y en contraste con lo que ocurre en cloroplastos, no se han encontrado homólogos del gen FtsZ en el genoma mitocondrial de Arabidopsis, humanos, ratones, C. elegans y levaduras. En su lugar, en plantas superiores, se ha descripto la existencia de dos proteínas denominadas “dynamin-like proteins”, ADL2a y ADL2b, las cuales participarían en el proceso de división mitocondrial (Arimura and Tsutsumi, 2002; Arimura et al., 2004). Cada una de las mitocondrias presentes en la célula contiene su propio ADN, ARN y ribosomas. Este hecho da soporte a la teoría que supone que las mitocondrias descienden de bacterias aeróbicas que vivían simbióticamente con las células eucariotas primitivas (Lenhinger., 2005). Normalmente, el ADN presente en el total de mitocondrias de una célula somática no llega al 0.1 % del ADN celular total. El ADN mitocondrial codifica ciertas proteínas específicas de la membrana mitocondrial interna y algunos ARNt y ARNr mitocondriales. Si bien la mitocondria tiene su propio material genético, más del 98% de las proteínas que se localizan en esta organela están codificadas en el genoma nuclear que luego son sintetizadas en el citosol e importadas dentro de la mitocondria (Leaver, 1983; Unseld et al., 1997; Gray et al., 1999; Duby et al., 2002; Whelan and Glaser, 1997). El primer informe sobre el importe de proteínas hacia la mitocondria de plantas fue realizado en 1987 (Boutry et al., 1987). Estudios posteriores determinaron la existencia de dos maquinarias en la membrana externa de mitocondrias: (i) la traslocasa general de membrana externa (TOM, Translocase of the Outher Membrane) y (ii) la maquinaria de ensamblaje SAM (Sorting and Assembly Machinery”) (Rapaport, 2002; Stojanovski et al., 2007). Además las mitocondrias contienen una tercera maquinaria de importe y ensamblado de proteínas del espacio intermembrana (MIA, Machinery for Intermembrane space import and Assembly”) (Neupert and Herrmann, 2007; Bolender et al., 2008). 4 Introducción 1.2.Fosforilación oxidativa La fosforilación oxidativa es la síntesis de ATP impulsada por la transferencia de electrones al oxígeno, y la fotofosforilación, que es la síntesis de ATP impulsada por la luz, son los dos mecanismos más importantes de producción de ATP en las células, y generan conjuntamente la mayor parte del ATP sintetizado en los organismos aeróbicos (Lehninger et al., 1993; Hüttemann et al., 2008). En la fosforilación oxidativa, se produce la reducción de O2 a H2O con electrones cedidos por el NADH y el FADH2 que captan los electrones de las reacciones oxidativas del complejo de la piruvato deshidrogenasa, del ciclo del ácido cítrico, de la ruta de la ß-oxidación de ácidos grasos y de reacciones del catabolismo de aminoácidos. Estos electrones son canalizados en forma de pares electrónicos hacia los complejos proteicos que forman la cadena respiratoria o cadena de transporte de electrones mitocondrial (Lehninger et al., 1993). La cadena respiratoria mitocondrial está compuesta por cuatro complejos enzimáticos y dos transportadores electrónicos que en general son proteínas integrales de membrana, con grupos prostéticos capaces de aceptar y donar uno o dos electrones. Cada componente de la cadena puede aceptar electrones del transportador precedente y transferirlo al siguiente en una secuencia específica. Los complejos que forman esta cadena son: NADH deshidrogenasa (complejo I, CI), succinato deshidrogenasa (complejo II, CII), citocromo bc1 (complejo III, CIII), citocromo oxidasa (complejo IV, CIV), coenzima Q (ubiquinona) y el citocromo c (Hüttemann et al., 2008) (Figura 2). 5 Introducción Figura 2: Representación esquemática de la cadena de transporte de electrones de la membrana interna mitocondrial (Lenhinger., 2005). El CI se encuentra en la membrana interna mitocondrial y transfiere los electrones desde el NADH hacia la ubiquinona vía varios centros redox (FMN y centros Fe-S). Este complejo está formado por múltiples subunidades, alrededor de 30 a 46 en hongos y mamíferos. La mayoría de ellas están codificadas a nivel nuclear, mientras que algunas pocas subunidades están codificadas en el ADN mitocondrial. Por esto, se postula que es necesaria una fina regulación entre ambos genomas a fin de asegurar el correcto ensamblado y estequiometría del complejo (Rasmusson et al., 1998). En Arabidopsis, un total de 30 proteínas forman parte del CI, muchas de las cuales son homólogas a las encontradas en otros organismos. Recientemente han sido identificadas cuatro subunidades codificadas a nivel del genoma mitocondrial (ND1, ND5, ND7 Y ND9) y 17 subunidades codificadas a nivel nuclear (Heazlewood et al., 2003 ). La succinato deshidrogenasa (CII), es una enzima que participa tanto en el ciclo del ácido cítrico como en la cadena de transporte de electrones. Cataliza la reacción de oxidación de succinato a fumarato y la reducción de ubiquinona a ubiquinol. (Scheffler, 1998; Ackrell, 2000). El CII ha sido caracterizado en bacterias, protozoos, hongos y animales y en todos ellos se ha encontrado que está compuesto por cuatro subunidades: una flavoproteina (SDH1), una subunidad Fe-S (SDH2), y dos 6 Introducción subunidades de anclaje a membrana (SDH3 y SDH4) (Horsefield et al., 2004; Millar et al., 2004b). El sitio de unión al succinato está formado por el polipéptido SDH1, el cual se une covalentemente al FAD, actuando como aceptor de protones en el paso inicial de la oxidación del succinato. Esta subunidad flavoproteica, interactúa con la subunidad Fe-S que contiene tres centros Fe-S no hemínicos, [Fe2-S2], [Fe4-S4] and [Fe3-S4], que actúan como conductores de electrones desde la flavoproteína hacia la membrana (Scheffler, 1998; Ackrell, 2000; Hüttemann et al., 2008). En Arabidopsis se ha podido separar el CII de los otros complejos por la técnica de “Blue-native Page”, y se demostró que está compuesto por al menos 8 subunidades proteicas. Así, el CII de Arabidopsis contiene las cuatro subunidades clásicas, las cuales poseen secuencias similares a las encontradas en otros organismos y 4 subunidades específicas de este organismo (Eubel et al., 2003). Las cuatro subunidades adicionales fueron denominadas SDH5, SDH6, SDH7 y SDH8, y serían subunidades específicas de plantas (Millar et al., 2004b). Las ocho subunidades de SDH se encuentran codificadas en el genoma nuclear (Scheffler, 1998; Figueroa et al., 2001; Millar et al., 2004b), encontrándose que varias de ellas están codificadas por más de un gen. Por ejemplo, la subunidad SDH1, está codificada por dos genes nucleares SDH1-1 y SDH1-2; SDH2, está codificada por tres, llamados, SDH2-1, SDH 2-2 y SDH2-3; SDH3 está codificada por otros tres, la subunidad SDH4 está codificada por un único gen nuclear y SDH7 se encuentra codificada por dos genes nucleares (Millar et al., 2004b). La ubiquinona-citocromo c oxidoreductasa (CIII), consiste en 10 subunidades, incluídas las peptidasas mitocondriales, codificadas por 16 genes, ya que seis de estas subunidades, están codificadas por más de un gen en A. thaliana (Braun and Schmitz, 1992; Eubel et al., 2003). La mayoría de las subunidades que forman este complejo, están codificadas por el genoma nuclear (Kruft et al., 2001; Werhahn and Braun, 2002; Meyer et al., 2008), aunque existe una única subunidad, el citocromo b, que está codificada en el genoma mitocondrial (Unseld et al., 1997). 7 Introducción La citocromo c oxidasa (COX, CIV) está formada por 13 subunidades en mamíferos, tres de las cuales están codificadas en el genoma mitocondrial y diez son nucleares. Previamente se había informado la existencia de 7 u 8 subunidades de este complejo en plantas (Peiffer et al., 1990). Estudios recientes llevados a cabo por Eubel y col. (Eubel et al., 2003) y Millar y col. (Millar et al., 2004) han dilucidado la presencia de al menos 12 subunidades del CIV en plantas de Arabidopsis. En estos trabajos se han identificado ocho proteínas homólogas a las subunidades conocidas de COX de otros organismos y otras seis subunidades específicas de plantas (Millar et al., 2004). La mitocondria de plantas posee una bifurcación de la cadena de transporte de electrones clásica, dada por la presencia de cinco enzimas accesorias que no translocan protones: cuatro NADH deshidrogenasas insensibles a rotenona (NDH-DHS) y una oxidasa alternativa (Moller, 2001a) (Figura 3). Figura 3: Organización de la cadena de transporte de electrones de mitocondria de plantas y el complejo ATP sintasa (CV). Adicionalmente a los complejos respiratorios CI al CIV encontrados en mitocondrias de otros organismos, la mitocondria de plantas contiene una oxidasa alternativa y NAD(P)H deshidrogenasas en el lado interno y externo de la membrana interna de la organela. (Tomado de Taiz y Zeiger, Plant Physiology, 2ª Edición). La vía alternativa de respiración está compuesta por una única proteína llamada oxidasa alternativa (AOX) (Elthon and McIntosh, 1987), la cual existe como homodímero en la membrana mitocondrial interna (Umbach and Siedow, 1993), y se presenta en dos formas, una oxidada o menos activa, y otra reducida o activa (Umbach 8 Introducción and Siedow, 1993) (Figura 3). Cuando ocurre alguna inhibición de los complejos respiratorios, especialmente CIV, como así también alguna saturación en la cadena de citocromos, produciendo un aumento de la relación ATP/ADP y NADH/NAD+, los electrones se desvían de la cadena de transporte clásica hacia la vía alternativa, al nivel del pool de ubiquinona (Lambers, 1982; Laties, 1983). La enzima AOX cataliza la reducción de cuatro electrones del oxígeno hacia el agua, salteándose dos centros de conservación de energía (CIII y CIV) y, por lo tanto, no se genera el gradiente de protones que llevaría a la síntesis de ATP. Consecuentemente, la energía que es conservada como ATP en la vía clásica, se pierde como calor cuando los electrones pasan a través de la vía alternativa (Lambers, 1982). En Arabidopsis, AOX, esta codificada por cinco genes, cuatro de los cuales se denominan tipo AOX1 y uno tipo AOX2 (Considine et al., 2002). Se observó que la expresión de los diferentes genes que codifican para AOX depende del tipo tisular y el estadío de desarrollo del mismo (Saisho et al., 1997; Saisho et al., 2001; ThirkettleWatts et al., 2003). AOX1a se expresa en la mayoría de los tejidos, aunque AOX1c lo hace en cotiledones jóvenes y flores, teniendo mayores niveles de expresión en estas últimas. AOX1b, AOX1d y AOX2 se expresan en todos los tejidos pero en menores niveles. Se ha informado que AOX, tiene un papel antioxidante en la mitocondria de plantas, encontrándose mayores niveles de expresión en situaciones de estrés como el ocasionado por frío, ataque de patógenos, sequía, y daño celular. En estas situaciones los genes más afectados son los AOX1a y AOX1d (Millar et al., 2008 ). La otra bifurcación de la cadena de transporte clásica, se da a nivel del complejo I por la presencia de NADH y NADPH deshidrogenasas insensibles a rotenona. Estas enzimas transfieren los electrones a la ubiquinona desde la matriz o el espacio intermembrana donde se encuentra el pool de NAD(P)H (Rasmusson et al., 2004). Como hemos mencionado, estas deshidrogenasas son insensibles a rotenona (inhibidor del CI) y consecuentemente la respiración en las plantas puede continuar en presencia de este compuesto (Roberts et al., 1995). Sin embargo, bajo estas condiciones el CI no puede translocar protones y por lo tanto habría una deficiencia en la producción de ATP. 9 Introducción Por otro lado, se han identificado 7 genes de NDH-DHs en Arabidopsis y se los han dividido en tres clases: NDA (NDA1 y NDA2), NDB (NDB1, NDB2 y NDB4) y NDC (NDC1) (Michalecka et al., 2003). Usando experimentos de importe de proteínas marcadas radiactivamente, se reveló que NDA1, NDA2 y NDC1 están localizadas en el interior de la membrana interna, mientras que NDB1, NDB2 y NDB4 están localizadas en la parte externa de la membrana interna (Elhafez et al., 2006 ). A través de estudios de caracterización bioquímica en mitocondrias aisladas de plantas, se ha demostrado que las NADH deshidrogenasas internas, operan en paralelo con el CI (Rasmusson and Moller, 1991). Del lado externo de la membrana interna, el NADH y el NADPH, son oxidados por deshidrogenasas dependientes de Ca+2 (Roberts et al., 1995). A su vez, el NADPH interno puede oxidarse de una manera dependiente de Ca+2, mientras que la oxidación del NADH interno es la única actividad independiente de Ca+2 (Moller, 2001b). La diferente especificidad de cada una de las deshidrogenasas le permite a la planta poder oxidar el NAD(P)H durante distintas situaciones . Algunos de los complejos de la cadena transportadora de electrones están compuestos por centros Fe-S, formando proteínas ferro – sulfuradas, en las cuales el hierro está presente no en la forma de hemo (como en los citocromos), sino en asociación con átomos de azufre inorgánico, con residuos de cisteína de la proteína, o con ambos (Lenhinger., 2005). Así, el complejo NADH deshidrogenasa contiene 8 centros Fe-S (Herald et al., 2003), el Complejo II posee 3 centros Fe-S (Elorza et al., 2004) y el citocromo c un grupo (Millar et al., 2004a). Además se ha descripto la presencia de otras proteínas Fe-S en la matriz mitocondrial como la enzima aconitasa (Elorza et al., 2004), que interviene en el ciclo de Krebs . Uno de los tópicos de estudio en este trabajo de Tesis está relacionado con la caracterización de genes y sus productos proteicos que participan en el ensamblado de los centros Fe-S y biogénesis de ferrosulfoproteínas en plantas. Dado que la mayoría de las proteínas Fe-S mitocondriales está codificada por genes nucleares, el ensamblado de las mismas constituye un buen modelo para el estudio de los mecanismos de comunicación entre el núcleo y la mitocondria. 10 Introducción 1.3. Comunicación núcleo-mitocondria La coordinación de funciones entre diferentes compartimentos y organelas celulares es esencial para el crecimiento y desarrollo de la planta. En estos últimos años, se ha incrementado el estudio y los conocimientos sobre las señales generadas por organelas como el cloroplasto y la mitocondria y la adaptación de la respuesta transcripcional nuclear a esas señales. A este proceso se lo denomina señalización retrógrada (Pogson et al., 2008). Las mitocondrias y cloroplastos de plantas, tienen un origen bacteriano y un genoma propio y único. Sin embargo, durante la evolución, la mayoría de los genes endosimbióticos originales, han sido transferidos al núcleo, necesitando una señal que permita que la proteína sea sintetizada en el citosol y luego dirigida a la organela (Giege´ et al., 2005). Como se mencionó anteriormente, más del 98% de las proteínas mitocondriales están codificadas en el ADN nuclear y son importadas dentro de la mitocondria, donde cumplen su función (Leaver, 1983; Unseld et al., 1997; Whelan and Glaser, 1997; Gray et al., 1999; Duby et al., 2002). Para ello, los genes nucleares que codifican para proteínas mitocondriales han adquirido señales de transcripción adecuadas (promotores) y secuencias de importe necesarias para que los productos sean dirigidos a la organela (Margulis, 1995; Figueroa et al., 1999). Aproximadamente de las 1000 o más proteínas que se estiman que constituyen la mitocondria funcional de plantas (Millar et al., 2004a), únicamente 33 proteínas, 3 ARNr y 20 ARNt son codificadas en el genoma mitocondrial en Arabidopsis thaliana (Unseld et al., 1997; Duchêne and Maréchal-Drouard, 2001). Por lo descripto más arriba, debe existir una comunicación y regulación precisa entre el núcleo y la mitocondria para asegurar la expresión coordinada de ambos genomas. Esto es particularmente importante en la biogénesis de los complejos respiratorios de la membrana mitocondrial interna. Es claro, que la interrelación entre los factores de transcripción y coactivadores contribuyen a la expresión de genes 11 Introducción respiratorios tanto nucleares como mitocondriales. Además, la regulación de la biogénesis mitocondrial depende de proteínas que al interaccionar con el ARN de proteínas mitocondriales, influencian su metabolismo y expresión (Cannino et al., 2007). Estudios desarrollados por Brennicke y col. en Arabidopsis, han demostrado que la regulación de la expresión génica de mitocondrias, incluye un complejo proceso a nivel transcripcional y posttranscripcional, como el procesamiento del 5´y 3´ ARN, splicing de intrones, edición del ARN y regulación de la estabilidad del ARN (Binder and Brennicke, 2003). Estudios comparativos entre actividad transcripcional y los niveles de varios ARN codificados en el genoma mitocondrial de Arabidopsis, revelan una baja correlación entre la actividad relativa de los promotores y la abundancia de los transcriptos, sugiriendo una fuerte regulación a nivel postraduccional (Giegé et al., 2000). Con el objeto de ampliar los conocimientos sobre los mecanismos de comunicación núcleo – mitocondria, en nuestro laboratorio se ha desarrollado un modelo de estudio basado en la construcción de plantas que presentan una disfunción mitocondrial generada por la expresión de un gen "no-editado" u-ATP9 (Hernould et al., 1993; Zabaleta et al., 1996; Gómez-Casati et al., 2002). La deficiencia mitocondrial indujo la expresión de tres genes nucleares que codifican proteínas Fe-S del complejo CI (PSST, TYKY y NADHbp), como así también otros factores asociados a este complejo, como es ARR2 (Arabidopsis Response Regulator 2). Este gen se expresa predominantemente en granos de polen y activaría la transcripción de algunos genes que codifican subunidades del CI como PSST (Lohrmann et al., 2001). El modelo de disfunción mitocondrial de las plantas u-ATP9 mostró también la inducción en los niveles de expresión de algunos genes nucleares relacionados con la síntesis de proteínas Fe-S en mitocondrias, como el homólogo de frataxina de Arabidopsis (AtFH) (Busi, et al., 2004). 12 Introducción 1.4 Biogénesis de los grupos Fe-S La biogénesis de grupos Fe-S es una de las funciones esenciales de la mitocondria. Los grupos Fe-S son moléculas inorgánicas que forman parte de ferrosulfoproteínas celulares (Beinert et al., 1997). En los organismos vivos estos grupos se encuentran formando diferentes estructuras: (i) diméricas ([Fe2-S2]) que tienen estructura rómbica y (ii) tetraméricos ([Fe4-S4]), de estructura cúbica. Aunque los dos grupos descriptos son los más abundantes entre las proteínas Fe-S, también existen formas de mayor complejidad estequiométrica como por ejemplo el grupo [Fe8-S7] en la enzima nitrogenasa. En general, los grupos Fe-S están unidos a las cadenas polipeptídicas mediante enlaces que se establecen entre las cisteínas de la proteína y los átomos de azufre del cofactor (Figura 4). Una excepción la constituye la proteína Rieske (una de las subunidades del CIII mitocondrial) en la cual el átomo de Fe coordina con dos residuos de histidina en lugar de dos residuos de cys (Rieske et al., 1964; Iwata et al., 1996). Figura 4: Esquema de la estructura de los grupos Fe-S unidos a un polipéptido. (a) Estructura simple con un único átomo de Fe coordinado con cuatro residuos de cys. Existen algunos centros que incluyen dos S inorgánicos y dos átomos de S de cisteínas (b) Fe2-S2 y (c) Fe4-S4 (Lenhinger., 2005). Las ferrosulfoproteínas están involucradas en una gran cantidad de procesos celulares como reacciones enzimáticas, respiración celular, biogénesis de ribosomas, regulación de la expresión génica, fijación de nitrógeno y las respuestas a estrés. Una de las funciones más importantes está relacionada con la transferencia de electrones en los 13 Introducción complejos respiratorios mitocondriales y en el aparato fotosintético de cloroplastos (Balk and Lobréaux, 2005; Lill and Muhlenhoff, 2008). Las ferrosulfoproteínas, pueden participar en estos procesos debido al amplio rango de potencial redox que presentan, ya que varía de -0.65 V hasta +0.45 V. Durante el proceso de transferencia de electrones, un átomo de Fe del centro Fe-S es oxidado o reducido dependiendo del microambiente en que se encuentre y el tipo de proteína a la cual esté unido (Beinert, 2000). Algunos ejemplos de proteínas Fe-S son los complejos respiratorios mitocondriales CI, CII y CIII; la aconitasa, presente en la matriz mitocondrial y relacionada con el ciclo de Krebs (Beinert et al., 1997); la nitrogenasa, relacionada con la fijación de N2 (Peters et al., 1995; Rees and Howard, 2000); la aldehído oxidasa, involucrada en la biosíntesis de ácido indolacético y ácido absícico (Andrade et al., 2000), la ferredoxina que participa en el proceso fotosintético (Hall et al., 1966; Palmer and Sands, 1966); la Iron Responsive Protein IRP1, de localización citosólica (Lill et al., 2005); y la endonucleasa Ntg2p presente en el núcleo celular (Lukianova and David, 2005) (Tabla 1). 14 Introducción Tabla 1: Proteínas Fe-S y localización sub-celular en plantas superiores Proteína Aconitasa Adenosina 5´fosfosulfato reductasa (APS reductasa) Adrenodoxina (ferredoxina mitocondrial) Aldehído oxidasa Amidofosforibolsiltransferasa Biotina sintasa Clorofila a oxigenasa Colina monooxigenasa Cnx2 (síntesis molibdopterina) Acido dehidroxi dehidratasa ADN glicosilasa Endonucleasa III (NTH1) Ferredoxina Ferredoxina-tioredoxina reductasa Glutamato sintasa (Fd-GOGAT) Lipoato sintasa (relacionado con biotin sintasa) NADH- plastoquinona oxidoreductasa subunidades K, I, H (PsbG, NdhI, NdhH) NADH- ubiquinona oxidoreductasa (CI) Homólogo NBP35 (P-loop NTPasa) NAR1 (tipo hidroxilasa) Nitrito reductasa Feofórbido a oxigenasa (LLS1) Fotosistema I subunidades PsaA y PsaB Fotosistema I subunidade PsaC Proteína Rieske (PetC)(complejo citocromo b6f) Proteína Rieske (ubiquinol-citocromo c reductasa) Tipo inhibidor RNAsaL Sirohidroclorin ferroquelatasa Succinato deshidrogenasa subunidad 2 Sulfito reductasa Tic55, proteína plástidica de maquinaria de importe Xantina deshidrogenasa Centro Fe-S [Fe4-S4] [Fe4-S4] Localización a Mitocondria, citosol Plástido [Fe2-S2] 2[Fe2-S2] [Fe4-S4] [Fe2-S2], [Fe4-S4] [Fe2-S2] (tipo Rieske) [Fe2-S2] (tipo Rieske) Mitocondria Citosol Plástido Mitocondria Plástido Plástido Citosol (P) Plástido Núcleo Núcleo Plástido Plástido Plástido Mitocondria, plástido Plástido b [Fe4-S4] [Fe4-S4] [Fe4-S4] [Fe2-S2] [Fe4-S4] [Fe3-S4] [Fe2-S2], [Fe4-S4]? b b b b [Fe4-S4] [Fe2-S2], (tipo Rieske) [Fe4-S4] 2[Fe4-S4] [Fe2-S2] (tipo Rieske) [Fe2-S2] (tipo Rieske) b [Fe2-S2] [Fe2-S2], [Fe3-S4], [Fe4-S4] [Fe4-S4] [Fe2-S2] (tipo Rieske) 2[Fe2-S2] Mitocondria Citosol (P) Citosol (P) Plástido Plástido Plástido Plástido Plástido Mitocondria Citosol (P) Plástido Mitocondria Plástido Plástido Citosol a Fe-S proteínas encontradas en varias vías metabólicas en plantas, incluidas fotosíntesis, respiración, asimilación de nitrógeno y azufre, reparación al DNA y cofactores de biosíntesis. La mayoría de las proteínas Fe-S se encuentran en cloroplastos (plástidos) y mitocondria. (P) Predicho en base a secuencias u otra información b Tipo de centro Fe-S todavía no determinado. Tomada de Balk and Lobréaux, (2005). Los centros Fe-S se pueden ensamblar a apoproteínas in vitro en presencia de S=, Fe+2 y Fe+3 y ditiotreitol (Balk and Lill, 2004). Esta reacción no puede llevarse a cabo in vivo, ya que el azufre y el hierro en sus formas libres son extremadamente tóxicos para la célula. Por este motivo, es necesario que la biosíntesis de los grupos Fe15 Introducción S y su inserción a apoproteínas se lleve a cabo bajo un proceso controlado. In vivo, el ensamblado de los grupos Fe-S está asistido por una compleja maquinaria proteica de ensamblado que utiliza el aminoácido cisteína como fuente de sulfuro y se combina con el Fe, asociado a numerosas proteínas molde o scaffold, para formar los grupos Fe-S (Lill and Muhlenhoff, 2008). La maquinaria de ensamblado está representada en la naturaleza por tres sistemas, estructural y funcionalmente relacionados: NIF, SUF e ISC (Balk and Lobréaux, 2005). El sistema NIF fue el primero en ser descripto en la bacteria fijadora de nitrógeno Azotobacter vinelandii, asociado con la biogénesis de la nitrogenasa, de allí su nombre (NItrogen Fixation) (Zheng et al., 1993). La maquinaria SUF, llamada así por su participación en la movilización de sulfuro, se encuentra en numerosas bacterias, arqueas y en cloroplastos de plantas, pero está ausente en hongos y metazoos. Este sistema se encuentra estrechamente relacionado al estrés oxidativo y al sensado de hierro en la célula (Takahashi and Tokumoto, 2002; Loiseau et al., 2003). La maquinaria ISC (Iron-Sulfur Cluster) representa el sistema más utilizado para la biogénesis de los grupos Fe-S y maduración de ferrosulfoproteínas de las células vivas, y ha sido descripto en cianobacterias, hongos, insectos, plantas y animales. En algunas bacterias, incluida Escherichia coli, el sistema ISC y SUF están presentes en paralelo, mientras que en otras especies existe solamente uno de estos sistemas. En eucariotas la maquinaria ISC está localizada en las mitocondrias aunque en los últimos años se ha postulado la existencia de un sistema paralelo en el citosol y en núcleo (Kispal et al., 1999; Mühlenhoff and Lill, 2000). La localización del sistema ISC en la mitocondria estaría asociada al ambiente reductor provisto por estas organelas (evidenciado por la ausencia de puentes disulfuros en las proteínas), condiciones que favorecen el ensamblado de los grupos Fe-S. El mecanismo básico de biosíntesis de los grupos Fe-S requiere la presencia de 3 ó 4 proteínas claves, aunque en algunos organismos se han descripto hasta 10 y 12 polipéptidos involucrados (Balk and Lobréaux, 2005). Dentro de las proteínas claves se encuentra una cisteína desulfurasa y dos o tres proteínas scaffold, que actuarían como molde o soporte para la síntesis de los grupos Fe-S. En la Figura 5 se muestra un esquema de las etapas que serían comunes en los sistemas NIF, ISC y SUF. 16 Introducción Figura 5: Representación esquemática de los pasos básicos en la biosíntesis de centros Fe-S. Tomado de Balk y Lobreaux (2005) 1.4.1. Biogénesis de grupos Fe-S en plantas Recientemente, se ha descripto en Arabidopsis thaliana la existencia de genes homólogos a los que participan en la biogénesis de grupos Fe-S en otros organismos como levaduras y mamíferos (Balk and Lobréaux, 2005). Se postuló que en plantas existiría compartamentalización de las vías metabólicas que llevan a la síntesis de estos grupos y que dicho proceso está adaptado a las necesidades específicas de las células de organismos fotosintéticos. Los plástidos y las mitocondrias contienen vías genéticas y bioquímicamente diferentes para el ensamblado de centros Fe-S, sin embargo, se han encontrado numerosas proteínas homólogas en ambos compartimentos, sugiriendo que mecanismos similares estarían conservados en los pasos básicos del proceso de ensamblado (Balk and Lobréaux, 2005) (Figura 6, Tabla 2). 17 Introducción Figura 6: Compartamentalización de la biogénesis de proteínas Fe-S en la célula vegetal. Las proteínas cuya localización no ha sido confirmada experimentalmente se encuentran en gris. Por analogía con levaduras, la mitocondria podría importar un componente (X) que sería requerido para la biogénesis de proteínas Fe-S en el citosol y el núcleo (flechas discontinuas) (Balk and Lobréaux, 2005). 18 NFU, CNFU-V NFU3, CNFU-IVa NFU4, NFU-III NFU5, NFU-I NAP1, SUFB,ABC1 NAP7, SUFC At5g49940 At4g25910 At3g20970 At1g51390 At4g04770 At3g10670 Plástido(E) Plástido(E) Mit(E) Mit(E) Plástido(E) Plástido(E) Plástido(E) NFU, CNFU-IVb At4g01940 Plástido Mit(P) ISCA, SUFA At1g10500 Mit(E) At5g03905 ISU3 At3g01020 Mit(E) Citosol(P) ISU2 At4g04080 Mit(E) At2g36260 ISU1 At4g22220 Plástido(P) Plástido(E) Mit(E) Localización Mit(P) SUFE At4g26500 e At2g16710 NFS2, CpNifS At1g08490 Nombre de la proteína NFS1 a At5g65720 Locus Arabidopsis b Isa Isa1 Isa1 Isa Isu1,2 Isu1,2 Isu1,2 ---- SufC SufB ---- ---- NP_312283 Nfu1 NP_312283 Nfu1 NP_312283 Nfu1 NP_312283 Nfu1 NP_312283 NFU1 Isc/SufA Isc/SufA Isc/SufA Isc/SufA IscU IscU IscU SufE SufS/CsdB ---- c, e Homologos E.coli Levadura IscS Nfs1 d Slr0075(Sy) Slr0074 (Sy) C-term, NifU (Av) Ssl2667 (Sy) Slr1417 (Sy) N-term, NifU(Av) Slr1419 (Sy) Slr0077 (Sy) SII0387,Slr0704 (Sy) NifS(Av), Otros Tabla 2: Proteínas confirmadas y candidatas de ensamblado de proteínas Fe-S en Arabidopsis thaliana ABC/ATPasa ABC/ATPasa Scaffold Scaffold Scaffold Scaffold Scaffold Scaffold Scaffold Scaffold Scaffold Scaffold Scaffold Scaffold Co-enzima de SufS Cys desulfurasa grupo II Cys desulfurasa grupo I Función Molecular ADX1, MFDX2 ADX2, MFDX1 ADXR, MFDR At4g21090 At4g05450 At4g32360 ATM1, STA2 ATM2 ATM3, STA1 ERV1 HCF101 HCF101-L2 HCF101-L1 APO1 At4g28630 At4g28620 At5g58270 At1g49880 At3g24430 At5g50960 At4g19540 At1g64810 Plástido(P) At1g54500? ----- ---------- Nar1 ------ Cfd1 Mrp ----- ----- Nbp35 ----- Erv1 Atm1 Atm1 Atm1 Yfh1 Jac1 Ssq1 Arh1 Yah1 Yah1 ---- Mrp Mrp Mrp ----- ----- ----- ----- CyaY HscB HscA Fdx Fdx SufD RubA(Syc) NifH (Av) Slr0067 (Sy) Frataxina (Hs) Slr0076(Sy) Rubredoxina Tipo hidrogenasa Desconocida P-loop NTPasa P-loop NTPasa P-loop NTPasa P-loop NTPasa Sulfidril oxidasa Transportador ABC Transportador ABC Transportador ABC Chaperona Fe Co-Chaperona Chaperona (HSP70) Reductasa Ferrodoxina Ferrodoxina Proteína ABC Proteínas componentes de la maquinaria de ensamblado de proteínas Fe-S en plantas, por evidencia experimental o por homología con proteínas de levaduras o bacterias. b Localización subcelular está basada en resultados experimentales (E) o predichos por programas bioinformáticos (P) o alguna otra evidencia. a Citosol(P) Plástido(E) Mit(P) Citosol (P) Plástido (E) Mit(E) Mit(E) Mit(P) At4g16440 Rubredoxina Mit(P) FH At4g03240 Mit(E) Mit(P) Mit(P) Mit(E) Mit(E) Mit(E) Plástido(E) At5g06410 At5g09590 At4g37910 o NAP6, SUFD At1g32500 Todas las proteínas plastídicas excepto APO1 tienen homólogos in la cianobacteria Synechocystis , sugiriendo que la maquinaria de ensamblado de centros Fe-S es heredada de sus ancestros comunes, de acuerdo con la teoría endosimbiótica. El homologo de las proteínas IscS y IscU en E.coli están localizadas en mitocondria (Mit). Notar que la mitocondria no tiene homólogos de SUF, de acuerdo con su ausencia en el genoma de levaduras y mamíferos. d Homólogos seleccionados en otros organismos: Av, bacteria fijadora de nitrógeno Azotobacter vinelandii ; Sy, la cianobacteria Synechocystis sp. .PCC 6803; Syc la cianobacteria Synechochoccus sp. PCC 7002; Hs, Homo sapiens; C-term., C-terminal; N-term., N-terminal. e Indica ausencia de homólogos; las celdas vacías en las columnas significan que los loci o las proteínas no han sido anotadas o nombradas por el momento Tomada de Balk and Lobreaux (2005). c Introducción Takahashi y colaboradores han demostrado la existencia del proceso de ensamblado de centros Fe-S en el estroma del cloroplasto, dependiente de ATP y de NADPH (Takahashi et al, 1991). Se han identificado además, en el genoma de Arabidopsis dos genes que codifican para cisteínas desulfurasas: NFS1 y NFS2 (Kushnir et al., 2001; Pilon-Smits et al., 2002). Recientemente se inició la caracterización parcial de AtNfs1, AtNfs2 y AtSUFE, que codifican para las cisteínas desulfurasas mitocondrial y plastídica, y para el activador de estas enzimas, respectivamente (Pilon-Smits et al., 2002b; Xu and Møller, 2006). Otro de los genes descriptos en el genoma de Arabidopsis a partir de análisis bioinformáticos es el homólogo de Suf. Este gen codifica para una proteína de plástidos y se ha descripto como activador de las cisteínas desulfurasas cloroplásticas (Balk and Lobréaux, 2005; Ye et al., 2006). Por otra parte, también se han encontrado cinco genes NFU, tres de los cuales codifican para proteínas localizadas en plástidos (Léon et al., 2003). Hasta el momento no existen estudios in vivo donde se haya demostrado el ensamblado de proteínas Fe-S en mitocondrias de plantas, sin embargo, estas organelas contienen varios componentes que son homólogos a la maquinaria ISC de bacterias y levaduras. Por ejemplo, dos de las cinco proteínas NFU y la proteína “scaffold” IscA/Isa de Arabidopsis se encuentran localizadas en mitocondria. Por otro lado, el genoma de Arabidopsis contiene tres genes con alta similitud de secuencia con IscU/Isu que codifican proteínas con localización mitocondrial (Léon et al., 2005). Otra de las proteínas asociadas con la síntesis de grupos Fe-S es la proteína frataxina. Mediante análisis bioinformáticos, Huynen y col. (2001) predijeron la participación de frataxina en el ensamblaje de los grupo Fe-S a proteínas. Se postuló también que esta proteína estaría relacionada con daño oxidativo y disfunción mitocondrial (Koutnikova et al., 1997; Rotig et al., 1997). Recientemente en nuestro laboratorio se ha aislado y caracterizado el gen AtFH que codifica para la frataxina de Arabidopsis thaliana (Busi et al., 2004). La función y estructura de esta proteína en mitocondrias de plantas será objeto de estudio en este trabajo de Tesis. 22 Introducción Con lo mencionado hasta el momento, se postula que los componentes claves de la maquinaria ISC mitocondrial, como fue identificada en levaduras, estarían presentes también en la mitocondria de plantas, indicando que esta organela sería capaz de ensamblar sus propias proteínas Fe-S. A pesar de que las proteínas Fe-S son más abundantes en cloroplastos y mitocondrias, un gran grupo de proteínas Fe-S desarrollan sus funciones en el citosol y el núcleo. Cómo estas proteínas obtienen sus cofactores Fe-S es un punto de gran interés e investigación en este momento. Ya hemos mencionado la presencia, en eucariotas no fotosintéticos, de un sistema de ensamblado para proteínas Fe-S citosólicas (CIA), posiblemente las plantas cuenten con un sistema similar (Balk and Lobréaux, 2005). 1.5. Producción de especies reactivas de oxígeno Las especies reactivas del oxígeno (ROS) son un conjunto de moléculas derivadas del metabolismo celular en el cual el oxígeno cumple un papel fundamental. .- Entre las ROS se encuentran: (i) el anión superóxido (O2 ) que es un potente oxidante; (ii) el peróxido de hidrógeno (H2O2), que es el más estable de las ROS, pudiendo difundir a través de la membrana y causar daño oxidativo a proteínas distantes a la zona . de producción (Moller, 2001a); y (iii) el radical hidroxilo ( OH) que es la especie más reactiva. La alta reactividad de las ROS se debe a que poseen electrones desapareados que les hace reaccionar con otras moléculas orgánicas en procesos redox. El estrés oxidativo incluye una serie de procesos deletéreos que resultan del desbalance entre la formación excesiva de ROS y/o de especies reactivas de nitrógeno (RNS) y la limitada defensa antioxidante. Mientras que pequeñas fluctuaciones en los niveles de concentración de ROS juegan un papel importante en la señalización intracelular (Droge and 2002; Balaban et al., 2005), un aumento descontrolado de estos oxidantes lleva a una liberación de especies reactivas provocando daño a proteínas (Stadtman and Levine, 2000), lípidos (Rubbo et al., 1994), polisacáridos (Kaur and 23 Introducción Halliwell, 1994;) y ADN (LeDoux et al., 1999; Richter et al., 1988). Esta producción de ROS provoca daño mitocondrial, ocasionando un amplio rango de patologías que pueden llevar a apoptosis (Figura 7). Figura 7: Esquema de la producción de ROS mitocondrial (Murphy M., 2009). La producción de ROS por la mitocondria puede causar daño a proteínas mitocondriales, membrana y DNA, imposibilitando a la mitocondria a sintetizar ATP y otra gran cantidad de funciones metabólicas que se llevan a cabo en ella. El daño oxidativo en la mitocondria puede aumentar la tendencia de la mitocondria a liberar proteínas del espacio intermembrana como con la citocromo c (cyt c), al citosol, permeabilizando la membrana externa y de esta manera activar la maquinaria apoptótica de la célula. Además, la producción de ROS en la mitocondria produce la inducción de poros de permeabilidad transitoria (PTP), produciendo daño en la permeabilidad de la membrana. Por otro lado, la producción de ROS puede mediar la señalización redox, afectando reversiblemente las funciones de mitocondria, citosol y núcleo. Mitochondrion: mitocondria. Intermembrane space: espacio intermembrana. Outer membrane: membrana externa. Inner membrane: membrana interna. Respiratory chain: cadena respiratoria. Lipid peroxidation: peroxidación lipídica. Oxidative damage: daño oxidativo. Defective proteins: proteínas defectuosas. Mutations and deletions: mutaciones y deleciones. En 1966, se dio a conocer el primer trabajo en donde se involucraba a la cadena de transporte de electrones como productora de ROS (Jensen, 1966). Otros estudios demostraron que mitocondrias aisladas tenían la capacidad de producir H2O2 (Loschen et al., 1971; Chance et al., 1979; Jensen, 1966). Sin embargo, las mitocondrias no son el único sitio de producción de ROS en las células, se informó que también existe producción de ROS en cloroplastos, peroxisomas, retículo endoplásmico, núcleo y citosol (Moller, 2001a; del Río et al., 2003 ;Andreyev et al., 2005). 24 Introducción Dentro de las mitocondrias, los complejos CI y CIII de la cadena de transporte de electrones son dos de los principales sitios de producción de ROS. Se describió también, que la inhibición del CI por rotenona o del CIII por antimicina A lleva a un aumento en los niveles de ROS (Sugioka et al., 1988; Chen et al., 2003 ). Sin embargo, se ha descripto en células de mamíferos, que en algunas condiciones patológicas existiría una disminución de ROS en CI luego del tratamiento con rotenona (Becker et al., 1999). Existen diversos mecanismos para disminuír los niveles de ROS en las células. Las superóxido dismutasas (SODs), constituyen la primera línea de defensa contra las ROS, catalizando la dismutación del radical superóxido a peróxido de hidrógeno y oxígeno (Fridovich, 1995; del Río et al., 2003 ). De acuerdo al metal que utiliza la enzima como cofactor, las SODs se clasifican en tres grupos: Fe-SOD, la cual está localizada en los cloroplastos, Mn-SOD, localizada en mitocondrias y peroxisomas, y la Cu-Zn-SOD, localizada en el cloroplasto, citosol y espacio extracelular (Fridovich, 1986 ; Bannister et al., 1991; Kanematsu and Asada, 1991; Smith and Doolittle, 1992). El peróxido de hidrógeno producido se descompone a agua y oxígeno por acción de una enzima que contiene hemo, la catalasa (Kwon and Sun An, 2007). La catalasa, está localizada mayoritariamente en los peroxisomas, aunque se ha reportado la existencia de isoformas mitocondriales y apoplásticas (Scandalios et al., 1980). El peróxido de hidrógeno se remueve además por otros mecanismos en la mitocondria de plantas. Uno de estos mecanismos, es el ciclo glutatión – ascorbato el cual está compuesto por la ascorbato peroxidasa, la dehidroascorbato reductasa y la glutatión reductasa (Jimenez et al., 1997 ; Chew et al., 2003). La ascorbato peroxidasa reduce el peróxido de hidrógeno a agua usando ascorbato, que es reducido por una secuencia de reacciones a cargo de la monohidroascorbato reductasa y la dehidroascorbato reductasa que utilizan glutatión reducido. Por último la glutatión reductasa regenera el glutatión reducido (GSH) a su forma oxidada (GSSG) para continuar el ciclo. 25 Introducción Otras proteínas que intervienen en la respuesta a daño oxidativo son las peroxiredoxinas. Las peroxiredoxinas mitocondriales regulan los eventos de traducción de señales mediados por peróxido a la vez que lo reducen, pudiendo también detoxificar alquilhidroperóxidos y peroxinitrito (Dietz et al., 2006; Finkemeier et al., 2005). Debido a esta actividad, estas proteínas modularían la oxidación lipídica y la señalización relacionada con NO (Baier and Dietz, 2005; Rhee et al., 2005). Las peroxiredoxinas utilizan tioredoxina o glutatión como fuente de poder reductor. Además, en estas reacciones también intervienen las enzimas tioredoxina reductasa o la glutatión reductasa y el NADPH mitocondrial (Gelhaye et al., 2004; Sweetlove and Foyer, 2004). Se informó también que la actividad de la AOX cumple un papel importante en la reducción de los niveles de ROS en las células vegetales (Maxwell et al., 1999). Por otro lado, además de los procesos enzimáticos que hemos descripto, las plantas poseen otros mecanismos para contrarrestar los efectos del estrés sobre las proteínas como la sobreexpresión de chaperonas y otras proteínas estabilizadoras, como así también la acumulación de moléculas orgánicas de bajo peso molecular conocidas como osmolitos o solutos compatibles (Chen and Murata, 2002; Wang et al., 2004). Estos mecanismos influyen sobre el aumento de la estabilidad y la prevención de la agregación de las proteínas y la degradación irreversible de la molécula dañada. 1.6. Hierro y daño oxidativo. Una porción significativa del hierro intracelular en las células de mamíferos es dirigido a la mitocondria, dependiendo de los requerimientos metabólicos celulares (Schueck et al., 2001). La captación citosólica de hierro ocurre cuando el transportador extracelular transferrina se une al receptor de alta afinidad de superficie y es endocitado (Napier et al., 2005). El receptor de transferrina (TfR) es expresado ubicuamente en todas las células (Klausner et al., 1993.). Una vez que el complejo transferrina-TfR es internalizado en los endosomas, el hierro es liberado en el citosol y reducido del estado Fe3+ a Fe2+ soluble utilizando el transportador de metales divalentes (DMT-1) (Garrick et al., 2003). Este transportador está ampliamente expresado como una proteína integral 26 Introducción de membrana que transporta cationes de metales divalentes, incluido el Fe+2 (Gunshin et al., 1997; Fleming et al., 1998). Los mecanismos específicos que determinan y facilitan la captación de hierro por la mitocondria siguen sin definirse (Carraway et al., 2006). Para mantener la homeostasis celular del hierro, la expresión de transportadores citosólicos y de proteínas de almacenamiento (ferritina, DMT-1 y TfR) es rápidamente regulada por cambios en los niveles de hierro celular. El control posttranscripcional de la estabilidad del ARNm y su traducción ocurre a través de la interacción entre proteínas regulatorias de hierro (IRP) y elementos regulatorios de hierro (IRE) ubicados en regiones no transcriptas del ARNm. Cuando los niveles de hierro aumentan, estas interacciones causan un rápido descenso en la expresión de TfR y un aumento de la ferritina (Klausner et al., 1993.; Gunshin et al., 1997; Fleming et al., 1998; Carraway et al., 2006). El hierro es un nutriente esencial para las plantas. Su función es donar y aceptar electrones, hecho fundamental en la cadena de transporte de electrones de la respiración y fotosíntesis. Sin embargo, es tóxico cuando se acumula a altos niveles: puede actuar vía la reacción de Fenton generando radicales hidroxilo que dañan lípidos, proteínas y ácidos nucleicos (Figura 8). Las plantas deben entonces ser capaces de responder ante dos situaciones, la sobrecarga y la deficiencia de hierro (Connolly and Guerinot, 2002,). 27 Introducción Figura 8: Vía metabólica de formación de especies reactivas de oxígeno en plantas. La figura muestra la participación de otras enzimas importantes en esta vía metabólica: APX, ascorbato peroxidasa; GSH glutatión peroxidasa; SDO, superóxido dismutasa. (Tomado de Mori y colaboradosres (Mori and Schroeder, 2004)). Debido a la potencial toxicidad asociada a los altos niveles de hierro, las células lo almacenan en la proteína especializada ferritina. Las múltiples subunidades (más de 24) que componen la ferritina forman una estructura hueca que puede albergar más de 4.500 átomos de hierro en su interior. Así, la ferritina juega un papel importante en la homeostasis de hierro. Los estudios de las ferritinas de plantas han revelado diferencias importantes en su estructura, localización y regulación respecto a las de animales. Mientras que éstas últimas son encontradas en el citosol, las de plantas contienen péptidos tránsito que las dirigen a organelas (Proudhon et al., 1996). Además, mientras que la expresión regulada por hierro de la ferritina animal es controlada principalmente a nivel de traducción mediante el sistema IRE-IRP, los experimentos realizados en maíz y soja demostraron que la expresión está regulada tanto a nivel transcripcional (Lescure et al., 1991; Lobréaux et al., 1992; Wei and Theil, 2000; Petit et al., 2001) como post transcripcional (Fobis-Loisy et al., 1996). Un detalle significativo es que no se han encontrado regiones regulatorias IRE en las ferritinas de plantas (Briat et al., 1999). La mayor cantidad de hierro requerida en una célula eucariota es consumido en su gran mayoría por la vía de síntesis del grupo hemo y la biosíntesis de centros FeS. Estos dos procesos son llevados a cabo por la mitocondria y necesitan del transporte 28 Introducción activo del hierro reducido a través de la membrana interna mitocondrial (Lange et al., 1999). Hace varias décadas se ha postulado la presencia de proteínas capaces de captar hierro en la mitocondria (Flatmark and Romslo, 1975) y estudios recientes han identificado proteínas de la membrana mitocondrial interna que estarían involucradas en transporte activo de hierro mitocondrial (Kispal et al., 1997 ; Foury and Roganti, 2002; Park et al., 2002). Como se comentó más arriba, altas concentraciones de Fe pueden favorecer la producción de radicales ·OH, un potente oxidante que causa daño de moléculas incluyendo al ADN y proteínas. Por el contrario, la deficiencia de Fe interrumpe el normal ensamblado de ferro- y hemoproteínas de la cadena respiratoria mitocondrial (Attama et al, 2001; Lin et al, 2001; Nihei et al, 2001) hecho que conduce también a un desbalance de especies del oxígeno. Una deficiencia en el proceso de síntesis de grupos Fe-S puede provocar la acumulación de Fe3+ y S2-, tóxicos metabólicos que generan daño celular. La mitocondria contiene concentraciones micromolares de Fe biodisponible y uno de los mayores retos biológicos en la célula es mantener este hierro en forma soluble (Fe+2) y no tóxica dado al pH alcalino y la alta producción de anión superóxido y peróxido de hidrogeno que posee la mitocondria (Tangerås, 1985; Mori and Schroeder, 2004). Es esencial, entonces, que exista una fina regulación de la formación y el correcto ensamblado del hierro a las ferrosulfo- y hemoproteínas para que no se acumule hierro a niveles tóxicos. Se ha descripto que en levaduras y animales la proteína frataxina es esencial para la homeostasis celular de hierro y la supervivencia ante el daño oxidativo mientras que en plantas, este fenómeno no había sido abordado hasta el momento. A principios de 2009, se presentaron resultados que vinculan los niveles de hierro intracelulares y el aumento de óxido nítrico (NO) con la deficiencia de frataxina y estrés oxidativo (Martin et al., 2009). 29 Introducción 1.7. Frataxina: estructura y función El primer hallazgo de la existencia de frataxina, surgió a partir de los conocimientos de una enfermedad humana denominada Ataxia de Friedreich (FRDA). Ésta es una enfermedad cardio-neurodegenerativa con baja sobrevida, causada por la deficiencia de frataxina, una proteína codificada a nivel nuclear y de localización mitocondrial. La proteína proveniente de humanos contiene 169 aminoácidos y está codificada por un gen de 507 pb ubicado en el cromosoma 9 (Campuzano et al., 1996; Schapira, 1998; Becker et al., 2001). La deficiencia en la expresión del gen de frataxina es consecuencia de la presencia de una expansión del triplete GAA en el primer intrón (Campuzano et al., 1996). Esta expansión intrónica provoca una conformación inusual en la heterocromatina dando una estructura ȕ –ADN (triple hélice) poco común, que determina el silenciamiento y la subsecuente reducción de la expresión de frataxina (Sakamoto et al., 1999). La mayoría de los pacientes son homocigotas para la expansión GAA, aunque se han encontrado también algunos heterocigotas con una mutación puntual en uno de los alelos y una expansión del triplete GAA en el otro alelo del gen (Cossée et al., 1999). Esta enfermedad es progresiva y en los tejidos especializados de alta demanda energética, (neuronas de los ganglios de la raíz dorsal, células de músculo cardíaco y células ȕ del páncreas) se observa una disfunción mitocondrial y muerte celular (Puccio et al., 2001). La ataxia progresiva de los miembros, la hipertrofia cardíaca y la diabetes mellitus encontrada en estos pacientes se atribuyen a los bajos niveles de ATP producidos en estos tejidos (Lodi et al., 1999). Cuanto mayor sea la expansión del triplete GAA, se produce mayor deficiencia en la expresión de frataxina determinando mayor gravedad de la enfermedad (Campuzano et al., 1997). La frataxina es una proteína que ha sido ampliamente conservada a través de la evolución en bacterias, levaduras, mamíferos y plantas sin presentar mayores cambios estructurales. Esta conservación secuencial, y la presencia de un único y novedoso 30 Introducción plegamiento proteico (llamado dominio frataxina), sugiere que puede cumplir un papel similar en todos estos organismos (Dhe-Paganon et al., 2000; Musco et al., 2000). El dominio frataxina parece ser característico de esta proteína (y los homólogos relacionados) y no se han encontrado hasta la fecha otras proteínas en banco de datos con este dominio particular (Cho et al., 2000; Musco et al., 2000). La Figura 9 muestra la conservación de los elementos correspondientes a la estructura secundaria de frataxinas de diferentes organismos. No existen deleciones o inserciones significativas entre las regiones de Į hélice predichas y las láminas ȕ. 31 Figura 9: Alineamiento de la secuencia de la frataxina humana con homólogos procariotas y eucariotas. En verde agua se muestran los segmentos responsables de la estructura secundaria Į hélice, y en verde brillante los correspondientes a láminas ȕ. Los alineamientos fueron realizados por el programa Multalin. Los residuos iguales se encuentran en rojo. Los residuos altamente conservados en azul. Los residuos en mayúsculas en las secuencias consenso ocurren con una frecuencia de un 83%, los residuos en minúsculas ocurren entre un 67% y 83% de frecuencia (Dhe-Paganon et al., 2000). Introducción El dominio frataxina de la proteína de humanos (residuos 88-210, sin la secuencia N-terminal de importe a mitocondria) presenta una conformación de Įȕsandwich (Figura 10A). Doce residuos acídicos de la hélice Į1 y de la hoja ȕ1 forman una larga y continua zona de alta densidad de carga negativa en la superficie de la proteína creando un dipolo (Figura 10B) (Dhe-Paganon et al., 2000). Debido al tamaño y naturaleza de este dipolo, se postula una potencial interacción de esta proteína con otros ligandos u otras proteínas. Tanto la estructura global como su superficie aniónica, se encuentra conservada en todos los organismos estudiados (Gibson et al., 1996; Cañizares et al., 2000). Nueve de los 12 residuos con carga negativa, descriptos para la frataxina humana, están conservados en AtFH (homologo de frataxina en Arabidopsis thaliana). La presencia de esta superficie con carga negativa, más la conservación de residuos aminoacídicos, sugieren dos cosas: (i) frataxina podría interaccionar con otras proteínas para ejercer su función biológica y (ii) los aminoácidos cargados negativamente podrían ser claves en determinar esta función. Figura 10 A: Estructura de la frataxina. El diagrama muestra el plegamiento de la frataxina, una estructura compacta Įȕ-sandwich. Las Į-hélices están mostradas verde agua y láminas ȕ en verde brillante. Las láminas desde ȕ1 a ȕ5, forman un piso de láminas ȕ antiparalelo, que interacciona con las dos Į hélices. Las dos hélices están cercanamente en paralelo entre ellas y con el plano de la larga lámina ȕ formada. Se forma una pequeña lámina ȕ en el extremo C-terminal de ȕ5 y las láminas ȕ6 y ȕ7 (Dhe-Paganon et al., 2000). 33 Introducción Figura 10 B: Representación de la superficie molecular de frataxina. La superficie molecular fue calculada y sombreada de acuerdo con los potenciales electrostáticos (-10 kt/e, rojo y +10 kt/e, azul) con el programa GRASP (Nicholls et al., 1991). Los residuos individuales en la superficie acídica están marcados (Dhe-Paganon et al., 2000). Se han propuesto diversas funciones en las cuales frataxina podría intervenir, incluyendo homeostasis del hierro y respiración (Babcock et al., 1997), metabolismo del hemo (Lesuisse et al., 2003), ensamblado de centros Fe-S (Chen et al., 2002), fosforilación oxidativa y estrés oxidativo (Ristow et al., 2000) y acumulación de Fe en mitocondria bajo una forma hidrosoluble y no tóxica (Babcock et al., 1997; Isaya et al., 2004). Sin embargo, hasta el momento no está clara su función precisa. En eucariotas, frataxina es necesaria para el normal funcionamiento de la mitocondria (Wilson and Roof, 1997; Koutnikova et al., 1998). Levaduras deficientes en la expresión del homólogo de frataxina, YFH, presentaron una disminución en la respiración (Wilson and Roof, 1997), pérdida de DNA mitocondrial (Koutnikova et al., 1997) y acumulación de hierro en la mitocondria (Babcock et al., 1997). En extractos de mitocondrias de levaduras que carecen de frataxina, la formación de proteínas Fe-S está severamente reducida, sugiriendo su participación en la biogénesis de proteínas Fe-S (Huynen et al., 2001; Muhlenhoff et al., 2002). Se ha descripto recientemente que la frataxina actuaría como una chaperona de hierro y modularía la actividad de proteínas mitocondriales como por ejemplo, la aconitasa (Bulteau et al., 2004). Zhang y col (2005) han sugerido que frataxina jugaría un papel importante en la protección del Fe 34 Introducción biodisponible dentro de la mitocondria, facilitando su utilización no sólo en el ensamblaje de Fe-S proteínas, sino también para síntesis del grupo hemo. Otros trabajos sugieren que frataxina estaría involucrada en la conversión energética mitocondrial y la fosforilación oxidativa. Se informó que la sobreexpresión de frataxina en células de mamíferos causa una inducción del flujo del ciclo de Krebs con un incremento en la actividad respiratoria, aumento del potencial de membrana mitocondrial y elevado contenido de ATP celular (Ristow et al., 2000). La mayoría de los trabajos realizados sobre frataxina fueron llevados a cabo en bacterias, levaduras y mamíferos, no habiendo ninguna descripción de esta proteína en plantas. Como mencionamos más arriba, se aisló recientemente en nuestro laboratorio el gen de frataxina de Arabidopsis thaliana (AtFH, At4g03240). AtFH se encuentra codificado en el cromosoma 4, y está compuesto por cinco exones y cuatro intrones, dando un ARNm de 564 pb que codifica una proteína de 187 aminoácidos y de masa molecular 14kDa. El análisis con el programa PSORT o MITOPROT II sobre la secuencia aminoacídica de AtFH, permitió identificar un posible péptido tránsito para la localización mitocondrial, compuesto por los primeros 31 aminoácidos (Busi et al., 2004). Este resultado coincide con los resultados obtenidos con el homólogo de frataxina de levaduras, ya que estudios de localización celular mostraron que YFH se encuentra en mitocondrias (Cavadini et al., 2000). Además se describió que YFH es procesado por proteasas para dar luego la forma madura y funcional de la proteína (Branda et al., 1999). Es importante destacar que el homólogo de frataxina de humanos es doblemente procesado, dando lugar también a la forma madura de la proteína con posible localización en el espacio intermembrana mitocondrial (Cavadini et al., 2000). Se llevaron a cabo también ensayos de complementación con AtFH de células de Saccharomyces cerevisiae mutantes nulas para la expresión de frataxina (ǻyfh). La cepa mutante nula muestra baja tasa respiratoria y alta sensibilidad a agentes oxidantes. Los resultados de complementación mostraron que AtFH restauró la tasa respiratoria y provocó una disminución a la sensibilidad a agentes oxidantes en levadura complementada (Busi et al., 2004). Estos datos permiten postular que AtFH sería una 35 Introducción proteína funcional y que podría estar involucrada en la respiración mitocondrial y en respuesta a estrés oxidativo. Por lo tanto, aunque se han sugerido diversas funciones para frataxina, su función precisa queda aún por dilucidarse. Hasta el momento no existían reportes sobre la presencia o función de esta proteína en organismos vegetales. 1.8. Biogénesis de hemoproteínas Como describimos más arriba, la función de la mitocondria, depende de un continuo suministro de hierro para la formación de centros Fe-S (ISC) y para la vía de biosíntesis del hemo (O'Neill et al., 2005). Estos dos procesos son llevados a cabo dentro de la organela (Muhlenhoff et al., 2002; Lange et al., 2004). Los tetrapirroles son compuestos que juegan un papel vital en varios procesos celulares, incluídos la fotosíntesis y respiración. Las plantas superiores contienen cuatro clases de tetrapirroles: clorofilas, hemo, sirohemo y fitocromobilinas. Se ha descripto que todos los tetrapirroles derivan de una vía fotosintética común (Tanaka and Tanaka, 2007) (Figura 11). La vía de biosíntesis de tetrapirroles presenta numerosas ramificaciones. Las etapas que van desde glutamato a uroporfirinógeno III son comunes para la síntesis en plantas superiores de los cuatro tetrapirroles antes mencionados. Los tres pasos siguientes, hasta la producción de protoporfirina IX son comunes para la biosíntesis de clorofila, hemo y fitocromobilina. Finalmente, las últimas etapas son únicas para cada uno de los tetrapirroles descriptos (Tanaka and Tanaka, 2007). 36 Introducción 37 Introducción Figura 11: Biosíntesis de tetrapilloles. Los números indican las enzimas que catalizan cada reacción. 1: glutamil t-RNA sintetasa, 2: glutamil t-RNA reductasa, 3: glutamato 1- semialdehído aminotransferasa, 4: ALA dehidratasa o porfobilinógeno sintasa, 5: porfobilinógeno deaminasa, 6: uroporfirinógeno III sintasa, 7: uroporfirinógeno III decarboxilasa, 8: coproporfirinógeno III oxidasa (CPOX), 9: protoporfirinógeno IX oxidasa (PPOX), 10: magnesio quelatasa, 11: magnesio protoporfirina IX metiltransferasa, 12: magnesio protoporfirina IX monometilester ciclasa, 13: protoclorofilina oxidoreductasa, 14: divinilclorfilina reductasa, 15: clorofila sintasa, 16: clorofilina a oxigenasa, 17:clorofilasa, 18 y 19: clorofila b reductasa, 20: metiltransferasa, 21: oxidasa no identificada hasta el momento, 22: sirohidroclorin ferroquelatasa, 23: ferroquelatasa (FeCh), 24: hemo oxigenasa, 25: fitocromobilin sintasa. La mayor parte de la síntesis de tetrapirroles en plantas ocurre en los cloroplastos. Esta localización no es sorprendente para la rama de bifurcación de la biosíntesis de sirohemo y clorofilas, ya que los productos finales de esta rama funcionan exclusivamente en plástidos. A su vez, todos los pasos metabólicos identificados para la síntesis de fitocromobilina, también están localizados en el cloroplasto a pesar que la unión del cromóforo a la apoproteína ocurre en el citosol (Kohchi et al., 2001; Terry et al., 2002). La síntesis del grupo hemo es bastante más compleja, y su localización exacta es todavía incierta, particularmente en organismos vegetales. Se ha sugerido que las últimas tres enzimas de la vía de biosíntesis de hemo, CPOX (coproporfirinógeno oxidasa III), PPOX (protoporfirinógeno oxidasa IX) y FeCh (ferroquelatasa) se localizan tanto en cloroplastos como en mitocondrias, pero hasta el momento no existe un consenso al respecto (Tanaka and Tanaka, 2007). Recientemente, Williams y col. han reportado la presencia de dos isoformas de CPOX en maíz, una de localización cloroplástica y la otra mitocondrial (Williams et al., 2006). Por otro lado, Santana y col. describieron que uno de los dos genes homólogos de CPOX de Arabidopsis thaliana (At4g03205) contiene una deleción que causa un cambio en el marco abierto de lectura del gen, concluyendo que se trata de un pseudogen (Santana et al., 2002); mientras que la otra isoforma de CPOX (At1g03475) se localiza exclusivamente en el cloroplasto. Estos datos son consistentes con las observaciones realizadas por Smith y col. que muestran que la actividad de CPOX está localizada en cloroplastos de hojas etioladas de arvejas y no en mitocondrias (Smith et al., 1993). En contraste, la actividad de PPOX, fue detectada tanto en cloroplastos como en mitocondrias de arvejas y cebada (Jacobs 38 Introducción and Jacobs, 1987; Smith et al., 1993). De acuerdo con estos resultados, la presencia de al menos dos isoformas de PPOX ha sido informada en A. thaliana y tabaco (Narita et al., 1996; Li et al., 2003). A diferencia de lo que ocurre con PPOX, existen algunas controversias sobre la exacta localización de las isoformas de FeCh en mitocondrias de plantas. Cornah y col. han reportado que la mayor actividad de la ferroquelatasa en arvejas, ha sido asociada a cloroplastos (Cornah et al., 2002). Masuda y col., han encontrado que dos isoformas FeCh de pepino, fusionadas a GFP, se dirigían al cloroplasto y no a mitocondria (Masuda et al., 2003). Lister y col. describieron que dos isoformas de la ferroquelatasa de A. thaliana son importadas hacia el cloroplasto in vitro (Lister et al., 2004). Otros trabajos realizados por van Lis y col., informaron sobre la existencia de un único gen de FeCh en C. reinhardtii con localización cloroplástica, sugiriendo que en este organismo el hemo se produciría únicamente en el cloroplasto y se exportaría al citosol y a la mitocondria (van Lis et al., 2005). La información detallada en el párrafo anterior indicaría que la FeCh se localizaría únicamente en el cloroplasto, pero que una fracción de FeCh o una proteína no identificada aún, presentaría una actividad mínima en mitocondria. Por otro lado, si la proteína ferroquelatasa no existiera en mitocondria, no debería haber una necesidad biológica para que exista PPOX en esta organela (Tanaka and Tanaka, 2007). El grupo hemo es una de las moléculas más importantes en las células de plantas superiores. Aunque es menos abundante que las clorofilas, el hemo posee funciones muchos más diversas. Constituye el grupo prostético de citocromos fotosintéticos y respiratorios involucrados en traducción de energía; de oxidasas como la catalasa, peroxidasa y NADPH oxidasa; de hemoglobinas incluídas leghemoglobinas, involucradas en homeostasis del oxígeno; así como también se lo encuentra en los citocromos P450 interviniendo en la biosíntesis de varios metabolitos secundarios (Pal Singh et al., 2002). Por otro lado el hemo está implicado en el fino control de la síntesis de los tetrapirroles, a través de la inhibición de la enzima glutamil-tRNA reductasa, 39 Introducción involucrada en la producción de la primer molécula de la vía, ácido 5- aminolevulínico (Terry and Kendrick, 1999). Hemos discutido que la última enzima de la vía de biosíntesis del hemo es la ferroquelatasa, que cataliza la incorporación de Fe en estado ferroso dentro del anillo porfirínico, y es el último intermediario común entre la vía del hemo y de clorofilas. Es por esto, que FeCh sería importante en la regulación de esta bifurcación metabólica entre las dos vías, y tendría un papel relevante en la coordinación de la producción de hemo y aproproteínas (Lange et al., 2004). Diferentes trabajos asociaron a la proteína mitocondrial frataxina en el ensamblado de centros Fe-S (Rotig et al., 1997; Huynen et al., 2001; Duby et al., 2002; Puccio and Kœnig, 2002). Sin embargo, hasta el momento no está clara la participación de frataxina en la biosíntesis de hemo. El nexo entre el metabolismo de Fe y la proteína frataxina se estableció originalmente por la caracterización de unas cepas de levaduras a las cuales se les había delecionado el homologo de frataxina (YFH1) (Babcock et al., 1997). En estas levaduras mutantes se acumulaba Fe dentro de las mitocondrias formando un precipitado tóxico inorgánico (Lesuisse et al., 2003); al mismo tiempo, proteínas que contenían Fe, incluyendo las Fe-S proteínas y hemoproteínas decrecían o eran inactivas. Se informó que levaduras deficientes en frataxina contienen bajos niveles de citocromos. Además, los niveles de ferroquelatasa (Hem15) también están reducidos, posiblemente por una represión transcripcional, pero sin alterar los niveles de hemo celulares (Lesuisse et al., 2003). En células de mamíferos, se describió que la deficiencia de frataxina lleva a la regulación en menos de algunos transcriptos que codifican enzimas de la vía de síntesis del hemo (Schoenfeld et al., 2005). Recientemente, Zhang y col reportaron que levaduras mutantes en frataxina mostraban defectos en la síntesis de hemo (Zhang et al., 2005). A partir de los resultados mencionados se postuló que frataxina jugaría un papel importante en la protección del Fe biodisponible dentro de la mitocondria, facilitando su utilización no sólo en el ensamblaje de Fe-S proteínas, sino también para síntesis del grupo hemo. 40 Hipótesis y Objetivos Hipótesis y Objetivos Hipótesis de trabajo: En los últimos años se intensificó el estudio del ensamblaje de los centros FeS y su inserción en polipéptidos para producir proteínas funcionales. Sin embargo, existe muy poca información sobre la ocurrencia de estas vías en organismos vegetales, siendo los organismos más estudiados las levaduras, bacterias y mamíferos. Investigaciones recientes sugieren que en plantas, tanto la mitocondria como los plástidos tendrían su propia maquinaria para la producción de ferrosulfoproteínas. La proteína mitocondrial, AtFH de Arabidopsis thaliana estaría involucrada en múltiples funciones tales como conversión energética y fosforilación oxidativa, participaría en ensamblaje de clusters Fe-S a proteínas, biosíntesis de hemoproteínas y en el metabolismo del hierro. Objetivo: El objetivo general de este trabajo es identificar y caracterizar proteínas y ligandos que participan en la biogénesis de proteínas Fe-S y hemoproteínas mitocondriales. El estudio está enfocado a las proteínas que regularían el ensamblaje de los complejos respiratorios de la membrana interna de la mitocondria (debido a que muchos de ellos contienen ferrosulfoproteínas y hemoproteínas) que están poco caracterizadas, especialmente en plantas. Objetivos específicos: 1- Determinar el papel fisiológico de AtFH a través de pruebas de pérdida y ganancia de función: Caracterizar plantas deficientes en AtFH (mutantes insercionales por T-DNA y plantas transgénicas que expresan el gen de AtFH en orientación antisentido, as-AtFH) y plantas que sobreexpresen AtFH (s-AtFH). 42 Hipótesis y Objetivos 2- Clonar, expresar y purificar AtFH recombinante y determinar su función bioquímica in vitro. 3- Analizar el efecto de la deficiencia de AtFH sobre la respuesta transcripcional nuclear, especialmente de genes que codifican proteínas relacionadas con la biogénesis de mitocondrias. Determinar su papel en la biogénesis de proteínas que contienen grupos Fe-S, particularmente las de localización mitocondrial. 4- Evaluar la participación de AtFH en la respuesta a estrés oxidativo en plantas. 5- Estudiar la participación de AtFH en la síntesis del grupo hemo y su participación en la biogénesis de hemoproteínas 43 Materiales y Métodos Materiales y Métodos 3. Materiales y Métodos 3.1. Material vegetal y condiciones de crecimiento Plantas de A. thaliana var. Columbia-0 (Col-0) fueron utilizadas como línea tipo salvaje (wild type, wt). Las tres líneas mutantes, atfh-1 (SALK_021263), atfh-2 (SALK_ 094203) y atfh-3 (SALK_122008) fueron obtenidas de la T-DNA Express Collection en el Salk Institute (http://signal.salk.edu/cgibin/tdnaexpress). Las mismas contienen una inserción de T-DNA dentro de la secuencia del gen AtFH. En la línea atfh-1, el T-DNA se encuentra en la región no traducida (5´UTR), por delante del codón ATG de iniciación de la traducción. Las otras dos líneas atfh-2 y atfh-3 contienen la inserción de T-DNA en el primer intrón y el cuarto exón respectivamente. Las semillas de cada una de las líneas fueron germinadas directamente en tierra y mantenidas a 4°C al menos por 72 h. Las plantas fueron crecidas en condiciones de invernadero a 25°C bajo luz fluorescente (Grolux, Sylvania y Cool White, Philips) con una intensidad de 150 ȝmol m-2 s-1 utilizando un fotoperíodo de 16 h. luz/8 h. oscuridad. 3.2. Cepas bacterianas En este trabajo se utilizaron diferentes cepas de Escherichia coli y Agrobacterium tumefaciens: 1) E. coli XL1-Blue: recA1 endA1 gyrA96 thi-1 hsdR17 supE44 relA1 lac [F’proAB lacIZ ǻM15 Tn10 (Tet r)]. 2) E. coli BL21 CodonPlus-RIL: F- ompT hsdSB (r B- m-B) dcm+ Tetr Gal endA Hte [argU ileY leuW Camr. 3) A. tumefaciens: cepa GV3101; plásmido Ti: pTiC58; chromo C58; opines:Nopalina. 45 Materiales y Métodos 3.3. Vectores plasmídicos Los vectores utilizados en este trabajo y sus características se detallan en la Tabla M 1. Tabla M 1. Características de vectores utilizados pET24a Novagen Expresión de proteínas nºcatálogo: 69749-3 pDH51 (Pietrzak y col., 1986) Casete promotor terminador CaMV35S , ampR. y Cedido por el lab. Dr. Carrillo, UNR, Argentina pCAMBIA 1302 CAMBIA Expresión de proteínas en plantas, hyg R (plantas), KanR (bacteria) Cedido por el lab. del Dr. Araya, REGER, CNRS, Francia 3.4. Medios de cultivo, antibióticos y otros reactivos 3.4.1. Medios de cultivo bacteriano Medio LB (Luria Bertani) líquido: Tripteína 10 g; Extracto de levadura 5 g; NaCl 10 g; agua c.s.p. 1 litro. Medio LB agar: Idem medio LB líquido con el agregado de 15 g/l de agar. Medio MS (Murashige y Skoog): (Murashige y Skoog, 1962). Nº catálogo: M0404 (Sigma-Aldrich, St. Louis, USA). Medio MS cultivo de células: Murashige y Skoog shoot multiplication médium A (MSMA) (Murashige y Skoog, 1962). Nº catalogo: M7024 (Sigma-Aldrich, St. Louis, USA). 3.4.2. Medios líquidos y soluciones buffer 10 X TBE buffer: TRIZMA base 108 g; ácido bórico (H3BO3) 55 g; 0.5 M EDTA, pH 8 40 ml; agua c.s.p. 1 litro. TE buffer: TRIZMA base 10 mM; EDTA pH 8.0 1 mM; agua c.s.p. 800 ml; ajustar pH 8 con HCl; agua c.s.p. 1 litro. 46 Materiales y Métodos 20 X SSC: NaCl 3 M; acetato de sodio (C6H5Na3O7.2H2O) 0.3 M; agua c.s.p. 800 ml; HCl 1 M hasta pH 7.0; agua c.s.p. 1 litro. 10 X PBS: NaCl 80 g; KCl 2 g; Na2HPO4.7H2O 26.8 g; KH2PO4 2.4 g; agua c.s.p. 800 ml; HCl hasta pH 7.4; agua c.s.p. 1 litro. 3.4.3. Antibióticos y otros compuestos relacionados Los antibióticos, así como algunos compuestos usados en la selección de clones positivos, se detallan en la Tabla M 2. Tabla M 2. Antibióticos y otros compuestos relacionados utilizados en el estudio Compuesto Concentración stock Concentración en cultivo ampicilina 50 mg / ml 50 mg/l gentamicina 25 mg / ml 25 mg / l kanamicina 50 mg / ml 50 mg / l rifampicina 100 mg / ml 100 mg / l cloranfenicol 25 mg / ml 25 mg / l higromicina 50 mg/ ml 20 ȝg/ ml ( en placa) 1M 250 ȝM – 1 mM Isopropil ȕ-D-tiogalacto- piranósido (IPTG) 3.5. Oligonucleótidos Los oligonucleótidos fueron sintetizados por Sigma-Aldrich, St. Louis, USA. En la Tabla M3 se describe la secuencia de los distintos cebadores utilizados, el gen sobre el cual fueron diseñados y el sitio de restricción (si corresponde). 47 Materiales y Métodos Nombre Secuencia (5´-3´) Gen Sitio restricción NPTII Fw TGGATTGCACGCAGGTTCTCCG NPTII NPTII Rv CGAGGAAGCGGTCAGCCCATTC Oligo dT pd (T)12-18 Act2 Fw AATCTCCGGCGACTTGACAG Actina Act2 Rv AAACCCTCGTAGATTGGCACAG At3g18780 *Act2Fw TGCTGGAATCCACGAGACAACCTA Actina Act2Rv TGCTGAGGGAAGCAAGAATGGAAC At3g18780 *cbp Fw CCGGCCTATTCGTGTGGATTTTGA CBP cbp Rv CATAATTCGTTGGCGCAGCTTGAG At5g44200 *ucb Fw GACTCCTTACGAAGGCGGTGTTTT UCB ucb Rv GCTCAGGATGAGCCATCAATGCTA At5g25760 p-CaMV35S ATGTGATATCTCCACTGACGTAA p-CaMV 35S 35StermFw ATTTCAGCGGGCATGCCTGCAG t-CAMV35S CAL Fw AATTCGAAGATGGATGGG Calreticulina CAL Rv AACATAATGCTGTAAGGA At1g56340 AOX Fw ATTTTTTCAGAGACGATA Oxidasa AOX Rv GCGAATGTCAGAAGCAAA alternativa At5g64210 PEROX Fw ATCTTCAAGGGGAAGAAA Peroxirredoxina PEROX Rv GCCGACCATCTCTCAGAC At3g06050 Aco Fw GAAGTGATTCCAACAAAATCAA Aconitasa Aco Rv AACACCAGGCAATACCATACTC At4g26970 SDH21 Fw AAACATATGGCGGAGGCGGAAACAAAA Succinato SDH21 Rv AAACTCGAGACGCTGAAGTTGCTTGAT deshidrogenasa At2g18450 Fra5New AAACATATGGCTACAGCTTCAAGG Fw AtFH At4g03240 FRATEN Fw AAAGGATCCATGCTTTGTGTATTT At4g03240 fra2X Fw GGGCATATGGAGGAAGAGTTTCACAAA At4g03240 NdeI 48 Materiales y Métodos AtFH Fra-his Rv AAACTCGAGTGAGAGTTGGATTGGTTC XhoI At4g03240 Fra-stop Rv AAACTCGAGTTATGAGAGTTGGATTGGTTC At4g03240 XhoI as-AtFHbam Fw AAAGGATCCATGGCTACAGCTTCAAGG At4g03240 BamHI 5´UTR Fw GTGACAGAACCGCCATGGCTACA At4g03240 Sma I TGTTTTAAAGCCTTCTTTATGAGA At4g03240 PSST Fw GCCTAGTCCTCGCCAGTCTG PSST PSST Rv GCTGGAGTAGTCCATAGAGC At5g11770 TYKY Fw GATTTTGGCTCGCAGGTCACTG TYKY TYKY Rv GCGGACCATTTCTGTGAGGAAC At1g79010 NADH BP Fw GAAAGCTGTGGGCAGTGCAC NADH BP Rv TCTCGAGCTCTGGCCTAAAG 3´UTR Rv NADH binding protein At5g08530 Catalasa1 CAT1 Fw GATAAGCTACTCCAGACCCGGA CAT1 Rv CTGAGACCAGTAAGAGATCCA CAT2 Fw GATAAGCTGCTTCAAACCCGTG CAT2 Rv CTGAGACCAGTAAGAGATCCA CAT3 Fw ACGACAAGCTGCTCCAGTGTA CAT3 Rv CTGAGACCAGTAAGAGATCCA *AtFEQ1 Fw CAGTCAGTTTCGTGAGTGAGCACA Ferroquelatasa1 AtFEQ1 Rv TCTGAGTCAACCACTGCATTTGGG At5g26030 *AtFEQ2 Fw CAAACCTTGAAGCTCGACAGTCGT AtFEQ2 Rv CAAGATGCCCCACTGATGCAGAAA At1g20630 Catalasa2 At4g35090 Catalasa3 At1g20620 Ferroquelatasa2 At2g30390 Porfobilinógeno *AtHemB1 Fw TGCTGACGTAGTTAGTCCGAGTGA sintasa1 AtHemB1 Rv TCACGTGCCTCAATCAATGCCT At1g69740 *AtHemB2 Fw AGGCATTGCTAGAAGCACGAGAAG AtHemB2 Rv GGCGGATGCATAACAATGACTCCA Porfofilinógeno sintasa2 At1g44318 Coproporfirinógeno *AtHEMF2 Fw AACCTGGTCCGATTCCGTTCTTTG AtHEMF2 Rv ACGACCGAATGAAAGTGCTTGACG sintasa2 At4g03205 * Oligonucleótidos utilizados para la técnica de PCR en tiempo real. 49 Materiales y Métodos 3.6. Preparación y análisis de ADN 3.6.1. Propagación de ADN plasmídico 3.6.1.1. Preparación de bacterias Escherichia coli competentes Reactivos y soluciones utilizados: Medio LB-agar y LB-liquido. Solución A (MgCl2 100 mM), Solución B (CaCl2 100 mM) y Solución C (CaCl2 100 mM, 15% V/V glicerol). Se crecieron células de Escherichia coli en una placa de LB-agar durante 12 h. a 37°C. A partir de una de las colonias crecidas, se inoculó en 3 ml de medio LB. El cultivo se creció a 37°C con agitación durante 12 h. Posteriormente, 1 ml de este cultivo se utilizó para inocular 100 ml de medio LB y se incubó a 37°C con agitación hasta alcanzar la fase logarítmica de crecimiento (determinada por cuantificación de la absorbancia a 600 nm). Posteriormente, se centrifugó el cultivo a 4°C y 3000 × g durante 10 min. El precipitado resultante se resuspendió en 50 ml de solución B y se incubó en hielo durante 20 min. Posteriormente se centrifugó a 4°C y 2100 × g durante 10 min. Finalmente el precipitado se resuspendió en 1 ml de solución C y el homogenado conteniendo las células competentes se fraccionó en alícuotas de 100 ȝl que fueron guardadas a -80°C hasta su uso. 3.6.1.2. Transformación de bacterias competentes de E. coli. Se transformaron 100 ȝl de células de E. coli (XL1-Blue o BL21, según corresponda), con 10 ȝl de ADN proveniente de las reacciones de ligación o con 2 ȝl de ADN plasmídico (10-20 ng/μl). Las células se incubaron durante 30 min. a 4ºC. Posteriormente, la mezcla fue sometida a un shock térmico (45 s a 42ºC seguido de 15 min. en hielo). Se agregaron 600 ȝl de medio LB y se incubó por 1 h. a 37ºC con agitación. Finalmente, las bacterias transformadas fueron crecidas en medio LB-agar con el antibiótico correspondiente, durante 18 h a 37ºC. 50 Materiales y Métodos 3.6.2. Aislamiento de ADN plasmídico 3.6.2.1. Aislamiento de ADN plasmídico (kit comercial) Para la obtención de ADN plasmídico se utilizó el equipo comercial “GenElute™ Plasmid Miniprep Kit” (Sigma-Aldrich, St. Louis, USA), según las instrucciones del fabricante, obteniéndose ADN libre de ARN. Este método se utilizó para obtener ADN plasmídico con el que se llevaron a cabo procesos de secuenciación, clonado o digestión con enzimas de restricción. 3.6.2.2. Aislamiento de ADN plasmídico mediante lisis alcalina La extracción de ADN plasmídico para el análisis rutinario de colonias se realizó según el método de Sambrook con algunas modificaciones (Sambrook et al., 1989). Se centrifugó 3 ml de cultivo de células en fase estacionaria de crecimiento durante 1 min. a 12000 × g. A continuación, se resuspendió el precipitado en 200 ȝl de solución P1 (Glucosa 50mM, Tris-HCl 25mM, pH 8.0, EDTA 10mM, pH 8.0). Posteriormente se adicionó 300 ȝl de solución P2 (NaOH 0.2N, SDS 1% P/V) y se mezcló por inversión, hasta lograr la clarificación de la solución y se incubó durante 5 min. en hielo. Se agregó 300 ȝl de solución P3 (acetato de potasio 3M, pH 4.8), se mezcló rápidamente por inversión y se incubó durante 5 min. en hielo. Posteriormente se centrifugó durante 20 min. a 12000 × g. El sobrenadante fue transferido a otro tubo, se agregó 5 ȝl ARNasa A (1 ȝg/ȝl) y se incubó a 37ºC durante 30 min. Luego se agregó 400 ȝl de cloroformo, se agitó por inversión y se centrifugó a 13400 rpm durante 5 min. La fase superior se transfirió a otro tubo. Para lograr la precipitación del ADN plasmídico se agregó igual volumen de isopropanol, se mezcló por inversión y la mezcla fue incubada en hielo durante 25 min. Luego de centrifugar a 13400 rpm durante 25 min. se retiró el sobrenadante y se agregó 500 ȝl de etanol 70% y se centrifugó durante 5 min. Finalmente, se retiró el sobrenadante, se dejó secar el precipitado y el ADN se resuspendió en un volumen adecuado de agua estéril (aprox 40 μl). 51 Materiales y Métodos 3.6.3. Electroforésis de ADN en geles de agarosa Las moléculas de ADN con un tamaño comprendido entre 0.2 y 10 Kb se separaron en geles de agarosa 0.8% P/V con bromuro de etidio (5 mg/ml). Estos geles fueron sometidos a electroforesis horizontal en un campo eléctrico de dirección constante, utilizando el Buffer TBE 1 X. Los fragmentos fueron visualizados en un transiluminador UV y sus tamaños fueron determinados por comparación con marcadores de masa molecular comerciales (400 bp, 100 bp, Productos bio-Lógicos, UNQ) 3.6.4. Purificación de ADN a partir de geles de agarosa Los fragmentos de ADN de interés se aislaron de los geles de agarosa y se purificaron utilizando el kit comercial “GFXTM PCR DNA and Gel Band Purification kit” (GE Healthcare Bio-Sciences, Uppsala, Sweden). El ADN se purificó siguiendo las instrucciones del fabricante. 3.7. Manipulación enzimática de ADN 3.7.1. Reacción de ligación Las reacciones de unión de los fragmentos aislados de ADN a los vectores plásmídicos se llevó a cabo de la siguiente manera: Se utilizó una relación inserto:vector 3:1. Se mezcló el inserto con el vector más 1 ȝl de buffer de ligación 10X (Tris-HCl 60 mM pH 7.8, MgCl2 20 mM, DTT 20 mM, ATP 2 mM y PEG 10 % V/V) y 1 ȝl de T4 ADN ligasa. A continuación, se agregó H2O destilada c.s.p. 10 ȝl y por último, se incubó la reacción en un baño a 16°C durante 18 h. 3.7.2. Reacciones de digestión con enzimas de restricción Todas las reacciones de digestión realizadas siguieron un mismo procedimiento que consistió en tratar 50-100 ng de ADN lineal y el vector plasmídico con 10-15 U de enzima de restricción, 2 ȝl de buffer 10X específico para cada enzima, según las indicaciones del fabricante, y H2O destilada estéril c.s.p. 20 52 Materiales y Métodos ȝl. La mezcla de reacción se incubó durante 1 h. a la temperatura correspondiente para cada enzima. En el caso de las digestiones realizadas con dos enzimas de restricción el tiempo de incubación se incrementó a 3 h. 3.8. Reacción en cadena de la polimerasa (PCR) Todas las reacciones de PCR realizadas se llevaron a cabo utilizando buffer de PCR 1X (Promega, WI, USA), dNTPs 0.4 mM, 0.4 ȝM de cada oligonucleótido, MgCl2 2 mM, Taq Polimerasa 1 U, H2O c.s.p. 25 ȝl y ADNg (ADN genómico) o ADNc (ADN copia) como molde. El protocolo general de amplificación constó de un ciclo de desnaturalización inicial a 94°C durante 4 min., un número determinado de ciclos: fase de desnaturalización a 94°C 30 seg; fase de hibridación a la temperatura óptima para cada juego de oligonucleótidos determinado por gradiente de temperatura; fase de elongación 1 min. por cada 1000 pb a amplificar, a 72°C, más una extensión final de 10 min. a la misma temperatura. El número de ciclos se estableció en función del objetivo buscado en cada experimento. 3.9. Extracción de ARN total El ARN total de diferentes tejidos fue extraído usando el reactivo Trizol (Invitrogen Corp. Carlsbad, CA, USA), compuesto por una mezcla de fenol y tiocianato de guanidina. Cada muestra se homogeneizó con nitrógeno líquido, según la relación 50-100 mg de tejido: 1 ml de Trizol. Se agregó 200 μl de cloroformo y se agitó vigorosamente de forma manual durante 15 s. La mezcla se dejó reposar durante 3 min. a temperatura ambiente y luego se centrifugó 15 min. a 12000 × g a 4°C. Luego de esta etapa se observa una separación en la mezcla de dos fases, una inferior orgánica de color rojo y otra superior, acuosa e incolora, que contiene al ARN. Se transfirió la fase superior a otro tubo y se agregó 188 μl de alcohol isopropílico. Luego de agitar, se dejo reposar a temperatura ambiente durante 10 min. Posteriormente se centrifugó a 12000 × g durante 10 min. a 4°C y se descartó el sobrenadante (el ARN precipita en el fondo del tubo). Se lavó exhaustivamente el precipitado con etanol 70% y se agitó utilizando un vortex durante 2 min. Se 53 Materiales y Métodos centrifugó a 7000 × g durante 5 min. a 4°C y se descartó el sobrenadante. El ARN se resuspendió en agua tratada con DEPC (dietilpirocarbonato, para la inactivación de ARNasas). El material así obtenido se encuentra en condiciones de ser procesado por RT-PCR. 3.10. Cuantificación de ARN total La cantidad de ARN total se determinó espectrofotométricamente a 260 nm (1 unidad de absorbancia equivale a 40 μg de ARN). La pureza de las muestras obtenidas se determinó mediante la relación de absorbancia 260 nm/280 nm. 3.11. RT-PCR semicuantitativa La RT-PCR es un método específico y sensible muy utilizado para medir transcriptos de ARN y la detección de cambios en sus niveles de transcripción bajo diferentes condiciones experimentales, utilizando como referencia un gen constitutivo. El ARN de hojas y flores fue extraído según se detalló en la Sección 3.9. Para los estudios de RT-PCR, el ARN fue sometido a un tratamiento con ADNasa I para eliminar las impurezas de ADN, según se detalla a continuación: 5 μg ARN; 1 μl Buffer reacción; 2 U ADNasa I (Qiagen Inc., Valencia, CA, USA). La mezcla se incubó durante 25 min. a temperatura ambiente y luego se adicionó 1 μl de EDTA 25 mM y se incubó 20 min. a 70°C. Posteriormente, se realizó la síntesis de ADNc utilizando una transcriptasa reversa M-MLV RT (Moloney Murine Leukemia Virus Reverse Transcriptase, USB Corp. Cleveland, OH, USA). Para ello se cuantificó espectrofotométricamente el ARN y se procesó según el siguiente protocolo: 54 Materiales y Métodos Hexámeros Random pd(N)6 (Amersham Biosciences, UK) (1 μg/ul) 1 μl ARN 5 μg Buffer 5X M-MLV 5 μl dNTPs (10 mM) 0.5 μl Inhibidor de ARNasas (Amersham Biosciences, UK) 25 U M-MLV RT H2O DEPC c.s.p. 200 U Vol.final 25 μl La mezcla de reacción se incubó durante 1 h. a 42°C y luego a 70°C durante 15 min. Una vez terminada la RT se realizó la reacción en cadena de la polimerasa (PCR) a fin de evaluar los niveles de ARNm de cada gen. Los genes de ȕ-ACTINA o ARNr 18S (Ambion Inc. Austin, TX, USA) se utilizaron como control interno. El número de ciclos mínimo de PCR para los genes constitutivos fue de 24. Para poder comparar las muestras entre sí se hicieron distintas diluciones de las mismas a fin de tener en todas la misma cantidad de partida de ADNc.. En el caso del ARNr 18S se utilizó una relación de primers 18S: competímeros de 2:9, respectivamente, para modular la eficiencia de amplificación del ARNr 18S, de acuerdo con las instrucciones del fabricante. Para evaluar los niveles de los diferentes transcriptos, se realizaron reacciones de PCR a 16, 20, 24 y 28 ciclos utilizando como molde ADNc. Para cada ADNc se determinó el número apropiado de ciclos a fin de obtener los datos durante la fase exponencial de la reacción de PCR. 3.12. PCR en tiempo real (QPCR) A partir de ARN total extraído con Trizol (ver sección 3.9) se realizó una transcripción reversa para obtener ADNc utilizando el kit Superscript II Reverse transcriptase. El medio de reacción contenía ARN (2 a 20 μg), 2 μl de dNTPs 10 mM, 2 μl de oligo dT (23mer), 2.5 μg/ul y H2O csp 26 μl. El medio se incubó a 65°C por 10 min. y luego se enfrió en hielo. Posteriormente se agregó 8 μl de Buffer 5X, 4 μl de DTT y 2 μl de Superscript II en un volumen final de 40 μl. La mezcla de reacción se incubó 1h. a 42°C y luego 10 min. a 70°C. El ADNc se purificó utilizando el kit QIAquick PCR purification kit (Qiagen Inc. Valencia, CA, USA). 55 Materiales y Métodos La PCR cuantitativa se realizó en placas de 96 pocillos. El medio de reacción contenía 4.3 μl de primers fw y rv (7.5 μM), 10 ng de ADNc y 12.5 μl de mezcla de reacción (Maxima SYBR Green qPCR Master Mix 2X, Fermentas). Las reacciones se llevaron a cabo en un termociclador iCycler (BioRad, Hercules, CA, USA). Como control interno se utilizaron los genes de ȕ-ACTINA, CBP y UCB. 3.13. Extracción de ADN genómico El ADN genómico (ADNg) extraído de hojas de A. thaliana fue utilizado para la caracterización genotípica de las líneas mutantes por T-ADN. Se partió de aproximadamente 100 mg de tejido vegetal, el cual se trituró en N2 (l) en microtubos. Se agregó 200 μl de buffer bromuro de cetil-trimetil-amonio (CTAB) y se trituró hasta lograr la completa homogeneización del tejido. Posteriormente, se agregó 100 μl más de buffer CTAB y se incubó a 60ºC durante 30 min. Se centrifugó a 13400 rpm durante 5 min. y al sobrenadante se le agregó 300 μl de cloroformo. Después de agitar, se centrifugó nuevamente a 13400 rpm por 5 min. La fase acuosa (superior) fue transferida a otro tubo al que se le agregó un volumen igual de isopropanol y se precipitó el ADN por 20 min. a temperatura ambiente. El precipitado obtenido luego de centrifugar a 7000 × g por 10 min. fue lavado etanol 70% V/V y secado. Finalmente, el precipitado se resuspendió en 20 μl de agua destilada estéril y se cuantificó espectrofotométicamente. 3.14. Cuantificación de ADN La cantidad de ADN total se determinó espectrofotométricamente a 260 nm (1 unidad de absorbancia equivale a 50 μg/μl de ADN). La pureza de las muestras obtenidas se determinó mediante la relación de absorbancia 260 nm/280 nm. 3.15. Hibridación de ácidos nucleicos (Southern-blot) Se realizó una electroforesis en gel de agarosa 0.8% P/V en TBE 1X a 100 V. Los geles fueron tratados a 254 nm por 2 min. en un horno UV Crosslinker (BioRad 56 Materiales y Métodos Laboratories, Munich, Germany). Posteriormente, los geles se incubaron durante 30 min. en solución desnaturalizante (NaOH 0.5 N, NaCl 1.5 M) con agitación. Se lavó 3 veces con agua destilada y luego se incubaron durante 30 min. en solución de renaturalización (Tris-HCl 0.5 M, pH 7.4, NaCl 1.5 M) con agitación. Después de lavar con agua destilada los geles se colocaron sobre un puente de papel Whatman (Whatman Int. Ltd., Maidstone, England), embebido con buffer SSC 10X y en contacto con el mismo. Sobre el gel de agarosa se ubicó una membrana de nylon (Hybond N+, Amersham Biosciences, UK) y sobre ella, tres hojas de papel de filtro Whatman, más una pila de papel secante y se dejó transfiriendo 16 h. Finalmente, la membrana de nylon conteniendo el ADN fue irradiada a 254 nm por 2 min. en horno UV Crosslinker. El fragmento de ADN escogido como sonda se obtuvo por PCR utilizando oligonucleótidos específicos para el gen de interés. Cada sonda de ADN (aprox 100 ng, cc. final 10 ng/μl) fue desnaturalizada a 100°C durante 5 min. y luego enfriada 5 min. en hielo. Se agregó 10 μl del reactivo de marcado (conteniendo la enzima peroxidasa) ECL Gold (Amersham Biosciences, UK) y 10 μl de solución de glutaraldehído. La mezcla fue incubada 10 min. a 37°C y usada inmediatamente para la hibridación. La membrana de nylon conteniendo los fragmentos de ADN fue prehibridada a 42°C durante 1 h. en buffer ECL Gold (Amersham Biosciences, UK), NaCl 0.5M, agente bloqueante 5% (P/V) en agitación. Luego se agregó la sonda marcada y se mantuvo en agitación durante 18 h. a la misma temperatura. Posteriormente la membrana se lavó dos veces con una solución SSC 0.5X, SDS 0.5% P/V a 55°C durante 20 min. A continuación se lavó la membrana dos veces con solución SSC 2X durante 5 min. a temperatura ambiente. La membrana fue sumergida en solución de detección (mezcla de sc. 1, productor de peróxido de hidrogeno y sc. 2, luminol), durante 1 min. Luego de escurrido el exceso de solución, la membrana de nylon fue expuesta a una película fotográfica durante diferentes períodos de tiempo. 57 Materiales y Métodos 3.16. Preparación y análisis de proteínas 3.16.1. Extracción de proteínas Los diferentes tejidos de A. thaliana se cortaron en pequeños trozos, se congelaron en N2 (l) y se homogeneizaron con un émbolo hasta lograr un polvo. Se agregó un medio de extracción conteniendo: Tris-HCl 20 mM, pH 6.8; EDTA 1 mM; PMSF 1 mM. Las fracciones solubles se obtuvieron por centrifugación a 10000 x g durante 30 min., a 4ºC. 3.16.2. Determinación del contenido proteico La concentración de proteína total se determinó por el método de Bradford (1976). Se utilizó BSA como testigo. 3.16.3. Electroforesis de proteínas Tanto los extractos de tejido vegetal como bacterianos o proteínas purificadas se sometieron a una electroforesis en condiciones desnaturalizantes (SDS-PAGE), utilizando un equipo Mini Protean II Cell (BioRad, Hercules CA, USA) y siguiendo la técnica descripta por Laemmli (1970). La concentración de acrilamida en el gel de separación fue de 12-15% P/V según la muestra a analizar y en el gel de concentración fue de 5% P/V. Las muestras se incubaron a 100ºC durante 2 min. en presencia del siguiente buffer de muestra: Tris-HCl pH 6.8 12 mM; 5% V/V glicerol; 0.4% P/V SDS; 2.88 mM 2-mercaptoetanol y 0.02 % P/V azul de bromofenol. La electroforesis se realizó a 120 V (voltaje constante). Las bandas de proteína se revelaron por tinción con azul brillante de Coomassie R-250, seguido de desteñido con una solución de etanol: ácido acético: agua (3:1:6). 58 Materiales y Métodos 3.17. Obtención de anticuerpos policlonales Los anticuerpos policlonales contra AtFH recombinante de A. thaliana fueron obtenidos mediante inmunización de conejo luego de la inyección de cuatro dosis de 100 μg de la proteína recombinante purificada (Harlow and Lane, 1988). 3.18. Análisis por western blot Los extractos proteicos o proteínas purificadas se sometieron primero a una electroforesis desnaturalizante (SDS-PAGE) y luego a una electrotransferencia utilizando la celda Bio-Rad Mini-Trans-Blot transfer siguiendo el protocolo desarrollado por Burnette (Burnette, 1981). Los geles se transfirieron a membranas de nitrocelulosa durante 16 h. a 4°C y a 30 V (voltaje constante) en presencia del siguiente buffer de transferencia: Tris-HCl 20 mM; glicina 150 mM; 20 % V/V metanol; pH 8.0. Las membranas se incubaron en una solución de bloqueo compuesta por 3 % P/V BSA en PBS (NaCl 137 mM; KCl 2.7 mM; Na2HPO4 4.3 mM; KH2PO4 1.4 mM) durante 1 h. Luego, se realizaron 2 lavados de 15 min. con H2O y se incubó las membranas 1 h. con el anticuerpo primario correspondiente. Los anticuerpos primarios que se utilizaron fueron (i) anticuerpos policlonales de conejo anti-AtFH de Arabidopsis thaliana (Maliandi et al., 2007); (ii) anticuerpos monoclonales anti-Histag (Qiagen Inc, Valencia, CA, USA); (iii) anticuerpos policlonales anti-catalasa (cedidos gentilmente por el Profesor Mikio Nishimura, Department of Cell Biology, National Institute for Basic biology, Okazaki, Japan); (iv) anticuerpos policlonales anti-NAD9 (cedidos por el Dr. Daniel Gonzalez, UNL). Luego de la incubación con el anticuerpo primario correspondiente, las membranas se lavaron 3 veces con una solución 2 % P/V BSA en PBS durante 15 min., y se incubaron 1 h. con anti-IgG conjugada con la enzima fosfatasa alcalina. (Para los anticuerpos primarios monoclonales, se utilizaron anticuerpos secundarios anti-ratón y para los anticuerpos primarios policlonales se utilizaron anticuerpos secundarios anti-conejo). Finalmente, las membranas se lavaron 2 veces con PBS durante 10 min. y fueron incubadas por 5 min. con buffer sustrato conteniendo: Tris-HCl 100 mM pH 9,5; NaCl 100 mM; MgCl2 5 mM. La presencia de las bandas proteicas se reveló 59 Materiales y Métodos mediante el agregado de 66 μl de azul de nitrotetrazolio (NBT, 50 mg/ml) y 33 μl de 5-bromo-4-cloro-3-indoilfosfato (BCIP, 50 mg/ml) en 10 ml de buffer sustrato (la reacción se desarrolló al resguardo de la luz hasta la aparición de bandas de color púrpura y se detuvo con lavados de H2O destilada). Para estimar la abundancia relativa de cada banda proteica, en algunos casos se realizó un análisis densitométrico utilizando el programa Gel PRO Analyzer Version 3.0 (Media Cybernetics, Inc. Bethesda, MD, USA). 3.19. Aislamiento de plantas mutantes homocigotas A fin de aislar plantas homocigotas de las mutantes insercionales atfh-1, atfh2 y atfh-3, se comprobó en primer lugar, la ubicación del inserto en el gen AtFH mediante una reacción de PCR sobre ADNg y utilizando oligonucleótidos específicos flanqueando el lugar de inserción del T-DNA (Fraten Fw/ Fra-Sma Rv y Fra 5 New Fw / FraSma Rv para las líneas atfh-1 y Fra 5 New Fw/ FraSma Rv y NPTII Fw y Rv para las líneas atfh-2 y atfh-3). El ADNg fue extraído a partir de hojas utilizando el método modificado de extracción con CTAB (Sambrook et al., 1989) (ver sección 3.13). 3.20. Construcción de vectores binarios para transformación de plantas de A. thaliana A partir de ADNc obtenido por retrotranscripción utilizando ARN total, se realizó un ensayo de PCR con oligonucleóticos secuencia de AtFH en orientación específicos para amplificar la sentido (AtFHBam up: AAAGGATCCATGGCTACAGCTTCAAGG el sitio BamHI esta subrayado; Frastop: subrayado) AAACTCGAGTTATGAGAGTTGGATTGGTTC, el sitio XhoI esta y AtFH en orientación AAAGGATCCATGGCTACAGCTTCAAGG, FraSma: antisentido (AtFH Bamup: el sitio BamHI esta subrayado; AAACCCGGGTGAGAGTTGGATTGGTTC, el sitio SmaI esta subrayado). El fragmento de interés fue aislado y purificado y se sometió a digestión (ver sección 3.7.2) con las endonucleasas BamHI y XhoI para la construcción s- 60 Materiales y Métodos AtFH. Para la construcción as-AtFH se utilizaron las endonucleasas SmaI y BamHI. El vector pDH51, se sometió a digestión con las endonucleasas BamHI y SalI para la construcción s-AtFH. El sitio de clonado múltiple dentro del vector no contiene el sitio XhoI, por consiguiente se digirió con SalI ya que ambas generan extremos cohesivos compatibles. Para la construcción as-AtFH el vector se digirió con las endonucleasas SmaI y BamHI. El fragmento de DNA resultante para cada construcción, fue clonado en el vector pDH51 dentro del sitio de clonado múltiple, entre el promotor y terminador del CaMV35S. Se transformaron células de E. coli y se analizaron los clones positivos, que contienen el casete con el fragmento AtFH en orientación sentido (s-AtFH) o el fragmento AtFH en orientación antisentido (asAtFH). Estos casetes fueron liberados con EcoRI y clonado en el vector binario pCAMBIA 1302 que previamente había sido digerido con EcoRI. 3.21. Preparación de células competentes de Agrobacterium tumefaciens y su transformación 3.21.1. Preparación de células competentes Una colonia de A. tumefaciens GV3101 fue crecida 16 h. en medio LB líquido suplementado con antibióticos (rifanpicina 100 μg/ml y gentamicina, 25 μg/ml) a 30°C. Luego, utilizando 2 ml de este cultivo se inoculó 200 ml de medio LB y antibióticos y las células se crecieron con agitación hasta alcanzar una OD600 = 0.3 – 0.4. Posteriormente el cultivo se centrifugó a 3000 × g durante 20 min. a 4°C. El precipitado se lavó con 10 ml de buffer TE frío y se resuspendió en 5 ml de LB y antibióticos. La suspensión de células competentes se fraccionó en alícuotas de 500 ȝl, se congeló en N2 (l) y se mantuvieron a –70°C hasta su utilización o hasta tres meses. 3.21.2. Transformación de las células de A. tumefaciens A una alícuota de 500 ȝl células competentes se le agregó 0.5-1.0 ȝg de ADN plasmídico. Se incubó sucesivamente 5 min. en hielo, 5 min. en N2 (l) y 5 min. a 37°C. La suspensión se diluyó a 1 ml con medio LB y antibióticos rif-gen-kam y se 61 Materiales y Métodos incubó con agitación a 28°C durante 2- 4 h. Posteriormente, se inocularon 200 ȝl del cultivo en medio LB-agar y antibióticos y se incubaron las placas a 28°C durante 48 h. La transformación fue verificada por PCR sobre ADN plasmídico obtenido mediante minipreparaciones a partir de diferentes colonias (ver seccion 3.21.3). 3.21.3. Obtención de ADN plasmídico de A. tumefaciens Células de A. tumefaciens fueron inoculadas en 5 ml de medio LB suplementados con los antibióticos correspondientes y se crecieron por 16 h. a 30°C. Los cultivos se centrifugaron a 1500 × g por 15 min. a 4°C. Se descartó el sobrenadante y se resuspendió el precipitado en 200 ȝl de Solución I (glucosa 50 mM, EDTA 10 mM, Tris-HCl 25 mM, pH 8.0 y lisozima 5 mg/ml de) agitando en un vortex y luego se incubó durante 10 min. a temperatura ambiente. Se agregó 400 μl de Solución II (SDS 1% V/V, NaOH 0.2 N) preparada en el momento y se mezcló por inversión cuatro veces. El homogenado se incubó 10 min. a temperatura ambiente. Posteriormente, se agregó 60 μl de fenol alcalino (2 volúmenes de NaOH 0.2 N más 1 volumen de fenol equilibrado en Tris-HCl 1M, pH 8.0 y mezclado en el instante previo al uso). Se mezcló por inversión, por lo menos 16 veces. Se agregó 300 μl de acetato de sodio 3 M, pH 5.0 y se mezcló por inversión 20 veces. Se incubó durante 20 min. a –20°C y luego se centrifugó 5 min. a temperatura ambiente a 1500 × g. El sobrenadante fue recolectado y se adicionó un volumen igual de fenol equilibrado con Tris-HCl, 1M, pH 8.0. Luego se procedió a la extracción del ADN por separación de fases tras agitación por inversión aproximadamente 20 veces. Después de centrifugar a 12000 × g por 5 min., se tomó la fase superior y se agregó un volumen igual de cloroformo. Se agitó y luego se centrifugó a 12000 × g por 5 min. Luego de decantar, se separó la fase superior, se agregó 0.7 volúmenes de isopropanol para precipitar el ADN y se incubó a –20°C por 20 min. Después de centrifugar 10 min. a 12000 × g se descartó el sobrenadante y se lavó el precipitado con 500 μl de etanol 70% V/V. Finalmente se centrifugó 5 min. a 12000 × g, se descartó el sobrenadante y se dejó secar. El precipitado fue resuspendido en un volumen adecuado de agua destilada (aprox 40 μl). 62 Materiales y Métodos 3.22. Transformación de plantas de A. thaliana utilizando el método de infiltración por floral dip La transformación de plantas de Arabidopsis se realizó mediante la técnica de floral dip (Clough and Bent, 1998). Brevemente, se cultivaron plantas de Arabidopsis wt (var. Col-0) a 23ºC hasta alcanzar el estadio de floración (aprox 6 semanas). Para tener un buen número de brotes florales, las inflorescencias primarias fueron cortadas aproximadamente 4 días antes de la transformación para eliminar la dominancia apical y posibilitar la emergencia de múltiples botones florales secundarios. Dos días antes de la transformación se inició un cultivo de 3 ml de células de A. tumefaciens GV3101, transformadas con el plásmido binario con la construcción correspondiente. El cultivo se realizó a 28ºC en medio LB (gen-rif) y el antibiótico que permita la selección del plásmido de interés. Con estos pre-inóculos se inocularon 250 ml de medio LB suplementado con los antibióticos antes mencionados. Se crecieron en agitación a 30ºC por 24 h. Al día siguiente, las bacterias se cosecharon por centrifugación a 7000 × g por 10 min. y se resuspendieron en 250 ml de medio de infiltración (sacarosa 5% P/V, detergente SILWET® L-77-Ag 0.005% V/V) hasta lograr una OD600 = 0.8. El medio de infiltración se colocó en vasos de precipitado, y se sumergieron los brotes florales durante 15 s. Las macetas se colocaron en forma horizontal en bandejas de plástico y cubiertas con plástico transparente, de forma tal de mantener la humedad y posibilitar el paso de luz. Luego de dos días, las plantas se colocaron de forma vertical y se retiró la cubierta plástica. Aproximadamente luego de 3 a 5 semanas se cosecharon las semillas que fueron mantenidas a 4ºC hasta ser utilizadas. 3.23. Esterilización de semillas de Arabidopsis Las semillas de Arabidopsis fueron esterilizadas en Solución de Bleach (NaClO 5% V/V, SDS 1% P/V) mediante agitación en vortex durante 8 min. y luego se realizaron 3 lavados sucesivos con H2O destilada estéril. Las semillas fueron resuspendidas en TOP-agar (0.2 % P/V agar) y luego sembradas en placas con medio MS y cuando corresponda, con el antibiótico de selección. 63 Materiales y Métodos 3.24. Selección de plantas transformadas Las semillas esterilizadas se inocularon en placas de Petri conteniendo medio MS, agar 0.8% P/V, sacarosa 1% P/V y el/los antibióticos correspondientes, y se incubaron durante 48 h. a 4ºC en oscuridad. A los dos días, las placas se pasaron a invernadero y se crecieron a 23°C con iluminación. Las plantas transgénicas serán aquellas que crezcan en presencia de los antibióticos (resistentes). Luego de la aparición de las primeras hojas verdaderas, las plantas fueron pasadas a tierra (rusticación). 3.25. Clonado, expresión y purificación de proteínas 3.25.1. Clonado molecular de AtFH de A. thaliana El ADNc correspondiente al gen AtFH (564 pb) fusionado a histidina y con codón de terminación, fueron obtenidos por RT-PCR usando los oligonucleótidos: fra2x y Fra-his; fra2x y Fra-stop, respectivamente. Los productos de PCR resultantes fueron digeridos usando endonucleasas de restricción (ver sección 3.7.2) y clonados en el vector pET24a (Novagen, EMD Biosciences Inc, Madison, WI, USA). Los clones positivos fueron verificados por secuenciación de ADN. Así, se generaron los plásmidos recombinantes pATF01 (que codifica para AtFH-His6, conteniendo en el extremo C-terminal la fusion a histidina y pATF02 que codifica para AtFH-STOP, conteniendo el codón de terminación. 3.25.2. Expresión y purificación de las proteínas recombinantes AtFH-His6 y AtFH- STOP de A. thaliana Los plásmidos pATF01 y pATF02, se utilizaron para transformar células competentes de E.coli BL21 (DE3) y BL21 (DE3)-RIL (ver sección 3.6.1.2). Se inoculó medio LB suplementado con kanamicina (30 mg/l), con las células transformadas. Se crecieron a 37ºC en agitación (160 rpm) hasta obtener una DO600= 0.4-0.6. La expresión de la proteína recombinante se indujo con el agregado 1 mM de IPTG por aproximadamente 4-5 h. a 30 ºC. El tiempo óptimo de expresión fue 64 Materiales y Métodos seleccionado luego del análisis por SDS-PAGE de muestras tomadas a diferentes tiempos luego de la inducción con IPTG. Para realizar la purificación, los cultivos conteniendo las células transformadas fueron colectados por centrifugación a 3000 × g por 20 min. a 4ºC, se lavaron dos veces y se resuspendieron con 2 ml de buffer A (Na2HPO4 50 mM, NaCl 300 mM, imidazol 10 mM) por cada gramo de células. Luego los cultivos se sonicaron (130 Watt Ultrasonic Processor, Vibra Cell, Sonics & Materials UK) y centrifugaron a 12000 × g por 10 min. a 4°C. El extracto crudo se filtró a través de una membrana de acetato de celulosa de 0.2 μm. Para la purificación se utilizaron columnas comerciales de afinidad de 1 ml cargada con Ni+2 como ligando (Hi Trap chelating HP, Amersham Biosciences, UK) y una bomba peristáltica (Biologic LP, Bio-Rad, Hercules, CA, USA). En primer lugar, se lavó la columna con 10 ml de agua destilada y 10 ml de buffer A (flujo 1 ml/min). Posteriormente se cargó la muestra en la columna y se equilibró con 10 ml de buffer de unión, B (NaH2PO4 0.02 M, NaCl 0.5 M, imidazol 20 mM, pH 7.4). La elución de la proteína se realizó en un volumen de 25 ml utilizando un gradiente de imidazol 20 mM inicial (buffer de unión) - 500 mM final en buffer de unión. Las fracciones que contenían AtFH eluyeron a una concentración de imidazol de alrededor de 250 mM. Estas fracciones se recolectaron, se concentraron hasta 1 mg/ml y se guardaron a 20ºC. En estas condiciones la proteína fue estable por lo menos por 2 meses. Para la expresión y purificación de AtFH-STOP, células de E.coli BL21 (DE3)-RIL conteniendo el plásmido pATF02 fueron crecidas en medio LB a 37ºC hasta llegar a una DO600 = 0.5. La expresión de la proteína se indujo con 1 mM de IPTG. Las células se cultivaron con agitación por 4 h a 30 ºC, se centrifugaron y luego se resuspendieron en buffer C (Tris-HCl 50 mM, pH 7.5, EDTA 1 mM). Posteriormente las células se sonicaron (130 Watt Ultrasonic Processor, Vibra Cell, Sonics & Materials UK). La fracción soluble del extracto celular se cargó en una columna comercial de DEAE-Sepharosa fast flow (Amersham Biosciences, UK) equilibrada con buffer C. Luego se lavo con 5ml del mismo buffer. La proteína se eluyó usando un gradiente de buffer D (Tris-HCl 50 mM, pH 7.5, EDTA 1 mM, y KCl 600 mM). La fracción que contenía AtFH-STOP, se recolecto y concentró y luego diluyó 10 veces con buffer C. La nueva solución se sembró en una columna 65 Materiales y Métodos Mono–Q (Amersham Biosciences, UK) y se eluyó con un gradiente lineal de KCl (0450 mM) en buffer E (Tris-HCl 50 mM, pH 8.0 y EDTA 1 mM). La fracción conteniendo la proteína se recolectó y concentró. Luego se realizó una cromatografía en una columna de filtración molecular (Superosa 12) usando buffer E con KCl 150 mM. La proteína purificada fue concentrada y almacenada a – 20 ºC. 3.26. Ensayo de degradación oxidativa de 2-deoxiribosa Una mezcla de Fe (II) 15 μM, H2O2 8 μM, 2- deoxyribosa 5 mM (Fluka, St. Louis, USA) en buffer Hepes-KOH 10 mM, pH 7.0, fue incubada en ausencia o en presencia de AtFH-His6 y AtFH-STOP recombinantes por 30 min. a 25 ºC (vol. final 100 ȝl). Posteriormente, se agregó 50 μl de ácido fosfórico 4 % V/V y 50 μl de ácido tiobarbitúrico 1% P/V. La mezcla de reacción fue incubada por 10 min. a 100 ºC y posteriormente enfriada en hielo. Después de este procedimiento se agrego 75 μl de SDS 10% V/V. La cantidad de malondialdehído producida fue determinada espectrofotométricamente a 532 nm. 3.27. Determinación de Fe Se partió de 60-100 mg de hojas de Arabidopsis de plantas wt y atfh-1 de 6 semanas. Se homogeneizó el material con 500 μl de acetato de sodio 0.1 M y luego se centrifugó a 13400 rpm por 5 min. Se utilizó 250 μl de sobrenadante y al mismo se le agregó 100 μl hidroxilamina 10% P/V, 500 μl de acetato de sodio 0.1 M, 1.4 ml H2O y 250 μl fenantrolina 2 mM. Se dejo incubando la mezcla de reacción a temperatura ambiente por 10 min. hasta desarrollo de color y se cuantificó la absorbancia a 510 nm. La curva de calibración se realizó utilizando soluciones de distintas concentraciones de Fe (NH4)2(SO4)2 (2.10-6 M, 3.6.10-6 M, 7.2.10-6M, 1.44.10-5M, 2.88.10-5M, 3.6.10-5M, 7.2.10-5M, 1.44.10-4M, 2.88.10-4M) (Greenbers, et al., 1992). 66 Materiales y Métodos 3.28. Determinación de la concentración de ATP Se cuantificó el contenido total de ATP en hojas de roseta y flores de plantas wt, mutantes y transgénicas de 6 semanas (Teixeira et al., 2005). Las mismas fueron congeladas en N2 (l) y trituradas hasta lograr un polvo. Se utilizó aproximadamente 20 mg de tejido para cada determinación y cada muestra se homogeneizó con 60 μl de HCl 0.1 M en microtubos de 1.5 ml. El homogenado fue centrifugado a 20000 × g por 10 min. a 4°C. El sobrenadante se cargó en un micro-concentrador Microcon YM-3 (Millipore Corp, Bedford, MA, USA) a 14000 × g a 4°C a fin de concentrar y desalar la muestra, ya que altas concentraciones de sales en la muestra pueden causar un efecto inhibitorio en la actividad luciferasa y disminuir la sensibilidad de la técnica. Luego de neutralizar con buffer Tris-HCl pH 7.4, la solución filtrada fue usada para la medida del contenido de ATP por luminiscencia mediante la utilización del ATP bioluminescent assay kit (Sigma-Aldrich, St. Louis, MO, USA). El fundamento de la reacción se basa en la medida de la emisión de luz cuando el ATP es consumido al reaccionar con la D-luciferina, a través de una reacción catalizada por la enzima luciferasa. La cantidad de luz fue cuantificada utilizando un luminómetro (LD-400 Luminescence Detector-Beckman Coulter, Inc.) y es proporcional al ATP presente en la muestra. Los niveles de ATP se expresaron en nmol .g–1 PF. Todos los datos fueron sometidos a análisis de t-Student, con un nivel de probabilidad P <0.05. 3.29. Medidas de respiración El consumo de oxígeno fue medido a 25°C usando una cámara de aire cerrada unida a un electrodo tipo Clark (Hansatech Leaf Disc Electrode Unit, Hansatech, UK). Se cortaron hojas de plantas de aproximadamente 6 semanas de A. thaliana (entre 200 a 300 mg) y éstas fueron colocadas en la cámara en presencia del electrodo de oxígeno. La calibración se realizó mediante 2 puntos: para el límite superior se utilizó el pasaje de aire ambiental (21% O2) y el límite inferior se logró con el pasaje de N2 (g) (0% O2) a través de la cámara. El consumo de oxígeno fue monitoreado durante 15 min. 67 Materiales y Métodos 3. 29.1. Tratamiento con Rotenona Se cortaron entre 100-200 mg de hojas de plantas wt, atfh-1 y as-AtFH, y se sumergieron en 40 μM de rotenona. Se realizaron dos tiempos de incubación, 2 h y 15 h, y luego se midió el consumo de O2 como se detalla en la sección 3.29. La solución de trabajo de 40 μM de rotenona, se preparó a partir de una solución madre de 1 mM de rotenona, disuelta en agua, 0.5% metanol y 0.5 % etilenglicol para favorecer su solubilización. 3.30. Aislamiento de mitocondrias y determinaciones enzimáticas 3.30.1. Aislamiento de mitocondrias Las mitocondrias fueron aisladas como se describió previamente (Werhahn et al., 2001) con modificaciones (Kruft, 2001). Las muestras de tejido vegetal (aprox. 5 g. de hojas) fueron trituradas en mortero en buffer de extracción conteniendo: sacarosa 450 mM, EGTA 1.5 mM, BSA 0.2 % P/V, PVP (polivinilpolipirrolidona) 0.6 % P/V, DTT (ditiotreitol) 10 mM y MOPS (ácido 3-N-morpholinopropanesulfonico) 15 mM, pH 7.4. Luego se realizó una centrifugación diferencial consistente en 5 pasos: 4 pasos para purificar la mitocondria de las partículas con alto coeficiente de sedimentación (10 min. a 3500 x g, 2 veces, 5 min. a 3500 x g y 5 min. a 6000 x g) y un paso final para colectar la fracción enriquecida de mitocondrias luego de la centrifugación por 10 min. a 17000 x g. Posteriormente se realizaron 3 pasos de centrifugación en gradiente de Percoll, 18%, 29% y 45% V/V de Percoll en 0.3 M sacarosa y 10 mM MOPS, pH 7.2. Luego de la centrifugación por 45 min. a 70000 x g, la fracción enriquecida en mitocondria se aisló de la interfase entre 29% y 45% V/V de Percoll. Bajo estas condiciones la fracción de mitocondrias está deprivada esencialmente de contaminación citoplasmática o plastídica. Las mitocondrias aisladas fueron homogeinizadas en buffer conteniendo 300 mM manitol y 10 mM K2HPO4 (pH 7.4) y 0.5 % V/V Tritón X-100 a fin de solubilizar las organelas para determinación de actividades enzimáticas. 68 Materiales y Métodos 3.30.2 Medida de la actividad enzimática de aconitasa, succinato deshidrogenasa y malato deshidrogenasa La actividad aconitasa fue determinada mediante el aumento de la absorbancia a 240 nm. El medio de reacción contenía: D-Isocitrato 20 mM y TrisHCl 100 mM, pH 8 y mitocondrias solubilizadas (1 μg/μl de proteína total), en un volumen final de 500 μl. Las determinaciones se llevaron a cabo a 25°C durante 20 min., a intervalos de 30 s. (Li, et al., 1999). La actividad succinato deshidrogenasa se determinó mediante el descenso de la absorbancia a 600 nm. El medio de reacción fue el siguiente: succinato 100 mM, diclorofenol indofenol (DCPIP) 0.5 mM, azida 40 mM, buffer fosfato 10 mM, CaCl2 10 mM y BSA 0.5 mg/ml y mitocondria solubilizadas (1μg/μl de proteína total), en un volumen final de 500 μl. Las determinaciones se llevaron a cabo a 25°C durante 20 min., a intervalos de 30s. (Schirawski and Unden, 1998). La actividad malato deshidrogenasa fue cuantificada por descenso de la absorbancia a 340 nm. El medio de reacción contenía: imidazol 50 mM pH 6.9, NADH 0.15 mM y ácido oxálico 1 mM y una suspensión de mitocondrias (1μg/μl de proteína total), en un volumen final de 500 μl. Las determinaciones se llevaron a cabo a 25°C durante 20 min., a intervalos de 30 s. (Tripodi and Podesta, 2003). 3.31 Infiltración de hojas con H2O2 Aproximadamente 20 hojas de roseta de A. thaliana wt fueron infiltradas con una solución 500 ȝM de H2O2, con la ayuda de una jeringa de 1 ml (sin aguja). Se extrajo ARN a diferentes tiempos de incubación (t0, 2, 4, 8 y 24 h.), según el protocolo descripto en la sección 3.9. Como control, se usaron plantas infiltradas con agua destilada, procesadas de forma similar. 69 Materiales y Métodos 3.32. Detección histoquímica de superóxido en hojas de Arabidopsis La detección histoquímica de superóxido, se realizó en hojas de roseta de plantas de A. thaliana de 6 semanas, las cuales fueron infiltradas por vacío con una solución 6 mM NBT (Nitroblue Tetrazolium, Promega, WI, USA), 1 mM azida de sodio (Na3N) (Flohé and Otting, 1984; Beyer and Fridovich, 1987; Laloi et al., 2004). El NBT de color amarillo pálido reacciona con anión superóxido, produciendo formazán, el cual forma un precipitado de color azul oscuro aproximadamente a los 20 min. de iniciada la reacción. La clorofila fue removida de las hojas mediante calentamiento por 10 min. en presencia de etanol 95% V/V (Laloi et al., 2004). 3.33. Detección histoquímica de especies reactivas de oxígeno en hojas y raíces de plantas de Arabidopsis thaliana La detección histoquímica de ROS fue realizada de acuerdo a Hempel y col. (Hempel et al., 1999). Brevemente, hojas de roseta y raíces de plantas de Arabidopsis wt, mutantes y transgénicas de 6 semanas, fueron sumergidas en solución buffer fosfato salino (PBS) 1X conteniendo 5 μM H2DCFDA (dihidro-cloro fluoresceína diacetato). El tejido fue incubado por 2 min. a temperatura ambiente en oscuridad, y luego lavado 2 veces por 3 min. en PBS 1X. La fluorescencia fue visualizada inmediatamente utilizando un microscopio de fluorescencia Nikon Eclipse E800. 3.34. Determinación del contenido de hemo y actividad catalasa 3.34.1. Determinación de la concentración de hemina: Se partió de aproximadamente 1 g. de hojas de roseta de plantas de A. thaliana wt, atfh-1 y as-AtFH. El tejido se homogenizó utilizando un mortero. Posteriormente se agregó 2 ml de Na2HPO4 50 mM, pH 7.4 y se centrífugo a 11000 x g, a 4ºC por 10 min. Al sobrenadante se le agregó 1 ml de glicina-HCl 50 mM, pH 2; 0.2 ml de HCl 4N; 0.2 ml de NaCl 5M y 1 ml de cloroformo acidificado (pH 1-2). Se midió la concentración de hemina extraída en fase clorofórmica a través de un método espectrofotométrico. Para ello, se determinó la absorbancia a tres longitudes 70 Materiales y Métodos de onda, 388, 450 y 330 nm, y se obtuvo la absorbancia corregida (Lombardo et al., 2005). Se utilizó una solución de hemina (Sigma , St. Lousi, USA) como Standard. 3.34.2. Medida de la actividad catalasa en hojas, flores y mitocondrias aisladas y recuperación de la actividad catalasa por el agregado de hemina exógena. Se partió de 170 mg de hojas o aproximadamente 50 mg de flores de A. thaliana wt, plantas mutantes (atfh-1) y plantas transgénicas (as-AtFH) de 6 semanas, o una fracción de mitocondrias aisladas como se describió en la sección 3.30.1. El material vegetal fue triturado en N2 líquido para facilitar la ruptura del mismo, posteriormente se homogenizó en relación 1:10 P/V con el buffer de extracción (KH2PO4 50 mM, pH 7.8, Tritón X-100 0.5 % V/V, EDTA 0.5 mM) y se le adicionó PMSF 1 mM como inhibidor de proteasas. Luego de centrifugar el sobrenadante se utilizó para la determinación de la actividad catalasa, como se describió previamente (Aebi, H, 1984). Se tomó 5 ȝl de muestra del sobrenadante obtenido en la etapa anterior y se le adicionó 475 ȝl de buffer de determinación (KH2PO4 50mM, pH 7), y 20 ȝl H2O2 10 mM-50 mM (Coeficiente de extinción 38 M-1.cm-1, debe estar a una absorbancia de 0.38 a 1.9). Cada uno de los tratamientos se realizó por triplicado. La actividad catalasa se determinó como la disminución de absorbancia a 240 nm debido al consumo de H2O2 a 25ºC. La recuperación de la actividad catalasa se determinó luego de la incubación de una suspensión de mitocondrias aisladas en presencia de hemina 50 μM por 1h con agitación, a 25°C. El medio de reacción contenía 250 mM manitol, 50 mM KCl, 2 mM MgCl2, 20 mM HEPES pH 7.4, 1 mM K2HPO4, 1 mM DTT, 10 mM ATP, 20 μM ADP, 10 mM succinato de sodio, 50 μM hemina y 10 μl de suspensión de mitocondrias (aprox 10 mg/ml de proteína total). Luego de la incubación, las muestras se centrifugaron 20 min. a 3000 x g a 4ºC, se solubilizaron las mitocondrias en buffer 20 mM K2HPO4 pH 7 y agitación con vortex y se determinó la actividad catalasa en el sobrenadante. 71 Materiales y Métodos 3.35. Métodos bioinformáticos Los niveles de ARNm obtenidos a partir de los experimentos de RT-PCR se cuantificaron utilizando el programa Gel Pro Analyzer 3.1 (Media Cybernetics, Silver Spring, MD, USA). Las secuencias de nucleótidos y proteínas fueron analizadas usando los programas BLAST (Altschul y col., 1990; Altschul y col., 1997). 72 Resultados y discusión Capítulo I Resultados y discusión Capítulo I La mayor parte de la investigación sobre frataxina ha sido llevada a cabo en levaduras, bacterias y mamíferos (Babcock et al., 1997; Ristow et al., 2000). Esta proteína fue inicialmente descripta en humanos, en pacientes portadores de una enfermedad autosómica recesiva denominada Ataxia de Friedrich (Campuzano et al., 1996; Schapira, 1998; Becker et al, 2001). Como mencionamos anteriormente, la frataxina es una proteína mitocondrial codificada en el núcleo. El gen codificante ha sido conservado en bacterias y animales sin haber sufrido grandes cambios estructurales, hecho que sugiere una función similar en todos los organismos (Huynen et al., 2001). Se ha propuesto su participación en el ensamblado de centros Fe-S (Huynen et al., 2001), en la homeostasis mitocondrial del hierro, metabolismo del hemo, fosforilación oxidativa (Ristow et al., 2000) y estrés oxidativo (Park et al., 2002). La deficiencia de la misma provoca un inadecuado influjo de hierro hacia la mitocondria, pérdida de síntesis de ATP y una deficiencia en la defensa antioxidante, ocasionando una acumulación de hierro mitocondrial y como consecuencia mayor daño oxidativo (Lill and Kispal, 2000; Park et al., 2002). Sin embargo, a pesar de tener asignadas múltiples funciones en la mitocondria, su función específica queda aún por elucidarse. Aunque su función parece ser indispensable en varios procesos celulares, hasta hace siete años, no existía información sobre esta proteína en organismos fotosintéticos, a excepción de la anotación de una secuencia relacionada en el banco de datos del proyecto genoma de Arabidopsis thaliana. Fue en nuestro laboratorio donde se identificó por primera vez el gen de frataxina de un organismo vegetal (AtFH, homólogo de frataxina de A. thaliana) y su producto proteico (AtFH). La expresión heteróloga de AtFH en una cepa de Saccharomyces cerevisiae, mutante nula para la expresión de frataxina, permitió comprobar su funcionalidad a través de la complementación del fenotipo (Busi et al., 2004). 74 Resultados y discusión 4.1. Caracterización de plantas de Arabidopsis thaliana deficientes en la expresión de AtFH 4.1.1. Estructura del gen AtFH de Arabidopsis thaliana La función de un gen puede estudiarse a partir de ensayos bioquímicos, celulares o de desarrollo. Para confirmar la presencia y función de un gen, debe recurrirse a la confirmación experimental, por ejemplo realizando supresión o sobreexpresión del mismo. Esto nos permite asociar la secuencia génica a un fenotipo a partir del cual puede deducirse el papel de dicho gen en el metabolismo o en el desarrollo de un organismo (Bouchez et al., 1998). El genoma de A. thaliana contiene un único locus (At4g03240) para el homólogo de frataxina (Busi et al., 2004) y el mismo se encuentra en el cromosoma 4. Estructuralmente está compuesto por cinco exones y cuatro intrones, originando un ARNm de 564 pb, y que codifica una proteína de 187 aminoácidos de masa molecular estimada de 14 kDa (Figura 12). El análisis bioinformático realizado con los programas PSORT (http://psort.ims.u-tokyo.ac.jp/) y MITOPROT II (http://ihg.gsf.de/ihg/mitoprot.html) sobre la secuencia aminoacídica deducida de AtFH, permitió identificar una corta secuencia de 31 aminoácidos que sugiere una localización mitocondrial (Busi et al., 2004). Estos resultados están de acuerdo con la localización descripta para la frataxina humana y de levaduras. Primer Fraten 5´UTR Primer Fra-Sma Primer Fra 5 New intrón exón Figura 12: Estructura del gen que codifica el homólogo de frataxina de Arabidopsis thaliana (AtFH). AtFH está compuesto por 5 exones y 4 intrones. Las flechas corresponden a los sitios de unión de los oligonucleótidos utilizados en los análisis moleculares. 5’UTR corresponde a la región 5’ no traducida. 75 Resultados y discusión 4.1.2. Selección de plantas mutantes homocigotas de A. thaliana con inserción de T-ADN en el gen AtFH Solamente un pequeño porcentaje de proteínas codificadas en los genomas de plantas y animales está suficientemente caracterizado respecto a su función celular. El papel de muchas de estas proteínas permanece completamente desconocido (aproximadamente 40%) o parcialmente comprendido (Gollery et al 2006, 2007; Horan et al 2008). A partir del conocimiento de la secuencia de ADN de diversos organismos vegetales se generaron diferentes bases de datos que contienen información de un gran número de genes. Sin embargo, en Arabidopsis thaliana todavía existe un grupo de genes a los cuales no se les ha identificado su función (Goodman et al., 1995; Cooke et al., 1996 ). Las comparaciones de secuencia en busca de homología pueden utilizarse para sugerir funciones similares en varios de los genes referidos, sin embargo, para poder asociar un gen a un determinado fenotipo y a partir de ahí poder dilucidar su función en el metabolismo o en el desarrollo de un organismo, se debe realizar la supresión o sobreexpresión de su secuencia codificante (Martienssen, 1998). El silenciamiento de genes vía la supresión sentido y antisentido ha sido un método bastante empleado durante los últimos 20 años (Baulcombe, 2007). Sin embargo, este método requiere que se generen para cada gen, varias líneas transgénicas independientes, debido a que la supresión de genes esenciales podría llevar a un fenotipo letal. En plantas, fue necesario desarrollar una técnica en la cual se pudiese insertar una secuencia conocida dentro de su genoma para ocasionar la mutación (Figura 13). La mutagénesis utilizando la inserción de trasposones y de T-ADN (porción del plasmido Ti inductor de tumores), aporta varias ventajas como es el uso de genes reporteros o genes marcadores de selección presentes en el plásmido que permiten rápidamente la selección de los individuos mutantes (Fedoroff and Smith, 1993; 76 Resultados y discusión Wilson et al., 1996). Esta técnica fue utilizada inicialmente para levaduras pero actualmente se utiliza también en plantas y organismos animales (Hayes, 2003). La técnica de mutagénesis insercional permitió obtener plantas de Arabidopsis con genes knockout para más del 70% de su genoma (Alonso et al., 2003). Una de las desventajas de esta técnica es que aún no se conoce el mecanismo preciso de integración del T-ADN dentro del genoma de plantas (Iida and Terada, 2004). Elemento insertado Gen de interés ATG STOP Figura 13: Mutagénesis por inserción. La inserción de un elemento móvil de ADN dentro de un gen interrumpe la actividad del mismo a nivel transcripcional, a nivel de la traducción o ambos. Los insertos pueden ser directamente elegidos para usar como marcadores de selección (por ejemplo, confiriendo resistencia a kanamicina o Basta), por la secuencia de inserción (transposones o T-ADN). La presencia de un gen o enhancer con un casete reportero permite que la expresión del gen en el sitio de inserción pueda ser monitoreada. A fin de identificar las posibles funciones del gen de frataxina en plantas superiores, se obtuvieron del “Arabidopsis Biological Resourse Center” (ABRC) tres líneas de plantas mutantes insercionales en el gen de AtFH: SALK_ 021263, SALK_094203 y SALK_ 122008. Estas líneas se denominaron como atfh-1, atfh-2 y atfh-3, respectivamente. La línea atfh-1 tiene la inserción del T-DNA en la región no traducida (5´UTR) por delante del codón ATG de iniciación de la traducción (Figura 14A), de acuerdo con el sitio de inserción predicho por secuenciación (http://www.arabidopsis.org). Las otras dos líneas mutantes, atfh-2 y atfh-3 tienen la inserción en el primer intrón y cuarto exón, respectivamente (Figura 14B y C). 77 Resultados y discusión (A) intrón T-DNA exón 5´UTR (B) 5´UTR T-DNA intrón exón (C) 5´UTR intrón exón T-DNA Figura 14: Esquema de la estructura del gen AtFH de plantas atfh-1 (SALK_021263) (A); líneas atfh-2 (SALK_094203) (B) y plantas atfh-3 (SALK_122008) (C). En cada una de las líneas se muestra en gris claro la inserción del T-ADN: región 5’UTR (en las líneas atfh-1); 1er. intrón (líneas atfh-2) y 4to. exón (líneas atfh-3). La ubicación del inserto en el gen AtFH en las diferentes líneas fue comprobada por análisis de PCR con oligonucleótidos específicos para los sitios flanqueantes al lugar de inserción del T-ADN (primer 5’ Fraten, Fra 5 New y 3’ FraSma) (Figura 13 y 15). Las plantas mutantes homocigotas atfh-1 fueron identificadas fácilmente (Figura 15A). Utilizando los primers Fraten y FraSma se obtuvo la amplificación de una banda de ADN de aproximadamente 1740 pb en las plantas wt, mientras que en 78 Resultados y discusión las plantas atfh-1 no se obtuvo ninguna amplificación. La presencia de una banda de ADN de 1086 pb luego de la amplificación utilizando los primers Fra5New y FraSma en las plantas wt como en la línea atfh-1 nos permitió corroborar que la inserción del T-ADN se encuentra en la región 5’UTR del gen AtFH (Figura 15A). En contraste con estos resultados, no pudimos obtener plantas homocigotas para las líneas atfh-2 y atfh-3 (Figura 15B). Para comprobar si estas dos líneas efectivamente contenían la inserción de T-ADN en su genoma, se realizó un análisis por PCR utilizando primers específicos para amplificar el gen de NPTII (de resistencia a Kanamicina) perteneciente a la secuencia del T-ADN. Los resultados en ambas líneas mostraron la amplificación de un producto de PCR del tamaño esperado (710 pb), confirmando que todas las plantas analizadas eran mutantes insercionales de T-ADN (Figura 15C). La presencia del producto de amplificación del gen AtFH y de NPTII, sugiere que solo uno de los alelos del gen AtFH se encuentra interrumpido por el T-ADN en el genoma de las líneas analizadas. Los resultados obtenidos indican que las plantas atfh-2 y atfh-3 son heterocigotas para el gen de AtFH. 79 Resultados y discusión (A) Fraten Fra Sma Fra5 New Fra Sma 1086bp 1740bp wt atfh-1 wt (B) atfh-1 (C) Fra5 New Fra Sma NPTII up NPTII down 710 pb 1086 pb 1 atfh-2 2 atfh-2 3 atfh-2 1 atfh-2 wt 2 atfh-2 3 atfh-2 wt 710 pb 1086 pb 1 atfh-3 2 atfh-3 3 atfh-3 wt 1 atfh-3 2 atfh-3 3 atfh-3 wt Figura 15: (A) Análisis de los productos de amplificación por PCR sobre el ADN de la línea mutante atfh-1, utilizando los primers Fraten/FraSma (panel izquierdo) y Fra5New/FraSma (Panel derecho). (B) Análisis por PCR de los productos de amplificación sobre ADN de las líneas mutantes atfh-2 y atfh-3 (plantas 1-3), usando los primers Fra5New/FraSma y (C) utilizando los primers NPTII up/down. El tamaño del ADN amplificado está indicado en cada figura. 4.1.3. Construcción de líneas transgénicas de Arabidopsis thaliana que expresan AtFH en orientación antisentido y sentido Con el objeto de obtener otra línea de plantas con expresión disminuida de AtFH construimos una línea transgénica que expresa el transcripto en orientación antisentido (denominada as-AtFH). Además, a fin de sobreexpresar el gen de AtFH generamos otra línea con el gen en orientación sentido (denominada s-AtFH). A partir del ADNc de AtFH, se realizaron las reacciones de PCR con cebadores específicos conteniendo sitios para las enzimas de restricción BamHI y XhoI para la construcción sentido, y SmaI y BamHI para la antisentido (ambos fragmentos de 564bp). Estos fragmentos fueron clonados en el vector pDH51 (Figura 80 Resultados y discusión 16A) dentro de un casete que contiene al promotor y terminador CaMV35S, generando así los vectores pDH51-as-AtFH y pDH51-s-AtFH (Figura 16 B y C, respectivamente). Los casetes de expresión CaMV35S-as-AtFH y CaMV35S-s-AtFH obtenidos de esta última construcción fueron subclonados en el vector pCAMBIA 1302 entre los sitios EcoRI, obteniéndose de esta manera los vectores pCAMBIA-as-AtFH y pCAMBIA-s-AtFH (Figura 16 D). Ambos vectores se utilizaron para la transformación de plantas wt mediante la técnica de floral dip (ver Materiales y Métodos), utilizando Agrobacterium tumefaciens (Clough et al., 1998). (A) KpnI SmaI BamHI XbaI lI Sal I Pst I Sph I (B) as- AtFH Sma I Sma I Bam HI BamH I (C) s- AtFH BamH I BamH I Xho Sal I 81 Resultados y discusión (D) SMC NPTII CaMV35S pCAMBIA 1302 Eco RI Eco RI CaMV35S as-AtFH Eco RI Eco RI CaMV35S Casete CaV35S-as-AtFH t- CaMV35S s-AtFH t- CaMV35S Casete CaV35S-s-AtFH Figura 16: (A) Esquema de la región del plásmido pDH51 conteniendo el promotor CaMV35S, el sitio de múltiple clonado y el sitio terminador t-CAMV35S. (B) y (C) Esquema del vector plasmídico pDH51 conteniendo la secuencia correspondiente a as-AtFH y s-AtFH, respectivamente. Plac : promotor lac de pUC18. Cajas “CAT” y “TATA” en la región promotora de la unidad de expresión están marcadas por la secuencia original CAAT y TATATAA y la secuencia señal de poliadenilación por AATAAA. Las flechas marcadas (ARN) muestran el sitio de inicio de la transcripción. SMC: sitio de múltiple clonado derivado de pUC18. (D) Esquema del vector pCAMBIA-as-AtFH y pCAMBIA-sAtFH. NPTII gen de resistencia a kanamicina. Las plantas transgénicas de Arabidopsis que contenían la construcción asAtFH o s-AtFH se seleccionaron por crecimiento en medio solido MS- higromicina (antibiótico para la selección de plantas). De esta forma, se pudo obtener otra línea de plantas independiente (as-AtFH) que junto a la línea homocigota atfh-1 nos permitirán realizar experimentos de pérdida de función. La línea s-AtFH, será necesaria como prueba positiva de las funciones del gen y complementa los experimentos de pérdida de función. 4.1.4. Efecto de la deficiencia de AtFH en plantas de Arabidopsis 4.1.4.1. Análisis fenotípico de plantas de Arabidopsis deficientes en la expresión de AtFH Nuestro conocimiento de la biología básica de las plantas y la habilidad para manipularlas genéticamente, dependen de la investigación sobre el desarrollo, la genética y los procesos metabólicos fundamentales de estos organismos. Uno de los mayores desafíos en la biología molecular de plantas es el aislamiento e identificación 82 Resultados y discusión de genes de los cuales no se conocen las funciones bioquímicas (Goodman et al., 1995). Desde hace unos años, Arabidopsis thaliana, ha ganado popularidad como sistema modelo para el estudio de la biología de plantas, debido a que posee un ciclo de crecimiento rápido, relativamente bajo contenido de repeticiones de DNA, un genoma chico, y es una planta de un tamaño pequeño (Meyerowitz, 1987, 1994). En primer lugar, con el objeto de determinar si existe expresión de ARNm de AtFH en las plantas atfh-1 y as-AtFH, se realizó una reacción RT-PCR con cebadores específicos sobre ARN total extraído de hojas de ambas líneas y se comparó con la línea wt. Los resultados mostraron una disminución en la expresión del AtFH de aproximadamente un 70% en las plantas atfh-1 y as-AtFH (Figura 17A). Estos resultados indican que en la línea atfh-1, la inserción del T-ADN en la región 5’UTR del gen AtFH no bloquea la expresión del mismo, pero sí reduce significativamente su transcripción. Así, la línea atfh-1 no es una mutante nula para la expresión de AtFH, sino que es una mutante deficiente (knockdown). Los niveles proteicos de AtFH se determinaron mediante análisis por western blot y revelado con anticuerpos específicos anti-AtFH (ver Materiales y Métodos Sección 3.17). Los resultados muestran una disminución de aproximadamente un 50% en los niveles de proteína AtFH tanto en extractos provenientes de mitocondrias de plantas atfh-1 (Figura 17B) como en extractos totales provenientes de hojas de plantas atfh-1 y as-AtFH (Figura 17C). 83 Resultados y discusión (A) wt atfh-1 wt as-AtFH AtFH AtFH ȕ-ACTINA 18 S (B) (C) wt atfh-1 as-AtFH kDa 17 AtFH Figura 17: (A) Análisis por RT-PCR de la expresión del gen de AtFH en plantas atfh-1 (panel izquierdo) y as-AtFH (panel derecho). El RNA total fue obtenido de hojas de plantas de A. thaliana de 22 días. La expresión del gen ARNr 18S y ȕ-ACTINA fueron utilizados como control interno. (B) Análisis de western blot de la proteína AtFH de extractos mitocondriales de plantas wt y mutantes atfh1 usando anticuerpos anti-AtFH recombinante de A. thaliana. (C) Análisis por western blot de los niveles de proteína AtFH en extractos de hojas de roseta de plantas wt, atfh-1 y as-AtFH. Las plantas deficientes en AtFH mostraron un retardo en el crecimiento en las distintas etapas del desarrollo. Sin embargo, no se observaron diferencias significativas en la morfología de raíces, tallos, hojas y flores (Figura 18). 84 Resultados y discusión wt atfh-1 a wt as-AtFH atfh-1 as-AtFH c b Figura 18: Análisis fenotípico de plantas de A. thaliana deficientes en la expresión de AtFH, atfh-1 y as-AtFH comparadas con la línea tipo salvaje (wt). (a) Plantas de 14 días crecidas en medio MS; (b) Plantas de 21 días; (c) Plantas de 45 días. En cuanto al desarrollo reproductivo, las plantas atfh-1 y as-AtFH mostraron alteraciones significativas en la morfología de silicuas y semillas. Se observó una disminución en el peso fresco de las silicuas de alrededor de 30% y 60% para atfh-1 y as-AtFH respecto a la wt. Ambas líneas deficientes también presentaron un menor número de semillas por silicua, 32 ± 5 en las plantas atfh-1 y 19 ± 7 en la línea asAtFH, mientras que las plantas wt mostraron alrededor de 45 ± 6 semillas por silicua (Figura 19). wt atfh-1 as-AtFH Figura 19: Morfología de las silicuas de plantas de A. thaliana wt, atfh-1 y as-AtFH de 60 días. Estos resultados están de acuerdo con los descriptos previamente por nuestro laboratorio, en donde se informó que los niveles de expresión de AtFH se encuentran inducidos entre 3 a 4 veces en órganos reproductivos (Busi et al., 2004). 85 Resultados y discusión Es importante mencionar que los órganos reproductivos de organismos vegetales tienen una alta demanda energética y que dependen de la correcta función mitocondrial. Cabe destacar que la mitocondria tiene un papel central en procesos de alto requerimiento energético como la esporogénesis y la maduración del polen y en procesos como la floración y fructificación (Brennicke et al., 1999). Los resultados obtenidos también concuerdan con el aumento en los niveles de ARNm de frataxina observado en tejidos de alta demanda energética de mamíferos, como el hígado, riñón, tejido adiposo y células cardíacas (Koutnikova et al., 1997). Nuestros resultados revelan además que la mutación en la región 5’UTR del gen AtFH correlaciona con una deficiencia en el proceso de fructificación y formación de semillas. A partir de los datos mencionados, y teniendo en cuenta que frataxina es una proteína mitocondrial, se puede inferir que la deficiencia de AtFH podría causar un desequilibrio energético, provocando una disfunción mitocondrial que afectaría algunos procesos de alta demanda energética, como lo es la fructificación en plantas. Esto avalaría una de las posibles funciones postuladas para frataxina, que es su participación en el metabolismo energético mitocondrial. 4.1.5. Análisis fenotípico de las líneas mutantes atfh-2 y atfh-3 Como mencionamos previamente, no se pudieron aislar plantas homocigotas de las líneas atfh-2 y atfh-3. El análisis fenotípico de las silicuas de las plantas heterocigotas reveló la existencia de aproximadamente un 25% de óvulos abortados distribuidos a lo largo de la silicua (Figura 20). Por otro lado el genotipo de las plantas provenientes de semillas viables no es significativamente diferente a la relación 2:1 (plantas resistentes: plantas sensibles a kanamicina). Estos resultados sugieren que la disrupción de AtFH resultaría en una letalidad a nivel del cigoto o embrión. Este resultado coincide con la letalidad embrionaria informada en ratones knockout que presentaban una deleción en el cuarto exón del gen de frataxina (Cossée et al., 1999). 86 Resultados y discusión wt atfh-2 atfh-3 Figura 20. Silicuas inmaduras de plantas wt y plantas heterocigotas atfh-2 y atfh- 3. Las flechas indican los óvulos abortados en el estado de cotiledón durante el desarrollo embrionario. Estudios posteriores llevados a cabo en plantas de A. thaliana (mutantes S594203 y S-622008), que contienen la inserción de T-ADN en la región codificante de AtFH, en el 2º intrón y 4º intrón respectivamente, revelaron que la deleción de este gen causa la detención del desarrollo del embrión en el estadio globular (4 días post polinización) (Vazzola et al., 2007). Estos datos, junto con la variación de los niveles de expresión de AtFH a lo largo del crecimiento y desarrollo del embrión, permiten sugerir además que AtFH cumple un papel importante durante la embriogénesis. A partir de los datos obtenidos, podemos postular que AtFH es una proteína esencial en plantas, cuya función es fundamental en procesos de alto requerimiento energético como la embriogénesis, floración y fructificación. 4.1.6. Caracterización de las plantas de Arabidopsis thaliana que expresan el gen AtFH en orientación sentido (s-AtFH) 4.1.6.1. Fenotipo de plantas transgénicas s-AtFH El análisis fenotípico de las plantas s-AtFH reveló un retardo marcado en el crecimiento respecto a las líneas wt en distintas etapas del desarrollo vegetativo 87 Resultados y discusión (Figura 21 A). Este retardo en el crecimiento fue más evidente que el observado para las líneas deficientes atfh-1 y as-AtFH (Figura 21 B). Además, luego de 15 días de vida, las plantas s-AtFH mostraron un aspecto clorótico, no pudiendo sobrevivir más allá de los 20 días. Estos resultados fueron llamativamente diferentes a los observados para las líneas deficientes ya que a pesar de tener un crecimiento más retrasado respecto a las wt, éstas alcanzan el estadio adulto y la etapa reproductiva. (A) (B) wt wt atfh-1 as-AtFH s-AtFH s-AtFH Figura 21: (A) Fenotipo de plantas wt y s-AtFH luego de 20 días de crecimiento. (B) Comparación del fenotipo de plantas wt, atfh-1, as-AtFH y s-AtFH luego de 20 días de crecimiento. Luego del análisis fenotípico, se llevó a cabo la determinación de los niveles de transcripción de AtFH por PCR en tiempo real (Figura 22A). El análisis de AtFH en plantas de 5, 10, 15 y 20 días reveló una disminución progresiva en sus niveles de expresión, lo que sugiere la existencia de algún mecanismo de represión a nivel transcripcional . Por otra parte, los niveles de proteína AtFH analizados por western blot mostraron una disminución de aproximadamente 30% (Figura 22B). 88 Resultados y discusión (A) (B) wt s-AtFH AtFH Figura 22: (A) Análisis por PCR en tiempo real de la expresión de AtFH (endógeno) en plantas wt (barras blancas) y s-AtFH (barras negras) de 5, 10, 15 y 20 días. (B) Análisis por western blot de los niveles de AtFH en extractos de hojas de plantas wt y s-AtFH de 10 días. Los resultados obtenidos muestran que no hubo sobreexpresión de AtFH en las plantas s-AtFH. En contraste, se observó una disminución en los niveles de AtFH endógeno, con una disminución de la cantidad de proteína al igual que las líneas atfh1 y as-AtFH. Sin embargo, el fenotipo de la línea s-AtFH fue más marcado que las líneas deficientes en AtFH, ya que las plantas tuvieron una sobrevida de no más de 20 días. El silenciamiento de genes por ARN (“RNA silencing”) se refiere colectivamente a diversos eventos basados en el procesamiento del ARN que resultan en una inhibición secuencia específica de la expresión génica, ya sea a nivel transcripcional, de la estabilidad del ARNm o de la traducción. Estos procesos cursan a través de tres eventos bioquímicos característicos: la formación de dobles hebras de 89 Resultados y discusión ARN (dsARN), el procesamiento de ellas generando pequeñas unidades de dsARN de 20 – 26 nucleótidos con extremos cohesivos (“staggered”) y la acción inhibitoria de ARN simple hebra seleccionadas que actúan interfiriendo la complementariedad de forma parcial o total entre ARN y ADN (Brodersen and Voinnet, 2006). El silenciamiento generado por ARN exógenos provoca la ruptura de los transcriptos, posterior a la formación de dsARN. El silenciamiento post-transcripcional (PTGS) fue descubierto en plantas transgénicas de Petunia y se evidenció como una pérdida de la expresión de ambos transgenes, ya sea en orientación sentido o antisentido, como así también de la del gen endógeno homólogo (Napoli et al., 1990 ). A la vista de los resultados obtenidos con las plantas s-AtFH, podemos sugerir la existencia de un mecanismo de silenciamiento ante la expresión del transgen en orientación sentido, generando la menor expresión de AtFH endógeno y menor cantidad de proteína, efecto contrario al buscado. 90 Capítulo II Resultados y discusión Capítulo II: En los últimos años se han realizado numerosos trabajos basados en modelos animales y de levaduras que mostraban deficiencia en la expresión de frataxina (Rötig et al., 2002). De esta forma, a partir de estudios in vivo se sugirió que frataxina es requerida para la homeostasis del hierro, producción de proteínas Fe-S, fosforilación oxidativa y protección contra agentes oxidantes (Rötig et al., 2002). Sin embargo, son pocos los estudios bioquímicos sobre la caracterización de esta proteína. La alta similitud secuencial y estructural de frataxinas de diversos orígenes permite sugerir que cumpliría la misma función en todos los organismos. Recientemente, Park y col. describieron que la frataxina recombinante de levaduras (YFH) posee actividad Ferrooxidasa (Park et al., 2002). Se informó además que YFH y la frataxina humana se ensamblan en un oligómero de aproximadamente 840 kDa que acumula hierro en forma soluble. A partir de estos datos se postuló que la frataxina sería una chaperona de Fe2+ (O'Neill et al., 2005). Sin embargo, no está claro el papel que cumpliría esta proteína en la protección al daño oxidativo causado por metales. Para tener un mayor conocimiento acerca de la actividad biológica de frataxina, es necesario llevar a cabo también la caracterización bioquímica de la proteína. Para ello, hemos desarrollado un método eficiente para la producción de AtFH recombinante de Arabidopsis thaliana. 5.1. Clonado, expresión y purificación de AtFH recombinante El ADNc de AtFH se obtuvo por RT-PCR utilizando cebadores específicos, como se detalla en Materiales y Métodos. El ADNc fue digerido con enzimas de restricción y clonado en el vector pET24a, generando el plásmido pATF01 que codifica para AtFH fusionada a un fragmento de 6 histidinas (AtFH-His6). Así, el vector pATF01 se utilizó para la transformación de diferentes cepas de E. coli. 92 Resultados y discusión La expresión de proteínas funcionales en células heterólogas, es el principio básico de la biotecnología moderna. Desafortunadamente, muchas proteínas son dificultosas para ser expresadas fuera de su contexto original (Gustafsson et al., 2004). La expresión de proteínas recombinantes en células de E. coli es el sistema más ampliamente usado para la obtención de proteínas heterólogas debido a su alta tasa de crecimiento, capacidad para el cultivo continuo, bajo costo y amplia caracterización (Swartz, 2001). Con el objeto de determinar el huésped adecuado para la expresión de AtFH-His6 se transformaron dos tipos de células competentes de E. coli: BL21 (DE3)-RIL y BL21 (DE3). Las células BL21 (DE3)-RIL, llamadas también Codon-plus, contienen copias extras de genes que codifican para los ARNst argU, ileY y leuW. Estos ARNst tienen la capacidad de reconocer los codones raros AGA/AGG (arginina), AUA (isoleucina), y CUA (leucina), respectivamente, representados con baja frecuencia en las células de E. coli. La utilidad de esta cepa radica en que se ha reportado que la presencia de codones raros en la construcción recombinante tiene una influencia significativa en los niveles de expresión, afectando la cantidad y calidad de las proteínas producidas en el huésped (Yan et al., 2004 ; Gustafsson et al., 2004). En las células BL21 (DE3)-RIL se detectó una proteína de masa molecular aparente de 15 kDa, de acuerdo con la masa molecular esperada para AtFH-His6, mientras que no se detectaron niveles de expresión significativos de la proteína recombinante en la cepa BL21 (DE3) (Figura 23). Esto puede deberse a que el mRNA de AtFH contiene 12 codones raros (2AGG, 2 CUA, 4 AGA, y 4 AUA). De esta forma, la presencia de los ARNst adicionales en la cepa BL21 (DE3)-RIL podría ser responsable de los altos niveles de expresión de AtFH. 93 Resultados y discusión Figura 23. Análisis de la expresión de AtFH recombinante en células BL21 (DE3) (A) y BL21 (DE3)-RIL (B). Las proteínas solubles totales fueron corridas en un gel de SDS-PAGE y reveladas por tinción con Coomasie blue. Fila 1, proteínas bacterianas antes de la inducción con IPTG; filas 2, 3 y 4, corresponden a proteínas bacterianas luego de 3, 6 y 18 hs. de la inducción con IPTG; fila 5, marcado de peso molecular. El análisis densitométrico usando el programa Gel Pro analyzer reveló que los niveles de expresión de AtFH en las células BL21 (DE3)-RIL fue de aproximadamente un 22 % de la proteína celular total (Figura 23B). El procedimiento de aislamiento de AtFH-His6, se llevó a cabo mediante la purificación en un paso, usando columnas de afinidad comerciales, HiTrap Chelating HP column, equilibradas con 0.1 M NiSO4. La proteína se eluyó utilizando un gradiente de Imidazol 20 – 500 mM. El análisis por SDS-PAGE de las diferentes fracciones eluidas de la columna cromatográfica reveló la presencia de una única banda proteica de 15 kDa en el rango 200-250 mM de imidazol (Figura 24A). A partir de 500 ml de medio de cultivo se obtuvieron aproximadamente 1.5 g de células de E. coli y 3.2 mg de la proteína recombinante purificada. La presencia de AtFH-His6 se comprobó mediante análisis por western blot utilizando anticuerpos tetra-His comerciales, que reaccionan contra el tag de histidinas C-terminal de la proteína (Figura 24B). Utilizando este procedimiento de cromatografía en un solo paso, hemos logrado purificar el homólogo de frataxina en Arabidopsis con alto rendimiento y sin productos de degradación. 94 Resultados y discusión Figura 24. (A) Análisis por SDS-PAGE de las fracciones eluidas de la cromatografía en columna de Ni++. Los números en la parte superior de la figura indican la concentración de imidazol de cada una de las fracciones. (B) Análisis por western blot de la fracción eluida a 200 mM de imidazol usando anticuerpos anti-His6.. Los números indican la masa molecular de las proteínas marcadoras utilizadas. 5.3. Atenuación de la reacción de Fenton por AtFH Para analizar la funcionalidad de la proteína recombinante AtFH producida en bacterias decidimos evaluar su capacidad para atenuar la reacción de Fenton. Como se sugirió previamente, frataxina podría funcionar como chaperona de hierro y de esta forma, su presencia podría atenuar el daño oxidativo por metales. Así, se llevó a cabo el ensayo de atenuación midiendo la inhibición de la producción de malondialdehído luego de la adición de ácido tiobarbitúrico como se describió previamente (Park et al., 2002). En la Tabla 3 se muestra que en presencia de AtFH-His6 recombinante la producción de malondialdehído disminuye aproximadamente un 55 % respecto al control en ausencia de AtFH. Los controles que se utilizaron fueron analizados y procesados de igual manera que la muestra incógnita. La concentración de proteína que se utilizó en este experimento estaba en exceso respecto al Fe y H2O2 (en ambos casos relación 2:1). Con este resultado, se evaluó la posibilidad que la disminución en la producción de malondialdehído se deba a que la proteína recombinante secuestrara el Fe a través del 95 Resultados y discusión tag de histidinas y de esta manera atenuara la reacción de Fenton. Para descartar esta hipótesis, se expresó AtFH recombinante con codón de terminación (AtFH-STOP) en células BL21 (DE3)-RIL, y se purificó usando cromatografía de DEAE-sepharose y columna de intercambio iónico Mono-Q, seguida de filtración en gel (Superose 12). Bajo estas condiciones, se obtuvo la proteína ATFH-STOP con una pureza de un 95% estimada por electroforesis de SDS-PAGE (Figura 25). 1 2 3 kDa -48 -24 -18 -14 Figura 25: Análisis por SDS-PAGE de la proteína AtFH-stop. Las calles 1, 2 y 3 indican distintas fracciones de elución de la columna Superose12. Los números en la columna derecha indican la masa molecular (kDa) de los marcadores utilizados. De esta manera, utilizando estos tres pasos cromatográficos se obtuvieron 0.2 mg de AtFH-stop por litro de medio de cultivo, de la proteína purificada a partir de aproximadamente, 3 g de células. Cuando la proteína AtFH-STOP purificada se adicionó al medio de reacción, la producción de malondialdehído se inhibió aproximadamente dos veces como se había visto para el caso de la proteína recombinante AtFH- His6 (Tabla3). Esto indicó que la presencia de la secuencia de fusión a histidina no tuvo ningún efecto sobre la reacción de atenuación. 96 Resultados y discusión Tabla 3: Degradación oxidativa de 2- deoxíribosa a malondialdehído Condición a MDA (nmol) dRibosa + Fe(II) 0.093 +/- 0.018 dRibosa + Fe(II) + AtFH- His6 0.078 +/- 0.012 dRibosa + Fe (II) + AtFH-stop 0.062 +/- 0.008 dRibosa + Fe(II) + H2O2 0.365 +/- 0.042 dRibosa + Fe(II) + H2O2 + AtFH- His6 0.171 +/- 0.031 dRibosa + Fe(II) + H2O2 + AtFH-stop 0.152 +/- 0.020 dRibosa + H2O2 0.031 +/- 0.005 a La mezcla de reacción se incubó en presencia o ausencia de frataxina recombinante fusionada a histidina (AtFH-His6) o con codón de terminación (AtFH-STOP) Por lo tanto la proteína AtFH-His6 no solamente es útil para el procedimiento de purificación en un solo paso, sino también para ser utilizada en estudios funcionales sin necesidad de cortar el péptido de fusión His6. Los resultados obtenidos indican que AtFH es una proteína funcional, con capacidad de atenuar la reacción de Fenton in vitro. A partir de estos datos, podemos postular que AtFH estaría involucrada en la homeostasis de Fe, la protección contra el daño oxidativo por metales y en el mantenimiento del estado redox celular. 97 Capítulo III Resultados y discusión Capítulo III: En las mitocondrias se llevan a cabo procesos fundamentales para la vida celular, como la respiración a través de la cadena de transporte de electrones de la membrana interna, que genera el gradiente electroquímico necesario para la producción de ATP (fosforilación oxidativa) y la oxidación de ácidos tricarboxílicos o ciclo de Krebs. Tres de los cuatro complejos respiratorios mitocondriales, CI, CIII y CIV, están formados por proteínas codificadas en el genoma nuclear y mitocondrial, a diferencia del CII que es el único que posee todas sus subunidades codificadas en el núcleo (Scheffler, 1998; Figueroa et al., 2001; Heazlewood et al., 2003; Millar et al., 2004). Para el correcto ensamblado de estos complejos multiproteicos, la expresión de los respectivos genes nucleares y mitocondriales debe estar finamente regulada (Brennicke et al., 1999). Parte de este control y la responsabilidad ante demandas energéticas específicas son llevadas a cabo a nivel transcripcional. El complejo I de la cadena respiratoria (CI, NADH:ubiquinona oxidoreductasa) tiene como función, transferir electrones desde el NADH generado en la matriz hacia la ubiquinona a través de los centros redox (FMN y centros Fe-S). En este proceso, los protones son translocados a través de la membrana interna para generar la fuerza protón-motriz necesaria para la síntesis de ATP. Esta es una función importante dentro de la mitocondria, ya que sirve para mantener los niveles normales de tasa respiratoria y para una producción de ATP eficiente durante el desarrollo de la planta (Rasmusson et al., 1998). De las 30 subunidades que componen el CI en Arabidospsis, 17 se encuentran codificadas en el genoma nuclear, y luego son traducidas en el citoplasma e importadas hacia la mitocondria, y otras 4 subunidades están codificadas a nivel del genoma mitocondrial (Heazlewood et al., 2003 ). Además, el CI posee una subunidad que une un grupo flavin mononucleótido y 5 ferrosulfoproteínas que en combinación resultan en un total de 8 centros Fe-S (Heazlewood et al., 2003 ). 99 Resultados y discusión Como describimos previamente, se creó en el laboratorio un modelo de disfunción mitocondrial basado en la utilización de líneas con deficiencia en la subunidad 9 de la ATP sintasa mitocondrial (u-ATP9) (Gómez-Casati et al., 2002; Busi et al., 2006). Utilizando este modelo, se informó la inducción de tres genes nucleares que codifican proteínas Fe-S mitocondriales del CI, PSST, TYKY y NADHbp, además del aumento en los niveles de expresión de AtFH (Gómez-Casati et al., 2002); (Busi et al., 2004). Diversos trabajos han descripto la utilización de mutantes e inhibidores del CI y la deficiencia de distintas subunidades de este complejo ha sido asociado a disfunción mitocondrial (Duarte and Videira, 2000; Noctor et al., 2004; Garmier et al., 2008a). Asimismo, se informó que la deficiencia de frataxina provoca una pérdida de la función mitocondrial en células de mamíferos (fibroblastos, células neuronales), levaduras (Saccharomyces cerevisiae) y nematodos (Caenorhabditis elegans) (Schapira, 2000; Pandolfo, 2002; Rea et al., 2007). Recientemente se resolvió la estructura cristalina del dominio hidrofílico del CI de Thermus thermophilus (Sazanov and Hinchliffe, 2006). Los resultados mostraron la presencia de 9 centros Fe-S y una subunidad nueva llamada Nqo15, de estructura similar a frataxina. A partir de estos datos se ha propuesto que Nqo15 estaría involucrada en la reparación de los grupos Fe-S del CI. Si bien no se describió que frataxina forme parte del CI en otros organismos, recientemente se describió que en E. coli, el homólogo de frataxina (CyaY) no es un componente estructural del CI pero interactúa de forma transitoria con el mismo (Pohl et al., 2007). Además se determinó que en células mutantes de E. coli con deficiencia en CyaY la cantidad de proteína correspondiente al CI está disminuída en un 70%. Estos resultados están de acuerdo con la posible función de frataxina en la biogénesis de proteínas Fe-S, sin embargo, son escasos los trabajos sobre la posible asociación de frataxina con el CI o el efecto de la deficiencia de frataxina sobre el mismo en organismos eucariotas. A partir de lo expuesto, decidimos investigar el efecto de la deficiencia de AtFH sobre el CI en plantas de Arabidopsis. 100 Resultados y discusión 6.1. Efecto de la deficiencia de AtFH sobre la función mitocondrial en Arabidopsis thaliana A fin de evaluar el estado energético en las plantas de Arabidopsis deficientes en AtFH, se decidió medir los niveles de ATP y el consumo de oxígeno en hojas y flores de plantas atfh-1 y as-AtFH. Encontramos una disminución marcada del contenido de ATP total en ambos tejidos. Las plantas atfh-1 mostraron un descenso en los niveles de ATP de aproximadamente 32 % en hojas y flores; mientras que la línea as-AtFH mostró niveles 46% y 50% menores también en hojas y flores, respectivamente, respecto a la línea wt (Figura 26). (A) (B) Figura 26. Determinación de niveles de ATP en hojas (A) y flores (B) de plantas wt, atfh-1 y as-AtFH de Arabidopsis. PF: peso fresco. El contenido de ATP total se determinó por bioluminiscencia. 101 Resultados y discusión Se determinó además la tasa respiratoria de las plantas atfh-1 y as-AtFH. Los resultados mostraron un descenso de 28% y 26% en el consumo de oxígeno en hojas de plantas atfh-1 y as-AtFH, respectivamente (Figura 27A), mientras que se observó una disminución de aproximadamente 40% en el consumo de oxígeno en flores de ambas líneas deficientes (Figura 27B). Estos datos se correlacionan con los menores niveles de ATP total observados. (A) (B) Figura 27. Determinación del consumo de O2 (ȝmol O2. min-1. g-1 PS) en hojas (A) y flores (B) de plantas wt, atfh-1 y as-AtFH. 102 Resultados y discusión En conjunto, la menor tasa respiratoria y la disminución en los niveles de ATP en las plantas atfh-1 y as-AtFH, sugieren que la deficiencia de AtFH lleva a una disfunción mitocondrial. Estos datos están de acuerdo con las alteraciones morfológicas observadas en ambas líneas en los órganos reproductivos, que son dependientes de la correcta función de la mitocondria. Nuestros datos coinciden con lo descripto para pacientes o modelos celulares de Ataxia de Friedreich, que exhiben bajos niveles de ATP en distintos tipos de células (musculares y hepáticas) (Ristow et al., 2000; Thierbach et al., 2005; Koutnikova et al., 1997; Wilson and Roof, 1997). Los resultados obtenidos refuerzan lo postulado previamente, sobre la posible función de frataxina como activadora de la conversión energética y la fosforilación oxidativa (Ristow et al., 2000). Estudios realizados en mutantes en frataxina de Saccharomyces cerevisiae y Caenorhabditis elegans permitieron asociar la deficiencia de esta proteína a una disminución en la respiración celular (Babcock et al., 1997; Zarse et al., 2007); mientras que trabajos realizados en líneas sobreexpresantes de frataxina, han demostrado que actúa como un activador de la fosforilación oxidativa, provocando un aumento en el potencial de membrana mitocondrial y un aumento en el contenido celular de ATP (Ristow et al., 2000). En nuestro laboratorio, se comprobó la recuperación de la tasa respiratoria en mutantes nulas ¨yfh de Saccharomyces cerevisiae cuando se complementaron con el gen AtFH, lo que sugiere un aumento en la actividad de la cadena de transporte de electrones generada por esta proteína (Busi et al., 2004). La disfunción mitocondrial en las líneas de Arabidopsis deficientes en AtFH, representada por la baja tasa respiratoria y bajos niveles de ATP, podría deberse a una alteración en la actividad de los complejos respiratorios que contienen grupos Fe-S (CI, CII y CIII), ya que una de las posibles funciones de frataxina es su participación en la biogénesis de estos grupos (Chen et al., 2002). Esto avalaría la participación de AtFH como activador de la fosforilación oxidativa también en organismos vegetales. 103 Resultados y discusión 6.2. Efecto de AtFH sobre la transcripción y actividad del CI mitocondrial 6.2.1. Determinación de los niveles de expresión de genes del CI en plantas con deficiencia en AtFH Con el objeto de evaluar la respuesta del genoma nuclear frente a la disfunción mitocondrial inducida por la deficiencia de frataxina decidimos analizar los niveles de expresión de tres genes nucleares que codifican para subunidades Fe-S del CI: PSST, TYKY, NADHbp en las plantas atfh-1 y as-AtFH. También se evaluó la expresión del gen Nad9, que forma parte del CI y también codifica para una subunidad con centro Fe-S, pero se diferencia de los anteriores en que está codificada en el genoma mitocondrial. Luego de la extracción de ARN total y la reacción de retrotranscripción utilizando hexámeros, se llevaron a cabo reacciones de PCR semicuantitativa con oligonucleótidos específicos para cada gen (ver Materiales y Métodos). La Figura 28A muestra los niveles de expresión de: Nad9, PSST, TYKY, NADHbp. La cuantificación por densitometría de la abundancia relativa de los transcriptos para cada uno de estos genes muestra una inducción de Nad9 (entre 1.5 y 2.1 veces) en las plantas atfh-1 y as-AtFH. Se observó también inducción en los niveles de transcripción de PSST (alrededor de 2.8 y 2.5 veces, respectivamente). Además, los niveles de NADHbp se encontraron aumentados entre 4.1 y 4.9 veces en las plantas atfh-1 y as-AtFH, respectivamente. En contraste, se observó una leve disminución del transcripto que codifica para TYKY en las plantas atfh-1 y as-AtFH (Figura 28 B). 104 Resultados y discusión (A) wt atfh-1 as-AtFH Nad9 PSST TYKY NADHbp E-ACTINA (B) Figura 28: (A) Análisis de la expresión de los genes del CI, Nad9, PSST, TYKY, NADHbp a partir de hojas de plantas mutantes atfh-1, transgénicas as-AtFH y wt . La expresión del gen de ȕ-ACTINA, se utilizó como control. (B) Análisis de la expresión relativa de los genes mostrados en (A) por densitometría utilizando el programa Gel-Pro Analyzer. Barras blancas: líneas wt, barras grises, plantas atfh-1 y barras negras, plantas as-AtFH. También realizamos el análisis de expresión de los genes descriptos en la figura anterior en órganos florales. En general, el comportamiento fue similar a lo observado previamente en hojas. Los niveles de transcripción de Nad9 fueron 2.3 y 1.5 veces superiores en las plantas atfh-1 y as-AtFH, respectivamente. Se encontró una inducción de 1.6 veces de PSST en plantas atfh-1, mientras que la expresión de NADHbp fue 2.3 y 2.9 veces superior en las plantas atfh-1 y as-AtFH, 105 Resultados y discusión respectivamente. Por otra parte no se observaron diferencias significativas en la transcripción del ARNm de TYKY (Figura 29). (A) wt atfh-1 as-AtFH Nad9 PSST TYKY NADHbp E-ACTINA (B) Figura 29: (A) Análisis de la expresión de los genes del CI, Nad9, PSST, TYKY, NADHbp a partir de flores de plantas mutantes atfh-1, transgénicas as-AtFH y wt. La expresión del gen de ȕ-ACTINA, se utilizó como control. (B) Análisis de la expresión relativa de los genes mostrados en (A) por densitometría utilizando el programa Gel-Pro Analyzer. Barras blancas: líneas wt, barras grises plantas atfh-1 y barras negras plantas as-AtFH. Los resultados mostrados en las Figura 28 y 29, están de acuerdo con lo observado previamente en plantas u-ATP9 que tienen una disfunción mitocondrial 106 Resultados y discusión (Gómez-Casati et al., 2002). Se postuló que en estas plantas la disfunción mitocondrial podría generar alguna señal induciendo de esta forma la transcripción de ciertos genes nucleares del CI que codifican proteínas relacionadas con la biogénesis de la organela. Resultados recientes obtenidos en el laboratorio con microarreglos de ADNc sobre plantas u-ATP9 mostraron también la inducción de algunos factores como ARR2 (Arabidopsis Response Regulator 2) (Busi y col, manuscrito enviado). Además resultados preliminares de PCR en tiempo real mostraron también una inducción de ARR2 en plantas atfh-1 y as-AtFH (Gómez Casati y col, resultados inéditos). Se describió que ARR2 es un activador transcripcional de algunos genes como PSST, una de las subunidades Fe-S del CI de arabidopsis (Lohrmann et al., 2001). Se propuso además que ARR2 estaría involucrado en un sistema complejo de dos componentes modulando un gran número de vías de transducción de señales relacionadas con estímulos hormonales (Hass et al., 2004). Por lo tanto, la inducción de la expresión de los genes nucleares PSST y NADHbp y el gen mitocondrial Nad9 puede ser interpretada como una compensación nuclear y una adaptación mitocondrial, respectivamente, en respuesta a la disfunción de la organela, asociada a alteraciones en la respiración y menor contenido de ATP. Por otro lado, mencionamos que el gen Nad9 es de codificación mitocondrial, sin embargo, tiene similar comportamiento que los genes codificados en el núcleo. A fin de cuantificar los niveles de la proteína NAD9, llevamos a cabo un análisis de western blot con anticuerpos específicos contra esta subunidad (cedidos por el Dr. Daniel González, UNL). Los resultados muestran que no existe una alteración significativa en los niveles de NAD9 en plantas atfh-1 y as-AtFH respecto a los niveles de NAD9 encontrados en plantas wt (Figura 30). wt atfh-1 as-AtFH NAD9 Figura 30: Análisis de western blot de la proteína NAD9 en extractos mitocondriales de plantas wt y líneas deficientes atfh-1 y as-AtFH utilizando anticuerpos anti-NAD9 recombinante. 107 Resultados y discusión Se postuló que la biogénesis mitocondrial depende de la fina coordinación de la transcripción en los genomas nuclear y mitocondrial en respuesta a señales de desarrollo, ambientales y metabólicas. Sin embargo, recientemente se llevaron a cabo, estudios sobre el papel de ambos genomas en la biogénesis de los complejos respiratorios (Giege´ et al., 2005). Se observó que dicha coordinación ocurre a nivel del ensamblaje de los complejos más que a nivel transcripcional. Si bien nuestros resultados mostraron una inducción en la transcripción de Nad9, los niveles similares de proteína NAD9 observados por western blot pueden explicarse por la existencia de un alto control a nivel postranscripcional sobre las proteínas codificadas en la mitocondria (Giegé et al., 2000; Giege´ et al., 2005). 6.2.2. Efecto de la deficiencia de AtFH sobre la actividad del CI El uso de mutantes o de inhibidores del CI es importante para lograr entender en profundidad la función de este complejo respiratorio. Se ha descripto que varios compuestos químicos como la rotenona y otros insecticidas inhiben la actividad del CI (Teeter et al., 1969). Estos inhibidores bloquean la cadena de transporte de electrones entre un centro Fe-S y la ubiquinona (Lümmen, 1998). Se ha informado también que la inhibición de la actividad del CI produce un aumento en la biogénesis de proteínas mitocondriales (Lister et al., 2004; Millar et al., 2004). Con el propósito de averiguar si la deficiencia de AtFH produce modificaciones en la actividad del CI, se midió el consumo de O2 en plantas deficientes en AtFH (atfh-1 y as-AtFH) y plantas wt con y sin el agregado de rotenona (Figura 31). De esta forma, se estimó la actividad del CI como la diferencia entre el consumo de O2 total y el consumo de O2 en presencia de rotenona como se describió previamente (Sabar et al., 2000). La Figura 31 muestra que luego del agregado de rotenona, las plantas wt muestran una tasa de consumo de O2 de alrededor de 1.7 μmol. min-1. g-1 PS (aproximadamente 60% del consumo de O2 en ausencia de rotenona). De esta forma, 108 Resultados y discusión se determinó que el consumo de O2 sensible a rotenona es de 1.1 μmol. min-1. g-1 PS (Tabla 4). Estos datos coinciden con datos previos obtenidos en diferentes organismos vegetales que reflejaron una inhibición de la respiración por rotenona en el rango del 40-60% (Menz et al., 1992; Polygalova et al., 2007; Garmier et al., 2008b). De esta forma, estimamos que la actividad del CI corresponde a la actividad sensible a rotenona y sería aproximadamente del 40% del consumo total de O2 (Barras grises, Figura 31). Se determinó también el consumo de O2 en las líneas deficientes en AtFH, atfh-1 y as-AtFH. Los resultados están de acuerdo con los datos obtenidos previamente en hojas sin tratamiento (Figura 27), donde se observa una disminución de alrededor de 40% en la tasa respiratoria. La incubación de hojas de Arabidopsis en presencia de rotenona provocó una disminución de alrededor de 20% en el consumo de O2 en ambas líneas deficientes en AtFH. Así, estimamos que la actividad respiratoria sensible a rotenona es de alrededor de 0.4 y 0.3 μmol. min-1. g-1 PS para las líneas atfh-1 y as-AtFH respectivamente (Figura 31, Tabla 4). Estos resultados permiten deducir que la actividad del CI respiratorio está disminuida alrededor de 4 veces en las plantas atfh-1 y as-AtFH respecto a las plantas wt, lo que sugeriría una disfunción (al menos parcial) de la actividad del CI mitocondrial causada por la deficiencia de AtFH. 109 Resultados y discusión Figura 31. Determinación del consumo de O2 (ȝmol O2. min-1. g-1 PS) en hojas de plantas wt, atfh-1 y as-AtFH (barras negras). Las barras grises representan la velocidad de consumo de O2 sensible a rotenona. PS: peso seco. Tabla 4: Determinación de la velocidad de consumo de O2 en ausencia y presencia de rotenona _____________________________________________________________________ Línea velocidad de consumo de O2 (μmol . min-1. g-1 PS) - rotenona + rotenona Diferencia _____________________________________________________________________ wt 2.8 ± 0.2 1.7 ± 0.2 1.1 ± 0.4 atfh-1 2.0 ± 0.2 1.6 ± 0.1 0.4 ± 0.3 as-AtFH 2.1 ± 0.1 1.8 ±0.1 0.3 ± 0.2 _____________________________________________________________________ 110 Resultados y discusión En resumen, observamos en las hojas de plantas con deficiencia en AtFH un aumento en los niveles de transcripción de dos genes nucleares, PSST, NADHbp y el gen mitocondrial Nad9, encontrándose niveles similares de NAD9. Sin embargo estos datos no se correlacionan con la disminución observada en la actividad del CI. Tomados juntos estos datos indican que a nivel bioquímico, la ausencia de frataxina sería equivalente al tratamiento con rotenona, ya que ambas alteraciones provocan un funcionamiento anormal del CI con las consecuencias respiratorias ya detalladas. El CI es el más complicado y menos comprendido de los complejos participantes de la fosforilación oxidativa, debido a que es el que tiene mayor cantidad de subunidades y las mismas se encuentran codificadas a nivel nuclear y mitocondrial (Schulte, 2001; Videira and Duarte, 2002; Brandt, 2006; Duarte and Videira, 2007; Maas et al., 2008). La búsqueda de homología ha revelado un alto grado de conservación de ciertos módulos del CI entre varios organismos que se ve reflejada en una gran semejanza estructural entre el complejo proveniente de arqueas, cianobacterias, bacterias y animales, aunque la unidad de entrada de los electrones varíe (Vogel et al., 2007 ). El CI bacteriano está compuesto por 14 subunidades muy conservadas y que son consideradas la estructura mínima requerida para ser funcional (Friedrich et al., 1993; Leif et al., 1993). Los estudios filogenéticos realizados han generado modelos que describen la evolución modular del CI (Leif et al., 1993; Friedrich and Weiss, 1997; Finel, 1998; Friedrich and Scheide 2000; Mathiesen and Hagerhall, 2003; Friedrich and Bottcher, 2004). En estos modelos se propone que este complejo se origina por fusión de ensamblados proteicos preexistentes constituyendo módulos para la transferencia de electrones y el transporte de protones. En más detalle, se postula que CI tiene su origen en una hidrogenasa soluble dependiente de niquelhierro (con homología a PSST/NuoB y NuoD). Esta hidrogenasa ganó un sitio de unión a quinonas (NuoH) y por la adquisición de NuoC (de función desconocida), de una ferredoxina (TYKY/NuoI) y de un translocador de iones (NuoL) se transformó en una proteína de membrana. Esta estructura fue posteriormente expandida por la triplicación de las subunidades translocadoras, NuoM y NuoN. Después que el 111 Resultados y discusión complejo perdió el sitio activo para níquel-hierro y su habilidad para reaccionar con la molécula de hidrógeno, se adquirió el módulo deshydrogenasa (compuesto por NuoE, NuoG y NuoF/NADHbp) y las subunidades de membrana NuoA, NuoK y NuoJ (Vogel et al., 2007 ) (Figura 32). E. coli A. thaliana NuoB PSST NuoF NADHbp NuoI TYKY NuoC NAD9 Bacterias Plantas Figura 32: Esquema del ensamblado del CI bacteriano y de plantas desde el punto de vista de la evolución modular (Modificado de Vogel et al., 2007 ). Como mencionamos antes, además de las subunidades descriptas en el párrafo anterior, el genoma mitocondrial vegetal codifica a NAD7 y NAD9, homólogos a NuoD y NuoC respectivamente (Rasmusson et al., 1998). El conocimiento sobre la formación y ensamblaje del CI mitocondrial en plantas superiores se basó principalmente en estudios realizados en mutantes de maíz, tabaco y Arabidopsis (Vogel et al., 2007 ). Plantas con mutaciones en distintas subunidades 112 Resultados y discusión presentaron un incorrecto funcionamiento del CI. Aunque sólo un gen estuviera afectado y originara la disfunción, y que no se observaran variaciones significativas en los niveles de trasncripción de genes mitocondriales, numerosos polipéptidos fueron detectados solamente en trazas o con falencias en el ensamblado y la unión a la membrana, demostrando que es fundamental la presencia de todos los componentes del CI para la estabilidad de las proteínas (Marienfeld and Newton, 1994; Brangeon et al., 2000). En plantas de tabaco mutantes CMSI y CMSII (cytoplasmic male sterile I and II) está ausente la subunidad mitochondrial NAD7 (Pla et al., 1995; Gutierres et al., 1999). El fenotipo presente en estas plantas es similar y es provocado por la ausencia de niveles detectables de otras subunidades, tanto mitocondriales como nucleares. Aunque al momento de evaluar estos transcriptos se observaron niveles normales o inducidos, al igual que en las plantas deficientes en frataxina, el hecho de no obtener proteínas o complejos funcionales sugiere la proteólisis de las proteínas no integradas (Gutierre et al., 1997) (Pineau et al., 2005). Por lo tanto, es posible suponer que el correcto ensamblado de los complejos respiratorios (en este caso CI) dependería de mecanismos post-transcripcionales, y en ellos frataxina cumpliría un papel fundamental. 113 Capítulo IV Resultados y discusión Capítulo IV Como mencionamos anteriormente, otro de los complejos mitocondriales que posee centros Fe-S es el complejo respiratorio II, CII, que cataliza la reducción de ubiquinona a ubiquinol. Este complejo multiproteico, también llamado succinato dehidrogenasa, está asociado a la membrana interna mitocondrial y es parte también del ciclo del TCA, catalizando la oxidación de succinato a fumarato. El CII ha sido caracterizado en bacterias y eucariotas (Lemire and Oyedotun, 2002; Yankovskaya et al., 2003). Se ha demostrado que contiene cuatro subunidades: dos proteínas periféricas de membrana, una flavoproteína (SDH1), una proteína Fe-S (SDH2) y dos proteínas pequeñas integrales de membrana (SDH3 y SDH4). En Arabidopsis thaliana, todas las subunidades de SDH están codificadas en el genoma nuclear (Scheffler, 1998; Figueroa et al., 2001; Figueroa et al., 2002; Millar et al., 2004), y sorprendentemente, varias de las subunidades del CII están codificadas por más de un gen. Por el momento se han reportado que tres de los genes, llamados SDH2-1, SDH2-2 y SDH2-3, codifican para la subunidad Fe-S, y dos genes nucleares, designados como SDH1-1 y SDH1-2, codifican para la flavoproteína (Figueroa et al., 2001; Figueroa et al., 2002). SDH2-1 y SDH2-2 tienen estructuras similares, codifican proteínas cercanamente idénticas y tienen patrones de expresión similares, encontrándose su mayor expresión en flores (Figueroa et al., 2001; Elorza et al., 2004). Este resultado, estaría de acuerdo con que SDH2-1 y SDH2-2 probablemente provengan de un reciente evento de duplicación y serían redundantes. A diferencia de esto, se ha reportado una alta expresión tejido específico para SDH2-3. Este gen es estructuralmente diferente y ha divergido hace tiempo de SDH2-1 y SDH2-2; se expresa altamente en embriones durante el desarrollo de la semilla y sus transcriptos se acumulan en semillas secas (Elorza et al., 2006). El sitio de unión de succinato, está formado por la proteína SDH1 que está covalentemente unida a la molécula de FAD, actuando como receptor de iones hidrógenos en el paso inicial de la oxidación del succinato. Esta subunidad de flavoproteína interactúa con la subunidad SDH2, que transfiere electrones hacia la membrana, a través de los tres centros Fe-S. Las dos proteínas integrales de 115 Resultados y discusión membrana, SDH3 y SDH4, anclan los subcomplejos SDH1 y SDH2 del lado de la matriz de la membrana mitocondrial interna, y contienen sitios de unión para la ubiquinona y hemo (Yankovskaya et al., 2003). Otra de las ferrosulfoproteínas encontradas en mitocondrias es la aconitasa, enzima que participa en el ciclo de Krebs catalizando la conversión de citrato a isocitrato en la matriz mitocondrial. Esta proteína pertenece a la familia de las deshidratasas que contienen centros Fe-S, cuya actividad depende de que dicho centro se encuentre en la forma cubana de [Fe4-S4] (Beinert et al., 1996; Gardner, 2002). La enzima purificada es altamente susceptible a la inactivación por formación de centros [Fe3-S4] (Flint et al., 1993). La pérdida de la actividad aconitasa es un indicador de la generación de superóxido y de daño oxidativo en gran variedad de enfermedades degenerativas y envejecimiento (Yan et al., 1997; Schapira, 1999). Esto sugiere que aconitasa funcionaría como un sensor del estado redox celular. Hemos mencionado que una de las posibles funciones de frataxina es su participación en la biogénesis de proteínas Fe-S (ISCs). Esta hipótesis fue recientemente confirmada en levaduras mutantes en frataxina (¨yfh) en las cuales la función de varias proteínas Fe-S se ve severamente afectada (Chen et al., 2002). Sin embargo, poco se conoce acerca del mecanismo por el cual estos grupos inorgánicos se ensamblan en proteínas biológicamente activas (Napier et al., 2005). De acuerdo a esta hipótesis, nosotros postulamos que es posible que la deficiencia de frataxina altere la función de los complejos respiratorios (y posiblemente de otras proteínas mitocondriales) que contienen centros Fe-S en plantas. Previamente demostramos, que AtFH es necesaria para la actividad del CI mitocondrial. A fin de conocer la posible participación de AtFH en la biogénesis de otras ferrosulfoproteínas y complejos de la cadena respiratoria, decidimos investigar el efecto de la deficiencia de AtFH sobre el CII y la aconitasa. 116 Resultados y discusión 7.1. Efecto de AtFH sobre la transcripción de genes que codifican proteínas Fe-S mitocondriales. Para conocer más sobre la posible participación de AtFH en la biogénesis de proteínas Fe-S mitocondriales, se llevó a cabo la determinación de los niveles de expresión de los ARNm que codifican para SDH2-1 y la enzima aconitasa (ACO) en las plantas wt y atfh-1. La Figura 33 muestra el análisis por RT-PCR de ambos transcriptos. Los resultados reflejan que ACO se encuentra inducido en las plantas atfh-1 alrededor de 1.5 veces. Asimismo, se observó un incremento en los niveles de transcripción de SDH2-1 de alrededor de 2 veces en la línea deficiente en AtFH, respecto a las plantas wt (Figura 33). (A) (B) 2,5 atfh-1 ACO SDH2-1 18 S Expresión relativa wt 2 1,5 1 0,5 0 ACO SDH2-1 Figura 33: (A) Análisis por RT-PCR de la expresión del gen de aconitasa (ACO) y subunidad SDH2-1 del CII en plantas wt y atfh-1. El RNA total aislado fue retrotranscipto y luego amplificado usando primers específicos. El producto de PCR fue separado en una electroforesis en gel de agarosa y transferido a membrana de nitrocelulosa e hibridado usando sondas específicas. La expresión del gen ARNr 18S fue utilizado como control interno. (B) Análisis de la expresión relativa de los genes mostrados en (A) por densitometría utilizando el programa Gel-Pro Analyzer. Como se mencionó más arriba, existen 3 genes en Arabidopsis que codifican para la subunidad SDH2: SDH2-1, SDH2-2 y SDH2-3. Con el objeto de determinar si existen diferencias en la expresión de los tres genes, se evaluó la transcripción de los mismos por PCR en tiempo real (Figura 34). Los resultados confirman la inducción 117 Resultados y discusión del gen SDH2-1 observada por RT-PCR. Además se observó una inducción de alrededor de 2 veces en los niveles de transcripción de SDH2-2. Los niveles de expresión del gen SDH2-3 fueron indetectables (no mostrado), lo que está de acuerdo con su expresión en tejido embrionario, durante la maduración de la semilla (Elorza et al., 2006). 4 Expresión relativa 3,5 3 2,5 2 1,5 1 0,5 0 SDH2-1 SDH2-2 Figura 34. Análisis por PCR en tiempo real de los niveles de SDH2-1 y SDH2-2 en plantas wt (barras blancas) y atfh-1 (barras grises). Para evaluar si el aumento en la expresión del ARNm de ACO y SDH2-1 en las plantas atfh-1 se correspondía con un aumento en la actividad proteica, determinamos la actividad enzimática aconitasa y SDH en mitocondrias aisladas de las líneas wt y atfh-1 (Figura 35). Los resultados mostraron una disminución en la actividad de ambas enzimas en la línea deficiente en AtFH. En particular, la actividad aconitasa de la línea mutante fue prácticamente indetectable, mientras que la actividad SDH se encontró disminuida alrededor de un 30 % respecto a la actividad SDH mostrada por la línea wt. Para comprobar si la reducción en los niveles de actividad de estas dos ferrosulfoproteínas se correlacionaba con la disminución en la actividad de otras enzimas mitocondriales, se ensayó además la actividad malato dehidrogenasa (MDH) en las preparaciones de mitocondrias aisladas. La MDH es una enzima que participa 118 Resultados y discusión en una de las reacciones del ciclo del TCA y no contiene grupos Fe-S en su estructura. La Figura 35 (panel izquierdo) muestra que la actividad MDH medida fue similar en mitocondrias provenientes de plantas wt y atfh-1. Así, los resultados indican que la deficiencia de AtFH se correlaciona con la menor actividad de dos ferrosulfoproteínas (aconitasa y SDH), mientras que la actividad de otra enzima que no posee centros Fe-S no se encontró alterada ante la deficiencia de AtFH. Los resultados sugieren que AtFH estaría involucrada en la modulación de la actividad de Actividad relativa proteínas mitocondriales con centros Fe-S. Aconitasa wt atfh-1 SDH wt atfh-1 MDH wt atfh-1 Figura 35: Determinación de la actividad enzimática de proteínas con centros Fe-S: aconitasa (ACO) y succinato dehydrogenasa (SDH) y como control de proteína mitocondrial sin centro Fe-S, malato dehidrogenasa (MDH). En cada ensayo enzimático se utilizaron 10μg de proteína total de extractos mitocondriales de las líneas wt y atfh-1. La actividad de cada enzima en plantas wt fue utilizada como valor de referencia. Los datos mostrados indican niveles aumentados de ARNm de ACO y SDH2-1 en las plantas deficientes en AtFH; sin embargo no se corresponden con un aumento en la actividad enzimática aconitasa y SDH. A partir de estos datos se puede postular que AtFH podría tener un rol en el proceso de maduración de la proteína como por ejemplo, la biogénesis de los centros Fe-S o en el ensamblado de estos grupos a las apoproteínas. Así, los datos obtenidos en el presente trabajo, concuerdan con una de las funciones postuladas para frataxina como es su participación en la biogénesis de grupos Fe-S y ferrosulfoproteínas en mitocondrias. Cabe destacar que este trabajo de Tesis constituye el primer informe sobre el requerimiento de frataxina 119 Resultados y discusión para la actividad completa de dos proteínas con centros Fe-S en organismos vegetales. Nuestros resultados están de acuerdo con lo descripto recientemente sobre la función activadora de la frataxina de levaduras sobre la aconitasa (Bulteau et al., 2004). Se describió que YFH interactúa con la aconitasa de una manera dependiente de citrato, reduciendo así los niveles de oxidación que ocasionan la inactivación de la enzima. De esta forma, frataxina convierte la forma inactiva de la aconitasa [Fe3-S4] en la forma activa [Fe4-S4] de la proteína proveyendo un cuarto átomo de Fe para completar el cluster. Así, frataxina funcionaría como una chaperona de Fe, protegiendo el desensamblado del grupo [Fe4-S4] y promoviendo la reactivación de la enzima (Bulteau et al., 2004). Por otra parte, la participación de frataxina parecería ser importante pero no esencial para la actividad SDH, ya que la misma disminuyó solamente un 30%. Recientemente se ha informado la interacción física existente entre la frataxina de levaduras y la SDH (Gonzalez-Cabo et al., 2005). Además se ha observado una reducción en la actividad del complejo succinato deshidrogenasa en levaduras mutantes que carecían de frataxina. Estos resultados sugieren un posible rol de frataxina en la regulación de la entrada de los electrones a la cadena respiratoria a través del CII. Entonces, frataxina podría afectar la estabilidad o ensamblado del complejo SDH regulando de esta manera su actividad (Gonzalez-Cabo et al., 2005). Finalmente, la respuesta diferencial observada en la disminución de la actividad de aconitasa y SDH, podría reflejar que el proceso de ensamblado de los centros Fe-S o su regulación, ocurre de manera diferente para cada una de estas proteínas. 120 Resultados y discusión 7.2. AtFH y stress oxidativo 7.2.1. Plantas deficientes en frataxina muestran un aumento en el contenido de Fe y en la producción de especies reactivas de oxígeno (ROS) El estrés oxidativo es un estado de la célula en el cual se encuentra alterada la homeostasis óxido-reducción intracelular, es decir el balance entre pro-oxidantes y antioxidantes, resultando en una excesiva formación de especies reactivas de oxígeno (ROS) y /o especies reactivas de nitrógeno (RNS) (Moller, 2001a). La producción de especies reactivas del oxigeno (ROS) es una inevitable consecuencia de la vida en condiciones aeróbicas. Los cloroplastos y los peroxisomas de plantas son los sitios de mayor producción de ROS y probablemente en células no fotosintéticas esto lo realiza la mitocondria (Moller and Kristensen, 2004). Durante la vida celular, ocurren pequeñas fluctuaciones en los niveles de concentración de ROS, debido a una producción endógena durante determinadas situaciones del desarrollo, como es la maduración de semillas, o como resultado normal del metabolismo de la fotosíntesis y la respiración. Estas ROS producidas en baja concentración actuarían como posibles moléculas señales (Droge, 2002), mientras, que un aumento descontrolado de las mismas puede causar la liberación de más radicales libres como . el radical hidroxilo ( OH) que se forma en presencia de iones metálicos como Fe o Cu . (II), el radical superóxido (O2 -), y el oxígeno singlete. Existen varios mecanismos de defensa en la mitocondria, que incluyen moléculas antioxidantes, enzimas de detoxificación y sistemas de reparación que limitan la producción de ROS. Cuando estos mecanismos se encuentran sobrepasados, se produce el daño celular. En general los ROS juegan un papel importante en la inactivación de endonucleasas y consecuentemente producen daño al DNA (Hagar et al., 1996), pueden modificar proteínas, haciéndolas más susceptibles a ataques proteolíticos (Stadtman and Levine, 2000) y atacar lípidos (Rubbo et al., 1994) y polisacáridos (Kaur and Halliwell, 1994;). 121 Resultados y discusión Se ha informado que frataxina normalmente forma complejos multiméricos con Fe, y que la ausencia de la misma, causa un aumento en los niveles de Fe mitocondrial. El Fe cataliza la reacción de Fenton produciendo radical hidroxilo a partir de peróxido de hidrógeno, con el consecuente daño a las macromoléculas mitocondriales (Babcock et al., 1997; Karthikeyan et al., 2002; Puccio and Kœnig, 2002). En trabajos realizados en levaduras mutantes en frataxina, se ha visto un aumento de 10 veces la concentración de Fe mitocondrial, acompañado de un aumento en la producción de ROS (Babcock et al., 1997). Esto llevó a la aparición de un fenotipo “petite “en la mayoría de las colonias examinadas con pérdida de la función mitocondrial, (Foury and Cazzalini, 1997; Wilson and Roof, 1997; Puccio and Koenig, 2000). Otros estudios reportaron además la presencia de daño a nivel del ADN mitocondrial (Bidichandani et al., 1999; Karthikeyan et al., 2002; Lesuisse et al., 2003). Estudios realizados en pacientes con Ataxia de Friedreich, mostraron depósitos de hierro en corazón, sugiriendo una sobrecarga del mismo (Lamarche et al., 1980), como así también signos de daño oxidativo incluyendo múltiples defectos en proteínas Fe-S y pérdida del ADN mitocondrial (Rotig et al., 1997; Bradley et al., 2000). Previamente demostramos que AtFH recombinante fue capaz de atenuar la reacción de Fenton in vitro (ver capítulo II). A partir de esta observación y de lo mencionado más arriba, decidimos evaluar si la deficiencia de AtFH puede provocar un aumento en el contenido de Fe en plantas de Arabidopsis. Para ello, llevamos a cabo la determinación del contenido de Fe soluble total en hojas de plantas wt y atfh1. Los resultados indican que el contenido de Fe en las plantas deficientes en AtFH es aproximadamente 55% mayor que en plantas wt (Figura 36). Así nuestros resultados sugieren que AtFH podría tener un papel importante en el metabolismo del Fe también en organismos vegetales. 122 Resultados y discusión 200 % μmol Fe . g -1 PF 180 160 140 120 100 80 60 40 20 0 wt atfh-1 Figura 36: Determinación espectrofotométrica del contenido de Fe soluble en muestras de hojas de plantas wt y atfh-1. La cantidad de Fe se expresa en μmol Fe soluble . g-1 de peso fresco (PF). A fin de evaluar si la deficiencia de AtFH produce una mayor cantidad de ROS en los tejidos vegetales, se determinó por histoquímica la producción del radical . superóxido (O2 ) y de ROS totales en plantas de Arabidopsis wt y en las mutantes atfh-1. Para determinar la presencia de anión superóxido se infiltraron hojas de ambas líneas con Nitrobluetetrazolium (NBT) que produce un precipitado de color azul en presencia de radical superóxido (Beyer and Fridovich, 1987; Jakubowski and Bartosz, 2000). Luego de la incubación de hojas wt con NBT no se observa tinción alguna, mientras que la incubación de hojas provenientes de la mutante atfh-1 con NBT produjo una precipitación de color azul, especialmente en tricomas (Figura 37A). Los resultados indican que la deficiencia de AtFH lleva a una acumulación de radical superóxido, especialmente en tricomas, estructuras utilizadas para la detoxificación y eliminación de ROS. Por otra parte, se determinó la cantidad de ROS total en hojas y raíces de plantas wt y atfh-1. Los resultados muestran un incremento en la fluorescencia en ambos tejidos provenientes de plantas mutantes luego de la incubación con 2´,7´- 123 Resultados y discusión diclorofluorescein diacetato (H2DCFDA) (Figura 37 B). Estos datos indican que tanto en raíces como en hojas (especialmente en tricomas) de plantas deficientes en AtFH, existe un incremento en los niveles de ROS totales. wt (A) atfh-1 Raíces (B) wt atfh-1 Tricomas wt atfh-1 Figura 37: (A) Determinación histoquímica de anión superóxido en hojas de plantas wt y atfh-1 luego de la infiltración con NBT. (B) Determinación de la acumulación de ROS total en raíces y hojas de plantas wt y atfh-1 utilizando la sonda fluorescente H2CFDA. En conjunto, los resultados permiten postular que AtFH tendría un papel importante en la prevención de la acumulación de altos niveles de Fe y ROS dentro de la célula. Como se describió previamente, el aumento del contenido de Fe (u otros metales) podría causar daño oxidativo vía la reacción de Fenton. Esta hipótesis está de acuerdo con los resultados que muestran que AtFH recombinante tiene la capacidad de atenuar la reacción de Fenton in vitro (Ver Tabla 3, página 99). 124 Resultados y discusión Se ha sugerido que la deficiencia de frataxina sería responsable de la alteración del influjo de hierro (Radisky et al., 1999), defecto en la biosíntesis de centros Fe-S (Foury, 1999; Muhlenhoff et al., 2002), pérdida de la síntesis de ATP (Ristow et al., 2000), y /o inadecuada defensa antioxidante (Chantrel-Groussard et al., 2001), todas condiciones que al final conducen a la acumulación de hierro y al aumento del daño oxidativo. Así, la participación de frataxina en procesos tan diversos podría explicarse únicamente si la función básica de esta proteína fuese la de proveer un mecanismo de almacenamiento de hierro de manera biodisponible y no tóxica (Patel and Isaya, 2001). 7.2.2. Análisis molecular de marcadores de estrés oxidativo Las plantas son especialmente susceptibles a daño oxidativo ya que se produce un aumento en la producción de ROS cuando están sometidas, por ejemplo, a estrés ambiental como altas temperaturas, escasez de agua, polución, etc. (Smirnoff, 1998). Posiblemente las plantas han desarrollado una red dinámica de defensa antioxidante para poder reducir la acumulación de ROS, eliminarlas y detoxificar, y reparar moléculas oxidadas. La red de defensa antioxidante en plantas mejor caracterizada se encuentra en cloroplastos, donde la cadena de transporte electrónico fotosintética puede generar una gran cantidad de ROS por una disipación incompleta de energía (Sweetlove et al., 2002). Las mitocondrias de plantas también producen ROS como resultado del metabolismo aeróbico y por ello, han desarrollado mecanismos que tratan de limitar su acumulación (Millar et al., 2001; Moller, 2001b; Moller and Kristensen, 2004). Como ya se describió, dos de los principales sitos de producción de ROS en mitocondrias son el CI y CIII. Además, un incremento de ROS puede darse por la presencia del radical ubisemiquinona, que puede transferir un único electrón hacia el oxígeno para producir anión superóxido (Gutteridge and Halliwell, 1989). La actividad de la oxidasa alternativa (AOX) reduce la acumulación de ROS mitocondrial disminuyendo los niveles del pool de quinonas (Maxwell et al., 1999). 125 Resultados y discusión En Arabidopsis, AOX, está codificada por 5 genes, cuatro del tipo AOX1 y uno del tipo AOX2 (Considine et al., 2002). La expresión de diferentes genes se ha visto que es tanto tejido como desarrollo específicos (Saisho et al., 1997; Saisho et al., 2001; Thirkettle-Watts et al., 2003). AOX1 se expresa en la mayoría de los tejidos, con la expresión de AOX1c en cotiledones jóvenes y en tejido floral. AOX1b, 1d y AOX2 presentan menores niveles de expresión. La naturaleza de AOX de inducirse por estrés en una amplia variedad de plantas estaría dada, en Arabidopsis, por la mayor respuesta frente a estrés de los genes AOX1a y AOX1d. Análisis por microarreglos en donde se evalúa la respuesta a varios tratamientos de estrés que alteran la función mitocondrial y/o plantas con niveles de AOX alterados, revelan que las AOXs co-expresan con genes que típicamente funcionan afuera de la mitocondria. Existen varias vías de inducción de AOX y cada una de ellas produce una magnitud diferente de inducción; a su vez esa inducción depende de los niveles endógenos de AOX, y por último, puede deberse no solamente al estrés oxidativo sino también ser independiente del mismo, según los genes de AOX que se investiguen y el tejido analizado (Clifton et al., 2006). Por otro lado, se ha descripto que otros dos genes, peroxiredoxina (At3g06050) y calreticulina (At1g56340) estarían involucrados en respuesta a estrés y daño oxidativo en mitocondrias de plantas (Sweetlove et al., 2002). La peroxirredoxina (At3g06050), pertenece a la familia de las tiorredoxinas dependientes de peroxidasas. Esta proteína ha sido observada en el proteoma mitocondrial de Arabidopsis y posee una secuencia de importe a mitocondria (Kruft et al., 2001). Recientemente ha sido enfocada en la investigación en plantas y animales, en el contexto de desarrollo, adaptación, y enfermedad, debido a que sus funciones están involucradas en la defensa antioxidante, reduciendo los peróxidos tóxicos dentro de la célula y en señalización redox (Finkemeier et al., 2005). La peroxirredoxina, es una proteína muy ubicua, encontrada en bacterias (Tartaglia et al., 1990), levaduras (Chae et al., 1994), animales (Kim et al., 1988) y plantas superiores (Baier and Dietz, 1996; Baier and Dietz, 1997). 126 Resultados y discusión La calreticulina (At1g56340), generalmente, ha sido considerada como una proteína no mitocondrial, ya que no posee secuencia de importe a mitocondria, pero sin embargo se la encuentra inducida en condiciones de estrés. Esta proteína ha sido implicada en la homeostasis del Ca+2 mitocondrial en mamíferos, llevando el Ca +2 a mitocondria desde el retículo endoplasmático (Simpson et al., 1997). Durante el estrés oxidativo se producen cambios en la homeostasis del Ca+2 mitocondrial alterando la permeabilidad de la membrana, lo cual genera poros de transición comprometidos en muerte celular programada observados en mamíferos (Pfeiffer et al., 2001). A partir de estos datos, decidimos investigar los niveles de expresión de estos tres marcadores (oxidasa alternativa, peroxiredoxina y calreticulina) en plantas wt y las mutantes deficientes atfh-1. Los resultados mostraron una inducción en la transcripción del ARNm de los tres marcadores en las plantas atfh-1 (Figura 38). Paralelamente se llevó a cabo el análisis por la técnica de PCR en tiempo real, y los resultados reflejaron una inducción de 5 veces en el ARNm de AOX; mientras que PEROX y CAL se encontraron inducidos 3 y 3.5 veces, respectivamente, en las plantas deficientes en AtFH (Figura 39). Figura 38. (A) Determinación de los niveles de ARNm genes de respuesta a estrés por RT-PCR en plantas wt y atfh-1: oxidasa alternativa (AOX, At5g64210), peroxiredoxina (PEROX, At3g06050) y calreticulina (CAL, At1g56340). Las muestras fueron separadas por electroforesis en gel de agarosa, y transferida e hibridadas usando sondas específicas. La expresión del gen RNAr 18S fue utilizada como control interno. 127 Resultados y discusión 7 Expresión relativa 6 5 4 3 2 1 0 AOX PEROX CAL Figura 39. Análisis por PCR en tiempo real de los niveles de AOX, PEROX y CAL en hojas de plantas wt (barras blancas) y atfh-1 (barras grises). El aumento de los transcirptos de AOX y de PEROX, estarían indicando que se estaría registrando la presencia de ROS y sería necesaria una activa producción de proteínas antioxidantes para tratar de minimizar la formación de las mismas. A su vez, el aumento de los niveles de ARNm de CAL, reflejaría el mayor estado de estrés oxidativo en que se encuentra la célula. Estos resultados son consistentes con los mostrados previamente, los cuales se muestra aumento de la producción de ROS y por lo tanto con el estado de estrés oxidativo que ocasiona la deficiencia de AtFH. 7.2.3. Respuesta de AtFH frente a agentes oxidantes Se ha reportado que la adición de H2O2, menadiona y antimicina A a suspensiones de cultivos celulares causan cambios significativos en el proteoma mitocondrial (Sweetlove et al., 2002). Estos cambios se habrían originado por un aumento de proteínas blancos mitocondriales con función antioxidante o de respuesta a estrés, o una asociación selectiva con proteínas que poseen un rol potencial en estrés o en la degradación de proteínas mitocondriales (Sweetlove et al., 2002). Para investigar el papel de frataxina en el estrés oxidativo, hojas de roseta de plantas de 25- 30 días fueron tratadas con concentraciones sub-letales de H2O2 (500 μM). Luego de 4 h. de tratamiento, se determinaron los niveles de ARNm de AtFH 128 Resultados y discusión mediante RT-PCR. Los resultados muestran un incremento en los niveles de transcripción de AtFH de 4 a 5 veces en las plantas tratadas respecto a los controles (Figura 40). (B) (A) 5 Expresión relativa 4,5 4 3,5 3 2,5 2 1,5 1 0,5 0 C H 2 O2 Figura 40. (A) Expresión de AtFH en plantas wt luego del tratamiento con concentraciones sub-letales de H2O2. Los análisis de expresión se realizaron por RT-PCR, seguidos por hibridación con sondas específicas para AtFH. El RNA total fue extraído de hojas luego de 4 horas de infiltración con 0.5 mM de H2O2. Como control se infiltraron hojas con agua destilada. La expresión del gen RNAr 18 S fue usado como control interno. (B) Análisis de la expresión relativa de los genes mostrados en (A) por densitometría utilizando el programa Gel-Pro Analyzer. Este aumento en los niveles de ARNm de AtFH sugiere su posible participación en la defensa oxidante en plantas, de acuerdo con los resultados en los que se encontró que la deficiencia de AtFH va acompañada de un aumento de la producción de ROS y de marcadores de estrés oxidativo. Los resultados concuerdan con lo observado previamente en levaduras, donde la complementación de células de S. cerevisiae ¨yfh con AtFH restaura parcialmente la sensibilidad a agentes oxidantes (Busi et al., 2004). En conjunto, nuestros resultados refuerzan la hipótesis del papel de frataxina en la protección ante el daño oxidativo causado por metales y reparación de los grupos Fe-S. 129 Capítulo V Resultados y discusión Capítulo V: 8.1. Participación de AtFH en la biogénesis de hemoproteínas A pesar de que inicialmente no se detectaron síntomas de porfirias (patologías asociadas a anomalías en la síntesis del hemo) en pacientes con Ataxia de Friedreich, cuando el gen de frataxina no había sido aun identificado, se reportó un defecto en la conversión de protoporfirina IX hacia la formación del hemo (Morgan et al., 1979). Posteriormente se observó que en pacientes con esta patología el fenotipo de la serie roja era normal, y por lo tanto, se sugirió que la síntesis del hemo puede ocurrir en ausencia de frataxina (Becker et al., 2002). Más recientemente Lesuisse y colaboradores (2003), demostraron que los niveles de Fe- protoporfirina IX se encontraban reducidos en levaduras mutantes deficientes en frataxina (ǻyfh). Investigaciones realizadas por Lange y su grupo (2004), mostraron una disminución en la síntesis del hemo en levaduras portadoras de mutaciones en varios componentes de la vía de biosíntesis de ISC, incluída frataxina. En otros trabajos realizados en células de ratones deficientes en frataxina, se observó un aumento en los niveles de protoporfirina IX, una reducción en los niveles de hemo a y hemo c y una disminución de la actividad de la citocromo oxidasa, sugiriendo un defecto entre la protoporfirina IX y el hemo a (Schoenfeld et al., 2005). A partir de ésto se postuló que frataxina podría tener un papel en la regulación del Fe biodisponible dentro de las células. La alteración de la biodisponibilidad del Fe tendría alguna influencia sobre la síntesis del grupo hemo y la biogénesis de hemoproteínas celulares. Así, utilizando nuestro modelo de plantas atfh-1 y as-AtFH, decidimos investigar el efecto de la deficiencia de frataxina sobre el metabolismo del grupo hemo y la biogénesis de hemoproteínas en plantas. 8.1.1. La deficiencia de AtFH provoca una disminución en el contenido de hemo La forma más funcional del hierro encontrada en la naturaleza es el grupo hemo, el cual juega un rol catalítico y regulatorio dentro de la célula (Zhu et al., 131 Resultados y discusión 2002). Se informó que el hemo puede controlar la actividad específica de factores de transcripción y otras proteínas en una variedad de vías metabólicas (McCoubrey et al., 1997; Goessling et al., 1998; Ogawa et al., 2001; Tang et al., 2003). Este control podría ocurrir por unión del cofactor a “motivos regulatorios de hemo” (HRM) presentes en estos factores. Es por este motivo que fluctuaciones endógenas en el hemo pueden ser muy importantes en la regulación del metabolismo celular (Zhang and Guarente, 1995; Atamna et al., 2002; Zhu et al., 2002). El grupo hemo es el quelato formado por la protoporfirina IX con un átomo de Fe (II). Sufre autooxidación rápidamente dando como producto ferri-protoporfirina. Este complejo férrico tiene un residuo cargado positivamente que se puede neutralizar con un ión cloruro, formando un complejo piramidal – cuadrado pentacoordinado usualmente llamado hemina (Batlle, 1997). Se ha informado que el hemo, bajo la forma de hemina actúa como un sistema regulador intracelular de utilización de oxígeno (Pal and Joshi-Purandare, 2001). Por lo tanto se ha sugerido que el hemo controla la síntesis, transporte y ensamblado de proteínas mitocondriales de origen nuclear que están involucradas en respiración (Lombardo et al., 2005). La cuantificación de los niveles de hemo en las líneas deficientes en la expresión de AtFH reveló una disminución en la concentración del mismo de aproximadamente un 60% (Figura 41). 132 Resultados y discusión Figura 41. Determinación del contenido de hemo en plantas wt, atfh-1 y as-AtFH. El contenido de hemo se determinó cuantitativamente a partir de la extracción con cloroformo acidificado en extractos de hojas de plantas de Arabidopsis. Estos resultados son consistentes con otros trabajos en los que se ha informado que la deficiencia de hemo mitocondrial puede ser una consecuencia de la deficiencia de frataxina (Lesuisse et al., 2003; Yoon and Cowan, 2004; Schoenfeld et al., 2005), que ocasionaría la disminución de la actividad de enzimas ISC en la mitocondria. Cabe aclarar que dos de las enzimas que intervienen en la síntesis del hemo en mamíferos, ferroquelatasa y adrenodoxina, requieren cofactores ISC para su actividad (Lu and Cortopassi, 2007). En mamíferos, se describió que la síntesis de hemo y la actividad ferroquelatasa se encuentran claramente asociadas a mitocondrias (Lange et al., 1999). En contraste, la síntesis de hemo y la localización de la ferroquelatasa en mitocondrias de plantas están bastante discutidas. Algunos trabajos informaron de la presencia de actividad de esta enzima en mitocondrias de plantas (Porra and Jones, 1963; Little and Jones, 1976) y de la existencia de una actividad biosintética de hemo en estas organelas en arvejas (Cornah et al., 2002) y tabaco (Papenbrock et al., 2001). Se demostró por experimentos de importe de proteínas a organelas, que la ferroquelatasa I de Arabidopsis es importada a mitocondrias y cloroplastos, mientras que la ferroquelatasa II sólo tendría localización cloroplástica (Smith et al., 1994; Chow et al., 1998). Sin embargo experimentos de localización por fusión a GFP (green fluorescent protein) mostraron que ambas ferroquelatasas de Arabidopsis 133 Resultados y discusión tendrían localización plastídica (Masuda et al., 2003). A partir de estos datos se sugirió la existencia de otra proteína responsable de la producción de hemo mitocondrial. Como hemos descripto, la localización de las enzimas involucradas en la síntesis de tetrapirroles en plantas está siendo investigada y es un área de debate que ayudará a explicar la vía de biosíntesis de hemo en plantas. 8.1. 2. Efecto de la deficiencia de AtFH sobre la actividad catalasa. A partir de los datos donde se encontraron menores niveles de protoporfirina IX en mutantes de levadura ǻyfh y deficiencias en la síntesis de hemo en mutantes en los genes de la vía de biosíntesis de ISC (Lesuisse et al., 2003; Lange et al., 2004), decidimos investigar el papel de AtFH en la biogénesis de catalasa, una proteína con grupo hemo. La catalasa, como ya hemos mencionado en la introducción, es un tetrámero que contiene grupo hemo, está localizada principalmente en peroxisomas y cataliza la dismutación del H2O2 a H2O y O2. La familia multigénica de catalasas en Arabidopsis, está representada por 3 genes codificantes de subunidades individuales, que se asocian para formar al menos seis isoenzimas (Zhong and McClung, 1996; Michael and McClung, 2002). Las isoenzimas y los ARNm muestran patrones de expresión diferentes (órgano específico). Los tres ARNm se expresan en hojas de plantas wt de acuerdo a lo informado por Frugoli y colaboradores (1996), siendo la isoforma CAT2 (citosólicas y/o peroxisoma/glioxisoma) la más abundante, mientras que CAT1 (citosólicas y/o peroxisoma/glioxisoma) y CAT3 (mitocondrial) lo hacen con niveles de expresión inferiores (Frugoli et al., 1996). A fin de evaluar el efecto de la deficiencia de AtFH sobre la actividad de esta hemoproteína, se midió actividad catalasa total en hojas y flores de plantas wt, mutantes atfh-1 y transgénicas as-AtFH. En ambas líneas deficientes se observó un 134 Resultados y discusión descenso del 20% en la actividad catalasa en extractos de hojas, mientras que en los extractos de flor se observó una reducción de un 14% para la línea atfh-1 y un 40 % para la línea as-AtFH. (Figura 42). (A) (B) Figura 42. Actividad específica de catalasa en hojas de roseta (A) y flores (B) de Arabidopsis thaliana wt, atfh-1 y as-AtFH. Una unidad (U) se define como la cantidad de enzima que cataliza el consumo de 1 ȝmol min -1 de H2O2 A partir de estos resultados, decidimos analizar los niveles de transcripto de cada uno de los genes que codifican las catalasas de Arabidopsis mediante RT-PCR. Así, medimos la expresión de CAT1, CAT2 y CAT3 en hojas y flores de la línea wt y las plantas deficientes en AtFH. Los resultados no mostraron modificaciones significativas en los niveles de expresión del ARNm para cada isoforma, en ambos tejidos analizados (Figura 43A y B). Por lo tanto, los datos indicarían que AtFH tendría algún papel en el control de la actividad catalasa, y que posiblemente este control no ocurra a nivel transcripcional, sino a nivel de la proteína. 135 Resultados y discusión (A) Hoja 1 2 Flor 3 1 2 3 CAT1 CAT2 CAT3 ȕ- ACTINA 1,2 1,2 1 1 Expresión relativa Expresión relativa (B) 0,8 0,6 0,4 0,2 0,8 0,6 0,4 0,2 0 0 CAT1 CAT2 CAT2 CAT1 CAT2 CAT3 Figura 43: (A) Análisis por RT-PCR de la expresión de CAT1 (At1g20630), CAT2 (At4g35090) y CAT3 (At1g20620), en plantas wt (calle1), atfh-1 (calle 2) y as- AtFH (calle 3). Se utilizó como molde ADNc proveniente de hojas de roseta y flores. La expresión del gen ȕ-ACTINA fue usada como control interno. (B) Análisis de la expresión relativa de los genes mostrados en (A) por densitometría utilizando el programa Gel-Pro Analyzer (gráfico de la derecha hoja e izquierda flor). Barras blancas: wt, barras grises: atfh-1, barras negras as-AtFH. Para determinar si las variaciones observadas en la actividad catalasa total se deben a diferencias en el contenido de proteína, realizamos un análisis por western blot utilizando anticuerpos anti-catalasa (cedidos por el Profesor Mikio Nishimura, Department of Cell Biology, National Institute for Basic biology, Okazaki, Japan). Los ensayos se llevaron a cabo en hojas y flores de plantas wt, atfh-1 y as-AtFH. Los datos no mostraron diferencias significativas en el contenido proteico de cada una de 136 Resultados y discusión las líneas ensayadas (Figura 44). Así, los resultados sugieren que no habría diferencias en el contenido total de proteína catalasa ante la deficiencia de AtFH. Hoja wt atfh-1 as-AtFH Flor wt atfh-1 as-AtFH Figura 44. Análisis por western-blot revelado con anticuerpos anti-catalasa de A. thaliana. Se sembró 100 ȝg de proteína en cada calle a partir de extractos de hoja y flor. La flecha señala la banda correspondiente a la masa molecular de 55 kDa. Con el objetivo de confirmar que la disminución en la actividad catalasa es debida a una deficiencia del grupo hemo, se suplementaron suspensiones de mitocondrias aisladas con 0.025 μmoles de hemina y se determinó nuevamente la actividad de esta enzima. La Figura 45 muestra que la actividad catalasa en mitocondrias de plantas atfh-1 y as-AtFH se recupera con el agregado de hemina exógena (Figura 45). Los resultados indican que la actividad catalasa en plantas atfh1 y as-AtFH disminuye aproximadamente un 40% y 51%, respectivamente, mientras que luego del agregado de hemina, la actividad catalasa total se recupera a valores cercanos a los mostrados por la línea wt. 137 Resultados y discusión Figura 45. Actividad catalasa en mitocondrias aisladas de plantas wt, atfh-1 y as-AtFH en ausencia (barras blancas) y presencia (barras negras) de 50 μM de hemina. Una unidad (U) se define como la cantidad de enzima que cataliza el consumo de 1 ȝmol min -1 de H2O2. Estos resultados sugieren que la disminución en la actividad catalasa se debe a la ausencia de un grupo hemo funcional en las plantas deficientes en AtFH. Asimismo, nuestros datos permiten pensar en una alteración de la actividad de la catalasa 3, que es la isoforma que tiene localización mitocondrial. Por ello, nuestros datos permiten avalar la hipótesis que AtFH tendría un papel importante en la producción del grupo hemo interviniendo así en la biogénesis de hemoproteínas como la catalasa. Estos resultados coinciden con lo postulado previamente a partir de estudios en levaduras, donde se sugirió que la frataxina mantendría el Fe biodisponible para su utilización en la síntesis de grupos Fe-S y hemo en el interior celular (Lesuisse et al., 2003). Recientemente se informó que el agregado exógeno de hemina, rescató el fenotipo de deficiencia de frataxina en células neuronales incrementando la actividad de algunas Fe-S proteínas y de la citocromo oxidasa (hemoproteína) (Napoli et al., 2007). Esto además está de acuerdo con reportes recientes donde se informó que la eritropoyetina, que es sabido estimula la síntesis de hemo, puede recuperar el fenotipo observado en células deficientes en frataxina (Sturm et al., 2005). 138 Resultados y discusión 8.1.3. Efecto de la deficiencia de AtFH en enzimas claves de la síntesis del hemo Mediante el análisis de microarreglos de ADNc de linfoblastos humanos derivados de pacientes con FRDA y ratones mutantes en frataxina se informó una deficiencia en los niveles de ARNm de la enzima coproporfirinógeno oxidasa (CPOX). Además se detectó un aumento en los niveles de protoporfirina IX, indicando la existencia de deficiencias en la síntesis del grupo hemo (Schoenfeld et al., 2005). Posteriormente, se sugirió que la frataxina podría afectar la especificidad de la ferroquelatasa hacia los iones hierro o zinc, alterando la incorporación de los mismos dentro de la protoporfirina IX (Lesuisse et al., 2003; Schoenfeld et al., 2005). El aumento de la actividad Zn- quelatasa en células deficientes en frataxina, sugieren que una posible función para esta última podría ser la de ceder hierro hacia la ferroquelatasa, y que bajo las condiciones de deficiencia de frataxina, la ferroquelatasa se vuelve menos selectiva hacia la incorporación de hierro dentro de la protoporfirina IX (Schoenfeld et al., 2005). Otros estudios han demostrado por análisis de QRT PCR la disminución significativa de los trascriptos de ALAS1 (Ala dehydratasa), CPOX (coproprfirinógeno oxidasa) y ferroquelatasa en células de miocardio de ratas deficientes en frataxina (Puccio et al., 2001). Para comprender mejor el efecto de la deficiencia de AtFH sobe la vía de biosíntesis del hemo, evaluamos los niveles de transcriptos de varias enzimas claves de esta vía metabólica. En primer lugar, investigamos los niveles de ARNm de los dos genes que codifican para las ferroquelatasas de Arabidopsis: ferroquelatasa I (AtFeCh1, At5g26030) y ferroquelatsa II (AtFeCh2, At2g30390). AtFeCh1, se expresa en todos los tejidos de la planta, sin embargo su mayor expresión se encuentra en raíces y flores y aumenta notablemente en presencia de sacarosa, daño del tejido y estrés oxidativo (Pal Singh et al., 2002). Por otro lado, AtFeCh2 se expresa en toda la parte aérea de la planta, en mucho mayor medida en flores y no se expresa en raíces. Su expresión se ve reducida notablemente en tejidos dañados y en estrés oxidativo (Pal Singh et al., 2002). 139 Resultados y discusión Los niveles de AtFeCh1 y 2 se encontraron inducidos alrededor de 1.5 a 2 veces en las hojas de plantas atfh-1 y as-AtFH (Figura 46). En contraste, el ARNm de AtFeCh1 no mostró variaciones significativas en flores de ambas líneas deficientes, mientras que se observó una disminución de alrededor de 25% y 50% en flores de las plantas atfh-1 y as-AtFH, respectivamente (Figura 46). 7 Expresión relativa 6 5 4 3 2 1 0 wt atfh-1 as-AtFH AtFeCh1 wt atfh-1 AtFeCh2 as-AtFH wt atfh-1 as-AtFH AtFeCh1 Hoja wt atfh-1 as-AtFH AtFeCh2 Flor Figura 46: Análisis por Q-PCR de la expresión de genes implicados en la síntesis del hemo: ferroquelatasa I (AtFeCh1, At5g26030) y ferroquelatsa II (AtFeCh2, At2g30390) en hojas y flores de plantas wt (barras blancas), atfh-1 (barras grises) y as-atfh (barras negras). El ARN total fue retrotranscripto y amplificado usando primers específicos. Los datos se normalizaron a los niveles de expresión de AtFeCh1 y AtFeCh2 en hojas de plantas wt. Los resultados indican diferencias a nivel de la transcripción de los genes AtFeCh1 y 2 en los órganos estudiados ante la deficiencia de AtFH. Esto puede deberse a diferencias a nivel de la regulación transcripcional de estos dos genes, que podrían responder a factores tejido u órgano específicos. Por otro lado, también se midieron los niveles de transcripto para otras enzimas que participan en la síntesis de hemo, porfobilinógeno sintasa-I (AtHemB1, At1g69740) y porfobilinógeno sintasa-II (AtHemB2, At1g44318). Esta enzima cataliza la síntesis de porfobilinógeno mediante la condensación asimétrica de dos moléculas de ácido aminolevulínico. Todos los tetrapirroles incluído el hemo, 140 Resultados y discusión clorofilas y vitamina B12, comparten como precursor común el porfobilinógeno (Tanaka and Tanaka, 2007). La Figura 47 muestra los resultados del análisis de la expresión de AtHemB1 y AtHemB2. En hojas, se encontró una disminución de alrededor del 30% y del 70% en los niveles de AtHemB1, en plantas atfh-1 y as-AtFH, respectivamente. De forma similar, los niveles de AtHemB2 disminuyeron alrededor de 60% en ambas líneas deficientes en AtFH. En contraste, no se observaron diferencias significativas en la cantidad de AtHemB1 en las flores de las diferentes líneas, mientras que los niveles de AtHemB2 se incrementaron 4 y 12 veces en las plantas atfh-1 y as-AtFH, respectivamente (Figura 47). 5 4,5 Expresión relativa 4 3,5 3 2,5 2 1,5 1 0,5 0 wt atfh-1 as-AtFH AtHemB1 wt atfh-1 AtHemB2 as-AtFH wt atfh-1 as-AtFH AtHemB1 Hoja wt atfh-1 as-AtFH AtHemB2 Flor Figura 47: Análisis por Q-PCR de Porfobilinogeno sintasa-I (AtHemB1, At1g69740), y Porfobilinogeno sintasa-II (AtHemB2, At1g44318) en plantas wt (barras blancas), atfh-1(barras grises) y as-AtFH (barras negras) tanto en hojas como en flor. El RNA total fue retrotranscripto y amplificado usando primers específicos. Los datos se normalizaron a los niveles de expresión de AtHemB1 y AtHemB2 en hojas de plantas wt. Los resultados reflejan la alteración de la expresión de los dos genes que codifican para AtHemB e indican que esta alteración es tejido específica, de forma similar a lo que ocurre con los genes que codifican para AtFeCh. 141 Resultados y discusión Finalmente, decidimos investigar otro de los genes claves en la biosíntesis del hemo, el de la coproporfirinógeno oxidasa (AtHemF2, At4g03205). Esta enzima ha sido identificada como uno de los pasos regulatorio de la vía (Woodarda and Dailey, 2000). Utilizando PCR en tiempo real, se observó una disminución de alrededor del 60% y 80% en los niveles de transcripto AtHemF2 en hojas de plantas atfh-1 y as-AtFH, respectivamente. En contraste, en órganos florales se encontró una leve disminución en los niveles de AtHemF2 en plantas atfh-1, mientras que no se encontraron diferencias significativas en los niveles de este transcripto en las plantas as-AtFH cuando se comparó con las plantas wt (Figura 48). 3,5 Expresión relativa 3 2,5 2 1,5 1 0,5 0 wt atfh-1 as-AtFH wt atfh-1 AtHemF2 AtHemF2 Hojas Flores as-AtFH Figura 48: Coproporfirinógeno oxidasa (AtHemF2, At4g03205.2) en plantas wt(barras blancas), atfh1(barras grises) y as-atfh (barras negras), tanto en hojas (A) como en flor (B). El RNA total fue retrotranscripto y amplificado usando primers específicos. La expresión relativa igual a 1 corresponde al nivel de transcripción de AtHemF2 en hojas de plantas wt. En conjunto, los resultados presentados muestran que la deficiencia de AtFH en Arabidopsis conduce a una alteración en varios transcriptos de la vía de síntesis de hemo. Esto sugeriría que al menos en hoja, la acumulación de este compuesto podría generar una retroinhibición hacia la coproporfirinógeno oxidasa reduciendo de esta manera los niveles de transcripción de AtHemF2 como se observa en la Figura 48. Existiría entonces una vía de síntesis de hemo mitocondrial? Una posibilidad es que la distribución dual o no de las ferroquelatasas sea diferente entre las distintas 142 Resultados y discusión especies de plantas (Papenbrock et al., 2001, Masuda et al., 2003). Otra posibilidad es la existencia de otra enzima mitocondrial que catalize la inserción de los metales divalentes en la protoporfirina IX. Es sabido que dicha inserción puede llevarse a cabo por reacciones tanto enzimáticas como no enzimáticas. Por ejemplo, los iones ferrosos pueden ser insertados de manera no enzimática en la porfirina en presencia de tiol reductores y ácidos grasos, pero estas reacciones no ocurrirían in vivo (Taketani and Tokunaga, 1984). Si asumimos la presencia de otra proteína mitocondrial, con baja afinidad por los metales como la ferroquelatasa (Papenbrock et al., 2001; Cornah et al., 2002), es posible que tal enzima sea la responsable de la menor producción de hemo en la mitocondria. En este caso, la secuencia nucleotídica y aminoacídica de esta proteína podría ser significativamente diferente de la ferroquelatasas conocidas (Masuda et al., 2003). Como se mencionó en párrafos precedentes, si bien hay discrepancias sobre la localización de algunas de las enzimas de la vía de síntesis del hemo en plantas, nuestros datos nos permiten postular que la deficiencia de AtFH mitocondrial provocaría la alteración de la transcripción de genes del metabolismo hemo, lo que concuerda con los menores niveles de hemo encontrados en las plantas atfh-1 y asAtFH. La deficiencia de hemo funcional, alteraría también la actividad de algunas hemoproteínas como la catalasa, hecho reflejado en la menor actividad catalasa total que encontramos en diferentes tejidos como hojas y flores de las plantas atfh-1 y asAtFH, como así también en extractos de mitocondrias aisladas. En conjunto, nuestros resultados permiten postular que AtFH tendría un rol importante en la biogénesis de hemoproteínas dentro de la célula. 143 Conclusiones Conclusiones Conclusiones x La caracterización fenotípica de las plantas mutantes insercionales atfh-1, atfh-2, atfh-3 y transgénicas as-AtFH y s-AtFH, reveló que frataxina es una proteína esencial en plantas. A partir de los resultados expuestos podemos inferir que AtFH sería indispensable en procesos de alto requerimiento energético como por ejemplo la embriogénesis, la floración y la fructificación, avalando una de las posibles funciones postuladas para ella, que es su participación en el metabolismo energético mitocondrial. x Con el objeto de caracterizar la proteína a nivel bioquímico, se clonaron, expresaron y purificaron las proteínas AtFH-His6 y AtFH-STOP de Arabidopsis thaliana. Los estudios funcionales de ambas proteínas recombinantes mostraron que tienen la capacidad de atenuar la reacción de Fenton in vitro. Los resultados nos permiten concluír que AtFH es una proteína funcional y que estaría involucrada en la homeostasis del Fe a través de la protección contra el daño oxidativo causado por este metal y que tendría un papel importante en el mantenimiento del estado redox celular. x La deficiencia de AtFH induce un aumento de los niveles de ROS tanto en hojas como en raíz, generando modificaciones en la expresión de genes vinculados con detoxificación y respuesta a estrés oxidativo. Este resultado está de acuerdo con el aumento en la concentración del Fe presente en estas plantas, con capacidad para catalizar la reacción de Fenton y así aumentar la producción de ROS. Por otro lado, hemos visto un aumento en la expresión de AtFH luego del tratamiento con H2O2. En conjunto, estos datos nos permiten confirmar que AtFH está comprometida en la respuesta a estrés oxidativo en organismos vegetales. x La disminución en la concentración de ATP, junto con un descenso en el consumo de O2 de las plantas atfh-1 y as-AtFH indican que la deficiencia de frataxina lleva a una disfunción mitocondrial, acorde al papel propuesto para AtFH en el metabolismo energético celular y la fosforilación oxidativa. 145 Conclusiones Conjuntamente, se observó una inducción en la expresión de dos genes nucleares, PSST y NADHbp y del gen mitocondrial Nad9, que codifican para subunidades Fe-S del CI respiratorio. Ésto sugeriría que existe una compensación nuclear y una adaptación mitocondrial, respectivamente, en respuesta a la disfunción de la organela. Si bien los genes mencionados mostraron una inducción ante la deficiencia de AtFH y no se observaron cambios en la cantidad de proteína NAD9, se encontró que la actividad del CI mitocondrial en las plantas AtFH deficientes estaba disminuída. Por ésto, frataxina sería una proteína indispensable, posiblemente involucrada en el correcto ensamblaje de los grupos Fe-S a apoproteínas o tendría un papel relevante en la reparación de los grupos Fe-S dañados. Los resultados indican que AtFH cumple un papel importante en el correcto funcionamiento de la cadena de transporte electrónico mitocondrial. x En las líneas mutantes para AtFH se observaron bajos niveles de actividad de las proteínas Fe-S mitocondriales SDH y aconitasa, y altos niveles en la expresión de los ARNm que codifican para SDH2-1, SDH2-2 y ACO, mientras que no se encontró alterada la actividad de proteínas sin grupos Fe-S como la MDH. Estos resultados indican que AtFH estaría involucrada en la modulación de la actividad y biogénesis de algunas ferrosulfoproteínas mitocondriales como el CII y la aconitasa. x Si bien en plantas existen discrepancias sobre la localización de algunas de las enzimas de la vía de síntesis del hemo, nuestros datos nos permiten postular que la deficiencia de AtFH mitocondrial provocaría la alteración de la transcripción de genes de la biosíntesis de hemo, lo que concuerda con los menores niveles de este último encontrados en las plantas atfh-1 y as-AtFH. La deficiencia de hemo alteraría también la función de algunas hemoproteínas como la catalasa, hecho reflejado en los menores niveles de actividad de esta enzima medidos en hojas y flores, como así también en extractos de mitocondrias aisladas de las plantas atfh1 y as-AtFH. En conjunto, nuestros resultados permiten postular que AtFH tendría un papel importante en la biogénesis del grupo hemo y de hemoproteínas dentro de la célula vegetal. 146 Resumen Resumen Resumen Dos procesos fundamentales que llevan a cabo los organismos vegetales son la fotosíntesis y la respiración. Dichos procesos requieren la participación de diferentes proteínas y complejos multiproteicos, muchos de ellos formados por ferrosulfoproteínas y hemoproteínas. La maduración de ferro-sulfoproteínas es una de las funciones esenciales de la mitocondria, ya que las proteínas Fe-S constituyen una familia de moléculas involucradas en varias funciones celulares de gran importancia. Son constituyentes de los complejos respiratorios mitocondriales CI, CII y CIII y también están presentes en la matriz mitocondrial (aconitasa). En los últimos años se intensificó el estudio del ensamblaje de los centros Fe-S y su inserción en polipéptidos para producir proteínas funcionales. Sin embargo, existe muy poca información sobre la ocurrencia de estas vías en organismos vegetales. A partir de estudios en células de mamíferos y levaduras, se postuló la participación de frataxina en el ensamblado de centros Fe-S, metabolismo energético mitocondrial, respiración y homeostasis del hierro. Frataxina es una proteína mitocondrial y está altamente conservada desde bacterias hasta mamíferos y plantas, sin presentar mayores cambios estructurales, especialmente en el dominio Cterminal. Esto sugiere que esta proteína cumpliría funciones similares en todos estos organismos. El objetivo de este trabajo de Tesis es la caracterización de proteínas que participarían en la biogénesis de proteínas Fe-S y hemoproteínas mitocondriales. El estudio está enfocado a investigar la función del homólogo de frataxina de Arabidopsis thaliana (AtFH). El análisis fenotípico y molecular de plantas deficientes en AtFH reveló que esta proteína es esencial en plantas y está involucrada en diferentes procesos de alto requerimiento energético como son la embriogénesis, la floración y la fructificación. La deficiencia de AtFH conduce a una disfunción mitocondrial asociada con una menor tasa respiratoria y la disminución del contenido de ATP celular. Por otro lado, 148 Resumen AtFH es requerida para la actividad completa de proteínas Fe-S mitocondriales como el CI, CII y la aconitasa, sugiriendo que esta proteína está involucrada en la respiración mitocondrial y la fosforilación oxidativa. Estudios bioquímicos realizados con la proteína AtFH recombinante in vitro, sugirieron su participación en la protección contra daño oxidativo, homeostasis del Fe y mantenimiento del estado redox celular. Estos datos están de acuerdo con el aumento de especies reactivas de oxígeno observado en las plantas deficientes en AtFH. Además, las plantas deficientes en AtFH mostraron una alteración en la transcripción de ARNm de genes involucrados en la biosíntesis de hemo, como así también menores niveles de hemo total. Estos datos confluyen con los menores niveles de actividad catalasa encontrados en las plantas con deficiencia en AtFH. En conjunto, nuestros resultados nos permiten sugerir que AtFH está involucrada además en la biosíntesis del grupo hemo y la biogénesis de hemoproteínas en plantas. 149 Referencias Referencias Referencias 1) 2) 3) 4) 5) 6) 7) 8) 9) 10) 11) 12) 13) 14) 15) 16) 17) 18) 19) 20) 21) 22) 23) 24) 25) 26) 27) 28) 29) Ackrell, B. (2000). Progress in understanding structure-function relationships in respiratory chain complex II. FEBS Letters 466, 1-5. Aebi H. (1984). Catalase in vitro. Methods in Enzymology. 105, 121-126. Alonso, J., Stepanova, A., and (2003). T-DNA mutagenesis in Arabidopsis. Methods Mol Biol. 236, 177188.Babcock, M., de Silva, D., Oaks, R., Davis-Kaplan, S., Jiralerspong, S., Montermini, L., Pandolfo, M., and Kaplan, J. (1997). Regulation of Mitochondrial Iron Accumulation by Yfh1p, a Putative Homolog of Frataxin. Science 276, 1709-1712. Andrade, S.L.A., Brondino, C.D., Feio, M.J., Moura, I., and Moura, J.J.G. (2000). Aldehyde oxidoreductase activity in Desulfovibrio alaskensis NCIMB 13491 EPR assignment of the proximal [2Fe2S] cluster to the Mo site. European Journal of Biochemistry 267, 2054-2061. Andreyev, A., Kushnareva, Y., and Starkov, A. (2005). Mitochondrial metabolism of reactive oxygen species. Biochemistry (Mosc) 70, 200-214. Arimura, S.-i., and Tsutsumi, N. (2002). A dynamin-like protein (ADL2b), rather than FtsZ, is involved in Arabidopsis mitochondrial division. Proc Natl Acad Sci USA 99, 5727-5731. Arimura, S.-i., Yamamoto, J., Aida, G.P., Nakazono, M., and Tsutsum, N. (2004). Frequent fusion and fission of plant mitochondria with unequal nucleoid distribution. Proc Natl Acad Sci USA 101, 7805-7808. Atamna, H., Killilea, D., Killilea, A., and Ames, B. (2002). Heme deficiency may be a factor in the mitochondrial and neuronal decay of aging.. Proc Natl Acad Sci USA 99, 14807-14812. Babcock, M., de Silva, D., Oaks, R., Davis-Kaplan, S., Jiralerspong, S., Montermini, L., Pandolfo, M., and Kaplan, J. (1997). Regulation of Mitochondrial Iron Accumulation by Yfh1p, a Putative Homolog of Frataxin. Science 276, 1709-1712. Baier, M., and Dietz, K.J. (1996). Primary structure and expression of plant homologues of animal and fungal thioredoxin-dependent peroxide reductases and bacterial alkyl hydroperoxide reductases. Plant Mol Biol 31, 553-564. Baier, M., and Dietz, K.J. (1997). The plant 2-Cys peroxiredoxin BAS1 is a nuclear-encoded chloroplast protein: its expressional regulation, phylogenetic origin, and implications for its specific physiological function in plants. The Plant Journal 12, 179-190. Baier, M., and Dietz, K. (2005). Chloroplasts as source and target of cellular redox regulation: a discussion on chloroplast redox signals in the context of plant physiology. J Exp Bot 56, 1449-1462. Balaban, R.S., Nemoto, S., and Finkel, T. (2005). Mitochondria, oxidants, and aging. . Cell. 120483-495 Balk, J., and Lill, R. (2004). The Cell's Cookbook For Iron-Sulfur Clusters: Recipes For Fool's Gold? Chem BioChem 5, 1044-1049. Balk, J., and Lobréaux, S. (2005). Biogenesis of iron-sulfur proteins in plants. Trends in Plant Science 10, 324-331. Bannister, W., Bannister, J., Barra, D., Bond, J., and Bossa, F. (1991). Evolutionary aspects of superoxide dismutase: the copper/zinc enzyme. Free Radic Res Commun. 12-13, 349-361. Batlle, A. (1997). Biosíntesis del hemo en Porfirias y Porfirinas. Aspectos Clínicos, bioquímicos y biología molecular. Acta Bioquímica clínica. Latinoamericana 3, 145-171 Baulcombe, D. (2007). Molecular biology. Amplified silencing. Science 315, 199-200. Becker E, R.D. (2001). Frataxin: its role in iron metabolism and the pathogenesis of Friedreich's ataxia. Int J Biochem Cell Biol 33, 1-10. Becker, L., vanden Hoek, T., Shao, Z., Li, C., Schumacker, P., and (1999). Generation of superoxide in cardiomyocytes during ischemia before reperfusion. Am J Physiol. 277, 2240-2246. Becker, E.M., Greer, J.M., Ponka, P., and Richardson, D.R. (2002). Erythroid differentiation and protoporphyrin IX down-regulate frataxin expression in Friend cells: characterization of frataxin expression compared to molecules involved in iron metabolism and hemoglobinization. Blood 99, 3813-3822. Beech, P., and Gilson, P. (2000). FtsZ and organelle division in Protists. Protist 151, 11-16. Beech, P., Nheu, T., Schultz, T., Herbert, S., Lithgow, T., Gilson, P., and McFadden, G. (2000). Mitochondrial FtsZ in a chromophyte alga. Science 287, 1276-1279. Beinert, H. (2000). A tribute to sulfur. Eur J Biochem 267, 5657-5664. Beinert, H., Holm, R.H., and Munck, E. (1997). Iron-Sulfur Clusters: Nature's Modular, Multipurpose Structures. Science 277, 653-659. Beinert, H., Kennedy, M., and Stout, C. (1996). Aconitase as Iron minus sign Sulfur Protein, Enzyme, and Iron-Regulatory Protein. Chem Rev. 96, 2335-2374. Beyer, W.F. and Fridovich, I. (1987) Assaying for superoxide dismutase activity: some large consequences of minor changes in conditions. Anal Biochem 161, 559-566. Bidichandani, S., Purandare, S., Taylor, E., Gumin, G., Machkhas, H., Harati, Y., Gibbs, R., Ashizawa, T. and Patel, P. (1999). Somatic sequence variation at the Friedreich ataxia locus includes complete contraction of the expanded GAA triplet repeat, significant length variation in serially passaged lymphoblasts and enhanced mutagenesis in the flanking sequence. Hum Mol Genet. 8, 2425-2436. Binder, S., and Brennicke, A. (2003). Gene expression in plant mitochondria: transcriptional and posttranscriptional control.. Philos Trans R Soc Lond B Biol Sci 358, 181-189. 151 Referencias 30) Bolender, N., Sickmann, A., Wagner, R., Meisinger, C., and Pfanner, N. (2008). Multiple pathways for sorting mitochondrial precursor proteins. EMBO Rep. 9, 42-49. 31) Bouchez D, H.H. (1998). Functional genomics in plants. Plant Physiol. 118, 725-732. 32) Boutry, M., Nagy, F., Poulsen, C., Aoyagi, K., and Chua, N. (1987). Targeting of bacterial chloramphenicol acetyltransferase to mitochondria in transgenic plants. Nature 328, 340-342. 33) Branda, S., Cavadini, P., Adamec, J., Kalousek, F., Taroni, F., and Isaya, G. (1999). Yeast and human frataxin are processed to mature form in two sequential steps by the mitochondrial processing peptidase. J Biol Chem. 274, 22763-22769. 34) Bradley, J., Blake, J., Chamberlain, S., Thomas, P., Cooper, J., and Schapira, A. ( 2000). Clinical, biochemical and molecular genetic correlations in Friedreich's ataxia. Hum Mol Genet 9, 275-282. 35) Brandt, U. (2006). Energy converting NADH:quinine oxidoreductase (complex I). Annu. Rev. Biochem 75, 69-92. 36) Brangeon, J., Sabar, M., Gutierres, S., Combettes, B., Bove , J., Gendy, C., Chetrit, P., Des FrancsSmall, C.C., Pla, M., Vedel, F., and De Paepe, R. (2000). Defective splicing of the first Nad4 intron is associated with lack of several complex I subunits in the Nicotiana sylvestris NMS1 nuclear mutant. Plant J. 21, 269-280. 37) Braun, H.-P., and Schmitz, U.K. (1992). Affinity purification of cytochrome reductase from potato mitochondria. European Journal of Biochemistry 208, 761-767. 38) Brennicke, A., Zabaleta, E., Dombrowski, S., Hoffmann, M., and Binder, S. (1999). Transcription signals of mitochondrial and nuclear genes for mitochondrial proteins in dicot plants.. J Hered 90, 345-350. 39) Briat, J.-F., Lobréaux, S., Grignon, N., and Vansuyt, G. (1999). Regulation of plant ferritin synthesis: how and why? . Cell Mol Life Sci 56, 155-166. 40) Brodersen, P., and Voinnet, O. (2006 ). The diversity of RNA silencing pathways in plants. Trends Genet 22, 268-280. 41) Bulteau, A.-L., O'Neill, H.A., Kennedy, M.C., Ikeda-Saito, M., Isaya, G., and Szweda, L.I. (2004). Frataxin Acts as an Iron Chaperone Protein to Modulate Mitochondrial Aconitase Activity. Science 305, 242-245. 42) Busi, M., Zabaleta, E., Araya, A., and Gomez-Casati, D. (2004). Functional and molecular characterization of the frataxin homolog from Arabidopsis thaliana. FEBS Letters 576, 141-144. 43) Busi, M., Gómez-Casati, D., Perales, M., Araya, A., and Zabaleta, E. (2006). Nuclear-encoded mitochondrial complex I gene expression is restored to normal levels by inhibition of unedited ATP9 transgene expression in Arabidopsis thaliana. Plant Physiol Biochem 44, 1-6. 44) Burnette, W. (1981). Western Blotting: Electrophoretic transfer of proteins from SDS-polyacrylamide gels to unmodified nitrocellulose and radiographic detection with antibody and radioiodinated protein A. Anal. Biochem 112, 195-203. 45) Campuzano, V., Montermini, L., Lutz, Y., Cova, L., Hindelang, C., Jiralerspong, S., Trottier, Y., Kish, S.J., Faucheux, B., Trouillas, P., Authier, F.J., Durr, A., Mandel, J.L., Vescovi, A., Pandolfo, M., and Koenig, M. (1997). Frataxin is reduced in Friedreich ataxia patients and is associated with mitochondrial membranes. Hum. Mol. Genet. 6, 1771-1780. 46) Campuzano V, M.L., Moltò MD, Pianese L, Cossée M, Cavalcanti F, Monros E, Rodius F, Duclos F, Monticelli A, Zara F, Cañizares J, Koutnikova H, Bidichandani SI, Gellera C, Brice A, Trouillas P, De Michele G, Filla A, De Frutos R, Palau F, Patel PI, Di Donato S, Mandel JL, Cocozza S, Koenig M, Pandolfo M. (1996). Friedreich's ataxia: autosomal recessive disease caused by an intronic GAA triplet repeat expansion. Science 271, 1374-1375. 47) Cannino, G., Di Liegro, C., and Rinaldi, A. (2007 ). Nuclear-mitochondrial interaction. Mitochondrion 7, 359-366. 48) Cañizares, J., Blanca, J.M., Navarro, J.A., Monrós, E., Palau, F., and Moltó, M.D. (2000). dfh is a Drosophila homolog of the Friedreich's ataxia disease gene Gene 256, 35-42. 49) Carraway, M.S., Suliman, H.B., Madden, M.C., Piantadosi, C.A., and Ghio, A.J. (2006). Metabolic capacity regulates iron homeostasis in endothelial cells. Free Radical Biology & Medicine 41, 1662-1669. 50) Cavadini, P., Adamec, J., Taroni, F., Gakh, O., and Isaya, G. (2000). Two-step Processing of Human Frataxin by Mitochondrial Processing Peptidase. PRECURSOR AND INTERMEDIATE FORMS ARE CLEAVED AT DIFFERENT RATES. J. Biol. Chem. 275, 41469-41475. 51) Cavalcanti, F., Tammaro, A., De Michele, G., Filla, A., Cocozza, S., Labuda, M., Montermini, L., Poirier, J., and Pandolfo, M. (1999). Friedreich's ataxia: point mutations and clinical presentation of compound heterozygotes. Annual Neurology 45, 200-206. 52) Chae, H.Z., Chung, S.J., and Rhee, S.G. (1994). Thioredoxin-dependent peroxide reductase from yeast. J. Biol. Chem. 269, 27670-27678. 53) Chance, B., Sies, H., and Boveris, A. (1979). Hydroperoxide metabolism in mammalian organs. Physiol. Rev. 59, 527-605. 54) Chantrel-Groussard, K., Geromel, V., Puccio, H., Koenig, M., Munnich, A., Rötig, A., and Rustin, P. (2001). Disabled early recruitment of antioxidant defenses in Friedreich's ataxia. Hum Mol Genet 10, 20612067. 55) Chen, O., Hemenway, S., and Kaplan, J. (2002). Inhibition of Fe-S cluster biosynthesis decreases mitochondrial iron export: evidence that Yfh1p affects Fe-S cluster synthesis. Proc Natl Acad Sci USA 99, 12321-12326. 152 Referencias 56) Chen, Q., Vazquez, E., Moghaddas, S., Hoppel, C., and Lesnefsky, E. ( 2003 ). Production of reactive oxygen species by mitochondria: central role of complex III. J. Biol Chem 278, 36027-36031. 57) Chen, T., and Murata, N. (2002). Enhancement of tolerance of abiotic stress by metabolic engineering of betaines and other compatible solutes. Curr Opin Plant Biol 5, 250-257. 58) Chew, O., Rudhe, C., Glaser, E., and Whelan, J. (2003). Characterization of the targeting signal of dualtargeted pea glutathione reductase.. Plant Mol Biol. 53, 341-356. 59) Cho, S., Lee, M., Yang, J., Lee, J., Song, H., and Suh, S. (2000). Crystal structure of Escherichia coli CyaY protein reveals a previously unidentified fold for the evolutionarily conserved frataxin family. Proc Natl Acad Sci USA. 97, 8932-8937. 60) Chow, K., Singh, D., Walker, A., and Smith, A. (1998). Two different genes encode ferrochelatase in Arabidopsis: mapping, expression and subcellular targeting of the precursor proteins. Plant J. 15, 531-541. 61) Clifton, R., Millar, A., and Whelan, J. (2006). Alternative oxidases in Arabidopsis: a comparative analysis of differential expression in the gene family provides new insights into function of nonphosphorylating bypasses.. Biochim Biophys Acta. 1757, 730-741. 62) Clough SJ, B.A. (1998). Floral dip: a simplified method for Agrobacterium-mediated transformation of Arabidopsis thaliana. Plant J. 16, 735-743. 63) Connolly , E.L., and Guerinot, M.L. (2002,). Iron stress in plants. Genome Biology 3, 1024.10211024.1024. 64) Considine, M.J., Holtzapffel, R.C., Day, D.A., Whelan, J., and Millar, A.H. (2002). Molecular Distinction between Alternative Oxidase from Monocots and Dicots. Plant Physiol. 129, 949-953. 65) Cornah, J., Roper, J., Pal Singh, D., and Smith, A. (2002). Measurement of ferrochelatase activity using a novel assay suggests that plastids are the major site of haem biosynthesis in both photosynthetic and nonphotosynthetic cells of pea (Pisum sativum L.). Biochem J. 362, 423-432. 66) Cooke, R., Raynal, M., Laudié, M., Grellet, F., Delseny, M., Morris, P., Guerrier, D., Giraudat, J., Quigley, F., Clabault, G., Li, Y., Mache, R., Krivitzky, M., Gy, I., Kreis, M., Lecharny, A., Parmentier, Y., Marbach, J., Fleck, J., Clément, B., Philipps, G., Hervé, C., Bardet, C., Tremousaygue, D., and Höfte, H. (1996 ). Further progress towards a catalogue of all Arabidopsis genes: analysis of a set of 5000 non-redundant ESTs. Plant J. 9, 101-124. 67) Cossée, M., Dürr, A., Schmitt, M., Dahl, N., Trouillas, P., Allinson, P., Kostrzewa, M., NivelonChevallier, A., Gustavson, K., Kohlschütter, A., Müller, U., Mandel, J., Brice, A., Koenig, M., Cavalcanti, F., Tammaro, A., De Michele, G., Filla, A., Cocozza, S., Labuda, M., Montermini, L., Poirier, J., and Pandolfo, M. (1999). Friedreich's ataxia: point mutations and clinical presentation of compound heterozygotes. Annual Neurology 45, 200-206. 68) del Río, L., Corpas, F., Sandalio, L., Palma, J., and Barroso, J. ( 2003). Plant peroxisomes, reactive oxygen metabolism and nitric oxide. IUBMB Life 55, 71-81. 69) Dhe-Paganon, S., Shigeta, R., Chi, Y.-I., Ristow, M., and Shoelson, S.E. (2000). Crystal Structure of Human Frataxin. J. Biol. Chem. 275, 30753-30756. 70) Dietz, K.-J., Jacob, S., Oelze, M.-L., Laxa, M., Tognetti, V., Nunes de Miranda, M., Baier, M., and Finkemeier, I. (2006). The function of peroxiredoxins in plant organelle redox metabolism. Journal of Experimental Botany 57, 1697-1709. 71) Droge, W., and (2002). Free radicals in the physiological control of cell function. Physiol. Rev 82, 47-95. 72) Duarte, M., and Videira, A. (2000). Respiratory chain complex I is essential for sexual development in neurospora and binding of iron sulfur clusters are required for enzyme assembly. Genetics 156, 607-615. 73) Duarte, M., and Videira, A. (2007). Mitochondrial NAD(P)H dehydrogenases in filamentous fungi. In Complex I and Alternative NADH Dehydrogenases (González-Siso, M. I., ed). Transworld Research Network, Kerala, India., 55-68. 74) Duby, G., Foury, F., Ramazzotti, A., Herrmann, J., and Lutz, T. (2002). A non-essential function for yeast frataxin in iron-sulfur cluster assembly. Hum. Mol. Genet. 11, 2635-2643. 75) Duchêne, A., and Maréchal-Drouard, L. (2001). The chloroplast-derived trnW and trnM-e genes are not expressed in Arabidopsis mitochondria.. Biochem Biophys Res Commun. 285, 1213-1216. 76) Elhafez, D., Murcha, M., Clifton, R., Soole, K., Day, D., and Whelan, J. (2006). Characterization of mitochondrial alternative NAD(P)H dehydrogenases in Arabidopsis: intraorganelle location and expression. Plant Cell Physiol 47, 43-54. 77) Elorza, A., Leon, G., Gomez, I., Mouras, A., Holuigue, L., Araya, A., and Jordana, X. (2004). Nuclear SDH2-1 and SDH2-2 Genes, Encoding the Iron-Sulfur Subunit of Mitochondrial Complex II in Arabidopsis, Have Distinct Cell-Specific Expression Patterns and Promoter Activities. Plant Physiol. 136, 4072-4087. 78) Elorza, A., Roschzttardtz, H., Gómez, I., Mouras, A., Holuigue, L., Araya, A., and Jordana, X. (2006). A nuclear gene for the iron-sulfur subunit of mitochondrial complex II is specifically expressed during Arabidopsis seed development and germination.. Plant Cell Physiol 47, 14-21. 79) Elthon, T., and McIntosh, L. (1987). Identification of the alternative terminal oxidase of higher plant mitochondria. Proc Natl Acad Sci USA. 84, 8399-8403. 80) Eubel, H., Jansch, L., and Braun, H.-P. (2003). New Insights into the Respiratory Chain of Plant Mitochondria. Supercomplexes and a Unique Composition of Complex II. Plant Physiol. 133, 274-286. 81) Fedoroff, N., and Smith, D. (1993). A versatile system for detecting transposition in Arabidopsis. Plant J. 3, 273-289. 153 Referencias 82) Figueroa, P., Gómez, I., Holuigue, L., Araya, A., and Xavier, J. (1999). Transfer of rps14 from the mitochondrion to the nucleus in maize implied integration within a gene encoding the iron-sulphur subunit of succinate dehydrogenase and expression by alternative splicing. Plant J 18, 601-609. 83) Figueroa, P., León, G., Elorza, A., Holuigue, L., and Jordana, X. (2001). Three different genes encode the iron-sulfur subunit of succinatedehydrogenase in Arabidopsis thaliana. Plant Mol Biol 46, 241-250 84) Figueroa, P., Léon, G., Elorza, A., Holuigue, L., Araya, A., and Jordana, X. (2002). The four subunits of mitochondrial respiratory complex II are encoded by multiple nuclear genes and targeted to mitochondria in Arabidopsis thaliana.. Plant Mol Biol 50, 725-734. 85) Finel, M. (1998). Organization and evolution of structural elements within complex I. Biochim. Biophys. Acta 1364 112-121. 86) Finkemeier, I., Goodman, M., Lamkemeyer, P., Kandlbinder, A., Sweetlove, L.J., and Dietz, K.-J. (2005). The Mitochondrial Type II Peroxiredoxin F Is Essential for Redox Homeostasis and Root Growth of Arabidopsis thaliana under Stress. J. Biol. Chem. 280, 12168-12180. 87) Flatmark, T., and Romslo, I. (1975). Energy-dependent accumulation of iron by isolated rat liver mitochondria. Requirement of reducing equivalents and evidence for a unidirectional flux of Fe(II) across the inner membrane. J Biol Chem. 250, 6433-6438. 88) Fleming, M.D., Romano, M.A., Su, M.A., Garrick, L.M., Garrick, M.D., and Andrews, N.C. (1998). Nramp2 is mutated in the anemic Belgrade (b) rat: evidence of a role for Nramp2 in endosomal iron transport. . Proc. Natl. Acad. Sci. USA 95, 1148-1153. 89) Flint, D., Tuminello, J., and Emptage, M. (1993). The inactivation of Fe-S cluster containing hydrolyases by superoxide. J Biol Chem. 268, 22369-22376. 90) Flohé, L., and Otting, F. (1984). Superoxide dismutase assays. Methods Enzymol 105, 93-104. 91) Fobis-Loisy, I., Aussel, L., and Briat, J.-F. (1996). Post-transcriptional regulation of plant ferritin accumulation in response to iron as observed in the maize mutant ys1. FEBS Letters 397, 149-154. 92) Foury, F., and Cazzalini, O. (1997). Deletion of the yeast homologue of the human gene associated with Friedreich's ataxia elicits iron accumulation in mitochondria.. FEBS Lett. 411, 373-377. 93) Foury, F. (1999). Low iron concentration and aconitase deficiency in a yeast frataxin homologue deficient strain.. FEBS Lett 456, 281-4 94) Foury, F., and Roganti, T. (2002). Deletion of the mitochondrial carrier genes MRS3 and MRS4 suppresses mitochondrial iron accumulation in a yeast frataxin-deficient strain. J Biol Chem. 277, 2447524483. 95) Fridovich, I. (1986). Biological effects of the superoxide radical. Arch Biochem Biophys 247, 1-11. 96) Fridovich, I. (1995). Superoxide radical and superoxide dismutases. Annu Rev Biochem 64, 97-112. 97) Friedrich, T., Weidner, U., Nehls, U., Fecke, W., Schneider, R., and Weiss, H. (1993). Attempts to define distinct parts of NADH:Ubiquinone oxidoreductase (Complex I). J. Bioenerg. Biomembranes 25, 331-337. 98) Friedrich, T., and Weiss, H. (1997). Modular evolution of the respiratory NADH: ubiquinone oxidoreductase and the origin of its modules. J. Theor. Biol 187, 529-540. 99) Friedrich, T., and Scheide , D. (2000). The respiratory complex I of bacteria, archaea and eukarya and its module common with membrane-bound multisubunit hydrogenases. FEBS Lett. 479, 1-5. 100) Friedrich, T., and Bottcher, B. (2004). The gross structure of the respiratory complex I: a Lego system,. Biochim. Biophys. Acta 1608, 1-9. 101) Frugoli, J.A., Zhong, H.H., Nuccio, M.L., McCourt, P., McPeek, M.A., Thomas, T.L., and McClung, C.R. (1996). Catalase Is Encoded by a Multigene Family in Arabidopsis thaliana (L.) Heynh. Plant Physiol. 112, 327-336. 102) Gardner, P. (2002). Aconitase: sensitive target and measure of superoxide.. Methods Enzymol. 349, 9-23. 103) Garmier, M., Carroll, A., Delannoy, E., Vallet, C., Day, D., Small, I., and Millar, A. (2008a). Complex I dysfunction redirects cellular and mitochondrial metabolism in Arabidopsis. Plant Physiol. 148, 13241341. 104) Garmier, M., Carroll, A., Delannoy, E., Vallet, C., Day, D., Small, I., and Millar, A. (2008b). Complex I dysfunction redirects cellular and mitochondrial metabolism in Arabidopsis. Plant Physiol. 148, 13241341. 105) Garrick, M.D., Dolan, K.G., Horbinski, C., Ghio, A.J., Higgins, D., and Porubcin, M. (2003). DMT1: a mammalian transporter for multiple metals. Biometals 16, 41-54. 106) Gelhaye, E., Rouhier, N., Gérard, J., Jolivet, Y., Gualberto, J., Navrot, N., Ohlsson, P., Wingsle, G., Hirasawa, M., Knaff, D., Wang, H., Dizengremel, P., Meyer, Y., and Jacquot, J. (2004). A specific form of thioredoxin h occurs in plant mitochondria and regulates the alternative oxidase. Proc Natl Acad Sci USA. 101, 14545-14550. 107) Gibson, T., Koonin, E., Musco, G., Pastore, A., and Bork, P. (1996). Friedreich's ataxia protein: phylogenetic evidence for mitochondrial dysfunction. Trends Neuroscience 19, 465-468. 108) Giegé, P., Hoffmann, M., Binder, S., and Brennicke, A. (2000). RNA degradation buffers asymmetries of transcription in Arabidopsis mitochondria. EMBO Rep. 1, 164-170. 109) Giege´, P., Sweetlove, L.J., Cognat, V., and Leaver, C.J. (2005). Coordination of Nuclear andMitochondrialGenomeExpressionduring Mitochondrial Biogenesis in Arabidopsis. The Plant Cell 17, 1497–1512. 154 Referencias 110) Greenbers Arnold E. Estándar Methods for the examination of water and wastewater, 1992. Publication office American Public Health Association,18th edición, U.S.A. 111) Gómez-Casati DF, B.M., Gonzalez-Schain N, Mouras A, Zabaleta EJ, Araya A. (2002). A mitochondrial dysfunction induces the expression of nuclear-encoded complex I genes in engineered male sterile Arabidopsis thaliana. FEBS Letters 532, 70-74. 112) Gonzalez-Cabo, P., Vazquez-Manrique, R.P., Garcia-Gimeno, M.A., Sanz, P., and Palau, F. (2005). Frataxin interacts functionally with mitochondrial electron transport chain proteins. Hum. Mol. Genet. 14, 2091-2098. 113) Goodman, H., Ecker, J., and Dean, C. (1995). The genome of Arabidopsis thaliana. Proc Natl Acad Sci U S A. 92, 10831-10835. 114) Goessling, L., Mascotti, D., and Thach, R. (1998). Involvement of heme in the degradation of ironregulatory protein 2. J Biol Chem. 273, 12555-12557. 115) Gray, M.W., Burger, G., and Lang, B.F. (1999). Mitochondrial Evolution. Science 283, 1476-1481. 116) Gunshin, H., Mackenzie, B., Berger, U.V., Gunshin, Y., Romero, M.F., Boron, W.F., Nussberger, S., Gollan, J.L., and Hediger, M.A. (1997). Cloning and characterization of a mammalian proton-coupled metal-ion transporter. Nature 388, 482-488. 117) Gutierre, S., Sabars, C., Lelandais, P., Chetrit, P., Diolez, H., Degand, M., Boutry, F., Vedel, Y., de Kouchkovsky, and De Paepe, R. (1997). Lack of mitochondrial and nuclear-encoded subunits of complex I and alteration of the respiratory chain in Nicotiana sylvestris mitochondrial deletion mutants. Proc. Natl. Acad. Sci. USA 94, 3436-3441. 118) Gutierres, S., Combettes , B., De Paepe, R., Mirande, M., Lelandais, C., Vedel, F., and Chetrit, P. (1999). In the Nicotiana sylvestris CMSII mutant, a recombination-mediated change 5ƍ to the first exon of the mitochondrial Nad1 gene is associated with lack of the NADH:ubiquinone oxidoreductase (complex I) NAD1 subunit. Eur. J. Biochem 261, 361-370. 119) Gutteridge, J., and Halliwell, B. (1989). Iron toxicity and oxygen radicals. Baillieres Clin Haematol 2, 195-256. 120) Gustafsson, C., Govindarajan, S., and Minshull, J. (2004). Codon bias and heterologous protein expression. Trends Biotechnol. 22, 346-353. 121) Hagar, H., Ueda, N., and Shah, S. (1996). Role of reactive oxygen metabolites in DNA damage and cell death in chemical hypoxic injury to LLC-PK1 cells. Am J Physiol 271, 209-215. 122) Hall, D., Gibson, J., and Whatley, F. (1966). Electron spin resonance spectra of spinach ferredoxin. Biochem Biophys Res Commun. 23, 81-84. 123) Harlow, E., and Lane, D. (1988). Antibodies: A laboratory manual. Cold Spring Harbor Laboratory, New York. 124) Hass, C., Lohrmann, J., Albrecht, V., Sweere, U., Hummel, F., Yoo, S., Hwang, I., Zhu, T., Schafer, E., Kudla, J., and Harter, K. (2004). The response regulator 2 mediates ethylene signalling and hormone signal integration in Arabidopsis. EMBO J 23, 3290-3302. 125) Hayes, F., and (2003). Transposon-based strategies for microbial functional genomics and proteomics. Annu Rev Genet. 37, 3-29. 126) Heazlewood, J.L., Howell, K.A.A., and Millar, H. (2003). Mitochondrial complex I from Arabidopsis and rice: orthologs ofmammalian and fungal components coupled with plant-specific subunits. Biochimica et Biophysica Acta 1604, 159- 169. 127) Hempel, S.L., Buettner, G.R., O'Malley, Y.Q., Wessels, D.A., and Flaherty, D.M. (1999). Dihydrofluorescein diacetate is superior for detecting intracellular oxidants: comparison with 2',7'dichlorodihydrofluorescein diacetate, 5(and 6)-carboxy-2',7'-dichlorodihydrofluorescein diacetate, and dihydrorhodamine 123. Free Radical Biology and Medicine 27, 146-159. 128) Herald, V., Heazlewood, J., Day, D., and Millar, A. ( 2003 ). Proteomic identification of divalent metal cation binding proteins in plant mitochondria.. FEBS Letters 537, 96-100. 129) Hernould, M., Suharsono, S., Litvak, S., Araya, A., and Mouras, A. (1993). Male-sterility induction in transgenic tobacco plants with an unedited atp9 mitochondrial gene from wheat. Proc Natl Acad Sci USA 90, 2370-2374. 130) Horsefield, R., Iwata, S., and Byrne, B. (2004). Complex II from a structural perspective. Curr Protein Pept Sci 5, 107-118. 131) Hüttemann, M., Lee, I., Pecinova, A., Pecina, P., Przyklenk, K., and Doan, J. (2008). Regulation of oxidative phosphorylation, the mitochondrial membrane potential, and their role in human disease. J Bioenerg Biomembr. 40. 132) Huynen, M.A., Snel, B., Bork, P., and Gibson, T.J. (2001). The phylogenetic distribution of frataxin indicates a role in iron-sulfur cluster protein assembly. Hum. Mol. Genet. 10, 2463-2468. 133) Iida, S., and Terada, R. (2004). A tale of two integrations, transgene and T-DNA: gene targeting by homologous recombination in rice. Curr Opin Biotechnol 15, 132-138. 134) Isaya G, O.N.H., Gakh O, Park S, Mantcheva R, Mooney SM. (2004). Functional studies of frataxin. Acta Paediatr Suppl 93, 68-71. 135) Inskeep, W.P., and Bloom, P.R. (1985). Extinction Coefficients of Chlorophyll a and b in N,NDimethylformamide and 80% Acetone. Plant Physiol. 77, 483-485. 155 Referencias 136) Iwata, S., Saynovits, M., Link, T., and Michel, H. (1996). Structure of a water soluble fragment of the 'Rieske' iron- sulfur protein of the bovine heart mitochondrial cytochrome bc1 complex determined by MAD phasing at 1.5 A resolution. Structure 4, 567-579. 137) Jacobs, J., and Jacobs, N. (1987). Oxidation of protoporphyrinogen to protoporphyrin, a step in chlorophyll and haem biosynthesis. Purification and partial characterization of the enzyme from barley organelles. Biochem J. 244, 219-224. 138) Jakubowski, W., and Bartosz, G. (2000). 2,7-dichlorofluorescin oxidation and reactive oxygen species: what does it measure? . Cell Biol Int 24, 757-760. 139) Jensen, P. (1966). Antimycin-insensitive oxidation of succinate and reduced nicotinamide-adenine dinucleotide in electron-transport particles. II. Steroid effects. Biochim Biophys Acta. 122, 167-174. 140) Jimenez, A., Hernandez, J., Del Rio, L., and Sevilla, F. (1997). Evidence for the Presence of the Ascorbate-Glutathione Cycle in Mitochondria and Peroxisomes of Pea Leaves. Plant Physiol. 114, 275-284. 141) Kanematsu, S., and Asada, K. (1991). Chloroplast and cytosol isozymes of CuZn-superoxide dismutase: their characteristic amino acid sequences.. Free Radic Res Commun. 12-13, 383-390. 142) Karthikeyan, G., Lewis, L., and Resnick, M. (2002). The mitochondrial protein frataxin prevents nuclear damage. . Hum Mol Genet 11, 351-362. 143) Kaur, H., and Halliwell, B. (1994). Detection of hydroxyl radicals by aromatic hydroxylation.. Methods Enzymol. 233, 67-82. 144) Kim, K., Kim, I., Lee, K., Rhee, S., and Stadtman, E. (1988). The isolation and purification of a specific "protector" protein which inhibits enzyme inactivation by a thiol/Fe(III)/O2 mixed-function oxidation system. JBiolo Chem 263, 4704-4711. 145) Kispal, G., Csere, P., Guiard, B., and Lill, R. (1997). The ABC transporter Atm1p is required for mitochondrial iron homeostasis.. FEBS Lett. 418, 346-350. 146) Kispal, G., Csere, P., Prohl, C., and Lill, R. (1999). The mitochondrial proteins Atm1p and Nfs1p are essential for biogenesis of cytosolic Fe/S proteins. EMBO J. 18, 3981–3989. 147) Klausner, R.D., Rouault, T.A., and Harford, J.B. (1993.). Regulating the fate of mRNA: the control of cellular iron metabolism. Cell 72, 19-28. 148) Kohchi, T., Mukougawa, K., Frankenberg, N., Masuda, M., Yokota, A., and Lagarias, J. (2001). The Arabidopsis HY2 gene encodes phytochromobilin synthase, a ferredoxin-dependent biliverdin reductase. Plant Cell Physiol 13, 425-436. 149) Koutnikova, H., Campuzano, V., and Koenig, M. (1998). Maturation of wild-type and mutated frataxin by the mitochondrial processing peptidase. Hum. Mol. Genet. 7, 1485-1489. 150) Koutnikova, H., Campuzano, V., Foury, F., Dollé, P., Cazzalini, O., and Koenig, M. (1997). Studies of human, mouse and yeast homologues indicate a mitochondrial function for frataxin. Nat Genet 16, 345-351. 151) Kruft, V., Eubel, H., Jansch, L., Werhahn, W., and Braun, H.-P. (2001). Proteomic Approach to Identify Novel Mitochondrial Proteins in Arabidopsis. Plant Physiol. 127, 1694-1710. 152) Kushnir S, B.E., Storozhenko S, Davey MW, Papenbrock J, De Rycke R, Engler G, Stephan UW, Lange H, Kispal G, Lill R, Van Montagu M. (2001). A mutation of the mitochondrial ABC transporter Sta1 leads to dwarfism and chlorosis in the Arabidopsis mutant starik. Plant Cell 13, 89-100. 153) Kwon, S.I., Lee,Hyoungseok ., and Sun An, C. (2007). Differential Expression of Three Catalase Genes in the Small Radish (Rhaphanus sativus L. var. sativus). Mol Cells 24, 37-44. 154) Laloi, C., Mestres-Ortega, D., Marco, Y., Meyer, Y., and Reichheld, J.-P. (2004). The Arabidopsis Cytosolic Thioredoxin h5 Gene Induction by Oxidative Stress and Its W-Box-Mediated Response to Pathogen Elicitor. Plant Physiol. 134, 1006-1016. 155) Lamarche, J., Côté, M., and Lemieux, B. (1980). The cardiomyopathy of Friedreich's ataxia morphological observations in 3 cases. Can J Neurol Sci. 7, 389-396. 156) Lambers, H. (1982). Cyanide resistant respiration: A nonphosphorylatingelectron transport pathway acting as an energy overflow. Plant Physiol. 55, 478-485. 157) Lange, H., Kispal, G., and Lill, R. (1999). Mechanism of Iron Transport to the Site of Heme Synthesis inside Yeast Mitochondria. J. Biol. Chem. 274, 18989-18996. 158) Lange, H., Muhlenhoff, U., Denzel, M., Kispal, G., and Lill, R. (2004). The Heme Synthesis Defect of Mutants Impaired in Mitochondrial Iron-Sulfur Protein Biogenesis Is Caused by Reversible Inhibition of Ferrochelatase. J. Biol. Chem. 279, 29101-29108. 159) Laties, G.G. (1983). Membrane-Associated NAD-Dependent Isocitrate Dehydrogenase in Potato Mitochondria. Plant Physiol. 72, 953-958. 160) Leaver. (1983). Protein synthesis by isolated plant mitochondria. Methods Enzymol 97, 476-484. 161) LeDoux, S., Driggers, W., Hollensworth, B., and Wilson, G. (1999). Repair of alkylation and oxidative damage in mitochondrial DNA. 434, 149-159. 162) Lehninger, A., Nelson, D.L., and Cox, M.M. (1993). Principles of Biochemistry. 2nd edition. Worth Publishers, Estados Unidos 163) Lenhinger. (2005). Principles of Biochemistry, 4th edition. Worth Publishers, Estados Unidos 164) Leif, H., Weidner, U., Berger, A., Spehr, V., Braun, M., van Heek, P., Friedrich, T., Ohnishi, T., and Weiss, H. (1993). Escherichia coli NADH dehydrogenase I, a minimal form of the mitochondrial complex I. Biochem. Soc. Trans. 21, 998-1001. 165) Lemire, B., and Oyedotun, K. (2002). The Saccharomyces cerevisiae mitochondrial succinate:ubiquinone oxidoreductase. Biochim Biophys Acta. 1553, 102-116. 156 Referencias 166) Léon S, T.B., Briat JF, Lobréaux S. (2005). Mitochondrial localization of Arabidopsis thaliana Isu Fe-S scaffold proteins. FEBS Letters 579, 1930-1934. 167) Léon S, T.B., Ribot C, Briat JF, Lobréaux S. (2003). Iron-sulphur cluster assembly in plants: distinct NFU proteins in mitochondria and plastids from Arabidopsis thaliana. Biochem J. 371, 823-830. 168) Lescure, A.-M., Proudhon, D., Pesey, H., Ragland, M., Theil, E., and Briat, J.-F. (1991). Ferritin gene transcription is regulated by iron in soybean cell cultures. Proc Natl Acad Sci USA 88, 8222-8226. 169) Lesuisse, E., Santos, R., Matzanke, B.F., Knight, S.A.B., Camadro, J.-M., and Dancis, A. (2003). Iron use for haeme synthesis is under control of the yeast frataxin homologue (Yfh1). Hum. Mol. Genet. 12, 879-889. 170) Li, X., Volrath, S., Nicholl, D., Chilcott, C., Johnson, M., Ward, E., and Law, M. (2003). Development of protoporphyrinogen oxidase as an efficient selection marker for Agrobacterium tumefaciens-mediated transformation of maize. Plant Physiol. 133, 736-747. 171) Lill, R., and Kispal, G. (2000). Maturation of cellular Fe-S proteins: an essential function of mitochondria. Trends Biochem Sci. 25, 352-356. 172) Lill, R., and Mühlenhoff, U. (2008). Maturation of Iron-Sulfur Proteins in Eukaryotes: Mechanisms, Connected Processes, and Diseases. Annual Review of Biochemistry 77, 669-700. 173) Lill, R., Mühlenhoff, U. (2005). Iron-sulfur-protein biogenesis in eukaryotes. Trends Biochem Sci 30, 133141. 174) Lister, R., Chew, O., Lee, M.-N., Heazlewood, J.L., Clifton, R., Parker, K.L., Millar, A.H., and Whelan, J. (2004). A Transcriptomic and Proteomic Characterization of the Arabidopsis Mitochondrial Protein Import Apparatus and Its Response to Mitochondrial Dysfunction. Plant Physiol. 134, 777-789. 175) Little, H., and Jones, O. (1976). The subcellular localization and properties of the ferrochelatase of etiolated barley. Biochem J. 156, 309-314. 176) Lobréaux, S., Massenet, O., and Briat, J.-F. (1992). Iron induces ferritin synthesis in maize plantlets. Plant Mol Biol 19, 563-575. 177) Lodi, R., Cooper, J., Bradley, J., Manners, D., Styles, P., Taylor, D., and Schapira, A. (1999). Deficit of in vivo mitochondrial ATP production in patients with Friedreich ataxia. Proc Natl Acad Sci USA. 96, 11492-11495. 178) Logan, D.C. (2006). The mitochondrial compartment. J. Exp. Bot. 57, 1225-1243. 179) Lohrmann, J., Sweere, U., Zabaleta, E., Bäurle, I., Keitel, C., Kozma-Bognar, L., Brennicke, A., Schäfer, E., Kudla, J., and Harter, K. (2001). The response regulator ARR2: a pollen-specific transcription factor involved in the expression of nuclear genes for components of mitochondrial complex I in Arabidopsis.. Mol Genet Genomics 265, 2-13. 180) Loiseau, L., Ollagnier-de-Choudens, S., Nachin, L., Fontecave, M., and Barras, F. (2003). Biogenesis of Fe-S Cluster by the Bacterial Suf System: SufS and SufE form a new type of cysteine desulfurase. J. Biol. Chem. 278, 38352-38359. 181) Lombardo, M.E., Araujo, L.S., Ciccarelli, A.B., and Batlle, A. (2005). A spectrophotometric method for estimating hemin in biological systems. Analytical Biochemistry 341, 199-203. 182) Loschen, G., Flohe, L., and Chance, B. (1971). Respiratory chain linked H2O2 production in pigeon heart mitochondria. FEBS Lett 18, 261-264. 183) Lu, C., and Cortopassi, G. (2007). Frataxin knockdown causes loss of cytoplasmic iron-sulfur cluster functions, redox alterations and induction of heme transcripts. Arch Biochem Biophys. 457, 111-122. 184) Lukianova, O., and David, S. (2005). A role for iron-sulfur clusters in DNA repair. Curr Opin Chem Biol. 9, 145-151. 185) Lümmen, P. (1998). Complex I inhibitors as insecticides and acaricides.. Biochim Biophys Acta. 1364, 287-296. Review. 186) Mackenzie, S., and McIntosh, L. (1999). Higher Plant Mitochondria. Plant Cell 11, 571-586. 187) Maliandi, M.V., Busi, M.V., Clemente, M., Zabaleta, E.J., Araya, A., and Gomez-Casati, D.F. (2007). Expression and one-step purification of recombinant Arabidopsis thaliana frataxin homolog (AtFH). Protein Expression and Purification 51, 157-161. 188) Margulis, L and Sagan D. (1995). What Is Life? Publisher, Simon & Schuster, New York. 189) Marienfeld, R., and Newton, K.J. (1994). The maize NCS2 abnormal growth mutant has a chimeric Nad4-Nad7 mitochondrial gene and is associated with reduced complex I function. Genetics 138, 855-863. 190) Martin, M., Rodríguez-Colman, M.J., Gómez-Casati, D.F., Lamattina, L., and Zabaleta, E.J. (2009). Nitric oxide accumulation is required to protect against iron-mediated oxidative stress in frataxin-deficient Arabidopsis plants. FEBS Letters 583, 542-548. 191) Martienssen, R.A. (1998). Functional genomics: probing plant gene function and expression with transposons. Proc Natl Acad Sci U S A. 95, 2021-2026. 192) Mathiesen, C., and Hagerhall, C. (2003). The ‘antiporter module’ of respiratory chain complex I includes the MrpC/NuoK subunit-a revision of the modular evolution scheme. FEBS Lett. 549, 7-13. 193) Masuda, T., Suzuki, T., Shimada, H., Ohta, H., and Takamiya, K. (2003). Subcellular localization of two types of ferrochelatase in cucumber. . Planta 217, 602-609. 194) Maas, M., Krause, F., Dencher, N., and Sainsard-Chanet, A. (2008). Respiratory Complexes III and IV Are Not Essential for the Assembly/Stability of Complex I in Fungi. J Mol Biol. 387, 259-269. 157 Referencias 195) Maxwell, D.P., Wang, Y., and McIntosh, L. (1999). The alternative oxidase lowers mitochondrial reactive oxygen production in plant cells. Proc Natl Acad Sci USA 96, 8271-8276. 196) McCoubrey, W., Huang, T., and Maines, M. (1997). Heme oxygenase-2 is a hemoprotein and binds heme through heme regulatory motifs that are not involved in heme catalysis.. J Biol Chem. 272, 1256812574. 197) McFadden, G.I., and Ralph, S.A. (2003). Dynamin: The endosymbiosis ring of power? Proceedings of the National Academy of Sciences of the United States of America 100, 3557-3559. 198) Menz, R., Griffith, M., Day, D., and Wiskich, J. (1992). Matrix NADH dehydrogenases of plant mitochondria and sites of quinone reduction by complex I. Eur J Biochem 208, 481-485. 199) Meyer, E., Taylor, N., and Millar, A. (2008). J Proteome Res. Resolving and identifying protein components of plant mitochondrial respiratory complexes using three dimensions of gel electrophoresis. 7, 786-794. 200) Meyerowitz, E.M. (1987). Arabidopsis thaliana Annu. Rev. Plant Biol.Genet 21, 93-111 201) Meyerowitz, E.M. (1994). in Arabidopsis, eds. Meyerowitz, E. M. & Somerville,C. R. Cold Spring Harbor Lab. Press,Plainview, NY, 21-36. 202) Michael, T., and McClung, C. (2002). Phase-specific circadian clock regulatory elements in Arabidopsis. Plant Physiol. 130, 627-638. 203) Michalecka, A.M., Svensson, S., Johansson, F.I., Agius, S.C., Johanson, U., Brennicke , A., Binder, S., and Rasmusson, A.G. (2003). Arabidopsis Genes Encoding Mitochondrial Type II NAD(P)H Dehydrogenases Have Different Evolutionary Origin and Show Distinct Responses to Light Plant Physiol. 133, 642-652. 204) Millar, A.H., Eubel, H., Jänsch, L., Kruft, V., Heazlewood, J.L., and Braun, H.P. (2004). Mitochondrial cytochrome c oxidase and succinate dehydrogenase complexes contain plant specific subunits. Plant Mol Biol 56, 77-90. 205) Millar, H., Small, I.D., Day, D., and Whelana, J. (2008 ). Mitochondrial Biogenesis and Function in Arabidopsis. The Arabidopsis Book. American Society of Plant Biologists. 206) Millar, A.H., Sweetlove, L.J., Giegé, P., and Leaver, C.J. (2001). Analysis of the Arabidopsis mitochondrial proteome. Plant Physiol. 127, 1711-1727. 207) Moller.I (2001). Plant mitochondria and oxidative stress: electron transport,NADPH turnover, and metabolism of reactive oxygen species. Annu. Rev. PlantPhysiol. Plant Mol. Biol 52, 561-591. 208) Moller, I. (2001). PLANT MITOCHONDRIA AND OXIDATIVE STRESS: Electron Transport, NADPH Turnover, and Metabolism of Reactive Oxygen Species.. Annu Rev Plant Physiol Plant Mol Biol 52, 561591. 209) Moller, I.M., and Kristensen, B.K. (2004). Protein oxidation in plant mitochondria as a stress indicator. Photochem Photobiol Sci 3, 730-735. 210) Moran, R. (1982). Formulae for determination of chlorophyllous pigments extracted with N, Ndimethylformamide. Plant Physiol. 69, 1376-1381. 211) Morgan, R., Naglie, G., Horrobin, D., and Barbeau, A. (1979). Erythrocyte protoporphyrin levels in patients with Friedreich's and other ataxias. Can J Neurol Sci. 6, 227-232. 212) Mori, I.C., and Schroeder, J.I. (2004). Reactive Oxygen Species Activation of Plant Ca2+ Channels. A Signaling Mechanism in Polar Growth, Hormone Transduction, Stress Signaling, and Hypothetically Mechanotransduction. Plant Physiol. 135, 702-708. 213) Muhlenhoff, U., Richhardt, N., Ristow, M., Kispal, G., and Lill, R. (2002). The yeast frataxin homolog Yfh1p plays a specific role in the maturation of cellular Fe/S proteins. Hum. Mol. Genet. 11, 2025-2036. 214) Mühlenhoff, U., and Lill, R. (2000). Biogenesis of iron-sulfur proteins in eukaryotes: a novel task of mitochondria that is inherited from bacteria. Biochimica et Biophysica Acta (BBA) - Bioenergetics 1459, 370-382. 215) Muhlenhoff, U., Richhardt, N., Gerber, J., and Lill, R. (2002). Characterization of Iron-Sulfur Protein Assembly in Isolated Mitochondria. A REQUIREMENT FOR ATP, NADH, AND REDUCED IRON. J. Biol. Chem. 277, 29810-29816. 216) Musco G, S.G., Kolmerer B, Adinolfi S, Martin S, Frenkiel T, Gibson T, Pastore A. (2000). Towards a structural understanding of Friedreich's ataxia: the solution structure of frataxin. Structure 8, 695-707. 217) Napier, I., Ponka, P., and Richardson, D. (2005). Iron trafficking in the mitochondrion: novel pathways revealed by disease. Blood 105, 1867-1874. 218) Napoli, C., Lemieux, C., and Jorgensen, R. (1990 ). Introduction of a Chimeric Chalcone Synthase Gene into Petunia Results in Reversible Co-Suppression of Homologous Genes in trans. Plant Cell 2, 279-289. 219) Narita, S., Tanaka, R., Ito, T., Okada, K., Taketani, S., and Inokuchi, H. (1996). Molecular cloning and characterization of a cDNA that encodes protoporphyrinogen oxidase of Arabidopsis thaliana. . Gene. 182, 169-175. 220) Neupert, W., and Herrmann, J. (2007). Translocation of proteins into mitochondria. Annu Rev Biochem 76, 723-749. 221) Nicholls, A., Sharp, K., and Honig, B. (1991). Protein folding and association: insights from the interfacial and thermodynamic properties of hydrocarbons.. Proteins 11, 281-296. 222) Noctor, G., Dutilleul, C., De Paepe, R., and Foyer, C. (2004). Use of mitochondrial electron transport mutants to evaluate the effects of redox state on photosynthesis, stress tolerance and the integration of carbon/nitrogen metabolism. J Exp Bot 55, 49-57. 158 Referencias 223) O'Neill, H.A., Gakh, O., Park, S., Cui, J., Mooney, S.M., Sampson, M., Ferreira, G.C., and Isaya, G. (2005). Assembly of Human Frataxin Is a Mechanism for Detoxifying Redox-Active Iron. Biochemistry 44, 537-545. 224) Orrenius, S., Zhivotovsky, B., and Nicotera, P. (2003). Regulation of cell death: the calcium-apoptosis link. Nat Rev Mol Cell Biol 4, 552-565. 225) Ogawa, K., Sun, J., Taketani, S., Nakajima, O., Nishitani, C., Sassa, S., Hayashi, N., Yamamoto, M., Shibahara, S., Fujita, H., Igarashi, K., and (2001). Heme mediates derepression of Maf recognition element through direct binding to transcription repressor Bach1. EMBO J. 20, 2835-2843. 226) Pal, J., and Joshi-Purandare, M. (2001). Dose-dependent differential effect of hemin on protein synthesis and cell proliferation in Leishmania donovani promastigotes cultured in vitro. J.Biosci. 26, 225-231. 227) Pal Singh, D., Cornah, J.E., Hadingham, S., and Smith, A.G. (2002). Expression analysis of the two ferrochelatase genes in Arabidopsis in different tissues and under stress conditions reveals their different roles in haem biosynthesis Plant Molecular Biology 4-5, 773-788. 228) Palmer, G., and Sands, R. (1966). On the magnetic resonance of spinach ferredoxin. J Biol Chem 241, 253. 229) Pandolfo, M. (2002). Frataxin deficiency and mitochondrial dysfunction. Mitochondrion. 2, 87-93. 230) Papenbrock, J., Mishra, S., Mock, H.-P., Kruse, E., Schmidt, E.-K., Petersman, A., Braun, H.-P., and Grimm, B. (2001). Impaired expression of the plastidic ferrochelatase by antisense RNA synthesis leads to a necrotic phenotype of transformed tobacco plants. Plant J. 28, 41-50. 231) Porra, R., and Jones, O. (1963). Studies on ferrochelatase. Biochem J. 87, 181-185. 232) Park, S., Gakh, O., Mooney, S.M., and Isaya, G. (2002). The Ferroxidase Activity of Yeast Frataxin. J. Biol. Chem. 277, 38589-38595. 233) Patel, P., and Isaya, G. (2001). Friedreich ataxia: from GAA triplet-repeat expansion to frataxin deficiency.. Am J Hum Genet 69, 15-24. 234) Peiffer, W., Ingle, R., and S., F.-M. (1990). Structurally unique plant cytochrome c oxidase isolated from wheat germ, a rich source of plant mitochondrial enzymes. Biochemistry, 29, 8696-8701. 235) Pfeiffer, D.R., Gunter, T.E., Eliseev, R., Broekemeier, K.M., and Gunter, K.K. (2001). Release of Ca2+ from mitochondria via the saturable mechanisms and the permeability transition. IUBMB Life. 52, 205-212. 236) Peters, J., Fisher, K., and Dean, D. (1995). Nitrogenase structure and function: a biochemical-genetic perspective.. Annu Rev Microbiol. 49, 335-366. 237) Petit, J., Van Wuytswinkel, O., Briat, J.-F., Lobreaux, S., and (2001). Characterization of an irondependent regulatory sequence involved in the transcriptional control of AtFer1 and ZmFer1 plant ferritin genes by iron. J Biol Chem 276, 5584-5590. 238) Pilon-Smits, E.A.H., Garifullina, G.F., Abdel-Ghany, S., Kato, S.-I., Mihara, H., Hale, K.L., Burkhead, J.L., Esaki, N., Kurihara, T., and Pilon, M. (2002). Characterization of a NifS-Like Chloroplast Protein from Arabidopsis. Implications for Its Role in Sulfur and Selenium Metabolism. Plant Physiol. 130, 1309-1318. 239) Pineau, B., Mathieu , C., Gerard-Hirne, C., De Paepe, R., and Chetrit, P. (2005). Targeting the NAD7 subunit to mitochondria restores a functional complex I and a wild type phenotype in the Nicotiana sylvestris CMS II mutant lacking Nad7. J. Biol. Chem. 280, 25994-26001. 240) Pla, M., Mathieu, C., De Paepe, R., Chetrit, P., and Vedel, F. (1995). Deletion of the last two exons of the mitochondrial Nad7 gene results in lack of the NAD7 polypeptide in a Nicotiana sylvestris CMS mutant. Mol. Gen. Genet. 248, 79-88. 241) Pogson, B., Woo, N., Förster, B., and Small, I. (2008). Plastid signalling to the nucleus and beyond.. Trends Plant Sci 13, 602-609. 242) Pohl, T., Uhlmann, M., Kaufenstein, M., and Friedrich, T. (2007). Lambda Red-mediated mutagenesis and efficient large scale affinity purification of the Escherichia coli NADH:ubiquinone oxidoreductase (complex I). Biochemistry 46, 10694-10702. 243) Polygalova, O., Bufetov, E., and Ponomareva, A. (2007). Wheat root cells functioning under inhibition of I and II complexes of mitochondrial respiratory chain. Tsitologiia 49, 664-670. 244) Proudhon, D., Wei, J., Briat, J.-F., and Theil, E. (1996). Ferritin gene organization: differences between plants and animals suggest possible kingdom-specific selective constraints. J Mol Evol. 42, 325-336. 245) Puccio, H., and Kœnig, M. (2002). Friedreich ataxia: a paradigm for mitochondrial diseases Current Opinion Genet. Dev. 12, 272-277. 246) Puccio, H., Simon, D., Cossee, M., Criqui-Filipe, P., Tiziano, F., Melki, J., Hindelang, C., Matyas, R., Rustin, P., and Koenig, M. (2001). Mouse models for Friedreich ataxia exhibit cardiomyopathy, sensory nerve defect and Fe-S enzyme deficiency followed by intramitochondrial iron deposits. Nat Genet 27, 181186. 247) Puccio, H., and Koenig, M. (2000). Recent advances in the molecular pathogenesis of Friedreich ataxia. Hum Mol Genet 9, 887-892. 248) Radisky, D., Babcock, M., and Kaplan, J. (1999). The yeast frataxin homologue mediates mitochondrial iron efflux. Evidence for a mitochondrial iron cycle. J Biol Chem. 274, 4497-4499. 249) Rapaport, D. (2002). Biogenesis of the mitochondrial TOM complex. Trends Biochem Sci. 27. 250) Rasmusson, A., Soole, K., and Elthon, T. (2004). Alternative NAD(P)H dehydrogenases of plant mitochondria. Annu Rev Plant Biol 55, 23-39. 159 Referencias 251) Rasmusson, A.G., and MØLler, I.M. (1991). Effect of calcium ions and inhibitors on internal NAD(P)H dehydrogenases in plant mitochondria. European Journal of Biochemistry 202, 617-623. 252) Rasmusson, A.G., Heiser, V., Zabaleta, E., Brennicke, A., and Grohmann, L. (1998). Physiological, biochemical and molecular aspects of mitochondrial complex I in plants. Biochimica et Biophysica Acta (BBA) - Bioenergetics 1364, 101-111. 253) Rea, S., Ventura, N., and Johnson, T. (2007). Relationship between mitochondrial electron transport chain dysfunction, development, and life extension in Caenorhabditis elegans. e259. PLoS Biol 5. 254) Rees, D., and Howard, J. (2000 ). Nitrogenase: standing at the crossroads.. Curr Opin Chem Biol 5, 559566. Review. 255) Rhee, S., Chae, H., and Kim, K. (2005). Peroxiredoxins: a historical overview and speculative preview of novel mechanisms and emerging concepts in cell signaling.Free Radic Biol Med. 38, 1543-1552. 256) Richter, C., Park, J., and Ames, B. (1988). Normal oxidative damage to mitochondrial and nuclear DNA is extensive. Proc Natl Acad Sci USA. 85, 6465-6467. 257) Rieske, J.S., Maclennan, D.H., and Coleman, R. (1964). Isolation and properties of an iron-protein from the (reduced coenzyme Q)-cytochrome C reductase complex of the respiratory chain. Biochem. Biophys. Res. Commun 15, 338-344. 258) Ristow, M., Pfister, M.F., Yee, A.J., Schubert, M., Michael, L., Zhang, C.-Y., Ueki, K., Michael, M.D., II, Lowell, B.B., and Kahn, C.R. (2000). Frataxin activates mitochondrial energy conversion and oxidative phosphorylation. Proc Natl Acad Sci USA 97, 12239-12243. 259) Roberts, T., Fredlund, K., and Møller, I. (1995). Direct evidence for the presence of two external NAD(P)H dehydrogenases coupled to the electron transport chain in plant mitochondria. FEBS Lett. 373. 307-309. 260) Rotig, A., de Lonlay, P., Chretien, D., Foury, F., Koenig, M., Sidi, D., Munnich, A., and Rustin, P. (1997). Aconitase and mitochondrial iron-sulphur protein deficiency in Friedreich ataxia. Nat Genet 17, 215-217. 261) Rötig, A., Sidi, D., Munnich, A., and Rustin, P. (2002). Molecular insights into Friedreich's ataxia and antioxidant-based therapies. Trends Mol Med 5, 221-224. 262) Rubbo, H., Radi, R., Trujillo, M., Telleri, R., Kalyanaraman, B., Barnes, S., Kirk, M., and Freeman, B. (1994). Nitric oxide regulation of superoxide and peroxynitrite-dependent lipid peroxidation. Formation of novel nitrogen-containing oxidized lipid derivatives. J Biol Chem. 269, 26066-26075. 263) Sabar, M., De Paepe, R., and de Kouchkovsky, Y. (2000). Complex I impairment, respiratory compensations, and photosynthetic decrease in nuclear and mitochondrial male sterile mutants of Nicotiana sylvestris. Plant Physiol. 124, 1239-1250. 264) Saisho, D., Nambara, E., Naito, S., Tsutsumi, N., Hirai, A., and Nakazono, M. (1997). Characterization of the gene family for alternative oxidase from Arabidopsis thaliana. Plant Mol Biol 35, 585-596. 265) Saisho, D., Nakazono, M., Lee, K., Tsutsumi, N., Akita, S., and Hirai. (2001). The gene for alternative oxidase-2 (AOX2) from Arabidopsisthaliana consists of five exons unlike other AOX genes and is transcribed at an early stage during germination. Genes Genet. 76, 89-97. 266) Sakamoto, N., Chastain, P.D., Parniewsk, P., Ohshima, K., Pandolfo, M., Griffith, J.D., and Wells, R.D. (1999). Sticky DNA: Self-Association Properties of Long GAA·TTC Repeats in R·R·Y Triplex Structures from Friedreich’s Ataxia. Mollecular Cell 3, 465-475. 267) Sambrook, J., Fritsch, E., and Maniatis, T. (1989). Molecular cloning: A laboratory manual, 2nd edn. (Nolan C(ed)). Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY. 268) Santana, M., Tan, F., and Smith, A. (2002). Molecular characterization of coproporphyrinogen oxidasa from Glycine max and Arabidopsis thaliana. Plant Physiol.biochem. 40, 289-298. 269) Sazanov, L., and Hinchliffe, P. (2006). Structure of the hydrophilic domain of respiratory complex I from Thermus thermophilus. Science. 311, 1430-1436. 270) Scandalios, J., Chang, D., McMillin, D., Tsaftaris, A., and Moll, R. (1980). Genetic regulation of the catalase developmental program in maize scutellum: Identification of a temporal regulatory gene. Proc Natl Acad Sci USA. 77, 5360-5364. 271) Schapira. A (1998). Mitochondrial dysfunction in neurodegenerative disorders. Biochim Biophys Acta 1366, 225-233. 272) Schapira, A. (1999). Mitochondrial involvement in Parkinson's disease, Huntington's disease, hereditary spastic paraplegia and Friedreich's ataxia.. Biochim Biophys Acta. 1410, 159-170. 273) Schapira, A. (2000). Mitochondrial disorders. Curr Opin Neurol 13, 527-532. 274) Scheffler, I. (1998). Molecular genetics of succinate:quinone oxidoreductase in eukaryotes. Prog Nucleic Acid Res Mol Biol 60, 267-315. 275) Schoenfeld, R.A., Napoli, E., Wong, A., Zhan, S., Reutenauer, L., Morin, D., Buckpitt, A.R., Taroni, F., Lonnerdal, B., Ristow, M., Puccio, H., and Cortopassi, G.A. (2005). Frataxin deficiency alters heme pathway transcripts and decreases mitochondrial heme metabolites in mammalian cells. Hum. Mol. Genet. 14, 3787-3799. 276) Schueck, N.D., Wootner, M., and Koeller, D.M. (2001). The role of the mitochondrion in cellular iron homeostasis. Mitochondrion. 1, 51-60. 277) Schulte, U. (2001). Biogenesis of respiratory complex I. J. Bioenerg. Biomembr 33, 205-212. 160 Referencias 278) Simpson, P.B., Mehotra, S., Lange, G.D., and Russell, J.T. (1997). High Density Distribution of Endoplasmic Reticulum Proteins and Mitochondria at Specialized Ca2+ Release Sites in Oligodendrocyte Processes. J. Biol. Chem. 272, 22654-22661. 279) Smirnoff. (1998). Plant resistance to environmental stress. Curr Opin Biotechnol 9, 214-219. 280) Smith, A., Santana, M., Wallace-Cook, A., Roper, J., and Labbe-Bois, R. (1994). Isolation of a cDNA encoding chloroplast ferrochelatase from Arabidopsis thaliana by functional complementation of a yeast mutant. J Biol Chem. 269, 13405-13413. 281) Smith, A., Marsh, O., and Elder, G. (1993). Investigation of the subcellular location of the tetrapyrrolebiosynthesis enzyme coproporphyrinogen oxidase in higher plants. Biochem J. 292, 503-508. 282) Smith, M., and Doolittle, R. (1992). A comparison of evolutionary rates of the two major kinds of superoxide dismutase. J Mol Evol. 34, 175-184. 283) Stadtman, E., and Levine, R. (2000). Protein oxidation.. Ann N Y Acad Sci. 899, 191-208. 284) Stojanovski, D., Guiard, B., Kozjak-Pavlovic, V., Pfanner, N., and Meisinger, C. (2007). Alternative function for the mitochondrial SAM complex in biogenesis of alpha-helical TOM proteins. J Cell Biol 179, 881-893. 285) Sugioka, K., Nakano, M., Totsune-Nakano, H., Minakami, H., Tero-Kubota, S., and Ikegami, Y. (1988). Mechanism of O2- generation in reduction and oxidation cycle of ubiquinones in a model of mitochondrial electron transport systems. Biochim Biophys Acta. 936, 377-385. 286) Swartz, J. (2001). Advances in Escherichia coli production of therapeutic proteins. Curr Opin Biotechnol. 12, 195-201. 287) Sweetlove, L.a.F., CH. (2004). Roles for reactive oxygen species andantioxidants in plant mitochondria. , Vol 1, Advances in In DA Day, AH Millar, J Whelan, eds, Plant Mitochondria: From Genome to Function Photosynthesis and Respiration. Kluwer Academic Press, Dordrecht, The Netherlands, pp 307-320. 288) Sweetlove, L., Heazlewood, J.L., Herald, V., Holtzapffel, R., Day, D.A., Leaver, C.J., and Millar, A.H. (2002). The impact of oxidative stress on Arabidopsis mitochondria. Plant J. 32, 891-904. 289) Taiz, L., and Zeiger, E. (1998). Plant Physiology., 2nd Ed. 290) Takahara, M., Takahashi, H., Matsunaga, S., Miyagishima, S., Takano, H., Sakai, A., Kawano, S., and Kuroiwa, T. (2000). A putative mitochondrial ftsZ gene is present in the unicellular primitive red alga Cyanidioschyzon merolae. . Mol Gen Genet. 264, 452-460. 291) Takahashi, Y., and Tokumoto, U. (2002). A Third Bacterial System for the Assembly of Iron-Sulfur Clusters with Homologs in Archaea and Plastids. J. Biol. Chem. 277, 28380-28383. 292) Takahashi Y, M.A., Fujita Y, Matsubara H. (1991). Roles of ATP and NADPH in Formation of the Fe-S Cluster of Spinach Ferredoxin. Plant Physiol. 95, 104-110. 293) Tanaka, R., and Tanaka, A. (2007). Tetrapyrrole Biosynthesis in Higher Plants. Annual Review of Plant Biology 58, 321-346. 294) Taketani, S., and Tokunaga, R. (1984 ). Non-enzymatic heme formation in the presence of fatty acids and thiol reductants. Biochim Biophys Acta. 798, 226-230. 295) Tang, X., Xu, R., Reynolds, M., Garcia, M., Heinemann, S., and Hoshi, T. (2003). Haem can bind to and inhibit mammalian calcium-dependent Slo1 BK channels.. Nature 425, 531-535. 296) Tangerås, A. (1985). Mitochondrial iron not bound in heme and iron-sulfur centers and its availability for heme synthesis in vitro.. Biochim Biophys Acta. 843, 199-207. 297) Tartaglia, L.A., Storz, G., Brodsky, M.H., Lai, A., and Ames, B.N. (1990). Alkyl hydroperoxide reductase from Salmonella typhimurium. Sequence and homology to thioredoxin reductase and other flavoprotein disulfide oxidoreductases. J. Biol. Chem. 265, 10535-10540. 298) Teeter, M., Baginsky, M., and Hatefi, Y. (1969). Ectopic inhibition of the complexes of the electron transport system by rotenone, piericidin A, demerol and antimycin A. Biochim Biophys Acta. 172, 331333. 299) Terry, M., Linley, P., and Kohchi, T. (2002). Making light of it: the role of plant haem oxygenases in phytochrome chromophore synthesis. Biochem Soc Trans 30, 604-609. 300) Terry, M.J., and Kendrick, R.E. (1999). Feedback Inhibition of Chlorophyll Synthesis in the Phytochrome Chromophore-Deficient aurea and yellow-green-2 Mutants of Tomato. Plant Physiol. 119, 143-152. 301) Teixeira, R., Knorpp, C., and Glimelius, K. (2005). Modified sucrose, starch, and ATP levels in two alloplasmic male-sterile lines of B. napus. J. Exp. Bot. 56, 1245-1253. 302) Thierbach, R., Schulz, T., Isken, F., Voigt, A., Mietzner, B., Drewes, G., von Kleist-Retzow, J., Wiesner, R., Magnuson, M., Puccio, H., Pfeiffer, A., Steinberg, P., and Ristow, M. (2005). Targeted disruption of hepatic frataxin expression causes impaired mitochondrial function, decreased life span and tumor growth in mice. Hum Mol Genet. 14, 3857-3864. 303) Thirkettle-Watts, D., McCabe, T.C., Clifton, R., Moore, C., Finnegan, P.M., Day, D.A., and Whelan, J. (2003). Analysis of the Alternative Oxidase Promoters from Soybean. Plant Physiol. 133, 1158-1169. 304) Umbach, A.L., and Siedow, J.N. (1993). Covalent and Noncovalent Dimers of the Cyanide-Resistant Alternative Oxidase Protein in Higher Plant Mitochondria and Their Relationship to Enzyme Activity. Plant Physiol. 103, 845-854. 305) Unseld, M., Marienfeld, J.R., Brandt, P., and Brennicke, A. (1997). The mitochondrial genome of Arabidopsis thaliana contains 57 genes in 366,924 nucleotides. Nat Genet 15, 57-61. 161 Referencias 306) van Lis, R., Atteia, A., Nogaj, L., and Beale, S. (2005). Subcellular localization and light-regulated expression of protoporphyrinogen IX oxidase and ferrochelatase in Chlamydomonas reinhardtii. Plant Physiol. 139, 1946-1958. 307) Vazzola, V., Losa, A., Soave, C., and Murgia, I. (2007). Knockout of frataxin gene causes embryo lethality in Arabidopsis. FEBS Letters. 581, 667-672. 308) Videira, A., and Duarte, M. (2002). From NADH to ubiquinone in Neurospora mitochondria. Biochim. Biophys. Acta 1555, 187-191. 309) Vitha, S., McAndrew, R.S., and Osteryoung, K.W. (2001). FtsZ Ring Formation at the Chloroplast Division Site in Plants. J. Cell Biol. 153, 111-120. 310) Vogel, R., Smeitink, J., and Nijtmans, L. (2007). Human mitochondrial complex I assembly: a dynamic and versatile process. Biochim Biophys Acta. 1767, 1215-1227. 311) Wang, W., Vinocur, B., Shoseyov, O., and Altman, A. (2004). Role of plant heat-shock proteins and molecular chaperones in the abiotic stress response.. Trends Plant Sci 9, 244-252. 312) Wei, J., and Theil, E. (2000). Identification and characterization of the iron regulatory element in the ferritin gene of a plant (soybean). J Biol Chem. 275, 17488-17493. 313) Werhahn, W., and Braun, H. (2002). Biochemical dissection of the mitochondrial proteome from Arabidopsis thaliana by three-dimensional gel electrophoresis. Electrophoresis 23, 640-646. 314) Whelan, J., and Glaser, E. (1997). Protein import into plant mitochondria. Plant Mol Biol. 33, 771-789. 315) Williams, P., Hardeman, K., Fowler, J., and Rivin, C. (2006). Divergence of duplicated genes in maize: evolution of contrasting targeting information for enzymes in the porphyrin pathway. Plant J. 45, 727-739. 316) Wilson, R., and Roof, D. (1997). Respiratory deficiency due to loss of mitochondrial DNA in yeast lacking the frataxin homologue. Nat Genet. 16, 352-357. 317) Wilson, K., Long, D., Swinburne, J., and Coupland, G. (1996). A Dissociation insertion causes a semidominant mutation that increases expression of TINY, an Arabidopsis gene related to APETALA2.. Plant Cell 8, 659-671 318) Woodarda, S.I., and Dailey, H.A. (2000). Multiple Regulatory Steps in Erythroid Heme Biosynthesis 384, 375-378. 319) Xu, X., and Møller, S. (2006). AtSufE is an essential activator of plastidic and mitochondrial desulfurases in Arabidopsis. EMBO J. 25, 900-909. Yan, L., Levine, R., and Sohal, R. (1997). Oxidative damage during aging targets mitochondrial aconitase. Proc Natl Acad Sci USA. 94, 11168-72. 320) Yan, H., Wang, W., He, Y., Zhao, Z., Gao, Y., Zhang, Y., and Sun, S. (2004). Construction, expression, and characterization of a recombinant annexin B1-low molecular weight urokinase chimera in Escherichia coli. Acta Biochim Biophys Sin 36, 184-190. 321) Yankovskaya, V., Horsefield, R., Törnroth, S., Luna-Chavez, C., Miyoshi, H., Léger, C., Byrne, B., Cecchini, G., and Iwata, S. (2003). Architecture of succinate dehydrogenase and reactive oxygen species generation. Science 299, 700-704. 322) Ye, H., Abdel-Ghany, S.E., Anderson, T.D., Pilon-Smits, E.A.H., and Pilon, M. (2006). CpSufE Activates the Cysteine Desulfurase CpNifS for Chloroplastic Fe-S Cluster Formation. J. Biol. Chem. 281, 8958-8969. 323) Youle, R., and Karbowski, M. (2005). Mitochondrial fission in apoptosis. Nat Rev Mol Cell Biol. 6, 657663. 324) Yoon, T., and Cowan, J.A. (2004). Frataxin-mediated Iron Delivery to Ferrochelatase in the Final Step of Heme Biosynthesis. J. Biol. Chem. 279, 25943-25946. 325) Zabaleta, E., Mouras, A., Hernould, M., Suharsono, and Araya, A. (1996). Transgenic male-sterile plant induced by an unedited atp9 gene is restored to fertility by inhibiting its expression with antisense RNA. Proc Natl Acad Sci USA 93, 11259-11263. 326) Zarse, K., Schulz, T., Birringer, M., and Ristow, M. (2007). Impaired respiration is positively correlated with decreased life span in Caenorhabditis elegans models of Friedreich Ataxia. FASEB J 21, 1271-1275. 327) Zhang, L., and Guarente, L. (1995). Heme binds to a short sequence that serves a regulatory function in diverse proteins.. EMBO J. 14, 313-320. 328) Zhang, Y., Lyver, E.R., Knight, S.A.B., Lesuisse, E., and Dancis, A. (2005). Frataxin and Mitochondrial Carrier Proteins, Mrs3p and Mrs4p, Cooperate in Providing Iron for Heme Synthesis. J. Biol. Chem. 280, 19794-19807. 329) Zheng, L., White, R.H., Cash, V.L., Jack, R.F., and Dean, D.R. (1993). Cysteine desulfurase activity indicates a role for NIFS in metallocluster biosynthesis. Proceedings of the National Academy of Sciences of the United States of America 90, 2754-2758. 330) Zhong, H., and McClung, C. (1996). The circadian clock gates expression of two Arabidopsis catalase genes to distinct and opposite circadian phases. Mol Gen Genet. 251, 196-203. 331) Zhu, Y., Lee, H., and Zhang, L. (2002). An examination of heme action in gene expression: heme and heme deficiency affect the expression of diverse genes in erythroid k562 and neuronal PC12 cells.. DNA Cell Biol 21, 333-346. 162