Del gene a la proteína Capítulo 17 Campbell y Reece, 2002 Sexta
Anuncio

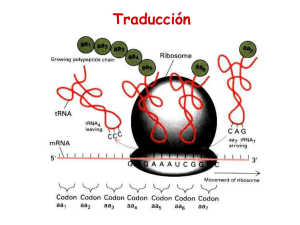
Del gene a la proteína Capítulo 17 Campbell y Reece, 2002 Sexta edición George Beadle y Edward Tatum demostraron la relación entre gene y proteína usando Neurospora crassa • Ver experimentos en la Figura 17.1 • Así se estableció la hipótesis de un gene una enzima • Todas las enzimas son proteína, pero no todas las proteínas son enzimas, por lo tanto se modificó el concepto a un gene una proteína • Algunas proteínas están compuestas de dos o más cadenas polipéptidas (e.I. hemoglobina), por lo tanto con el tiempo la hipótesis cambió a un gene un polipéptido En la Figura 17.2 se comparan los términos transcripción y traducción en las células eucariota y procariota Transcripción es el proceso mediante el cual se sintetiza de una molécula de RNA a partir del DNA • Es el mismo lenguaje, de DNA a RNA, de nucleótidos a nucleótidos • La molécula de RNA que se forma es un RNA mensajero (mRNA) • Es mensajero porque lleva la información desde el DNA hasta el ribosoma donde se sintetizan las proteínas • En los organismos eucariota la transcripción ocurre en el núcleo y en los procariota ocurre en el citoplasma Traducción es el proceso mediante el cual se sintetiza un polipéptido partir del mRNA • Hay un cambio en lenguaje, de nucleótidos a amino ácidos • Ocurre en los ribosomas • El proceso es diferente en organismos eucariota y procariota Comparación entre procariotas y eucariotas: Procariotas: • La transcripción y traducción pueden ocurrir casi simultáneamente Dr. Iván Ferrer-Rodríguez Chap 17 1 Eucariotas: • Ambos procesos ocurren en compartimientos separados • Se forma primero un transcripto primario o un pre-mensajero • El pre-mensajero es procesado y se forma el mRNA En resumen, el DNA codifica para un mRNA y éste codifica para proteínas Comparación entre el DNA, RNA y proteínas Polímero: DNAÆ mRNAÆ Proteínas • Azúcar desoxirribosa ribosa no aplica • Monómero nucleótidos (4) nucleótidos (4) amino ácidos (20) • Cantidad cuatro: A,T,G,C cuatro: A,U,G,C veinte • Molécula ácido nucleico, ácido nucleico, polipéptido hebra doble hebra sencilla Transcripción en detalle: Hay 4 nucleótidos que codifican para 20 amino ácidos ¿Cómo ocurre esto? • Si un solo nucleótido codificara para un amino ácido, entonces se podrían Nucleótidos (4) Amino ácidos (20) codificar sólo 4 amino ácidos 4 • 41 • 42 16 • 43 64 • Si dos nucleótidos codificaran para un amino ácido, entonces se podrían codificar 16 amino ácidos • Si tres nucleótidos codificaran para un amino ácido, entonces se podrían codificar 64 amino ácidos • Esto permitió a los científicos descifrar que la información pasaba del DNA a las proteínas en forma de en tripletes • Por ejemplo, hacen falta 300 nucleótidos para sintetizar un polipéptido de 100 amino ácidos • A un triplete de bases en el mRNA se le llama codón Dr. Iván Ferrer-Rodríguez Chap 17 2 • Para un gene dado, sólo una hebra se transcribe y produce un mRNA • Esta hebra se llama la hebra templado porque provee el templado para la síntesis del mRNA • El mRNA es complementario al gene que lo codifica, no es idéntico • Cada nucleótido en el mRNA se añade por complementariedad, pero no hay timina (T) en el RNA, éste es sustituido por uracil (u) • El mRNA es sintetizado por una polimerasa de RNA, que sólo tiene actividad en dirección 5'Æ3' ¿Cómo se descifró el código genético? • En los años 60, Marshall Nirenberg realizó unos experimentos que ayudaron a descifrar el código genético • Sintetizó una molécula artificial de mRNA • Esta molécula era un polímero de nucleótidos conteniendo solamente uracil (un poli u) • Añadió el poli u a un tubo de ensayo con todos los componentes necesarios para la síntesis de un polipéptido • No importaba cuando detuviera la traducción, siempre obtenía un polipéptido conteniendo un solo amino ácido, fenilalanina (phe) • Así logro descifrar que uuu codifica para fenilalanina • Luego se fueron descifrando los codones restantes • Tienen que aprender a utilizar la tabla presentada en la Figura 17.4 • Hay 64 posibles codones • • 61 codones codifican para amino ácidos • 3 codones son señales para que finalice la traducción (UAA, UAG, UGA) El codón AUG tiene función dual: • Codifica para el amino ácido metionina • Es un codón de iniciación, da la señal para el ensamblaje del ribosoma y que comience la traducción Dr. Iván Ferrer-Rodríguez Chap 17 3 • El código es redundante, pero NO es ambiguo • Más de un codón pueden codificar para un amino ácido (redundante) • Un codón sólo codifica para un amino ácido (no es ambiguo) • ¿Qué amino ácido es codificado por el codón por UUU? • El código es casi universal (antes se decía que era universal), es casi igual para todos los organismos vivientes • CCG codifica para el amino ácido prolina en todos los organismos que se conocen • Por que todos los organismo usan el mismo código, es posible ponerle un gene de un organismo a otro • La Figura 17.5 muestra una planta de tabaco a la que le pusieron un gene de una luciérnaga y la planta libera energía en forma de luz • Para que se pueda producir una proteína a partir de un mRNA tiene que haber un marco de lectura abierto (Open Reading Frame = ORF) • Analizar la siguiente secuencia genética: DNA 3'TAC AAA GAG TAA CAC AGT GGG TGG CGT GTG TTT TCC ATT5' ¿Cuál es la molécula de DNA complementaria? ¿Qué RNA mensajero se produce de esta molécula de DNA? ¿Qué proteína se produciría a partir del mRNA? Las respuestas están en un cuadro al final de la clase Síntesis de mRNA en detalle (Figura 17.6) • Consiste de 3 etapas: iniciación, elongación y terminación • Una unidad de transcripción es el segmento de DNA que es convertido en mRNA • El promotor es la región del DNA a la que se pega la enzima para iniciar la transcripción. El promotor contienen el punto de iniciación ("start point") Dr. Iván Ferrer-Rodríguez Chap 17 4 • El punto de iniciación es la secuencia de nucleótidos donde comienza la síntesis de RNA • La iniciación incluye el enlace de la polimerasa al promotor, el que se desenrolle del DNA y el inicio de la síntesis de RNA en el punto de iniciación • La elongación incluye el movimiento de la polimerasa a través de la hebra de DNA en dirección 5'Æ3'; la enzima va abriendo el DNA y luego de la síntesis de RNA, el DNA se va reuniendo • La terminación ocurre cuando la polimerasa transcribe la secuencia terminador, que es una secuencia de nucleótidos que da la señal para que termine la transcripción; el punto de terminación es la secuencia de nucleótidos donde termina la transcripción • Al final de la transcripción, se libera el mRNA y la polimerasa se desmonta del DNA y el DNA regresa a su estado original La enzima responsable de la síntesis de RNA a partir del DNA es la Polimerasa de RNA • Sólo funciona en dirección 5'Æ3' • Los organismos procariota tienen una sola polimerasa de RNA para todos los RNA • • La polimerasa reconoce el promotor Los organismos eucariota tienen 3 polimerasas de RNA: I, II, III (polimerasa II sintetiza el mRNA) • La polimerasa se pega al promotor sólo si se pegan primero unas proteínas llamadas factores de transcripción • El promotor usualmente contiene una secuencia conocida como la caja TATA, a la cual se pegan los factores de transcripción (Figura 17.7) • Estos factores de transcripción junto a la polimerasa se conocen como el complejo de iniciación de transcripción Modificaciones post-transcripcionales en el RNA (Figura 17.8): En eucariotas el pre-mensajero es modificado antes de salir del núcleo • terminal 5' - gorro, tope, tapa (secuencia líder) - se le añade una trifosfato de guanosina • esto previene degradación del RNA por enzimas hidrolíticas Dr. Iván Ferrer-Rodríguez Chap 17 5 • • • ayuda a atarse al ribosoma terminal 3' - gorro, tope, tapa (secuencia remolque, "trailer") se le añaden a este terminal entre 30-200 adenina, el poli A • esto previene degradación del RNA por enzimas hidrolíticas • ayuda a atarse al ribosoma • facilita la exportación del mRNA desde el núcleo hasta el ribosoma La secuencia líder y la secuencia trailer no se traducen. Otro procesamiento del RNA (Figura 17.9): • En un mRNA eucariota pueden haber secuencias que codifican para amino ácidos llamadas exones y secuencias que no codifican para amino ácidos llamadas intrones • La Figura 17.9 muestra el gene de β-globina, una de las cadenas polipéptidas en la hemoglobina • tiene 146 amino ácidos • tiene 3 exones • tiene 2 intrones • Todo se transcribe y forma un RNA pre-mensajero • Antes del RNA pre-mensajero salir del núcleo, se remueven los intrones, se juntan los exones y se mantienen las secuencias no codificadoras líder y "trailer" • El algunos genes, diferentes exones codifican para dominios diferentes de la proteína (Figura 17.11) • Cada dominio de la proteína se dobla independiente • Cada dominio lleva a cabo una función diferente • El dominio se define como una región discreta estructural y funcional de la proteína, o en otras palabras, como un fragmento de la proteína que tiene características particulares Remoción de intrones (Figura 17.10) • Luego de la transcripción, el RNA inmaduro o transcripto primario conteniendo los intrones se combina con unas proteínas llamadas snRNPs (small nuclear Dr. Iván Ferrer-Rodríguez Chap 17 6 ribonucleoproteins) y otras proteínas para formar un complejo llamado "spliceosoma" • Estos snurps están compuestos de RNA y proteínas y están localizados en el núcleo de la célula • El RNA en el snRNPs reacciona con el RNA en los terminales del intrón, el intrón es cortado y removido y se unen los dos exones • Esta idea llevó al descubrimiento de las ribozimas, moléculas de RNA que funcionan como enzimas Síntesis de proteínas a partir del mRNA: • El concepto básico de traducción está dado en la Figura 17.12 • El mRNA va a ir hasta los ribosomas donde ocurre traducción • En la medida que el mRNA pasa a través del ribosoma, los codones son leídos uno a uno y traducidos en amino ácidos • La molécula que conecta el mRNA con el amino ácido se llama RNA de transferencia (tRNA) • El tRNA es como un intérprete que traduce de nucleótidos (mRNA) a amino ácidos (proteína) • Su función es transferir los 20 amino ácidos desde el lugar de almacenamiento en el citosol hasta el ribosoma • Las células sintetizan los amino ácidos a partir de otros compuestos o los reciclan de los alrededores • Consiste de ~ 80 nucleótidos que se dobla y generan una estructura tridimensional La estructura en detalle del tRNA está dada en la Figura 17.13 El tRNA tiene: • Terminal 3' - lugar de enlace con el amino ácido que va a ser transportado hasta el ribosoma • Terminal 5' • 4 regiones donde la molécula se torna de doble hebra • Tres lazos (loops) Dr. Iván Ferrer-Rodríguez Chap 17 7 • En uno de los lazos, el del lazo opuesto al terminal 3', hay una secuencia única de nucleótidos que es complementaria al codón, esta secuencia de tres nucleótidos se conoce como el anticodón • La secuencia del anticodón normalmente se escribe en dirección 3'Æ5' para que alinee adecuadamente con la secuencia del codón • Hay un tRNA específico para cada uno de los 20 amino ácidos y cada uno tiene un anticodón particular La unión de los amino ácidos con sus respectivos tRNA es catalizada por una enzima llamada sintetasa de aminoacil tRNA (Figura 17.14) 1) Lo primero que ocurre es que el sitio activo de la enzima enlaza el amino ácido y trifosfato de adenosina (ATP) 2) El ATP pierdo dos grupos fosfato (P) y se pega al amino ácido en forma de monofosfato de adenosina (AMP) 3) El tRNA correspondiente se enlaza al amino ácido y desplaza el AMP 4) La enzima libera el amino ácido activado (aminoacil tRNA) La anatomía del ribosoma en detalle está dada en la Figura 17.15 • • Tiene dos subunidades, una grande y una pequeña, compuestas por proteínas y RNA ribosomal (rRNA) • En los eucariotas estas subunidades se sintetizan en los nucleolos • Las subunidades se unen cuando se va a traducir un mRNA • Normalmente el rRNA es el tipo de RNA más abundante en las células Los ribosomas eucariotas y procariotas son similares en estructura y función, pero los ribosomas eucariotas son un poco más grandes y difieren un poco en composición • Algunos antibióticos como la tetraciclina y estreptomicina afectan los ribosomas procariota, pero no tiene efecto en los ribosomas eucariota • Esto es lo que permite que estos antibióticos puedan utilizarse para tratar infecciones bacterianas Dr. Iván Ferrer-Rodríguez Chap 17 8 • Tiene un lugar para enlazar al mRNA • Tiene tres lugares de enlace de tRNA llamados E, P, A • Un tRNA encaja bien en el sitio A cuando el anticodón y el codón engranan bien, aquí en este sitio estará el próximo amino ácido que va a ser añadido a la cadena • • En el sitio P mantiene el péptido creciente, aquí hay un tRNA conteniendo el péptido que se está formando • • La "A" implica que en este lugar se enlaza el amino ácido activado, (aminoacil tRNA), o sea el tRNA que tiene enlazado un amino ácido y que lo trae para añadirlo al péptido La "P" implica que en este lugar está el "peptidyl tRNA", el péptido Los tRNA salen del ribosoma a través del sitio E y van a buscar otros amino ácidos • La "E" implica que desde este lugar salen (" exit") los amino ácidos La traducción puede dividirse entres etapas: iniciación, elongación y terminación La iniciación de traducción está descrita en la Figura 17.17 • La iniciación reúne el mRNA, el tRNA cargando el primer amino ácido y las dos subunidades del ribosoma • Lo primero que ocurre es que se enlaza el mRNA con la subunidad pequeña del ribosoma • En eucariotas, el gorro en el terminal 5' (5' cap) le indica al ribosoma en que región del mRNA tiene que enlazarse • Luego se enlaza un tRNA con el anticodón UAC, que parea con el codón de iniciación AUG, que codifica para metionina • Se enlaza la subunidad grande, el tRNA inicial queda en el sitio P, el sitio A queda disponible para que se enlace el próximo tRNA • Este ensamblaje requiere energía que es suplida por GTP; además se requieren unas proteínas conocidas como factores de iniciación de traducción La elongación (Figura 17.18) Dr. Iván Ferrer-Rodríguez Chap 17 9 • Se van añadiendo amino ácidos, uno a uno • Para la adición de los amino ácidos se requieren energía (GTP) y unas proteínas llamadas factores de elongación • La adición ocurre en tres pasos que están descritos en la Figura 17.18: 1) Reconocimiento del codón - un aminoacil tRNA se enlaza al codón en el sitio A 2) Formación del enlace peptídico - el ribosoma cataliza la formación de un enlace peptídico entre el terminal amino del amino ácido nuevo y el terminal carboxilo del péptido creciente; luego del enlace el péptido creciente queda localizado en el sitio A 3) Transposición (translocation) - el tRNA en el sitio A, junto al mRNA se mueve al sitio P; el tRNA en el sitio P se mueve al sitio E y es liberado del ribosoma Terminación de traducción (Figura 17.19) 1) Cuando un codón de terminación llega al sitio A, una proteína llamada "factor de liberación", en lugar de un tRNA, entra al ribosoma 2) El factor de liberación rompe el enlace entre el tRNA en el sitio P y el último amino ácido que se añadió al polipéptido; el polipéptido se libera del ribosoma 3) Se disocian las dos subunidades del ribosoma y los otros componentes En la Figura 17.25 se muestra un resumen de transcripción y traducción en la célula eucariota • El Dogma central establece el orden en que fluye la información: DNAÆRNAÆProteínas • Un gene puede transcribirse muchas veces y un mRNA puede traducirse muchas veces • Algunos genes son convertidos en mRNA, pero NO en proteínas; algunos ejemplos son los genes que codifican para rRNA y tRNA • Transcripción y traducción son similares en eucariotas y procariotas; la diferencia principal es el procesamiento por el que pasa el mRNA en el núcleo de la célula eucariota Tienen que aprenderse la Tabla 17.1 "Tipos de RNA en la célula eucariota" Dr. Iván Ferrer-Rodríguez Chap 17 10 Las mutaciones pueden afectar la estructura y función de las proteínas • Las mutaciones son cambios en el material genético de una célula o un virus • Algunas mutaciones sólo afectan un par de bases en el gene (un nucleótido y el nucleótido en la hebra complementaria), por lo que se conocen como mutaciones de punto • Si la mutación de punto ocurre en un gameto, la misma puede transmitirse a la progenie • Si la mutación tiene un afecto adverso en el fenotipo de un humano o animal, la condición mutante se conoce como un desorden genético o enfermedad hereditaria • Un ejemplo de esto es la enfermedad "sickle cell", en la cual hay una mutación de punto en el gene que codifica para uno de los polipéptidos de la hemoglobina (Figura 17.23) • Hay una mutación de punto donde se cambia una A por una U • Este cambio provoca un cambio de amino ácidos, ácido glutámico es sustituido por valina • Se produce una hemoglobina alterada que provoca que la célula se torne con una depresión en el centro (ver figura 14.15) Hay diferentes tipos de mutaciones de punto: 1) Sustituciones - son mutaciones de punto donde ocurre un cambio en un solo par de bases (un nucleótido y su par complementario). Hay varios tipos (Figura 17.24): • Sustitución silente - es la que no cambia el amino ácido • Mutación con sentido equivocado - provoca cambio en el amino ácido • Mutación sin sentido - cambia el amino ácido por un codón de terminación 2) Inserciones y deleciones - es la adición o pérdida de uno o más nucleótidos en el gene • Éstas cambian el marco de lectura de los codones y se llaman mutaciones de cambio en el marco de lectura Dr. Iván Ferrer-Rodríguez Chap 17 11 • • Normalmente estas mutaciones tienen un efecto desastroso porque cambian el marco de lectura (ORF) en el mRNA Las mutaciones pueden ocurrir por: • errores en la replicación del DNA • errores en la reparación del DNA • recombinación genética • Estas mutaciones se conocen como mutaciones espontáneas • Hay agentes químicos y físicos que interactúan con el DNA y que provocan mutaciones; éstos se conoces como mutágenos; • Algunos ejemplos de mutágenos son: luz ultravioleta y rayos X Respuesta: DNA 5'ATG TTT CTC ATT GTG TCA CCC ACC GCA CAC AAA AGG TAA3' DNA 3'TAC AAA GAG TAA CAC AGT GGG TGG CGT GTG TTT TCC ATT5' mRNA 5'AUG UUU CUC AUU GUG UCA CCC ACC GCA CAC AAA AGG UAA3' Proteína met phe leu ile val ser pro thr ala his lys arg stop tRNA 3'UAC AAA GAG UAA CAC AGU GGG UGG CGU GUG UUU UCC stop5' Dr. Iván Ferrer-Rodríguez Chap 17 12