IV USCAP & AACR HIGHLIGHTS
Nombre y apellido del autor/a: JUAN JOSÉ RÍOS MARTÍN
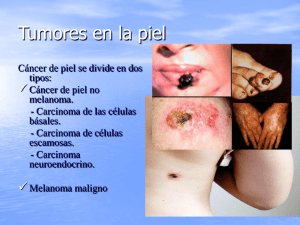
Título del trabajo: ACTUALIZACIÓN EN CARCINOMA DE CÉLULAS DE MERKEL
Resumen:
- El estudio inmunohistoquímico es necesario para realizar el diagnóstico del carcinoma
neuroendocrino cutáneo primario (CCM) ya que otras neoplasias de células pequeñas pueden simular
su cuadro histológico. INSM1 es un factor de transcripción expresado en tejidos con diferenciación
neuroendocrina y puede ser de utilidad frente a los marcadores citoplásmicos habituales. Los autores
estudian la expresión de INSM1 en 15 casos de CCM y 17 casos de diferentes neoplasias con
morfología similar al CCM. 14/15 casos (93%) expresaron el marcador nuclear resultando en un alta
sensibilidad y una fácil interpretación (1).
- En dos trabajos se pone de manifiesto que los CCM son genéticamente muy heterogéneos. El
primero de ellos (2) estudia una serie de 16 casos de CCM que no expresan citoqueratina 20 (este dato
se ha relacionado con una patogenia no asociada al poliomavirus), 10 de los cuales no muestran por
PCR la presencia del poliomavirus. La mayor tasa de mutaciones en genes relacionados con la
oncogénesis es encontrada en estos últimos, destacando que las alteraciones en los genes supresores
RB1 y TSC1 estaban restringidas a este grupo. El segundo de los trabajos (3) estudia 8 casos mediante
secuenciación completa del exoma y corrobora la gran variabilidad de genes mutados en esta
neoplasia. Mutaciones inactivadoras de RB y p53 aparecen en un número significativo de casos y
también activación de vías de señalización tales como WNT y JAK/STAT. Este último hallazgo podría
ser de interés para potenciales terapias dirigidas.
Referencias:
1 (489) Patrick S Rush et al. INSM1: A Novel Nuclear Marker in Merkel Cell Carcinoma (Cutaneous
Neuroendocrine Carcinoma)
2 (459) Angela MB Collie et al. Next Generation Sequencing of Cytokeratin-Negative Merkel Cell Carcinoma
3 (490) Roxana Sánchez Pacheco et al. Merkel Cell Carcinoma – Molecular Heterogeneity, as Revealed By
Integrated Genomic Analysis