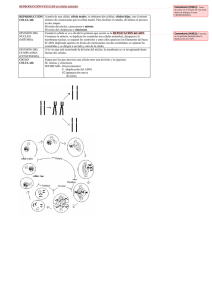
Inestabilidad Cromosómica - Universidad del Bío-Bío
Anuncio

1 “Principios de Genética Toxicológica” Marta A. Carballo y Marta Mudry ( Eds.) “Inestabilidad Cromosómica” por Enrique Zamorano – Ponce, D.Sc. Laboratorio de Genética Toxicológica (GENETOX), Departamento de Ciencias Básicas, Facultad de Ciencias, Universidad del Bío-Bío, casilla 447 Chillán-CHILE . Teléfono: 56-42-203075 Fax: 56-42-203046 e-mail: [email protected] 2 La idea que en estos momentos se tiene de la célula es muy distinta de la que se concebía hasta hace algún un tiempo atrás. Hoy, la unidad morfológica y fisiológica de organización de los seres vivos se nos muestra como una estructura dinámica, en la que simultáneamente se llevan a cabo decenas de procesos en distintos lugares dentro de ella, los que regulados por complejos mecanismos de control, permiten el funcionamiento equilibrado de la misma: “Cuando se observa una célula bajo el microscopio, parece sosegada. Pero debajo de esa quietud externa se esconde una frenética actividad bioquímica”. (Lau y Bartel, 2003) Efectivamente, mientras en el citoplasma, una compleja maquinaria molecular puede por ejemplo- estar traduciendo los mensajeros para la síntesis de proteínas como Ku70, Ku80, Artemisa y Xrcc4, involucradas tanto en fenómenos de reparación de fracturas de ADN de hebra doble, como en la protección (capping) de telómeros (Bailey y Goodwin, 2004); en el interior del núcleo estarán probablemente ocurriendo -en simultáneo- otros procesos complejísimos como la interacción de condensina II con el ADN para contribuir con los primeros pasos en el ensamblaje cromosómico (Ono, et al., 2004), o el secuestro de un ARNm por ADN no codificador para promover una regulación epigenética de la expresión de la información genética (Wayt Gibbs, 2004). Es decir, la célula es un todo coordinada y evoca en su diseño estructural y funcional los conceptos de unidad, ya que es un todo y el de unicidad ya que es única. Sin embargo, diversos factores alteran el patrón normal de regulación de señales internas e inducen a la célula a expresar un fenotipo inestable, que se evidencia desde un punto de vista citogenético como anomalías en la estructura o en el número de cromosomas del individuo. A este fenómeno se lo ha denominado colectivamente como inestabilidad cromosómica CIN (del inglés: Chromosome INstability), un apartado de importancia dentro del gran tema de la inestabilidad genómica. A casi 30 años desde que Nowell (1976) acuñara el término tras analizar células neoplásicas en las que el fenotipo CIN es un biomarcador frecuente, el cúmulo de descubrimientos en torno a las bases biológicas que explican el fenómeno avanza rápidamente y las causas, que hace algunos años se veían como inconexas, en estos momentos empiezan a relacionarse entre sí (Gollin, 2004). 3 Toda causa, por definición lleva a su efecto. La causa inmediata del fenotipo CIN debe encontrarse en algún accidente o conjunto de ellos que inducen a la célula a expresar anomalías cromosómicas de tipo estructural o numérica. En este capítulo se analizan algunas de esas causas y en torno a ello se relevan las publicaciones más recientes y significativas entorno al tema. ¿Qué es la inestabilidad cromosómica? Puede ser definida como un estado de formación continua de nuevas mutaciones cromosómicas, a un grado más elevado que en células normales (Gisselsson, 2003). Entre las causas que han sido involucradas en la aparición de fenotipo CIN, en este capítulo se describen las siguientes: i fallos en puntos de control del ciclo celular, ii anomalías centrosómicas numéricas o estructurales, iii defectos en la segregación, iv malfuncionamiento de telómeros y v recombinación homóloga vinculada a reparación de roturas de ADN de hebra doble. A. Fallos en los puntos de control del ciclo celular: Los notables cambios que acontecen en la célula, particularmente en el núcleo durante las diferentes etapas del ciclo celular proliferativo, son ejemplos notables de la gran precisión arquitectónica y de geometría biológica subyacente a la organización espacial del genoma. En el proceso de cuatro etapas por las que atraviesa un cromosoma durante el ciclo celular, incluyendo: duplicación, condensación, segregación y descondensación; cada una de ellas representa un factor determinante de igual importancia a la hora de entender los delicados mecanismos que controlan el tránsito a través de las diversas etapas del ciclo celular, así como las causas que pueden explicar el fenómeno de descontrol que afecta –bajo determinadas circunstancias- el normal desarrollo del proceso, expresándose durante la mitosis siguiente como alteraciones cromosómicas de tipo estructural o numéricas. Tradicionalmente las diversas manifestaciones del fenotipo CIN de tipo numérico se ha atribuido a fallos en el punto de control entre Metafase y Anafase, sin embargo un número importante de evidencias indican que puede no ser esa la única causa y que fallos en los puntos de control G1-S y G2-M pueden explicar un fenotipo CIN de índole numérico (aneuploidía), cosa que recién empieza a entenderse gracias al aislamiento e identificación de nuevas proteínas 4 que hasta hace poco eran desconocidas y que de alguna manera se las está relacionando a la etiología del fenotipo CIN. Se sabe que el avance a través de las diversas etapas del ciclo celular es regulado de manera muy estricta. La división celular normal es un proceso que avanza coordinado con el crecimiento celular, mientras que la iniciación de la síntesis de ADN o fase “S” se ordena de tal manera de prevenir una amplificación inapropiada del ADN que pueda ocasionar inestabilidad en el genoma. Para asegurarse de que se cumple con esos importantes requerimientos divisionales, las células han desarrollado un mecanismo de supervivencia basado en una intrincada red de vías de señalización por quinasas, que conducen a estos “puntos de control” durante el desarrollo del ciclo celular proliferativo. Se ha identificado tres puntos de control en las transiciones siguientes: i G1 – S, ii G2 – M y iii Metafase y Anafase. i. G1 – S El punto de transición G1-S ha sido muy bien estudiado en levaduras en las que se ha identificado un punto de inicio denominado START (o Punto de Restricción en células de mamíferos) que requiere de un Factor Promotor de S (FPS), constituido por el producto del gen CDC28 (S. cerevisiae) o cdc2 (S. pombe); una quinasa dependiente de ciclina y ciclina. El FPS es entonces análogo al Factor Promotor de Mitosis (FPM) que, como veremos más adelante, controla el ingreso en M. En levaduras existe una sola quinasa que participa en los dos puntos de control, en tanto que en células de mamíferos se ha identificado a dos distintas: una para cada punto. En START una ciclina G1 se une a CDC28/cdc2quinasa y forma el FPS activo. La Cdk entonces fosforila proteínas claves que se necesitan para el progreso dentro de S. Una vez que las ciclinas han activado a Cdk, el nivel de ciclinas decrece como resultado de una proteolisis dependiente de ubiquitina 1. (Fig 1) Hay que decir, sin embargo que las células en un organismo multicelular se enfrentan a un conjunto distinto de problemas que una levadura o una célula huevo fertilizada. En un organismo multicelular, algunas células se reproducen rápido, otras lo hacen lento y la salud 1 Proteolisis dependiente de ubiquitina. La ubiquitinación de una proteína es una herramienta muy versátil que poseen las células eucariontes para controlar la estabilidad, funciones y localización subcelular de proteínas específicas. La proteolisis dependiente de ubiquitina es la inactivación de una proteína por unión a ella de ubiquitina (ub), una pequeña molécula que actúa como una etiqueta que es reconocida por una maquinaria proteica que transporta la proteína ubiquitinada hacia un proteosoma para su degradación. APCactivo (a) APC une ubiquitina a securina del complejo cohesina (b) Securina ubiquitinada es degradada por separasa (c) procede separación de las cromátidas b Cdc20 APCinactivo a Cdh1 APC/Cdh1activo c G0 G1 Anafase Skp2 Metafase M Skp2 degradado Prometafase Profase No se ingresa en “S” Ciclo Celular FPS Degradación mediada por ubiquitina FPM Cdc25 S G2 Complejo Cohesina. Mantiene unidas las hebras de ADN tras su síntesis en “S”. Ciclina mitótica Punto de control G1 -S Complejo Cohesina Cdk mitótico Punto de control G2 -M Securina Ciclina mitótica degradada Punto de control M - A Securina degradada Ciclina G1 Cromátidas hermanas Ubiquitina Cdk-G1 Ciclina G1 degradada Cdh1 Figura 1. Modelo General de Regulación del ciclo celular. Los punto de control G1 – S y G2 -M se lleva a cabo por activación de complejos proteicos constituidos por Ciclina y Cdk. Para la activación de Cdk se requiere de unión a ciclina cuya disponibilidad (niveles intracelulares) se controla por regulación transcripcional y/o por proteolisis específica. Las ciclinas son blanco de degradación dependiente de ubiquitina por la vía de proteosomas. SCF reconoce ciclinas como substrato cuando ellas están fosforiladas, mientras que APC agrega ubiquitina a ciclinas cuando factores específicos son activados por fosfatasas. 5 general del organismo requiere que ninguno de esos tipos celulares predomine. Ello hace que las células deban no sólo responder a señales internas, sino también a señales que provienen del entorno y que tienen que ver con el contacto entre las células, factores de crecimiento, hormonas, etc., de tal forma que si bien, el entramado regulatorio del ciclo celular en organismos multicelulares sigue vías muy parecidas a las que conocemos en levaduras, no cabe duda de que el nivel de complejidad y de regulación es significativamente mayor. Existen ejemplos claros de proteínas regulatorias que participan directa o indirectamente en la regulación del tránsito por el ciclo celular en células de mamíferos y que se las ha relacionado con algunos tipos de fallos que conducen a la aparición de fenotipo CIN. Dado que la adquisición de un volumen determinado y la expresión de una organización interna precisa, son cruciales como conditio sine qua non para que la célula ingrese en “S”, adquieren relevancia especial la familia de genes que codifican para GTPasas Rho, incluyendo a Cdc42 además de Rho y Rac, involucradas todas en la morfogénesis celular, al inducir ciertos tipos de actinas citoesqueletales y regular el correcto alineamiento y estabilización de microtúbulos durante la interfase del ciclo celular (Kamai et al., 2004). Se ha demostrado recientemente que Cdc42 y su efector Mdia3 están involucrados también en la bi-orientación y estabilización de la unión de microtúbulos al cinetócoro del cromosoma (Yasuda et al., 2004), así como en la alineación y segregación de los cromosomas. De tal forma que fallos en su regulación, induce a la aparición de fenotipo CIN que puede conducir ya sea a tumorogénesis o apoptosis (Narumiya et al., 2004; Nojima, 2004). Es decir, se inicia la identificación de genes, cuyos productos tienen que ver con la morfogénesis celular durante G1 e involucrados en procesos de unión de los microtúbulos a los cinetócoros de los cromosomas, de tal forma que su mal funcionamiento puede traer consigo para la célula problemas no sólo de índole morfológica, sino además funcionales que impedirán un normal proceso de segregación cromosómica durante la anafase de la mitosis siguiente. Se sabe que las transiciones en el ciclo celular son conducidas por ondas de degradación de reguladores del ciclo dependiente de ubiquitina. Los complejos SCF (del inglés: Skp1-Cullin-F-box) y APC/C (Complejo Promotor de Anafase, del inglés APC por: Anaphase Promoting Complex) o también llamado Ciclosoma, representan dos clases de ligasas de ubiquitina cuyas actividades regulan primariamente las transiciones G1-S y Metafase-Anafase respectivamente. Los blancos principales del complejo SCF son: p21 y p27, dos inhibidores de Cdk (quinasa dependiente de ciclina) durante la fase S; mientras que el blanco principal -pero 6 no único- del complejo APC/C es Securina, una subunidad del denominado complejo cohesina cuya ubiquitinación por APC/C lo hace blanco de Separasa o Separina que la degrada catalíticamente permitiendo la separación de las cromátidas hermanas durante la anafase. APC en este caso se une a un activador específico, Cdc20, para poder llevar a cabo su función. Sin embargo, si bien la función de APC/C-Cdc20 en la transición metafase-anafase es relativamente clara, existe un número creciente de evidencias que señalan que APC/C conteniendo a un segundo activador llamado Cdh1, (APC/C-Cdh1), también juega un rol importante en la fase G1 del ciclo celular (Ang et al., 2004; Sudo et al., 2004). Se ha demostrado que la oncoproteína Skp2 (un activador de S ubicado en la caja F) promueve la transición G1-S actuando como factor de especificidad para SCF de tal forma que actúe sobre reguladores claves para que éstos sean degradados por proteosomas. Skp2 es una proteína lábil cuyos niveles oscila de una manera dependiente del ciclo celular. La acumulación de Skp2 se presenta desregulada a menudo en células cancerosas.. Se ha demostrado además que la acumulación de SCF-Skp2 precisa una inactivación previa de APC/C-Cdh1 (Wei et al., 2004) lo que se ha confirmado a través del silenciamiento de Cdh1 por ARNi2 (Bashir et al., 2004). (fig 1). Estos dos últimos estudios ponen en evidencia nuevos mecanismos de control en los cuales la maquinaria mitótica se comunica con proteínas que regulan el tránsito a través de la fase G1 del ciclo celular Papin et al., 2004). Otras líneas de evidencias indican que el correcto inicio de la duplicación de los centrosomas, que comienza precisamente durante la transición G1-S, es un factor determinante para lograr el ingreso en S. Se ha visto que fallos en este punto de control que lleva a fenómenos de amplificación de centrosomas, induce la aparición de fenotipo CIN (Farrugia et al., 2004). Elegantes experimentos realizados en el laboratorio del profesor Hinchcliffe, en la Universidad de Notre Dame (USA), han demostrado que la extracción de los centrosomas a partir de células de mamíferos en cultivo mediante microcirugía, induce la aparición de carioplastos, que son incapaces de ingresar en S. Ello parece indicar la importancia de la presencia de centrosomas en el inicio de la fase S (Hinchcliffe, 2003) y es una evidencia 2 El ARNi o ARN de interferencia constituye un nivel de regulación génica descubierto hace pocos años en células eucarióticas. Se basa en que la presencia de ARN de doble hebra en la célula, elimina la expresión de un gen que posee la misma secuencia. Se trata de una vía altamente conservada de silenciamiento génico postranscripcional (Scherr et al., 2004) en la cual la nucleasa “Dicer” corta RNA de doble hebra en pequeñas piezas de RNA de interferencia o RNAi (Fukagawa et al., 2004) Descrito sólo en el año 1998, hoy a este proceso se lo considera como una herramienta importante para estudiar la función génica en células vivas (Kent et al., 2004, terapia génica (Ichim et al.,2004), expresión génica durante el desarrollo (Maine, 2004). 7 adicional de la compleja orquestación de actividades que se lleva a cabo durante el progreso a través de las etapas del ciclo celular, particularmente en la transición G1-S que recién empiezan a entenderse Gollin (2004) y cuyos fallos en su regulación pueden explicar fenotipos de inestabilidad cromosómica al interferir con el normal proceso de detención que experimenta cualquier célula al detectarse fallos en procesos claves que tienen relación con la transmisión fidedigna de la información genética a la descendencia que es –diríamos- uno de los propósitos claves en la mantención de una delicada homeostasis en el proceso de reproducción celular . ii. G2 - M Otro intrincado sistema bioquímico de control ha sido descrito en la transición G2 / M (Fig 1). El clásico modelo de control en este punto indica que también se trata de una proteína quinasa Cdk que se une a ciclina B para formar un complejo denominado como “Factor Promotor de Mitosis”. La activación del FPM depende de Cdc25, una fosfatasa que elimina grupos fosfatos inhibidores unidos a aminoácidos ubicados en el sitio clave para el funcionamiento de la Cdk como es su sitio activo. De esa forma, se activa el complejo que va a fosforilar una serie de substratos como la histona H1 involucrada en la condensación cromosómica, laminina ubicada en la lámina nuclear y cuya fosforilación gatilla la disolución de la envoltura nuclear, entre otros. Sin embargo, hoy ya se sabe que el punto de control gatilla la señal de ingreso, sólo si el funcionamiento de otros genes es el adecuado. Investigaciones orientadas a encontrar reguladores de estos puntos de control han permitido identificar al gen de control CHFR (del inglés: checkpoint with forkhead-associated and ring finger) que coordina los primeros estadíos de la mitosis, deteniendo la condensación cromosómica en respuesta a un estrés mitótico e interactuando con Plk-1 (polo- like kinase) para cumplir su trabajo (Erson, 2004). Plk-1 realiza múltiples funciones esenciales durante el ciclo celular. Se ha visto que aquellas células deficientes en Plk-1 son incapaces de separar sus centrosomas, fallan en formar un huso bipolar y experimentan detención en prometafase procesos dependientes de Mad-2 / BubR1 (Van Vugt et al., 2004). (para revisión de Plks el lector puede ir a: Barr et al., 2004). Por otro lado, el silenciamiento de CHFR ha sido asociado con metilación aberrante en su sitio promotor y desacetilación de histonas, dos modificaciones epigenéticas que lo inactivan y pueden originar fenotipos de inestabilidad cromosómica (Kang et al., 2004). 8 Estudios muy recientes han permitido identificar además a otras proteínas que también participan en el punto de control de tránsito G2/M como: Aurora-A y Nek2. Las quinasas Aurora, de las cuales se han identificado tres familias Aur A, Aur B y Aur C, corresponden a una nueva familia de quinasas de serina y treonina que poseen un sitio catalítico altamente conservado. Se encuentran localizadas en los centrosomas de células interfásicas, en los polos del huso mitótico y en el cuerpo medio el aparato mitótico. Los substratos identificados para aurora quinasa A y B son: la proteína motora parecida a la kinesina (involucrada en el movimiento cromosómico), proteínas del huso mitótico, histona H3, proteínas cinetocóricas y la proteína supresora de tumores p53. Se ha demostrado que estas proteínas aseguran la formación de un huso bipolar, correcta segregación de cromosomas y la completación de la citocinesis (Crane et al., 2004). Su regulación se lleva a cabo ya sea por fosforilación, por unión de moléculas específicas o por proteolisis dependiente de ubiquitina. La desregulación de las quinasas Aurora induce fallos en el ensamblaje del huso bipolar, en la función de control G2/M y en la división celular originando malsegregación de cromosomas individuales o poliploidización acompañado por amplificación centrosómica (Meraldi et al., 2004 a). Estas evidencias sugieren fuertemente que la regulación eficiente de ciertas proteínas importantes por su implicación en los mecanismos de control de ciclo celular, están de alguna manera involucradas también en la correcta segregación de los cromosomas durante anafase y que la desregulación de su expresión puede inducir a la célula a mostrar fallos en los procesos que aseguran la segregación equitativa de los cromosomas entre las células hijas y con ello, a originar desbalances numéricos muy comunes por lo demás en células tumorogénicas. En los próximos años seguramente se identificarán otras proteínas involucradas en estos procesos y se empezará a armar un puzzle hasta ahora no terminado sobre las complejas causas que inducen a la célula a expresar inestabilidad cromosómica (Patel et al., 2004). iii. Metafase-Anafase Durante la división celular en eucariontes, la célula primero duplica su ADN en la fase S y después empaca el ADN en cromátidas hermanas en mitosis y entonces segrega los cromosomas entre las células hijas. La correcta separación de las cromátidas hermanas descansa en un delicado equilibrio de dos procesos opuestos: la cohesión entre las cromátidas hermanas y las fuerzas de separación que promueven las fibras del huso mitótico. La cohesión entre las cromátidas hermanas es establecida por un complejo proteico de Cohesina, durante la duplicación del ADN y persiste hasta la segregación cromosómica (Fig 1). En la mitosis, las 9 cromátidas hermanas se unen a las fibras del huso mitótico a través de los cinetócoros; complejos proteináceos asociados con ADN centromérico. Es vital para las cromátidas hermanas unir a los microtúbulos que emanan desde los dos polos de la célula (bi-orientación), estableciéndose un fenómeno de tensión a lo largo de los dos cinetócoros de un par de cromátidas hermanas. Después de que todas las cromátidas hermanas se encuentran biorientadas y sus cinetócoros se encuentran en tensión, una gran ligasa de ubiquitina E3 llamado el Complejo Promotor de Anafase o Ciclosoma (Melloy y Holloway, 2004), parecido a FPS pero con un conjunto específico de subunidades (Fig 2), en asociación con uno de sus cofactores de unión a substrato Cdc20 que se activa a fines de metafase, adiciona ubiquitina a la proteína Securina. La destrucción catalítica de la subunidad Scc1 del complejo cohesina por la separasa, destruye la cohesión entre las cromátidas hermanas y gatilla la etapa de anafase. (Fig 3). Los microtúbulos del huso ayudados por proteínas motoras de localización cinetocórica como CENP-E, una proteína parecida a kinesina, tiran los dos conjuntos de cromátidas hacia polos opuestos de la célula y como resultado cada célula hija recibe información genética idéntica (Amon, 2001; Uhlman, 2004; Hagstrom y Meyer, 2004). Recientemente se ha demostrado que en levaduras Ipl1, una proteína quinasa parecida a proteínas de la familia Aurora, se requiere para establecer la tensión y la biorientación de las cromátidas hermanas. Interesante resulta el hecho que esta misma proteína también está implicada en el punto de chequeo del huso. En particular IpI1 puede detectar falta de tensión en los cinetócoros. Intriga el hecho que la misma proteína requerida para el proceso de biorientación sea también responsable de controlar el estado de esa biorientación en estados posteriores del ciclo celular. Recientemente se ha demostrado que Aurora B de mamíferos, análogo a IpI1 de levaduras, está involucrada en la unión bipolar de los cromosomas y en una correcta función del punto de control del huso mitótico, aunque hasta hoy no se ha dilucidado cómo puede ello ser controlado. Se piensa que el mecanismo de detección de tensión a nivel cinetocórico debe involucrar a CENP-E. Además, la falta de tensión puede también ser detectada por un subconjunto específico de proteínas de chequeo como IpI1/ AuroraB y Mps1. No se sabe cómo estas proteínas podrían estar interactuando con las otras proteínas de control que se sitúan río debajo de este mecanismo propuesto (Fig.3) Los componentes moleculares de este punto de control mitótico fueron identificados inicialmente en S. cerevisiae y posteriormente se encontraron proteínas de control homólogas en otros organismos, incluidos mamíferos. Esas incluyen a proteínas de la familia Mad (del Apc4,5,7,10 Cdc16,23,27 Cul1 Skp1 Roc1 Cdc20 Se une a blancos tempranos substrato Apc11 Apc1 Cdc34 Cdc4p Apc2 Cdhl1 se une a blancos tardíos Cdh1 Cdc20 Ubc10 Substrato Cdc4p SCF APC Figura 2. Constitución multimérica de los complejos SCF (Skp1.Cullin-F-box) regulador del tránsito G1 –S en comparación a APC (Anaphase Promoting Complex). SCF reconoce a ciclina como substrato, mientras que APC ubiquitina ciclinas cuando se activan factores específicos.(tomado de Tyers and Jorgenson, 2000). Skp (del inglés S-phase kinase associated Proteins): corresponden a una familia de proteínas estructuralmente relacionadas que fueron identificadas por primera vez debido a su habilidad de unirse a ciclinas, que comparten un dominio en común que se une específicamente a proteínas de la caja F (F-box). Cullin corresponden a un dominio de proteínas estructuralmente relacionadas que fueron descubiertas por su habilidad de unirse a ciclinas, juegan un rol importante en la regulación del ciclo celular formando parte del complejo SKp1-Cullin-F-box (SCF). Las proteínas del dominio F-box, corresponden a una familia de proteínas que comparten el dominio F box en común, involucradas en interacciones proteína-proteína, juega un rol importante en la ubiquitinación de proteínas vía unión a proteínas del dominio Skp. Cinetócoro sin tensión de ocupación CENP-E Aurora B / lpl 1 Bud R1(Mad3 )- Bub3 Rod - Zw 10 Mps1 Bub1-Bub3 Mad1 - Mad2 P BubR1- Bub3 - Cdc20 MCC Mad2- Cdc20 APC-Cdc20 Degradación vía proteosoma Ub Ub Securina Ub Separasa cohe sin Separación de cromátidas hermanas Figura 3. Vía del control de huso. Esta vía es activada por la inexistencia de microtúbulos y de tensión en el cinetócoro cromosómicos. El mecanismo de detección del defecto es desconocido pero puede involucrar a Aurora B y CENP-E. Este último interactúa con BubR1 pero se desconoce cómo afecta la función de BubR1. En levaduras Aurora B puede actuar río arriba de Mps1 en la detección de la tensión. Se desconoce si es CENP-E o Aurora B la que regula Bub1-Bub3. En humanos Bub1 fosforila in vitro a la proteína humana Mad1. Así Mps1 y Bub1-Bub3 deben estar localizadas en su acción río arriba en relación a Mad1Mad2. Bajo la activación del punto de control ambos, esto es BubR1-.Bub3 y Mad2 interactúan con Cdc20 directamente inhibiendo su capacidad de activar a APC/C. La interacción pasajera entre BubR1-Bub3-Cdc20 y Mad2-Cdc20 lleva a la formación del complejo BubR1-Bub3-Mad2-Cdc20 (Complejo MMC) que es un inhibidor más eficiente de APC/C. Cuando todos los cinetócoros están unidos bipolarmente el punto de control es inactivado y APC ubiquitina a securina. La degradación de securina permite la activación de separasa o separina que rompe a Scc1, una subunidad del complejo cohesina que mantiene unida a las cromátidas hermanas. Con ello se gatilla la segregación de los cromosomas y con ello se inicia la anafase. (Tomado de Yu, 2002) 10 inglés: Mitotic arrest deficient), como: Mad1, Mad2, Mad3 / BubR1, Bub1, Bub 2, Bub3 y Mps1 (Chang y Yen, 2003). Con excepción de Bub2, esas proteínas forman una compleja red de señalización para bloquear la actividad de APC/C-Cdc20 de unirse a proteínas claves para su degradación mediada por ubiquitina (Sudakin y Yen, 2004), previniendo de esa forma una segregación cromosómica precoz y asegurando así una repartición segura del material genético (Bharadwaj y Yu, 2004). En levaduras, Bub2 parece no estar involucrado en la regulación de APC/C-Cdc20 y en la segregación cromosómica. En vez de ello, esta proteína regula negativamente las funciones de la red de salida de mitosis (Mitotic Exit Network o MEN) inhibiendo a APC/C-Cdh1 que media la degradación de ciclinas mitóticas y otros importantes reguladores del ciclo celular. Un único cinetócoro no unido a microtúbulo dentro de una célula es suficiente para activar el punto de control del huso. Así, debe producir una señal inhibitoria que difunde para bloquear la actividad del APC/C en la célula. Recientes estudios genéticos, bioquímicos y estructurales han iluminado la naturaleza de esta señal difusible que inhibe al APC/C. En la visión actual plasmada en dos modelos, el APC/C es inhibido a través de unión estequiométrica directa al complejo de control mitótico (MCC) que contiene BubR1, Bub3, Mad2 y Cdc20. Pudiese el MMC estar organizado en dos subcomplejos: BubR1-Bub3-Cdc20 y Mad2-Cdc20. Los dos modelos difieren en el mecanismo de ensamblaje del MCC. En el primer modelo (Fig 4), un cinetócoro no unido cataliza la formación de los complejos inhibidores de control que pueden difundir para inhibir al APC/C. La señal difusible puede ser MMC o bien alguno de sus dos subcomplejos. En el segundo modelo, el MCC está presente a través del ciclo celular y su formación no ocurre en los cinetócoros. Bajo la influencia de la activación del punto de control, una señal aún no identificada, llega al cinetócoro y sensibiliza al APC/C para permanecer inhibido por MCC. Se ha encontrado efectivamente que el complejo APC/C es inhibido por Mad2 cuando los cromosomas aún no están unidos a las fibras del huso, actuando como un supresor génico ya que las células MAD2 +/- entran en anafase y muestran inestabilidad cromosómica (Michel et al., 2004). Empleando ARNi para MAD2, se ha encontrado una severa reducción de los niveles de esta proteína, lo que resulta en fallos mitóticos, y muerte celular significativa por la formación defectuosa del huso mitótico, condensación cromosómica incompleta y salida prematura de Mad2 Mad2 Mad1 Bub3 Bub3 Cdc2 Mad1 Bub1 Mad2 Bub1 Mad2 Cinetócoro unido Mad1 Mad1 CENP-E P Bub3 Mad2 M i c r o t ú b u lo CENP-E Cinetócoro no- unido P Cdc20 BubR1 MCC Bub3 BubR1 Bub3 BubR1 Mad2 Cdc20 APC Cdc20 ? Figura 4. APC Bub + BubR1 + Mad2 Activo APC Inactivo Prometafase Cdc20 Metafase Anafase Un modelo para la formación de los complejos inhibidores de APC en el cinetócoro. En este modelo Mad2 es reclutada en el cinetócoro por Mad1 y Cdc20 es separada del cinetócoro por Bubr1-Bub3. Mad 1 también gatilla cambios de conformación en Mad2. Aún no se ha determinado el mecanismo por el cual Mad2 se disocia de Mad1. La fosforilación de Mad1 por Bub1 o Mps 1, podría jugar algún papel en este proceso. Mad2 disociado de Mad1 podría retener alguna conformación de unión más afín con Cdc20 que se ha unido a Bubr1-Bub3 conformando el MMC. Este complejo de control mitótico o sus subcomplejos podrían difundir lejos del cinetócoro para asociarse con APC/C e inhibir su actividad. Cuando Los cinetócoros son capturados en su totalidad por los microtúbulos del huso Mad1 y Mad ya dejan de localizarse en los cinetócoros. Las concentraciones de BubR1-Bub3 y otras proteínas de control también disminuyen en su localización cinetocórica. El rompimiento de MMC conduce a la activación de APC/C, degradación de securina y separación de las cromátidas hermanas (Tomado de Yu 2002) 11 mitosis conduciendo a multinucleación. Se sabe que la ciclina B es degradada prematuramente en células tratadas por breve período con RNA de MAD2 pero no en células MAD +/-, lo que sugiere una explicación a la malformación de huso y catástrofe mitótica que se observa en células knockout para MAD2. (Michel et al, 2004). Otro inhibidor del complejo APC/C ha sido recientemente descrito en células HeLa y se ha llamado Emi1 (del inglés Early Mitotic Inhibitor 1) que interfiere con el funcionamiento del complejo APC/C. Los niveles de Emi1 oscilan en el ciclo celular. Se acumula en fase “S” y es rápidamente degradado en prometafase (Moshe et al., 2004). Se ha evidenciado que Emi1 secuestra a Mad2 y evita así la activación del APC/C La degradación de Emi1 en mitosis temprana, llevada a cabo por el complejo ubiquitin-ligasa o SCF , es necesaria para la activación del complejo APC/C. Plk1 que se acumula durante la mitosis estimula marcadamente la unión de Emi1 a APC/C. Por otro lado, el complejo APC/C es activado por dos proteínas regulatorias: Cdc20 y Cdh1. Al respecto, se ha visto que la activación del complejo APC-Cdh1 juega un rol crucial tanto en la detención de G1 dependiente del inhibidor Cdk y en la detención en G2 inducida por daño en el ADN (Sudo et al., 2004). Hallazgos muy recientes indican que bajo ciertas circunstancias Mad2 adopta dos conformaciones de plegamiento en equilibrio (llamadas N1Mad2 y N2-Mad2). N2-Mad2 es un inhibidor más potente del complejo APC/C. Se ha visto que la sobreexpresión de un mutante de Mad 2 que específicamente secuestra N2-Mad2, bloquea parcialmente las señales de control en células vivas. Por otro lado las dos conformaciones de mad2 se interconvierten lentamente in vitro. Aparentemente este doble estado conformacional es crítico para el punto control del huso (Luo, 2004). B. Anomalías centrosómicas El centrosoma es un organelo esencial en las células animales constituido por una pareja de centríolos y el denominado “Material PeriCentriolar (MPC) que involucra a diversas proteínas, algunas de las cuales ya empiezan a ser identificadas y caracterizadas en: C. elegans (Pelletier et al., 2004), en Drosophila (Martínez-Campos et al., 2004) y humanos (Kim et al., 2004; Thompson et al., 2004). El MPC ha sido descrito ya sea como una nube de material que rodea a todo el centrosoma o como un racimo de proteínas dividido en dos subconjuntos, unas que adhieren a la superficie lateral de los centriolos y otro que se extiende hacia fuera de 12 esta región a modo de una nube de material. Investigaciones recientes indican un tercer modo de organización que corresponde a un subconjunto de proteínas del MPC que se integran entre ellas para organizar un tubo (PCM tube) con un extremo abierto y otro cerrado y que se duplica en coordinación con la duplicación del centrosoma. Respecto a este último tipo de organización del MPC, se ha documentado que: i el tubo de MPC representa una porción del MPC asociada directamente con el centriolo, ii el tubo de MPC tiene una asociación específica y reproducible con la estructura polar del centriolo, iii el tubo es un sitio de organización microtubular citoplasmático y posee una estructura que influencia el patrón de ensamblaje inicial de microtúbulos dentro de la región yuxta-centriolar y iv el tubo de MPC posee una relación estructural con el centriolo, lo que permite la expresión simultánea de las funciones de éste y del MPC; por ejemplo ciliogénesis, organización de microtúbulos citoplasmáticos (Ou et al., 2003). Al centrosoma se lo ha denominado como el Centro Organizador de Microtúbulos (COMt), ya que desde ese lugar se produce la nucleación de microtúbulos para organizar el citoesqueleto en células interfásicas y, el huso bipolar divisional. Se duplican sólo una vez por célula en cada ciclo celular de una manera semiconservativa (Fig 5 ) , de tal modo que errores en este proceso pueden conducir a la aparición de fenotipo CIN reflejado en la formación de husos aberrantes o en la segregación errónea de cromosomas (Nigg, 2003). Se ha demostrado que la amplificación anormal de los centrosomas es una causa principal de defectos mitóticos en células cancerosas (D’Assoro et al, 2002). La presencia de más de dos centrosomas (amplificación centrosómica) induce defectos en la formación de un huso bipolar normal con un consiguiente incremento en la frecuencia de errores en la transmisión cromosómica. (Fukasawa, 2002). Existen varios mecanismos responsables de la amplificación centrosómica, incluyendo duplicación descontrolada de los centrosomas y bloqueo de citocinesis seguido por el tránsito hacia el próximo ciclo celular (Fukasawa, 2002). El efecto de agentes genotóxicos como la irradiación y la exposición a ciertos agentes quimioterapéuticos inducen amplificación centrosómica tanto directamente o induciendo alteraciones genéticas de ciertos genes cuyos productos están involucrados en la homeostasis numérica de los centrosomas. Uno de esos genes es p53; la pérdida o la inactivación mutacional de p53 conduce a amplificación centrosómica (Tarapore y Fukasawa, 2002). Sin embargo, así como las anomalías numéricas y estructurales de los centrosomas han sido implicadas en la formación de husos multipolares, mal segregación cromosómica e inestabilidad cromosómica, Separación centrosómica M G2 El ciclo del centrosoma S Maduración centrosómica Elongación Centriolar Figura 5. G1 Duplicación Centriolar El Ciclo del Centrosoma. El ciclo de duplicación centrosómica puede ser subdividido en varios pasos discretos. Durante la mitosis el centrosoma en cada polo contiene un par de centriolos. Esos dos centriolos muestran usualmente una conspicua conformación ortogonal. Al final de la mitosis esta asociación ortogonal se pierde (lo que se conoce como desorientación centriolar. La duplicación centriolar ocurre durante la fase S del ciclo celular. A nivel morfológico esto se caracteriza por la formación de procentriolos en el extremo proximal de cada centriolo parental, de tal forma que la duplicación centriolar es semiconservativa desde el punto de vista del centrosoma pero conservativa desde el punto de vista del centriolo. Los procentriolos se elongan hasta que alcanzan la máxima longitud. En el límite G2 –M se produce la maduración centrosómica. Esta maduración implica el intercambio de muchos componentes y culmina con el reclutamiento de complejos anillos de tubulina gama, un requisito para la actividad de nucleación microtubular. En respuesta a la activación de proteínas motoras dependiente de microtúbulo los centrosomas se separan entre ellos e instruyen la formación del huso bipolar.( Tomado Nigg, 2002) 13 debe decirse que la causalidad no ha sido definitivamente establecida. Se ha dado a conocer que las anomalías centrosómicas no necesariamente correlacionan con inestabilidad cromosómica numérica y que incluso la presencia de metafases multipolares no es suficiente para predecir si una célula o un tumor es cromosómicamente inestable (Duensing et al., 2003). En células de ratón se puede inducir amplificación centrosómica tras la inactivación mutacional de p53. Sin embargo, en células humanas, el sólo silenciamiento de p53 no es causa suficiente para inducir la amplificación centrosómica (D’Assoro et al., 2004). Ello indica claramente que en células humanas deben estar involucrados otros mecanismos igualmente importantes, que aseguran la integridad numérica de los centrosomas y con ello la integridad genómica. La ciclina E, una subunidad regulatoria de Cdk2 que juega un rol fundamental en la duplicación centrosómica, se ha encontrado frecuentemente sobreexpresada en algunos cánceres humanos. Recientemente se ha demostrado que la sobreexpresión de ciclina E, junto a inactivación de p53 induce efectivamente amplificación centrosómica, no así cada uno por sí solo. Debido a que las ciclinas en células humanas son proteínas tremendamente reguladas, estos hallazgos indican que ese patrón de regulación de la expresión de ciclina E, juega un rol adicional en la homeostasis numérica de los centrosoma en células humanas y que la deregulación de ciclina E, junto con la inactivación de p53 resulta en amplificación centrosómica e inducción de fenotipo CIN (Kawamura, 2004). Líneas paralelas de evidencias refuerzan la idea que el centrosoma es un integrador funcional de las vías que disparan el inicio de la mitosis proceden de estudios realizados en células tumorales mediante el empleo de la técnica de inhibición por ARNi en los que se demuestra que la fosforilación de Cdc25-B en Serina 353 (S353) -en el centrosoma- depende de Aurora quinasa A (producto de un oncogen necesario para el ensamblaje de un aparato mitótico funcional y la regulación de la ploidía celular) y es fundamental en el control de ingreso en mitosis. Experimentos de microinyección de anticuerpos anti-S353 inducen a retraso mitótico, mientras que la sobreexpresión de un mutante fosfomimético de S353 potencia el efecto inductor de mitosis de Cdc25-B (Dutertre et al., 2004). Además, la estructura y función de los centrosomas son parámetros regulados de una manera dependiente de ciclo celular. Primero, los centrosomas se duplican semiconservativamente. Posteriormente, una vez en mitosis los centrosomas duplicados son objeto de un cambio dramático en la capacidad de nuclear microtúbulos y se separan físicamente hacia los polos opuestos del núcleo donde ellos organizan el huso bipolar en conjunto con los cromosomas condensados. Hasta el momento los mecanismos que controlan 14 esos procesos permanecen desconocidos. Sin embargo, mediante técnicas que utilizan anticuerpos específicos se ha evidenciado que la fosforilación de proteínas centrosómicas juega un rol fundamental en esos procesos. Un de esas proteínas es Nek2, integrante de una nueva familia de proteínas quinasas muy relacionada a NIMA (del inglés: Never in mitosis gene A) una proteína quinasa aislada a partir de Aspergilus nidulans, el hongo filamentoso. En este hongo, NIMA coopera con Cdk1 una proteina quinasa que promueve la entrada en mitosis y la sobreexpresión de NIMA en una variedad de especies heterólogas promueve una condensación prematura de los cromosomas y en algunos casos la formación del huso. Esto sugiere una conservación evolutiva de las vías que involucran a las quinasas relacionadas a NIMA. La familia de proteínas Nek humanas incluye a 11 miembros a la fecha (Bowers y Boylan, 2004), sin embargo, las relaciones funcionales entre las proteínas quinasas Nek en vertebrados en general y las NIMA de aspergillus permanecen sin descifrarse hasta el momento. Nek2 experimenta cambios a través del ciclo en abundancia y actividad. Es altamente expresada en células germinales y existen algunos datos que señalan su participación en la condensación del cromosoma meiótico. Sin embargo, la sobreexpresión de Nek 2 en células somáticas no tiene efecto sobre la condensación cromosómica, sino que induce una separación prematura de los centrosomas que recuerda la separación de centrosomas que ocurre en el límite G2-M. Ensayos de inmunofluorescencia y de fraccionamiento celular, evidencian una localización asociada a centrosoma para esta proteína, lo que sugiere que -en general- este tipo de quinasas está involucrada en el ciclo centrosómico. Estos resultados han permitido sugerir que la separación de los centrosomas es un fenómeno dependiente de fosforilación de proteínas aún no identificadas por Nek2 que induce la pérdida de cohesión de los centrosomas duplicados y que ello podría representar los primeros pasos en la separación efectiva de los centrosomas durante las etapas tempranas de la mitosis. Como sustento de esta hipótesis se ha identificado la proteína C-Nap1 (del ingles: centrosome Nek2associated protein 1) que corresponde a un péptido de 281 Kda que se sitúa en el extremo proximal de los centriolos. Todo indica que la fosforilación de esta proteína por parte de Nek 2 es la señal que induce la separación de centrosomas (fig 6).. Anomalías en este proceso naturalmente conllevan a fenómenos de amplificación centrosómica, lo que genera desbalances numéricos en la distribución de cromosomas debido a un mal funcionamiento centrosómico. Modelo de Separación Centrosómica G2 Nek2 Mitosis Proteínas Motoras C-Nap1 Figura 6. Modelo de separación de centrosoma. La fosforilación de la proteína C-nap1 por Nek dos sería un paso crucial que gatillaría la separación de los centrosomas durante la profase mitótica 15 C. Defectos en Segregación Cada una de nuestras células hereda su información en la forma de cromosomas a partir de una célula madre. Con el objeto de que ambas hijas obtengan una cantidad equitativa de esa información, las células necesitan asegurarse que los cromosomas replicados son separados y distribuidos durante la división celular. Como se decía previamente errores en el proceso conducen a aneuplodías, células con cromosomas de menos o supernumerarios. Se sabe que la duplicación del ADN está relacionada a la segregación de las cromátidas hermanas en la mitosis por el fenómeno denominado como Cohesión de Cromátidas Hermanas (CCH). Se ha identificado un complejo, cohesina, que mantiene juntas las hebras de ADN duplicadas después de su síntesis formando un anillo proteináceo que abarca a ambas cromátidas hermanas (Fig. 7). En la anafase una proteasa, separasa, destruye a cohesina, lo que gatilla el funcionamiento de la maquinaria de separación. Cohesina es un complejo proteico de 4 subunidades que se requiere –como se decía- para mantener unidas las cromátidas de cromosomas replicados. Contiene un heterodímero de proteínas de mantenimiento cromosómico SMC (del inglés Structural Mantenaince of Chromosomes, de las cuales ya se han identificado 6 en células de mamíferos con un peso molecular que varía entre los 110 y los 170 kDa y que juegan un rol crucial en numerosos eventos nucleares incluyendo la propia organización de los cromosomas durante la mitosis. Son conservadas en eucariontes: Smc1 y Smc3 forman un heterodímero requerido para la progresión de la metafase en células mitóticas. Se sabe que estas dos proteínas se encuentran en vertebrados formando un complejo con Scc1 y Scc3 pertenecientes a otra familia de proteínas. Cohesina posee una localización en la cromatina dependiente de la fase del ciclo celular que se analice, lo que era esperable para un factor de cohesión. En organismos superiores gran parte de la cohesina disocia en profase aunque persisten algunas cantidades importantes en centrómeros que disocia en Anafase. Esta liberación profásica de la cohesina es necesaria para la resolver la separación de las cromátidas. Si la cohesina no se remueve, las cromátidas son incapaces de separarse. Experimentos de inmunoprecipitación e inmunoflourescencia en cromatina revelan que la cohesina se ubica a través de los brazos cromosómicos y que se concentra en el centrómero que es el sitio de cohesión más fuerte, algo esperable para un complejo que conecta las cromátidas hermanas y las mantiene unidas. (Fig. 8). La disociación de cohesina en la anafase se gatilla mediante la degradación catalítica de su subunidad Scc1 por Separasa. El rompimiento de Scc1 es necesario para la separación de Smc1 Smc3 Scc3 Scc1 Modelo de cohesión de cromátidas hermanas por el complejo cohesina. Los Figura 7. modelos actuales plantean la idea de que el compleo “abraza ambas cromátidas rodeándolas. Scc1 (Securina) es el blanco de ataque por separasa lo que determina la separación de las cromátidas. (Tomado de Hagstrom y Meyer, 2003) Securina Separasa Separasa Metafase Anafase Disociación de masa de Cohesina Condensación Cromosómica Securina Profase Figura 8. Control de cohesión entre cromátidas hermanas. En vertebrados el cúmulo de cohesina disocia durante la profase. Una pequeña cantidad de cohesina permanece sobre los cromosomas predominantemente a nivel del centrómero. Esta cohesina es finalmente atacada por separasa en la transición metafase-anafase. La activación de separasa en este punto es llevado a cabo por la destrucción de securina por APC/C (Tomado de Amon,2001) 16 las cromátidas hermanas y es una entrada en anafase sin posibilidad retorno. Separasa es inhibida por Securina hasta que el APC le une ubiquitina y la destruye. El APC en cambio, es incapaz de llevar a cabo su acción hasta que el punto de control del huso permita apagar la señal de “anafase en espera”. Ese y otros mecanismos aseguran que cohesina permanezca en su sitio hasta que la célula esté lista para ingresar en Anafase. Se deduce entonces que defectos en el complejo cohesina conduce a una separación de cromátidas prematura, mal funcionamiento cinetocórico y del punto de control del huso mitótico y consecuentemente a la aparición de fenotipo CIN (Hoque e Ishikawa, 2002; Yu, 2002). Sin embargo, la segregación defectuosa de los cromosomas que conducen a fenotipo CIN puede originarse por la desregulación de una serie de otras proteínas que según se van identificando y caracterizando se descubre que están involucradas también en la posible etiología de inestabilidad cromosómica. Veamos algunos casos. Usando la técnica de interferencia con ARNi en células humanas en cultivo se ha demostrado que la proteína H2A.Z, una variante de H2A presente desde metazoos hasta humanos, es también esencial en la transmisión fidedigna de los cromosomas. En ausencia de H2A.Z el genoma se torna inestable y esta inestabilidad es originada por defectos en el proceso de segregación cromosómica. Los estudios de localización de H2A.Z revelan que en esas células esta proteína se ubica en sectores heterocromáticos ricos en HP1α que existen a lo largo de los brazos cromosómicos pero no en la región centromérica. Recordemos que HP1α es una proteína no histónica muy conservada que localiza en regiones heterocromáticas del cromosoma. Si se elimina H2A.Z, se rompen las interacciones normales que se establecen entre la cromatina y HP1α en la región de los brazos y notablemente también en las regiones pericéntricas. De esta forma se sugiere que H2A.Z controla la localización de HP1α y consecuentemente H2A.Z es esencial para la segura y correcta transmisión de los cromosomas (Rangasamy et al., 2004). Por otra parte se ha evidenciado que la iniciación de la duplicación del ADN requiere del ensamblaje de un complejo preduplicativo (ORC) en la cromatina durante G1 del ciclo celular. En células humanas, la subunidad Orc-2 localiza dentro del núcleo y en centrosomas a través de todo el ciclo celular. Además Orc-2 se localiza unida fuertemente a la proteína de heterocromatina Hp1α . La eliminación de Orc-2 causa fenotipos múltiples. Un subconjunto de células que evidencian una fase S defectuosa con poco antígeno nuclear de proliferación 17 nuclear (PCNA, del inglés Proliferating Cell Nuclear Antigen). Otro subconjunto de células, muestra cambios en la localización de Hp1α y otro que muestra detención en fase S con cromosomas anormalmente condensados, proceso anómalo de congresión y múltiples centrosomas. Esos resultados indican que Orc-2 está involucrada en la duplicación cromosómica, estructura cromosómica, en el control del número de centrosomas, lo que sugiere que esta proteína coordina todos los estados del ciclo de herencia cromosómica. Anomalías en la regulación de esta proteína se asocia a la aparición de fenotipo CIN numérico por fallos en la segregación (Prasanth et al., 2004). En esta compleja maquinaria proteica involucrada directa o indirectamente en la correcta segregación de los cromosomas durante los procesos divisionales no debe dejar de mencionarse nuevamente a las Quinasas Aurora, implicadas en el ciclo del centrosoma, ensamblaje del huso, condensación cromosómica, punto de control del huso y citocinesis. Se ha evidenciado que una desregulación de estas proteínas induce a fallos en el ensamblaje del huso mitótico, en el punto de control del huso y en la división celular originando segregación defectuosa de cromosomas individuales o poliploidización acompañada de amplificación centrosómica. (Meraldi et al., 2004b) La segura alineación de cromosomas en la metafase así como la subsecuente segregación hacia los polos –como se ve- es un proceso complejo que implica una maquinaria molecular muy elaborada y caracterizada parcialmente. Las kinesinas, proteínas motoras que hidrolizan ATP según se mueven a lo largo de los microtúbulos, incluyen a 155 proteínas aisladas a partir de 11 especies distintas distribuidas en tres grupos. Un grupo de ellas juega un rol importante en la movilización de estructuras a través del citoplasma (vesículas –organelos); otro funciona directamente en la movilización de cromosomas y un tercero que participa en ambos procesos (Dagenbach y Endow, 2004). Avances recientes en la identificación y caracterización de proteínas motoras involucradas en la segregación indican que la cromoquinesina humana Hkif4A, una proteína de 140 kDa que incluye en su estructura un dominio motor parecido a kinesina, juega un rol esencial en este proceso. Esta proteína se localiza en el nucleoplasma durante la interfase y sobre los cromosomas condensados durante la mitosis. Se acumula en la zona media durante anafase y en el anillo de clivaje durante la citocinesis. La eliminación de esta proteína mediada por ARNi resulta en una organización prometafásica defectuosa, mal alineación de los cromosomas en metafase, defectos en el huso y segregación cromosómica también defectuosa. Se ha demostrado también que Hkif4A 18 interactúa con los complejos de condensina I y II, de tal forma que la eliminación de Hkif4A conlleva a una hipercondensación cromosómica, lo que sugiere además que esta proteína es requerida para la mantención de la arquitectura cromosómica. (Mazumdar et al., 2004). Como se observa los defectos segregacionales pueden observarse tras la desregulación de varias proteínas que empiezan a caracterizarse, no obstante el mecanismo subyacente permanece sin conocerse hasta el momento aunque es evidente que la segregación errónea de los cromosomas hijos hacia los polos es un proceso complejo en el que las fallas en el funcionamiento del huso o en el cinetócoro no son las únicas causalidades que explican el fenómeno en general. La interferencia con ARNi demuestra que hay varias proteínas involucradas a través del ciclo celular, cuya desregulación es un factor desencadenante del fenómeno CIN. D. Malfuncionamiento de telómeros La naturaleza lineal del cromosoma eucariótico impone algunos desafíos a la célula ya que la presencia de extremos precisa de estructuras especializadas para prevenir su degradación o fusión, fenómenos que conducen a inestabilidad cromosómica que es a menudo asociada con el desarrollo del cáncer. Los complejos de ADN y proteínas que cubren los extremos de los cromosomas de esos fenómenos se denominan telómeros (Rodees et al., 2002). Su principal función es prevenir que los extremos del cromosoma sean reconocidos y procesados como fracturas en el ADN lo que normalmente conduce a una detención en el ciclo celular así como a reacciones de fusión para reunir los extremos rotos. La unión de los cromosomas a través de sus extremos conduce a la formación de cromosomas dicéntricos que irremediablemente expresan una distribución aberrante de la información genética entre las células hijas, así como a la formación de nuevos extremos no protegidos que generan otros ciclos de inestabilidad cromosómica. Los telómeros también son críticos para prevenir el gradual acortamiento cromosómico que se origina por el clásico problema de la “duplicación de los extremos del ADN”, es decir la imposibilidad de las ADN polimerasas convencionales para duplicar el extremo de un ADN linear. El ADN telomérico consiste de repeticiones in tandem a menudo cortas que contienen grupos de tres o cuatro guaninas. A continuación se muestran las secuencias mejor caracterizadas de secuencias teloméricas terminales. 19 ORGANISMO SECUENCIA Tetrahymena TTGGGG Oxytricha TTTTGGGG Giardia TAGGG Physarum TTAGGG Kluyveromyces TCGGATTTGATTAGGTATGTGGTGT Neurospora TTAGGG Caenorphabditis TTAGGG Vertebrados TTAGGG En casi todos los eucariontes estudiados, el ADN telomérico (ADNt) consiste en repeticiones in tandem de pequeñas secuencias nucleotídicas con una distribución asimétrica de los pares G:C, pues las G se acumulan en una de las hebras (hebra G) y se encuentran agrupadas. La hebra G está orientada de 5' a 3' hacia el extremo del telómero y forma el extremo 3' del ADN cromosómico. En la zona más extrema no está apareada formando un segmento final de hebra simple o monofibrilar con una longitud que varía según la especie. La longitud del telómero es variable, en Oxytricha y Euplotes (microorganismos ciliados) es apenas de 38 pb mientras en ratones alcanza 150 kb. Cada organismo posee una longitud media característica. Un telómero funcional requiere un mínimo de longitud de ADN y la presencia de una caperuza (capping) protectora constituida de proteínas muchas de las cuales ya han sido identificadas y caracterizadas. Esas proteínas se expresan constitutivamente y son necesarias para la protección estabilidad, replicación y/o elongación de los telómeros. En ausencia de telomerasa, los telómeros se acortan gradualmente. La longitud de los telómeros en humanos es parcialmente controlada por un mecanismo de retroalimentación en el cual la elongación del telómero por la Telomerasa es limitada por al acumulación del complejo TRF1 (del inglés Telomere Repeat Factors) en el extremo del cromosoma. Uno de los constituyentes de TRF1 es una tanquirasa denominada como Tin2 que modula a poliADP ribosa polimerasa y regula así la longitud del telómero (Ye y de Lange, 2004). Existe un cúmulo de evidencias que indican que el mal funcionamiento de los telómeros cromosómicos conducen a la manifestación de fenotipo CIN (Yan y Cao, 2004; Mathieu et al., 2004; Plug-De Maggio et al.,2004; Gollin, 2004). Por otra parte TRF1 y TRF2 reclutan en el telómero una serie de otras proteínas involucradas en la reparación de fracturas en el ADN como Ku y el complejo Mre11, Rad50, Nbs1 (Zhu et al, 2000) (Fig 9) . Tanquirasa Nucleosomas El complejo telomérico humano consiste de ADN y proteínas asociadas. Los Figura 9. telómeros humanos consisten en aproximadamente 7000 -10.000 pares de bases de ADN de hebra doble. La mayor parte de este ADN está empaquetado en nucleosomas, sin embargo en el extremo de cada cromosoma existe un ADN telomérico de unas 100 pares de bases que se une a dos proteínas teloméricas TRF1 y TRF2. TRF1 forma un complejo con otras dos proteínas Tin2 y Tanquirasa que contiene PARP (PoliADP ribosa polimerasa). TRF2 recluta a Rap1. Además de los complejos que se muestran en la figura, otras proteínas como Ku y el complejo Mre11-Rad50Nbs1, implicado en el proceso de reparación de ADN de hebra doble también, están localizadas en el telómero. El segmento ADN de hebra simple rico en Guanina que sobra (100 nucleótidos de largo) une a Pot1. (Tomado de Rhodes et al., 2002). 20 Recientemente, mediante el uso de imágenes 3D combinado a FISH y análisis cuantitativo, se ha estudiado la localización de los telómeros durante el ciclo celular. Se ha observado que los telómeros se organizan en una manera dependiente de la etapa del ciclo celular. Notablemente, en G2 tardío los telómeros se asocian a un disco telomérico, que no se había descrito hasta hoy. Estos discos teloméricos se desorganizan en células tumorales observándose agregados teloméricos, que causan aberraciones cromosómicas irreversibles. Ello plantea la posibilidad de analizar estos agregados teloméricos como signos de inestabilidad cromosómica en núcleos interfásicos (Chuang et al., 2004). Estudios citogenéticos y moleculares han indicado que aquellos cromosomas con incluso un extremo desprotegido son genéticamente inestables en tanto no se restituya la integridad del telómero afectado. Durante este periodo de inestabilidad cromosómica ocurren ciclos de fractura y fusión (BFB del inglés: Breakage_Fusion_Breakage) que culminan en aneuplodías. Los ciclos BFB y la inestabilidad cromosómica promueven fusiones entre cromátidas a través del fenómeno de unión de extremos no homólogos. Durante la mitosis, la separación de los centrómeros en cromosomas dicéntricos produce un puente ana-telofásico, seguido por fractura cromosómica y la eventual fusión de extremos dañados lo que promueve ciclos BFB adicionales. Durante la adquisición de nuevos telómeros se pueden originar ciclos recurrentes de amplificación génica en esos cromosomas reordenados lo que sugiere que las fracturas de ADN de hebra doble cercanas al punto de fractura son importantes a la hora de promover amplificación génica. E. Recombinación homóloga vinculada a Reparación de Fracturas de ADN de hebra doble . La inducción de fracturas de ADN de hebra doble por exposición a agentes dañinos, o como intermediarios en procesos celulares normales, constituyen un riesgo para la integridad del genoma. Si no se reparan apropiadamente, estas fracturas pueden traducirse en aberraciones cromosómicas las cuales a su vez pueden conducir a la muerte celular o crecimiento celular descontrolado. Para mantener la integridad del genoma, a través del curso de la evolución se han desarrollado múltiples vías de reparación de fracturas de hebra doble: 21 Recombinación Homóloga (RH), Unión de extremos no homólogos (NHEJ) y reunión de hebras simples (SSA). Las fracturas de hebra doble pueden originarse en el ADN por varios mecanismos, a saber: Conversión de una fractura de hebra simple en fractura de hebra doble, radiación ionizante (rayos X, rayos gamma), por fuerzas mecánicas durante la mitosis que tiran moléculas de ADN enredadas, por acción incompleta de topoisomerasas, por la acción de endonucleasas en horquillas de replicación estancadas o en otros sitios específicos. En el propósito de entender los mecanismos que explican el fenotipo CIN estructural expresado como deleción, conversión génica, duplicaciones in tandem o Intercambio entre cromátidas hermanas ICH) este apartado se focaliza en el análisis de los procesos involucrados en el origen de cada una de esas alteraciones en la estructura cromosómica. Se han descrito dos formas importantes de reparación recombinacional que tienen que ver con fracturas de hebra doble. i. Recombinación no-homóloga (NHEJ: non homologous end joining) que requiere de poca o ninguna secuencia de homología y es mediada por una unión directa de los extremos rotos y prevalece durante G1. (Vía rápida) (Fig 10 a) y ii. recombinación homóloga que se lleva a cabo durante S y G2 del ciclo celular y se sustenta en una complementariedad entre la cromátida intacta y la dañada como base de intercambio de hebras y reparación (Vía lenta) La fractura de hebra doble origina dos extremos “sucios”, esto es, que contienen no sólo una interrupción del eje azúcar–fosfato, sino posiblemente bases nitrogenadas rotas, e incluso enlaces cruzados en la zona de la rotura. Estas anomalías deben ser corregidas antes de que ellos sean procesados. Al parecer esta labor de limpieza la realiza un complejo proteico Mre11 en el que participan Mre11/Rad50/Nbs1 (MRN) localizado precisamente en los sitios de fractura doble, con actividad endo y exonucleasa (D’Amours y Jackson, 2002). El componente Rad50 del MRN posee dos colas intramoleculares sobreenrrolladas y altamente flexibles que funcionan como dos brazos que sugieren un rol adicional para el complejo MRN, como es facilitar el proceso de encuentro entre los dos extremos rotos. Se ha estimado que el hecho de aumentar por esta vía (brazos de Rad50) el radio de búsqueda de extremos rotos incrementa la posibilidad de que dos extremos rotos se encuentren en 16 veces (Helleday, 2003). Recientemente se ha implicado a este complejo (Mre11) en el mantenimiento de telómeros y en (a) NHEJ 5’ 3’ 5’ 3’ 5’ 3’ (b) ataque DNAss 5’- 3’ 3’ (c) Unión de hebras simples 5’ 3’ 3’ 3’ 5’ 3’ 5’ (i) Entrecruzamiento 5’ 3’ 5’ 3’ (h) 2° invasión 3’ 5’ 5’ 3’ (f) Unión de hebras simples (d) Primera invasión 5’ 3’ 3’ 5’ (e) migración de ramas 5’ 3’ 3’ 5’ (g) NHEJ (j) sin entrecruzamiento 5’ 3’ 5’ 3’ Duplicación in tandem 3’ 5’ Figura 10. Reparación por recombinación de una fractura de ADN de hebra doble. (a) La unión de extremos no homólogos (NHEJ) está involucrada en una rápida pero a menudo inexacta reparación de una fractura de hebra doble que puede resultar en deleción. (b) Los extremos del ADN pueden ser blanco de exonucleasas que dejan extremos 3’ sobrantes (c) Regiones repetidas pueden permanecer descubiertas las que pueden usarse para reunir hebras simples que conducen a deleción de ADN que separa las repeticiones. (d) Uno de los extremos de hebra simple del ADNss puede iniciar un proceso de invasión de una secuencia homóloga de ADN (roja). La hebra invasora se alarga pasado el sitio de fractura. (e) La migración de las ramas de la unión holliday (HJ) puede liberar a la hebra invasora desvelando secuencias homólogas de ADNss que sobran en el extremo opuesto del ADN. (f) Síntesis dependiente de reunión de hebras simples (SSA) puede usar esta homología en reparación lo que resulta en conversión génica sin deleción. (g) Síntesis dependiente de unión de extremos no homólogos une el extremo de ADN extendido sin usar la secuencia de homología. Ello resultará en duplicación in tandem . (h) Si las ramas de la Unión Holliday (HJ) no migran puede ocurrir invasión de hebra por el segundo extremo de ADNss originando una HJ doble. En células de mamíferos, esas HJ no se resuelven por entrecruzamiento (i, puntas de flechas llenas) . En vez de ello las HJ pueden resolverse sin entrecruzamiento (j; puntas de flechas vacías) causando un evento de conversión génica. (Tomado de Helleday, 2003) 22 la activación del punto de control de daño en el ADN. Este complejo posee una nucleasa estimulada por ATP para procesar extremos de ADN heterogéneos en el proceso de unión (Assenmacher y Hopfner, 2004). Pudiese este complejo actuar como un sensor de fracturas y actuar en la activación y propagación de las vías de señalización que gobiernan los puntos de control del ciclo celular en respuesta a un daño en el ADN. Además el complejo Mre11 influye en la reparación recombinacional entre las cromátidas hermanas (Stracker et al., 2004). En sustento de las ideas anteriores se ha demostrado recientemente que en respuesta a estrés duplicativo, las células relocalizan y activan proteínas de reparación de ADN y las involucradas en la detención del ciclo como la Proteína de Duplicación A, (PDA); un complejo de tres subunidades requerido para la duplicación del ADN y para la reparación de roturas (Robison et al., 2004). La unión de extremos no homólogos o NHEJ en fracturas de ADNds se inicia con la unión del heterodímero constituido por las proteínas Ku70 y K80 las cuales a modo de un verdadero anillo, se sitúan alrededor de los extremos rotos del ADN cumpliendo una función estructural que facilita la reunión de esos extremos. (Walker et al 2004). Para unir los extremos la subunidad catalítica DNA-PK (proteína Quinasa dependiente de ADN) se une al heterodímero Ku70-Ku80 y cataliza la unión de los extremos del ADN usando inositol-6-fosfato como cofactor. Nuevamente los extremos del ADN pueden necesitar un ajuste, una reacción catalizada por Ligasa IV o Artemisa que se une a DNA-PK y exhibe actividad exonucleasa que abre horquillas y otras estructuras que no pueden ser reunidas fácilmente (Ma et al., 2002). Si existen pequeñas zonas de homología en los extremos del ADN ellas pueden además ayudar en la alineación de las hebras. Cuando los extremos están alineados, se deben producir grupos terminales 3’OH y 5’-fosfato unibles por una polinucleótido Quinasa. Los extremos son finalmente unidos por la ligasa IV o Artemisa que existe asociada con Xrcc4. La recombinación homóloga (RH) es el intercambio (Crossing over) o reemplazo (conversión génica) de una región de ADN por su secuencia de ADN homóloga a partir de un cromosoma homólogo o de una cromátida hermana. La RH ocurre ocasionalmente entre secuencias de ADN similares o dentro de un mismo cromosoma. El paso inicial de la RH se piensa que es el ataque exonucleásico 5’ a 3’ del extremo del ADN roto para producir un ADN de hebra simple (ADNss). (Fig 10b). Aquí pueden ocurrir dos 23 cosas: i. Reparación por re-unión de extremos de hebra simple o ii. Invasión de hebras en la reparación de los dos extremos i. Reparación por reunión de extremos de hebra simple Si no se cubren las secuencias repetidas dentro del ADNss que sobresale originado por acción exonucleásica, RAD52 y la Proteína de Replicación A (PRA) pueden iniciar un proceso de reparación entre las secuencias repetidas. RAD52 se une a los extremos 3’ de los ADNss, en tanto que PRA se une a todo el filamento de hebra simple que sobresale. Cuando esas secuencias repetidas se unen, las regiones adyacentes quedan fuera formando un sustrato para XRCC1/XPF que aparentemente juega un rol importante en la unión de hebras simples. En mamíferos la reunión de extremos de ADNss es frecuente entre secuencias repetidas. El producto que resulta de la reunión de extremos de ADNss es una deleción haciendo de éste un mecanismo proclive a errores. (Fig 10c) ii. Invasión de hebras en la reparación de dos extremos En este caso la proteína clave para lograr ello es RAD51, el homólogo eucariótico de RecA en E.coli. RAD51 al igual que RecA forma un filamento nucleoproteico en regiones de ADNss y cataliza la búsqueda de secuencias homólogas, apareamiento de hebras e intercambio de éstas. Se ha comprobado que los ratones knockout para RAD52 viven y son saludables, mientras que los ratones knockout para RAD51 son inviables. De la misma forma ratones knockout para otros genes involucrados en RH, tales como: XRCC2, RAD51B, RAD51D, RAD50, Mre11 o Nbs1 son letales, lo que indica que es vital contar con las vías de RH intactas para poder llevar a buen término el proceso. Una secuencia de ADN homóloga es necesaria para la invasión de hebras dependiente de RAD51 (fig 10d). Las cromátidas hermanas que están presentes durante S y G2 participan en este evento al menos 100 veces más frecuentemente que los cromosomas homólogos. Para que ocurra la invasión de hebras es necesario que RAD51 disponga de PRA en el ADNss que sobresale. Siguiendo la invasión de moléculas de ADN homólogas por RH se inicia la síntesis de ADN en el extremo 3’ por una ADN polimerasa. La síntesis avanza más allá del sitio original de fractura de doble hebra. Se deja una unión Holliday (HJ del inglés Holliday Junction) en el sitio de la invasión, que puede migrar en ambas direcciones. Si la migración se produce en la 24 dirección de la replicación (fig 10e). puede revertirse la invasión dejando un extremo de ADN que ha sido alargado más allá de la fractura original. Este ADNss compartirá homología con el otro extremo y puede ser reparado por unión de hebras simples (Fig 10 f) El producto final que resulta de la reunión de extremos de hebra simple no contiene deleción en tanto que la forma regular de reparación por reunión de extremos de hebra simple rotos que utiliza secuencias repetidas, sí. Sin embargo, la síntesis dependiente de unión de hebras simples resulta en conversión génica que corresponde a una vía de reparación libre de errores aunque puede resultar en pérdida de heterocigosidad. Alternativamente, puede ocurrir que el extremo de ADN alargado sea reparado por unión de extremos no homólogos (fig 10g). El resultado de este proceso es una duplicación in tandem en el sitio de la fractura de doble hebra. Como una alternativa para la liberación del extremo invasor, el segundo extremo roto puede invadir la misma molécula de ADN homóloga (Fig 10h). Esto resultará en la formación de una HJ doble que puede ser resuelta ya sea por crossing over (fig 10i) o sin crossing over ( fig 10j). Los fenómenos de entrecruzamiento en mamíferos son poco frecuentes y no se ha descrito nunca un ICH cuando se analizan los productos de la reparación por recombinación homóloga de fracturas de doble hebra. Como quiera que sea, el producto formado por la resolución de HJ dobles por un mecanismo que no implica entrecruzamiento, es una conversión génica. En pocas palabras, los productos que se obtienen de la recombinación de extremos rotos son deleciones, conversiones génica o duplicaciones in tandem. La explicación más probable de este solapamiento entre la reparación de fracturas de hebra doble ya sea por unión de extremos no homólogos y RH, debe darse en el contexto en el cual las fracturas ocurren. RH es favorecida muy probablemente a fines de fase S y durante G2 cuando están presentes las cromátidas hermanas, mientras que la unión de extremos no homólogos es favorecida durante G1 cuando los cromosomas homólogos están distantes y conduciría a una irremediable pérdida de heterocigocidad. 25 La RH ha demostrado ser de suma importancia en la reparación de daño durante la replicación desde la levadura hasta el hombre. Las fracturas de hebra doble se pueden generar después de la duplicación, por conversión de fracturas de hebra simple en fracturas de hebra doble. La diferencia más importante entre la fractura de hebra doble clásica y una fractura de hebra doble asociada a duplicación es que -en esta última- existe sólo un extremo para iniciar la RH (Fig 11a ). Después que el gap de ADNss ha sido rellenado (Fig 11b) el extremo de ADN libre puede invadir esta molécula intacta de ADN (Fig 11c y d ) y retomar la duplicación (fig 11e). Siguiendo este evento de recombinación, la hebra líder se posicionará en el lado opuesto y dejará atrás de la horquilla una HJ. Un evento de intercambio entre cromátidas hermanas (ICH) ocurrirá subsecuentemente sin conversión génica. (Fig 11f) Este ICH es visualizado gracias a que el ADN sintetizado de novo permanece unido a su templado (cohesina) y después de la segunda mitosis este sitio de fractura puede ser visualizado elegantemente mediante la técnica citológica para visualizar los ICH. (Fig 11g). Evidencias adicionales sustentan la idea de que los ICH se originan por fracturas de hebra simple que persisten proviene de experimentos con células defectivas en la reparación de fracturas de hebra simple, por ejemplo células deficientes en XRCC1 o PARP1 (PoliADP ribosa polimerasa1) que muestran altos niveles de ICH espontáneos. Es evidente que la RH también juega un rol importante en la reparación de horquillas de duplicación atascadas (Stalled Replication Forks). Se han descrito varias causas de estancamiento de la horquilla de duplicación, por ejemplo, el normal proceso de avance de la maquinaria de duplicación puede encontrarse con secuencias especiales complementarias de ADN que son susceptibles de formar horquillas en la región de hebra única simple de la hebra retrasada según las hebras bifurcadas de ADN van abriéndose. Este fenómeno puede explicar la inestabilidad de la secuencia repetidas CTG que muestra cambios dramáticos en el número de copias en enfermedades neurológicas como la enfermedad de Huntington. Otra causa de estancamiento en el avance de la maquinaria de duplicación es la presencia de lesiones de ADN no reparadas como los fotodímeros originados por luz UV. La horquilla atascada puede ser cortada por alguna endonucleasa de hebra simple, y rescatada por recombinación. Para evitar que las endonucleasas rompan la horquilla de hebra simple, hay necesidad de que exista una forma de recomenzar la duplicación y saltarse la lesión. Una forma ha sido propuesta por Michel en la cual la hebra de ADN sintetizado de novo se desenrrolla de su 5’ 3’ 5’ 3’ (a) Horquilla de duplicación asociada a fractura de doble 5’ 3’ 3’ 5’ 5’ 3’ 5’ 3’ (b) Ataque exonucleásico 5’a 3’ ( c ) Formación de un templado cromatídico i t t 3’ 5’ 3’ 5’ 5’ 3’ Segunda Mitosis ICH 5’ 3’ 3’ 5’ 3’ 5’ 3’ 3’ 5’ 5’ 3’ (d) Invasión de hebra Figura 11. (e) Horquilla de duplicación restituida (f) Resolución de HJ Reparación por Recombinación Homóloga (RHH) de una horquilla de duplicación colapsada. (a) Una horquilla de duplicación asociada a una fractura de hebra doble posee un extremo libre para iniciar un ataque exonucleásico (b) el extremo 3’ del ADNss que sobra es cubierto con Rad51 y otras proteínas de recombinación homóloga implicadas en la invasión de hebras. (c) El espacio de hebra simple sobre el templado de ADN es llenado antes (d) de la invasión de hebras. La síntesis de la hebra líder puede continuar en el templado invadido de ADN de tal forma que se restablece la horquilla de duplicación (e). (f) una HJ simple queda por detrás de la horquilla de duplicación lo que se resuelve probablemente sin entrecruzamiento. Ya que el templado y el ADN sintetizado de novo se han fusionado (seguir líneas en f), se visualizará ICH en la segunda mitosis (g). Las flechas indican las direcciones de la síntesis de ADN. Las líneas negras corresponden al templado y las rojas al ADn sintetizado de novo. Las flechas llenas indican entrecruzamiento y las vacías indican no entrecruzamiento. (Tomado de Helleday, 2003). 26 templado (quizás después que ha pasado la ADN polimerasa y se ha disociado). Este proceso requiere al menos de una helicasa. Las dos nuevas hebras se podrían aparear entre ellas formando el típico estructura de cuatro brazos que se ha denominado como Unión Holliday (UH) (del inglés: Holliday junction) en honor a quien las describióy cuya formación da lugar a las recientemente denominadas como estructuras en “pie de pollo” (Helleday, 2003). La formación de este tipo de estructuras seguramente es facilitada por otras proteínas (Fig 12). Un gran número de evidencias en bacterias indica que las horquillas de replicación atascadas pueden ponerse en reversa para formar estas estructuras en pie de pollo que pueden ser reparadas por síntesis translesión o por recombinación. Es posible que esto mismo pueda ocurrir en células de mamíferos pero no existen hasta el momento evidencias directas de que ello sea así, sin embargo el hecho de que la RH sea eficazmente inducida a nivel de horquillas estancadas en células de mamíferos en ausencia de fracturas dobles sustenta el modelo bacteriano indicando que las estructuras en pie de pollo son también substrato para la RH en células de mamíferos. El modelo actual de salto (bypass) y restauración de la horquilla de duplicación estancada implica reversión de la horquilla causada posiblemente por un fenómeno torsional positivo en el ADN o bien por acción enzimática. (Fig 13a ). Primero, una hebra se devolverá para formar una estructura en pie de pollo (Fig 13b ). Esta y otras estructuras intermediarias son substrato del complejo endonucleasa humano Mus81-Eme1 que es inducido cuando las células son tratadas con inhibidores de la duplicación. El rompimiento de estas estructura produce una doble fractura idéntica a la producida en una horquilla de duplicación colapsada (Fig 13c). Este evento de reparación por recombinación homóloga resulta en un ICH (Fig 13d). Sin embargo, la horquilla de duplicación puede continuar en reversa y producir una estructura en pie de pollo que incluye una HJ en la unión de cuatro ramas (Fig 13e). Tras la síntesis y/o reparación de este extremo de ADN que sobresale, esta HJ puede migrar hacia atrás y resumir la duplicación sin recombinación. Sin embargo la HJ puede ser blanco de una actividad resolvasa (Fig 13f) y formar una fractura de doble hebra. La RH podría de esta forma podría reparar esta fractura por el mismo mecanismo en la reparación de un extremo y de esta forma restituir el proceso de duplicación (Fig 13g). Alternativamente, la RH puede activarse en el extremo de ADN que emerge antes de que la HJ (a) Fractura ds clásica 2- Extremos de la fractura (b) horquilla colapsada 1- extremo de hebra doble 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ (c) horquilla estancada Pie de Pollo 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ Figura 12. Estructura de los substratos para recombinación en células de mamíferos. (a) Una fractura de doble hebra clásica deja dos extremos libres que pueden iniciar recombinación. (b) Una fractura de hebra simple puede convertirse en fractura de hebra doble durante la duplicación lo que colapsa la horquilla dejando un extremo de ADN libre que puede ser usado para recombinación. (c) La horquilla de duplicación puede toparse con un bloqueo (T-T) en su camino lo que conduce a un atascamiento de la horquilla. Bajo esas condiciones la horquilla puede ponerse en reversa originando estructura con forma de “pie de pollo” que pueden servir como substrato para recombinación, (Tomado de Helleday, 2003). a) Horquilla estancada 5’ 3’ b) Medio pie de pollo e) Pie de pollo j) Union Holliday con 4 brazos de ADN 5’ 3’ 5’ 3’ 3’ 5’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ Resolución de HJ c) Horquilla colapsada f) Formación DSB h) Duplicación restituida 3’ 5’ 5’ 3’ 3’ 5’ 5’ 3’ HR 5’ 3’ 3’ HR d) Reparación de un extremo/SCE 5’ 3’ 5’ g) Duplicación restituida 3’ i) Conversión génica 3’ 5’ 5’ 5’ 3’ Figura 13. 5’ 3’ 5’ 3’ Reparación por recombinación homóloga en una horquilla atascada. (a) una horquilla de duplicación se atasca por un obstáculo en el templado de ADN. (b) La horquilla se puede poner en reversa facilitado por una torsión positiva en el ADN o por acción enzimática. Se forma un intermediario mitad de un pie de pollo. (c) Esta y otras estructuras intermediarias pueden ser atacados por endonucleasas originando una horquilla colapsada con un extremo libre. (d) esta puede ser reparada por recombinación homóloga como se muestra en la figura 12 y resultará en un ICH. (e) La horquilla de duplicación también puede ponerse en reversa para formar un pie de pollo completo con una HJ. Aquí, el pie de pollo es representado como una horquilla en reversa o como un intermediario de recombinación que son idénticos. (f) La HJ puede ser cortada para formar un extremo de ADNds que puede ser reparado por recombinación homóloga para restaurar la duplicación (g). Alternativamente, el extremo libre de ADN que sale fuera del pie de pollo puede ser usado directamente en recombinación homóloga para restituir la duplicación (h). Esto evita la formación de una fractura de hebra doble. (i) Resolución de la HJ sin entrecruzamiento que conduce a conversión génica. Las flechas señalan la dirección de la síntesis de ADN, las líneas negras corresponden al templado y las líneas rojas a ADN sintetizado de novo. Las puntas de flechas vacías indican sin entrecruzamiento. No se muestra el proceso que implica entrecruzamiento. (Tomado de Helledey, 2003). 27 se destruya y restitutir la duplicación (Fig 13h). Todo indica que el resultado de la reparación por recombinación homóloga es siempre un evento de conversión génica. Importa que analicemos brevemente algunas propiedades generales de la HJ: a. Se pueden armar todos los pares de bases b. El punto de entrecruzamiento puede “desplazarse” rompiendo un conjunto de puentes de hidrógeno y simultáneamente armando otros(recordemos que este proceso no requiere de energía y así las HJ pueden desplazarse en ausencia de cualquier “ayuda” de proteínas. c. Así, la HJ puede desplazarse todo el recorrido y resolver esta situación. Se sabe que en procariontes existe un complejo set de proteínas que facilitan el desplazamiento de la UH. Un ejemplo de ello es catalizado por las proteínas bacterianas RuvA y RuvB. RuvA reconoce la forma de la HJ. Ruv B se une a RuvA y forma un anillo alrededor de una de los brazos del ADN. RuvB es una helicasa que puede tirar el ADN a través de su centro. Dos hexámeros de RuvB sobre ambos lados de la HJ promoverán así un rápido desplazamiento de ella. Se ha demostrado que el complejo RuvAB se comporta como un verdadero complejo proteico motor, capaz de conducir la migración de las HJ a una velocidad promedio de 43 pares de bases/segundo a lo largo de muchos kilobases de secuencias homólogas (Dawid et al., 2004) Mientras RuvA y Ruv B promueven el desplazamiento de la HJ y su disolución, otra forma alternativa de solucionar este tipo de estructura es –como se mencionaba- mediante la actividad de una resolvasa. La proteína bacteriana RuvC, es una resolvasa que corta en posiciones simétricas en los dos segmentos de ADN homólogos. El corte de la HJ puede originar dos productos alternativos dependiendo de cuál par de hebras es atacada por la enzima. En el caso de las horquillas de duplicación atascadas en ambos casos origina una molécula de ADN intacta y un extremo roto. La proteína RecA ha sido un modelo de estudio para el entendimiento de cómo una proteína cataliza la recombinación genética homóloga. Algunas proteínas parecidas a RecA, incluyendo a Rad51 humana parecen funcionar como filamentos helicoidales formados sobre el ADN. 28 Recientemente se ha visto que p53 posee un efecto inhibitorio de intercambio de hebras, así como sobre el proceso de puesta en reversa de horquillas estancadas. Se postula que p53 podría así cumplir una función importante en la estabilización genómica previniendo el reordenamientos genéticos promovidos por Rad51, lo que sustenta la hipótesis que p53 contribuye a la estabilidad genómica por una modulación independiente de transcripción de recombinación homóloga. (Yu et al.,2004; Yoo et al., 2004 ; Kumari et al., 2004) Se ha comprobado además que Rad51 esta involucrada en mantenimiento de telómeros (Tarsounas et al., 2004) y que su actividad ATPásica asociada es modulada por Ca++ (Brugreev y Mazin, 2004). Recientes análisis del filamento cristalizado de Rad51 muestra 6 ejes de simetría y la alternancia de interfases proteína –proteína es levemente distinta lo que sugiere que la unidad funcional de esta proteína puede ser un dímero (Conway et al., 2004). Recientes hallazgos también demuestran que Rad51 se requiere para el procesamiento de las UH en células de mamíferos. Ello se ha visto en células que portan mutación para los genes de recombinación Rad51C o XRCC3 las cuales poseen bajos niveles de actividad resolvasa. Además la eliminación de Rad51C de extractos humanos fraccionados causa pérdida de actividad de desplazamiento y actividad resolutiva pero esas funciones pueden restaurarse por complementación con una variedad de complejos parálogos de Rad51 que contienen Rad51C (Liu et al., 2004). Además se ha demostrado que la sobreexpresión de Rad51 promueve aneuplodía y múltiples reordenamientos cromosómicos (Richardson et al., 2004) y produce fallos en el tránsito por el ciclo celular y conduce a apoptosis (Yoo y McKee, 2004) Como se observa, la recombinación homóloga (RH) es esencial para la supervivencia celular en mamíferos. Los productos de la RH pueden ser: Intercambios entre Cromátidas Hermanas (ICH), conversión génica, deleciones o duplicaciones in tandem. La RH está involucrada en la reparación de fracturas de hebra doble y lesiones que ocurren en el ADN en la horquilla de replicación. Una fractura de hebra doble puede resultar en deleción, duplicación in tandem o conversión génica después de reparación por recombinación de los dos extremos. Por el contrario los ICH pueden ser el resultado de reparación por recombinación de un extremo en la horquilla de duplicación estancada, es decir, una fractura de hebra simple se convierte en fractura de hebra doble en la horquilla de duplicación puede ocurrir en ausencia de un intermediario y puede resultar ya sea en ICH o conversión génica.(Helleday, 2003) 29 El proceso de reiniciar la duplicación de la información genética por recombinación, proceso en el que está involucrada PARP1 (Poli ADP ribosa polimerasa (Yang et al, 2004) puede dejar atrás una UH lo cual puede resolverse de dos maneras. Una de ellas origina entrecruzamiento entre cromátidas hermanas replicadas. Ello es genéticamente invisible ya que ambas copias poseen la misma información genética, sin embargo –como se decía previamente- pueden ponerse en evidencia a través del ensayo de Intercambio de Cromátidas Hermanas (ICH). Para ello las células son marcadas con el análogo de timina, BrdU. Después ellas son trasladadas a un medio libre de BrdU por dos generaciones. Bajo esas circunstancias, una de las hebras del ADN de las cuatro en un par de cromátidas hermanas deberían marcarse con BrdU. Los ICH se evidencian por modificaciones en ese patrón uniforme de tal forma que parte de cada cromátida hermana posee BrdU. Recientemente se han presentado evidencias que indican que el complejo Mre11 (Mre11, Rad50, Nbs1) juega un rol fundamental en cada uno de los punto del metabolismo de fracturas cromosómicas. El complejo actúa como sensor de fracturas e interviene directamente en la activación y propagación de señales que gobiernan las funciones de los puntos de control en respuesta a daño en el ADN. Además, Mre11 influencia la reparación recombinacional del ADN promoviendo la recombinación entre cromátidas hermanas (Stracker, 2004). Conclusión finales. Desde que Novell hace poco más de un cuarto de siglo describiera el fenómeno de inestabilidad cromosómica, se han realizado ingentes estudios para determinar sus posibles causas. Actualmente se piensa que el fenotipo de inestabilidad cromosómica se origina por un fallo o un conjunto de ellos que conducen a una pérdida en la regulación de ciertas proteínas asociadas con: puntos de control del ciclo celular, segregación de cromosomas, ciclo del centrosoma, integridad de telómeros y reparación recombinacional. En este capítulo se han soslayado además algunos factores de naturaleza epigenética que también puede afectar la regulación normal de la expresión de proteínas y con ello originar fenotipos de inestabilidad. Procesos de hiper o hipoacetilación, metilación, y fosforilación de histonas juegan un rol importante a través del denominado “Código Histona” (Momparler, 2003; Oligny, 2003; Hake et al., 2004; Honda et al., 2004; Takay et al., 2004; Wang et al., 2004). Sin embargo a pesar del progreso experimentado en los últimos años, los mecanismos involucrados aún no se esclarecen del todo 30 y persisten muchas interrogantes que hacen del tema de inestabilidad cromosómica un tópico de mucho interés para dilucidar las bases cromosómica ligadas a la progresión tumorogénica. Agradecimiento El autor expresa su gratitud al Sr. Gerardo Quezada Silva por su excelente colaboración en la gráfica del presente capítulo. El autor agradece además, el financiamiento para actualización bibliográfica asignado al proyecto de investigación 012407-2 de la Universidad del Bío-Bío. Bibliografía Amon,A. 2001. Together until Separin do us part. Nat Cell Biol 3(1), 12-14 Ang XL, Harper JW. 2004 Interwoven ubiquitination oscillators and the control of the cell cycle transitions. Sci STKE, 242, p31 Assenmacher N, Hopfner KP. 2004. MRE11/RAD50/NBS1: complex activities. Chromosoma DOI: 10.1007/s00412-004-0306-4 Bailey SM, Goodwin EH. 2004. DNA and telomeres: beginnings and endings. Cytogenet Genome Res 104(1-4), 109-115 Barr FA, Silljé HHW, Nigg EA. 2004. Pol-like kinases and the orchestration of cell division. Nature Reviews Molecular Cell Biology 5, 429-441 Bashir Y, Dorrello NV, Amador V, Guardavaccaro D, Pagano M. 2004. Control of the SCF (Sk2Cks1) ubiquitin ligase by the APC/C(Cdh1) ubiquitin ligase. Nature, 428 (6779),190-193 Bharadwaj R, Yu H. 2004. The spindle checkpoint, aneuploidy, and cancer. Oncogene,23(11), 2016-2027. Bowers AJ, Boylan JF. 2004. Nek8, a NIMA family kinase member, is orverexpressed in primary human breast tumor. Gen ,328,135-142 31 Bugreev DV, Mazin AV. 2004. Ca++ activates human homologous recombination protein Rad51 by modulating its ATPase activity. Proc Natl Acad Sci 101(27), 9983-9993 Conway AB, Lynch TW, Zhang Y, Fortin GS, Fung CW, Symington LS, Rice PA. 2004. Crystal structure of a Rad51 filament. Nat Struct Mol Biol in press. Crane R, Gadea B, Littlepage L, Wu H, Ruderman JV. 2004. Aurora A, meiosis and mitosis. Biol Cell, 96(3), 215-229 Chang GK, Yen TJ. 2003. The mitotic checkpoint: A signaling pathways that allows a single unattached kinetochore to inhibit mitotic exit. Prog Cell Cycle Res 5, 431-439 Chuang TC, Moshir S, Garini Y, Chuang AY, Young IT, Vermolen B, Doel RvR, Mougey V, Perrin M, Braun M, Kerr PD, Fest T, Boukamp P, Mais S. 2004. The three dimensional organization of telomeres in the nucleus of mammalian cells. BMC Biol 2(1), 12-24 Dagenbach EM, Endow SA. 2004. A new kinesin tree. J.Cell Sci, 117(pt1), 3-7 D’Amour D, Jackson SP.2002. The Mre11 complex : at the cross road of DNA repair and checkpoint signaling. Nat.Rev Mol Cell Bio 3, 449-459 D’Assoro AB, Busby R, Suino K, Delva E, Almodovar-Mercado GJ, Johnson H, Folk C, Descamps S, Prigent C. 2002. Aurora-A,-B et –C à l’aube d’une nouvelle connexion entre l’amplification des centrosomes, l’aneuploïdie et le cancer. Medicine/Science 18, 474-480 D’Assoro AB, Busby R, Suino K, Delva E, Alomodovar-Mercado GJ, Hohnson H, Folk C, Farrugia DJ, Vasile V, Stivala F, Salisbury JL. 2004. Genotoxic stress leads to centrosome amplification in breast cancer cell lines that have incactive G1/S cell cycle checkpoint. Oncogene 23(23), 4068-4075 Dawid A, Croquette V, Grigoriev M, Heslot F. 2004. Single-molecule study of RuvAB-mediated holliday junctions migration. Proc Natl Acad Sci (USA) 101(32), 1611-11616. 32 Duensing S, Lee BH, Cin PD, Müngen K. 2003. Excessive centrosome abnormalities without ongoing numerical chromosome instability in a Burkitt’s lymphoma. Molecular Cancer 2:30. doi:10.1186/1476-4598-2-30 Dutertre S, Cazales M, Quaranta M, Froment C, Trabut V, Dozier C, Mirey C, Bouche JP, TheisFebvre N, Schmitt E, Monsarrat B, Prigent C, Ducommun B. 2004. Phosphorilation of CDC25B by Aurora-A at the centrosome contributes to the G2-M transition. J.Cell Sci. 117 (Pt12) 25232531. Erson AE, Petty AM. 2004. CHFR-associated early G2/M checkpoint defects in breast cancer cells. Mol Carcinog 39(1),26-33 Farrugia DJ, Vasile V, Stivala F, Salisbury JL. 2004. Genotoxic stress leads to centrosome amplification in breast cancer cell lines that have an inactive G1/S cell cycle chechk point. Oncogene, 23(23), 4068-4075 Fukagawa T, Nogami M, Yoshikawa M, Ikeno M, Okazaki T, Takami Y, Nakayama T, Oshimura M. 2004. Dicer is essential for formation of the heterochromatin structure in vertebrate cells. Nat.Cell Biol, in press. Fukasawa K. 2004. Introduction. Centrosome. Oncogene, 21(49), 6140-6145 Gisselsson D. 2003. Chromosomal Instability in Cancer- How, when, and why. Adv Cancer Res, 87, 1-29 Gollin SM. 2004. Chromosomal Instability. Curr Opin Oncol 16(1), 25-31. Hagstrom KA, Meyer BJ. 2003. Condensin and cohesin: more than chromosome compactor and glue. Nat Rev Genet,4, 520-534 Hake SB, Xiao A, Allis CD. 2004. Linking the epigenetic “language” of covalent histone modifications to cancer Br J Cancer 90(4), 761-769. 33 Helleday T. Pathways for mitotic homologous recombination in mammalian cells. 2003. Mutat Res. 532(1-2), 103-115. Hinchchliffe EH. 2003. Cell cycle: seeking permission from the mother centriole. Curr Biol 13(16), 646-648. Honda T, Tamura G, Waki T, Kawata S, Nishizuka S, Motoyama T. 2004. Promoter hypermethylation of the Cfhr gene in neoplastic and non-neoplastic gastric epithelia. Br.J Cancer 90(10), 2013-2016. Hoque MT, Ishikawa F. 2002. Cohesin defects lead to premature sister chromatid separation, kinetochore dysfunction, and spindle-assembly checkpoint activation. J. Biol. Chem 277(44), 42306-42314. Ichim TE, Li M, Qian H, Popov IA, Rycerz K, Zheng X, White D, Zhong R, Min WP. 2004. RNA interference: a potent tool for gene-specific therapeutics Am J Transplant 4(8), 1227-1236. Kamai T, Yamanishi T, Shirataki H, Takagi K, Asami H, Ito Y, Yoshida K. 2004. Overexpression of RhoA, Rac1 and Cdc42 GTPases is associated with progression in testicular cancer Clin Cancer Res, 10(14), 4799-4805 Kang HC, Kim IJ, Park JH, Shin Y, Park HW, Ku JL, Yang HK, Lee KU, Choe KJ, Park JG. 2004. Promoter hypermethylation and silencing of CHFR mitotic stress checkpoint gene in human gastric cancers. Oncol Rep 12(1), 129-133 Kawamura K, Izumi H, Ma Z, Ikeda R, Moriyama M, Tanaka T, Nojima T, Levin LS, FujikawaYamamoto K, Susuki K, Fukasawa K. 2004. Induction of centrosome amplification and chromosome instability in human bladder cancer cell by p53 mutation and cyclin E overexpression. Cancer Res 64(14), 4800-4809 Kent, OA and MacMillan, AM. 2004. RNAi: running interference for the cell. Org.Biomol.Chem 2(14), 1957-1961 34 Kim JC, Badano JL, Sibold S, Esmail MA, Hill J, Hoskins BE, Leitch CC, Venner K, Ansley SJ, Ross AJ, Leroux MR, Katsanis N, Beales PL. 2004. The Bardet-Biedl protein BBS4 targets cargo to the pericentriolar region and is required for microtubule anchoring and cell cycle progression Nat Genet, 36(5), 462-470 Kumari A, Schultz N, Helleday T. 2004. p53 protects from replication-associated DNA doublestrand breaks in mammalian cells. Oncogene 23(13), 2324-2329 Lau NC, Bartel DP. 2003. Interferencia de ARN. Investigación y Ciencia, octubre, 6-13 Liu Y, Masson JY, Shah R, O’Regan P, West SC. 2004. Rad51 is required for Holliday junction processing in mammalian cells. Science 303(5655), 243-246 Luo X, Tang Z, Xia G, Wassmann K, Matsumoto T, Rizo J, Yu H. 2004. The Mad2 spindle checkpoint protein has two distinct natively folded states. Nat Struct Mol Biol 11(4), 338-345 Ma Y, Pannicke U, Schwarz K, Lieber MR, 2002. Hairpin opening and overhang processing by an Artemis/DNA dependent protein kinase complex in non-homologous end joining and V(D)J recombination. Cell, 781-794 Maine EM 2001 RNAi As a tool for the undestanding germline development in Caenorhabditis elegans: uses and caution. Dev. Biol, 239(2) 177-189. Martínez-Campos M, Basto R, Baker M, Raff JW. 2004. The Drosophila pericentrin-like protein is essential for cilia/flagella function, but appears to be dispensable for mitosis J.Cell Bio 165(5), 673-683 Mathieu N, Pirzio L, Freulet-Marriere MA, Desmaze C, Sabatier L. 2004. Telomeres and chromosomal instability. Cell Mol Life Sci. 61(6), 641-656 Mazumdar M, Sundareshan S, Misteli T. 2004. Human chromokinesis KIF4A functions in chromosome condensation and segregation. J Cell Biol (in press) 35 Melloy PG, Holloway SL. 2004. Changes in the localization of Saccharomyces cerevisiae anaphase promoting complex upon microtubule depolymerization and spoindle checkpoint activation. Genetics 167(3),1079-1094. Meraldi P, Draviam VM, Sorger PK. 2004a. Timing and checkpoint in the regulation of mitotic progression. Dev Cell (), 7(1), 45-60. Meraldi P, Honda R, Niggs EA. 2004b. Aurora kinases link chromosome segregation and cell division to cancer susceptibility Curr Opin Genet Dev 14(1), 29-36. Michel L, Díaz-Rodríguez E, Narayan G, Hernando E, Murty VV, Benezra R. 2004. Complete loss of the tumor supresor MAD2 causes premature cyclin B degradation and mitotic failure in human somatic cellz. Proc Natl Acad Sci USA 101(13), 4459-4464 Momparler RL. 2003. Cancer epigenetics Oncogene 22(42), 6479-6483. Moshe Y, Boulaire J, Pagano M, Hershko A. 2004. Role of Polo-like kinase in the degradation of early mitotic inhibitor 1, a regulator of the anaphase promotin complex/cyclosome Proc Natl Acad Sci (USA), 101(21), 7937-7942. Narumiya S, Oceguera-Yañez F, Yasuda S. 2004. A new look at Rho ATPases in cell cycle: Role in kinetochore-microtubule attachment. Cell Cycle, 3(7), 465-475. Nigg EA. 2002. Centrosome aberrations: cause or consequence of cancer progression? Nat Reviews Cancer 2, 815-825 Nojima H. 2004. G1 and S phase checkpoints, chromosome instability and cancer. Methods Mol Biol; 280, 3-50. Nowell PC. 1976. The clonal evolution of tumor cell populations. Science,194, 23-28 Oligny LL. 2003. Cancer and epigenesist: a developmental perspective. Adv Pediatr 50, 59-80. 36 Ono T, Fang Y, Spector DL, Hirano T. 2004. Spatial and temporal regulation of condensina I y II in mitotic chromosomes assembly in human cells. Mol Biol Cell 15(7), 3296-3308 Ou YY, Zhang M, Chi S, Matyas JR, Rattner JB. 2003. Higher order structure of the PCM adjacent to the centriole. Cell Motil Cutoskeleton 55(2), 125-133 Papin C, Rouget C, Lorca T, Castro A, Mandart E. 2004. XCdh1 is involved in progesteroneinduced oocyte maduration. Dev Biol 272(1), 66-75 Patel D, Incassati A, Wang N, McCance DJ. Human papillomavirus type 16 E6 y E7 cause polyploidy in human keratinocytes and up-regulation of G2-M-phase protein. Cancer Res (2004) 64(4), 1299-1306 Pelletier L, Ozlu N, Hannak E, Cowan C, Habermann B, Ruer M, Muller-Reichert T, Hyman AA. 2004. The Caenorhabditis elegans centrosomal protein SPD-2 is required for both pericentriolar material recruitment and centriole duplication. Curr Biol 14(10), 863-873 Plug-De Maggio AW, Sunsdvold T, Wurscher MA, Koop JI, Klingelhutz AJ, McDougall JK. 2004. Telomere erosion and chromosomal stability in cells expressing the HPV oncogene 16E6. Oncogene 23(20), 3561-3571. Prasanth SG, Prasanth KV, Siddiqui K, Spector DL, Stillman B. 2004. Human Orc2 localizes to centrosomes and heterochromatin during chromosome inheritance. EMBO J 23(13), 2651-2663 Rangasamy D, Greaves I, Tremethick DJ. 2004. RNA interference demonstrate a novel role for H2A.Z in chromosome segregation. Nat. Struct Mol Biol 11(7), 650-655 Rhodes D, Fairall L, Simonsson T, Court R, Cahpman L. 2002. Telomere Architecture. EMBO reports 3(12), 1139-1145 Richardson C, Stark JM, Ommundsen M, Jasin M. 2004. Rad51 overexpression promotes alternative double-strand break repair pathways and genome instability. Oncogene 23(2), 546553 37 Robison JG, Elliott J, Dixon K, Oakley GG. 2004. Replication protein A and the Mre11.Rad50.Nbs1 complex co-localize and interact at sites od stalled replication forks. J Biol Chem 279(33), 34802-34810 Scherr M, Steinmann D, Eder M. 2004. RNA interference (RNAi) in hematology Ann Hematol 83(1), 1-8. Stracker TH, Theunissen JW, Morales M, Pettrini JH. 2004. The Mre11 complex and the metabolism of chromosome breaks: the importance of communicating and holding things together. DNA Repair 3(8-9), 845-854 Sudakin V, Yen TJ. 2004. Purification of the mitotic checkpoint complex, an inhibitor of the APC/C from HeLa cells Methods Mol Biol, 281, 199-212 Sudo T, Ueno NT, Saya H. 2004. Functional analysis of APC-Cdh1. Methods Mol Biol. 281, 189198 Takai N, Desmond JC, Kumagai T, Gui D, Said JW, Whittaker S, Miyakawa I, Koeffler HP. 2004. Histone deacetylase inhibitors have a profound antigrowth activity in endometrial cancer cells. Clin Cancer Res, 10(3), 1141-1149. Tarapore P, Fukasawa K. 2002 Loss of p53 and centrosome hyperamplification. Oncogene,21(49), 6234-6240 Tarsounas M, Munoz P, Claas A, Smiraldo PG, Pittman DL, Blasco MA, West SC. 2004. Telomere maintenance requieres the RAD51 recombination repair protein. Cell 117(3), 337-347. Thompson HM, Cai H, Chen J, Euteneuer U, McNiven MA. 2004. Dynamin 2 binds gammatubulin and participates in centrosome cohesion. Nat.Cell Biol 6(4), 335-342 Tyers M , Jorgenson P. 2000. Proteolisis and the cell cycle : with this ring I thee destroy. Curr op in Genet and Develop 10, 54-64 Uhlmann F. 2004. The mechanism of sister chromatid cohesion. Exp Cell Res 15; 296(1):80-5. 38 Van Vugt MA, Van De Weerdt BC, Vader G, Janssen H, Calafat J, Klompmaker R, Wolthuis RM, Medema RH. 2004. Polo like kinase-1 is required for bipolar spindle formation but is dispensable for APC/Cdc20 activation and initiation of cytokinesis. J Biol Chem (in press) Walker JR, Corpina RA, Goldberg J. 2004. Structure of Ku heterodimer bound to DNA and its implications for double strand break repair Nature 412, 607-614. Wang X, Haber JE.2004.Role of Saccharomyces single–stranded DNA-binding protein RPA in the strand invasion step of double strand break repair PLoS Biol 2(1), E21 Wang Y, Fischle W, Cheung W, Jacobs S, Khorasanizadeh S, Allis CD. 2004. Beyond the double helix: Writing and reading the histones code. Novartis Found Symp 259, 3-17 Wayt Gibbs, W. 2004. El genoma oculto Investigación y Ciencia, enero, 23-28 Wei W, Ayad NG, Wan Y, Zhang GJ, Kirschner MW, Kaelin WG jr. 2004. Degradation of the SCF component Skp2 in cell cycle pahes G1 by the anaphase promoting complex. Nature 428(6979), 194-198 Yan GR, Cao Y. 2004. Recent progress in molecular mechanism of chromosome instability and their association with carcinogenesis. Ai Zheng, 23(3), 353-357 Yang YG, Cortes U, Patnaik S, Jasón M Wang ZQ. 2004. Ablation of PARP1 does not interfere with the repair od DNA double-strand breaks, but compromises the reactivation of stalled replication forks. Oncogene 23(21), 3872-2882. Yasuda S, Oceguera-Yanez F, Kato T, Okamoto M, Yonemura S, Terada Y, Ishizaki T, Narumiya S. 2004. Cdc42 and mDia3 regulate microtubule attachment to kinetochores. Nature. 428(6984), 767-71. Ye JZ, de Lange T. (2004) TIN is a tankyrase 1PARP modulator in the TRF1 telomere lenght control complex. Nat Genet 36(6), 618-623 39 Yoo D, Wang Y, Stapleford K, Wiemuller L, Chen J. 2004. P53 inhibits strand exhange and replication fork regression promoted by human Rad51 J Mol Biol, 336(3), 639-654. Yoo S, McKee BD. 2004. Overexpression of Drosophila Rad51 protein (DmRad51) disrupt cell cycle progression and leads to apoptosis Chromosoma 113(2), 92-101. Yu H. 2002. Regulation of APC-Cdc20 by the spindle checkpoint. Curr Opin Cell Biol,14, 706714 Yu X, VanLoock MS, Yang S, Reese JT, Egelman EH. 2004. What is the structure of rec-DNA filament? Curr Prot Pept Sci, 5(2), 73-79 Zhu XD, Kuster B, Mann M, Petrini JH, Lange,T. 2000 Cell-cycle-regulated association of RAD50/MRE11/NBS1 with TRF2 and human telomeres. Nature Genet. 25, 347–352.