Jerarquía Taxonómica - Laboratorio de Genética Molecular
Anuncio

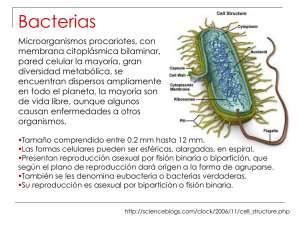
Laboratorio de Genética Molecular División de Estudios de Posgrado e Investigación Facultad de Odontología, UNAM CURSO DE MICROBIOLOGÍA Alta especialidad en implantología Cuarta parte Dra. Laurie Ann Ximénez-Fyvie Mtra. Adriana Patricia Rodríguez-Hernández Métodos para el estudio de microorganismos Identificación fenotípica •Tinción de Gram •Morfología celular •Motilidad •Tolerancia al oxígeno •Morfología de colonia •Tipificación bioquímica •Perfiles de proteínas celulares Identificación genética •Hibridaciones DNA-DNA •PCR •Secuenciación 16S rRNA Identificación fenotípica Definición Secuencia consecutiva de métodos de “microbiología tradicional” que en conjunto llevan a la identificación de especies bacterianas en cultivo; mediante la determinación y posterior seguimiento de sus características fenotípicas y metabólicas a través de diagramas de flujo de identificación aceptados Aplicación • Identificación y cuantificación de especie cultivable previamente caracterizadas o de microorganismos predeterminados a partir de muestras clínicas o cultivos puros Ventajas • Permite la realización de pruebas de susceptibilidad antimicrobiana posteriores o en paralelo a la identificación • Es posible realizar la cuantificación de las especies evaluadas Desventajas En comparación a la identificación genética: • Más laborioso • Mayor tiempo y costo • Difícil manejo de muestras bucales y de otros sitios con proporciones elevadas de especies fastidiosas • Subestimación de especies fastidiosas • No permite la identificación de especies no-cultivables Identificación fenotípica- Diagrama de flujo Tinción de Gram Morfología celular Motilidad Tolerancia al O2 Morfología de colonia Tipificación bioquímica Perfiles de proteínas celulares Identificación fenotípica- Tinción de Gram Gram positivo Gram negativo Identificación fenotípica- Morfología celular Coco Bacilo Espirilo Pleomórfico Identificación fenotípica- Motilidad Contraste de Fases Campo Obscuro En Agar Tubo de Punción •Sin motilidad •De nado (Swimming) •De deslizamiento (Glidding) •En espasmos (Twitching) Identificación fenotípica (Tolerancia al oxígeno) •Anaerobio estricto (en ausencia de O2) •Microaerofílico (en conc. bajas de O2) •Capnofílico (en presencia de CO2) •Anaerobio facultativo (en presencia/ausencia de O2) •Aerobio estricto (en presencia de O2) Identificación fenotípica- Morfología de colonia •Color •Tamaño •Forma •Textura •Consistencia •Periferia •Adherencia al agar •Formación de fosas •Propiedades hemolíticas •Fluorescencia con luz UV Identificación fenotípica- Tipificación bioquímica •Fermentación de carbohidratos •Productos terminales •Catalasa •Oxidasa •Coagulasa •Reacciones de aglutinación Identificación fenotípica- Perfiles de proteínas celulares SDS-PAGE (sodium dodecyl sulfate - polyacrylamide gel electrophoresis) Métodos genéticos- Hibridaciones DNA-DNA Definición Método no-dependiente del cultivo bacteriano para la identificación de especies; mediante la detección de fragmentos cortos de DNA marcados (sondas) tras su enlace a moléculas de DNA complementarias en una muestra (templete) Aplicación • Identificación y cuantificación de microorganismos predeterminados a partir de muestras clínicas o cultivos puros Ventajas En comparación al resto de las pruebas de identificación: • Capacidad para evaluar mayor número de especies y muestras En comparación a la identificación fenotípica: • Menor tiempo y costo • Fácil manejo de muestras bucales y de otros sitios con proporciones elevadas de especies fastidiosas • Permite la identificación de especies no-cultivables En comparación a otras pruebas genéticas: • Es posible cuantificación las especies evaluadas Desventajas En comparación a la identificación fenotípica: • No permite la realización de pruebas de susceptibilidad antimicrobiana en paralelo a la identificación En comparación a otras pruebas genéticas: • Menor sensibilidad y especificidad Métodos genéticos- Hibridaciones DNA-DNA •Sonda: fragmento de DNA marcado con una molécula reportadora que permite su detección después de la hibridación •Molécula Reportadora Radioactiva: Isótopos con emisiones (generalmente P32) No-Radioactiva: Digoxigenina, Fluorescina, Biotina, etc. •Especificidad: determinada por el diseño de la sonda •Sensibilidad: determinada por el tipo y concentración de la sonda •Southern Blot Blanco = DNA Sonda = DNA •Northern Blot Blanco = RNA Sonda = DNA •Western Blot Blanco = Proteína Sonda = Anticuerpo Métodos genéticos- Hibridaciones DNA-DNA “MiniSlot” Canales abiertos Membrana de nylon “MiniBlotter” Canales de hibridación Membrana de nylon Filtros Socransky et al. Biotechniques 1994 Métodos genéticos- Hibridaciones DNA-DNA Socransky et al. Biotechniques 1994 Métodos genéticos- Hibridaciones DNA-DNA Especie Actinomyces georgiae Actinomyces israelii Actinomyces meyeri Actinomyces naeslundii stp. 1 Actinomyces odontolyticus Actinomyces viscosus Aggregatibacter actinomycetemcomitans Campylobacter gracilis Campylobacter rectus Campylobacter showae Capnocytophaga gingivalis Capnocytophaga ochracea Capnocytophaga sputigena Dialister pneumosintes Eikenella corrodens Eubacterium nodatum Eubacterium saburreum Filifactor alocis Fusobacterium nucleatum Fusobacterium periodonticum ATCC 49285 12102 35568 12104 17929 43146 * 33236 33238 51146 33624 27872 33612 33048 23834 33099 33271 35896 † 33693 Especie Neisseria mucosa Parvimonas micra Porphyromonas asaccharolytica Porphyromonas gingivalis Prevotella intermedia Prevotella loescheii Prevotella melaninogenica Prevotella nigrescens Propionibacterium acnes Selenomonas noxia Streptococcus anginosus Streptococcus constellatus Streptococcus gordonii Streptococcus intermedius Streptococcus mitis Streptococcus oralis Streptococcus sanguinis Tannerella forsythia Treponema denticola Veillonella parvula * Serotipos a: 43717 & b: 43718 † Subespecies nucleatum: 25586, polymorphum: 10953 & vincentii: 49256 ATCC 19696 33270 25260 33277 25611 15930 25845 33563 6919 43541 33397 27823 10558 27335 49456 35037 10556 43037 35405 10790 Métodos genéticos- PCR Definición Método no-dependiente del cultivo bacteriano para la identificación de especies; mediante la amplificación de porciones específicas de DNA en una muestra (templete) Aplicación • Identificación de microorganismos predeterminados a partir de muestras clínicas o cultivos puros Ventajas Desventajas En comparación al resto de las pruebas de identificación: • Mayor sensibilidad En comparación a la identificación fenotípica: • Menor tiempo y costo • Fácil manejo de muestras bucales y de otros sitios con proporciones elevadas de especies fastidiosas • Permite la identificación de especies no-cultivables En comparación a la identificación fenotípica: • No permite la realización de pruebas de susceptibilidad antimicrobiana en paralelo a la identificación En comparación a la identificación fenotípica e hibridaciones DNA-DNA: • No permite la cuantificación de especies (excepto PCR en tiempo real) En comparación a hibridaciones DNA-DNA: • Bajo número de especies evaluadas Métodos genéticos- PCR •Ciclos repetitivos de desnaturalización, alineamiento de primers y extensión para producir múltiples copias de una secuencia determinada de DNA •Especificidad: determinada por el diseño de los primers •Sensibilidad: hipotéticamente 1 célula PCR Métodos genéticos- Secuenciación de la fracción 16S rRNA Definición Método no-dependiente del cultivo bacteriano para la identificación de especies; mediante la determinación de la secuencia en el DNA de la fracción 16S rRNA en una muestra (templete) y su posterior comparación con secuencias publicadas Aplicación • Identificación de cualquier microorganismo a partir de muestras clínicas o cultivos puros Ventajas Desventajas En comparación al resto de las pruebas de identificación: • Mayor especificidad En comparación a la identificación fenotípica: • Menor tiempo y costo • Fácil manejo de muestras bucales y de otros sitios con proporciones elevadas de especies fastidiosas • Permite la identificación de especies no-cultivables En comparación a la identificación fenotípica: • No permite la realización de pruebas de susceptibilidad antimicrobiana en paralelo a la identificación En comparación a la identificación fenotípica e hibridaciones DNA-DNA: • No permite la cuantificación de especies En comparación a hibridaciones DNA-DNA: • Bajo número de especies evaluadas Métodos genéticos- Secuenciación de la fracción 16S rRNA •Comparación con bancos de secuencias conocidas •Especificidad: hipotéticamente “absoluta” •Sensibilidad: hipotéticamente 1 célula Secuenciación 16S rRNA