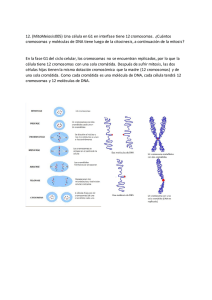
2 La naturaleza del material genético y el código genético. Objetivo
Anuncio

2 La naturaleza del material genético y el código genético. Objetivo: Describir la estructura molecular de ADN y del ARN y la función de los ácidos nucleicos. 2.1 Organización celular. “En muchos aspectos conocemos mejor la estructura del universo que los mecanismos de las células vivientes. Es posible calcular la edad del sol y predecir cuando va a apagarse, pero no podemos explicar como es que un ser humano puede vivir 80 años y un ratón solo 2. Actualmente conocemos las secuencias genómicas de éste y muchas otras especies, pero no podemos predecir como una célula se comporta si mutamos un gen nuevo (o poco estudiado). Las estrellas pueden ser 1043 veces mas grandes, pero una sola célula es mas compleja y con mas productos sorprendentes de las leyes de física y química. A través de la herencia y la selección natural, operando desde inicio de la vida en la tierra hasta el día de hoy, las células vivas se han ido refinando progresivamente y aumentando su maquinaria molecular, y registrando los resultados de sus experimentos como instrucciones genéticas que transfieren a su progenie. Entre mas se estudian las células a nivel molecular, mas resta por descubrir…” Alberts 2008. The molecular biology of the cell. Cromosomas visibles solo durante división celular. Estructura química del ADN descrita en 1953 con características necesarias para funcionar como material genético (heredable): Cromatina es el material genético de la célula en reposo. Complementariedad A-T G-C Variablidad A-T G-C TCTA- DNA Nucleosoma DNA y complejo de histonas (H2A, H2B, H3 y H4) con dos copias c/u. Solenoide Nucleosoma= ocho proteínas histónicas + una fibra de ADN de 200 pares de bases Cromatina condensada=Nucleosoma + 1 histona En el hombre hay 23 pares (cariotipo): 22 autosomas (no sexual) 1 heterosoma (sexual) Hombre XY Mujer XX Cada uno viene por duplicado (diploide): 1 gameto masculino + 1 gameto femenino Haploide solo una copia (durante la meiosis). Características comunes de todas las células •Todas tienen una membrana celular que separa el caos fuera de la célula con el alto grado de organización interno celular. Una célula sin membrana no es una célula. •Toda vida celular contiene DNA como material genético. Todas las células contienen variedades de moléculas de RNA y Proteínas (la mayoría enzimas). •Todas las células están compuestas de los mismos químicos básicos: carbohidratos, proteínas, ácidos nucleicos, grasas y vitaminas. Asas •Todas las células regulan el flujo de nutrientes y desechos que entran y salen. •Todas las células se reproducen y son el resultado de la reproducción. Giro (30 rosetas) * Dominio funcional o unidad de replicación. Cromosoma totalmente condensado (metafase). •Todas las células requieren una fuente de energía. •Todas las células están altamente reguladas mediante sistemas elaborados de detección que les permite estar alertas de cualquier reacción que ocurre a su interior y de las condiciones del ambiente que las rodea. Esta información es continuamente procesada para tomar decisiones metabólicas. 1 Células PROCARIOTAS, células primitivas Son menos complejas Anabaena azolla Cianobacteria, 2.8 billones de años Bacterias Células EUCARIOTAS son mas complejas estructural y bioquímicamente Formas libres Representan un estadio mas avanzado de la evolución al fusionar en una sola célula diversas células procariotas interdependientes. Tienen un nivel de organización mas elevado ya que contienen organelos o estructuras separadas por membranas de otros componentes citoplasmáticos. Tejido de la cebolla Células eucariotas Virus: - No célula!??? - Infectan toda clase de organismos vivos. - Contienen sus propios elementos genéticos (DNA o RNA) - Utiliza sistema de traducción del hospedero para sintetizar sus proteínas. La idea central de la evolución biológica es que todos los seres vivientes sobre la Tierra comparten un ancestro común Spuntik 2008!! T4 Infecta E. coli Los seres humanos no evolucionaron de los chimpancés. Humanos y chimpancés son primos evolutivos y comparten un ancestro común reciente que no fue ni chimpancé ni humano. (Science 3 March 2006.311(5765):1283 – 1287) 2 Dos tipos de células: Principales organelos (célula Eucariota) MITOCONDRIAS En la célula animal son estructuras productoras de ATP = Energía. Funciones: – Respiración. – Biosíntesis de esteroides y algunos ac grasos. – Síntesis de ATP. Contienen DNA y maquinaria para la síntesis de proteínas, similar a la de procariotas (Ribosomas 70S). Tienen doble membrana (como gram -). División por fisión binaria. El humano las hereda completamente de la madre de modo que mutaciones en el DNA mitocondrial dan indicios únicamente del linaje materno. Núcleo (membrana nuclear) Nucleoide (sin membrana) Organelos abundantes y con membrana Organelos escasos y sin membrana DNA linear, sin DNA extracromosomal DNA circular, con DNA extracromosomal Ribosomas grandes (80 S) Ribosomas pequeños (70 S) CLOROPLASTOS En la célula vegetal convierte la luz solar en energía y permite la fotosíntesis fijando dióxido de carbono en forma de azúcar. División por fisión binaria. Tienen su propio DNA, circular, enrollado, sin histonas y con múltiples copias (similar al de cianobacterias). Posen sus propios RNA´s (t y r) Genomas y pliegues membranales similar al de eubacterias. Genoma pequeño sin intrones. Diversas copias del genoma por mitocondria. No tiene histonas asociadas. Una célula con muchas mitocondrias. APARATO DE GOLGI Secreta substancias (glicosilación de proteínas o lípidos, selección, destinación, almacenamiento y distribución de lisosomas y síntesis de polisacáridos de la matriz extracelular). 1 Núcleo (envoltura nuclear). 2 Poro nuclear. 3 Retículo endoplasmatico granuloso (REG). 4 Retículo endoplasmatico liso (REL). 5 Ribosoma sobre el REG. 6 Proteínas transportadas. 7 Vesícula golgiana. 8 Aparato de Golgi. 9 Cara cis del aparato de Golgi. 10 Cara trans del aparato de Golgi. 11 Sacos membranales de un dictyosoma. Aparato de golgi, Regula el trafico vehicular mediante vesículas, modificaciones posttraduccionales (O-Glycosilacion, Sulfatación y Fosforilación). RE Retículo endoplasmatico, no es organelo RER (3), sitio de la traducción y transporte de proteínas. REL (4), síntesis de lípidos, metabolismo de glúcidos (N-Glycosilacion) o almacén de calcio. Polyribosomas y Retículo Endoplasmatico La aparición de la envoltura nuclear permitió que los eucariontes aislaran los procesos genéticos principales, como la auto-duplicación del ADN o la síntesis de ARN. Además esto posibilitó que el ARNm se modifique dentro del núcleo antes de ser traducido en los ribosomas. Estas modificaciones no ocurren en los procariontes, ya que a medida que la ARN polimerasa sintetiza el ARN, simultáneamente el extremo 5’ se une al ribosoma y comienza la traducción. 3 Ribosomas y proteínas Poliribosomas=polysomas Son agrupaciones de ribosomas asentados en el mRNA para sintetizar proteínas. En ocasiones se encuentran libres en el citoplasma (polyribosomas libres). Los ribosomas son pequeñas partículas de ribonucleoproteinas (20x30 nm). Se conforman de dos subunidades. Rutas de secreción proteica • Proteína es una cadena de aminoácidos (aa). • Hay 20 aa, cada uno con diferente comportamiento químico. • Esta cadena no permanece linear sino que se pliega según las propiedades de cada aa. • La conformación de una proteína determina su función. • La secuencia de los diferentes aa produce proteínas con diferentes conformaciones y funciones. • Los genes determinan la secuencia de aa en una proteína. • Cambios en el gen (mutaciones) pueden cambiar la secuencia de aa de la proteína y alterar su conformación y su función. NUCLEO Es el organelo más grande de la célula eucariota. Contiene los cromosomas, DNA y enzimas necesarias para sintetizarlo y transcribirlo. Dirige el desarrollo y funcionamiento de la célula controlando las reacciones químicas del citoplasma y almacena la información necesaria para la división celular. Los eritrocitos (glóbulos rojos) maduros de los mamíferos carecen de núcleo (y de mitocondrias). Existen algunas células eucariotas con 2 o mas núcleos (hongos y ciliados). Rodeado por una envoltura nuclear (6) de doble membrana que se fusiona a intervalos regulares y forma los poros nucleares (3) que permiten intercambios nucelocitoplasmaticos como la salida de mRNA. Separa las reacciones químicas de aquellas del citoplasma. NUCLEO… La membrana externa es continua con el Retículo Endoplasmatico Granuloso (REG, 7) y presenta ribosomas sobre su lado citoplasmático (2). El espacio perinuclear entre las dos membranas (1) es continuo al REG. La membrana interna esta cubierta por una red de proteínas (lamina o nucleoesqueleto) que participan en la organización de los movimientos de la cromatina durante las diferentes fases del ciclo celular. 4 Núcleo, sede de la replicación (duplicación del ADN) y la transcripción (síntesis de ARN), mientras que la traducción ocurre en el citoplasma. En las células procariotas todos esos procesos coinciden en el mismo compartimento celular. NUCLEO… En su interior se encuentran uno o varios nucleolos (4) cubiertos por una matriz fibrosa llamada nuceloplasma (liquido de consistencia gelatinosa donde se disuelven sustancias como nucleótidos trifosfatos, enzimas, proteínas y factores de trascripción) y sitentiza el rRNA. El ADN (5) se encuentra en forma de cromatina (complejo ADN-proteínas o cromosomas). Eucromatina es la forma menos compacta, genes que son frecuentemente exprimidos. Plásmidos: DNA extracomosomal de pequeño tamaño en forma circular Heterocromatina facultativa es la forma compacta, genes que no son exprimidos. Heterocromatina constitutiva es la forma que fabrica los telomeros, centromeros de los cromosomas. Plásmido o DNA extracromosomal Núcleo Almacena de forma organizada los genes en los cromosomas. Trasporta factores de regulación y productos génicos a través de los poros nucleares. Produce los mensajes que se traducirán en proteínas. Produce los ribosomas en la región nucleolar o nucleolo. Organiza el desenrollamiento del DNA durante la replicación y la transcripción. Lamina nuclear Provee soporte interno espacio o cisterna perinuclear (REG) (MET Hepatocito. A, Eucromatina; b, RER; c, Mitoncndria). Heterocromatina: Condensada y transcripcionalmente inactiva (constitutiva y facultativa). Localización nuclear periférica. Representa ~10%. Membrana nuclear Eucromatina: No condensada y trascripcionalmente activa o en replicación. Localización nuclear central. 5 Se importan dentro del núcleo: Complejos de poro nuclear (CPN) • Las proteínas sintetizadas en el citoplasma necesarias para ensamblar los ribosomas. El número de CPN es variable, incrementándose a medida que aumenta la actividad celular. En una célula de mamífero hay entre 3000 a 4000 complejos de poro. CPN constituido por un gran número de proteínas de disposición octamérica. • Los factores de transcripción requeridos en la activación o inactivación de los genes. Está formado por: • Los factores de empalme necesarios en el proceso de maduración de los ribosomas. L · Ocho columnas proteicas, que forman las paredes laterales del poro. Para poder ingresar en el núcleo, las proteínas sintetizadas en el citoplasma deben contener la señal de localización nuclear (nuclear signal localization, NSL). Las moléculas y macromoléculas ensambladas y exportadas desde el núcleo al citoplasma incluyen: · Un anillo externo, formado por ocho unidades proteicas. · Un anillo interno, también con estructura octamérica. · Proteínas de anclaje que fijan cada columna al espacio perinuclear. · Proteínas radiales que se proyectan desde las columnas hacia la luz del poro, a manera de diafragma • Las subunidades ribosomales · Proteínas fibrilares fijas al anillo interno y externo. En la cara nuclear convergen para formar una canastilla o cesta. A lo largo de estas fibrillas se ubican nucleoporinas que intervienen en el transporte de sustancias a través del poro. • ARNm. Ellos salen a través del complejo de poro con una proteína especial que posee una señal nuclear de exportación (nuclear export signal, NES). 15 – 20 nm • ARN de transferencia • Factores de transcripción que son devueltos al citoplasma para ser reutilizados. · Un poro central o abertura. Exportación de ARN Las moléculas de mayor tamaño requieren de una proteína “transbordadora” o carioportina. Los ARN maduros se asocian a proteínas llamadas transportinas, las cuales actúan como transbordadores permitiendo el pasaje de ARN al citoplasma. La superfamilia de carioportinas esta integrada por: -importinas (proteínas) -exportinas (proteínas) El ARNm es llevado fuera del núcleo. Los ARNm maduros que presentan la poli A se asocian con varias proteínas, formando una partícula de ribonucleoproteína (RNP). -transportinas (ARN maduros) (Nup) Las importinas son heterodímeros, formados por dos subunidades (α y ß) El poro se dilata y la proteína es traslocada activamente Actividad en el poro nuclear (CRBP) Estas partículas se mueven linealmente a través de la canasta nuclear. Al igual que las importinas, las RNP son recicladas hacia el núcleo. En el citoplasma, las CRBP (cytoplasmic RNA-binding proteins) reemplazan a las RNP para guiar a los ARNs a sus destinos citosólicos correctos. Estructura y función del nucleolo Sitio de síntesis de las subunidades ribosómicas NE : Envoltura nuclear C : Citoplasma o citosol NO : Organizador Nucleolar PG : Pars Granulosa PF : Pars Fibrosa PF- región donde se transcribe el RNA ribosomal con varios cromosomas encargados de la organización nucleolar. PG- es la región donde se acumulan las proteínas ribosómicas ó ribonucleoproteínas. 6 Organizador nucleolar NO: Cromosomas PF: rRNA transcrito PG: Acumulación de ribonucleoproteínas Ribonucleoproteínas: Forman las dos subunidades ribosomales (grande y pequeña). Son transportadas por separado fuera del núcleo y se ensamblan en el citoplasma, lo cual previene la síntesis de proteínas dentro de el. Se adhieren Endoplasmatico. al Retículo •Los cromosomas ocupan lugares específicos. •Los genes que codifican productos relacionados, aunque estén localizados en diferentes cromosomas, pueden estar ubicados próximos en el núcleo interfásico. Por ejemplo, los cromosomas humanos 13, 14, 15, 21 y 22 poseen un gran número de genes que codifican para ARNr. Dichos cromosomas están agrupados de tal forma que los genes de los ARNr están todos juntos y confinados en el nucléolo, el lugar donde se sintetizan, procesan y ensamblan los ARNr. Esta separación física asegura que los ARNr puedan ser eficientemente ensamblados dentro de las subunidades ribosomales. Durante la síntesis del rRNA, se observa la forma de un árbol navideño. La parte alta del árbol es el sitio de inicio. Hacia la parte baja, las “ramas” son mas largas. Cada una es una cadena creciente de rRNA. El codigo DNA esta siendo transcrito y los nucleotidos añadidos a la cadena de RNA. Lamina nuclear •Se localiza anexa a la membrana nuclear interna. •Consiste de una capa de filamentos finos que rodean al núcleo excepto en el poro nuclear. •Funciona como elemento estabilizador. Implicadas en la organización funcional del núcleo. Características: Consiste de filamentos de 30-100 nm de espesor. Estos filamentos son polímeros de lamina de entre 60-75 kDa. 2 La naturaleza del material genético y el código genético. Objetivo: Describir la estructura molecular de ADN y del ARN y la función de los ácidos nucleicos. Laminas tipo A se localizan internamente, junto al nucleoplasma. Laminas tipo B se localizan junto a la membrana nuclear enlazadas a proteínas membranles. 2.1 División celular. Juegan un rol en el ensamblaje y desensamblaje antes y después de la mitosis. Una vez fosforiladas, se desencadena el desamblaje de la lamina y la envoltura nuclear se rompe en vesículas. La de fosforilación revierte este efecto y permite que el núcleo se reconstituya. La célula eucariota separa los cromosomas nucleares en dos juegos idénticos. Mitosis G1 : S: G2 : M: Ciclo celular: Crecimiento y división en dos células hijas Adquisición de ATP e incremento de tamaño. Replicación del DNA. Adquisición de ATP e incremento de tamaño pre-división. Mitosis, separación nuclear y citoplásmica (Citocinesis). * Las fases G1, S y G2 son etapas englobadas en la interfase (90% del ciclo). Para ello deben ser alineados y replicados de manera ordenada. + Seguida por la citocinesis, que divide los núcleos, el citoplasma, los organelos y la célula membranal en dos células hijas. = Mitosis 7 Interfase La célula se dedica a la actividad metabólica y a almacenar energía para la mitosis (las próximas cuatro fases que incluyen la división nuclear). Cromosomas no son claramente visibles en el núcleo, aunque una mancha oscura llamada nucleolo puede ser visible. La célula puede contener un par centriolos (o la organización centros de microtúbulos en plantas), ambos son sitios organización de microtúbulos. de de las de El DNA celular esta marcado con DAPI (blue), las mitocondrias están marcadas con MitoTracker Red CMXRos (rojo) y los microtubulos marcados con verde con Alexa Fluor 488 adjuntos a anticuerpos secundarios. “Señales" moleculares en microtubulos dirigen el trafico al interior de la célula guiando proteinas de carga que viajan a lo largo de la célula a través de proteinas fibrosas llamadas microtubulos. Algunas enzimas modifican los microtubulos añadiendo o removiendo moleculas de sus superficies. Dineina Credito: Nicolle Rager Fuller, National Science Foundation Tubulina GTP Profase Cromatina en el núcleo comienza a condensarse y se hace visible en el microscopio óptico como cromosomas. El nucleolo desaparece. GTP Centríolos empiezan a moverse a los extremos opuestos de la célula y las fibras se extienden desde el centrómero. Algunas fibras cruzan la célula para formar el huso mitótico. 8 Prometafase La membrana nuclear se disuelve, marcando el comienzo de prometafase. Proteínas se enlazan a los centrómeros creando el cinetocoro. Microtubulos se unen a cinetocoros y los y los cromosomas comienzan el movimiento. (Lodish, et al., 1995. Molecular Cell Biology, 3rd ed.) Metafase Spindle fibres (Uso mitotico) alinea los cromosomas a lo largo de la mitad del núcleo celular. Esta línea se denomina la placa de metafase. Esta organización ayuda a asegurar que en la próxima fase, cuando los cromosomas se separan, cada nuevo núcleo recibirá una copia de cada cromosoma. Los cromosomas son altamente condensados y el huso mitótico parece (visible como una serie de túbulos verde en las imágenes digitales de células en metafase PtK2 de hígado de ratón). Los dos polos del huso y las fibras del huso son claramente visibles, como son los asters, que migran radialmente fuera de la placa de metafase. El huso mitótico contiene las fibras (microtúbulos) responsables de la translocación y la separación de los cromosomas. 9 •Cada huso está compuesto por un conjunto de microtúbulos ensambladas a partir de moléculas de tubulina disponibles de la red microtubular citoplásmica que son desmontadas para la división celular. •Los microtúbulos convergen en cada extremo del eje hasta el punto denominado el polo del huso o centro mitótico. •En células animales cada unidad del huso contiene un par de centriolos, los cuales se auto-replican. Están rodeados por un arreglo radial de microtúbulos Aster (estrella). •Las células vegetales, en cambio, no contienen centriolos, y su eje mitótico no esta claramente definidos en los polos. Las plantas carecen de la serie radial astral característica de las células animales. Anafase Los pares de cromosomas separados en el cinetocoro se mueven a los lados opuestos de la célula. El movimiento resulta de una combinación de movimiento del cinetocoro a lo largo de los microtúbulos del huso que se acortan por la perdida de monómeros de tubulina y de la interacción física de los microtúbulos polares que se alargan. Es un proceso muy rápido que dura solo unos minutos, es la etapa mas corta de la mitosis. La migración de las cromátidas a los polos opuestos del huso miótico ocurre a una tasa de aproximadamente un micrómetro por segundo en la mayoría de las células animales. Las fibras que comprenden el huso mitóico son de dos tipos: Fibras polares se extienden del centro del polo del huso hacia la placa metafasica Fibras del cinetocoro (cromosomales) van del cromosoma individual condensado hacia los polos. La posición espacial del centrómero con respecto a los brazos del cromosoma es muy clara por microscopia ya que son arrastrados hacia el polo del huso. Morfológicamente los cromosomas pueden ser divididos en tres clases: Cromosomas metacéntricos: tienen el centrómero cerca del centro resultando en dos brazos de longitud equivalente. Presentan forma de V cuando se observan durante la anafase. Cromosoma telocéntrico: tiene el centrómero cerca de un extremo y parece migrar como un solo brazo. Cromosoma acrocéntrico: La mayoría de los cromosomas, con el centrómero localizado a ¾ partes (entre el centro y el extremo de la cromátida) proporciona una estructura en forma de L. Cuando los cromosomas han migrado completamente al polo, los microtubulos del cinetocoro comienzan a desaparecer y los microtubulos polares continúan elongandose. Anafase esta caracterizada por dos procesos distintos: Anafase A (o anafase temprana), los microtubulos del cinetocoro se acortan para translocar las cromátidas lejos de la placa metafasica hacia los polos (a) y (b). Anafase B (anafase tardía), separación de los polos del huso provocada por el alargamiento de la red de microtúbulos polares (c a f). Este es el paso entre anafase y telofase temprana Al final de la anafase, cada huso polar (spindle pole) ha adquirido un set equivalente de cromosomas hermanos. Comienza el proceso de citocinesis con la formación de un anillo contráctil de filamentos de actina y myosina-II posicionado entre la membrana plasmática paralela a la placa metafasica. Posteriormente este anillo convergirá empujando la membrana hacia el centro para dividir la célula. La duración de cada uno varia entre organismos. 10 Telofase Actina •Los cromosomas hijos llegan al polo del huso y son eventualmente redistribuidos en la cromatina. •Cromosomas se descondensan en cromatina y son menos definidos. •Las redes de microtubulos polimerizados del huso miótico son redistribuidos en los componentes del citoesqueleto. •Comienza la síntesis del RNA en el núcleo. •Citocinesis que comenzó en anafase tardía continúa en telofase. •Las mitocondrias citoplasma. se distribuyen en el •Midbody o cuerpo medio contiene remanentes de los micro túbulos polares del huso miótico, mantiene un puente entre ambas células varias horas después de que finaliza esta fase. •La membrana nuclear comienza a reformarse alrededor de cada grupo de cromosomas. •El núcleo reaparece. Su membrana proviene de remanentes de la envoltura nuclear parental y porciones del sistema de endomembrana contenido en cada una de las células hijas. •Una vez completa la telofase y que la membrana celular (o pared celular) esta formada, el núcleo ha madurado a un estado pre-miótico. •El paso final es el inicio de la ruptura de la membrana de ambas células. Citocinesis •Etapa final de la división celular. En procariotas la división se da por Fisión binaria •Comienza durante la anafase tardía o telofase temprana con la formación de membrana nuclear, de los nucleolos y decondensación de cromosomas. •El citoplasma se divide por anillo de actina y miosina que se contracta, dividiendo el citoplasma y su contenido en dos células hijas. Formando un surco perpendicular al huso miótico (cuerpo medio o midbody). •Las mitocondrias se reparten en el citoplasma •Uso miótico se distribuye en citoesqueleto. Las plantas, debido a la rigidez de la pared, requieren que se sintetice una placa nueva entre las dos células hijas. E. coli dividiéndose por fisión binaria (bacilo gram -) 11 Meiosis en eucariotas (animales y esporas de plantas). Mitosis Meiosis reduce el número de cromosomas a la mitad durante la formación de gametos. Ocurre únicamente durante la gametogénesis, cuando los gametos o células sexuales (ej. óvulo o esperma) se forman. Meiosis comprende dos divisiones nucleares sucesivas que producen cuatro células haploides. primera división (meiosis I) es la división de reducción. segunda división (meiosis II) separa las cromátidas. Meiosis Durante la primera "reducción meiótica" los pares de cromosomas son repartidos de modo que cada gameto (o espora) contiene uno de cada par cromosómico, es decir, es haploide. Cuando se unen los gametos durante la fecundación, el cigoto resultante recibe una cromátida de cada uno de los padres. Los procesos esenciales de la meiosis consisten en: - Reducción del número de cromosomas. - Segregación al azar de los cromosomas. - Recombinación genética por intercambio de segmentos cromosómicos. Interfase I-cada cromátida individual se duplica = mitosis La división celular Mitótica produce nuevas células genéticamente idénticas a las células madre. Meiosis aumenta la variación genética en la población debido a que durante la meiosis I, cada par de cromosomas (cromosomas homólogos) se reúne en un proceso conocido como Synapsis. Cromátidas de cromosomas homólogos pueden intercambiar partes en un proceso llamado cruce o recombinación homologa. Cada célula diploide en meiosis puede producir 2n combinaciones cromosómicas diferentes. donde n es el número de haploides. En los seres humanos es el número es 223 que es más de ocho millones de combinaciones diferentes. Metafase I-cromosomas alineados en placa Metacéntrica = mitosis Meiosis I Anafase I- separa los cromosomas homólogos = mitosis Telofase I- envoltura nuclear y citocinesis = mitosis Intercinecis Profase II – cada dyad (1/2 a tetrad) esta compuesta de un par de cromatides hermanas conectadas por un centromero. Metafase II - similar a Metafase I. Dyads alineados en placa metacentrica. Anafase II – separa dyads en cromatides individuales. Cada cromatide hermana termina en lados opuestos de la celula. Meiosis II Telofase II – envoltura nuclear alrrededor de cada set de DNA y el citoplasma se divide nuevamente. Como resultado se forman 4 celulas haploides a partir de una diploide. Meiosis I Apareamiento y entrecruzamiento de los cromosomas homólogos Complejo Sinaptonémico 12 Quiasmas Nódulo de recombinación P3 P1 P2 P4 P5 M1 A1 T1 2P1 2M 2A 2T Meiosis I M I T O S I S M E I O S I S Meiosis II VIDEOS 13 2 La naturaleza del material genético y el código genético. Objetivo: Describir la estructura molecular de ADN y del ARN y la función de los ácidos nucleicos. de Espermatogénesis-proceso formación de espermatozoides por meiosis (en animales, por mitosis en plantas) en órganos especializados conocidos como gónadas (que en los machos se denominan testículos). Formación de gametos (haploides, n) a partir de la células diploides de la línea germinal. Gametogénesis Luego de la división, las células se diferencian transformándose en espermatozoides Las cuatro células derivadas de la meiosis se diferencian en espermatozoides. Primer cuerpo polar Segundo cuerpo polar Espermátidas u ovótida 2.3 Diferenciación celular. Primer cuerpo polar dividido Ovogénesis-proceso de formación de un óvulo por meiosis (en animales, por mitosis en el gametofito de las plantas) en órganos especializados conocidos como ovarios. El citoplasma y organelas forman una célula más grande: el óvulo y las otras tres (llamadas cuerpos polares) no desarrollan. • Las células de casi todos los organismos pluricelulares proceden de la división repetida de una única célula precursora: el óvulo fecundado. • La inmensa mayoría de células animales y vegetales conservan toda la información genética. • Además de las diferencias morfológicas, las células difieren en su habilidad de moverse, en su organización interna (procariotas vs eucariotas), metabólismo. El huevo humano es una sola célula de ~200 µm, y el esperma también es una sola célula. De la unión de ellos se originan los 10 trillones de células que forman el cuerpo humano. Lactobacteria (Lactococcus lactis). Archaebacteria (Methanosarcina). Células sanguíneas (eritrocitos, leucocitos y plaquetas). Huevos de dinosaurio fósil. Alga verde (Volvox aureus). Neurona del cerebelo. Epitelio intestinal. Células vegetales (esqueleto rígido de celulosa). (Lodish, 2004) (Lodish, 2004) • El carácter final de una célula esta determinado por toda la secuencia de influencias a las que ha estado sometida durante el transcurso del desarrollo. •Es reversible para eritrocitos y otro tipo de células (gen pax5) “reprogramación celular”. • Vertebrado > 200 tipos celulares, difieren en estructura y función. Desarrollo embrionario • La especialización depende de alteraciones de la expresión génica y no de la perdida o adquisición de genes. Estado de dos células • Los grupos de genes son activados o inhibidos en respuesta a señales tanto externas como internas. Estado de cuatro células Estado de ocho células (Lodish, 2004) Cada célula en el estado ocho células del embrión del ratón tiene el potencial de originar cualquier parte del animal completo. Las células con esta capacidad se conocen como células embriónicas (ES, esteam cells). ES pueden ser cultivadas en el laboratorio y bajo condiciones controladas desarrollarse en varios tipos de células diferenciadas. Tiene cerca de 6,000 células cubriendo su superficie, la mayoría de ellas son indistinguibles por microscopia de luz simple. Vemos anticuerpos marcados con un marcador fluorescente enlazado a proteínas que están en el núcleo; cada pequeña esfera corresponde a un núcleo. La expresión diferencial en genes aparece rápidamente en embriones tempranos, en el cual todas las células son similares hasta que son marcadas para detectar proteínas codificadas por genes particulares. 1 Tipos celulares • Epitelio • Tejido conjuntivo • Músculo • Tejido nervioso (Lodish, 2004) Genes similares han sido conservados durante la evolución, regulan muchos procesos del desarrollo en diversos animales. Ambos comparten genes que controlan procesos similares, tales como el crecimiento del corazón y los ojos y la organización del plano corporal, indicando una función conservada desde tiempos antiguos. 2 Distintos tipos celulares presentan patrones similares de bandas. Los tipos celulares de un organismo pluricelular se diferencian unos de otros porque sintetizan y acumulan distintos juegos de moléculas de RNA y de proteínas. Los genes y las zonas de genes se duplican y las nuevas copias divergen de la original mediante mutación y recombinación, para realizar nuevas funciones adicionales. Por ello es que un mismo organismo a menudo se encuentran parecidos familiares entre genes que codifican proteínas que realizan funciones relacionadas. El conjunto de cromosomas de todas las células del cuerpo son idénticos. La comparación de genomas de diferentes células muestran que los cambios de expresión génica no van acompañados de cambios en las secuencias de DNA. Estos genes están relacionados evolutivamente y su existencia revela una estrategia básica a través de la cual se ha originado la complejidad creciente de los organismos. Hay 6 Etapas en las que se puede controlar la expresión génica en eucariotas Distintos tipos celulares sintetizan distintos conjuntos de proteínas Cuantas diferencias existen entre dos tipos celulares dados??? Se sabe que : 1. Procesos comunes en las células, por lo tanto muchas proteínas en común Ej. Actina, ribosomas, enzimas del metabolismo 2. Hay células especializas , con proteínas especializadas. Ejs. Hemoglobina solo en eritrocitos Células ciliadas del oído o del ojo. 3. Al comparar 2,000 proteínas de diferentes tipos celulares de un organismo, habrá muy pocas diferencias. Lo que varia es la proporción de éstas (menos de 5 veces). Las células pueden controlar la síntesis de proteínas: 1- Control transcripcional- Regulando el momento y la frecuencia de la transcripción de genes concretos. 2- Control o procesamiento del RNA- Controlando el modo de maduración o procesamiento de los transcritos primarios de RNA. 3- Control del transporte de RNA- Seleccionando los mRNA maduros que van a ser exportados al citoplasma. 4- Control traduccional- Seleccionando los mRNA citoplasmáticos que van a ser traducidos por ribosomas. 5- Control de la degradación de los mRNA- Desestabilizando selectivamente algunas moléculas de mRNA citoplasmático. 6- Control de la actividad proteica- Activando, inactivando o ubicando de modo selectivo las proteinas ya sintetizadas. Control combinatorio Unas cuantas proteínas reguladoras pueden generar muchos tipos celulares diferentes durante el desarrollo. Ejemplo hipotético: Embrión izquierda sintetiza proteína par, embrión derecha sintetiza proteína impar. Se han generado 8 tipos celulares (G a N) con 5 prot. reguladoras. Si se continua se podrían generar 10,000 tipos celulares con 25 proteínas reguladoras distintas. 3 Una proteína reguladora dada no tiene necesariamente una función única y claramente definida, como por ejemplo la de activación de un grupo determinado de genes o la de determinación de un cierto tipo celular. Principales líneas de investigación en Diferenciación Celular Puede tener diferentes funciones que se solapan con las de las otras proteínas reguladoras. •Totipotencialidad y células troncales- Morfogénesis y polaridad. • Orígenes de replicación de DNA- Organización y estructura. La producción de diferentes tipos de células no es suficiente para hacer un organismo, los diversos tipos de células deben organizarse y ensamblarse en todos los tejidos y órganos. En el organismo en desarrollo, las células crecen y se dividen en algunos momentos y en otros no, se ensamblan y comunican, previenen o reparan errores en el proceso de desarrollo, coordinan cada tejido con el resto. En el organismo adulto, la división celular normalmente se detiene en la mayoría de los órganos. Si una parte de un órgano tal que el hígado es dañado o removido, la división celular se activa hasta que el órgano es regenerado. • Receptores celulares - Análisis de los mecanismos moleculares que regulan su expresión y funcionamiento en procesos de diferenciación neuronal y en oncogénesis. • Factores de crecimiento transmembranales- Estructura y biología. • Complejo principal de histocompatibilidad- Regulación de la expresión. (embrionales flexibles). (sin capacidad para formar gametos). (varios tipos pero limitada). ¿Qué clases de células troncales existen? 4 Rutas de reprogramación celular: a, Es posible que una célula madura de un linaje X se desdiferencie hacia un progenitor no comprometido a partir del cual puede formar una ruta de diferente para eventualmente ser una célula de un linaje diferente (Y). Cobaleda et al. encontraron que células maduras de Pax5 B deficientes pueden formar células T maduras. b, Alternativamente células X maduras, pueden transdiferenciarse directamente en precursores de células Y maduras. Una tercer ruta puede implicar desdiferenciacion y transdiferenciacion simultaneas. Nature. Volume 449(7161), 27 September 2007, pp 410-411 5 Vías de la muerte celular Célula normal La célula cambia su patrón de expresión de genes dependiendo de las señales que reciba Condensación de cromatina. Ruptura del DNA en 200 pb. Formación de protuberancias (cuerpos apoptoticos). Ruptura del DNA en tamaños aleatorios Destrucción de la membrana. Desintegración de la célula en el medio. Fagocitosis. Necrosis Muerte violenta, implica varias células. No respeta la membrana celular. Apoptosis Muerte programada de una célula ó suicidio celular. Respeta la membrana celular en sus primeras etapas. 6 2 La naturaleza del material genético y el código genético. Objetivo: Describir la estructura molecular del ADN, ARN y la función de los ácidos nucleicos. El ácido desoxiribonucléico y el ácido ribonucléico son macromoléculas catenarias que actúan en el almacenamiento y transferencia de la información genética. ADN fue aislado por primera vez del núcleo, pero también existen ácidos nucleicos en otras partes de la célula como formas libres en procariotas. Miescher 1869 llamó nucleína al ADN por su participación en el ciclo celular. Descubrió esta sustancia con alto contenido de fósforo y nitrógeno en el núcleo de células sanguíneas en acumulaciones de pus. Flemming en 1882 descubrió que esta sustancia a la que llamó cromatina, se separaba en tiras a manera de estambre durante diferentes fases del ciclo celular. 2.4 Acido deoxiribonucleico: ADN. Los nucleótidos son las unidades monómeras de los ácidos nucleicos. Cada tipo de ácido nucleico se distingue por la secuencia de bases heterocíclicas característica de sus nucleótidos estructurales. Las unidades monómeras del ADN se llaman desoxirribonucléotidos. Cada nucleótido contiene tres componentes característicos: - Una base nitrogenada heterocíclica derivada de la purina o pirimidina - Una pentosa - Una molécula de ácido fosfórico RNA U = Uracilo (2,4-dioxopirimidina) C = Citosina (4-amino-2-oxopirimidina) T = Timina (5-metil-2,4-dioxipirimidina) A = Adenina ( 6-aminopurina) Existen otras bases nitrogenadas no tan comunes presentes en los ácidos nucleicos: 5,6-dihidrouracilo 1-metiluracilo 3-metiluracilo 2-metiladenina 7-metilguanina G = Guanina (2-amino-6-oxopurina) ARN 9 ADN Los azucares de los ácidos nucléicos se encuentran en forma de furanosas (anillos de 5 carbonos) y se unen a las bases nitrogenadas por medio de un enlace ß-N-glucosilo establecido entre el carbono 1 de la pentosa y el nitrógeno 9 de las bases púricas o el nitrógeno 1 de las bases pirimidínicas. 1 Debido a la presencia de la base nitrogenada los nucleótidos muestran una fuerte absorción en la zona de los 250-280 nm de la luz ultravioleta, lo que resulta muy útil para su análisis cuantitativo. Cuando el grupo fostato de los nucleótidos se separa por hidrólisis, la estructura residual recibe el nombre de nucleósido (desoxirribonucleósidos y ribonucleósidos). Enlace fosfodiester Todos los ribo y desoxirribonucleótidos aparecen en forma de 5’ monofosfatos, 5’ difosfatos y 5’ trifosatos; DNA Los ácidos monofosfórico, disfosfóricos y trifosfóricos de los nucleósidos se denominan genéricamente NMP, NDPs ó NTPs. N= A, T, G ó C. Los nucleótidos se unen mediante un puente o enlace fosfodiester entre el carbono 5’ y el carbono 3 ‘ para formar los ácidos nucleicos. ATP y ADP, cAMP son transportadores de fosfatos en las rutas de energía química y funcionan también como coenzimas trasportando moléculas sillares (UDP-Glucosa). Por convención internacional la secuencia de nucleótidos en la cadena de ácidos nucleicos se abrevia con el código de una sola letra para cada nucleótido y en dirección 5’- 3’ de izquierda a derecha. 5´- GATC - 3´ Tanto el DNA como el RNA comparten ciertas propiedades químicas y físicas debido a que en ambos las sucesivas unidades nucleotídicas se hallan unidas covalentemente de idéntica manera por medio de los enlaces fosfodiester. Las bases nitrogenadas constituyen cadenas laterales diferenciadas. Erwin Chargaff descubre entre 1949 y 1951, la regularidad de las proporciones de las bases nitrogenadas en diferentes especies. Los resultados obtenidos se resumen en las "leyes de equivalencia de las bases nitrogedas": 1. En todo tipo de dsDNA, la cantidad de Adenina es la misma que la de Timina, mientras que la cantidad de Guanina es igual a la de Citosina: A = T , G = C , (A + G) /( C + T) ≅ 1 2. El DNA de diferentes tejidos de la misma especie, tiene la misma composición de bases nitrogenadas. La cantidad de DNA en células de diferentes especies es proporcional a la complejidad de esta y refleja la cantidad de información genética que lleva (mayor contenido, mayor complejidad). 3. La composición de bases nitrogenadas en el DNA varía de una especie a otra. 4. La composición del DNA de una especie, no cambia con la edad o con la nutrición. Deducciones sobre la estructura de doble hélice 5. La composición de cualquier DNA puede ser descrita mediante la proporción de sus bases G + C. El intervalo va de 20 al 80 % en diferentes especies. Especies mas relacionadas tienen composición similar. 1) La molécula de DNA está formada por dos cadenas de polinucleótidos enrolladas alrededor de un mismo eje, formando una doble hélice. Esta estructura tiene semejanza a una escalera de "caracol". Journal of Biological Systems (2004) 12, no. 3, 261-271 2 2) Las dos cadenas son antiparalelas ya que una de las cadenas empieza por el extremo donde se encuentra el residuo 5', mientras que la otra lo hace por el extremo donde se encuentra el residuo 3'. Puentes de hidrogeno 3) Las bases nitrogenadas de los nucleótidos se orientan hacia el interior de la doble hélice, mientras que los grupos fosfato y las moléculas de azúcar se orientan hacia el exterior. Fosfatos tienen carga negativa Por tanto, DNA tiene una carga neta negativa ! 4) Las bases de ambas cadenas están unas frente a otras y se unen a través de puentes de hidrógeno: dos entre la adenina y la timina (A=T) y tres entre la guanina y la citosina (G ≡ C). 5) La longitud de cada vuelta en la hélice es de 3.4 ηm. Un nanómetro es igual a 10-9 m. 6) La distancia entre un par de nucleótidos y otro es de 0.34 ηm, por lo tanto, en cada vuelta debe haber 10 pares de nucleótidos. Interior es hidrofobico La doble hélice mantiene un ancho constante debido a que purinas (A, G) siempre aparean con pirimidinas (C, T) mediante las bases complementarias A=T y G≡C. Cadena polinucleotida es responsable de la transferencia de la información genética y por lo tanto de la evolución. Nucleotidos pueden contener uno ó mas grupos fosfato. De acuerdo a esto pueden ser designados como nucleotido monofosfatos, nucleotido difosfatos y nucleotido trifosfatos. Enlace fosfodiester 5´- 3´ Formación del enlace fosfodiester 5´- 3´ 1.2 nm P2O7 4-+ H20 2.2 nm Para formar la cadena polinucleotida, los nucleotidos trifosfatos son enlazados covalentemente. En esta reacción, el oxígeno del grupo 3´hidroxilo en la cadena “ataca” el fosfato del nucleotido trifosfato eliminando H2O y liberando dos residuos fosfatos exteriores (pirofosfatos=PPi= P2O7 4-). 3 Pares de bases (pb) por giro Rotación por par de bases Altura del giro Diámetro de la hélice Humedad relativa Bases 11.0 +34.7° 2.56 nm 2.3 nm - 10.4 +34.6° 3.38 nm 1.9 nm + 12.0 -30.0° 5.71 nm 1.8 nm + A Fosfatos Desoxirribosas B Z DNA circular: bacterias y organelos eucariotas DNA monohebra: conformación espacial +∆G, then energy--in the form of work--would have to be added to the reacting system to make the reaction go. -∆G, means that such a reaction will be favored and will release energy. ∆G =0, neither the forward nor the reverse reaction prevails (equilibrium). (Lewin, 2010) 4 Algunas propiedades químicas útiles del DNA 1. Secuencias palindrómicas en DNA dúplex DNA esta cargado negativamente. Los enlaces de fosfato confieren carga neta negativa, propiedad importante para el análisis electroforetico. 2. DNA puede desnaturalizarse y renaturalizarse. Elevando la temperatura o utilizando productos químicos, es posible romper los enlaces hidrogeno entre las bases de cada cadena y “derretir (melt)” la doble hélice en dos cadenas polinucleotidas. Este proceso se llama desnaturalización. Este proceso es reversible -si las condiciones son favorables, las dos cadenas de DNA se reanillan. La Renaturalización de las dos cadenas ocurre eficientemente solo cuando las secuencias de las bases son complementarias, ej. Cuando los pares A-T y G-C se pueden formar. La Reasociación de las cadenas sencillas complementarias también se llama hibridación de los ácidos nucleicos. 3. DNA es soluble en agua. DNA se disuelve fácilmente en agua. De forma rutinaria se utiliza un buffer Tris (para controlar el pH) y el agente quelante EDTA (para atrapar cationes que son cofactores para atrapar enzimas que pueden atacar el DNA) para solubilizar los ácidos nucleicos. DNA es insoluble en etanol. 4. DNA absorbe luz ultravioleta. Las purinas y pirimidinas absorben luz en el rango ultravioleta del espectro, con una absorbancia máxima de 260 nm. La cantidad de luz absorbida es proporcional a la cantidad de DNA en solución. Para DNA de doble cadena, una lectura de A260 de 1.0 corresponde a una concentración de 0.05 mg/ml. Entonces, midiendo la absorbancia del DNA en una solución de concentración desconocida, se puede calcular la concentración del DNA en solución. 5. DNA puede ser teñido y cuantificado con bromuro de etidio. EtBr es una molécula anillada que se inserta entre los pares de bases. Esta inserción es útil para visualizar el DNA debido a que el EtBr fluoresce cuando se expone a la luz ultra violeta, el DNA normalmente invisible se puede observar y fotografiar. Adicionalmente como la cantidad de fluorescencia es proporcional a la masa total del DNA en solución, la cantidad de DNA puede ser estimada comparando la fluorescencia de una muestra con una serie de estándares. Qué tan grandes son los genomas Procariotas? Propiedades del DNA en solución (1x106) Molécula extremadamente delgada y larga (fragmentos recuperables de 10 millones de pb). Los fosfatos orientados hacia la periferia se ionizan completamente a pH mayor a 4.0 A pHs fisiológicos (4 – 11) el DNA es estable, fuera de esos limites se desnaturaliza. Desnaturaliza con: (a) pH extremo (b) Altas temperaturas (en función de su contenido porcentual de bases) (c) Disminución de la constante dieléctrica del medio acuoso. (d) Exposición a urea, formamida o solventes similares. Absorbe a 260 nm (Efecto hipercrómico) Coeficiente de sedimentación (densidad de la molécula=masa y forma de la particula) Aplicaciones Hibridación (p.e. Southern blotting, dot blot) (Lewin B. Genes VII) Millones de pb Tamaño del genoma La proporción del genoma ocupada por DNA no repetitivo varia mucho. Procariotas, solo contiene DNA no repetitivo. Eucariotas inferiores, la mayoría del DNA es no repetitivo; <20% cae en moderado o altamente repetitivo. En células animales mas de la mitad del DNA usualmente es ocupado por secuencias moderada y altamente repetitivas. Rango de valores-C encontrados en diferentes taxones. Incrementa el tamaño mínimo del genoma en cada grupo conforme incrementa la complejidad. Conforme aumentan las cantidades absolutas de DNA en eucariotas superiores, observamos amplias variaciones en los tamaños del genoma en algunos taxa. (Lewin B. Genes VII) En plantas y anfibios, el DNA no repetitivo puede ser reducido a una minoría del genoma, con componentes moderada y altamente repetitivos abarcando mas del 80%. (Lewin B. Genes VII) 5 Biorad DNA size ruler = Marcador de peso molecular = escalera Electroforesis en gel de agarosa A. Marcador de peso molecular de 20 bp B. 100 bp C. 100 bp D. 500 bp E. 1 kb F. EZ Load precision molecular mass ruler RNA (ácido ribonucléico) 2 La naturaleza del material genético y el código genético. Objetivo: Describir la estructura molecular del ADN, ARN y la función de los ácidos nucleicos. Polímero de nucleótidos La pentosa de los nucleótidos es la Ribosa: en la posición 2' del anillo del azúcar hay un grupo hidroxilo (OH) libre Polímero monohebra de ribonucleótidos Químicamente más inestable que el DNA 2.4 Acido ribonucleico: ARN. Se hidroliza fácilmente en disolución acuosa Presenta Adenina, Guanina, Citosina y Uracilo Mucho más abundante que el DNA en la célula RNA El RNA participa como intermediario en la síntesis de proteínas al leer la secuencia de DNA, interpretarla, transportar la señal y traducirla en una secuencia de péptidos. Trascripción Replicación RNA polimerasa Traducción DNA ¿Qué es la transcripción? En este proceso, un complejo enzimático, sintetiza una hebra de RNA que tiene una secuencia de bases complementaria a una zona del DNA que se ha leído. En la transcripción no se copia todo el cromosoma como en la replicación, tan solo una sección de él. Los productos principales de la transcripción son tres tipos de RNA: RNAm, RNAt y RNAr. No obstante, el transcrito que contiene la información de secuencias de bases para la síntesis proteica es el RNAm. 6 Las RNA polimerasas Las RNA polimerasas son complejos grandes de proteínas funcionales (enzimas), formadas por 12 subunidades. Se conocen tres tipos de RNA polimerasas en eucariotas: • RNA polimerasa I (RNA Pol I): Transcribe los genes de las moléculas RNAr de tipo 28 S, 18 S, y 5.8 S (en nucleolo). Es la RNA polimerasa con mayor actividad en las células. U • RNA polimerasa II (RNA Pol II; RNAP II): Transcribe los genes que codifican para proteínas en forma de RNAm (en nucleoplasma). U • RNA polimerasa III (RNA Pol III): Transcribe todos los genes de las moléculas de RNAt, los genes de las moléculas de RNAr del tipo 5 S, así como los genes de las moléculas de RNA pequeño o RNAs (en nucleoplasma). En procariotas existe un solo tipo de RNA polimerasa. RNA: Es el ácido nucleico más abundante en la célula, y puede purificarse fácilmente. Una célula típica contiene 10 veces más RNA que DNA. RNAt: Las moléculas de RNA de transferencia tienen entre 75 y 90 nucleótidos, y su peso molecular es de unos 25,000 daltones. Se conocen unos 60 RNAt distintos, y se encuentran en todas las células. Intervienen en la síntesis de proteínas, ya que van unidos a un aminoácido. Pueden presentar nucleótidos poco usuales (ácido pseudouridílico, ácido inosílico) e incluso bases características del DNA como la timina. Constituyen aproximadamente el 15% del ARN celular total RNAr: El RNA ribosómico está presente en los ribosomas, orgánulos intracelulares implicados en la síntesis de proteínas. Este ácido ribonucleico se sintetiza en el nucleolo y constituyen el 80% del RNA celular total y participan en la estructura de los ribosomas Ribosoma Subunidad Pequeña Subunidad Grande Procariota (70 S) 30 S (RNAr 16 S, además 50 S (RNAr´s 23 S y 5 S, 21 proteínas) además 35 proteínas) Eucariota (80 S) 40 S (RNAr 18 S, además 60 S (RNAr´s 28 S, 5.8 S y 5 S, 30 proteínas) además 45 proteínas) Los aminoácidos son transportados hacia el sitio de síntesis en los ribosomas. La molécula responsable de este transporte es el RNA de transferencia (RNAt) En el caso de los ARNm eucarióticos, cada ARNm contiene información sólo para una cadena polipeptídica, de ahí que se hayan designado monocistrónicos, mientras que en los procariotas, el ARNm puede existir en forma de moléculas policistrónicas (cada RNAm codifica para mas de un gen). En el citoplasma, los ARNm van a tener una duración relativamente corta, determinada, en parte, por las necesidades concretas de la célula. Se ha observado que algunos ARNm son sintetizados y almacenados en un estado inactivo o latente en el citoplasma, preparados para dar una respuesta rápida en la síntesis proteica. Ver tabla de tipos y características de los ARN celulares ¿Qué es la traducción? (Brazo aceptor) (Brazo TΨC) Ψ, pseudouridina Cuando el RNAt esta cargado con su correspondiente aa, se llama aminoacil RNAt. La aminoacilARNt sintetasa (una enzima) cataliza el enlace entre los ARNt específicos y los aminoácidos que concuerdan con sus anticodones. RNAm: El RNA mensajero es de tamaño variable, dependiendo del tamaño del péptido que codifica. Cada célula produce pequeñas cantidades de miles de mensajeros diferentes, que serán traducidos a proteínas que la célula requiere. Muchos RNAm son comunes en todas las células, codificando para los llamados genes o proteínas de mantenimiento ("housekeeping" proteins). Por otro lado, y en respuesta a los procesos de diferenciación celular, hay RNAm´s que son específicos para cierto tipo de células. (Brazo extra) Proceso en el que la información transcrita del DNA a RNA (RNAm) es interpretada por el complejo de síntesis proteica (ribosomas o poliribosomas formados de RNAr). Esta interpretación consiste en incorporar, uno a uno, los aminoácidos específicos (transportado por un RNAt) de acuerdo a la secuencia codificada durante la transcripción. De esta manera, una secuencia específica de ribonucleótidos se “traduce” en una secuencia específica de aminoácidos que conforman una proteína en particular. 7 •Cada tipo de RNAt es producto de un gen distinto y acarrea (en su extremo 3′ ) uno de los 20 aminoácidos (así que la, mayoría de los aminoácidos tienen mas de un RNAt responsable de su transferencia). •Tienen entre 74 - 95 nucleótidos, diferencias en brazo D y brazo extra. •Regiones complementarias hacen que tengan estructura terciaria de doble hélice. •Las regiones no complementarias forman 3 loops o burbujas. •En la burbuja o loop central, 3 bases no apareadas forman el anticodon. zoom •El apareamiento correcto de bases entre el anticodon y el codon (en la cadena de RNAm), permite la transferencia del aminoácido adecuado a la cadena peptidica en crecimiento. El RNA ribosomal (RNAr), sintetizado en el organizador nucleolar, junto con cierto tipo de proteínas estructurales constituyen a los ribosomas. Las subunidades ribosomales se distinguen por el tipo de RNAr que los compone. Las diferencias principales están asociadas a su tamaño, por lo que pueden ser caracterizados por su coeficiente de sedimentación (S = Coeficiente de sedimentación de Svedberg). 8 El RNA heterogéneo nuclear (hnRNA) se conoce también como transcrito primario y es la cadena “cruda” de RNA que posteriormente será procesada en otros tipos de RNA. El transcrito primario (hnRNA) es editado o modificado con la participación de una serie de ribonucleoproteínas nucleares, “cortando” partes de la señal y haciendo por tanto que la traducción quede limitada a solo parte de la cadena transcrita originalmente. Estas modificaciones tienen por objeto aumentar la vida media de estas moléculas en el citoplasma. Dogma Central de la Biología Molecular Principales Diferencias en la Transcripción y Traducción entre Procariotas y Eucariotas 9 EL CÓDIGO GENÉTICO 2 La naturaleza del material genético y el código genético. Objetivo: Describir la estructura molecular del ADN, ARN y la función de los ácidos nucleicos. •Para 1960 se conocían ya las estructuras del ADN y ARN. •Se sabía que el ADN dirigía la síntesis del ARN (Trascripción). •y que el ARN dirigía la síntesis de proteínas a través del RNAt (Traducción). •Se sabía que el DNA se encontraba en el núcleo, mientras que el RNA era particularmente abundante en el citoplasma, donde se sintetizan las proteínas. 2.6 El código genético. Proteínas hechas de una combinación de 20 aminoácidos: - Marshal Warren Nirenberg et al., 1961: RNAm sintético constituido solamente por nucleótidos de uracilo poli-U. eléctricamente neutro la primera palabra del código: UUU = fenilalanina 64 combinaciones posibles de tripletes -1 base/codón = 4 aa’s -2 bases/codón = 16 aa’s -3 bases/codón = 20 aa’s carga positiva de un lado y negativa del otro Descifran el código genético en junio de 1966 Formando codones o tripletes del RNAm para todos los aminoácidos Se disocia en agua produciendo cationes hidrógeno En disolución acuosa aporta iones OH- El ADN consta de 4 nucleótidos: GCAT EL RNA consta también de 4 nucleótidos: GCAU 1961-1966 Nirenberg, Khorana, Holley, Ochoa y Grunger-Manago RNA polimerasa. Como se sospechaba cada triplete (codon) codificaba para un aminoácido. Ya que hay 4 ribonucleótidos entonces hay 64 codones. UUU-UUU-UUU-UUU CCC-CCC-CCC-CCC AAA-AAA-AAA-AAA UAU-UAU-UAU-UAU 1 2 = = = = Hay: 4 nucleótidos en DNA. 64 posibles codones (43). 61 RNAt amino-acetilados o “cargados” que corresponden a 61 codones. 20 aminoácidos. Experimento desarrollado por Marshall Nirenberg y colaboradores para descifrar el código genético Phe-Phe-Phe-Phe Pro-Pro-Pro-Pro Lys-Lys-Lys-Lys Tyr-Tyr-Tyr-Tyr UUU-AAA-UAU = Phe-Lys-Tyr UUU-AAA-UAU-UUU-AAA-UAU = Phe-Lys-Tyr-Phe-Lys-Tyr UUU-AAA-UAA-UUU-AAA-UAU = Phe-Lys 3 UAA = Codon-STOP Ver experimento « Cracking the genetic code » 1 Código genético “universal” • Degenerado 43 = 64 codones: 61 para aa 3 nonsense STOP: UAA ("ocre") UAG ("ambar") UGA (“ópalo") START: Met ATG ó AUG ¾Hay excepciones al código restringidas a algunos procariotas y las mitocondrias Excepciones al codigo Las mitocondrias de animales y microorganismos (no plantas) utilizan UGA para codificar triptofano y no como un finalizador de síntesis. En procariotas el codón de inicio también puede ser: GUG y UUG. Por ejemplo, Escherichia coli, Enterobacteriaceae, emplea en un una bacteria de la familia La mitocondria animal utiliza AUA para metionina, no isoleucina. Las mitocondrias de vertebrados utilizan AGA y AGG para finalizar síntesis (STOP). En las levaduras, las mitocondrias, asignan todos los codones que comienzan con CU para sintetizar treonina en lugar de leucina. En cambio, las mitocondrias de las plantas usan el código universal, lo que ha permitido a las angiospermas transferir genes mitocondriales al núcleo de manera muy natural. EL CÓDIGO GENÉTICO ES NO SOLAPADO O SIN SUPERPOSICIONES Distribución de los codones de inicio: 83% de los casos ATG (AUG en el ARN) ATG, 3542 GTG, 612 14% GTG (GUG en el transcrito) TTG, 130 3% TTG (UUG en el ARN) ATT y CTG RNAt y su influencia en el marco de lectura El marco de lectura de un RNA mensajero es generalmente invariable, comenzando con un codón AUG y terminando en alguno de los codones de terminación. un nucleótido solamente pertenece a un único triplete OK! La lectura de este marco de lectura se realiza de corrido por lo que si hay una inserción o una deleción se produce un cambio en el marco de lectura del punto de la mutación hacia delante. Esto produce que se traduzca el mensajero a una proteína completamente distinta. Un supresor de marco de lectura restablece el marco de lectura original compensando las deleciones y las inserciones en un gen. Sin embargo, también algunas moléculas de RNAt con propiedades aberrantes funcionan como supresores extragénicos de errores en el marco de lectura. Algunos errores originados del copiado del DNA quedan como repeticiones de ciertas bases y esto puede ser solventado por la presencia de anticodones aberrantes que reconocen 4 bases en lugar de 3. 2 El numero de codones para cada aminoácido no se correlaciona con su frecuencia de uso en proteínas Aminoacil-RNAt-sintetasas (ARS): Activación del RNAt Existen dos tipos de ARS que tienen diferentes puntos de anclaje sobre la molécula de RNAt, dependiendo de la secuencia de esta última. Es así como la conformación espacial del RNAt habilita a alguna de las dos clases de ARS a unirse y pegar el aminoácido correspondiente. Clase I: Incorpora el aminoácido en el carbono 2’ de la ribosa en la adenina terminal 3’ Clase II: Incorpora el aminoácido en el carbono 3’ de la ribosa en la adenina terminal 3’ 3’ 5’ ARS Clase I Los aa mas sencillos son los mas representados → minimiza efecto por mutaciones E.g. CUC por CUG es Leucina o bien CUU por AUU es isoleucina, un aa relacionado. A C C ARS Clase II 9 Más de un tipo de RNAt puede acarrear el mismo aminoácido Sitios de mayor especificidad involucrados en el reconocimiento y acoplamiento de las ARS. Sitios conservados que permiten que diferentes tipos de RNAt puedan portar el mismo aminoácido. Secuencia consenso (común) a todos los RNAt 3 Lectura de Prueba: Precisión RNAt-aa´s RNA polimerasa III Membrana nuclear RNA polimerasa I El reconocimiento del RNAt correcto esta principalmente controlado por la afinidad de la aminoacil-RNAt-sintetasa por su RNAt específico y además la aminoacilación del residuo de aa cuando no es el correcto es muy lenta. En resumen, la selección del aminoácido correcto esta favorecido sobre alguno incorrecto en un orden promedio de 250:1 DNA RNAt RNAhn RNA Polimerasa II aminoácidos RNAr Aminoacil-RNAt anticodon Tasa de error Riboproteína Ribonucleótidos Activación de la ARS con un aa incorrecto: 1/225 Incorporación del aa incorrecto al RNAt: 1/270 Tasa global de error: 1/60,000 Cadena polipeptídica en síntesis Poro nuclear codon Lectura de Prueba (Corrección mediante Proofreading) Ribosoma RNAm Mediante un cambio de conformación espacial en la ARS se promueve la hidrólisis del aa incorrecto o bien, una vez formado el complejo ARS-aa-RNAt, el aa incorrecto es hidrolizado 5 6 4 1 3 9 2 19 7 8 10 14 11 12 18 17 16 13 4 Que son las proteínas ? 2 La naturaleza del material genético y el código genético. Son macromoléculas de masa molecular elevada, formadas por aminoácidos unidos mediante enlaces peptídicos. Pueden estar formados por una o varias cadenas. Objetivo: Describir la estructura molecular de ADN, ARN y la función de los ácidos nucleicos. 2.7 Estructura proteica. La secuencia de aa de una proteína siempre se presenta en dirección N a C, leyendo de izquierda a derecha. Formados por un carbono alfa unido a un grupo carboxilo, un grupo amino, un hidrógeno y una cadena R de composición variable, que determina las propiedades de los diferentes aminoácidos; existen cientos de cadenas R por lo que se conocen cientos de aminoácidos diferentes. -grupo amino (NH3+ también escrito como NH2) -grupo carboxilo (COO- también escrito como COOH) -Cadena R Aminoácidos esenciales Se los considera esenciales no porque sean los únicos necesarios para la síntesis proteica, sino porque deben estar incluidos en la dieta. Cada especie, tiene su grupo de aminoácidos escenciales propios. Nomenclatura de los péptidos La estructura primaria de una proteína es simplemente el orden de sus aminoácidos. Oligopeptidos- Si el # de aa es < 10 Dipeptidos- Si el # de aa es 2 Tripeptidos- Si el # de aa es 3 Tetrapeptidos- Si el # de aa es 4 Polipeptidos ó cadenas polipeptídicas- Si el # de aa es > 10 Aquaporin 2 Como consecuencia del establecimiento de enlaces peptídicos entre los distintos aa que forman la proteína se origina una cadena principal o "esqueleto" a partir del cual emergen las cadenas laterales de los aminoácidos - Proteína- Número de aa > 50. Estructura primaria de la Insulina: consta de dos cadenas de aa enlazadas por puentes disulfuro entre las cisteínas (dos genes diferentes) 1 La estructura secundaria de una proteína es la que adopta espacialmente. tipos: hélice alfa y lámina beta. Hélice alfa Las laminas beta… Paralelas o antiparalelas La cadena polipeptídica principal forma la estructura central, y las cadenas laterales se extienden por fuera de la hélice. El grupo carboxilo (CO) de un aminoácido n se une por puente hidrógeno al grupo amino (NH) de otro aminoácido que está tres residuos mas allá ( n + 4 ). De esta manera cada grupo CO y NH de la estructura central (columna vertebral o "backbone") se encuentra unido por puente hidrógeno. La estructura terciaria es la estructura plegada y completa en tres dimensiones de la cadena polipeptídica. Es específica de cada molécula, además, determina su función Estructura cuaternaria - Solo está presente si hay mas de una cadena polipeptídica. Con varias cadenas polipeptídicas, la estructura cuaternaria representa su interconexión y organización. El plegamiento terciario no es inmediato, primero se agrupan conjuntos de estructuras denominadas dominios que luego se articulan para formar la estructura terciaria definitiva. Este plegamiento está facilitado por uniones denominadas puentes disulfuro, -S-S- que se establecen entre los átomos de azufre del aminoácido cisteína. From outside the cell, the four units of the water-channel protein AQP1 are visible, each with a pore that penetrates the cell membrane. Modelo de reloj de arena Isoformas A side view of a pore (blue dots) in the waterchannel protein AQP1, which pierces the cell membrane. Cell exterior is at top, interior at bottom. The pore is about 2.8 angstroms across at its narrowest. Según su composición, las proteínas se pueden clasificar en dos tipos que son proteínas simples o proteínas conjugadas. Por un lado tenemos las proteínas que son proteínas simples y son aquellas que, por hidrolisis, producen solamente µ-aminoácidos. Por otro lado, están proteínas que son proteínas conjugadas. Estas proteínas contienen además de su cadena polipeptídica un componente que no es un aminoácido, denominado grupo prostético. Este componente puede ser un ácido nucleico, un lípido, un azúcar o simplemente un ión inorgánico. Ejemplos de proteínas que son proteínas conjugadas son la mioglobina, la hemoglobina y los citocromos. Según su forma se clasifican en: Proteínas fibrosas, insolubles en agua, como la alfa queratina o el colágeno Proteínas globulares, solubles en agua. 2 Los cuatro niveles estructurales de la hemoglobina Estructura y función Las proteínas pueden ser clasificadas en familias de acuerdo a su estructura y función. > del 25 % de similitud de identidad de aa sugiere estructura similar. > del 30 % de similitud de aa (estructura) sugiere función similar. 50-60% de homología La similitud en la conformación tridimensional de proteínas de una determinada familia es usualmente sorprendente. Aquí se ven dos proteasas para serina (pancreáticas). En verde aa similares. En rojo el sitio activo donde se enlaza y rompe el substrato (diferentes aa) mediante hidrólisis. = familia pero ≠ función (AQP1 vs AQP3). En la naturaleza ~2000 tipos de estructuras, se conocen con certeza 800 (50 de ellas representan ¾ de todas las estructuras estimadas). Nomenclatura de proteínas Todas las proteínas se enlazan a otras moléculas La interacción física de una proteína con otra molécula determina sus propiedades biológicas. Esto se hace mediante enlaces altamente específicos (fueres o débiles). AMP 3 Red de interacciones entre proteínas en una célula de levadura. Como trabaja una enzima ? Cada línea conectando dos puntos (proteínas) indica una interacción proteína-proteína. Rompe los enlaces mediante hidrólisis (añade agua a la molécula), distorsiona su geometría y distribución de electrones ∴ incrementa el numero de colisiones rompiendo el enlace. RESUMEN PROTEINAS 3 Replicación, trascripción traducción del material genético. y Objetivo: Describir como una célula duplica su material genético precisamente a partir del ADN y explicar como decodifica y utiliza la información contenida en su genoma. 3.1 Replicación y reparación del ADN. Tres etapas fundamentales de la replicación: Uno de los mecanismos mas importantes de toda célula es la Replicación del DNA → continuación de la vida. Fenómeno extremadamente complicado en el que participan gran cantidad de enzimas. Debe asegurarse que las células hijas sean idénticas a la célula madre y que estas no sean defectuosas. I Iniciación II Elongación III Terminación I Iniciación: Reconocimiento del sitio de origen. Separar y estabilizar cadenas paténtales (Helicasa). Iniciar síntesis del DNA en horquilla de replicación por complejo proteico llamado Primosoma. II Elongación: Formación del complejo de síntesis de cadenas hijas o Replisoma (complejo activo DNA polimerasa III) III Terminación: Unión o reacción de terminación de la síntesis al final del replicón. Bloqueo (tus-Prot) inhibiendo helicasa, Metilación (Hemimetilacion) Finalmente, los cromosomas deben ser separados uno de otro para que pasen independientemente a cada una de las células hijas. 4 Pero como ocurre si…. La replicación tiene las siguientes características: 1) Las DNA polimerasas no pueden desenrollar el dúplex de DNA. 1) Ocurre en el citoplasma en procariotas y en el núcleo en eucariotas. 2) Las cadenas en el dúplex son antiparalelas y las DNA polimerasas solo pueden añadir nucleótidos en dirección 5’- 3’. 2) Es semiconservativa, es decir que conserva una cadena del DNA original. 3) Requieren de la presencia de un “cebador” de DNA o RNA para adicionar nucleótidos. Es decir dependen de la preexistencia de un fragmento patrón para iniciar el copiado de las cadenas paténtales. 3) Bidireccional y simultanea. 4) Necesita cebadores de RNA. 5) La adición de nucleótidos sólo ocurre en dirección 5’-3’. 6) Ocurre sólo una vez por ciclo celular. 7) Se replica el genoma completo. Replicón es la unidad del DNA que posee los elementos de control para la replicación (inicio y terminación). Cada replicón se activa tan sólo una vez en cada ciclo celular (*virus). Los replicones son numerosos en eucariotas, mientras que el genoma viral o el genoma bacteriano pueden ser considerados como replicones únicos. I. Iniciación • oriC: región de 240 pb con la capacidad de replicación independiente y controlada. Contiene una serie de secuencias repetidas (9-pb y 13-pb) que son características de cualquier secuencia de origen de la síntesis. Estas secuencias proporcionan un sitio de anclaje para las proteínas que inician la replicación. Existe también una secuencia rica en AT adyacente a oriC que aparentemente facilita el desenrollamiento del dúplex para exponer los templetes (cadenas base) sencillos a ser copiados. Las características comunes de estos sitios de origen de la replicación: • Secuencias repetidas cortas. • Sitios de reconocimiento para proteínas multiméricas de iniciación. • Sitios flanqueados por secuencias ricas en TA. oriC: E. coli La replicación tiene tres tipos de origen (secuencias iniciadoras): • oriC (E. coli). • SV40 (simian virus 40). • ARS (secuencias de replicación autónomas) en levadura. Hay tres tipos de DNA Polimerasa dependiente de DNA que se han aislado y purificado a partir de E. coli : 1) DNA Polimerasa I: llena huecos y participa en la reparación del DNA. 2) DNA Polimerasa II: realiza el control de calidad, es decir que se asegura que los nucleótidos sean los correspondientes, llena huecos y facilita la síntesis en cadenas dañadas (perpetuando las mutaciones). 3) DNA Polimerasa III: es la enzima funcional en las cuñas de replicación en las que sintetiza el DNA nuevo. En mamíferos son: α, β, γ, δ, ε Topoisomerasa (girasa) Al finalizar la síntesis de una dúplex circular, el resultado es de dos cadenas concatenadas, es decir, un anillo dentro de otro anillo. La topoisomerasa juega un papel importante de nuevo al abrir parte de la cadena, originando una mella por la que la otra cadena puede liberarse y así separar físicamente los cromosomas para permitir que cada uno de ellos pase a cada una de las dos células hijas. Hay tanto en eucariotas como en procariotas. Varios tipos dependiendo ligando y tupo de corte. 5 II. Elongación. Replicación en Procariotas y Eucariotas Formación del complejo de síntesis de cadenas hijas o Replisoma. Una de las cadenas es replicada discontinuamente: Fragmentos Okazaki (Cadena retrasada) (Cadena líder) La replicación es bidireccional y ocurre simultáneamente en ambas cadenas, por lo que la horquilla o burbuja de iniciación se va haciendo más larga conforme avanza la replicación al sintetizar nuevo DNA. Cada extremo de la horquilla representa una cuña creciente (growing fork) donde ambas cadenas se sintetizan, no obstante una cadena es sintetizada de forma continua a partir de un solo cebador de RNA. La cadena que va en dirección 5’ – 3’ crece en la misma dirección en la que crece la horquilla y por ello se le denomina la cadena líder. Una de las cadenas es replicada discontinuamente: Fragmentos Okazaki III II <15 nucleótidos fragmentos resultantes RNA-DNA (1000-2000 nucleótidos) La síntesis de la otra cadena se complica mucho ya que la dirección global de su crecimiento debe ser en dirección contraria a la que trabaja la DNA polimerasa. Este requerimiento incompatible con las funciones de la enzima que polimeriza el DNA es solucionado mediante un proceso que involucra un copiado discontinuo de la cadena retrasada mediante el uso de muchos cebadores de RNA. DNA polimerasa III: Síntesis de la cadena líder y la cadena retrasada. DNA polimerasa III es una proteína bastante grande (>600 kDa) y altamente compleja, formada por 10 polipéptidos diferentes. Posee una estructura de dímero asimétrico y contiene dos copias de la mayoría de las subunidades y dos sitios catalíticos para la adición de nucleótidos. La DNA polimerasa III procesa hasta 500,000 nucleótidos sin desprenderse, actividad indispensable para el crecimiento de la cadena líder. El “núcleo” o interior del complejo esta formado por las subunidades α, ε, θ contiene los sitios activos para la adición de nucleótidos. La habilidad de la DNA polimerasa para comportarse como un centro procesador se lo da la subunidad β que forma una estructura cerrada alrededor de la doble hélice y posteriormente asociarse y anclar el centro activo al final del cebador. Las subunidades restantes tienen como función convertir la parte activa en un centro procesador (processive enzyme). Sin éstas subunidades la enzima es mas bien distributiva, es decir, se desprende después de sintetizar entre 10 y 50 nucleótidos. Carga-abrazadera Portador de Abrazadera: γ, δ, δ',χ, ψ (complejo γ) abrazadera Abrazadera: β Centros catalíticos: Centro catalítico α, ε, θ Velocidad natural: 500-1000 nucleótidos por segundo 6 De las 6 subunidades restantes, 5 (γ,δ,δ’,χ,ψ) conforman el llamado complejo γ, que controla la transferencia de la subunidad β al sustrato (DNA-cebador), mientras que la subunidad τ dimeriza el complejo activo al interior de la polimerasa. Síntesis cadena LIDER Síntesis cadena RETRASADA Abrazadera: β Estudios bioquímicos han mostrado que el centro activo de la polimerasa libera un fragmento Okazaki completo pero no se disocia de la cuña de crecimiento. En contraste, la subunidad β y la primasa se disocian del la cuña de crecimiento y se reasocian más adelante en la cadena retrasada. Por tanto la subunidad β y primasa actúan de manera distributiva sobre la cadena retrasada. Líder Retrasada Portador de Abrazadera: γ, δ, δ',χ, ψ (complejo γ) Centros catalíticos: α, ε, θ τ mantiene el complejo dimerizado Estabilizador del Dímero: τ Replisoma asímetrico ó modelo de trombón DNA polimerasas: Procariotas - Eucariotas Video Replicación del DNA http://www.mcb.harvard.edu/Losick/images/TromboneFINALd.swf Todos estos eventos deben estar estrechamente correlacionados y coordinados para que la cuña de crecimiento avance a una velocidad comprobada de 500-1000 nucleótidos por segundo al momento de que ambas cadenas están siendo replicadas. Las DNA polimerasas I, II y III de E. coli, corresponden en función a las α, β, γ de eucariotas. Las DNA polimerasa III y su equivalente γ son las que llevan el peso de la labor de síntesis en las bacterias y en las mitocondrias, mientras las otras son más bien parte del sistema de reparación. Por su parte, la DNA polimerasa δ y la DNA polimerasa α se encargan en eucariotas de la síntesis de la hebra líder y de la hebra retardada, respectivamente, durante la replicación. III. Terminación Una vez que el ARN ha sido removido y reemplazado, los fragmentos de Okazaki deben ser enlazados. El extremo 3'-OH de un fragmento es adyacente al extremo 5'-fosfato del fragmento previo. LA DNA ligasa es la responsable de unir ambos fragmentos. Las ligasas están presentes tanto en procariotas como en eucariotas. La reacción se lleva en dos pasos e implica la formación de un complejo enzima-AMP. *****Péptido de terminación: tus-Prot TER son sitios específicos de terminación de la síntesis en los que se ancla tus-Prot. Esta proteína previene que la helicasa desdoble el dúplex de DNA interrumpiendo la cuña de replicación. El AMP del complejo enzimático se enlaza al 5fosfato de la mella formando un enlace fosfodiester en la extremidad 3´-0H liberando la enzima y el AMP. 7 Reparación del DNA Cambios en la secuencia de DNA pueden ser inducidos por: • Efectos ambientales tales como la radiación, químicos mutagénicos y descomposición térmica de los nucleósidos. • Errores de lectura de la polimerasa al momento de la replicación del DNA. Si estos errores no fueran corregidos, al menos en su mayor parte, las células somáticas acumularían tantas alteraciones en su secuencia que no podrían realizar sus funciones. Asimismo las células germinales portarían demasiadas diferencias y por tanto la progenie no podría formarse o llegaría un momento en que no seria viable. Mecanismo de reparación de la molécula que transfiere los caracteres hereditarios de generación en generación es indispensable para la vida. No obstante, y en el contexto evolutivo, salta a la vista que los errores o cambios en las secuencias no son removidos en su totalidad. Si existiera un mecanismo perfecto en el que ninguna alteración o cambio pudiera ser transferido a la siguiente generación, todos los organismos serían prácticamente idénticos. Si bien la reproducción sexual introduce variación en el acervo genético de las poblaciones, si la secuencia de ambos progenitores fuera idéntica, la recombinación durante la meiosis y por tanto los gametos formados de esta manera portaría exactamente la misma información. Los cambios aparecen, el sistema de reparación intracelular trata de corregir los daños, los cambios no corregidos pasan a la siguiente generación, si estos daños no permiten el correcto funcionamiento el organismo no pasa las “pruebas de selección natural” y los nuevos cambios desaparecen del acervo genético. Si los cambios no alteran la sobrevivencia del organismo, este podrá reproducirse y los cambios se acumularán en su progenie y en la progenie de su progenie. Las bases enzimáticas por las que se corrige una secuencia de bases no están plenamente conocidas en E. coli. Se sabe que la DNA polimerasa introduce o “se equivoca” en una base cada 10,000 adiciones. Dado que el tamaño de los genes en esta bacteria es de 1000 pb, esto quiere decir que uno de cada 10 genes podría tener una mutación o bien una tasa de 0.1 mutación por gen por generación. Sin embargo la tasa de mutación experimentalmente medida es mucho menor (0.00001 o 0.000001). El descubrimiento de la función de lectura de prueba descubierta inicialmente en la DNA polimerasa I y posteriormente en la III. Una cadena sintética de poli-dA fue usada como cebador y además contenía una base mal apareada en el extremo 3’. Al introducir DNA polimerasa I aislada de E. coli la base modificada era removida mediante actividad 3’-5’ exonucleásica. La mayor parte de las polimerasas descritas en bacterias poseen la actividad exonucleásica, sin embargo solo las DNA polimerasas d y e de los eucariotas poseen dicha actividad. El que la actividad sea compartida por varios tipos de polimerasas refleja la importancia de la actividad de lectura de prueba para evitar daño genético excesivo, manteniendo la fidelidad del copiado. Tipo de Lesión en la cadena de DNA Ejemplo - Causa Base faltante (Gap) Remoción térmica de purinas por ácidos o calor (10,000 purinas por dia por célula): A, G Remoción de bases alteradas por las DNA glucosilasas. Base alterada Radiación Iónica etilmetano) Base incorrecta Mutaciones en la subunidad exonucleásica 3’-5’ Protuberancias por deleción o inserción Agentes intercalantes (acridinas) introducen o remueven nucleotidos durante la replicación o recombinación. Pirimidinas ligadas o asociadas Dímeros ciclobutílicos, principalmente de Timina causados por radiación UV. Rupturas de cadena Radiación Iónica o químicos que rompen los enlaces fosfodiester (bleomicina) Ligamiento cruzado de cadena Ligamiento covalente de dos cadenas por agentes bifuncionales alquilantes (mitomicina). Fragmentos 3’ deoxiribosa Disrupción de la estructura de la deoxiribosa por radicales libres haciendo que se rompa la cadena. o alquilación (sulfato de Por su parte la DNA polimerasa III corrige conforme va avanzando la síntesis, retardandola momentáneamente hasta reemplazar la base dañada. Sistemas de reparación • Algunas enzimas revierten directamente ciertos tipos de daño al ADN. • Existen rutas para cortar bases, reparar bases faltantes y corregir errores mediante remoción y reemplazo. • Hay sistemas que funcionan mediante recombinación para reconstituir la copia no dañada utilizada para remplazar una secuencia dañada. • La ruta de los extremos no-homologos reincorpora extremos de doble cadena rotos. • Diversos tipos de DNA polimerasas pueden estar implicados en la restitución de tramos de ADN. 8 Sistemas de reparación por escisión en E. coli El « volteado » de base es utilizada por metilases y glicosilasas • El sistema UVR realiza incisiones a 12 bases de separación de ambas partes del ADN dañado, elimina el ADN entre ellos, y resintetza nuevo ADN. El sistema de reparación por escisión UVR incluye tres genes, uvrA, B, C, que codifican para los componentes de una reparación endonucleasa. La methylase invierte la citosina completamente fuera de la hélice, esta entra en una cavidad en la enzima donde es modificada. Luego, se devuelve a su posición normal en la hélice. Cuando se producen errores durante la replicación en E. coli, es posible distinguir la cadena original de ADN. Inmediatamente después de la replicación del ADN metilado, únicamente la cadena original parental porta los grupos metilos. En el período, mientras que la hebra recién sintetizada espera la introducción del grupo metilo, las dos cadenas se pueden distinguir. Desajustes pueden ser reparados de forma preferente para > 1 kb en torno a un sitio GATC. El resultado es que la nueva cadena síntetizada se ha corregido a partir de la hebra parental. Los mutantes dam- de E. coli presentan un aumento de la tasa de mutación espontánea. 9