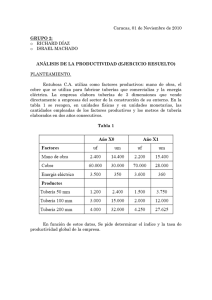
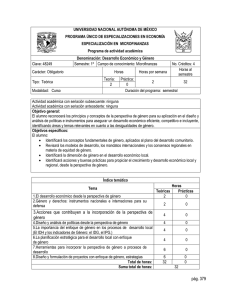
“Estudio Estructural y Funcional del Inhibidor de Serina – Proteasas
Anuncio

UNIVERSIDAD NACIONAL DE MAR DEL PLATA FACULTAD DE CIENCIAS EXACTAS Y NATURALES “Estudio Estructural y Funcional del Inhibidor de Serina – Proteasas tipo Germina presente en plantas de Trigo” Lic. Andrea Yamila Mansilla Director: Dr. Rubén Danilo Conde Instituto de Investigaciones Biológicas Tesis para optar por el título de Doctor en Ciencias Químicas Esta tesis fue desarrollada mediante Becas de Postgrado I y II otorgadas por el CONICET. Las mismas fueron dirigidas por el Dr. Rubén D. Conde y codirigidas por la Dra. Carmen I. Segarra, quienes sumaron sus conocimientos sobre Bioquímica y Biología Molecular, y Fisiología Vegetal y Fitopatología. Intentar significa arriesgar, arriesgar significa probablemente perder, perder significa aprender, aprender significa siempre ganar. (Anónimo) AGRADECIMIENTOS Al Dr. Conde, por permitirme realizar esta tesis bajo su dirección y darme lugar en su laboratorio. Por la libertad con la que me dejo trabajar, siempre contando con su apoyo, sabiduría y conocimientos (“wikiconde”). Por ser un ejemplo como persona y científico, por el buen ambiente en el que se trabaja y por priorizar a las personas ante todo. A Carmen, mi codirectora... Por estar siempre cuando la necesité, tanto en la discusión de los experimentos como en las correcciones de esta tesis. Por acompañarme, aconsejarme, por ser un ejemplo de lucha y por contribuir a que todos tengamos una facultad y una educación mejor. Por guiar mis primeros pasos en este mundo de la investigación y verme crecer… desde que me independicé de carpeta en la computadora hasta la finalización del doctorado. A Cristina Cordo y a todo su grupo de trabajo del CIDEFI (Facultad de Agronomía de la Universidad Nacional de La Plata), porque sin ellos no hubiera sido posible la realización de los ensayos a campo. Por su ayuda, predisposición y entusiasmo para trabajar. A Claudia Casalongué por estar siempre predispuesta a discutir nuestros experimentos, por el aporte de sus ideas para la realización de esta tesis, por preocuparse en mi futuro, por su alegría y energía. A la gente linda del laboratorio 3-4, a los que están y a los que se fueron, y que constantemente me ayudaron con sus consejos y buena onda. En especial a Marce, que aún estando lejos me brindó su ayuda y sus aportes son siempre un verdadero lujo. A Diego y Vicky, por alegrarse y compartir los días en que los experimentos daban bien y por levantarme el ánimo cuando todo salía mal (esos días si que abundaban!), por las charlas de los pasillos, por los mates, por sus consejos, por todo… Porque además de ser compañeros son muy buenos amigos y aunque la vida nos lleve por caminos diferentes sé que estarán siempre conmigo. A todos los integrantes del IIB por los momentos compartidos, por la ayuda con algún experimento o con el uso de algún equipo, por su compañerismo. A Mary, Tere y Vero, por la buena onda que tienen. Tere, gracias especialmente por responder siempre mis dudas acerca de las finanzas!!! A mis amigas… Vero, gracias por estar siempre, tanto en los momentos felices como en los difíciles de mi vida. A Rosy, Yesi, Lau y Ali, porque a pesar de que no nos veamos tanto como queremos se que están y que puedo contar con ellas. Por muchos años más de mates, charlas y anécdotas. A mis padres y a mi hermana, por estar orgullosos y confiar en mí, porque sin su apoyo nada de esto hubiera sido posible. A mis sobrinitos, porque son dos soles en mi vida. A Cristian por estar al lado mío, por su amor incondicional, porque la luchamos juntos, por aguantarme siempre, por darme fuerza en los momentos complicados, por admirarme y por tratar de prestar atención cuando quería explicarle algún experimento… (perdón!) A la Universidad Nacional de Mar del Plata y al CONICET porque permitieron la realización de esta tesis. ÍNDICE TEMÁTICO Índice .........................................................................................................................i Abreviaturas..............................................................................................................iv Anexo: Nomenclatura de los aminoácidos ...............................................................vi RESUMEN .............................................................................................................. 1 INTRODUCCIÓN .................................................................................................. 3 1. La proteólisis en las plantas .............................................................................. 3 1.1. Proteasas ........................................................................................................ 3 1.2. Las serina proteasas y los mecanismos de defensa de las plantas ................. 5 1.3. Inhibidores de Proteasas ................................................................................ 6 1.3.1. Familia Serpina ........................................................................................... 8 1.3.2. Familia Bowman-Birk ................................................................................ 8 1.3.3. Familia Kunitz ............................................................................................ 9 1.4. Funciones fisiológicas de los inhibidores de proteasas ................................ 10 1.4.1. Participación en los mecanismos de defensa frente a estrés biótico o abiótico ............................................................................................................. 10 2. Proteínas Germinas y GLPs ............................................................................. 11 2.1. Germinas y GLPs como miembros de la superfamilia de las Cupina ......... 13 2.2. Actividades Enzimáticas de las GLPs .......................................................... 14 2.3. GLPs como proteínas estructurales y receptores .......................................... 14 2.4. GLPs en estrés biótico y abiótico ................................................................. 15 3. Proteínas Multifuncionales .............................................................................. 18 OBJETIVOS .......................................................................................................... 20 MATERIALES Y MÉTODOS ............................................................................. 21 1. Material Biológico .......................................................................................... 21 1.1. Plantas .......................................................................................................... 21 1.2. Hongos ......................................................................................................... 21 2. Obtención del Fluido Intercelular (FI) ............................................................ 22 3. Obtención de la fracción FI70 ........................................................................ 22 4. Extracción de proteínas unidas a la pared celular ........................................... 22 5. Extracción de proteínas de embriones maduros de semillas de trigo ............. 23 6. Medición de actividades enzimáticas ............................................................. 23 6.1. Actividad proteolítica .................................................................................. 23 6.2. Actividades inhibitorias de la proteólisis ..................................................... 23 7. Métodos utilizados para la cuantificación y análisis de proteínas .................. 24 7.1. Cuantificación de proteínas ......................................................................... 24 7.2. Separación en geles de poliacrilamida de una dimensión............................ 24 7.2.1. SDS-PAGE ............................................................................................... 24 7.2.2. SDS-PAGE-Tricina .................................................................................. 24 i 7.3. Separación en geles de poliacrilamida bidimensionales .............................. 25 7.4. Técnicas de tinción de geles ........................................................................ 26 7.4.1. Tinción de proteínas con Coomassie Brillant Blue .................................. 26 7.4.2. Tinción de proteínas con Coomassie coloidal .......................................... 26 7.4.3. Tinción de proteínas con Nitrato de plata ................................................. 26 7.4.4. Tinción negativa con Zn – imidazol ......................................................... 27 7.5. Ensayos de Western blot .............................................................................. 27 7.6. Digestiones químicas o enzimáticas en gel .................................................. 28 7.6.1. Digestión con Bromuro de Cianógeno...................................................... 28 7.6.2. Digestión con hidroxilamina ..................................................................... 28 7.6.3. Digestión con tripsina ............................................................................... 29 7.7. Detección de actividades proteolíticas en gel .............................................. 29 7.8. Detección de actividades inhibitorias de la proteólisis en gel ..................... 29 7.9. Detección de actividad superóxido dismutasa (SOD) en gel ....................... 30 7.10. Detección de actividad pirofosfatasa/fosfodiesterasa (AGPPasa) en gel .. 30 8. Espectrometría de masas ................................................................................. 30 8.1. LC-MS/MS .................................................................................................. 30 8.2. MALDI-TOF/TOF ....................................................................................... 31 8.3. Análisis de los datos de espectrometría de masas........................................ 31 9. Preparación del anticuerpo primario específico de IPG ................................. 32 10. Modificación química de aminoácidos ......................................................... 32 11. Modelado 3D de IPG .................................................................................... 32 12. Métodos para la extracción, cuantificación y análisis de ARN .................... 33 12.1. Extracción de ARN total ............................................................................ 33 12.2. Cuantificación del ARN............................................................................. 33 12.3. Síntesis del ADN copia .............................................................................. 33 13. Métodos generales de clonado y transformación de E. coli ....................... 34 13.1. Amplificación de fragmentos de ADN mediante la técnica de reacción en cadena de la polimerasa (PCR) ..................................................................... 34 13.2. Electroforesis en geles de agarosa ............................................................. 34 13.3. Purificación de fragmento de ADN a partir de geles de agarosa ............... 34 13.4. Reacciones de ligación de fragmentos de ADN ........................................ 34 13.5. Transformación de células E. coli competentes con ADN ........................ 35 13.6. Mini-preparaciones de ADN ...................................................................... 35 13.7. Digestión del ADN con EcoR1 .................................................................. 36 13.8. Purificación del plásmido para su secuenciación ...................................... 36 14. Tratamientos de estrés .................................................................................. 36 14.1. Tratamientos de estrés abiótico.................................................................. 36 14.1.1 Estrés salino ............................................................................................. 36 14.1.2. Estrés por altas temperaturas .................................................................. 37 14.2. Estrés biótico: Ensayos de biocontrol de Trichoderma harzianum sobre el hongo patógeno Septoria tritici ........................................................................... 37 ii CAPÍTULO I CARACTERIZACIÓN ESTRUCTURAL Y FUNCIONAL DE IPG Introducción del Capítulo I .................................................................................. 38 RESULTADOS ...................................................................................................... 39 1. Digestión Química y Enzimática de IPG ......................................................... 39 1.1. Digestión de IPG con Bromuro de Cianógeno ............................................. 39 1.2. Digestión de IPG con tripsina....................................................................... 43 1.3. Digestión de IPG con hidroxilamina ............................................................ 45 2. Análisis de IPG a través de geles bidimensionales.......................................... 47 3. Identificación de la secuencia de su ADNc ..................................................... 50 4. Actividad AGPPasa de IPG ............................................................................. 54 5. Determinación de la mínima secuencia de IPG con actividad inhibitoria ....... 55 6. Identificación de aminoácidos esenciales de IPG............................................ 58 6.1. Modificación de histidinas ........................................................................... 58 6.2. Modificación de arginina .............................................................................. 59 6.3. Modificación de cisteínas ............................................................................. 61 7. Modelo estructural de IPG ............................................................................... 63 DISCUSIÓN ........................................................................................................... 69 CAPÍTULO II EXPRESIÓN DE IPG DURANTE EL DESARROLLO DE LA PLANTA DE TRIGO Y FRENTE A DISTINTAS SITUACIONES DE ESTRÉS Introducción del Capítulo II ................................................................................. 73 RESULTADOS ...................................................................................................... 74 1. Estrés abiótico ................................................................................................. 74 1.1. Estrés salino .................................................................................................. 75 1.2. Estrés por altas temperaturas ........................................................................ 85 2. Estrés biótico: inoculación con el hongo fitopatógeno Septoria tritici y el hongo biocontrolador Trichoderma spp. ............................................................. 89 2.1. Estudios realizados en la etapa final del macollaje ..................................... 91 2.2. Estudios realizados en espigazón ................................................................. 95 DISCUSIÓN .......................................................................................................... 100 CONCLUSIONES ................................................................................................ 105 BIBLIOGRAFÍA .................................................................................................. 107 iii ABREVIATURAS AAs: Aminoácidos ADN: Ácido desoxirribonucleico ADNc: Ácido desoxirribonucleico copia AGPPasa: Adenosina glucosa pirofosfatasa/fosfodiesterasa APS: Persulfato de amonio ARN: Ácido ribonucleico BCIP: 5-bromo-4-cloro-3-Indolil fosfato BNPP: bis (4-nitrofenil) fosfato BSA: Sero albúmina bovina Buffer: Solución reguladora de pH (del inglés) CHAPS: 3-(cloroamido propil)- dimetilamonio-1-propanosulfato DEPC: Dietilpirocarbonato DTT: Ditiotreitol EDTA: Acido etilendiaminotetraacético ESI: Ionización por electrospray FI: Fluido Intercelular FI70: FI calentado a 70 °C GLP: Proteínas tipo germina (del inglés germin-like protein) HR: Respuesta hipersensible IPG: Inhibidor de proteasas tipo germina JA: Ácido Jasmónico LB: Medio de cultivo Luria-Bertani MALDI: Desorción-ionización en matriz inducida por láser MS/MS: Espectrometría de masas en tándem MS: Espectrometría de masas NBT: Azul de nitrotetrazolio NEM: N-etilmaleimida OXO: Oxalato oxidasa PAGE: Electroforesis en geles de poliacrilamida PCR: Reacción en cadena de la polimerasa RT-PCR: Reacción en cadena de la polimerasa con transcriptasa reversa SDS: Dodecil sulfato de sodio iv SOD: Superóxido dismutasa TBST: Buffer tris salino con Tween-20 TEMED: N,N,N,N'-tetrametilnediamina TOF: tiempo de vuelo (del inglés time of flight) v ANEXO Nomenclatura de los aminoácidos Aminoácido Código de 3 letras Código de 1 letra Alanina Ala A Arginina Arg R Asparagina Asn N Ácido aspártico Asp D Cisteína Cys C Glutamina Gln Q Ácido glutámico Glu E Glicina Gly G Histidina His H Isoleucina Ile I Leucina Leu L Lisina Lys K Metionina Met M Fenilalanina Phe F Prolina Pro P Serina Ser S Treonina Thr T Triptófano Trp W Tirosina Tyr Y Valina Val V vi Resumen Resumen Las plantas están normalmente sujetas a una gran variedad de situaciones adversas, y por ello han desarrollado estrategias que les permiten sobreponerse a las mismas. En el caso del ataque de patógenos, la proteólisis juega un rol muy importante en los mecanismos de defensa. En nuestro laboratorio se encontró que el apoplasto de la hoja de trigo presenta una actividad proteolítica del tipo serina que participa en los mecanismos de defensa de la planta contra la infección del hongo fitopatógeno Septoria tritici. Luego, se demostró que esta actividad colocaliza con un inhibidor de serina proteasas que la regula. La secuencia aminoacídica del extremo N-terminal de dicho inhibidor permitió establecer que pertenece a la familia de proteínas tipo germinas (GLPs, del inglés germin-like proteins), por lo que se lo denominó IPG (Inhibidor de Proteasas tipo Germina). IPG, al igual que otras GLPs descriptas, es una glicoproteína que presenta también actividad superóxido dismutasa (SOD). Cabe mencionar que IPG es la primer GLP descripta con actividad de inhibidor de proteasas. Por esta razón, se propuso determinar si IPG es una nueva proteína no descripta hasta el momento. Con este fin, se obtuvieron secuencias aminoacídicas parciales de IPG mediante técnicas de proteómica. Las mismas permitieron aislar su ADNc y, consecuentemente, obtener su secuencia aminoacídica completa. Se encontró que la misma es 99% similar a una GLP de cebada que presenta actividad de adenosina glucosa pirofosfatasa/fosfodiesterasa (AGPPasa). Luego se constató que IPG posee esta actividad enzimática, por lo que se la puede considerar como una proteína multifuncional. Dado que IPG no presentó similitud de secuencia con los inhibidores de proteasas conocidos, se buscó el sitio responsable de esta acción. Mediante la modificación química de aminoácidos, se demostró que su única arginina es necesaria para inhibir proteasas. Además, mediante modelado tridimensional, se determinó que dicho aminoácido es periférico, lo que facilitaría su interacción con el sustrato. Por otra parte, y debido a que muchas proteínas de la familia de las GLPs han sido vinculadas con la resistencia de las plantas al estrés biótico y abiótico, se evaluó el comportamiento de IPG frente a distintas situaciones de estrés. En el caso de los estreses abióticos estudiados, salinidad y calor, se observó que IPG se relocaliza en la pared celular con el correspondiente aumento de sus actividades enzimáticas (SOD e inhibidor de proteasas). En el caso del estrés salino, se notó que dicha relocalización va acompañada de modificaciones bioquímicas y morfológicas en las plantas que indican 1 Resumen mayor tolerancia. Con respecto al estrés biótico, se estudió en ensayos a campo el rol de IPG en la interacción entre el hongo patógeno S. tritici y el hongo biocontrolador Trichoderma harzianum. Previamente, se había notado que los síntomas de la enfermedad provocados por el hongo S. tritici disminuyen en plántulas de trigo crecidas en invernáculo e inoculadas con cepas de Trichoderma. En ese caso, se observó un aumento de la actividad proteolítica del fluido intercelular (FI), a la que previamente se le había asignado capacidad antifúngica, y una concomitante disminución de la actividad inhibitoria de IPG, su regulador. En el presente estudio se evaluó a campo si las actividades enzimáticas relacionadas con la resistencia a la septoriosis mediada por el hongo biocontrolador T. harzianum también se modifican en hojas de estadios ulteriores de desarrollo del trigo. Se encontró que, a pesar de que IPG es una proteína minoritaria en esos estadios, continúa regulando la actividad proteolítica del FI. Esto sugiere que el estudio del mecanismo de regulación que ejerce IPG sobre la actividad proteolítica antifúngica de la hoja de trigo puede ser un aporte importante para el control biológico de la septoriosis a campo. Además, dada la presencia de IPG en estadios avanzados del desarrollo del trigo y sus características multifuncionales, podría considerarse como una potencial herramienta con fines agronómicos. 2 Introducción Introducción 1. La proteólisis en las plantas La proteólisis es esencial para el desarrollo y la fisiología de las plantas. Es responsable de la respuesta a estrés y de la limpieza celular mediante la eliminación de proteínas anormales, y por otra parte, suministra los aminoácidos necesarios para sintetizar nuevas proteínas. Las proteasas son reguladores claves de la homeostasis, crecimiento y desarrollo de las plantas. El hecho de que el genoma de Arabidopsis thaliana codifique para aproximadamente 600 proteasas, señala la importancia de la proteólisis en las plantas. Las proteasas son capaces de hidrolizar el amino terminal de proteínas (aminoproteasas, diproteasas o triproteasas), el carboxilo terminal (carboxiproteasas) o los enlaces peptídicos internos (endoproteasas) (Beers et al., 2004; Schaller, 2004; van der Hoorn and Jones, 2004). Además la proteólisis es fundamental para generar y catabolizar péptidos bioactivos como hormonas o zimógenos. En el presente trabajo de tesis se centró la información en una proteína con actividad de inhibidor de serina proteasas, por lo que se hará una revisión de las proteasas en general y el modo de acción de las serina proteasas en particular, de los inhibidores de proteasas y de las funciones de ambas como proteínas de defensa en las plantas. 1.1. Proteasas Las enzimas proteolíticas o proteasas, son enzimas que catalizan la ruptura hidrolítica de enlaces peptídicos específicos en sus proteínas blanco. Estas enzimas están ampliamente distribuidas en plantas, animales y microorganismos (Christeller, 2005; Haq et al., 2004; Joanitti et al., 2006; Lawrence and Koundal, 2002; Mosolov et al., 2001b; Mosolov and Valueva, 2005; Neurath, 1989; Ryan, 1990; Supuran et al., 2002; Valueva and Mosolov, 2004). En organismos superiores, cerca del 2% de los genes codifican para estas enzimas (Barrett et al., 2001). Las proteasas desempeñan un papel clave en muchos procesos biológicos, siendo indispensables para el mantenimiento y la supervivencia de los organismos. Las proteasas se clasifican como cisteína, aspartato, serina y metalo proteasas, según las características de su sitio activo (Barrett, 1986). A su vez, las proteasas de un tipo catalítico pueden ser agrupadas en familias según su especificidad al sustrato (Rawlings and Barrett, 1993). Por ejemplo, las serina proteasas se dividen en tres grupos según el tipo de residuo del sitio de ruptura (P1). Las proteasas tipo tripsina tienen 3 Introducción residuos cargados positivamente como lisina o arginina preferentemente en la posición P1. Las proteasas del tipo elastasa, en cambio, tienen residuos pequeños e hidrofóbicos en la posición P1 y las del tipo quimotripsina poseen residuos grandes e hidrofóbicos como fenilalanina, tirosina o leucina (Leung et al., 2000). El sitio activo de estas enzimas consiste en una triada catalítica de serina, histidina y aspártico (Ser/His/Asp). La unión del sustrato en el sitio activo forma un complejo de Michaelis, exponiendo el grupo carbonilo del enlace amida a un ataque nucleofílico del hidroxilo de la serina de la triada catalítica, en medio básico generado por el imidazol de la histidina (Fig. 1a). El intermediario tetraédrico resultante es estabilizado por puente de hidrogeno entre el NH de la serina 195 (para la quimotripsina) y la glicina (Gly193), que forma el agujero oxianión. Una transferencia del protón de la His57 a la amina del intermediario facilita la expulsión del fragmento C-terminal del sustrato dando un complejo acilo (Fig. 1b). El complejo acil-enzima es atacado por agua, con la formación de un nuevo intermediario tetraédrico (Fig. 1c) con una subsecuente ruptura vía catálisis acida de His57 para dar el fragmento amino terminal del sustrato y regenerar la Ser195 (Fig. 1). Figura 1. Mecanismo catalítico general de las serina proteasas. (a) interacciones por puente de hidrogeno con la histidina y el aspártico de la triada catalítica, activando el grupo OH de la serina para el ataque nucleofílico al enlace peptídico, formando el primer intermediario tetraédrico hemiacetálico. (b) transferencia del protón de la His57 a la amina del intermediario lo que facilita la expulsión del fragmento C-terminal del sustrato para dar el complejo covalente acilo. (c) ataque del agua al complejo para formar el segundo intermediario tetraédrico (d) que colapsa por catálisis acida de la His57 para regenerar la Ser195 y el extremo N-terminal del sustrato (Leung et al., 2000). 4 Introducción 1.2. Las serina proteasas y los mecanismos de defensa de las plantas La mayor cantidad de información que vincula a las serina proteasas con los mecanismos de defensa de las plantas se ha obtenido del estudio de las interacciones planta-patógeno. Se ha descripto que, cuando se produce el ataque de un patógeno, se inducen las serina proteasas de la planta lo que desencadena una respuesta denominada hipersensible (HR). Esta HR es el principio del proceso de defensa de la planta, que provoca necrosis y muerte celular para restringir el crecimiento del patógeno. Dicha respuesta, esta compuesta por un estallido oxidativo causado por las especies reactivas de oxigeno producidas por las células. Además, el ataque de patógenos desencadena respuestas bioquímicas en las plantas, incluyendo la acumulación de un grupo típico de proteínas llamadas PRPs (Proteínas Relacionadas con la Patogénesis) (Antão and Malcata, 2005). Estos mecanismos de defensa han sido estudiados por Tornero et al. (1996), quienes se centraron en la respuesta de las plantas de tomate a la infección por viroides. En dicha interacción planta-patógeno se observó la inducción de un conjunto de PRPs, de las cuales una de ellas mostró actividad endoproteolítica y, debido a su peso molecular de 69 kDa, fue llamada P69. Esta enzima es sintetizada como una preproproteína, y comparte homología con algunos dominios de las serina proteasas del tipo subtilisinas. La enzima madura se acumula en los espacios intercelulares de las hojas de las plantas infectadas con viroides. Por otra parte, los autores informaron que la P69 se induce en la planta no sólo en respuesta a patógenos como hongos y nematodos, sino también por moléculas como etileno y ácido salicílico, las cuales son consideradas como mediadoras de las respuestas de defensa en plantas. El rol de P69 podría estar asociado a la interacción de la superficie celular con el ambiente extracelular, directamente a través de la degradación de la matriz extracelular, o indirectamente activando la ruta de transducción de señales (Tornero et al., 1997). Con respecto a esta última función, se ha asociado a las serina proteasas en los mecanismos de transducción de señales (Antão and Malcata, 2005). Por ejemplo, se estableció que una xilanasa de Trichoderma viride (TvX) induce un estallido oxidativo y produce la muerte celular en un cultivo de células de tabaco (Yano et al., 1999); además se encontró que los inhibidores de serina proteasas inhiben dicha respuesta, lo que sugiere que estas enzimas pueden ser parte de una vía de señalización. Esta hipótesis fue apoyada por un estudio entre el hongo Cochliobolus victoriae y la planta de avena (Coffeen and Wolpert, 2004). En dicho sistema biológico, una toxina del hongo induce la HR en A. 5 Introducción sativa, la cual se caracteriza por cambios morfológicos y bioquímicos, y uno de ellos es la proteólisis de la Rubisco, un proceso regulado por una cascada de señalización, en el que están involucradas dos serina proteasas. Estas serina proteasas son específicas de dicha cascada y se determinó que estas proteasas permanecen en el FI como enzimas activas después de la inducción provocada por la infección con C. victoriae o estrés térmico. En nuestro laboratorio, se determinó que la actividad proteolítica de la primer hoja de trigo se modifica diferencialmente durante la inoculación de plántulas de trigo con el hongo fitopatógeno Septoria tritici, según se trate de un cultivar susceptible o resistente. Dicha actividad está asociada a dos isoformas de serina proteasas del FI. La misma aumenta en el cultivar resistente Pigüé y decrece en el cultivar susceptible Isla Verde y contribuye a los mecanismos de defensa del trigo a la septoriosis (Segarra et al., 2002). A pesar de que las enzimas proteolíticas son indispensables para las células y organismos, pueden ser dañinas cuando se encuentran en exceso. Por ello estas actividades deben estar reguladas y controladas (Rawlings et al., 2004b). Las formas de controlar a estas enzimas son varias y se pueden mencionar por ejemplo, la síntesis como pre-proteínas inactivas, la especificidad de sustrato y la interacción con proteínas que puedan inhibirlas. En el caso de la tripsina, por ejemplo, se dan los tres niveles de control, dado que se sintetiza como un precursor inactivo (tripsinógeno), el que requiere un procesamiento proteolítico para ser activado. Una vez que esta activada, la tripsina actúa específicamente sobre el enlace peptídico del carboxilo de los aminoácidos lisina o arginina y los controles suplementarios en su actividad se dan a través de la interacción con la antitripsina, el inhibidor de la forma activa (Laskowski and Qasim, 2000). 1.3. Inhibidores de Proteasas Los inhibidores de proteasas son muy comunes y están ampliamente distribuidos en todos los organismos. Han sido aislados y caracterizados de plantas, animales y microorganismos (Christeller, 2005; Haq et al., 2004; Mosolov et al., 2001a; Mosolov and Valueva, 2005; Supuran et al., 2002). Los inhibidores de proteasas de plantas son generalmente proteínas pequeñas, localizados en los tejidos de almacenamiento, como tubérculos y semillas, pero también han sido encontrados en las partes aéreas de las plantas (De Leo et al., 2002). Son 6 Introducción inducidos en plantas en respuesta a lesiones o ataque por insectos o patógenos (Ryan, 1990). En las plantas, los inhibidores de proteasas más descriptos son los que actúan como proteínas anti-metabólicas que interfieren con el proceso digestivo de insectos. Su capacidad defensiva se debe a la inhibición de las proteasas presentes en el tracto digestivo de los insectos o de las que son secretadas por los microorganismos, causando una reducción en la disponibilidad de los aminoácidos necesarios para el crecimiento y desarrollo (Lawrence and Koundal, 2002). Los inhibidores de proteasas han sido agrupados en distintas familias o subfamilias y en diferentes clanes, según sus secuencias o estructuras de los dominios respectivamente. Un dominio inhibitorio se define como el segmento de secuencia aminoacídica que contiene un solo sitio activo. Basados en la homología de secuencia de los dominios inhibitorios, los inhibidores de proteasas han sido clasificados en 48 familias (Rawlings et al., 2004a). Once de estas familias poseen inhibidores complejos, es decir que presentan más de un dominio inhibitorio, y contienen entre 2 y 15 dominios inhibitorios. Basados en su estructura terciaria, 31 de las 48 familias han sido asignadas a 26 clanes, indicando así que una gran proporción de familias no tiene relación en su estructura tridimensional. A diferencia de las proteasas, las familias de inhibidores no pueden ser agrupadas según el tipo catalítico de las enzimas que inhiben, porque algunas familias contienen inhibidores que actúan sobre distintas clases de proteasas (Rawlings et al., 2004a). Por ejemplo, los inhibidores de una familia que generalmente inhiben serina proteasas pueden tener integrantes que inhiben a cisteína o aspartilo proteasas (Heibges et al., 2003). De las familias de inhibidores existentes, en este trabajo describiremos a tres de ellas, cuyas características y modos de acción son muy diferentes: Bowman-Birk, Kunitz y Serpinas. Los inhibidores de las dos primeras familias son los más abundantes en plantas y comparten el mecanismo de acción canónico, pero presentan marcadas diferencias en cuanto a su estructura y composición aminoacídica. Los inhibidores de la familia de las Serpinas fueron seleccionados ya que poseen un mecanismo de acción muy particular denominado “suicida”. 7 Introducción 1.3.1. Familia Serpina Las serpinas vegetales han sido purificadas y caracterizadas a partir de semillas de cereales (Laskowski and Kato, 1980; Tsybina et al., 2004; Yoo et al., 2000), del polen y de los exudados del floema de Cucurbita maxima (Wieczorek et al., 1985). Como característica general, poseen una masa molecular de entre 39 a 43 kDa y la secuencia primaria es conservada entre los miembros de la familia. La mayoría inhibe serina proteasas, aunque se han descripto inhibidores de caspasas (Ray et al., 1992) y cisteína proteasas (Irving et al., 2002a; Irving et al., 2002b; Schick et al., 1998). En el caso de una serpina de cebada, se ha demostrado la capacidad de inhibir tripsina y quimotripsina en el mismo sitio activo. Debido al mecanismo de acción, los inhibidores de esta familia son inhibidores irreversibles, generalmente denominados “suicidas”. Esto se debe a que el inhibidor presenta un bucle en su sitio activo, que es reconocido por el sitio catalítico de la proteasa sustrato y se forma un complejo de inhibición cuando la proteasa rompe el enlace sensible (P1-P1') presente en el bucle de la serpina. Este complejo inicial, luego sufre un cambio conformacional en el que la serina proteasa es translocada lo que produce la distorsión de su sitio activo y la pérdida de actividad. (Huntington et al., 2000). 1.3.2. Familia Bowman-Birk Los inhibidores de esta familia se encuentran generalmente en semilla, pero también se los ha encontrado en hojas al producirse una lesión (Eckelkamp et al., 1993). Los inhibidores de las dicotiledóneas consisten en una sola cadena peptídica con masa molecular de 8 kDa. Poseen dos dominios homólogos con un sitio activo cada uno para las proteasas afines. Estos inhibidores interactúan independientemente pero de manera simultánea con dos proteasas, iguales o diferentes (Birk, 1985; Raj et al., 2002). El primer sitio activo suele ser específico para tripsina, quimotripsina y elastasa (Qi et al., 2005). La configuración del sitio activo esta estabilizado por la presencia de siete puentes disulfuro conservados (Chen et al., 1992; Lin et al., 1993). En cambio en monocotiledóneas hay de dos tipos. Unos consisten en una simple cadena peptídica de 8 kDa y un solo sitio activo, y el otro grupo tiene 16 kDa de masa molecular y 2 sitios activos (Prakash et al., 1996; Tashiro et al., 1987). Los inhibidores Bowman-Birk poseen un único bucle formado por nueve aminoácidos, conectados por un puente disulfuro que adopta una conformación canónica (Bode and Huber, 1992). Dicho bucle se denomina de “unión al sustrato” ya 8 Introducción que en ese lugar se une la proteasa (Lee and Lin, 1995), y el mecanismo de acción es canónico (mecanismo estándar de unión enzima-sustrato). 1.3.3. Familia Kunitz Los miembros de esta familia son en su mayoría activos contra serina proteasas, pero también pueden inhibir otras proteasas (Ritonja et al., 1990). Los inhibidores de esta familia están ampliamente distribuidos en plantas y se han descripto en legumbres, cereales y solanáceas (Ishikawa et al., 1994; Laskowski and Kato, 1980). Se ha descripto un inhibidor tipo Kunitz con actividad antifúngica en las raíces de Pseudostellaria heterophylla (Wang and Ng, 2006) y también se ha relacionado su producción con situaciones de estrés (Ledoigt et al., 2006; Park et al., 2005; Plunkett et al., 1982). La masa molecular de estos inhibidores generalmente es de 18-22 kDa, poseen dos puentes disulfuros y un solo sitio activo. Sin embargo, no todos los inhibidores de la familia tienen estas características y se ha propuesto una nueva clasificación según el contenido de cisteínas (Oliva et al., 2010). Es decir, se dividen en cuatro grupos: los que no tienen Cys (ID Merops I03.12, http://merops.sanger.ac.uk), los que tienen una sola Cys (ID Merops I03.16, http://merops.sanger.ac.uk), los que poseen dos (inhibidores de Bauhinia tipo II) y aquellos con más de dos residuos de Cys (inhibidores de Bauhinia tipo I), que participan en el puente disulfuro Cys-Cys. Con respecto al sitio activo de los inhibidores de tripsina de esta familia, por lo general poseen una lisina (Lys) o arginina (Arg), tal como es el caso de los inhibidores de Acacia confusa (Hung, 1994), Cassia obtusifolia (Liao et al., 2007) y también el inhibidor de tripsina de Enterolobium contortisiliquum (ECTI) (Batista et al., 1996) y Leucaena leucocephala (LITI) (Souza-Pinto et al., 1996). En el caso de los inhibidores de la elastasa, esta posición es ocupada por un residuo de alanina (Ala) (Bode and Huber, 2000). Algunos inhibidores poseen un segundo sitio reactivo independiente (Dattagupta et al., 1999), mientras que otros presentan diferentes alteraciones en esta región, tal como es el caso del inhibidor de garbanzos, HGPI (Srinivasan et al., 2005), en el que la Arg o Lys fue sustituida por el motivo de secuencia glicina/glutamatoisoleucina-serina (G/E-I-S). Los inhibidores de esta familia son canónicos, es decir, poseen un mecanismo de acción clásico de unión al sustrato, y el complejo que forman con la proteasa se disocia muy lentamente (Ritonja et al., 1990). 9 Introducción 1.4. Funciones fisiológicas de los inhibidores de proteasas Los inhibidores pueden participar activamente en la regulación de procesos proteolíticos, actuar como proteínas de reserva, y servir como un importante elemento en el sistema de defensa de las plantas contra plagas y microorganismos fitopatógenos (Mosolov and Valueva, 2005). 1.4.1. Participación en los mecanismos de defensa frente a estrés biótico o abiótico Es de gran importancia para la supervivencia de las plantas generar una respuesta adecuada a nivel celular frente a diversos estímulos externos. Debido a que las plantas no pueden moverse en busca de condiciones más favorables, han desarrollado respuestas fisiológicas y bioquímicas para superar condiciones de estrés o ataque de patógenos. Las proteasas y sus inhibidores están relacionados con la ruptura de proteínas, y su rol es esencial en la adaptación a las diversas condiciones ambientales (Vierstra, 1993) constituyendo un mecanismo molecular muy importante involucrado en la respuesta a estrés. La mayoría de los estudios realizados en los inhibidores de proteasas se han enfocado a su función frente al daño mecánico y ataque de patógenos e insectos. Los daños mecánicos o producidos por insectos determinan la acumulación de inhibidores de proteasas en las hojas de la mayoría de las plantas (Brown and Ryan, 1984). La síntesis y compartimentalización vacuolar de los inhibidores de proteasas en hojas de plantas ha sido descripta por Ryan desde los años 80 (Ryan, 1980), cuando describió que estos inhibidores no actúan sobre las proteasas de la planta sino que bloquean las proteasas digestivas de los animales y hongos. Numerosos estudios han demostrado la capacidad de los inhibidores de proteasas de plantas de suprimir la actividad de la enzimas del tracto digestivo de insectos (Gatehouse JA et al., 2000; Ryan, 1990) y que una dieta rica en inhibidores de serina y cisteína proteasas hace que se suprima el crecimiento, desarrollo y reproducción de los insectos (Broadway and Duffey, 1986; Gatehouse and Boulter, 1983; Kuroda et al., 1996). Los inhibidores de serina proteasas tienen un efecto más pronunciado sobre insectos lepidópteros, en los que estas enzimas juegan un rol importante en evitar que se degraden las proteínas alimento (Ryan, 1990). Los inhibidores de cisteína proteasas afectan a coleópteros, que tienen cisteína proteasas activas en el intestino (Benchekroun et al., 1995; Lecardonnel et al., 1999). Por ello, se puede concluir que los inhibidores ejercen su acción contra los insectos suprimiendo la 10 Introducción asimilación normal de las proteínas alimento (Gatehouse JA et al., 2000; Ryan, 1990). El rol de los inhibidores en defensa de los insectos fue demostrado con el uso de plantas transgénicas (Hilder et al., 1987; Irie et al., 1996; Mosolov and Valueva, 2005). Por otra parte, también se ha descripto su participación frente a situaciones de estrés abiótico. Por ejemplo se ha descripto la inducción del gen de un inhibidor Bowman-Birk en plantas de Arabidopsis thaliana sometidas a estrés oxidativo inducido por Al+3 (Richards et al., 1994) y en raíces de trigo afectadas por metales pesados (Snowden et al., 1995). También se ha reportado la inducción de un inhibidor tipo Kunitz de soja (SKTI) cuando las plantas son sometidas a sequía (Downing et al., 1992). El estrés no solo induce inhibidores de serina proteasas, se ha descripto el aumento de expresión de inhibidores de cisteína proteasas en órganos vegetativos de cebada en respuesta a anaerobiosis, iluminación deficiente y bajas temperaturas (Gaddour et al., 2001). Sin embargo en estos casos no se conocen los detalles de los mecanismos moleculares involucrados. En los casos descriptos anteriormente, la respuesta de defensa está asociada al aumento de la actividad de los inhibidores de proteasas. Sin embargo, en el caso del inhibidor de proteasas IPG (objeto de estudio de la presente tesis) se observó su disminución en el FI de la hoja de trigo del cultivar resistente Pigüé luego de la infección con S. tritici (Segarra et al., 2003). Esta disminución determina un aumento de la actividad proteolítica, cuya actividad antifúngica fue demostrada (Segarra et al., 2002). 2. Proteínas Germinas y GLPs A partir de una búsqueda de proteínas específicas de la germinación, en 1980 fue identificada una proteína de trigo que no reveló homología con ninguna proteína conocida hasta ese momento. Tampoco se le pudo atribuir una función especifica por lo que se le asignó un nuevo nombre: Germina (Thompson and Lane, 1980). Esta proteína se caracteriza por ser una proteína glicosilada, multimérica, resistente a la temperatura y a la proteólisis, localizada en la pared celular y con actividad oxalato oxidasa OXO (Dratewka-Kos et al., 1989; Jaikaran et al., 1990; Lane et al., 1986; Lane et al., 1993; Woo et al., 2000). La síntesis de esta proteína coincide con el inicio del crecimiento del embrión durante la germinación. Años mas tarde se descubrieron numerosas proteínas con distintos porcentajes de identidad con respecto a la germina de trigo, que se clasificaron en germinas o GLPs (del inglés Germin Like Proteins). 11 Introducción Las germinas constituyen un grupo muy homogéneo que comprende proteínas con más del 90% de identidad con la germina de trigo y poseen 95% de identidad entre ellas. Las germinas han sido extensamente estudiadas en trigo y cebada. Hasta el momento no han sido detectadas en las dicotiledóneas en las que se las ha buscado (Grzelczak et al., 1985). Es posible, entonces que sean proteínas específicas de las gramíneas. Las GLPs, contrariamente a las germinas, son un grupo de proteínas muy heterogéneo, están presentes en musgos, gimnospermas y en prácticamente todas las angiospermas incluyendo las gramíneas. En este grupo, las identidades de aminoácidos oscilan entre un 25 a casi el 100%. Las comparaciones con las germinas revelan identidades que van desde el 30% hasta un valor máximo de el 70%. La ausencia de proteínas con rango de identidad entre el 70 y el 90% con las germinas permite definirlas como grupos muy distintos (Bernier and Berna, 2001). Figura 2. Representación esquemática de la organización típica de germinas y GLPs. En color azul claro se indica una conservación de secuencia moderada, el azul oscuro (boxes A, B y C) indica una alta conservación de secuencia y el rojo representa una región altamente variable. Los aminoácidos conservados que definen los boxes A, B y C se señalan debajo. Las dos cisteínas que forman un puente disulfuro interno y los cuatro aminoácidos implicados en la unión al ion metálico se marcan en rojo. pep, péptido señal; X, cualquier aminoácido hidrofóbico. Extraído de Bernier y Berna (2001) La Figura 2 muestra la organización típica de las germinas y GLPs (Bernier and Berna, 2001) . Todas las germinas y GLPs están compuestas aproximadamente por 220 aminoácidos incluyendo un péptido señal. La forma madura de estas proteínas está compuesta por 3 oligopéptidos altamente conservados que conforman los boxes A, B y C. La secuencia del box A contiene una de las dos cisteínas involucradas en la formación del puente disulfuro interno que estabiliza el extremo N-terminal. Los boxes B y C contienen tres residuos de histidina y uno de glutamato involucrados en la unión al metal y responsables de la estructura β-barril, característica de la superfamilia de las cupinas a la que pertenecen. Otra característica de estas proteínas es el sitio de 12 Introducción glicosilación (NXS/T) entre el box A y B. Además la mayoría de las GLPs, pero no las germinas, poseen el tripéptido RGD o el relacionado KGD. Las proteínas animales tales como fibronectina y vitronectina que contienen este péptido son proteínas de adhesión a la matriz extracelular que interactúan con integrinas y así participar en el intercambio de información entre el exterior y el interior de las células. Originalmente se creía que las germinas eran pentámeros, pero los datos cristalográficos han demostrado que son hexámeros (Woo et al., 2000; Woo et al., 1998). Dichos hexámeros se organizan como trímero de dímeros, y cada dímero es estructuralmente equivalente al monómero de las proteínas de almacenamiento 7S (vicilinas). La interacción entre los dímeros para formar el hexámero es predominantemente hidrofóbica y se establece entre los dominios α- hélice del cterminal. En cuanto a las GLPs también se las ha descripto in vivo como complejos homohexaméricos con una masa molecular aparente de aproximadamente 100 kDa en SDS-PAGE seminativos (Christensen et al., 2004; Vallelian-Bindschedler et al., 1998; Zhang et al., 1995). 2.1. Germinas y GLPs como miembros de la superfamilia de las Cupinas La identificación de la superfamilia de las cupinas se basó originalmente en el hecho de que la germina de trigo compartía 9 aminoácidos (HI/THPRATEI) con una proteína relacionada al estrés, una esferulina, producida durante el hambreado del Myxomicete Physarum polycephalum. Esta similaridad se extendió a las proteínas globulinas de reserva, como vicilinas y leguminas. El conocimiento de la estructura 3D de estas proteínas permitió un alineamiento detallado basado en dichas estructuras, que reveló un gran grupo de proteínas de eucariotas, procariotas, arqueas y virus que tienen la forma de β-barril (“cupa” en latín significa pequeño barril) (Dunwell, 1998). El dominio cupina característico comprende dos motivos conservados, cada uno correspondiente a dos laminas β, separados por una región menos conservada compuesta de otras dos laminas β con un región variable entre medio. El tamaño total de la región entre motivos varia desde un mínimo de 11 aminoácidos en algunas enzimas microbianas, 50 aminoácidos en algunas proteínas de reserva y mas de 100 aminoácidos para ciertos factores de transcripción y dioxigenasas (Dunwell et al., 2001; Dunwell et al., 2000). Se les ha asociado una gran diversidad de funciones enzimáticas como: dioxigenasas, decarboxilasas, hidrolasas, isomerasas y epimerasas; así como 13 Introducción también otras no enzimáticas tales como: unión de hormonas, factores de transcripción nuclear y almacenamiento en semilla. 2.2 Actividades Enzimáticas de las GLPs Cuatro diferentes actividades enzimáticas han sido asociadas a esta familia de proteínas: oxalato oxidasa (OXO), superóxido dismutasa (SOD), ADP glucosa pirofosfatasa/fosfodiesterasa (AGPPasa) e inhibidor de serina proteasas. La actividad OXO ha sido vinculada mayormente con las germinas y no con las GLPs, sin embargo, se ha descripto una GLP de Silene vulgaris (Bringezu et al., 1999) que posee dicha actividad. La actividad SOD ha sido detectada en varias GLPs. Los primeros antecedentes provienen de una GLP aislada de cultivos de células in vitro del musgo Bárbula Unguiculata (Yamahara et al., 1999) y de la proteína principal del néctar del tabaco, llamada Nectarina I (Carter and Thornburg, 2000). Ambas poseen al manganeso como cofactor. Luego fue descripta esta actividad para las GLP de cebada (Christensen et al., 2004), arveja (Gucciardo et al., 2007), azalea (Kondo et al., 2008) y uva (Godfrey et al., 2007). En hojas de cebada se aislaron dos isoformas de AGPPasa (Rodríguez-López et al., 2001) que resultaron ser distintos oligómeros de una GLP de cebada descripta por Vallelian-Bindschedler et al. (1998). Finalmente, en nuestro laboratorio se aisló una GLP que presentó actividad de inhibidor de serina proteasas (Segarra et al., 2003) y actividad SOD. Esta actividad de inhibidor de serina proteasas no ha sido detectada hasta el momento en ninguna otra GLP, por lo que es de interés determinar la secuencia aminoacídica de esta proteína para establecer si se trata de un nuevo miembro de la familia dada su nueva actividad. El primer capitulo de la presente tesis abordará este estudio y tratará de establecer el sitio activo para dicha función. 2.3. GLPs como proteínas estructurales y receptores Las GLPs se extraen fácilmente de la matriz extracelular debido a que están asociadas débilmente mediante enlaces iónicos. Sin embargo, algunas de estas proteínas se insolubilizan tras un estrés. En cebada, un tratamiento térmico resulta en una mayor resistencia a un patógeno por un nuevo mecanismo que no involucra la formación de la papila o muerte celular hipersensible. Las modificaciones de la pared celular, 14 Introducción incluyendo la insolubilización de unas pocas proteínas, parece ser responsable de dicha resistencia. Después del tratamiento térmico o de la exposición al peróxido de hidrógeno o a una infección, dos GLPs no pueden ser recuperadas en el FI (VallelianBindschedler et al., 1998). La insolubilización por estrés no es una propiedad general de las germinas y GLPs, dado que este fenómeno no se observó por ejemplo ni con la germina de trigo, ni con AtGER3 de Arabidopsis expresada en plantas transgénicas de tabaco (Membre et al., 2000). En cultivos celulares de lupino blanco, se observó todo lo contrario. El tratamiento de estas células con CuCl2 produjo un cambio en la distribución de GLP, resultó ser más abundante en el filtrado del cultivo con respecto a las células control, en las que la mayor cantidad de GLP estaba asociada a la fracción de pared celular (Wojtaszek, 1997). También se les ha asignado la función de receptor, tal como se ha descrito para dos GLPs, denominadas ABP19 y ABP2, de Prunus persica L. cv. Akatsuki que se unen a auxinas (Ohmiya et al., 1998) y para otra GLP de Pisum sativum, que posee la secuencia RGD, que funcionaría como receptor de la ricadesina, proteína que media la unión de Rhizobium y Agrobacterium a las células de las plantas hospedantes (Swart et al., 1994). 2.4. GLPs en estrés biótico y abiótico Las plantas se defienden del ataque de los patógenos por una gran variedad de mecanismos que incluyen la producción de compuestos antimicrobianos específicos, el entrecruzamiento de lignina y de proteínas de la pared celular, síntesis de carbohidratos que refuercen la pared celular, y la muerte celular hipersensible (Dunwell et al., 2000). Una de las primeras funciones sugeridas para la germina fue su participación en la respuesta a patógenos (Lane et al., 1986; Lane, 1994). Dicho rol fue directamente demostrado por Schweizer et al. (1999) mediante ensayos de expresión transitoria del gen de la germina (gf 2.8) en la epidermis de trigo. La penetración de Blumeria graminis se redujo en las células que expresaban el gen intacto de la germina de trigo. La expresión del gen de una GLP de trigo (TaGLP2a) o de la germina modificada genéticamente sin actividad OxO produjo un efecto similar, aunque más débil. Esto demostró que la actividad OxO no es lo único que determina la resistencia al patógeno (Schweizer et al., 1999). Además se determinó que al expresar el gen de la germina gf 2.8, el producto se insolubiliza en el lugar de la penetración del hongo con un concomitante aumento en la producción de H2O2. Así, se propuso que las germinas y 15 Introducción GLPs juegan un rol estructural, reforzando la pared celular durante el ataque del patógeno. Por otro lado, cuando expresaron una GLP de cebada HvGER2a (VallelianBindschedler et al., 1998) no se vieron efectos sobre la penetración del patógeno. Posteriormente, Christensen et al. (2004) demostraron que los miembros de la subfamilia 4 de las GLP son componentes fundamentales de la resistencia basal en gramíneas y luego Zimmerman et al. (2006) realizaron un estudio completo de la función de 6 subfamilias de GLP en cebada. En dicho trabajo se demostró la compleja interacción de las proteínas HvGER en la regulación de la resistencia basal contra B. graminis. Por otra parte, se estudió la organización y funcionalidad de 8 secuencias promotoras de la subfamilia 4 de las GLPs, y se demostró que, tanto en trigo como en cebada estos promotores son inducidos ante estrés biótico (Himmelbach et al., 2010). En arroz, se identificaron 12 miembros de la familia de las GLPs definidos dentro de un mismo marcador de selección de resistencia a enfermedades (Manosalva et al., 2009). Experimentos de interferencia de ARN (RNAi) determinaron que la expresión de estos 12 genes de GLP contribuyen directamente a la resistencia ante el ataque de dos patógenos fúngicos diferentes Magnaporthe oryzae y Rhizoctonia solani (Manosalva et al., 2009). También se ha descripto la participación de una GLP en la defensa contra insectos herbívoros (Lou and Baldwin, 2006). En plantas de tabaco, Nicotiana attenuata, el ataque por parte del especialista herbívoro Manduca sexta, o la adición de secreciones orales de las larvas en las heridas de la planta, aumenta el nivel de transcripto de NaGLP. En plantas con este gen silenciado las larvas de dos herbívoros de N. attenuata provocaron mayor daño que en plantas control. Se constató una disminución en la producción de H2O2, glucósidos terpeno e inhibidores de proteasas. Esto sugiere que NaGLP participa en la respuesta de defensa de N. attenuata por la producción de H2O2 y por la vía de señalización del etileno. En nuestro laboratorio se demostró que la GLP del FI de la hoja de trigo con actividad de inhibidor de serina proteasas (IPG) regula la actividad de dos proteasas extracelulares que modifican su actividad frente al ataque del hongo patógeno Septoria tritici (Segarra et al., 2002; Segarra et al., 2003) . Dando de esta forma otro ejemplo de la participación de las GLPs en los mecanismos de defensa de la planta al ataque de los patógenos. La participación en las respuestas de defensa de las germinas y GLPs no se limita al estrés biótico. En plántulas de cebada sometidas a estrés salino, se encontró que 16 Introducción dos proteínas Gs1 y Gs2 aumentaban en raíces pero disminuían en coleóptilos (Hurkman et al., 1991). Dichas proteínas pertenecen a la familia de las germinas y se propuso que dicho aumento podría estar relacionado con una función protectora. Años más tarde, Hurkman y Tanaka (1996) demostraron que la expresión del gen de dicha germina es regulado de manera tejido-específica y modulada por los tratamientos con NaCl y hormonas vegetales (ácido indol acético, abscísico, salicílico y jasmónico). Por otra parte, en el musgo Barbula unguiculata, se encontró un aumento en la expresión del gen de una GLP con actividad SOD y que dicho aumento es causado por la disociación de la proteína BuGLP de la pared celular al medio en las células en fase logarítmica (Nakata et al., 2002). Recientemente se ha demostrado que el estrés salino provoca un cambio en la localización de la germina en los embriones de trigo, encontrándose predominantemente en las células del coleóptilo y disminuyendo en las células de la coleorriza (Caliskan, 2009). En la halófita facultativa Mesembryanthemum cristallinum (planta que habita en el hielo) se identifico una GLP que estaría involucrada en el balance hídrico de la planta (Michalowski and Bohnert, 1992). Berna y Bernier (1999) estudiaron la expresión del gen de la germina (gf 2.8) y demostraron que la expresión del mismo es estimulada por algunos estreses abióticos, especialmente a iones metálicos pesados como Cd+2, Cu+2 y Co+2 y daño mecánico (wounding). También se correlacionó dicho aumento con la liberación de H2O2 en el apoplasto. En la planta halófita Atriplex lentiformis se encontró que la proteína GLP (AlGLP) con actividad SOD es regulada diferencialmente por daño mecánico y acido abscísico (Tabuchi et al., 2003). Los autores sugieren que el aumento en la expresión de AlGLP es para aumentar los niveles de H2O2 y de esa forma modificar la estructura de la pared celular en respuesta al daño mecánico. En la mayoría de los casos, la participación de las germinas y GLPs en los mecanismos de defensa frente al estrés biótico o abiótico se relaciona con las actividades enzimáticas OXO y SOD. Hasta el momento el hallazgo de que una GLP del apoplasto de la hoja de trigo con actividad inhibitoria de proteasas es único. Como se mencionó anteriormente dicha actividad se relacionó con la respuesta de defensa de la planta frente al ataque del patógeno Septoria tritici, pero no se ha determinado aún su participación frente a otros estreses bióticos o abióticos. Por lo que, parte de la presente tesis abordará dicho estudio. 17 Introducción 3. Proteínas Multifuncionales La proteína IPG de trigo estudiada en este trabajo de tesis resultó ser una GLP con al menos tres funciones enzimáticas, lo que la caracteriza como una proteína multifuncional. La multifuncionalidad constituye un paradigma emergente que amplia el concepto tradicional de que una secuencia de aminoácidos determina un plegamiento o estructura única y ésta a su vez, una sola función. Si bien la visión tradicional permitió un notable avance en el desarrollo de la bioquímica moderna, la gran cantidad de información acumulada sobre la relación estructura /función ha dado base a un nuevo concepto sobre la plasticidad de las proteínas. Las proteínas multifuncionales permiten aumentar el número de funciones “reutilizando” a las proteínas para un fin distinto. De este modo, un simple polipéptido puede cumplir distintos roles en diferentes momentos y/o lugares, aumentando la complejidad de los organismos sin un aumento en el numero de genes. Hay un número cada vez mayor de proteínas a las que se les asigna más de una función en el organismo y que pueden o no estar relacionadas en una misma vía metabólica. Un ejemplo bien conocido es el de la fosfoglucosa isomerasa. Las funciones de esta enzima dependen de su localización dentro o fuera de la célula. Dentro de la célula es una enzima citosólica, ubicua, que cataliza el segundo paso de la glucólisis, la interconversión de glucosa-6-fosfato a fructosa-6-fosfato. Sin embargo, al ser secretada por la célula cumple al menos 4 funciones distintas. Posee actividad de neuroleuquina (Chaput et al., 1988; Faik et al., 1988), que es una citocina que hace que las células B maduren en células secretoras de anticuerpos, y un factor de crecimiento neuronal que promueve la supervivencia de algunas neuronas espinales embrionarias y algunos nervios sensoriales (Gurney et al., 1986a; Gurney et al., 1986b). Además, también funciona como factor de motilidad autocrino, que es una citocina que estimula la migración celular (Watanabe et al., 1996). Por último, es también un mediador de diferenciación y maduración que puede producir la diferenciación de las células humanas de la leucemia mieloide (Xu et al., 1996). Otras proteínas tienen funciones distintas dependiendo de su estado de oligomerización, es decir, tienen una función como monómero y otra como multímero. Por ejemplo, la subunidad de 37 kDa de la gliceraldehído-3-fosfato deshidrogenasa humana es una enzima glicolítica que, como un tetrámero, convierte gliceraldehído-3fosfato a 1,3-difosfoglicerato. Sin embargo como un monómero, es una uracil ADN 18 Introducción glicosilasa nuclear (Meyer-Siegler et al., 1991). Esta actividad es importante para la eliminación del uracilo que está presente en el ADN por el uso erróneo de dUTP durante la síntesis de ADN o desaminación de residuos de citosina. En una revisión reciente del tema de proteínas multifuncionales (Huberts and van der Klei, 2010) se reflexiona acerca de lo difícil que resulta evaluar que tan abundantes son este grupo de proteínas multifuncionales en los organismos. Sin embargo, la gran cantidad de proteínas que se han identificado con esta característica sugieren que se trata de un fenómeno general, hallado en todos los reinos en los que se clasifican los organismos vivos (Woese and Fox, 1977). Se necesita profundizar el estudio de este tipo de proteínas para entender mejor la esencia de su función y contestar varios interrogantes que se han planteado en relación a qué lleva a una proteína a desempeñar una función en particular o a cómo evolucionan las funciones de las proteínas multifuncionales. En este sentido, su descubrimiento permite considerar la existencia de otro nivel de complejidad en la célula. 19 Objetivos Objetivos General Contribuir al estudio estructural y funcional de la proteína IPG de hoja de trigo y, en general, de proteínas tipo germina. Específicos: En el presente trabajo de tesis se plantearon los siguientes objetivos: 1) Identificación de la secuencia aminoacídica completa de IPG. 2) Identificación de la secuencia de su ADNc. 3) Caracterización estructural-funcional de IPG. Es decir, las propiedades estructurales de su actividad inhibitoria de serina-proteasas. 4) Análisis de la expresión de IPG en distintos órganos de la planta de trigo y frente a distintos procesos fisiológicos. 20 Materiales y Métodos Materiales y Métodos 1. Material Biológico 1.1. Plantas Plántulas de trigo (Triticum aestivum L.) del cultivar Relmo Centinela fueron crecidas en un cuarto de cultivo a 25 °C e iluminadas con lámparas Grolux (Sylvania inc., MA, USA) a una radiación promedio de 250 µmol/m2 por segundo y durante 14 horas/día. Como sustrato se utilizó vermiculita regada con solución nutritiva de Hoagland (1950). Para ensayos a campo se utilizaron plantas de trigo susceptibles a septoriosis del cultivar Buck Guapo y Buck CL51 (Centro de Investigaciones de Fitopatología – CIDEFI- Facultad de Ciencias Agrarias y Forestales, Universidad Nacional de La Plata) 1.2. Hongos El aislamiento de S. tritici provino de plantas de trigo ProINTA Molinero naturalmente infectadas. Para la esporulación, este se cultivó en tubos de inclinación con el medio de agar de malta (30 g/l de extracto de malta, 5 g/l de peptona, 2 g/l de extracto de levadura y 20 g/l de agar en agua destilada). Los conidios se obtuvieron inundando el medio de cultivo cubierto por la colonia mucosa del hongo contenido en la caja de Petri con 5 ml. de agua destilada estéril y quitándolos con una barra doblada de vidrio. La suspensión resultante se filtró con una tela de malla tramada y el filtrado se ajustó a 1 x 106 esporas/ml. Inmediatamente antes de la inoculación de las plantas, se mezcló con 50 µl/l de 0.05% (v/v) Tween 20 en solución acuosa. Se utilizaron dos aislamientos de Trichoderma harzianum: Th.cc5 y Th.118. Ambas cepas se cultivaron en medio de agar de papa glucosado (APG 2%) con fotoperíodo de 12 horas. de luz (3500 lux emitida por tubos fluorescentes de luz fría con un adicional de luz cercana al UV de 365 nm) a 20 ºC por 7 días. Los conidios de cada aislamiento se cosecharon inundando los recipientes con agua destilada estéril y luego frotando la superficie media con una barra estéril de vidrio. Después de filtrar a través de 2 telas de malla tramadas, la concentración de propágulos se ajustó a 1 x 10 8 conidios/ml con el agregado de Tween 20 en solución acuosa (0.05% v/v). Las semillas de trigo recubiertas con Trichoderma spp. se prepararon según (Perelló and Mónaco, 2007). Dos horas antes de sembrar, las semillas se colocaron en solución de hipoclorito de sodio al 5% (v/v) por 5 minutos. Diez ml de la suspensión de cada aislamiento de T. harzianum se colocaron en 90 ml de agar agua al 2,5%. Estas suspensiones fueron 21 Materiales y Métodos mezcladas con 85 gramos de semillas de trigo desinfectadas por 20 min. Finalmente, esta solución se agitó en agitador magnético durante 20 minutos. Después de drenar el líquido en exceso, las semillas recubiertas con Trichoderma spp. se secaron bajo corriente de aire estéril por 3 horas. Las semillas control se trataron con una suspensión de agar agua y agua destilada sin Trichoderma spp. 2. Obtención del Fluido Intercelular (FI) El FI se obtuvo a partir de hojas sometidas a diferentes condiciones experimentales tal como lo describieron Pinedo et al. (1993). Cinco gramos de hojas de trigo (80 – 100 hojas) se sumergieron a 4 °C en 30 ml de solución de extracción de FI que contenía buffer fosfato de sodio 50 mM, pH 7.5; 2-mercaptoetanol 0,1% (v/v); cloruro de sodio 0.6 M y Tween-20 0,13% (v/v). Las hojas se sometieron a vacío durante tres períodos de 10 segundos separados por intervalos de 30 segundos cada vez. Luego se secaron en papel de filtro, se colocaron sobre filtros de vidrio fritado ubicados en tubos y se centrifugaron a 400 x g durante 20 minutos. En promedio, a partir de cada gramo de peso fresco se obtuvieron 0,25 ml de FI. Las muestras fueron controladas de manera tal de asegurar la inexistencia de contaminación con proteínas intracelulares (Pinedo et al., 1993). 3. Obtención de la fracción FI70 El FI fue calentado a 70 °C durante 30 minutos y purificado por centrifugación a 16000 x g durante 15 minutos. Esta fracción se denominó FI70. Las proteínas presentes en esta fracción fueron precipitadas con acetona 90 % (v/v) a -20°C. El sedimento se solubilizó en buffer fosfato de sodio 25 mM pH 7.5 o buffer desnaturalizante, concentrándose entre 4 y 5 veces. 4. Extracción de proteínas unidas a la pared celular Las proteínas de la pared celular se extrajeron según lo descripto por VallelianBindschedler et al. (1998) con algunas modificaciones. Aproximadamente 0.5 gr de hojas de trigo a las que se le había extraído el FI fueron maceradas con N2 (l). El polvo obtenido fue lavado 3 veces con acetona para eliminar los pigmentos y luego se incubó por una hora en una solución de extracción que contenía SDS 1 % (p/v) y NaCl 200 mM. Posteriormente se recuperó el sobrenadante por centrifugación a 3000 x g y se concentró con acetona (apartado 3). 22 Materiales y Métodos 5. Extracción de proteínas de embriones maduros de semillas de trigo Aproximadamente 50 semillas de trigo fueron imbibidas en agua o solución salina durante 0, 1, 5, 18 y 24 horas. Posteriormente, se extrajeron los embriones de las semillas y fueron homogeneizados en 0.5 ml de buffer Hepes/KOH 20 mM pH 7.5, conteniendo CH3COOK 100 mM, (CH3COO)2Mg 3.6 mM y 2-mercaptoetanol 3 mM por 15 minutos, tal como lo describen Cuming y Lane (1979). Dicho homogenato fue centrifugado a 3000 x g durante 20 minutos y el sobrenadante se concentró con acetona tal como se describió para las proteínas del FI70 (apartado 3.). 6. Medición de actividades enzimáticas 6.1. Actividad proteolítica La actividad proteolítica se determinó según Pinedo et al. (1993) en una mezcla de reacción conteniendo 0.25 µg de tripsina ó 25-100 µg de proteínas del FI, caseína 2 mg/ml y buffer fosfato de sodio 50 mM pH 7.5 en volumen final 500 µl. La reacción se desencadenó por la adición de caseína. La mezcla fue incubada durante 1 hora a 37 °C. La reacción fue detenida por el agregado de 50 µl de acido tricloroacético 50% (p/v) y guardada a 4 °C. El material soluble conteniendo los productos de la proteólisis fue colectado por centrifugación a 2000 x g durante 20 minutos. Alícuotas de 200 µl se neutralizaron con 200 µl de KOH 0.3 M, agregándose luego 400 µl de buffer borato de potasio 0.4 M pH 9.7 y una solución de fluorescamina 0.3 mg/ml acetona (Udenfriend et al., 1972). La absorbancia de los derivados de fluorescamina fue medida a 390 nm, utilizándose 100-200 nmoles de leucina como estándar. Todas las mediciones se realizaron por triplicado. La actividad proteolítica fue expresada en términos de unidad por hoja, µg de tripsina o %, según corresponda. Cada unidad corresponde a 1 nmol de leucina liberado por hora. 6.2. Actividades inhibitorias de la proteólisis Cuando fue indicado, en la reacción descripta para medir la actividad proteolítica (apartado 6.1.), antes de la adición de la caseína, la mezcla fue pre incubada durante 30 minutos a 37 °C con 20-50 µg de FI70. La actividad inhibitoria fue expresada en términos de unidad por hoja, µg de tripsina o %, según corresponda. Cada unidad corresponde a la cantidad de inhibidor necesaria para inhibir la actividad proteolítica en un 50%. 23 Materiales y Métodos 7. Métodos utilizados para la cuantificación y análisis de proteínas. 7.1. Cuantificación de proteínas La concentración de proteínas se determinó utilizando el método del ácido bicinconínico (Smith et al., 1985), utilizando como patrón una solución de albúmina de suero bovino (BSA, Sigma). 7.2. Separación en geles de poliacrilamida de una dimensión 7.2.1. SDS-PAGE La electroforesis de proteínas en presencia de SDS (SDS-PAGE) se llevó a cabo según el método descripto por Laemmli (1970), empleando el sistema de geles verticales Miniprotean III (BioRad). En todos los casos, se utilizó una relación de acrilamida: bis-N’,N’-metil-bisacrilamida 30:1 % (p/v). El gel de separación se preparó en solución Tris-HCl 375 mM pH 8.8 conteniendo SDS 0,1 % (p/v) y una concentración final de acrilamida de 12 % (p/v). El gel de concentración se preparó en solución TrisHCl 125 mM pH 6.8 conteniendo SDS 0,1 % (p/v) y acrilamida 5 % (p/v). En todos los casos se adicionó persulfato de amonio (APS) 0,1 % (p/v), como iniciador de la polimerización del gel y TEMED 0,4 % (v/v) como catalizador de la formación de radicales libres a partir del APS. Las muestras se disolvieron en buffer de muestra que contenía Tris-HCl 0.13 M pH 6.8, azul de bromofenol 12% (p/v), glicerol 25% (p/v), SDS 5% (p/v) y 2mercaptoetanol 12.5% (v/v). Cuando fue indicado, las muestras se calentaron durante 3 minutos a 100 °C. La separación electroforética se realizó a 4°C en buffer Tris-HCl 25 mM pH 8.3, glicina 192 mM y SDS 0,1 % (p/v), aplicando una intensidad de corriente constante de 20 mA/gel. Una vez finalizada la separación electroforética, las proteínas presentes en el gel se visualizaron por distintos métodos de tinción. 7.2.2. SDS-PAGE-Tricina Los geles de tricina descriptos por Schägger and von Jagow (1987) constan de tres geles de poliacrilamida de distintas densidades: un gel superior o concentrador, un gel intermedio o espaciador y un gel inferior o separador, con tamaño de poro estrecho 24 Materiales y Métodos que separa a las proteínas en función de su tamaño. Los geles de tricina son muy resolutivos para proteínas de bajo peso molecular (1-100 kDa). En esta tesis se trabajó también con los geles de poliacrilamida-urea descriptos por Schägger (2006), en los cuales la urea reduce la movilidad electroforética de las proteínas en general y en particular de las proteínas de baja masa molecular. Para este tipo de geles se utilizó una relación acrilamida: bis-N`,N’-metilbisacrilamida 46.5:3 % (p/v). El gel de separación se preparó en solución Tris-HCl 1M pH 8.45 conteniendo SDS 0,1 % (p/v), urea 6 M y una concentración final de acrilamida de 16% (p/v). El gel espaciador se preparó en solución Tris-HCl 1M pH 8.45 conteniendo SDS 0,1 % (p/v), glicerol 8 % (v/v) y acrilamida 10 % (p/v). Por último, el gel concentrador se preparó en solución Tris-HCl 0.75 M pH 8.45 conteniendo SDS 0.075 % (p/v) y acrilamida 4%. En todos los casos se adicionó APS 0.1% (p/v) de y TEMED 0.4% (v/v). La separación electroforética se realizó a 4°C a corriente constante de 10 mA/gel. El buffer catódico consistía en una solución Tris 0.1 M, Tricina 0.1 M y SDS 0.1% (p/v). El buffer anódico era Tris-HCl 0.2M pH 8.9. 7.3. Separación en geles de poliacrilamida bidimensionales Las proteínas del FI70 se separaron por su punto isoeléctrico y masa molecular mediante el empleo de geles bidimensionales. En la primera dimensión las proteínas se separaron por punto isoeléctrico mediante la técnica de NEPHGE (non-equilibrium pH gel electrophoresis) descripta por O´Farrell (1977). La mezcla de polimerización contenía: acrilamida 5.4% (p/v), CHAPS 2% (p/v), urea 9 M y 6,8% (v/v) de una mezcla de anfolitos con los siguientes rangos de pH: 3-10 (1%), 4-6.5 (2%), 5-8 (3%) y 2-11 (0.8%). El sedimento seco de FI70 producto de la concentración con acetona (20 µg proteína) fue disuelto en 10 µl de una solución de CHAPS 4% (p/v), DTT 0.2 M, urea 18 M y luego mezclado con 10 µl de anfolitos (rango de pH 3-10). La corrida electroforética de la primera dimensión se llevo a cabo a 4 °C a un voltaje de 500 V por 10 minutos y luego a 700 V por 3,5 horas. Se utilizó H3PO4 0.05% en el cátodo y NaOH 100 mM en el ánodo. Para la segunda dimensión las proteínas se separaron según su masa molecular en geles SDS-PAGE 12%, previamente las proteínas fueron reducidas y alquiladas. Brevemente, antes de la segunda dimensión los capilares de acrilamida son tratados 4 25 Materiales y Métodos minutos con el buffer C (SDS 3% (p/v), DTT 50 mM, Tris-HCl 0.5 M pH 6.8) y luego otros 4 minutos con buffer D (SDS 3%, iodoacetamida 0.2 M, TRIS-HCl 0.5 M pH 6.8). Inmediatamente los capilares son apoyados sobre un gel en placa 12% (1 mm espesor, 90 mm ancho, 75 mm longitud) y corridos a 4 °C en las mismas condiciones descriptas para un SDS-PAGE convencional (apartado 7.2.1.) 7.4. Técnicas de tinción de geles 7.4.1. Tinción de proteínas con Coomassie Brillant Blue Los geles se sumergieron en una solución de Coomassie brillant blue R-250 0.25% (p/v) (Sigma) en una mezcla metanol: ácido acético: agua (50:10:40). Para la decoloración de los geles se empleó una mezcla metanol: ácido acético: agua (50:10:40). 7.4.2. Tinción de proteínas con Coomassie coloidal Se siguió el método descripto por Neuhoff (1988). Brevemente, los geles se sumergieron en solución fijadora conteniendo etanol 30% (p/v) y acido fosfórico 2% (v/v) durante 12 horas. Luego fueron lavados 3 veces durante 30 minutos con agua destilada y equilibrados por 1 hora con una mezcla metanol: sulfato de amonio: ácido fosfórico: agua (25:16:5:54). Finalmente se agregó a la mezcla anterior Coomassie Blue G-250 1 g/L de y se incubó con esta suspensión coloidal durante 2 días. 7.4.3. Tinción de proteínas con Nitrato de plata Se siguió el método descripto por Blum et al. (1987). Los geles fueron lavados tres veces con etanol 50% (v/v) por 20 minutos. Posteriormente se sumergieron 1 minuto en tiosulfato de sodio 0.02% (p/v) y se lavaron 4 veces por períodos de 20 segundos con agua destilada. Luego se incubaron 20 minutos en oscuridad con la solución de tinción que consiste en AgNO3 0.01 M y formaldehído 0.125% (v/v). Los geles fueron lavados con agua destilada para retirar el exceso de esta solución y se incubaron en solución reveladora. Esta última contiene Na2CO3 6% (p/v), formaldehído 0.125% (v/v) y tiosulfato de sodio 0.04 % (p/v). Una vez visualizadas las proteínas, se agrega una solución de metanol 50% (v/v) y ácido acético 12% (v/v) para detener la tinción. 26 Materiales y Métodos 7.4.4. Tinción negativa con Zn – imidazol Esta técnica de tinción desarrollada por Fernández-Patrón et al. (1992) es altamente sensible y reversible. El método esta basado en la precipitación selectiva del imidazolato de zinc en el gel excepto en las zonas donde hay proteínas. Debido a que las proteínas no son fijadas ni modificadas, es un método útil para eluir las proteínas de interés de los geles de poliacrilamida. Luego de la electroforesis los geles son lavados con agua e incubados por 20 minutos en una solución de imidazol 0.2 M conteniendo SDS 0.1% (p/v). Posteriormente se lavan los geles con agua destilada y se coloca una solución de ZnSO4 0.2 M. Al cabo de 1 minuto el fondo del gel se torna de color blanco debido a la precipitación del complejo Zn-imidazol-SDS y las bandas de proteínas permanecen sin coloración. Para eluir una proteína de interés de la banda del gel, se debe cortar la banda e incubar dos veces por 5 minutos con una solución de EDTA 100 mM en buffer fosfato de sodio 50 mM pH 7.5, para eliminar el ión zinc unido a la proteína. Posteriormente se procede a la elución de la proteína realizando una ruptura mecánica de la banda de gel para generar micro partículas. Se agrega buffer fosfato de sodio 50 mM pH 7.5 y se agita vigorosamente durante 20 minutos. Se centrifuga a 5000 x g durante 10 minutos y el sobrenadante contiene la proteína de interés con aproximadamente un 80 % de rendimiento. 7.5. Ensayos de Western blot Las proteínas, previamente separadas mediante SDS-PAGE, se transfirieron a membranas de nitrocelulosa (BioRad) empleando un equipo de transferencia semi-seco (Invitrogen). La transferencia se realizó en buffer Tris-HCl 25 mM, glicina 150 mM y metanol 20 % (v/v) a temperatura ambiente durante 20 minutos, aplicando una intensidad de corriente constante de 200 mA/gel. Las membranas de nitrocelulosa se bloquearon por incubación durante 12 horas a 4 °C en buffer TBST (Tris-HCl 50 mM pH 8.0, NaCl 150 mM, Tween-20 0.5 % (v/v)) conteniendo 5 % (p/v) leche descremada en polvo. Una solución de TBST pero con el agregado de sólo 3 % (p/v) de leche descremada en polvo se utilizó para la dilución del anticuerpo primario y se mantuvo en contacto con las membranas a temperatura ambiente durante 2 horas con agitación suave. El exceso de anticuerpo se eliminó mediante cuatro lavados de 15 minutos con buffer TBST. Luego se adicionó el 27 Materiales y Métodos anticuerpo secundario anti-IgG conjugado con la enzima fosfatasa alcalina o fluorescente (Cy TM 5; Invitrogen) utilizando la misma solución anterior. Al cabo de 2 horas de incubación a temperatura ambiente se efectuaron cuatro lavados de 15 minutos con TBST. La visualización se realizó dependiendo al anticuerpo secundario utilizado. Para el caso del anticuerpo secundario conjugado a fosfatasa alcalina, la actividad enzimática se reveló incubando la membrana en 5 ml de buffer AP (Tris-HCl 0,1 M pH 9.5, NaCl 0,1 M y MgCl2 5 mM) adicionado con 7,5 μl de BCIP 50 mg/ml (Sigma) y 11 μl de NBT 50 mg/ml (Sigma). Para el caso del anticuerpo fluorescente la visualización se realizó escaneando la membrana en el canal rojo del STORM 840 y analizadas con el programa Image-Quant 5.2 (Molecular Dynamics). 7.6. Digestiones químicas o enzimáticas en gel 7.6.1. Digestión con Bromuro de Cianógeno Se siguió el protocolo descripto por Córdoba et al. (1997). La proteína de interés es separada en un SDS-PAGE 12% teñido de acuerdo con Rosenfeld et al. (1992) con Coomassie Brilliant Blue 0.2% (p/v), metanol 20% (v/v) y acido acético 0.5 % (v/v) por 20 minutos y luego desteñida con metanol 30% (v/v). La banda es cortada del gel, transferida a un tubo eppendorf y lavada dos veces con 150 µl de una solución acuosa de acetonitrilo 50% (v/v) por 20 minutos a temperatura ambiente. Posteriormente, se seca la pieza de gel utilizando un evaporador centrifuga SAVANT y luego se rehidrata con 150 µl de una solución de BrCN (16 µg/µl en acido fórmico 70% (v/v)) en oscuridad a temperatura ambiente por 48 horas. El líquido se evapora totalmente con una corriente de N2 (g) bajo campana y se agregan 2 veces 150 µl de agua para eliminar el exceso de reactivo, que luego se evaporan en el SAVANT. Los péptidos resultantes de la digestión son eluídos en buffer de muestra. 7.6.2. Digestión con hidroxilamina Para la digestión con hidroxilamina se siguió el protocolo descripto por Saris et al. (1983). La banda correspondiente a la proteína de interés fue cortada del SDSPAGE 12% teñido de acuerdo con Rosenfeld et al. (1992), transferida a un tubo eppendorf y lavada dos veces con 150 µl de una solución acuosa de acetonitrilo 50% (v/v) por 20 minutos a temperatura ambiente. Posteriormente, la pieza de gel es secada utilizando un evaporador centrifuga SAVANT y sumergida en la solución de ruptura 28 Materiales y Métodos compuesta por: hidrocloruro de hidroxilamina 2M, hidrocloruro de guanidinio 6M, Tris 15 mM y el pH es ajustado a 9.3 por el agregado de LiOH 4.5 M. La digestión se lleva a cabo a 45 ºC durante 3 horas y se agrega buffer de muestra para detener la reacción y eluir los péptidos. 7.6.3. Digestión con tripsina La proteína de interés es separada en un SDS-PAGE teñido de acuerdo con Rosenfeld et al. (1992). La banda fue cortada del gel, transferida a un tubo eppendorf y lavada dos veces con 150 µl de una solución de acetonitrilo 50% (v/v) en NH4HCO3 200 mM de por 20 minutos a temperatura ambiente. Posteriormente, se seca la pieza de gel utilizando el evaporador centrifuga SAVANT y luego se rehidrata con una solución de tripsina (250 µg/ml en NH4HCO3). La digestión se lleva a cabo durante 12 horas a 37 °C y luego el líquido es completamente evaporado en el SAVANT. 7.7. Detección de actividades proteolíticas en gel La actividad proteolítica del FI de las hojas fue examinada mediante SDSPAGE, conteniendo gelatina 0.1% tal como lo describieron Heussen y Dowdle (1980). Las electroforesis se corrieron a 4 °C a 10 mA/gel. Luego de la electroforesis, los geles fueron lavados para remover el SDS, realizándose tres lavados de 10 minutos cada uno con buffer fosfato de sodio 250 mM pH 7.5, Tritón X-100 1% (v/v). Luego se realizó un lavado de 10 minutos con buffer fosfato de sodio 250 mM pH 7.5, CaCl2 10 mM. Finalmente fueron incubados a 37 °C durante 3 horas en buffer fosfato de sodio 250 mM pH 7.5 y teñidos con Coomassie Blue R-250 (apartado 7.4.1.). Las bandas claras revelaron la actividad proteolítica, en tanto que la gelatina no degradada apareció como un fondo azul oscuro. 7.8. Detección de actividades inhibitorias de la proteólisis en gel La actividad inhibitoria de la proteólisis del FI70 fue determinada mediante SDS-PAGE con acrilamida 12% y gelatina 0.1% copolimerizada, seguido por la incubación con una solución de tripsina 1 mg/ml (Sigma) durante 12 horas a 37 °C. Previo a la incubación con la tripsina se realizaron los lavados descriptos anteriormente (apartado 7.7.). Luego de la incubación con la tripsina, los geles fueron teñidos con Coomassie Blue (apartado 7.4.1.) las bandas de proteínas que exhiben actividad 29 Materiales y Métodos inhibitoria se visualizaron por su color azul oscuro, contrastando con el fondo claro del gel. 7.9. Detección de actividad superóxido dismutasa (SOD) en gel La identificación de actividad SOD en el FI o FI70 se realizó mediante SDSPAGE (Beauchamp and Fridovich, 1971). La muestra proteica se disolvió en buffer de muestra omitiendo el agregado de 2-mercaptoetanol. Se sembraron 20 µg de FI70 y finalizada la electroforesis los geles se incubaron en oscuridad por 1 hora en presencia de buffer fosfato de sodio 50 mM pH 7.5; NBT 0.25 mM; Riboflavina 33 µM; TEMED 30 mM. Luego se expusieron a una lámpara de 400 W durante 30 a 60 minutos. Cuando se visualizaron las bandas claras en un fondo oscuro, los geles se lavaron y se mantuvieron en agua destilada. 7.10. Detección de actividad pirofosfatasa/fosfodiesterasa (AGPPasa) La identificación de la actividad AGPPasa se realizó mediante un SDS-PAGE y utilizando la metodología descripta por Rodríguez-López et al. (2001). Brevemente, 20 µg de FI fueron disueltos en buffer de muestra sin 2-mercaptoetanol y finalizada la electroforesis el gel fue incubado con bis (4-nitrofenil) fosfato (BNPP; Sigma) 5 mM y MgCl2 5 mM. Cuando se visualizaron las bandas amarillas debidas a la producción del 4-nitrofenol, los geles se escanearon. 8. Espectrometría de masas 8.1. LC-MS/MS Los péptidos obtenidos de la ruptura en gel de IPG con BrCN fueron analizados por LC/MS/MS en la Universidad de Alicante, España. Para ello, las bandas de gel se lavaron con NH4HCO3 25 mM y agua para eliminar las impurezas del colorante y del SDS, antes de su reducción con DTT 10 mM y alquilación con iodoacetamida 100mM en NH4HCO3 50 mM. A continuación estas bandas se digirieron con tripsina porcina modificada (Promega, Madison WI) en NH4HCO3 25 mM durante 6-7 horas a 37º C. Los péptidos resultantes de la digestión se extrajeron en una solución de bicarbonato de amonio, luego en acetonitrilo 70% (v/v) y finalmente en ácido fórmico 1% (v/v). Dichos péptidos trípticos fueron separados mediante un sistema de cromatografía nano LC (Agilent 1100) y analizados con un espectrómetro de trampa de iones XCT plus (Agilent) equipado con una fuente nano-ESI (MS/MS) 30 Materiales y Métodos 8.2. MALDI-TOF/TOF Los spots obtenidos del gel bidimensional fueron analizados por espectrometría de masas en el CEQUIBIEM (FCEyN – UBA). Las piezas de gel fueron tratadas de manera similar a la descripta en el apartado anterior y analizadas por MALDI-TOF (matrix assisted laser desorption ionization–time of flight) en el espectrómetro Ultraflex II (Bruker). Algunos picos masas fueron analizados nuevamente (TOF-TOF). 8.3. Análisis de los datos de espectrometría de masas. Se utilizó el programa MASCOT (Matrix-Science) para analizar los datos provenientes de la espectrometría de masas (MALDI-TOF) y de la espectrometría en tándem MALDI-TOF/TOF y ESI-MS/MS. Este programa compara los datos de relación masa/carga obtenidos experimentalmente, con los patrones de péptidos y proteínas de las bases de datos. La búsqueda se realizó en la base de datos no redundante de NCBI (National Center for Biotechnology Information), en un principio utilizando todas las entradas y posteriormente restringiendo la búsqueda al reino viridiplantae. Los parámetros de búsqueda fueron: tolerancia de desvío de masa molecular entre 50-100 ppm o 0.1 Da a 2.5 Da para caso de MS/MS, máximo de 2 sitios no digeridos por la tripsina (missed cleavages) y las modificaciones variables permitidas en la búsqueda fueron carbamidometilación de cisteínas y oxidación de las metioninas. Se utilizaron otros programas para confirmar los resultados del MASCOT MS/MS como: Phenix (GeneBio), OMMSA (Open Mass Spectrometry Search Algorithm) y Peaks (de novo sequencing, Bioinormatics Solutions Inc.). Para comparar manualmente los picos m/z obtenidos experimentalmente para IPG con los teóricos de alguna proteína de base de datos se utilizó el programa MSDigest del Protein Prospector (http://prospector.ucsf.edu). Además se utilizó el programa GlycoMOD (http://www.expasy.ch/tools/glycomod) para determinar si la masa de algún péptido de IPG evidencia una posible glicosilación, al comparar con la digestión teórica de la AGPPasa. 31 Materiales y Métodos 9. Preparación del anticuerpo primario específico de IPG El FI70 seco fue disuelto en buffer de muestra y calentado a 100 °C durante 3 minutos. Luego fue separado por SDS-PAGE y teñido con Zinc-imidazol (apartado 7.4.4.). La banda mayoritaria de 18-21 kDa (aproximadamente 800 µg proteína) fue cortada del gel y mezclada con 2 ml de buffer fosfato de sodio 25 mM pH 7.5 y 2 ml de adyuvante completo de Freund's. Esta suspensión fue inyectada subcutáneamente a una coneja blanca adulta. Dos inyecciones de refuerzo con adyuvante incompleto fueron realizadas a los 30 y 60 días luego de la primera inyección, ambas con 600 µg de antígeno. Cuatro semanas más tarde, se colectó la sangre (aproximadamente 30 ml) de la vena femoral del conejo. El suero fue separado por centrifugación a 2000 x g por 15 minutos y guardado a -20 °C. El titulo del anticuerpo fue determinado por dot blot según Hawkes (1986). 10. Modificación química de aminoácidos Los residuos de arginina fueron modificados con ninhidrina 100 mM por 30 minutos en buffer fosfato de sodio 100 mM pH 9.1 a 37 °C tal como lo describió Chaplin (1976) y con concentraciones crecientes de 2,3-butanodiona en buffer fosfato de sodio 100 mM de pH 8.5 en oscuridad a 37 °C de acuerdo con Kumar et al. (2004). El dietilpirocarbonato (DEPC), un reactivo que modifica el grupo imidazol de las histidinas, fue utilizado en una concentración de 10 mM por 30 minutos en buffer fosfato de sodio pH 6.8 (Romaniouk and vijay, 1997). Los residuos de cisteínas fueron modificados por el tratamiento con Netilmaleimida (NEM) 100 mM de por 1 h en buffer fosfato de sodio 200 mM pH 6.8 a 37 ºC. Simultáneamente se realizaron los controles omitiendo el agregado de los agentes modificadores. 11. Modelado 3D de IPG La estructura 3D de IPG fue modelada en Swiss-model workspace (Arnold et al., 2006) usando como molde la estructura cristalina de la proteína oxalato oxidasa de cebada [Código PDB 2et7; (Opaleye et al., 2006)]. Esta proteína tiene un 40% de identidad con IPG y el e-value del modelo fue 3.84041e-27. El modelo resultante fue graficado usando el programa PyMOL (The PyMOL Molecular Graphics System, Versión 1.2r3pre, Schrödinger, LLC.). La calidad del modelo tridimensional de IPG fue analizada mediante el programa Verify3D (Eisenberg et al., 1997). Además se 32 Materiales y Métodos evaluaron los ángulos diedros psi y phi de cada uno de los aminoácidos a través del gráfico de Ramachandran. Dicho gráfico fue realizado con el programa RAMPAGE (Lovell et al., 2003). 12. Métodos para la extracción, cuantificación y análisis de ARN 12.1. Extracción de ARN total El ARN total se extrajo a partir de 100-200 mg de la primera hoja de trigo de 12 días. El tejido se maceró con N2 líquido y la extracción de ARN total se realizó utilizando el reactivo TRIZOL (Invitrogen Life Technologies) siguiendo las indicaciones del proveedor. 12.2. Cuantificación del ARN La cuantificación del ARN se realizó determinando la absorbancia de las muestras en 260 nm en espectrofotómetro Ultrospec 1100. La concentración de ARN se calculó teniendo en cuenta que una absorbancia a 260 nm de 1.0 equivale a una solución de 40 μg/ml de ARN. Para analizar la contaminación de las muestras con proteínas se determinó su absorbancia en 280 nm. Se consideraron muestras de ARN libre de proteínas a aquellas que presentaron una relación de A260/A280 cercana a 2. 12.3. Síntesis del ADN copia Se realizó la transcripción reversa a ADNc por acción de la enzima Transcriptasa Reversa del virus de la leucemia murina de Moloney (MMLV-RT). Rápidamente, 2 µg de ARN y 1 μl de oligo dT18 0.25 μg/μl (volumen final de 14.2 μl) fueron incubados a 70 °C durante 10 minutos en un ciclador térmico. Este es el paso de desnaturalización de los oligómeros y del ARN. Posteriormente, las muestras son puestas en hielo durante 5 minutos y luego se le agregan 5.8 µl de una solución que contiene 0.8 µl de enzima RT (200U/μl), 4 μl del buffer 5x de la enzima RT y 1 μl 10 mM dNTPs. Las muestras se colocan en el ciclador térmico a 40º C durante 90 minutos y luego el programa incuba 15 minutos a 70º C para detener la reacción por inactivación de la enzima. 33 Materiales y Métodos 13. Métodos generales de clonado y transformación de E. coli 13.1. Amplificación de fragmentos de ADN mediante la técnica de reacción en cadena de la polimerasa (PCR) Las reacciones de amplificación por PCR se realizaron en un volumen final de 20 μl conteniendo como templado ADNc 40-50 ng, 1.25 μM de cada cebador, MgCl2 1.25 mM, dNTPs 0,2 mM, buffer 1X para la enzima Taq y 1U de enzima Taq polimerasa. Las reacciones se llevaron a cabo en un termociclador utilizando un ciclo de desnaturalización de 5 minutos a 95 ºC y luego 30 ciclos de desnaturalización, apareamiento y elongación (1 minuto para cada etapa del ciclo). La temperatura de desnaturalización fue de 95 ºC, la de elongación de 72 ºC y para la temperatura de apareamiento se realizó un gradiente desde 46 °C hasta 56 °C. Este gradiente se realizó para determinar la temperatura óptima de apareamiento entre los cebadores y el ADNc. Por último, se realizó una elongación final de 5 minutos a 72 ºC. 13.2. Electroforesis en geles de agarosa Los geles de agarosa 1% (p/v) se prepararon en solución TBE 1X (Tris-borato 90 mM pH 8.3 y EDTA 2 mM) y se adicionó 0,5 μg/ml bromuro de etidio. Las muestras de ADN a sembrar se mezclaron con 1/5 volumen de 5X solución de siembra para ADN (glicerol 50 % (v/v), azul de bromofenol 1 % (p/v), SDS 0,1 % (p/v) y EDTA 40 mM pH 8.0). La separación electroforética se realizó en buffer TBE 1X empleando un voltaje constante de 10 V/cm lineal de gel. Los fragmentos de ADN se visualizaron en un transiluminador a 310 nm. 13.3. Purificación de fragmento de ADN a partir de geles de agarosa Los fragmentos de ADN se separaron en geles de agarosa y las bandas de interés identificadas mediante visualización sobre transiluminador UV se escindieron del gel. Dichos fragmentos se purificaron utilizando el kit comercial Wizard (Promega). 13.4. Reacciones de ligación de fragmentos de ADN Las reacciones de ligación intermolecular de fragmentos de ADN se realizaron en un volumen final de 10 μl utilizando 1 U de la enzima ADN-ligasa del bacteriófago T4 (Promega) y el buffer de reacción suministrado por el proveedor de la enzima. En 34 Materiales y Métodos cada reacción se utilizaron 50 ng de vector pGEMT-easy (Promega) y dos relaciones molares de inserto: vector, 1:1 y 1:3. 13.5. Transformación de células E. coli competentes con ADNc Cien μl de células DH5α (Invitrogen) se incubaron en presencia de 50-100 ng de los productos de las reacciones de ligación durante 1 hora a 4 ºC. Luego las células se sometieron a un shock térmico incubándolas durante 20 segundos a 42 ºC. A continuación se adicionó 0.95 ml de medio LB fresco (triptona 10 g/l, extracto de levadura 5 g/l y NaCl 10 g/l) y se incubó a 37 ºC durante 1 hora. Las células se colectaron por centrifugación a 4000 x g durante 1 minuto, se resuspendieron en 50 μl de medio LB y se sembraron con espátula Drigalski sobre placas de Petri conteniendo LB sólido suplementado con el antibiótico adecuado. Las placas se incubaron a 37 ºC durante 20 horas. 13.6. Mini-preparaciones de ADN La preparación de ADN plasmídico a partir de células de E. coli transformadas se realizó según el método de lisis alcalina descrito por Birnboim y Doly (1979) con algunas modificaciones. Las células transformadas con los plásmidos correspondientes se cultivaron en medio LB conteniendo ampicilina (100 μg/ml). Los cultivos se crecieron a 37 ºC con agitación hasta saturación. Para cada minipreparación de ADN plasmídico, 2 ml del cultivo se centrifugaron a 4000 x g durante 5 minutos y el sedimento se resuspendió en 100 μl de buffer I (Tris-HCl 50 mM pH 8.0, sacarosa 20% (p/v), EDTA 1 mM). Luego de incubar 5 minutos a temperatura ambiente se adicionaron 100 μl de buffer II (NaOH 0.2 M, SDS 1 % (p/v)) y se mezcló por inversión. Posteriormente, se adicionaron 100 μl de buffer III (CH3COONa 3M pH 4.5) y la mezcla se incubó nuevamente en hielo durante 5 minutos. Luego de centrifugar a 17000 x g durante 5 minutos a 4 ºC, se recuperó el sobrenadante. El ADN plasmídico se precipitó mediante el agregado 0,7 volúmenes de 2-propanol. La mezcla se incubó durante 30 minutos a temperatura ambiente, y posteriormente se centrifugó a 17000 x g a 4 ºC durante 4 minutos. El precipitado se lavó con 1 ml de etanol 70 % (v/v) y se secó a temperatura ambiente. El ADN plasmídico se resuspendió en 40 μl de agua destilada estéril. 35 Materiales y Métodos 13.7. Digestión del ADN con EcoR1 La digestión de ADN con EcoR1 se realizó de acuerdo a las condiciones indicadas por el proveedor. Se utilizó 1 U de enzima por cada μg de ADN plasmídico a digerir. La digestión se llevó a cabo durante 2-3 horas a 37 ºC en un volumen final de 20 μl. 13.8. Purificación del plásmido para su secuenciación La purificación del ADN plasmídico se realizó con el kit Illustra (GE Healthcare) según las instrucciones del proveedor. Se realizó para dos colonias en las que se había confirmado la presencia del inserto de interés. Posteriormente, se enviaron a secuenciar automáticamente en el secuenciador ABI310 (Applied Biosystems) usando cebadores universales M13. 14. Tratamientos de estrés 14.1. Tratamientos de estrés abiótico 14.1.1 Estrés salino Aproximadamente 300 semillas de trigo, provenientes de la misma partida, fueron sembradas en bandejas plásticas conteniendo vermiculita y regadas con agua corriente de red. Cinco días después se iniciaron los tratamientos que consistieron en regar las plántulas con agua o con una solución de NaCl 200 mM por 12 días. Pasado este período se evaluó en primera y segunda hoja: longitud, peso fresco, contenido de clorofila y de prolina. El contenido de clorofila se determinó con el medidor Minolta Spad 502, que determina la cantidad relativa de clorofila presente mediante la medición de la absorción de la hoja en dos regiones de longitud de onda (rojo e infrarrojo cercano) y calcula el valor numérico SPAD proporcional a la cantidad de clorofila presente en la hoja. La prolina se cuantificó según el método descripto por Bates (1973) con algunas modificaciones. Se realizó un macerado de 0.5 g de hojas con N2 (l) y luego se resuspendió en 5 ml de una solución acuosa de acido sulfosalicílico 3% (p/v). El material soluble se colectó por centrifugación a 2000 x g por 20 minutos. Un volumen de esta fracción se mezcló con volúmenes iguales de acido acético glacial y una solución de ninhidrina 2.5 % (p/v) en ácido acético y se incubó a 100 °C por 30 minutos. Posteriormente, se midió la absorbancia a 520 nm con un espectrofotómetro Ultrospec 1100 y el contenido de prolina se determinó a partir de una curva estándar de prolina (Sigma). 36 Materiales y Métodos 14.1.2. Estrés por altas temperaturas Plántulas de trigo de 11 días fueron sometidas a tratamientos a 40 °C durante 0, 1, 2, 3 y 4 horas. Las plántulas se procesaron a las 24 horas de iniciado este tratamiento luego de ser mantenidas en las mismas condiciones de luz y temperaturas que las descriptas en el apartado 1. 14.2. Estrés biótico: Ensayos de biocontrol de Trichoderma harzianum sobre el hongo patógeno Septoria tritici. Los ensayos de biocontrol de Trichoderma spp. sobre S. tritici fueron realizados en el campo experimental de la Facultad de Ciencias Agrarias y Forestales de La Plata, durante los meses de Julio a Noviembre de 2009 y 2010. El diseño experimental fue en bloques al azar con 14 tratamientos y 3 repeticiones: 2 aislamientos de T. harzianum (cc5 y c118), cada uno aplicado con la técnica de recubrimiento de semillas y como pulverizado de suspensión de esporas en dos momentos fenológicos del cultivo: macollaje y espigazón; estableciendo también dos controles: uno libre de inóculo y el otro inoculado sólo con S. tritici. Las plantas se inocularon con S. tritici en plántula, en macollaje y espigazón. En todos los casos las plantas inoculadas estuvieron sujetas a exposición bajo cámara húmeda por 48 horas. Para plántula, la cámara húmeda se practicó con bolsas de polietileno, mientras que para los dos estadios restantes se produjo una alta humedad relativa asperjando el follaje con agua cada dos horas. Además, en macollaje y espigazón, las plantas fueron asperjadas con una suspensión de ambas cepas de Trichoderma 24 horas antes que la inoculación del patógeno. Las plantas control estuvieron sujetas a exposición bajo cámara húmeda durante 48 horas o fueron asperjadas solamente con agua según el estadio. A los 21 días de la inoculación con S. tritici para cada estadio, se evaluaron parámetros de severidad de la enfermedad (porcentaje de necrosis y de cobertura picnidial en hoja). En el estadio de macollaje se evaluó la primera hoja de cada macollo y en espigazón la hoja bandera. En los ensayos realizados durante 2010 algunas plantas fueron tratadas con el fungicida Amistar (Syngenta; 0.35 % v/v) previo a la inoculación con S. tritici. 37 Capítulo I ¨Caracterización estructural y funcional de IPG¨ Introducción Capítulo I En nuestro grupo de trabajo se aisló un inhibidor de serina proteasas del FI de hoja de trigo. Este inhibidor regula la actividad proteolítica extracelular involucrada en los mecanismos de defensa del trigo frente al ataque del hongo fitopatógeno Septoria tritici (Segarra et al., 2002). Para caracterizar al inhibidor de serina proteasas, se determinó la secuencia del extremo N-terminal (Segarra et al., 2003), que resultó ser idéntica a la de uno de los tres oligopéptidos (box A) altamente conservados de las germinas y GLPs (Bernier and Berna, 2001). Debido a esto, se lo denominó Inhibidor de Proteasas tipo Germina (IPG). IPG, al igual que otras GLPs descriptas, es una glicoproteína que en su forma oligómerica (66-69 kDa) presenta también actividad superóxido dismutasa (SOD) (Segarra et al., 2003). Se han asociado diversas funciones enzimáticas a las germinas y GLPs, como oxalato oxidasa, superóxido dismutasa y adenosina glucosa pirofosfatasa/ fosfodiesterasa. Sin embargo, IPG es la primera GLP que presenta actividad de inhibidor de serina proteasas. Por ello, uno de los objetivos de la presente tesis fue determinar la secuencia aminoacídica completa y la de su ADNc, para determinar si se trata de un nuevo miembro de las GLPs o si es una proteína ya descripta, a la que no se le había detectado dicha actividad inhibitoria. Además se propuso determinar su sitio activo. 38 Resultados Resultados Capítulo I 1. Digestión Química y Enzimática de IPG Con el objetivo de determinar la estructura primaria de IPG, se procedió a la digestión química y enzimática de la proteína, ya que según la cantidad y el tamaño de los péptidos resultantes se puede obtener información valiosa acerca de la composición aminoacídica de la proteína. Se seleccionaron el bromuro de cianógeno (BrCN) y la hidroxilamina (NH2OH) para la digestión química y la tripsina para la digestión enzimática. En el presente trabajo, algunos de los péptidos obtenidos se analizaron por espectrometría de masas para determinar secuencias parciales de los mismos, ya sea por comparación de espectros en las bases de datos o por análisis de masas en tándem. 1.1. Digestión de IPG con Bromuro de Cianógeno El monómero de IPG (masa molar aproximada de 21 kDa) fue fragmentado con bromuro de cianógeno (BrCN) en gel (Cordoba et al., 1997). La elección de este reactivo se basó en que la reacción es muy específica, con pocas reacciones secundarias y adaptables a pequeña o gran escala. El BrCN rompe el enlace carboxilo terminal de los residuos de metionina y dado que este es uno de los aminoácidos menos abundantes en una proteína, se genera un número pequeño de péptidos de tamaños hasta 10 – 20 kDa. Los péptidos resultantes de la digestión química de IPG, fueron separados mediante SDS-PAGE 16% (Figura 3). Para extraer los fragmentos obtenidos por digestión química del gel, dicha porción fue incubada en buffer de muestra durante toda la noche. Dado que la eficiencia de la extracción no fue total, se sembró tanto el buffer de muestra como la propia pieza de gel. 39 Resultados Capítulo I Figura 3. Análisis mediante SDS-PAGE 16 % (Schagger and von Jagow, 1987) de los fragmentos de IPG obtenidos por digestión con BrCN. Calles 1 y 2: pieza de gel tratada con BrCN. Calle 3: Buffer de muestra en el que fue incubada la pieza de gel. Calle 4: Marcadores de masa molecular. Las letras a-g se corresponden con los productos de digestión. En total se obtuvieron siete bandas cuyas masas moleculares aproximadas fueron: a, 28 kDa; b, 24 kDa; c, 17.4 kDa; d, 14 kDa; e, 10.9 kDa; f, 8.4 kDa y g, 4.7 kDa. Dado que la masa molecular de la banda a supera a la del monómero de IPG, la misma podría deberse a la formación de un agregado. Las masas moleculares de las demás bandas evidencian la ocurrencia de una digestión parcial, ya que la sumatoria de las mismas excede en masa a IPG. En las calles donde se sembraron las piezas de gel (Figura 3, calles 1 y 2) solo se vieron dos bandas que corresponderían a las bandas f y g. Todas las bandas, salvo la a, fueron cortadas del gel y analizadas por espectrometría de masas. Esta técnica está basada en la ionización de péptidos y en su separación de acuerdo a la relación masa/carga. Los péptidos que se ionicen deben ser de masas entre 800 y 4000 Da, para que sean detectados por el equipo. Para ello, se suele realizar una digestión de los péptidos o proteína de interés con tripsina o quimotripsina. Para generar una concentración alta en la fase gaseosa de los péptidos ionizados se han desarrollado dos métodos: espectrometría de desorción-ionización en matriz inducida por láser (MALDI) e ionización por electrospray (ESI). En MALDI la muestra se co-precipita con un compuesto orgánico que absorbe la luz del láser y transmite la energía a la muestra para su ionización. En ESI, las disoluciones que 40 Resultados Capítulo I contienen la proteína o péptidos fluyen a través de una fina punta metálica mantenida a un potencial eléctrico y así se vaporizan en finas gotas (spray). Normalmente, los iones que se generan con esta ionización están multicargados (+2, +3) mientras que en MALDI los iones poseen carga +1. Después de haber generado los iones en fase gaseosa, se evalúa la relación carga/masa. El análisis más usado es el de tiempo de vuelo (time of flight, TOF), en donde los iones se aceleran en un campo eléctrico hacia un detector. Los iones más ligeros experimentan una mayor aceleración y por lo tanto llegan primero al detector. Así se generan espectros de masas (MS), donde el conjunto de picos masa/carga generan una huella peptídica que permite identificar a una proteína por comparación con los espectros de las bases de datos. Se puede profundizar el análisis realizando una espectrometría de masa en tándem (MS/MS). En ese caso, los péptidos ionizados son seleccionados para ser fragmentados por colisión con partículas gaseosas (CID), disociación por captura electrónica (ECD) o disociación por transferencia electrónica (ETD) y así generar la disrupción de las uniones peptídicas. Dependiendo del enlace que se rompa y del extremo en el que queda la carga (N o Cterminal) se generan dos series de iones: a, b y c ó x, y ó z. Las masas son medidas por el detector y se genera un espectro MS/MS. Nuevamente, se puede comparar este espectro con los de las bases de datos o se puede obtener la secuencia de novo del péptido. Dicha secuenciación de novo se logra identificando los iones de una misma serie y de la diferencia de las masas de los picos se deduce la masa del aminoácido correspondiente. En la presente tesis se utilizaron ambas técnicas de ionización (MALDI y ESI) para el análisis de IPG por espectrometría de masas. En la próxima sección se describirá el estudio de IPG a partir de geles bidimensionales en el cual se utilizaron las técnicas de MALDI-TOF y TOF-TOF. En cambio, el estudio de los péptidos obtenidos por digestión con BrCN (Figura 3) se realizó la ionización por electrospray (ESI) acoplada a cromatografía líquida para la separación de los péptidos (LC-MS/MS). Esta espectrometría de masas en tándem (MS/MS) se llevo a cabo en el servicio externo de la Universidad de Alicante (España). Los datos se analizaron con los programas: Mascot (MS/MS ión search, Matrix Science Ltd.), Phenix (GeneBio), OMMSA (Open Mass Spectrometry Search Algorithm) y Peaks (de novo sequencing, Bioinormatics Solutions Inc.). Con todos ellos se obtuvo el mismo resultado, identificándose un péptido proveniente de la banda b cuya secuencia fue: VTFLDDAQVK (Tabla 1). 41 Resultados Capítulo I Tabla 1. Análisis por espectrometría de masa en tándem de la banda b del SDS-PAGE de la figura 1 con el programa MASCOT MS/MS. Dicha secuencia fue procesada en BLAST (Altschul et al., 1990) y resultó ser idéntica a la de una proteína GLP con actividad Adenosina Glucosa Pirofosfatasa / Fosfodiesterasa de trigo (AGPPasa, CAC85479). Se realizó la digestión teórica con BrCN de esta proteína, utilizando el programa PeptideCutter (Expasy), para determinar si las masas teóricas coincidían con las de los péptidos encontrados experimentalmente (Tabla 2) Tabla 2. Digestión teórica con BrCN de la proteína AGPPasa perteneciente a las GLPs. La proteína AGPPasa (CAC85479) fue digerida con BrCN utilizando el programa PeptideCutter. A pesar de que los valores teóricos obtenidos no coinciden exactamente con los hallados experimentalmente, nos permiten proponer el siguiente esquema de organización de IPG (Figura 4). Figura 4. Esquema propuesto de la organización de IPG. Los valores experimentales de los péptidos obtenidos por la digestión de IPG con BrCN se muestran arriba de las flechas. Se marcan los dos residuos de metionina de la proteína (Met) y el sitio de glicosilación (G). NH2 y COOH representan los extremos amino y carboxilo terminal, respectivamente. 42 Resultados Capítulo I Cabe mencionar que los programas de digestión de proteínas no toman en cuenta las modificaciones postraducción como la glicosilación. Por lo tanto, el péptido de 10.9 kDa probablemente sea el correspondiente al extremo N-terminal y que su masa sea mayor a la teórica de 7.4 kDa debido a que se encuentra glicosilado. La masa experimental del péptido interno es muy cercana a la teórica (4.7 vs 4.8 respectivamente). Finalmente, el péptido del extremo carboxilo terminal difiere en 1.2 kDa con el teórico (8.4 kDa encontrado experimentalmente y 7.2 kDa el calculado teóricamente). El esquema propuesto en la Figura 4 permite también justificar las masas obtenidas como producto de la digestión parcial de IPG. Es decir, la suma de las masas halladas experimentalmente para el primer y segundo péptido es de 15.6 kDa, valor muy cercano al encontrado experimentalmente de 17.4 kDa. Además, la suma del segundo y tercer péptido es de 13.1 kDa, valor que se aproxima al experimental de 14 kDa. En conclusión, en la digestión química de IPG con BrCN se obtuvieron péptidos de tamaños similares a los que se obtendrían teóricamente para la proteína GLP con actividad AGPPasa (Rodríguez-López et al., 2001). 1.2. Digestión de IPG con tripsina La tripsina es una endoproteinasa miembro de la familia de las serina proteasas, con alta especificidad por los enlaces peptídicos del extremo carboxilo de los aminoácidos arginina y lisina. Si bien IPG es capaz de inhibir a la tripsina, en este ensayo se utilizó una concentración de tripsina diez veces superior a la utilizada en las experiencias de inhibición (Segarra et al., 2003), según las condiciones descriptas por Rosenfeld et al. (1992). Para analizar el patrón de los péptidos resultantes de la ruptura enzimática, los mismos fueron analizados por SDS-PAGE 16% - urea (Schagger, 2006), que permite una mejor resolución de los mismos (Figura 5). 43 Resultados Capítulo I Figura 5. Análisis mediante SDS-PAGE 16 % - urea de los fragmentos de IPG obtenidos por digestión con tripsina. Como producto de la digestión de IPG con tripsina se visualizaron nueve bandas cuyas masas moleculares son aproximadamente: 23.1; 22.4; 15.3; 13.5; 11.6; 9.4; 6.8; 4.2 y 3.1 kDa. Ya que la sumatoria de las masas obtenidas supera a la masa de IPG, se podría afirmar que ocurrió una digestión parcial. Debido a que la digestión con BrCN de IPG y posterior análisis por MS/MS arrojó como resultado una secuencia peptídica perteneciente a la GLP con actividad AGPPasa, se realizó la digestión teórica de esta proteína con tripsina utilizando el programa PeptideCutter. El objetivo fue determinar si estos productos de digestión de la AGPPasa se corresponden con los obtenidos experimentalmente para IPG. El resultado se muestra en la Tabla 3. Tabla 3. Digestión teórica de la proteína AGPPasa perteneciente a las GLPs. La proteína AGPPasa (CAC85479) fue digerida con tripsina utilizando el programa PeptideCutter. 44 Resultados Capítulo I Los sitios teóricos de corte de la tripsina para la GLP AGPPasa son nueve, originándose productos menores a 5.6 kDa. Dado que algunos residuos de arginina o lisina están muy próximos, es probable que en algunos casos los mismos sean omitidos, lo que justificaría la digestión parcial obtenida experimentalmente. En consecuencia, dada esta digestión parcial y el gran número de péptidos obtenidos, es prácticamente imposible determinar una correspondencia entre los datos experimentales y los productos teóricos de la digestión de la AGPPasa. 1.3. Digestión de IPG con hidroxilamina La reacción con hidroxilamina (NH2OH) ha sido utilizada para digerir ADN y desacetilar proteínas a pH neutro (Golderer and Gröbner, 1991). Sin embargo, a pH alcalino, la hidroxilamina puede usarse para cortar el enlace entre asparragina-glicina (Asn-Gly o N-G). Con este tipo de digestión química generalmente se obtienen péptidos grandes, ya que esta dupla de aminoácidos es poco común de encontrar (Bornstein and Balian, 1977). Por ello, se realizó la digestión de IPG con este reactivo químico y los productos de digestión obtenidos fueron separados en SDS-PAGE 16% - urea para lograr una buena separación de los mismos. El resultado de la digestión se muestra en la Figura 6. Figura 6. Análisis mediante SDS-PAGE 16 % - urea de los fragmentos de IPG obtenidos por digestión con hidroxilamina. 45 Resultados Capítulo I Se obtuvieron tres productos de digestión cuyas masas moleculares aproximadas fueron: a, 7.6 kDa; b, 5.2 kDa y c, 3.8 kDa. También se observaron bandas de mayor masa molecular que corresponden a la digestión parcial de IPG. Debido a que la digestión con BrCN de IPG y posterior análisis por MS/MS arrojó como resultado una secuencia peptídica perteneciente a la GLP con actividad AGPPasa, se realizó la digestión teórica con hidroxilamina de dicha proteína utilizando el programa PeptideCutter (Tabla 4). Se encontró que la cantidad y la masa de los péptidos teóricos se corresponden con los obtenidos experimentalmente para IPG. Tabla 4. Digestión teórica con hidroxilamina de la proteína AGPPasa perteneciente a las GLPs. La proteína AGPPasa (CAC85479) fue digerida con hidroxilamina utilizando el programa PeptideCutter. 46 Resultados Capítulo I 2. Análisis de IPG a través de geles bidimensionales Las digestiones químicas y enzimáticas descriptas en la sección anterior fueron realizadas en gel a partir de la banda de poliacrilamida que contenía IPG, aislada de un SDS-PAGE monodimensional. Dicho abordaje experimental tiene la desventaja de que en la porción de gel analizada podría haber más de una proteína con masa molecular similar a IPG. Debido a ello, se realizó un gel bidimensional que, además de separar las proteínas por masa molecular, también las separa por punto isoeléctrico (pI), por lo que es poco probable encontrar en un spot más de una proteína. Para ello, una alícuota del FI calentado a 70 °C (FI70) se sembró en un gel 2D, pudiendo detectarse 6 diferentes spots: dos de 18 kDa, pI 5.5 y 6.2; tres de 20 kDa, pI 5.1, 5.7 y 6.2 y uno de 21 kDa, pI 6.2 (Figura 7). Figura 7. FI70 analizado mediante 2D SDS-PAGE, teñido con Coomassie blue coloidal. Debido a que los seis spots tienen la masa molecular aproximada del monómero de IPG (18 a 21 kDa; Segarra et al., 2003) todos ellos fueron analizados por MALDITOF en el servicio del Centro de Estudios Químicos y Biológicos por Espectrometría de Masas (CEQUIBIEM, UBA). Como se mencionó anteriormente en la descripción general de la técnica de espectrometría de masas, los spots proteicos son digeridos con tripsina para obtener masas que puedan ser detectadas por el equipo. Del análisis de las masas correspondientes a los fragmentos trípticos, se observó que existen al menos 4 comunes 47 Resultados Capítulo I a los 6 spots (Figura 8). Por análisis de huella peptídica en las bases de datos no se pudo asignar identidad a ningún spot. Por ello, se analizó el fragmento de 2201 Da por MS/MS, debido a que resultó el más intenso y es común a los espectros de los 6 spots. El resultado obtenido en el programa MASCOT permitió identificar un péptido interno de 23 aminoácidos (AAVTPAFVGQFPGVNGLGISAAR) que también se encuentra en la proteína AGPPasa (Tabla 5) Figura 8. Espectro MALDI-TOF del spot 2 del gel bidimensional. Los asteriscos marcan los picos m/z comunes a los seis spots analizados del gel 2D. Tabla 5. Análisis por espectrometría de masa en tándem del pico m/z 2201 con el programa MASCOT MS/MS. Estos resultados indicarían que los 6 spots son isoformas de IPG. En el caso de los spots de igual masa y distinto pI, esta diferencia podría deberse a la modificación de algunos de sus aminoácidos. En el caso de los spots del mismo pI pero distinta masa molecular, esto podría deberse a un diferente grado de glicosilación. Se analizó la digestión tríptica teórica de la proteína AGPPasa (Tabla 3) para determinar por qué con los datos de las masas trípticas de los spots del gel 48 Resultados Capítulo I bidimensional no se pudo identificar a la proteína. En la digestión teórica obtenemos 2 péptidos de m/z > 4000 Da (4446 y 5594 kDa) que no encontramos en los datos experimentales debido a las limitaciones de la técnica (m/z altos no son detectados por el equipo). Existe también otro péptido teórico de m/z 2397.1833 que no se encuentra en los espectros lo que puede deberse a que dicho péptido esté glicosilado (la secuencia AGVSAGDFYYHGLAAAGNTSNLIK presenta el sitio de glicosilación NTS) Cabe mencionar que el programa Mascot utilizado para analizar los datos no toma en cuenta este tipo de modificación postraduccional. Por otra parte se realizó un gel bidimensional copolimerizado con gelatina en la segunda dimensión para identificar el/los spot responsables de la actividad inhibitoria de IPG (Figura 9). Figura 9. Análisis del FI70 mediante 2D SDS-PAGE, teñido con Coomassie blue coloidal. (A) y (B): con y sin gelatina copolimerizada respectivamente. Se detectó actividad inhibitoria de la proteólisis en uno de los spots de pI 6.2. Dada la masa molecular del mismo se podría suponer que se trata del spot 2 detectado por la tinción con Coomassie coloidal en el gel B (Figura 9B). En los demás spots no se visualizó actividad inhibitoria y esto podría deberse tanto a particularidades de las isoformas como a un problema cuantitativo, dado que por la cantidad de proteína que se siembra en este tipo de geles, la misma puede resultar insuficiente en los spots minoritarios. 49 Resultados Capítulo I 3. Identificación de la secuencia del ADNc de IPG Debido a que con la espectrometría de masas solo se obtuvo la secuencia de dos péptidos de IPG, se propuso obtener la secuencia completa a partir del ADNc. Para ello se diseñaron los respectivos cebadores para la Reacción en Cadena de la Polimerasa (PCR). Una de las secuencias utilizadas fue la correspondiente a la región N-terminal de IPG (ltqdfcvadl), obtenida previamente por secuenciación de Edman (Segarra et al., 2003) y altamente conservada en las GLPs. La otra secuencia utilizada, cercana al Cterminal y obtenida por espectrometría de masas (vtflddaqvk), en cambio, no se encuentra en ninguna de las tres regiones conservadas en las GLPs (boxes A, B o C; Bernier y Berna, 2001). Para obtener la secuencia de nucleótidos, se realizó la traducción reversa utilizando el código de codones más frecuentes en trigo (programa SMS Reverse Translate). A partir de la extracción de ARN de la primera hoja de trigo, se realizó la síntesis de ADNc a través de la reacción mediada por la enzima Transcriptasa Reversa seguida por PCR (RT-PCR), para amplificar el mensajero de IPG. Las condiciones de amplificación por PCR fueron: 30 ciclos con temperatura de apareamiento en gradiente desde 46 a 56 ºC. La utilización del gradiente de temperatura fue para determinar la temperatura óptima de apareamiento entre los cebadores y el ADNc (Figura 10) Figura 10. Electroforesis en gel de agarosa de los productos de amplificación por PCR de IPG utilizando diferentes temperaturas de apareamiento. Calles 1 a 6: gradiente de temperatura de apareamiento: 46.4 °C, 48.4 °C, 50.3 °C, 52.3 °C, 54.°C y 56 °C. Calle 7: escala de ADN 1kb. Se obtuvo un producto de 540 pb que se corresponde con el tamaño esperado y la temperatura óptima de apareamiento entre los cebadores y el cADN es de 56 °C 50 Resultados Capítulo I (Figura 10, calle 6), por lo que esas condiciones fueron las usadas en los ensayos posteriores. Con el objetivo de determinar la secuencia del producto amplificado, se cortó la banda de 540 pb del gel de agarosa y se purificó. El producto purificado de 540 pb se ligó al vector pGEM-T Easy y se transformaron bacterias competentes de E. coli DH5α. Se obtuvieron 62 colonias en total y para confirmar la presencia del inserto de interés, se realizaron mini preparaciones de ADN plasmídico de 6 colonias elegidas al azar tratadas con la enzima de restricción EcoR1. Dicha enzima de restricción posee dos sitios de corte en el plásmido, que flanquean el inserto. Dos de las colonias analizadas contenían el inserto de 540 pb, por lo que los plásmidos de dichas colonias se purificaron y se enviaron a secuenciar a un servicio externo. La secuencia de ADN obtenida para ambos clones fue la misma y se tradujo tomando en cuenta los 6 posibles marcos de lectura, siendo la secuencia aminoacídica del marco de lectura 1 la posteriormente analizada en BLASTP (Figura 11). Figura 11. Secuencia parcial obtenida a partir del ADNc de IPG. La secuencia peptídica de IPG deducida se muestra debajo de la secuencia de ADNc. Las flechas indican la secuencia de los cebadores utilizados para la reacción RT-PCR y su orientación muestra el sentido de los mismos (Fw y Rv). En negrita se señala la secuencia de los boxes conservados de las GLPs (Bernier y Berna, 2001). 51 Resultados Capítulo I La secuencia nucleotídica analizada en BLASTn (Basic Local Alignment Search Tool nucleotide) muestra un 99% de identidad con el ARN mensajero de la GLP AGPPasa (N° de acceso GenBank AJ319659.1, Figura 12). Además, la secuencia aminoacídica deducida y procesada en BLASTp (Basic Local Alignment Search Tool protein) arroja el mismo resultado (N° de acceso GenBank CAC85479.1, Figura 13). Esto confirma los resultados obtenidos anteriormente de espectrometría de masas. Figura 12. Alineamiento entre la secuencia de ADNc obtenida para IPG y el ARNm de la GLP AGPPasa de trigo (emb AJ319659.1). Los asteriscos (*) indican nucleótidos idénticos, en negrita se señalan los nucleótidos diferentes entre ambas secuencias y los guiones indican la diferente longitud de las mismas. 52 Resultados Capítulo I Figura 13. Alineamiento entre la secuencia parcial de aminoácidos deducidos para IPG y la secuencia completa de la AGPPasa de trigo (emb CAC85479.1). Los asteriscos (*) indican aminoacidos idénticos y en negrita se señala el aminoácido diferente entre ambas secuencias. Ambas secuencias solo difieren en dos nucleótidos, lo que lleva a la modificación de un solo aminoácido en la proteína madura (Ser por Gly). Esto podría deberse a un error en la secuenciación, sin embargo se encontró en la base de datos de trigo KOMUGI (Integrated Wheat Science Database) un ADNc 100% idéntico (CJ704957), por lo que descartamos esa posibilidad. 53 Resultados Capítulo I 4. Actividad AGPPasa de IPG Debido a que la secuencia de IPG resultó ser idéntica en un 99% a la AGPPasa descripta por Rodríguez-López et al. (2001), se analizó si IPG poseía esta actividad enzimática. Para ello, se realizó un ensayo de actividad pirofosfatasa en gel con las proteínas provenientes del FI de trigo. Se detectó una banda de actividad con una movilidad relativa similar a la del oligómero de IPG (~66 kDa). Para confirmar su identidad, esta banda de actividad fue eluída en buffer de muestra y hervida tres minutos para obtener la forma monomérica de IPG siendo luego analizada por western blot. El anticuerpo específico contra el monómero de IPG reconoció la banda proveniente de actividad fosfodiesterasa (Figura 14B) Figura 14. Actividad fosfodiesterasa de IPG. (A) Una alícuota de FI fue separada en un SDS-PAGE en condiciones no reductoras y se analizó su actividad in gel. La actividad enzimática fue detectada por la incubación del gel con 5 mM bis(4-nitrofenil) fosfato y 5 mM MgCl2 hasta la aparición de un producto amarillo (4-nitrofenol) (B). Western blot de la banda identificada en (A), luego de su elución en buffer de muestra conteniendo 2-mercaptoetanol y calentada a 100 °C. Por lo tanto IPG podría considerarse como una proteína multifuncional con al menos tres actividades enzimáticas: inhibidor de serina proteasas, superóxido dismutasa y fosfodiesterasa. 54 Resultados Capítulo I 5. Determinación de la mínima secuencia de IPG con actividad inhibitoria Aunque menos que el oligómero de 66 kDa, el monómero de IPG de 18-21 kDa conserva actividad inhibitoria (Segarra et al., 2003). Para avanzar en el conocimiento del sitio activo responsable de dicha actividad inhibitoria, los péptidos resultantes de la ruptura con BrCN, tripsina e hidroxilamina fueron analizados mediante SDS-PAGE 16% copolimerizado con gelatina, para determinar si alguno de esos fragmentos conservaba la actividad inhibitoria de la proteólisis (El-Shamei et al., 1996). En el caso de la digestión de IPG con BrCN, sólo se detectó actividad en el fragmento correspondiente a IPG sin digerir (Figura 15). Esto permite suponer que el sitio activo de IPG podría encontrarse situado alrededor de alguna de las dos metioninas que posee la proteína o que debido a la escasa masa obtenida de algunos de los péptidos no sea posible detectar la actividad en este tipo de zimogramas. Figura 15: Análisis de los fragmentos de IPG obtenidos por digestión con BrCN mediante SDS-PAGE 16% teñido con Coomassie coloidal. A, SDS-PAGE teñido con Coomassie coloidal; B, SDS-PAGE copolimerizado con gelatina seguido por digestión con tripsina y teñido con Coomassie coloidal. Para la digestión de IPG con tripsina, se obtuvieron dos péptidos que mantienen la actividad inhibitoria (Figura 16 B). Sin embargo, dada la gran cantidad de péptidos obtenidos y la digestión parcial que ocurre (Figuras 5 y 16A), no se pudo asignar identidad a ninguno de los dos. 55 Resultados Capítulo I Figura 16: Análisis de los fragmentos de IPG obtenidos por digestión con tripsina mediante SDS-PAGE 16% teñido con Coomassie coloidal. (A) y (B) sin y con gelatina copolimerizada respectivamente. A: calle 1, Marcadores de masa molecular; calle 2, FI70 hervido con buffer de muestra; calle 3, pieza de gel tratada con tripsina; calle 4, Buffer de muestra en el que fue incubada la pieza de gel. B: Calle 1, FI70 hervido con el buffer de muestra; calle 2, pieza de gel tratada con tripsina; calle 3 y 4, Buffer de muestra en el que fue incubada la pieza de gel. C: las calles 3 y 4 del gel B (circulo blanco) fueron teñidas con plata. Este tipo de análisis se repitió en geles 16%-urea-gelatina para lograr una mayor separación de los péptidos y poder asignarle identidad, pero no se obtuvieron resultados positivos. Se tuvieron problemas similares en el análisis de actividad de los péptidos obtenidos por digestión con hidroxilamina, en donde seguramente el exceso de hidroxilamina interfería en la corrida de los geles. Como estrategia alternativa se planteó eluir del gel de poliacrilamida los péptidos obtenidos de la digestión con hidroxilamina y medir la actividad de inhibidor de proteasas por el método de la fluorescamina (Udenfriend et al., 1972) ya que esta técnica es de mayor sensibilidad. Para la elución de los péptidos del gel se propuso utilizar la técnica de tinción negativa de geles con Zinc-Imidazol (Fernandez-Patrón et al., 1992). Dicha técnica es altamente sensible y reversible, y permite la elución eficiente de la proteína o péptido de interés conservando su actividad biológica. Para ello, en primer lugar se analizó si la banda correspondiente a IPG, eluída de un gel de poliacrilamida mediante esta técnica, conserva su actividad inhibitoria. Aproximadamente 30 µg de proteínas provenientes del FI70 fueron separados en un SDS-PAGE 12% y teñidos con Zinc-imidazol. La banda correspondiente a IPG fue cortada, eluída y analizada nuevamente en SDS-PAGE 12% para evaluar el rendimiento. Se encontró que el rendimiento de la elución fue del 85% (Figura 17) y la medición de actividad inhibitoria por el método de la fluorescamina arrojó resultados similares a los reportados por Segarra et al. (2003) para el monómero de IPG. 56 Resultados Capítulo I Figura 17: Análisis del rendimiento de elución de IPG por el método de Zinc-imidazol. SDS-PAGE 12% teñido con Zn-Imidazol (A) y Coomassie blue R250 (B). La banda correspondiente a IPG señalada con una flecha fue cortada, eluida y analizada en otro gel (B) para evaluar el rendimiento de la elución. Al constatar que la técnica es eficiente y que no provoca la pérdida de actividad de IPG, se prosiguió a analizar los péptidos que se obtienen por digestión con hidroxilamina y teñirlos por este método. Para ello, la pieza de gel teñida por Zn-Im que contenía a IPG fue incubada con hidroxilamina y luego se eluyeron de dicha pieza los productos obtenidos. Posteriormente se separaron en un gel 16%-urea y se tiñeron nuevamente con Zn-Im. Probablemente por interferencias entre los distintos reactivos utilizados, no se pudo visualizar ninguno de los productos de la digestión. Se realizaron distintos controles para descartar que esto se debiera a problemas por la doble tinción con Zn-Im y/o a interferencias con la urea del gel. Sin embargo no se pudo determinar a que se debieron estos resultados negativos. 57 Resultados Capítulo I 6. Identificación de aminoácidos esenciales de IPG IPG es la primera GLP a la que se le asigna actividad de inhibidor de proteasas. Dado que su secuencia no presenta identidad con ninguna de las familias de inhibidores de proteasas descriptos, el conocimiento del sitio activo resulta importante para la caracterización de esta novel actividad. Debido a que con las metodologías de ruptura enzimática y química no se pudo determinar la secuencia mínima necesaria para mantener la actividad inhibitoria, se realizaron modificaciones químicas de distintos aminoácidos para determinar el sitio activo de IPG. Este tipo de estrategia ha sido ampliamente utilizada para determinar sitios activos de proteínas (Means and Feeney, 1990; Vallejos, 1981). 6.1. Modificación de histidinas El grupo imidazol de las histidinas puede ser modificado por la alquilación del nitrógeno. Uno de los reactivos utilizados para dicho proceso es el dietilpirocarbonato (DEPC) según la siguiente reacción: Figura18. Reacción de alquilación de la histidina de una proteína (P) por DEPC En las GLPs, las histidinas son aminoácidos muy importantes involucrados junto con el glutamato en la unión al manganeso y en la actividad SOD (Woo et al., 2000). Por ello se modificó químicamente este aminoácido para así determinar si el sitio activo del inhibidor coincide con el de la actividad SOD. Para determinar si la modificación química de las histidinas produce un cambio en la actividad de IPG se realizó un gel de actividad inhibitoria (Figura 19B). En paralelo se corrió un SDS-PAGE teñido con Coomassie Blue para comprobar que IPG muestra el mismo tamaño molecular luego del tratamiento químico (Figura 19A). Como se observa en la Figura 19, el tratamiento de IPG con DEPC no produjo modificaciones en la actividad inhibitoria, por lo que podemos concluir que el sitio activo de la actividad inhibitoria no coincide con el de la actividad SOD. 58 Resultados Capítulo I Figura 19. Análisis de la actividad inhibitoria de IPG tratada con DEPC. A, SDS-PAGE teñido con Coomassie blue; B, SDS-PAGE copolimerizado con gelatina seguido por digestión con tripsina. IPG fue preincubado con DEPC 10 mM en buffer fosfato pH 6.8 (calle 1) o solo en buffer fosfato pH 6.8 (calle 2) como control. 6.2. Modificación de arginina El aminoácido arginina es difícil de modificar con la mayoría de los reactivos químicos debido a su extrema basicidad, requiriéndose para ello un pH tan alto que puede modificar la estructura de la proteína. Reactivos como 2,3-butanodiona o 1,2ciclohexanodiona reaccionan con el grupo guanidinio de las argininas para producir una estructura de dioles vecinales (Figura 20A). Otro reactivo utilizado para modificar específicamente al aminoácido arginina es la ninhidrina, según lo descripto por Chaplin (1976). El producto que se forma es el que se muestra en la Figura 20B, donde se puede observar como se bloquea el grupo imino de la arginina. A B Figura 20. Reacciones de modificación de la arginina de una proteína (P). A, con 2,3-butanodiona; B, con ninhidrina. 59 Resultados Capítulo I El tratamiento de IPG con ninhidrina produjo la pérdida total de actividad inhibitoria de IPG (Figura 21). Figura 21. Modificación de la actividad inhibitoria de IPG con ninhidrina. A, SDS-PAGE teñido con Coomassie blue como control de tamaño de IPG; B, SDS-PAGE copolimerizado con gelatina seguido por digestión con tripsina. IPG fue preincubado con ninhidrina 100 mM en buffer fosfato pH 9.1 (calle 1) o solo en buffer fosfato pH 9.1 (calle 2) como control. Para confirmar este resultado se realizó un nuevo tratamiento utilizando concentraciones crecientes de 2,3-butanodiona (Figura 22). Figura 22. Efecto de concentraciones crecientes de 2,3-butanodiona (0, 0.1, 0.5 y 1M) sobre la actividad inhibitoria de IPG. (A) IPG analizado en SDS-PAGE copolimerizado con gelatina y luego incubado con tripsina. (B) IPG analizado en SDS-PAGE como control de tamaño. Este nuevo experimento ratificó la importancia de la arginina ya que se evidenció una relación inversa entre la capacidad de inhibir proteasas de IPG y la concentración del reactivo. 60 Resultados Capítulo I 6.3. Modificación de cisteínas Una de las características más importante de la cisteína es su capacidad para estabilizar la estructura de las proteínas mediante la formación de puentes disulfuros. Los grupos sulfhidrilos de las cisteínas son probablemente los grupos más reactivos encontrados en las proteínas y debido a ello, los reactivos químicos que modifiquen este aminoácido son de fundamental interés. Uno de los reactivos que se utiliza para modificar la cisteína es la N-etil maleimida (NEM) que produce la alquilación de la cisteína tal como se muestra en la Figura 23 Figura 23. Reacción de modificación de la cisteína de una proteína (P) con N-etil maleimida. El NEM es un reactivo alquilante que reacciona con los sulfhidrilos para formar enlaces tioester estables (Smyth et al., 1960). Uno de los inconvenientes de esta reacción es su fuerte dependencia con el pH. Las reacciones de la maleimida son especificas para el grupo –SH en un rango de pH entre 6.5 y 7.5 (Smyth et al., 1964). A pH mas altos existen reacciones con grupos aminos libres (Brewer and Riehm, 1967). Debido a la fuerte dependencia con el pH de la especificidad de reacción del NEM con la cisteína, se realizó la modificación de IPG con este reactivo a pH 5, 6.8 y 9.1. El objetivo de dicho experimento fue poder diferenciar la reacción específica del NEM con la cisteína de la reacción inespecífica que se produce con aminoácidos básicos (Figura 24), y así determinar el efecto sobre la actividad de IPG. A pH 5, en donde no esta descripto que el NEM pueda modificar cisteínas u otros aminoácidos, la actividad de IPG se mantiene similar al control (Figura 24B, calle 1). Sin embargo, a pH 6.8, en donde la reacción del NEM es exclusiva con la cisteína, la actividad inhibitoria de IPG disminuye a la mitad (Figura 24B, calle 2). Esto indicaría que alguna de las 3 cisteínas de IPG (Cys 6, 12 y 21) son importantes para la actividad inhibitoria o que la modificación de este tipo de aminoácido provoca algún cambio conformacional en la proteína que hace que pierda dicha actividad. Esta última hipótesis se basa en la importancia de los puentes disulfuros para estabilizar la estructura terciaria 61 Resultados Capítulo I de las proteínas. A pH alcalino (pH 9.1) esta descripto que el NEM reacciona con aminoácidos básicos. Como se puede observar en la Figura 24B (calle 3), cuando la reacción se realizó a este pH la capacidad de IPG para inhibir tripsina desaparece totalmente. Con este resultado y el obtenido anteriormente de modificación de arginina con 2,3butanodiona, podríamos suponer que a este pH alcalino el NEM reaccionó inespecíficamente con la arginina y por ello la actividad inhibitoria de IPG desaparece totalmente. Figura 24. Modificación de la actividad inhibitoria de IPG con NEM a distintos pHs. A, SDS-PAGE teñido con Coomassie blue; B, SDS-PAGE copolimerizado con gelatina seguido por digestión con tripsina. IPG fue preincubado con NEM 100 mM en buffer fosfato pH 5 (calle 1), pH 6.8 (calle 2) y pH 9.1 (calle 3). Como control se muestra la preincubación de IPG en buffer fosfato pH 6.8 (calle 4), aunque se realizaron los controles a todos los pH utilizados. 62 Resultados Capítulo I 7. Modelo estructural de IPG Los resultados obtenidos con las modificaciones químicas de aminoácidos demostraron que la única arginina de IPG (Arg 69) es requerida para inhibir serina proteasas. Considerando que la arginina está involucrada en la asociación específica con tripsina (Laskowski and Qasim, 2000), se intentó determinar la localización espacial de este aminoácido. Debido a que se contaba con la secuencia aminoacídica completa de IPG y que no existen datos cristalográficos de dicha proteína, se planteó realizar una predicción de estructura tridimensional utilizando herramientas bioinformáticas. La predicción de la estructura tridimensional a partir de la secuencia primaria de la proteína, es una estrategia muy utilizada en los últimos años. Existen diferentes métodos: ab-initio u homología. Los métodos ab-initio se fundamentan enteramente en principios físico-químicos, y los de homología están basados en la información de secuencias y estructuras contenida en bases de datos. El modelado por homología, involucra la utilización de de una estructura como templado o molde que sirve para predecir la estructura de la secuencia problema. Los avances en la metodología de simulación y el conocimiento de las fuerzas que manejan el plegamiento de proteínas tienen un alto impacto en los métodos ab-initio, mientras que la rápida expansión de las bases de datos a partir de la obtención de nuevas secuencias y estructuras han incrementado la utilización del modelado por homología. Actualmente estas técnicas se interrelacionan, permitiendo la optimización de los modelos obtenidos (Al-Lazikani et al., 2001). Para la construcción del modelo tridimensional de IPG se utilizó el servidor Swiss-model workspace que genera un modelo por homología. Este programa produce el alineamiento de secuencia entre IPG y alguna proteína presente en las bases de datos de estructura 3D resuelta por cristalografía. Se considera que las parejas de proteínas que presentan una identidad de secuencia mayor al 30% tienen estructura tridimensional similar (Sander and Schneider, 1991), y por ello se pueden usar como templado. Para IPG, el mejor alineamiento que se obtuvo fue con la proteína perteneciente a la familia germina, con actividad oxalato oxidasa (Opaleye et al., 2006) y código PDB 2et7A (Figura 25). El porcentaje de identidad de secuencia es del 40%, y el e-value del modelo fue de 3.84041e-27. El modelo obtenido se muestra en la Figura 26. 63 Resultados Capítulo I Figura 25. Alineamiento de estructura primaria y secundaria entre IPG y 2et7A, utilizada como templado. En el alineamiento de estructura secundaria se señala la de tipo βlámina (s) y α-hélice (h). Figura 26. Modelo tridimensional de IPG. En el modelo se pueden observar los cuatro pares de laminas β que forman el barril tipo remolino o jellyroll, característico de los monómeros de germinas. También se señala la única arginina (Arg69) que se encuentra periféricamente situada y los aminoácidos involucrados en el sitio activo SOD (His81, His83, His128 y Glu88). 64 Resultados Capítulo I El modelo estructural obtenido fue validado por el gráfico de Ramachandran, el programa Verify3D y el cálculo de energía total. El gráfico de Ramachandran permite analizar la calidad del modelo evaluando los ángulos diedros psi y phi de cada uno de los aminoácidos (Figura 27). Figura 27. Mapa de Ramachandran para la estructura modelada de IPG. El gráfico fue realizado con el programa RAMPAGE (Lovell et al., 2003). Este gráfico indica si los residuos presentan ángulos diedros que sean estéricamente favorables para los diferentes elementos de la estructura secundaria (hélices α, láminas β, giros, etc.). Las zonas favorables, permitidas y no permitidas aparecen marcadas en la figura con colores diferentes y los aminoácidos que caen en dichas zonas están representados por puntos. El cuadrante superior izquierdo representa las láminas β, el cuadrante superior derecho e inferior izquierdo las hélices α que giran a la izquierda y derecha respectivamente. El modelo de IPG obtenido indica que el 93.4% de los residuos está favorecidos estéricamente, el 5.4% está en la región permitida y sólo dos residuos en la zona no permitida. Por otra parte, el programa Verify3D analiza la compatibilidad del modelo 3D con la secuencia de aminoácidos de IPG realizando comparaciones del entorno químico del 65 Resultados Capítulo I modelo y con estructuras cristalográficas en bases de datos. Para ello todos los aminoácidos, salvo los diez primeros y diez últimos, reciben una puntuación: valores negativos indican mala calidad del modelo, valores cercanos a cero indican zonas dudosas y valores positivos cercanos a 1, zonas de muy buena calidad. Para el modelo de IPG se obtuvieron todos valores positivos, tal como se observa en la Figura 28, con pocas zonas dudosas. Figura 28. Resultado del análisis del modelo de IPG con el programa Verify3D. En el eje de coordenadas x se marca la posición del aminoácido evaluado y en el eje y el puntaje asignado por el programa. Finalmente, el valor de energía total calculada para el modelo fue de -2822.932 KJ/mol y el valor negativo de energía indica que la estructura es estable. Por lo tanto, se podría afirmar que el modelo es de buena calidad y representa una buena aproximación a la conformación tridimensional de la proteína. A partir del modelo obtenido de IPG (Figura 26) se observa que la Arginina 69 es periférica, lo que permite explicar su rol en la interacción con el sustrato. Con respecto a la actividad SOD, el modelo predice que el glutamato (Glu88) y las tres histidinas (His81, His 83, His 128) responsables de la unión al manganeso, se sitúan en las laminas β antiparalelas vecinas y forman un conjunto de grupos imidazol tal como lo describieron Gane et al. (1998) para otra germina. 66 Resultados Capítulo I Debido a que el oligómero de IPG posee mayor actividad de inhibidor de proteasas que el monómero, se intentó determinar si en la estructura oligomérica la arginina también se encuentra expuesta. Como se mencionó en la introducción, se ha determinado por cristalografía que las germinas son homohexámeros ordenados como trímero de dímeros (Woo et al., 2000). En el caso de la germina utilizada como templado para la predicción de estructura de IPG, los autores de dicho trabajo (Opaleye et al., 2006) propusieron un modelo de interacción de monómeros. Utilizando un programa de visualización de estructuras (Pymol), se determinó la posición de la arginina análoga a la Arg69 de IPG en el modelo de interacción de monómeros propuesto por Opaleye. Figura 29: Modelo hexamérico de la germina con actividad OXO. En el modelo propuesto por Opaleye et al. (2006) se señalan con distintos colores los seis monómeros de la proteína. En color negro se señala la ubicación de la Arg76 de cada monómero (análoga a Arg69 de IPG). Como se puede observar en el modelo hexamérico (Figura 29), la arginina en posición análoga a la Arg69 de IPG en el modelo de trímeros de dímeros, se localiza externamente y por lo tanto también resulta accesible en la interacción con el sustrato. A pesar de que ni la secuencia aminoacídica, ni la estructura tridimensional de IPG, presenten similitud con los inhibidores de proteasas descriptos, la composición del sitio activo permite establecer una analogía entre ambos ya que la mayoría de los inhibidores tipo Kunitz poseen arginina en el sitio activo (Ceciliani et al., 1994; Haldar et al., 1996; Joubert et al., 1985). Además, tal como se observa en la Figura 30, las 67 Resultados Capítulo I estructuras terciarias del inhibidor de tripsina Ascaris (Grasberger et al., 1994) y el inhibidor bifuncional de tripsina y α-amilasa (Strobl et al., 1995) muestran que la arginina perteneciente al sitio activo se encuentra en un bucle expuesto, de la misma manera que la Arg69 en el modelo de IPG. Figura 30. Estructura de dos inhibidores de proteasas que poseen arginina en el sitio activo. (A) Inhibidor de tripsina Ascaris (Grasberger et al., 1994). (B) Inhibidor bifuncional de tripsina y α-amilasa (Strobl et al., 1995). En ambos casos se señala la arginina expuesta del sitio activo (P1). 68 Discusión Discusión Capítulo I La mayoría de las GLPs han sido descubiertas indirectamente en la búsqueda o análisis de determinadas actividades enzimáticas y secuencias. Muchas de las proteínas de esta familia presentan la particularidad de exhibir más de una actividad biológica (Bernier and Berna, 2001). En el caso particular de IPG, la búsqueda se inició tratando de asignarle una identificación dentro de los inhibidores de proteasas. Sin embargo cuando se obtuvo la secuencia de su N-terminal se descubrió su pertenencia a esta familia (Segarra et al., 2003). Posteriormente la obtención de su secuencia aminoacídica completa a partir de su ADNc permitió la identificación de los boxes A, B y C que caracterizan a la familia de las germinas y GLPs (Bernier and Berna, 2001). A partir de estos datos se constató que IPG no pertenecía a ninguna de las familias características de inhibidores de proteasas. Dado que nunca se había descripto una actividad de inhibidor de proteasas en las GLP, se pensó que IPG podría ser un nuevo miembro en este grupo de proteínas. Sin embargo, se encontró que IPG resulto ser idéntica a la GLP de cebada con actividad adenosina glucosa pirofosfatasa/fosfodiesterasa (AGPPasa) descripta por Rodríguez-López et al. (2001) y análoga a HvGER2a caracterizada previamente por Vallelian-Bindschedler et al. (1998). La forma oligomérica de IPG también posee actividad SOD (Segarra et al., 2003) y la estructura tridimensional obtenida por modelado bioinformático presenta el sitio de unión a manganeso idéntico al que se ha descripto para otras germinas y GLPs con esta actividad (Woo et al., 2000; Yamahara et al., 1999). A pesar de que IPG resultó análoga a HvGER2a, en estudios realizados por Zimmermann et al. (2006) la actividad SOD no fue detectada en esta proteína, lo que podría deberse a una baja estabilidad de la misma por tratarse de una proteína recombinante que no forma oligómeros. En los últimos años se han encontrado numerosas proteínas que presentan más de una función o actividad enzimática y que pueden o no estar relacionadas en una misma vía metabólica. Entre otros muchos ejemplos de proteínas multifuncionales (Khandelwal et al., 2007; Oba et al., 2003; Stec et al., 2000; Urban et al., 2005; Zhang et al., 2003), Sharma et al. (2004) describieron una proteína de tabaco perteneciente a la familia RIP (Ribosome-inactivating proteins) que presenta actividad de N-glicosidasa característica de la familia y también actividad Fe-SOD. Ambas actividades juegan un rol fundamental en las respuestas de defensa a estrés en plantas. Para el caso de IPG, se 69 Discusión Capítulo I han detectado al menos tres actividades enzimáticas: inhibidor de proteasas, AGPPasa y SOD. Se ha descripto que los inhibidores de proteasas, además de regular procesos proteolíticos y actuar como proteínas de reserva en semillas, pueden participar en mecanismos de defensa contra microorganismos fitopatógenos e insectos (Mosolov and Valueva, 2005). La actividad AGPPasa controla el nivel de los azúcares nucleótidos por lo que podría cumplir un rol importante en el fortalecimiento de la pared celular regulando la síntesis de los polisacáridos en la misma (Baroja-Fernández et al., 2000). Por último, la actividad SOD, además de proteger a las células de los efectos tóxicos del anión superóxido producido en el estrés oxidativo, genera peróxido de hidrogeno que puede actuar como molécula señal en mecanismos de defensa (Kovtun et al., 2000), catalizar reacciones de cross-linking involucradas en la síntesis de lignina (Lane et al., 1993; Mehdy, 1994; Olson and Varner, 1993; Wei et al., 1998) o actuar como agente antimicrobiano (Apostol et al., 1989; Custers et al., 2004; Legendre et al., 1993; Walters, 2003). En el caso de IPG, las tres actividades enzimáticas que posee no están relacionadas en una misma vía metabólica pero podrían participar de manera conjunta en los mecanismos de defensa de las plantas frente a situaciones de estrés biótico o abiótico. Recientemente Shetty et al. (2009) mostraron que en el FI de hojas de un cultivar de trigo resistente a la septoriosis, se acumulan isoformas de β-1,3-glucanasas luego de la inoculación con el hongo Septoria tritici. El posterior análisis de una de ellas por espectrometría de masas permitió identificarla como la AGPPasa de cebada descripta por Rodríguez-López et al. (2001) y por lo tanto idéntica a IPG de trigo. Por lo que ésta también podría ser una nueva función de IPG en relación a los mecanismos de defensa del trigo frente a la septoriosis. Cabe recordar que Segarra et al. (2002) demostraron que la actividad inhibitoria de IPG disminuye en un cultivar resistente de trigo luego de la infección con Septoria tritici. Esta disminución posibilita el aumento de una actividad proteolítica del FI cuya acción antifúngica fue descripta. Este ejemplo permite hipotetizar que un mismo estrés, en este caso la infección con S. tritici, puede modificar diferencialmente dos vías de acción distintas para una misma proteína. Dado que la actividad de inhibidor de proteasa de tipo serina de IPG fue descripta por vez primera en nuestro laboratorio, uno de los objetivos de esta tesis fue hallar el sitio activo responsable de dicha actividad. Una de las estrategias fue determinar si alguno de los fragmentos obtenidos por digestión química o enzimática 70 Discusión Capítulo I mantenía la actividad inhibitoria. Con dicho abordaje experimental, no se obtuvieron resultados positivos y una de las explicaciones posibles es que la escasa masa de los péptidos resultantes de la digestión dificulta la detección de la actividad en geles. Por ello se trató de eluir los péptidos del gel y realizar la medición de actividad con una técnica más sensible. Esa estrategia no rindió péptidos biológicamente activos. Finalmente, la estrategia utilizada para la identificación del sitio activo fue la modificación de algunos residuos de la proteína con reactivos químicos específicos (Vallejos, 1981) para así determinar si se producen cambios en su actividad inhibitoria. La modificación química de proteínas es un método relativamente simple y rápido que brinda información del rol de un aminoácido particular en una proteína. El gran desarrollo de la biología molecular ha convertido a la mutagénesis dirigida en una herramienta accesible y de uso frecuente. Sin embargo la modificación química selectiva, a pesar de su utilidad, no es aplicada regularmente. Cabe mencionar que cuando se utiliza la modificación química, hay que tener en cuenta las condiciones de la reacción (concentración de los reactivo, tiempo, temperatura, pH, entre otras) debido a que en condiciones inapropiadas el reactivo puede reaccionar con más de un tipo de aminoácido o puede alterar el plegamiento de la proteína. Hay muchos trabajos que demuestran la composición del sitio activo de una enzima modificando químicamente algún aminoácido: la arginina presente en el sitio activo de la acetolactato sintasa de tabaco (Kim et al., 2003), los residuos esenciales de histidina y arginina de la enzima Glucosa-6-Fosfato dehidrogenasa de Streptomyces aureofaciens (Montavon and Kruger, 1993) y el rol indispensable de la arginina, lisina y tirosina en el sitio activo de la enzima glutamato deshidrogenasa de Rumex dentatus (El-Shora and Youssef, 2008) En este trabajo, se modificó la histidina con DEPC con el objetivo de determinar si la acción inhibitoria sobre las serina proteasas coincide con el sitio activo de la SOD (Woo et al., 2000). Por otra parte, se modificó arginina debido a la especificidad de unión a tripsina en los inhibidores de serina proteasas (Laskowski and Qasim, 2000). Debido a que la actividad inhibitoria de IPG no cambio con DEPC, la idea de que las histidinas formasen parte del sitio activo fue descartada. Contrariamente, IPG no inhibió tripsina cuando la única arginina fue modificada, por lo que se puede concluir que este aminoácido es esencial para dicha actividad. Aunque IPG no presente similitud de secuencia con las familias de inhibidores de proteasas descriptos, este hallazgo es interesante debido a que la mayoría de los inhibidores tipo Kunitz poseen 71 Discusión Capítulo I arginina en su sitio activo (Batista et al., 1996; Ceciliani et al., 1994; Haldar et al., 1996; Joubert et al., 1985; Kim et al., 1985; Ozawa and Laskowski, 1966; Yamamoto et al., 1983). Además, en las estructuras terciarias de algunos inhibidores de proteasas la arginina aparece en un bucle expuesto, tal como ocurre en el modelo 3D realizado para IPG. Debido a que la mayoría de los inhibidores de proteasas poseen varios puentes disulfuros que estabilizan el sitio activo, también se realizó la modificación de los residuos de cisteína con NEM. Al pH específico de modificación de cisteína, la actividad de IPG disminuyó a la mitad y desapareció totalmente a pH básico, en el cual también ocurre la modificación de aminoácidos básicos como la arginina. Por ello, se puede concluir que si bien las cisteínas son importantes para mantener la actividad inhibitoria de IPG, la arginina es el aminoácido principal del sitio activo. Para avanzar en la caracterización del sitio activo responsable de la actividad de inhibidor de proteasa, serán necesarios futuros estudios que se centren en la mutagénesis dirigida o en la utilización de péptidos sintéticos que permitan definir el sitio activo completo de IPG. 72 Capítulo II “Expresión de IPG durante el desarrollo de la planta de trigo y frente a distintas situaciones de estrés” Introducción Capítulo II Muchas proteínas de la familia de las GLPs han sido vinculadas con la resistencia de las plantas al estrés biótico y abiótico. Para IPG, se ha demostrado su participación en los mecanismos de defensa frente a septoriosis en plántulas de trigo (Segarra et al., 2002). En el presente capítulo de tesis, se evaluará el comportamiento de IPG frente a distintas situaciones de estrés, y se tratará de establecer si los cambios bioquímicos y morfológicos estudiados evidencian cierta tolerancia del cultivo al estrés. Las condiciones de estrés abiótico evaluadas fueron salinidad y calor, y la interacción entre el hongo patógeno S. tritici y el hongo biocontrolador Trichoderma harzianum fue estudiada como parte del estrés biótico. Por otra parte y debido a que se han identificado GLPs en diferentes estadios del desarrollo de la planta como en inducción floral, maduración del fruto, embriogénesis somática y cigótica, desarrollo de la semilla, entre otros (Dunwell et al., 2000), se decidió evaluar diferentes localizaciones de IPG. Hasta el momento IPG sólo fue hallada en el FI de primera hoja de plántulas de trigo y en el presente trabajo se exploró su localización en embriones de semillas maduros, segunda hoja de plántulas de trigo, primera hoja de macollos y hoja bandera de plantas de trigo en estadio espigazón. Además, dichas localizaciones se relacionaron con los estreses bajo estudio. 73 Resultados Resultados Capítulo II 1. Estrés abiótico El estrés abiótico provoca cambios morfológicos, fisiológicos, bioquímicos y moleculares en las plantas que afectan negativamente el crecimiento y productividad de las mismas (Wang et al., 2001). Las plantas sufren una exposición continua a diversos estreses abióticos en su ambiente natural, y a veces se combinan con estreses bióticos. Para sobrevivir en esas condiciones las plantas han desarrollado mecanismos complejos para percibir las señales externas lo que permite una respuesta óptima a las condiciones ambientales. La respuesta de protección, en general, está regulada por fitohormonas como el ácido salicílico (SA), el ácido jasmónico (JA), el etileno (ET), y el ácido abscísico (ABA). Estas moléculas endógenas de bajo peso molecular, actúan de manera sinérgica o antagónica, y son las que conectan el estrés abiótico con el biótico (Bostock, 2005; Lorenzo and Solano, 2005; Mauch-Mani and Mauch, 2005). Por otra parte, la generación de especies reactivas de oxígeno se ha propuesto como un proceso clave que se comparte entre las respuestas de estrés bióticos y abióticos (Apel and Hirt, 2004; Torres and Dangl, 2005). Las especies reactivas de oxígeno, como el anión superóxido (O2.-), peróxido de hidrogeno (H2O2), radical hidroxilo (.OH) y el oxigeno singulete (1O2), pueden alterar el metabolismo normal de los organismos a través del daño oxidativo a lípidos (Fridovich, 1986; Wise and Naylor, 1987), ácidos nucleicos y proteínas (Imlay and Linn, 1988). Una vez producido el O2.- puede rápidamente dismutar para producir H2O2 y O2. Las plantas poseen mecanismos antioxidantes que las protegen contra las ROS. La metaloenzima superóxido dismutasa (SOD) convierte O2.- en H2O2 , y las catalasas y peroxidasas catalizan la descomposición del H2O2 (Chang et al., 1984). Cuando las plantas son sometidas a condiciones de estrés ambiental como alta intensidad de luz, temperaturas extremas, sequía, salinidad elevada, tratamiento con herbicida o deficiencias de minerales, el equilibrio entre la producción de especies reactivas de oxígeno y la actividad de las enzimas antioxidantes se ve alterado, resultando en un daño oxidativo (Spychalla and Desborough, 1990; Wise and Naylor, 1987). Se ha demostrado que las plantas con altos niveles de antioxidantes, ya sean constitutivos o inducidos, tienen una mayor resistencia al daño oxidativo (Spychalla and Desborough, 1990). El estudio de cómo las plantas responden a los diferentes estreses a nivel de genes y proteínas es importante para desarrollar cultivares tolerantes a las condiciones 74 Resultados Capítulo II desfavorables. En general, la identificación de genes que se expresen diferencialmente en condiciones de estrés es de mucha utilidad para entender los mecanismos de defensa de la planta. El desarrollo de técnicas de gran alcance como el análisis por microarrays proporciona una gran cantidad de información acerca de los genes implicados en las respuestas al estrés ambiental. Además, dichos enfoques genómicos deben complementarse con análisis cualitativos y cuantitativos del proteoma de la planta. Como se ha mencionado anteriormente, las GLPs han sido asociadas a mecanismos de defensa de las plantas frente a estreses bióticos y abióticos. En el caso de IPG, se ha demostrado su participación en los mecanismos de defensa frente al ataque del hongo patógeno Septoria tritici (Segarra et al., 2002) sin embargo no se ha evaluado su participación en los mecanismos de tolerancia a estrés abiótico. Además, como se demostró en el capítulo anterior, IPG es una proteína multifuncional, con al menos tres actividades enzimáticas. Por ello, en la primera parte de este capítulo, se evaluará la vinculación de IPG con los mecanismos de tolerancia al estrés salino y al estrés por altas temperaturas desde el punto de vista de su abundancia y de la modificación de sus actividades enzimáticas. 1.1. Estrés salino La salinidad del suelo es un problema agrícola importante, sobre todo porque la mayoría de los cultivos poseen baja tolerancia a la sal. La respuesta de las plantas al estrés salino es un fenómeno complejo que involucra procesos bioquímicos y fisiológicos, así como también cambios morfológicos y de desarrollo (Flowers et al., 1977; Greenway and Munns, 1980). La identificación de genes cuya expresión permita a las plantas adaptarse al estrés o tolerar la sal, es esencial para los programas de mejoramiento genético. Una de las estrategias que se siguen para determinar los mecanismos moleculares implicados en el estrés salino, es identificar genes que cambien su expresión como resultado del estrés salino. En ese aspecto, se ha demostrado que el estrés salino provoca cambios en la regulación génica en general y, en particular, de dos isoformas de GLPs en raíces de cebada (Hurkman and Tanaka, 1996). Sin embargo, poco se sabe sobre el posible papel de estas proteínas en el estrés salino (Berna and Bernier, 1999; Hurkman et al., 1994; Hurkman and Tanaka, 1996; Hurkman et al., 1991). Recientemente se ha demostrado que el estrés salino no provoca cambios en la expresión del gen de una germina (gf 2.8), pero sí ocurre una 75 Resultados Capítulo II relocalización de la misma (Caliskan, 2009), encontrándose predominantemente en las células del coleóptilo y disminuyendo en las células de la coleorriza de los embriones. Sin embargo aún falta dilucidar los mecanismos por los que se altera el sitio de expresión de dicha germina. En el presente trabajo de tesis, se evaluó el comportamiento de IPG en las plántulas de trigo sometidas a estrés salino y se trató de determinar si participa en los mecanismos de tolerancia a dicho estrés. En primer lugar se determinó el contenido de IPG en el FI de primera y segunda hoja de plántulas de trigo de 17 días, sometidas a estrés salino durante 12 días. Tal como se observa en la Figura 31, los niveles de IPG en el FI de la primera hoja permanecieron constantes (A) y disminuyeron en la segunda (B) cuando se analizaron a través de un western-blot. Figura 31. Efecto del estrés salino sobre la abundancia de IPG en el FI. Proteínas del FI de la primera hoja (A) o de la segunda hoja (B) fueron separadas en SDS-PAGE, transferidas a una membrana de nitrocelulosa e inmuno-detectadas con el anticuerpo específico de IPG. Calle 1: plantas control, Calle 2: plantas tratadas con 200 mM NaCl. La figura es representativa de tres experimentos independientes. En cada calle se sembró el FI que corresponde a 100 mg de peso fresco de hojas Se ha descripto que en hojas de cebada sometidas tanto a estrés biótico como abiótico una GLP del FI se insolubiliza o, dicho de otra manera, se relocaliza en pared celular (Vallelian-Bindschedler et al., 1998). En dicho trabajo, para determinar que tipo de unión posee la GLP con la pared celular, se realizó una separación de las proteínas diferenciando a las que están unidas mediante enlaces iónicos, uniones no covalentes o covalentes. Se determinó que la GLP de cebada se encontraba en la fracción de proteínas unidas de manera no covalente a la pared celular. Para determinar si algo similar ocurre con IPG en el FI de la hoja de trigo, se obtuvo el FI de las hojas y posteriormente se extrajeron las proteínas unidas de manera no covalente a la pared celular, utilizando un buffer conteniendo SDS, tal como lo describen VallelianBindschedler et al. (1998). El resultado de dicha extracción se muestra en la Figura 32, 76 Resultados Capítulo II en la que se observa que los niveles de IPG son mayores en la primer hoja cuando es sometida a estrés salino (Figura 32 A). En cambio, en la segunda hoja la diferencia entre control y estrés no fue estadísticamente significativa (Figura 32 B). Figura 32. Efecto del estrés salino sobre la abundancia de IPG en la pared celular. Proteínas unidas a la pared celular extraídas de la primera hoja (A) o de la segunda hoja (B) fueron separadas en SDS-PAGE, transferidas a una membrana de nitrocelulosa e inmunodetectadas con el anticuerpo específico de IPG. Calle 1: control, Calle 2: 200 mM NaCl. La figura es representativa de tres experimentos independientes. En cada calle se sembró el volumen de extracto que corresponde a 50 mg de peso fresco de hojas. Como se ha mencionado anteriormente, IPG es un inhibidor de proteasas con actividad SOD y dichas actividades enzimáticas han sido vinculadas con la tolerancia al estrés abiótico. En el caso de inhibidores de proteasas, no son muchos los trabajos que han demostrado esta relación, pero por ejemplo, se ha descripto un inhibidor de proteasas de trigo, del tipo Bowman-Birk, que juega un rol importante en la regulación del crecimiento de las plantas y transporte de Na+ en plantas sometidas a estrés salino (Shan et al., 2008). También se ha demostrado la acumulación de inhibidores de tripsina y α-amilasa en hojas de Amaranthus hypochondriacus en respuesta a estrés salino, entre otros tipos de estreses abióticos (Sanchez-Hernandez et al., 2004). Con respecto a la actividad SOD, son muchos los trabajos que demuestran su importancia en plantas sometidas a estrés, debido a su capacidad para eliminar a las especies reactivas de oxígeno (Esfandiari et al., 2007; Mandhania et al., 2006; Rout and Shaw, 2001). Por ello, se evaluó si la actividad de inhibidor de proteasas o la actividad SOD de IPG se modificaban por el estrés salino. En el primer caso la actividad inhibitoria fue medida in vitro (Udenfriend et al., 1972) y se detectó que dicha actividad disminuye en el FI y aumenta en la pared, tanto para primera como para segunda hoja (Figura 33). 77 Resultados Capítulo II Figura 33. Efecto del estrés salino sobre la actividad inhibitoria de IPG en FI y pared celular. (A) Primera hoja y (B) segunda hoja. La actividad inhibitoria fue expresada en porcentaje, considerando el 100% a que aquella que provoca una inhibición total sobre tripsina. En cada medición se utilizó el volumen de muestra correspondiente a 100 mg de peso fresco de hoja. La actividad SOD se analizó mediante un zimograma (Beauchamp and Fridovich, 1971). En el FI de plantas sometidas a estrés salino la actividad SOD disminuye, tanto para primera como para segunda hoja (Figura 34, FI A y B). Por el contrario, en la pared celular de primera hoja sometida a estrés salino se observó un leve aumento con respecto al control (Figura 34, Pared, A). Figura 34. Efecto del estrés salino sobre la actividad SOD de IPG. Las proteínas del FI o unidas a la pared celular fueron separadas en SDS-PAGE en condiciones no reductoras y analizadas para su actividad SOD. (A) Primera hoja, (B) Segunda hoja, (1) control, (2) 200 mM NaCl. En cada calle se sembró el volumen de muestra correspondiente a 500 mg de peso fresco de hoja. El peróxido de hidrógeno generado por la dismutación del anión superóxido y catalizado por SOD en la pared de la primera hoja, podría actuar tanto como molécula señal en mecanismos de defensa (Kovtun et al., 2000) o catalizando reacciones de 78 Resultados Capítulo II cross-linking involucradas en la síntesis de lignina y por ende en el fortalecimiento de la pared celular (Lane et al., 1993). El estrés salino produce un retraso en el crecimiento de las plantas (Hernández et al., 1995; Takemura et al., 2000). La respuesta inmediata al estrés, es la reducción en la velocidad de expansión de la superficie de la hoja, lo que lleva a un detenimiento total cuando la concentración de sal aumenta (Wang and Nil, 2000). El estrés salino también provoca una disminución considerable en el peso fresco y seco no sólo de hojas sino de tallos y raíces (Ali-Dinar et al., 1999; Hernández et al., 1995). En el caso de las plántulas de trigo evaluadas en el presente trabajo en las condiciones de estrés salino, no se observaron cambios significativos en la longitud de la primera hoja de trigo pero si en la segunda hoja (Figura 35, A y B). Además en la primer hoja, la disminución del peso fresco fue menor que en la segunda (Figura 35 C). Figura 35. Efecto del estrés salino sobre la longitud y peso fresco de las hojas de trigo. (A) Plántulas enteras (primera y segunda hoja) de trigo control o sometidas a estrés salino (200 mM NaCl). En las mismas se determinó longitud (B) y peso fresco (C). Los datos son el promedio de al menos 5 experimentos independientes. Barras con letras distintas son significativamente diferentes entre sí, con p < 0,05 (test de T). Las barras representan el SD. 79 Resultados Capítulo II En general, el contenido total de clorofila y carotenoides disminuyen en las hojas sometidas a estrés salino. (Khavari-Nejad and Chaparzadeh, 1998; Parida et al., 2004) Las hojas más viejas comienzan a desarrollar clorosis y, si la duración del estrés es prolongada, directamente se caen (Agastian et al., 2000; Gadallah, 1999; Hernández et al., 1995; Hernández et al., 1999). Sin embargo, Wang and Nil (2000) han descripto que el contenido de clorofila aumenta bajo condiciones de estrés salino en Amaranthus. En las plantas analizadas en la presente tesis se encontró que el contenido de clorofila en la primera hoja de las plántulas sometidas a estrés es mayor que en las hojas control. Estas diferencias no se observaron en la segunda hoja (Figura 36 A). Figura 36. Efecto del estrés salino sobre el contenido de clorofila (A) y de Prolina libre (B) de las hojas de trigo. El contenido de clorofila de hojas control y tratadas con 200 mM NaCl se midió con el minolta-SPAD según lo descripto en Materiales y Métodos. El contenido de prolina libre se determinó según lo descripto por Bates et al. (1973) Los datos son el promedio de al menos 3 experimentos independientes. Barras con letras distintas son significativamente diferentes entre sí, con p < 0,05 (test de T). Las barras representan el SD. Por otra parte y debido a que la acumulación de prolina es un mecanismo corriente en las plantas halófitas (Hernández et al., 2000; Lee and Liu, 1999), se determinó el nivel de este osmolito en hojas control y estresadas. Como se observa en la Figura 36 B hay un aumento del 200 % en el contenido de prolina libre de la primera hoja tratada con 200 mM de NaCl, mientras que en la segunda hoja no hay diferencias significativas. Además los niveles de prolina en la segunda hoja son más bajos que en la primera. Dado que la acumulación de prolina otorga una mayor tolerancia al estrés salino (Kuznetsov and Shevyakova, 1997), estos resultados sugieren que en la primera hoja de plántulas de trigo se han activado algunos de los mecanismos de tolerancia a dicho estrés que no pudieron mantenerse en la 80 Resultados Capítulo II segunda hoja, y ello puede estar relacionado con el aumento de IPG en la pared celular (Figura 32A). Para determinar si el aumento de IPG en la pared celular se debe a una insolubilización de IPG del FI, es decir una relocalización en la pared, o a una disminución en la cantidad que se exporta al apoplasto, se realizó un nuevo ensayo modificando el método de extracción de proteínas. Para ello, previo al tratamiento con SDS para extraer las proteínas unidas de manera no covalente a la pared, se realizó una incubación con un buffer fosfato pH 7.5 y así se extrajeron las proteínas solubles intracelulares. El resultado de dicho experimento se muestra en la Figura 37. Figura 37. Efecto del estrés salino sobre la abundancia de IPG en pared celular: unida no covalentemente (P) y en su forma soluble (S). Luego de la obtención del FI de la primera hoja las proteínas restantes fueron extraídas en buffer fosfato (S) y con SDS (P). (A) Western blot contra el anticuerpo especifico de IPG; (B) Densitometría de la banda inmuno detectada en el western blot. Se consideró el 100% a la densitometría de las calles control. En cada calle se sembró el volumen de FI que corresponde a 50 mg de peso fresco de hoja. Tanto en las plántulas control como en las sometidas a estrés salino, hay mayor cantidad de IPG en la fracción de proteínas solubles que en la insoluble unida a pared celular (Figura 37A). Si se compara sólo la fracción de proteínas de pared, hay mayor cantidad de IPG en las plántulas tratadas con 200 mM NaCl, que va acompañado de una disminución en la fracción soluble, tal como lo muestra la densitometría (Figura 37B). Sin embargo esta disminución no alcanza para explicar el mayor aumento en pared, por lo que para comprobar las razones de esta relocalización se deberían realizar otros experimentos para cuantificar la cantidad de IPG con péptido señal de modo de determinar si se encuentra ubicado intracelularmente o en el apoplasto. 81 Resultados Capítulo II Debido a que la proteína germina fue aislada de embriones maduros de semillas (Thompson and Lane, 1980), y que uno de los objetivos de la presente tesis es determinar si IPG se expresa en otros órganos o estadios de la planta, se decidió evaluar la presencia de IPG en embriones maduros de semilla de trigo. Además, y en relación con la caracterización del estrés salino, se intentó determinar si su contenido se modifica frente al estrés salino. En primer lugar se evaluó el efecto del estrés salino en la germinación de las semillas de trigo (Figura 38). Figura 38. Efecto del estrés salino sobre la germinación de las semillas de trigo. Se expresó el % de germinación en función de las horas de incubación en las soluciones de NaCl de 0 a 200 mM. Como se puede observar en la Figura 38, el aumento en la concentración salina retarda la germinación. A las 18 h de incubación en agua (control, 0 mM NaCl) el 90% de las semillas están germinadas, mientras que en la incubación en 200 mM de NaCl, sólo el 10% de las semillas germinaron. En tiempos más largos (a partir de las 24 h) y hasta los 150 mM de NaCl, el estrés salino parece no influenciar significativamente la germinación. En cambio, una concentración de 200 mM de NaCl, retarda mucho más la germinación, y 60 h post-imbibición no se llega al valor del control. Una vez determinado el efecto de la salinidad en la germinación, se planteó establecer si IPG se encuentra en embriones maduros de semilla de trigo, y de ser así, si hay modificaciones en su contenido en condiciones de estrés salino. Para ello se extrajo el embrión a diferentes horas post-imbibición de las semillas. Los tiempos usados fueron: 0 (secos), 1 h, 5 h, 18 h y 24 h, en los que se imbibieron en agua (control) o en 82 Resultados Capítulo II solución de NaCl 200 mM. Se utilizó esta concentración de cloruro de sodio debido a que con ella se obtuvo el efecto más marcado de retardo en la germinación (Figura 38). Las proteínas totales se extrajeron de los embriones según Cuming y Lane (1979) y se realizó un western blot para determinar si IPG esta presente en dicho extracto (Figura 39). Figura 39. Efecto de la salinidad sobre la abundancia de IPG en embriones de semillas de trigo. (A) Western-blot y (B) densitometría de la banda inmunodetectada. Semillas de trigo fueron imbibidas durante 0, 1, 5, 18 y 24 hs en agua (C) o 200 mM NaCl (S) y luego se realizó la extracción de proteínas del embrión. Las muestras proteícas (20 µgr en cada calle) fueron hervidas durante 3 minutos en buffer de muestra con 2-mercaptoetanol y separadas en SDSPAGE, transferidas a una membrana de nitrocelulosa e inmuno-detectadas con el anticuerpo específico de IPG. Se puede observar que el anticuerpo reconoce específicamente una banda de aproximadamente 50 kDa (Figura 39A). Dicha masa molar, se aproxima más al oligómero de IPG que al monómero que debería estar presente dado que las muestras fueron totalmente desnaturalizadas para dicho experimento. Cabe mencionar que el anticuerpo de IPG es policlonal, por lo que puede interaccionar con proteínas similares estructuralmente. Entonces, una posible explicación del resultado anterior es que IPG no se encuentre en el embrión y el anticuerpo esté reconociendo una proteína semejante. Hay que recordar que en la superfamilia de las cupinas, a la que germinas y GLPs pertenecen, se encuentran las proteínas globulinas de reserva, como vicilinas y leguminas, muy similares estructuralmente a IPG. Otra posibilidad es que en este órgano IPG se encuentre formando dímeros y no monómeros, y éstos necesiten condiciones diferentes para su desnaturalización. Eso justificaría el mayor peso molecular obtenido. Para comprobar alguna de estas hipótesis, se necesitarían otro tipo de ensayos. Se podría realizar 83 Resultados Capítulo II espectrometría de masas, MALDI-TOF, a la banda inmunodetectada y determinar si el pico m/z de 2201 kDa, característico de IPG (ver resultados Capítulo I), se encuentra en dichos espectros. Otro experimento que podría realizarse sería un Northern-blot con la sonda de ADNc obtenida y se esa manera determinar certeramente si IPG se expresa en embriones maduros de semilla de trigo. De todas formas en la banda inmunodetectada, así se trate de IPG o de alguna proteína de la familia, no se observaron modificaciones significativas entre los tratamientos control y 200 mM NaCl, en los distintos tiempos de imbibición ensayados. Sin embargo, sí se pudo determinar que a medida que aumenta el tiempo de imbibición de la semilla, aumenta la cantidad de la proteína identificada por el anticuerpo con respecto a las proteínas totales, tal como lo muestra el gráfico de barras de la densitometría (Figura 39B). 84 Resultados Capítulo II 1.2. Estrés por altas temperaturas El estrés por altas temperaturas es otro de los estreses abióticos con mayor impacto sobre el rendimiento y la calidad de los cultivos en muchas partes del mundo (Neilson et al., 2009). La temperatura media mundial está aumentando con el tiempo y por lo tanto el estudio de cómo las plantas responden al estrés por alta temperatura a nivel de genes y proteínas es importante para desarrollar cultivares tolerantes al calor. A nivel génico, por ejemplo, muchos estudios han demostrado la regulación positiva de las transcripciones de las proteínas de choque térmico (HSP, Heat Shock Proteins) (Larkindale and Vierling, 2008; Rizhsky et al., 2002; Simoes-Araujo et al., 2002). También se han descripto aumentos en las transcripciones de miembros de la familia de factores de transcripción DREB2, el gen AsEXP1 que codifica para una expansina, genes que codifican para la sintasa galactinol y enzimas antioxidantes (Busch et al., 2005; Lim et al., 2006; Rizhsky et al., 2002; Rizhsky et al., 2004; Xu et al., 2007). Larkindale y Vierling (2008) también encontraron que la disminución en las transcripciones de los genes relacionados con la muerte celular programada, metabolismo básico, y los de respuestas a estrés bióticos son igualmente importantes para la aclimatación al calor. Sin embargo, debido a que los niveles de abundancia de ARNm y de las proteínas no siempre se correlacionan, los enfoques genómicos deben complementarse con análisis cualitativos y cuantitativos del proteoma de la planta. Con respecto al análisis proteómico de las plantas bajo estrés térmico, se ha demostrado que la expresión diferencial de las proteínas HSPs es una importante estrategia de adaptación de las plantas al estrés (Feder and Hofmann, 1999; Howarth, 1991; Vierling, 1991). Las HSPs, con masas moleculares que van desde 10 hasta 200 kDa, están involucradas en la transducción de señales durante el estrés por calor y tienen la función de chaperonas, ayudando al correcto pliegue de las proteínas celulares, y a la protección de los efectos adversos de alta temperatura a sitios funcionales (Vierling, 1991). Las chaperonas moleculares son proteínas que se unen y estabilizan la conformación de otra proteína. Además, son responsables del transporte y degradación en los procesos celulares normales, y también en la estabilización de las proteínas y membranas, y ayudan al replegado de las mismas en condiciones de estrés. Las HSPs no son las únicas proteínas importantes en la defensa al estrés por altas temperaturas. Por ejemplo, se ha encontrado que la expresión de ubiquitina, proteínas tipo LEA (Late Embryogenesis Abundant) o dehidrinas juegan un papel importante en la tolerancia al estrés. La principal función de la ubiquitina y la síntesis de 85 Resultados Capítulo II conjugados de ubiquitina, parece ser la protección de las estructuras celulares y subcelulares contra el daño oxidativo y la deshidratación (Ortiz and Cardemil, 2001). Las proteínas LEA pueden prevenir la agregación y se ha demostrado que, por ejemplo, protegen a la citrato sintasa de la desecación en condiciones de estrés por calor y sequía (Goyal et al., 2005). En el caso de las dehidrinas, su función está relacionada con las vías de degradación de las proteínas, reduciendo al mínimo los efectos adversos de la deshidratación y el estrés oxidativo durante el estrés por calor (Schöffl et al., 1999). Otras proteínas encargadas de la protección oxidativa inducida por el calor son la tiorredoxina h, glutatión S-transferasa y dehidroascorbato reductasa (Lee et al., 2007), Cu / Zn SOD citosólica (Herouart et al., 1994) y Mn-peroxidasa (POD) (Brown et al., 1993) (Brown et al. 1993), cuyas expresiones son estimuladas por el estrés. En el caso particular de la actividad SOD, en un estudio sobre Chenopodium murale, se informó que la proteína de la fracción del estroma era más tolerante al calor que la Cu / Zn-SOD de los tilacoides, y se determinó que dicha proteína era la responsable de la estabilidad de los cloroplastos bajo estrés por calor (Khanna-Chopra and Sabarinath, 2004). Sin embargo, se ha descripto un descenso en la expresión de una Cu / Zn-SOD a causa del calor en hojas de arroz (Lee et al., 2007). También Sato et al. (2001) reportaron que la actividad SOD no fue afectada en las plántulas de arroz expuestos al calor (42 ° C). Estos resultados sugieren que las proteínas SOD podrían responder al estrés térmico en forma compleja o que, isoformas diferentes podrían ser reguladas diferencialmente. Por lo tanto, la respuesta al estrés térmico es muy compleja y son muchos los factores que intervienen para brindar tolerancia a las plantas. Debido a que IPG es un inhibidor de proteasas que también presenta actividad SOD, se evaluó el comportamiento de dicha proteína en el estrés por altas temperaturas. Entonces, para evaluar el efecto de altas temperaturas sobre la actividad inhibitoria de la proteólisis de IPG, plántulas de trigo de 11d fueron sometidas a tratamientos de 40º C durante 3 y 4 horas, y luego mantenidas durante 24 horas en las mismas condiciones de luz y temperatura que las descriptas previamente como normales (ver materiales y métodos). Debido a que la actividad de IPG regula la actividad proteolítica presente en el apoplasto FI (Segarra et al., 2002), también se cuantificó esta actividad enzimática en el FI. 86 Resultados Capítulo II Figura 40. Efecto del estrés por altas temperaturas sobre la actividad proteolítica del FI e inhibitoria de IPG FI70. 0, 3 y 4 indican las horas de tratamiento a 40 °C a las que fueron sometidas las plántulas de trigo. La actividad proteolítica e inhibitoria se expresaron como porcentaje con respecto a las plantas control (100%). Los datos de actividad proteolítica representan resultados de al menos 3 experimentos independientes. En cada medición se utilizó el volumen de muestra correspondiente a 100 mg de peso fresco de hoja. Luego de las tres horas de tratamiento, la actividad inhibitoria de IPG aumenta ~30 %. Dicho aumento se correlaciona con una disminución de la actividad proteolítica total del FI (~60%). La actividad de IPG se modifica frente al estrés por altas temperaturas del mismo modo que el reportado previamente frente a estrés biótico durante la infección por Septoria tritici de un cultivar susceptible (Segarra et al., 2002). La inducción de las proteínas de shock térmico es parte de la respuesta del trigo al estrés por altas temperaturas (McElwain and Spiker, 1992). Debido a que IPG aumenta su actividad en estas condiciones, podría existir una relación entre ambos eventos, tendientes a promover la termotolerancia en las plantas de trigo. Para avanzar en este estudio y determinar si existe una correlación entre dicho aumento de actividad y el contenido de IPG en el FI, se realizaron ensayos de western blot. El western blot mostró que los niveles de IPG en el FI no se modificaban con el tratamiento de 3 horas a 40 °C (Figura 41B). Sin embargo, se determino un leve aumento en la fracción de proteínas unidas a la pared celular luego del tratamiento a 40 °C (Figura 41A), tal como lo descripto para el estrés salino. 87 Resultados Capítulo II Figura 41. Efecto del estrés por altas temperaturas sobre la abundancia y localización de IPG. Proteínas unidas a la pared celular (A) o del FI (B) fueron separadas en SDS-PAGE, transferidas a una membrana de nitrocelulosa e inmuno-detectadas con el anticuerpo específico de IPG. Calle 1: Control, Calle 2: tratadas por 3 hr a 40 °C. En cada calle se sembró el volumen de extracto que corresponde a 50 mg de peso fresco de primera hoja. 88 Resultados Capítulo II 2. Estrés biótico: inoculación con el hongo fitopatógeno Septoria tritici y el hongo biocontrolador Trichoderma spp. Como se mencionó anteriormente, en nuestro laboratorio se determinó que dos actividades proteolíticas extracelulares de la hoja de trigo se modifican frente al ataque del hongo patógeno Septoria tritici, aumentando hasta 400% en el cultivar resistente o disminuyendo en el cultivar susceptible a dicha enfermedad. Además, dichas actividades proteolíticas del FI inhiben la germinación de las conidiosporas del hongo y su actividad esta regulada por IPG. Posteriormente se comprobó que IPG participa en el control biológico de la septoriosis a través de la previa inoculación de plántulas de trigo con el hongo biocontrolador Trichoderma spp. Se constató que en las plántulas de trigo inoculadas con cepas de Trichoderma que lograron disminuir los síntomas de la enfermedad provocados por el hongo S. tritici, hubo un aumento de la actividad proteolítica y una disminución de la actividad inhibitoria de IPG respecto de las plantas control (Cordo et al., 2007). Dichos experimentos fueron realizados en `plántulas de trigo de un cultivar susceptible, en condiciones controladas de invernadero. Sin embargo, para que Trichoderma spp. pueda ser utilizado por los productores rurales como hongo biocontrolador de la septoriosis, hay que demostrar su efectividad en estadios avanzados del desarrollo de la planta de trigo mediante ensayos a campo. En la Figura 42 se esquematiza el proceso de crecimiento y desarrollo de la planta de trigo, en donde se pueden diferenciar 4 etapas principales: (1) macollaje, (2) encañado, (3) espigazón y floración, y finalmente (4) llenado de granos. Figura 42. Esquematización del proceso de crecimiento y desarrollo de las plantas de trigo. 89 Resultados Capítulo II La etapa de macollaje se inicia cuando aparecen brotes secundarios desde el eje principal, llamados macollos. Dependiendo de las condiciones de cultivo, pueden originarse varios macollos; éstos, luego de desplegar las primeras hojas, generan su propio sistema de raíces adventicias. Los macollos, por lo tanto, aunque formando siempre parte de la planta que los originó, comienzan a independizarse progresivamente de ésta, hasta llegar a comportarse como una planta individual. El desarrollo de la caña o etapa de encañado comienza cuando aparece una pequeña protuberancia que circunda al eje principal en la parte subterránea. Dicha protuberancia corresponde a lo que en definitiva será el primer nudo aéreo que dará lugar a la formación de la espiga en su parte apical. La etapa de encañado finaliza cuando el último nudo en la parte alta de la planta da paso a la espiga a través de la vaina de la hoja bandera, y ahí comienza la etapa de espigazón. Una vez que la espiga está completamente expresada en el extremo del tallo, se considera finalizada la etapa de espigazón. Sólo a partir de ese momento y en forma relativamente rápida, comienza la floración, fase que dura entre 1 y 2 semanas como promedio. La etapa de floración se considera terminada cuando todos los estambres de una espiga se hacen visibles asomándose a través de las espiguillas. La etapa de llenado de granos, se extiende desde que ocurre la fecundación hasta el momento en que se alcanza la madurez fisiológica, con aproximadamente 35% de humedad en los granos. Los granos inicialmente presentan un contenido acuoso; posteriormente, al ir madurando, pasan sucesivamente por los siguientes estados: lechoso, masa blanda, masa dura (madurez fisiológica), grano duro y madurez de cosecha. La duración de la etapa de llenado de granos presenta, en general, una correlación positiva con el rendimiento. Durante toda la etapa de llenado de granos, es fundamental, que tanto la espiga como las dos hojas superiores, permanezcan completamente sanas debido a su importancia decisiva en el tamaño de los granos y por lo tanto en el rendimiento final de las plantas. Por lo tanto, con el objetivo de analizar el efecto biocontrolador de Trichoderma spp. a campo, se realizaron experimentos en plantas de trigo provenientes de estadios más desarrollados, evaluándose la severidad de la enfermedad (porcentaje de necrosis y cobertura picnidial de la hoja analizado en el trabajo final de grado de Gustavo S. Walker Esponda de la Facultad de Agronomía de la UNLP) y correlacionándolos con las actividades proteolíticas del FI y la actividad inhibitoria de IPG. El estudio de dichas actividades se realizó en el FI de hojas provenientes de plantas en la etapa final de 90 Resultados Capítulo II macollaje y en la hoja bandera del estadio espigazón. Debido a que hasta el momento sólo se conoce que IPG está presente en hojas de plántulas de trigo, estos experimentos permitirán conocer si IPG también se expresa en hojas de macollos y hoja bandera. 2.1. Estudios realizados en la etapa final del macollaje. Plantas de trigo de un cultivar susceptible a la septoriosis que habían sido pretratadas con dos cepas del hongo biocontrolador Trichordema spp. (Th.cc5 y Th.118), fueron inoculadas con el hongo fitopatógeno Septoria tritici. Posteriormente, se extrajo el FI de la primera hoja de cada macollo y se analizó si las actividades proteolíticas descriptas anteriormente en plántulas están presentes en dicha fracción, al igual que IPG y su actividad inhibitoria. Además, para relacionar el comportamiento de dichas actividades enzimáticas con la resistencia a la septoriosis mediada por T. harzianum, se determinó el porcentaje de necrosis de la hoja provocado por S. tritici y la cobertura picnidial. La actividad proteolítica del FI fue analizada por SDS-PAGE copolimerizado con gelatina (Figura 43). No se observaron modificaciones significativas en ninguno de los tratamientos, salvo para las plantas pre-tratadas con Th.cc5 en las que la actividad proteolítica disminuyó levemente con respecto al control (Figura 43, calle 3). Figura 43. Efecto de S. tritici y Trichoderma spp. sobre la actividad proteolítica del FI examinado mediante SDS-PAGE copolimerizado con gelatina. En cada calle se sembró el FI correspondiente a 150 mg de peso fresco de primera hoja del macollo. 1, trigo; 2, trigo inoculado con S. tritici; 3, trigo proveniente de semillas recubiertas con Th.cc5; 4, trigo proveniente de semillas recubiertas con Th.118; 5, trigo proveniente de semillas recubiertas con Th.cc5 e inoculado con S. tritici; 6, trigo proveniente de semillas recubiertas con Th.118 e inoculado con S. tritici. 91 Resultados Capítulo II Aunque no se obtuvieron diferencias significativas en la actividad proteolítica de los tratamientos, se realizó un western-blot con el anticuerpo específico de IPG para determinar si IPG se expresa en la primera hoja de macollos de trigo (Figura 44). Figura 44. Efecto de S. tritici y Trichoderma spp. sobre la abundancia del monómero de IPG en el FI de primera hoja de macollos. En cada calle se sembró el FI correspondiente a 500 mg de peso fresco, el gel se transfirió a membrana de nitrocelulosa y se incubó con anticuerpo específico anti-IPG. 1, trigo; 2, trigo inoculado con S. tritici; 3, trigo proveniente de semillas recubiertas con Th.cc5; 4, trigo proveniente de semillas recubiertas con Th.118; 5, trigo proveniente de semillas recubiertascon Th.cc5 e inoculado con S. tritici; 6, trigo proveniente de semillas recubiertas con Th.118 e inoculado con S. tritici. Tal como se observa en el western-blot, IPG se expresa en la primera hoja de macollos de trigo. Las modificaciones observadas en el contenido de IPG no se corresponden con los cambios en las actividades proteolíticas analizadas (Figura 43), a excepción de calle 3, en la cual se observa un aumento en la cantidad de IPG que correlaciona con la leve disminución en la actividad proteolítica evidenciada en ese mismo tratamiento en la Figura 43. Con respecto a la severidad de la septoriosis, no se observaron diferencias significativas en el porcentaje de necrosis con los tratamientos (Tabla 6). Sin embargo, el porcentaje de cobertura picnidial en la hoja disminuyó significativamente en las plántulas cuyas semillas habían sido recubiertas con la cepa de Trichoderma Th.118, lo que podría ser adjudicado a un efecto de biocontrol de dicha cepa sobre la septoriosis. Tabla 6. Medida de la cobertura necrótica y picnidial causada por S. tritici en plantas de trigo pretratadas con Trichoderma spp. Los valores fueron expresados en porcentaje y representan el promedio de tres réplicas independientes. Las promedios seguidos por igual letra no son diferentes significativamente (P<0.05) según el test de diferencias mínimas significativas (LSD). 92 Resultados Capítulo II A diferencia de los resultados obtenidos para plántulas por Segarra et al. (2002) y Cordo et al. (2007), en las plantas de trigo inoculadas con S. tritici que presentan síntomas de la enfermedad (Tabla 6), la actividad proteolítica no disminuyo en el FI con respecto a las plantas control (Figura 43). Además, debido a que en ningún tratamiento hubo una modificación de dicha actividad enzimática, no se puede correlacionar la disminución en la severidad de la enfermedad producida por Th.118 con una respuesta bioquímica de defensa asociada a la proteólisis. El experimento descripto anteriormente, fue repetido al año siguiente (2010) con algunas modificaciones. Una de ellas fue que se asperjaron las cepas de Trichoderma harzianum en macollaje (que hasta el presente se aplicaba solamente por recubrimiento en semillas). La otra modificación que se introdujo fue agregar tratamientos a los que se les aplicó fungicida. El objetivo de dicha modificación fue tratar de evitar infecciones de las plantas de trigo con otros patógenos, que de otra manera no se pueden evitar en los ensayos a campo. Previamente se verificó que el fungicida utilizado no modificase ninguna de las actividades enzimáticas evaluadas (dato no mostrado). El resultado de estos nuevos tratamientos se muestra en la Figura 45. Figura 45. Efecto de S. tritici y Trichoderma spp. sobre la actividad proteolítica del FI (A) o inhibitoria de IPG (B). Ambas fueron examinadas mediante SDS-PAGE copolimerizado con gelatina. En cada calle se sembró el FI correspondiente a 150 mg de peso fresco de primera hoja de macollaje. 1, trigo; 2, trigo inoculado con S. tritici; 3 y 3´, trigo proveniente de semillas recubiertas y asperjado en macollaje con Th.cc5 y Th.118 respectivamente; 4 y 4´, trigo proveniente de semillas recubiertas y asperjado en macollaje con Th.cc5 y Th.118 e inoculado con S. tritici; 5 y 5´, idem 3 y 3´ con fungicida; 6 y 6´, idem 4 y 4´ con fungicida; 7, trigo con fungicida e inoculado con S. tritici. Como se puede observar en la Figura 45A, la actividad proteolítica presente en el FI disminuye cuando la planta es inoculada con Septoria tritici (calle 2) con respecto al control (calle 1). Sin embargo, cuando las plantas son pretratadas con ambas cepas de Trichoderma y luego inoculadas con S. tritici (calles 4 y 4´), la actividad proteolítica es semejante a la de las plantas control. 93 Resultados Capítulo II Por otra parte, la disminución de la actividad proteolítica de la calle 2 se correlacionó con el aumento en la actividad inhibitoria de IPG para ese mismo tratamiento (Figura 45 B). En el caso de las plantas a las que se les había aplicado fungicida en plántula y macollaje (calles 5, 6 y 7 del gel de la Figura 45) los resultados son confusos y las modificaciones observadas son opuestas a las obtenidas para los tratamientos sin fungicida. Con respecto a la severidad de la septoriosis en estos experimentos, en general se obtuvieron valores muy bajos de necrosis y cobertura picnidial respecto de los valores habituales para esta enfermedad. A pesar de ello, se observó una disminución del 50% en la necrosis provocada por S. tritici cuando las plantas provenían de semillas recubiertas con la cepa Th.cc5 y asperjadas con la misma cepa de Trichoderma (Tabla 7) y una leve disminución (25%) cuando la cepa utilizada fue la Th.118. Tabla 7. Medida de la cobertura necrótica y picnidial causada por S. tritici en plantas pretratadas con Trichoderma spp. Los valores fueron expresados en porcentaje y representan el promedio de tres réplicas independientes. 94 Resultados Capítulo II Debido a que los tratamientos con ambas cepas de T. harzianum redujeron la necrosis de las hojas provocada por S. tritici y que, las actividades proteolíticas del FI aumentaron a valores semejantes al control en dichos tratamientos, se podría decir que ambas cepas de Trichoderma provocaron una respuesta bioquímica de defensa o de biocontrol sobre S. tritici. Estos resultados concuerdan con los obtenidos previamente en plántulas de trigo (Cordo et al., 2007). Por otra parte, en los tratamientos en los cuales además se aplicó fungicida, es casi imposible establecer una correlación del efecto biocontrolador de Trichoderma sobre S. tritici con la actividad proteolítica del FI e inhibitoria de IPG. Es por esto, que si bien la aplicación de fungicida resulta útil a los efectos de prevenir a campo otras enfermedades distintas a la septoriosis, su uso representa una variable adicional que, sin dudas, complejiza aún más el análisis de las respuestas de defensa. 2.2. Estudios realizados en espigazón La actividad proteolítica del FI fue analizada en la hoja bandera de plantas de trigo en estadio espigazón. Las semillas habían sido recubiertas y las plantas luego asperjadas con dos cepas distintas de Trichoderma (Th.cc5 y Th.118) tanto en macollaje como en espigazón, e inoculadas con Septoria tritici según se indica en la Tabla 8. Estos ensayos fueron llevados a cabo durante el año 2009. Tabla 8. Tratamientos realizados a las plantas de trigo analizadas en estadio espigazón. 95 Resultados Capítulo II Figura 46: Efecto de S.tritici y Trichoderma spp. sobre la actividad proteolítica del FI examinado mediante SDS-PAGE copolimerizado con gelatina. En cada calle se sembró el FI correspondiente a 150 mg de peso fresco de hoja bandera. T1 a T14 los distintos tratamientos (ver tabla 8). Como se observa en la Figura 46, la actividad proteolítica disminuye en casi todos los tratamientos con respecto a las plantas control (T1), salvo para los tratamientos T6, T9 y T13 en los que se observó un ligero aumento. Es decir, el recubrimiento de las semillas con la cepa Th.118 (T6), recubrimiento de las semillas y posterior asperjado en macollaje con Th.cc5 (T9) y nuevo asperjado con esta misma cepa en espigazón (T13) hace que los niveles de actividad proteolítica vuelvan a ser semejantes a los de las plantas control aunque hayan sido infectadas con S. tritici. De la misma manera que en macollaje, se analizó si IPG se expresa en hoja bandera de espigazón mediante ensayos de western-blot y de esa manera se analizaron también las modificaciones en su contenido (Figura 47). Figura 47 Efecto de S. tritici y Trichoderma spp. sobre la abundancia del monómero de IPG en el FI examinado mediante Western blot. En cada calle se sembró el FI correspondiente a 500 mg de peso fresco de hoja bandera, el gel se transfirió a membrana de nitrocelulosa y se incubó con anticuerpo específico anti-IPG. T1 a T14 los distintos tratamientos (ver Tabla 6). Se pudo determinar que IPG está presente en el FI proveniente de la hoja bandera y que su contenido varía en los distintos tratamientos. Si bien las muestras que presentan mayor actividad proteolítica (T6, T9 y T13) son las que poseen menor cantidad de IPG, para el resto de los tratamientos no se pueden establecer correlaciones claras entre las variaciones que se observan en el western blot con las actividades proteolíticas. Por ello, se realizó un gel copolimerizado con gelatina y luego incubado 96 Resultados Capítulo II con tripsina para detectar directamente la actividad inhibitoria de IPG en los distintos tratamientos (Figura 48). Figura 48. Efecto de S.tritici y Trichoderma spp. sobre la actividad inhibitoria de IPG examinado mediante SDS-PAGE copolimerizado con gelatina e incubado con tripsina. En cada calle se sembró el FI correspondiente a 2 gr de peso fresco de hoja bandera. T1 a T13 los distintos tratamientos (ver Tabla 8). La actividad inhibitoria de la proteólisis de IPG fue muy baja en todos los tratamientos, y en algunos casos no llega a visualizarse. Sin embargo, se pudo determinar que en el tratamiento T2 hay un aumento de la actividad de IPG con respecto a T1, que se correlaciona con la disminución de actividad proteolítica observada anteriormente (Figura 46). Dados los bajos valores de actividad inhibitoria de IPG obtenidos, se decidió comparar la abundancia de IPG entre la primera hoja de plántulas y la hoja bandera de espigazón. Para ello se realizó un SDS-PAGE sembrando el FI correspondiente a igual cantidad de peso fresco de hoja de ambos estadios y se inmunodetectó la banda correspondiente a IPG (Figura 49). Figura 49. Comparación de la abundancia de IPG entre primer hoja de plántula de 12d y hoja bandera de espigazón de trigo. El FI de 100 mg de hojas de ambos estadios fue separado en SDS-PAGE y (A) teñido con coomassie blue o (B) transferido a una membrana de nitrocelulosa e inmuno-detectadas con el anticuerpo específico de IPG. Calle 1, FI de primera hoja de trigo de 12 días; Calle 2, FI de hoja bandera de trigo de 130 días. 97 Resultados Capítulo II IPG es casi indetectable con el anticuerpo específico en el FI de hoja bandera (Figura 49 2B), mientras que en el FI de plántulas es reconocido fuertemente. Esta diferencia no se debe a una carga deficiente en proteínas ya que, tal como se observa en el gel teñido con Coomassie (Figura 49 A), la siembra de proteínas totales es aún mayor en hoja bandera. Con estos resultados se puede concluir que a pesar de que IPG es una proteína minoritaria en el FI de hoja bandera de espigazón, su actividad inhibitoria sólo regula las actividades proteolíticas que se modifican cuando el trigo es infectado con S. tritici (T2). Para determinar si las modificaciones en las actividades proteolíticas del FI se relacionan con los mecanismos de defensa de la planta mediados por T. harzianum, evidenciados por una disminución de los síntomas de la septoriosis, se evaluó la cobertura picnidial y necrótica en la hoja bandera (Tabla 9). Tabla 9. Determinación de la cobertura necrótica y picnidial causada por S. tritici en plantas pretratadas con Trichoderma spp. Los valores fueron expresados en porcentaje y representan el promedio de tres réplicas independientes. Las promedios seguidos por igual letra no son diferentes significativamente (P<0.05) según el test de diferencias mínimas significativas (LSD). Como se observa en la Tabla 9, se registran diferencias en los distintos tratamientos, aunque en esta oportunidad el trigo inoculado con S. tritici (T2) manifestó valores muy bajos de severidad (porcentaje de necrosis: 6,67% y de cobertura picnidial: 0,83%). Cabe destacar que en este ensayo hubo una infección adicional observada al principio del estadio espigazón provocada por el hongo patógeno Blumeria graminis f.sp. tritici (dato no mostrado). Dicha infección podría haber actuado como una preinoculación del cultivo, lo que suprimiría los síntomas posteriores de la enfermedad provocados por S. tritici, tal como se ha reportado para otros sistemas experimentales 98 Resultados Capítulo II (Jorgensen et al., 1996; Loughman et al., 1993). A pesar de ello, se puede comparar la efectividad en el biocontrol de la septoriosis de las dos cepas de Trichoderma utilizadas. En los tratamientos en los que además del recubrimiento de la semilla se realizó aplicación aérea de Th.cc5 (T9 y T13), la necrosis y la cobertura picnidial es mucho menor que en los casos donde se aplicó la cepa Th.118 (T10 y T14), por lo que se podría afirmar que la cepa Th.cc5 es más efectiva en el control de la enfermedad. Además, cabe recordar que para los tratamientos T9 y T13 se había determinado un aumento en las actividades proteolíticas del FI, por lo que tal como lo reportado para plántulas (Cordo et al., 2007), se puede afirmar que existe una relación entre el biocontrol ejercido por la cepa Th.cc5 y el aumento de la proteólisis. 99 Discusión Discusión Capítulo II Muchas de las proteínas de la familia de las GLPs han sido vinculadas con la resistencia de las plantas a situaciones de estrés biótico y abiótico. Debido a ello, en esta segunda parte de la tesis se analizó el comportamiento de IPG frente a dichas situaciones de estrés. Uno de los trabajos más relacionado con IPG y el estrés, es el de Vallelian-Bindschedler et al. (1998) en el que caracterizan a una GLP de hoja de cebada HvGER2a (que resultó idéntica a IPG) frente a estrés por altas temperaturas, tratamiento con H2O2 e infección con el hongo patógeno Erysiphe graminis f. sp. hordei. En las tres situaciones estudiadas, se determinó que HvGER2a desaparece del FI luego del estrés y se relocaliza en pared celular, pero la abundancia del ARNm difiere según el tratamiento. En el caso del estrés térmico hay un aumento en la expresión del ARNm, con el tratamiento con H2O2 no hay modificaciones y con la infección fúngica se detecta una disminución. Este ejemplo ilustra la complejidad de los mecanismos de tolerancia a distintos tipos de estrés lo que hace difícil descifrar la participación de esta proteína a partir del mero análisis de los niveles del mensajero y su correspondiente producto. Para IPG, el inhibidor de proteasas tipo germina del FI de hoja de trigo, se ha demostrado su participación en los mecanismos de defensa frente a septoriosis (Segarra et al., 2002). Para avanzar en su caracterización y determinar si participa en respuestas a otros tipos de estrés, se analizó su comportamiento en condiciones de estrés salino en primera y segunda hoja de plántulas de trigo. En la primera hoja se encontró que IPG desaparece del FI y aumenta en la fracción de proteínas unida a la pared celular, tal como en el ejemplo de la GLP de cebada HvGER2a mencionado anteriormente (Vallelian-Bindschedler et al., 1998). Más allá del rol de IPG en la pared, su insolubilización, podría ser indicador de estrés en las hojas de trigo. Con respecto a sus actividades enzimáticas, la actividad inhibitoria de la proteólisis de IPG disminuyó en el FI de ambas hojas y aumentó en la pared, mientras que la actividad SOD sólo aumentó en la fracción de proteínas unida a la pared celular de la primera hoja. Ha sido demostrado que el estrés salino produce un desbalance iónico que provoca toxicidad, estrés osmótico y oxidativo debido a la generación de especies reactivas de oxígeno (Alscher et al., 1997; Cheeseman, 1988; Noctor and Foyer, 1998). Estos efectos, dañinos para las plantas, son contrarrestados por las enzimas antioxidantes: SOD, peroxidasas, glutatión reductasa y catalasa entre otras. La actividad SOD protege a las células de los efectos tóxicos del anión superóxido producido en el estrés oxidativo, ya 100 Discusión Capítulo II que cataliza la reacción de dismutación en peróxido de hidrógeno y oxígeno. En plántulas de trigo de cultivares sensibles o tolerantes al estrés salino, se ha demostrado que la actividad SOD disminuye o aumenta respectivamente en cada caso (Mandhania et al., 2006), por lo que los autores proponen su participación en la tolerancia de las plántulas al estrés salino. De acuerdo a los resultados obtenidos en la presente tesis, se podría afirmar que en las condiciones de estrés salino estudiadas, el aumento en la actividad SOD observado también estaría involucrado en la tolerancia a dicho estrés. Por otra parte, el peróxido de hidrogeno generado podría estar catalizando reacciones de cross-linking involucradas en la síntesis de lignina y por ende en el fortalecimiento de la pared celular (Lane et al., 1993). Previamente en nuestro laboratorio, se analizó la naturaleza de las señales involucradas en la regulación de las actividades proteolíticas e inhibitorias de la proteólisis de IPG. Se observó que el ácido jasmónico (JA) provoca el mismo tipo de respuesta que la generada por S. tritici sobre cultivares resistentes o susceptibles en relación a la actividad de IPG (Segarra et al., 2003), por lo que se podría afirmar que dicha hormona mimetiza el efecto del patógeno (Casalongué et al., 2003). Por otra parte, se ha descripto que cuando las plantas son sometidas a estrés salino se acumulan proteínas de respuesta a JA (Moons et al., 1997). Entonces, la acumulación de IPG en la pared durante la respuesta al estrés salino podría ser un proceso dependiente de JA. De esa manera esta hormona podría ser el punto de conexión entre el estrés abiótico y biótico en la regulación de IPG. Además, en el cultivar de trigo estudiado se evaluaron cambios bioquímicos y morfológicos que evidencien tolerancia al estrés salino. Se encontró que la longitud de la primera hoja no se modifica con este estrés pero que aumenta el contenido de clorofila y prolina. En cambio, en la segunda hoja de trigo se determinó que tanto la longitud como contenido de prolina y clorofila disminuyen. Se ha descripto que bajo condiciones de estrés salino el contenido de clorofila de las hojas generalmente disminuye (Parida and Das, 2005). Sin embargo para el mismo tipo de estrés, Wang y Nil (2000) han reportado un aumento de la clorofila en las hojas de Amaranthus, que en este caso interpretan como signo de tolerancia. También se ha señalado la importancia de la prolina en la respuesta de las plantas al estrés salino (Khedr et al., 2003). Werner y Finkelstein (1995) han descripto que mutantes de Arabidopsis deficientes en la síntesis de prolina, si bien son capaces de germinar en medio salino no pueden continuar con su desarrollo normal. Okuma et al. (2000) encontraron que la aplicación exógena de 101 Discusión Capítulo II prolina mejora el crecimiento de cultivos de células de tabaco estresadas y adjudicaron dicho efecto al rol de la prolina como osmo-protector de enzimas y membranas. Estos resultados junto con la observación de la relocalización de IPG en pared celular y el aumento de sus actividades enzimáticas en este compartimento, ponen de manifiesto la participación de esta proteína multifuncional en la respuesta de tolerancia observada frente al estrés salino. En el caso del estrés por altas temperaturas, también se observó la relocalización de IPG en pared celular. A diferencia del estrés salino, en el estrés térmico se determinó un aumento en la actividad inhibitoria de IPG en el FI. No son muchos los trabajos que vinculan a los inhibidores de proteasas con al tolerancia al estrés abiótico. Sin embargo se ha descripto que el gen del inhibidor de cisteína proteasas de castaño, denominado CsC, es inducido fuertemente en las raíces y hojas de plántulas sometidas a estrés salino y bajas temperaturas, así como también en las raíces luego del estrés por calor (Pernas et al., 2000). Dichos autores sugieren que CsC probablemente estaría involucrado en el mecanismo de respuesta a dichos estreses y su regulación mediada por ácido abscísico o jasmónico dependiendo del tipo de estrés. Por otra parte, se ha descripto la inducción de un inhibidor de proteasas de Brassica napus, bajo condiciones de estrés por sequía y calor. Dicho inhibidor contiene en su secuencia aminoacídica un motivo característico de los inhibidores tipo Kunitz (Satoh et al., 2001). Dado que sólo se cuantificó la actividad inhibitoria en el FI, la misma se debería medir también en pared celular ya que se observó la relocalización de IPG en este compartimento. También se debería analizar si sus otras actividades enzimáticas se modifican en estrés por calor. Hasta el momento los resultados obtenidos sólo indicarían una concordancia con aquellos reportados por Vallelian-Bindschedler et al. (1998) para la GLP HvGER2a de cebada, la cual bajo el mismo estrés se relocaliza en la pared celular. En ese caso, se proponía dicho evento como un marcador de estrés térmico. Con respecto a la caracterización de IPG en estrés biótico, previamente en nuestro laboratorio se había determinado que actividades proteolíticas extracelulares de la hoja de trigo se modificaban frente al ataque del hongo patógeno Septoria tritici, inhibían la germinación de las conidiosporas del hongo y eran reguladas por el inhibidor IPG (Segarra et al., 2002; Segarra et al., 2003). Por ello se postuló su participación en los mecanismos de defensa frente a este hongo patógeno. La “mancha de la hoja” o septoriosis del trigo, provocada por S. tritici, es una de las enfermedades de mayor distribución en la región triguera de América del Sur. Esta enfermedad es por 102 Discusión Capítulo II naturaleza endémica y, según las condiciones climáticas, su aparición es cíclica (Cunfer, 1999). Cuando el ataque foliar es intenso se reduce la superficie fotosintética, se producen granos de tamaño reducido y por lo tanto se afecta la calidad del cultivo de trigo (Cordo and Marechal, 1990). Para controlar la enfermedad se ha recurrido habitualmente al uso de fungicidas (Cook et al., 1999). Sin embargo debido a la alta contaminación que producen los agroquímicos, se están buscando alternativas ecológicas para el control de las enfermedades, como por ejemplo el control biológico. En relación a estas estrategias alternativas de manejo de la septoriosis, Cordo et al. (2007) demostraron que las plántulas de trigo pretratadas con Trichoderma harzianum mostraban menos síntomas luego de la infección con S. tritici, y esto se correlacionaba con una disminución en la actividad inhibitoria de IPG y un aumento de la actividad proteolítica del FI, cuya actividad antifúngica ya había sido demostrada (Segarra et al., 2002). En el presente trabajo de tesis se evaluó en ensayos a campo la eficiencia y mecanismo de acción de Trichoderma spp para controlar la mancha de la hoja de trigo. Desde el punto de vista experimental, este tipo de ensayos presentan el inconveniente de la aparición de variables difíciles de controlar (temperatura, humedad ambiente, disponibilidad de agua) y presencia de patógenos naturales diferentes al estudiado. Además, para tener las replicas necesarias de los experimentos, se necesita mínimamente de tres años. A pesar de estos inconvenientes, se pudo determinar tanto en macollaje como en espigazón que la actividad proteolítica del FI disminuye respecto del control en el cultivar de trigo susceptible cuando las plantas son infectadas con S. tritici, resultados semejantes a los obtenidos previamente (Segarra et al., 2002). Dichos valores de actividad proteolítica vuelven a ser semejantes al control cuando las semillas fueron recubiertas, y las plantas asperjadas en macollaje y luego en espigazón con Trichoderma spp. Además, en algunos tratamientos, se demostró que las modificaciones de la actividad proteolítica se debían a su regulación por la actividad inhibitoria de IPG, tal como se había observado previamente en plántulas (Cordo et al., 2007). A diferencia de los ensayos en plántulas, en los cuales IPG es la proteína mayoritaria de la primera hoja de trigo, en espigazón esta proteína es casi indetectable en el FI de la hoja bandera. A pesar de ello, su actividad inhibitoria sigue siendo fundamental en la regulación de las proteasas. Estos resultados sugieren que, tal como se ha descripto en otros sistemas experimentales (Hanson and Howell, 2004; Koike et al., 2001), T. harzianum determina en plantas de trigo inoculadas con S. tritici una respuesta bioquímica de defensa. Estos resultados permiten afirmar que el control biológico mediado por T. harzianum a través 103 Discusión Capítulo II de la regulación de las actividades proteolíticas del FI constituye una herramienta potencial de gran importancia en relación al manejo de la septoriosis. Con respecto a la localización de IPG en distintos órganos y estadios de desarrollo del cultivo de trigo, se confirmó su presencia en hojas de plántulas, de macollos y en la hoja bandera del estadio espigazón. Además, se comprobó que en este último estadio, la abundancia de IPG es mucho menor que en los anteriores. También se trató de determinar, a través de ensayos de western-blot, si IPG estaba presente en embriones maduros de semilla de trigo, tal como se describió para las primeras germinas halladas (Thompson and Lane, 1980). En este caso, el resultado no fue concluyente, dado que el anticuerpo reconoció específicamente una proteína de masa molecular diferente a la del monómero de IPG. Para confirmar si se trata de una isoforma de IPG debería realizarse un Northern-blot con la sonda de ADNc de IPG, y así determinar fehacientemente si el mensajero se expresa en dicho órgano. En conclusión, los resultados presentados sugieren la participación de IPG tanto en los mecanismos de defensa de las plantas de trigo frente a la septoriosis como también en la tolerancia a distintos tipos de estrés abiótico. Sin embargo para confirmar este rol, se deberían analizar plantas transformadas genéticamente en las cuales el gen de IPG se encuentre silenciado o sobreexpresado, y así evaluar su aporte e importancia en las respuestas a distintas condiciones de estrés. Los resultados que se obtuviesen de estos experimentos, no sólo determinarían un notable avance en el conocimiento de las proteínas tipo germinas, sino que también constituirían un aporte fundamental para el mejoramiento del cultivo de trigo en relación a las demandas agronómicas. 104 Conclusiones Conclusiones Se obtuvo la secuencia aminoacídica de IPG, lo que permitió identificarla como una GLP con actividad pirofosfatasa/fosfodiesterasa. Pese a que su actividad como inhibidor de proteasas resultó novel en las GLPs, no se trata de una nueva proteína dentro de esta familia. IPG es una proteína multifuncional con al menos tres actividades enzimáticas: inhibidor de proteasas, superóxido dismutasa y pirofosfatasa/fosfodiesterasa. La Arg69 de IPG es necesaria para conservar la actividad de inhibidor de serina proteasas, y dicho aminoácido es periférico en la estructura tridimensional predicha mediante herramientas bioinformáticas. Este hallazgo permitiría relacionar a IPG con los inhibidores de proteasas del tipo Kunitz ya que presentan el mismo aminoácido expuesto en la estructura del sitio activo. Se analizó la presencia de IPG en distintos estadios del desarrollo del trigo y mediante ensayos de western-blot se comprobó su presencia en primera y segunda hoja de plántulas y en hojas provenientes de plantas en macollaje y espigazón. En los dos estreses abióticos estudiados, salinidad y calor, se observó una relocalización de IPG en la pared celular de las hojas de trigo y un aumento de sus actividades enzimáticas en dicho compartimento. Además, en el caso del estrés salino, se pudo establecer una correlación entre la relocalización de IPG y la tolerancia de las plántulas de trigo evidenciada por la modificación de los parámetros fenotípicos y bioquímicos estudiados. 105 Conclusiones Se constató, mediante ensayos a campo, que en las plantas de trigo susceptibles a la septoriosis hubo una disminución de la actividad inhibitoria de IPG cuando las mismas fueron pretratradas con el hongo biocontrolador Trichoderma harzianum. Se observó asimismo un concomitante aumento de la actividad proteolítica del FI, cuya capacidad antifúngica fue previamente demostrada. Estos resultados correlacionaron con la disminución de los síntomas de la enfermedad, lo que permite asignar a IPG un rol en el control biológico de la septoriosis mediado por T. harzianum. 106 Bibliografía Bibliografía Agastian P, Kingsley SJ, Vivekanandan M. 2000. Effect of Salinity on Photosynthesis and Biochemical Characteristics in Mulberry Genotypes. Photosynthetica 38, 287-290. Al-Lazikani B, Jung J, Xiang Z, Honig B. 2001. Protein structure prediction. Current Opinion in Chemical Biology 5, 51-56. Ali-Dinar H, Ebert G, Lüdders P. 1999. Growth, chlorophyll content, photosynthesis and water relations in guava (Psidium guajava L.) under salinity and different nitrogen supply. Gartenbauwissenschaft 64, 54-59. Alscher RG, Donahue JL, Cramer CL. 1997. Reactive oxygen species and antioxidants: Relationships in green cells. Physiologia Plantarum 100, 224-233. Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ. 1990. Basic local alignment search tool. J Mol Biol 215, 403-410. Antão CM, Malcata FX. 2005. Plant serine proteases: biochemical, physiological and molecular features. Plant Physiology and Biochemistry 43, 637-650. Apel K, Hirt H. 2004. Reactive oxygen species: metabolism, oxidative stress, and signal transduction. Annu Rev Plant Biol 55, 373-399. Apostol I, Heinstein PF, Low PS. 1989. Rapid Stimulation of an Oxidative Burst during Elicitation of Cultured Plant Cells : Role in Defense and Signal Transduction. Plant Physiology 90, 109-116. Arnold K, Bordoli L, Kopp J, Schwede T. 2006. The SWISS-MODEL workspace: a web-based environment for protein structure homology modelling. Bioinformatics 22, 195-201. Baroja-Fernández E, Zandueta-Criado A, Rodríguez-López M, Akazawa T, Pozueta-Romero J. 2000. Distinct isoforms of ADPglucose pyrophosphatase and ADPglucose pyrophosphorylase occur in the suspension-cultured cells of sycamore (Acer pseudoplatanus L.). FEBS Letters 480, 277-282. Barrett AJ, ed. 1986. The classes of proteolyic enzymes. Boca Raton, Florida: CRC Press. Barrett AJ, Rawlings ND, O'Brien EA. 2001. The MEROPS database as a protease information system. J Struct Biol 134, 95-102. Bates LS, Waldren RP, Teare ID. 1973. Rapid determination of free proline for waterstress studies. Plant and Soil 39, 205-207. Batista IFC, Oliva MLV, Araujo MS, Sampaio MU, Richardson M, Fritz H, Sampaio CAM. 1996. Primary structure of a Kunitz-type trypsin inhibitor from Enterolobium contortisiliquum seeds. Phytochemistry 41, 1017-1022. Beauchamp C, Fridovich I. 1971. Superoxide dismutase: Improved assays and an assay applicable to acrylamide gels. Analytical Biochemistry 44, 276-287. 107 Bibliografía Beers EP, Jones AM, Dickerman AW. 2004. The S8 serine, C1A cysteine and A1 aspartic protease families in Arabidopsis. Phytochemistry 65, 43-58. Benchekroun A, Michaud D, Nguyen-Quoc B, Overney S, Desjardins Y, Yelle S. 1995. Synthesis of active oryzacystatin I in transgenic potato plants. Plant Cell Reports 14, 585-588. Berna A, Bernier F. 1999. Regulation by biotic and abiotic stress of a wheat germin gene encoding oxalate oxidase, a H2O2-producing enzyme. Plant Mol Biol 39, 539-549. Bernier F, Berna A. 2001. Germins and germin-like proteins: Plant do-all proteins. But what do they do exactly? Plant Physiology and Biochemistry 39, 545-554. Birk Y. 1985. The Bowman-Birk inhibitor. Trypsin- and chymotrypsin-inhibitor from soybeans. International Journal of Peptide and Protein Research 25, 113-131. Birnboim HC, Doly J. 1979. A rapid alkaline extraction procedure for screening recombinant plasmid DNA. Nucleic Acids Res 7, 1513-1523. Blum H, Beier H, Gross HJ. 1987. Improved silver staining of plant proteins, RNA and DNA in polyacrylamide gels. Electrophoresis 8, 93-99. Bode W, Huber R. 1992. Natural protein proteinase inhibitors and their interaction with proteinases. Eur J Biochem 204, 433-451. Bode W, Huber R. 2000. Structural basis of the endoproteinase-protein inhibitor interaction. Biochim Biophys Acta 1477, 241-252. Bornstein P, Balian G. 1977. Cleavage at Asn-Gly bonds with hydroxylamine. Methods Enzymol 47, 132-145. Bostock RM. 2005. Signal crosstalk and induced resistance: straddling the line between cost and benefit. Annu Rev Phytopathol 43, 545-580. Brewer CF, Riehm JP. 1967. Evidence for possible nonspecific reactions between Nethylmaleimide and proteins. Analytical Biochemistry 18, 248-255. Bringezu K, Lichtenberger O, Leopold I, Neumann D. 1999. Heavy metal tolerance of Silene vulgaris. Journal of Plant Physiology 154, 536-546. Broadway RM, Duffey SS. 1986. Plant proteinase inhibitors: Mechanism of action and effect on the growth and digestive physiology of larval Heliothis zea and Spodoptera exiqua. Journal of Insect Physiology 32, 827-833. Brown JA, Li D, Alic M, Gold MH. 1993. Heat Shock Induction of Manganese Peroxidase Gene Transcription in Phanerochaete chrysosporium. Appl Environ Microbiol 59, 4295-4299. Brown WE, Ryan CA. 1984. Isolation and characterization of a wound-induced trypsin inhibitor from alfalfa leaves. Biochemistry 23, 3418-3422. 108 Bibliografía Busch W, Wunderlich M, Schöffl F. 2005. Identification of novel heat shock factordependent genes and biochemical pathways in Arabidopsis thaliana. The Plant Journal 41, 1-14. Caliskan M. 2009. Salt stress causes a shift in the localization pattern of germin gene expression. Genet Mol Res 8, 1250-1256. Carter C, Thornburg RW. 2000. Tobacco nectarin I. Purification and characterization as a germin-like, manganese superoxide dismutase implicated in the defensE of floral reproductive tissues. J Biol Chem 275, 36726-36733. Casalongué C, Segarra C, Pinedo M, Conde R. 2003. Jasmonic acid in wheat leaves: control of proteolytic and inhibitor protease activities. American Society of Plant Biologist Congress. Honolulu - USA. Ceciliani F, Bortolotti F, Menegatti E, Ronchi S, Ascenzi P, Palmieri S. 1994. Purification, inhibitory properties, amino acid sequence and identification of the reactive site of a new serine proteinase inhibitor from oil-rape (itBrassica napus) seed. FEBS Letters 342, 221-224. Coffeen WC, Wolpert TJ. 2004. Purification and Characterization of Serine Proteases That Exhibit Caspase-Like Activity and Are Associated with Programmed Cell Death in Avena sativa. Plant Cell 16, 857-873. Cook, Hims, Vaughan. 1999. Effects of fungicide spray timing on winter wheat disease control. Plant Pathology 48, 33-50. Cordo CA, Marechal LR. 1990. Enzimas hidrolíticas detectadas en cultivo filtrado de Septoria tritici (Teleomorfo, Mycosphaerella graminicola). Summa phytopathol 16, 111120. Cordo CA, Monaco CI, Segarra CI, Simon MR, Mansilla AY, Perelló AE, Kripelz NI, Bayo D, Conde RD. 2007. Trichoderma spp. as elicitors of wheat plant defense responses against Septoria tritici. Biocontrol Science and Technology 17, 687-698. Cordoba OL, Linskens SB, Dacci E, Santome JA. 1997. 'In gel' cleavage with cyanogen bromide for protein internal sequencing. J Biochem Biophys Methods 35, 110. Cuming AC, Lane BG. 1979. Protein Synthesis in Imbibing Wheat Embryos. European Journal of Biochemistry 99, 217-224. Cunfer BM. 1999. Stagonospora and Septoria pathogens of cereals: The infection process. In: Van Ginkel MM, A; Krupinsky, J., ed. Stagonospora and Septoria disease of cereals: a compilation of global research. Mexico: CIMMYT, 41-45. Custers JH, Harrison SJ, Sela-Buurlage MB, van Deventer E, Lageweg W, Howe PW, van der Meijs PJ, Ponstein AS, Simons BH, Melchers LS, Stuiver MH. 2004. Isolation and characterisation of a class of carbohydrate oxidases from higher plants, with a role in active defence. Plant J 39, 147-160. 109 Bibliografía Chang H, Siegel BZ, Siegel SM. 1984. Salinity-induced changes in isoperoxidases in taro Colocasia esculenta. Phytochemistry 23, 233-235. Chaplin MF. 1976. The use of ninhydrin as a reagent for the reversible modification of arginine residues in proteins. Biochem J 155, 457-459. Chaput M, Claes V, Portetelle D, Cludts I, Cravador A, Burny A, Gras H, Tartar A. 1988. The neurotrophic factor neuroleukin is 90% homologous with phosphohexose isomerase. Nature 332, 454-455. Cheeseman JM. 1988. Mechanisms of Salinity Tolerance in Plants. Plant Physiology 87, 547-550. Chen P, Rose J, Love R, Wei CH, Wang BC. 1992. Reactive sites of an anticarcinogenic Bowman-Birk proteinase inhibitor are similar to other trypsin inhibitors. Journal of Biological Chemistry 267, 1990-1994. Christeller JT. 2005. Evolutionary mechanisms acting on proteinase inhibitor variability. FEBS J 272, 5710-5722. Christensen AB, Thordal-Christensen H, Zimmermann G, Gjetting T, Lyngkjaer MF, Dudler R, Schweizer P. 2004. The germinlike protein GLP4 exhibits superoxide dismutase activity and is an important component of quantitative resistance in wheat and barley. Mol Plant Microbe Interact 17, 109-117. Dattagupta JK, Podder A, Chakrabarti C, Sen U, Mukhopadhyay D, Dutta SK, Singh M. 1999. Refined crystal structure (2.3 A) of a double-headed winged bean alpha-chymotrypsin inhibitor and location of its second reactive site. Proteins: Structure, Function and Genetics 35, 321-331. De Leo F, Volpicella M, Licciulli F, Liuni S, Gallerani R, Ceci LR. 2002. PLANTPls: A database for plant protease inhibitors and their genes. Nucleic Acids Research 30, 347-348. Downing WL, Mauxion F, Fauvarque MO, Reviron MP, De Vienne D, Vartanian N, Giraudat J. 1992. A Brassica napus transcript encoding a protein related to the Kunitz protease inhibitor family accumulates upon water stress in leaves, not in seeds. Plant Journal 2, 685-693. Dratewka-Kos E, Rahman S, Grzelczak ZF, Kennedy TD, Murray RK, Lane BG. 1989. Polypeptide structure of germin as deduced from cDNA sequencing. J Biol Chem 264, 4896-4900. Dunwell JM. 1998. Cupins: a new superfamily of functionally diverse proteins that include germins and plant storage proteins. Biotechnol Genet Eng Rev 15, 1-32. Dunwell JM, Culham A, Carter CE, Sosa-Aguirre CR, Goodenough PW. 2001. Evolution of functional diversity in the cupin superfamily. Trends Biochem Sci 26, 740746. 110 Bibliografía Dunwell JM, Khuri S, Gane PJ. 2000. Microbial Relatives of the Seed Storage Proteins of Higher Plants: Conservation of Structure and Diversification of Function during Evolution of the Cupin Superfamily. Microbiol Mol Biol Rev 64, 153-179. Eckelkamp C, Ehmann B, Schöpfer P. 1993. Wound-induced systemic accumulation of a transcript coding for a Bowman-Birk trypsin inhibitor-related protein in maize (Zea mays L.) seedlings. FEBS Letters 323, 73-76. Eisenberg D, Luthy R, Bowie JU. 1997. VERIFY3D: assessment of protein models with three-dimensional profiles. Methods Enzymol 277, 396-404. El-Shamei Z, Wu JW, Haard NF. 1996. Influence of wound injury on accumulation of proteinase inhibitors in leaf and stem tissues of two processing tomato cultivars. Journal of Food Biochemistry 20, 155-171. El-Shora HM, Youssef MM. 2008. Identification of catalytically essential amino acid residues and immobilization of glutamate dehydrogenase from Rumex cotyledons. Asian J. Biochem. 3, 320-329. Esfandiari E, Shekari F, Shekari F, Esfandiar M. 2007. The effect of salt stress on antioxidant enzymes activity and lipid peroxidation on the wheat seedlings. Not Bot Hort Agrobot Cluj 35, 48–56. Faik P, Walker JI, Redmill AA, Morgan MJ. 1988. Mouse glucose-6-phosphate isomerase and neuroleukin have identical 3' sequences. Nature 332, 455-457. Feder ME, Hofmann GE. 1999. Heat-shock proteins, molecular chaperones, and the stress response: evolutionary and ecological physiology. Annu Rev Physiol 61, 243-282. Fernandez-Patrón C, Castellanos-Serra L, Rodriguez P. 1992. Reverse staining of sodium dodecyl sulfate polyacrylamide gels by imidazole-zinc salts: sensitive detection of unmodified proteins. Biotechniques 12, 564-573. Flowers TJ, Troke PF, Yeo AR. 1977. The Mechanism of Salt Tolerance in Halophytes. Annual Review of Plant Physiology 28, 89-121. Fridovich I. 1986. Biological effects of the superoxide radical. Arch Biochem Biophys 247, 1-11. Gadallah MAA. 1999. Effects of Proline and Glycinebetaine on Vicia Faba Responses to Salt Stress. Biologia Plantarum 42, 249-257. Gaddour K, Vicente-Carbajosa J, Lara P, Isabel-Lamoneda I, Díaz I, Carbonero P. 2001. A constitutive cystatin-encoding gene from barley (Icy) responds differentially to abiotic stimuli. Plant Mol Biol 45, 599-608. Gane PJ, Dunwell JM, Warwicker J. 1998. Modeling based on the structure of vicilins predicts a histidine cluster in the active site of oxalate oxidase. J Mol Evol 46, 488-493. Gatehouse AMR, Boulter D. 1983. Assessment of the antimetabolic effects of trypsin inhibitors from cowpea (Vigna unguiculata) and other legumes on development of the 111 Bibliografía bruchid beetle Callosobruchus maculatus. Journal of the Science of Food and Agriculture 34, 345-350. Gatehouse JA, Gatehouse AMR, Brown D. 2000. Control of phytophagous insect pests using serine proteinase inhibitors. In: Michaud D, ed. Recombinant Protease Inhibitors in Plants. Texas, USA: Landes Bioscience 9-26. Godfrey D, Able AJ, Dry IB. 2007. Induction of a grapevine germin-like protein (VvGLP3) gene is closely linked to the site of Erysiphe necator infection: a possible role in defense? Mol Plant Microbe Interact 20, 1112-1125. Golderer G, Gröbner P. 1991. ADP-ribosylation of core histones and their acetylated subspecies. Biochemical Journal 277, 607-610. Goyal K, Walton LJ, Tunnacliffe A. 2005. LEA proteins prevent protein aggregation due to water stress. Biochem J 388, 151-157. Grasberger BL, Clore GM, Gronenborn AM. 1994. High-resolution structure of Ascaris trypsin inhibitor in solution: direct evidence for a pH-induced conformational transition in the reactive site. 2, 669-678. Greenway H, Munns R. 1980. Mechanisms of Salt Tolerance in Nonhalophytes. Annual Review of Plant Physiology 31, 149-190. Grzelczak Z, Rahman S, Kennedy T, Lane B. 1985. Germin. Compartmentation of the protein, its translatable mRNA and its biosynthesis among roots, stems and leaves of wheat seedlings. Can J Biochem Cell Biol 63, 1003–1013. Gucciardo S, Wisniewski JP, Brewin NJ, Bornemann S. 2007. A germin-like protein with superoxide dismutase activity in pea nodules with high protein sequence identity to a putative rhicadhesin receptor. Journal of Experimental Botany 58, 1161-1171. Gurney ME, Apatoff BR, Spear GT, Baumel MJ, Antel JP, Bania MB, Reder AT. 1986a. Neuroleukin: a lymphokine product of lectin-stimulated T cells. Science 234, 574-581. Gurney ME, Heinrich SP, Lee MR, Yin HS. 1986b. Molecular cloning and expression of neuroleukin, a neurotrophic factor for spinal and sensory neurons. Science 234, 566-574. Haldar UC, Saha SK, Beavis RC, Sinha NK. 1996. Trypsin inhibitors from ridged gourd (Luffa acutangula Linn.) seeds: purification, properties, and amino acid sequences. J Protein Chem 15, 177-184. Hanson LE, Howell CR. 2004. Elicitors of Plant Defense Responses from Biocontrol Strains of Trichoderma viren. Phytopathology 94, 171-176. Haq SK, Atif SM, Khan RH. 2004. Protein proteinase inhibitor genes in combat against insects, pests, and pathogens: Natural and engineered phytoprotection. Archives of Biochemistry and Biophysics 431, 145-159. Hawkes R. 1986. The dot immunobinding assay. Methods Enzymol 121, 484-491. 112 Bibliografía Heibges A, Salamini F, Gebhardt C. 2003. Functional comparison of homologous members of three groups of Kunitz-type enzyme inhibitors from potato tubers (Solanum tuberosum L.). Molecular Genetics and Genomics 269, 535-541. Hernández J, Jimenez A, Mullineaux P, Sevilla F. 2000. Tolerance of pea (Pisum sativum L.) to long-term salt stress is associated with induction of antioxidant defences. Plant, Cell &#38; Environment 23, 853-862. Hernández J, Olmos E, Corpas F, Sevilla F, del Rio L. 1995. Salt-induced oxidative stress in chloroplasts of pea plants. Plant Science 105, 151-167. Hernández JA, Campillo A, Jiménez A, Alarcón JJ, Sevilla F. 1999. Response of antioxidant systems and leaf water relations to NaCl stress in pea plants. New Phytologist 141, 241-251. Herouart D, Van Montagu M, Inze D. 1994. Developmental and environmental regulation of the Nicotiana plumbaginifolia cytosolic Cu/Zn-superoxide dismutase promoter in transgenic tobacco. Plant Physiol 104, 873-880. Heussen C, Dowdle EB. 1980. Electrophoretic analysis of plasminogen activators in polyacrylamide gels containing sodium dodecyl sulfate and copolymerized substrates. Analytical Biochemistry 102, 196-202. Hilder VA, Gatehouse AMR, Sheerman SE, Barker RF, Boulter D. 1987. A novel mechanism of insect resistance engineered into tobacco. Nature 330, 160-163. Himmelbach A, Liu L, Zierold U, Altschmied L, Maucher H, Beier F, Müller D, Hensel G, Heise A, Schützendübel A, Kumlehn J, Schweizer P. 2010. Promoters of the barley germin-like GER4 gene cluster enable strong transgene expression in response to pathogen attack. Plant Cell 22, 937-952. Howarth CJ. 1991. Molecular responses of plants to an increased incidence of heat shock. Plant, Cell & Environment 14, 831-841. Huberts DH, van der Klei IJ. 2010. Moonlighting proteins: an intriguing mode of multitasking. Biochim Biophys Acta 1803, 520-525. Hung CH. 1994. Inactivation of Acacia confusa trypsin inhibitor by site-specific mutagenesis. FEBS Letters 353, 312-314. Huntington JA, Read RJ, Carrell RW. 2000. Structure of a serpin-protease complex shows inhibition by deformation. Nature 407, 923-926. Hurkman WJ, Lane BG, Tanaka CK. 1994. Nucleotide sequence of a transcript encoding a germin-like protein that is present in salt-stressed barley (Hordeum vulgare L.) roots. Plant Physiol 104, 803-804. Hurkman WJ, Tanaka CK. 1996. Effect of Salt Stress on Germin Gene Expression in Barley Roots. Plant Physiol 110, 971-977. Hurkman WJ, Tao HP, Tanaka CK. 1991. Germin-Like Polypeptides Increase in Barley Roots during Salt Stress. Plant Physiol 97, 366-374. 113 Bibliografía Imlay JA, Linn S. 1988. DNA damage and oxygen radical toxicity. Science 240, 13021309. Irie K, Hosoyama H, Takeuchi T, Iwabuchi K, Watanabe H, Abe M, Abe K, Arai S. 1996. Transgenic rice established to express corn cystatin exhibits strong inhibitory activity against insect gut proteinases. Plant Mol Biol 30, 149-157. Irving JA, Pike RN, Dai W, Bromme D, Worrall DM, Silverman GA, Coetzer TH, Dennison C, Bottomley SP, Whisstock JC. 2002a. Evidence that serpin architecture intrinsically supports papain-like cysteine protease inhibition: engineering alpha(1)antitrypsin to inhibit cathepsin proteases. Biochemistry 41, 4998-5004. Irving JA, Shushanov SS, Pike RN, Popova EY, Bromme D, Coetzer TH, Bottomley SP, Boulynko IA, Grigoryev SA, Whisstock JC. 2002b. Inhibitory activity of a heterochromatin-associated serpin (MENT) against papain-like cysteine proteinases affects chromatin structure and blocks cell proliferation. J Biol Chem 277, 1319213201. Ishikawa A, Ohta S, Matsuoka K, Hattori T, Nakamura K. 1994. A family of potato genes that encode Kunitz-type proteinase inhibitors: Structural comparisons and differential expression. Plant and Cell Physiology 35, 303-312. Jaikaran AS, Kennedy TD, Dratewka-Kos E, Lane BG. 1990. Covalently bonded and adventitious glycans in germin. J Biol Chem 265, 12503-12512. Joanitti GA, Freitas SM, Silva LP. 2006. Proteinaceous protease inhibitors: Structural features and multiple functional faces. Current Enzyme Inhibition 2, 199-217. Jorgensen, H. J L, Andresen, H, Smedegaard P, V. 1996. Control of Drechslera teres and other barley pathogens by preinoculation with Bipolaris maydis and Septoria nodorum. St. Paul, MN, ETATS-UNIS: American Phytopathological Society. Joubert FJ, Heussen C, Dowdle EB. 1985. The complete amino acid sequence of trypsin inhibitor DE-3 from Erythrina latissima seeds. Journal of Biological Chemistry 260, 12948-12953. Khandelwal A, Chandu D, Roe CM, Kopan R, Quatrano RS. 2007. Moonlighting activity of presenilin in plants is independent of gamma-secretase and evolutionarily conserved. Proc Natl Acad Sci U S A 104, 13337-13342. Khanna-Chopra R, Sabarinath S. 2004. Heat-stable chloroplastic Cu/Zn superoxide dismutase in Chenopodium murale. Biochem Biophys Res Commun 320, 1187-1192. Khavari-Nejad RA, Chaparzadeh N. 1998. The effects of NaCl and CaCl2 on photosynthesis and growth of alfalfa plants. Photosynthetica 35, 461-466. Khedr AHA, Abbas MA, Wahid AAA, Quick WP, Abogadallah GM. 2003. Proline induces the expression of salt-stress-responsive proteins and may improve the adaptation of Pancratium maritimum L. to salt-stress. Journal of Experimental Botany 54, 2553-2562. 114 Bibliografía Kim S-H, Hara S, Hase S, Ikenaka T, Toda H, Kitamura K, Kaizuma N. 1985. Comparative Study on Amino Acid Sequences of Kunitz-Type Soybean Trypsin Inhibitors, Tia, Tib, and Tic. J Biochem 98, 435-448. Kim S-H, Park E-J, Yoon S-S, Choi J-D. 2003. An active site arginine residue in tobacco acetolactate synthase. Seoul, COREE, REPUBLIQUE DE: Korean Chemical Society. Koike N, Hyakumachi M, Kageyama K, Tsuyumu S, Doke N. 2001. Induction of Systemic Resistance in Cucumber against Several Diseases by Plant Growth-promoting Fungi: lignification and Superoxide Generation. European Journal of Plant Pathology 107, 523-533. Kondo K, Yamada K, Nakagawa A, Takahashi M, Morikawa H, Sakamoto A. 2008. Molecular characterization of atmospheric NO2-responsive germin-like proteins in azalea leaves. Biochem Biophys Res Commun 377, 857-861. Kovtun Y, Chiu WL, Tena G, Sheen J. 2000. Functional analysis of oxidative stressactivated mitogen-activated protein kinase cascade in plants. Proc Natl Acad Sci U S A 97, 2940-2945. Kumar RS, Suresh CG, Pundle A, Prabhune A. 2004. Evidence for the involvement of arginyl residue at the active site of penicillin G acylase from Kluyvera citrophila. Biotechnol Lett 26, 1601-1606. Kuroda, M, Ishimoto, Suzuki, K, Kondo, H, Abe, Kitamura, Arai, S. 1996. Oryzacystatins exhibit growth-inhibitory and lethal effects on different species of bean insect pests, Callosobruchus chinensis (Coleoptera) and Riptortus clavatus (Hemiptera). Tokyo, JAPON: Japan Society for Bioscience Biotechnology and Agrochemistry. Kuznetsov VV, Shevyakova NI. 1997. Stress responses of tobacco cells to high temperature and salinity. Proline accumulation and phosphorylation of polypeptides. Physiologia Plantarum 100, 320-326. Laemmli UK. 1970. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature 227, 680-685. Lane B, Grzelczak Z, Kennedy T, Hew C, Joshi S. 1986. Preparation and analysis of mass amounts of germin: demonstration that the protein which signals the onset of growth in germinating wheat is a glycoprotein. Biochemistry and Cell Biology 65, 354– 362. Lane BG. 1994. Oxalate, germin, and the extracellular matrix of higher plants. FASEB J 8, 294-301. Lane BG, Dunwell JM, Ray JA, Schmitt MR, Cuming AC. 1993. Germin, a protein marker of early plant development, is an oxalate oxidase. J Biol Chem 268, 1223912242. Larkindale J, Vierling E. 2008. Core genome responses involved in acclimation to high temperature. Plant Physiol 146, 748-761. 115 Bibliografía Laskowski M, Kato I. 1980. Protein inhibitors of proteinases. Annu Rev Biochem 49, 593-626. Laskowski M, Qasim MA. 2000. What can the structures of enzyme-inhibitor complexes tell us about the structures of enzyme substrate complexes? Biochimica et Biophysica Acta (BBA) - Protein Structure and Molecular Enzymology 1477, 324-337. Lawrence PK, Koundal KR. 2002. Plant protease inhibitors in control of phytophagous insects. Electronic Journal of Biotechnology 5, 121-143. Lecardonnel A, Chauvin L, Jouanin L, Beaujean A, Prévost G, Sangwan-Norreel B. 1999. Effects of rice cystatin I expression in transgenic potato on Colorado potato beetle larvae. Plant Science 140, 71-79. Ledoigt G, Griffaut B, Debiton E, Vian C, Mustel A, Evray G, Maurizis JC, Madelmont JC. 2006. Analysis of secreted protease inhibitors after water stress in potato tubers. International Journal of Biological Macromolecules 38, 268-271. Lee CF, Lin JY. 1995. Amino acid sequences of trypsin inhibitors from the melon Cucumis melo. J Biochem 118, 18-22. Lee D-G, Ahsan N, Lee S-H, Kang KY, Bahk JD, Lee I-J, Lee B-H. 2007. A proteomic approach in analyzing heat-responsive proteins in rice leaves. Proteomics 7, 3369-3383. Lee T-M, Liu C-H. 1999. Correlation of decreased calcium contents with proline accumulation in the marine green macroalga Ulva fasciata exposed to elevated NaCl contents in seawater. Journal of Experimental Botany 50, 1855-1862. Legendre L, Rueter S, Heinstein PF, Low PS. 1993. Characterization of the Oligogalacturonide-Induced Oxidative Burst in Cultured Soybean (Glycine max) Cells. Plant Physiology 102, 233-240. Leung D, Abbenante G, Fairlie DP. 2000. Protease inhibitors: current status and future prospects. J Med Chem 43, 305-341. Liao H, Ren W, Kang Z, Jiang JH, Zhao XJ, Du LF. 2007. A trypsin inhibitor from Cassia obtusifolia seeds: Isolation, characterization and activity against Pieris rapae. Biotechnology Letters 29, 653-658. Lim CJ, Yang KA, Hong JK, Choi JS, Yun DJ, Hong JC, Chung WS, Lee SY, Cho MJ, Lim CO. 2006. Gene expression profiles during heat acclimation in Arabidopsis thaliana suspension-culture cells. J Plant Res 119, 373-383. Lin G, Bode W, Huber R, Chi C, Engh RA. 1993. The 0.25-nm X-ray structure of the Bowman-Birk-type inhibitor from mung bean in ternary complex with porcine trypsin. European Journal of Biochemistry 212, 549-555. Lorenzo O, Solano R. 2005. Molecular players regulating the jasmonate signalling network. Curr Opin Plant Biol 8, 532-540. 116 Bibliografía Lou Y, Baldwin IT. 2006. Silencing of a germin-like gene in Nicotiana attenuata improves performance of native herbivores. Plant Physiol 140, 1126-1136. Loughman R, Wilson RE, Thomas GJ. 1993. The influence of disease complexes involving Leptosphaeria (Septoria) nodorum on detection of resistance to three leaf spot diseases in wheat. Euphytica 72, 31-42. Lovell SC, Davis IW, Arendall WB, 3rd, de Bakker PI, Word JM, Prisant MG, Richardson JS, Richardson DC. 2003. Structure validation by Calpha geometry: phi,psi and Cbeta deviation. Proteins 50, 437-450. Mandhania S, Madan S, Sawhney V. 2006. Antioxidant defense mechanism under salt stress in wheat seedlings. Biologia Plantarum 50, 227-231. Manosalva PM, Davidson RM, Liu B, Zhu X, Hulbert SH, Leung H, Leach JE. 2009. A germin-like protein gene family functions as a complex quantitative trait locus conferring broad-spectrum disease resistance in rice. Plant Physiol 149, 286-296. Mauch-Mani B, Mauch F. 2005. The role of abscisic acid in plant-pathogen interactions. Curr Opin Plant Biol 8, 409-414. McElwain EF, Spiker S. 1992. Molecular and physiological analysis of a heat-shock response in wheat. Plant Physiol 99, 1455-1460. Means GE, Feeney RE. 1990. Chemical modifications of proteins: history and applications. Bioconjugate chemistry 1, 2-12. Mehdy MC. 1994. Active Oxygen Species in Plant Defense against Pathogens. Plant Physiol 105, 467-472. Membre N, Bernier F, Staiger D, Berna A. 2000. Arabidopsis thaliana germin-like proteins: common and specific features point to a variety of functions. Planta 211, 345354. Meyer-Siegler K, Mauro DJ, Seal G, Wurzer J, deRiel JK, Sirover MA. 1991. A human nuclear uracil DNA glycosylase is the 37-kDa subunit of glyceraldehyde-3phosphate dehydrogenase. Proc Natl Acad Sci U S A 88, 8460-8464. Michalowski CB, Bohnert HJ. 1992. Nucleotide Sequence of a Root-Specific Transcript Encoding a Germin-Like Protein from the Halophyte Mesembryanthemum crystallinum. Plant Physiol 100, 537-538. Montavon P, Kruger NJ. 1993. Essential Arginyl Residue at the Active Site of Pyrophosphate:Fructose 6-Phosphate 1-Phosphotransferase from Potato (Solanum tuberosum) Tuber. Plant Physiol 101, 765-771. Moons A, Prinsen E, Bauw G, Van Montagu M. 1997. Antagonistic Effects of Abscisic Acid and Jasmonates on Salt Stress-Inducible Transcripts in Rice Roots. Plant Cell 9, 2243-2259. 117 Bibliografía Mosolov VV, Grigoryeva LI, Valueva TA. 2001a. Involvement of proteolytic enzymes and their inhibitors in plant protection (review). Applied Biochemistry and Microbiology 37, 115-123. Mosolov VV, Grigoryeva LI, Valueva TA. 2001b. Plant proteinase inhibitors as polyfunctional proteins (a review). Prikl Biokhim Mikrobiol. 37, 643-650. Mosolov VV, Valueva TA. 2005. Proteinase inhibitors and their function in plants: a review. Prikladnaia biokhimiia i mikrobiologiia 41, 261-282. Nakata M, Shiono T, Watanabe Y, Satoh T. 2002. Salt stress-induced dissociation from cells of a germin-like protein with Mn-SOD activity and an increase in its mRNA in a moss, Barbula unguiculata. Plant and Cell Physiology 43, 1568-1574. Neilson KA, Gammulla CG, Mirzaei M, Imin N, Haynes PA. 2009. Proteomic analysis of temperature stress in plants. Proteomics 10, 828-845. Neuhoff V, Arold N, Taube D, Ehrhardt W. 1988. Improved staining of proteins in polyacrylamide gels including isoelectric focusing gels with clear background at nanogram sensitivity using Coomassie Brilliant Blue G-250 and R-250. Electrophoresis 9, 255-262. Neurath H. 1989. Proteolytic processing and physiological regulation. Trends Biochem Sci 14, 268-271. Noctor G, Foyer CH. 1998. ASCORBATE AND GLUTATHIONE: Keeping Active Oxygen Under Control. Annu Rev Plant Physiol Plant Mol Biol 49, 249-279. O'Farrell PZ, Goodman HM, O'Farrell PH. 1977. High resolution two-dimensional electrophoresis of basic as well as acidic proteins. Cell 12, 1133-1141. Oba Y, Ojika M, Inouye S. 2003. Firefly luciferase is a bifunctional enzyme: ATPdependent monooxygenase and a long chain fatty acyl-CoA synthetase. FEBS Lett 540, 251-254. Ohmiya A, Tanaka Y, Kadowaki K, Hayashi T. 1998. Cloning of genes encoding auxin-binding proteins (ABP19/20) from peach: significant peptide sequence similarity with germin-like proteins. Plant Cell Physiol 39, 492-499. Okuma E, Soeda K, Tada M, Murata Y. 2000. Exogenous proline mitigates the inhibition of growth of Nicotiana tabacum cultured cells under saline conditions. Soil Science and Plant Nutrition 46, 257–263. Oliva MLV, Silva MCC, Sallai RC, Brito MV, Sampaio MU. 2010. A novel subclassification for Kunitz proteinase inhibitors from leguminous seeds. Biochimie 92, 1667-1673. Olson P, Varner J. 1993. Hydrogen peroxide and lignification. The Plant Journal 4, 887-892. 118 Bibliografía Opaleye O, Rose RS, Whittaker MM, Woo EJ, Whittaker JW, Pickersgill RW. 2006. Structural and spectroscopic studies shed light on the mechanism of oxalate oxidase. J Biol Chem 281, 6428-6433. Ortiz C, Cardemil L. 2001. Heat-shock responses in two leguminous plants: a comparative study. J Exp Bot 52, 1711-1719. Ozawa K, Laskowski M. 1966. The Reactive Site of Trypsin Inhibitors. Journal of Biological Chemistry 241, 3955-3961. Parida A, Das AB, Mittra B. 2004. Effects of salt on growth, ion accumulation, photosynthesis and leaf anatomy of the mangrove, &lt;i&gt;Bruguiera parviflora&lt;/i&gt. Trees - Structure and Function 18, 167-174. Parida AK, Das AB. 2005. Salt tolerance and salinity effects on plants: a review. Ecotoxicology and Environmental Safety 60, 324-349. Park Y, Bo HC, Kwak JS, Kang CW, Lim HT, Cheong HS, Hahm KS. 2005. Kunitz-type serine protease inhibitor from potato (Solanum tuberosum L. cv. Jopung). Journal of Agricultural and Food Chemistry 53, 6491-6496. Perelló A, Mónaco C. 2007. Status and progress of biological control of wheat (Triticum aestivum L.) foliar diseases in Argentina. Fitosanidad 11, 85-105. Pernas M, Sánchez-Monge R, Salcedo G. 2000. Biotic and abiotic stress can induce cystatin expression in chestnut. FEBS Letters 467, 206-210. Pinedo M, Segarra C, Conde RD. 1993. Occurrence of two endoproteinases in wheat leaf intercellular washing fluid. Physiologia Plantarum 88, 287-293. Plunkett G, Senear DF, Zuroske G, Ryan CA. 1982. Proteinase inhibitors I and II from leaves of wounded tomato plants: Purification and properties. Archives of Biochemistry and Biophysics 213, 463-472. Prakash B, Selvaraj S, Murthy MRN, Sreerama YN, Rajagopal Rao D, Gowda LR. 1996. Analysis of the amino acid sequences of plant Bowman-Birk inhibitors. Journal of Molecular Evolution 42, 560-569. Qi RF, Song ZW, Chi CW. 2005. Structural features and molecular evolution of Bowman-Birk protease inhibitors and their potential application. Acta Biochimica et Biophysica Sinica 37, 283-292. Raj SSS, Kibushi E, Kurasawa T, Suzuki A, Yamane T, Odani S, Iwasaki Y, Ashida T. 2002. Crystal structure of bovine trypsin and wheat germ trypsin inhibitor (I2b) complex (2:1) at 2.3 Ã… resolution. Journal of Biochemistry 132, 927-933. Rawlings ND, Barrett AJ. 1993. Evolutionary families of peptidases. Biochem J 290 ( Pt 1), 205-218. Rawlings ND, Tolle DP, Barrett AJ. 2004a. Evolutionary families of peptidase inhibitors. Biochem J 378, 705-716. 119 Bibliografía Rawlings ND, Tolle DP, Barrett AJ. 2004b. MEROPS: the peptidase database. Nucleic Acids Res 32, D160-D164. Ray CA, Black RA, Kronheim SR, Greenstreet TA, Sleath PR, Salvesen GS, Pickup DJ. 1992. Viral inhibition of inflammation: Cowpox virus encodes an inhibitor of the interleukin-1[beta] converting enzyme. Cell 69, 597-604. Richards KD, Snowden KC, Gardner RC. 1994. Wali6 and wali7. Genes induced by aluminum in wheat (Triticum aestivum L.) roots. Plant Physiol 105, 1455-1456. Ritonja A, Krizaj I, Mesko P, Kopitar M, Lucovnik P, Strukelj B, Pungercar J, Buttle DJ, Barrett AJ, Turk V. 1990. The amino acid sequence of a novel inhibitor of cathepsin D from potato. FEBS Letters 267, 13-15. Rizhsky L, Liang H, Mittler R. 2002. The combined effect of drought stress and heat shock on gene expression in tobacco. Plant Physiol 130, 1143-1151. Rizhsky L, Liang H, Shuman J, Shulaev V, Davletova S, Mittler R. 2004. When defense pathways collide. The response of Arabidopsis to a combination of drought and heat stress. Plant Physiol 134, 1683-1696. Rodríguez-López M, Baroja-Fernandez E, Zandueta-Criado A, Moreno-Bruna B, Munoz FJ, Akazawa T, Pozueta-Romero J. 2001. Two isoforms of a nucleotide-sugar pyrophosphatase/phosphodiesterase from barley leaves (Hordeum vulgare L.) are distinct oligomers of HvGLP1, a germin-like protein. FEBS Lett 490, 44-48. Romaniouk A, vijay IK. 1997. Structure-function relationships in glucosidase I: amino acids involved in binding the substrate to the enzyme. Glycobiology 7, 399-404. Rosenfeld J, Capdevielle J, Guillemot JC, Ferrara P. 1992. In-gel digestion of proteins for internal sequence analysis after one- or two-dimensional gel electrophoresis. Analytical Biochemistry 203, 173-179. Rout NP, Shaw BP. 2001. Salt tolerance in aquatic macrophytes: possible involvement of the antioxidative enzymes. Plant Sci 160, 415-423. Ryan CA. 1980. Wound-regulated synthesis and vacuolar compartmentation of proteinase inhibitors in plant leaves. Current topics in cellular regulation 17, 1-23. Ryan CA. 1990. Proteinase inhibitors in plants genes for improving defenses against insects and pathogens Annual Review of Phytopathology 28, 425-449. Sanchez-Hernandez C, Martínez-Gallardo N, Guerrero-Rangel A, ValdesRodríguez S, Delano-Frier J. 2004. Trypsin and a-amylase inhibitors are differentially induced in leaves of amaranth (Amaranthus hypochondriacus) in response to biotic and abiotic stress. Physiologia Plantarum 122, 254-264. Sander C, Schneider R. 1991. Database of homology-derived protein structures and the structural meaning of sequence alignment. Proteins 9, 56-68. Saris CJM, van Eenbergen J, Jenks BG, Bloemers HPJ. 1983. Hydroxylamine cleavage of proteins in polyacrylamide gels. Analytical Biochemistry 132, 54-67. 120 Bibliografía Sato Y, Murakami T, Funatsuki H, Matsuba S, Saruyama H, Tanida M. 2001. Heat shock-mediated APX gene expression and protection against chilling injury in rice seedlings. J Exp Bot 52, 145-151. Satoh H, Uchida A, Nakayama K, Okada M. 2001. Water-Soluble Chlorophyll Protein in Brassicaceae Plants is a Stress-Induced Chlorophyll-Binding Protein. Plant and Cell Physiology 42, 906-911. Schagger H. 2006. Tricine-SDS-PAGE. Nat Protoc 1, 16-22. Schagger H, von Jagow G. 1987. Tricine-sodium dodecyl sulfate-polyacrylamide gel electrophoresis for the separation of proteins in the range from 1 to 100 kDa. Anal Biochem 166, 368-379. Schaller A. 2004. A cut above the rest: the regulatory function of plant proteases. Planta 220, 183-197. Schick C, Pemberton PA, Shi G-P, Kamachi Y, Cataltepe S, Bartuski AJ, Gornstein ER, Bromme D, Chapman HA, Silverman GA. 1998. Cross-Class Inhibition of the Cysteine Proteinases Cathepsins K, L, and S by the Serpin Squamous Cell Carcinoma Antigen 1: A Kinetic Analysis†Biochemistry 37, 5258-5266. Schöffl F, Prandl R, Reindl A. 1999. Molecular responses to heat stress. Molecular Responses to Cold, Drought, Heat and Salt Stress in Higher Plants. Austin , Texas: ShinozakiK, Yamaguchi-ShinozakiK, eds, 81-98. Schweizer P, Christoffel A, Dudler R. 1999. Transient expression of members of the germin-like gene family in epidermal cells of wheat confers disease resistance. Plant J 20, 541-552. Segarra CI, Casalongué CA, Pinedo ML, Cordo CA, Conde RD. 2002. Changes in Wheat Leaf Extracellular Proteolytic Activity after Infection with Septoria tritici. J Phytopathol 150, 105-111. Segarra CI, Casalongué CA, Pinedo ML, Ronchi VP, Conde RD. 2003. A germinlike protein of wheat leaf apoplast inhibits serine proteases. Journal of Experimental Botany 54, 1335-1341. Shan L, Li C, Chen F, Zhao S, Xia G. 2008. A Bowman-Birk type protease inhibitor is involved in the tolerance to salt stress in wheat. Plant, Cell and Environment 31, 1128-1137. Sharma N, Park S-W, Vepachedu R, Barbieri L, Ciani M, Stirpe F, Savary BJ, Vivanco JM. 2004. Isolation and Characterization of an RIP (Ribosome-Inactivating Protein)-Like Protein from Tobacco with Dual Enzymatic Activity. Plant Physiology 134, 171-181. Shetty NP, Jensen JD, Knudsen A, Finnie C, Geshi N, Blennow A, Collinge DB, Jorgensen HJ. 2009. Effects of beta-1,3-glucan from Septoria tritici on structural defence responses in wheat. J Exp Bot 60, 4287-4300. 121 Bibliografía Simoes-Araujo JL, Rodrigues RL, de AGLB, Mondego JM, Alves-Ferreira M, Rumjanek NG, Margis-Pinheiro M. 2002. Identification of differentially expressed genes by cDNA-AFLP technique during heat stress in cowpea nodules. FEBS Lett 515, 44-50. Smith PK, Krohn RI, Hermanson GT, Mallia AK, Gartner FH, Provenzano MD, Fujimoto EK, Goeke NM, Olson BJ, Klenk DC. 1985. Measurement of protein using bicinchoninic acid. Anal Biochem 150, 76-85. Smyth DG, Blumenfeld OO, Konigsberg W. 1964. Reactions of N-ethylmaleimide with peptides and amino acids. Biochem J 91, 589-595. Smyth DG, Nagamatsu A, Fruton JS. 1960. Some Reactions of N-Ethylmaleimide1. Journal of the American Chemical Society 82, 4600-4604. Snowden KC, Richards KD, Gardner RC. 1995. Aluminum-Induced Genes (Induction by Toxic Metals, Low Calcium, and Wounding and Pattern of Expression in Root Tips). Plant Physiol 107, 341-348. Souza-Pinto JC, Oliva ML, Sampaio CA, Damas J, Auerswald EA, Limaos E, Fritz H, Sampaio MU. 1996. Effect of a serine proteinase inhibitor from Leucaena leucocephala on plasma kallikrein and plasmin. Immunopharmacology 33, 330-332. Spychalla JP, Desborough SL. 1990. Superoxide Dismutase, Catalase, and alphaTocopherol Content of Stored Potato Tubers. Plant Physiol 94, 1214-1218. Srinivasan A, Giri AP, Harsulkar AM, Gatehouse JA, Gupta VS. 2005. A Kunitz trypsin inhibitor from chickpea (Cicer arietinum L.) that exerts anti-metabolic effect on podborer (Helicoverpa armigera) larvae. Plant Molecular Biology 57, 359-374. Stec B, Yang H, Johnson KA, Chen L, Roberts MF. 2000. MJ0109 is an enzyme that is both an inositol monophosphatase and the 'missing' archaeal fructose-1,6bisphosphatase. Nat Struct Mol Biol 7, 1046-1050. Strobl S, Muhlhahn P, Bernstein R, Wiltscheck R, Maskos K, Wunderlich M, Huber R, Glockshuber R, Holak TA. 1995. Determination of the three-dimensional structure of the bifunctional alpha-amylase/trypsin inhibitor from ragi seeds by NMR spectroscopy. Biochemistry 34, 8281-8293. Supuran CT, Scozzafava A, Clare BW. 2002. Bacterial protease inhibitors. Med Res Rev 22, 329-372. Swart S, Logman TJ, Smit G, Lugtenberg BJ, Kijne JW. 1994. Purification and partial characterization of a glycoprotein from pea (Pisum sativum) with receptor activity for rhicadhesin, an attachment protein of Rhizobiaceae. Plant Mol Biol 24, 171183. Tabuchi T, Kumon T, Azuma T, Nanmori T, Yasuda T. 2003. The expression of a germin-like protein with superoxide dismutase activity in the halophyte Atriplex lentiformis is differentially regulated by wounding and abscisic acid. Physiologia Plantarum 118, 523-531. 122 Bibliografía Takemura T, Hanagata N, Sugihara K, Baba S, Karube I, Dubinsky Z. 2000. Physiological and biochemical responses to salt stress in the mangrove, Bruguiera gymnorrhiza. Aquatic Botany 68, 15-28. Tashiro M, Hashino K, Shiozaki M, Ibuki F, Maki Z. 1987. The complete amino acid sequence of rice bran trypsin inhibitor. Journal of Biochemistry 102, 297-306. Thompson EW, Lane BG. 1980. Relation of protein synthesis in imbibing wheat embryos to the cell-free translational capacities of bulk mRNA from dry and imbibing embryos. J Biol Chem 255, 5965-5970. Tornero P, Conejero V, Vera P. 1996. Primary structure and expression of a pathogen-induced protease (PR-P69) in tomato plants: Similarity of functional domains to subtilisin-like endoproteases. Proc Natl Acad Sci U S A 93, 6332-6337. Tornero P, Conejero V, Vera P. 1997. Identification of a new pathogen-induced member of the subtilisin-like processing protease family from plants. J Biol Chem 272, 14412-14419. Torres MA, Dangl JL. 2005. Functions of the respiratory burst oxidase in biotic interactions, abiotic stress and development. Curr Opin Plant Biol 8, 397-403. Tsybina T, Dunaevsky Y, Musolyamov A, Egorov T, Larionova N, Popykina N, Belozersky M. 2004. New protease inhibitors from buckwheat seeds: Properties, partial amino acid sequences and possible biological role. Biological Chemistry 385, 429-434. Udenfriend S, Stein S, Böhlen P, Dairman W, Leimgruber W, Weigele M. 1972. Fluorescamine: a reagent for assay of amino acids, peptides, proteins, and primary amines in the picomole range. Science 178, 871-872. Urban C, Xiong X, Sohn K, Schroppel K, Brunner H, Rupp S. 2005. The moonlighting protein Tsa1p is implicated in oxidative stress response and in cell wall biogenesis in Candida albicans. Mol Microbiol 57, 1318-1341. Valueva TA, Mosolov VV. 2004. Role of inhibitors of proteolytic enzymes in plant defense against phytopathogenic microorganisms. Biochemistry (Moscow) 69, 13051309. Vallejos R. 1981. Chemical modification of chloroplast coupling factor 1. In: S SBaSR, ed. Energy Coupling in Photosynthesis. North Holland/New York: Elsevier, 129-139. Vallelian-Bindschedler L, Mosinger E, Metraux JP, Schweizer P. 1998. Structure, expression and localization of a germin-like protein in barley (Hordeum vulgare L.) that is insolubilized in stressed leaves. Plant Mol Biol 37, 297-308. van der Hoorn RA, Jones JD. 2004. The plant proteolytic machinery and its role in defence. Curr Opin Plant Biol 7, 400-407. Vierling E. 1991. The Roles of Heat Shock Proteins in Plants. Annual Review of Plant Physiology and Plant Molecular Biology 42, 579-620. 123 Bibliografía Vierstra RD. 1993. Protein Degradation in Plants. Annual Review of Plant Physiology and Plant Molecular Biology 44, 385-410. Walters DR. 2003. Polyamines and plant disease. Phytochemistry 64, 97-107. Wang HX, Ng TB. 2006. Concurrent isolation of a Kunitz-type trypsin inhibitor with antifungal activity and a novel lectin from Pseudostellaria heterophylla roots. Biochemical and Biophysical Research Communications 342, 349-353. Wang W, Vinocur B, Shoseyov O, Altman A. 2001. Biotechnology of plant osmotic stress tolerance: physiological and molecular considerations. Acta Horticulturae 560, 285–292. Wang Y, Nil N. 2000. Changes in chlorophyll, ribulose bisphosphate carboxylaseoxygenase, glycine betaine content, photosynthesis and transpiration in Amaranthus tricolor leaves during salt stress. The Journal of Horticultural Science & Biotechnology 75, 623-627. Watanabe H, Takehana K, Date M, Shinozaki T, Raz A. 1996. Tumor cell autocrine motility factor is the neuroleukin/phosphohexose isomerase polypeptide. Cancer Res 56, 2960-2963. Wei Y, Zhang Z, Andersen CH, Schmelzer E, Gregersen PL, Collinge DB, Smedegaard-Petersen V, Thordal-Christensen H. 1998. An epidermis/papillaspecific oxalate oxidase-like protein in the defence response of barley attacked by the powdery mildew fungus. Plant Molecular Biology 36, 101-112. Werner JE, Finkelstein RR. 1995. Arabidopsis mutants with reduced response to NaCl and osmotic stress. Physiologia Plantarum 93, 659-666. Wieczorek M, Otlewski J, Cook J, Parks K, Leluk J, Wilimowska-Pelc A, Polanowski A, Wilusz T, Laskowski M. 1985. The squash family of serine proteinase inhibitors. Amino acid sequences and association equilibrium constants of inhibitors from squash, summer squash, zucchini, and cucumber seeds. Biochem Biophys Res Commun 126, 646-652. Wise RR, Naylor AW. 1987. Chilling-enhanced photooxidation : evidence for the role of singlet oxygen and superoxide in the breakdown of pigments and endogenous antioxidants. Plant Physiol 83, 278-282. Woese CR, Fox GE. 1977. Phylogenetic structure of the prokaryotic domain: the primary kingdoms. Proc Natl Acad Sci U S A 74, 5088-5090. Wojtaszek P. 1997. Oxidative burst: an early plant response to pathogen infection. Biochem J 322 ( Pt 3), 681-692. Woo EJ, Dunwell JM, Goodenough PW, Marvier AC, Pickersgill RW. 2000. Germin is a manganese containing homohexamer with oxalate oxidase and superoxide dismutase activities. Nat Struct Biol 7, 1036-1040. 124 Bibliografía Woo EJ, Dunwell JM, Goodenough PW, Pickersgill RW. 1998. Barley oxalate oxidase is a hexameric protein related to seed storage proteins: evidence from X-ray crystallography. FEBS Lett 437, 87-90. Xu J, Tian J, Belanger FC, Huang B. 2007. Identification and characterization of an expansin gene AsEXP1 associated with heat tolerance in C3 Agrostis grass species. J Exp Bot 58, 3789-3796. Xu W, Seiter K, Feldman E, Ahmed T, Chiao JW. 1996. The differentiation and maturation mediator for human myeloid leukemia cells shares homology with neuroleukin or phosphoglucose isomerase. Blood 87, 4502-4506. Yamahara T, Shiono T, Suzuki T, Tanaka K, Takio S, Sato K, Yamazaki S, Satoh T. 1999. Isolation of a germin-like protein with manganese superoxide dismutase activity from cells of a moss, Barbula unguiculata. J Biol Chem 274, 33274-33278. Yamamoto M, Hara S, Ikenaka T. 1983. Amino Acid Sequences of Two Trypsin Inhibitors from Winged Bean Seeds (Psophocarpus tetragonolobus (L)DC.). J Biochem 94, 849-863. Yano A, Suzuki K, Shinshi H. 1999. A signaling pathway, independent of the oxidative burst, that leads to hypersensitive cell death in cultured tobacco cells includes a serine protease. Plant Journal 18, 105-109. Yoo BC, Aoki K, Xiang Y, Campbell LR, Hull RJ, Xoconostle-Cázares B, Monzer J, Lee JY, Ullman DE, Lucas WJ. 2000. Characterization of Cucurbita maxima phloem serphin-1 (CmPS-1). A developmentally regulated elastase inhibitor. Journal of Biological Chemistry 275, 35122-35128. Zhang J, Zhang Y, Inouye M. 2003. Thermotoga maritima MazG Protein Has Both Nucleoside Triphosphate Pyrophosphohydrolase and Pyrophosphatase Activities. Journal of Biological Chemistry 278, 21408-21414. Zhang ZG, Collinge DB, Thordal-Christensen H. 1995. Germin-like oxalate oxidase, a H2O2-producing enzyme, accumulates in barley attacked by the powdery mildew fungus. Plant Journal 8, 139–145. Zimmermann G, Baumlein H, Mock HP, Himmelbach A, Schweizer P. 2006. The multigene family encoding germin-like proteins of barley. Regulation and function in Basal host resistance. Plant Physiol 142, 181-192. 125