Transcripción Y Procesamiento
Anuncio
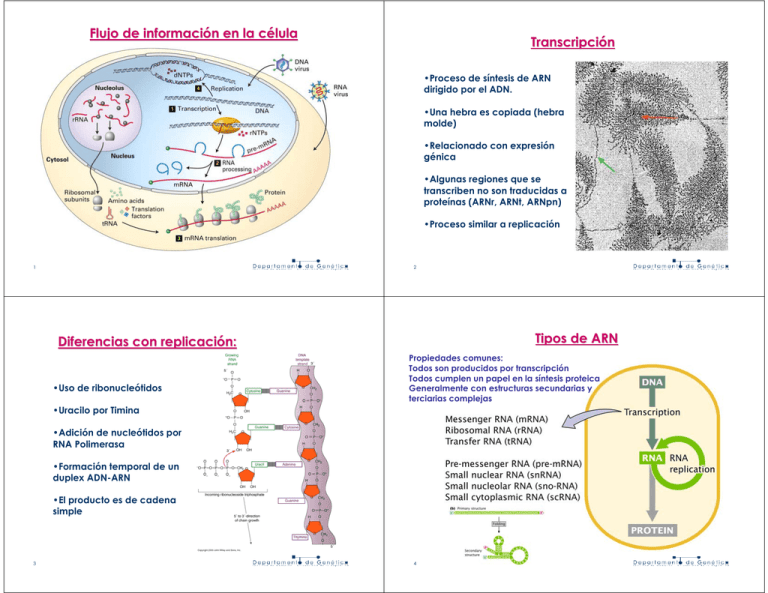
Flujo de información en la célula Transcripción •Proceso de síntesis de ARN dirigido por el ADN. •Una hebra es copiada (hebra molde) •Relacionado con expresión génica •Algunas regiones que se transcriben no son traducidas a proteínas (ARNr, ARNt, ARNpn) •Proceso similar a replicación 1 2 Tipos de ARN Diferencias con replicación: •Uso de ribonucleótidos Propiedades comunes: Todos son producidos por transcripción Todos cumplen un papel en la síntesis proteica Generalmente con estructuras secundarias y terciarias complejas •Uracilo por Timina •Adición de nucleótidos por RNA Polimerasa •Formación temporal de un duplex ADN-ARN •El producto es de cadena simple 3 4 Características de la transcripción ARN celulares, abundancia y función Dirigido por una ARN Polimerasa La transcripción está fuertemente regulada. Apenas un 0.01% de los genes se está expresando en una célula eucariota en un momento dado. 66 % % 80% 80% 10% 10% 44 % % Una hebra de ADN sirve de molde La enzima no requiere cebador La enzima elonga una cadena en dirección 5`-3` Formación de enlace 3´-5´fosfodiéster La síntesis sólo se inicia en secuencias específicas llamadas promotores Desenrrollamiento parcial del ADN 5 6 ARN polimerasa bacteriana Nomenclatura transcripcional Una hebra de ADN sirve de molde (hebra molde) •Unica ARNPolimerasa bacteriana •Multímero de varias subunidades •subunidad σ70 se une a secuencias específicas en las posiciones –10 y –35 del promotor El transcripto es igual a la hebra codificante y complementario a la hebra molde •la subunidad α queda en posición upstream •las subunidades b y b’ se asocian con el sitio de inicio (Sense) CCTTACTTACTGTTACGCCG GGAATGCCTGACAATGCGGC • el sitio de inicio de la transcripción es +1 •downstream = dirección de la transcripción (Antisense) CCUUACUUACUGUUACGCCG 7 8 Promotores bacterianos Modelo de la ARN Polimerasa bacteriana El modelo muestra la interacción de la holoenzima con el promotor formando el complejo abierto. Hebra molde en gris 9 10 Transcripción en bacterias: iniciación El proceso de la transcripción 1. Reconimiento de la hebra molde Unión de la polimerasa al promotor (Formación de un complejo cerrado) Formación de una burbuja de ADN de cadena simple (complejo abierto) 2. Iniciación Síntesis de los primeros 9 o 10 nucleótidos Varios eventos abortivos, donde el ARN es liberado Pasado este punto (10 nucleótidos) se libera sigma y comienza la longación 11 12 Terminación transcripcional en bacterias Transcripción en bacterias: elongación Reconocimiento de señales particulares que indican donde terminar la síntesis La burbuja de transcripción se desestabiliza y colapsa liberándose el ARN Rho dependiente Rho independiente proteína Rho se une al ARN bucle autocomplementario Varios eventos abortivos, donde el ARN es liberado Pasado este punto (10 nucleótidos) se libera sigma y comienza la elongación Adición de nucleótidos al extremo 3´ hidroxilo de la cadena creciente 13 14 Diferencias entre la transcripción procariota y eucariota ARN polimerasas eucariotas INCAPACES DE RECONOCER EL PROMOTOR ARNm policistrónico transcripción y traducción acopladas ARN Polimerasa única 15 ARNm monocistrónico transcripción y procesamiento acoplados 3 ARN Polimerasas diferentes Polimerasa Pol I Pol II Pol III 16 localización nucleolo nucleoplasma nucleoplasma genes transcritos ARNr ARNm , ARNpn ARNt, ARNr 5S inhibición por a-amanitina insensible muy sensible sensible La polimerasa II eucariota es similar a la bacteriana..... 17 .....aunque más compleja 18 Transcripción en eucariotas Iniciación en eucariotas Proteínas ARN Polimerasas Factores de Transcripción Modificadores Todas las polimerasas eucariotas necesitan Factores Transcripcionales basales para reconocer los promotores e iniciar la transcripción Polimerasa II Factores generales Factores específicos Modificadores Señales 19 Promotores Potenciadores 20 Promotores regulados eucariotas Factores transcripcionales Proteínas de unión a sitios específicos de ADN •Secuencias de reconocimiento específico para Factores de Transcripción Se asocian a Promotores y potenciadores Estructura modular: dominios de activación •Estructura modular 21 dominios de unión al ADN 22 Los Factores Transcripcionales son proteínas de unión al ADN Potenciadores Activación de la Transcripción Reconocen secuencias específicas en promotores y potenciadores Se ensamblan cooperativamente sobre el ADN mediante interacciones proteína-proteína Potenciadores (enhancers) reguladores en cis Función a distancia Independientemente de orientación Sitios de union a factores de transcripción 23 24 Los factores de transcripción específicos colaboran en el ensamblado del complejo de iniciación Existen activadores, represores y mediadores Factores específicos colaboran para colocar los FBasales en el promotor y éstos posicionan las polimerasas La mayoría de los genes regulados por factores transcripcionales múltiples 25 26 Los factores basales se ensamblan secuencialmente y posicionan la ARN polimerasa Elongación en eucariotas, el papel del CTD CTD- CARBOXI TERMINAL DOMAIN de la RNA Polimerasa II c/repetidos de 7 péptidos (26-52aa) Unicos a pol II Su fosforilación es esencial para la elongación La fosforilación depende de uno de los factores generales 27 28 PROCESAMIENTO DE ARN EN EUCARIOTAS El ciclo transcripcional eucariota Todo ARN se procesa. • Los ARNr se transcriben juntos y por corte se generan fragmentos menores. • Los ARNt se procesan y sufren modificaciones de bases. • Los ARNm sufren 4 tipos de modificaciones: - Capping. - Cola poli A. - Corte y empalme de intrones (Splicing) - Editing. 29 30 Unidad Transcripcional Ribosomal Transcripción Y Procesamiento de ARNr 31 • Genes dispuestos en tándem: aproximadamente 200 copias de genes de ARNr en Genoma Humano (Cromosomas con satélites). • Transcriptos como un gran precursor policistrónico de 13000 nt (ARNr 45S). •En interfase se ven como Nucleolos: zonas de transcripción y unión a proteínas ribosómicas. •Procesados secuencialmente (degradación de las secuencias intermedias) •Los transcriptos son metilados 32 Transcripción y Procesamiento del ARNt Modificaciones de bases en el ARNt • Transcriptos como mono o policistrónicos •Los transcriptos son procesados secuencialmente •Formación del trébol •Degradación de extremos y adición de brazo aceptor CCA •Eliminación de intrones •Modificación de bases 33 34 Procesamiento de los ARN mensajeros Capping • Estructura de la Caperuza 5´ 35 36 El mecanismo de la Poliadenilación Poliadenilación en extremo 3´ Proceso secuencial dependiente de : •Reconocimiento de secuencias en el transcripto •Interacción ARN-proteinas específicas •Interacción proteína-proteína 37 38 Hibridación ADNADN-ARN Corte y empalme (splicing (splicing)) de intrones y exones el gen de la ovoalbúmina 39 40 El proceso de corte y empalme •Ensamblado secuencial de partículas riboproteicas (SNURPs) •Apareamiento de bases con el mRNA Tipos de intrones •Corte y empalme •Liberación del intrón U1 snRNP U2 snRNP Pre mRNA intron GU A AG Salida del U4, U6, U5 snRNP intron Ligacion de los exones 41 42 Reconocimiento de señales de procesamiento El spliceosoma •Estructura riboproteica encargada del procesamiento de los intrones •Apareamiento de bases del ARN precursor con el ARN del SNURP •Ensamblado secuencial de partículas riboproteicas (SNURPs) 43 44 El splicing consiste en dos transesterificaciones sucesivas Ribozimas TIPO I Algunos genes de ARNr • ARN que elimina sus propios intrones • ARN que elimina intrones de otros ARN • ARN que cataliza su propia replicación Estructura terciaria de un intron tipo I de un ARN de Tetrahymena que se autoelimina del resto del ARN. TIPO II Mitocondria y cloroplastos 45 46 Una visión moderna de la expresión génica 47






