RNA: Química y estabilidad RNA
Anuncio
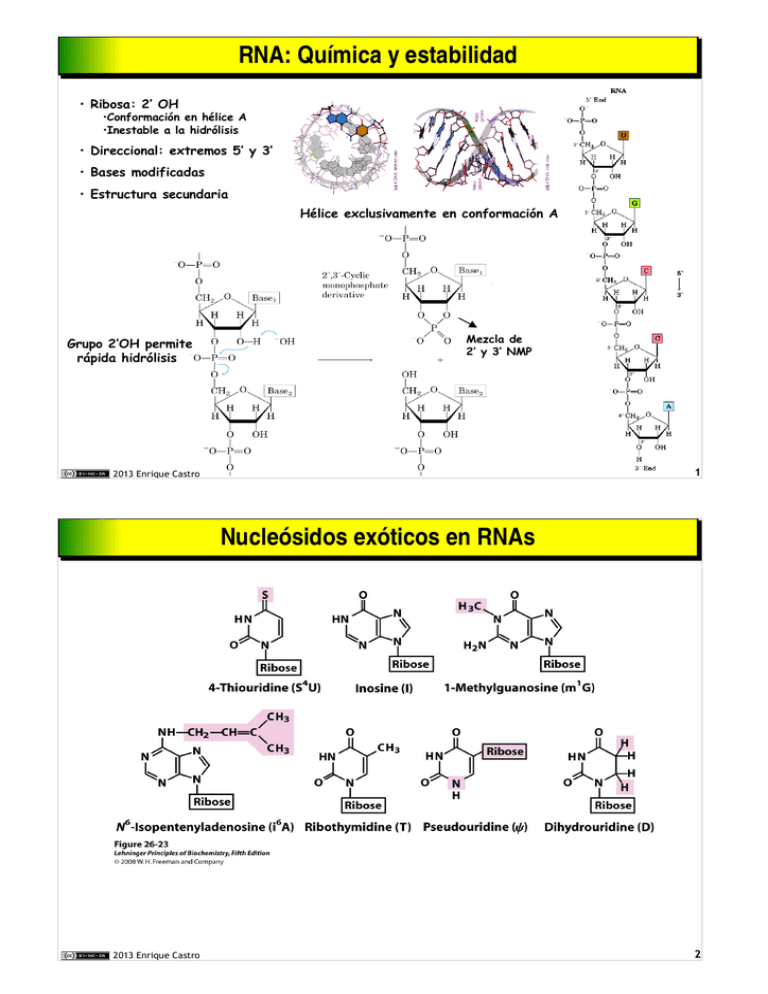
RNA: Química y estabilidad RNA: Química y estabilidad • Ribosa: 2’ OH •Conformación en hélice A •Inestable a la hidrólisis • Direccional: extremos 5’ y 3’ • Bases modificadas • Estructura secundaria Hélice exclusivamente en conformación A Grupo 2’OH permite rápida hidrólisis Mezcla de 2’ y 3’ NMP 1 2013 Enrique Castro Nucleósidos exóticos en RNAs Nucleósidos exóticos en RNAs 2013 Enrique Castro 2 RNA: estructura y tipos RNA: estructura y tipos RNA ribosómico (rRNA) • Cadena simple • Alta estructura secundaria (hélices en bucles intracatenarios) • Estructural (ribosoma) • Catalítico 120 nts (5S) 1542 (16S) 2904 (23S) 80 % RNA total rRNA 16S RNA de transferencia (tRNA ) • Cadena simple • Estructura secundaria media (hélice en bucles intracatenarios) 73-90 nts 15 % RNA total • Abundancia de bases modificadas • Adaptador del código genético RNA pequeño nuclear (snRNA ) • Cadena simple • Alta estructura secundaria (hélice en bucles intracatenarios) <300 nts 3 % ? RNA total • Ricos en U • Estructural (partículas snRNP, espliceosoma) • Catalítico (eliminación de intrones) RNA heterogéneo nuclear (hnRNA ) • Cadena simple • Baja estructura secundaria • Informativo: transcritos primarios de genes lncRNA • Cadena simple, intergénico, ¿espurio? RNA intrónico • No-informativo RNA mensajero (mRNA ) 75-3000 nts 2 % RNA total • Informativo: codificación de proteínas (madurado a partir de hnRNA) 2013 Enrique Castro 3 Convenios y nomenclatura: transcripción y traducción Convenios y nomenclatura: transcripción y traducción con sentido Hebra codificante No molde 5’3’ sin sentido Hebra molde No codificante 3’5’ mRNA 5’3’ Secuencia de hebra codificante Complementario a molde 3’5’ Proteína NC Traducido de mRNA 5’3’ 2013 Enrique Castro Hebra codificante 5’3’ 4 Transcripción: proceso general Transcripción: proceso general Transcripción: polimerización de RNA • • • • Reconocimiento del promotor Ensamblaje sobre inicio Activación del PIC: desalojo del promotor Terminación: corte y poliadenilación Terminación Promotor Sitio inicio Procesamiento del transcrito primario (pre-mRNA) • • • • Caperuza 5’ Corte y poliadenilación 3’ Eliminación de intrones (splicing) Edición Transporte nucleo-citoplasmático 5 2013 Enrique Castro RNA polimerasa : mecanismo enzimático RNA polimerasa : mecanismo enzimático Caracteríscticas de RNA polimerasas • • • • • Oligómero de alto peso molecular (6-14 subunidades) Requiere molde de DNA Utiliza NTP, libera PPi No necesita cebador (3'-OH libre): 2 sitios NT Carece de 3’exonucleasa (corrección) RNAn + NTP ↔ RNAn+1 + PPi 2013 Enrique Castro 6 Inhibidores de RNA polimerasas Inhibidores de RNA polimerasas Rifampicina: se une a RNApol β (bacterias) impide desalojo el promotor Actinomicina D: intercala entre GC adyacentes distorsiona dúplex impideprogreso de la burbuja (RNApol) (pro/eucariotas) Carece de 3'OH impide crecimiento (pro/eucariotas) α-amanitina: bloquea RNApol II>RNApol III (eucariotas) 7 2013 Enrique Castro Topología Topología del del inicio inicio de de transcripción transcripción Reconocimiento del promotor • RNApol se une inespecíficamente al DNA (baja afinidad) • Recorre el dúplex buscando el promotor • Compejo multiproteico reconoce el promotor Complejo promotor cerrado • (Unión RNApol/DNA enrollado, Kd = 10-6-10-9 M ) • RNApol se desplaza hacia la caja Pribnow. Iniciación • 2 sitios de unión de NTPs en la RNApol Sitio de iniciación: prefiere A,G Sitio de elongación: 2º nucleótido • El 3'-OH del nucleótido en el lugar de iniciación ataca y se une al α-P del NTP en el otro sitio • La RNApol transloca 1 pb y repite ciclo, alargando la cadena naciente de RNA Complejo promotor abierto • Actividad helicasa desenrolla ≈12-15 pb hasta +2 - +5. • burbuja de transcripción. Unión de muy alta afinidad, Kd = 10-14 M 2013 Enrique Castro Desalojo del promotor • Cuando el transcrito tiene 6-10 nts • Factores de reconocimiento del promotor disociados 8 Burbuja de transcripción Burbuja de transcripción Elongación: Núcleo de RNA polimerasa (sin GTFs) RNA pol es procesiva RNA pol no precisa helicasa (desenrrolla al avanzar) Fidelidad media (10-4 errores) (adecuado para transcritos de <10000 nt) Velocidad 20-50 bases/segundo. (más lento en zonas ricas en G/C) Topoisomerasas preceden y siguen a la burbuja de transcripción (alivio de tensiones superhelicoidales) 13-15 pb 17-19 pb 17-19 pb 8-9 pb 6-7 nt Elongación: topoisomerasas Topoisomerasas preceden y siguen a la burbuja de transcripción (alivio de tensiones superhelicoidales) 9 2013 Enrique Castro RNA polimerasas eucarióticas RNA polimerasas eucarióticas Tres polimerasas diferentes RNApol I: pre-rRNA (nucleolo) RNApol II: general (mRNA) RNApol III: genes RNA (tRNA, snRNA etc) Sensibilidad a α-amanitina Pol II > polIII >> polI, RNApol bact 10 µM 100µM Proteínas grandes multiméricas ≈12 subunidades (500-700 kD) 5 comunes 4-7 específicas de I-III 2 subunidades grandes RPB1, RPB2 (130-220KD) homólogas a β,β’ de E. coli Estructura y función conservadas Interacción con promotores A través de RPB4 Requiere factores adicionales específicos de promotor RNApol III reconoce señales 3’ 2013 Enrique Castro Factores de Transcripción 2 subunidades homólogas a la α de E. coli RPB3 (20-40 kD) 10 Transcripción en eucariotas: estructura del mRNA Transcripción en eucariotas: estructura del mRNA Genes discontinuos Exones/intrones Procesamiento: splicing Estructura del mRNA eucariótico: Caperuza 5’ (estabilización) 5’ UTR (regulación) Región codificante 3’UTR (regulación) Cola de poli-A 3'(estabilización) Caperuza 5’ Región codificante (ORF) 5’ UTR Cola poli-A 3’ 3’ UTR 11 2013 Enrique Castro RNA polimerasa II RNA polimerasa II Transcripción de genes de proteínas Reconoce variedad de promotores Ampliamente regulable 2 subunidades grandes RPB1 (220 kD), RPB2 (150 kD) homólogas a β β’ de E. coli Requiere al menos 7 factores de transcripción generales Dominio carboxi-terminal (CTD) fosforilable Dominio CTD de RPB1 52 repeticiones de YSPTSPS Hasta 50 nm P P P P Funciones de CTD: Regulación • Unión al promotor: desfosforilado • Iniciación: fosforilado • Unión de proteínas accesorias RPB1: unión a NTPs (catalítica) RPB2: unión a DNA Regulación por fosforilación múltiple Procesamiento del pre-mRNA 2013 Enrique Castro 12 Promotores eucarióticos: promotor proximal (core promoter) Promotores eucarióticos: promotor proximal (core promoter) Exones intrones y UTRs Elementos potenciadores Estructura de un gen de mamífero: -200 -10 / -50 kb +10 / +50 kb -30 •Caja TATA •Iniciador •Caja GC Promotor proximal start 2013 Enrique Castro 13 Elementos reguladores Cis y Trans Elementos reguladores Cis y Trans Elementos reguladores Cis = secuencias promotoras (DNA) Elementos reguladores Trans = factores de transcripción (Prot) Elementos trans (proteínas) se unen a elementos cis (secuencias DNA) 2013 Enrique Castro 14 Factores de transcripción asociados a RNApol II Factores de transcripción asociados a RNApol II 15 2013 Enrique Castro Pasos clave en la transcripción del DNA por RNApol II Pasos clave en la transcripción del DNA por RNApol II Ensamblaje: Construcción secuencial y ordenada del complejo de iniciación Reconocimiento del promotor Unión de RNApolII (CTD desfosforilado) Iniciación: Hasta el complejo de preiniciación Apertura del complejo Desenrollado del molde y activación de la polimerización Activación de la RNApol Fosforilación del CTD Hasta el desalojo del promotor Elongación: Actividad de TFIIH Disociación de factores generales Reclutamiento de factores de elongación Reclutamiento por CTD de RNPs para procesado (capping, splicing) Crecimiento del transcrito por polimerización de NTPs sobre el molde Mientras no termine Terminación: Reclutamiento del complejo de poliadenilación (reconocimiento de la señal AUAAA) Corte y liberación del transcrito: desactivación de RNApol Poliadenilación 3’ 2013 Enrique Castro Acoplamiento de corte y poliadenilación con terminación 16 Ensamblaje de RNApol II sobre el promotor Ensamblaje de RNApol II sobre el promotor 1. Reconocimiento de promotor • unión de TBP • unión de TFIID ( y/o TFIIA) TFIID= •TBP o similar •11 TAFs 3. Reclutamiento de RNApol II • unida a TFIIF • CTD desfosforilado 2. Unión de TFIIB • Recluta RNApol TFIIF inhibe la unión no específica a sitios no promotores 9 subunidades Se une a TFIIB Complejo de iniciación cerrado 5. Apertura • Actividad helicasa de TFIIE y TFIIH 2013 Enrique Castro Complejo de iniciación abierto 4. Formación del complejo cerrado • unión de TFIIE • unión de TFIIH Complejo de pre-iniciación (PIC) 17 Ensamblaje de RNApol II en promotor: TBP­TFIID Ensamblaje de RNApol II en promotor: TBP­TFIID TBP reconoce secuencia poli-A en promotor Se une al DNA en conformación agudamentemente curvada 2013 Enrique Castro 18 Inicio de la transcripción por RNApol II Inicio de la transcripción por RNApol II TBP TFIIA TFIIB TFIIF RNApol II TFIIE Ensamblaje sobre core promoter TFIIH reutilización Señal de terminación Helicasa de TFIIH: Precisa Mediator Complejo enhanceosoma • PIC abierto • Molde expuesto pTEFb Elongación Quinasa de TFIIH: • Fosforilación múltiple en CTD • Activación de RNApol II • Disociación de TFIID/TBP-TFIIB 2013 Enrique Castro Desalojo del promotor marca inicio Factores generales disociados Liberación de TFIIE/H 19 Procesamiento del mRNA eucariótico Procesamiento del mRNA eucariótico • Simultáneo a la elongación • Factores reclutados por p-CTD PIC Reclutamiento de la capping enzyme Caperuza formada en la la transición iniciación-elongación (mRNA naciente 20-40 Nts) Requiere desfosforilación en Ser-5 Reclutamiento de la maquinaria de splicing (RNPs) Reclutamiento de la maquinaria de corte y poliadenilación capping enzyme maquinaria de splicing (RNPs) Requiere fosforilación en Ser-2 por pTEF2b(quinasa) mRNA/RNPs 2013 Enrique Castro 20 Procesamiento del mRNA: Formación de la caperuza 5’ Procesamiento del mRNA: Formación de la caperuza 5’ Funciones de la caperuza: Protección de 5’exonucleasas Exportación del núcleo Reconocimiento por ribosomas Capping enzyme Síntesis: Durante iniciación-elongación (25-30 nts) Enzima ligada a CTD-P de RNApol II 21 2013 Enrique Castro Terminación de transcripción: corte y poliadenilación Terminación de transcripción: corte y poliadenilación Señal de terminación en DNA/transcrito: Reclutamiento de endonucleasa Corte del transcrito termina la acción de RNApolII Catalizado por componentes RNP G/U Sitio de corte 10-30 nts 20-40 nts G/U RNApolII con RNA acortado: desensamblaje AAUAAA Rica en G/U muy conservada muy degenerada Une CPSF Une CstF Structural biology of poly(A) site definition. Yang Q, Doublié S. Wiley Interdiscip Rev RNA. 2011 2(5):732-47. 80-250 nts 2013 Enrique Castro 22 Procesamiento del mRNA: poliadenilación 3’ Procesamiento del mRNA: poliadenilación 3’ CPSF forma complejo inestable con AAUAAA Previene terminación por RNApol II Corte señala terminación a RNApol II Unión de CFI y CFII complejo estable unido a señales de terminación CStF se une a G/U Liberación de factores Se recluta poli-A polimerasa Poliadenilación lenta Reclutamiento de PABII Unión de PAP activa endonucleasa: corte 3’ de AAUAAA PABII estimula a PAP Poliadenilación rápida 23 2013 Enrique Castro Intrones eucarióticos: gen y mRNA de la ovoalbúmina de pollo Intrones eucarióticos: gen y mRNA de la ovoalbúmina de pollo Intrones: bucles extra (no codificante) mRNA maduro DNA genómico DNA genómico del gen de ovoalbúmina hibridado a su mRNA Exones: Elemento funcional Dominio estructural Variabilidad/combinatoria 30-100 aa Intrones: Señales de control Variabilidad/combinatoria 100-20.000 nts 3’UTR 5’UTR 2013 Enrique Castro 1158 nts (386 aa) 24 Estructura Estructura de de los los intrones intrones Señales identificadoras 1: Límite 5’: GU 2: Punto de ramificación (A), a 20-50 pb de 3’ 3: Región rica en pirimidinas (≈15 pb) 4: Límite 3’: AG 1 2 3 4 Intrón Punto de ramificación Exón 5’ A/C A G GU A/G A G U 70 60 80 Frecuencia de aparición, % Exón 3’ 20-50 pb 100 U A/G A C/U 95 70 80 45 90 80 100 80 Señal GU: Sitio de empalme 5’ Zona rica en Pir 100 – 20000 pb C AG G 80 100 60 Señal AG: Sitio de empalme 3’ Corte y empalme: A partir de bordes 5’GU y AG3’ Punto de ramificación es esencial Exclusivamente en el núcleo El sustrato de splicing es transcrito+caperuza+poli-A Catalizado por partículas snRNP (espliceosoma) snRNAP reclutadas por CTD-P de RNApol II 25 2013 Enrique Castro Splicing: Corte y empalme de intrones Splicing: Corte y empalme de intrones 2 reacciones de trans-esterificación sucesivas Exón 1 (5') Pre-mRNA Autocorte: • Tipo I algunos rRNA, rRNA • Tipo II (plantas, hongos) No autocorte: 1ª trans-esterificación. 2’-OH de A ataca pGU 5’ • Genes estructurales • tRNAs Requieren catalizador: snRNP “Lazo”: blucle cerrado covalentemente. Enlace 2’A-5’G 2ª transesterificación. 3’-OH de exón 1 ataca AGpX Exones ligados en orden Degradado rápidamente 2013 Enrique Castro Exón 1 (5') Exón 2 (3') 26 Procesamiento de intrones; estructura y función de snRNPs Procesamiento de intrones; estructura y función de snRNPs Partículas snRNP Catalizan splicing ≈ 5 snRNAs U1, U2, U4, U5, U6 (U11,U12) 100-200 nts. muy conservados ≈ 50 proteínas Ensamblaje requiere ATP Reclutados por CTD-P de RNApol II RNPn : generales y específicas Motivos de unión a RNA (RBD, RGG, KH) RNP1, RNP2 U2: unión al punto de ramificación (o bien U12) U1: reconocimiento de 5’GU (o bien U11) Py Centro de ramificación Borde 5' Funciones de Un snRNA Rica Pir Borde 3' Funciones de RNPs Hibridación: reconocimiento de señales Forman el lazo al reunir los extremos 5’ y 3’ Catálisis de transesterificación: ribozimas 2013 Enrique Castro Previene degradación snRNA Previene plegamientos secundarios de RNA Reconoce secuencias señal de control Recluta otras proteínas Transporte nucleocitoplasmático 27 Corte y empalme en el espliceosoma Corte y empalme en el espliceosoma U1 snRNP U2 snRNP ADP+Pi reutilización Otros espliceosomas usan U11 y U12 Funciones de snRNA: U1: une a extremos 5’ y 3’ U2: une a ramificación (catalítico) U4: mantiene inhibido U6 U5: se une a 5’ U6: catalítico Reordenamiento interno (rehidridacion) Funciones EJC Marcan RNA como procesado Unión complejo de transporte Reclutan exosoma (NMD) U6 hibridado con U2 y centro de empalme 5’ simultáneamente Lazo RNA degradado en Exosoma Depositado EJC Carmody & Wenter (2009) J. Cell Sci. 122, 1933-1937 2013 Enrique Castro (20-24 nt upstream borde 5' ) EJC 28 Procesamiento alternativo de intrones Procesamiento alternativo de intrones Promotores alternativos Hibridación: reconocimiento de señales Forman el lazo al reunir los extremos 5’ y 3’ Catálisis de transesterificación: ribozimas Poliadenilación alternativa Hibridación: reconocimiento de señales Forman el lazo al reunir los extremos 5’ y 3’ Catálisis de transesterificación: ribozimas Exon skipping (4 no incluido) 29 2013 Enrique Castro Procesamiento alternativo de intrones: modos Procesamiento alternativo de intrones: modos Poliadenilación alternativa Hibridación: reconocimiento de señales Forman el lazo al reunir los extremos 5’ y 3’ Catálisis de transesterificación: ribozimas Isoformas proteicas a medida de tejido 2013 Enrique Castro 30 Transcripción (RNApol I) y procesamiento de pre­rRNA Transcripción (RNApol I) y procesamiento de pre­rRNA SL1:multimérico Transcripción de: Activación e iniciación NO requiere ATP rRNA cluster (18S, 5.8S, 28S) Exclusivamente en nucleolo TBP Ensamblaje UBF se une a UCE Se une SL1 (TBP) TIF-IB fosforilado une RNApol I TIF-IB entrega RNApol I a SL1 RNApol I UBF rRNA promoter +1 -150 -100 pb Terminación por factor específico unido en 3’ UCE +1 Core element Procesamiento Intensa metilación 2'OH Modificación covalente de bases Corte por snoRNPs snoRNAs: from genes & introns Metilación 2'OH (≈100 b) Conversión DHU, ψ snoRNPs (snoRNAs) 31 2013 Enrique Castro Transcripción Transcripción por por RNApolIII RNApolIII Transcripción de: A Ensamblaje en promotor Señales de control intragénicas (3') 4 tipos (según transcrito) TBP en TFIIIB (flexión de DNA) Terminación por señal poli-U Unión TFIIIC a A-box y B-box Tipo I Promotor tRNA tRNAs rRNA 5S RNAs pequeños (snRNAs, snoRNAs, 7S RNA SRP) RNAs SINES +1 tRNA TBP B +1 rRNA 5S C Unión TFIIIA a ICR Elementos de control internos Tipo II Promotor rRNA (no horquilla) TBP Procesamiento de tRNAs Metilación Ribozimas de corte Sitio aceptor añadido Splicing no espliceosomal tRNA-nucleotidil transferasa. molde interno: 3 sitios de unión Schramm & Hernandez (2002) Genes Dev. 16:2593-2620 ribozima Intrón 2013 Enrique Castro No en espliceosoma mecanismo especial 32 Exportación de mRNAs al citosol Exportación de mRNAs al citosol Complejo mRNP mRNA maduro (5'-cap, 3'-poli-A) Proteínas CBC Proteínas EJC Proteínas hnRNP sin intrones desligado del espliceosoma Con EJCs hnRNP A1 EJCs snRNPs exclusivas nucleares (no se exportan) NPC transporta activamente Las proteínas son exportadas. mRNA va ligado hnRNP A1 & EJCs (retorna al núcleo) No exportado = degradado (e.g. talasemias) Sistema de vigilancia exosoma (3’ exonucelasas) mRNPs PAB1 33 2013 Enrique Castro Importación de proteínas RNPs Transporte núcleo­citoplasma Transporte núcleo­citoplasma Importación de factores citoplásmicos Importación de RNPs de ensamblaje en citoplasma Proteínas RNP snRNPs Caperuza adicional Importación activa snRNA snRNPs nucleolo núcleo 5S RNA Exportación de partículas pre-ribosómicas Importación de proteínas 18 S Partículas pre-ribosómicas 2013 Enrique Castro 28 S RNA 5.8 S RNA 5 S RNA Subunidades ribosómicas 34 Transporte citoplasma­núcleo: y proteínas G pequeñas (ran) Transporte citoplasma­núcleo: y proteínas G pequeñas (ran) Transporte unidireccional: ran hnRNP A1 Exportación sólo unido a Ran-GTP (NLS hidrófoba) Ran-GTP transportada exclusivamente núcleo →citoplasma Distribución asimétrica de Ran-GEF/Ran-GAP • Β-Importina une alta afinidad Ran-GTP • [Ran GTP] alte en nucleoplasma • Complejo αβ-importina disociado (competición) [Ran-GTP] GEF: intercambio GDP/GTP Gradiente de Ran-GDP Gradiente de Ran-GTP [Ran-GDP] • Ran-GDP al núcleo (a favor de gradiente) Ran-GAP: • β-Importina no une Ran-GDP • β-Importina une α-importina & carga • Complejo αβ-importina formado Hidrólisis de GTP unido 35 2013 Enrique Castro Mecanismos de supervisión de RNA: núcleo Mecanismos de supervisión de RNA: núcleo Diferencias: Ran-GTP une transportador vacío Importación es activa Requiere factores adicionales (NTF2) Cada etapa puede fallar Mecanismos QC en cada etapa • 5' exonucleasa Rat1 • Debranching enzyme • 3' Exonucleasa exosómica 2013 Enrique Castro 36 Mecanismos de supervisión de RNA: citoplasma Mecanismos de supervisión de RNA: citoplasma UPF Vigilancia/Control de calidad • NMD codones stop prematuros • NSD non-stop • NGD no-go EJC UAA Ribosoma pionero elimina EJCs Ribosoma EJC Ribosoma detenido en stop Une eRF1, eRF2 Si queda cerca de EJC en 3' Une UPF1-3 Recluta endonucleasa exosómica mRNA codón stop prematuro mRNA sin codón stop Ski7 une al sitio A ribosoma Libera mRNA Recluta exosoma Ribosomas detenidos en poli-A RNA con estructura secundaria Endonucleasa Dom34/Hbs1 Ribosomas detenidos (no-go) Recambio constitutivo RNA dúplex (virus) / RNAi • Dicer/RISC 37 2013 Enrique Castro Degradación de RNA Degradación de RNA ➢ 3' exonucleasa poli-A • Actividad des-adenilasa (Ccr4/Caf1) • Inhibida por PAB1 • Actividad lenta pero constante Degradación lenta progresiva de cola poli-A ➢ Complejo Descaperuzante • • • • Elimina cap Inhibida por eIF-4E/eiF4G Reclutada cuando poli-A corta Permite Actividad 5' exonucleasa Xm1 Decapping actua sólo si previamente acortada poli-A ➢ Exosoma • • • • Complejo multiproteico hexamérico 3' exo- & endo-nucleasa Degradación rápida y completa de RNAs Nuclear/citoplasma 2013 Enrique Castro 38 Metabolismo citosólico del mRNA: vía rápida por señales AU Metabolismo citosólico del mRNA: vía rápida por señales AU Reclutamiento de endonucleasa específica Señales codificadas en 5’UTR y 3’UTR 2013 Enrique Castro 39