Procesamiento de mRNA
Anuncio
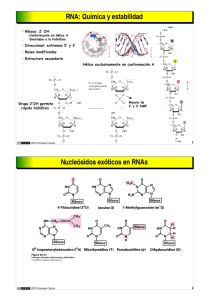
Procesamiento de mRNA M. en C. Elva Carolina Chávez Hernández aprendegenetica.blogspot.mx Tipos de RNA En eucariontes las RNA polimerasas están especializadas El RNA es procesado para su maduración El procesamiento puede ser CO- ó POST- transcripcional dependiendo del tipo de RNA RNAt RNAm RNAr • • • Adición del 5’ cap Corte y empalme (splicing) Poliadenilación en 3’ • Metilación de la ribosa. • Remoción de partes intermedias del pre-RNAr • • • Remoción de extremos Modificación de las bases. Adición de -CCA El procesamiento del pre-mRNA para obtener un mRNA maduro es co-transcripcional CTD Capping del extremo 5’ Remoción de intrones Splicing Poliadenilación del extremo 3’ El dominio carboxilo terminal de subunidad mayor de la RNA polimerasa II (CTD), recluta a los factores necesarios para el procesamiento del mRNA Capping del extremo 5’ Fosfatasa GTP + RNA Guanil transferasa “Cap” (7mGpppN) Metil transferasa La adición del cap en el 5’ es un punto de control para que proceda la elongación de la transcripción Fosforilación de Ser5 de CTD Reclutamiento de factores que “arrestan” a RNAPII Se “libera” la RNAPII Elongación productiva Reclutamiento de cinasas por las enzimas de capping Fosforilación de CTD-Ser2 y de factores que arrestan a RNAPII Reclutamiento de enzimas de capping Capping del extremo 5’ del transcrito ¿Para qué añadir el 5’ Cap? 1. Le da estabilidad al mRNA en el extremo 5’ (evita corte por exonucleasas de RNA). 2. Permite la exportación nuclear del mRNA, o en su caso la retención en el núcleo. 3. Posibilita el inicio de la traducción en mRNAs eucariontes. 4. Promueve la degradación de mRNAs cuando están las señales celulares apropiadas. Remoción de intrones (splicing) Proceso co-transcripcional (elongación) Remoción de intrones (splicing) Intrón: Secuencia del pre-mRNA que se remueve en el splicing. No forma parte del mRNA maduro. Exón: Secuencia del pre-mRNA que permanece en el mRNA maduro. UTR: región no traducible del mRNA, puede ser 5’UTR o 3’UTR. Las UTRs son parte de los exones. ORF: marco abierto de lectura (open reading frame), secuencia del mRNA que contiene los codones que codifican la secuencia de aminoácidos de una proteína. Exón 1 Intrón 1 Exón 2 Intrón 2 Exón 3 pre-mRNA Corte y empalme mRNA maduro 5’ UTR ORF 3’ UTR Tipos de splicing: Por Spliceosoma Autocatalítico Intrones del Grupo II Autocatalítico Intrones del Grupo I ¡El RNA puede tener actividad catalítica! Ribozimas Intrones autocatalíticos Grupo I Grupo II Requiere guanosina o guanilato externo. Requiere una adenina dentro de su secuencia. El Intrón se libera como cadena con ambos extremos libres El Intrón se libera como lariat. Un solo extremo 3’ libre. Por Spliceosoma Secuencias necesarias para el splicing Existen secuencias específicas en los límites exón-intrón (en el sitio 5’ y 3’ para el splicing); así como una Adenina en el contexto de secuencia (sitio de ramificación) que promueve la catálisis. Reacción de Splicing El Spliceosoma El spliceosoma es una “maquinaria dinámica” formada por más de 100 proteínas y 5 snRNAs (RNAs pequeños nucleares) que se unen en partículas pequeñas ribonucleoproteícas (snRNPs) U1 reconoce el sitio 5’ de splicing U2 se une al sitio de formación de la rama Un complejo con U4, U5 y U6 se une al spliceosoma Se libera U1 y U4, primera reacción de transesterificación y formación del lariat Segunda reacción de transesterificación: unión de los exones y liberación del intrón y el spliceosoma. La interacción entre los snRNAs y el pre-mRNA permite la remoción de intrones por el spliceosoma U1 y U6 interactúan con el sitio 5’ del intrón U2 interactúa con el sitio de formación de la rama La interacción entre U6 y U2 acercan ambos sitios 1a reacción de transesterificación U5 acerca los extremos de los exones 2a reacción de transesterificación El splicing alternativo permite que a partir de un gen se produzcan distintas proteínas similares DNA pre-mRNA mRNA Proteína Tipos de splicing alternativo Splicing constitutivo Salto de exón Retención de intrón Exones mutuamente excluyentes Sitio de splicing 5’ alternativo Sitio de splicing 3’ alternativo Consecuencia de mutaciones en intrones Poliadenilación del extremo 3’ del mRNA El pre-mRNA es cortado 11-30 nt rio abajo de la secuencia consenso en la 3’ UTR Poliadenilación en el extremo 3’ del pre-mRNA genera la cola del poli (A) La poliadenilación del extremo 3’ está acoplada a la terminación de la transcripción La poliadenilación del extremo 3’ está acoplada a la terminación de la transcripción La poliadenilación del extremo 3’ está acoplada a la terminación de la transcripción La poliadenilación del extremo 3’ está acoplada a la terminación de la transcripción Mucho procesamiento después... ¿Cuál es la importancia del procesamiento de mRNA? La salida del mRNA al citoplasma es coordinada por proteínas unidas a la molécula: CBC: Complejo de unión al 5’ Cap (Cap Binding Complex) EJC: Complejo de unión de exones (Exon Junction Complex) PABP: Proteína de unión a la cola de poli(A)(Poly(A) Binding Protein) Resumen sobre Transcripción Procariontes Eucariontes 1. Todas las especies de RNA son sintetizadas por la misma especie de RNA polimerasa. 1. Hay 3 diferentes RNA polimerasas responsables de la transcripción de diferentes moléculas de RNA 2. El mRNA se traduce durante la transcripción. 2. El mRNA es procesado antes de ser transportado a citoplasma (adición de CAP, cola de poliA, splicing) 3. Los genes son segmentos contiguos de DNA alineados ininterrumpidamente con el RNA traducido a proteína. 3. Los genes frecuentemente se interrumpen por intrones. 4. Los mRNAs son frecuentemente policistrónicos (operones) 4. Los mRNAs son monocistrónicos 5. La RNA polimerasa solo requiere a sigma para reconocer el promotor 5. Las RNA polimerasas requieren múltiples factores de transcripción adicionales para reconocer el promotor. 6. No hay procesamiento co- transcripcional del mRNA, pero sí hay procesamiento posttranscripcional de tRNA y rRNA 6. El mRNA sufre procesamiento extenso durante la transcripción. Hay procesamiento post-transcripcional de tRNA y rRNA.