BIOSINTESIS DE PROTEINAS
Anuncio

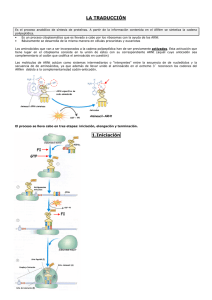
BIOSINTESIS DE PROTEINAS TRADUCCIÓN: Secuencia de ARNm → Secuencia de aminoácidos = Proteína CÓDIGO GENÉTICO: - Universal - 4 bases codifican para → 20 aminoácidos (AA) - Secuencia 3 bases ARNm: codon → 1 aminoácido (AA) - Los 2 primeros nucleótidos son imprescindibles. - Muy importante la identificación del nucleótido de inicio. - Frecuencia de error =<10-4 ESQUEMA GENERAL: ADN → Transcripción ARNm (lect. 5` a 3`, sínt. 3` a 5`) → Proteinas (sínt. NH2 a COO-) Traducción - Ribosomas: ARNr y proteinas - ARNt, asociado a un aminoácido ARN DE TRANSFERENCIA: - Nexo entre ARNm y proteína - Específico para cada AA Estructura: - Estr. 2ria en Hoja de trébol (mantenida evolutivamente) - 73 – 93 nucleótidos - Alg. Alg bases son no comunes: Dihidrouridina Dihidrouridina, Inosina Inosina,… - Nucleótidos apareados: dobles cadenas - Nucleót. no apareados: lazos (TψC, DHU, Anticodon) -Tallo aceptor: extr.3` no apareado (CCA) es el sitio de unión del AA. -Estr. tridimensional: L con el tallo aceptor y el anticodon enfrentados. - Teoría del balanceo (Ej: alanina: GCU, GCC y GCA) ACTIVACIÓN DEL ARNt y AMINOACIL-ARNt-SINTETASA AA-ARNt-Sintetasa AA ARNt Sintetasa - Realiza la traducción prop. dicha - AA-ARNt: unión éster -2`o3`de la ribosa de la adenina del ARNt y COO- del AA: COO 1)- AA + ATP → AA-AMP + PPi 2) AA-AMP 2)AA AMP + ARNt + PPi → AA-ARNt AA ARNt + AMP + 2 Pi Reacción general (1+2)- AA+ ATP + ARNt → AA-ARNt + AMP + 2 Pi -La La mayoría del ARNt esta unido a AA. -Prueba de Lectura: sitio activo (discrimina AA mayores) y sitio de prueba (elimina AA menores). - Reconoce secuencia del tallo aceptor y alg. veces también del anticodon. RIBOSOMAS -Complejo Complejo supramolecular de ARNr y Proteínas - ARNr función catalítica principal y Proteínas función estructural. - Ribosoma (E.Coli): (E Coli): subs 30S y 50S → 70S 30S: ARN 16S (1500 nucl.) y 21 prot 50S: ARN 5S y 23S y 33 prot. Eucariotas: 40S y 60S= 80S - Poseen 3 sitios de unión para ARNt: A, P y E TRADUCCIÓN: 3 pasos: - Iniciación - Elongación - Terminación INICIACIÓN -Ensamble de ambas subs. del ribosoma, AA-ARNt de iniciación y ARNm. ARNm -Requiere un ARNt específico con Met (formil-Met en Proc.) que reconoce el codon de iniciación AUG: Secuencia Shine-Dalgarno ((Proc)) o 1er AUG desde 5`-3` ((Euc). ) - Eucariotas son monocistrónicos y Procariotas son policistrónicos: una moléc. ARNm → más de una proteina. p - 3 factores de Iniciación: IF3 e IF1 se unen a la sub 30S,, IF2 ((tipo de Prot. G) se une a GTP y permite la unión del AA-ARNt de inic. al sitio P de la sub.30S, posterior unión de sub 50S y liberación de factores ELONGACIÓN -El ARNm se lee de 5` a 3` y la sínt. de Prot. crece desde NH2 a COO-Comienza en el sitio P con el ARNt de inic. y el sitio A libre. -Intervienen 2 fact. de elongación: EF-Tu (tipo de Prot. G) que se une a GTP y a un AA-ARNt; EF-Ts cataliza el intercambio de GDP por GTP del EF-Tu. -El AA-ARNt se une al sitio A, une la Met de iniciación y de allí se mueve hacia el sitio P desplazando a la otra molec. de ARNt sin el AA hacia el sitio E →Crecim. de la cadena polipeptídica. -Enlace peptídico: peptidil transferasa asociada a la sub 50S. -Interacción codon-anticodon determina el AA-ARNt a ocupar el sitio A. -El desplaz. del ARNt desde A a P desplaza al ribosoma a través del ARNm EF-Tu-GTP-AAEF T GTP AA ARNt TERMINACIÓN -Secuencia finalizadora del codon del ARNm: UAA, UAG, UGA - 3 fact. de terminación (Proc): RF1 (reconoce UAA, UAG), RF2 (reconoce UAA y UGA) y RF3 (tipo Prot. G) que une GTP y refuerza las uniones de los otros fact. con el codon de ARNm. - El heterodímero altera la activ. de la peptidil transferasa: se hidrata el sitio donde actúa la enzima. - En Euc. hay un solo factor: RRF. RRF ANTIBIÓTICOS: Ó muchos actúan inhibiendo la sínt. de Proteínas Liberación de RRF, de subunidades del ribosoma y de la cad. polipeptídica. PRINCIPALES DIFERENCIAS ENTRE SINTESIS DE PROTEINAS EN EUCARIOTAS (E) Y PROCARIOTAS (P). 1- Tamaño de los ribosomas: E- 80S (40-60) P- 70S (30-50) 2- AA-ARNt iniciador: E- Met P- formil-Met 3- Reconocimiento de la Iniciación:E- 1er AUG desde 5`-3`. P- Secuencia Shine-Dalgarno y AUG. 4- ARNm: E- monocistrónico P- policistrónico 5- Factores de Iniciación: E: varios (> de 3). P: 3. 6- Fact. de Terminación: E: 1. P: 3. COSTO ENERGÉTICO DE LA SÍNT. SÍNT DE PROTEÍNAS Por cada AA incorporado a la cadena polipeptídica: 1 ATP → 1 AMP + 1Pi +1Pi --------- Activación ARNt (AA-ARNt-sintetasa) 1 GTP → 1 GDP + 1 Pi …..Iniciación Iniciación (IF2) (1 x prot) 2 GTP → 2 GDP + 2 Pi …..Elongación (EF-Tu y EFg) 1 GTP → 1 GDP + 1 Pi …..Terminación (RF3) (1 x prot) ATP: formación de nuevos enlaces GTP: cambios conformacionales MODIFICACIONES DE PROTEÍNAS: Cotraduccionales (prot. unidas al ribosoma, durante la traducción) o postraduccionales. Desformilación Glicosilación Fosforilación Acetilación Ca bo ac ó Carboxilación Metilación Hidroxilación Acilación Etc… Glicosilación: unión covalente de uno o varios glicosilos. Se glucosilan proteínas que se van a secretar o son de membrana, y nunca se da en los procariotas. Co o Postraduccional. - favorecen o estabilizan la conformación final de la proteína extracelular o de membrana - aumentan la vida media de la proteína incrementando su estabilidad y su resistencia a la digestión por las proteasas. - aumentan la solubilidad en un medio acuoso - aportan las estructuras que van a reconocer algunos receptores (por ejemplo ejemplo, los antígenos) Fosforilación Postraduccional Afecta OH de Ser, y Thr y Tyr Acetilación: Introduce un acetilo en grupo amino (Met y Lys) Carboxilación -En C beta de Asp pog gamma de Glu. -Carboxi-Asp o carboxi-Glu: quelantes de Ca imprescindibles en la coagulación Metilación - Extremo NH2 de Lys y o COO- de Glu. - SAM (S-Adenosil-Lmetionina: donador de metilos) PROTEÍNAS ASOCIADAS A MEMBRANAS: Se sintetizan a través de ribosomas asociados a membranas: - Procariotas: Membr. Plasmática. - Eucariotas: Ret.Endoplásmico →Ap.Golgi→Vesículas de excreción. Ej. Síntesis de una proteína viral asociada a la membrana. En este proceso algunas de las proteínas celulares son covalentemente modificadas con otra pequeña proteína (peso molecular 11 kDa) llamada SUMO ( (small ll ubiquitin-related bi iti l t d modifier) difi ) Proyecto proteoma humano Genoma – constituido por ADN, es el conjunto de genes Transcriptoma - genes se transcriben en forma de ARN mensajero Proteoma -conjunto de todas las proteínas obtenidas directamente del ARNm o tras diversas d e sas ttransformaciones a s o ac o es Metaboloma -conjunto de los diferentes metabolitos existentes, con diversas naturalezas químicas Genómica → Proyecto Genoma Humano → identificación y secuenciación de alrededor de 40.000 genes Sin embargo, embargo las proteínas son las verdaderas expresiones funcionales del genoma Dogma: 1 gen=1 proteína → 1 gen =varias proteínas – -mecanismos de maduración de ARN y modificación postraduccionales de las proteínas . -Relación entre número de proteínas y genes: 1-2 en bacterias 3 en levaduras 6-8 en Humanos (posiblemente)- 40000 genes presentes y más de 100.000 proteínas conocidas en la actualidad. PPH: desde abril del 2001 - más de mil millones de dólares de financiación- Objetivos: creación de un Catálogo general de proteínas humanas (variantes posibles, interacciones entre proteínas y proteínas o entre proteínas y ácidos nucleicos, mecanismos de acción, etc.)