Síntesis de proteínas
Anuncio

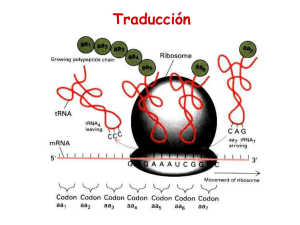
SECCIÓN L Síntesis de proteínas L 1 Aspectos de la síntesis de proteínas Notas clave Interacciones codón-anticodón En la hendidura del ribosoma, las tres bases del codón en el mRNA y el anticodón en el par de tRNA de una manera antiparalela. Si se modifica la base del 5′-anticodón, el tRNA puede interactuar por lo general con más de un codón. Balanceo La hipótesis del balanceo explica cómo se forman pares de bases no estándar entre bases 5′-anticodón modificadas y 3′-codón. Cuando el nucleósido que se balancea es inosina, el tRNA puede formar pares de bases con tres codones: los que terminan en A, C o U. Sitio de unión a ribosomas Este sitio (RBS) es una secuencia ascendente a partir del codón de inicio en el mRNA bacteriano que forma pares de bases con una secuencia complementaria cerca de la terminación 3′ del rRNA 16S para posicionar el ribosoma para el inicio de la síntesis de proteínas. También se le conoce como secuencia Shine-Dalgarno (sus descubridores). tRNA iniciador Un tRNA especial (tRNA iniciador) reconoce al primer codón AUG y se le usa para iniciar la síntesis de proteínas en procariotas y eucariotas. En bacterias, la metionilo-tRNA sintetasa carga primero al tRNA con metionina. Luego, la transformilasa convierte a la metionina en N-formilmetionina. En eucariotas, no se modifica la metionina en el iniciador tRNA. Hay diferencias estructurales entre el iniciador tRNA de E. coli y el que inserta residuos Met internos. Polisomas Los polirribosomas (polisomas) se forman en un mRNA cuando se anexan ribosomas sucesivos, se empieza la traducción y luego se mueven en dirección 5′→3′ a lo largo del mRNA. Un polisoma es un complejo de múltiples ribosomas en varias etapas de traducción en una molécula de mRNA. Temas relacionados (J1) Procesamiento de rRNA y ribosomas (J2) Procesamiento del tRNA y otros RNA pequeños (K2) Estructura y función de tRNA (L2) Mecanismo de la síntesis de proteínas Interacción codón-anticodón Balanceo El anticodón es uno de los bucles de tallo del tRNA e interactúa con una tripleta complementaria de bases en el mRNA, el codón, cuando se acercan los dos en la hendidura del ribosoma (sección J1). La interacción es de naturaleza antiparalela (figura L1-1). Cuando se encontró que algunas moléculas muy purificadas de tRNA interactuaban con más de un codón, esta capacidad se correlacionó con la presencia de nucleósidos modificados en la posición 5′-anticodón, sobre todo la inosina (sección K2). La inosina (I) se forma mediante procesamiento posterior a la transcripción (sección J2) de la adenosina cuando ocurre en esta posición. Esto lo realiza la deaminasa de adenosina específica de tRNA, que convierte al grupo 6-amino de la base adenina en uno ceto (hipoxantina). Crick sugirió la hipótesis del balanceo para explicar la redundancia del código genético. Se dio cuenta mediante la construcción de un modelo que la base del 5′-anticodón podía moverse más que las otras dos bases y formar pares de bases no estándar, siempre y cuando las distancias entre las unidades de ribosa fueran menores de lo normal. En el cuadro L1-1 se muestran sus predicciones específicas y las observaciones reales. No se permiten pares de bases purina-purina ni pirimidina-pirimidina, porque la distancia entre las ribosas sería incorrecta. Ningún tRNA podría reconocer más de tres codones, por lo que se necesitarían cuando menos 32 tRNA para decodificar los 61 codones, excluidos los de detención. Los tRNA pueden reconocer uno, dos o tres codones, depen- 147 148 SECCIÓN L – SÍNTESIS DE PROTEÍNAS 3′ metionina co muestra conservación con el rRNA 16S bacteriano, esta sección está ausente. En cambio, el capuchón en el extremo 5′ puede tener una función similar en eucariotas. tRNA iniciador Anticodón Codón Figura L1-1. Interacción codón-anticodón. diendo de su base de balanceo (base 5′-anticodón). Si ésta es C, sólo reconoce el codón con terminación G. Si es G, reconoce dos codones con terminación en U o C, que se modifican más adelante, forma pares con A o G. El nucleósido de balanceo no es nunca A, porque se convierte en I que luego forma pares con A, C o U. Sitio de unión a ribosomas En el mRNA bacteriano, una secuencia conservada, con abundancia de purinas, por lo general 5′-AGGAGGU-3′, se encuentra en 8 a 13 nt ascendente a partir del primer codón que se traduce (el codón de inicio). Esta secuencia forma pares de bases con la terminación 3′ del rRNA 16S en la pequeña subunidad del ribosoma (5′-ACCUCCU-3′). Se le llama sitio de unión a ribosomas o secuencia de ShineDalgarno, que recibe este nombre por sus descubridores, y se cree que coloca al ribosoma de manera correcta en relación con el codón de inicio. Aunque el rRNA 18S eucarióti- En eucariotas y procariotas, la metionina siempre es el primer aminoácido incorporado en una cadena de proteínas, aunque en bacterias y en mitocondrias y cloroplastos esta Met se modifica a N-formilmetionina. Un tRNA iniciador especial, diferente del que forma pares con codones AUG en el resto de la región codificante, es el que reconoce al codón de inicio AUG. En E. coli hay diferencias sutiles entre estos dos tRNA (figura L1-2). Hay bases que no han formado pares en la terminación del tronco aceptor del aminoácido que se requieren para la formilación. También hay tres pares de bases G-C en el brazo del anticodón que son necesarios para la entrada en el sitio P del ribosoma (sección L2). Por último, el tRNA iniciador permite más flexibilidad en la formación de pares de bases (balanceo) porque carece de la A alquilada en el bucle del anticodón y, por tanto, sólo reconoce AUG como codón de inicio, pero también GUG y en muy pocas ocasiones UUG, que ocurre con mucho menos frecuencia en los mRNA bacterianos. El tRNA no iniciador es menos flexible y sólo puede formar pares con los codones AUG. La misma metionil-tRNA sintetasa carga con Met a ambos tRNA (sección K2) para dar la metionil-tRNA (Met-tRNA), pero la enzima transformilasa modifica sólo al Met-tRNA iniciador (Met-tRNAfMet) para dar N-formilmetionil-tRNAfMet (fMet-tRNAfMet ). La unión al grupo N-formil se parece a un enlace péptido y puede ayudar a que este tRNA iniciador entre en el sitio P del ribosoma, un requisito esencial para el inicio, mientras que todos los otros tRNA entran en el sitio A (sección L2). Polisomas Cuando un ribosoma ha empezado la traducción de una molécula de mRNA (sección L2) y se ha desplazado de 70 a 80 nt en sentido descendente a partir del codón de inicio, un segundo ribosoma puede integrarse en el sitio de inicio y empezar a traducir el mRNA. Cuando este segundo ribo- Cuadro L1-1. Predicciones originales del balanceo. Base 5′-anticodón A Lectura prevista de la base 3′-codón Observaciones U A convertida en I mediante anticodón deaminasa No hay balanceo, formación normal de pares de bases C G G CyU G y G modificada, pueden formar pares con C y U U AyG No se encuentra U como base 5′-anticodón I Ay Balanceo como se predijo. Inosina (I) puede Cy reconocer 3′-A, -C o -U U L1 – ASPECTOS DE LA SÍNTESIS DE PROTEÍNAS (a) Met formil 149 (b) Met Adenosina alquilada Anticodón Anticodón Figura L1-2. Los metionina-tRNA de E coli. (a) El tRNA fMet-tRNAfMet iniciador; (b) el metionil-tRNA Met-tRNAMet. soma se retira, un tercero puede empezar, y así sucesivamente. A los ribosomas múltiples en un solo mRNA se les denomina polisomas (forma corta de polirribosomas) y puede haber hasta 50 en algunos mRNA, aunque no pueden acercarse a más de 80 nt. L 2 Mecanismo de la síntesis de proteínas Notas clave Revisión general La síntesis de proteínas recorre tres etapas: • Inicio: el ensamblado de un ribosoma en un mRNA. • Elongación: ciclos repetidos de entrega de aminoácidos, formación de enlaces péptidos y movimiento a lo largo del mRNA (translocación). • Terminación: la liberación de la cadena polipeptídica. Inicio En las bacterias, para el inicio se necesitan las subunidades grandes y pequeñas de ribosomas, el mRNA, el tRNA iniciador, tres factores de inicio (IF) y GTP. IF1 e IF3 se unen a la subunidad 30S y evitan la fijación a la subunidad grande. IF2 + GTP pueden unirse entonces y ayudar al tRNA iniciador a que se una después. Esta pequeña subunidad puede ahora anexarse a un mRNA mediante su sitio de unión a ribosomas. El tRNA iniciador puede formar pares de bases con el codón de inicio AUG, que libera IF3, creando así el complejo de inicio 30S. Luego se une la subunidad grande, desplazando IF1 e IF2 + GDP, creando el complejo de inicio 70S, que es el ribosoma ensamblado por completo en la posición correcta del mRNA. Elongación La elongación comprende los tres factores de elongación (EF), EF-Tu, EF-Ts y EF-G, además de GTP, tRNA cargados y el complejo de inicio 70S (o su equivalente). Consta de tres pasos: • Se entrega un tRNA cargado como un complejo con EF-Tu y GTP. Se hidroliza al GTP y se libera • • EF-Tu-GDP, que puede reutilizarse con la ayuda de EF-Ts y GTP (mediante el ciclo de intercambio EF-Tu-EF-Ts). La peptidil transferasa, una actividad de la subunidad grande de rRNA, elabora un enlace peptídico mediante transferencia del polipéptido en el sitio P al aminocil-tRNA en el sitio A sin necesidad de energía adicional. Con la energía de la hidrólisis de GTP, la translocasa (EF-G) mueve el ribosoma un codón a lo largo del mRNA, expulsando el tRNA sin carga y transfiriendo el polipéptido en crecimiento de regreso hacia el sitio P. Terminación Los factores de liberación (RF1 o RF2) reconocen los codones de detención y, con ayuda de RF3, causan que la peptidil transferasa transfiera al polipéptido finalizado a una molécula de agua, liberándola. El factor de reciclaje de los ribosomas ayuda a disociar las subunidades ribosómicas del mRNA. Temas relacionados (J2) Procesamiento del tRNA y de otros RNA pequeños (K2) Estructura y funcionamiento del tRNA Revisión general No toda la secuencia mRNA se traduce en proteínas, sólo la región desde el codón de inicio (e incluido éste) hasta el de terminación. A las regiones ascendentes y descendentes de estos codones se les denomina regiones no traducidas de 5′ (5′-UTR) y no traducidas de 3′ (3′-UTR), respectivamente, y son importantes para regular la traducción. El mecanismo real de síntesis de proteínas puede dividirse en tres etapas: • Inicio: el ensamblado de una ribosa en una molécula de mRNA. 150 (L1) Aspectos de la síntesis de proteínas (L3) Inicio en eucariotas • Elongación: ciclos repetidos de adición de aminoácidos. • Terminación: liberación de la nueva cadena de proteínas. Éstas se ilustran en las figuras L2-1 a L2-3 y requieren las actividades de diversos factores. En bacterias, se usan las abreviaturas IF o EF para los factores de inicio y elongación, respectivamente, mientras que en eucariotas se usan eIF y eEF. Hay detalles diferentes en el mecanismo entre bacterias y eucariotas, y casi todos se presentan en la etapa de inicio. En esta sección se describe el mecanismo en las bacterias y en la siguiente (L3) se tratan las diferen- L2 – MECANISMO DE LA SÍNTESIS DE PROTEÍNAS cias en eucariotas. La síntesis de proteínas en arqueas es un mosaico, que muestra características de ambos. Inicio El propósito de este paso es ensamblar un ribosoma completo en una molécula de mRNA en el punto de inicio correcto, el codón de inicio. Los componentes que intervienen son las subunidades ribosómicas grandes y pequeñas, el mRNA, el tRNA iniciador en su forma cargada (sección L1), tres factores de inicio y GTP. Los factores de inicio IF1, IF2 e IF3 son apenas una décima parte más abundantes que los ribosomas. A continuación se describe la secuencia de eventos (figura L2-1): • IF1 e IF3 se unen a una subunidad 30S libre. En ausencia de mRNA, IF3 ayuda a evitar la formación de un ribosoma inactivo al detener la unión de una subunidad grande. Más tarde ayuda a que el iniciador tRNA cargado co- Factores de inicio Subunidad 30S Región complementaria 16S tRNA iniciador Complejo de inicio 30S Subunidad 50S Sitio A Sitio P 151 Complejo de inicio 70S Figura L2-1. Inicio de síntesis de proteínas en E. coli. 152 • • • • SECCIÓN L – SÍNTESIS DE PROTEÍNAS rrecto entre en el sitio de unión apropiado (sitio P). IF1 bloquea el sitio al que se uniría el tRNA no iniciador. IF2 forma un complejo con GTP y luego se une a una subunidad pequeña para ayudar al enlace del tRNA iniciador cargado. La subunidad 30S y los IF asociados se unen a una molécula de mRNA al reconocer el sitio de unión del ribosoma (RBS) en el mRNA (sección L1). El tRNA iniciador se une después al complejo en el sitio P (consúltese más adelante) al formar pares de bases de su anticodón con el primer codón AUG del mRNA. En este punto, puede liberarse IF3 para que puedan cumplirse sus funciones de mantener separadas las subunidades, ayudar en su unión al mRNA y dirigir al tRNA iniciador. A esto se le denomina complejo de inicio 30S. Ahora la subunidad 50S puede unirse, desplazando IF1 e IF2. En este paso se hidroliza el GTP. Al complejo formado al final de la fase de inicio se le llama complejo de inicio 70S. Como se muestra de manera simplificada de las figuras L2-1 a L2-3, el ribosoma ensamblado tiene dos sitios de fijación a tRNA. Son los sitios A y P para sitios aminoacilo y peptidilo. El sitio A es donde se unen las moléculas entrantes de aminoacil-tRNA y el P es donde suele encontrarse la cadena de polipéptidos en crecimiento. Estos sitios se localizan en la hendidura de la subunidad pequeña (sección J1) y contienen codones adyacentes a los que se les está traduciendo. Un resultado importante del inicio es la colocación del tRNA iniciador en el sitio P. Es el único tRNA que hace esto, porque todos los demás deben entrar en el sitio A (sección L1). La colocación correcta del codón AUG iniciador fija el marco de lectura por el resto del proceso de traducción (sección K1). Elongación Con la formación del complejo de inicio 70S, el ciclo de elongación puede empezar. Se subdivide en tres pasos: i) entrega de aminoacil-tRNA; ii) formación de un enlace peptídico, y iii) translocación. Estos tres pasos se muestran en la figura L2-2, empezando con un sitio P ocupado y uno A vacío. Comprende tres factores de elongación, EF-Tu, EF-Ts y EF-G; todos ellos unen GTP o GDP. EF-Ts y EF-G son casi tan abundantes como los ribosomas, pero EF-Tu es casi 10 veces más abundante. i) Entrega de aminoacil-tRNA. Se requiere EF-Tu para la entrega de aminoacil-tRNA al sitio A y, en este paso, se consume energía en la forma de hidrólisis de GTP a GDP+Pi. El complejo liberado EF-Tu-GDP se regenera con la ayuda de EF-Ts. En el ciclo de intercambio EFTu-EF-Ts, EF-Ts desplaza a GDP y después GTP desplaza a EF-Ts. El complejo resultante, EF-Tu-GTP, ahora tiene la capacidad de unir otro aminoacil-tRNA y de entregarlo al ribosoma. Todos los aminoacil-tRNA pueden formar este complejo con EF-Tu, excepto el tRNA iniciador. Formación de un enlace peptídico. Después de la entrega de aminoacil-tRNA, se ocupan los sitios A y P y los dos aminoácidos que han de unirse quedan en posiciones cercanas. La actividad peptidil transferasa de la subunidad 50S puede formar ahora un enlace peptídico entre estos dos aminoácidos sin necesidad de más energía, porque ésta, en la forma de ATP, se usó para cargar el tRNA (sección K2). Lo interesante es que el sitio catalítico de la peptidil transferasa es parte del rRNA 23S (o 28S) en la subunidad grande y no requiere una proteína. Por tanto, este rRNA es una ribozima (sección J2). iii) Translocación. Un complejo de EF-G (translocasa) y GTP se une al ribosoma y, en un paso que consume energía, se expulsa al tRNA del sitio P, se desplaza al peptidil-tRNA del sitio A al P y el mRNA se mueve un codón en relación con el ribosoma. Se liberan GDP y EF-G y este último es reciclado. Ahora hay un codón nuevo en el sitio A vacío. En bacterias, se desplaza primero al tRNA descargado al sitio E (sitio de salida), que se localiza sobre todo en la subunidad 50S, y se le expulsa cuando se une el siguiente aminoacil-tRNA. De esta manera, el ribosoma mantiene contacto con el mRNA vía 6 bp, lo que puede cambiar las posibilidades de desplazamiento de marco: el deslizamiento de una o dos bases, que cambiaría el marco de lectura y la secuencia de proteínas resultantes (sección E2). No hay evidencia de un sitio E en eucariotas. ii) Una vez que se ha completado un ciclo del proceso de elongación de tres pasos, se repite hasta que uno de los tres codones terminación (o detención) aparece en el sitio A. De esta manera, se traduce el mRNA 5′→3′ y la proteína se sintetiza de la N-terminal a la C-terminal. Terminación Aparte de los pocos casos especializados en que pueden interpretarse como un aminoácido (sección K1), las especies de tRNA no suelen reconocer a los codones de detención. En cambio, las proteínas llamadas factores de liberación interactúan con estos codones y causan la liberación de toda la cadena de polipéptidos (figura L2-3). Los factores de liberación de clase I, RF1 y RF2, reconocen a los codones UAA y UAG, y UAA y UGA, respectivamente. Causan que la peptidil transferasa transfiera el polipéptido a agua en lugar de hacerlo a un aminoacil-tRNA; de este modo se libera la nueva proteína. El factor de liberación clase 2, RF3, promueve la disociación de RF1 o RF2 del ribosoma. Para retirar al tRNA sin carga del sitio P y liberar al mRNA, se necesita EF-G junto con factor reciclador del ribosoma (RRF) para la disociación completa de las subunidades. IF3 puede unir ahora la subunidad pequeña para evitar que se reformen los ribosomas 70S inactivos. Los ribosomas pueden estancarse en un mRNA si una base está dañada y resulta irreconocible (sección E1), si el L2 – MECANISMO DE LA SÍNTESIS DE PROTEÍNAS mensaje no tiene codón de detención o si no hay tRNA correcto suficiente para un codón en particular. Existen mecanismos para tratar con los problemas de mRNA dañados o truncados (acortados), a los que se les está traduciendo en proteínas defectuosas. En bacterias, se usa un RNA pequeño llamado tmRNA (RNA mensajero de transferencia), que tiene propiedades de tRNA y mRNA, para liberar al ribosoma estancado y asegurar la degradación de la proteína defectuosa. En primer lugar, el tmRNA se comporta como tRNA al entregar alanina al sitio A y permitir que tenga lugar la formación de un enlace peptídico. Luego ocurre la translocación, colocando otra parte del tmRNA en el sitio Sitio A vacío Peptidil tRNA en sitio P Codones Unión de aminocil-tRNA específico al sitio A Complejo AA-tRNA-GTP-EF-Tu Codones Complejo EF-Tu-EF-Ts Formación de enlace peptídico Ciclo de intercambio EF-Tu-Ts Centro de peptidil transferasa Codones Translocación de peptidil-RNA del sitio A al P 153 Complejo EF-G-GTP Codones Figura L2-2. Etapa de elongación de síntesis de proteínas en E. coli. 154 SECCIÓN L – SÍNTESIS DE PROTEÍNAS Codón de terminación Codones Factor de liberación 1 o 2 Factor de liberación 3 Codones Peptidil transferasa Cadena polipeptídica liberada Codones Factor de liberación Subunidad 50S Subunidad 30S Figura L2-3. Terminación de síntesis de proteínas en E. coli. A, donde se comporta como un mRNA que dirige la traducción de 10 codones y uno de detención contenido dentro de su propia secuencia. Por tanto, la proteína liberada tiene una etiqueta de 10 aminoácidos codificados por el tmRNA en su C-terminal, lo que la convierte en blanco para la degradación rápida. Existen mecanismos diferentes en eucariotas (sección L3). L 3 Inicio en eucariotas Notas clave Revisión general La mayor parte de las diferencias en el mecanismo de síntesis de proteínas entre bacterias y eucariotas se presenta en la etapa de inicio. Además, los eucariotas sólo tienen un factor de liberación (eRF) y su tRNA iniciador no sufre N-formilación, como en las bacterias. Rastreo El complejo eucariótico de la subunidad 40S de ribosoma se une al capuchón en la terminación 5′ del mRNA y luego se mueve 5′→3′, rastreando el mRNA para localizar el codón de inicio, AUG. Éste no siempre es el primero que encuentra, porque debe estar en el contexto de secuencias apropiadas. Puede usarse más de un AUG para iniciar, lo que lleva a proteínas diferentes. Inicio Abarca cuatro pasos principales que requieren cuando menos 12 eIF que caen en grupos funcionales diferentes. Entre ellos ensamblan el complejo de preinicio 43S, se unen al mRNA o reclutan a la subunidad 60S mediante el desplazamiento de otros factores. En contraste con las bacterias, el inicio requiere la unión del tRNA iniciador con la subunidad 40S antes de que se una al mRNA. Entre los controles importantes se incluyen la fosforilación de eIF2, que aporta el tRNA iniciador, y las proteínas unidoras eIF4E, que inhiben la unión de eIF4G, lo que bloquea el reclutamiento del complejo 43S. Elongación Esta etapa de la síntesis de proteínas es, en esencia, idéntica a la descrita para las bacterias (sección L2). Los factores EF-Tu, EF-Ts y EF-G tienen equivalentes eucarióticos directos a los que se les denomina eEF1α, eEF1βγ y eEF2, respectivamente, que realizan las mismas funciones. Terminación Los eucariotas sólo tienen un factor de liberación de clase 1 (eRF1) para la terminación de la síntesis de proteínas. Puede reconocer a los tres codones de detención. El factor de liberación de clase 2, eRF3, causa que eRF1 abandone el ribosoma. Se ponen en operación mecanismos de vigilancia para liberar a los ribosomas estancados o degradar a los mRNA defectuosos. Temas relacionados (J2) Procesamiento de tRNA y otros RNA pequeños (K2) Estructura y función del tRNA Revisión general Aparte de la síntesis de proteínas en las mitocondrias y los cloroplastos (que se cree que se originaron en bacterias simbióticas), los detalles de la traducción en eucariotas difieren un poco de lo descrito en la sección L2. Casi todas estas diferencias se encuentran en la fase de inicio donde interviene una cantidad mayor de factores de inicio eucarióticos (eIF). El encuentro del codón de inicio correcto requiere un proceso de rastreo porque no hay secuencia de unión a ribosomas. Aunque hay dos especies diferentes de tRNA para la metionina (una de ellas es el tRNA iniciador), la anexada no se convierte en N-formilmetionina. En el cuadro L3-1 se presenta una comparación de los factores que intervienen en bacterias y eucariotas. Rastreo La ausencia de un RBS reconocible en el mRNA eucariótico significa que el mecanismo para seleccionar el codón de inicio debe ser diferente. Marilyn Kozak propuso un modelo de rastreo en que la subunidad 40S, que ya porta el tRNA iniciador, se anexa a la terminación 5′ con capu- (L2) Mecanismo de la síntesis de proteínas chón del mRNA y rastrea en dirección 5′→3′ hasta que encuentra un AUG apropiado. Éste no siempre es el primero, porque debe estar en el contexto de la secuencia correcta (5′-gccRccAUGG-3′), donde R representa la purina y las minúsculas indican preferencia leve. La cercanía de la secuencia real que rodea al AUG de esta secuencia de consenso de Kozak determina la probabilidad de que se presente el inicio. Por tanto, es posible iniciar en más de un AUG con probabilidades diferentes que llevan a la síntesis de proteínas relacionadas con secuencias con terminación N diferentes. Inicio En la figura L3-1 se muestran los pasos y los factores para el inicio de la síntesis de proteínas en los eucariotas. Hay cuatro pasos principales: i) ensamblado del complejo de preinicio 43S mediante un complejo multifactores (MFC); ii) reclutamiento del complejo de preinicio 43S mediante el mRNA a través de interacciones en su terminación 5′; iii) rastreo para localizar el codón de inicio, y iv) reclutamiento de la subunidad 60S para formar el complejo 155 156 SECCIÓN L – SÍNTESIS DE PROTEÍNAS Cuadro L3-1. Comparación de factores de síntesis de proteínas en bacterias y eucariotas. Bacterias Eucariotas Función IF1, IF3 eIF1, eIF1A, eIF3, eIF5/eIF2, elF2B Union a subunidades pequeñas/entrega de tRNA iniciador IF2 eIF4B, eIF4F, elF4H Unión a mRNA eIF5B Desplazamiento de otros factores y reclutamiento de subunidades grandes EF-Tu eEF1α Entrega de tRNA aminoacil al ribosoma EF-Ts eEF1βγ Reciclado de EF-TU o eEF1α EF-G eEF2 Translocación eRF1 Reconocimiento de codón de terminación y liberación de cadenas de polipéptidos (clase 1) eRF3 Disociación de factores de clase 1 (clase 2) Factores de inicio Factores de elongación Factores de terminación RF1 RF2 RF3 de inicio 80S. Cuando menos 12 factores de inicio más o menos definidos intervienen en la síntesis de proteínas en eucariotas, y algunos tienen funciones análogas a los tres IF bacterianos. Pueden agruparse de varias maneras, pero es lógico hacerlo de acuerdo con el paso en que actúan: • Los que intervienen en el ensamblado del complejo de preinicio 43S, como eIF1, eIF1A, eIF2, eIF3 y eIF5. • Los que se unen al mRNA para reconocer el capuchón en la terminación 5′ y fusionar la estructura secundaria, como eIF4B y eIF4F. Este último es un complejo heterotrímero de una RNA helicasa a la que se le denomina eIF4A, una proteína de unión a capucha (eIF4E) y una proteína de estructura: eIF4G. • Los que reclutan a la subunidad 60S al desplazar otros factores, como el eIF5B, que libera otros cinco factores para que pueda unirse a la subunidad 60S. Tienen lugar los siguientes eventos, empezando con el complejo binario eIF2-GTP, formado por el eIF2B al reciclar el eIF2-GTP liberado en una etapa tardía durante el inicio. El tRNA iniciador se une para hacer un complejo ternario de tres componentes, el tRNAi iniciador, eIF2 y GTP. En levaduras, el complejo ternario forma parte de un MFC que contiene eIF1, eIF2-GTP-tRNAi, eIF3 y eIF5. La unión del MFC a la subunidad 40S libre es auxiliada por el eIF1A y al complejo resultante se le denomina complejo de preinicio 43S. En algunos eucariotas el complejo ternario puede unirse más tarde (a la subunidad 40S libre que contiene eIF1, eIF1A, eIF3 y eIF5). Obsérvese este orden diferente de ensamblado en eucariotas donde el tRNA iniciador se une a la subunidad pequeña antes de que lo haga el mRNA (compárese con la figura L2-1). Antes de que este complejo grande pueda unirse al mRNA, el último debe haber interactuado con eIF4B y eIF4F (que reconoce el capuchón en 5′ gracias a su subunidad eIF4E) y, usando energía del ATP, se le ha desenrollado, además de que la subunidad eIF4A debe retirársele su estructura secundaria. El eIF4H es de ayuda en esto. La subunidad de estructura eIF4G del eIF4F puede unirse a PABI, la proteína unidora citoplásmica poli(A) (sección J3) y estimular la traducción al hacer circular el mRNA (no de manera covalente, sino espacial a través de contactos proteína-proteína). La estimulación con PABI de la traducción también proporciona una revisión de que el ribosoma está uniéndose a un mRNA intacto que tenga un capuchón en la terminación 5′ y una cola 3′-poli(A). El segundo paso importante ocurre cuando el complejo de preinicio 43S se ha unido al complejo mRNA mediante interacciones entre eIF4G y eIF3. En el tercer paso, el ATP se hidroliza mientras se rastrea el mRNA para encontrar el codón de inicio AUG. Éste suele ser el primero, aunque no siempre es el caso (consúltese la explicación en párrafos anteriores). En el cuarto paso, eIF5B debe desplazar a eIF1, eIF2, eIF3 y eIF5 para permitir que la subunidad 60S se una y que el GTP se hidrolice. Se liberan eIF1A y eIF5B cuando el primero ha promovido la unión de la subunidad 60S para formar el complejo de inicio 80S completo. El eIF2B recicla el complejo liberado eIF2-GDP y la fosforilación de la subunidad-α de eIF2 regula el ritmo de reciclado (y, por tanto, el de inicio de síntesis de proteínas). Ciertos eventos, como la infección vírica y la producción resultante de interferón, causan inhibición de la síntesis de proteínas al promover la fosforilación de eIF2. Otro punto de regulación comprende unión de eIF4E/proteínas inhibidoras que pueden bloquear por completo el ensamblado de eIF4F en algunos mRNA (sección L4). L3 – INICIO EN EUCARIOTAS Algunos virus de RNA usan un proceso independiente de capuchón para ensamblar ribosomas en sitios de entrada internos ribosómicos (IRE) en sus RNA policistróni- 1 1A 2 elF2 3 elF3 5 elF5 5B cos (secciones M1 y M4). Los IRES también se encuentran en algunos genes celulares, en especial los que intervienen en la supervivencia bajo estrés. elF1 elF1A 3 1 2 5 1 3 elF5B GTP 5 GTP 2 Subunidad ribosómica 40S 1A Complejo multifactorial tRNA i 1 3 5 2 GTP 1A Complejo de preinicio 43S cap mRNA * Codón de inicio elF4B elF4F Poli(A) An AUG Ciclo eIF2B 1 3 ATP Unidad ribosómica 40S 5 GTP 2 * ADP + Pi 1A An AUG ATP Rastreo + Monosoma inactivo ADP + Pi 1 Subunidad ribosómica 60S 3 5 2 * GTP 1A An AUG Complejo de preinicio 48S 5B 1 Subunidad ribosómica 60S * 3 5 GDP + Pi 2 5B Sitio A 1A An AUG Sitio P 1A * AUG 157 An Complejo de inicio 80S Figura L3-1. Inicio de síntesis de proteínas en eucariotas. 5B 158 SECCIÓN L – SÍNTESIS DE PROTEÍNAS Elongación El ciclo de elongación en la síntesis de proteínas en bacterias y eucariotas es muy similar. Se requieren tres factores con propiedades similares a su contraparte bacteriana (cuadro L3-1). eEF1α, eEF1βγ y eEF2 tienen funciones equivalentes, como se describió en la sección L2 para EF-Tu, EF-Ts y EF-G. Terminación En eucariotas, un factor único de liberación clase 1, eRF1, reconoce a los tres codones de terminación y realiza las labores ejecutadas por RF1 (o RF2) en bacterias. El factor de liberación clase 2, ERF3, causa la liberación de eRF1 y requiere GTP para estar activo. Los mecanismos de vigilancia del mRNA operan en eucariotas para liberar ribosomas atascados o degradar mRNA defectuosos que tal vez hayan surgido debido a mutaciones (sección E2). La degradación imparable libera ribosomas atascados que han traducido un mRNA al que le falta un codón de detención y esté atrapado en la terminación 3′. El producto de la traducción es defectuoso porque tiene polilisina en su C-terminal debido a la traducción de la cola poli(A). Un factor proteínico (Ski7) ayuda a disociar el ribosoma y reclutar una exonucleasa 3′→5′ para degradar el mRNA defectuoso. La proteína defectuosa marcada con polilisina también se degrada con rapidez. En NMD, se reconocen los mRNA que contienen codones de detención prematuros debido a la presencia de complejos proteínicos depositados en las uniones exónexón por corte y empalme en el núcleo (sección J3). En los mRNA normales, estos complejos son desplazados por el primer ribosoma que traduzca el mRNA, y después se alcanza el codón de detención. En los mRNA defectuosos, se alcanza el codón de detención prematuro antes de que se desplacen todos los complejos y esto causa la eliminación del capuchón del mRNA y la degradación 5′→3′ consecuente. NMD también se usa para regular la expresión de transcriptos normales. L 4 Traducción, su control y eventos posteriores Notas clave Control de la traducción en bacterias En bacterias, puede afectarse el nivel de traducción de cistrones diferentes mediante: i) la unión de proteínas que evitan el acceso de ribosomas; ii) la estabilidad relativa a las nucleasas de regiones del mRNA policistrónico, y iii) la unión de moléculas de mRNA cortas antisentido. Control de la traducción en eucariotas En eucariotas, la unión de las proteínas también puede enmascarar al mRNA y evitar la traducción, y las repeticiones de secuencias como 5′-AUUUA-3′ pueden hacer inestable al mRNA y disminuir su frecuencia de traducción. Los microRNA con acción en trans en el complejo silenciador inducido por RNA pueden tener como destino mRNA específicos para degradación por la unión a secuencias complementarias en UTR-3′. Las secuencias con acción en cis como el elemento de respuesta al hierro en los mRNA transferrina y ferritina son importantes para el control del transporte y almacenaje de hierro. Poliproteínas A un solo producto de la traducción que se divide para generar dos o más proteínas separadas se le conoce como poliproteína. Muchos virus las producen. Orientación a un destino en proteínas Las secuencias de péptidos terminales cortas o internas en proteínas determinan la ubicación subcelular, como núcleo, mitocondrias, etc. La secuencia de señal de las proteínas secretadas causa que el ribosoma traductor se una a factores que le permitan su anclaje a una membrana y la transferencia de la proteína a través de ésta conforme se le sintetiza. Una peptidasa de señal suele retirar después la secuencia de señal. Plegado y modificación de proteínas Las chaperonas auxilian a las proteínas recién sintetizadas a plegarse para evitar el plegado erróneo y la agregación. Las alteraciones más comunes de los polipéptidos recién creados son la división y modificación química. Ocurre la división para retirar péptidos de señal, liberar fragmentos maduros de las poliproteínas, retirar péptidos internos y recortar las N- y C-terminales. Todas las cadenas laterales de aminoácidos, excepto seis, pueden experimentar cualquiera de las cuantiosas modificaciones químicas posteriores a la traducción. Degradación de proteínas Las proteínas dañadas, modificadas o con inestabilidad inherente son marcadas para degradación mediante la anexión covalente de múltiples moléculas de ubiquitina. Luego, el complejo proteasa 26S, el proteasoma, degrada a la proteína ubiquitinilada. Los defectos en la degradación de proteínas son responsables de varias enfermedades humanas. Temas relacionados (H4) Genes de RNA Pol II: promotores y potenciadores (J3) Procesamiento del mRNA, hnRNP y snRNP Control de la traducción en bacterias Debido a las diferencias del mRNA entre bacterias y eucariotas (p. ej., policistrónico contra monocistrónico, sección G1) y la ausencia de membrana nuclear en las primeras, la traducción se controla en formas diferentes. En su aspecto más simple, la traducción del mRNA bacteriano policistrónico comprende el movimiento del ribosoma de la terminación 5′ a la 3′. La subunidad ribosómica 30S permanece anexada al mRNA hasta el final, con ensamblado total del ribosoma, inicio y terminación de cada proteína codificada de manera secuencial, a medida que se encuentra cada RBS, codón de inicio y de detención. Sin embargo, es posible que (J4) Procesamiento alterno del mRNA (L2) Mecanismo de la síntesis de proteínas (L3) Inicio en eucariotos proteínas reguladoras de fijación a RNA, que están unidas cerca, obstruyan a los RBS, mientras que estructuras secundarias como bucles de tronco pueden oscurecer de manera temporal al RBS de un cistrón descendente; en el último caso, esta estructura inhibitoria se daña cuando un ribosoma traduce el cistrón ascendente, lo que permite la unión interna de una subunidad 30S. La formación de una estructura de bucle de tronco puede inhibir la acción de enzimas cuya función es degradar el mRNA, sobre todo en la terminación 5′, donde inhibe la acción de RppH, la enzima que inicia la degradación (sección J3). Las bacterias también emplean pequeñas moléculas de RNA reguladoras con se159 160 SECCIÓN L – SÍNTESIS DE PROTEÍNAS cuencias parciales complementarias de regiones de mRNA específicas, como el RBS. La estructura dúplex resultante inhibe la traducción. Mecanismos como éste permiten la elaboración de cantidades diferentes de productos de la traducción a partir de los cistrones de un mRNA policistrónico. Varios operones que codifican proteínas ribosómicas de E. coli muestran una forma interesante de control de la traducción donde una región del mRNA tiene estructura terciaria que se parece al sitio de unión de una proteína ribosómica codificada por el mRNA. Si hay rRNA insuficiente disponible para que se una el producto de la traducción, se une a su propio mRNA y evita la traducción adicional. Control de la traducción en eucariotas Puede lograrse el control global de la traducción en eucariotas mediante fosforilación de factores clave en el inicio de la traducción. Por ejemplo, la represión global de la traducción eucariótica se presenta cuando se fosforila la subunidad α de eIF2 (sección L3). También se presenta debido a la unión de una proteína eIF4EBP1 (un miembro del grupo 4E-BP de represores de la traducción) a eIF4E, lo que evita el ensamblado del ribosoma activo (sección L3). Los factores de crecimiento, las hormonas y los estimuladores de la división celular pueden activar una cinasa (mTor) que fosforila eIF4EBP1, causando disociación de eIF4E, que activa la traducción. Aunque los eucariotas suelen controlar la cantidad de proteínas específicas al modificar el nivel de transcripción del gen (sección H4), al procesar el RNA (secciones J3 y J4), o ambas acciones, el control específico de la traducción ocurre también en el citoplasma. La presencia de varias copias de elementos con abundancia de AU como 5′-AUUUA-3′, por lo general en la región no codificante 3′, marca al mRNA para degradación rápida, a menudo mediante el exosoma (sección J3) y así se limita la traducción. Otra forma de control de la traducción se relaciona con las proteínas del grupo 4E-BP que se unen de manera indirecta al mRNA específico y evitan la traducción. A este RNA se le denomina “mRNA enmascarado”. En circunstancias apropiadas, es posible traducir el mRNA cuando la proteína se disocia. La regulación de la traducción individual o por grupos de RNA incluye pequeños ncNRA con acción en trans como microRNA (miRNA), RNA corto de interferencia (siRNA) y RNA asociados con piwi (piRNA). Estos RNA son responsables, por lo menos en parte, del fenómeno de silenciamiento del RNA, la disminución regulada de la expresión genética mediante RNA exógeno de doble hebra (dsRNA) observado por primera vez en plantas. miRNA y siRNA comparten muchas propiedades y funciones, y la diferencia entre ellos no queda clara por completo. Los piwi son proteínas reguladoras de fijación a ácido nucleico y representan una clase especial de miRNA encontrados en las células germinativas. Puede haber cuando menos 1 000 genes de miRNA en el genoma humano; algunos de ellos se encuentran en los intrones de genes codificadores de proteínas que regulan, mientras que otros se encuentran entre genes estructurales. Algunos se han conservado por evolución y tienen un desarrollo regulado. A la mayor parte de los miRNA se les sintetiza mediante RNA Pol II como precursores más grandes, sujetos a procesamiento como inclusión de capuchones, poliadenilación y división (sección J2). Estos miRNA primarios (primiRNA) tienen estructuras secundarias extensas y algunas pueden aportar varias especies de miRNA maduros, que son cortos (21 a 23 nt) y de hebra doble. Estos RNA inhiben la traducción al unirse a una región complementaria en el mRNA de destino, por lo general en el 3′-UTR, un proceso conocido como interferencia del RNA (RNAi). En plantas, la coincidencia es casi perfecta, mientras en animales sólo es parcial, lo que significa que el miRNA de un animal puede tener como destino a varios mRNA. El miRNA es parte de un complejo de RNP denominado RISC, que incluye la proteína de procesamiento Dicer (sección J2) y proteína argonauta que promueve que se reprima la traducción al inhibir o escindir de manera directa al mRNA o al hacerlo blanco de degradación aumentada (sección J3). Se calcula que los miRNA regulan la expresión de más de la mitad de los genes en el genoma humano, y se ha correlacionado la sobreexpresión de algunos de ellos con ciertos tipos de cáncer. Es probable que el RNAi haya evolucionado para proteger a las células eucarióticas de los virus de RNA (sección M4) pero ahora se ha adaptado para cumplir un papel más amplio en el control de la expresión genética. En la actualidad se les explota en experimentos para descubrir las funciones de genes específicos mediante RNAi inactivado sintético (sección S2). Un ejemplo interesante del control de la traducción en mamíferos que se relaciona con las secuencias con acción en cis no codificadoras dentro del mRNA es la regulación del contenido celular de hierro. Este elemento resulta esencial para la actividad de algunas proteínas, pero es dañino en exceso. Se le transporta al interior de las células mediante el receptor de transferrina y se le almacena unido a la ferritina. Los mRNA para cada una de estas proteínas contienen una secuencia no codificante llamada elemento de respuesta al hierro (IRE) que puede formar estructuras de bucle en tronco a las que puede unirse una proteína detectora de hierro (ISP). Sin embargo, la posición de IRE y la acción de la ISP son muy diferentes en cada mRNA. La IRE en el mRNA del receptor de la transferrina es 3′-UTR y cuando se une ISP, lo que sucede cuando el hierro es escaso, estabiliza al mRNA, aumentando la traducción del receptor para tratar de mejorar la captación de hierro. Sin embargo, cuando éste es abundante la ISP se disocia de 3′-IRE y desenmascara las secuencias desestabilizadoras, para que las nucleasas puedan atacarlos, lo que produce la degradación del mRNA, reduce la traducción y disminuye la captación de hierro. Por el contrario, bajo estas mismas condiciones de abundancia de hierro, aumenta la traducción del mRNA L4 – TRADUCCIÓN, SU CONTROL Y EVENTOS POSTERIORES ferritina. Esto ocurre porque a niveles bajos de hierro, el IRE localizado en el 5′-UTR de la mRNA ferritina se une con ISP, reduciendo la capacidad del ribosoma para traducirla. Cuando se eleva la concentración de hierro, la ISP se disocia de 5′-IRE y procede la traducción, aumentando el nivel de ferritina necesaria para el almacenamiento. Este sistema de control de la traducción regula con rapidez y de manera sensible la concentración intracelular de hierro. Existen muchos otros ejemplos de control de la traducción que dependen de la unión de proteínas en la cercanía de secuencias desestabilizadoras, lo que lleva a la supresión de su efecto. Poliproteínas A transcriptos de bacteriófagos y víricos (sección M2) y muchos mRNA de hormonas en eucariotas (p. ej., la proopiomelanocortina) se les traduce para producir una sola cadena de polipéptidos; más adelante, proteasas específicas la dividen para producir varias proteínas maduras a partir de un solo producto de traducción. Al polipéptido progenitor se le denomina poliproteína. Orientación a un destino en proteínas Secuencias específicas de aminoácidos más o menos cortas dentro de las proteínas suelen determinar la ubicación final de éstas en la célula. También determinan si se secretan, se importan en el núcleo o se dirigen a otros organelos. La secreción de proteínas por procariotas y eucariotas re- quiere una secuencia de señal en la proteína naciente, además de proteínas exportadoras específicas. En bacterias, intervienen varios sistemas de secreción; los patógenos utilizan algunos de ellos para secretar toxinas dentro del organismo infectado. En el caso de las proteínas secretadas por eucariotas, una partícula de RNP, la partícula de reconocimiento de señales (SRP), que contiene RNA 7SL, es la que reconoce la señal (figura L4-1). Cuando un ribosoma citosólico empieza a traducir un mRNA que codifica una proteína destinada a secreción, SRP se une al ribosoma y al polipéptido que emerge y detiene la traducción al evitar la unión de factores de elongación. SRP reconoce a los ribosomas con una cadena de proteínas naciente que contienen una secuencia de señal (péptido de señal) compuesto por 13 a 36 aminoácidos y que contiene por lo menos un residuo con carga positiva seguido por un centro hidrofóbico de 7 a 13 residuos seguido por un residuo pequeño y neutral, a menudo Ala. Luego, la SRP (anexada al ribosoma detenido) se une al receptor de SRP (proteína de anclaje) en el lado citosólico del retículo endoplásmico (ER) y luego se le libera para reciclaje cuando el ribosoma se anexe a proteínas receptoras de ribosomas del complejo de translocación (translocón) en el ER. El ribosoma puede seguir con la traducción y se empuja a la cadena naciente de polipéptidos a través de la luz (interior) del ER. A medida que lo atraviesa, la peptidasa de señal retira al péptido de señal. Cuando se libera la proteína en el ER, por lo general se le modifica, a menudo mediante glucosila- SRP Receptor de SRP Complejo de receptor ribosómico-translocador de proteínas Citosol Ciclo de la SRP Membrana del RER Luz del RER 161 Peptidasa Péptido de señal de señal Figura L4-1. Secreción de proteínas en eucariotas. 162 SECCIÓN L – SÍNTESIS DE PROTEÍNAS ción; al parecer, patrones diferentes de glucosilación controlan el destino final de la proteína. La formación correcta de enlaces de disulfuro (sección A3), que se encuentran con más frecuencia en proteínas secretadas, recibe ayuda de la proteína isomerasa disulfuro en el ER. Otras secuencias peptídicas son responsables por la localización intracelular de proteínas. Secuencias de N- o Cterminales pueden causar la importación de las proteínas a mitocondrias, cloroplastos y peroxisomas, mientras que la secuencia interna –Lys-Lys-Lys-Arg-Lys (o cualquier combinación de cinco aminoácidos consecutivos) puede actuar como señal de localización nuclear, causando que la proteína que la contiene (p. ej., histona) sea importada en el núcleo. Plegado y modificación de las proteínas Un polipéptido recién traducido no siempre genera de inmediato una proteína funcional (sección A3). Se necesita el plegado en la estructura terciaria correcta y esto empieza antes de completarse la síntesis. Aunque esta estructura es inherente a la secuencia primaria in vitro y muchos polipéptidos se pliegan correctamente de manera espontánea, la naturaleza de traducción colaborativa del plegado y la concentración elevada de proteínas en la célula requiere que las proteínas chaperonas auxilien en el proceso de plegado, sobre todo para evitar los errores debidos a agregación de secuencias hidrofóbicas de exposición reciente durante la traducción. Muchas chaperonas son también HSP (p. ej., Hsp70, GroEL/GroES), porque evitan y revierten la agregación causada por desnaturalización proteínica a temperaturas elevadas. Aparte del plegado correcto y la posible formación de enlaces disulfuro, es posible que se necesiten otras alteraciones para que desarrollen la actividad a la que están orientadas. Estas modificaciones posteriores a la traducción incluyen división y covalencia. La primera es muy común, en especial el recorte por la acción de amino y carboxipeptidasas, pero también se presenta la remoción de péptidos internos. En la mayoría de los casos, los enlaces disulfuro mantienen unidas las secuencias que flanquean al péptido escindido, como en el caso de la insulina; sin embargo, se conocen casos donde estas secuencias se combinan mediante corte y empalme de proteínas en un solo polipéptido, de manera análoga al corte y empalme con el premRNA. En estos casos, al fragmento escindido se le denomina inteína y, a las secciones unidas y con información, exteínas. A las secuencias de señal también suele separárseles de las proteínas secretadas, y cuando las proteínas conforman una poliproteína, es necesario dividir este precursor para liberar las proteínas componentes. Por ejemplo, a la pequeña ubiquitina de 8.5 kDa se le elabora como una poliproteína que contiene varias copias unidas de una terminación a otra, y es necesario procesarlas para generar las moléculas individuales. Las modificaciones químicas son muchas y variadas y tienen lugar en las N- y C-terminales, y en casi todas las cadenas laterales de 20 aminoácidos, con la excepción de Ala, Gly, Ile, Leu, Met y Val. Entre las modificaciones se incluyen acetilación, hidroxilación, fosforilación, metilación, glucosilación y aun la adición de nucleótidos y cadenas proteicas pequeñas como la ubiquitina (consúltese más adelante) y las SUMO y NEDD8 relacionadas. Es común la hidroxilación de Pro en el colágeno y suelen acetilarse algunas de las proteínas histona (secciones C2 y C3). Estas modificaciones tienen cuantiosas funciones en la regulación y señalización celular, mediando a menudo en interacciones proteína-proteína. La fosforilación reversible controla la actividad de muchas enzimas, como la glucógeno fosforilasa y algunos factores de transcripción. Degradación de proteínas Las proteínas tienen vidas medias (t1/2) muy diferentes. Estructurales como el colágeno tienen t1/2 medida en semanas o meses mientras que las reguladoras tienden a recambiarse con rapidez (t1/2 = minutos u horas). Las células deben tener la capacidad de degradar y disponer de proteínas defectuosas y dañadas. En eucariotas, el residuo N-terminal tiene un papel muy importante en la estabilidad inherente. Ocho aminoácidos con N-terminales (Ala, Cys, Gly, Met, Pro, Ser, Thr, Val) se correlacionan con estabilidad (t1/2 > 20 h), ocho (Arg, His, Ile, Leu, Lys, Phe, Trp, Tir) con t1/2 corta (2 a 30 min) y cuatro (Asn, Asp, Gln, Glu) se desestabilizan después de modificaciones químicas. Una proteína dañada, modificada o con un residuo con N-terminal desestabilizante en sentido inherente se ubiquitinila mediante la unión covalente de residuos Lys y de glicinas con C-terminal de moléculas de ubiquitina. Un complejo proteasa 26S (proteasoma) reconoce y digiere a la proteína ubiquitinilada en una reacción que requiere ATP, liberando ubiquitina intacta para reciclaje. Los aminoácidos liberados pueden reutilizarse para elaborar nuevas proteínas. Los defectos en su degradación pueden causar compuestos proteínicos insolubles que se acumulan en las células y se relacionan con la patogénesis de algunos cánceres y trastornos neurodegenerativos, como las enfermedades de Alzheimer, Parkinson y Huntington (sección A3).