En procariontes En eucariontes
Anuncio

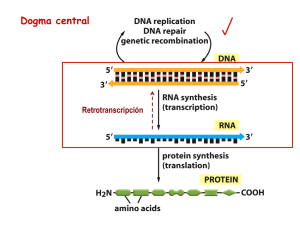
Tema V. Transcripción en eucariontes Procariontes Eucariontes 1. Todas las especies de RNA son sintetizadas por la misma especie de RNA polimerasa. 1. Hay 3 diferentes RNA polimerasas responsables de la transcripción de diferentes moléculas de RNA 2. El mRNA se traduce durante la transcripción. 2. El mRNA es procesado antes de ser transportado a citoplasma (adición de CAP, cola de poliA, remoción de intrones 3. Los genes son segmentos contiguos de DNA alineados ininterrumpidamente con el RNA traducido a proteína. 3. Los genes frecuentemente se interrumpen por intrones. 4. Los mRNAs son frecuentemente policistrónicos 4. Los mRNAs son monocistrónicos Diferentes tipos de RNA polimerasas Las tres RNA polimerasas en eucariontes: 1. RNA Polimerasa I sintetiza RNA ribosomal (rRNA) 5.8S, 18S y 28S. 2. RNA Polimerasa II sintetiza RNAs mensajeros (mRNA), RNAs pequeños nucleolares (snoRNA), micro RNAs (miRNA), RNAs pequeños interferentes (siRNA) y la mayoría de RNAs pequeños nucleares (snRNA). 3. RNA polimerasa III sintetiza RNAs de transferencia (tRNAs), rRNA 5S y algunos RNAs pequeños nucleares (snRNAs). RNA polimerasa II de levadura Arthur Kornberg, premio Nobel Medicina 1959 por el descubrimiento de la DNA polimerasa III bacteriana Transcripción por la RNA polimerasa II - 12 subunidades formando un complejo de mas de 500 kDa - múltiples factores accesorios (factores de transcripción) RNA pol II cola que se fosforila RNA pol bacteriana Promotor tipo II basal de eucariontes Caja TATA 25 pb río arriba del Inr (+1) D Factores de transcripción generales: TFIID A TBP: proteína de unión a caja TATA TAFs B TFIIA TFIIB E Ayudan a posicionar al complejo de factores sobre el promotor F TFIIF Proporciona la ocupación de +30 bp Promotor tipo II basal de eucariontes TFIIE H Une a RNA pol II al complejo montado sobre el promotor TFIIJ TFIIH Promueve el escape del promotor y fosforila el CTD de RNA pol II brindando progresividad a la transcripción Sitios consenso en promotores eucariontes La unión al sitio TATA distorsiona la cadena Proteínas activadoras de la transcripción Activadores: se unen a secuencias intensificadoras en el promotor del gen. Incrementan los niveles de transcripción Coactivadores: integran señales de activadores y/o represores a la maquinaria de transcripción con los factores de transcripción basal Represores: se unen a secuencias silenciadoras en el promotor del gen. Disminuyen los niveles de transcripción Factores de transcripción basal: Estos factores posicionan a la ARN polimerasa sobre el gen y comienzan la transcripción en respuesta a la señal de activadores y/o represores Tema V. Procesamiento del RNA mRNA: solo en eucariontes tRNA y rRNA: tanto en eucariontes como en procariontes Otros RNAs no codificantes: tanto en eucariontes como en procariontes Procesamiento del mRNA en eucariontes • Adición del CAP en el extremo 5’ • Splicing. Exclusión de intrones • Poliadenilación en el extremo 3’ 1. Adición del CAP 1) La fosfatasa remueve un fosfato del 5´ 2) Una guanil transferasa agrega GMP 3) Una metil transferasa agrega el grupo metilo 2. Reacción de corte en la unión exón-intrón Existen secuencias específicas en los límites exón-intrón R= purinas Y= pirimidinas Moléculas de RNA son las responsables del corte El apareamiento de las moléculas de snRNA requiere complementación de bases con el pre-mRNA snRNPs: U1, U2, U4, U5, U6 BBP U2AF El splicing (corte y empalme de intrones) lo realiza el snRNA de U6 snRNP: Actividad de ribozima Puede existir procesamiento alternativo de intrones en el pre-mRNA 5 mRNAs maduros 5 proteínas diferentes 3. Poliadenilación Proteínas específicas reconocen las secuencias de poliadenilación El mRNA es cortado y la enzima Poli-A polimerasa (PAP) agrega A’s Proteínas de unión a la cola de poliA se unen al extremo 3’ hasta que el mensaje sale del núcleo El alargamiento del RNA está acoplado su procesamiento La salida del mensaje del núcleo es coordinada El receptor de exporte nuclear dirige la salida RNA ribosomal Es el RNA más abundante de la célula. Tiene función tanto estructural como catalítica en la síntesis de proteínas. Existen diferentes tipos de acuerdo a su coeficiente de sedimentación asociados a las subunidades ribosomales: En procariontes Subunidad ribosómica grande 23S 5S Subunidad ribosómica pequeña 16S En eucariontes 28.5S 5.8S 5S 18S Procesamiento del rRNA en procariontes Procesamiento del rRNA en eucariontes El nucléolo es el lugar de síntesis de rRNA y pre-ribosomas Nature Reviews Molecular Cell Biology 2, 514-520 El ribosoma Alberts et al., 3rd ed., p 232 Ribosomas procariontes y eucariontes Cumbre cabeza Protuberancia central Tallo plataforma base El rRNA juega un papel muy importante en la traducción rRNA 16S rRNA 23S RNA de transferencia Es el RNA más pequeño, con unos 75 nt de longitud, en promedio. Su función es transportar los aminoácidos en forma activada hasta el ribosoma donde ocurre la síntesis de proteínas. Existe al menos un tipo de tRNA para cada uno de los 20 aminoácidos. Sin embargo, algunos aminoácidos tienen varios tRNAs. Todos los tRNAs presentan en su 3’ la secuencia CCA, independientemente del aminoácido que transporta. El extremo 3’ consitutye el sito aceptor del aminoácido. Procesamiento de tRNA en procariontes Procesamiento del extremo 5’: Rnasa P (Ribonucleoproteína compuesta por RNA 377 nt y proteína 20 kDa) Acción catalítica: RNA (ribozima) Procesamiento: co-transcripcional Procesamiento del extremo 3’: 1) endonucleasa elimina un grupo de bases 2) exonucleasa, RNAsa D elimina los restantes nucleótidos 3) El triplete CCA característico de estas moléculas es añadido por una tRNA nucleotidil transferasa Bases modificadas que se encuentran en los tRNA • Pseudouracilos (y) • 4 tiouridina • 2 metilguanina • 2 isopententenil adenina • Dihidrouridina (D) • Inosina Procesamiento del intron en los tRNA en eucariontes Una endonucleasa corta en los sitios donde hay protuberancias y una DNA ligasa sella esos cortes Los tRNA tienen una longitud de aproximadamente 80 nucleótidos Inhibidores de la transcripción eucarionte + N H Acridina Actinomicina D Sar L-Pro Sar L-meVal D-Val O L-Pro D-Val L-Thr O L-meVal O L-Thr C C N Se intercala entre bases G y C NH2 O O CH3 O CH3 Las rifampicinas son antibióticos producidos por Streptomyces mediterranei, con buena actividad contra bacterias Gram-positivas y contra Mycobacterium tuberculosis. Su mecanismo de acción estriba en la inhibición del inicio de la transcripción, uniéndose de modo no covalente a la subunidad ß de la RNA polimerasa bacteriana. Rifampicina B a-amanitina Es un octapéptido bicíclico que se obtiene del hongo Amanita phalloides. Inhibe la transcripción de la RNA polimerasa II eucarionte.