Dogma central
Anuncio
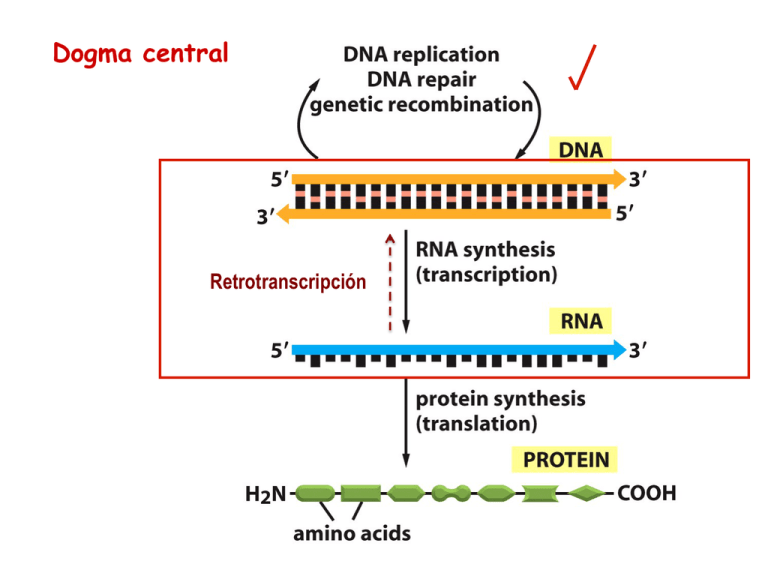
Dogma central Retrotranscripción Organización celular procarionte y eucarionte En eucariontes la transcripción se encuentra espacial y temporalmente separada de la traducción La estructura química del RNA Cadena sencilla Ribosa (2’ y 3’OH) Uracilo en lugar de timina El uracilo se aparea con la adenina DNA Transcripción 3’-TACCCTAAGTCC-5’ Procariontes RNA 5’-AUGGGAUUCAGG-3’ ¡ El RNA es una molécula multifuncional ! DNA Transcripción RNA Eucariontes mRNA rRNA tRNA snRNA ü RNA mensajero, codifica para proteína ü RNA ribosomal, componente estructural del ribosoma ü RNA de transferencia, adaptador entre mRNA y proteína ü RNAs pequeños nucleares, funcionan en corte y empalme de intrones snoRNA ü RNAs pequeños nucleolares, procesamiento y modificación scaRNA ü RNAs pequeños de cajal, modifican snoRNAs, snRNAs, de rRNA miRNAs, regulan traducción específica ü microRNAs, regulan la expresión genética miRNA siRNA Otros no codificantes ü RNAs pequeños interferentes, apagan la expresión de genes mediante degradación de mRNAs específicos y mediante modificación epigenética del DNA ü Otros RNAs no codificantes que funcionan en procesos diversos como síntesis de telómeros, inactivación del cromosoma X, transporte de proteínas, etc. El RNA es mono-catenario pero puede formar regiones de doble cadena Dúplex Región de cadena sencilla Tallo - asa Protuberancia Burbujas internas Desapareamiento, simétrica, asimétrica Juntas Tres tallos, cuatro tallos Estructura secundaria y terciaria en el RNA ribosomal Estructura secundaria y terciaria en el RNA de transferencia Estructura secundaria en el RNA mensajero Los ribonucleótidos pueden presentar diversas modificaciones necesarias para cumplir su función Estas modificaciones son comunes en el tRNA Transcripción codificante molde RNA polimerasa Solo se utiliza una de las cadenas como molde El RNA transcrito es complementario a la secuencia de la cadena de DNA que se utilizó como molde y es idéntico a la cadena CODIFICANTE excepto por la presencia de U. La transcripción involucra tres etapas: • Inicio • Elongación • Terminación INICIO (Bacteria) Promotor: secuencias que reconoce la RNA polimerasa y proteínas asociadas a ella para iniciar la transcripción Caja Pribnow: TATAAT Secuencia – 35: TTGACA Elemento UP: AAAWWTWTTTTNNNAAANNN (opcional) W: A/G N: cualquier nucleótido ¿En cuál de las dos cadenas se leen estas secuencias? 5’ 3’ En la cadena codificante La dirección de la transcripción de los genes puede variar en el cromosoma Operón: Varios genes regulados por un solo promotor Los promotores tienen orientación e indican el sentido del movimiento de la RNA polimerasa Subunidades de la RNA polimerasa bacteriana Centro catalítico Especificidad promotor - no requiere cebador - utiliza Mg2+ como cofactor - utiliza NTPs como sustrato - transcribe en sentido 5’ → 3’ Factores sigma (σ) Sigma garantiza la unión estable de la RNA pol en el promotor Enzima núcleo HOLOENZIMA Existen diferentes estados en la Iniciación de la Transcripción Complejo cerrado Factor sigma Complejo abierto (12 nt) Síntesis de 10 nt de RNA Liberación de Sigma Escape del promotor Burbuja de transcripción y movimiento ELONGACIÓN 5’ 3’ Sustratos: NTPs (ATP, UTP, GTP, CTP) Elongación de la transcripción La polimerasa mantiene unos 10-12 nt desapareados en el DNA: lugar donde ocurre la síntesis de RNA Elongación de la transcripción Múltiples moléculas de RNA polimerasa se encuentran transcribiendo el Operón; así de una molécula de DNA codificante la célula fabrica la cantidad de moléculas de RNA que requiere para su función. Terminación de la transcripción bacteriana terminadores intrínsecos, independientes de la proteína ρ (Rho); se forman en el RNA transcrito por apareamiento, pausan a la Polimerasa y esta libera el RNA recién sintetizado Terminadores dependientes de Rho; La proteína Rho (helicasa) se une al RNA en regiones ricas en C (sitios rut) y avanza hasta la burbuja de transcripción donde desenrolla el complejo DNA:RNA y libera el transcrito sitios RUT: regiones ricas en C Inhibidor de la transcripción en bacteria: Rifampicina B Las rifampicinas son antibióticos producidos por Streptomyces mediterranei, con buena actividad contra bacterias Gram-positivas y contra Mycobacterium tuberculosis. Su mecanismo de acción estriba en la inhibición del inicio de la transcripción, uniéndose de modo no covalente a la subunidad ß de la RNA polimerasa bacteriana.