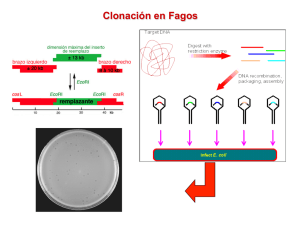
VECTORES Y CLONADO Martín Monte, viernes 27 de Marzo, 2009 Caracterización y estudio de la función de un gen (Tb podría ser un fragmento de DNA genómico para el estudio de splicing o del promotor de un gen) 1- Obtención del gen de interés mRNA RT 1ra cadena 2da cadena cDNA Vector PCR Screening Búsqueda Library Obtención de la secuencia Caracterización y estudio de la función de un gen Secuenciación Análisis de la secuencia: Identificación del fragmento secuenciado enfrentándolo a bases de datos de DNA Caracterización y estudio de la función de un gen Análisis de la secuencia: Caracterización del fragmento de DNA STOP ATG 5’UTR ORF 3’UTR cDNA Caracterización y estudio de la función de un gen Origen library Origen PCR Utilidad limitada (sonda, transc-traducc in vitro) Generalmente los cDNAs no se expresan cuando están clonados en vectores de libraries Se puede replicar en bacterias Utilidad muy limitada (sonda) No se expresan Se debe amplificar y secuenciar cada vez que se necesite más material No se puede replicar en bacterias Para continuar con el estudio del fragmento de DNA es necesario clonarlo en un vector adecuado Vectores • PLASMIDOS: construcciones de DNA derivados de plásmidos naturales. Límite clonado 0.110 Kb • BACTERIOFAGOS: Derivados del genoma de virus que infectan bacterias (fago l ) 8-25 Kb. Pueden ser empacados en fagos nuevamente. • COSMIDOS: Plásmido que contiene secuencias cos del fago l y pueden ser empacados en fagos 35-50 Kb • BACs (Bacterial Artificial Chromosomes) Derivados plásmido F de E. coli, 75-300 Kb • PAC s es como un BAC, pero deriva del plásmido-P1 . Lim. Clonado 100 Kb • YACs (Yeast artificial Chromosome): cromosoma de levaduras artificial, pretende imitar a un cromosoma, contiene Centrómero y Telómeros . Lim Clonado:100-1000 Kb plásmido BAC Fago l YAC Vectores Clonado en un vector adecuado al estudio que se pretende realizar: • Sistema: bacterias, levaduras, plantas, células de mamífero • Objetivo: • Clonado de fragmentos de DNA para hacer una library • Expresión de cDNA completo, fragmentos, necesidad de fusiones • Análisis de la actividad de un promotor • Expresión de la proteína para ver su función • Expresión de la proteína para producirla y purificarla Vectores Son largos fragmentos de DNA construidos a partir de plásmidos, o bacteriófagos naturales con la capacidad de replicación autónoma y que permiten el clonado de fragmentos de DNA Características: • Poseen secuencias que permiten su propagación autónoma (ORI) • Poseen una región para insertar el DNA a clonar (Multiple Cloning Site-MCSo polylinker) con sitios únicos para varias enzimas de restricción. • Marcadores de selección: permiten seleccionar exclusivamente las células que contiene el vector. PLASMIDOS: Molécula circular de DNA doble cadena extracromosomal que replica en forma autónoma dentro de la bacteria (1.6-300 Kb) FUNCIONES CODIFICADAS POR LOS PLASMIDOS • Resistencia a antibióticos • Degradación de compuestos orgánicos (Pseudomonas) • Producción de toxinas (ej E.coli, enterotoxinas) • Producción de bacteriocinas (colicinas, subtilicinas) • Inducción de tumores (plantas) • Sistemas de Restricción-modificación • Producción de antibióticos (ej Streptomyces) • Resistencia a metales pesados R6 pSC101 Stanley Cohen Tn3 Tetr Ampr Ori pMB9 Mary Betlach ColE1 Construido por Ray Rodriguez and Herbert Boyer PLASMIDOS: Sitio para clonado Gen de resistencia Origen de replicación PLASMIDOS: marcadores de selección Permiten la selección de aquellas células que poseen el vector : • Marcadores de resistencia a antibióticos Ej: Ampicilina , Kanamicina, Tetraciclina , Zeocina, Neomicina, Puromicina • Marcadores de auxotrofía (permiten síntesis de un componente esencial para el crecimiento celular) Ej: aminoácidos PLASMIDOS: marcadores de selección Ampicilina: agente beta-lactámico que interfiere con la síntesis de pared bacteriana Resistencia: Ampr (gen bla), codifica para una enzima con actividad b-lactamasa Tetraciclina: Actúan inhibiendo la síntesis proteica al unirse a la subunidad 30 S del ribosoma bacteriano e impide la unión del tRNA a este y el transporte de aminoácidos hasta la subunidad 50 S Resistencia: Tetr (gen tet) codifica para una proteína que se une a membrana bacteriana y impide transporte del antibiotico Kanamicina: Es un aminoglucósido que se une a ribosomas 30S , produce misreading del mRNA y codones stop. Resistencia: Kanr (gen kan) aminoglicosido fosfotransferasa modifica el antibiótico. PLASMIDOS: Orígenes de replicación • El origen de replicación del plásmido es la región encargada de su propagación • Puede haber más de un origen de replicación con diferente función en un plásmido Ejemplos: ColE1 Ori: bacterias f1 Ori: fago filamentoso 2uM Ori: levaduras SV40 Ori: células • El tipo de origen para bacterias (ECol1, pMB1, pSC101) está relacionado con la cantidad de copias de plásmido que se generarán en la bacteria, y divide a los plásmidos en alto (30-300) o bajo (1-30) número de copias. • Existen muchos orígenes de replicación para bacterias, muchos de ellos modificados en sus sistemas de control para lograr los de alto número de copias. • Vectores que utilizan el mismo Ori no pueden replicarse en la misma bacteria debido a que los reguladores que actúan en trans pueden regular ambos plásmidos. Esto se denomina Grupo de Incompatibilidad . Todos los plásmidos deben tener un origen de replicación en bacterias ya que su amplificación y purificación en bacterias es un paso obligado para su utilización Origen ColE1 o pMB1 -555 -265 -20 ori REPLICACION Corte por RNAasa H RNAII primer ori RNAII primer Corte por RNAasa H Rop NO REPLICACION RNA I Origen de Replicación Grupo de compatibilidad Número de copias Otras Características pMB1 pBR 322 y sus derivados (i.e, pET vectors) 15-20 pMB1 son similares a ColE1 pMB1 mutado Vectores pUC 500-700 mutación en el gen rop p15A pSC101 ColE1 pACYC y sus derivados pSC101 y sus derivados ColE1 10-12 5 aprox 15-20 f1 Se sintetiza DNA simple cadena SV40 Para replicación en eucariotas superiores Caracterización y estudio de la función de un gen Origen library Origen PCR Utilidad limitada (sonda, transc-traducc in vitro) Generalmente los cDNAs no se expresan cuando están clonados en vectores de libraries Se puede replicar en bacterias Utilidad muy limitada (sonda) No se expresan Se debe amplificar y secuenciar cada vez que se necesite más material No se puede replicar en bacterias Para continuar con el estudio del fragmento de DNA es necesario clonarlo en un vector adecuado Clonado de fragmentos de DNA en vectores Fragmento/inserto Vector MCS Principales Protocolos: 1) Corte de V e F con ER y Ligado 2) Recombinación entre V e F Fragmento clonado en vector Clonado de fragmentos de DNA en vectores DIGESTIÓN CON ENZIMAS DE RESTRICCIÓN SEGUIDA DE LIGACIÓN Pasos básicos: • Digestión por separado del vector y el fragmento con una o dos enzimas de restricción compatibles mediante una reacción in vitro • Reacción de ligación incubando el vector y el fragmento digeridos con una enzima con actividad DNA ligasa. • Transformación de bacterias competentes con el producto de la reacción de ligación utilizando antibióticos para seleccionar las colonias que hayan incorporado el plásmido • Screening de los plásmidos que hayan incorporado el inserto • Secuenciación para chequear el DNA recombinante (Vector + inserto) Clonado de fragmentos de DNA en vectores DIGESTIÓN CON ENZIMAS DE RESTRICCIÓN SEGUIDA DE LIGACIÓN 1) Elección del vector según el organismo y el propósito del clonado 2) Elección de la enzimas de restricción: considerar: 1- que las enzimas estén en el polylinker y en la orientación correcta 2- que no haya sitios internos en el fragmento 3- si fuera una fusión con un tag o proteína, controlar el marco de lectura Clonado de fragmentos de DNA en vectores DIGESTIÓN CON ENZIMAS DE RESTRICCIÓN SEGUIDA DE LIGACIÓN Utilización de un programa que indique las enzimas que cortan nuestro fragmento y las que no Clonado de fragmentos de DNA en vectores DIGESTIÓN CON ENZIMAS DE RESTRICCIÓN SEGUIDA DE LIGACIÓN Utilización de un programa que indique las enzimas que cortan nuestro fragmento y las que no BamHI MCS EcoRI MCS Clonado de fragmentos de DNA en vectores DIGESTIÓN CON ENZIMAS DE RESTRICCIÓN SEGUIDA DE LIGACIÓN 2) Elección de la enzimas de restricción: • Si no hay posibilidad de clonar en forma direccionada (dos enzimas diferentes que orientan el fragmento en una única dirección), también se puede recurrir : - Clonar con una sola enzima de restricción y luego controlar en que orientación entró el fragmento 200bp HindIII EcoRI HindIII EcoRI MCS MCS 600bp HindIII EcoRI MCS Clonado EcoRI Screening HindIII HindIII EcoRI MCS vector Ins 600bp Ins 200bp Clonado de fragmentos de DNA en vectores DIGESTIÓN CON ENZIMAS DE RESTRICCIÓN SEGUIDA DE LIGACIÓN 2) Elección de la enzimas de restricción: Si no hay posibilidad de clonar con las enzimas presentes en el MCS -polylinker también se puede recurrir a : - Utilizar enzimas diferentes pero que dejen el mismo extremo cohesivo XbaI NheI G CGATC XbaI EcoRI P HindIII inserto EcoRI NheI CTAGA T GC T A G A C G A T CT P Sitio híbrido NheI/XbaI No lo digiere ninguna enzima XbaI MCS HindIII EcoRI NheI/XbaI inserto MCS Clonado de fragmentos de DNA en vectores DIGESTIÓN CON ENZIMAS DE RESTRICCIÓN SEGUIDA DE LIGACIÓN 2) Elección de la enzimas de restricción: Si no hay posibilidad de clonar con las enzimas presentes en el MCS -polylinker también se puede recurrir a : - Formar extremos romos a partir de cohesivos EcoRI BamHI MCS 1 EcoRI P HindIII BamHI inserto EcoRV EcoRI DNA T4 Pol P P XbaI HindIII BamHI inserto EcoRV P EcoRV MCS 2 La digestión con EcoRV forma naturalmente extremos blunt XbaI MCS 2 Clonado de fragmentos de DNA en vectores DIGESTIÓN CON ENZIMAS DE RESTRICCIÓN SEGUIDA DE LIGACIÓN Generación de extremos romos a partir de extremos cohesivos utilizando la T4DNA polimerasa Clonado de fragmentos de DNA en vectores DIGESTIÓN CON ENZIMAS DE RESTRICCIÓN SEGUIDA DE LIGACIÓN Reacción de Ligación: El 5’-P es necesario para la ligación Clonado de fragmentos de DNA en vectores DIGESTIÓN CON ENZIMAS DE RESTRICCIÓN SEGUIDA DE LIGACIÓN Reacción de Ligación: para evitar que el vector se re-ligue cuando se corta con una sola enzima de restricción hay que defosforilar los extremos 5’ libres 1) Digestión con una enzima de restricción (EcoRI) EcoRI EcoRI MCS P P EcoRI EcoRI MCS MCS 2) Tratamiento del vector con una fosfatasa (fosfatasa alcalina) P inserto EcoRI MCS MCS P EcoRI inserto MCS Clonado de productos de PCR en vectores Productos de PCR Vectores Amplificación con primers sin sitios ER Taq 5’ 3’ OH-A T4 DNA Pol Pfu 5’ 3’ OH- Clonado T A-OH 3’ 5’ T T Taq + dATP -OH 3’ 5’ PNK + dATP 5’ P 3’ OH- -OH 3’ P 5’ Clonado blunt Vector de-P Amplificación con primers con sitios ER 5’ 3’ OH- -OH 3’ 5’ Digestión con ERs y clonado sticky Clonado de fragmentos de DNA en vectores REACCIÓN BASADA EN EVENTOS DE RECOMBINACIÓN Sequence and Ligase Independent Cloning Clonado de fragmentos de DNA en vectores TRANSFORMACIÓN y SELECCIÓN DE BACTERIAS TRANSFORMACIÓN (Bacterias competentes) Métodos químicos: Cl2Ca, DMSO, etc Eficiencia:107 UFC/ug DNA Métodos físicos: Electroporación (pulsos electricos ) Ef: 108 UFC/ug DNA SELECCIÓN Crecimiento en presencia de antibiótico Plato LB-Agar + Amp Clonado de fragmentos de DNA en vectores BÚSQUEDA DE VECTORES CON INSERTO (RECOMBINANTES) A Mapeo de restricción utilizando una enzima que corte una sola vez y permita ver la diferencia entre vector vacío y vector con inserto B Vector + ins Vector Mapeo de restricción utilizando un doble corte que permita ver la liberación del fragmento vector Ins 600bp Algunos vectores poseen la secuencia del gen beta-gal con el MCS en su interior. Si el inserto está presente, beta-gal no se expresa y el test para actividad beta-gal da negativo (blanco). Si el vector se religó beta-gal está intacto y el test da positivo (colonias azules) Clonado de fragmentos de DNA en vectores BÚSQUEDA DE VECTORES CON INSERTO (RECOMBINANTES) C Reacción PCR sobre el DNA de las colonias obtenidas en el plato de selección utilizando primers específicos para el inserto o primers que hibridicen sobre el MCS PCR D Réplica del plato de selección sobre membrana de nylon y búsqueda de las colonias con inserto utilizando una sonda marcada específica. Se revela por autoradiografía y se identifican las colonias positivas Clonado de fragmentos de DNA en vectores 1. Elección y preparación de vector e inserto 2. Reacción de ligación o recombinación 3. Transformación de bacterias competentes en un medio que permita seleccionar las colonias con vector (Antibióticos) 4. Screening: en la etapa de analizar el DNA es necesario hacer crecer algunas las colonias por separado en medio líquido con antibiótico y purificar plásmido a partir de una alícuota del cultivo. Analizar el DNA purificado (restricción, secuenciación, etc) y una vez elegido el DNA correcto, hay que volver al cultivo original para hacer stock de bacterias y amplificar el vector para su uso.
Anuncio
Descargar
Anuncio
Añadir este documento a la recogida (s)
Puede agregar este documento a su colección de estudio (s)
Iniciar sesión Disponible sólo para usuarios autorizadosAñadir a este documento guardado
Puede agregar este documento a su lista guardada
Iniciar sesión Disponible sólo para usuarios autorizados

![1 Si v = [ 36 -12 ] , w = [ 9 -3 ] , y S = 1wl. Indique cuáles opciones](http://s2.studylib.es/store/data/004950498_1-7f0b4c506a631255d680bd90dfa0a3e5-300x300.png)
