atp sintasa - WordPress.com
Anuncio

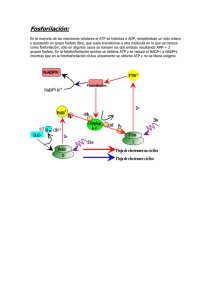
UNIVERSIDAD AUTÓNOMA METROPOLITANA UNIDAD CUAJIMALPA UEA: FISIOLOGIA MOLECULAR ATP SINTASA Académico: Manuel Gutiérrez Alumnas: B. Ximena Olivares Guadarrama Liliana Moctezuma Cruz Joselyn Alfaro Tania G. Rojas Pérez Citlali C. Madrid Díaz • Introducción • Estructura y ubicación • Mecanismo de acoplamiento reversible • Deficiencia e inhibición de ATP sintasa • Ejemplos • La ATP sintasa es un complejo enzimático encargado de proveer a la célula la energía necesaria para realizar todos sus procesos vitales mediante la síntesis de ATP, aunque también puede llevar a cabo la hidrólisis de éste, por lo que al complejo también se le nombra F1F0ATPasa. ATP sintasa, también conocida como F1F0 ATPasa, cataliza la formación de ATP a partir de ADP y Pi en los procesos conocidos como la fosforilación oxidativa (impulsada por oxidaciones en las células animales y microorganismos) y la fotofosforilación (impulsada por la luz en células vegetales). • El nombre se debe a que las subunidades pueden separarse en dos dominios estructurales llamados F1 y F0. • La síntesis de ATP está acoplada a un gradiente de potencial electroquímico de protones, que es generado por los complejos enzimáticos de la cadena respiratoria durante la oxidación de sustratos. • El movimiento de protones a través de ATP sintasa es necesario para la liberación del ATP de F1. • Se propone que la energía liberada durante el movimiento de protones a través de la membrana, lo cual produce un cambio conformacional de la ATP sintasa, que da lugar a la liberación del ATP unido fuertemente a la subunidad β. Existen numerosas enzimas que hidrolizan el ATP en la célula, sin embargo, el 90% de la síntesis en condiciones aerobias lo realiza la ATP sintasa. La ATP sintasa es un complejo enzimático ampliamente distribuido en la naturaleza. Se localiza en la membrana plasmática de las bacterias, la tilacoidal de los cloroplastos y la membrana interna de las mitocondrias. La ATP sintasa es una enzima altamente conservada en la escala filogenética, 72% de identidad entre las subunidades de mayor peso molecular. A pesar de su alto grado de complejidad estructural, la ATP sintasa de mitocondrias de bovino es la mejor conocida en la actualidad. F1 El nombre F1F0 ATP sintasa se debe a que las subunidades pueden separarse en dos dominios estructurales llamados F1 y F0, definidos por su polaridad, un hidrofóbico o F0 integrado a la membrana y un segmento hidrofílico o F1 que es el dominio catalítico de la enzima. A la ATP sintasa se le representa como un motor molecular rotativo que participan en la conversión de energía, y por lo tanto consta de un rotor y de un estator. FO • El rotor está compuesto por el tallo central de la enzima y el anillo c. Estas subunidades están involucradas en el movimiento de la enzima durante la catálisis, las subnidades γ, δ, ε, giran acordes con el segundo rotor en FO. • El estator asegura la estabilidad de los rotores. Lo forman el hexamero αβ y las subunidades del tallo periférico, las cuales se mantienen estáticas durante la síntesis de ATP y a su vez mantienen fijas a las subunidades catalíticas. complejo enzimático de los eucariontes tiene una estructura muy elaborada, presenta una composición de 16 subunidades diferentes: El F1 a, b, c, α, β, γ, δ, ε, A6L, d, f, F6, e, g, OSCP y un regulador interno o proteína inhibitoria IF1 El dominio catalítico F1 globular en la enzima mitocondrial es un conjunto de subunidades con la estequiometría α3β3γ1δ1ε1 Las tres subunidades α y las tres subunidades β están dispuestas alternativamente alrededor de una α-helice central en la subunidad γ. Subunidades γ, δ y ε forman un tallo central que une el subcomplejo (αβ)3 de F1 a la membrana del dominio Fo. ε y γ se encuentran vinculados a un anillo de subunidades c en el dominio Fo Esto sugiere que la enzima funciona por un mecanismo que implica la modulación cíclica de afinidad de nucleótidos en subunidades β catalíticas. Tallo central F1 y Fo también están conectados por un segundo elemento, el tallo periférico (estator). Su función no se ha demostrado, pero puede actuar como un estator para contrarrestar la tendencia del dominio (αβ)3 para seguir la rotación del tallo central Compuesto por una sola copia de las subunidades: OSCP, b, d, F6 y A6L ancladas a la subunidad a que se encuentra en el interior de la membrana. Se observa que la subunidad b es la principal formadora del tallo periférico. Algunos dominios de b también forman parte de FO La proteína OSCP (proteína que confiere sensibilidad al inhibidor oligomicina) y se encuentra en la parte más alta de la ATPasa mitocondrial. interactúa con las regiones Nterminales de las subunidades α, que sobresalen de la "corona" en la parte superior de F1 Las subunidades membranales de la F1F0- ATPasa ( dominio FO) son principalmente a, b y c (hidrofóbicas). La subunidad c es un oligómero en forma de cilindro hueco al que se le adosa la subunidad a y se ancla al rotor (γ, δ, ε) y al eje lateral de la subunidad b. a y c transportan los protones formando el “canal de protones” de la F1F0. La transferencia de protones causa el movimiento de las subunidades ε y γ El número de subunidades c varía entre 10 y 14, que están dispuestos en un anillo, con un grupo carboxilo conservado cerca de la mitad de una de las dos hélices hidrófobas que atraviesan la membrana. e, d, f, g y A6L son proteínas pequeñas (60 a 110 aa). La función de estas proteínas, denominadas supernumerarias, no es del todo clara pero se sabe que son necesarias para el ensamble del dominio F0 Existe un regulador endógeno de la ATP sintasa, evolutivamente exclusivo de eucariotes: proteína inhibitoria (IF1) Formada por 84 aa que previene la hidrólisis de ATP en condiciones de bajo potencial electroquímico. Se une a la F1 por su extremo amino en una interfase αβ que contiene ADP. Su extremo carboxilo sobresale del complejo y es capaz de interaccionar con el extremo carboxilo de otro complejo F1I y formar un dímero de F1I a través de una hélice enrollada del rotor central o de la subunidad b. Esta estructura es elástica y puede doblarse y comprimirse, por lo tanto puede realizar cambios conformacionales. El sitio responsable de la inhibición lo forman 6 aminoácidos, Phe17, Arg20, Glu21, Arg22, Glu25 y Phe28; esta región se une a la subunidad β formando el complejo F1-IF1 La estructura de la ATP sintasa de los mamíferos es más compleja que la ATP sintasa bacteriana. Se conservan las subunidades catalíticas, así como las subunidades que forman el rotor de la enzima. La mayor diferencia se encuentra en las subunidades que componen el estator de la enzima y algunas subunidades supernumerarias adicionales (como en levadura k e i) que se encargan de la dimerización de la enzima. Éste está compuesto por una subunidad b, d, y F6 y OSCP, ésta ultima es homóloga de la subunidad δ en bacteria. a) ATP sintasa mitocondrial, b) ATP sintasa de bacteria Motor molecular (ATP) PMF Energía mecánica = cambio de conformación Energía química de enlace (ATP) 100 ATP (3 protones) Síntesis de ATP Hidrólisis de ATP Deficiencia de la ATP sintasa. • Los trastornos de la fosforilación oxidativa mitocondrial (OXPHOS) Diversas enfermedades (tejidos que exigen energía, el sistema nervioso, el músculo esquelético y el corazón) • Los defectos genéticos generan mutaciones en el DNA mitocondrial (mtDNA) como en los genes nucleares. • Mutaciones en el DNA nuclear se involucran con más frecuencia en enfermedades pediátricas de deficiencia mitocondrial que las mutaciones del mtDNA. • Trastornos mendelianos del OXPHOS pueden afectar a todos los complejos de la cadena respiratoria, sobre todo del complejo I y IV. • Trastornos de la ATP sintasa se han reportado con menos frecuencia. Inhibidores de la ATP sintasa. Oligomicina. Inhibe la síntesis de ATP, interfiere en el transporte de protones. * Se fija en el tallo de la ATP sintasa, inhibiendo el canal de protones, e impidiendole la reentrada de protones en la matriz mitocondrial. * Como los gradientes de pH y eléctricos no se pueden disipar en presencia de la oligomicina, el transportador de protones se detiene por la dificultad para el bombeo de más protones contra los gradientes. * Causa una acumulación de protones en el espacio intermembrana. Referencias • Araceli Cano-Estrada y Diego González-Halphen (2011) F1F0-ATP SINTASA Y SUS DIFERENCIAS ESTRUCTURALES. UNAM. • Itoh H, Takahashi A, Adachi K, Noji H, Yasuda R, Yoshida M, Kinosita K (2004) Mechanically driven ATP synthesis by F1-ATPase. Nature. • Becker WM, Kleinsmith LJ, Hardin J. El mundo de la célula.6a Ed. España: Pearson educació;2007. • Alberts B, Bray D, Hopking K, Jonhson A, Lewis J, Raff M, Roberts K, Walter P.Introducción a la biología celular.3a Ed. México: Médica Panamericana;2011. Daniela Stock, et. al. The rotary mechanism of ATP synthase. Elsevier. Current Opinion in Structural Biology 2000, 10:672–679. Donald Voet, Judith G. Voet, Charlotte W. Pratt. Fundamentos de Bioquímica. La vida a nivel molecular. 2ª Edición. David L. Nelson, Michael M. Cox Lehninger. Principios de Bioquímica. 4ª Edición. Domínguez-Ramírez, Marietta Tuena de Gómez-Puyou. La F1F0 ATP sintasa: un complejo proteico con gran versatilidad estructural y functional. Revista Especializada en Ciencias Químico-Biológicas, vol. 8, núm. 1, junio, 2005, pp. 18-27, Universidad Nacional Autónoma de México. México. • • • •