Práctica 2 MODELIZACIÓN MOLECULAR Y DISEÑO DE FÁRMACOS
Anuncio
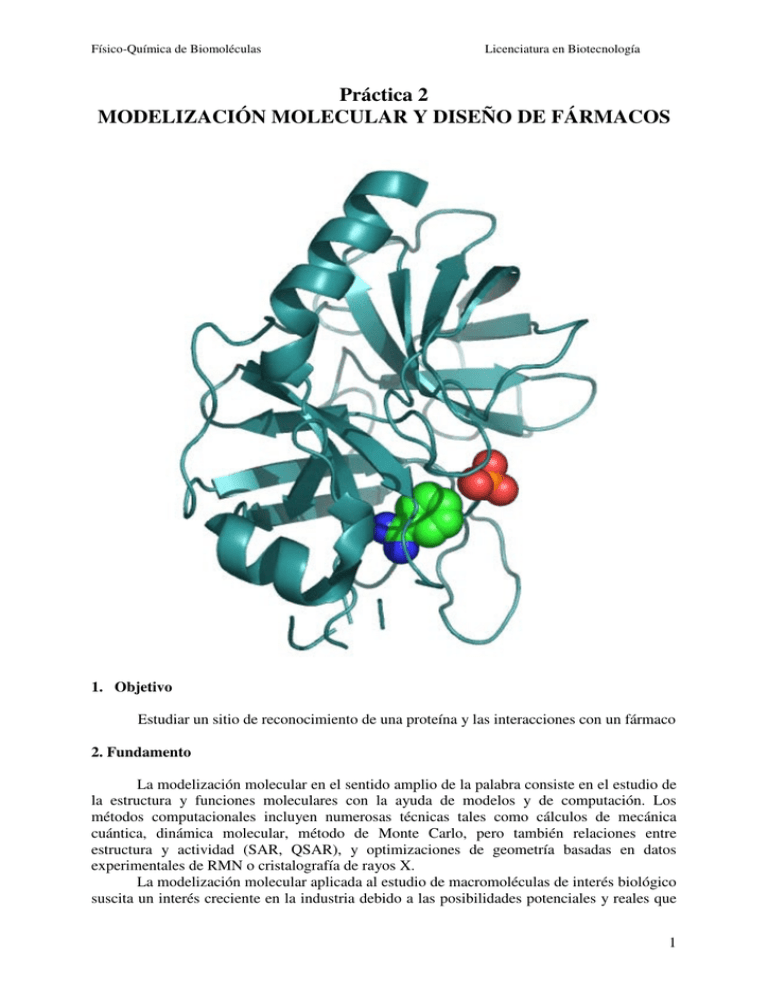
Físico-Química de Biomoléculas Licenciatura en Biotecnología Práctica 2 MODELIZACIÓN MOLECULAR Y DISEÑO DE FÁRMACOS 1. Objetivo Estudiar un sitio de reconocimiento de una proteína y las interacciones con un fármaco 2. Fundamento La modelización molecular en el sentido amplio de la palabra consiste en el estudio de la estructura y funciones moleculares con la ayuda de modelos y de computación. Los métodos computacionales incluyen numerosas técnicas tales como cálculos de mecánica cuántica, dinámica molecular, método de Monte Carlo, pero también relaciones entre estructura y actividad (SAR, QSAR), y optimizaciones de geometría basadas en datos experimentales de RMN o cristalografía de rayos X. La modelización molecular aplicada al estudio de macromoléculas de interés biológico suscita un interés creciente en la industria debido a las posibilidades potenciales y reales que 1 Físico-Química de Biomoléculas Licenciatura en Biotecnología ofrece. Limitaremos la discusión a aquellas técnicas que definen un sistema virtual de cierto número de moléculas, definen una función de energía de interacción entre estas moléculas o subunidades de estas moléculas para obtener a través de la suma de todas las interacciones una energía global del sistema. Existen fundamentalmente dos técnicas: la técnica de Monte Carlo, que se usa para generar una serie de estados posibles del sistema. Define movimientos al azar de una partícula del sistema (p.ej. rotación/traslación de una molécula), calcula la función de energía, y considera el movimiento válido si la nueva energía del sistema es más baja o si un número aleatorio a generar en este momento es superior a exp(-∆E/kT). La otra técnica clásica es la dinámica molecular que genera fuerzas intermoleculares a partir de las expresiones de las energías, y realiza simulaciones deterministicas a partir de las ecuaciones clásicas de Newton ( ma = f , donde a es la aceleración y f las fuerzas). La ventaja principal de estas dos técnicas es que permiten estudiar el comportamiento del sistema (modelo) a nivel molecular y que, al poder modificar de forma muy controlada ciertos parámetros tales como las fuerzas de ciertas interacciones, permite mejorar la comprensión del sistema. Por supuesto, también permite un ahorro en reactivos. Entre los inconvenientes cabe destacar que es necesario simplificar mucho los sistemas por lo que es fácil que el modelo que se esta estudiando esté muy lejos del sistema real que pretende representar y que su comportamiento cuantitativo y cualitativo sea distinto. La búsqueda del sitio de unión que se va a llevar a cabo en esta práctica esta relacionada con estas técnicas porque se define una función de energía que se detalla más adelante en este guión y cada posición relativa de un ligando con un receptor da un valor de energía total de interacción del sistema. La búsqueda viene a ser encontrar la posición y/o el ligando que da lugar a una energía más baja posible. Cuando se trata de desarrollar un fármaco, se estudian las interacciones entre la molécula propuesta y el sitio de interacción. Típicamente, se parte de una geometría inicial del sitio de interacción que puede haberse determinado por cristalografía de rayos X y se coloca el fármaco propuesto al que llamaremos ligando. Las interacciones se determinan calculando la energía de la interacción. Esta se obtiene a partir de un campo de fuerzas puramente clásico, aunque también es posible un cálculo mecano-cuántico semi-empírico o desde los “primeros principios”. Estas interacciones deben describirse con un campo de fuerzas muy fiable para permitir hacer una previsión relevante. Si se consigue un compuesto cuya interacción sea lo suficientemente fuerte como para que a bajas concentraciones de medicamento en el cuerpo, se forme el complejo (es decir, que la constante de estabilidad del complejo es suficientemente alta para que a concentraciones micromolares de fármaco la mayoría del fármaco esté interaccionando con el sitio receptor), es posible que el compuesto pueda servir de fármaco. Pero con esto solo se supera el primer paso. Todavía hacen falta muchos ensayos a largo plazo para determinar la bioactividad de la droga y su efecto sobre el cuerpo humano. Por ejemplo, aunque un compuesto interaccione fuertemente con el receptor, puede ser biológicamente inactivo. Una propiedad importante es que sea lo suficientemente lipofílico como para poder atravesar la membrana celular, pero no demasiado, ya que se quedaría fijado a ella. Por otra parte, puede desactivarse o ser más propenso a fijarse en otros sitios aún (¡hay muchos receptores posibles en el cuerpo y muchas vías metabolicas!): para provocar efectos inocuos o tóxicos. También es necesario, para ser apto como medicamento, que satisfaga otros condicionantes: por ejemplo, que pueda almacenarse (¡en una farmacia!) durante un tiempo razonable antes de su uso. Una vez que se encuentra un compuesto o varios compuestos adecuados (los denominados “lead compounds”), se examinan compuestos de la misma familia, por ejemplo aquellos que difieren solo por algún sustituyente, con el fin de encontrar el compuesto óptimo. 2 Físico-Química de Biomoléculas Licenciatura en Biotecnología En esta práctica nos centraremos en la etapa del reconocimiento molecular. Buscaremos los compuestos con mayor energía de interacción con el receptor. El sistema que vamos a estudiar es el receptor de la β-tripsina. La β-tripsina es una hidrolasa que cataliza, por tanto, la hidrólisis de enlaces peptídicos cuyos grupos carbonilo provienen de residuos de arginina o lisina. La benzamidina es un inhibidor competitivo de la tripsina dado que es un análogo de la cadena lateral guanidilo de la arginina (Figura 1). La benzamidina, al ocupar las cavidades que son los sitios de unión de las moléculas de tripsina, resulta ser un inhibidor competitivo para los residuos de arginina de los péptidos. Este análogo se fija a la enzima pero no reacciona. Aunque no sea una limitación fundamental para los programas de modelización molecular, muchos de ellos (como también el que usamos en esta Práctica) asumen que el receptor es rígido y que por tanto la cavidad no se deforma. Por este motivo, usaremos una estructura de la proteína cocristalizada con el inhibidor. En el caso de la β-tripsina, se sabe que la cavidad es bastante rígida, por lo que este método debe ser aplicable al examen de otros compuestos.1 La estequiometría de la interacción que consideramos es 1:1 y se puede describir mediante la siguiente reacción: ELsolv ↔Esolv+Lsolv G=-RT lnKi (1) Donde Esolv es la enzima solvatada, Lsolv el ligando solvatado y ELsolv el complejo enzimaligando solvatado. Observese que esta reacción reversible se ha escrito en el sentido de la disociación, aunque vayamos a llamar a su constante de reacción Ki (la “i” es de inhibición). Lo que buscamos determinar, a través de distintas posiciones y orientaciones de prueba, es el valor más bajo de entalpía libre. Éste nos dará también el valor de la constante de la reacción de formación del complejo. Dado que Ki es: [Esolv ][Lsolv ] , Ki = [ELsolv ] Ki se expresa en unidades de concentración (p.ej. mM). La concentración necesaria de inhibidor para que la mitad de los sitios enzimáticos estén ocupados es Ki, dado que en este caso [Esolv]= [ELsolv]. COO H3N H NH2 NH2 H3N C CH2 CH2 CH2 CH2 CH2 CH2 NH CH2 CH CH NH2 C COO H NH3 NH2 Figura 1: Benzamidina, arginina y lisina. La similitud estructural de benzamidina y arginina hace que compitan por los sitios de unión de la arginina. No se representan los H del anillo bencénico. 1 N.Ota, C. Stroupe, JMS. Ferreira-da-Silva, S.A. Shah, M. Mares-Guia, AT. Brunger, Proteins 37:641 (1999) 3 Físico-Química de Biomoléculas Licenciatura en Biotecnología La práctica esta enfocada a las posibilidades del programa ArgusLab, aunque lo más importante es entender la lógica de la modelización para comprender mejor su alcance y sus limitaciones. En efecto, existen innumerables programas para hacer “modelización molecular” de muchos tipos, y lo que cuenta, al fin y al cabo, es entender bien como de fiables pueden ser unas previsiones con el programa empleado. Aparte de unas funciones muy habituales en los programas de este tipo, tales como, por ejemplo, la posibilidad de construir moléculas y de visualizarlas, rotarlas, etc.., el programa ArgusLab es capaz de calcular energías y hacer optimizaciones de geometría con campos de fuerza y con métodos (cuánticos) semi-empíricos como el método AM1. También es capaz de calcular, con los métodos semi-empíricos, energías y poblaciones de orbitales moleculares, por lo que permite visualizar densidades electrónicas, orbitales, y calcular espectros UV/Vis. Este programa también permite el estudio de sitios de unión, basándose en el siguiente principio: se definen el ligando y el receptor, entonces se define una caja lo suficientemente grande como para abarcar la cavidad del receptor en la que se produce la unión. Se va a definir en esta caja una red de puntos con un espaciado entre puntos a elegir (típicamente 0.4 Å). Se van a probar entonces los ligandos colocando su centro de gravedad en cada uno de los puntos de la red. La energía de la interacción se calcula mediante una función llamada “Scoring function” (Puntuación). Se trata de que la energía de interacción sea lo más negativa posible, correspondiendo a una interacción fuerte entre ligando y receptor. Para cada punto de la red examinado, se permiten pequeñas traslaciones y se van probando orientaciones del ligando en búsqueda de la interacción más estable en torno a este punto de red. Opcionalmente, se permite al ligando adaptarse hasta cierto punto a la geometría de la cavidad mediante deformaciones de tipo torsión (si es un ligando flexible). Si el espaciado de red es pequeño, es común observar que las interacciones ligando-receptor son muy parecidas para puntos vecinos (misma orientación general del ligando, mismos enlaces de hidrógeno). Se dice que estas soluciones forman un “cluster de soluciones”. La “puntuación” es el aspecto más importante y más delicado del método. En este programa, como en la mayoría de los programas existentes, la Puntuación es una función empírica muy similar a los campos de fuerza, parametrizada con moléculas orgánicas y biológicas. Es muy rápida de evaluar, permitiendo así probar muchos puntos de red. La entalpía libre de la reacción (1) de formación del complejo solvatado se calcula en ArgusLab mediante la fórmula empírica (2) Gbind = Gvdw + Ghidrofóbico + GH-bond + Gdeformación + G0 (2) En otros programas, se usan otras fórmulas, pero el principio general suele ser el mismo. Esta función depende de la naturaleza y posición de los átomos de la proteína y del ligando, se ha diseñado como suma de interacciones de distinta naturaleza física y se le ha asignado una forma funcional que puede estar basada en consideraciones teóricas o no. La parametrización de un potencial empírico como el de ArgusLab se ha hecho por ajuste no lineal de los coeficientes del modelo a una serie de datos conocidos para conjuntos ligandoproteína. En la ec. (2), se define la entalpía libre como la suma de un término de van der Waals y un término de interacción hidrofóbica, de enlace de hidrógeno, de deformación y de un valor fijo que se obtiene al efectuar la regresión. Se pueden escribir estas interacciones como producto de un factor energético (C) y de otro adimensional que tiene en cuenta la geometría y la naturaleza de los átomos: Gvdw = CVDW VDW Ghidrofóbico = Chidrofóbico HP (3) (4) 4 Físico-Química de Biomoléculas Licenciatura en Biotecnología GH-bond = CH-bond HB Gdeformación = Crotor ROT G0 = Cregresión (5) (6) (7) El factor adimensional de la ec. (3) se obtiene sumando todas las interacciones ligando proteína según la siguiente expresión: VDW = ligand protein i dij,0 rij j 8 − 2 dij,0 4 rij + ligand ligand i j >i dij,0 rij 8 − 2 dij,0 4 rij (8) donde dij,0=ri+rj es la suma de los radios de van der Waals de los átomos i y j. Habitualmente, la energía potencial de estas fuerzas que existen hasta en gases nobles se expresa con un término atractivo en 1/r6 (fuerzas de dispersión, de London, de interacción dipolo inducidodipolo inducido, etc..) y un término repulsivo en 1/r12. En ArgusLab, se ha optado por parametrizarlo como fuerzas más “blandas” y de mayor alcance. como: HP = La función adimensional correspondiente a las interacciones hidrofóbicas se obtiene ligando proteina f(dij), i (9) j donde f(dij) = 1.0 si dij < dij,0 + 0.5Å = 2/3 (dij,0 + 2 – dij) si dij,0 + 0.5 Å < dij ≤ dij,0 + 2.0 Å =0 si dij > dij,0 + 2.0 Å Como se muestra en la fórmula (9), la interacción hidrofóbica se obtiene fundamentalmente contando los pares de sitios de interacción ligando-proteína hidrofóbicos en contacto. Esta interacción suele ser una de las contribuciones más importantes. Los puentes de hidrógeno de la ec. (5) se calculan a través de: θ1 d θ2 Figura 2: Notaciones que se usan para calcular la energía de un puente de hidrógeno. HB = ligando proteina HBij i (10) j HBij = f(dij) g(θ1,ij) g(θ2,ij) rij distancia entre átomos donante y aceptor θ1,ij , θ2,ij ver Figura Las funciones f y g varían de 1 a 0 de un modo sencillo (lineal por segmentos) dependiendo de lo cerca que estén del valor óptimo. Además, no se permite más de un determinado número de puentes de hidrógeno por cada aceptor y cada donante. 5 Físico-Química de Biomoléculas Licenciatura en Biotecnología Lo que cuenta es la distancia entre el átomo donante y el aceptor y las orientaciones relativas. En la fórmula de la ec. (10), no interviene la posición del hidrógeno. El único papel que desempeña el hidrógeno es convertir al átomo electronegativo correspondiente en átomo donante. La interacción de puentes de hidrógeno suele ser una de las contribuciones más importantes a la energía de interacción en ausencia de enlaces covalentes ligando-receptor. La ec. (6), que modeliza el coste energético debido a la deformación del ligando, se calcula como sigue: ROT = ligand ROTi i ROTi = 0 = 0.5 = 1.0 = 0.5 si el átomo i no esta involucrado en ninguna torsión. si el átomo i esta involucrado en 1 torsión. si el átomo i esta involucrado en 2 torsiones. si el átomo i esta involucrado en más de 2 torsiones. No se permite la deformación del receptor en ArgusLab. Finalmente, la ec. (7) es simplemente una constante que posee el mismo valor para todos los pares ligando-receptor en todos los casos. Da cuenta, de forma implícita, de la pérdida de entropía translacional y rotacional debido al hecho de que ligando y receptor están ligados. 3. Procedimiento experimental Realizaremos esta práctica con el programa ArgusLab que se descargará de la página WebCT de la asignatura: http://camelot.upo.es:8900/webct/public/home.pl Descargar también el programa OpenBabel (Babel) para la última parte de esta práctica así como un fichero zip que contiene las coordenadas de varias moléculas. Crear una carpeta de trabajo en el Escritorio en la que se ponen todos los ficheros. Esta práctica transcurre en varias etapas: Caracterizaremos la cavidad y veremos cómo interaccionan benzamidina y β-tripsina en la estructura cristalina (estudiaremos los puentes de hidrógeno). Luego, haremos una búsqueda del sitio de unión con el programa (docking calculation). Comprobaremos que la posición y orientación de la benzamidina en la cavidad es muy parecida a la dada por la estructura cristalina. Anotaremos el valor de energía de interacción. A continuación, dibujaremos la bencilamina, y haremos una búsqueda del sitio de unión. Finalmente, haremos una búsqueda automatizada de los sitios de unión de toda una base de datos. En lugar de usar una base de datos existente descargable de la red, construiremos la nuestra propia a partir de varios compuestos parecidos a la benzamidina. La búsqueda, al abarcar solo unos pocos compuestos seleccionados por su similitud con la benzamidina, durará entonces pocos minutos en lugar de días. 3.1 Caracterización de la cavidad y de la interacción con el ligando Estructura de partida: Usaremos la β-tripsina cocristalizada con benzamidina “3PTB” de la Protein Database Bank. La descargaremos de http://www.rcsb.org/pdb/Welcome.do Salvarlo bajo el nombre de 3PTB.ENT en la carpeta de nueva creación. 6 Físico-Química de Biomoléculas Licenciatura en Biotecnología Esta página ofrece además información sobre los ligandos estructuralmente parecidos a la benzamidina. A modo de ejemplo, cabe mencionar las estructuras de isoniacida, salicilamida y hidroxicarbamida. Estos 3 compuestos también se incluirán en la búsqueda de sitios de unión. Nota: Si no fuera posible acceder a la web externa, esta estructura está también disponible como fichero de ejemplo del programa ArgusLab (C:\Archivos de Programas\Arguslab\Tutorials\Docking\Beta Trypsin). Abrir el fichero con la estructura descargada de la base de datos. En la parte izquierda de la pantalla tiene que verse una estructura de ficheros que inicialmente sólo muestra “3ptb”. Si no fuese visible, hacerlo visible con Tools→Molecule Tree View o pulsando el icono . Ampliar la vista en árbol (tree view) de 3ptb pulsando sobre la carpeta “3ptb”. Aparecen entonces varias carpetas como p.ej. Atoms, Residues y Groups. Ampliar Residues→Misc. Aparece 1 BEN y 480 CA que son el ligando benzamidina y un catión de calcio, respectivamente. La vista será (salvo por el color de fondo que se ha cambiado usando Settings →Colors→Pick color): Figura 3: Vista del programa ArgusLab. Se representa 3PTB, una estructura compuesta de βtripsina, del ligando benzamidina complejado, así como aguas de cristalización y un catión de Ca2+. Ahora hay que definir la benzamidina como ligando y el entorno de la benzamidina como el receptor (binding site). El programa puede así calcular la energía de interacción entre el ligando y el receptor y se pueden definir varios ligandos en la misma cavidad sin que se estorben unos a otros (los ligandos no se “ven” unos a otros). En efecto, el programa 7 Físico-Química de Biomoléculas Licenciatura en Biotecnología considera secuencialmente la interacción ligando-receptor y calcula una energía (o una serie de energías cuando se vaya a hacer la búsqueda del sitio de unión idóneo) para cada par ligando-receptor. Marcar 1 BEN con el botón izquierdo del ratón para seleccionar el ligando. La benzamidina se pone de color amarillo. Esconder el receptor de la vista. En el menú Edit →Hide Unselected. Solo permanece visible la benzamidina. Centrar la vista en pantalla en torno a los elementos seleccionados (en este caso, la molécula de benzamidina). Para ello, View →Center Molecule in Window o con el icono . Añadir los hidrógenos mediante el icono . Seleccionar con el botón derecho del ratón: Set Render Mode → BallCylinder med Con la molécula todavía seleccionada, pulsar de nuevo el botón derecho del ratón en la parte de vista en árbol (tree view). Seleccionar la opción Make a Ligand Group from this Residue. En la carpeta “Groups”, ArgusLab crea un icono llamado “1 BEN” de tipo Ligando. La vista al término de estas operaciones debería ser la que aparece a continuación: Figura 4: Vista del programa ArgusLab. Se representa la benzamidina. En este punto (si no se ha hecho ya antes), salvar el fichero en formato nativo (ArgusLab). Salvar con regularidad este fichero a lo largo del progreso de la práctica. Vamos a renombrar el ligando. Para ello, seleccionar “1 BEN” de la carpeta Groups, pulsar el botón derecho del ratón, y seleccionar Modify Group. Introducir el nombre: “Ligando BD por 8 Físico-Química de Biomoléculas Licenciatura en Biotecnología DRX” (BD designará la benzamidina). El tipo de grupo no se debe modificar, debe seguir siendo un ligando. Vamos a crear ahora otro grupo que es el entorno del ligando previamente definido. Pulsar el botón derecho del ratón en el ligando que acabamos de renombrar y seleccionar Make a BindingSite Group for this Group. Esto crea un binding site formado por todos los residuos que tienen al menos un átomo a una distancia no mayor de 3,5 Å de alguno de los átomos del ligando de DRX. No sólo aparecen los residuos en la vista gráfica, sino que se pone de manifiesto también en la vista en árbol con la creación de un grupo “Ligando BD por DRX bindingsite”. Necesitamos cambiar la ampliación de la vista. En Groups, seleccionamos el ligando y su binding site. Usar shift y botón izquierdo del ratón o control y botón izquierdo del ratón. Como antes, se usa el botón que permite centrar la vista en la pantalla. Para hacernos una idea de tamaño y forma de la cavidad, vamos a construir una superficie de contorno de la cavidad. Así podremos entender e incluso predecir si un determinado ligando podría “caber” en la cavidad (debido a la forma y tamaño del ligando o, dicho de otra manera, debido a los impedimentos estéricos). Vamos a definir una caja que engloba a la cavidad y a calcular su superficie de contorno. Un modo de hacerlo es a través del menú de búsqueda de sitio de unión(¡!). Calculation →Dock a Ligand o bien . Este menú que está pensado para poder elegir un ligando y un binding site y definir una caja en la que se llevará a cabo la búsqueda del sitio de unión se va a utilizar de momento solamente para definir una caja. Donde pone “Binding Site Bounding Box” comprobar que está marcada la opción “Display Box”. Pulsar “Calculate Size”, de manera que el programa determine un tamaño de caja optimizado a las dimensiones de la cavidad. No necesitamos nada más de momento, de modo que salimos del menú con Cancel (anular) o con OK (¡Tener cuidado de NO salir con Start!). Sorprendentemente, ArgusLab mantiene activa la caja aún habiéndo salido del menú anterior con anular. Seleccionar Surfaces →Solvent Accessible Surface En este menú se define como “Bounding Box” ya no toda la molécula sino solo “Use Displayed Box”. A efectos del espacio ocupado, se deberá excluir, lógicamente, al ligando. Seleccionar “Surface Quality” de calidad media y la superficie de un color elegido por el usuario. El gris es muy adecuado. Rotar la molécula de manera que se vea asomar el ligando. Esta imagen se puede salvar en formato jpeg (File →Export). 9 Físico-Química de Biomoléculas Licenciatura en Biotecnología Podemos borrar la superficie que acabamos de crear: botón derecho del ratón encima de la superficie y seleccionar Remove Surface. Como objetivo siguiente, vamos a visualizar en esta geometría los puentes de hidrógeno formados. Seleccionar en “Groups” el “ligando BD por DRX”. Pulsar botón derecho del ratón y marcar Show Hydrogen Bonds Rotar y ampliar adecuadamente para ver en qué sitios la proteína “ancla” al ligando. Observar que el programa ignora los átomos de hidrógeno, aunque asume que la estructura está adecuadamente provista de hidrógenos. Por eso, los puentes de hidrógeno se indican entre dos átomos electronegativos: uno actúa como donante, el otro como receptor del puente de hidrógeno. Anotar en la Tabla siguiente los átomos implicados, las distancias de los enlaces de hidrógeno y el aminoácido implicado de la proteína. Átomo del ligando Átomo del receptor Distancia (d=donante, Aminoácido implicado a=aceptor) 3.2 Búsqueda del sitio de unión de la benzamidina Después de esta etapa de caracterización del sitio de unión obtenido por DRX, se procede a probar el campo de fuerzas haciendo una búsqueda del sitio de unión. Comprobaremos la validez del resultado comparándolo con la información cristalográfica. Seleccionar el “ligando BD por DRX”, sea en la carpeta “Groups” o en la de “Residues→Misc”. Copiarlo y pegarlo. Esto se puede hacer por ejemplo con Control-C y Control-V. En Residues→Misc aparecerá un nuevo ligando 810 BEN. Definirlo como ligando. Para ello, pulsar el botón derecho del ratón, Make a Ligand Group from this Residue. En la carpeta “Groups” debería haber aparecido otro ligando llamado 2 BEN. Renombrarlo: pulsar el botón derecho del ratón, Modify Group. Darle el nombre de “Ligando BD”. 10 Físico-Química de Biomoléculas Licenciatura en Biotecnología Le vamos a dar un aspecto diferente para que se vea mejor. Una vez seleccionado el ligando, pulsar el botón derecho del ratón →Set Render Mode→Cylinder med Ahora lanzamos el cálculo de la búsqueda del sitio de unión. Nota: el ligando no tiene por qué estar en la cavidad, no necesitamos moverlo ya que el programa define la caja de acuerdo a la geometría del receptor y va probando posiciones. Activar el menú de búsqueda con Calculation→Dock a Ligand o pulsando . Seleccionar el ligando apropiado (Ligando BD). En el apartado de la caja (Bounding Box), pulsar Calculate Size. Cerciorarse de que el algoritmo de búsqueda sea “Argusdock”, el tipo de cálculo “Dock” y el Ligando Flexible. Pulsar entonces Start y observar como va probando los sitios. Para cada punto posible de la red (es decir, en el que no se llegue a interacciones de energía altísimas por “chocar” algún átomo del ligando con el receptor), se muestra brevemente el ligando en su posición. Inspeccionar las estructuras, que deberían estar relativamente cercanas. Para apreciar cuantitativamente los resultados, seleccionar los dos ligandos en Groups con la tecla Control y el botón izquierdo del ratón. Ahora, posicionarse en la Carpeta Groups, pulsar el botón derecho del ratón y seleccionar Calc RMSD position between two similar Groups. Anotar el valor de la desviación típica obtenida que debería encontrarse en torno a 2,5 Å. La información que se ha generado durante el cálculo de búsqueda está disponible en dos sitios. En la vista en árbol, en “Calculations”, se puede ampliar la información y aparecen las distintas posiciones del ligando (“pose”) y el valor de la entalpía libre, ordenados por estabilidad decreciente. También se indica el tamaño del “cluster”, posiciones que después de minimizar han quedado tan cercanas que son equivalentes. Pulsar el botón derecho del ratón en “Pose 2” y seleccionar la opción Display. De manera general, cuando una propiedad en la vista en árbol tiene un icono de mechero Bunsen significa que se puede representar gráficamente. También es posible examinar la desviación típica. El segundo sitio desde el que se accede a información sobre los cálculos es en el fichero de registro (log) que se consulta pulsando el icono . Esto abre el fichero en un editor, generalmente el block de notas. Con cada nuevo cálculo se sobrescribe el fichero. 11 Físico-Química de Biomoléculas Licenciatura en Biotecnología 3.3 Búsqueda del sitio de unión de la bencilamina NH3 H2C Figura 5: Bencilamina. No se representan los H del anillo bencénico. Vamos a construir la bencilamina y a usarla como ligando. Por supuesto, hay muchas maneras de construirla, por ejemplo usando la utilidad de construir moléculas de ArgusLab (el ModelBuilder). De todas maneras, se propone aquí otra manera de hacerlo, propia a ArgusLab. Dado que la bencilamina es un compuesto relacionado con la benzamidina, se puede partir de esta última: Posicionarse en el residuo de benzamidina procedente del análisis de difracción de rayos X (DRX). Copiarlo con CTRL-C. Abrir un fichero nuevo con File→ New. Empastar la benzamidina (CTRL-V). Seleccionar los átomos que sobran y borrarlos (botón derecho del ratón, ...). Ahora faltan por completar los hidrógenos usando el icono . Se debería comprobar siempre el resultado, en especial: que el número de H adicionados por el programa es “razonable”, que las distancias de enlace son realistas, que la naturaleza del enlace es la deseada (enlace simple, doble, de resonancia, ...), y que la “carga formal” sea la correcta. Para seleccionar los enlaces, marcar la molécula entera. Una vez seleccionada, marcar el enlace. Pulsar el botón derecho del ratón y elegir el tipo de enlace deseado o fijar la longitud del enlace. Se puede ahora salvar el resultado como fichero bencilamina.pdb en formato pdb con Save as. Nota: ArgusLab solo salva lo que esta activo en este momento. Aunque haya varios sistemas abiertos y se vean uno debajo de otro en la vista en árbol, ArgusLab solo trabaja con un sistema a la vez, aunque se pueda pasar cómodamente de un sistema a otro. Regresar al sistema anterior pulsando en él en la vista en árbol y hacer la búsqueda de sitio de unión de la bencilamina. ¿Echáis algo de menos? Puede que en la ventana del cálculo de búsqueda de sitio de unión no aparezca el ligando que queréis. Será que no lo habéis definido como ligando. Regresar a la bencilamina y definirla como ligando. Ahora debería estar disponible para cálculos de “docking”. 3.4 Construcción de una base de datos para probar varios ligandos 12 Físico-Química de Biomoléculas Licenciatura en Biotecnología La búsqueda del sitio de unión más estable en los pasos anteriores sólo implicaba definir un tamaño de caja lo suficientemente grande. Dado que el tamaño de caja viene determinado por la cavidad, es posible calcularlo una única vez y probar con varios ligandos. Se podrían probar una serie de ligandos de manera automatizada sin intervención del usuario. Este es el principio de la búsqueda que nos proponemos ahora. ArgusLab permite hacer este tipo de búsquedas y necesita para ello un fichero de base de datos de formato SDF. Vamos a construir este fichero nosotros mismos con unas cuantas estructuras de ligandos. Para esto, usaremos el programa OpenBabel. Este programa es un convertidor de formatos de ficheros de coordenadas moleculares. También nos permite construir un fichero SDF a partir de varios ficheros PDB correspondientes a nuestros ligandos. Utilizaremos las estructuras de varios ligandos que se encuentran en la página WebCT. Abrir OpenBabel y crear la base de datos (este proceso es bastante intuitivo). Esta base de datos deberá contener los compuestos de la Figura 6 y al menos dos compuestos de la Figura 7 (se podrá prescindir de examinar la hidroxicarbamida): O O C C Cl NH2 CH3 C C C NH2 NH2 NH2 NH2 NH2 O CH2 C NH2 NH2 NH2 Figura 6: p-cloro-benzamidina, p-amino-benzamidina, p-metil-benzamidina y pamidino-fenilpiruvato. N C O O C NH2 HN OH OH NH NH2 O C NH2 13 Físico-Química de Biomoléculas Licenciatura en Biotecnología Figura 7: Hidroxicarbamida, isoniacida (hidracida del ácido isonicotínico; nombre IUPAC: 4-piridina-carbohidracida), salicilamida (IUPAC: 2-hidroxi-benzamida) Comprobar que Babel crea un fichero SDF de salida y que este fichero contiene coordenadas (estas deben aparecer en la ventana derecha de Babel). Tener cuidado de darle un nombre de fichero que acabe en “.sdf” o “.SDF”, dado que Babel no le añade automaticamente esta extensión al nombre. De carecer de esta extensión, no será posible cargar la base de datos en ArgusLab. Una vez que se disponga del fichero SDF que contiene los datos estructurales de todos los compuestos, realizar un cálculo de búsqueda de sitio de unión de una base de datos. Escoger el tamaño de caja que ArgusLab determine en base a la cavidad. El grid puede mantenerse en 0.4 Å. Una vez finalizado el cálculo, consultar los resultados en el fichero de texto que transcribe los resultados. Ir completando los resultados en la tabla siguiente: Energía Compuesto de inter- acción experimental (kcal/mol) benzamidina -6.52 bencilamina -3.91 p-cloro-benzamidina -5.82,3 p-amino-benzamidina -6.93 p-metil-benzamidina -6.23 p-amidino-fenilpiruvato +0.354 salicilamida ? hidroxicarbamida ? isoniacida ? Energía de inter- acción computada de Ki,exp en mol/l config. más estable (kcal/mol) 2 F.L. Gervasio, A. Laio, M. Parrinello, J.Amer.Chem.Soc. 127: 2600 (2005) J.W. Essex, D.L. Severance, J. Tirado-Rives, W.L. Jorgensen, J.Phys.Chem.B 101:9663 (1997) 4 Base de datos PDB, estructura 1TPP (Marquart et al., Acta Cryst. B 39:480 (1983)) 3 14 Físico-Química de Biomoléculas Licenciatura en Biotecnología Considerando sólo la estabilidad del complejo de interacción, sin consideraciones de lipofilia, etc... ¿Cuál es, según los datos experimentales, el ligando más apropiado para inhibir la hidrolasa? ¿Y según los cálculos? ¿Cómo se ajustan los datos experimentales y los calculados? Llevar a cabo el análisis de los resultados obtenidos. 7. Bibliografía Andrew R. Leach, Molecular Modelling, NJ USA: Kluwer Acad., 1998 Tamar Schlick, Molecular Modelling and Simulation: an Interdisciplinary Guide, Springer, New York, 2002, disponible como libro electrónico 15

