Archivo PDF, 4 slides por página, 126 KB
Anuncio

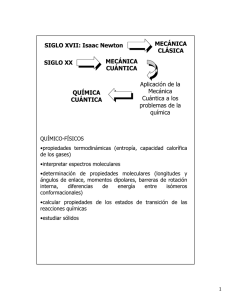
MECÁNICA CLÁSICA SIGLO XVII: Isaac Newton SIGLO XX QUÍMICA MECÁNICA CUÁNTICA QUÍMICA CUÁNTICA QUÍMICA Aplicación de la Mecánica Cuántica a los problemas de la química TEÓRICA QUÍMICA QUÍMICA COMPUTACIONAL CUÁNTICA MODELADO MODELADO MOLECULAR MOLECULAR QUÍMICO-FÍSICOS •propiedades termodinámicas (entropía, capacidad calorífica de los gases) •interpretar espectros moleculares •determinación de propiedades moleculares (longitudes y ángulos de enlace, momentos dipolares, barreras de rotación interna, diferencias de energía entre isómeros conformacionales) •calcular propiedades de los estados de transición de las reacciones químicas •estudiar sólidos QUÍMICOS ORGÁNICOS •estimar estabilidades relativas de las moléculas •calcular propiedades de los intermedios de reacción •estudiar mecanismos de las reacciones químicas •analizar espectros de RMN QUÍMICOS ANALÍTICOS QUÍMICA TEÓRICA: es la disciplina que estudia todos los métodos teóricos que pueden aplicarse a la química. QUÍMICA CUÁNTICA:es la disciplina que estudia la aplicación de la Mecánica Cuántica al estudio de las moléculas. QUÍMICA COMPUTACIONAL: es la disciplina que estudia el uso de métodos numéricos aplicados a la química. MODELADO MOLECULAR: es la disciplina que estudia la construcción de modelos de la estructura y el comportamiento molecular. MODELO: Conjunto de objetos y reglas predeterminadas que los relacionan, utilizado como descripción simplificada de un fenómeno real cuya complejidad desafía la descripción exacta. Alternativamente, puede constituir una descripción precisa de las propiedades que interesan del sistema real en estudio, mientras que contiene una descripción promediada, simplificada, o simplemente inexistente de las otras propiedades que constituyen su complejidad. Ejemplos: Modelo de puntos de Lewis Modelo de reacciones: SN2, SEAr •espectroscopía MODELADO MOLECULAR QUÍMICOS INORGÁNICOS •propiedades de los compuestos de coordinación de metales de transición OBJETIVO: SISTEMA QUÍMICO LEYES FÍSICAS MODELOS TÉCNICAS NUMÉRICAS EXPLICAR PREDECIR BIOQUÍMICOS •conformaciones de las moléculas biológicas •interacción enzima-sustrato •solvatación de moléculas biológicas •Caracterizar y predecir la estructura y estabilidad de los sistemas químicos •estimar diferencias energéticas entre diferentes estados •explicar caminos de reacción, mecanismos, procesos químicos generales a nivel atómico y molecular. 1 ¿CÓMO HACER MODELADO MOLECULAR? CONSTRUCCIÓN DE MODELOS MOLECULARES Colección de datos organizados Geometría de una molécula PROGRAMAS DE COMPUTACIÓN Estructura de datos ORGANIZACIÓN JERÁRQUICA: •Cálculo de energías asociadas a ellas moleculares y átomos enlaces ángulos diedros monómeros subunidades propiedades •cálculo de la estructura tridimensional de las moléculas DATOS: •descripción de la composición química •visualización de las moléculas y de sus propiedades •descripción de la geometría •análisis cuantitativo de los resultados del modelo •descripción del patrón de enlace Computacionalmente rayos x, RMN COMPOSICIÓN QUÍMICA: C, H, O, etc. Hibridación (MM) Tipo de átomo PATRÓN DE ENLACE: conectividad GEOMETRÍA: se especifica en forma de coordenadas para todos los átomos CONVERSIÓN 2D --> 3D vía una interfase gráfica Coordenadas cartesianas Matriz Z CONVERSIÓN 2D --> 3D: Hyperchem MÉTODOS DE CÁLCULO Empíricos Semiempíricos “Model builder” ab initio Funcionales de la densidad Energías Propiedades Propiedades moleculares dependientes de la conformación COORDENADAS: CARTESIANAS INTERNAS x,y,z para cada átomo Matriz Z distancias ángulos diedros MÉTODOS DE MANIPULACIÓN DE LA GEOMETRÍA MOLECULAR COORDENADAS CARTESIANAS: Minimización Dinámica Molecular Monte Carlo C Perturbación Diferentes conformaciones del sistema ANÁLISIS Parámetros geométricos 0.0 0.0 0.0 O 0.0 1.22 0.0 H 0.94 -0.54 0.0 H -0.94 -0.54 0.0 Superposición/comparación MÉTODOS GRÁFICOS Interpretación visual de las conformaciones y las propiedades moleculares Mapeo de superficie o contorno Ventajas: permite reconocer simetría Trayectorias Desventajas: son redundantes 2 CONDICIONES DE SIMETRÍA: COORDENADAS INTERNAS: MATRIZ Z O1 C2 O1 r1 H3 C2 r2 O1 t1 H4 C2 r2 O1 t1 H3 180.0 r1 1.20 r2 1.01 t1 119.0 dij: distancia de enlace, es simplemente la distancia entre los átomos enlazados i y j. θijk: ángulo de enlace, es el ángulo formado entre los enlaces ij y jk. El ángulo de enlace es siempre positivo y menor de 180º, es decir, es el menor de los dos ángulos posibles. φjkl: ángulo de torsión (también denominado ángulo diedro) entre los cuatro átomos i-j-k-l se define como el ángulo entre los dos planos, uno conteniendo a los átomos i, j y k y el otro conteniendo a los átomos j, k y l O1 O2 O1 r1 H3 O2 r2 O1 t1 H4 O1 r2 O2 t1 H3 180.0 r1 1.30 r2 0.95 t1 109.0 O1 O2 O1 r1 H3 O2 r2 O1 t1 H4 O1 r2 O2 t1 H3 f1 ÁNGULO DE TORSIÓN: o ángulo diedro entre cuatro átomos 1-2-3-4 se define como el ángulo entre los dos planos, uno conteniendo a los átomos 1, 2 y 3 y el otro conteniendo a los átomos 2, 3 y 4. CONDUCE A RESULTADOS INCORRECTOS r1 1.30 r2 0.95 t1 109.0 f1 180.0 ÁTOMOS DUMMY: COORDENADAS INTERNAS: MATRIZ Z O1 Ángulo de enlace = 180.0 C2 O1 1.20 Ángulo diedro indefinido H3 C2 1.01 O1 119.0 H4 C2 1.01 O1 119.0 H3 180.0 ÁTOMO DUMMY CONSTANTES Y VARIABLES: O1 líneas de Matriz Z O1 C2 O1 r1 C2 H3 C2 r2 O1 t1 XX3 C2 1.0 O1 90.0 H4 C2 r3 O1 t2 H3 f1 C4 C2 r2 XX3 90.0 O1 ---------------------------------------------------------------- línea en blanco H5 C4 r3 C2 t1 XX3 0.0 F1 180 H6 C4 r3 C2 t1 XX3 180.0 líneas de constantes O1 r1 180.0 ---------------------------------------------------------------- línea en blanco r1 1.20 líneas de variables r1 1.20 r2 1.01 r2 1.30 r3 1.01 r3 1.00 t1 119.0 t1 120.0 t2 119.0 3 X2 X1 X2 1.0 C1 X1 r1 X2 90.0 H11 C1 r2 X1 t1 X2 H12 C1 r2 X1 t1 X2 180.0 C2 X1 0.0 r1 X2 90.0 C1 120.0 H21 C2 r2 X1 t1 X2 H22 C2 r2 X1 t1 X2 180.0 C3 X1 0.0 r1 X2 90.0 C1 -120.0 H31 C3 r2 X1 t1 X2 0.0 H32 C3 r2 X1 t1 X2 180.0 r1 0.75 r2 1.10 t1 109.0 OPERANDO SOBRE LA GEOMETRÍA MOLECULAR CREACIÓN Y EDICIÓN MODEL BUILDING Dibujar moléculas en 2D Model Builder Coordenadas cartesianas Matriz Z Archivos PDB Aproximación a estructuras 3D CÁLCULOS Métodos Resultados INFORMACIÓN OBTENIDA Single Point Energía total de una configuración Optimización de geometría Una configuración estable Dinámica Molecular Simulación del cambio de la conformación molecular con el tiempo y la temperatura geométrica energética HOMO LUMO potenciales electrostáticos densidad de carga densidad de espín momento dipolar 4