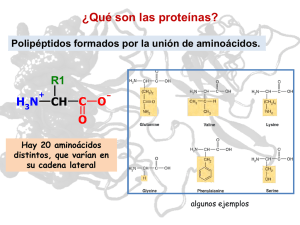
PROTEINAS Aminoácidos Estructura y clasificación de los aminoácidos. Códigos de abreviación Existen 20 aminoácidos diferentes de que forman parte de las proteínas. Todos ellos son α-aminoácidos y constan de un grupo amino, un grupo carboxilo, un hidrógeno y un grupo distintivo llamado R unidos a un mismo carbono denominado carbono-α. El carbono-α recibe este nombre por ser el carbono adyacente al carbono del grupo carboxilo, y el grupo diferenciador de los distintos aminoácidos (R) se denomina cadena lateral. Aminoácidos alifáticos Su característica fundamental es la hidrofobicidad de la cadena lateral (con excepción de Gly) Suelen ocupar el interior de las proteínas globulares, donde contribuyen a la estructura global de la proteína debido al efecto hidrofóbico (“micela proteica”) Reactividad química muy escasa Glicina tiene un destacado papel estructural: suele ser invariante en series filogenéticas Aminoácidos aromáticos La presencia de sistemas aromáticos hace que absorban luz UV en torno a 280 nm; la absorción UV de las proteínas se debe a su contenido en estos aminoácidos Fenilalanina y triptofano son hidrofóbicos Tioaminoácidos Importante papel estructural de la cisteína por la posibilidad de formar enlaces disulfuro con otro residuo de cisteína La cisteína participa en el centro activo de muchas enzimas La metionina es el aminoácido iniciador de la síntesis de proteínas (codon AUG) Prolina Importante papel estructural: su presencia dificulta la formación de estructura secundaria Por esa misma razón suele ser invariante en series filogenéticas Como tal o modificado por hidroxilación (4-hidroxiprolina) es muy abundante en el colágeno Hidroxiaminoácidos El grupo -OH de la serina es fundamental en el centro activo de muchas enzimas (serin proteinasas, p.e.) Forma enlaces glicosídicos con oligosacáridos en ciertas glicoproteínas El grupo -OH tanto de S como de T es susceptible de fosforilación: importante modificación postraduccional que regula la actividad de muchas proteínas Aminoácidos dicarboxílicos y sus amidas Todos ellos son importantísimos intermediarios en el metabolismo nitrogenado, sobre todo glu y gln Forman parte del centro activo de las glicosidasas y de las serin enzimas (tríada SHD) Proveen a la proteína de superficies aniónicas que sirven para fijar cationes (p.e., Ca++) Aminoácidos dibásicos Lisina sirve para formar intermediarios covalentes en catálisis enzimática (bases de Schiff) Lisina es el aminoácido que une determinadas coenzimas a la estructura de la proteína Arginina es un importante intermediario en el ciclo de la urea Histidina forma parte del centro activo de muchas enzimas, debido al carácter nucleófilo del imidazol Histidina contribuye al tamponamiento de los medios biológicos por tener un pKa cercano al pH intracelular Aminoácidos: estereoisomería COO + * H 3N C H * C H COO+ OH H OH CH 3 + - H OHH H OH CH3 L- alo-Treonina carbono quiral o asimétrico OH H D-Treonina H 3N C C C *C CH3 L-Treonina COO H3HN C -NH H3+ COO + - H3H N C -NH H3+ H C OH CH3 D-alo-Treonina Formación de Diastero isómeros COO + H 3 N *C H H *C CH COO+ 3 HH 3N C NH H 3+ CH3H C CH 2 CH2 CH 3 CH3 L-Isoleucina D-Isoleucina COO + HH 3 C H 3N C H CHH3 C CH H 3 COO + H 3HN H C NH H 3+ C CH 3 CH 2 CH 2 CH 3 CH 3 L-alo-Isoleucina D-alo-Isoleucina Formación de Diastero isómeros Clasificación de aminoácidos según polaridad • Aminoácidos apolares o hidrofóbicos: - ala (A), val (V), leu (L), ile (I), fen (F), trp (W), met (M), pro (P) • Aminoácidos polares sin carga eléctrica: - gly (G), tir (Y), ser (S), tre (T), cis (C), asn (N), gln (Q) • Aminoácidos polares con carga eléctrica: - Aniónicos (-): asp (P), glu (E) - Catiónicos (+): Lis (K), arg (R), His (H) Cadenas laterales con carga eléctrica Arginina Aspártico (ác.) Glutámico (ác.) Histidina Lisina 12.48 3.86 4.25 6.00 10.53 + + + 4. Ácidos y Bases Ácidos y Bases (teoría de Lewis) Bases: grupos moleculares con pares electrónicos libres (nucleófilos) Ácidos: grupos moleculares con afinidad hacia pares electrónicos libres (electrófilos) Ácidos y Bases (teoría de Bronsted-Lowry) Ácido: especie química que tiende a ceder protones Base: especie química que tiende a aceptar protones A- + H+ AH B + H+ BH+ Sustancia anfipróticas, una sustancia que como agua es tanto capaz de dar como de aceptar protones. Ácido Ác. Clorhídrico Ác. Fórmico Ác. Acético Ác. Carbónico: Disociación 1 Disociación 2 Ác. Fosfórico: Disociación 1 Disociación 2 Disociación 3 Amoníaco Metilamina Base conjugada HCl H-COOH CH3-COOH ClH-COOCH3-COO- H2CO3 HCO3- HCO3CO32- H3PO4 H2PO4HPO42NH4+ CH3NH3+ H2PO4HPO42PO43NH3 CH3NH2 Producto iónico del agua H2O H+ + OH[H+][OH-] Kd = [H2O] Kd[H2O] = [H+][OH-] = 10-14 Monopróticos Polipróticos HCL H2CO3 2. Determinar la concentración molar de los iones H+ y de iones OH- en las siguientes Soluciones: (a) Solución de HCl 0.0004 moles/L (b) Solución de KOH 0.002 moles/L Resp : (a) [H+] = 4 x 10-4 moles/L ; [OH-] = 2.5 x 10-11 moles /L (b) [OH-1] = 2 x 10-3 moles/L; [H+] = 5.0 x 10-12 moles / L Concepto de pH pH= -log10 [H+] - Logaritmo cambiado de signo de la concentración de hidrogeniones - Dado el producto iónico del agua, [H+][OH-] = 10-14 la neutralidad ácido-base tiene lugar cuando [H+] = [OH-] = 10-7 ; pH = 7 - Medio ácido: pH < 7 Medio básico: pH > 7 3. Suponga que la autoionización contribuye en forma insignificante a las concentraciones de iones hidrogeno e hidróxilo. Calcular el pH de las siguientes soluciones acuosas: 1. 2. 10-3 M HCl 10-4 M NaOH R. 1. 10-3 M HCl [H+] o [ H3O+] =10-3; por lo tanto el pH=3 2. 10-4 M NaOH [OH+] =10-4M puesto que [OH-] [ H3O+]=10-14 [ H3O+]= 10-10 por lo tanto el pH=10 Concepto de pKa pKa= -log10 Ka - Es la fuerza que tienen las moléculas de disociarse (es el logaritmo negativo de la constante de disociación de un ácido débil). - Es propio de un grupo, no de una molécula - Cuando pH = pKa, [ácido] = [base] - La capacidad tampón es máxima en las proximidades del pKa - Un ácido es tanto más fuerte cuanto más bajo es su pKa; una base es tanto más fuerte cuanto más alto es su pKa. pKa, the symbol for the acid dissociation constant at logarithmic scale pKa de algunos sistemas ácido-base Ác. Fórmico Ác. Acético Ác. Fosfórico: Disociación 1 Disociación 2 Disociación 3 Amoníaco Metilamina 3.75 4.76 2.14 7.20 12.40 9.25 10.60 AH A- + H+ [A-][H+] Ka = [AH] Ecuación de HendersonHasselbalch Es útil para predecir las propiedades de las soluciones amortiguadoras. [A-] logKa = log [H+] + log [AH] [A-] -log [H+] = -logKa + log [AH] [ base] pH = pKa + log [ácido] Soluciones Amortiguadoras Soluciones amortiguadoras son aquellas soluciones cuya concentración de hidrogen-iones varía muy poco al añadirles ácidos o bases fuertes. El objeto de su empleo, tanto en técnicas de laboratorio como en la finalidad funcional del plasma, es precisamente impedir o amortiguar las variaciones de pH y, por eso, suele decirse que sirven para mantener constante el pH. [ base] pH = pKa + log [ácido] La Ecuación de Henderson-Hasselbach permite calcular el pH de una mezcla amortiguadora conociendo su composición. En su deducción, para un amortiguador compuesto de un ácido débil y una sal de su base conjugada, se considera que la concentración de ácido libre es aproximadamente igual a la del ácido total, y la concentración del ión base conjugada coincide con la concentración de la sal. 4. Calcular el pH de la solución que resulta al mezclar dos soluciones acuosas que contienen, respectivamente, 2 moles de ácido acético y 6 moles de acetato de sodio. El pKa del ácido acético es 4.75 pH = pKa + log [base] [ácido] CH3COOH CH3COONa+ R. pH= 4.75+log 6 = 5.22 2 pH<pKa H+ activo, sustancia protonada pH>pKa H+ Inactivo, sustancia desprotonada Sistema carbónico-bicarbonato CO2 + H2O H2CO3 HCO3- + H+ carbónico bicarbonato - pKa relativamente alejado del pH fisiológico Base: [HCO3-] - Ácido: [CO2] - [CO2]: regulado por la respiración - [HCO3-]: regulado por excreción renal Sistema de los fosfatos H3PO4 H2PO4- Disociación 1 Disociación 2 pKa pKa Disociación 3 pKa 12.40 HPO4-- PO4--- 2.14 7.20 - La segunda disociación tiene un adecuado al tamponamiento fisiológico, pero su concentración es relativamente baja. Titulación de ácido acético CH3 COOH CH3 COO- + H+ 9 8 CH3 COO- 7 pH 6 5 pKa [Acido=Base] 4 3 CH3 COOH 2 1 0 1 Base añadida Titulación de ácido acético Comportamiento tampón 9 8 7 pH 6 5 4 Región donde el pH cambia muy poco 3 2 1 0 1 Base añadida El punto isoeléctrico es el pH al que una sustancia anfótera tiene carga neta cero pI = (pKa¹ + pKa²)/2 13 aa + Tirosina pKCOOH+pKNH3/2 Aminoácidos ácidos (Ácido Aspártico y Ácido Glutámico) pKCOOH+pKR/2 Aminoácidos básicos (Histidina, Lisina y Arginina) pKNH3+pKR/2 H3N+ I II COOH COO- COO- C H H2N C H C H CH3 H 3N + CH3 III CH3 Alanina pH < pKC pH > pKC pH > pKC pH < pKN pH < pKN pH > pKN I II COO- COOH H3N+ C H III H 3N + C H H 3N + IV COO- COO- C H H2N C H CH2 CH2 CH2 CH2 COOH COOH COO- COO- pH < pKC pH > pKC pH > pKC pH > pKC pH < pKL pH < pKL pH > pKL pH > pKL pH < pKN pH < pKN pH < pKN pH > pKN Titulación de aminoácido dicarboxílico 14 COOH H3N+ 12 C H IV CH2 10 COOH pH 8 III 6 4 II 2 I 0 0 1 2 Base añadida (uu. arbitrarias) 3 I II IV COO- COO- COO- C H H 2N C H H2N C H (CH2)4 (CH2)4 (CH2)4 (CH2)4 NH3+ NH3+ NH3+ NH2 COOH H3N+ III C H H3N+ pH < pKC pH > pKC pH > pKC pH > pKC pH < pKN pH < pKN pH > pKN pH > pKN pH < pKL pH < pKL pH < pKL pH > pKL Titulación de aminoácido dibásico 14 COOH H3N 12 + IV C H (CH2)4 10 NH3 III + pH 8 6 II 4 2 I 0 0 1 2 Base añadida (uu. arbitrarias) 3 RADICALES IONIZABLES ε-amino lisina (lys, K) Guanidio Arginina (arg, R) Imidazol Histidina (his, H) β-carboxilato Aspártico (asp, D) γ-carboxilato Glutámico (glu, E) A pH bajo, la mayor parte de los grupos disociables estarán protonados, y por lo tanto habrá un gran número de cargas positivas en la proteína A pH elevado, los grupos disociables no estarán protonados, con lo cual habrá mayor número de cargas negativas Cátodo (-) Ánodo (+) Oxitocina podría estar involucrada en la formación de relaciones de confianza y generosidad Enlace vacío Largo (nm) Fuerza en agua Fuerza en (kcal/mol) Covalente 0.15 90 90 Iónico 0.25 3 80 Hidrógeno 0.30 1 4 van der Waals 0.35 (por átomo) 0.1 0.1 Los enlaces de hidrógeno y de van der Waals son de poca fuerza, sin embargo son muy comunes e importantes en las interacciones intra e intermoleculares de proteínas y lípidos, definiendo, por ejemplo, la estructura terciaria de una proteína y la formación de bicapas lipídicas. Niveles estructurales en las proteínas Estructura primaria: Secuencia de aminoácidos Estructura secundaria: Plegamiento básico de la cadena debido a enlaces de hidrógeno entre grupos -CO- y -NHde la unión peptídica: hélices, láminas y giros Estructura terciaria: Estructura tridimensional de la proteína Estructura cuaternaria: Asociación de distintas subunidades, siendo cada una un polipéptido. 0RGANIZACIÓN PRIMARIA Estructura primaria de la insulina Cadena peptídica de 21 aas Cadena peptídica de 30 aas Organización secundaria Using fundamental chemical principles and a few experimental observations, Pauling and Corey predicted the existence of these secondary structures in 1951, several years before the first complete protein structure was elucidated Dada la rigidez del enlace peptídico, la conformación de las proteínas depende de la rotación de los enlaces N-Cα y Cα-C que unen dos enlaces peptídicos adyacentes y si pueden girar libremente. El ángulo de rotación del enlace N-Cα se denomina fi (f) y el del enlace Cα-C se denomina psi (y). La conformación de la cadena polipeptídica quedará perfectamente definida si se definen los ángulos fi y psi de cada uno de sus enlaces. Ángulos de conformación Φ fi Ψ psi La hélice alfa. Cuando 5 o más aminoácidos consecutivos de una proteína adoptan ángulo phi de ~ -60 y psi de ~ -50 aparece una hélice alfa. En las hélices alfa el carbonilo (-CO) del residuo i forma un puente de hidrógeno con el NH del residuo i+4 (salvo los 4 CO del extremo C de la hélice y los 4 NH del extremo N). α-hélice Es una estructura helicoidal dextrógira, es decir, que las vueltas de la hélice giran hacia la derecha. Adquieren esta conformación proteínas que poseen elevado número de aminoácidos con radicales grandes o hidrófilos, ya que las cargas interactúan con las moléculas de agua que la rodean. La estructura se estabiliza, gracias a la gran cantidad de puentes de hidrógeno que se establecen entre los aminoácidos de la espiral. Hélice 310 Φ = -49º Ψ = -26º Paso de rosca: 0.59 nm Traslación por residuo: 0.19 Residuos por vuelta: 3 Organización secundaria Radicales hacia afuera de la hélix Organización secundaria aa prolina impide formación de hélix Organización secundaria Existen dos factores (o mas bien aminoácidos que pertenezcan a la cadena polipeptídica) que pueden interrumpir la orientación helicoidal: (1) presencia de prolina la cual provoca una torsión de la cadena y, (2) la presencia de fuerzas electrostáticas localizadas de repulsión debido a un conjunto de grupos –R cargados positivamente (lisina y argina), o negativamente (ac- glutámico y aspártico). β-Laminar También se denomina hoja plegada o lámina plegada. Es una estructura en forma de zig-zag, forzada por la rigidez del enlace peptídico y la apolaridad de los radicales de los aminoácidos que componen la molécula. Se estabiliza creando puentes de Hidrógeno entre distintas zonas de la misma molécula, doblando su estructura. De este modo adquiere esa forma plegada. Representación de Ramachandran Φ fi Grados lámina beta hélice alfa Grados Ψ psi La hélice alfa. Cuando 5 o más aminoácidos consecutivos de una proteína adoptan ángulo phi de ~ -60 y psi de ~ -50 aparece una hélice alfa. La lámina beta. Cuando 2 o más aminoácidos consecutivos de una proteína adoptan ángulos phi de ~ -120 y psi de ~ +130, aparece una hebra beta Mioglobina C Proteína globular con alto contenido en α-hélice N Proteína globular con estructura β: Concanavalina A 1. non-linear 2. 3D dimensional 3. Formed and stabilized by hydrogen bonding, covalent (e.g. disulfide) bonding, hydrophobic packing toward core and hydrophilic exposure to solvent 4. A globular amino acid polymer folded and compacted is somewhat functional (catalytic) and energetically favorable Se distinguen dos tipos de estructura terciaria: 1. Proteínas con estructura terciaria de tipo fibroso en las que una de las dimensiones es mucho mayor que las otras dos. Son ejemplos el colágeno, la queratina del cabello o la fibroína de la seda), En este caso, los elementos de estructura secundaria (hélices α u hojas β) pueden mantener su ordenamiento sin recurrir a grandes modificaciones, tan sólo introduciendo ligeras torsiones longitudinales, como en las hebras de una cuerda. 2. Proteínas con estructura terciaria de tipo globular, más frecuentes, en las que no existe una dimensión que predomine sobre las demás, y su forma es aproximadamente esférica. En este tipo de estructuras se suceden regiones con estructuras al azar, hélice α hoja β, acodamientos y estructuras supersecundarias. Los enlaces covalentes pueden deberse a (1) la formación de un puente disulfuro entre dos cadenas laterales de Cys, o a (2) la formación de un enlace amida (-CO-NH-) entre las cadenas laterales de la Lys y un AA dicarboxílico (Glu o Asp). Los enlaces no covalentes pueden ser de cuatro tipos: (1) fuerzas electrostáticas entre cadenas laterales ionizadas, con cargas de signo opuesto, (2) puentes de hidrógeno, entre las cadenas laterales de AA polares (3) interacciones hidrofóbicas entre cadenas laterales apolares y (4) fuerzas de polaridad debidas a interacciones dipolo-dipolo Mioglobina C N Proteína globular con alto contenido en α-hélice Proteína globular con estructura β: Concanavalina A Existen regiones diferenciadas dentro de la estructura terciaria de las proteínas que actúan como unidades autónomas de plegamiento y/o desnaturalización de las proteínas. Estas regiones constituyen un nivel estructural intermedio entre las estructuras secundaria y terciaria reciben el nombre de dominios. Los dominios se pliegan por separado a medida que se sintetiza la cadena polipeptídica. Es la asociación de los distintos dominios la que origina la estructura terciaria. piruvato quinasa Fibrinógeno Proteína fibrosa con alto contenido en α-hélice ESTRUCTURA CUATERNARIA Cuando una proteína consta de más de una cadena polipeptídica, es decir, cuando se trata de una proteína oligomérica, decimos que tiene estructura cuaternaria. La estructura cuaternaria debe considerar: (1) el número y la naturaleza de las distintas subunidades o monómeros que integran el oligómero y (2) la forma en que se asocian en el espacio para dar lugar al oligómero. La figura corresponde a la hemoglobina. ESTRUCTURA CUATERNARIA En proteínas con estructura terciaria de tipo fibroso, la estructura cuaternaria resulta de la asociación de varias hebras para formar una fibra o soga. La miosina o la tropomiosina constan de dos hebras con estructura de hélice a enrolladas en una fibra levógira. La a-queratina del cabello y el fibrinógeno de la sangre presentan tres hebras en cada fibra levógira. El colágeno consta de tres hebras helicoidales levógiras que forman una fibra dextrógira. La fibroína de la seda presenta varias hebras con estructura de hoja b orientadas de forma antiparalela ESTRUCTURA CUATERNARIA Cuando varias proteínas con estructura terciaria de tipo globular se asocian para formar una estructura de tipo cuaternario, los monómeros pueden ser: Exactamente iguales, como en el caso de la fosfoglucoisomerasa o de la hexoquinasa. Muy parecidos, como en el caso de la lactato deshidrogenasa. Con estructura distinta pero con una misma función, como en el caso de la hemoglobina. Estructural y funcionalmente distintos, que una vez asociados forman una unidad funcional, como en el caso de la aspartato transcarbamilasa, un enzima alostérico con seis subunidades con actividad catalítica y seis con actividad reguladora. hemoglobina aspartato transcarbamilasa ESTRUCTURA CUATERNARIA Las fuerzas que mantienen unidas las distintas cadenas polipeptídicas son, en líneas generales, las mismas que estabilizan la estructura terciaria. Las más abundantes son las interacciones débiles (hidrofóbicas, polares, electrostáticas y puentes de hidrógeno), aunque en algunos casos, como en las inmunoglobulinas, la estructura cuaternaria se mantiene mediante puentes disulfuro. El ensamblaje de los monómeros se realiza de forma espontánea, lo que indica que el oligómero presenta un mínimo de energía libre con respecto a los monómeros Desnaturalización de una proteína Las proteínas se desnaturalizan cuando pierden su estructura tridimensional (conformación química) y así su característico plegamiento de su estructura. El proceso puede ser reversible o irreversible dependiendo de la fuerza del agente desnaturante Comportamiento de las proteínas en disolución Factores que influyen sobre la solubilidad de las proteínas • • • • Temperatura pH Fuerza iónica Constante dieléctrica del solvente La desnaturalización afecta a los distintos niveles Desnaturalización de la estructura cuaternaria: Las subunidades de proteína se separan o su posición espacial se altera. Desnaturalización de la estructura terciaria Implica la interrupción de: - Enlaces covalentes entre las cadenas laterales de los aminoácidos (como los puentes disulfuros entre las cisteínas). - Enlaces no covalentes dipolo-dipolo entre cadenas laterales polares de aas.; enlaces dipolo inducidos por fuerzas de Van Der Waals entre cadenas laterales no polares de aminoácidos Desnaturalización de la estructura secundaria Las proteínas pierden todos los patrones de repetición regulares como las alfa- hélices y adoptan formas aleatorias. La estructura primaria, la secuencia de aminoácidos ligados por enlaces peptídicos, no es interrumpida por la desnaturalización. • Factores desnaturalizantes • Los agentes que provocan la desnaturalización de una proteína se llaman agentes desnaturalizantes.. Se distinguen agentes físicos (calor) y químicos (detergentes, disolventes orgánicos, pH, fuerza iónica). • Como en algunos casos la desnaturalización es reversible, es posible precipitar proteínas de manera selectiva mediante cambios en: - la polaridad del disolvente, - la fuerza iónica, - el pH, - la temperatura. • Efecto de la polaridad del disolvente sobre la estructura de las proteínas • La polaridad del disolvente disminuye cuando se le añaden sustancias menos polares que el agua como el etanol o la acetona. Con ello disminuye el grado de hidratación de los grupos iónicos superficiales de la molécula proteica, provocando la agregación y precipitación. Los disolventes orgánicos interaccionan con el interior hidrofóbico de las proteínas y desorganizan la estructura terciaria, provocando su desnaturalización y precipitación. La acción de los detergentes es similar a la de los disolventes orgánicos. Fuerza iónicas sobre la estructura de las proteínas • Un aumento de la fuerza iónica del medio (por adición de sulfato amónico, urea o hidrocloruro de guanidinio, por ejemplo) también provoca una disminución en el grado de hidratación de los grupos iónicos superficiales de la proteína, ya que estos solutos (1) compiten por el agua y (2) rompen los puentes de hidrógeno o las interacciones electrostáticas, de forma que las moléculas proteicas se agregan y precipitan. En muchos casos, la precipitación provocada por el aumento de la fuerza iónica es reversible. Mediante una simple diálisis se puede eliminar el exceso de soluto y recuperar tanto la estructura como la función original. A veces es una disminución en la fuerza iónica la que provoca la precipitación. Así, las proteínas que se disuelven en medios salinos pueden desnaturalizarse al dializarlas frente a agua destilada, y se renaturalizan cuando se restaura la fuerza iónica original. Efecto del pH sobre la estructura de las proteínas • Los iones H+ y OH- del agua provocan efectos parecidos, pero además de afectar a la envoltura acuosa de las proteínas también afectan a la carga eléctrica de los grupos ácidos y básicos de las cadenas laterales de los aminoácidos. Esta alteración de la carga superficial de las proteínas elimina las interacciones electrostáticas que estabilizan la estructura terciaria y a menudo provoca su precipitación. La solubilidad de una proteína es mínima en su punto isoeléctrico, ya que su carga neta es cero y desaparece cualquier fuerza de repulsión electrostática que pudiera dificultar la formación de agregados. Efecto de la temperatura sobre la estructura de las proteínas • Cuando la temperatura es elevada aumenta la energía cinética de las moléculas con lo que se desorganiza la envoltura acuosa de las proteínas, y se desnaturalizan. Asimismo, un aumento de la temperatura destruye las interacciones débiles y desorganiza la estructura de la proteína, de forma que el interior hidrofóbico interacciona con el medio acuoso y se produce la agregación y precipitación de la proteína desnaturalizada. Los intercambiadores iónicos son matrices sólidas que contienen sitios activos (también llamados grupos ionogénicos) con carga electrostática (positiva o negativa). De esta forma, la muestra queda retenida sobre el soporte sólido por afinidad electrostática. Dependiendo de la relación carga/tamaño unos constituyentes de la mezcla serán retenidos con mayor fuerza sobre el soporte sólido que otros, lo que provocará su separación. Las sustancias que permanecen más tiempo libres en la fase móvil, avanzan más rápidamente con el fluir de la misma y las que quedan más unidas a la fase estacionaria o retenidas avanzan menos y por tanto tardarán más en salir o fluir Se trata de un tipo de electroforesis desnaturalizante en la que las muestras se desnaturalizan por calor en presencia de agentes desnaturalizantes (β-mercaptoetanol, que destruye los puentes disulfuro, SDS que desnaturaliza y recubre a la proteína con cargas netas negativas), y se separan como cadenas polipeptídicas aisladas. Inicialmente, las proteínas se separan según el punto isoeléctrico (pI) mediante isoelectroenfoque en geles de gradiente de pH inmovilizados en condiciones desnaturalizantes. El gradiente de pH de la primera dimensión se selecciona en cada caso para obtener la máxima resolución del grupo de proteínas de interés. En la segunda dimensión, las proteínas se separen en función de la masa molecular en un gel estándar SDS-PAGE ENZIMAS ESTABILIZADAS POR INMOVILIZACION MULTIPUNTUAL SOBRE SOPORTES GLIOXIL ENZIMA ACTIVIDAD Tripsina Quimotripsina Penicilina G acilasa de E. Coli Penicilina G acilasa de K. citrophila Ferredoxina NADP reductasa de Anabaena Lipasa de C. rugosa Glutamato racemasa Esterasa de B. stearothermofilus Termolisina de B. thermoproteolyticus >>> estable 75% 70% 70% 70% 60% 50% 70% 70% 100% ESTABILIZACION 10.000 60.000 8.000 7.000 1.000 150 1.000 1.000 100 animales, plantas y microorganismos como fuentes de enzimas Plantas fuentes potenciales de enzimas a escala industrial. Granos, remojar y germinar, se convierten en malta que contiene amilasas y proteasas usadas en la producción de cerveza y la destilación de bebidas alcohólicas. Siglo XIX, métodos simples para obtención de grandes cantidades de proteasas de la savia de plantas tropicales. animales, plantas y microorganismos como fuentes de enzimas Ablandadores de carne, digestión y limpieza de lentes de contacto. Papaina y quimiopapaina del árbol de la papaya. Ficina de higueras. Bromelaina del tallo de la planta de la piña. Enzimas Microbianas y sus aplicaciones Enzima Fuente Aplicación industrial Industria Invertasa Levadura Relleno de caramelos Confitería Glucosa Oxidasa Hongos Eliminación de glucosa y oxígeno, papeles para pruebas de la diabetes Alimentaria Farmacéutica Glucosa Isomerasa Bacterias Jarabe de cereales rico en glucosa Bebidas refrescantes Pectinasa Hongos Prensado, clarificación del vino Zumos de frutas Renina Hongos Coagulación de la leche Quesera Celulasa Bacterias Suavizante y abrillantador de tejidos; detergente Lavandería Lipasa Hongos Degradar la grasa Lechería, lavandería Lactasa Hongos Degradar la lactosa a glucosa y galactosa Lechería, alimentos DNA polimerasa Bacterias; Archea Replicación del DNA por PCR Investigación biológica y forense.
Anuncio
Documentos relacionados
Descargar
Anuncio
Añadir este documento a la recogida (s)
Puede agregar este documento a su colección de estudio (s)
Iniciar sesión Disponible sólo para usuarios autorizadosAñadir a este documento guardado
Puede agregar este documento a su lista guardada
Iniciar sesión Disponible sólo para usuarios autorizados