Redes de regulación
Anuncio

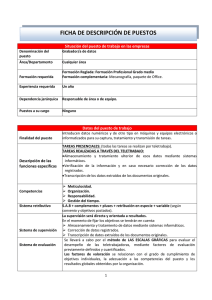
Redes de regulación Guillermo Carbajosa Resumen • Introducción a la transcripción y su regulación • Redes de regulación: Definición • Encontrando Sitios de Unión: • Conocidos (Pattern Matching) • Nuevos (Pattern Discovering) Resumen II • Propiedades de las redes de regulación • Propiedades generales • Motivos • Evolución • Dinámica Regulación de la transcripción • • Resultado de la interacción entre proteínas y DNA El conjunto de proteínas que se unan a su región promotora (directa o indirectamente) va a determinar la expresión de un gen: • • • En que tejidos En que fase del desarrollo Bajo que condiciones ambientales Transcripción en bacterias Transcripción en bacterias: Operones Transcripción en Eucariotas Otras fuentes de regulación • Elongación • Estabilidad del ARNm • Micro ARN • etc... Redes de regulación Redes de regulación II Definiendo redes de regulación Lee at al. 2002 Science 298:799-804 Computacionalmente • Sitios de unión al ADN conocidos: “Pattern Matching” • • “¿Qué genes regula un factor de transcripción?” Si sabemos la secuencia a la que se une un factor de transcripción, ¿podemos encontrar donde se une en un genoma? Sitios de unión al ADN desconocidos: “Pattern Discovering” • “¿Qué sitios de unión regulan un conjunto de genes?” Si sabemos que un conjunto de genes está co-regulado, ¿podemos encontrar los sitios de unión al ADN responsables de su regulación? Buscando sitios conocidos • Descripción de un sitio de unión • Secuencias consenso • Patrones • Matrices Secuencias consenso Sitios determinados Experimantalmente Secuencia Consenso ATCGTGCTATAGGTAAGT ATCGTGGTATACGTAAGT ATCGTGCTTTAGGTAAGA ATCCTGCTATTGCTAAGT ATCGTGCTATAGGTAAGT Secuencias consenso II ACGTA CGACGTAGATGACCTACGGATGCACGAACG CGACGTAGATGACCTACGGATGCACGAACG CGACGTAGATGACCTACGGATGCACGAACG CGACGTAGATGACCTACGGATGCACGAACG Patrones ATCGTGCTATAGGTAAGT ATCGAGGTATAGGTAAGT ATGGGGCTACAGGTAAGA ATGGCGCTATTGGTAAGT AT[CG][ACGT]G[CG]TA[CT][AT]GGTAAG[AT] ATSGNGSTAYWGGTAAGW W= R= K= S= Y= M= A A G C C A or or or or or or T G T G T C Matrices A C G T A A A A T T T T C C C C G G G C T T T T G G G G C G C C T T T T A A T A T T T T A A A T G C G G G G G C T T T T A A A A A A A A G G G G T T A T 4 0 0 0 0 0 0 4 0 4 0 0 0 1 3 0 0 0 0 4 0 0 4 0 0 3 1 0 0 0 0 4 3 0 0 1 0 0 0 4 3 0 0 1 0 1 3 0 0 1 3 0 0 0 0 4 4 0 0 0 4 0 0 0 0 0 4 0 1 0 0 3 A T G G C T C G A T T G G T A T G T 4+4+0+3+0+4+3+0+3+4+1+3+3+4+4+0+4+3=47 T A G C C A G T T T A T T A G C G T 0+0+0+1+0+0+3+4+1+4+3+0+0+0+0+0+4+3=23 Logos • Representación gráfica de un alineamiento • La altura de cada base es proporcional a su frecuencia • La altura de cada posición es proporcional a su conservación (medido en bits de información) ✴http://weblogo.berkeley.edu/logo.cgi Sitios especiales • • Promotores • • http://www.fruitfly.org/seq_tools/promoter.html • http://www.cbs.dtu.dk/services/Promoter/ http://www.softberry.com/berry.phtml? topic=bprom&group=programs&subgroup=gfindb Terminadores • http://www.softberry.com/berry.phtml? topic=findterm&group=programs&subgroup=gfindb • http://cbcb.umd.edu/software/transterm/ Bases de datos • Factores de transcripción, sitios de unión y sus matrices en eucariotas • Bases de datos y programas relacionados • PathoDB: una base de datos de formas mutadas relevantes de factores de transcripción y sitios de unión • S/Mart: información sobre las regiones unidas al anclaje y la matriz nuclear y las proteínas de la matriz nuclear • Transcompel: una base de datos de elementos regulatorios complejos que afectan a la transcripción en eucariotas ✴http://www.gene-regulation.com/ RegulonDB • Factores de transcripción, sus sitios de unión y sus operones en E. coli • Herramientas de visualización y análisis • Integrado en Ecocyc (www.ecocyc.org) ✴http://www.cifn.unam.mx/Computational_Genomics/regulondb/ Otras bases de datos • TRED: Humanos y ratón • CEPDB: C. elegans • SCPD: Levaduras • Promotores, sitios de unión, matrices, etc... ✴http://rulai.cshl.edu/software/index1.htm Encontrando sitios • Se necesita un conjunto de genes que se cree/sabe que está co-regulado • Se toman las regiones promotoras • Se buscan posibles regiones reguladoras Genes co-regulados • Perfiles transcripcionales (microarrays) • Otro tipo de asociaciones: • Rutas metabólicas • Misma clasificación funcional • Genes ortólogos (“huella filogenética”) Microarrays 1. Cada experimento compara 2 condiciones 2. Se necesitan varios experimentos 3. Para cada gen se obtiene un perfil 4. Los perfiles se comparan 5. Los genes se agrupan en función de sus perfiles Huella filogenética • • Se obtienen grupos de genes ortólogos • • Organismos distantes: no hay conservación Se asume que la regulación esta conservada, al igual que los sitios de unión Organismos cercanos: las secuencias no funcionales no han divergido aún Métodos • Motivos sobre-representados • • • • Computacionalmente caro Exhaustivo Ruidoso Basado en matrices • Rápido • • Consensus, MAME Gibbs sampling • • no exhaustivo, pueden obtenerse resultados distintos cada vez Filtrado por simetría Motivos Sobrerepresentados 1. Calcular la frecuencia de cada palabra de longitud n-bases en nuestras secuencias 2. Determinar aquellas que son significativamente más abundantes en nuestro grupo que en otros grupos Secuencia Esperado Observado AAAAA 2 3 AAAAC 3 2 AAAAG 5 3 ATGCA 13 17 ATGCC 15 143 ATGCG 17 14 2 0 ... ... TTTGC Gibbs Sampling 1. Selecciona al azar ACGTAGGTTC ACGTAGCAGT ACGGATGCGA ACGTAGCGTA 2. Eliminar la peor del alineamiento ACGTAGGTTC ACGTAGCAGT ACGGATGCGA ACGTAGCGTA 3. Sustituir por la mejor en la misma secuencia ACGTAGGATC ACGTAGCAGT ACGGATGCGA ACGTAGCGTA Repetir hasta que se estabiliza la calidad del alineamiento ACGTAGGATC ACGTAGCAGT ACGGATGCGA ACGTAGCGTA Agrupando sitios • Motivos que solapan • Motivos que se parecen AAGTCGGC AGTCGGCT GTCGGCTT -----------AAGTCGGCTT AGTCGGCA AGTCGGCT TGTCGGCT -----------AGTCGGCT Flujo de trabajo Pattern Discovery Pattern Matching Motive Clustering Propiedades de la red de regulación Propiedades Generales • Distribución exponencial • Distribución “Ley de potencias” • • • “Libre de escala” Robustez Caminos cortos (integración de señal) Evolución de las redes Duplicación y Evolución Duplicación de TFBS Red de Co-regulación • • • • gamma =~1 c=0.6 “libre de escala” “mundo pequeño” Simulando la evolución Simulando la evolución II • En ausencia de selección pueden aparecer redes de propiedades similares a las reales Motivos Motivos II • Algunas topologías (motivos) aparecen con mucha frecuencia Milo et al, 2002. Science 298:824 Motivos específicos Evolución de motivos No: En E coli no hay reguladores homólogos con el mismo motivo Evolución de motivos II Conant & Wagner,2003. Nat Genet. 34:264 Propiedades de los motivos Shen-Orr et al,2002. Nat Genet. 31:64 Circuitos Tyler et al. 2003 Current Opinion in Cell Biology 15:221 Las redes son dinámicas Luscombe et al., 2004 Nature 431:308 Las redes son dinámicas II Luscombe et al., 2004 Nature 431:308 ¡Gracias por vuestra atención! (y a Ildefonso Cases por la presentación) ¿Preguntas? Prácticas: • http://rsat.scmbb.ulb.ac.be/rsat/ • Misc>Tutorials • • 1. Sequence retrieval 3. Pattern matching • • 3.2. Patser 4. Pattern discovering • • 4.1.1. Oligo analysis 4.2.1. Gibbs Motif Sampler