2.3.3.3.2.- Análisis del ADN por PCR
Anuncio
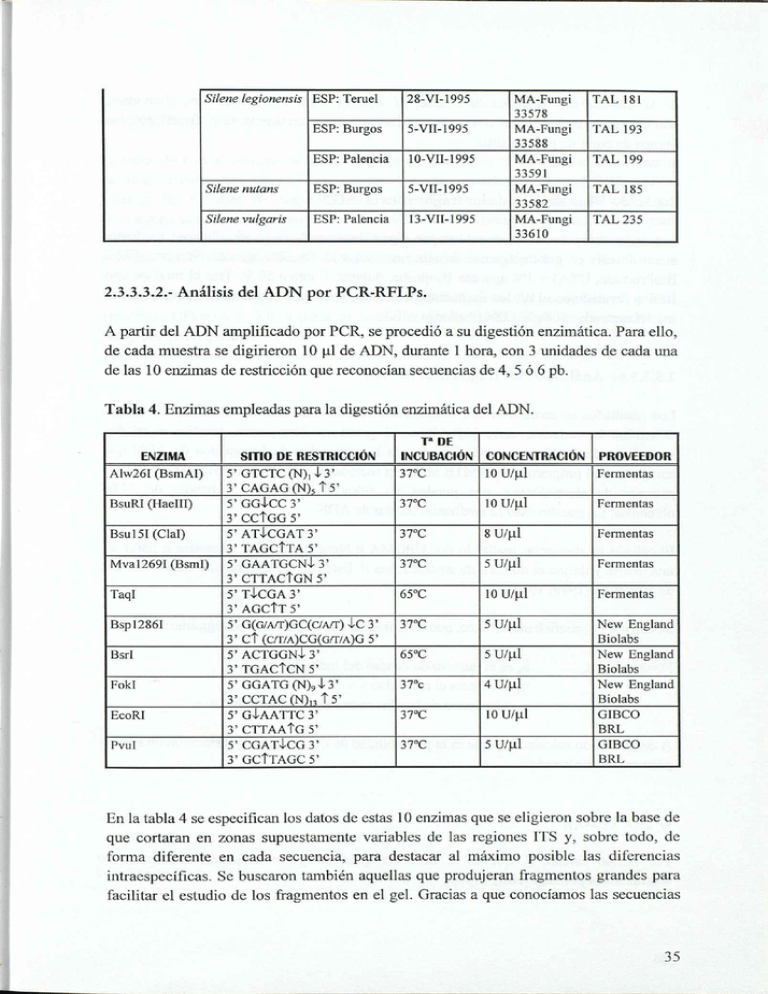
Silène legionensis ESP: Teruel 28-VI-1995 ESP: Burgos 5-VII-1995 ESP: Palencia 10-VII-1995 Silène nutans ESP: Burgos 5-VII-1995 Silène vulgaris ESP: Palencia 13-VII-1995 MA-Fungi 33578 MA-Fungi 33588 MA-Fungi 33591 MA-Fungi 33582 MA-Fungi 33610 TAL 181 TAL 193 TAL 199 TAL 185 TAL 235 2.3.3.3.2.- Análisis del A D N por P C R - R F L P s . A partir del ADN amplificado por PCR, se procedió a su digestión enzimàtica. Para elio, de cada muestra se digirieron 10 ul de ADN, durante 1 hora, con 3 unidades de cada una de las 10 enzimas de restricción que reconocían secuencias de 4, 5 ó 6 pb. Tabla 4. Enzimas empleadas para la digestión enzimática del ADN. SITIO DE RESTRICCIÓN ENZIMA Alw26I (BsmAI) BsuRI (HaelII) Bsul5I(ClaI) Mval269I (Bsml) Taql Bspl286I 5' GTCTC ( N ) , l 3 ' 3'CAGAG ( N ) , f 5' 5' G G l C C 3 ' 3' CCÎGG 5' 5' ATÍCGAT 3' 3' TAGCÎTA 5' 5' GAATGCN! 3' 3' CTTACÎGN 5' 5' TlCGA 3' 3' A G C Î T 5 ' 5' G(G/A/T)GC(C/A/T) i C 3' T* DE INCUBACIÓN CONCENTRACIÓN PROVEEDOR 37°C 10 u / u l Fermentas 37°C 10 U/ul Fermentas 37°C 8 U/ul Fermentas 37°C 5 U/ul Fermentas 65°C 10 u / u l Fermentas 37°C 5 U/ul 65°C 5 U/ul 37°c 4 U/ul 37°C 10 u / u l 37°C 5 U/ul New England Biolabs New England Biolabs New England Biolabs G1BCO BRL GIBCO BRL 3' C Î (CAT/A)CG(G/T/A)G 5' Bsrl Fokl EcoRI Pvul 5' 3' 5' 3' 5' 3' 5' 3' ACTGGNi 3' TGACtCN 5' GGATG (N) 4-3' CCTAC ( N ) , T 5 ' GIAATTC3' CTTAAÎG 5' CGATÍCG 3' GCÎTAGC 5' 9 3 En la tabla 4 se especifican los datos de estas 10 enzimas que se eligieron sobre la base de que cortaran en zonas supuestamente variables de las regiones ITS y, sobre todo, de forma diferente en cada secuencia, para destacar al máximo posible las diferencias intraespecíficas. Se buscaron también aquellas que produjeran fragmentos grandes para facilitar el estudio de los fragmentos en el gel. Gracias a que conocíamos las secuencias