Extracción y purificación de los ácidos nucleicos
Anuncio
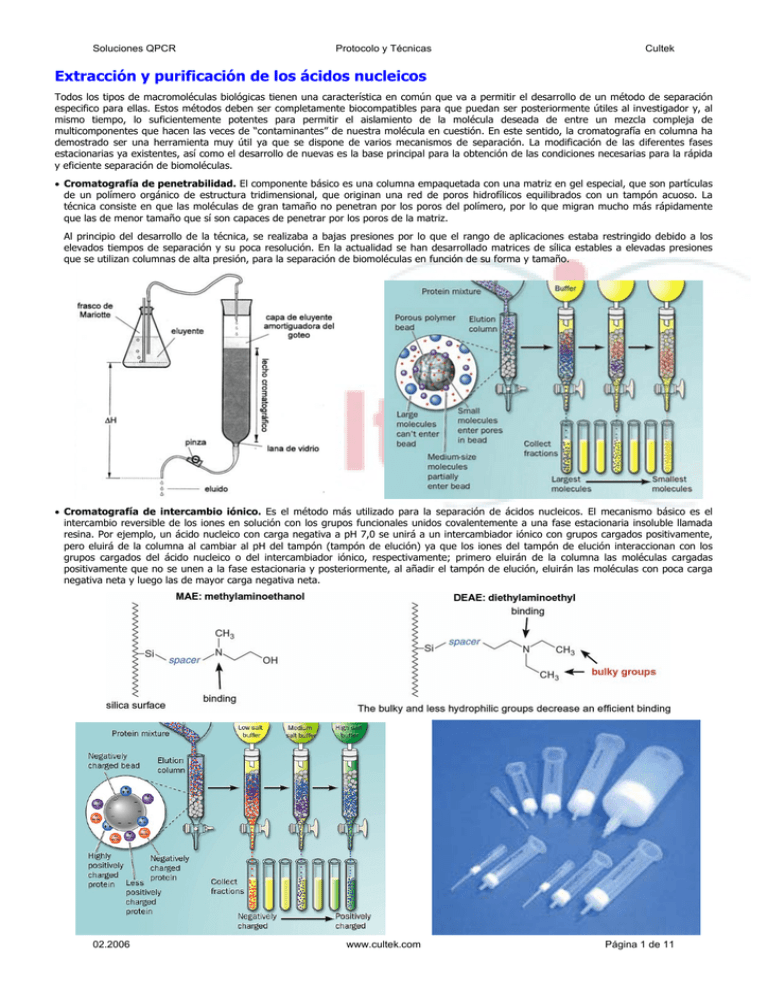
Soluciones QPCR Protocolo y Técnicas Cultek Extracción y purificación de los ácidos nucleicos Todos los tipos de macromoléculas biológicas tienen una característica en común que va a permitir el desarrollo de un método de separación especifico para ellas. Estos métodos deben ser completamente biocompatibles para que puedan ser posteriormente útiles al investigador y, al mismo tiempo, lo suficientemente potentes para permitir el aislamiento de la molécula deseada de entre un mezcla compleja de multicomponentes que hacen las veces de “contaminantes” de nuestra molécula en cuestión. En este sentido, la cromatografía en columna ha demostrado ser una herramienta muy útil ya que se dispone de varios mecanismos de separación. La modificación de las diferentes fases estacionarias ya existentes, así como el desarrollo de nuevas es la base principal para la obtención de las condiciones necesarias para la rápida y eficiente separación de biomoléculas. • Cromatografía de penetrabilidad. El componente básico es una columna empaquetada con una matriz en gel especial, que son partículas de un polímero orgánico de estructura tridimensional, que originan una red de poros hidrofílicos equilibrados con un tampón acuoso. La técnica consiste en que las moléculas de gran tamaño no penetran por los poros del polímero, por lo que migran mucho más rápidamente que las de menor tamaño que sí son capaces de penetrar por los poros de la matriz. Al principio del desarrollo de la técnica, se realizaba a bajas presiones por lo que el rango de aplicaciones estaba restringido debido a los elevados tiempos de separación y su poca resolución. En la actualidad se han desarrollado matrices de sílica estables a elevadas presiones que se utilizan columnas de alta presión, para la separación de biomoléculas en función de su forma y tamaño. • Cromatografía de intercambio iónico. Es el método más utilizado para la separación de ácidos nucleicos. El mecanismo básico es el intercambio reversible de los iones en solución con los grupos funcionales unidos covalentemente a una fase estacionaria insoluble llamada resina. Por ejemplo, un ácido nucleico con carga negativa a pH 7,0 se unirá a un intercambiador iónico con grupos cargados positivamente, pero eluirá de la columna al cambiar al pH del tampón (tampón de elución) ya que los iones del tampón de elución interaccionan con los grupos cargados del ácido nucleico o del intercambiador iónico, respectivamente; primero eluirán de la columna las moléculas cargadas positivamente que no se unen a la fase estacionaria y posteriormente, al añadir el tampón de elución, eluirán las moléculas con poca carga negativa neta y luego las de mayor carga negativa neta. 02.2006 www.cultek.com Página 1 de 11 Soluciones QPCR Protocolo y Técnicas Cultek Una modificación de este método es la extracción en fase sólida, en la que se utiliza una matriz intercambiadora de aniones empaquetada en columnas de polipropileno. La unión se produce bajo condiciones de baja salinidad y durante los pasos de lavado y elución se va incrementando la concentración de sales en los tampones correspondientes. Las impurezas se eliminan debido a su diferente capacidad de unión y se obtienen una rápida y eficiente purificación de los ácidos nucleicos (DNA y/o RNA). • Cromatografía de adsorción. Se basa en la adsorción y desorción de los ácidos nucleicos en presencia de sales caotrópicas. Bajo condiciones nativas, los ácidos nucleicos están recubiertos de una capa hidratante de moléculas de agua que mantienen la solubilidad del DNA en soluciones acuosas. Con la adición de iones caotrópicos a los ácidos nucleicos, se destruye esta ordenada estructura de moléculas de agua de la capa hidratante, por lo que las sales caotrópicas crean un entorno hidrofóbico alrededor del DNA. Bajo estas condiciones hidrofóbicas, los ácidos nucleicos se unen perfectamente a la membrana de sílica de las columnas, mientras que las proteínas, los metabolitos y otros contaminantes no se unen y, por lo tanto, son eliminados de la muestra durante los pasos de lavado. Posteriormente, los ácidos nucleicos se eluyen de la membrana de sílica mediante tampones de elución con baja concentración de sales (ligeramente alcalinos) o simplemente agua, ya que permiten recuperar la capa hidratante de los ácidos nucleicos, liberándolos así de la membrana. Con esta tecnología, no se produce ningún efecto sobre las moléculas de los ácidos nucleicos ya que la unión intramolecular de los mismos no es de naturaleza hidrofóbica. Con este rápido proceso de purificación mediante la tecnología de la adsorción-desorción sobre membranas de sílica, se obtiene ácidos nucleicos altamente purificados y listos para usar en procesos posteriores como PCR, RT-PCR, QPCR, secuenciacion, blotting, clonaje, etc. • Ultrafiltración. En esta tecnología, la muestra se carga directamente sobre la membrana de ultrafiltración y mediante vacío o centrifugación, se eliminan todos los contaminantes mientras que la muestra con los ácidos nucleicos permanecen retenidos en la superficie de la membrana. Después de un paso de lavado opcional, se recuperan los ácidos nucleicos añadiendo agua o un tampón adecuado. • Bolas magnéticas. Con esta tecnología, los ácidos nucleicos se unen selectivamente a bolas paramagnéticas en presencia de sales caotrópicas mientras que el resto de los contaminantes son eliminados de la muestra. Los ácidos nucleicos purificados se eluyen de la solución con las bolas paramagnéticas bajo condiciones de baja salinidad y están listos para ser usados en posteriores aplicaciones. Pasos generales de la extracción. Todos los métodos de preparación de las muestras pueden dividirse en una serie de pasos generales, de tal forma que la necesidad de cada paso dependen del microorganismo y de la muestra: Liberación de los ácidos nucleicos de las bacterias, hongos o virus. Puede ser un paso muy sencillo en el caso de los virus y algunas bacterias (Mycoplasma spp.) pero muy difícil en el caso de otras bacterias (Mycobacterium tuberculosis) y hongos. Hay una gran variedad de métodos para la liberación de los ácidos nucleicos de diferentes microorganismos, entre los que se encuentran el hervir en agua destilada o tampón de PCR, el uso de detergentes con o sin calor, hidróxido sódico con calor, ciclos de congelacióndescongelación, SDS-proteinasa K, ácido perclórico, enzimas (lisozima), sonicación, etc. aunque la utilización de enzimas no es muy recomendable ya que la propia muestra puede contener inhibidores de las mismas. Protección y estabilización de los ácidos nucleicos frente a la degradación. El RNA es mucho más difícil de estabilizar que el DNA. 02.2006 www.cultek.com Página 2 de 11 Soluciones QPCR Protocolo y Técnicas Cultek Eliminación de inhibidores de la amplificación. Para algunas muestras (esputo, sangre) pueden ser necesarios más pasos que para otras (orina, LCR). Concentración de la muestra y la diana en un pequeño volumen. Algunas muestras (esputo para M.tuberculosis y Legionella pneumophila y sangre para sepsis) requieren una mayor grado de concentración que otras (orina para Chlamydia o Gonococcus) para alcanzar la sensibilidad deseada Colocación de la diana en un medio acuoso compatible con el método de amplificación Los laboratorios generalmente utilizan diferentes métodos para aislar el DNA de diferentes bacterias (ya sean gramnegativas o grampositivas); esto es un problema ya que requiere la preparación y almacenamiento de una gran cantidad de reactivos y tampones y la utilización de diferentes protocolos de extracción. A continuación se muestra un protocolo universal de extracción de DNA genómico con el que se pueden extraer entre 100 y 400µg de DNA; consta de un módulo central (útil para la extracción de DNA de bacterias gramnegativas, incluyendo las encapsuladas y las productoras de polisacáridos) y un módulo opcional (permite la extracción de bacterias grampositivas): • Módulo central 8 Cultivar los microorganismos en el medio más adecuado o en 5-10mL de medio de cultivo. Si se utilizan placas de agar, recoger las colonias con un asa estéril y suspenderlas en 5mL de buffer de suspensión (3,3mL de NaCl 3M; 1,0mL de Tris 1M pH 8,0; 2,0mL de EDTA 0,5M pH 8,0; 58,5mL de sacarosa y ajustar el volumen a 100mL). Si se utiliza medio de crecimiento, centrifugar las colonias durante 5min a 3600×g a 4°C, eliminar el sobrenadante y resuspender el pellet en 5mL de buffer de suspensión. 8 Centrifugar la suspensión durante 5min a 3600×g a 4°C. Eliminar el sobrenadante. Resuspender el pellet en 3,78mL de buffer de suspensión. 8 Añadir 200µL de SDS al 20% y mezclar completamente mediante inversión. Incubar la mezcla a 37°C durante 30min. Añadir 20µL de proteinasa K (20mg/mL), mezclar y continuar incubando a 37°C durante otro 30min. 8 Añadir 720µL de NaCl (5M), mezclar completamente, añadir 600µL de CTAB (bromuro de hexadeciltrimetilamonio) y mezclar por inversión. Incubar a 65°C durante 10min y a 37°C durante 5min. 8 Añadir un volumen igual de PCI (25:24:1 fenol-cloroformo-alcohol isoamílico). Centrifugar a 1500×g durante 15min. Traspasar la fase acuosa superior a un nuevo tubo. Añadir un volumen igual de CI (24:1 cloroformo-alcohol isoamílico) y centrifugar durante 15min a 1500×g. 8 Traspasar la fase acuosa superior a un nuevo tubo. Añadir 2,5 volúmenes de etanol frío al 95%, mezclar por inversión y mantener a -70°C durante 30min. 8 Centrifugar durante 30min a 14500×g. Eliminar el sobrenadante y secar el pellet mediante vacío. Resuspender el pellet en 400µL de buffer TE (Tris HCl 10mM; EDTA 1mM pH 8,0) 8 Añadir 1µL de una dilución 1:3 en agua estéril de RNasa T1. Incubar la mezcla a 37°C durante 1h. 8 Centrifugar a 19900×g durante 2min. 8 Traspasar la fase acuosa superior a un nuevo tubo. Añadir 0,1 volúmenes de acetato sódico 3M y mezclar bien. Precipitar el DNA con 2,5 volúmenes de etanol frío al 95%. Mantener a -70°C durante 20-30min. 8 Centrifugar durante 8min a 19900×g. Eliminar el sobrenadante. Lavar el pellet con 500µL de etanol al 70% y volver a centrifugar durante 8min. 8 Descartar el sobrenadante, secar el DNA a vacío (15-30min) y disolver el pellet de DNA en 100-400µL de agua estéril. 8 Medir la concentración de DNA en un espectrofotómetro: diluir un alícuota 1:20 (15µL en 285µL de H2O), y leer a λ = 260nm. La A260 es igual a la concentración de DNA en gr/L. 02.2006 www.cultek.com Página 3 de 11 Soluciones QPCR Protocolo y Técnicas Cultek Esquema del módulo central del protocolo universal de extracción de DNA genómico. • Módulo opcional para bacterias grampositivas 8 Cultivar las bacterias en el medio más adecuado o en 5-10mL de medio de crecimiento. Si se utilizan placas de agar, recoger las colonias con un asa estéril y suspenderlas en 5mL de buffer de suspensión. Centrifugar las colonias durante 5min a 3600×g a 4°C. Eliminar el sobrenadante y resuspender en 1mL de solución de prelisis (0,25mL de Tris 2M pH 7,0; 3,1mL de lipasa pancreática; 0,3mL de taurocolato sódico; 0,5mL de CaCl2 0,1M; 5,85mL de sacarosa y 0,05gr de lisozima) e incubar a 37°C durante 45min. 8 Si se utiliza medio de crecimiento, centrifugar las colonias durante 5min a 3600×g a 4°C, eliminar el sobrenadante y resuspender el pellet en 5mL de buffer de suspensión. Centrifugar durante 5min a 3600×g a 4°C y eliminar el sobrenadante. Resuspender en 1mL de solución de prelisis e incubar a 37°C durante 45min. 02.2006 www.cultek.com Página 4 de 11 Soluciones QPCR Protocolo y Técnicas Cultek PCR en tiempo real. La PCR en Tiempo Real es, básicamente, una PCR convencional en la que los equipos de amplificación (termocicladores) llevan incorporados un sistema de detección de fluorescencia (fluorímetro), basándose la tecnología en la utilización de unas moléculas específicas denominadas fluoróforos y quenchers. Ambos factores, nos va a permitir monitorizar, en tiempo real, lo que esta ocurriendo dentro de cada tubo en cada ciclo de amplificación y va a sustituir a los pasos de amplificación, electroforesis y análisis de imagen de una PCR tradicional. Los factores que afectan a la PCR cuantitativa son, entre otros: optimización del protocolo de reacción para maximizar su eficiencia. Hay que tener en cuenta que una pequeña diferencia en la eficiencia de reacción por ciclo, puede originar una substancial diferencia en la cantidad final de producto; por ejemplo, si una reacción es tan solo un 90% tan eficiente como otra, la relación de producto final entre ambas reacciones después de 40 ciclos será alrededor de 65 a 1 evitar un número excesivo de ciclos de amplificación un adecuado procesamiento de las muestras que asegure la eliminación de los inhibidores que pueden disminuir la eficiencia de amplificación El parámetro fundamental en una PCR en Tiempo Real y en función del cual se van a realizar todos los cálculos analíticos y obtención de resultados, es el denominado Ciclo Umbral (Threshold Cycle o Ct), que se define como el ciclo a partir del cual la fluorescencia es estadísticamente significativa por encima del ruido de fondo (background). Las principales propiedades de este parámetro Ct son: Ct Ct Ct Ct Ct Ct Ct Ct separa los datos del ruido de fondo determina el ciclo inicial de amplificación es inversamente proporcional al número de copias inicial del template se suele establecer como 10 veces el ruido de fondo, aunque se puede modificar manualmente Una de las principales ventajas de la PCR en Tiempo Real frente a la PCR convencional (aparate de su mayor sensibilidad) es que a la hora de cuantificar, en la PCR convencional se hace en función del producto final y debido a que la propia PCR es una reacción enzimática, las variaciones que se pueden observar son muy significativas; sin embargo, en la PCR en Tiempo Real, dicha cuantificación se realiza en función del valor de Ct, que al ser al principio de la amplificación, no se ve afectado por todas esas variaciones, con lo que los resultados son completamente reproducibles y mucho más precisos. En la siguiente figura se muestran los resultados de 96 replicados de una muestra puestos en una misma placa de PCR y en ella se pueden observar las enormes variaciones que se pueden obtener en los resultados del producto final frente a nula variación observable en el valor de Ct en todos los replicados. 02.2006 www.cultek.com Página 5 de 11 Soluciones QPCR Protocolo y Técnicas Cultek Ct Como ya se ha mencionado anteriormente, la tecnología de la PCR en Tiempo Real se basa en la utilización conjunta de termocicladores con un fluorímetro incorporado y fluoróforos. Mecanismo de acción de un fluoróforo. Estos fluoróforos determinan dos tipo de mecanismos de detección: • No específicos. Detectan todos los DNA de doble cadena (dsDNA) producidos durante la reacción de amplificación (ya sea producto específico, producto inespecífico o dímeros de primers). Es el método estándar y consiste en añadir un agente intercalante de la doble cadena o un fluoróforo que emite fluorescencia cuando se une a ésta. El más utilizado es el SYBR Green I que se excita a 497nm y emite a 520nm. Para discriminar si las muestras con curva de amplificación positivas corresponden a productos específicos o a dímeros de primers o productos inespecíficos, se suele realizar una curva de desnaturalización (“melting curve”) al final de la reacción: la reacción se calienta lentamente desde 50°C hasta 95°C monitorizando continuamente la fluorescencia; la temperatura a la cual el DNA se desnaturaliza se observa como una drástica caída de la fluorescencia debido a la disociación del SYBR Green I. Los productos de PCR de diferente longitud y diferentes secuencias se desnaturalizan a diferentes temperaturas, observándose diferentes picos cuando se representa la derivada de la ⎛ dF ⎞ fluorescencia con respecto a la temperatura ⎜ − ⎟ frente a la temperatura. ⎝ dT ⎠ 02.2006 www.cultek.com Página 6 de 11 Soluciones QPCR Protocolo y Técnicas Cultek Una posibilidad para evitar este problema es desnaturalizar los productos de PCR incrementando la temperatura de la muestra. A la temperatura de melting (Tm) del producto, se puede observar una drástica reducción de la intensidad de fluorescencia, ya que los productos de PCR inespecíficos (cortos) se desnaturalizan a una temperatura muy inferior que los específicos (largos). Esto permite corregir las curvas de desnaturalización de la contribución de los productos de PCR inespecíficos, incrementando la sensibilidad de la técnica a una copia por reacción. En estas condiciones el SYBR Green I se puede utilizar para cuantificar e incluso para la detección de mutaciones puntuales (SNPs), ya que incluso los amplicones que difieren en un solo nucleótido, pueden desnaturalizarse a diferentes temperaturas y pueden detectarse por sus picos de desnaturalización (es posible distinguir muestras homocigotas -un solo pico- de muestras heterocigotas -dos picos-). • Específicos. Estos sistemas de detección son capaces de distinguir entre la secuencia de interés y los dímeros de primers o las amplificaciones inespecíficas. Todos ellos se basan en la utilización de quenchers y sondas marcadas con un amplio rango de fluoróforos con diferentes espectros de excitación y emisión. Un fluoróforo es una molécula que absorbe energía y pasa a un estado excitado; posteriormente, al volver al estado inicial emite el exceso de energía en forma de fluorescencia. Los quenchers son moléculas que aceptan la energía de un fluoróforo y la disipan en forma de calor o fluorescencia: Mecanismo de actuación de un quencher. Las dos formas de disipación de la energía de los quenchers definen los dos posibles mecanismos de actuación: 8 Quenching colisional: se produce cuando el fluoróforo está en contacto o muy próximo al quencher de tal forma que se produce la transferencia de energía al quencher, el cual la disipa en forma de calor. 8 Quenching FRET: se produce cuando el fluoróforo transfiere la energía al quencher (que en este caso puede ser otro fluoróforo) y esta energía es disipada en forma de fluorescencia a una mayor longitud de onda. La eficiencia del proceso depende de la distancia entre el fluoróforo y el quencher. Los fluoróforos y quenchers más utilizados se detallan en la siguiente tabla II, así como las longitudes de onda de excitación y emisión. Fluoróforos y quenchers más utilizados. Nombre Excitación (nm) Emisión (nm) Fluoróforos FAM 492 515 TET 521 536 JOE 527 548 HEX 535 556 TAMRA 555 580 ROX 575 602 Cy3 552 565 Cy3.5 581 596 Cy5 651 674 Cy5.5 675 694 Cy7 743 767 R6G 518 543 Texas Red 583 603 VIC 528 546 02.2006 Nombre Methyl Red ElleQuencher Dabcyl Dabsyl TAMRA www.cultek.com Excitación (nm) Quenchers 410 650,600 453 466 555 Emisión (nm) No No No No disponible disponible disponible disponible 580 Página 7 de 11 Soluciones QPCR Protocolo y Técnicas Cultek A continuación se describen algunas de las sondas más ampliamente utilizadas: 8 Sondas TaqMan. Son las sondas más utilizadas y suelen ser las sondas de elección (después del SYBR Green) de los investigadores que empieza a trabajar con la Q-PCR. Fueron inicialmente desarrolladas por Roche y Applied Biosystems y en ellas se une un fluoróforo al extremo 5’ de la sonda (tradicionalmente FAM) y un quencher al 3’ (tradicionalmente TAMRA pero también se utiliza Dabcyl, Methyl Red o ElleQuencher). Cuando el fluoróforo es excitado, transfiere su energía por quenching FRET al quencher. Cuando la Taq empieza amplificar a partir del primer unido al DNA diana, desplaza el extremo 5’ de la sonda que es degradado por la actividad exonucleasa 5’ Ö 3’ de la Taq. Este proceso libera el fluoróforo al medio separándolo del quencher, lo que ocasiona un aumento irreversible de la fluorescencia detectada. 8 Sondas Molecular Beacon. Difieren de las sondas anteriores tanto en su estructura como en su mecanismo de acción. Consisten en una estructura de horquilla en la cual el bucle es DNA monocatenario (ssDNA) complementario del amplicón; el brazo de la horquilla tiene una longitud aproximada de 6 bases, esta formada por C y G y es la encargada de mantener la estructura de la horquilla. El fluoróforo esta unido a uno de los extremos del brazo y el quencher (normalmente Dabcyl o Methyl Red) al otro extremo; el brazo de la horquilla mantiene al fluoróforo y al quencher próximos, de tal forma que el quenching que se observa es colisional. Durante la PCR, la sonda se une a la secuencia específica del DNA diana ya que el heterodúplex sonda-diana es termodinámicamente más estable que la estructura de horquilla de la sonda. Una vez que la sonda se ha unido a su diana, la horquilla se abre, separándose el fluoróforo y el quencher, con lo que se produce un incremento de la fluorescencia. 02.2006 www.cultek.com Página 8 de 11 Soluciones QPCR Protocolo y Técnicas Cultek Este incremento de fluorescencia que se observa es completamente reversible (al contrario que con las sondas TaqMan), ya que a elevadas temperaturas, la sonda se disocia de su diana y vuelve a la estructura de horquilla original. El brazo de la estructura añade especificidad a este tipo de sonda ya que el híbrido formado entre la sonda y la diana tiene que ser más estable que la del propio brazo para que se mantenga la unión sonda-diana. Todas aquellas dianas que no sean totalmente complementarias formarán el dúplex con la sonda pero se disociarán a una temperatura bastante menor que las que apareen perfectamente. El estudio de las curvas de disociación permite detectar hasta mutaciones puntuales. La principal desventaja de los Molecular Beacons es que su diseño es bastante complicado ya que, además de hacer un estudio de secuencia, como ocurre en el caso de las sondas TaqMan, para que la secuencia del bucle sea complementaria del amplicón, hay que hacer un estudio termodinámico para que la energía de unión bucle-amplicón sea termodinámicamente más estable que la del brazo del propio Molecular Beacon, ya que si no es así, aunque la región complementaria del bucle se encuentre presente en el amplicón, éste nunca se abrirá para unirse al amplicón. Además, aunque se haga un diseño perfecto, la señal suele ser bastante pobre, ya que muchas veces ocurre que cuando el Molecular Beacon se abre e hibrida con el amplicón, el fluoróforo y el quencher quedan lo suficientemente próximos para que el quencher siga capturando la fluorescencia del fluoróforo. 8 Sondas Scorpions. Difieren de las sondas anteriores en que su mecanismo de acción es intramolecular. La horquilla esta unida al extremo 5’ de un primer especifico por medio de un “bloqueante de la PCR”. Después de la extensión del primer durante la amplificación, la secuencia especifica de la sonda se une a la región complementaria dentro de la misma hebra de DNA. Esta hibridación abre la estructura de horquilla de tal forma que el quencher ya no queda próximo al fluoróforo y se observa un incremento en la fluorescencia. El “bloqueante de la PCR” evita que continúe la amplificación a lo largo de la sonda, lo que provocaría que se abriera la estructura de horquilla de la sonda (con el correspondiente incremento de fluorescencia) en ausencia de secuencias diana especificas. La unión intramolecular de la sonda es cinéticamente favorable y altamente efectiva. Además, no es necesaria la ruptura enzimática de la sonda, por lo que se reduce el tiempo necesario para obtener señal. No obstante, como el quencher y el fluoróforo permanecen en la misma hebra de DNA y relativamente próximos, a veces se puede producir el quenching incluso cuando la horquilla esté abierta. 02.2006 www.cultek.com Página 9 de 11 Soluciones QPCR Protocolo y Técnicas Cultek 8 Sondas Amplifluor. Este sistema consta de 2 pasos realizados en la misma reacción de PCR: uno de los primers específicos lleva una secuencia universal (llamada “secuencia Z”) en el extremo 5’, por lo que durante la amplificación la secuencia Z se añade al amplicón. A continuación se utiliza un segundo primer (llamado “Uniprimer”) complementario de la secuencia Z y que incorpora una horquilla en su extremo 5’ con un fluoróforo y un quencher. Esta horquilla se incorpora al producto de PCR cuando se produce la extensión del “Uniprimer”, de tal forma que cuando este nuevo producto de PCR se convierte en molde durante el siguiente ciclo de amplificación, la Taq abre la horquilla, lo cual separa el fluoróforo y el quencher que se encontraban próximos en la estructura inicial, observándose un incremento de la fluorescencia fácilmente detectable. 8 Sondas de hibridación. Han sido desarrolladas por Roche para su utilización con su equipo capilar (LightCycler). Se diseñan dos sondas para que se unan adyacentemente en el amplicón; uno de los primers incorpora un fluoróforo (FAM) en el extremo 3’ y el otro primer otro fluoróforo (LC Red 640 ó 705, ROX ó Cy5) en el 5’. Durante la reacción de PCR, los dos primers se unen a su secuencia especifica del amplicón; a continuación el sistema excita el FAM, el cual transmite su energía al otro fluoróforo mediante quenching FRET, observándose un incremento de la señal de fluorescencia proporcional al incremento del número de amplicones. Esta tecnología permite no solo realizar PCR cuantitativas sino la detección de mutaciones puntuales, para lo cual se híbrida uno de los primers sobre el punto de polimorfismo. El mal apareamiento del primer provocado por la mutación ocasiona que la sonda se disocie a una temperatura diferente que la sonda completamente complementaria del amplicón; esta disociación ocasiona una disminución de la fluorescencia ya que el quenching FRET no se produce. 8 Sondas Eclipse. Son pequeñas sondas lineales que incorporan la tecnología del “Minor Groove Binder” (MGB) y el quencher en el extremo 5’ y el fluoróforo en el 3’. Se piensa que el MGB protege a la sonda de la actividad exonucleasa de la Taq. En la forma “libre” de la sonda, el quencher y el fluoróforo quedan próximos por lo que no se observa emisión de fluorescencia. Cuando la sonda se une a su diana, se linealiza, por lo que desaparece el quenching y se observa un aumento de la fluorescencia. 02.2006 www.cultek.com Página 10 de 11 Soluciones QPCR 02.2006 Protocolo y Técnicas www.cultek.com Cultek Página 11 de 11