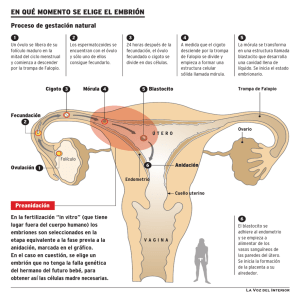
Genética poblacional y filogeografía de Caretta caretta
Anuncio
TESIS DE GRADO CIENTÍFICO DOCTOR EN CIENCIAS BIOLÓGICAS “Genética poblacional y filogeografía de Caretta caretta (Linnaeus, 1758) y Chelonia mydas (Linnaeus, 1758): Una perspectiva regional con énfasis en el suroeste de Cuba” Autor: Lic. Ariel Ruiz Urquiola Tutores: Dra. Georgina Espinosa López1 Dr. Federico Alberto Abreu Grobois2 1Facultad de Biología, Universidad de La Habana Mazatlán, Instituto de Ciencias del Mar y Limnología, Universidad Nacional Autónoma de México 2Unidad Centro de Investigaciones Marinas Universidad de La Habana La Habana, 2008 A la memoria de: Osniel Marínez Pelikián Lutgarda C.. González Géigel Pedro Blanco Blanco Armelio Frometa Fals A las causas de los imposibles La diversidad genética significa para las poblaciones silvestres Lo mismo que la Libertad para el Homo sapiens Un H. sapiens sin pensamiento es víctima de las circunstancias y con pensamiento lo es de sí mismo, es más libre AGRADECIMIENTOS Si fuera católico tendría que creer que este acto de defensa es un milagro y en consecuencia, tendría que agradecer además de a la santísima Trinidad, a las once mil vírgenes y no sé cuántos santos. Al creer en la naturaleza como máxima expresión de la divinidad entonces, creo que este acto de defensa es el fruto de energías impartidas desde muchos cuerpos y posiciones geográficas disímiles, y entonces tengo que agradecer al menos a los animados que de una forma u otra han contribuido a que este tercer tema fraguara en medio de tempestades, cuyo meollo no ha sido otra cosa que el compromiso más que con la verdad científica per se, con la clarificación de una problemática ambiental que ha sido en gran medida obsesión de mis últimos diez años, desde que era estudiante de la Facultad de Biología de la Universidad de La Habana: el uso sostenible de las tortugas marinas en Cuba. Si mis modestos esfuerzos, aunque con inversiones energéticas desmedidas de mi persona, no hubieran contribuido a aportar información útil para evaluar el uso consuntivo de las tortugas marinas en lo que a principios de este año se reconoce como veda permanente e indefinida a la pesquería de este recurso, jamás hubiera defendido una tesis académica de esta naturaleza. Dra. Georgina Espinosa, tutora de esta tesis y madraza del autor, quien ha representado en primera instancia un ejemplo genuino de la dignidad de un científico, albergando un profundo pensamiento y un compromiso exclusivo con los resultados de la investigación y de la repercusión de éstos en la sociedad, donde en definitivas cuentas, se han de tomar las decisiones derivadas. Cualquier calificativo positivo como profesora y ser humano sería incompleto para referirme a tan distinguido, y cada vez más raro ejemplar dentro de la sociedad del hombre nuevo, por eso, he de concluir sencillamente con una frase: gracias por existir y formar hombres de bien, aunque excepcionalmente y por fortuna, algunos no hayamos perdido la capacidad de recalcitrar. Dr. F. Alberto Grobois, también tutor de esta tesis y padrazo del autor, con quien he lidiado en contiendas académicas por la búsqueda, única y exclusivamente, de la mayor aproximación a la verdad científica más allá de cualquier posición de fuerza. A través de su guardarraya he podido abrirme paso en la comunidad científica internacional y como si fuera poco, encontrar abrigo en su guarida y con su querida y heterogénea familia. También le doy mis gracias por apoyar en la formación de mis estudiantes y ayudar a mi familia en la medida de sus posibilidades. Dr. Erik García, oponente en el acto de predefensa de esta tesis, por contribuir de forma significativa a mi formación como obrero de la ciencia desde tempranos momentos, cuando parecería un loquillo investigando raros lagartijos endémicos de nuestro archipiélago. Sus aportaciones han sido esenciales para mejorar la calidad de mis investigaciones, y ahora, del documento defendido. Muchas gracias por mantenerse como formador a pesar de las circunstancias. Dr. Vicente Berovides, también oponente en el acto de predefensa de esta tesis, por su excelente crítica para mejorar la calidad del documento defendido y por sus extenuantes preguntas, que aún me llevan a la reflexión. Dra. Clara González, oponente a petición en el acto de predefensa de esta tesis, en primera instancia por aún ser formadora de generaciones de biólogos. Las correcciones derivadas de la exhaustiva revisión del documento realizadas por su persona, han sido responsables de la notoria mejoría con que ahora se vuelve a presentar. También quiero agradecer por la conformación de un examen de especialidad, conjuntamente con los profesores Dr. Erik García y MC. Rina Pedrol, que me preparó sólidamente para la discusión de la tesis. Dra. Aymée Robainas, por contribuir en calidad de oponente y revisora de varias de las tesis de mis estudiantes, cuyas correcciones están reflejadas explícitamente en esta tesis. Además de admirarla como profesional, también fenotípicamente, he de reconocer sus valores excepcionales entre los que resalto la amistad, cualidad compartida con los distinguidos miembros de su familia cercana. Dra. Ana M. Suárez, sin su ayuda no hubiera llegado y más que pasar la meta, hacerlo con alegría a pesar de las circunstancias. Creo que su ejemplo en todos los campos me ha servido de pie de amigo, sobre todo para balancear la otra cara de la enfermedad. Dra. María Eugenia Alonso, en primera instancia por encausar mis mejores energías para hacer frente a la enfermedad y luego, por torearme en ausencia de mi madraza para llegar a pesar de la arbitrariedad, que siempre disocia. MC. Rina Pedrol, por su excelencia como pedagoga, ejercida con particular elegancia. Dra. María Elena Ibarra, por contribuir a abrir un espacio dentro del ámbito universitario para la investigación de las tortugas marinas. Estaría insatisfecho si no expresará también mi gratitud por la ayuda recibida en el plano familiar y por los logros de conjunto en tiempos pasados. Especialmente, a mis estudiantes cuyas tesis han tenido que ver directamente con el desarrollo de las investigaciones biológicas y de conservación de las tortugas marinas: MC. Idania Lee, MC. Emir Pérez, MC. Roberto C. Frías, MC. Maribel González, Lic. Javier Rodríguez, Lic. Frander B. Riverón, Lic. Mayumi Vega, Lic. Talia Pérez, Lic. Juan Solano, Lic. Yuzneydy Pereira, y Diseñadores Néstor Navarro y Marwin Sánchez. Sin el aporte de ellos, esta contribución sería no sólo incompleta, sino pobre. En particular, he de destacar la ayuda en la búsqueda de información y contribución científica del MC. Emir Pérez, por demás, hermano en los avatares de la vida a pesar de tener una formación ideológica diferente, quizás más encausada. Dra. Nancy N. FitzSimmons y Dr. James W. Wiley, por la revisión de parte de la información científica generada para esta tesis, facilitación de bibliografía y consejos impartidos para llegar. Dr. Rogelio Díaz, de quien siempre recibí buenos consejos sensus latior a pesar de discrepar en un principio sobre la permisividad del uso consuntivo de Eretmochelys imbricata. Creo que fue muy paciente conmigo. Parte de las muestras aportadas para este trabajo fueron colectadas por su persona, y generosamente ha permitido el uso de sus datos originales que le sirvieron para obtener el grado científico de Doctor, con una sola condicionante: el alumbramiento de información confiable para la actualización del plan de manejo de E. imbricata en Cuba. A su persona le debo gestos humanos para contrarrestar la enfermedad, que me permitieron continuar con mi actividad científica. Nunca lo podré olvidar. Lic. Madelín Ramos, cuyas manos formaron parte del procesamiento de varias de las muestras de esta tesis y también ayudaron en la colecta, en condiciones difíciles, junto a otros amigos como Barbarita, Elier y Daniel. Bibliotecaria Yuriem Lezcano del CIM-UH, por su enriquecedora revisión de las referencias bibliográficas. También por su apoyo en momentos difíciles. Bibliotecaria Clara Ramírez de la UNAM, por su ayuda profesional para optimizar la búsqueda de bibliografía, sobre todo de los clásicos. MC. Sylvia Leal, por su apoyo en la corrección de los artículos a publicar. También por su cariño. Lic. Gonzalo Nodarse, Téc. Biología Marina Erick Escobar, MC. Julia Azanza y personal de apoyo a la investigación, cuyos nombres a veces son sustituidos por los jefes de proyectos, por la facilitación de buena parte de las muestras procesadas en esta tesis. A los pescadores de la Empresa Pesquera Industrial de Camagüey: Miguel Camps, Juan Pajares, Amado Blanco, Jorge L. Fals, Idilberto Fals, Osvaldo Fals, el niño y sus esposas, en particular Norma, Clarita, Blanquita y Mayda, y a su dirección: en particular al Ing. Ángel Freyre (Director), Ernesto Aguilar (Subdirector de Operaciones Pesqueras), Juan Miguel (Subdirector económico) y a los entonces Jefes de Flota (Armelio Frometa) y transporte (Jorge Beber), al igual que a los pescadores de la unidad pesquera de Cocodrilo, pertenecientes a la Empresa Pesquera de la Isla de la juventud: Alexis Meneses, Fulgencio, Abel, Gerardo y Eltón, y a Mirta, la directora de entonces, por facilitar la infraestructura para la toma de muestras y por la información aportada en relación a la pesquería, aspectos biológicos y comercialización de las tortugas marinas. A mis colegas Dra. Graciela Olmedo y Dra. Olimpia Carrillo, cuya excelencia en su investigación arrojó luz sobre los conceptos de “tradición” y “valor nutricional”, aplicados como uno de los basamentos para mantener el uso consuntivo legal de las tortugas marinas en Cuba. A las profesoras del Instituto Superior de Diseño, Dis. Michel Miyarez y MC. Lucila Fernández, por la profesionalidad con la que asumieron la emprendedora y exhaustiva Campaña de Bien Público para la Conservación de las tortugas marinas en Cuba. Lic. Sergio Álvarez por su ayuda para que fluyera la presentación de la tesis durante el acto de predefensa y por su amistad. A mi enigmática MC. Olivia Millán, por nuestras pláticas exigentes sobre temáticas de genética y por compartir momentos especiales a la sombra de un té de café, en compañía de la querida MC. María de los Ángeles, MC. Nadia y Lic. Ricardo, a los cuales también debo mi subsistencia en tierras aztecas. Natalia, sin su abrigo no hubiera llegado. MC. Raquel Briseño por aunar a la mayoría del gremio de tortugas marinas y encausarnos por el camino más recto, por su sensatez y acertados juicios que con su glamour nos ha mejorado a todos. Hada Marydele Donnelly, MC. Fernando Bretos y Dr. David Gugenheim por el apoyo material y espiritual en pos de la capacitación y desarrollo de proyectos de jóvenes científicos cubanos. MC. Leandro Bombino, por tratar de llevar a vías de hecho un macroproyecto para la conservación de las tortugas marinas en los archipiélagos cercanos al Camagüey y por su ayuda extraordinaria para encausar algunos de nuestros trabajos de investigación, en la cual también ha estado involucrado el aliviar el peso de la enfermedad. Susana, Celia y Fausto, a quienes debo la impresión y fotocopia de la documentación requerida para la predefensa. Ahora de nuevo a Susana y nuevos colaboradores Olguita, Jorge y Berta. Carpintero Marcial Blanco, otro padrazo de la Ciénaga de Zapata con quien hice el amadrinador de tortugas marinas, instrumento de gran utilidad para el manejo de estos animales que ha sido objeto de atención para otros investigadores. Mayda Pelikián, madre de un gran amigo, devenido en un pescador responsable, y madraza del autor, por su enorme capacidad de recuperación que inspira a cualquier semilla. Por ayudarme a frenar el avance de la enfermedad. Tía-Mayra y Fela, otras madrazas pinareñas, sin las cuales no hubiera podido avanzar contra la enfermedad. A mis compañeros de trabajo más cercanos del CIM-UH, que prefiero no nombrar en mi paranoia, por el intercambio académico, por compartir gratos momentos y por ayudarme contra la enfermedad. MC. Patricia González y MC. Silvia Díaz, hermana y madre en difíciles circunstancias. Dra. Concepción Campa, sin su ayuda hoy no estaría aquí, tampoco los miembros más queridos de mi familia. A mis hermanos de aquí no mencionados con anterioridad: René, Jorge Luis, Liuba, Rosita, Maite, Alejandro, Maickel y Oscar, y a los desperdigados: Susana, Ernesto, Pedro Pablo, Adyari, Erwin, Omar, Daniel, Daylin, Gabriel, Margarita, Armando, Idabel e Hidalgo. Sin ellos tampoco estaríamos aquí. A mis amigos foráneos Olga, Jimena, Benjamín, Julien, Constanza, Cecilia, Martín, Rosalba, Germán, Carlos, Mary, Ingmar, Marfer, Antonio y Ruco, colaboradores en disímiles circunstancias. A los médicos Fleites, Jorge Luis, Areces, Rubio, Juanca, Tania, Barroso y Luis, a las farmacéuticas María Antonieta y Arays, y a las enfermeras Adalgiza, Giselle, Aidita, Lucy y Martica, sin la contribución de ellos tampoco estaríamos aquí. A los que ya no están y dedico esta contribución científica, que no voy a nombrar porque quiero que sigan en paz, por ellos también estoy aquí. A mi hermana, madre, padre y tío por perseverar ante lo extremo, que se contrapone a la existencia. En la balanza de cargas nefastas, ha sido el peso de ustedes el que me ha evitado perder el equilibrio y sacar ese extra en mi cuarto e indeseado tema, que sin lugar a dudas me hizo mejor persona y me fortaleció mi capacidad de denuncia, ya grande. Si algunos nombres han escapado de mis agradecimientos, sobre todo aquellos que de una forma u otra han estado relacionados con mi proyecto de investigación, las personas a las que distinguen no han de sentirse excluidos. En mis circunstancias no he podido dejar de asumir el reto de nombrar y asumo la responsabilidad de cualquier descuido. Por último y no por considerarlo menos importante, a la naturaleza cubana de quien se ha servido íntegramente este trabajo y quien me flechó desde muy temprano y para siempre. Índice Nomenclatura, abreviaturas, y algunas conceptualizaciones empleadas……………...………..…….i SÍNTESIS..........................................................................................................................................ii I. INTRODUCCIÓN..........................................................................................................................01 Hipótesis de trabajo.........................................................................................................................05 Novedad científica...........................................................................................................................05 Importancia teórica..........................................................................................................................06 Importancia práctica........................................................................................................................06 II. REVISIÓN BIBLIOGRÁFICA II.1 Estatus de las poblaciones de anidación en el Atlántico II.1.1 Chelonia mydas................... .................................................................................................08 II.1.2 Caretta caretta.......................................................................................................................11 II.2 El mtDNA como marcador molecular en la biología poblacional y evolutiva...................13 II.3 Marcadores moleculares: herramientas útiles para la conservación................................15 II.4 Descripción de la RNC del mtDNA II.4.1 Chelonia mydas.....................................................................................................................17 II.4.2 Caretta. caretta......................................................................................................................18 II.5 Estructura poblacional en el Atlántico II.5.1 Chelonia mydas.....................................................................................................................18 II.5.2 Caretta caretta.......................................................................................................................20 II.6 Diversidad genética en el Atlántico Chelonia mydas y Caretta caretta..................................................................................................24 II.7 Filogenia y Filogeografía II.7.1 Chelonia mydas.....................................................................................................................26 II.7.2. Caretta caretta......................................................................................................................29 III. MATERIALES Y MÉTODOS III.1 Obtención y procesamiento de las muestras.....................................................................32 III.2 Análisis de los datos.............................................................................................................35 IV. RESULTADOS IV.1 Análisis de secuencias IV.1.1 Chelonia mydas……............................................................................................................42 IV.1.2 Caretta caretta……..............................................................................................................45 IV.2 Estructura poblacional IV.2.1 Chelonia mydas……............................................................................................................48 IV.2.2 Caretta caretta……..............................................................................................................50 IV.3 Diversidad genética IV.3.1 Chelonia mydas……............................................................................................................56 IV.3.2 Caretta caretta……..............................................................................................................58 IV.4 Relaciones filogenéticas y filogeográficas IV.4.1 Chelonia mydas……............................................................................................................61 IV.4.2 Caretta caretta……..............................................................................................................64 V. DISCUSIÓN V.1 Análisis de secuencias.........................................................................................................69 V.2 Estructura poblacional.........................................................................................................72 V.3 Diversidad genética.............................................................................................................79 V.4 Relaciones filogenéticas y filogeográficas.......................................................................90 V.5 Implicaciones para la conservación..................................................................................94 VI. CONCLUSIONES...................................................................................................................97 VII. RECOMENDACIONES.........................................................................................................99 VIII. REFERENCIAS BIBLIOGRÁFICAS AUTOBIBLIOGRAFÍA Publicaciones que forman parte de la tesis Otras publicaciones relacionadas con el tema Secuencias inscritas en el Genbank Trabajos presentados en eventos científicos relacionados con el tema de tesis NOMENCLATURA, ABREVIATURAS, Y ALGUNAS CONCEPTUALIZACIONES ________________________________________ i Nomenclatura, abreviaturas, y algunas conceptualizaciones empleadas: A AMOVA C conducta de reproducción en el lugar de origen (Inglés. Natal homing) CpG Cuenca ddNTP DNA dNTP Fidelidad por el sitio de anidación Filopatría G Indel Linaje MA Mediterráneo, a mtDNA nDNA pb PCR Población RNC scnDNA SP Stepping-stone model T Adenina Análisis de la varianza molecular Citosina Término construido por el autor en calidad de análogo del término inglés “natal homing” aplicado a tortugas marinas por ser especies filopátridas y fieles al sitio de anidación según Carr (1967 & 1975). Gen. Formalmente isla CpG. Dícese de la región de DNA de al menos 200 pb, con un porcentaje de GC mayor de 50 y con un promedio de CpG observado/esperado mayor de 0.6 adaptado de Gardiner-Garden & Frommer (1987) y Takai & Jones (2002). Del lat. Concha: f. Territorio cuyas aguas afluyen todas a un mismo río, lago o mar. • cuenca atlántica; dícese de los territorios cuyas aguas confluyen finalmente en el océano Atlántico, i.e. costa este de América, islas del mar Caribe y Golfo de México, y costas oeste de África y Europa. • cuenca del mar Mediterráneo; dícese de los territorios cuyas aguas confluyen finalmente en el mar Mediterráneo. Didesoxinucleótidos Ácido desoxirribonucleíco Desoxinucleótidos Capacidad de discriminar el lugar de anidación respecto al primer sitio de puesta, una vez el individuo encuentra el área de reproducción (anidación) (adaptado de Carr (1967 & 1975)) Capacidad de discriminar la región donde se ubica el área de reproducción (anidación), una vez el individuo completa la migración (adaptado de Carr (1967 & 1975)) Guanina Inserción/ deleción – plural: indeles Del prov. linhatge o cat. Llinyatge: m. Ascendencia o descendencia de cualquier familia. Evol. Series de poblaciones ancestrales y descendientes a través del tiempo. Usualmente se refiere a la evolución de una sola especie o a varias especies descendientes de un ancestro común. Gen. Estirpe de variantes genéticas que comparten un ancestro común, donde median de uno a dos cambios entre las vecinas, rara vez tres. • mediterráneos americano – Viejo Mundo; dícese del linaje cuyos haplotipos pertenecen a colonias de anidación con costas en el mar Caribe, Golfo de México, costa atlántica de la península de la Florida y mar Mediterráneo. • Atlántico Sur; dícese del linaje cuyos haplotipos pertenecen a colonias de anidación de América del Sur con costas en el océano Atlántico. • Atlántico – península de la Florida; dícese del linaje cuyos haplotipos pertenecen a colonias de anidación con costas en el océano atlántico y ambas costas de la península de la Florida. Millones de años Del lat. Mediterraneus: adj. Dícese de lo que está rodeado de tierra e.g., mar Mediterráneo. Ú. t. c. s. m. p. us. Dícese también de lo que está en el interior de un territorio e.g., Ciudad Mediterránea. Perteneciente al mar Mediterráneo, o a los territorios que baña. • Mediterráneo del Viejo mundo; dícese de las colonias de anidación con costas en el mar Mediterráneo. • Mediterráneo americano; dícese de las colonias de anidación con costas en el mar Caribe, Golfo de México, y costa atlántica de la península de la Florida. ADN mitocóndrico ADN nuclar Pares de bases Reacción en cadena de la polimerasa Del lat. populatio, -onis: f. Acción y efecto de poblar. Conjunto de personas que habitan la Tierra o cualquier división geográfica de ella. Evol. Dícese de un grupo de individuos conespecíficos que ocupan una región geográfica más o menos definida y exhiben una reproducción continua de generación en generación; las interacciones ecológicas y reproductoras son más frecuentes entre estos individuos que con otros miembros de otras poblaciones de la misma especie. Región no codificadora copia simple de DNA nuclear, es decir, amplificación de un fragmento, locus o loci en una cadena. Sitio polimórfico – plural: SPs Traducción en este documento como modelo de pasarela. Timina Nota: Estas aclaraciones están hechas con la intención de que pueda arbitrarse el documento a partir del conocimiento de la línea de pensamiento del autor y sus asunciones, teniendo en cuenta la diversidad de criterios según la bibliografía consultada. SÍNTESIS________________________________________________________________________________________________ ii SÍNTESIS El manejo efectivo para la conservación de las tortugas marinas depende del entendimiento de las interconexiones demográficas entre las poblaciones. En este trabajo se determinaron la estructura poblacional, la diversidad genética y las relaciones filogenéticas y filogeográficas entre las principales colonias de anidación cubanas de Chelonia mydas y Caretta caretta, y se compararon en un contexto regional, a partir de la secuenciación de la primera mitad de la región no codificadora del DNA mitocóndrico, con vistas a un manejo sostenible. En las colonias de anidación cubanas de ambas especies estuvieron presentes los haplotipos más distribuidos y representados de los linajes de cada especie informados para la región. No obstante, las colonias de C. mydas también se destacaron por la presencia de haplotipos endémicos. Estas constituyeron poblaciones panmictas y estuvieron estructuradas significativamente con las restantes poblaciones de anidación de la región, como consecuencia de un porcentaje de variación mayor dentro que entre las colonias de anidación. Por consiguiente, las colonias de anidación cubanas constituyen poblaciones que han intercambiado migrantes limitadamente con las restantes del Mediterráneo americano. El grado de diferenciación genética (F- Φ ST) estuvo positivamente correlacionado con la distancia geográfica entre las colonias de anidación de la región, cumpliéndose el modelo pasarela congruente con los patrones de corrientes marinas imperantes en la región. Existieron fallos a la conducta de reproducción en el lugar de origen dentro de cada especie, que condujeron a dispersiones de los haplotipos más distribuidos y representados entre los que se encontraron los ancestros de cada linaje. Estos fallos pudieron ser provocados por las fluctuaciones climáticas durante el Pleistoceno, que conllevaron a sucesivas recolonizaciones de hábitat en función de las características óptimas para la anidación. Consecuentemente, las huellas de las expansiones demográficas han quedado difusas, a pesar de la estructura genética significativa y presencia de endemismo en la mayoría de las poblaciones de anidación. A partir del análisis filogeográfico de clados anidados se corroboró un flujo genético restringido por aislamiento por distancia, con fragmentación pasada y colonizaciones a grandes distancias o una de estas dos últimas variantes. Finalmente, las poblaciones de anidación cubanas, a pesar de compartir un pasado histórico común con las restantes poblaciones de anidación de la región, constituyeron unidades genéticas singulares consideradas Mus, que deben ser manejadas cuidadosamente para evitar pérdida de la variabilidad genética del genofondo de cada especie de tortuga marina. INTRODUCCIÓN__________________________________________________________________________________________ 1 I. INTRODUCCIÓN Las tortugas marinas: caguama, Caretta caretta (Linnaeus, 1758); carey, Eretmochelys imbricata (Linnaeus, 1766); golfina, L. olivacea (Eschscholtz, 1829); lora, Lepidochelys kempii (Garman, 1880); tinglado, Dermochelys coriacea (Vandelli, 1761); tortuga plana, Natator depressus (Garman, 1880); y tortuga verde, Chelonia mydas (Linnaeus, 1758); cuyas formas actuales datan de varios millones de años, presentaban poblaciones numerosas antes que el hombre desarrollara las flotas de navegación y perfeccionara las artes de pesca. Paradójicamente, con el devenir de la civilización, diezman los tamaños poblacionales y algunas colonias de anidación fueron extirpadas de sus regiones nativas (Meylan, 1999a; Meylan & Donnelly, 1999; Seminoff, 2002; Heppell et al., 2003; Witherington & Frazer, 2003). Es hasta hace poco, cuando la escasez o el colapso de los recursos naturales que el hombre venía aprovechando repercuten negativamente en la supervivencia de las sociedades, que el pensamiento humano toma conciencia colectiva sobre el uso sostenible de los recursos renovables (Caughley & Gunn, 1996). Los efectos negativos e irreversibles de la explotación desmesurada de la generalidad de estos recursos, ha implicado fragmentación de las poblaciones y deterioro de los hábitat. Razón por la cual, gran parte de los especialistas en tortugas marinas dudan de la sostenibilidad de un uso extractivo de estas especies (Campbell, 2003; Heppell et al., 2003) considerando: la magnitud de la reducción poblacional en correspondencia con la historia natural, las limitaciones y lagunas en el conocimiento disponible, las dificultades prácticas de implementar un manejo que abarque la distribución total de cada una de las poblaciones, la maduración sexual tardía y los diversos hábitat que utilizan en su complejo ciclo de vida. Es por esto que todas las tortugas marinas se encuentran inscritas como especies amenazadas de extinción (Baillie & Groombridge, 1996) y se prohíbe el comercio internacional de las mismas, manifiesto en la inclusión de éstas en el apéndice I de la Convención sobre el Comercio Internacional de Especies Amenazadas de Fauna y Flora Silvestres (CITES) a partir de la década de los setenta del pasado siglo (Meylan & Donnelly, 1999). Debido a que las tortugas marinas son fieles a la conducta de reproducción en el lugar de origen (Carr, 1967 & 1975; Pritchard, 1976; Limpus et al., 1992), verificada por la diferenciación genética significativa que existente entre las colonias de anidación (Meylan INTRODUCCIÓN__________________________________________________________________________________________ 2 et al., 1990; Bowen et al., 1992, 1993, 1994, 1995; Broderick et al., 1994; Norman et al., 1994; Encalada et al., 1996 y 1998; Bass et al., 1996), la marcación de los individuos en las áreas de anidación, alimentación y desarrollo, ya sea a través de marbetes (Limpus et al., 1992; Meylan, 1999b; Balazs, 2000;) y marcadores moleculares (FitzSimmons et al., 1995; Bowen et al., 2004), ha constituido una forma viable para estimar los tamaños poblacionales, comportamiento migratorio y demás patrones demográficos por regiones de anidación. La filopatría y la característica poco migratoria dentro de las áreas cálidas de alimentación de los estadios más avanzados de crecimiento y desarrollo de algunas poblaciones de C. mydas (Limpus & Walter, 1980; Balazs, 1982; Mendoca & Ehrhart, 1982; Green, 1993; Limpus et al., 1994b) y E. imbricata (Doi et al., 1992; Limpus, 1992; Broderick et al., 1994; Heppell & Crowder, 1996; Carrillo et al., 1999; Díaz-Fernández et al., 1999), también han conllevado a la defensa del uso sostenible de las tortugas marinas por parte de científicos, comunidades rurales y gobiernos (Márquez, 1990; Cornelius et al., 1991; Doi et al., 1992; Ruiz, 1994; Clifton et al., 1995; Campbell, 1998; Evans & Vargas, 1998; Anónimo, 1998; Carrillo et al., 1999; Díaz-Fernández, 1999; Mohadin, 2000). Considerando la variabilidad estacional y etológica en las poblaciones de estas especies, y la falta de integración regional en el conocimiento del recurso renovable compartido, las propuestas defendidas no han aportado datos suficientemente convincentes para lograr un consenso en la comunidad científica a favor del uso sostenible, independientemente de que se reconozca el carácter tradicional de la pesquería para muchos asentamientos costeros (Crouse, 1999; Meylan, 1999a y b; Campbell, 2002; Heppell et al., 2003). No obstante el uso continuado de estos recursos a escalas nacionales por asentamientos costeros y países (Thorbjarnarson et al., 2000a y b; Campbell, 2003), varias poblaciones de tortugas marinas muestran crecimientos sostenidos después de implementar programas de conservación durante décadas, como son los casos de L. olivacea del Pacífico mexicano (PeñafloresSalazar et al., 2001), algunas poblaciones de E. imbricata en el Caribe (Garduño et al, 1999; IUCN, 2002); de C. mydas en Estados Unidos de América (USA) (Seminoff, 2002) y Costa Rica (Bjorndal et al., 1999); y de L. kempii en México (Márquez et al., 1999). Un manejo adecuado de las pesquerías de tortugas marinas no tiene por qué afectar la supervivencia de las poblaciones (Anónimo, 1998). INTRODUCCIÓN__________________________________________________________________________________________ 3 En el Atlántico, el Mediterráneo americano ha constituido una importante región de anidación para C. mydas (Seminoff, 2002), C. caretta (Ehrhart et al., 2003) y E. imbricata (Mortimer & Donnelly, 2007). Particularmente, el archipiélago de Cuba es reconocido como un sitio importante de anidación, alimentación y pesquería de E. imbricata (Anónimo, 1998; Bass, 1999; Carrillo et al., 1999; Meylan, 1999a; Meylan & Donnelly, 1999; Moncada et al., 1999). Sin embargo, no se conoce una publicación científica que aborde la anidación de C. mydas y C. caretta de manera concluyente en Cuba. Razón por la cual, se clasifica la anidación del archipiélago cubano como secundaria (Seminoff, 2002; Ehrhart et al., 2003). No obstante, el occidente de Cuba ha sido reconocido a nivel nacional como un sitio de importancia para la anidación de C. mydas y C. caretta, destacándose áreas del archipiélago de los Canarreos como Cayo Largo del Sur, donde han sido informados más de 2000 nidos de C. mydas por temporada de anidación (Nodarse, com. pers.); Cayo Real de la Cayería de San Felipe, donde han sido informados 43 nidos de C. mydas durante 15 días del final de la temporada de anidación de 1998; y la playa “El Guanal” del sur de la Isla de la Juventud, donde han sido marcados 98 ovíferas de C. mydas en el intervalo de 1989 hasta 1996 (Nodarse et al., 2000a) y han sido informados 174 nidos de C. caretta en el período de anidación de 1989 (Nodarse et al., 2000b); así como la península de Guanahacabibes, donde han sido informados 638 anidaciones y han sido marcadas 172 ovíferas de C. mydas durante el 2002 (Azanza et al., 2003). Paradójicamente, se desconoce la estructura poblacional de las colonias de anidación dentro de las áreas de anidación cubanas, y por consiguiente, las relaciones de estas colonias con las restantes del Mediterráneo americano así cómo la contribución de éstas a las áreas marinas de agregación de la cuenca atlántica. Este aspecto adquiere extraordinaria importancia debido a que cientos de estos animales caen anualmente en redes de pescadores (Campbell, 2003), sin poder determinar adecuadamente los niveles de impacto en las poblaciones afectadas ni en los asentamientos costeros beneficiados, con vistas a lograr un manejo sostenible a nivel regional. Considerando la contribución significativa de cada colonia de anidación de tortugas marinas al genofondo de la metapoblación en el Mediterráneo americano, como consecuencia de la estructuración genética existente entre la generalidad de éstas, y la INTRODUCCIÓN__________________________________________________________________________________________ 4 problemática del declive de las tortugas marinas por diversos factores, se propone determinar comparativamente las relaciones genéticas entre las principales colonias de anidación de C. mydas y C. caretta del suroeste de Cuba en el contexto del Mediterráneo americano, empleando secuencias de la RNC mtDNA, con vista a un manejo sostenible. El objetivo general se desglosa en los siguientes objetivos específicos con sus respectivas tareas: 1. Determinar la estructura poblacional de colonias de anidación cubanas de C. mydas y C. caretta en un contexto regional. • Colecta y procesamiento biológico – molecular de muestras de tejido en áreas de anidación para obtener secuencias de la RNC del mtDNA. • Comparación de las secuencias con los haplotipos informados por la literatura y bases de datos para identificar la identidad de los individuos procesados. • Comparación estadística a los niveles colonia (incluyendo aquellas con las cuales las cubanas han intercambiado genes), área y región de anidación (mediante métodos de agrupamiento jerárquico), para determinar el nivel población. 2. Caracterizar la diversidad genética de colonias de anidación de C. mydas y C. caretta en el archipiélago cubano. • Exploración la diversidad genética con índices preestablecidos, que incluyen desde la propia heterogeneidad hasta el tamaño efectivo de ovíferas a los niveles colonia y población de anidación. • Comparación de la diversidad genética de las entidades cubanas respecto a las de la región del Mediterráneo americano con las cuales han intercambiado genes. 3. Dilucidar las relaciones filogenéticas y filogeográficas entre haplotipos de C. mydas y C. caretta, y entidades poblacionales determinadas en un contexto regional. • Construcción del camino evolutivo más probable de los genes y de éstos dentro de las poblaciones de anidación del Mediterráneo americano, mediante relaciones de ancestría a través de métodos de distancia y cladísticos. 4. Comparar la estructura poblacional, diversidad genética, y relaciones filogenéticas y filogeográficas determinadas entre C. mydas y C. caretta en un contexto regional. INTRODUCCIÓN__________________________________________________________________________________________ 5 • Integración de los resultados obtenidos en las tareas anteriores a través de una discusión interespecífica, considerando además resultados análogos informados para otras especies de tortugas marinas. Hipótesis de trabajo. Hipótesis nula: • Si la generalidad de las colonias de anidación del Mediterráneo americano de C. caretta y C. mydas, especies filopátridas y fieles al sitio de anidación, están integradas fundamentalmente por haplotipos comunes de los linajes de la cuenca atlántica como consecuencia de fallos a la filopatría entonces, considerando la geografía del archipiélago de los Canarreos así como los fallos a la fidelidad por el sitio de anidación, las colonias cubanas no mostrarán estructura genética significativa con poblaciones de anidación vecinas de la región y exhibirán un modelo evolutivo semejante a nivel interespecífico. Hipótesis alternativa: • Si la generalidad de las colonias de anidación del Mediterráneo americano de C. caretta y C. mydas, especies filopátridas y fieles al sitio de anidación, están estructuradas genéticamente en el espacio de forma significativa aunque están integradas fundamentalmente por haplotipos comunes de los linajes de la cuenca atlántica entonces, a pesar de la geografía del archipiélago cubano así como de los fallos a la filopatría y fidelidad por el sitio de anidación, las colonias cubanas mostrarán estructuración genética significativa con las poblaciones de anidación de la región y exhibirán un modelo evolutivo particular en al menos una especie según su corología. Novedad científica: • Por primera vez se describen clinas geográficas en áreas de anidación continentales de C. mydas y C. caretta. • Por primera vez se determinó espacialmente la estructura poblacional de las principales colonias de anidación de C. mydas y C. caretta del suroeste de Cuba y en el contexto del Mediterráneo americano. INTRODUCCIÓN__________________________________________________________________________________________ • 6 Por primera vez se caracterizó la diversidad genética de las principales colonias de anidación de C. mydas y C. caretta del suroeste de Cuba. • Por primera vez se integraron en la filogenia y filogeografía de C. mydas y C. caretta las principales colonias de anidación cubanas y sus genes en el contexto del Mediterráneo americano. Importancia teórica: A la luz de los resultados de esta investigación fue generada la información que faltaba sobre estructura y diversidad genética, para comprender el modelo poblacional y las relaciones genéticas (filogenia y filogeografía) entre las poblaciones de anidación de C. mydas y C. caretta y sus genes en el Mediterráneo americano. A partir del conocimiento de la estructura poblacional regional, pudo constatarse que el patrón de corrientes que circula por el mar Caribe, Golfo de México y costas atlánticas de USA, constituye una barrera efectiva para el flujo de migrantes entre colonias de anidación vecinas geográficamente de C. mydas y C. caretta e.g. colonias de anidación de México y Cuba, y de las costas oeste y este de la península de la Florida. Por consiguiente, el modelo pasarela podría no ajustarse, aunque sea el más probable, en dependencia de cómo sean tomadas las unidades a comparar. Importancia práctica: El conocimiento aportado a la estructura, diversidad y relaciones genéticas en C. mydas y C. caretta, a partir del estudio de las principales colonias de anidación del archipiélago cubano, constituye una información valiosa para el establecimiento de las unidades de manejo dentro y fuera del archipiélago cubano. De forma clara se precisan las continuidades genéticas de las colonias de anidación de C. mydas y C. caretta dentro del archipiélago de los Canarreos, que deberán tenerse en cuenta en programas de manejo - conservación. Los resultados presentados en esta tesis han formado parte de tres artículos científicos publicados en revistas arbitradas (Revista Cubana de Química (2004), Revista de Investigaciones Marinas (2008a y b)), uno en reemisión (Conservation Genetics (2008)), y tres secuencias inscritas en 1) GenBank (http://ncbi.nlm.nih.gov) y 2) The Archie Carr Center for Sea Turtle Research (ACCSTR) (http://www.accstr.com, [email protected]), y han formado parte del trabajo “Estudio y Conservación de las INTRODUCCIÓN__________________________________________________________________________________________ 7 Tortugas Marinas en Cuba. Siete Años de trabajo”, premiado como el Mejor Resultado en la protección del Medio Ambiente de La Universidad de La Habana en el 2005. También han sido presentadas 43 ponencias en varios eventos científicos nacionales e internacionales. -Molecular Evolution Meeting, Sorrento, Italia (2002). - Opciones ambientales para la Industria Pesquera, La Habana, Cuba (2003). -Taller internacional para la conservación de las tortugas marinas en Guanahacabibes, Pinar del Río, Cuba: Primera edición (2003) y Segunda edición (2005). -GEO Juvenil, La Habana, Cuba (2005). - Evento Nacional de la Náutica Recreativa, La Habana, Cuba (2006). - MARCuba, La Habana, Cuba (2006, 2003). -Annual Symposium on Sea Turtle Biology and Conservation: 28th Loreto, Mexico (2008); 26th Crete, Greece (2006); 25th Savanna, Georgia, USA (2005); 24th San José, Costa Rica (2004). -VIII Congreso Latinoamericano de Herpetología, Varadero, Cuba (2008). Este documento está conformado por 99 páginas, distribuidas en introducción, revisión bibliográfica (RB), materiales y métodos (MM), resultados (R), discusión, conclusiones y recomendaciones. En la RB, MM y R se insertan dos, dos y trece figuras respectivamente. En R también se insertan nueve tablas. Formando parte del documento no paginado se incluyen los acápites índice, nomenclatura – abreviaturas – conceptualizaciones, síntesis y referencias bibliográficas correspondientes a las citas (285), al final del documento. REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 8 II. REVISIÓN BIBLIOGRÁFICA II.1 Estatus de las colonias de anidación en el Atlántico. II.1.1 Chelonia mydas. Las colonias de anidación de C. mydas han declinado en todas las cuencas oceánicas como consecuencia de la sobreexplotación de los huevos e individuos, y en menor cuantía, de la pesquería incidental y la degradación de los hábitat marinos (Seminoff, 2002). Por consiguiente, la población global de C. mydas ha disminuido entre un 37 y 61% durante las últimas tres generaciones de la especie, i.e. 141 años atrás (Seminoff, op.cit.). Consecuentemente, se ha sido ratificada la condición de especie en Peligro (Endangered (EN)) según la Unión para la Conservación de la Naturaleza y los Recursos Naturales (IUCN) (Baillie & Groombridge, 1996; Hilton-Taylor, 2000). A su vez, y considerando que C. mydas constituyó un recurso pesquero dentro de la pesquería legal de un número significativo de países y que aún se pesca en unos pocos, la Convención sobre el Comercio Internacional de Especies en Peligro (CITES) la inscribió en el Apéndice I para evitar su uso como producto mercantil y de esa forma poder contribuir a su conservación. En la actualidad, las principales poblaciones de anidación de la cuenca atlántica, i.e. poblaciones de anidación con más de 500 ovíferas por año (Seminoff, 2002), se localizan en Isla Ascensión (Gran Bretaña) (Mortimer & Carr, 1987); Isla Trindade (Brasil) (Moreira et al., 1995; Moreira, 2001); Tortuguero (Costa Rica) (Carr et al., 1982; Bjorndal et al., 1999); Isla de Bioko (Guinea Ecuatorial) (Thomas et al., 1999); Archipiélago Bijagos (Guinea-Bissau) (Barbosa et al., 1998); Surinam (Schulz, 1982); sur de la península de la Florida (USA) (Ehrhart & Witherington, 1992; Meylan et al., 1994) y Turquía (Geldiay, 1987; Kasparek et al., 2001). En estas poblaciones se han conducido programas de seguimiento con asesoría científica desde hace algunas décadas por lo que se consideran poblaciones de anidación índices para evaluar las tendencias demográficas de C. mydas (Seminoff, 2002). Otras poblaciones de anidación clasificadas como secundarias en la actualidad, i.e. poblaciones de anidación con menos de 500 ovíferas por año, se suman a la lista de poblaciones índices debido a la información demográfica disponible (Seminoff, op.cit.): Isla de Aves (Venezuela) (Sole, 1994; Lagueux, 2001) y península de Yucatán (México) (López, 2000). Existen numerosas colonias de anidación secundarias en las islas del Caribe (Seminoff, 2002). No obstante, éstas han sido pobremente estudiadas de forma REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 9 general (Seminoff, op.cit.). A pesar de que existen pocas publicaciones científicas sobre la anidación de C. mydas en el archipiélago de Cuba, el occidente de éste es reconocido a nivel nacional como un importante sitio de anidación para la especie, destacándose áreas del archipiélago de Los Canarreos como Cayo Largo del Sur, donde han sido informados más de 2000 nidos por temporada de anidación (Nodarse, com. pers.); Cayo Real de la Cayería de San Felipe, donde han sido informados 43 nidos durante quince días en el último mes de la temporada de anidación de 1998 (Ruiz et al., 2003) y la playa “El Guanal” del sur de la Isla de la Juventud, donde han sido marcadas 98 ovíferas en el intervalo de 1989 hasta 1996 (Nodarse et al., 2000a), así como la península de Guanahacabibes, donde han ido informados 638 anidaciones y marcadas 172 ovíferas durante el 2002 (Azanza et al., 2003). Como C. mydas es filopátrida y fiel al sitio de anidación (Carr & Ogren, 1960; Carr, 1967, 1975; Carr et al., 1978; Carr et al., 1982; Dodd, 1988), las colonias de anidación de la cuenca atlántica constituyen una metapoblación (Encalada et al., 1996). Por consiguiente, los estudios poblacionales conducidos en cada área de anidación han sido representativos para los análisis de las tendencias poblacionales de la especie. Por otra parte, algunas de las poblaciones de anidación más importantes del Atlántico en tiempos pasados ya no lo son, e.g. la colonia de anidación de Islas Caimán (Gran Bretaña), considerada en el pasado entre las más grandes del mundo (Lewis, 1940; Parsons, 1962). En la actualidad, ésta ha sido prácticamente extirpada como consecuencia de la depredación humana y la pérdida de hábitat (Lewis, op.cit.; Parsons, op.cit.), desconociéndose el origen de la repoblación. En esta isla se ha comprobado a través de la presencia de los haplotipos más distribuidos y representados de los linajes de la cuenca atlántica, que la repoblación fue consecuencia de fallos a la conducta de reproducción en el lugar de origen (Lahanas et al., 1994). Los fallos de la especie a la conducta de reproducción en el lugar de origen han facilitado la fundación de nuevas colonias (Carr, 1967). Estos fallos, que han traído consigo la anidación en otras playas frecuentadas por la especie o la colonización de nuevas, también han sido documentados por Bowen et al. (1992), Encalada et al. (1996) y Bass et al. (1998). Otras tres poblaciones de anidación han experimentado una reducción del tamaño poblacional en el siglo pasado: Bioko (Thomas et al., 1999) e Isla de Aves (Sole, 1994). Para otras colonias de anidación de C. mydas como las cubanas, han sido REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 10 testimoniadas observaciones sobre la abundancia de las anidaciones en las crónicas de las Indias Occidentales. Eran tan abundantes, que conllevó a la aprobación del uso de las ovíferas como alimento para la dotación esclava de los ingenios azucareros. Por consiguiente, la clasificación acorde a la importancia de las poblaciones de anidación según la magnitud de ésta no debe primar sobre los análisis de tendencias poblacionales (Seminoff, 2002). Los fallos a la filopatría en C. mydas, han estado influenciados de alguna forma por la complejidad del ciclo biológico, que incluye amplias migraciones entre las áreas de desarrollo y de anidación siguiendo rutas que abarcan de cientos a miles de kilómetros (Meylan, 1982). Ambas clases sexuales migran (Carr, 1986; Mortimer & Portier, 1989), con un intervalo de remigración de 2.86 años de acuerdo con Hirth (1980), Dodd (1988) y Van Buskirk & Crowder (1994). Una vez que las hembras llegan a las áreas de anidación, anidan de dos a siete veces, poniendo cerca de 100 huevos con un intervalo de reanidación intra-estacional de doce días (Hirth, op.cit.; Dodd, op.cit.; Van Buskirk & Crowder, op.cit.). Tanto las ovíferas como los machos reproductores regresan a las áreas de alimentación distantes en un período de tiempo impreciso (Hirth, 1997). Después de un período de incubación de ocho semanas los neonatos emergen, abandonan las playas de anidación y comienzan una fase oceánica (Carr, 1987). Los juveniles de C. mydas reclutan los hábitat neríticos, que son ricos en pastos marinos, cuando alcanzan los 30 a 40 cm de largo curvo del carapacho o más (Bjorndal & Bolton, 1988; Keinath & Musick, 1991; Limpus et al., 1994a). A menudo estos hábitat son clasificados como de desarrollo. En éstos, los juveniles forrajean y crecen hasta alcanzar la madurez sexual (Musick & Limpus, 1997). Los estimados publicados de la primera edad de madurez sexual se encuentran en el intervalo de 27 a 33 años en el Atlántico (Frazer & Ladner, 1986), 30 o más años en Australia (Limpus & Walter, 1980) y de 9 a 58 años en Hawai (USA) (Balazs, 1982). Una vez que alcanzan la madurez comienzan a migrar entre las áreas de forrajeo y las áreas de anidación para el apareamiento. Estas migraciones comprenden intervalos de años múltiples e irregulares (Hirth, 1997). Durante los períodos de no apareamiento, los adultos residen en las áreas neríticas de alimentación que en ocasiones coinciden con hábitats de desarrollo de juveniles (Limpus et al., 1994b). Finalmente, el balance entre la fidelidad por el sitio de anidación y la dispersión a largas distancias son la clave fundamental para REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 11 entender la filogeografía de las poblaciones de anidación de C. mydas del Atlántico (Encalada et al., 1996.) II.1.2 Caretta caretta. La supervivencia de las poblaciones de anidación de C. caretta está afectada en la actualidad fundamentalmente por la alta demanda de su carne para el consumo humano en regiones en vías de desarrollo (Perran, 1982), la pérdida de áreas de anidación como consecuencia del desarrollo turístico, la contaminación marina con residuos químicos y productos no biodegradables que reciben los océanos, mares y zonas costeras; y la captura incidental, particularmente cuando se usa el arte de pesca palangre que, según National Research Council (1990), genera más muertes que las restantes fuentes antrópicas de mortalidad. Razones por las cuales, en la actualidad C. caretta está categorizada globalmente como especie en peligro de extinción (EN) según la UICN (Hilton-Taylor, 2000) e inscrita en el Apéndice 1 de CITES para evitar su uso como producto mercantil. C. caretta se distribuye globalmente, a lo largo de las latitudes tropicales y subtropicales, así como en áreas templadas (Pritchard & Trebbau, 1984). En el Atlántico, la generalidad de las colonias de anidación ha sido localizada en latitudes subtropicales y templadas (Pritchard, 1979; Francisco et al., 1999). Las mayores concentraciones de anidación se ubican al sureste de USA, comprendiendo aproximadamente el 90% del esfuerzo reproductor en esta cuenca oceánica (más de 10 000 ovíferas anuales), seguida por las poblaciones de anidación de Carolina del Norte (USA) e Islas de Cabo Verde, que reciben hasta 1000 ovíferas cada año (U.S. Fish & Wildlife Service, 2002). Las colonias de anidación de Quintana Roo (México) y Bahía (Brasil) han sido consideradas como secundarias, con alrededor de 300 a 400 ovíferas por año, respectivamente (Zurita et al. 1993). En el Mar Mediterráneo, Bahía Kiparissia (Grecia) es una de las mayores colonias de anidación con 300 a 400 ovíferas por año (Groombridge, 1990; Margaritoulis, 1998), seguida por las colonias de anidación de Turquía (Baran & Kasparek, 1989; Gerosa et al., 1998) y Chipre (Demetropoulos & Hadjichristoporou, 1989; Broderick & Godley, 1996). Agrupaciones más pequeñas, aproximadamente de hasta 100 ovíferas arriban anualmente a las playas del norte de la península de la Florida (USA), Banco Cayo Sal (Bahamas), REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 12 Yucatán (México), Sergipe y norte de Bahía (Brasil), sur de Bahía a Río de Janeiro (Brasil). Agregados de anidación pequeños, pero significativos, también han sido informados para Bahamas, Cuba y el occidente de África (Dodd, 1988). C. caretta, al igual que el resto de las tortugas marinas, manifiesta filopatría (Bowen & Avise, 1995) y la mayoría de las ovíferas son fieles al sitio de anidación (Bjorndal et al., 1983; Dodd, 1988), por lo que los estudios de las colonias de anidación son básicos para analizar las tendencias poblacionales de la especie. En la actualidad, algunas de las colonias de anidación específicas de la subpoblación norteña de la península de la Florida han sido catalogadas como colonias en declive, particularmente los pertenecientes a los estados de Georgia y Carolina del Sur (Frazer, 1986; Hopkins-Murphy & Murphy, 1988; National Research Council, 1990; NMFS & FWS, 1995; Turtle Expert Working Group, 2000). Hopkins-Murphy et al. (2001) informaron un 5% de declive por año entre 1980 – 1997 para la población de Carolina del Sur, y un 2.6% por año entre 1964 y 1995 para la colonia de anidación de Little Cumberland Island (Georgia) (Frazer, 1983; Turtle Expert Working Group, 1998). No obstante, la filopatría de C. caretta puede variar en el entorno de 100 a 400 km según estudios de marcación en la Florida (Bowen & Avise, 1995) e.g., de miles de individuos marcados en el sureste de USA, unas pocas que anidaban originalmente en Georgia fueron relocalizadas anidando en el sur de la Florida (Bowen et al., 1993). Por lo que estas relocalizaciones ocasionales han podido indicar una “habilidad” de C. caretta para colonizar nuevas áreas de anidación en un tiempo evolutivo relativamente corto (Encalada et al., 1998). Estos fallos a la filopatría han traído consigo la anidación en otras playas frecuentadas por la especie o la colonización de nuevas playas (Encalada et al., op.cit.). Los fallos filopátridos en C. caretta han estado influenciados de algún modo por la complejidad del ciclo biológico, que incluye migraciones de más de 2600 km entre las áreas de forrajeo y las de anidación. C. caretta es una especie transoceánica, i.e., en el Atlántico los neonatos del sureste de USA, una vez emergidos, entran en la corriente de la Florida y posteriormente a la corriente del Golfo siendo transportados hasta el Atlántico oriental (Bolten et al., 1998). Luego de pasar por las islas oceánicas de Azores y Madeira, REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 13 y Canarias, son llevados por la Corriente Ecuatorial del Norte de regreso al Atlántico occidental, completando el "giro Atlántico Norte". En el Pacífico, las poblaciones anidadoras del Japón migran hacia las regiones de alimentación del Pacífico Oriental siguiendo la Corriente Kuroshio, y posteriormente la corriente Pacífico del Norte (Musick & Limpus, 1997), retornando a sus playas de origen por la Corriente Ecuatorial del Norte. Ambas clases sexuales migran, por lo menos distancias pequeñas, desde las áreas de alimentación a las áreas de apareamiento. Posteriormente, los machos regresan a las áreas de alimentación y las hembras a las de anidación (Miller, 1985; Limpus & Miller, 1993) con un intervalo de remigración de 2.59 años según Hirth (1980), Dodd (1988) y Van Buskirk & Crowder (1994). C. caretta reanida en un intervalo de distancia de 290 km aproximadamente, sugiriendo que la especie es capaz de transitar grandes distancias a pesar de ser pocos los individuos que realizan los movimientos (Bjorndal et al., 1983). II.2 El mtDNA como marcador molecular en la biología poblacional y evolutiva. El DNA mitocóndrico (mtDNA) ha sido usado con frecuencia para dilucidar identidades poblacionales y caminos evolutivos de linajes de animales por poseer un conjunto de características específicas entre las que se encuentran: 1) herencia materna en asociación con la falta de recombinación, 2) rápida tasa de divergencia por mutación. El mtDNA es una molécula haploide sin recombinación y en la generalidad de los casos, solo una fracción de la población, la mitad (hembras) si la proporción entre los sexos es (1:1) trasmite sus genes a la descendencia. Por consiguiente, los genomas de mtDNA diferenciados no serán contenidos en el mismo cigoto a no ser que ocurra un fenómeno de heteroplasmia producto de una pequeña contribución del macho, o se produzcan mutaciones en el citosol durante la reproducción sexual. Incluso si los eventos de recombinación ocurrieran, es improbable que los productos de recombinación representen nuevos genotipos y no serán reconocidos como recombinantes (Harrison, 1988), i.e. no existe recombinación efectiva. Todo lo cual ha condicionado una moderada tasa de variabilidad en el mtDNA y una filogenia matriarcal. Como consecuencia, el tamaño efectivo estimado con este marcador es menor que el que ha sido estimado para los alelos de los genes nucleares (FitzSimmons et al., 1996), y los procesos estocásticos serán particularmente importantes para determinar las frecuencias de los genotipos del mtDNA (Birky et al., 1983; Avise et al., 1984). A su vez, estas frecuencias deberían ser más REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 14 fuertemente influenciadas por los efectos de la deriva genética y los cuellos de botella poblacionales (Karl et al., 1992), por lo que el mtDNA puede actuar como un excelente identificador de los patrones de colonización, incluyendo los eventos fundadores y por ende, de los caminos evolutivos de los linajes (Harrison, 1988). Razones por lo cual el mtDNA ha sido considerado como una unidad genealógica no recombinante con múltiples alelos o haplotipos (Avise, 2004) Estudios comparativos tempranos sobre la evolución del mtDNA en primates han sugerido una tasa de divergencia de secuencias de 5 a 10 veces superior a la encontrada en scnDNA (Brown et al., 1979 y 1982). Sin embargo, esta característica contrasta con la baja variabilidad deducida a partir de su herencia materna y la falta de recombinación, a lo que se suma que es una molécula especialmente compacta con pocas secuencias intergenes como los intrones, y exceptuando la RNC, presenta pocas inserciones o duplicaciones (Brown et al., op.cit.). No obstante, se ha demostrado que durante una fase inicial rápida de divergencia, el mtDNA acumula sustituciones nucleotídicas a una tasa de 0.5-1.0% por linaje por millón de años, la cual es considerablemente más rápida que el promedio para scnDNA (Wilson et al., 1985; Moritz et al., 1987). Una deficiencia de los mecanismos de reparación ha sido sugerida como base para explicar la mayor tasa de sustituciones nucleotídicas en el mtDNA respecto al nDNA en vertebrados (Brown, 1983; Cann et al.; 1987; Moritz et al., 1987). Estas razones justifican que exista una considerable variabilidad entre los diferentes taxa respecto a la tasa de evolución relativa y absoluta del mtDNA (Britten, 1986; Moritz et al., 1987a), por lo que un reloj molecular universal sobre la base del mtDNA no puede ser asumido (Harrison, 1988). La tasa de evolución de secuencias varían a lo largo de la molécula de mtDNA e.g., las secuencias de los genes que codifican para los RNA ribosomales grandes y pequeños son altamente conservadas, mientras que en la RNC se acumulan rápidamente las sustituciones de base y los reordenamientos (Brown, 1983; Moritz et al., 1987). Debido a esta alta variabilidad intramolecular, la comparación de secuencias de diferentes regiones de la molécula de mtDNA puede proveer información muy útil para analizar diferentes niveles de divergencia desde la escala más básica, la población, hasta categorías taxonómicas más integradoras. Para taxa recientemente divergentes la sustitución de REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 15 bases ha ocurrido primariamente en las secuencias intergenes, en la RNC o en sitios dentro de la región codificadora que no resulten en un reemplazamiento de aminoácidos (Brown, op.cit.; Moritz et al., op.cit.). Por consiguiente, la variación intramolecular del mtDNA también adquiere importancia notable desde la perspectiva de la conservación de los recursos bióticos porque una reducción drástica de ésta podría identificar causas recientes que afectan el equilibrio de la naturaleza como el creciente impacto antrópico y facilitar la toma de decisiones efectivas (Allendorf & Luikart, 2007). El análisis de los genotipos de mtDNA dentro y entre poblaciones ha proveído una información importante para el estudio de la estructura poblacional, la dispersión, la variación geográfica, la filogenia y la zoogeografía (Avise, 1986; Moritz et al., 1987). En adición, la tasa de mutación relativamente rápida de la RNC ha permitido la resolución de poblaciones a escala fina, pues ayuda a discriminar pequeñas diferencias genéticas que han ido apareciendo en el tiempo evolutivo. Todo lo cual ha viabilizado las reconstrucciones de filogenias (Encalada et al., 1996) y de eventos biogeográficos (Bowen et al., 1992). Por esta razón, la secuenciación de la RNC ha sido convertida en una de las opciones como método para la definición de poblaciones con mtDNA (Harrison, 1988). No obstante, este marcador tiene la limitación de no incluir la variación aportada por la clase sexual macho en la generalidad de los taxa, por lo que una combinación con marcadores nucleares multilocus sería ideal si se trata de evaluar la población y procesos demográficos en su conjunto (Allendorf & Luikart, 2007). II.3 Marcadores moleculares: herramientas útiles para la conservación. La determinación del nivel población y los modelos evolutivos en una especie son esenciales para la conservación de la diversidad biológica (Allendorf & Luikart, 2007). La identificación y protección de poblaciones locales intraespecie constituyen foco esencial para la conservación porque ayudan a maximizar el potencial y minimizar los riesgos de extinción (Hughes et al., 1997; Hilborn et al., 2003; Luck et al., 2003). Por consiguiente, la población local a menudo es considerada la unidad funcional del ecosistema (Allendorf & Luikart, 2007). Es por esto, que de la correcta delimitación del nivel poblacional dependerá, en gran medida, el éxito de los programas de manejo y monitoreo (Allendorf & Luikart, op.cit.). Dentro de una metapoblación, la conservación suele ser aún más REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 16 compleja por la existencia de otros niveles como las unidades evolutivas de significación (ESUs, Ryder (1986)) o segmentos poblacionales distintos (DPS) (Allendorf & Luikart, 2007), a menudo reconocidos con estatus taxonómico de subespecie. Cuatro niveles de biodiversidad se reconocen como básicos para la conservación: genes, poblaciones, especie y ecosistema (Hughes et al., 1997; Luck et al., 2003). Mientras la especie revela los patrones históricos de un pasado evolutivo, el ecosistema soporta la biota en el presente y los genes representan la garantía de la biodiversidad en la tercera escala temporal o futuro (Bowen, 1999). Por consiguiente, la conservación de poblaciones genéticamente diferentes es imprescindible para garantizar la supervivencia de especies y el funcionamiento del ecosistema no sólo en el presente sino para el futuro (Luck et al., 2003). La descripción de las relaciones genéticas intraespecies y la reconstrucción de las filogenias son cruciales en el diseño de una estrategia de conservación (Allendorf & Luikart, 2007), dando lugar a la identificación de las ESUs aplicado a un conjunto de poblaciones que meritan ser atendidas en un aparte y de forma prioritaria dentro de una estrategia de conservación a partir de reconocerse un aislamiento y divergencia adaptativa. Parecido a las ESU, las unidades de manejo (MUs) no muestran evolución independiente ni fuerte divergencia adaptativa a pesar de que las poblaciones o demos que integren cada una estén estructurados significativamente entre sí (Moritz, 1994). Por consiguiente, la MU se convierte en una entidad más manejable en términos prácticos que la ESU, porque espacialmente es considerablemente más pequeña y a menudo, pertenece a la jurisdicción política-administrativa de un país o de unos pocos entre los cuales existen relaciones fronterizas para la protección de la integridad de cada nación por lo que los planes de manejo suelen viabilizarse i.e., dos ESU son reconocidas para C. mydas, una en el Atlántico y la otra en el Indopacífico (Karl & Bowen,1999). Dentro de cada una de estas ESU se han reconocido más de diez Mus, con frecuencia asociadas a áreas de anidación pertenecientes a un país. REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 17 II.4 Descripción de la RNC del mtDNA. II.4.1 Chelonia mydas. El genoma mitocóndrico de tortugas en general se ha caracterizado por bajas tasas de evolución y diversidad inusuales con respecto a los demás vertebrados (Avise et al., 1992). En el caso particular de C. mydas, Encalada et al. (1996) han reportado una tasa de cambio evolutivo para la región no codificadora del mtDNA de 0.012 – 0.024 sustituciones por locus por millón de años, basados en el reloj molecular calibrado con RFLP de 0.2 – 0.4% de divergencia por millón de años para Testudines (Avise et al., 1992a; Bowen et al., 1993). La RNC de C. mydas tiene una longitud de ca. 981 pb aproximadamente, flanqueada por los genes de tRNA treonina (Thr) y prolina (Pro) (Kumazawa & Nishida, 1999) (Fig. 1). Contiene una región hipervariable de aproximadamente 640 pb, tres secuencias conservadas: CSB-1, CSB.2 y CSB-3 de 23, 15 y 17 pb respectivamente, así como un grupo de repeticiones del dinucleótido TA, que abarca alrededor de 50 pb. tRNA-Thr→ ATT CTAGTAGCTT AACCCTAAAG CATTGGTCTT GTAAACCAAA GATTGAAAAC GGACTTAAAC CTTCATCCCC GGTCCCCAAA 1 ACACAGGAA± ±±AAGTGTCC A±CACAAACT 61 GAACAATACC CGCAATACCT ATCTATGTAT 121 TATGACCAGT AATGTTAACA GTTGATTTGG 181 T±AAATATTT TAACAACATG AATATTAAGC 241 TAAAATTAAA CTATTATACT CAACCATGAA 301 TAGCTATTCA CGAGAAATAA GCAACCCTTG 361 CCCATTCAGT CTGTGGCGTA CATAATTTGA 421 CACATACAAG TAACGACGTT CATTCGTTCC 481 CTATACATTA AATTTATAAC CTGGCATACG 541 TCTCTCTGTG TTCTCAGGCC CACATAACTG 601 CGTTTAAATA TGATTGGCCG TGCAAACTGA 661 GACATAGAAT TTCACAATTA AACCTAAACA 721 TACTTTTTAG CTAAACCCCC CTACCCCCGT 781 TACTTCTCGC CAAACCCCTA AATCCGAGAC 841 AGCATCACAC AAATCAATAG GATACTTACA 901 AACACCTCTA CCACACCTCA ACCAATATAT 961 ATTATATATA TTATATATAT AATATGTTAT 1021AAGATGCCAA GATGGGTAAT AAACATACCC TATAACCTTC CTAGAATAAT ACCGGAATCC TATAATTAAA AACTACCTAA ATTCTCTGCC TATTGTACAT CTACTTATTT CCCTAAACAT ±AAAAATCAT AGAGGATTAA AAGTGAAATG TATCGTCACA GTAATTGGTT TTAGTAAGAT ACAACATTAC TCTATTCTGG CCTCTGGTTA CCTTTAAAAG GCCTTTGGTT GTAGTTTTAC TTGCATATAG ATACCTGCCG ATTCAGTGAA TTAATGGTAT TATTAAGTTA ATGATCTACA ACCTAACTCA TAAAGTCAAC ACCAGCCCGC TGACCAAACT GACATAATAT CTAATATTTA AAAAGTACTA ATATATATTA TACATTATAT TGTAGCTTAT TATATTAAAG CAAAAACA →tRNA-Phe CAAAAGAGAA←tRNA-Pro CTATCCTTTG GTGCCCAACA ACCAATAGCA TGAATTTACA ACATAGGACA ATTTCCTAAA CAGTTTCAAG GTTTTTCAGG G±AATGAGTT TAGTTTTTTT ACTGGACTTA ATGCTTATAA TTATTAACTG TATAGCCATT CAACTGCATA TACAATTCAA ATATATATAT CACGGCACTG Fig. 1. Secuencia nucleotídica de la RNC del mtDNA en C. mydas, flanqueada por genes de tRNA, con de 78 SPs según compilación de resultados de: Allard et al. (1994); Lahanas et al. (1994, 1998); Norman et al. (1994); Encalada et al. (1996); Dutton et al. (1996); Bjorndal & Bolten (1988). La numeración de los sitios polimórficos se realiza tomando como referencia la base primera base (1) de la RNC del mtDNA según Kumazawa & Nishida (1999). Secuencias repetidas (TA). .. SP ± indel (analizado como un SP). REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 18 II.4.2 Caretta caretta. La RNC del mtDNA de C. caretta varía en longitud desde 1230 hasta 1630 pb aproximadamente (Fig. 2), flanqueada por los genes de tRNA prolina (Pro) y fenilalanina (Phe) (Laurent et al., 1998). Contiene los principales dominios conservados, i.e. las tres secuencias CSB-1, CSB-2 y CSB-3 y la secuencia putativa TAS, y una región repetitiva (TATATTATAT) en el extremo 3’. Esta repetición varía en una longitud de 300 a 700 pb y condiciona la heterogeneidad en la dimensión de la RNC (Laurent et al., op.cit.). Considerando la tasa de cambio evolutivo para la RNC del mtDNA de C. mydas (0.012 – 0.024 sustituciones por locus por millón de años) según Avise et al. (1992), Bowen et al. (1993) y Encalada et al. (1996), se han analizado los tiempos de divergencia de secuencias de C. caretta (Encalada et al., 1998). 1 9 69 129 189 249 309 369 429 489 549 609 669 729 789 849 909 tRNA–Pro→ A TCAAAANNNN CCTTCATCTC CGGTCCCCAA AACCGGAATN CTTCCAATTA AACTACCCTT GAAGCGCCAA CATGTAAATT TACCTATATT CTCTGCCGTG CCCAACAGAA AATACCTATC TATGTATTAT CGTACATCAA CTTATTTACC ACTAGCATAT GTTGTCGATT AATCTGACCT TAAACATAAA AACTTATTAA TTTTGCATAA TTACATGACT ATTATACAGG TAATAGGAAT GAAATGATAT AGGACATAAA TATTCTCAAC CATGAATATC GTTACAGTAA TAGGTTATTT CTTAGTTCAG GAAATAAGCA ATCCTTGTTA GTAAGATACA ATATTACCAG TTTCAAGTCC GTCGTACATA ACTGATCTAT TCTGGCCTCT GGTTGGTTTT TTCAGGCACA AAAGTTCATT CGTTCCTCTT TAAAAGGCCT CTGGTTGCAA GTAAATGAGT AAATTTATAA CCTGGCATAC GGTGGTTTTA CTTGCATGTG GTAGTCTTTT GTGTTCTCAG GCCCACATAA CTGATACCTG CCGAATTGAT GAAACTGAGC AATGATTGGC CGTGCAGAAT ACTTAATGGT ATTATTTAAT TAATGCTTTT TTTTTATAAA AACTCACAAC AGTTATTTAC AAGCTAAAAC CCATTACAAC AGTTAAACCC CCCCACCCCC ATAAACTAAC ATTATGCCCG AATAGCTATT CAAACCCCTA AATCCGAGAC TAACTAAACT GACACAACAT TAATCGCATA AAACTAATGA AACACTTACA CTAATACCTA AAAAGTACTA AAAACAATTC TACTACACCC AACTAACCAA ACATTATATT ATATATACAA TGTTATTGTA AAGCACGGCA CTGAAGATGC CAAGATGGGT AAACACACA →tRNA–Phe AGGGTTCAAA TGACGCAAAA TAATATCCAT GATCAGTAAT ACTGTTTTAG ATTAAACCAT CTCATCACGA ATTAAGTCAT TTAAGGCAGT TCTATACATT TTTTCTCTTT CTACGTTCAA AGGACATATA CATACTTTTT CACTTCTCGT AGCATTACAC ATCACACCTC GCTTATCACA Fig. 2. Secuencia nucleotídica de la RNC del mtDNA de C. caretta, flanqueada por genes de tRNA, con un total de 56 SPs según compilación de los resultados de Bolten et al. (1998), Encalada et al. (1998), Laurent et al. (1998), Dutton et al. (1996), Bowen et al. (1995). La numeración de los sitios polimórficos se realiza tomando como referencia la base primera base (1) de la RNC del mtDNA según Laurent et al. (1998). Secuencias repetidas (TA). .. SP ± indel (analizado como un SP). REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 19 II.5 Estructura poblacional en el Atlántico. II.5.1 Chelonia mydas. Allard et al. (1994) realizaron un análisis de las colonias de anidación de la Florida (USA) y Costa Rica, mediante técnicas de secuenciación de la RNC del mtDNA, con el propósito de determinar cuán distintas eran estas dos colonias en términos de genealogía de mtDNA, como predice la conducta de reproducción en el lugar de origen. Encontraron 3 SPs, que definieron cuatro haplotipos de los cuales dos fueron exclusivos de la Florida, y otro de Costa Rica. El cuarto fue compartido por ambas poblaciones. Las frecuencias haplotípicas de estas colonias fueron significativamente diferentes. La colonia de anidación de la Florida resultó ser cinco veces más polimórfica y de mayor diversidad nucleotídica que la de Costa Rica. Sin embargo, las variaciones intrapoblacionales fueron solamente del 0.01% en la primera, sugiriendo un ancestro común relativamente reciente, lo cual se opone a lo predicho por la teoría de la neutralidad en relación al tamaño actual de las poblaciones. Por lo que los autores puntualizaron que cualquier explicación con relación a este fenómeno debe tener implícito el tamaño de la población de anidación de la Florida (350 a 400 ovíferas), así como el tiempo relativamente corto transcurrido desde el establecimiento de esta población en el área (menor de 10 000 años). Lahanas et al. (1994) haciendo uso de la secuenciación de la RNC del mtDNA examinaron la evolución molecular y la genética poblacional de C. mydas en el “Gran Caribe”. Reconocieron seis haplotipos definidos a partir de 12 SPs. Las colonias de Isla de Aves (Venezuela) y Surinam presentaron un haplotipo común. Las cuatro poblaciones estudiadas se subdividieron en los grupos Occidental y Oriental (Costa Rica/ Florida e Isla de Aves/ Surinam respectivamente), puesto que se obtuvieron diferencias significativas entre las frecuencias haplotípicas de todos los pares de colonias, excepto para las comparaciones entre estos dos grupos de colonias citadas anteriormente. La diferenciación entre las poblaciones del este y el oeste fue por lo menos seis veces mayor que dentro de cada región. Además, las dos regiones estuvieron caracterizadas por tasas de migración (0.1-0.2 migrantes/ generación) menores que las requeridas para contrarrestar los efectos de la deriva genética (Barton & Slatkin, 1986; Slatkin & Barton 1989). REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 20 Encalada et al. (1996) emplearon la secuencia de la RNC del mtDNA para dilucidar la estructura genética y la filogeografía en colonias de anidación de C. mydas del océano Atlántico y el mar Mediterráneo. Encontraron veinte polimorfismos en 19 SPs, que definieron 18 haplotipos. Las frecuencias haplotípicas de las colonias de México y la Florida, Surinam e Isla de Aves, Isla Ascensión y Guinea Bissau no mostraron diferencias significativas, lo cual fue interpretado como resultado de un intercambio de genes continuo entre estos pares de colonias o consecuencia de un aislamiento reciente. Sin embargo, entre estos pares los autores encuentran sendas diferencias significativas, lo cual les permite reconocerlos como poblaciones de anidación con independencia demográfica. II.5.2 Caretta caretta. Encalada et al. (1998) realizaron una prospección genética de C. caretta de áreas de anidación atlánticas y mediterráneas, secuenciando la RNC del mtDNA (5’- 380 pb), para dilucidar la estructura poblacional y patrones filogeográficos de la especie. Analizaron las mismas diez áreas de anidación prospectadas genéticamente con RFLP por Bowen et al. (1994): Quintana Roo, México; Bahía, Brasil; Bahía Kiparissia, Grecia; USA: noroeste de la Florida, Georgia, Carolina del Sur y Carolina del Norte, sureste y suroeste de la península de la Florida). Comparando la composición haplotípica por pares de áreas/ colonias de anidación a través de la Prueba X2, los autores concluyeron que en las cuencas atlántica y mediterránea hay seis cohortes (unidades) demográficas independientes: México; Brasil; Grecia; USA: noroeste de la Florida, sur de la Florida, y desde el noreste de la Florida hasta Carolina del Norte. La existencia de una discontinuidad filogeográfica entre áreas de anidación del noreste de la Florida hasta Carolina del Norte y sur de la Florida, evidenció una ruptura entre Cabo Cañaveral y Jacksonville (Encalada et al., 1998). Los autores plantearon que esta ruptura concordó con las evidencias: 1) estudios de campo han indicado una relativa continuidad en las anidaciones de C. caretta a lo largo de la costa sur de la Florida, desde Dade hasta Brevord Counties, decreciendo la densidad de los nidos hacia Cabo Cañaveral; y 2) semejante disrupción filogeográfica ha sido encontrada para otros organismos costeros (Avise, 1992; Karl & Avise 1992; Lamb & Avise 1992). A pesar de que las comparaciones genéticas entre el área de anidación noroeste de la Florida y las colonias de anidación que integran la unidad noreste de la Florida hasta Carolina del Norte no mostraron significación, Encalada et al. (1998) concluyeron que ambas áreas de REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 21 anidación constituían unidades demográficas independientes basado en: 1) la alta significación de la comparación entre éstas (X2=17.53; p<0.001), i.e. sumando la composición haplotípica de las colonias de anidación en la unidad noreste de la Florida hasta Carolina del Norte, y 2) la diferenciación genética significativa de estas unidades, distantes geográficamente, con las áreas de anidación vecinas de la “unidad demográfica” (población de anidación) sur de la Florida. Encalada et al. (op.cit.) reafirmaron esta conclusión con la aplicación de un Análisis de la Varianza Molecular jerarquizado en el cual se atribuye el 64% de la variación total a las diferencias significativas entre las seis unidades demográficas genéticamente definidas a partir de un limitado número de migrantes intercambiados. Por otra parte, Encalada et al. (op.cit.) plantearon que la baja diversidad genética entre las colonias de anidación localizadas en la unidad noreste de la Florida hasta Carolina del Norte proporciona una resolución inadecuada para detectar divisiones poblacionales, siendo probable la existencia de una división demográfica dentro de esta región. Finalmente Encalada et al. (op.cit.) plantearon que la estructura poblacional encontrada en su estudio concordó con el patrón predominante revelado por los estudios de marcaciónrecaptura (Bowen et al., 1993). Encalada et al. (1998) puntualizaron que la transferencia de algunas ovíferas entre playas de anidación se refleja quizás en los altos estimados de razones de migración entre colonias de anidación. Mientras la conducta de reproducción en el lugar de origen predomine, estas relocalizaciones raras podrían indicar una habilidad para colonizar nuevos hábitat en una gran escala de tiempo evolutivo y una condición de desequilibrio resultado de un reciente evento de colonización, o unas de éstas variantes (Encalada et al., op.cit.). Laurent et al. (1998) realizaron una prospección genética de C. caretta en áreas de anidación en el Atlántico Norte y en el mar Mediterráneo, empleando la RNC del mtDNA (5’- 420 pb), para estimar la estructura poblacional de las colonias de anidación de las costas del mar Mediterráneo y evaluar la contribución de éstas a los agregados de pesquería. Ellos analizaron las colonias de anidación de USA: Georgia/ Carolina, y sur de la Florida, mar Mediterráneo: Grecia, Chipre, Turquía y cuenca del sureste. Laurent et al. (op.cit.) integraron las muestras provenientes de colonias de anidación de Libia, Italia e REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 22 Israel en la unidad cuenca del sureste, como consecuencia de los pequeños tamaños muestrales de cada una y de estar ubicadas geográficamente en la misma cuenca. Empleando la probabilidad de Fisher por pares de colonias de anidación y el Φ ST, los autores encontraron que las colonias de anidación de las costas del Atlántico occidental y del mar Mediterráneo estaban significativamente estructuradas, excepto las colonias de anidación del sur de la península de la Florida y Grecia, donde el Φ ST fue no significativo. Según los autores, estos resultados se ratificaron con la significación de un AMOVA jerarquizado entre las regiones de anidación americana (Georgia/ Carolina y sur de la Florida) y del mar Mediterráneo (Grecia, Chipre, Turquía y cuenca del sureste). El 41% de la variación genética total ocurrió entre grupos ( Φ CT =0.41; p<0.001), lo cual indicó la existencia de dos agregados diferentes de C. caretta. En las costas del mar Mediterráneo existieron dos agrupaciones ( Φ ST =0.33; p<0.001), una integrada por la generalidad de las colonias de anidación y la otra por Turquía (Laurent et al., 1998). Dentro de la cuenca del Mediterráneo, el 33% de la variación total fue encontrado entre las áreas de anidación, lo cual indicó una estructuración poblacional significativa. Los autores plantearon que la falta de divergencia genética entre las áreas de anidación de Chipre, Grecia y cuenca del sureste necesariamente no tiene que implicar panmixia debido a que diferencias potenciales pudieron permanecer indetectables, como consecuencia de las limitaciones del tamaño muestral o la pérdida de resolución del marcador. Además, Laurent et al. (op.cit.) informaron que las diferencias significativas en los tamaños de ovíferas de diferentes áreas de anidación de Chipre, Grecia y Libia según Laurent et al. (1995) y Broderick & Godley (1996), soportan fuertemente la estructuración poblacional. Laurent et al. (1998) destacaron que dentro de las áreas de anidación es posible que ocurra diferenciación poblacional entre sitios de anidación adyacentes, teniendo en cuenta los resultados obtenidos en Turquía por Schroth et al. (1996), mediante la aplicación de análisis sobre la base de marcadores nucleares y mitocondriales. Francisco et al. (1999) dilucidaron la identidad poblacional de las colonias de anidación de Volusia County. Estos autores compararon las frecuencias haplotípicas determinadas en la RNC del mtDNA (5’- 380 pb) de las colonias de anidación norte y sur de Jetty respectivamente, a través de la Prueba X2. Francisco et al. (op.cit.) concluyeron que REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 23 ambas colonias de anidación pertenecen a una misma población de anidación, considerada como una unidad de manejo independiente. Posteriormente, esta población (sumando las contribuciones de las colonias de anidación norte y sur de Jetty) fue comparada con las unidades demográficas independientes determinadas en Norteamérica por Encalada et al. (1998), para contribuir a la definición del límite de la estructura genética de la costa este de la península de la Florida (poblaciones de anidación sureñas y norteñas). Francisco et al. (1999) concluyeron que la estructura genética entre las unidades de manejo independientes Volusia y sur de la Florida es dudosa (X2=10.44, p=0.042). Francisco & Bowen (2001) realizaron un análisis de genética poblacional con las colonias de anidación de la península de la Florida sobre la base del estudio de secuencias de la RNC del mtDNA (5’- 380 pb) para evaluar la estructura de las colonias/ áreas de anidación de la península y cayos adyacentes, i.e. Dry Tortugas. Ellos diseñaron los análisis estadísticos teniendo en cuenta la existencia de barreras biogeográficas latitudinales tanto en la costa oeste como en la este de la península de la Florida según Avise (1992) y Briggs (1974), y entre el oeste y este del sur de la propia península según Muss et al. (2001). En el caso particular de la población de anidación noreste de la Florida hasta Carolina del Norte, Francisco & Bowen (2001) incluyeron a las colonias de anidación de Volusia County (muestra sumada) sobre la base de su ubicación al norte del límite biogeográfico de cabo Cañaveral según Briggs (1974) y Avise (1992). Francisco & Bowen (2001) discutieron la incertidumbre en la determinación de la posición de Volusia, al norte o al sur de la península, debido a que cuando se compara Volusia con Amelia Island no se obtuvo significación de la Prueba X2 al igual que con Melbourne Beach. Por lo que los autores plantearon que como consecuencia de que Volusia presenta haplotipos con frecuencias intermedias es posible que no pertenezca a ninguna de las dos unidades de manejo. Francisco & Bowen (op.cit.) incrementaron los tamaños muestrales de algunas colonias de anidación peninsulares analizadas previamente por Encalada et al. (1998), i.e. Melbourne Beach y Sarasota County, esgrimiendo que el tamaño muestral del estudio anterior fue un factor que pudo limitar el poder estadístico para detectar divisiones poblacionales entre las áreas de anidación. Al estudio se sumó la colonia de anidación peninsular de Cabo San Blas (Francisco & Bowen, 2001). Finalmente ratificaron las REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 24 unidades demográficas propuestas por Encalada et al. (1998), agregando otra integrada por las colonias de anidación de Dry Tortugas. II.6 Diversidad genética en el Atlántico. Chelonia mydas y Caretta caretta. En la generalidad de los estudios llevados a cabo en la cuenca atlántica con C. mydas, se han encontrado altos valores de diversidad genética del mtDNA para las colonias de anidación con menores tamaños censales de hembras, i.e. las colonias de anidación de la península de la Florida (Allard et al., 1994; Lahanas et al., 1994), Isla de Aves (Lahanas et al., op.cit.; Encalada et al., 1996) y México (Encalada et al., op.cit.). Este resultado ha contrastado con los menores valores de diversidad encontrados para las colonias de anidación con mayores tamaños censales de hembras, i.e. las colonias de Costa Rica (Allard et al., 1994; Lahanas et al., 1994; Encalada et al., 1996) y Surinam (Lahanas et al., op.cit.; Encalada et al., op.cit.). Lahanas et al. (op.cit.) plantearon que los menores valores de diversidad genética caracterizan a las grandes poblaciones, contrario a lo que ocurre con las poblaciones pequeñas. En éstas la alta diversidad se debe a dos causas diferentes: en el primer caso, a la existencia de un haplotipo único que se diferencia del resto en once mutaciones y en el segundo, a la existencia de varios haplotipos que difieren entre sí solamente en una o dos mutaciones. Para explicar la alta diversidad genética en poblaciones de anidación de pequeño tamaño y en el caso particular de la colonia de anidación de la península de la Florida, Allard et al. (1994) propusieron tres hipótesis. La primera planteó un aumento en la tasa de mutaciones del mtDNA en la población, lo que se contrapone con los bajos coeficientes de evolución y diversidad del mtDNA en las tortugas respecto al resto de los vertebrados (Avise et al., 1992). La segunda hipótesis se basó en que la mayor diversidad es el resultado de la mezcla reciente o todavía en proceso, de hembras provenientes de otras poblaciones del Atlántico occidental que ha podido facilitarse por el patrón de corrientes oceanográficas locales y geomorfología de la propia península. Estas alteraciones dentro de la conducta filopátrida son esenciales para la fundación de nuevas colonias en ambientes cambiantes. En la tercera hipótesis se consideró que la colonia de la Florida derivó de una población ancestral mayor. De acuerdo con la misma, los niveles actuales de diversidad del mtDNA reflejan una población históricamente grande en las vecindades de la colonia actual. Los registros REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 25 históricos de grandes poblaciones en las cercanías de la península de la Florida, por ejemplo en localidades del Caribe como las ubicadas en Islas Caimán y Dry Tortugas (Parsons, 1962), han apoyado la existencia de esta supuesta gran población (Allard et al., 1994). Finalmente Allard et al. (1994) señalan la importancia para la conservación del genofondo de las colonias de anidación de la Florida en el plan de manejo de la especie. La alta diversidad genética de la colonia de anidación de la Isla de Aves ha sido explicada por la hipótesis de la mezcla (Lahanas et al., 1994), evidenciada por la presencia de dos haplotipos correspondientes cada uno a los linajes de C. mydas de la cuenca atlántica, los cuales se compartieron con las colonias de anidación de la región. En correspondencia, Lahanas et al. (op.cit.) llegaron a la conclusión de que la colonia de esta área tiene una antigüedad superior a los 300 000 años, lo cual es poco probable debido a la inestabilidad de estas islas. Por tanto, estos autores valoraron como más probable la hipótesis de que el origen de los haplotipos fue a partir de una mezcla entre los linajes “occidental y oriental” [linajes de la cuenca atlántica]. Ellos acentúan que la mayor diversidad genética en el par de colonias de anidación con menores tamaños poblacionales resulta contradictorio con la teoría de neutralidad génica (Kimura, 1983). Ellos reconocen que existen fallos a la conducta de reproducción en el lugar de origen (Carr, 1967 y 1975) [Hipótesis de mezcla], los cuales tienen mayores probabilidades que ocurran de colonias de anidación grandes hacia otras de menor tamaño, donde el impacto de la inmigración ocasional es mayor. También señalan que la baja diversidad genética puede ser consecuencia de efectos fundadores o de cuellos de botella como plantean Nei & Graur (1984) y Avise et al. (1988). Respecto al efecto fundador, encuentran fundamento en que mientras la población crece exponencialmente, las mutaciones se acumulan aritméticamente a muy baja tasa lo cual tiene un efecto connotado sobre la diversidad genética del mtDNA de C. mydas considerando largos tiempo generacional y ciclo reproductor, y relativa baja tasa de mutación del marcador en cuestión. En la especie C. caretta, los valores de diversidad genética del mtDNA para las colonias de anidación del Atlántico, son comparables, aunque ligeramente más bajos que aquellos informados para C. mydas y Eretmochelys imbricata (Bass et al., 1996; Encalada et al., 1998). Particularmente en la península de la Florida, la diversidad nucleotídica dentro de REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 26 poblaciones ha sido alta (Francisco & Bowen, 2001), como consecuencia de tener representados dos linajes filogenéticamente diferentes (Encalada et al., 1998). Sin embargo, el intervalo de la diversidad haplotípica en las poblaciones de anidación de la península de la Florida ha sido muy variable: desde 0 hasta 0.714 (Francisco & Bowen, 2001). II.7 Filogenia y Filogeografía. II.7.1 Chelonia mydas. Chelonia comúnmente ha sido considerado un género monotípico integrado por C. mydas. Sin embargo, producto de la variación de la talla, forma y coloración de los individuos según la región de distribución, han sido determinadas subespecies (Carr, 1975; Pritchard & Trebbau, 1984; Márquez, 1990): C. m. viridis en el Caribe, C. m. mydas en el Atlántico Sur, C. m. japonica en el Índico-Pacífico Oeste, C. m. carinegra en el Golfo de California, y C. m. agassizi en el Pacífico Este. Para algunos autores la tortuga del Pacífico C. m. agassizi constituye una especie. No obstante, no hay suficientes evidencias que argumenten esta designación e incluso algunas la contradicen (Mrosovsky, 1983; Bowen & Karl, 1997). Estudios genéticos recientes con mtDNA han fundamentado la existencia de linajes de C. mydas dentro de los sistemas Atlántico – Mediterráneo e Índico-Pacífico, pero no han argumentado el estatus taxonómico de C. m. agassizi (Bowen et al., 1992, 1993). Bowen et al. (1992), utilizando técnicas de RFLP para dilucidar la filogeografía global de Chelonia, encontraron separación filogenética entre las dos mayores cuencas oceánicas: El océano Atlántico y el mar Mediterráneo, y los océanos Índico y Pacífico. Todos los individuos en estos dos grupos fueron separados por cinco o más cambios por sitios de restricción. Los autores plantearon que ese patrón genético es consistente con los límites climáticos y geográficos que comúnmente definen la distribución de C. mydas, y que las poblaciones de estas dos regiones probablemente estuvieron separadas por las condiciones de bajas temperaturas que aún persisten alrededor de los extremos sur continentales de África y especialmente de América, considerando que no existen actualmente barreras físicas para el movimiento dentro de las cuencas oceánicas Atlántico – Mediterráneo o Indo-Pacífico. Para esta especie no es suficiente la influencia de la corriente de Agujas proveniente del Ecuador (Tomczack, 2000) por el sureste de África REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 27 que contrarresta el efecto de la corriente circumpolar Antártica. El efecto de la corriente cálida sobre la fría ha condicionado el paso de varias especies marinas desde el océano Índico hasta el Atlántico (Briggs, 1974). Encalada et al. (1996) realizaron un estudio para resolver la filogeografía de C. mydas en el Atlántico y el Mediterráneo utilizando la secuenciación de la RNC del mtDNA. Ellos encontraron dos linajes en el Atlántico, uno correspondiente al oeste del Caribe y el Mediterráneo y el otro que incluye el este del Caribe, el Atlántico Sur y las colonias del oeste de África [un linaje se refiere a mediterráneos americano – Viejo mundo, y el otro, a Atlántico Sur]. Ambos linajes estuvieron separados por al menos seis mutaciones, mientras que los haplotipos dentro de cada grupo difirieron en un máximo de cinco. Los autores puntualizaron que el caso de intercambio de haplotipos entre los dos principales grupos filogeográficos por un simple individuo de México e Isla de Aves, podría indicar el bajo nivel de intercambio genético entre regiones diferentes geográficamente, ilustrando la estructura dinámica de las colonias de C. mydas en escalas de tiempo evolutivo. Basados en el reloj molecular de RFLP provisional (Avise et al., 1992; Bowen et al., 1993) y utilizando la información sobre el incremento en la divergencia de la RNC del mtDNA encontrado en su estudio, Encalada et al. (1996) calcularon una tasa de evolución de 0.012-0.024 sustituciones por locus por millón de años para la misma. Sobre la base de esta información propusieron un tiempo de divergencia estimado para los linajes de C. mydas de la cuenca atlántica en ca. 0.3-0.7 millones de años, y la divergencia con respecto al grupo externo del Pacífico en aproximadamente 1.8-3.7 millones de años. Además señalaron que sus resultados y los obtenidos por Bowen et al. (1992) indicaron que las colonias de anidación de C. mydas del Atlántico y las del Pacífico se aislaron probablemente durante el Pleistoceno temprano (ca. 2.0 MA atrás). Encalada et al. (1996) propusieron que la genealogía obtenida en su estudio permitió discernir entre dos posibles modelos para la distribución de los linajes del mtDNA en la cuenca atlántica. Uno explicó la distribución disyunta de organismos en términos de hábitat antiguamente continuo y separado por cambios geológicos o climáticos (escenario vicariante) y el otro enfatizó la habilidad de los organismos para dispersarse más allá de los límites tradicionales de su distribución histórica (escenario dispersionista). REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 28 Los hábitat de C. mydas se han restringido a zonas tropicales y subtropicales. En los tiempos de los episodios de máxima glaciación, el avance de la capa de hielo continental y la consecuente reducción del nivel del mar (aproximadamente 100 m por debajo del nivel actual (Bowen, 1978), redujeron probablemente los hábitat de forrajeo y de anidación para C. mydas, restringiéndolos a las zonas tropicales. Los registros climáticos para la glaciación de Wisconsin, i.e. última glaciación (18 000 años atrás), han indicado que el hielo marino se extendió al menos diez grados de latitud más cercanos al Ecuador del que lo está hoy día (Gates, 1993). Bajo estas condiciones de bajas temperaturas y elevada aridez, no existió disponibilidad de hábitat aptos para la anidación de C. mydas en los sitios de anidación contemporáneos de las latitudes altas del Atlántico donde se localizan las actuales colonias de anidación de las penínsulas de la Florida y Yucatán, así como de Chipre (Encalada et al., 1996). Las regiones ecuatoriales pudieron haber servido como refugios para la especie durante las glaciaciones, con subsecuentes colonizaciones en las altas latitudes durante los períodos interglaciales (Encalada et al., op.cit.). Por consiguiente, la dispersión a largas distancias durante los retiros glaciares (con los consecuentes aumentos de temperatura y la disponibilidad de alimento y condiciones de anidación), ha estado fuertemente implicada con la colonización de las colonias de anidación del oeste del Caribe y del Mediterráneo. Aprovechando estas condiciones durante los períodos interglaciales precursores de los haplotipos CM-A13 y 14 probablemente colonizaron el mar Mediterráneo, como ha sido indicado por los registros climáticos y las cercanas relaciones entre los haplotipos de Chipre y el Caribe (Encalada et al., op.cit.). Aunque la más reciente capa glaciar comenzó a retirarse hace aproximadamente 18 000 años, el evento Younger Dryas representó un fenómeno de enfriamiento global que ocurrió entre once y 10 000 años atrás (Engstrom et al., 1990; Kudrass et al., 1991; Gates, 1993). Por lo que los eventos de colonización al noroeste del Caribe y el Mediterráneo ocurrieron más probablemente dentro de los últimos 10 000 años. (Encalada et al., 1996). Dentro de la región ecuatorial precursores de haplotipos CM-A5, 6 y 7 pudieron haber colonizado la costa noreste de América del Sur (colonias actuales de Surinam y Guyana) (Encalada et al., op.cit.). Moll (1983) sugirieron que durante los tiempos de la disminución de los niveles del mar los recursos de forrajeo disponibles para C. mydas en Brasil e Isla REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 29 Ascensión se fraccionaron al norte y al sur, induciendo entonces la colonización de estas áreas. Aunque este escenario vicariancista puede considerarse para la colonización de las áreas de alimentación del norte durante las glaciaciones, la dislocación gradual de los agregados de alimentación no es probable que se tenga en consideración para la colonización de colonias distantes a altas latitudes (Encalada et al., 1996). Finalmente, considerando la información derivada de los estudios del mtDNA según Meylan et al. (1990), Bowen et al. (1992) y Encalada et al. (1996), se concluye que la filogenia matriarcal global de C. mydas ha sido moldeada por la separación de las cuencas oceánicas y por la conducta de reproducción en el lugar de origen. II.7.2. Caretta caretta. La reciente historia evolutiva de C. caretta en el Atlántico y el Mediterráneo ha estado fuertemente vinculada a la historia climática y geológica de la región (Bowen et al., 1994). Con la técnica de RFLP aplicada al marcador mtDNA se han reconocido dos linajes, separados por una distancia nucleotídica neta dA=0.009 la cual permitió estimar un tiempo de divergencia promedio de 3 MA (Bowen et al., op.cit.). Ambos linajes estuvieron representados en los océanos Atlántico e Índico, mientras que en el Pacífico sólo se encontró uno. La estructuración entre la generalidad de las colonias de anidación ha sugerido un connotado aislamiento entre las cuencas oceánicas en la escala ecológica del tiempo (Encalada et al., 1998). Sin embargo, en la escala evolutiva han estado conectadas. Durante los períodos fríos que se sucedieron en el Pleistoceno, las poblaciones de la especie en cada océano quedaron aisladas geográficamente (Bowen et al., 1994). El linaje representado por los haplotipos CC-A1-3 y 6 evolucionó en la cuenca atlántica, mientras que el otro lo hizo en la indo-pacífica. Subsecuentes aumentos de la temperatura durante los períodos interglaciales facilitaron la expansión del hábitat para la especie hacia latitudes más altas, y el establecimiento de un corredor marino alrededor de Sudáfrica. La transferencia efectiva entre cuencas oceánicas a través del corredor también pudo facilitarse debido a que la especie posee amplia plasticidad para soportar aguas templadas con relación a las restantes tortugas marinas. REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 30 En estos períodos un precursor del haplotipo CC-A6 del linaje de la cuenca atlántica, pudo colonizar la cuenca índica, y un precursor de los haplotipos CC-A4 y 5 del linaje indopacífico pudo colonizar la cuenca atlántica sobre la base de que el primer linaje está ampliamente distribuido en el Atlántico y representado escasamente en el Indo-Pacífico por un solo haplotipo, y de que el segundo está ampliamente distribuido en el IndoPacífico y en el Atlántico con un haplotipo común y otro raro (Bowen et al., op.cit.). La baja diversidad de los linajes en las cuencas oceánicas invadidas indica que las colonizaciones ocurrieron probablemente en los últimos 20 000 años. La colonización del Mediterráneo debió ser aún más reciente (Bowen et al., 1994), después de concluir la última glaciación, debido a que el desarrollo embrionario sólo es posible a temperaturas mayores o igual que 25oC según Buckley et al. (1982). Sin embargo, la especie pudo establecerse durante los períodos glaciales en el sur de la Florida, no así en las actuales locaciones de Georgia y Carolina del Norte, donde el establecimiento definitivo debió ser concomitante con la colonización del mar Mediterráneo. Esto se corrobora con la baja diversidad genética de las colonias establecidas en estas latitudes como consecuencia de sucesivos cuellos de botellas (Bowen et al., 1993). Con la técnica de secuenciación aplicada al marcador RNC del mtDNA (380 pb) Encalada et al. (1998) reconocieron dos linajes en la cuenca atlántica. Ambos linajes se separaron por una d(A)=0.05, la cual permitió estimar un tiempo de divergencia promedio de 3.2 MA (similar al determinado por Bowen et al. (1994), teniendo en cuenta que la divergencia entre secuencias de la RNC es seis veces mayor que el análisis de RFLP sobre mtDNA (Encalada et al., 1998). El primer linaje está representado estrictamente en la cuenca atlántica, mientras que el otro lo está en las cuencas mediterráneas americana y del Viejo mundo. Inusualmente, haplotipos de linajes independientes anidan en la misma playa. Dada la estructuración significativa entre las colonias del sur y del nordeste de la península de la Florida hasta el estado de Carolina del Norte, fue encontrada una discontinuidad filogeográfica la cual ha sido reportada para otras especies (Avise, 1992; Karl & Avise, 1992; Lamb & Avise, 1992). Durante las glaciaciones del Pleistoceno los linajes pudieron sobrevivir en latitudes ecuatoriales, con climas más estables (Encalada et REVISIÓN BIBLIOGRÁFICA_________________________________________________________________________________ 31 al., 1998). Subsecuentemente, durante los períodos interglaciales la especie pudo conquistar latitudes más altas. Un linaje ecuatorial, precursor del haplotipo A, pudo haber colonizado las latitudes más norteñas como por ejemplo, las caribeñas. Desde esta área, consecuentes colonizaciones pudieron ocurrir hacia las costas occidental y oriental de la Florida, y al norte, donde la severidad climática pudo ser la máxima responsable de sucesivos cuellos de botellas que se evidencian con la baja diversidad genética de las colonias de anidación actualmente establecidas en estas latitudes. Por otra parte, Encalada et al. (op.cit.) plantearon que una trasplantación del precursor del haplotipo B hacia el Atlántico occidental también pudo ocurrir a partir de un ancestro indopacífico, vía corredor del sur de África, como sugiere Bowen et al. (1994). Las colonias del norte de la península de la Florida y de los estados actuales de Georgia y Carolina del Norte no debieron establecerse hasta que concluyó la última glaciación, al igual que la trasplantación del haplotipo B hacia la cuenca mediterránea del Viejo mundo. La amplia distribución de este haplotipo evidencia que la especie ocasionalmente ha sido propensa a realizar largas migraciones y colonizaciones en períodos evolutivos relativamente cortos. MATERIALES Y MÉTODOS__________________________________________________________________________________ 32 III. MATERIALES Y MÉTODOS. III. 1 Obtención y procesamiento de las muestras. Muestras de tejidos de las N extremidades II correspondientes embriones a de 57(1) 56(1) I nidos diferentes, pero concomitantes en estadio de fueron colectadas en playas del suroeste del archipiélago cubano 1(11) HIFL 28(1) A 17(2) 18(3) 16(1) 15(1) 5(1) 1(7) 2(1) 3(8) 27(1) el desarrollo, 3(12) 1(3) 48(2) B SWCUB 48(3) 3(8) 3(5) QRMEX (Figs. 3 y 4): • Chelonia mydas: once en playa Boba [21º 58’N; 83º 36’O] (Cayo Real, Cayería Felipe), San 4(1) durante TORCR agosto de 1998, y 17 3(40) en la península de Guanahacabibes durante el 2000: tres en playa Antonio [21° 91’N; catorce 84° en 65’O] y playa Caleta de los Piojos [21° 82’N; 84° 87’O]. Las muestras de estas dos últimas colonias de anidación fueron juntadas en una sola 0 km 200 400 600 Fig. 3. Distribución y representación de los haplotipos (frecuencias absolutas y relativas representadas respectivamente por el número entre paréntesis y por matices en el pastel) de colonias de anidación de C. mydas en el Mediterráneo americano. El análisis se basó en secuencias de la RNC del mtDNA (ca. 490 pb). Linajes de la cuenca atlántica (Encalada et al., 1996): I- Caribe oriental – Atlántico Sur; IICaribe occidental – Mar Mediterráeo. Áreas/ colonias de anidación: HIFL- Hutchinson Island (USA); QRMEX- Quintana Roo (México); SWCUB- suroeste de Cuba/ A- Península de Guanahacabibes, B- Cayería San Felipe; TORCR- Tortuguero (Costa Rica). Los datos de HIFL corresponden a Allard et al. (1994), QRMEX a Encalada et al. (1996) y TORCR a Lahanas et al. (1998) y Allard et al. (1994). Colonias de anidación: CL- Cayo Largo, GU- Guanahacabibes, IJ- Isla de la Juventud y SF- San Felipe. Cuando aparecen dos gráficos por colonia de anidación, el superior se corresponde con los datos deC. mydas, mientras que el inferior con los de C. caretta. Haplotipos: CM-A1 – 4 (Allard et al., 1994); CM-A5 (Lahanas et al., 1994); CM-A15 – 18 (Encalada et al., 1996); CM-A27 y 28 (Bjorndal & Bolten, inscritos en el GenBank 2001); CMA48, 56 y 57 (Espinosa et al., inscritos en el GenBank 2003). Por conveniencia sólo se representan los números ordinales que identifican a cada haplotipo delante del paréntesis. localidad denominada península de Guanahacabibes, debido a que ha sido MATERIALES Y MÉTODOS__________________________________________________________________________________ 33 documentado el intercambio de ovíferas entre playas dentro la misma estación de anidación y entre años a través del programa de marcación – recaptura implementado por el Centro de Investigaciones Marinas en la península hace casi una década (Azanza, com. pers.). Adicionalmente ambas playas pertenecen a un mismo accidente geográfico con características paisajísticas similares del tipo llanura sumergida de la plataforma insular sometida a un oleaje interno fuerte de los subtipos (Mateo, 1989): 1) con débil cobertura vegetal, sobre roca y arena carbonatada y 2) ídem, pero sólo sobre roca. • Caretta caretta: seis en Cayo Real [21º 58’N; 83º 36’O](Cayería San Felipe), en junio y agosto de 1998; diez en la península de Guanahacabibes [21º 56’N; 84º 30’O] durante las temporadas de anidación comprendidas entre el 2000 y 2006; doce en El Guanal [21º 30’N; 82º 41’O] (Playa Larga, Isla de la Juventud) desde junio hasta agosto de 2006; ocho en playa Los Cocos [21º 38’N; 81º 28’O] (Cayo Largo del Sur) en marzo y junio de 2006. Las muestras fueron conservadas en etanol (90%), a temperatura ambiente. La extracción del DNA fue realizada a partir del método propuesto por Hillis et al. (1996). Se amplificó la mitad 5’ de la RNC del mtDNA (ca. 493 y 380 pb en C. mydas y C. caretta respectivamente) a través de la reacción en cadena de la polimerasa (PCR), empleando los pares de cebadores L15926 (Kocher et al., 1989) y TCR6 (Norman et al., 1994) en C. mydas, y TCR5 y TCR6 (Norman et al., op.cit.) en C. caretta para hacer comparables los fragmentos con los publicados para cada linaje. La PCR fue desarrollada en 50 µL de volumen de reacción, integrado por 2 unidades de Taq polimerasa, 1x PCR tampón II (Promega), 2.5 mM MgCl2, 0.2 mM de dNTP y 0.2 µM de cada cebador por 10-500 ng de DNA genómico. El proceso fue realizado en un termociclador automático (MJ Research), utilizando un paso de desnaturalización inicial de 4 min a 94 oC, seguido de 40 ciclos de 94 oC por 1 min, 45 oC por 45 s, 72 oC por 1 min; y 10 min a 72 oC de extensión final de la reacción. El producto de la PCR fue visualizado en una electroforesis sobre gel de agarosa con bromuro de etidio y la cantidad fue estimada a partir de comparaciones con un marcador de bajo peso molecular (Gibco BRL). MATERIALES Y MÉTODOS__________________________________________________________________________________ I II N 1(8) I BI CR 2(1) 2(1) 7(1) 1(19) 1(20) 7(1) 3(1) 1(4) CI 1(12) AI TB 2(2) SJ NWFL 1(8) NEFL-NC 10(2) 3(1) 13(1) 2(13) 1(32) CSB 10(1) NJt 14(1) SJt 2(4) 1(9) 11(1) 7(1) 20(1) 2(3) 1(4) 1(9) 7(2) 14(1) 3(3) SWFL 1(9) 1(42) ) 2(1) EA CpL TI 2(8) SEFL MB 2(17) 1(25) 1(15) SC HI 2(5) 1(4) 5(1) 3(1) KI 1(5) 2(9) PE 1(3) 2(6) 10(2) 9(2) 1(4) DT 12(1) 2(50) 2(3) 10(1) 14(3) 8(1) GU 1(3) 2(7) IJ CL 2(9) QRMEX 10(5) 9(1) 8(1) 2(5) SF 14(3) SWCUB 2(11) 3(2) 0 km 200 400 600 Fig. 4. Distribución y representación de los haplotipos (frecuencias absolutas y relativas representadas respectivamente por el número entre paréntesis y por matices en el pastel) de C. caretta en áreas/ colonias de anidación (representadas con siglas) del Gran Caribe y la costa este de USA. El análisis se basó en secuencias de la RNC del mtDNA (ca. 380 pb). Linajes de la cuenca atlántica (Encalada et al., 1996; Laurent et al., 1998): I- Atlántico Sur y Norte; II- Gran Caribe y Mar Mediterráneo. Áreas / colonias de anidación: Carolina del Sur/ CR- Cape Romain; DT- Dry Tortugas; Georgia/ CI- Cumberland Island; NC- Carolina del Norte/ BI- Balhead Island, CpL- Cape Lookout y TI- Topsail Island; NEFL- noreste de la Florida/ Amelia Island; NWFL- noroeste de la Forida/ CSB- Cape San Blas, EA- Eglin Air Force Base, SJ- St Joseph's y TB- Tyndall Beach; QRMEX- Quintana Roo (México); SEFL- sureste de la Florida/ MB- Melbourne Beach, HI- Hutchinson Island, NJt- norte de Jetty, PE- Port Everglades, y SJtsur de Jetty; SWCUB- suroeste de Cuba/ CL- Cayo Largo, GU- Guanahacabibes, IJ- Isla de la Juventud y SF- San Felipe; SWFLsuroeste de la Florida/ KI- Key Island y SC- Sarasota County. La generalidad de los datos de las colonias de anidación corresponden a Encalada et al. (1998), excepto en NJt y SJt (Francisco et al., 1999); MB y SC (Francisco & Bowen, 2001; Encalada et al., 1998); CSB y DT (Francisco & Bowen, 2001). Las líneas discontinuas indican las barreras biogeográficas de las costas oeste y este (Briggs, 1974) y de forma escalonada entre éstas (Muss et al., 2001) de la península de la Florida. Haplotipos: CC-A1; 2; 3; 5 – 10 (Encalada et al., 1998; Bolten et al., 1998); CC-A12 – 14 (Bolten et al., 1998) y CC – A20 (Bowen, inscritos en el Archie Carr Center). Por conveniencia sólo se representan los números ordinales que identifican a cada haplotipo delante del paréntesis. 34 MATERIALES Y MÉTODOS__________________________________________________________________________________ 35 El producto de la PCR fue purificado usando columnas Quick spin G50 (Boehringer Mannheim) y fue secuenciado cíclicamente (Sanger et al., 1977) usando el kit Thermo Sequenase (Amersham) con ddNTPs marcados con P33. El producto de secuenciación de cada muestra fue corrido en una electroforesis vertical en gel de poliacrilamida al 6%. Los geles fueron expuestos a placas fotosensibles. En el caso de C. caretta la secuenciación fue automática (compañía Macrogen), debido a que en el momento del procesamiento de estas muestras se dispuso de un procedimiento más eficiente en términos económicos. Estas secuencias fueron rectificadas visualmente con los cromatogramas mediante el programa BioEdit Sequence Alignment Editor (Hall, 1999). Cada muestra fue secuenciada en ambas direcciones para solucionar ambigüedades de posiciones nucleotídicas. III.2 Análisis de los datos. Las secuencias obtenidas en las colonias de anidación del suroeste cubano fueron identificadas y caracterizadas a través de un alineamiento con los haplotipos que tipifican el linaje restringido al hemisferio Norte para C. mydas (CM-A1 – 4, Allard et al., 1994; CMA13 – 18, Encalada et al., 1996; CM-A22, 26 – 31 y 34, Bjorndal & Bolten, inscritos en el GenBank 2001 y http://accstr.ufl.edu/cmmtdna.html), utilizando el programa Clustal X 1.8 (Thompson et al., 1997). Para esta selección fue considerada la disyunción genética entre los linajes de C. mydas de las cuencas Atlántica e Indopacífica (Bowen et al., 1992) y entre los linajes del Caribe occidental – Mar Mediterráneo y del Caribe oriental – Atlántico Sur (Encalada et al., 1996) (ver Fig. 3). Los alineamientos se rectificaron visualmente. Para C. caretta el alineamiento fue hecho con los haplotipos que tipifican los linajes restringidos uno, al Atlántico – península de la Florida y el otro, a los mediterráneos americano – Viejo mundo (CC-A1 – 10, Encalada et al. (1998); CC-A11 – 17, Bolten et al. (1998); CC-A18, Bass et al. (2004); CC-A26 – 32, Carreras et al. (2006); CC-A37 y 40, Roberts et al. (2005); CC-A44, Reece et al. (2005); CC-A48, Laurent et al. (1998); CC-A19 – 23, 24 y 25, 36 y 41, 42, 43, 45, 49 y 50 según Bowen, Bass, Bolten, Carreras, Shamblin, Mazzarella, Monzon, Roberts & Casale respectivamente (inscritos en el Archie Carr Center for Sea Turtle Research (ACCSTR): http://accstr.ufl.edu/cmmtdna.html)). Para esta selección fue considerada la disyunción genética entre los linajes de las cuencas del Atlántico e Indo-Pacífico (Bowen et al., 1994) y entre las colonias de anidación de los mediterráneos americano y del Viejo mundo (Encalada et. al., 1998; Laurent et al., 1998) MATERIALES Y MÉTODOS__________________________________________________________________________________ 36 (ver Fig. 4), así como la ubicación geográfica de Cuba, próxima a las colonias de anidación de la península de la Florida y cayos adyacentes, que a su vez estuvieron integradas por haplotipos de ambos linajes. La numeración de los SPs fue realizada tomando como referencia la primera base de la RNC del mtDNA (C. mydas según Kumazawa & Nishida (1999) (ver Fig. 1); C. caretta según Laurent et al. (1998) (ver Fig. 2)). En el caso en que existió un indel de más de una base, el SP se identificó con el número correspondiente al primer nucleótido, manteniendo en el próximo SP una correspondencia entre la numeración del nucleótido de la secuencia original y el SP en cuestión. A los efectos del procesamiento estadístico, cada indel (independiente de su tamaño) se recodificó como si fuera una sola transversión. La posición de los haplotipos de las colonias de anidación del suroeste cubano fue dilucidada mediante la construcción de un árbol con las menores mediciones entre los haplotipos del linaje (Rohlf, 1973). Para determinar la estructuración genética entre las colonias de anidación del suroeste cubano y las estudiadas previamente en el Atlántico el análisis fue limitado a aquellas colonias que al menos compartieron un haplotipo con las del suroeste cubano; i.e., C. mydas (ver Fig. 3): Tortuguero, Costa Rica; Quintana Roo (X’Cacel e Isla Cozumel), México; Florida, USA; según Allard et al. (1994), Encalada et al. (1996) y Lahanas et al. (1998); C. caretta (ver Fig. 4): USA: Melbourne Beach, Hutchinson Island, Port Everglades, norte y sur de Jetty, Key Island, Sarasota County, Cape San Blas, St. Joseph's, Tyndall Beach, Eglin Air Force Base, Georgia, Dry Tortugas, y México: Quintana Roo; según Encalada et al. (1998), Francisco et al. (1999) y Francisco & Bowen (2001). El grado de diferenciación genética (FST convencional para C. mydas, debido a que prácticamente existe un solo tipo de cambio (transición) como consecuencia de estar representada por dos linajes naturalmente emparentados; Φ ST para C. caretta, utilizando el método de Tamura (Tamura, 1992), debido a la existencia de diversos tipos de cambios como consecuencia de estar representada por dos linajes naturalmente no emparentados) fue estimado según Slatkin (1991) y los intervalos de confianza fueron obtenidos a través de 1000 permutaciones aleatorias, utilizando el programa Arlequin ver. 3.11 (Excoffier et al., 2005). Asumiendo un modelo de isla, el F- Φ ST fue utilizado para estimar el número MATERIALES Y MÉTODOS__________________________________________________________________________________ 37 absoluto de migrantes intercambiados entre dos poblaciones por generación. Las frecuencias haplotípicas entre localidades fueron comparadas utilizando la prueba de independencia X2 (Sokal & Rohlf, 1981), corregida con el procedimiento de Bonferroni (Rice, 1989) con 1000 simulaciones (Roff & Bentzen, 1989), a través del programa CHIRXC (Zaykin & Pudovkin, 1993). En el caso de la colonia de anidación de C. mydas de Tortuguero fue preferido comparar los resultados con los de Allard et al. (1994) y Lahanas et al. (1998), teniendo en cuenta que los tamaños muestrales utilizados por estos autores fueron similares a los de las otras colonias de anidación y que la composición haplotípica no fue afectada significativamente (X2(8)=13.79, p=0.11; a partir los datos originales y tabulados en Bjorndal et al. (2005) para los años 2001, 2002 y 1988-1996) cuando el tamaño de la muestra fue aproximadamente diez veces mayor, de acuerdo con Bjorndal et al. (op.cit.). Del análisis en C. mydas fue excluida la Isla de Aves (Venezuela) debido a que Lahanas et al. (1994) demostró que esta colonia de anidación fue colonizada recientemente por los haplotipos más ampliamente distribuidos y mejor representados en el Mediterráneo americano y áreas de anidación adyacentes de Surinam. La estructura genética de las colonias de anidación del suroeste cubano fue analizada jerárquicamente para comprobar si las colonias de anidación cubanas de cada especie constituyeron una población panmicta dentro de la macro estructura genética de la región, aplicando un análisis de la varianza molecular (AMOVA) (Excoffier et al., 1992) con 1000 permutaciones aleatorias, mediante el programa Arlequin ver. 3.11. Tres escenarios hipotéticos fueron considerados para C. mydas: las colonias de anidación del suroeste cubano junto con las otras colonias de anidación del Mediterráneo americano son localidades geográficas dentro de una población panmicta (Mediterráneo americano) (I); las colonias de anidación del suroeste cubano son localidades dentro de una población panmicta cubana y conjuntamente con las restantes poblaciones del Mediterráneo americano forman una metapoblación (II), y las colonias de anidación del suroeste cubano y la de la península de la Florida son localidades dentro de una población panmicta y conjuntamente con las restantes poblaciones del Mediterráneo americano forman una metapoblación (III). Para C. caretta, también fue probado el escenario hipotético de la existencia de una población panmicta entre las colonias de anidación del Mediterráneo americano (I), que comparten al menos un haplotipo con las cubanas, y fue comprobado si MATERIALES Y MÉTODOS__________________________________________________________________________________ 38 las colonias de anidación del suroeste de Cuba constituyen una población panmicta dentro de la macro estructura genética reconocida para la región (escenario hipotético II), considerando: 1) las poblaciones panmictas (unidades demográficas independientes) propuestas por Encalada et al. (1998), Francisco & Bowen (2001) y Bowen (2004), i.e. noroeste de la Florida (Cape San Blas, St. Joseph's, Tyndall Beach, Eglin Air Force Base), sur de la Florida (Melbourne Beach, Hutchinson Island, Port Everglades, norte y sur de Jetty, Key Island y Sarasota County), noreste de la Florida hasta Carolina del Norte (Georgia en representación de las restantes del norte de la Florida y Carolina del Norte y del Sur), Dry Tortugas y México, y 2) los resultados de la diferenciación por pares de colonias de anidación. Adicionalmente, fue aplicado otro AMOVA jerarquizado (escenario hipotético III), semejante al segundo, pero segregando las colonias de anidación del suroeste y sureste de la Florida. La muestra dentro del área de anidación noreste de la Florida hasta Carolina del Norte fue simplificada a la colonia de anidación de Georgia (selección al azar), considerando que una agrupación de muestras integradas por el mismo haplotipo puede provocar una significación del Φ SC (error de tipo I). También fue analizada la contribución por locus a la estructura poblacional siguiendo el algoritmo propuesto en el mismo programa Arlequin ver. 3.11. Aquellos loci que incidieron significativamente en los índices de fijación fueron reflejados en el texto, considerando los niveles jerárquicos de los escenarios hipotéticos. La prueba de Mantel (también del programa Arlequín ver. 3.11) fue aplicada para determinar la dependencia entre las medidas de diferenciación genética (F- Φ ST y dA de Nei) y la distancia geográfica por pares de colonias de anidación con 10 000 permutaciones aleatorias (Smouse et al., 1986). Una vez dilucidada la estructura genética de las colonias de anidación, la diversidad genética de éstas fue caracterizada. Para ello fueron calculados los porcentajes de endemismo haplotípico (número de haplotipos endémicos/ total de haplotipos) y de individuos endémicos (número de individuos con haplotipos endémicos/ total de individuos). Además fueron estimados los índices de diversidad genética ( Ĥ ) (Nei, 1987) y nucleotídica ( πˆ n) (Tajima, 1983; Nei, op.cit.) y fue corroborado que las mutaciones detectadas en la región no codificadora estudiada en cada población fueran MATERIALES Y MÉTODOS__________________________________________________________________________________ 39 selectivamente neutrales, mediante los estadísticos D de Tajima (Tajima, 1989, 1996) y Fs de Fu (Fu, 1997) con 1000 simulaciones (Stewart, 1977) mediante el programa Arlequin ver. 3.11. Considerando que en marcadores haploides selectivamente neutrales, el tamaño efectivo de la población puede expresarse como un medio de la razón de la correlación de genes de diferentes individuos en la misma población ( θˆ ) versus la tasa de mutación (µ) a partir de la formulación original (Watterson, 1975), este parámetro fue calculado por estación reproductora para cada colonia de anidación teniendo en cuenta una media de remigración de 3 años (Van Buskirk & Crowder, 1994; Miller, 1997) y una tasa de mutación de 1.2% por locus por MA por pares de linajes, informada para C. mydas según Avise et al. (1998) a partir de la metodología de Avise et al. (1992) y según datos de Bowen et al. (1992), adaptados a secuencias de mtDNA según Encalada et al. (1996) y de 0.83 % para C. caretta (Encalada et al., 1998). Esta tasa de mutación se correspondió a la cota superior de 3.0 MA y no al promedio del intervalo de tiempo propuesto para la constitución del istmo interoceánico (3.0-2.5 MA) según Lundelius (1987) y empleado por Avise et al. (1992) y fue corregida con el tiempo promedio de vida de una generación (47 y 45 años para C. mydas y C. caretta respectivamente), calculado según Pianka (1974) y empleando una media de maduración sexual de 37.5 y 35 años para C. mydas (Limpus & Chaloupka, 1997; Bjorndal et al., 2000) y C. caretta (ALRTM, 2003) respectivamente, y una media de vida reproductora de 19 y 20 años para C. mydas (Chaloupka et al., 2004) y C. caretta (ALRTM, 2003) respectivamente. Theta fue calculado según el número de sitios segregativos ( θˆ S) y la diversidad nucleotídica ( θˆ π), mediante el programa Arlequin ver. 3.11. Para analizar la demografía histórica de las poblaciones fue calculada la distribución del número observado de diferencias entre pares de haplotipos mediante el modelo de expansión súbita (Rogers & Harpending, 1992). Su significación fue examinada usando la desviación estándar de la suma de cuadrados entre las desigualdades observadas y esperadas (Ssd) a través del programa Arlequin ver. 3.11. Mediante este modelo fueron obtenidos el tamaño efectivo de la colonia de anidación antes de la expansión (N ( θˆ 0)) y el intervalo temporal en años donde ocurrió la expansión (t). En esta última variable fueron MATERIALES Y MÉTODOS__________________________________________________________________________________ 40 referidos los valores considerando el intervalo de confianza (IC) para alpha igual a 0.05 (Li, 1977). En el caso de que Theta tomó valor cero al inicio de la expansión, el tamaño efectivo al inicio de la expansión fue calculado con el valor medio del IC del estadístico con igual alpha. En el análisis de expansión demográfica también fue tenido en cuenta la significación de la Prueba de Fu. Las relaciones filogenéticas entre los haplotipos informados para las poblaciones de anidación de los linajes de la cuenca atlántica (Encalada et al., 1996) fueron inferidas utilizando los métodos de distancia UPGMA (Sneath & Sokal, 1973) y NJ (Saitou & Nei, 1987), a través del programa MEGA ver. 3.0 (Kumar et al., 1993). Fueron reconstruidos dos filogramas enraizados de haplotipos (por conveniencia para la representación gráfica, sólo fue mostrado el resultado del UPGMA) con los haplotipos correspondientes a las poblaciones de anidación que comparten al menos un haplotipo con las cubanas), empleando como grupo externo al haplotipo CMU40436 y B de linajes del Pacífico de C. mydas (Dutton et al., 1996) y C. caretta (Hatase et al., 2002) respectivamente. El soporte estadístico para los nodos fue estimado mediante un "bootstrapping" no paramétrico (Felsenstein, 1985) utilizando 1000 pseudo-réplicas. Los tiempos de divergencia entre los haplotipos fueron calculados a partir del intervalo superior de tiempo propuesto para la constitución del istmo de Panamá (Lundelius, 1987). También fue calculada la divergencia de secuencias mediante las distancias dentro y entre las poblaciones de anidación (Nei, 1987: fórmulas 10.19 y 10.20 respectivamente) y el número neto de sustituciones nucleotídicas entre dos poblaciones (Nei & Li, 1979). Éste último fue utilizado para estimar los tiempos de divergencia entre las poblaciones de anidación (Nei, 1987: fórmula 10.22). La interpretación de los árboles de distancia no constituye per se una estimación formal para verificar hipótesis filogeográficas, además de no incluir una metodología para la interpretación de los modelos observados (Templeton, 2004). Por esta razón también fue aplicado el Análisis Filogeográfico de Clados Anidados (NCPA) (Templeton et al., 1987) con los haplotipos de las colonias de anidación que al menos compartieron un gen con las del suroeste de Cuba. Para ello fueron tenidos en cuenta los haplotipos encontrados en el presente estudio y aquellos publicados para el linaje mediterráneos americano – Viejo Mundo, con sus respectivas frecuencias según datos originales de Allard et al. (1994), MATERIALES Y MÉTODOS__________________________________________________________________________________ 41 Encalada et al. (1996) y Lahanas et al. (1998) para C. mydas, y de Encalada et al. (1998), Laurent et al. (1998), Francisco et al. (1999) y Francisco & Bowen (2001) para C. caretta. Por lo tanto, en este análisis fueron incluidas las poblaciones de anidación de Lara (Chipre) para C. mydas y C. caretta, además de Grecia, Turquía y Brasil para esta última, porque sus haplotipos pertenecieron al mismo linaje y en algunos casos coincidieron con la representación de las colonias de anidación cubanas. Primeramente, el número máximo de conexiones entre pares de secuencias fue calculado bajo un criterio de “parsimonia”, a partir del número de mutaciones en relación con la probabilidad de parsimonia (Templeton et al., 1992), así como el haplotipo de mayor peso como grupo externo (Castelloe & Templeton, 1994). Este proceso resultó en un dendrograma no enraizado, con nueve pasos correspondientes al 95% del límite de conexiones, a través del programa TCS ver. 1.21 (Clement et al., 2000). Seguidamente, dos series de clados anidados jerárquicamente fueron definidos según las reglas de anidación propuestas por Templeton et al. (1992) y Templeton & Sing (1993), los cuales fueron analizados estadísticamente en términos de relaciones entre la genealogía de los haplotipos y la distancia geográfica de conexión más probables entre las colonias, con el programa GEODIS ver 2.5 (Posada et al., 2000). Los clados con valores significativos de distancias de clados fueron interpretados mediante el empleo de la clave de inferencia para el NCPA (Templeton, 2004), versión 11 noviembre 2005. El valor de probabilidad (p) para cada estadístico fue obtenido cuando el valor aleatorio de las permutaciones fue menor o igual al observado, excepto en los estadísticos F- Φ CT y FΦ SC donde el valor de p fue obtenido cuando la proporción fue mayor e igual porque se asume para el primer caso, que las poblaciones son reales y las agrupaciones regionales son artificiales y para el segundo caso, que las regiones son reales y las poblaciones dentro de cada región son artificiales. La significación de los estadísticos fue considerada cuando los valores de p fueron menores que 0.05. RESULTADOS____________________________________________________________________________________________ 42 IV. RESULTADOS. IV.1 Análisis de secuencias. IV.1.1 Chelonia mydas. Los haplotipos encontrados en el suroeste 23 linaje mediterráneos americano – 3). Se informan tres nuevos haplotipos endémicos para las colonias de anidación CM-A48, 56 y 57, acorde con 37 36* 25 294 470* 470* 171 x 385* 384 44 9 nomenclatura 233 Turtle 38 410 33 433 431 propuesta Research e inscritos en el GenBank con 27 96 AJ543730, AJ543732 AJ543735. Otros y Linaje II 22 149 365 13 93 272 28 14 16 371 250 250 17 250 250 166* 56 18 48 470* 172* 2 93 57 149 96 x los números de accesión: 49 384 278* por el Archie Carr Center for Sea 43 488 204* 284 la 173 410 367 32 20 6 410 410 367 173 365 39 133 7 171 133 45 50 5 448 35 133 46 470* 55 42 11 365 410 365 8 366* 40 230* 470* 374* 373 113 cubanas, designados como 36 113 428* 437* 272 10 x Viejo Mundo (Fig. 5; ver Fig. 24 12 x al Cuba x pertenecen de 21 41 Linaje I 15 365 184* 3 438* 30 294 39* 233 373 31 113 149 1 93 294 26 93 52 357* 53 29 dos 4 34 51 47 haplotipos (CM-A27 y 28; Fig. 5. Dendrograma no enraizado de máxima parsimonia basado en inscritos 2001), en el las diferencias nucleotídicas por pares de haplotipos de linajes de C. GenBank, mydas en la cuenca atlántica. El análisis se basó en haplotipos de la encontrados en la colonia de anidación de la península Guanahacabibes, de también podrían ser endémicos si sus RNC del mtDNA (ca. 493 pb). Círculo, indica al iésimo haplotipo; a rayas, haplotipos de origen desconocido; # fuera del círculo representa una mutación (transición), con un cuadrado una transversión, con una x un indel, y con un asterisco (*) un cambio diagnóstico. Linajes de la cuenca atlántica (Encalada et al., 1996): I- Caribe oriental – Atlántico Sur; IICaribe occidental – Mar Mediterráeo. Haplotipos: CM-A1, 2, 4, 8 – 18 (Encalada et al., 1996); CM-A3 (Allard et al., 1994); CMA22 (Bass y Witzell, 2000); CM-A23 – 34 (Bjorndal & Bolten, inscritos en el GenBank 2001); CM-A35 – 46 y 50 – 53 (Formia, inscritos en el Archie Carr Center); CM-A48, 56 y 57 (Espinosa et al., inscritos en el GenBank 2003); CM-A49 (Bagley, 2003); CM-A54 (Meylan, inscritos en el Archie Carr Center); CM-A55 (Naro-Maciel et al., 2007). descriptores (Bjorndal y Bolten) los encontraron en un área marina de agregación. Solamente uno de los haplotipos endémicos (CM-A48), presente en áreas de desarrollo de la Florida (Bagley, 2003), se comparte entre las dos colonias de anidación del suroeste de Cuba. RESULTADOS____________________________________________________________________________________________ 43 Los cinco haplotipos endémicos encontrados en las colonias de anidación del suroeste cubano están interconectados en una red donde existen uno o dos cambios por pares de haplotipos vecinos (ver Fig. 5), con respecto a los dos haplotipos más ampliamente distribuidos y mejor representados (CM-A1 y 3; i. e., centros de diversificación) en el linaje y en las colonias de anidación del Mediterráneo americano. El número de cambios que existe entre los haplotipos del linaje con respecto a los haplotipos CM-A1 y 3, considerando el camino más parsimonioso, es independiente del centro de diversificación (X2(4)=5.67, p=0.24). Cuando fueron discriminados los cambios con respecto a la posición (interna o externa) de los haplotipos, tampoco se encuentra dependencia en el número de cambios entre los haplotipos internos y los centros de diversificación (X2(4)=0). Sin embargo, se encuentra dependencia entre el número de cambios entre los haplotipos externos y los centros de diversificación (X2(4)=9.52, p=0.03), siendo más frecuente un cambio con respecto a CM-A3 y dos con respecto a CM-A1. Por consiguiente, se designa a CM-A1 como haplotipo ancestral del linaje II. Un tercer centro de diversificación se Frecuencia relativa de mutaciones (%) encuentra alrededor del haplotipo CM-A13 del Mediterráneo del Viejo Mundo. 30 a) Linaje I 25 b) Linaje II c) Linajes I y II 20 15 10 5 0 50 100 150 200 250 300 350 400 450 500 I-50 I-150I-200I-250I-300 I-400I-450I-500 as par Longitud secue (p es de mtDNA la 50 100 150 200 250 300 350 400 450 500 I-150 I-50I-100 I-150I-200 I-150 0 I-500 I-400 I-450 L secuen d eldmtDNA itud d itud itud b) 50 100 150 200 250 300 350 400 450 500 I-400I-450I-500 I-50I-100I-150I-200I-250I-300 Longitud de secuencias (pb) ngitud secue s pars del mtDNA (p Fig. 6. Distribución de mutaciones en linajes de la cuenca atlántica de C. mydas. El análisis se basó en haplotipos de la RNC del mtDNA (ca. 493 pb). Transición, líneas inclinadas a la izquierda, por debajo de una línea negra continua y gruesa; Transversión, líneas inclinadas a la derecha; indel, a cuadros; cambios diagnósticos de haplotipo y de haplotipo dentro de linaje en gris y gris y negro respectivamente; cambios diagnósticos entre linajes en líneas negras oscuras. Del total de sustituciones (31), aproximadamente el 94% son transiciones repartidas con mayor frecuencia entre los 350 y 400 pb (Fig. 6b), excepto entre los 300 y 350 pb, existiendo similar balance entre purinas y pirimidinas, así como entre los sentidos de las sustituciones en relación al camino más parsimonioso respecto al ancestro CM-A1: T→C RESULTADOS____________________________________________________________________________________________ 44 (6) y C→T (8); A→G (7) y G→A (8). Las restantes sustituciones corresponden a una transversión en el SP 365, que también constituye una transición para otras relaciones como ocurre en el haplotipo CM-A4, y a un indel de seis pb (CAATGG) en el SP 470. A su vez, el 31% aproximado de estas sustituciones representan cambios diagnósticos para los haplotipos (ver Fig. 5). La inserción antes mencionadas lo es de CM-A48, mientras que las transiciones (8) en los SPs 39, 166, 172, 184, 357, 374, 438 y 459 lo son de CM-A31, 34, 2, 15, 29, 22, 30 y 54 respectivamente. De éstas, las sustituciones entre purinas y pirimidinas están en proporción 5:3. El sentido de las sustituciones diagnósticas en relación al camino más parsimonioso con respecto al haplotipo ancestral es diferente entre las transiciones de pirimidinas: T→C (3) y C→T (0); A→G (3) y G→A (2). Entre los haplotipos correspondientes a las colonias de anidación del Mediterráneo americano se determina un máximo de 17 SP. Tabla 1. Estos SP se clasifican en 15 transiciones, una transversión en la posición 284 y el indel anteriormente mencionado en la posición 470. La transversión pertenece al haplotipo CM-A5, raro en la colonia de anidación de Quintana Roo. sólo persisten 4 SPs (93, 149, 250 y el indel). Para el otro linaje, que no está representado en las colonias cubanas, de anidación el mayor porcentaje de mutaciones A CM-A2 C A CM-A3 T A G G A T T C G G G T T C G G A T T C 493 cubano C 470 suroeste CM-A1 433 atlántica. En la muestra del 431 cuenca 410 la de Sitios polimórficos 371 linajes los 367 entre 365 diagnósticos 284 SPs 272 constituyen 433, 250 y la base 1 de la región de control del mtDNA (Kumazawa y Nishida, 1999). ±, indel de CAATGG. Haplotipos: CM-A1 – 4 (Allard et al., 1994); CM-A5 (Lahanas et al., 1994); CM-A15 – 18 (Encalada et al., 1996); CM-A27 y 28 (Bjorndal & Bolten, inscritos en el GenBank 2001); CM-A48, 56 y 57 (Espinosa et al., inscritos en el GenBank 2003). 184 431 americano. La numeración de los sitios polimórficos se realiza tomando como referencia 172 posiciones las 149 de 133 transiciones Tabla 1. Polimorfismo en la mitad 5’ de la RNC del mtDNA (ca. 490 pb) las de C. mydas, determinado en colonias de anidación del Mediterráneo 96 y 93 cambio Haplotipo Este T C G G T A G - A T C G G T A G - A T C G G T A G - A CM-A4 T A G G A T T C T T G G T A G - A CM-A5 C G A G A T C C G C A G C G A - G CM-A15 T A G G A C T C T C G G T A G - A CM-A16 C A G A A T C T T C G G T A G - A CM-A17 C A G A A T C C T C G A T A G - A CM-A18 C G G G A T T C T C G G T A G - A CM-A27 C A G G A T C C T C G G T A G - A CM-A28 T A G G A T C C T C G G T A G - A CM-A48 C A G G A T T C T C G G T A G + A CM-56 C A G A A T T C T C G G T A G - A CM-57 T A G A A T T C T C G G T A G - A RESULTADOS____________________________________________________________________________________________ 45 también estuvo representado por transiciones repartidas, con mayor frecuencia, entre los 150 y 500 pb (Fig. 6a), excepto entre los 300 y 350 pb. Las transversiones y los indeles se encontraron sólo entre 350 y 500 pb. IV.1.2 Caretta caretta. Los haplotipos encontrados en el suroeste de Linaje I 21 península de la Florida y el otro, mediterráneos 22 3 23 CC-A1, 2, 8, 10, 12 y 14. El CC-A12, informado 15 189* 280* 294* 326 150* americano – Viejo Mundo (Fig. 7; ver Fig. 4), i.e. 27 1 435* 40 suroeste cubano están interconectados, dentro de cada linaje, en una red donde existen generalmente uno o dos cambios por pares de haplotipos vecinos (ver Fig. 7), con respecto a los dos haplotipos más 49 206 470 161 9 29 268* 393* 320* 172* 428* 26 272* 142* 229 2 369* 470 16 161 468 206 468 206 42 43 30 207 423* 365* del 50 46 48 31 44 471* Los haplotipos encontrados en las colonias de anidación 25 Linaje II 28 24 326 47 473* 472* 18 199* 4 205* 41 según Bolten et al. (1998), resulta ser endémico 188 11 354 272 246* 320* para las áreas de forrajeo de Azores y Madeira de la Cayería San Felipe. 14 229* 37 17 20 19 271* x x Cuba pertenecen a dos linajes: uno, Atlántico – 8 473* 298 188 10 431 422 161 173* 3 13 298 6 7 12 468 431 422 298 188 5 x 36 32 45 distribuidos y mejor representados (CC-A1 y 2; i. Fig. 7. Dendrograma no enraizado de máxima e., centros de diversificación) en el linaje y en las parsimonia basado en las diferencias colonias de anidación del Mediterráneo americano. En el linaje del Atlántico – península de la Florida se encuentran tres centros de diversificación alrededor de los haplotipos CCA1, 11 y 4, con mayores probabilidades de ancestría para el caso de los dos primeros centros de diversificación. El número de cambios entre los haplotipos de este linaje con respecto a los haplotipos CC-A1 y 11, considerando el nucleotídicas por pares de haplotipos de linajes de C. caretta en la cuenca atlántica. El análisis se basó en haplotipos de la RNC del mtDNA (ca. 380 pb). Círculo, indica al iésimo haplotipo – a rayas, haplotipos de origen desconocido; # fuera del círculo representa una mutación (transición), con un cuadrado una transversión, con una x un indel, y con un asterisco (*) un cambio diagnóstico dentro de cada linaje. Linajes de la cuenca atlántica (Encalada et al., 1996; Laurent et al., 1998): I- Atlántico Sur y Norte; II- Gran Caribe y Mar Mediterráneo. Haplotipos: CC-A1 – 10 (Encalada et al., 1998); CC-A11 – 17 (Bolten et al., 1998); CC-A18 (Bass et al., 2004); CC-A26 – 32 (Carreras et al., 2006); CC-A37 y 40 (Roberts et al., 2005); CC-A44 (Reece et al., 2006); CC-A48 (Laurent et al., 1998); CC-A19 – 23, 24 y 25, 36 y 41, 42, 43, 45, 49 y 50 según Bowen, Bass, Bolten, Carreras, Shamblin, Mazzarella, Monzon, Roberts y Casale respectivamente (inscritos en el Archie Carr Center). camino más parsimonioso, es dependiente del centro de diversificación (X2(3)=14.79, RESULTADOS____________________________________________________________________________________________ 46 p<0.001). Cuando un análisis similar al anterior se realiza discriminando los cambios con respecto a la posición (interna o externa) de los haplotipos, no se encuentra dependencia para el número de cambios entre los haplotipos internos y los centros de diversificación (X2(2)=0). Sin embargo, se encuentra dependencia para el número de cambios existentes entre los haplotipos externos y los centros de diversificación (X2(3)=19.33, p<0.001), siendo más frecuente un cambio con respecto a CC-A1 y dos con respecto a CC-A11. Por consiguiente, designamos a CC-A11 como haplotipo ancestral del linaje I. En el linaje mediterráneos americano – Viejo Mundo sólo existe un centro de diversificación alrededor del haplotipo ancestral CC-A2, lo cual simplificó su designación como haplotipo ancestral Del total de sustitucion es del linaje Atlántico – península de la Florida 25 a) Linaje I el son b) Linaje II c) Linajes I y II 20 15 10 5 0 100 150 200 250 300 350 400 ngitud aproximada 88% 30 I-150I-200-250I-300I-350I-400 (14), mente Frecuencia relativa de mutaciones (%) del linaje. s par NC del m 100 150 200 250 300 350 400 450 500 100 150 200 250 300 350 400 450 500 I-150I-200I-250-300I-350I-400I-450I-500 L ecuen gitud d s parc l de es deNC DNA ( I-150I-200I-250I-300I-350-400I-450I-500 Longitud de secuencias (pb) gitud de ) ( L ecues pars deNC tDNA ecue s de Fig. 8. Distribución de mutaciones en linajes de la cuenca atlántica de C. caretta. El análisis se basó en haplotipos de la RNC del mtDNA (ca. 380 pb). Transición, representadas en líneas inclinadas a la izquierda, por debajo de una línea negra continua y gruesa; Transversión, líneas inclinadas a la derecha; indel, a cuadros; cambios diagnósticos de haplotipo y de haplotipo dentro de linaje en gris y gris y negro respectivamente; cambios diagnósticos entre linajes en líneas negras oscuras o retículos en blanco y negro. transiciones, repartidas con mayor frecuencia entre los 150 y 350 pb (Fig. 8a), con igual balance entre purinas y pirimidinas, pero no entre los sentidos de las sustituciones en relación al camino más parsimonioso respecto al ancestro CC-A11: T→C (4) y C→T (2); A→G (4) y G→A (2). Las restantes sustituciones corresponden a dos transversiones en los SPs 150 y 205. A su vez, el 64% aproximado de estas sustituciones representan cambios diagnósticos para los haplotipos (ver Fig. 7). Las transversiones antes mencionadas lo son de CC-A23 y 47, mientras que las transiciones (7) en los SPs 189, 199, 229, 246, 280, 294 y 320 lo son de CC-A21, 24, 37, 27, 17, 22 y 41 respectivamente. De éstas, las sustituciones entre purinas y pirimidinas están en proporción 3:4. El sentido RESULTADOS____________________________________________________________________________________________ 47 de las sustituciones diagnósticas en relación al camino más parsimonioso con respecto al haplotipo ancestral es diferencial entre las transiciones de purinas en comparación con las de pirimidinas: T→C (2) y C→T (2); A→G (3) y G→A (0). Dos de estas transiciones (SPs 189 y 320) se han fijado en al menos uno de los otros linajes. En el linaje mediterráneos americano – Viejo Mundo del total de sustituciones (39), aproximadamente el 82% de las mutaciones se corresponde con transiciones repartidas, con mayor frecuencia entre los 150 y 500 pb (Fig. 8b) excepto entre los 300 y 350 pb. La mayor parte de las transiciones han ocurrido entre purinas respecto a pirimidinas, con similar balance entre los sentidos de las sustituciones en relación al camino más parsimonioso respecto al ancestro CC-A2: T→C (6) y C→T (6); A→G (9) y G→A (11). Las restantes sustituciones corresponden a cuatro transversiones y dos indeles en los SPs 435 y 471-3, y 431 y 473 respectivamente. A su vez, el 44% aproximado de estas sustituciones representan cambios diagnósticos para los haplotipos (ver Fig. 7). Las transversiones antes mencionadas lo son de CC-A40 y 18, el indel en el SP 473 lo es de CC-A5, mientras que las transiciones (11) en los SPs 142, 172, 173, 268, 271, 272, 320, 365, 369, 393, 423 y 428, lo son de CC-A20, 30, 63, 44, 8, 26, 31, 48, 19, 50, 28, y 29 respectivamente. De éstas, las sustituciones entre purinas y pirimidinas están en proporción 5:7. El sentido de las sustituciones diagnósticas en relación al camino más parsimonioso con respecto al haplotipo ancestral es diferencial entre las transiciones de purinas y pirimidinas: T→C (5) y C→T (2); A→G (1) y G→A (4). Cuatro de estas transiciones (SPs 142, 173, 271 y 369) se fijaron en al menos uno de los otros linajes. Entre los haplotipos correspondientes a las colonias de anidación del Mediterráneo americano y la costa este de USA se determina un máximo de 26 SPs. Tabla. 2. Estos SPs se clasifican en 22 transiciones, una interna dentro del indel 465 en la posición 468 y otros cuatro indeles. Los indeles de las posiciones 163, 404 y 465 constituyen SPs diagnósticos entre los linajes representados, al igual que las transiciones de las posiciones 145, 147, 163, 214, 340, 356, 424 – 27, y 437. Los SPs 320 y 369 pudieran ser diagnósticos entre linajes, pero constituyen transiciones diagnósticas de los haplotipos CC-A19 y 31 pertenecientes al linaje II. En la muestra del suroeste cubano persisten la generalidad de los sitios polimórficos, menos los SPs 206, 298, 422 y el diagnóstico 473. RESULTADOS____________________________________________________________________________________________ 48 Tabla 2. Polimorfismo en la mitad 5’ de la región de control del mtDNA (ca. 380 pb) de C. caretta, determinado en colonias de anidación del Mediterráneo americano. La numeración de los sitios polimórficos se realiza tomando como referencia la base 1 de la región de control del mtDNA (Laurent et al., 1998). ±, indel de GCAAGT, con una transición interna en el SP 358. Haplotipos: CC-A1 – 10 (Encalada et al., 1998); CC-A11 – 14 (Bolten et al., 1998); CC-A20 (Bowen, inscrito en el Archie Carr Center). 37 51 53 63 78 96 104 161 162 188 210 230 244 246 259 294 312 314 315 317 327 355 363 35 358 32 Haplotipo Sitios polimórficos CC-A1 T G T T T A G A A C G G C C G C A - A A T A C - CC-A2 C A C C - G G A G T A G T T A T G G A G C G T + A A CC-A3 C A C C - G G A G T A A T T A T G G A G C G T + A A CC-A5 C A C C - G G A G T A G T T A T G G A G C G T + A - CC-A7 C A C C - G G A G T A G T T A T G G G G C G T + A A CC-A8 C A C C - G G A G C A G T T A T G G A G C G T + A A CC-A9 C A C T - G G G G T A G T T A T G G A G C G T + A A CC-A10 C A C C - G G A G T A G T T A T G G A G C G T + G A CCA11 T G T T T A G A A C A G C C G C A - A A T A C - CCA12 C A C C - A G A G T A G T T A T G G A G C G T + G A CC-A13 C A C C - G A A G T A G T T A T G G A G C G T + A A CC-A14 T G T T T A A A A C A G C C A C A - A A T A C - CC-A20 T A C C - G G A G T A G T T A T G G A G C G T + A A A A A IV.2 Estructura poblacional. IV.2.1 Chelonia mydas. Las colonias de Tabla 3. Comparación por pares de colonias de anidación de C. mydas del anidación suroeste no del cubano están estructuradas significativamente Mediterráneo americano, a partir de haplotipos de la RNC del mtDNA (ca. 490 pb). Las localidades están organizadas latitudinalmente en los titulos de la tabla. Arriba de la diagonal: comparación de las frecuencias haplotípicas empleando la Prueba X2 corregido por el procedimiento de Bonferroni a través del método Monte Carlo (1000 simulaciones). El primer número se corresponde con el valor del estadígrafo, los grados de libertad (entre paréntesis) seguido de la probabilidad (/ valor). Debajo de la diagonal: número absoluto de migrantes intercambiados en la parte superior, prueba de diferenciación por pares de colonias de anidación (FST) basada en permutaciones no paramétricas, seguida de la probabilidad (/ valor). Los datos incongruentes entre ambas pruebas estadísticas por pares de colonias de anidación se representan en negrita, y en itálica, aquellos cuya probabilidad fue significativa. Los datos de las colonias con superíndices 1, 2 y 3 son según Allard et al. (1994), Encalada et al. (1996) y Lahanas et al. (1998), respectivamente. (Tabla 3). Localidades Tampoco se Tortuguero1, 3 (Costa Rica) Quintana Roo1 (México) encuentra estructura significativa entre colonias 19.96(7) / <0.001 0.44 0.53/ <0.001 Guanahacabibes 0.86 (Cuba) 0.37/ <0.001 genética las Tortuguero2, 3 Quintana Roo2 Guanahacabibes (Costa Rica) (México) (Cuba) de anidación de la San Felipe (Cuba) Cuba suroeste Florida1 (USA) 25.82(7) / <0.001 12.03(2) / 0.007 21.60(7) / <0.001 25.37(3) / <0.001 16.16(11) / 0.04 17.55(7) / 0.002 23.69(11) / 0.002 12.51(7) / 0.03 6.20(6)/ 0.43 13.16 0.04/ 0.12 San Felipe (Cuba) 1.03 0.33/ 0.005 1.86 0.21/ 0.001 Cuba suroeste 3.48 0.12/ 0.02 4.03 0.11/ 0.006 Florida 2 (USA) 0.56 0.47/ <0.001 12.54 0.04/ 0.14 11.51(7) / 0.04 16.23 0.03/ 0.22 12.73(3) / 0.004 14.92(7) / 0.004 14.36 0.03/ 0.18 1.95 0.20/ 0.01 5.06 0.09/ 0.03 península de Guanahacabibes y las peninsulares del continente (Florida y Quintana Roo), RESULTADOS____________________________________________________________________________________________ 49 ya que aunque la probabilidad del X2 es significativa en ambas interacciones, la cota superior del intervalo de confianza es igual al valor crítico de no significación (0.03 – 0.05). Del análisis se infiere un flujo de migrantes en el orden de las docenas de individuos por generación. Sin embargo, la colonia de anidación de San Felipe está estructurada significativamente con las colonias de anidación peninsulares del continente, por lo que el flujo de migrantes entre estas colonias es pequeño. Cuando se juntan las colonias de anidación en un sola entidad, se mantiene la estructura genética significativa con las restantes colonias de anidación del Mediterráneo americano. Similar comportamiento también ocurre con la colonia de anidación de Tortuguero. 0,6 (FST) 0,5 por pares de colonias de anidación está correlacionado con la distancia geográfica (r=0.93, p=0.02), con baja dispersión de las interacciones (Fig. 9), y no con la distancia neta promedio basada en el número de sustituciones nucleotídicas (r=0.83, p=0.07). Las distancias neta promedio basada en el número de sustituciones geográfica nucleotídicas tampoco correlacionadas (r=0.74, p=0.08). y están Índice de diferenciación poblacional ( F ST ) El grado de diferenciación genética 0,4 0,3 0,2 y= 3x E28 0. .07 -0 0,1 0,0 -0,1 0 200 400 600 800 1000 1200 1400 1600 1800 2000 2200 2400 Distancia geográfica en km 95% confianza Fig. 9. Regresión lineal entre la diferenciación genética (FST estimado a partir de haplotipos de la RNC del mtDNA, ca. 493 pb) versus la distancia geográfica por pares de colonias de anidación de C. mydas localizadas en el Mediterráneo americano. Líneas: continua, curva de regresión lineal; discontinua, límite de confianza. La estructuración genética significativa encontrada entre las colonias de anidación integradas bajo la unidad poblacional suroeste cubano y las restantes del Mediterráneo americano, así como entre las colonias de esta región, se confirma por un valor significativo de FST en todas las estructuras hipotéticas jerarquizadas (Tabla 4), con un porcentaje de variación casi cuatro veces mayor dentro que entre colonias de anidación. A esta variación contribuye significativamente el 35% de los SPs en las tres estructuras hipotéticas, que no son loci diagnósticos (93: FST=0.36, p<0.001; 96: FST=0.18, p=0.003; 149: FST=0.08, p=0.02; 250: FST=0.12, p=0.009 y 371: FST=0.06, p=0.03) entre haplotipos y RESULTADOS____________________________________________________________________________________________ el diagnóstico locus 50 470 Tabla 4. Resultados del análisis jerárquico de varianza molecular bajo tres niveles hipotéticos de estructura geográfica en colonias (FST=0.18, p=0.001) del haplotipo de anidación de C. mydas del Mediterráneo americano. El análisis endémico CM-A48 colonias de de las anidación del suroeste cubano. se basó en haplotipos de la RNC del mtDNA (ca. 490 pb). Áreas de anidación/ colonias de anidación: QRMEX- Quintana Roo (México); sureste de la Florida (USA)/ HIFL- Hutchinson Island; SWCUB- suroeste de Cuba/ GU- Guanahacabibes, SF- San Felipe; TORCR- Tortuguero (Costa Rica). Los datos de HIFL corresponden a Allard et al. (1994), QRMEX a Encalada et al. (1996) y TORCR a Allard et al. (1994) y Lahanas et al. (1998). p, probabilidad. Fuente de variación Al agruparse jerárquicamente las muestras de las colonias de anidación del suroeste cubano (estructura obtiene el hipotética mismo significación y II), nivel p F estadísticos Varianza % total I. Estructura hipotética: colonias de anidación del SWCUB (GU y SF) y las de la región (HIFL, QRMEX y TORCR) constituyen una población panmicta Entre poblaciones 0.07 25.55 Dentro de las poblaciones 0.22 74.45 semejante <0.001 Entre regiones 0.06 20.20 0.20 0.30 Entre poblaciones/ regiones 0.02 5.93 0.07 0.22 Dentro de poblaciones 0.22 73.87 0.26 <0.001 III. Estructura hipotética: colonias de anidación del SWCUB y HIFL constituyen una unidad demográfica respecto a QRMEX y TORCR porcentaje de variación dentro de las colonias (ver Tabla 4), 0.26 II. Estructura hipotética: colonias de anidación del SWCUB constituyen una unidad demográfica respecto a HIFL, QRMEX y TORCR se de Partición observada Entre regiones 0.05 16.74 0.17 0.20 Entre poblaciones/ regiones 0.03 11.02 0.13 0.04 Dentro de poblaciones 0.22 72.24 0.28 <0.001 pero sin la contribución del locus 371 (FST=0.07, p=0.06). En esta hipótesis el FSC y el FCT no son significativos. Sin embargo, cuando las colonias de anidación del suroeste cubano se agrupan con la colonia de anidación de la península de la Florida (estructura hipotética III) el FSC es significativo, como consecuencia de la contribución significativa del SP no diagnóstico 93 (FSC=0.02, p<0.001). Consecuentemente con este resultado, la estructura hipotética II es aceptada. IV.2.2 Caretta caretta. Tabla 5. Comparación por pares de colonias de anidación de C. caretta Las colonias de anidación en el suroeste de Cuba, a partir de haplotipos de la RNC del mtDNA (ca. del suroeste de Cuba no están estructuradas significativamente entre sí ( Φ ST(gl=35)=-0.006, p=0.41). Tabla 5. Tampoco encuentra se estructura genética entre las colonias de anidación pertenecientes a una 380 pb). Las colonias de anidación están organizadas longitudinalmente en los titulos de la tabla. Arriba de la diagonal: comparación de las frecuencias haplotípicas empleando la 2 Prueba X corregido por el procedimiento de Bonferroni, a través del método Monte Carlo (1000 simulaciones). El primer número se corresponde con el valor del estadígrafo, entre paréntesis los grados de libertad, seguido de la probabilidad (/ valor). Debajo de la diagonal: número absoluto de migrantes intercambiados en la parte superior, prueba de diferenciación por pares de colonias de anidación (ФST) basada en permutaciones no paramétricas, seguida de la probabilidad (/ valor). Colonias de anidación Cayo Largo 0.38(1)/ 0.65 Cayo Largo Isla de la Juventud San Felipe Guanahacabibes Isla de la Juventud Inf -0.08/ 0.65 12.61 0.11/ 0.38 3.99 0.11/ 0.23 7.03 0.07/ 0.11 7.03 0.08/ 0.11 San Felipe Guanahacabibes 6.34(4)/ 0.14 7.88(4)/ 0.05 6.19(2),/ 0.06 6.12(2)/ 0.05 7.04(4)/ 0.05 10.25 0.05/ 0.24 RESULTADOS____________________________________________________________________________________________ 51 misma población de anidación del Mediterráneo americano, excepto entre Eglin Air Force Base versus Cape San Blas pertenecientes al noroeste de la Florida según la prueba X2. Tabla 6. Tabla 6. Comparación por pares de colonias de anidación de C. caretta en Mediterráneo americano, a partir de haplotipos de la RNC del mtDNA (ca. 380 pb). Las localidades están organizadas latitudinalmente en los titulos de la tabla. Arriba de la diagonal: comparación de las frecuencias haplotípicas empleando la Prueba X2 corregido por el procedimiento de Bonferroni, a través del método Monte Carlo (1000 simulaciones). El primer número se corresponde con el valor del estadígrafo, entre paréntesis los grados de libertad, seguido de la probabilidad (/ valor). Debajo de la diagonal: número absoluto de migrantes intercambiados en la parte superior, prueba de diferenciación por pares de colonias de anidación (ФST) basada en permutaciones no paramétricas, seguida de la probabilidad (/ valor). Los datos incongruentes entre ambas pruebas estadísticas por pares de colonias de anidación se representan en negrita, y en itálica, aquellos cuya probabilidad fue significativa. Colonias de anidación: QRMEX- Quintana Roo (México); SWCUB- suroeste de Cuba; USA: CSB- Cape San Blas, DT- Dry Tortugas, EA- Eglin Air Force Base, GE- Georgia, HI- Hutchinson Island, KI- Key Island, MB- Melbourne Beach, NJt- norte de Jetty, PE- Port Everglades, SC- Sarasota County, SJ- St Joseph's, SJt- sur de Jetty, y TB- Tyndall Beach. La generalidad de los datos de las colonias de anidación corresponden a Encalada et al. (1998) – Francisco y Bowen (2001), excepto en NJT y SJT (Francisco et al., 1999). Sureste de Florida Colonias QRMEX SWCUB 17.34 (7)/ 0.002 QRMEX 2.76 SWCUB 0.15/ 0.01 GE HI MB SJT NJT KI SC CSB SJ TB EA 19.72(5)/ 0 12.53(6)/ 0.01 12.94(5)/ 0.01 34.38(9)/ 0 18.45(5)/ 0 27.13(6)/ 0 12.07(5)/ 0.01 24.86(7)/ 0 14.73(5)/ 0.005 23.61(5)/ 0 18.76(6)/ 0.001 37.33(6)/ 0 58.77(5)/ <0.001 15.30(6)/ 0.006 8.97(6)/ 0.21 8.42(5)/ 0.20 27.24(8)/ 0 19.35(5)/ 0 32.20(7)/ 0 10.44(6)/ 0.07 27.13(7)/ 0 10.86(5)/ 0.11 24.15(5)/ 0 23.38(7)/ 0 41.74(6)/ 0 63.84(5)/ 0 11.62(4)/ 0.07 10.71(3)/ 0.04 38.59(7)/ 0 26.42(3)/ 0 43.15(5)/ 0 12.67(4)/ 0.02 41.76(6)/ 0 14.7(3)/ 0.02 33.98(3)/ 0 33.98(5)/ 0 57.1(4)/ 0 82.05(3)/ 0 1.18(2)/ 1.00 7.51(6)/ 0.39 4.87(3)/ 0.15 8.82(5)/ 0.10 2.19(3)/ 0.78 7.91(5)/ 0.13 1.67(2)/ 0.78 5.97(2)/ 0.04 6.38(4)/ 0.15 15.22(3)/ 0 29.11(2)/ 0 1.57(5)/ 0.73 2.04(2)/ 0.51 3.31(4)/ 0.63 0.80(2)/ 1 3.51(4)/ 0.50 1.25(1)/ 1 3.04(1)/ 0.14 4.49(3)/ 0.22 10.29(2)/ 0.003 20.66(1)/ 0.001 491(6)/ 0.62 8.21(48)/ 0.31 6.79(6)/ 0.46 7.61(6)/ 0.25 0.67(5)/ 1.00 3.02(5)/ 0.70 10.32(6)/ 0.16 10.12(5)/ 0.03 22.46(5)/ 0 1.28(4)/ 1.0 5.04(3)/ 0.14 5.24(5)/ 0.44 0.82(2)/ 0.75 1.33(2)/ 0.80 4.94(4)/ 0.40 6.22(3)/ 0.05 12.68(2)/ 0.001 6.63(4)/ 0.12 7.24(6)/ 0.22 1.27(4)/ 0.81 1.45(4)/ 0.87 7.39(5)/ 0.26 7.77(5)/ 0.08 14.28(4)/ 0 5.12(4)/ 0.25 1.39(2)/ 0.75 6.13(2)/ 0.02 5.30(3)/ 0.15 16.01(3)/ 0 30.00(2)/ 0 1.91(4)/ 0.87 3.76(4)/ 0.50 1.12(4)/ 0.95 9.20(4)/ 0.02 22.36(4)/ 0 1.30(1)/ 0.33 3.00(3)/ 0.55 6.37(2)/ 0.04 13.44(1)/ 0.007 3.54(3)/ 0.49 1.97(2)/ 0.68 4.20(1)/ 0.10 5.04(3)/ 0.14 15.51(3)/ 0.007 21.93 0.02/ 0.05 4.39 0.10/ 0.007 PE 1.22 0.29/ 0.02 Inf. -0.04/ 0.51 HI 0.55 0.48/ 0.001 11.51 0.75 Inf 0.04/ 0.20 0.40/ 0.007 -0.07/ 0.63 2.21 0.18/ 0.04 4.80 0.09/ 0.13 Inf -0.03/ 0.45 MB 0.58 0.47/ 0 2.00 0.20/ 0 0.58 0.46/ 0 SJT 0.29 0.63/ 0 1.39 0.26/ 0.004 0.32 0.61/0 3.15 0.14/ 0.07 Inf Inf -0.01/0.31 -0.04/ 0.59 NJT 0.39 0.56/ 0 1.27 0.28/ 0 0.39 0.56/ 0 2.40 0.17/ 0.05 21.09 0.02/ 0.23 1.22 Inf. 0.29/ 0.009 -0.01/ 0.33 0.60 0.45/ 0 SC CSB 1.72 0.23/ 0.01 0.61 0.45/ 0 Inf Inf -0.09/ 0.89 -0.07/ 0.69 9.28 0.05/ 0.22 0.28 2.55 0.38 21.80 0.64/ 0.005 0.38/ 0.08 0.57/ 0.002 0.02/ 0.33 0.11 0.16/ 0 SJ 2.62 0.16/ 0.007 0.58 0.46/ 0 3.27 0.77/ 0 0.86 0.37/ 0.03 0.29 2.54 0.39 22.29 0.63/ 0.002 0.17/ 0.09 0.56/ 0.001 0.02/ 0.23 TB Noroeste de Florida PE DT KI Suroeste de Florida DT EA 0.07 0.87/ 0 0.35 0.58/ <0.001 0.12 0.82/ 0 GE 0.02 0.95/ 0 0.18 0.73/ 0 0.06 0.89/ <0.001 Inf Inf -0.01/ 0.53 -0.05/ 1.00 5.93 0.08/ 0.10 2.86 3.85 0.11/ 0.09 0.15/ 0.05 Inf Inf Inf Inf 12.62 -0.05/ 0.62 -0.02/ 0.64 -0.03/ 0.53 -0.006/ 0.31 0.04/ 0.22 Inf -0.11/ 1.0 Inf -0.09/ 1.00 Inf Inf 70.13 -0.11/1.00 -0.08/ 0.81 0.007/ 0.36 Inf -0.09/ 0.93 2.22 0.18/ 0.15 8.29 0.06/ 0.13 Inf Inf -0.10/ 0.69 -0.07/ 0.43 0.48 0.33/ 0.02 0.09 0.08/ 0.09 17.55 0.03/ 0.33 Inf Inf Inf Inf 78.03 -0.11/ 0.41 -0.09/ 0.72 -0.10/ 0.70 -0.07/ 0.44 0.006/ 0.30 Inf -0.10/1.00 Inf -0.16/ 1.00 16.53 0.03/ 0.18 0.40 0.77 2.28 3.10 0.56/ 0.001 0.39/ 0.01 0.18/ 0.009 0.14/ 0.09 0.14 0.79/ 0 0.23 0.68/ 0 0.95 0.34/ 0 3.86 0.11/ 0.05 0.50 0.50/ 0 1.73 0.22/0.006 1.62 0.24/ 0.10 Inf -0.04/ 0.57 1.63 0.23/ 0.06 0.74 1.23 0.40/ 0.002 0.29/ 0.001 0.19 0.72/ 0 0.68 0.42/ 0 0.36 0.58/ 0.007 2.59 0.16/ 0.12 0.37 0.58/ 0.004 2.39(2)/ 0.41 22.14 0.02/ 0.16 El suroeste de Cuba sólo no está estructurado significativamente con las colonias de anidación Port Everglades y Hutchinson Island (sureste de la Florida). Tampoco están estructurados significativamente Key Island del suroeste de la Florida, ni Cape San Blas del noroeste de la Florida. También existe una incongruencia entre la significación de ambas pruebas para el suroeste de Cuba y Tyndall Beach, obteniéndose estructura genética significativa con el X2. Otras incongruencias ocurren entre Dry Tortugas versus Quintana Roo y Port Everglades. En la primera interacción el X2 es significativo a diferencia de la segunda, donde la significación es aportada por el FST. No obstante, Dry Tortugas y Quintana Roo están estructuradas con la generalidad de las colonias de anidación (85.71 y 92.86 %, respectivamente). Entre las colonias de las áreas de RESULTADOS____________________________________________________________________________________________ 52 anidación que integran el sur de la Florida no hay estructura genética significativa. Tampoco se encuentra significación entre las colonias de anidación del noroeste de la Florida versus la generalidad (71.88%) de las colonias de anidación de las otras áreas de la península. Las frecuencias relativas de los haplotipos presentes en las colonias de anidación de USA, a partir de datos originales de Francisco & Bowen (2001), Francisco et al. (1999) y Encalada et al. (1998), constituyen una variación clinal latitudinal de los haplotipos más distribuidos y representados de los linajes presentes en ambas costas de la península de la Florida (ver Fig. 4), i.e. el haplotipo CC-A1 del linaje Atlántico – península de la Florida aumenta con el incremento de la latitud mientras que el CC-A2 del linaje mediterráneos americano – Viejo Mundo disminuye. Esta variación tiene un punto de inflexión en la colonia de anidación más sureña Dry Tortugas, donde el haplotipo CC-A2 alcanza la frecuencia mayoritaria y en las colonias de anidación de Carolina del Sur y del Norte donde el haplotipo CC-A1 tiene frecuencia unitaria. En cada costa de la península de la Florida está correlacionado el grado de diferenciación genética ( Φ ST) con la distancia geográfica (costa del Golfo México: r=0.70, p<0.001; costa atlántica: r=0.73, p<0.001) y con la distancia neta promedio basada en el número de sustituciones nucleotídicas (costa del Golfo México: r=0.91, p<0.001; costa atlántica: r=0.92, p<0.001), a pesar de cierta dispersión de las interacciones (Fig. 10). La distancia geográfica también está correlacionada con la distancia neta promedio basada en el número de sustituciones nucleotídicas en ambas costas (costa del Golfo México: r=0.71, p<0.001; costa atlántica: r=0.79, p<0.001) (ver Fig. 10). Solamente se mantiene dependencia entre el Φ ST y la distancia neta promedio basada en el número de sustituciones nucleotídicas cuando se incluyen todas las colonias de anidación de USA (r=0.90, p<0.001), a pesar de cierta dispersión de las interacciones (Fig. 11). [ Φ ST versus distancia geográfica (r=0.18, p=0.05); distancia geográfica versus neta promedio basada en el número de sustituciones nucleotídicas (r=0.13, p=0.10)]. Sin embargo, cuando se aplican las pruebas de correlación analizando todas las colonias de anidación del Mediterráneo americano vuelve a obtenerse significación para el Φ ST versus distancia geográfica y dA, y para estas últimas (r=0.31, p=0.001; r=0.91, p<0.001; r=0.31, p<0.001; respectivamente), aunque con coeficientes de correlación menos altos en dos de las interacciones como consecuencia de una mayor dispersión de las interacciones. RESULTADOS____________________________________________________________________________________________ costa oeste 1,0 costa este 1,0 0,8 53 0,8 y= 0,6 8E 0.5 -3 .04 -0 0,6 0,2 7 0.0 Fst x.7 1 y=0 0,4 0,2 0,0 0,0 -0,2 -0,4 -100 0 100 200 300 400 500 600 700 dgeog 800 900 1000 -0,2 0 200 Distancia geográfica en km 1,2 400 600 800 1000 1200 1400 1600 dgeog 1,0 1,0 x .8 19 y= 0,8 + 08 0. 0,8 x .92 24 y= 0,6 .04 +0 0,6 0,4 Fst Fst Índice de diferenciación poblacional (ФSTFst) 0,4 0,4 0,2 0,2 0,0 0,0 -0,2 -0,4 -0,005 0,000 0,005 0,010 0,015 0,020 0,025 0,030 0,035 0,040 0,045 -0,2 -0,01 0,00 0,01 0,02 0,03 0,04 0,05 0,06 dneta promedio basada en el número de sustituciones nucleotídicas dneta Distancia neta (dA) 0,06 0,045 0,05 0,035 0,04 0,030 y= 0,025 x -4 9E 0.2 -0 .01 0,03 dneta dneta Distancia neta promedio (dA) 0,040 0,020 0,02 0,015 0,010 y= 0,005 -4x 6E 0.2 04 0.0 0,01 0,00 0,000 -0,005 -100 0 100 200 300 400 500 dgeog 600 700 800 900 1000 -0,01 -200 0 200 Distancia geográfica en km 400 600 800 dgeog 1000 1200 1400 1600 1800 95% confianza Fig. 10. Regresión lineal entre la diferenciación genética ( Φ ST estimado a partir de haplotipos de la RNC del mtDNA, ca. 380 pb) versus las distancias geográfica y neta promedio basada en el número de sustituciones nucleotídicas, y entre ambas distancia, por pares de colonias de anidación de C. caretta localizadas en la península de la Florida. Líneas: continua, curva de regresión lineal; discontinua, límite de confianza. RESULTADOS____________________________________________________________________________________________ 54 Mediterráneo americano 1,2 0,06 1,0 Distancia neta promedio (dA) 0,05 0,8 0,04 -3x + 0 .14 0,03 dneta E y=0.14 0,4 y=0.6 0,02 0,2 4 + 0.00 7E-5x 0,01 0,0 0,00 -0,2 -0,4 0 300 600 900 1200 1500 1800 dgeog 2100 2400 2700 -0,01 0 300 Distancia geográfica en km 600 900 1200 1500 1800 2100 2400 1,2 .4 19 y= 1,0 + 6x 8 0.0 2700 dgeog 1,2 1,4 USA 1,0 2 y= 6x 1.9 + 0.0 6 0,8 0,8 0,6 0,6 Fst Fst Índice de diferenciación poblacional (ФST)Fst 0,6 0,4 0,4 0,2 0,2 0,0 0,0 -0,2 -0,2 -0,4 -0,01 0,00 0,01 0,02 0,03 dneta 0,04 0,05 0,06 -0,4 -0,01 0,00 0,01 0,02 0,03 0,04 0,05 0,06 95% confianza Distancia neta promedio basada en el número de sustituciones nucleotídicas (dA) Fig. 11. Regresión lineal entre la diferenciación genética ( Φ ST estimado a partir de haplotipos de la RNC del mtDNA, ca. 380 pb) versus las distancias geográfica y neta promedio basada en el número de sustituciones nucleotídicas por pares de colonias de anidación de C. caretta localizadas en el Mediterráneo americano. Líneas: continua, curva de regresión dneta lineal; discontinua, límite de confianza. La estructura genética encontrada se confirma por un valor significativo de Φ ST en todas las hipótesis de estructuración jerárquicas (Tabla 7), con un porcentaje de variación casi dos veces mayor dentro que entre colonias de anidación. A esta variación contribuyen significativamente el 84% de los SPs integrados por los loci no diagnósticos (32: Φ ST=0.38; 35: Φ ST=0.38; 37: Φ ST=0.38; 51: Φ ST=0.35; 53: Φ ST=0.38; 63: Φ ST=0.37; 78: Φ ST=0.17; 104: Φ ST=0.38; 161: Φ ST=0.36; 162: Φ ST=0.42; 210: Φ ST=0.38; 230: Φ ST=0.38; 244: Φ ST=0.42; 246: Φ ST=0.38; 259: Φ ST=0.38; 294: Φ ST=0.38; 314: Φ ST=0.38; 315: Φ ST=0.38; 317: Φ ST=0.38; 327: =0.38; 355: Φ ST=0.33; todos con p<0.001) entre haplotipos. De éstos, la mayoría de los loci son diagnóstico entre linajes excepto los SPs 78, 162 y 244, y los loci 32, 63 y 161 que son “diagnósticos” solamente RESULTADOS____________________________________________________________________________________________ dentro 55 linaje Tabla 7. Resultados del análisis jerárquico de varianza molecular bajo del tres niveles hipotéticos de estructura geográfica en colonias de mediterráneos americano – anidación de C. caretta del Mediterráneo americano. El análisis se basó Viejo Mundo de los haplotipos CC-A20, 12 y 8 respectivamente. A su vez, los dos primeros haplotipos son endémicos colonias de de las anidación Melbourne Beach ( USA) y San Felipe (suroeste cubano). Al agruparse las muestras de las colonias de anidación del suroeste cubano en una población respecto a las preestablecidas, i.e. en haplotipos de la RNC del mtDNA (ca. 380 pb). Áreas/ colonias de anidación: QRMEX- Quintana Roo (México); SWCUB- suroeste de Cuba/ CL- Cayo Largo del Sur, GU- Guanahacabibes, IJ- Isla de la Juventud, y SF- San Felipe; USA: DT- Dry Tortugas; NEFL-NC- noreste de la Florida - Carolina del Norte/ GE- Georgia; NWFLnoroeste de la Forida/ CSB- Cape San Blas, EA- Eglin Air Force Base, SJ- St Joseph's y TB- Tyndall Beach; SEFL- sureste de la Florida/ HI- Hutchinson Island, MB- Melbourne Beach, PE- Port Everglades, NJt- norte de Jetty, y SJt- sur de Jetty; SWFL- suroeste de la Florida/ KI- Key Island y SC- Sarasota County. Los datos de QRMEX se correspoden a Encalada et al. (1998), y los demás de USA a estos autores y a Francisco & Bowen (2001), y Francisco et al. (1999). p, probabilidad. Partición observada p Φ estadísticos Varianza % total I. Estructura hipotética: colonias de anidación del SWCUB (CL, GU, IJ y SF) y las de la región (CSB, DT, EA, GE, HI, KI, MB, NJt, PE, QRMEX, SC, SJ, SJt y TB) constituyen una población panmicta Entre poblaciones 1.95 37.14 0.37 <0.001 Dentro de las poblaciones 3.31 62.86 II. Estructura hipotética: colonias de anidación del SWCUB constituyen una unidad demográfica respecto a SFL (HI, MB, PE, NJt, SJt, KI y SC), NEFL-NC (GE), NWFL (CSB, EA, SJ, y TB), DT y QRMEX Entre regiones 2.38 39.38 0.39 <0.001 Entre poblaciones/ regiones 0.18 3.01 0.05 0.001 Dentro de poblaciones 3.46 57.61 0.42 <0.001 III. Estructura hipotética: colonias de anidación del SWCUB constituyen una unidad demográfica respecto a SEFL (HI, MB, PE, NJt y SJt), SWFL (KI y SC), NEFL-NC (GE), NWFL (CSB, EA, SJ, y TB), DT y QRMEX Entre regiones 2.02 36.91 0.37 0.002 Entre poblaciones/ regiones 0.18 3.22 0.05 0.54 Dentro de poblaciones 3.28 59.87 0.40 <0.001 Fuente de variación sur, noroeste y noreste de la Florida; Dry Tortugas; y Quintana Roo; se obtienen porcentajes de variación semejantes dentro de las colonias de anidación, entre colonias de anidación por regiones y entre regiones (ver Tabla 7), a los cuales contribuyen los mismos loci anteriormente citados exceptuando el locus 78 para Φ SC y Φ CT. El locus 96 se vuelve significativo ( Φ CT=0.06, p=0.001) para el caso del índice de diferenciación entre regiones. A pesar de que la probabilidad del índice de diferenciación entre grupos rechaza la hipótesis nula, el índice de diferenciación entre poblaciones dentro de los grupos también rechaza la hipótesis nula. Por consiguiente, el escenario hipotético II se desecha. Sin embargo, cuando se segregan las colonias de anidación del oeste y este del sur de la Florida (escenario hipotético III), el índice de diferenciación entre poblaciones dentro de grupos acepta la hipótesis nula manteniéndose significativo sólo el locus no diagnóstico 312. Adicionalmente, el locus 188 se vuelve significativo ( Φ CT=0.05, p=0.02) para el caso del índice de diferenciación entre regiones. Por consiguiente, este escenario es escogido como la estructura poblacional más probable. RESULTADOS____________________________________________________________________________________________ 56 IV.3 Diversidad genética. IV.3.1 Chelonia mydas. La población de anidación del suroeste cubano se caracteriza por un alto porcentaje de endemismo haplotípico (71.43%) y un moderado porcentaje de individuos endémicos (32.14%) en relación a las poblaciones de anidación de la especie. Dentro de la población de anidación del suroeste de Cuba, la colonia de anidación de la península de Guanahacabibes presenta una diversidad genética que aproximadamente duplica la de la colonia de anidación de la Cayería San Felipe (Tabla 8), donde sólo se encontraron dos haplotipos. Esta colonia tiene una diversidad genética similar a la población de anidación de la Florida. A pesar de que la diversidad haplotípica de la colonia de anidación de Guanahacabibes es tan alta como la de la población de anidación de Quintana Roo, para un número igual de haplotipos, la diversidad nucleotídica es aproximadamente la mitad y el número de sitios segregativos es casi un cuarto. Esta proporción se mantiene cuando los datos de las colonias de anidación del suroeste cubano son integrados en una población de anidación. Solamente disminuye la diversidad haplotípica. Tabla 8. Medidas de diversidad genética e índices demográficos de colonias/ poblaciones de anidación de C. mydas localizadas en el Mediterráneo americano. El análisis se basó en haplotipos de la RNC del mtDNA (ca. 490 pb). Los datos de las colonias con superíndices 1, 2 y 3 son según Allard et al. (1994), Encalada et al. (1996) y Lahanas et al. (1998) respectivamente. El tamaño efectivo de las colonias/ poblaciones de anidación está calculado por estación reproductora. Los tamaños censales de hembras por estación reproductora están tomados según compilación de Seminoff (2002), excepto para la colonia de anidación de Guanahacabibes (Ibarra, 2005). D, Prueba de Tajima; Fs, Prueba de Fu; H, número de haplotipos; Ĥ , diversidad haplotípica; IC, intervalo de confianza para un alpha igual a 0.05; N, tamaño muestral; Ne(f), tamaño efectivo estimado de la colonia de anidación; Nef, tamaño censal de hembras de la colonia de anidación; N ( θˆ 0), tamaño censal antes de la expansión; p, probabilidad; S, suma de cuadrados; s, sitios segregativos; Sd, desviación estándar; superíndice a, indica parámetro del modelo de expansión súbita estimado sin la contribución del haplotipo A5; t, tiempo donde comenzó la expansión expresado en 1000 años (KA); θˆ , estimador de theta: subíndices S y 0 se corresponden con el número de sitios segregativos y el tamaños inicial de la población durante la expansión respectivamente; πˆ , diversidad nucleotídica; τ , tiempo de la expansión medido en 1/2u de generaciones; !, el procedimiento del mínimo cuadrado para ajustar el modelo de distribución desigual y la distribución observada no convergió después de los 1800 pasos. Distribución desigual Ne(f) Nef (Modelo de expansión súbita) Región de D (p)/ N H N Ĥ ±Sd( Ĥ ) πˆ n±Sd( πˆ n) s Fs (p) Histórico colecta Ssd (p) t(IC) θˆ S/ θˆ π / actual ( θˆ 0) τ Guanahacabibes (Cuba) 17 7 0.7647 ± 0.0943 0.0024 ± 0.0018 4 -0.02 (0.48)/ 709/ 708 -3.34 (0.002) / 213 ! San Felipe (Cuba) 11 2 0.4364 ± 0.1333 0.0018 ± 0.0015 2 0.85 (0.81)/ 409/ 525 2.01 (0.83) ? 2.9 0.16 (0.02) - - 28 7 0.6481 ± 0.0890 0.0022 ± 0.0017 4 0.07 (0.58)/ 616/ 635 -2.67 (0.03) ? 1.5 0.03 (0.13) 131 (21-312) 62 Península Florida1 24 (USA) 3 0.5616 ± 0.0468 0.0012 ± 0.0011 2 0.28 (0.72)/ 408/ 364 366/ 759 0.8 0.29 (0.51) 0.04 (0.002) - - Cuba suroeste 2 a a Quintana Roo (México) 20 7 0.8158 ± 0.0575 0.0051 ± 0.0032 14 -1.36 (0.08)/ -0.56 (0.38) 2,364/ 1,500 82/ 298 ! 1.8a 0.02 (0.22) 154 (40-335) 49a Tortuguero1, 3 (Costa Rica) 41 2 0.0488 ± 0. 0459 0.0001 ± 0.0002 -1.12 (0.14)/ -1.47 (0.03) 140/ 29 25,000/ 3.0 58,000 0 (0.16) 253 (37-254) 4 1 RESULTADOS____________________________________________________________________________________________ 57 La variación observada en las secuencias parciales analizadas es selectivamente neutral para cada población de anidación del Mediterráneo americano (ver Tabla 8). En todas las poblaciones de anidación, la composición de A+T es aproximadamente el triple que la de G+C. La media y la variación sobre la media de cada base nucleotídica son: A (35.89±0.03), T (30.74±0.08), G (13.80±0.03) y C (19.56±0.07). Entre las poblaciones de anidación los porcentajes de composición nucleotídica son similares. Los tamaños efectivos de las poblaciones de anidación estimados según theta, ya sea por el número de sitios segregativos o por la diversidad nucleotídica (π), son del mismo orden de magnitud (ver Tabla 8). La población de anidación de Tortuguero es la excepción, i.e., el tamaño efectivo considerando theta a partir del número de sitios segregativos es aproximadamente el cuádruplo. El tamaño efectivo estimado según theta a partir de la diversidad nucleotídica ( θˆ π) para la colonia de anidación de la península de Guanahacabibes es tres veces mayor que el tamaño censal actual de hembras. Para la población de Quintana Roo la desproporción es cinco veces mayor. En las otras dos poblaciones de anidación la desproporción es en sentido inverso: la población de anidación de la Florida tiene un tamaño censal actual de hembras que duplica el tamaño efectivo ( θˆ π), mientras que en la población de anidación de Tortuguero el tamaño censal actual de hembras es dos mil veces mayor que el tamaño efectivo ( θˆ π). Sólo en la población de la Florida, el tamaño efectivo ( θˆ π) equivale al tamaño censal histórico. La prueba de distribución desigual del número observado de diferencias por pares de secuencia se cumple en la población del suroeste cubano y en la de Tortuguero (Fig. 12). En ambos casos la curva es unimodal con moda en cero diferencias. En la primera y contrariamente a la segunda, la expansión fue dos veces más reciente y a partir de un tamaño efectivo moderadamente pequeño, a pesar de que los intervalos de confianza del tiempo de expansión se superponen en cierta medida entre las poblaciones de anidación (ver Tabla 8). No obstante, este resultado no se corresponde con la prueba de Fu (ver Tabla 8), sensible a la expansión demográfica, considerando que la significación de ésta a nivel 5% se obtiene para valores de probabilidad menores de 0.02 (Schneider et al., 2000). Cuando se analiza el procedimiento del mínimo cuadrado para ajustar la distribución RESULTADOS____________________________________________________________________________________________ desigual esperada y la anidación Guanahacabibes de y la población de anidación de Quintana Roo, no hay convergencia. Cuando la contribución haplotipo del CM-A5 se Frecuencia relativa (%) de 90 90 70 70 50 50 30 30 0 prueba de distribución desigual de frecuencias. Sin embargo, la prueba 3 0 1 Frecuencia modelada 95% confianza Quintana Roo1 50 10 no significación de la 2 70 modelo súbita se cumple por la 1 90 30 expansión 10 10 elimina en esta última, el de Tortuguero2, 3 Cuba suroeste observada en la colonia 58 0 1 2 3 4 5 Número de diferencias Fig. 12. Distribución desigual del número de diferencias observado entre pares de haplotipos de poblaciones de anidación de C. mydas del Mediterráneo americano. El análisis se basó en haplotipos de la RNC del mtDNA (ca. 490 pb). La N de las poblaciones con superíndices 1, 2 y 3 son según Encalada et al. (1996), Allard et al. (1994) y Lahanas et al. (1998) respectivamente. La distribución desigual representada en la población de anidación mexicana, excluye al haplotipo CM-A5. de Fu no muestra significación estadística (Fs=-0.80; p=0.29). En la colonia de anidación de San Felipe no se cumple el modelo de expansión súbita, pues la desviación de la suma de cuadrados es significativa al igual que en la población de anidación de la Florida. IV.3.2 Caretta caretta. La población de anidación del suroeste cubano se caracteriza por bajos porcentajes de endemismo haplotípico (16.67%) y de individuos endémicos (2.78%) en relación a las poblaciones de anidación de la especie. Dentro de la población de anidación del suroeste de Cuba, la colonia de anidación de la Cayería San Felipe presenta una diversidad genética que aproximadamente duplicó la de las restantes colonias de anidación del Archipiélago de los Canarreos (Tabla 9), en correspondencia con un mayor número de haplotipos. Contrariamente, la diversidad nucleotídica es más pequeña en un orden fraccionario, en correspondencia con un pequeño número de sitios segregativos. La diversidad genética de las colonias de anidación del suroeste cubano es alta cuando se integran en una población de anidación al igual que en las demás poblaciones de la región, salvo el noreste de la Florida hasta Carolina del Norte. RESULTADOS____________________________________________________________________________________________ 59 Tabla 9. Medidas de diversidad genética e índices demográficos de colonias/ poblaciones de anidación de C. caretta localizadas en el Mediterráneo americano. El análisis se basó en haplotipos de la RNC del mtDNA (ca. 380 pb). Los datos de las unidades demográficas con superíndices 1, 2 y 3 son según Encalada et al. (1998), Francisco et al. (1999) y Francisco & Bowen (2001) respectivamente. El tamaño efectivo de las colonias/ poblaciones de anidación está calculado por estación reproductora. Los tamaños censales de hembras por estación reproductora están tomados según compilación de Ehrhart et al. (2003). D, Prueba de Tajima; Fs, Prueba de Fu; H, número de haplotipos; Ĥ , diversidad haplotípica; IC, intervalo de confianza para un alpha igual a 0.05; N, tamaño muestral; Ne(f), tamaño efectivo estimado de la colonia de anidación; Nef, tamaño censal de hembras de la colonia de anidación; N ( θˆ 0), tamaño censal antes de la expansión; p, probabilidad; S, suma de cuadrados; Sd, desviación estándar; s, sitios segregativos; t, tiempo donde comenzó la expansión expresado en 1000 años (KA); θˆ , estimador de theta: subíndices S y 0 se corresponden con el número de sitios segregativos y el tamaños inicial de la población durante la expansión respectivamente; πˆ , diversidad nucleotídica; τ , tiempo de la expansión medido en 1/2u de generaciones; !, el procedimiento del mínimo cuadrado para ajustar el modelo de distribución desigual y la distribución observada no convergió después de los 1800 pasos. Distribución desigual Ne(f) Nef (Modelo de expansión súbita) Región de D (p)/ N H N Ĥ ±Sd( Ĥ ) πˆ n±Sd( πˆ n) s Fs (p) Histórico colecta Ssd (p) t(IC) θˆ S/ θˆ π - actual ( θˆ 0) τ Cayo Largo del Sur 8 (Cuba) 2 0.5357 ± 0.1232 0.0286 ± 0.0166 19 2.02 (0.99)/ 9.97 (0.99) 8,667/ 12,711 ? 6.0 0.37 (0.002) - - 10 2 0.4667 ± 0.1318 0.0263 ± 0.0149 20 1.50 (0.95)/ 11.01 (0.99) 8,356/ 11,689 ? 3.1 0.29 (0.009) - - Isla de la Juventud 12 (Cuba) 2 0.4091 ± 0.1333 0.0218 ± 0.0123 19 1.03 (0.88)/ 11.04 (1) 7,467/ 9,689 ? 3.0 0.23 (0.009) - - San Felipe (Cuba) 6 4 0.8000 ± 0.1721 0.0032 ± 0.0027 3 -0.44 (0.34)/ -1.45 (0.04) 1,556/ 1,422 ? ! - - - Cuba suroeste 36 6 0.5333 ± 0.0890 0.0205 ± 0.011 21 1.75 (0.95)/ 7.98 (0.98) 6,000/ 9,111 100 6.0 0.12 (0.12) 42 (7-36) 1,239 20 5 0.6526 ± 0.0927 0.0024 ± 0.0019 5 -1.14 (0.14)/ -1.43 (0.10) 1,689/ 1,022 <33 ! - - - 58 4 0.2541 ± 0.0735 0.0079 ± 0.0046 21 -1.20 (0.09)/ 5.35 (0.96) 5,378/ 3,289 3.0 0.03 (0.07) 11 (1-15) 242 44 5 0.6480 ± 0.0429 0.0280 ± 0.0144 23 3.26 (0.99)/ 15.02 (0.99) 6,267/ 12,444 1,700 6.0 0.20 (0.02) - - 114 10 0.5595 ± 0.0302 24 3.50 (1)/ 12.52(0.98) 5,377/ 11,778 18,967 6.2 0.25 (0.005) - - 47 4 0.3959 ± 0.0818 0.0199 ± 0.0105 22 1.64 (0.96)/ 13.52 (1) 5,911/ 8,844 3.0 0.12 (0.06) 11 (2-17) 264 100 2 0.0200 ± 0.0195 0.0011 ± 0.0011 20 -2.59 (0.001)/ 1.81 (0.70) 4,578/ 474 3.0 0.0005 (0.01) - - Guanahacabibes (Cuba) 1 Quintana Roo (México) Dry Tortugas (USA) 3 Florida suroeste (USA) 1,3 1,2,3 Florida sureste (USA) Florida noroeste (USA) Florida noreste (USA) 0.0265 ±0.0135 1,3 1 2,067 La variación observada en las secuencias parciales analizadas es selectivamente neutral para cada población de anidación del Mediterráneo americano y la costa atlántica de la Florida (ver Tabla 9), exceptuando al noreste de la Florida según el estimador de Tajima. En todas las poblaciones de anidación la composición de A+T es aproximadamente el doble de G+C. La media y la variación sobre la media de cada base nucleotídica son: A (34.01±0.09), T (34.79±0.10), G (14.95±0.36) y C (16.25±0.18). Entre las poblaciones de anidación los porcentajes de composición nucleotídica son similares. Los tamaños efectivos de las poblaciones de anidación estimados según theta, son mayores considerando la diversidad nucleotídica que el número de sitios segregativos en la generalidad de los casos (ver Tabla 9), excepto en las poblaciones del noreste de la RESULTADOS____________________________________________________________________________________________ 60 Florida y Dry Tortugas donde es a la inversa, y en la colonia de anidación de San Felipe y en la población de anidación de Quintana Roo donde son del mismo orden de magnitud. Los tamaños efectivos estimados según theta a partir de la diversidad nucleotídica ( θˆ π) para las poblaciones de anidación del suroeste cubano, Quintana Roo y el suroeste de la Florida son ca. noventa, treinta y siete veces mayores que el tamaño censal actual de hembras respectivamente. En las poblaciones de anidación del sureste y noreste de la península de la Florida la desproporción es en sentido inverso, siendo el tamaño censal actual de hembras del noreste ca. el cuádruplo del tamaño efectivo ( θˆ π). Dry Tortugas2 Frecuencia relativa (%) 90 Florida noroeste1,2 90 70 70 50 50 30 30 10 10 0 1 2 3 0 20 1 2 3 Cuba suroeste 90 4 20 21 Frecuencia modelada 95% confianza 70 50 30 10 0 1 2 3 4 5 6 18 19 20 Número de diferencias Fig. 13. Distribución desigual del número de diferencias observado entre pares de haplotipos de poblaciones de anidación de C. caretta del Mediterráneo americano. El análisis se basó en haplotipos de la RNC del mtDNA (ca. 380 pb). Los datos de las unidades demográficas con superíndices 1 y 2 son según Encalada et al. (1998) y Francisco y Bowen (2001) respectivamente. La prueba de distribución desigual del número observado de diferencias por pares de secuencia de frecuencias se cumple en las poblaciones de anidación del suroeste cubano, Dry Tortugas y noroeste de la península de la Florida (Fig. 13). La curva es unimodal con moda en cero diferencias. En la población de anidación del suroeste cubano la expansión ocurrió cuatro veces antes, previa a la última glaciación, y a partir de un tamaño efectivo grande, a pesar de que los intervalos de confianza del tiempo de expansión se superponen en cierta medida entre las poblaciones de anidación (ver Tabla 9). No obstante, este resultado no se corresponde con la prueba de Fu (ver Tabla 9), sensible a RESULTADOS____________________________________________________________________________________________ 61 la expansión demográfica. Cuando se analiza el procedimiento del mínimo cuadrado para ajustar la distribución desigual esperada y la observada en la población de anidación de Quintana Roo, no hay convergencia. IV.4 Relaciones filogenéticas y filogeográficas. IV.4.1 Chelonia mydas. Las relaciones filogenéticas entre los haplotipos determinados dA/2 (%) T (años) 100% 0.54 450,000 0.48 400,000 0.42 350,000 en las poblaciones de anidación, 0.36 300,000 estimadas a partir 0.30 250,000 de 0.24 200,000 0.18 150,000 0.12 100,000 0.06 50,000 métodos de distancia, permite distinguir sólo dos agrupaciones (Fig. 14). Una integrada por el haplotipo CM-A5, 3 3 27 2, 3 28 56 57 1 48 Cuba suroeste 1 2 3 Florida 3 15 1 1 18 16 17 5 1 Quintana Roo Fig. 14. Diagrama sobre las relaciones filogenéticas entre haplotipos y poblaciones perteneciente linaje 4 Tortuguero al de anidación de C. mydas del Mediterráneo americano, estimadas con métodos de Atlántico reconstrucción sobre la base de matrices de distancia de secuencias de la RNC del mtDNA (ca. 490 pb). Reconstrucción sobre la base del número neto de sustituciones nucleotídicas Sur (Encalada et al., 1996), y la otra que incluye a todos los haplotipos del entre dos poblaciones (Nei & Li, 1979) y p estimada mediante el método Tamura (Tamura, 1992). Sólo se indica aquel porcentaje de bootstrap mayor que 50 %. Los trazos continuos definen la divergencia entre los haplotipos y los discontinuos entre las poblaciones. Los trazos oscuros representan haplotipos no compartidos entre poblaciones de anidación y los grises, haplotipos compartidos por varias poblaciones de anidación. La N de las poblaciones con superíndices 1, 2 y 3 son según Encalada et al. (1996), Allard et al. (1994) y Lahanas et al. (1998) respectivamente. dA/2, valor medio de aglomeración; T, tiempo de divergencia. Haplotipos: CM-A1 – 4 (Allard et al., 1994); CM-A5 (Lahanas et al., 1994); CM-A15 – 18 (Encalada et al., 1996); CM-A27 y 28 (Bjorndal y Bolten, inscritos en el GenBank 2001); CM-A48, 56 y 57 (Espinosa et al., inscritos en el GenBank 2003). linaje mediterráneos americano –Viejo Mundo, cuya topología interna es variable en correspondencia con los bajos valores de bootstrap determinados en cada reconstrucción. La generalidad de los haplotipos diverge de su ancestro más cercano entre los 180 000 y los 93 000 A, exceptuando al haplotipo CM-A5, que lo hace tempranamente en el orden de los 800 000 A. RESULTADOS____________________________________________________________________________________________ 62 Entre las poblaciones de anidación se reconocen tres agrupaciones, cuya divergencia es al menos tres veces más reciente que la de los haplotipos determinados en las mismas (ver Fig. 14). La población de Tortuguero está separada de las restantes por una dA=0.06%, a partir de la cual se estima un tiempo de divergencia en el orden de los 30 000 A. La población de Quintana Roo está separada de la población del suroeste cubano y de la Florida por una dA dos veces y media menor que la divergencia de la población de anidación de Tortuguero. Con este dato se estima un tiempo de divergencia de 11 000 años aproximados para la población de anidación de Quintana Roo. Las poblaciones de anidación del suroeste cubano y de la Florida están separadas por una dA dos veces menor que la que separa a Quintana Roo de éstas. Esta distancia representa aproximadamente unos 5000 años de divergencia. Las colonias de anidación del suroeste cubano aún no evidencian internamente señales de divergencia. Las posiciones de las poblaciones de anidación de Quintana Roo y Tortuguero se intercambian cuando las relaciones filogenéticas entre las poblaciones de anidación se reconstruyen con el método de NJ (Saitou & Nei, 1987). En el NCPA, la anidación de los clados (0 – pasos) concluye en agrupaciones de haplotipos con orígenes diferentes exceptuando los clados 1 – 3 y 1 – 4, cuyos haplotipos son endémicos de la población de anidación del suroeste cubano (Fig. 15a). En el clado más interno (1 – 1) se encuentra el segundo haplotipo más distribuido y mejor representado del linaje (CM-A1), agrupado con otros haplotipos endémicos de las poblaciones de anidación del suroeste cubano, Florida y Quintana Roo. Este haplotipo es reconocido como ancestro del linaje mediterráneos americano – Viejo Mundo. El clado 1 – 2 tiene una integración similar, pero con el haplotipo más ampliamente distribuido y mejor representado (CM-A3) agrupado con otros haplotipos endémicos de las poblaciones de anidación de Quintana Roo y Tortuguero. En el clado restante del nivel (1 – 5) se agrupan haplotipos endémicos de las poblaciones de Quintana Roo y Lara. Entre la generalidad de los clados procesados estadísticamente se encuentra una asociación geográfica significativa (Fig. 15b), en correspondencia con una dependencia entre la representatividad y distribución de los haplotipos en las colonias de muestreo. RESULTADOS____________________________________________________________________________________________ a) 57 1-2 excepción, ya que la de no 17 1-5 nula 48 aceptada. En el clado 1 – se infiere un 15 distancias larga para de las que resultan: el segundo haplotipo 2 E 18 E 48 I–E representado dentro del y en el clado del clado, cuanto a y larga la en menos las externas dentro del clado anidado. En el clado 1 – 5 también se infiere un flujo genético restringido comprometido espacio en con dispersión, que 27 el alguna abarcó largas distancias áreas intermedias sobre no ocupadas por la especie, o en el tiempo con un flujo Clados de 1 – paso Clado Dc Dn Dn L 533.55 490.97 453.57 460.07 72.22 P 1,818.83 P 2,096.72 P 543.41 1-2 I 1,138.88 56 y 57 dentro de la misma población 1-3 I 0 1,650.71 27 y 28 dentro de la misma población 1-4 I 0 1,650.71 1-5 I 6,379.44 2 – 3 – 4 (No) 3I E 4 E 15 I–E 1,146.16 0 0 1,146.16 1,136.56 772.22 951.57 274.67 X2=12.47, p=0.15 P Ho Aceptada distancia media entre las posiciones internas 1-4 1-1 I anidado, pequeña para el haplotipo CM-A48 dentro Haplotipos Dc L 560.61 0 0 P 0 L 560.61 X2=23.26, p<0.001 más ampliamente distribuido y clado 28 4 flujo restringido con aislamiento b) Clado 1I por distancia, a partir de la E significación 16 14 x asociación geográfica es 1 13 2 1 3 hipótesis 63 56 1-1 18 El clado 1 – 2 es la 1-3 13I E 14 E 16 E 17 I–E P 0 0 0 0 0 P 4,306.12 2,870.75 9,569.17 L 9,985.22 P -3,796.46 X2=12.00, p=0.008 2 – 3 – 5 – 6 – 7 – 8 (Sí) L 8,816.07 L X2=143.23, p<0.001 2 – 3 – 5 – 15 (No) Fig. 15. Análisis filogeográfico de clados anidados del linaje de C. mydas representado en poblaciones de anidación de los mediterráneos americano y del Viejo Mundo, sobre la base de haplotipos de la RNC del mtDNA (ca. 490 pb). a) Árbol de haplotipos con diseño de clados anidados. b) Resultados del NCPA. Cuadrado, identifica al haplotipo ancestral; círculo, identifica al iésimo haplotipo; – representa una mutación, con un cuadrado una transversión y con un asterisco (*), una adición (CAATGG); figuras geométricas con líneas finas indican la relación entre haplotipos mediada por una mutación; con líneas gruesas indican 1 paso entre los clados, equivalente al total del cladograma. El tamaño de las figuras geométricas es proporcional a la representatividad del haplotipo. Dc y Dn, distancias promedio dentro del clado y en el clado anidado respectivamente; I y E, posición interna o externa del clado; I – E, distancia promedio entre los clados interior menos exterior dentro de un clado anidado; superíndices P y L, junto con datos sombreados, indican que la distancia es significativamente pequeña o larga. En el lugar de cada Ho rechazada se refieren números correspondientes al empleo de la clave de inferencia biológica según Templeton (2004). Haplotipos: CM-A1 – 4 (Allard et al., 1994); CM-A13 – 18 (Encalada et al., 1996); CM-A27 y 28 (Bjorndal & Bolten, inscritos en el GenBank 2001); CM-A48, 56 y 57 (Espinosa et al., inscritos en el GenBank 2003). RESULTADOS____________________________________________________________________________________________ 64 genético pasado seguido de extinciones en poblaciones intermedias, a partir de la significación de las distancias pequeñas del haplotipo CM-A13 dentro del clado y del clado anidado, larga del haplotipo CM-A17 en el clado anidado y pequeña distancia media entre las posiciones internas menos las externas dentro del clado anidado. Entre los clados anidados totalmente (clados de 1 – paso) se infiere una fragmentación pasada y colonización o movimientos a largas distancias una de estas dos últimas variantes, a partir de la significación de distancias pequeñas dentro de los clados 1 – 1 y 1 – 2 y en éstos anidados, y largas dentro del clado 1 – 5 y en éste anidado. IV.4.2 Caretta caretta. Las relaciones filogenéticas entre los haplotipos determinados en las poblaciones de anidación, estimadas a partir de métodos de distancia, permite distinguir sólo dos agrupaciones (Fig. 16). Una integrada por los haplotipo CC-A1, 11 y 14, pertenecientes al linaje Atlántico – península de la Florida, y la otra que incluye a la mayoría de los haplotipos de origen conocido del linaje mediterráneos americano – Viejo Mundo, cuya topología interna es variable en correspondencia con los bajos valores de bootstrap determinados en cada reconstrucción. La generalidad de los haplotipos diverge de su ancestro más cercano entre los 158 000 y los 500 000 años dentro de cada linaje. Entre ambos linajes la divergencia es en el orden de los 3 MA. Entre las poblaciones de anidación se reconocen cinco agrupaciones, de las cuales los grupos distintivos de poblaciones de anidación de la península de la Florida y el Mediterráneo americano divergen dos veces más reciente que cuando ocurrió la diversificación de los haplotipos dentro de cada linaje (ver Fig. 16). Las agrupaciones de poblaciones de anidación sureñas y norteñas dentro del grupo de la península de la Florida divergen en el mismo orden en que los haplotipos divergieron dentro de cada linaje. Las demás poblaciones de anidación divergen antes de la diversificación de los haplotipos dentro de cada linaje. Las poblaciones de anidación norteñas y sureñas de la península de la Florida están separadas por una dA=0.74%, a partir de la cual fue estimado un tiempo de divergencia en el orden de los 440 000 A. Dentro de cada agrupación latitudinal, las poblaciones de anidación están separadas por una dA=0.52% mucho mayor entre las poblaciones de anidación norteñas respecto a la dA=0.02% RESULTADOS____________________________________________________________________________________________ dA/2 (%) DT3 X SEFL1,2,3 SWCUB 1 1 14 1 11 14 SWFL1,3 1 14 NEFL1 NWFL1,3 1 1 65 T (años) 0.06 100,000 0.12 78% 200,000 0.18 0.24 300,000 0.30 400,000 0.36 500,000 0.42 1.12 1,300,000 2.76 3,000,000 100% 1.12 1,300,000 0.42 500,000 0.36 400,000 0.30 0.24 300,000 0.18 200,000 0.12 100,000 0.06 2 3 8 10 9 QRMEX1 2 10 9 DT3 2 8 12 10 SWCUB 2 3 5 7 10 11 13 20 SEFL1,2,3 2 3 7 SWFL1,3 2 NEFL1 2 3 7 NWFL1,3 Fig. 16. Diagrama sobre las relaciones filogenéticas entre haplotipos y unidades demográficas de C. caretta del Mediterráneo americano, estimadas con métodos de reconstrucción sobre la base de matrices de distancia de secuencias de la RNC del mtDNA (ca. 380 pb). Reconstrucción sobre la base del número neto de sustituciones nucleotídicas entre dos poblaciones (Nei & Li, 1979) y p estimada mediante el método Tamura (Tamura, 1992). Sólo se indica aquel porcentaje de bootstrap mayor que el 50 %. Los trazos continuos definen la divergencia entre los haplotipos y los discontinuos entre las poblaciones. Los trazos oscuros representan haplotipos no compartidos entre poblaciones de anidación y los grises, haplotipos compartidos por varias poblaciones de anidación. La N de las unidades demográficas con superíndices 1, 2 y 3 son según Encalada et al. (1998), Francisco et al. (1999) y Francisco & Bowen (2001) respectivamente. dA/2, valor medio de aglomeración; T, tiempo de divergencia. Haplotipos: CC-A1 – 10, Encalada et al. (1998); CC-A11 – 14, Bolten et al. (1998); CC-A20, según Bowen (inscrito en el Archie Carr Center). RESULTADOS____________________________________________________________________________________________ 66 encontrada entre las poblaciones de anidación sureñas. A partir de estos valores de aglomeración son estimados tiempos de divergencia entre las poblaciones de anidación dentro de cada agrupación latitudinal en los órdenes 150 000 y 12 000 años respectivamente. Dentro del Mediterráneo americano, la población de anidación del suroeste cubano diverge primero que Quintana Roo y Dry Tortugas con dA=0.21 y 0.02% respectivamente, lo cual representa tiempos de divergencia de 126 000 y 12 000 años respectivamente. Las colonias de anidación del suroeste cubano aún no evidencian internamente señales de divergencia. En el NCPA, la anidación de los clados (0 – pasos) concluye en agrupaciones de haplotipos con orígenes diferentes (Fig. 17a). En el clado interno (1 – 1) se encuentra el haplotipo más distribuido y mejor representado del linaje mediterráneos americano – Viejo Mundo (CM-A2), agrupado con otros haplotipos entre los que se encuentran los endémicos CC-A5, 13 y 20 endémicos de Port Everglades y norte de Jetty pertenecientes a la población de anidación del sureste de la Florida, y del suroeste de Cuba, respectivamente. Este haplotipo es reconocido como ancestro del linaje. Otros clados integrados por haplotipos de este linaje son los consecutivos 2 y 3. El clado 1 – 2 está integrado sólo por el haplotipo CC-A9, representado en las poblaciones de anidación de Dry Tortugas y Quintana Roo. El clado 1 – 3 está integrado por los haplotipos CC-A10, ampliamente distribuido pero mal representado, y por el CC-A12, endémico del suroeste de Cuba. Los clados restantes (1 – 4 y 5) están integrados por haplotipos del linaje Atlántico – península de la Florida. El clado 1 – 4 está integrado por los haplotipos CCA11, pobremente distribuido y representado que resulta el ancestro del linaje; y CC-A1, mejor distribuido y representado del linaje. El otro clado (1 – 5) está integrado por dos haplotipos alopátridos disyuntos, i.e. CC-A4 endémico y único haplotipo de la población de anidación de Bahía (Brasil) y por el CC-A14 del Mediterráneo americano. La anidación de los clados (1 – pasos) concluye en dos agrupaciones de haplotipos correspondiente a cada linaje (ver Fig. 17a). El clado 2 – 1 integra a los clados de un paso pertenecientes al linaje mediterráneos americano – Viejo Mundo, mientras que el clado 2 – 2 integra a los clados pertenecientes al linaje Atlántico – península de la Florida. Entre éstos media un mínimo de 16 pasos. RESULTADOS____________________________________________________________________________________________ a) 9 1-1 3 5 6 x x 1-2 7 2-1 12 1-3 1-5 10 8 67 13 4 14 20 2-2 11 1-4 16 2 b) Clado 2I E 3 E 5 E 6 E 7 E 8 E 13 E 20 I–E Haplotipo Dc 5,811.10 L 7,453.00 0 L 8,807.20 1,018.00 397.00 0 0 -115.86 1 Dn 5,825.50 L 6,566.58 4,358.64 5,883.97 4,877.00 4,822.84 4,380.33 4,356.43 P -198.93 X2=259.32, p=0.03 Clado 1-1I Clados de 1 - paso Dc Dn 5,892.42 L Clados de 2 - pasos Clado Dc Dn 5,830.51 L 1 – 2 – 11(sí) E 9 10 I E 12 I–E E 506.40 E 2,560.18 L 3,742.99 1-2 2,764.42 0 2,764.42 4,375.78 2,640.45 1,524.73 1,115.73 X2=5.45, p=0.58 1-3 I–E Ho aceptada P P 4,735.53 L 1,166.92 X2=40.34, p=0.25 1 11I I 893.69 0 892.37 607.26 E 5,795.40 E 1,543.12 2-1 L 5,218.58 P 3,203.00 L 1 – 2 – 3 – 4 (No) X2=7.76, p=0.82 1-4I P 891.51 1,234.04 P Ho aceptada 4I E 14 I–E P 0 P 666.54 P -666.54 X2=19.00, p<0.001 1 – 19 (No) L 4,629.69 L 5,343.98 S -714.29 E L L 1-5 4,968.46 5,125.71 I – E -4,076.95P -3,891.67P X2=191.27, p<0.001 1 – 2 – 11 (Sí) – 12 (No) 2-2 P 2 X =304.40, p<0.001 1 – 2 – 11 (Sí) – 12 (Sí) Fig. 17. Análisis filogeográfico de clados anidados de los linajes de C. caretta representado en poblaciones de anidación de los mediterráneos americano y del Viejo Mundo, sobre la base de haplotipos de la RNC del mtDNA (ca. 380 pb). a) Árbol de haplotipos con diseño de clados anidados. b) Resultados del NCPA. Para interpretar la simbología y nomenclatura ver Figura 14. Haplotipos: ver Figura 15. RESULTADOS____________________________________________________________________________________________ 68 Entre la generalidad de los clados procesados estadísticamente se encuentra una asociación geográfica significativa (Fig. 17b), en correspondencia con una dependencia entre la representatividad y distribución de los haplotipos en las colonias de muestreo. En los clados de un paso 1 – 3 y 4, las hipótesis nulas de no asociación geográfica son aceptadas. En el clado 1 – 1 se infiere una expansión contigua del rango de distribución de los haplotipos, a partir de la significación de las distancias que resultaron: largas para el haplotipo CC-A3, larga dentro del clado para el haplotipo CC-A6, y pequeña en cuanto a la distancia media entre las posiciones internas menos las externas en el clado anidado. En el clado 1 – 5 también se infiere una fragmentación alopátrida, a partir de la significación de las distancias pequeña dentro del clado y grande en el clado de los haplotipos integrantes, así como distancias pequeñas entre las posiciones internas menos las externas dentro del clado anidado. En los clados de dos pasos, se infiere un flujo de genes restringido con aislamiento por distancia entre las colonias de anidación para el clado 2 – 1, a partir de la significación de las distancias largas para el clado 1 – 1 y entre las posiciones internas menos las externas en el clado anidado, y pequeñas para el clado 1 – 3. En el clado 2 – 2 también se infiere una expansión contigua del rango de distribución de C. caretta a partir de la significación de las distancias pequeñas para el clado 1 – 4 y entre las posiciones internas menos las externas en el clado anidado, y largas para el clado 1 – 5. Este resultado también se obtiene entre los clados anidados totalmente (clados de 2 – pasos), a partir de la significación de distancias largas para el clado 2 – 1 y pequeñas para el clado 2 - 2. DISCUSIÓN______________________________________________________________________________________________ 69 V. DISCUSIÓN. V.1 Análisis de secuencias. El alto porcentaje de transiciones que se encuentra en las secuencias de la primera mitad de la RNC del mtDNA, ha sido informado para C. mydas (Lahanas et al., 1994; Encalada et al., 1996), C. caretta (Encalada et al., 1998; Laurent et al., 1998), E. imbricata (Frías et al., 2006; Bowen et al., 2007) y otras especies marinas (Brown et al., 1993; Wenink et al., 1994) y particularmente, para la RNC del mtDNA en póngidos (Brown et al., 1982), el hombre (Tamura & Nei, 1993) y en roedores (Brown et al., 1986). Una proporción similar también ha sido informada para el DNA nuclear (Anagnostopoulos et al., 1999; Graur & Li, 2000; Nachman & Crowell, 2000) y para pseudogenes en varias especies (Graur & Li, op.cit.; Nachman & Crowell, op.cit.). Existen dos vías principales por las cuales ocurren las mutaciones puntuales (Topal & Fresco, 1976). En la primera, las transiciones devienen de asociaciones erróneas de purina – pirimidina, y pueden ocurrir en cualquier cadena. En la segunda, las transversiones devienen de asociaciones erróneas purina – purina, y sólo pueden ocurrir si la purina reside en la cadena molde. Por consiguiente, si la transversión tiene baja probabilidad de ocurrencia, entonces, cuando aparece, debe ser diagnóstica como ocurre en el haplotipo CM-A36 y entre linajes de C. mydas, en los haplotipos CCA18, 23, 40 y 47 de C. caretta, y EI-ABI1 y EI-EATL1 de E. imbricata (Bowen et al., 2007). Los porcentajes equitativos de transiciones entre purinas y pirimidinas que se encuentran en estas tortugas marinas concuerdan con la abundancia de A y T, y con la predisposición a la conversión de CpG. La desproporción entre las sumas de A y T respecto a G y C ha sido informada previamente para C. mydas por Lahanas et al. (1994) y para otras especies marinas (Saccone et al., 1987; Arnason et al., 1993; Brown et al., 1993; Sang et al., 1994). En pseudogenes, la riqueza de A y T ha concordado con la tendencia al cambio de C y G a T y A respectivamente. Este fenómeno también puede ocurrir en la RNC del mtDNA porque no está sujeta a restricción funcional (Graur & Li, 2000). La transición de C a T puede facilitarse a través de la conversión de residuos de C metilada a residuos de T por desaminación (Coulondre et al., 1978; Razin & Riggs, 1980), la cual ha sido más frecuente en CpG por el alto porcentaje de metilación de C (Razin & Riggs, op.cit.). Como consecuencia y considerando la selectividad neutral sobre esta región, aumenta la probabilidad de transiciones recientes en las bases más abundantes (A y T). Por otro lado, DISCUSIÓN______________________________________________________________________________________________ 70 la abundancia de A y T ha sido una consecuencia de la predisposición histórica a la conversión de CpG. Entonces se cumplen las dos igualdades fCT= fGA y fTC= fAG. Por esta razón, los cambios diagnósticos que se encuentran en los haplotipos de C. mydas y C. caretta son consecuencia de transiciones generalmente en los sentidos A→G y T→C. Estos cambios concomitan con la edad del haplotipo y su frecuencia se relaciona con la riqueza del contenido de A y T más que con la predisposición a la conversión de CpG. En C. mydas se infiere que el indel correspondiente al SP 470 es una inserción porque no se presenta en el haplotipo ancestral, y su frecuencia es exclusiva de un solo haplotipo derivado en el linaje mediterráneos americano – Viejo Mundo. Otras dos inserciones han sido informadas para el mismo SP en haplotipos del linaje Atlántico Sur de esta especie: CM-A7, como una repetición en tándem de diez pb [CCTTTGGTTG] (Lahanas et al., 1994); CM-A25 y CM-A42, como repeticiones de diez [CAATGGGTTG] y cuatro [CAGG] pb (Formia & Bruford, 2002). Estas inserciones sólo han sido encontradas en uno y dos individuos respectivamente. Consecuentemente, el haplotipo CM-A7 es considerado raro (Lahanas et al., 1994). Curiosamente, esta adición presente en el haplotipo CM-A7 también ha sido encontrada en los haplotipos CM-PE1 y 2, endémicos y bien representados en el linaje del Pacífico dentro del Atolón de la Micronesia (Moritz et al., 2002). En esta misma posición se ha informado otra repetición en tándem para el haplotipo EI-A25 de E. imbricata (Díaz-Fernández, 1999) del linaje “Caribe B” (Okayama et al., 1999). En C. caretta también se infiere que los indeles correspondientes a los SPs 431 y 473 del linaje mediterráneos americano – Viejo Mundo son inserciones porque tampoco se presentan en el haplotipo ancestral y ocurren en sólo tres haplotipos del linaje. Otro indel en la posición 163, no informado por ser diagnóstico entre los linajes de la cuenca atlántica, también constituye una adición para el linaje Atlántico – península de la Florida porque no se comparte con el linaje naturalmente emparentado del Pacífico. Sin embargo, los indeles de uno y seis pares de bases en los SPs 404 y 465, tampoco informados por la misma razón antes esgrimida, no pueden clasificarse porque sólo se presentan en el linaje mediterráneos americano – Viejo Mundo. Las duplicaciones en el mtDNA han tenido una distribución geográfica y filogenética restringida también en otras especies (Moritz & Brown, 1987; Wallis, 1987). Moritz & Brown (1987) proponen que estas duplicaciones tienen una corta vida en relación a la escala de tiempo evolutivo. Otra inserción de diez pb DISCUSIÓN______________________________________________________________________________________________ 71 se encuentra en la secuencia del haplotipo CM-A25 de C. mydas (Bjorndal & Bolten, inscrito en el Archie Carr Center for Sea Turtle Research). En C. mydas, al igual que en E. imbricata, son frecuentes las repeticiones nucleotídicas continuas en los treinta pb antecesores a la inserción. También ocurre en las antecesoras a las adiciones y al indel de seis pares de bases del linaje mediterráneos americano – Viejo Mundo de C. caretta. Estas áreas son ricas en GT. En microsatélites de otras especies, la aparición de nuevas variantes genéticas ha sido explicada por el mal apareamiento de las bases, provocado por el deslizamiento de la cadena durante la replicación en áreas ricas en GT (Goldstein & Schlotterer, 1999). En levaduras se ha observado, que la enzima incrementa la frecuencia de mutaciones en tramos con abundancia de GT (Strand et al., 1993). Adicionalmente, en fragmentos de secuencias con repeticiones se han incrementado las mutaciones puntuales de 130 – 240 veces, porque la DNA polimerasa delta pierde la capacidad de corregir errores (Morrison et al., 1993). Por consiguiente, la presencia de indeles en C. mydas, C. caretta y otras especies de tortugas marinas como E. imbricata, podría ser más probable en un área específica de la RNC con repeticiones, y su composición debería también ser similar. De hecho una de estas inserciones es común en linajes alopátridos de C. mydas de las cuencas atlántica y pacífica. La frecuencia significativa de los haplotipos endémicos que portan las inserciones en C. mydas, tanto en las colonias del suroeste cubano como en la Micronesia, es otra evidencia de que en las islas pueden fijarse variantes raras durante las fluctuaciones demográficas, y cambiar sus frecuencias por acción de la deriva genética en ausencia de migraciones significativas. C. mydas presenta una distribución similar de mutaciones en los dos linajes de la cuenca atlántica a diferencia de C. caretta, en la que predominan los cambios diagnósticos de forma general (de haplotipos, haplotipos dentro de linajes y entre linajes (ver Figs. 6 y 8). Los linajes de la cuenca atlántica de C. caretta no están relacionados naturalmente, es decir, no comparten un ancestro común como sucede en C. mydas. Por consiguiente, entre estos linajes pueden ocurrir homoplasias y fijarse un mayor número de mutaciones, apareciendo transversiones e indels diagnósticos de linajes en la medida en que aumenten las distancias evolutivas entre éstos como sucede en roedores (Brown et al., 1986). Este argumento ha sido la única razón sólida para explicar el por qué hay mayores cambios, incluso transversiones etc. en C. caretta respecto a C. mydas. Realmente estos DISCUSIÓN______________________________________________________________________________________________ 72 mecanismos de distribución geográfica de la variación genética (vicarianza en C. mydas y vicarianza y dispersión en C. caretta) son la causa del curso y tipo de mutaciones respecto al ancestro más cercano. Adicionalmente, cualquier fuerza evolutiva que actúe sobre cualquier locus afectará la historia de la molécula completa, por lo que la fijación de una mutación ventajosa por selección, puede causar la fijación de otros polimorfismos a través del proceso de autostop (hitchhiking) genético (Maynard & Haigh, 1974) o coadaptación (Wallace 1991). El hecho de que exista una variación clinal de los haplotipos más distribuidos y representados en los linajes de C. caretta podría estar asociada a una influencia climática de la latitud, a pesar de que se supone neutralidad para la RNC, como ha ocurrido en Crassostrea virginica (Reeb & Avise, 1990) y en Fundulus heteroclitus (Gónzalez-Villaseñor & Powers, 1990). En vertebrados exotérmicos la tasa de sustitución promedio es más baja que en endotermos de semejante tamaño (Martin & Palumbi,1993), y en invertebrados haplogrupos de Drosophila son dependientes de la temperatura (Doi et al., 1999). Razones por la que no debe asumirse una misma tasa de mutación para calibrar el reloj molecular de diferentes especies (Ballar & Rand, 2005). V.2 Estructura poblacional. A partir del análisis de los resultados de estructuración genética de las colonias de anidación de C. mydas y C. caretta del suroeste cubano se concluye que éstas constituyen poblaciones de anidación panmictas. Las colonias de anidación del sureste de Cuba de E. imbricata también constituyen una población panmicta (Espinosa et al., 2004; Bowen et al., 2007a). Cuando se analiza la estructura entre las colonias de anidación cubanas y las del Mediterráneo americano y costa atlántica de la península de la Florida para cada especie, se encuentran estructuras genéticas significativas entre éstas. [Estas colonias de anidación han sido consideradas poblaciones para C. mydas por Allard et al. (1994), Lahanas et al. (1994), Encalada et al. (1996) y Bjorndal et al. (2005); para C. caretta se ha mantenido el estatus de población para la colonias de anidación de Quintana Roo (Encalada et al., 1998), mientras que las colonias de anidación de USA se han integrado en poblaciones (Francisco et al., 2001); y para la otra especie de tortuga marina que anida de forma apreciable en el archipiélago cubano E. imbricata por Bass et al. (1996), DíazFernández et al. (1999), Espinosa et al. (2004) y Bowen et al. (2007a)]. DISCUSIÓN______________________________________________________________________________________________ 73 Usando dos pruebas estadísticas y considerando la presencia o ausencia del haplotipo CM-A5 (perteneciente al otro linaje) en la población mexicana de C. mydas, se encuentra estructura genética significativa entre la colonia de anidación de la Florida y Quintana Roo. En contraste con este resultado, Encalada et al. (1996) no encuentra estructuración genética significativa entre estas colonias de anidación. Los resultados de las dos pruebas de estructuración genética difieren cuando se comparan las interacciones por pares de colonias o poblaciones de anidación de C. mydas del suroeste de Cuba con las de la Florida y Quintana Roo. En C. caretta también hubo incongruencias entre la significación de la prueba X2 con respecto a la prueba Φ ST por pares de colonias de anidación. Estas incongruencias dependieron de la resolución de cada estadístico en relación con el tamaño de muestra y la diversidad genética, principalmente con la haplotípica en los casos en los que el X2 fue significativo. Para tamaños de muestras dispares con haplotipos pertenecientes a linajes diferentes y una desproporción de las frecuencias haplotípicas, el X2 se vuelve más sensible respecto al F- Φ ST para detectar diferencias. Como era de esperar la prueba de F- Φ ST fue menos precisa que la de X2, a pesar de que esta última solamente considera el número y la frecuencia de los haplotipos. El F- Φ ST se expresa en términos de tiempos de coalescencia probando la hipótesis nula por permutaciones de haplotipos entre poblaciones (Slatkin, 1991). Por consiguiente, para la colonia de anidación de C. mydas de la península de Guanahacabibes, respecto a las poblaciones de anidación de la Florida y Quintana Roo, y para las interacciones entre las colonias de anidación de C. caretta del suroeste de Cuba versus Tyndall Beach, Quintana Roo versus Dry Tortugas, y Cape San Blas versus Eglin Air Force Base, la media del tiempo de coalescencia de dos genes delineados en la misma población será cercana a la media del tiempo de coalescencia de dos genes delineados en dos poblaciones a comparar, resultando en la no significación del F- Φ ST. Estos resultados han sido consecuencia de que los haplotipos más representados en C. mydas y C. caretta se compartan entre las colonias de anidación y de que los restante haplotipos endémicos aparecen en baja frecuencia. Justificación similar se puede asociar al comportamiento de la colonia anidación de C. mydas de San Felipe respecto a la población de anidación de la Florida. No obstante, la colonia de anidación de San Felipe está estructurada significativamente con la población de anidación de Quintana Roo, debido a que solamente comparten uno de los dos haplotipos mejor representados. Contrariamente, a pesar de que en la DISCUSIÓN______________________________________________________________________________________________ 74 interacción de las colonias de anidación de C. caretta de Dry Tortugas versus Port Everglades están representados haplotipos de ambos linajes, la media del tiempo de coalescencia de dos genes delineados en una población distará de la media del tiempo de coalescencia de dos genes delineados en ambas poblaciones, resultando en la significación del F- Φ ST. Estas incongruencias quedan resueltas con la aplicación de un AMOVA, que además de tener en cuenta el número de mutaciones entre los haplotipos, considera la varianza de las frecuencias haplotípicas generadas en las estructuras jerárquicas (Excoffier et al., 1992). Los resultados de esta prueba respaldan en C. mydas la estructura hipotética II, aunque el FCT no haya sido significativo. La diferenciación regional en esta especie ha sido más evidente cuando el grado de diferenciación entre los haplotipos se tiene en cuenta, considerando que la distancia molecular ha sido mayor por pares de haplotipos pertenecientes a regiones diferentes que para aquellos dentro de la misma región (Excoffier et al., op.cit.). Como los haplotipos de C. mydas en las colonias de anidación del Mediterráneo americano pertenecen al mismo linaje y entre éstos existen pocos cambios, la precisión del FCT disminuye. Adicionalmente, estas colonias de anidación comparten el haplotipo más ampliamente distribuido y mejor representado, por lo que no se ha considerado el comportamiento estadístico del índice FCT en las estructuras hipotéticas II y III. En C. caretta se aprueba la estructura hipotética II, con una combinación perfecta entre el comportamiento estadístico de Φ SC y Φ CT, porque a pesar de que la mayor variación también se produce dentro de las poblaciones, esta es dos veces menor que en C. mydas como consecuencia de la presencia de dos linajes no emparentados naturalmente y con distribuciones distintas, que en la península de la Florida llegan a constituir clinas. Por otra parte, en E. imbricata, la población de anidación cubana junto con las de Puerto Rico, Islas Vírgenes de USA, Antigua, Barbados y Venezuela, quedan dentro del grupo Caribe oriental, sin panmixia pero distinguible significativamente del otro (Caribe occidental, que junta las poblaciones de anidación de México, Belice y Costa Rica) (Bowen et al., 2007). Estas agrupaciones tienen su causa en un porcentaje mayor entre que dentro de las poblaciones de anidación (Bass et al., 1996), lo cual contrasta con la proporción de los componentes de varianza informada para C. mydas y C. caretta. DISCUSIÓN______________________________________________________________________________________________ 75 La existencia de estructura genética significativa entre las poblaciones de anidación de Cuba y Quintana Roo (México) para C. mydas y C. caretta, la presencia de dos linajes en la población de anidación cubana para C. caretta, y la no existencia de estructura genética significativa con algunas de las colonias del sur de la Florida en C. caretta, acentúan patrones de colonización relacionados más con los que ocurrieron en la península de la Florida que con los que ocurrieron en México, a pesar de que las poblaciones de anidación de Cuba y México están en la misma cuenca del Mar Caribe y son las más cercanas geográficamente. La corriente de Yucatán provee un flujo mayoritario de agua hacia el Golfo de México que hacia el estrecho de la Florida, donde el flujo es pequeño y episódico a través de la contracorriente cubana (Ochoa et al., 2001). A su vez, la existencia de la corriente de lazo, que en un extremo da paso directo a la corriente de la Florida, genera un flujo cuasi permanente de recirculación alrededor de Cuba (Gyory et al., 2005). Por consiguiente, ha existido un flujo directo entre las aguas cercanas a Cuba con las del sur de la Florida, principalmente con el sureste, y limitado con las de México. Todo lo cual ha influido en la conformación de poblaciones de anidación perfectamente distinguibles en las áreas de anidación de Cuba y México, y parecidas entre Cuba y el sureste de la Florida para ambas especies. Este sistema de corrientes ha sido una barrera efectiva para limitar el flujo de migrantes no sólo entre las poblaciones de anidación de Cuba y México, sino también entre las poblaciones de México y el noroeste de la Florida de C. caretta. La variación clinal de los haplotipos más distribuidos y representados de C. caretta en ambas costas de la península de la Florida ha ofrecido dificultades para el establecimiento de los límites de las poblaciones de anidación en esta región. La corriente de la Florida se desvía de la costa cerca de los 32oN generando remolinos, que decrecen cuesta abajo (Olson et al., 1983). Francisco & Bowen (2001) consideran que las colonias de anidación de Volusia County debieron formar parte de la población de anidación del noreste de la Florida hasta Carolina del Norte por estar situada al norte de Cabo Cañaveral, límite biogeográfico reconocido según Briggs (1974) y Avise (1992). Sin embargo, Bowen (2004) describe una estructura genética significativa entre esta área de anidación y la población de anidación del sur de la Florida. En el presente estudio se restringe aún más la pertenencia a la nueva población de anidación del sureste de la Florida. Al parecer, la efectividad de la barrera marina para C. caretta está un poco más al norte (ca. 31oN), en la DISCUSIÓN______________________________________________________________________________________________ 76 irregularidad topográfica conocida como “Charleston Bump”. En esta zona, se ha descrito un carácter bimodal de la corriente de la Florida manifiesto en estados débiles y fuertes de desviación, pero con predominio de varios meses de los fuertes (Bane & Dewar, 1988). Quizás, como la colonia de anidación de Cumberland Island se localiza cerca de la zona, pueda recibir migrantes de las colonias de latitudes más bajas, aunque de forma limitada. Por consiguiente, esta colonia de anidación y la vecina de Amelia Island constituyen el límite inferior de la población de anidación del noreste de la Florida hasta Carolina del Norte. Para el resto de las colonias de anidación de esa población, la barrera marina “Charleston Bump” es muy efectiva, ya que solamente están integradas por el haplotipo CC-A1 a pesar de tener anidaciones apreciables (TEWG, 2000) en comparación con otras colonias del Gran Caribe. Sin embargo, a pesar de que en la costa noroeste de USA el haplotipo CC-A2 no se anula, la indisponibilidad de sitios para la anidación (barrera biogeográfica) en la zona central de la costa del oeste de la península de la Florida, delimita las poblaciones de anidación del norte y del sur. En el caso del sur de la Florida, a pesar de que no existe estructura genética por pares de colonias de anidación, exceptuando las interacciones con Dry Tortugas, se definen con el análisis de la varianza molecular jerarquizado tres poblaciones de anidación en lugar de las dos clásicas (Encalada et al., 1998; Francisco & Bowen, 2001). Todo lo cual reconoce la efectividad del desagüe de los Everglades (barrera biogeográfica) para limitar el flujo amplio de migrantes entre las colonias de anidación del suroeste, Dry Tortugas y sureste de la Florida. Entre la generalidad de las colonias de anidación analizadas del Atlántico Sur también se ha encontrado estructura genética significativa para ambas especies, con la excepción en C. mydas de los pares de colonias de anidación de las islas Guinea Bissau – Ascensión, Ascensión – Bioko y Príncipe – Sao Tomé. En el primer caso, la posible causa se cree que fue por un aislamiento reciente (Encalada et al., 1996); en el segundo, la colonización reciente de la Isla de Bioko (Formia et al., 2002a); y en el tercero, el resultado de los fallos a la conducta de reproducción en el lugar de origen (Formia et al., op.cit.). Similar estructuración genética poblacional ha tenido lugar dentro de la cuenca indopacífica (Norman et al., 1994; Moritz et al., 2002), donde sólo las colonias de anidación de C. mydas del Cabo Noroeste del continente australiano y las de las islas Lacepede, cercanas a éste, no están estructuradas significativamente, probablemente como consecuencia de DISCUSIÓN______________________________________________________________________________________________ 77 la existencia de colonias de anidación intermedias entre éstas o porque fueron colonizadas por la misma población originaria, seguido de una segregación reciente. En el Pacífico Norte, también se informa estructura genética significativa entre las colonias de anidación de C. caretta del Archipiélago de Japón (Hatase et al., 2002) y entre éstas y las de Australia (Bowen et al., 1995). Esta estructuración se corresponde con variaciones morfométricas referidas para las colonias de anidación en cuestión (Hatase et al., 2002). Considerando la correlación obtenida entre el grado de diferenciación genética y la distancia geográfica por pares de colonias de anidación de C. mydas y C. caretta, y teniendo en cuenta el patrón de distribución ecológica (discreta) en las áreas de anidación, el modelo de flujo genético que mejor se ajusta es el de paserala. Este modelo explica la aparición de altos niveles de significación de estructura entre las colonias de anidación más distantes. Estos niveles son la causa del comportamiento estadístico de la correlación cuando se involucran todas las colonias de anidación de la región del Mediterráneo americano. Es por esto que a pesar de que el coeficiente de correlación no es alto cuando se analizan las interacciones con la distancia geográfica, como consecuencia de la dispersión de las interacciones, se obtuvieron diferencias significativas. Evidentemente, esta dispersión debe incrementarse cuando se consideren sólo las poblaciones validadas en el presente trabajo porque está explícita la similitud genética entre colonias de anidación contiguas de USA lo cual se hace extensivo al nivel poblacional. Todo lo cual contribuye a esclarecer los conceptos de colonia y población de anidación en términos de la diferenciación genética. Las colonias se corresponden principalmente con la agregación espacial de las tortugas marinas en áreas de anidación específicas, a menudo identificadas con el nombre vernáculo de un accidente geográfico, e.g., el nombre de la playa. La existencia de poblaciones discretas (demos) con diversos grados de estructuración ha sido consecuencia de un aislamiento reproductivo dependiente de barreras para la dispersión, como lo son la distancia en asociación con la conducta de reproducción en el lugar de origen. Todo lo cual justifica la clasificación de metapoblación (Harrison, 1991) para cada especie. Esta metapoblación es un sistema de demos en parche con demos no equilibrados. Entre los demos en parche el flujo genético ha estado en función de la distancia geográfica y ha sido afectado por eventos de extirpación y recolonización en tiempos histórico y ecológico. Entre los demos no equilibrados el flujo DISCUSIÓN______________________________________________________________________________________________ 78 genético debió ocurrir en tiempo histórico como resultado de eventos de colonización. Contrariamente, en E. imbricata la tasa de migración no es dependiente de la distancia geográfica (Bass et al., 1996; Reece et al., 2005). Como la distribución de haplotipos y linajes permite reconocer dos agrupaciones en el Mediterráneo americano (Bowen et al., 2007), una que incluye las poblaciones de anidación continentales del sur y otra a las isleñas del norte, poblaciones muy distantes geográficamente dentro de un grupo, como las de México y Costa Rica por una parte y Cuba y Antigua por otra, van a diferir menos genéticamente que las más cercanas geográficamente pero pertenecientes a agrupaciones diferentes e.g. México y Cuba, y Costa Rica y Puerto Rico. No obstante, Reece et al. (2005), asumen dentro de cada agrupación un modelo de aislamiento por distancia, cuyas huellas pudieron alterarse durante los cambios drásticos del Pleistoceno. Los eventos de recolonización en C. mydas, que afectaron la estructuración genética de las colonias de anidación del Mediterráneo americano y la costa atlántica de USA, debieron haber ocurrido antes de la diversificación de(l) (los) linaje(s). Esta interpretación está en correspondencia con los resultados de la diversidad genética (alto endemismo y baja a moderada representatividad de los haplotipos derivados) y filogenéticos de clados anidados (evidencias de colonizaciones, extirpaciones y recolonizaciones en un escenario con fragmentación histórica del hábitat). Estos resultados también son congruentes con el bajo flujo genético entre la población de anidación de Tortuguero y las restantes del Mediterráneo americano con las que al menos se comparte un haplotipo según Allard et al. (1994), Lahanas et al. (1994) y Bjorndal et al. (2005). Allard et al. (1994) proveen una alternativa apropiada para explicar los bajos valores de migrantes basada en la retención de los haplotipos compartidos a partir de un ancestro común, más que por un limitado flujo de genes. En C. caretta, la dispersión de un linaje lejanamente emparentado con el linaje vicariante del Atlántico (ancestro CC-A2), la existencia de un porcentaje de variación mayor dentro que entre las colonias de anidación, la presencia de una clina de haplotipos en la península de la Florida, y el bajo porcentaje de endemismo en las poblaciones de anidación, evidencian que esta especie ha fallado históricamente más a la conducta de reproducción en el lugar de origen. Probablemente, los fundadores del linaje dispersado DISCUSIÓN______________________________________________________________________________________________ 79 hayan tenido cierto éxito en la colonización de tierras templadas de las altas latitudes americanas durante los períodos interglaciales, debido al desafío además de largas distancias, de temperaturas menos cálidas al sur de África. El efecto fundador de este nuevo linaje sería más apreciable en las áreas no ocupadas por C. caretta (altas latitudes), que en donde hubieran existido colonias de anidación previamente establecidas correspondiente al linaje vicariante. Una vez asentado el nuevo linaje tanto al oeste como al este de la península de la Florida, comenzó una introgresión entre las colonias de anidación donde ambos linajes se encuentran en frecuencia mayoritaria, dando lugar al surgimiento de una clina geográfica latitudinal. Estos eventos han ocurrido paralelamente en ambas costas, lo cual ha provocado falta de estructura genética significativa entre algunas de las colonias de anidación del noroeste y noreste de la Florida, y entre todas las colonias del suroeste y sureste. Razón por la cual, la prueba de Mantel no da significativa cuando se aplica a todas las colonias de USA sin distinción de costa. No obstante, Encalada et al. (1998) plantean que la relocalización de ovíferas a grandes distancias puede explicar el alto estimado de migración entre las poblaciones de sitios opuestos de la península. Sin embargo, los eventos de relocalización mejor documentados han ocurrido entre playas de anidación adyacentes involucrando unas pocas decenas de kilómetros, mientras que las que ocurren a largas distancias son menos frecuentes (Richardson, 1982). En estudios de marcación se evidencian movimientos ocasionales de ovíferas entre playas de anidación (Bjorndal et al., 1983; Dodd, 1988), acentuándose las conductas filopatría y fidelidad por el sitio de anidación de la especie en tiempo ecológico. La similitud de la distribución de haplotipos entre playas de anidación ampliamente separadas, quizás sea un remanente de un proceso histórico de colonización que un indicio de un una migración paulatina. V.3 Diversidad genética. El alto endemismo haplotípico en la población de anidación de C. mydas del suroeste cubano, algo mayor que el informado para E. imbricata (Bowen et al., 2007), es consecuencia de la conducta de reproducción en el lugar de origen, reforzada por la condición de insularidad y la emergencia de la isla durante los períodos interglaciales antes de que divergieran los linajes de C. mydas (último tercio del Pleistoceno, previo la última glaciación), al igual que por el mantenimiento de las condiciones para el éxito de la DISCUSIÓN______________________________________________________________________________________________ 80 anidación durante las glaciaciones. Incluso en las colonias de anidación del suroeste cubano de C. caretta, especie con bajo endemismo en las colonias de anidación y menor fidelidad por el sitio de anidación, se presenta un haplotipo endémico (CC-A12). El hecho de que en C. caretta se haya probado una conducta migratoria transoceánica (Bowen et al., 1995; Bolten et al., 1998) y exista una menor distribución de colonias de anidación en la cuenca atlántica respecto a C. mydas y E. imbricata, y que estas colonias están próximas a los circuitos principales de corrientes marinas, podría ser una consecuencia de que esta especie hace su ciclo de vida a merced de las corrientes marinas. Quizás sea esta conducta errante la razón por la que falle más a la conducta de reproducción en el lugar de origen y exista una amplia distribución de haplotipos en contraposición con el endemismo. La diversidad haplotípica moderadamente alta de la colonia de anidación de C. mydas en la cayería San Felipe, a pesar de que sólo comparte dos haplotipos con la colonia de anidación de Guanahacabibes, proporciona evidencias para considerar a esta colonia como un sumidero. La población de anidación de la Florida de C. mydas también ha sido considerada un sumidero de acuerdo con Allard et al. (1994), quienes consideran que ésta se formó a través de la contribución de otras poblaciones de la región. Esta contribución debió estar influenciada por factores geomorfológicos y oceanográficos, así como por la proximidad geográfica a las áreas de anidación fuentes, existentes en tiempo ecológico o histórico. Es posible también que la población actual sea parte de una gran población que haya existido en el pasado (Avise et al., 1988; Allard et al., 1994). Sin embargo, para C. caretta la Cayería de San Felipe se comporta como fuente. Otras poblaciones de anidación insulares de C. mydas y C. caretta [también en E. imbricata], que no constituyeron parte de las poblaciones continentales durante las glaciaciones ni fueron completamente extirpadas durante los períodos interglaciales, muestran un alto endemismo haplotípico. Las islas volcánicas de Sao Tomé y Príncipe, cuyo origen está datado entre 13 y 31 MA respectivamente (Lee et al., 1994), no quedaron sumergidas durante los períodos interglaciales ni sus hábitats fueron afectados drásticamente por la última glaciación. Consecuentemente, en estas islas sobrevivieron colonias de anidación de C. mydas y se fijaron mutaciones y haplotipos. La isla de Sao DISCUSIÓN______________________________________________________________________________________________ 81 Tomé constituye probablemente una población fuente (Formia et al., 2002a), no sólo porque presenta una alta diversidad haplotípica sino porque también es la más alejada de las poblaciones del continente e islas adyacentes. Otras islas oceánicas, pero de la cuenca pacífica como los atolones de la Micronesia (Norman et al., 1994; Moritz et al., 2002) también exhiben un alto endemismo haplotípico y moderada representatividad de individuos endémicos en C. mydas. En las islas Ogasawara (Japón) y Fragata Francesa (Hawai), y en el Atolón de Mopelia (Polinesia Francesa), todos los haplotipos de C. mydas son endémicos (Norman et al., 1994). El endemismo de estas islas está probablemente relacionado además de con la condición de emergencia durante períodos interglaciales, con un aislamiento geográfico respecto a las masas terrestres continentales, y a otras islas con colonias de anidación establecidas. Otro argumento que ratifica esta explicación es que los haplotipos identificados en las poblaciones de anidación de estas áreas pertenecen a linajes raros poco diversificados y distribuidos (linajes remanentes). Adicionalmente, estos haplotipos se diferencian por tres cambios como mínimo respecto al posible ancestro del linaje más diversificado y distribuido de la cuenca indopacífica (datos provenientes del análisis de los resultados de Norman et al. (op.cit.) y Moritz et al. (2002)). En las colonia de anidación de C. caretta del archipiélago de Japón también existe un alto endemismo haplotípico (Hatase et al., 2002), pero en este caso el endemismo se corresponde con que estas áreas de anidación son las únicas dentro del circuito migratorio de la especie. En E. imbricata, la generalidad de las poblaciones de anidación localizadas en la islas del Mediterráneo americano también tienen alto endemismo haplotípico (Reece et al., 2005). Podría resultar contradictorio que la población de anidación de C. mydas de las playas continentales de Michoacán (México) esté integrada solamente por haplotipos endémicos (Chassin-Noria et al., 2004). Probablemente, si se pudieran obtener datos moleculares de otras colonias de anidación distantes de éstas en la costa pacífica de América Central (e.g. Costa Rica), disminuiría el por ciento de endemismo como consecuencia de compartirse los haplotipos más distribuidos y mejor representados entre la mayoría de las colonias de anidación. La ausencia de estructura genética significativa en las colonias de anidación de Michoacán también podría ser una evidencia de la pérdida de variabilidad genética y alteración de las frecuencias haplotípicas como consecuencia de la DISCUSIÓN______________________________________________________________________________________________ 82 sobreexplotación de C. mydas en la región. La sobreexplotación ha conllevado a la extirpación de numerosas colonias y al predominio probabilístico en tiempo ecológico, de los haplotipos más distribuidos y mejores representados en tiempo histórico. Otro caso interesante de endemismo continental también se da en la población de anidación de E. imbricata de Yucatán (Bass et al., 1996; Díaz-Fernández et al., 1999), pero las causas están propiamente en el circuito particular de corrientes marinas del sur del Golfo de México (Reece et al., 2005), que entre otras causas, ha posibilitado también la especiación de Lepidochelys kempii. Paradójicamente, la Isla Ascensión tiene un alto porcentaje de endemismo haplotípico en C. mydas a pesar de presentar condiciones aptas para la anidación después de la última glaciación (Bowen et al., 1992). Este porcentaje probablemente ha sido una consecuencia de una rápida y reciente colonización según Bowen et al. (op.cit.). La posición geográfica de la Isla de Ascensión en el medio de la cuenca del Atlántico Sur, con una fuerte influencia de la corriente Ecuatorial del Sur y la contracorriente, facilitó la colonización. La baja presión por depredadores en esta isla ha permitido una rápida tasa de crecimiento en la población (Formia et al., 2002 b). Entonces, ¿es real este porcentaje de endemismo? El tamaño de la muestra de esta población es aproximadamente el triple de las restantes poblaciones de anidación, con una alta representatividad de individuos endémicos (>30%), lo cual aumenta la probabilidad de colectar haplotipos raros dentro de la población. Por consiguiente, aumenta la diversidad nucleotídica pero no la haplotípica. Esta ha sido la razón por la cual Encalada et al. (1996) obtienen una diversidad haplotípica similar a la informada por (Formia et al., 2002 b), con un tamaño de muestra menor, mientras que la diversidad nucleotídica fue menor. Probablemente estos haplotipos raros, que aparentemente son endémicos, pudieran estar fijados en poblaciones fuentes del Atlántico Sur. Dado que éstos se presentan en baja frecuencia, la probabilidad de detectarlos es mínima en muestras pequeñas. También es posible que hayan desaparecido dentro de las poblaciones originales. En este caso se perdería la condición de fuente de la población de anidación de Isla Ascensión. Adicionalmente, C. mydas experimenta fallos filopátridos condicionados por factores oceanográficos y climáticos como el curso de los vientos (Hays et al., 2003), por lo que esta isla podría comportarse como una población sumidero de las restantes poblaciones de anidación (fuentes) de la región. DISCUSIÓN______________________________________________________________________________________________ 83 Un efecto similar puede ocurrir con la población de anidación continental de C. mydas en Tortuguero, cuyo endemismo haplotípico es aparentemente alto (Bjorndal et al., 2005). En esta población el tamaño de la muestra analizada fue de varios cientos de individuos, lo cual provoca que aumente la probabilidad de colectar variantes raras o detectar fallos a la conducta de reproducción en el lugar de origen. La mezcla de linajes obtenida por Bjorndal et al. (op.cit.) es evidencia de estos fallos, los cuales provocan un incremento de la diversidad nucleotídica respecto a los valores de diversidad nucleotídica calculados por Encalada et al. (1996). Este argumento concuerda con la baja representatividad de los haplotipos endémicos en la población de anidación de Tortuguero. En tal sentido podría considerarse que la población original pudo experimentar drásticas variaciones demográficas, sobreviviendo el haplotipo más representado (ancestro del linaje) y otros raros que pudieron fijarse. Adicionalmente, la población de anidación de Tortuguero puede tener un comportamiento dual de fuente y sumidero debido a su posición geográfica intermedia entre las poblaciones de anidación y linajes de la especie en el norte y sur de América. Sin embargo, el hecho de que la población de anidación de Tortuguero actúe como sumidero no está respaldado por los resultados estadísticos de expansiones demográficas históricas obtenidos en el presente análisis. Contrariamente, la población de anidación de C. mydas en la Isla de Aves no presenta endemismo haplotípico debido a su reciente colonización (Lahanas et al., 1994) por los haplotipos más distribuidos y representados en las poblaciones vecinas, que se comportan como fuentes. La inestabilidad geológica de la isla pudo afectar la supervivencia de las poblaciones de anidación establecidas (Lahanas et al., op.cit.). La existencia de una moderada diversidad haplotípica y alta diversidad nucleotídica con tan sólo dos haplotipos, reafirma una reciente colonización. Esta colonización fue llevada a cabo por los haplotipos más distribuidos y mejor representados de los linajes presentes en las poblaciones cercanas geográficamente a la isla. Otras islas cuyas poblaciones de anidación de C. mydas no tienen haplotipos endémicos como Bioko y Poilao, pudieron haber sido colonizadas recientemente a partir de una población de anidación más diversa (Formia et al. 2002a). La ausencia de haplotipos endémicos podría estar relacionada con una alta dinámica costera en función de un relieve moderadamente bajo en la Isla de Bioko y bajo DISCUSIÓN______________________________________________________________________________________________ 84 en la isla de Poilao, más que con sus recientes orígenes; i.e. volcánico para Bioko (Aka et al., 2001). Otros factores relacionados con la ausencia de haplotipos endémicos son la proximidad de estas islas al continente y la vulnerabilidad de las poblaciones de C. mydas presentes en ellas al impacto de la depredación humana (Formia et al., 2002a). De hecho, los haplotipos representados en estas islas son de los más distribuidos en el linaje del Atlántico Sur. El bajo porcentaje de endemismo haplotípico en islas alcanza su mayor expresión en las islas continentales de la cuenca indopacífica ecuatorial. Éstas estuvieron conectadas recientemente a tierras continentales de Asia o de Australia, durante la glaciación del Pleistoceno (Brown & Lomolino, 1998). La población de anidación de C. mydas de Chipre no se ha considerado para reforzar la relación entre la condición de insularidad y el endemismo, a pesar de estar integrada por haplotipos endémicos (Encalada et al., 1996; Kaska, 2000), ya que probablemente se pierda la condición de endemismo haplotípico cuando sean analizadas muestras de las colonias de anidación cercanas de Turquía. La colonización de la cuenca del mar Mediterráneo ha sido reciente (Bowen et al., 1992; Encalada et al., 1996), como consecuencia del restablecimiento de las condiciones favorables para la anidación de C. mydas después de la glaciación de Würm. Por esta razón, la antigüedad geomorfológica de sus áreas de anidación no ha sido evaluada en este análisis. Como los haplotipos de la población de anidación de Chipre tienen una representatividad similar al resto de las del Atlántico (una variante altamente representada y otra escasa), y considerando las relaciones filogenéticas dentro del linaje, al menos el haplotipo “ancestral” de esta población debió surgir en el Mediterráneo americano y posteriormente haberse fijado en la cuenca del mar Mediterráneo. La existencia en la actualidad de dos linajes de C. mydas con distribución parapátrida, uno en el Atlántico Norte y otro en el Sur (Encalada et al., 1996), a diferencia de C. caretta y E. imbricata donde son simpátridos, y la baja diversidad nucleotídica evidenciada en las poblaciones de anidación donde no existe mezcla de linajes, son indicios suficientes que apoyan la conducta de reproducción en el lugar de origen determinada para C. mydas (Carr, 1967; Carr et al., 1978; Bowen et al., 1992; Limpus et al., 1992). No obstante, existen fallos históricos en C. mydas que se evidencian no sólo por la mezcla de linajes en DISCUSIÓN______________________________________________________________________________________________ 85 algunas poblaciones de anidación, sino también por la amplia distribución y alta representatividad de unos pocos haplotipos dentro de cada linaje en las poblaciones de anidación donde están representados. En este estudio se trata de los haplotipos CM-A1 y 3. Estos haplotipos separados sólo por un cambio, son respectivamente el ancestro y un centro de diversificación dentro del linaje mediterráneos americano – Viejo Mundo. En adición, existen indicios fehacientes de colonización de las poblaciones del linaje mediterráneos americano – Viejo Mundo por el haplotipo más representado en las poblaciones norteñas del Atlántico Sur (CM-A5), cuya frecuencia va disminuyendo con la distancia geográfica en las poblaciones de anidación más distantes con respecto a la población fuente (Matapica, Surinam) (ver Lahanas et al., 1994; Encalada et al., 1996; Bjorndal et al., 2005). Este haplotipo junto con el más distribuido y representado en el linaje mediterráneos americano – Viejo Mundo (CM-A3) recolonizó Isla de Aves (Lahanas et al., 1994). Estos fallos históricos son los responsables del incremento notable de la diversidad nucleotídica en las poblaciones donde convergen ambos linajes (Lahanas et al., op.cit.). No obstante, la generalidad de los haplotipos tiene distribución restringida a una o unas pocas poblaciones de anidación (alto endemismo), lo cual acentúa el carácter limitado e histórico de los fallos a la conducta de reproducción en el lugar de origen en correspondencia con el modelo de metapoblación probado en el presente estudio para el Mediterráneo americano. Chelonia mydas muestra alta fidelidad por el sitio de anidación (Carr, 1967 & 1975; Pritchard, 1976; Meylan et al., 1990; Limpus et al., 1992) al igual que C. caretta, aunque esta última falla en el orden de los cinco kilómetros (Bjorndal et al., 1983; Limpus, 1985). Sin embargo, cuando se analizan genéticamente las colonias de anidación de C. mydas y C. caretta del suroeste cubano, no se encuentra estructura genética significativa, a pesar de estar distantes en el orden de los cientos de kilómetros. Similarmente Chassin-Noria et al. (2004) no encuentran estructura genética significativa para colonias de anidación de C. mydas separadas en el orden de los 45 km en playas continentales de Michoacán. Bjorndal et al. (2005) tampoco encuentran significación dentro de poblaciones de tortugas en secciones de la playa de Tortuguero donde ocurre la anidación, separadas en el orden de los 29 km. En C. caretta no se encuentra estructura genética significativa entre colonias de anidación pertenecientes a una misma población de anidación distribuidas en un DISCUSIÓN______________________________________________________________________________________________ 86 intervalo de 150 – 700 km (ver Encalada et al. (1998), Francisco & Bowen (2001) y Hatase et al. (2002)). Chassin-Noria et al. (2004) considerando los resultados de Encalada et al. (1996) y FitzSimons et al. (1997) concluyen que la conducta de reproducción en el lugar de origen en C. mydas opera sólo a grandes escalas geográficas. En contraste, con el empleo de minisatélites multilocus se encontraron bajos niveles de dispersión de las ovíferas de C. mydas respecto a los sitios de anidación en Tortuguero (Peare & Parker, 1996), respaldados por una correlación negativa significativa entre la similitud genética por pares de ovíferas y la distancia entre sus nidos, ya sea dentro de una o entre dos temporadas. No obstante, Peare & Parker (op.cit.) encuentran altos niveles de dispersión de ovíferas respecto a los sitios de anidación en Melbourne Beach, Florida. Mediante un programa de marcación física realizado en esta localidad se demostró fidelidad por el sitio de anidación dentro y entre épocas reproductoras (Johnson, 1994), por lo que Peare & Parker (1996) concluyen que los altos niveles de dispersión de las ovíferas en Melbourne (Florida) han sido consecuencias del aumento de la perturbación antrópica en relación al continuo desarrollo del lugar. Los fallos a la filopatría de C. mydas son comunes y en el orden de la docena de kilómetros. En programas de marcación física donde se han obtenido curvas de saturación en las playas de anidación, se ha constatado que los fallos a la filopatría durante las remigraciones son del orden del 15% y hasta el 36% en un radio de 60 km aproximadamente, en el sur de la Gran Barrera Coralina Australiana (Limpus et al., 1994c) y en la península de Guanahacabibes (Azanza, com. pers.) respectivamente. El caso más extremo del fallo durante el proceso de remigración ha sido referido para una ovífera entre dos islas del Índico Occidental separadas en el orden de los 2000 km (Le Gall & Hughes, 1987). Considerando la existencia de una barrera geográfica entre estas áreas de anidación (isla continental Madagascar) más que la distancia que las separa, el fallo producido a la conducta de reproducción en el lugar de origen debe ser clasificado como filopátrido y no a la fidelidad por el sitio de anidación. De cualquier forma, este tipo de fallo es un récord, puesto que en otras áreas de anidación con varios programas intensos de marcación física no se ha detectado. En C. caretta, a pesar de que existe el consenso de que es poco filopátrida, los fallos a la filopatría dependen mucho de la zona de estudio. En Australia, durante nueve temporadas de anidación de un programa de marcación – DISCUSIÓN______________________________________________________________________________________________ 87 recaptura, el 98% de las ovíferas se recapturaron en el mismo lugar (Limpus, 1985) mientras que en Cumberland Island (Georgia, USA) el 51% retornó dentro de los ca. 17 km, el 7% dentro de ca. 50 km y el 42% lo hizo a distancias mayores del sitio de marcación (Bell & Richardson, 1978). Por otra parte, los fallos durante las reanidaciones son más bajos, en el orden del 5.3 % para C. mydas en un área de anidación de ca. 50 km dentro de la península de Guanahacabibes (Azanza, com. pers.). Ya se han analizado las relaciones entre el endemismo y 1) las distinciones establecidas para la conducta de reproducción en el lugar de origen según Carr (1975), 2) la limitada incidencia de los fallos a la fidelidad por el sitio de anidación durante una misma estación reproductora en comparación con las remigraciones, y 3) la influencia de la geomorfología y características oceanográficas de las áreas de anidación. No obstante, se considera que el poder resolutivo y el modo de empleo del marcador molecular son tan importantes como los aspectos ya mencionados para analizar la condición de endemismo en las colonias/ poblaciones de anidación de tortugas marinas. La tasa de mutación de la RNC del mtDNA de tortugas marinas es baja (Avise et al., 1992a). Razón por lo cual se considera que para evaluar la fidelidad por el sitio de anidación debe utilizarse un marcador más variable y por ende, que modele de manera más realista la dinámica de la población en una escala de tiempo ecológico, como es el caso de los microsatélites. En el modo de empleo de este marcador nuclear debe de cuidarse además el número de alelos por locus en relación al tamaño de la muestra (Kalinowski, 2002), el número de loci (Kalinowski, 2005). También se conoce que los machos son filopátridos (FitzSimons et al., 1997), pero no se tiene la certeza de que sean fieles al sitio de anidación en las colonias de anidación cubanas. Por consiguiente, para estudiar la fidelidad por el sitio de anidación de las ovíferas de tortuga marina, a partir de un marcador molecular, se deben buscar loci hipervariables, como los microsatélites, que permitan identificar a cada individuo mediante su genotipaje; por lo que el marcador podría carecer de valor en estudios genéticos a nivel poblacional. Igualmente sucedería en el caso de probar la filopatría de una especie de tortuga marina en la colonia de anidación en un tiempo ecológico y no histórico, donde se impone la limitante temporal en el esfuerzo de la medición, pues la media y la moda de remigración son para C. mydas de 2.86±0.23 (Van Buskirk & Crowder, 1994) y 5 (Limpus et al., 1994c) DISCUSIÓN______________________________________________________________________________________________ 88 años respectivamente y para C. caretta de 2.59±0.15 años (Dodd, 1988). Todo lo cual determina la necesidad de grandes esfuerzos para probar la conducta de filopatría. El tamaño efectivo de la población también es dependiente de la diversidad genética (Avise et al., 1988). Por esta razón se obtiene, que el tamaño efectivo de la población de anidación de C. mydas de Quintana Roo es mayor que el evidenciado en la población de anidación de Tortuguero, al igual que en la generalidad de las poblaciones de anidación de C. caretta en relación a las de Quintana Roo, Dry Tortugas y el noreste de la Florida. En las poblaciones de anidación de Tortuguero y noreste de la Florida de C. mydas y C. caretta respectivamente, el tamaño efectivo ha sido dependiente del método utilizado. Cuando la diversidad nucleotídica es baja, el valor del estimador theta se subestima. Consecuentemente, los tamaños efectivos de estas poblaciones de anidación no se corresponden con los tamaños censales de hembras. Comúnmente el tamaño efectivo es menor que el tamaño censal (Nei & Imaizumi, 1966) como consecuencia e.g., de la superposición de generaciones, y por consiguiente de la estratificación de edades y del incremento probabilístico de la consanguinidad. El gran tamaño efectivo de la población de anidación de C. mydas en Quintana Roo puede ser una consecuencia de las entradas a partir de fuentes vecinas como ha ocurrido con la colonización de Isla de Aves (Lahanas et al., 1994). Lo mismo sucede con las poblaciones de anidación de C. caretta de la península de la Florida y con el suroeste de Cuba donde están representados ambos linajes. Por otro lado, mientras mayor sea el tamaño censal de la población sumidero y menor la tasa de mutación el efecto de la colonización será menos apreciable (Lahanas et al., op.cit.), como ha ocurrido en la población de anidación de C. mydas en Tortuguero. Los cuellos de botella recientes e históricos sobre las poblaciones naturales (Nei & Graur, 1984) y el efecto fundador evidenciado en la colonización y recolonización de playas de anidación por ovíferas que fallan a la fidelidad por el sitio de anidación (Lahanas et al., 1994), podrían también reducir drásticamente la diversidad genética y por ende disminuir el valor del tamaño efectivo de la población. Las fluctuaciones demográficas en las poblaciones de anidación analizadas podrían evidenciarse mediante el cumplimiento del modelo de expansión súbita a través de la distribución desigual de frecuencias, como ocurre con la generalidad de las poblaciones DISCUSIÓN______________________________________________________________________________________________ 89 del linaje de C. mydas del Atlántico Sur (Formia et al., 2002a). Sin embargo, en las poblaciones de anidación analizadas en ambas especies sólo se cumple para unas pocas y durante el Pleistoceno, mucho antes de la última glaciación, excepto en las poblaciones de C. caretta en Dry Tortugas y el noroeste de la Florida donde al parecer ocurrió después. Consecuentemente, la actual distribución y representatividad de algunas poblaciones de anidación de C. mydas y C. caretta podrían ser más antiguas que las informadas por Encalada et al. (1996, 1998) respectivamente. La no correspondencia entre la prueba de Fu y el modelo de expansión súbita en todos los casos, no proporciona una dirección acertada sobre como estudiar la demografía histórica de las poblaciones de anidación en estas especies. La distribución del número observado de diferencias entre pares de haplotipos mediante el modelo de expansión súbita (Rogers & Harpending, 1992) es menos potente que la prueba de Fu (Ramos-Onsins & Rozas, 2002), a pesar de que ésta tiene un comportamiento mejor en muestras grandes (Fu, 1997). No obstante, ¿podrían las poblaciones de C. mydas y C. caretta del Atlántico Norte ajustarse al modelo de expansión súbita? Si el marcador molecular es selectivamente neutral debe esperarse que poblaciones en equilibrio no estén bajo selección o que las fuerzas evolutivas estén compensadas. Es por esto que, si existiera migración no tendría efectos significativos sobre la distribución de las frecuencias génicas, i.e. una población fuente cuasicerrada. La historia natural de las tortugas marinas, apoyada por los resultados filogenéticos y filogeográficos, se basa en un efecto de colonizaciones y recolonizaciones. Consecuentemente la(s) huella(s) de las fluctuaciones demográficas puede(n) ser borradas como ha ocurrido en algunas poblaciones de la especie humana (Excoffier & Schneider, 1999). Los resultados relacionados con el F- Φ ST indican que la diferenciación entre las poblaciones de anidación de C. mydas y C. caretta se debe a la distribución (redistribución) de los genes dentro de la población, más que a los cambios de éstos (mutación). Por consiguiente, la migración ha sido la principal fuerza evolutiva que trazó los genes en las poblaciones de anidación de estas especies. Esta fuerza evolutiva se demuestra por fallos históricos en el retorno hacia los sitios natales de reproducción. Durante el intervalo de remigración una hembra de tortuga marina puede viajar miles de kilómetros (Luschi et al., 2003). Si esta hembra falla al retornar a su sitio de anidación, podría contribuir genéticamente a alguna de las colonias de anidación cercanas dentro de DISCUSIÓN______________________________________________________________________________________________ 90 su ruta de migración. Sin embargo, las colonias de anidación tienen algún grado de endemismo, sobre todo en C. mydas, y en las colonias de anidación localizadas en las islas, y sus procesos evolutivos se han ajustado al modelo de flujo genético pasarela (aislamiento por distancia dentro de grupos según Reece et al., 2005). Estas evidencias demuestran que los fallos a la filopatría están sesgados con la distancia. Si la ovífera no encuentra condiciones mínimas para anidar en el área donde nació entonces buscará otra área de anidación cercana. Estos fallos pueden ser considerados una consecuencia de la supervivencia más que fallos inherentes a la conducta de reproducción en el lugar de origen de la especie. De hecho, Encalada et al. (1996) dudaron de la capacidad de supervivencia de C. mydas y C. caretta en áreas de anidación en altas latitudes durante los períodos de glaciación, donde no anida E. imbricata ni en la actualidad (Mortimer & Donnelly, 2007). No obstante, los fallos a la filopatría ocurren e.g., las variaciones clinales no sólo de C. caretta en la península de la Florida, sino la del haplotipo CM-A5 de C. mydas, cuya frecuencia aumenta con la disminución en la latitud y por consiguiente, con la disminución de la distancia geográfica entre las poblaciones de anidación considerando la población fuente de Surinam (ver Lahanas et al. (1994) y Encalada et al. (1996)). Teniendo en cuenta todo lo anteriormente dicho, no debe de esperarse que el modelo de expansión súbita se ajuste para las poblaciones de anidación de C. mydas y C. caretta bajo los escenarios demográficos siguientes: I) un fuerte evento fundador de los haplotipos más distribuidos y mejor representados en cada uno de los linajes; II) redistribución de individuos como consecuencia de cambios drásticos que ocurrieron durante el Pleistoceno; III) una ruptura de las colonias como consecuencia de la colonización europea y IV) una combinación de todos los escenarios anteriores. V.4 Relaciones filogenéticas y filogeográficas. En las colonias de anidación cubanas de C. caretta se presentan dos linajes, ampliamente representados en la cuenca atlántica al igual que ocurre con E. imbricata (Okayama et al., 1999). Únicamente en C. caretta éstos no están emparentados naturalmente (Encalada et al., 1998; Bowen et al., 2007). Contrariamente, en C. mydas, sólo se presenta un linaje de los dos naturalmente emparentados de la cuenca atlántica (Encalada et al., 1996; Bowen et al., op.cit.). DISCUSIÓN______________________________________________________________________________________________ 91 La elección del haplotipo CM-A1 como ancestro del linaje de C. mydas de los mediterráneos americano – viejo mundo y costa atlántica de la península de la Florida, contrasta con el hecho de que el haplotipo CM-A3 es el más distribuido y representado en las poblaciones de anidación del Mediterráneo americano. El número de cambios significativamente alto entre los haplotipos externos y CM-A1, al igual que la posición intermedia del mismo considerando los otros dos centros de diversificación, han influido en la identificación de este haplotipo como ancestro del linaje. En el linaje análogo de C. caretta, la identificación del haplotipo ancestral fue sencilla porque existe un solo centro de diversificación. Sin embargo, en el otro linaje el análisis es más complejo porque existen tres centros de diversificación y sólo el haplotipo CC-A1 está ampliamente distribuido y representado. Considerando que este linaje tiene una amplia distribución en las altas latitudes, disminuyendo su representatividad hacia las zonas subtropicales, que ha sido un colonizador reciente, y que su diversificación muestra claramente dos sub-linajes (Bowen & Kart, 2007) a partir de los cuales ocurrió la derivación de los haplotipos concomitantes en el tiempo; el haplotipo ancestral dentro de éste debe ser aquel que tenga una posición intermedia e interna, i.e. el haplotipo CC-A11. La divergencia reciente (durante el Pleistoceno Superior) entre los haplotipos de las poblaciones de anidación del Mediterráneo americano para estas tortugas marinas está en correspondencia con las pocas mutaciones que separan a la generalidad de éstos del haplotipo ancestral dentro de cada linaje. Esta característica es común al linaje del Atlántico Sur de C. mydas (Formia et al., 2002a) y también en E. imbricata (Okayama et al., 1999; Bowen et al., 2007). No obstante, esta divergencia ocurrió mucho antes de la separación de las actuales poblaciones de anidación en C. mydas. En C. caretta la definición de las identidades poblacionales aparentemente ocurrió antes de la diversificación de los haplotipos, lo cual implicaría un apreciable porcentaje de endemismo haplotípico de los haplotipos menos representados dentro de cada linaje, aunque supuestamente la especie fallara frecuentemente a la conducta de reproducción en el lugar de origen en un intervalo de distancia del orden de los miles de kilómetros. La presencia de dos linajes no emparentados naturalmente en estas poblaciones de anidación ha sido la causa fundamental del incremento en la divergencia de secuencias. DISCUSIÓN______________________________________________________________________________________________ 92 Las poblaciones de anidación de estas especies comparten con mayor representatividad los haplotipos más distribuidos del linaje (incluido el ancestro) como consecuencia de: I) eventos históricos de dispersión (colonización y recolonización), a partir de los cuales se fundaron las colonias de anidación con los haplotipos más distribuidos y mejor representados en la región, II) fluctuaciones demográficas en demos establecidos que facilitaron la diversificación y la fijación de variantes haplotípicas reconocidas como endemismos de baja representatividad, y III) eventos de dispersión en tiempo ecológico (recolonización) entre colonias de anidación vecinas localizadas a lo largo de costas continentales afectadas por semejantes procesos oceanográficos. Las primeras dos causas responden a eventos que ocurrieron durante la época geológica, que devinieron en la composición actual y la estructura poblacional con mayores diferencias dentro que entre poblaciones de anidación de la región como consecuencia de un flujo genético restringido con aislamiento por distancia. Por supuesto, no se han obviado las dispersiones en tiempo ecológico provocadas por fallos a la conducta filopátrida de la especie, que atentan en contra del endemismo poblacional. Sin embargo, este tipo de dispersión ha sido menos frecuente y de ritmo lento entre las colonias de anidación localizadas en áreas de anidación separadas ya sea geomorfológicamente o por condiciones oceanográficas particulares, pudiéndose evidenciar en clinas a lo largo de las costas oeste y este de la península de la Florida para C. caretta (haplotipos CC-A1 y 2) y en la costa norte de Suramérica hasta Mesoamérica para C. mydas (haplotipo CM-A5) y en la baja diversidad nucleotídica en la generalidad de las poblaciones de anidación que sólo están integradas por un linaje o por una representatividad mayoritaria de uno de los linajes que se presenta. Resulta interesante que en la cuenca del mar Mediterráneo sólo se encuentren haplotipos de los linajes del Mediterráneo americano para ambas tortugas marinas, lo cual concuerda con el sistema de corrientes marinas (corrientes de la Florida, el Golfo y las de Azores y Portugal, además de las corrientes de Canarias y contracorriente Subtropical), que conecta ambos mediterráneos después de la constitución del istmo de Panamá. En C. mydas son endémicos, mientras que en C. caretta se comparten en su mayoría con el Mediterráneo americano. No es de extrañar la representación del mismo linaje en C. mydas porque en el Mediterráneo americano es evidente la presencia histórica del linaje homónimo, con recientes colonizaciones del linaje Atlántico Sur cuyo aporte es DISCUSIÓN______________________________________________________________________________________________ 93 despreciable en el análisis. La interpretación de la asociación geográfica de los haplotipos en los clados integrados por las poblaciones de anidación de C. mydas en Chipre y Quintana Roo, revela una antigua relación entre el centro de diversificación CM-A13 y las poblaciones de anidación americanas. No obstante, el tiempo de diversificación de los haplotipos de Chipre no difiere del resto de los haplotipos del linaje y es más antiguo que la fecha en que los hábitat quedaron disponibles para la anidación de C. mydas dentro de la cuenca del mar Mediterráneo, i.e. después de la glaciación de Würm (Encalada et al., 1996). Adicionalmente, en la cuenca del mar Mediterráneo no anidan el ancestro (CM-A1) ni el haplotipo más distribuido y representado del linaje (CM-A3). Consecuentemente, el haplotipo CM-A13, debió ser originado en alguna población de anidación del Mediterráneo americano. Entonces se puede interpretar, que la presencia de haplotipos endémicos en la población de anidación de Chipre se debe a la fijación de genes en la cuenca del mar Mediterráneo. Actualmente, el haplotipo CM-A13 es probablemente raro dentro de las colonias de anidación estudiadas o poco frecuente en otras poblaciones que aún no han sido estudiadas con marcadores moleculares en el Mediterráneo americano, una combinación de estas dos variantes o simplemente pertenece a colonias de anidación que se han extinguido en la actualidad. De hecho, este haplotipo fue encontrado en hábitats de desarrollo en la parte este-central de la Florida (Bagley, 2003). Estas alternativas podrían explicar de manera más convincente la presencia de haplotipos endémicos en otras poblaciones localizadas en áreas de anidación con hábitat inestables para el éxito reproductor durante las glaciaciones, más que la concepción de una gran población ancestral (panmicta) en la Florida como sugiere Allard et al. (1994). En C. caretta el análisis se hace complejo, pues asalta la polémica de no estar representados ambos linajes en la cuenca del mar Mediterráneo. La colonización de esta cuenca según Bowen et al. (1993, 1994), debió ser después de concluir la última glaciación (10 000 A) en correspondencia con la limitante de las bajas temperaturas para el éxito reproductor de la especie, la representación única del linaje mediterráneos americano – Viejo Mundo, y la divergencia por una sola mutación entre el haplotipo endémico CC-A6 del ancestro (CC-A2). No obstante, la especie pudo establecerse durante los períodos glaciales en el sur de la Florida, no así en las actuales locaciones de Georgia, Carolina del Sur y Carolina del Norte, donde el establecimiento del linaje Atlántico DISCUSIÓN______________________________________________________________________________________________ 94 – península de la Florida debió ser concomitante con la colonización de la cuenca del mar Mediterráneo (Bowen et al., 1993; Encalada et al., 1998). Esto se corrobora con la baja diversidad genética de las colonias establecidas en estas latitudes como consecuencia de sucesivos cuellos de botellas (Bowen et al., op.cit.; Encalada et al., op.cit.). Considerando la hipótesis filogeográfica de Encalada et al. (1998), surge la interrogante de por qué, si el linaje Atlántico – península de la Florida sólo se presenta con radiación exitosa en las colonias de latitudes más altas y estaba establecido previamente en la cuenca atlántica, no fue el colonizador de la cuenca del mar Mediterráneo. Sólo queda pensar en que una vez concluida la Glaciación de Würm, cuando el mar comenzó a reestablecer los niveles actuales, hubo un gran influjo hacia el mar Mediterráneo que arrastró numerosos migrantes correspondientes a las colonias de anidación posicionadas en mejores condiciones en el Mediterráneo americano y por consiguiente, con mayores tamaños poblacionales. Estos debieron pertenecer probablemente al haplotipo más distribuido y representado, también ancestro del linaje mediterráneos americano – Viejo Mundo. Esta explicación concuerda con la presencia actual del haplotipo CC-A1 en las pesquerías del mar Mediterráneo, luego de aumentar los tamaños censales en las colonias de anidación norteñas del Mediterráneo americano. En C. mydas debió ocurrir de forma similar, pero como es una especie más tropical debió experimentar una baja tasa de supervivencia, que por deriva fijó aleatoriamente algún haplotipo representado en los migrantes. V.5 Implicaciones para la conservación. Las poblaciones de anidación cubanas de C. mydas, al igual que las de E. imbricata (Espinosa et al., 2004), constituyen una población de variantes genéticas relictas dentro del Mediterráneo americano, con alto endemismo genético y moderada representatividad de individuos endémicos. Las colonias de anidación de C. caretta del suroeste cubano también constituyen una población de anidación independiente, con la presencia de un haplotipo endémico. Por consiguiente, ambas poblaciones deben ser consideradas como MUs, al igual que otras en el Mediterráneo americano (Karl & Bowen, 1999), siguiendo los criterios de Moritz (1994) y Allendorf & Luikart (2007). Estas MUs deberán tenerse en cuenta operativamente en las estrategias de los planes de manejo tanto nacionales como regionales de estas especies, cuya función contribuya a la preservación de la diversidad genética y de la supervivencia de estas poblaciones de anidación. DISCUSIÓN______________________________________________________________________________________________ 95 Las colonias de anidación que conforman a estas poblaciones, al igual que otras poblaciones de tamaño discreto, han funcionado como reservorios genéticos durante eventos que han provocado la extirpación de grandes poblaciones de anidación como sugiere Lahanas et al. (1994) para C. mydas. Consecuentemente, deben recibir igual atención en los programas de conservación de estas especies, aunque los tamaños censales de ovíferas de las poblaciones de anidación cubanas no clasifiquen entre los más representativos de las áreas de anidación de la cuenca atlántica según Dodd (1988) y Seminoff (2002). Algunos de los haplotipos endémicos de la población del suroeste cubano eran de origen desconocido (C. mydas: CM-A48 según Bagley (2003); CM-A27 y 28 según Bjorndal & Bolten (inscritos en el GenBank 2001); C. caretta: CC-A12 (Bolten et al., 1998). Los haplotipos de C. mydas fueron encontrados en áreas de desarrollo del este central de la Florida por Bagley (2003), y el de C. caretta en similares áreas pero de Madeira por Bolten et al. (1998). Los haplotipos CM-A27 y 28 también se encontraron en áreas de forrajeo en Carolina del Norte (Bass, 1999). Conociéndose la población de anidación a la que pertenecen estos haplotipos se podrá calcular de forma más realista la contribución de las poblaciones de anidación a los hábitat marinos de agregación de estas especies, algunos de éstos usados consultivamente (Campbell, 2003). Consecuentemente, este resultado también contribuye además de al conocimiento del nicho, al de la ruta migratoria de cualquier individuo identificado con un haplotipo endémico cubano durante su ciclo de vida en el Mediterráneo americano. Todo esto provee una oportunidad para la colaboración científica nacional e internacional y para el manejo sostenible de estas especies y sus hábitat. Finalmente, los resultados de este trabajo también aportan información útil para el diseño de áreas marinas protegidas (MPAs) en Cuba encaminadas a la conservación de estas tortugas marinas en declive (Seminoff, 2002), integrando los hábitat que usa en función del nicho y considerando el potencial evolutivo de sus demos dentro del genofondo de la metapoblación regional. Estos criterios también son relevantes para el diseño de MPAs (Bowen & Roman, 2005), aunque la generalidad de éstas son planeadas siguiendo sólo DISCUSIÓN______________________________________________________________________________________________ 96 criterios ecológicos en función de ecosistemas particulares. El diseño de MPAs que contemple la conservación de especies migratorias como las tortugas marinas requiere, por la complejidad del ciclo de vida de éstas, integración de conocimientos en los campos de la filogenia, ecología y evolución para garantizar su efectividad; por lo que el modelo Orlog (Bowen & Roman, op.cit.) podría ser una guía eficaz para lograr la meta. CONCLUSIONES__________________________________________________________________________________________ 97 VI. CONCLUSIONES. 1. Las colonias de anidación de C. caretta y C. mydas del suroeste de Cuba constituyen una población panmicta, como consecuencia de un amplio flujo absoluto de migrantes (fallos a la conducta de reproducción en el lugar de origen en tiempo histórico y ecológico) dentro de áreas geográficas con historia y patrones físicos comunes, por consiguiente el archipiélago de los Canarreos y la península de Guanahacabibes deben de tenerse en cuenta como una unidad territorial para el diseño de áreas protegidas marinas en función de la conservación de las tortugas marinas. 2. Las poblaciones de anidación de C. caretta y C. mydas del suroeste de Cuba muestran estructura genética significativa respecto a las poblaciones de anidación del Mediterráneo americano, como consecuencia de un flujo de migrantes restringido entre colonias de anidación vecinas (fallos históricos a la conducta de reproducción en el lugar de origen, que llegan a describir clinas) en correspondencia con el modelo evolutivo pasarela, por lo que constituyen identidades poblacionales con independencia en el tiempo ecológico, que deben ser manejadas particularmente para evitar pérdida de la variabilidad genética dentro de cada especie. 3. Las poblaciones de anidación de C. caretta y C. mydas del suroeste de Cuba tienen un endemismo relativamente alto, mayor en C. mydas, porque a pesar de que las especies fallan a la conducta de reproducción en el lugar de origen, los fallos suelen ser más frecuentes en áreas costeras continuas (continentales), afectadas por patrones oceanográficos comunes, que entre áreas discontinuas (islas), que no formaron partes de otros territorios durante los períodos interglaciales del Pleistoceno. 4. La población de anidación del suroeste de Cuba de C. caretta presenta alta diversidad genética en comparación con las demás poblaciones de la cuenca atlántica, mientras que la de C. mydas sólo tiene alta diversidad haplotípica, como consecuencia de dispersiones de los haplotipos más distribuidos y representados de los linajes, que constituyeron fallos históricos a la conducta de reproducción en el lugar de origen que han “borrado” las huellas de expansiones demográficas. La baja diversidad nucleotídica en la población de anidación de C. mydas es CONCLUSIONES__________________________________________________________________________________________ 98 consecuencia directa de la presencia histórica de un solo linaje en el Mediterráneo americano. 5. Las poblaciones de C. caretta y C. mydas del suroeste de Cuba son tan antiguas como las continentales, y las de otras islas que no quedaron sumergidas durante el Pleistoceno, porque tienen en cada especie no sólo los haplotipos más distribuidos y representados sino diversos haplotipos (algunos endémicos) de los linajes existentes en el Mediterráneo americano, cuyas frecuencias son significativamente diferentes. RECOMENDACIONES_______________________________________________________________________________________________ 99 VII. RECOMENDACIONES. • Emplear el concepto de poblaciones de anidación cubanas de C. mydas y C. caretta, sobre la base de los resultados de esta tesis en las políticas de manejo y en el diseño o rediseño de áreas protegidas marinas en el archipiélago de Cuba. • Determinar las contribuciones genéticas de las poblaciones de anidación cubanas de C. mydas y C. caretta a las áreas marinas de agregación de la cuenca atlántica, así como a las zonas de pesquerías cubanas para cuantificar la presencia de las poblaciones de anidación cubanas en los agregados marinos con vistas a evaluar el impacto del manejo sostenible de las poblaciones fuentes. • Estudiar comparativamente la estructura, diversidad y relaciones genéticas de las colonias de anidación de C. mydas y C. caretta en los archipiélagos Jardines de la Reina y del Rey con las poblaciones de anidación caracterizadas en el presente trabajo. • Emplear microsatélites como marcador molecular para el estudio de la estructura de las colonias de anidación cubanas de tortugas marinas en tiempo ecológico, lo cual incluiría la aportación genética de los machos, así como de las contribuciones de estas colonias a las áreas marinas de agregación alrededor del archipiélago. REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ VIII. REFERENCIAS BIBLIOGRÁFICAS. Aka, F. T., M. Kusakabe, K. Nagao & G. Tanyileke. 2001. Noble gas isotopic compositions and water/ gas chemistry of soda springs from the islands of Bioko, São Tome and Annobón, along with Cameroon volcanic line, West Africa. Applied Geochemistry 16: 323-338. Allard, M. W., M. M. Miyamoto, K. A. Bjorndal, A. B. Bolten & B. W. Bowen. 1994. Support for natal homing in green turtles from mitochondrial DNA sequences. Copeia 1994: 34– 41. Allendorf, F. W., & G. Luikart. 2007. Conservation and the genetics of populations. Blackwell Publishing, UK. ALRTM (Atlantic Loggerhead Recovery Team Meeting). 2003. National Conservation Training Center, Shepherdstown, WV: 22-26 september. Anagnostopoulos, T., P. M. Green, G. Rowley, C. M. Lewis & F. Giannelli. 1999. DNA variation in a 5-MB region of the X chromosome and estimates of sex-specific typespecific mutation rates. Am. J. Hum. Genet. 64: 508-517. Anónimo. 1998. Transferencia, con nota aclaratoria, de la Población cubana de Tortugas Carey (Eretmochelys imbricata) del Apéndice I al apéndice II de la CITES. Rev. Cubana Inv. Pesq. 1 (22): 1-31. Arnason, U., A. Gullberg & B. Widengren. 1993. Cetacean mitochondrial DNA control region: Sequences of all extant baleen whales and two sperm whale species. Mol. Biol. Evol. 10: 960-970. Avise, J. C. 1986. Mitocondrial DNA and the evolutionary genetics of higher animals. Phil. Trans. Royal Soc. Lond. B 312: 325-342. Avise, J. C. 1992. Molecular population structure and biogeographic history of a regional fauna: A case history with lessons for conservation biology. Oikos 63:62-76. Avise, J. C. 2004. Molecular markers, natural history, and evolution. 2nd ed. Champman and Hall, New Cork. Avise, J. C., B. W. Bowen, T. Lamb, A. B. Meylan, & E. Bermingham. 1992. Mitochondrial DNA evolution at a turtle’s pace: evidence for low genetic variability and reduced microevolutionary rate in the Testudines. Mol. Biol. Evol. 9: 457-473. Avise, J. C., D. Walker & G. Johnss. 1998. Speciation durations and Pleistocene effects on vertebrate phylogeography. Proc. R. Soc. Lond. B. 265: 1707-1712. REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ Avise, J. C., J. E. Neigel & J. Arnold. 1984. Demographic influences on mitochondrial DNA lineage survivorship in animal populations. J. Mol. Evol. 20: 99-105. Avise, J. C., R. M. Ball, Jr. & J. Arnold. 1988. Current versus historical population sizes in vertebrate species with high gene flow: a comparison based on mitochondrial DNA lineages and inbreeding theory for neutral mutations. Mol. Biol. Evol. 5: 331-344. Azanza, J., M. E. Ibarra, G. Espinosa, R. Díaz & G. Sansón. 2003. Conducta de anidación de la tortuga verde (Chelonia mydas) en las playas Antonio y Caleta de los Piojos de la Península de Guanahacabibes, Pinar del Río, Cuba. Rev. Inv. Mar. 24 (3): 231-240. Bagley, D. A. 2003. Characterizing juvenile green turtles (Chelonia mydas) from three east central Florida developmental habitats. MS thesis, University of Central Florida, Orlando, Florida, USA. Baillie, J. & B. Groombridge. 1996. IUCN Red List of Threatened Animals. Gland, Switzerland: IUCN. Balazs, G. H. 1982. Growth rates of immature green turtles in the Hawaiian Archipelago. 117-125. En: K. A. Bjorndal (ed.), Biology and Conservation of Sea Turtles. Smithsonian Institution Press, Wash., DC. USA. Balazs, G. H. 2000. Factores a considerar en el marcado de Tortugas marinas. 116-125. En: Técnicas de Investigación y Manejo para la conservación de las Tortugas Marinas. K. L. Eckert, K. A. Bjorndal, F. A. Abreu-Grobois y M. Donnelly (eds), UICN/ CSE, Grupo de Especialistas en Tortugas Marinas Publicación 4 (Traducción al español). Ballar, J. W. O., & D. M. Rand. 2005. The population biology of mitochondrial DNA and its phylogenetic implications. Annu. Rev. Ecol. Evol. Syst. 36: 621-42. Bane, J. M. Jr., & W. K. Dewar. 1988. Gulf Stream bimodality and variability downstream of the Charleston Bump. J. Geophysical Research 93: 6695-6710. Baran, I. & M. Kasparek. 1989. Marine Turtles Turkey: Recommendations for Conservation and Management. Report for WWF, Gland, Switzerland. Barbosa, C. A., C. Broderick & P. Catry. 1998. Marine turtles, in the Orango National Park (Bijagós Archipelago, Guinea-Bissau). Mar. Turtle Newsl. 81: 6-7. Barton, N. H., & M. Slatkin. 1986. A quasi-equilibrium theory of the distribution of rare alleles in subdivided population. Heredity 56: 409-415. REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ Bass, A. L. 1999. Report on the analysis of green turtles from the Pamlico – Albemarle Estuarine Complex, North Carolina. Department of Fisheries and Aquatic Sciences, University of Florida, USA. Bass, A. L., & W. N. Witzell. 2000. Demographic composition of immature green turtles (Chelonia mydas) from the east central Florida coast: Evidence from mtDNA markers. Herpetologica 56(3): 357-367. Bass, A. L., C. J. Lagueux & B. W. Bowen. 1998. Origin of green turtles, Chelonia mydas, at “Sleeping Rocks” off the northeast coast of Nicaragua. Copeia 1998: 1064-1069. Bass, A. L., D. A. Good, K. A. Bjorndal, J. I. Richardson, Z-M. Hillis, J. A. Horrocks & B. W. Bowen. 1996. Testing models of female reproductive migratory behavior and population structure in the Caribbean Hawksbill turtle, Eretmochelys imbricata with mtDNA sequences. Mol. Ecol. 5: 321-328. Bass, A. L., S. P. Epperly & J. Braun-McNeill. 2004. Multi-year analysis of stock composition of a loggerhead turtle (Caretta caretta) foraging habitat using maximum likelihood and Bayesian methods. Cons. Gen. 5: 783-796. Bell, R., & J. I. Richardson. 1978. An analysis of tag recoveries from loggerhead sea turtles (Caretta caretta) nesting on Little Cumberland Island, Georgia. 20-24. En: G. E. Henderson (ed.), Proceeding of the Florida and interregional conference on sea turtles, 24-25 July 1976, Jensen Beach, Florida. Florida Marine Research Publications No. 33. Birky, C. W., Jr., T. Maruyama & P. Fuerst. 1983. An approach to population and evolutionary genetic theory for genes in Mitochondria and Chloroplasts, and some results. Genetics 103: 513-527. Bjorndal, K. A. & A. B. Bolten. 1988. Growth rates of immature green turtles, Chelonia mydas, on feeding ground in the southern Bahamas. Copeia 1988: 555. Bjorndal, K. A., A. B. Bolten & M. Y. Chaloupka. 2000. Green turtle somatic growth model: evidence for density dependence. Ecol. Appl. 10: 269-282. Bjorndal, K. A., A. B. Bolten & S. Troëng. 2005. Population structure and genetic diversity in green turtles nesting at Tortuguero, Costa Rica, based on mitochondrial DNA control region sequences. Mar. Biol. 147: 1449-1457. Bjorndal, K. A., A. B. Meylan & B. J. Turner. 1983. Sea turtles nesting at Melbourne Beach, Florida. I. Size, growth and reproductive biology. Biol. Cons. 26: 65-77. REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ Bjorndal, K. A., J. A. Wetherall, A. B. Bolten & J. A. Mortimer. 1999. Twenty-six years of nesting data from Tortuguero, Costa Rica: an encouraging trend. Cons. Biol. 13: 126134. Bolten, A. B., K. A. Bjorndal, H. R. Martins, T. Dellinger, M. J. Biscoito, S. E. Encalada & B. W. Bowen. 1998. Transatlantic developmental migrations of loggerhead sea turtles demonstrated by mtDNA sequence analysis. Ecol. Appl. 8 (1): 1-7 Bowen, B. W. & J. C. Avise. 1995. Conservation genetics of marine turtles. 190-237. En: Avise, J. C. & J. L. Hamrick (eds.), Conservation Genetics: Case Histories from Nature. Chapman and Hall, New York. Bowen, B. W. 1999. Preserving genes, species, or ecosystems? Healing the fractured foundations of conservation policy. Mol. Ecol. 8: S5-S10. Bowen, B. W. 2004. Conservation implications of complex population structure in the loggerhead turtle (Caretta caretta). Final report to the Seatheast Fisheries Science Center. Requisition NFFN7020-4-00050. Bowen, B. W., & J. Roman. 2005. Gaia’s Handmaidens: the Orlog Model for conservation biology. Cons. Biol. 1037-1043. Bowen, B. W., & S. A. Karl. 1997. Population Genetics, Philogeography, and Molecular Evolution. 29-50. En: Lutz, P. L. & J. A. Musick (eds.), The Biology of Sea Turtles. CRC Press, Boca Ratón, Florida, USA. Bowen, B. W., & S. A. Karl. 2007. Population genetics and phylogeography of sea turtles. Mol. Ecol. doi: 10.1111/j.1365-294X.2007.03542.x. Bowen, B. W., A. B. Meylan, J. P. Ross, C. J. Limpus, G. H. Balazs y J. C. Avise. 1992. Global population structure and natural history of green turtle (Chelonia mydas) in terms of matriarchal phylogeny. Evolution 46: 865-881. Bowen, B. W., A. L. Bass, Shaio-Mei Chow, M. Bostrom, K. A. Bjorndal, A. B. Bolten, T. Okuyama, B. M. Bolker, S. Epperly, E. Lacasella, D. Shaver, M. Dodd, S. R. Hopkinsmurphy, J. A. Musick, M. Swingle, K. Rankin-Baransky, W. Teas , W. N. Witzell & P. H. Dutton. 2004. Natal homing in juvenile loggerhead turtles (Caretta caretta). Mol. Ecol. 13: 3797-3808. Bowen, B. W., F. A. Abreu-Grobois, G. H. Balazs, N. Kamezaki, C. J. Limpus & R. J. Ferl. 1995. Trans-Pacific migrations of the loggerhead turtle (Caretta caretta) demonstrated with mitochondrial DNA markers. Proc. Natl. Acad. Sci. USA 92: 3731-3734. REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ Bowen, B. W., N. Kamezaki, C. J. Limpus, G. H. Hughes, A. B. Meylan & J. C. Avise. 1994. Global phylogeography of the loggerhead turtle (Caretta caretta) as indicated by mitochondrial DNA haplotypes. Evolution 48: 1820-1828. Bowen, B. W., W. S. Grant, Z. Hillis-starr, D. J. Shaver, K. A. Bjorndal, A. B. Bolten & A. L. Bass. 2007. Mixed-stock analysis reveals the migrations of juvenile hawksbill turtles (Eretmochelys imbricata) in the Caribbean Sea. Mol. Ecol. 16: 49-60. Bowen, B. W., W. S. Nelson & J. C. Avise. 1993. A molecular phylogeny for marine turtles: Trait mapping, rate assessment, and conservation relevance. Proc. Natl. Acad. Sci. USA 90: 5574-5577. Bowen, D. Q. 1978. Quaternary Geology. A stratigraphic framework for multidisciplinary work. Pergamon Press, Oxford, United Kingdon. Briggs, J. C. 1974. Marine Zoogeography. McGraw-Hill, New York, EE.UU. Britten, R. J. 1986. Rates of DNA sequence evolution differ between taxonomic groups. Science 231: 1393-1398. Broderick, A. C. & B. J. Godley. 1996. Population and nesting ecology of the green turtle, Chelonia mydas, and the loggerhead turtle, Caretta caretta, in northern Cyprus. Zoology in the Middle East 13: 27-46. Broderick, D., C. Moritz, J. D. Miller, M. Guinea, R. I. T. Prince & C. J. Limpus. 1994. Genetic studies of the Hawksbill turtle Eretmochelys imbricata: evidence for multiple stocks in Australian waters. Pacific Cons. Biol. 1: 123-31. Brown, G. G., G. Gadaleta, G. Pepe, C. Saccone & E. Sbisà. 1986. Structural conservation and variation in the D-loop-containing region of vertebrate mitochondrial DNA. J. Mol. Biol. 192: 503-511. Brown, J. H., & M. V. Lomolino. 1998. Biogeography (second edition). Sinauer Associates, Inc. Publishers Sunderland, Massachusetts. Brown, J. R., A. T. Beckenbach & M. J. Smith. 1993. Intraespecific DNA sequence variation of the mitochondrial control region of white sturgeon (Acipencer transmontanus). Mol. Biol. Evol. 10: 326-341. Brown, W. M. 1983. Evolution of mitochondrial DNA. 62-68. En: M. Nei & R. K. Koehn (eds.), Evolution of genes and Proteins,. Sinauer Associates, Sutherland, MA. Brown, W. M., E. M. Prager, A. Wang & A. C. Wilson. 1982. Mitochondrial DNA sequences of primates: Tempo and mode of evolution. J. Mol. Evol. 18: 225-239. REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ Brown, W. M., M. George & A. C. Wilson. 1979. Rapid evolution of animal mitochondrial DNA. Proc. Natl. Acad. Sci. USA 76: 1967-1971. Buckley, H. A., L. R. Johnson, N. J. Shackleton & R. A. Blow. 1982. Late glacial to recent cores from the eastern Mediterranean. Deep Sea Research 29: 739-766. Campbell, L. 1998. Use them or lose them? Conservation and the consumptive use of marine turtle eggs at Ostional, Costa Rica. Environ. Cons. 25: 305-319. Campbell, L. 2003. Contemporary culture, use, and conservation of sea turtles. 307-338 En: P. L. Lutz, J. A. Musik & Wyneken (eds.), The biology of sea turtles, Vol II, CRC Press. USA. Campbell, L. M. 2002. Sustainable use of marine turtles: views of conservation experts. Ecol. Appl. 12(4): 1229-1246. Cann, R. L., M. Stoneking & A. C. Wilson. 1987. Mitochondrial DNA and human evolution. Nature 325: 31-36. Carr, A. 1967. So Excellent a Fish: A Natural History of Sea Turtles. Scribner, New York, USA. Carr, A. 1975. The Ascension Island green turtle colony. Copeia 3: 547-555. Carr, A. 1986. Rips, FADS, and little loggerheads. Bioscience 36(2): 92-100. Carr, A. 1987. New perspectives on the pelagic stage of sea turtle development. Cons. Biol. 1: 103-121. Carr, A. F., & L. Ogren. 1960. The ecology and migrations of sea turtle, IV: The green turtle in the Caribbean Sea. Bull. Am. Mus. Nat. His. 162: 1-48. Carr, A., A. Meylan, J. Mortimer, K. A. Bjorndal & T. Carr. 1982. Surveys of sea turtle population and habitats in the Western Atlantic. U. S. Department or Commerce NOAA Tech Memo. NMFS-SEFC-91. Carr, A., M. H. Carr & A. B. Meylan. 1978. The ecology and migrations of sea turtles. The west Caribbean green turtle colony. Bull. Am. Mus. Nat. Hist. 162: 1-46. Carreras, C., S. Pont & F. Maffucci. 2006. Genetic structure of immature loggerhead sea turtles (Caretta caretta) in the Mediterranean reflects water circulation patterns. Mar. Biol. 149: 1269-1279. Carrillo, E; G. J. W. Webb & S. Ch. Manolis. 1999. Hawksbill turtles (Eretmochelys imbricata) in Cuba: an assessment of the historical harvest and its impacts. Chel. Cons. Biol. 3(2): 264-280. REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ Castelloe J., & Templeton A. R. 1994. Root probabilities for intraspecific gene trees under neutral coalescent theory. Mol. Phyl. Evol. 3: 102-113. Caughley, G., & A. Gunn. 1996. Conservation Biology in theory and practice. Blackwell science, Inc, USA. Chaloupka, M. Y., C. J. Limpus & J. D. Miller. 2004. Green turtle somatic growth dynamics in a spatially disjunct Great Barrier Reef metapopulation. Coral Reefs 23: 325-335. Chassin-Noria, O., A. Abreu-Grobois, P. H. Dutton & K. Oyama. 2004. Conservation genetics of the east Pacific green turtle (Chelonia mydas) in Michoacan, Mexico. Genetica 1891: 1-12. Clement M., D. Posada & K. A. Crandall. 2000. TCS: a computer program to estimate gene genealogies. Mol. Ecol. 9 (10): 1657-1660. Clifton, K., D. O. Cornejo & R. S. Felger. 1995. Sea turtle on the Pacific Coast of Mexico. 199-209. En: K. Bjorndal (ed.), Biology and Conservation of Sea Turtles, Smithsonian Institute Press. Washington, D. C. Cornelius, S. E., M. A. Ulloa, J. C. Castro, M. Mata del Valle & D. C. Robinson. 1991. Management of olive ridley sea turtles (Lepidochelys olivacea) nesting at Playas Nancite and Ostional, costa Rica. 111-135. En: J. G. Robinson & K. H. Redford (eds.), Neotropical Wildlife Use and Conservation, University of Chicago Press, Chicago, USA. Coulondre, C., J. H. Miller, P. J. Farabaugh & W. Gilbert. 1978. Molecular basis of base substitution hotspots in Escherichia coli. Nature 274: 775-780. Crouse, D. T. 1999. Population modeling and implications for Caribbean Hawksbill sea turtle management. Chel. Cons. Biol. 3(2): 185-188. Demetropoulos, A., & M. Hadjichristophorou. 1989. Sea turtle conservation in Cyprus. Mar. Turtle Newsl. 44: 4-6. Díaz-Fernández, R., T. Okayama, T. Uchiyama, E. Carrillo, G. Espinosa, R. Márquez, C. Diez & H. Koike. 1999. Genetic sourcing for the hawksbill turtle Eretmochelys imbricata in the Northern Caribbean region. Chel. Cons. Biol. 3(2): 296-300. Dodd, C. K., Jr. 1988. Synopsis of the biological data on the loggerhead sea turtle Caretta caretta (Linnaeus, 1758). U. S. Fish Wildl. Serv. Biol. Rep. 88: 110. Doi, A, H. Suzuki & E. T. Matsuura. 1999. Genetic analysis of temperature-dependent transmission of mitochondrial DNA in Drosophila. Heredity 82: 555-60. REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ Doi, T., R. Márquez, H. Kimono & N. Azeno. 1992. Diagnosis and conservation of the hawksbill turtle population in the Cuban Archipelago. Tech. Rep., Japan Bekko Association, Japan. Dutton, P. H., S. K. Davis, T. Guerra & D. Owens. 1996. Molecular Phylogeny for Marine Turtles Based on Sequences of the ND4-Leucine tRNA and Control Regions of Mitochondrial DNA. Mol. Phyl. Evol. 3(5): 511-521. Ehrhart, L. M., & B. E. Witherington. 1992. Green Turtle. 90-94. En: P. E. Moler (ed.), Rare and Endangered Biota of Florida. Vol. III. Amphibians and reptiles. Univ. of Florida Press, Gainesville, Florida, USA. Ehrhart, Ll. M., D. A. Bagley & W. E. Redfoot. 2003. Loggerhead turtles in the Atlantic Ocean: geographic distribution, abundance, and population status. 157-174. En: A. B. Bolten & B. E. Witherington (eds.), Loggerhead sea turtle, Smithsonian Institution, USA. Encalada, S. E., K. A. Bjorndal, A. B. Bolten, J. C. Zurita, B. Schroeder, E. Possardt, C. J. Searsand & B. W. Bowen. 1998. Population structure of loggerhead turtle (Caretta caretta) nesting colonies in the Atlantic and Mediterranean as inferred from mitochondrial DNA control region sequences. Mar. Biol. 130: 567-575. Encalada, S. E., P. N. Lahanas, K. A. Bjorndal, A. B. Bolten, M. M. Miyamoto & B. W. Bowens. 1996. Phylogeography and population structure of the Atlantic and Mediterranean green turtle (Chelonia mydas): a mitochondrial DNA control region sequence assessment. Mol. Ecol. 5: 473-483. Engstrom, D. R., B. C. S. Hansen & H. E. Wright Jr. 1990. A possible Younger Dryas record in southeastern Alaska. Science 250: 1383-1385. Espinosa, G., A. Ruiz, J. Azanza, R. Frías, R. Díaz & M. Ramos. 2004. Population genetic from turtle in Cuban shelf using mitochondrial DNA. Rev. Cub. Quím. 16 (3): 317. Evans, K. E., & A. R. Vargas. 1998. Sea turtle egg commercialization in Isla de Canas, Panamá. 45. En: Byles, R. y Y. Fernández (Eds.), Proceeding of the 16th Annual Symposium on Sea Turtle Biology and Conservation, NOAA Tech. Mem. NMFSSEFSC-412: 158. Excoffier, L., & S. Schneider. 1999. Why hunter-gatherer populations do not show signs of Pleistocene demographic expansions. Proc. Natl. Acad. Sci. USA 96: 10597-10602. Excoffier, L. G. Laval & S. Schneider. 2005. Arlequin ver. 3.0: An integrated software package for population genetics data analysis. Evol. Bioinf. Onl. 1: 47-50. REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ Excoffier, L., P. Smouse & J. Quattro. 1992. Analysis of molecular variance inferred from metric distances among DNA haplotypes: Application to human mitochondrial DNA restriction data. Genetics 131: 479-491. Felsenstein, J. 1985. Confidence limits on phylogenies: An approach using the bootstrap. Evolution 39: 783-791. FitzSimmons, N. N., C. Moritz & S. S. Moore. 1995. Conservation and dynamics of Microsatellite loci over 300 million years of marine turtle evolution. Mol. Biol. Evol. 12(3): 432-440. FitzSimmons, N. N., C. Moritz, C. J. Limpus, L. Pop & R. Prince. 1997. Geographic structure of mitochondrial and nuclear gene polymorphisms in Australian green turtle populations and male-biased gene flow. Genetics 147: 1843-1854. FitzSimmons, N. N., J. D. Miller, C. Moritz, C. J. Parmenter, C. J. Limpus & R. Prince. 1996. Comparative genetic structure of green, loggerhead, and flatback populations in Australia based on variable mtDNA and nDNA regions. 25-32. En: B. W. Bowen & Witzell W. N. (eds.), Proceedings of the International Symposium on Sea Turtle Conservation Genetics, NOAA Tech. Mem. NMFS-SEFSC-396: 173. Formia, A., & M. W. Bruford. 2002. Mixed stock analysis of the endangered green turtle (Chelonia mydas) fishery in the Gulf of Guinea, Central Africa. 177-224. En: Formia (Author, Thesis of Doctor of Philosophy), Population and genetic structure of the green turtle (Chelonia mydas) in west and Central Africa; implications for management and conservation, School of Biosciences Cardiff University. Formia, A., B. J. Godley, J.-F. Dontaine & M. W. Bruford. 2002a. Mitochondrial DNA diversity and phylogeography of endangered green turtle (Chelonia mydas) populations in Africa. Cons. Genet. 7: 353-369. Formia, A., M. W. Bruford, B. J. Godley, A. C. Broderick, F. Glen & G. C. Hays. 2002b. Genetic structure across an entire oceanic island turtle rookery: sampling and diversity in Ascension Island green turtles. 136-175. En: Formia (Author, Thesis of Doctor of Philosophy), Population and genetic structure of the green turtle (Chelonia mydas) in west and Central Africa; implications for management and conservation, School of Biosciences Cardiff University. REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ Francisco, A., & B. W. Bowen. 2001. Identification of loggerhead turtle (Caretta caretta) stock structure in the southeastern united status and adjacent regions using nuclear DNA markers. Thesis to the graduate. University of Florida. 71. Francisco, A., A. Bass & B. Bowen. 1999. Genetic Characterization of Loggerhead Turtles (Caretta caretta) Nesting in Volusia County. Department of Fisheries and Aquatic Sciences, Florida, USA. Frazer, N. B. 1983. Survivorship of adult female loggerhead sea turtles, Caretta caretta, nesting on Little Cumberland Island, Georgia, USA. Herpetologica 39: 436-447. Frazer, N. B. 1986. Kemp’s decline: special alarm or general concern? Mar. Turtle Newsl. 37: 5-7. Frazer, N. B., & R. C. Ladner. 1986. A growth curve for green sea turtles, Chelonia mydas, in the U. S. Virgin Island. Copeia 1986: 798-802. Frías, R., A. Ruiz, Y. Pérez & G. Espinosa. 2006. Resolución de secuencias de la región de control de DNA mitocondrial en poblaciones cubanas de Eretmochelys imbricata. Rev. Cub. Quím. XIX (1): ISSN 0258-5595. Fu, Y-X. 1997. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection. Genetics 147: 915-925. Garduño, M., V. Guzmán, E. Miranda, R. Briceño-Dueñas & F. A. Abreu-Grobois. 1999. Increases in hawksbill turtle (Eretmochelys imbricata) nestings in the Yucatán Peninsula, México (1977-1996): data in support of successful conservation? Chel. Cons. Biol. 3(2): 286-295. Gates, D. M. 1993. Climate Change and its Biological Consequences. Sinauer Associates, Inc., Sunderland, Massachusetts, USA. Geldiay, R. 1987. Marine turtles in Turkey. Council of Europe, Convention of European Wildlife and Natural Habitats. Secretariat Memorandum, Appendix IV, 10-11. Strasbourg. Gerosa, G., M. Aureggi, P. Casale & S. V. Yerli. 1998. Green turtle nesting at Akyatan beach Turkey, 1994-1997. Marine. Zoology in the Middle East 24: 45-74. Goldstein D. B., & C. Schlötterer. 1999. Microsatellites Evolution and Applications. Oxford University Press. REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ González-Villaseñor, LI., & D. A. Powers. 1990. Mitochondrial-DNA restriction site polymorphisms in the teleost Fundulus heteroclitus support secondary integration. Evolution 44: 27-37. Graur, D., & W. H. Li. 2000. Fundamentals of Molecular Evolution. Sinauer Associates, Inc., Massachusetts. Green, D. 1993. Growth rates of wild immature green turtles in the Galapagos Islands, Ecuador. J. Herpetol. 27(3): 338. Groombridge, B. 1990. Marine Turtles in the Mediterranean: Distribution, Population Status, Conservation. Council of Europe Environment Conservation and Management Division. Conservation, Nature and Environment Series, No. 48. Strasbourg. Gyory, J., A. J. Mariano & E. H. Ryan. 2005. The Loop Current. Ocean Surface Currents. http://oceancurrents.rsmas.miami.edu/caribbean/loop Current.htm. Hall, T. 1999. BioEdit version 5.0.6. North Carolina State University, Department of Microbiology. Harrison, R. G. 1988. Mitochondrial DNA as a Genetic Marker in Population Evolutionary Biology. Section of Ecology and Systematics Corson Hall, Cornell University. Harrison, S. 1991. Local extinction in a metapopulation context: An empirical evaluation. 73-88. En: M. E. Gilpin & I. Hanski (eds.), Metapopulation dynamics: empirical and theorical investigations. Biol. J. Linn. Soc. 42: 1-336. Hatase, H., M. Kinoshita, T. Bando, N. Kamezaki, K. Sato, Y. Matsuzawa, K. Goto, K. Omuta, Y. Nakashima, H. Takeshita & W. Sakamoto. 2002. Population structure of loggerhead turtles, Caretta caretta, nesting in Japan: bottlenecks on the Pacific population. Mar. Biol. 141: 299-305. Hays, G. C., S. Akesson, A. C. Broderick, F. Glen, B. J. Godley, F. Papi & P. Luschi. 2003. Island – finding ability of marine turtles. Proc. R. Soc. Lond. B (Suppl.) 270: S5-S7. Heppell, S. S., & L. B. Crowder. 1996. Analysis of a fisheries model for harvest of Hawksbill Sea Turtles (Eretmochelys imbricata). Cons. Biol. 10(3): 874-880. Heppell, S. S., L. B. Crowder, D. T. Crouse, S. P. Eppely & N. B. Frazer. 2003. Population models for Atlantic loggerheads : past, present, and future. 255-273. En: A. Bolten & B. Witherington (eds.), The Biology and Conservation of loggerhead sea turtles. Smithsonian Institution Press, Washington D. C. REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ Hilborn, R., T. P. Quinn, D. E. Schindler & D. E. Rogers. 2003. Biocomplexity and fisheries sustainability. Proc. Nat. Acad. Sci. USA 100: 6564-6568. Hillis, D., C. Moritz & B. K. Mable. 1996. Molecular Systematics. Sinauer Associates, Sunderland, Massachusetts, USA. Hilton-Teylor, C. (ed.) 2000. 2000 IUCN Red List of Threatened Species. IUCN, Gland, Switzerland and Cambridge, UK. xviii+ 61 pp. Hirth, H. F. 1980. Some aspects of de nesting behaviour and reproductive biology of sea turtle. Am. Zool. 20: 507. Hirth, H. F. 1997. Synopsis of the biological data on the green turtle, Chelonia mydas (Linnaeus 1758). United States Fish and Wildlife Service Biological Report 97-1. Hopkins-Murphy, S. R., & T. M. Murphy. 1988. Status of the loggerhead turtle in South Carolina. 35-37. En: B. Schroeder (ed.), The Eighth Annual Workshop on Sea Turtle Conservation and Biology, NOAA Technical Memorandum NMFS-SEFSC-214. Hopkins-Murphy, S. R., T. M. Murphy, C. P. Hope, J. W. Coker & M. E. Hoyle. 2001. Population Trends and Nesting Distribution of the Loggerhead (Caretta caretta) in South Carolina 1980-1997. Final Report to the US Fish and Wildlife Service. South Carolina Department of Natural Resources, Charleston, USA. Hughes, J. B., G. C. Daily & P. R. Ehrlich. 1997. Population diversity: its extent and extinction. Science 278: 689-692. Hughes, J. B., G. C. Daily & P. R. Ehrlich. 1997. Population diversity: its extent and extinction. Science 278: 689-692. Ibarra, M. E., R. Díaz, G. Espinosa, J. Azanza, R. Díaz & Colectivo de Estudiantes de la Facultad de Biología. 2005. Informe Técnico con los resultados de la octava temporada del Proyecto Universitario para el Estudio y Conservación de las tortugas marinas en Guanahacabibes. Centro de Investigaciones Marinas, Universidad de La Habana. IUCN. 2002. Hawksbill turtles in the Caribbean region: basic biological characteristics and population status. CITES Wider Caribbean Range State Hawksbill Turtle Dialogue meetings. Johnson, S. 1994. Reproductive ecology of the Florida green turtle (Chelonia mydas). M. Sc. Thesis, University of Florida. Kalinowski, S. T. 2002. How many alleles per locus should be used to estimate genetic distance? Heredity 88: 62-65. REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ Kalinowski, S. T. 2005. Do polymorphic loci require large sample size to estimate genetic distance? Heredity 94: 33-36. Karl, S. A., & B. W. Bowen. 1999. Evolutionary significant units versus geopolitical taxonomy: molecular systematics of an endangered sea turtle (genus Chelonia). Cons. Bio. 13: 990-999. Karl, S. A., & J. C. Avise. 1992. Balancing selection at allozyme loci in oysters; implications from nuclear RFLPs. Science. 256: 100- 102. Kaska, Y. 2000. Genetic Structure of Mediterranean Sea Turtle Populations. Turk J. Zool. 24: 191-197. Kasparek, M., B. J. Godley & A. C. Broderick. 2001. Nesting of the green turtle, Chelonia mydas, in the Mediterranean: a review of status and conservation needs. Zoology in the Middle East 24: 45-74. Keinath, J. A., & J. A. Musick. 1991. Atlantic green turtle Chelonia mydas (Linnaeus). 448450. En: K. Terwilliger (ed.), Endangered and threatened species in Virginia. MacDonald y Woodward Pub. Co., Blacksburg, VA, USA. Kimura, M. 1983. The neutral theory of molecular evolution. Cambridge University Press, USA. Kocher, T. D., W. K. Thomas, A. Meyers, S. V. Edwards, S. Paabo, F. X. Villablanca & A. C. Wilson. 1989. Dynamics of mitochondrial DNA evolution in animals: amplification and sequencing with conserved primers. Proc. Natl. Acad. Sci. USA 86: 6196-6200. Kudrass, H. R., H. Erienkeuser, R. Vollbrecht & W. Weiss. 1991. Global nature of the Younger Dryas coling event inferred from oxygen isotope data from Sulu Sea cores. Nature 349: 406-409. Kumar, S., K. Tamura & M. Nei. 1993. MEGA: molecular evolutionary genetics analysis, ver. 1.01. Pennsylvania State University, University Park. Kumazawa, Y., & M. Nishida. 1999. Complete Mitochondrial DNA Sequences of the Green Turtle and Blue-Tailed Mole Skink: Statistical Evidence for Archosaurian Affinity of Turtles. Mol. Biol. Evol. 16(6): 784-792. Lagueux, C. 2001. Status and distribution of the green turtle, Chelonia mydas, in the Wider Caribbean Region. 32-35. En: K. L. Eckert & F. A. Abreu Grobois (eds.), Proceedings of the Regional Meeting: Marine Turtle Conservation in the Wider Caribbean Region: A REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ Dialogue for Effective Regional Management. Santo Domingo, 16-18 November 1999. WIDECAST, IUCN-MTSG, WWF, and UNEP-CEP. Lahanas, P. N., K. A. Bjorndal, A. B. Bolten, S. E. Encalada, M. M. Miyamoto, R. A. Valverde & B. W. Bowen. 1998. Genetic composition of green turtle (Chelonia mydas) feeding ground population: evidence for multiple origins. Mar. Biol. 130: 345-352. Lahanas, P. N., M. M. Miyamoto, K. A. Bjorndal & A. B. Bolten. 1994. Molecular evolution and population genetics of Greater Caribbean green turtles (Chelonia mydas) as inferred from mitochondrial DNA control region sequences. Genetica 94: 57-66. Lamb. T., & J. C. Avise. 1992. Molecular and population genetic aspects of mitochondrial DNA variability in the diamondback terrapin, Malaclemys terrapin. J. Hered. 83: 262269 Laurent, L., M. N. Bradai, D. A. Hadoud & H. E. Gomati. 1995. Marine Turtle nesting activity assessment on Libyan coasts. Phase 1: survey of the coasts between the Egyptian border and Sirte. Regional Activity Centre for Specially Protected Areas (UNEP), Tunis. Laurent, L., P. Casale, M. N. Bradai, B. J. Godley, G. Gerosa, A. C. Broderick, W. Schroth, B. Schierwater, A. M. Levy, D. Freggi, E. M. Abd El-Mawala, D. A. Hadoud, H. E. Gomati, M. Domingo, M. Hadjichristophorou, L. Kornaraky, F. Demirayak & CH. Gautier. 1998. Molecular resolution of marine turtle stock composition in fishery bycatch: a case study in the Mediterranean. Mol. Ecol. 7: 1529-1542. Le Gall, J. Y., & G. R. Hughes. 1987. Migrations de la tortue verte Chelonia mydas dans L'Océan Indien Sud-Ouest observées à partir des marquages sur les sites de ponte Europa et Tromelin (1970-1985). Amphibia - Reptilia 8: 277-282. Lee, D. C., A. N. Halliday, J. G. Fitton & G. Poli. 1994. Isotopic variations with distance and time in the volcanic islands of the Cameroon line: evidence for a mantle plume origin. Earth and Planetary Science Letters 123: 119-138. Lewis, C. B. 1940. The Cayman Islands and marine turtles. Bull. Inst. Jam. Sci. Ser. 2: 5665. Li, W. H. 1977. Distribution of nucleotide differences between two randomly chosen cistrons in a finite population. Genetics 85: 331-337. Limpus, C. J., & D. G. Walter. 1980. The growth of immature green turtles (Chelonia mydas) under natural conditions. Herpetologica 36: 162-165. REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ Limpus, C. J. 1985. A study of the loggerhead sea turtle, Caretta caretta, in eastern Australia. Ph.D. Dissertation, University of Queensland, St Lucia, Australia. Limpus, C. J. 1992. The hawksbill turtle, Eretmochelys imbricata, in Queensland: population structure within a southern Great Barrier Reef feeding ground. Wild. Res. 19: 489-506. Limpus, C. J., & J. D. Miller. 1993. Family Cheloniidae. 1-16. En: C. J. Glasby, G. J. B. Ross & P. L. Beesley (eds.), Fauna of Australia, vol. 2A, Amphibia & Reptilia., Australian Government Publishing Service, Canberra, Australia. Limpus, C. J., & M. Chaloupka. 1997. Nonparametric regression modelling of green sea turtle growth rates (southern Great Barrier Reef). Mar. Ecol. Prog. Ser. 149: 23-34. Limpus, C. J., J. D. Miller, C. J. Parmenter, D. Reimer, N. McLachlan & R. Webb. 1992. Migration of green (Chelonia mydas) and loggerhead (Caretta caretta) turtles to and from eastern Australian rookeries. Wildl. Res. 19: 347-358. Limpus, C. J., P. Eggler & J. D. Millar. 1994c. Long interval remigration in eastern Australian Chelonia. 85. En: B. Schroeder & B. Witherington (eds.), Proceedings of the Thirteenth Annual Workshop on Sea Turtle Biology and Conservation, NOAA Tech. Memo. NMFS-SEFSC.341, Miami, Florida. Limpus, C. J., P. J. Couper & M. A. Read. 1994b. The green turtle, Chelonia mydas, in Queensland: population structure in a warm temperate feeding area. Mem. Queensland Mus. 35: 139-154. Limpus, C. J., Walker, T. A. & West, J 1994a. Posthatchling specimens and records from the Australian region. 86. En: R. James (ed.), Proceeding Marine Turtle Conservation Workshop, Australian National Parks and Wildlife Service, Canberra, Australia. López, K. G. 2000. Aspectos reproductivos de la tortuga blanca Chelonia mydas en la playa de Las Coloradas Yucatán. Tesis de Maestría. Cinvestav IPN Unidad Mérida, México. Luck, G. W., G. C. Daily & P. R. Ehrlich. 2003. Population diversity and ecosystem services. Trends Eco. Evol. 18: 331-336. Lundelius, E. L. 1987. The North American quaternary sequence. 211-235. En: M. O. Woodburne (ed.), Cenozoic Mammals of North America. Univ. of California Press. Berkeley, CA, USA. REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ Luschi, P., G. C. Hays & F. Papi. 2003. A review of long-distance movements by marine turtles, and the possible role of ocean currents. Oikos 103: 293-302 Margaritoulis, D. 1998. Nesting of the loggerhead sea turtle, Caretta caretta, on the shores of Kiparissia bay, Greece. Mesogee 48: 59- 65. Márquez, R. 1990. FAO Species Catalogue; Sea Turtles of the World. An annotated and illustrated catalogue of the sea turtle species known to date. FAO Fisheries Synopsis 125 (11): 81. Márquez, R., J. Díaz, M. Sánchez, P. Burchfield, A. Leo, M. Carrasco, J. Peña, C. Jiménez & R. Bravo. 1999. Results of the Kemp’s ridley nesting beach conservation efforts in México. Mar. Turtle Newsl. 85: 2-4. Martin, A. P., & S. R. Palumbi. 1993. Body size, metabolic rate, generation time, and the molecular clock. Proc. Natl. Acad. Sci. USA 90: 4087-4091 Mateo, J. 1989. Paisaje. Pp XII 1.2-3. En: Col. Aut., Nuevo Atlas Nacional de Cuba, La Habana, Cuba. Maynard, J., & J. Haigh. 1974. The hitchhiking effect of a favourable gene. Genet. Res. 23: 23-35. Mendoca, M. T., & L. M. Ehrhart. 1982. Activity, population size and structure of immature Chelonia mydas and Caretta caretta in Mosquito Lagoon, Florida. Copeia 1: 161. Meylan, A. B., & M. Donnelly. 1999. Status justification for listing the Hawksbill turtle (Eretmochelys imbricata) as Critically Endangered on the 1996 IUCN Red List of Threatened Animals. Chel. Cons. Biol. 3(2): 200-224. Meylan, A. B. 1982. Sea turtle migration–evidence from tag returns. 91-100. En: K. A. Bjorndal (ed.), Biology and Conservation of Sea Turtles, Smithsonian Institution Press, Washington D. C., USA. Meylan, A. B. 1999a. Status of the Hawksbill turtle (Eretmochelys imbricata) in the Caribbean region. Chel. Cons. Biol. 3(2): 177-184. Meylan, A. B. 1999b. International movements of immature and adult Hawksbill turtles (Eretmochelys imbricata) in the Caribbean region. Chel. Cons. Biol. 3(2): 189-194. Meylan, A. B., B. W. Bowen & J. C. Avise. 1990. A genetic test of the natal homing versus social facilitation models for green turtle migration. Science 248: 724-728. Meylan, A. M., B. Schroeder & A. Mosier. 1994. Marine turtle nesting activity in the state of Florida, 1979-1992. 83. En: K. A. Bjorndal, A. B. Bolten, D. A. Johnson & P. J. Eliazar REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ (eds.), Proceedings of the Fourteenth Annual Symposium on Sea Turtle Biology and Conservation, NOAA Technical Memorandum NMFS-SEFSC-351. Miller, J. D. 1985. Embriology of marine turtles. 269-328. En: C. Gans, F. Billett & P. F. A. Maderson (eds.), Biology of the Reptilia, vol. 14A., John Wiley and Sons, New York. Miller, J. D. 1997. Reproduction in sea turtles. 51-81. En: P. L. Lutz & J. A. Musick (eds.), The biology of sea turtle, CRC Press, N.Y., USA. Mohadin, K. 2000. Sea turtle research and conservation in Suriname: history, constraints and achievements. 5-8. En: Kelle, L. et al. (eds), Proceeding of the 3rd Meeting on the sea turtle of the Guianas. Moll, D. 1983. A proposed origin of nesting location divergent and a shared feeding range in Brazilian green turtle populations. Copeia 1983: 121-125. Moncada, F, E. Carrillo, A. Sanz & G. Nodarse. 1999. Reproduction and nesting of the hawksbill turtle, Eretmochelys imbricata, in the Cuban Archipelago. Chel. Cons. Biol. 3(2): 257-263. Moreira, L. 2001. Estimativa do número de desovas da tartaruga verde-Aruana?, Chelonia mydas (Linnaeus, 1758) da Ilha da trinidade, Espírito santo Brasil. Masters Theis. Universidade Federal do Espírito Santo. Moreira, L., C. Baptistotti, J. Scalfone, J. C. Thomé & A. P. L. S. de Almeida. 1995. Occurrence of Chelonia mydas on the Island of Trinidade, Brazil. Mar. Turtle Newsl. 70: 2. Moritz C., T. Dowling & W. M. Brown. 1987. Evolution of Animal Mitochondrial DNA: Relevance for Population Biology and Systematics. Annu. Rev. Ecol. Syst. 18: 269-292. Moritz, C., & W. M. Brown. 1987. Tandem duplications in animals mitochondrial DNAs: Variation in incidence and gene contents among lizards. Proc. Natl. Acad. Sci. USA 34: 7183-7187. Moritz, C. 1994. Defining “evolutionarily significant units” for conservation. Trends Ecol. Evol. 9: 373-375. Moritz, C., D. Broderick, K. Dethmers, N. FitzSimmons & C. Limpus. 2002. Population genetics of Southeast Asian and Western Pacific green turtles, Chelonia mydas. Final Report to UNEP/CMS. Morrison, A., A. L. Johnson, L. H. Johnson & A. Sugino. 1993. Pathway correcting DNA replication errors in Saccharomyces cerevisiae. EMBO Journal 12: 1467-1473. REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ Mortimer, J. A., & A. Carr. 1987. Reproduction and migration of the Ascension Island green turtle (Chelonia mydas). Copeia 1987: 103-113. Mortimer, J. A., & K. M. Portier. 1989. Reproductive homing and internesting behaviour of the green turtle (Chelonia mydas) at Ascension Island, South Atlantic Ocean. Copeia 4: 962-977 Mortimer, J. A., & M. Donnely (eds.). 2007. IUCN Red List status assessment hawksbill turtle (Eretmochelys imbricata). Marine Turtle Specialist Group Review. Mrosovsky, N. 1983. Conserving Sea Turtles. British Herpetological Society, London, Reino Unido. Musick, J. A., & C. J. Limpus. 1997. Habitat utilization and migration in juvenile sea turtles. 137-164. En: P. L. Lutz & J. A. Musick (eds.), The Biology of Sea Turtles. CRC Press, Boca Raton, Florida. Muss, A., B. R. Robertson, C. A. Stepien, P. Wirtz & B. W. Bowen. 2001. Phylogeography of Ophioblennius: The role of ocean currents and geography in reef fish evolution. Evolution 55: 561- 572. Nachman, M. W., & S. L. Crowell. 2000. Estimate of the mutation rate per nucleotide in humans. Genetics 156: 297-304. Naro-Maciel, E., J. H. Becker, E. H. S. M. Lima, M. A. Marcovaldi & R. DeSalle. 2007. Testing dispersal hypotheses in foraging green sea turtles (Chelonia mydas) of Brazil. J. Hered. 98: 29-39. NRC (National Research Council). 1990. Decline of the Sea turtles. Causes and Prevention. National academy Press, Washington D.C. Nei M., & Y. Imaizumi, 1966, Genetic structure of human populations. II. Differentiation of blood group gene frequencies among isolated populations. Heredity 21: 183-190. Nei, M. 1987. Molecular Evolutionary Genetics. Columbia University Press, New York. Nei, M., & D. Graur. 1984. Extent of protein polymorphism and the neutral mutation theory. Evol. Biol. 17: 73-118. Nei, M., & W. H. Li. 1979. Mathematical model for studying genetic variation in terms of restriction endonucleases. Proc. Natl. Acad. Sci. USA. 76: 5269-5273. NMFS & FWS (National Marine Fisheries Service & Fish and Wildlife Service). 1995. Status Reviews for Sea Turtles Listed Under the Endangered Species Act of 1973. Silver Spring, Maryland, USA. 1-23 REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ Nodarse, G., F. Moncada, A. Meneses & C. Rodríguez. 2000a. Long-term monitoring of nesting of the green sea turtle (Chelonia mydas) in the southwest platform of Cuba. 6869. En: F. A. Abreu-Grobois, R. Briseño-Dueñas, R. Márquez-Millan & L. Sarti-Martinez (eds.), Proceedings of the Eighteenth Annual Symposium on Sea Turtle Biology and Conservation, NOAA Tech. Memo. NMFS-SEFSC-436. Nodarse, G., F. Moncada, A. Meneses & C. Rodríguez. 2000b. Monitoring of loggerhead (Caretta caretta) nesting on the south coast of the Isla de la Juventud. 369. En: A. Mosier, A. Foley & B. Brots (eds.), Proceedings of the twentieth Annual Symposium on Sea turtle Biology and Conservation, NOOA Tech. memo. NMFS- SEFSC- 477. Norman, J. A., C. Moritz & C. J. Limpus. 1994. Mitochondrial DNA control region polymorphisms: genetic markers for ecological studies of marine turtles. Mol. Ecol. 3: 363-373. Ochoa, J., H. Sheinbaum, A. Badan, J. Candela & D. Wilson. 2001. Geostrophy via potential vorticity inversion in the Yucatan Channel. J. Mar. Research 59: 725-747. Okayama, T., R. Díaz., Y. Baba, M. Halim, O. Abe, N. Azeno, & H. Koike. 1999. Genetic diversity of the hawksbill turtle in the Indo/Pacific and Caribbean regions. Chel. Cons. Biol. 3(2): 362-367. Olson, D. B., O. B. Brown & S. R. Emerson. 1983. Gulf Stream frontal statistics from Florida Straits to Cape Hatteras derived from satellite and historical data. J. Geophysical Research 88: 4569-4577. Parsons, J. J. 1962. The Green Turtle and Man. University of Florida Press, Gainesville. Peare, T., & P. G. Parker. 1996. Local genetic structure within two rookeries of Chelonia mydas (the green turtle). Heredity 77: 619-628. Peñaflores-Salazar, C., J. Vasconcelos-Pérez, E. Albavera-Padilla & M. C. Jiménez Quiroz. 2001. Especies sujetas a protección especial. Tortuga golfina. 1001-1021. En: M. A. Cisneros, L. F. Meléndez, E. Zárate, M. T. Gaspar, L. C. López, C. Saucedo & J. Tovar (eds). Sustentabilidad y Pesca Responsable en México. Evaluación y manejo. Publicado en CD. Instituto Nacional de la Pesca/ SEMARNAT. México. Perran, J. 1982. Historical Decline of Loggerhead, Ridley, and Leatherback Sea Turtles. 189-195. En: K. A. Bjorndal (ed.), Biology and Conservation of Sea Turtles. Smithsonian Institution Press, Washington, D.C. Pianka, E. R. 1974. Evolutionary Ecology. New York. Harper and Row. REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ Posada, D., K. A. Crandall & A. R. Templeton. 2000. Geodis: a program for the cladistic nested analysis of the geographical distribution of genetic haplotypes. Mol. Ecol. 9: 487488. Pritchard, P. C. H. & P. Trebbau. 1984. The turtles of Venezuela. Contrib. Herpetol. 2, Society for the Study of Amphibians and Reptiles. Fundación de Internados Rurales, Caracas, Venezuela. Pritchard, P. C. H. 1976. Post-nesting movements of marine turtles (Cheloniidae and Dermochelyidae) tagged in the Guianas. Copeia 1979: 749. Pritchard, P. C. H. 1979. Encyclopedia of Turtles. T. F. H. Publications, Inc. Jersey City, NJ. Ramos-Onsins, S. E., & J. Rozas. 2002. Statistical properties of new neutrality tests against population growth. Mol. Biol. Evol. 19(12): 2092-2100. Razin, A., & D. Riggs. 1980. DNA methylation and gene function. Science 210: 604–610. Reeb, C. A., & J. C Avise. 1990. A genetic discontinuity in a continuously distributed species: mitochondrial DNA in the American oyster, Crassostrea virginica. Genetics 124: 397-406. Reece, J. S, T. A. Castoe & C. L. Parkinson. 2005. Historical perspectives on population genetics and conservation of three marine turtle species. Cons. Gen. 6: 235-251. Rice, W. M. 1989. Analyzing tables of statistical test. Evolution 43: 223-225. Richardson, J. I. 1982. A population model for adult female loggerhead sea turtles (Caretta caretta) nesting in Georgia. Ph.D. Dissertation, Univ. of Georgia, Athens, Georgia. Roberts, M. A., C. J. Anderson, B. Stender, A. Segars, J. D. Whittaker, J. M. Grady & J. M. Quattro. 2005. Estimated contribution of Atlantic Coastal loggerhead turtle nesting populations to offshore feeding aggregations. Cons. Gen. 6: 133-139. Roff, D. A., & P. Bentzen. 1989. The statistical analysis of mitochondrial DNA polymorpisms: X2 and the problem of small samples. Mol. Biol. Evol. 6: 539-545. Rogers, A. R., & H. Harpending. 1992. Population growth makes waves in the distribution of pairwise genetic differences. Mol. Biol. Evol. 9: 552-569. Rohlf, F. J. 1973. Algorithm 76. Hierarchical clustering using the minimum spanning tree. Comp. J. 16: 93-95. Ruiz, A., Y. Pereira, E. Quezada, E. Pérez, M.l González, J. Solano, E. Fonseca, B. Hernández, M. A. González, S. Tobeñas & M. E. Ibarra. 2003. Análisis de la fase final REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ de incubación de Chelonia mydas en Cayo Real, Archipiélago de los Canarreos, Cuba. 182. En: Libro resumen en CD del VI Congreso de Ciencias del Mar, MARCuba, Cuba. Ruiz, G. A. 1994. Sea turtle nesting population at Playa La Flor, Nicaragua: an olive ridley “arribada” beach. En: K. A. Bjorndal, A. B. Bolten, D. A. Johnson & P. J. Eliazar (eds.), Proceeding of the 14th Annual symposium on Sea Turtle Biology and Conservation, NOAA Technical Memorandum NMFS-SEFSC-351: 129. Ryder, O. A. 1986. Species conservation and systematics: the dilemma of subspecies. Trend. Ecol. Evol. 1: 9-10. Saccone, C., M. Attimonelli & E. Sbisá. 1987. Structural element highly preserved during the evolution of the d-loop-containing region in vertebrate mitochondrial DNA. J. Mol. Evol. 26: 205-211. Saitou, N., & M. Nei. 1987. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Mol. Biol. Evol. 4: 406-425. Sang, T.-K., H.-Y. Chang, C.-T. Chen & C.-F. Hui. 1994. Population structure of the Japanese eel, Anguilla japonica. Mol. Biol. Evol. 11: 250-260. Sanger, F., S. Niklen & A. R. Coulson. 1977. DNA sequencing with chain – terminating inhibitors. Proc. Natl. Acad. Sci. USA 74: 5463-5467. Schroth, W., B. Streit & B. Schierwater. 1996. Evolutionary handicap for turtles. Nature, Lond. 384: 521-522. Schulz, J. P. 1982. Status of sea turtle populations nesting in Surinam with note on sea turtles nesting in Guyana and Frech Guyana. 435-438. En: K. A. Bjorndal (ed.), Biology and Conservation of Sea Turtles, Smithsonian Institution Press: Washington, D. C. Seminoff, J. A. (ed.). 2002. Green turtle (Chelonia mydas). Marine Turtle Specialist Group Review Draft. 2002 IUCN Red List status Assessment. Slatkin, M., & N. H. Barton. 1989. A comparison of three indirect methods for the estimating average levels of gene flow. Evolution 43: 1349-1368. Slatkin, M. 1991. Inbreeding coefficients and coalescence times. Genet. Res. Camb. 58: 167-175. Smouse, P. E., J. C. Long & R. R. Sokal. 1986. Multiple regression and correlation extensions of the Mantel Test of matrix correspondence. Syst. Zool. 35: 627-632. Sneath, P. H. A., & R. R. Sokal. 1973. Numerical Taxonomy. W. H. Freeman and Co., San Francisco, USA. REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ Sokal, R. R., & F. J. Rohlf. 1981. Biometry, 2nd Ed. W. H. Freeman and Co., San Francisco, USA. Sole, G. 1994. Migration of the Chelonia mydas population from Aves Island. 283-286. En: K.A. Bjorndal, A. B. Bolten, D. A. Johnson & P.J. Eliazar (eds.), Proceedings of the Fourteenth Annual Symposium on Sea Turtle Biology and Conservation, NOAA Tech. Memo. NMFS-SEFSC-351: 323. Stewart, F. M. 1977. Computer algorithm for obtaining a random set of allele frequencies for a locus in an equilibrium population. Genetics 86: 482-483. Strand, M., T. A. Prolla, R. M. Liskay & T. D. Petes. 1993. Destabilization of traces of simple repetitive DNA in yeast by mutations affecting DNA mismatch repair. Nature 365: 274-276. Tajima, F. 1983. Evolutionary relationship of DNA sequences in finite populations. Genetics 105: 437-460. Tajima, M. 1989. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism. Genetics 123: 585-595. Tajima, M. 1996. The amount of DNA polymorphism maintained in a finite population when the neutral mutation rate varies among sites. Genetics 143: 1457-1465. Tamura, K., & M. Nei. 1993. Estimation of the number of nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees. Mol. Biol. Evol. 11: 261-277. Tamura, K. 1992. Estimation of the number of nucleotide substitutions when there are strong transition – transversion and G+C contein biases. Mol. Biol. Evol. 9: 678-687. Templeton, A. R. 2004. Statistical phylogeography: Methods of evaluating and minimizing inference errors. Mol. Ecol. 13: 613-630. Templeton, A. R., & C. F. Sing. 1993. A cladistic analysis of phenotypic associations with haplotypes inferred from restriction endonuclease mapping. IV. Nested analyses with cladogram uncertainty and recombination. Genetics 134: 659-669. Templeton, A. R., E. Boerwinkle & C. F. Sing. 1987. A cladistic analysis of phenotypic associations with haplotypes inferred from restriction endonuclease mapping and DNA sequence data. I. Basic theory and an analysis of alcohol dehydrogenase activity in Drosophila. Genetics 117: 343-351. REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ Templeton, A. R., K. A. Crandall & C. F. Sing. 1992. A cladistic analysis of phenotypic associations with haplotypes inferred from restriction endonuclease mapping and DNA sequence data. III. Cladogram estimation. Genetics 132: 619-633. Thomas, J., J. Castroviejo & J. A. Raga. 1999. Sea turtles in Myanmar : Past and Present. Mar. Turtle Newsl. 88: 10-11. Thompson, J. D., T. J. Gibson, F. Plewniak, F. Jeanmougin & D. G. Higgins 1997. The ClustalX windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Research 24: 4876-4882. Thorbjarnarson, J. B., S. G. Platt & S. T. Khaing. 2000a. Human use of turtles: a worldwide perspective. 33-84. En: Klemens, M. W. (ed.), Turtle Conservation, Smithsonian Institution Press, Washington D. C. Thorbjarnarson, J. B., S. G. Platt & S. T. Khaing. 2000b. Sea turtles in Myanmar: past and present. Mar. Turtles Newsl. 88: 10-11. Tomczack, M. 2000. An introduction to physical oceanography. UICN. 1996. Red Data Book Gland. http//IntroOc/index.htm. Topal, M. D., & J. R. Fresco. 1976. Complementary base pairing and the origin of substitution mutations. Nature 263: 285-289. TEWG (Turtle Expert Working Group). 1998. An Assessment of the Kemp’s Ridley Sea Turtle (Lepidochelys kempii) and Loggerhead (Caretta caretta) Sea Turtle Populations in the Western North Atlantic. NOAA Technical Memorandum NMFS-SEFSC-409, Miami, USA. TEWG (Turtle Expert Working Group). 2000. Assessment Update for the Kemp’s Ridley and Loggerhead Sea Turtle Populations in the Western North Atlantic. NOAA Technical Memorandum NMFS- SEFSC-444, Miami, USA. U.S. Fish & Wildlife Service, North Florida Field Office. 2002. Loggerhead Sea Turtle (Caretta caretta) [http://www.fws.gov]. Diciembre del 2002. Van Buskirk, J., & L. B. Crowder. 1994. Life- history variation in marine turtles. Copeia 1994: 66-81. Wallace, B. 1991. Coadaptation revisited. J. Hered. 82: 82-95 Wallis, G. P. 1987. Mitochondrial DNA insertion polymorphism and germ line heteroplasmy in the Trituris. cristatus complex. Heredity 58: 229-238. REFERENCIAS BIBLIOGRÁFICAS________________________________________________________________________ Watterson, G. 1975. On the number of segregation sites in genetical models without recombination. Theor. Popul. Biol. 7: 256-276. Wenink, P. W., A. J. Baker & M. G. J. Tilanus. 1994. Mitochondrial control-region sequences in two shorebirds species, the turnstone and the dunlin, and their utility in population genetic studies. Mol. Biol. Evol. 11: 22-31. Wilson, A. C., R. L. Cann, S. M. Carr, M. George, U. B. Gyllensten, K. M. Helm-Bychowski, R. G. Higuchi, S. R. Palumbi, E. M. Prager, R. D. Sage & M. Stoneking. 1985. Mitochondrial DNA and two perspectives on evolutionary genetics. Biol. J. Linn. Soc. 26: 375-400. Witherington, B. E., & N. B. Frazer. 2003. Social and economic aspects of sea turtle conservation. 355-384. En: P. L. Lutz, J. A. Musik & Wyneken (eds.), The biology of sea turtles, Vol II. CRC Press. USA. Zaykin, D. V., & A. I. Pudovkin. 1993. Two programs to estimate significance of X2 values using pseudo-probability tests. J. Hered. 84: 152. Zurita, J. C., R. Herrera & B. Prezas. 1993. Tortugas marinas del Caribe. 735-750. En: S. I. Salazar- Vallejo & N. E. González (eds.), Biodiversidad Marina y Costera de México. Com. Nal. Biodiversidad y CIQRO, México City. AUTOBIBLIOGRAFÍA Publicaciones que forman parte de la tesis: -Espinosa, G., A. Ruiz, J. Azanza, R. Frías, R. Díaz, & M. Ramos. 2004. Population genetic from turtle in Cuban shelf using mitochondrial DNA. Rev. Cub. Quím. 16 (3): 317-318. -Ruiz, A., M. Vega, F.B. Riverón, F.A. Abreu, J. Solano, T. Pérez, E. Pérez, J. Azanza, R. Frías, R. Díaz, M.E. Ibarra y G. Espinosa. 2008a. Estructura genética de poblaciones de Caretta careta en el Gran Caribe y la costa atlántica de Estados Unidos, con énfasis en colonias de anidación del suroeste cubano. Rev. Invest. Mar. 29(2):151-160. -Ruiz, A., E. Pérez, F. B. Riverón, F. A. Abreu, T. Pérez, J. Solano, M. González, R. Frías, R. Díaz y G. Espinosa. 2008b. Caracterización molecular de la región no codificadora del DNA mitocóndrico en Caretta caretta y Chelonia mydas del suroeste de Cuba: Una perspectiva regional. Rev. Invest. Mar. 29(3):pp pendiente. Publicaciones que integrarán contenidos de la tesis: -Ruiz, A., J. Azanza, E. Pérez, F. A. Abreu, F. B. Riverón, M. González, R. Díaz, J. M. Alvarez, M. Jager, M. E. Ibarra, & G. Espinosa (Corresponding author). 2008. Population genetic structure of American Mediterranean green turtles (Chelonia mydas) based on mitochondrial DNA sequences, with an emphasis on rookeries from southwestern Cuba. Conservation Genetics (bajo re-revisión). - Ruiz, A., Y. Pereira, E. Quezada, E. Pérez, M. González, J. Solano, E. Fonseca, B. J. Hernández, M. A. González, S. Tobenas, & M. E. Ibarra. Proceso de anidación de tortuga verde (Chelonia mydas) en Cayo Real, Archipiélago de los Canarreos, Cuba. (en preparación). Otras publicaciones relacionadas con el tema: -Azanza, J., Y. Ruisanchez, M. E. Ibarra, A. Ruiz, & C. Y. Luis. 2006. Indicadores del éxito reproductivo de la tortuga verde (Chelonia mydas) en tres playas de la Península de Guanahacabibes, Pinar del Río, Cuba. Rev. Inv. Mar. 27 (1): 69-79. -Frías, R., A. Ruiz, Y. Pérez, & G. Espinosa. 2006. Resolución de secuencias de la región de control de DNA mitocondrial en poblaciones cubanas de Eretmochelys imbricata. Rev. Cub. Quím. XIX (1): ISSN 0258-5595. -Equipo editorial. 2006. GeoJuvenil, Cuba. Editorial Abril. 165 pp. Secuencias inscritas en el Genbank: -Espinosa, G., J. Azanza, A. Ruiz, M. Jager, J. M. Alvarez, & J. Deutsch. 2003. AJ543730. Chelonia mydas mitochondrial partial D-loop. GenBank (http://ncbi.nlm.nih.gov). -Espinosa, G., J. Azanza, A. Ruiz, M. Jager, J. M. Alvarez, & J. Deutsch. 2003. AJ543732. Chelonia mydas mitochondrial partial D-loop. GenBank (http://ncbi.nlm.nih.gov). -Espinosa, G., J. Azanza, A. Ruiz, M. Jager, J. M. Alvarez, & J. Deutsch. 2003. AJ543735. Chelonia mydas mitochondrial partial D-loop. GenBank (http://ncbi.nlm.nih.gov). Trabajos presentados en eventos científicos relacionados con el tema de tesis: -Espinosa, G., J. Azanza, A. Ruiz, M. Jager, R. Díaz, E. García, & J. M. Álvarez. 2002. Population genetics and molecular evolution of mtDNA non-coding region in Cuban nesting colony of Green Turtle Chelonia mydas. Molecular Evolution Meeting, Sorrento, Italy. Libro de Resúmenes. -Ruiz, A., Y. Pereira, E. Quezada, E. Pérez, M. González, J. Solano, E. Fonseca, B. Hernández, M. A. González, S. Tobenas, & M. E. Ibarra. 2002. Proceso de anidamiento de Chelonia mydas en Cayo Real, Archipiélago de los Canarreos, Cuba. Taller Internacional “Proyecto Universitario para el estudio y conservación de las tortugas marinas, Cuba. -Espinosa, G., A. Ruiz, J. Azanza, A. Ramos, R. Díaz, E. García, & M. E. Ibarra. 2003. Genética de poblaciones de tortugas marinas utilizando un marcador de mtDNA. Primer Taller Nacional de Tortugas Marinas. Centro de Inspección y Control Ambiental, Ministerio de Ciencia, Tecnología y Medio Ambiente. -Ruiz, A., J. Azanza, A. Ramos, R. Díaz, E. García, M. E. Ibarra, & G. Espinosa. 2003. Filogeografía de tortugas marinas utilizando un marcador de mt DNA. Primer Taller Nacional de Tortugas Marinas. Centro de Inspección y Control Ambiental, Ministerio de Ciencia, Tecnología y Medio Ambiente. -Azanza, J, M. E. Ibarra, G. Espinosa, R. Díaz, A. Ruiz, & G. González. 2003. Factores que afectan la conducta de anidación de Chelonia mydas en la Península de Guanahacabibes, Pinar del Río, Cuba. MARCuba, Cuba. Libro de Resúmenes. -Azanza, J., A. Ruiz, M. González, M. E. Ibarra, & G. Espinosa. 2003. Estructura genética de dos colonias de anidación cubanas de Chelonia mydas empleando un marcador de mtDNA. MARCuba, Cuba. Libro de Resúmenes. -Ruiz, A., Y. Pereira, E. Quezada, E. Pérez, M.l González, J. Solano, E. Fonseca, B. Hernández, M. A. González, S. Tobenas, & M. E. Ibarra. 2003. Análisis de la fase final de incubación de Chelonia mydas en Cayo Real, Archipiélago de los Canarreos, Cuba. MARCuba, Cuba. Libro de Resúmenes. -Ruiz, A., J. Azanza, J. Solano, M. E. Ibarra, & G. Espinosa. 2003. Estructura genética de una muestra de colonias de anidación cubanas de Caretta caretta empleando un marcador de mtDNA. MARCuba, Cuba. Libro de Resúmenes. -Ibarra, M. E., J. Azanza, R. Díaz, G. Espinosa, A. Ruiz, F. Moncada, G. Nodarse, & Colectivo de estudiantes de la Facultad de Biología. 2003. Proyecto universitario para el estudio y conservación de las tortugas marinas en Cuba: resultados de 6 años de trabajo. Mesa Redonda: Opciones ambientales para la Industria Pesquera, Cuba. -Azanza, J., Ruiz, A., M. Pumariega, R. Díaz, M. E. Ibarra, & G. Espinosa. 2004. Implicaciones de la genética poblacional de dos muestras cubanas de colonias de anidación de Chelonia mydas en la filogeografía de la especie. 24th Annual Symposium on Sea Turtle Biology and Conservation, Costa Rica. -Ruiz, A., J. Solano, J. Azanza, T. Pérez, R. Díaz, M. E. Ibarra, & G. Espinosa. 2004. Implicaciones de la genética poblacional de una muestra cubana de colonias de anidación de Caretta caretta en la filogeografía de la especie. 24th Annual Symposium on Sea Turtle Biology and Conservation, Costa Rica. -Colectivo de autores. 2005. Tercera reunión nacional del equipo editorial GEO Juvenil Cuba (Este proceso se realizó paralelo al evento nacional ECOJOVEN 2005). -Colectivo de autores. 2005. Proceso de consulta con el equipo editorial GEO Juvenil Cuba, para la edición del informe final del libro. -G. Espinosa, A. Ruiz, J. Azanza, R. Díaz, & M. E. Ibarra. 2005. Dinámica poblacional de tortugas marinas en el archipiélago cubano, empleando marcadores moleculares. Segundo taller internacional para la conservación de las tortugas marinas en Guanahacabibes, Cuba: La participación de las comunidades locales en la conservación. -J. Azanza, M. E. Ibarra, R. Díaz-Fernández, G. González, Y. Comabella, P. González, T. Rodríguez, D. A. Hernández, A. Rosado. I. Hernández, & A. Ruiz. 2005. Avances de la Educación Ambiental del Proyecto Universitario para el estudio y conservación de las tortugas en Cuba. Segundo taller internacional para la conservación de las tortugas marinas en Guanahacabibes, Cuba: La participación de las comunidades locales en la conservación. -J. Azanza, Y. Ruisanchez, A. Ruiz, C. J. Luis, D. Ríos, & M. E. Ibarra. 2005. Repercussion of abiotic factors in nesting process of Chelonia mydas in nesting areas of the west of Cuba. 25th Annual Symposium on Sea Turtle Biology and Conservation, Savanna, Georgia, USA. -J. Azanza, Y. Ruisanchez, A. Ruiz, C. J. Luis, D. Ríos, & M. E. Ibarra. 2005. Reproductive success indicators of green turtles (Chelonia mydas) in Guanahacabibes Peninsula, Pinar del Río, Cuba. 25th Annual Symposium on Sea Turtle Biology and Conservation, Savanna, Georgia, USA. -J. Azanza, M. E. Ibarra, R. Díaz, A. Ruiz, Y. Ruisánchez, & C. Y. Luis. 2006. Reproductive succes of Chelonia mydas in nesting areas of Guanahacabibes Peninsula, Cuba. 26th Annual Symposium on Sea Turtle Biology and Conservation, Crete, Greece. -A. Ruiz, J. Azanza, M. González, R. Díaz, M. E. Ibarra, & G. Espinosa. 2006. Population genetic of rookeries of green turtle in the west of the Cuban Archipelago, using a mtDNA marker. 26th Annual Symposium on Sea Turtle Biology and Conservation, Crete, Greece. -A. Ruiz, & M. E. Ibarra. 2006. Monitoreo de tortugas marinas: una vía para lograr simbiosis entre el turismo y la conservación. Evento Nacional de la Náutica Recreativa, Cuba. -L. R. Bombino, A. Ruiz, & E. González. 2006. Indagación sobre estatus de las tortugas marinas al norte de la provincial de Camagüey, Cuba. MARCuba, Cuba. Libro de Resúmenes. -M. Camps, J. L. Fals, I. Lee, A. Ruiz, J. Azanza, A. M. Rodríguez, G. Espinosa, & M. E. Ibarra. 2006. Caracterización del agregado de Caretta caretta en los pesqueros legales localizados en el archipiélago de Jardines del Rey (Cuba). MARCuba, Cuba. Libro de Resúmenes. -J. L. Fals, I. Lee, M. Camps, A. Ruiz, J. Azanza, A. M. Rodríguez, G. Espinosa, & M. E. Ibarra. 2006. Caracterización del agregado de Chelonia mydas en los pesqueros legales localizados en el archipiélago de Jardines del Rey (Cuba). MARCuba, Cuba. Libro de Resúmenes. -I. Lee, J. L. Fals, M. Camps, A. Ruiz, J. Azanza, A. M. Rodríguez, G. Espinosa, & M. E. Ibarra. 2006. Caracterización del agregado de Eretmochelys imbricata en los pesqueros legales localizados en el archipiélago de Jardines del Rey (Cuba). MARCuba, Cuba. Libro de Resúmenes. -E. Pérez, A. Ruiz, I. Lee, A. Sanz, M. E. Ibarra, & G. Espinosa. 2006. Maduración reproductiva de tortugas marinas en los agregados de pesquería legal del Archipiélago Jardines del Rey (Cuba). MARCuba, Cuba. Libro de Resúmenes. -G. Espinosa, A. Ruiz, R. C. Frías, R. Díaz, F. B. Riverón, A. J. Sosa, M. Vega, I. Lee, & M. E. Ibarra. 2006. Variación temporal de la identidad genética de la población de anidación de Eretmochelys imbricata en Doce Leguas (Archipiélago Jardines de la Reina, Cuba). MARCuba, Cuba. Libro de Resúmenes. -A. Ruiz, F. A. Abreu, R. C. Frías, R. Díaz, F. B. Riverón, A. J. Sosa, M. Vega, I. Lee, M. E. Ibarra, & G. Espinosa. 2006. Contribución temporal de la población de anidación de Eretmochelys imbricata del Archipiélago Jardines de la Reina al agregado pesquero del Archipiélago Jardines del Rey (Cuba). MARCuba, Cuba. Libro de Resúmenes. -N. Navarro, M. Sánchez, M. Canmarno, M. Miyares, L. Fernández & A. Ruiz (corresponding author). 2008. Public Awareness Campaign for the conservation of sea turtles in Cuba. 28th Annual Symposium on Sea Turtle Biology and Conservation, Loreto, Baja California Sur, México. Libro de resúmenes. -Lee, I., J. L. Fals, M. Camps, J. Azanza, E. Pérez, J. Rodríguez, A. M. Rodríguez, G. Espinosa, M. E. Ibarra & A.Ruiz (corresponding author). 2008. Characterization of the fishing stock of sea turtles in Jardines del Rey (Cuba). 28th Annual Symposium on Sea Turtle Biology and Conservation, Loreto, Baja California Sur, México. Libro de resúmenes. -Pérez, E., A. Ruiz (corresponding author), I. Lee, A. Sanz, G. Espinosa & M E. Ibarra. 2008. Relationship among size and reproductive condition in the fishing stock of Eretmochelys imbricata in Jardines del Rey (Cuba). 28th Annual Symposium on Sea Turtle Biology and Conservation, Loreto, Baja California Sur, México. Libro de resúmenes. -Ruiz, A., R. C. Frías, F. A. Abreu, J. Azanza, I. Lee, R. Díaz, M. E. Ibarra & G. Espinosa (corresponding author). 2008. Variación temporal de la población de anidación de Eretmochelys imbricata en el Archipiélago Jardines de la Reina y su contribución temporal al agregado pesquero del Archipiélago Jardines del Rey (Cuba). 28th Annual Symposium on Sea Turtle Biology and Conservation, Loreto, Baja California Sur, México. Libro de resúmenes. -Ruiz, A, M. Vega, F. B. Riverón, F. A. Abreu, J. Solano, T. Pérez, E. Pérez, J. Azanza, R. Frías, R. Díaz, M. E. Ibarra & G. Espinosa (corresponding author). 2008. Genetic structure of loggerhead populations in the Greater Caribbean and Atlantic western shore based on mitochondrial DNA sequences, with an emphasis on rookeries from southwestern Cuba. 28th Annual Symposium on Sea Turtle Biology and Conservation, Loreto, Baja California Sur, México. Libro de resúmenes. -Rodríguez, J., I. Lee, J. L. Fals, M. Camps, E. Pérez & A. Ruiz (autor por correspondencia). 2008. ¿Las especies de tortugas marinas constituyen un solo recurso pesquero?. VIII Congreso Latinoamericano de Herpetología, Varadero, Cuba. Libro de Resúmenes. -Rodríguez, J., I. Lee, J. L. Fals, M. Camps, E. Pérez & A. Ruiz (autor por correspondencia). 2008. Fluctuación temporal de las frecuencias de tortugas marinas (Caretta caretta, Chelonia mydas y Eretmochelys imbricata) en las capturas de la zona D (Jardines del Rey, Cuba). VIII Congreso Latinoamericano de Herpetología, Varadero, Cuba. Libro de Resúmenes. -Lee,I., M. Navarro, E. Pérez, R. Ramos, G. González & A. Ruiz. 2008. Costo- beneficio del uso histórico de las tortugas marinas en Nuevitas (Camagüey), Cuba. VIII Congreso Latinoamericano de Herpetología, Varadero, Cuba. Libro de Resúmenes. -Lee, I., J. L. Fals, M. Camps, E. Pérez, J. Rodríguez & A. Ruiz (autor por correspondencia). 2008. Características fenotípicas de las capturas históricas de tortugas marinas (Caretta caretta, Chelonia mydas y Eretmochelys imbricata) en la zona d (Jardines del Rey, Cuba). VIII Congreso Latinoamericano de Herpetología, Varadero, Cuba. Libro de Resúmenes. -Ruiz, A., F. A. Abreu, G. Ramírez & G. Espinosa. 2008. Implicaciones del número de poblaciones y del tamaño censal en la estimación de contribuciones de fuentes a sumideros de tortugas marinas. VIII Congreso Latinoamericano de Herpetología, Varadero, Cuba. Libro de Resúmenes. -Ruiz, A., F. B. Riverón, T. Pérez, F. A. Abreu, R. Díaz & Georgina Espinosa López (autor por correspondencia). 2008. Filogenia y filogeografía de tortugas marinas del archipiélago cubano en un contexto regional. VIII Congreso Latinoamericano de Herpetología, Varadero, Cuba. Libro de Resúmenes. -Ruiz, A, F. B. Riverón, M. Vega, M. González, J. Solano, R. Frías, E. Pérez, F. A. Abreu, R. Díaz & G. Espinosa (autor por correspondencia). 2008. Genética poblacional de tortugas marinas del archipiélago cubano en un contexto regional. VIII Congreso Latinoamericano de Herpetología, Varadero, Cuba. Libro de Resúmenes. -Lee, I., J. L. Fals, M. Camps, E. Pérez, J. Rodríguez & A. Ruiz (autor por correspondencia). 2008. Características fenotípicas de las capturas históricas de tortugas marinas (Caretta caretta, Chelonia mydas y Eretmochelys imbricata) en la zona d (Jardines del Rey, Cuba). VIII Congreso Latinoamericano de Herpetología, Varadero, Cuba. -Lee,I., M. Navarro, E. Pérez, R. Ramos, G. González & A. Ruiz. 2008. Costo- beneficio del uso histórico de las tortugas marinas en Nuevitas (Camagüey), Cuba. VIII Congreso Latinoamericano de Herpetología, Varadero, Cuba. -Rodríguez, J., I. Lee, J. L. Fals, M. Camps, E. Pérez & A. Ruiz (autor por correspondencia). 2008. Fluctuación temporal de las frecuencias de tortugas marinas (Caretta caretta, Chelonia mydas y Eretmochelys imbricata) en las capturas de la zona D (Jardines del Rey, Cuba). VIII Congreso Latinoamericano de Herpetología, Varadero, Cuba. -Rodríguez, J., I. Lee, J. L. Fals, M. Camps, E. Pérez & A. Ruiz (autor por correspondencia). 2008. ¿Las especies de tortugas marinas constituyen un solo recurso pesquero?. VIII Congreso Latinoamericano de Herpetología, Varadero, Cuba. -Frias, R. C., G. Espinosa, E. Pérez, F. B. Riverón, F. A. Abreu, T. Pérez, J. Solano, M. González, R. Díaz & A. Ruiz (autor por correspondencia). 2008. Secuencias parciales de la región no codificadora del DNA mitocóndrico en Caretta caretta, Chelonia mydas y Eretmochelys imbricata. VIII Congreso Latinoamericano de Herpetología, Varadero, Cuba. -Espinosa, G., A. Ruiz, F. A. Abreu, R. Frias, J. Azanza, I. Lee, R. Díaz, M. E. Ibarra, G. Ramírez, R. Briceño. 2008. El DNA mitocóndrico como marcador molecular para estimar las contribuciones de fuentes a sumideros en la tortuga carey (Eretmochelys imbricata). VIII Congreso Latinoamericano de Herpetología, Varadero, Cuba. -Pérez, E., I. Lee, N. Almaguer, A. Sanz, G. Espinosa & A. Ruiz (autor por correspondencia). Eretmochelys 2008. imbricata Morfofisiología (Testudinata: folicular comparativa Cheloniidae), Alligator en reptiles: mississipiensis (Crocodylia: Alligatoridae) y Anolis porcatus (Squamata: Polychrotidae). VIII Congreso Latinoamericano de Herpetología, Varadero, Cuba. -Pérez, E., I. Lee, N. Almaguer, A. Sanz, G. Espinosa & A. Ruiz (autor por correspondencia). 2008 ¿La presencia de espermatogénesis es sinónimo de madurez sexual en tortugas Herpetología, Varadero, Cuba. marinas? VIII Congreso Latinoamericano de