Tema 10. Regulación de la expresión génica
Anuncio

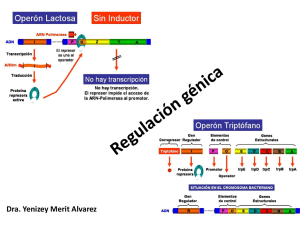
Tema 10. Regulación de la expresión génica Genética CC.MM. Contenidos • Expresión génica en procariotas • Expresión génica en eucariotas • Genética del desarrollo 1 Regulación de la expresión génica en procariotas La lógica de la regulación génica • Una propiedad fundamental de los seres vivos es su capacidad para adaptarse rápidamente al ambiente mediante la activación o inactivación de genes. • Esta habilidad permite que las células de un organismo puedan sintetizar proteínas en el momento y lugar precisos. • Esto evita un derroche energético, pues no es preciso sintetizar todas las proteínas que una célula es capaz de producir, sino únicamente las que se necesitan en cada momento. – Genes regulados: Aquellos cuya actividad es controlada en respuesta a las necesidades de una célula u organismo. – Genes constitutivos: Son los genes que están siempre activos, con independencia de las condiciones ambientales. 2 Regulación génica en procariotas • La actividad génica se controla mediante la regulación de la transcripción. • Proteínas reguladoras interaccionan con secuencias reguladoras del gen (operadores) para inducir o reprimir la expresión génica. – Control positivo: La unión de una proteína activadora induce la expresión génica. – Control negativo: La unión de una proteína represora impide la expresión génica. Regulación génica en procariotas Control positivo Control negativo • Las proteínas reguladoras existen en dos conformaciones alternativas. Sólo en una de ellas pueden unirse al DNA. • El cambio de una conformación a otra (cambio alostérico) es debido a la unión de sustancias llamadas efectores. 3 Regulación génica en procariotas • Los genes que codifican proteínas con funciones relacionadas se suelen organizar en operones. Un operón es una unidad genética de expresión coordinada de genes. • El operón está constituido por un grupo de genes estructurales adyacentes que se transcriben juntos en un mismo mRNA y cuya regulación está determinada por un elemento de control de la transcripción común. Metabolismo de la lactosa en E. coli • Reacciones catalizadas por la β-galactosidasa y la lactosa permeasa Cuando la lactosa es la única fuente de carbono, tres enzimas son sintetizadas de forma simultánea: – β-galactosidasa: Hidroliza la lactosa en glucosa y galactosa. También determina la transformación de lactosa en alolactosa. – Lactosa permeasa: Se localiza en la membrana citoplasmática y transporta la lactosa al interior de la célula. – Transacetilasa: Su función no es bien conocida. • En presencia de glucosa, E. coli sólo contiene unas 3 moléculas de β-galactosidasa, pero este número se incrementa 1000 veces cuando la lactosa es la única fuente de carbono. 4 El operón lactosa (lac) • • • Promotor (Plac+). Operador (lacO+). Tres genes estructurales: β-galactosidasa (lacZ+), lactosa permeasa (lacY+), transacetilasa (lacA+). • Gen regulador lacI+. Gen constitutivo. Posee su propio promotor y terminador. Se expresa débilmente produciendo unas pocas moléculas de proteína represora. El operón lac: ausencia lactosa • • • Control negativo. El gen regulador lacI+ codifica una proteína represora que se une al operador (lacO+) e impide la transcripción (la RNA polimerasa se une al promotor, pero no puede iniciar la transcripción). El represor puede liberarse eventualmente durante cortos periodos de tiempo, permitiendo que se produzcan unas pocas moléculas de enzimas. 5 El operón lac: presencia de sólo lactosa • • • • Algunas moléculas de lactosa se transforman en alolactosa por la β-galactosidasa. La alolactosa (efector) se une al represor, que modifica su conformación y se libera. Los represores de nueva síntesis son también modificados y no pueden unirse. Se inicia la síntesis de un mRNA policistrónico a partir del cual se forman los enzimas. El operón lac: control por catabolito • Si coexisten la lactosa y la glucosa, el operón lac se expresa a un bajo nivel hasta que se utiliza toda la glucosa (la fuente de energía más eficiente). • Control positivo debido a la proteína activadora CAP. • Si sólo hay lactosa, existe mucho AMP cíclico (cAMP). El cAMP (efector) se une al activador CAP, y el complejo CAP-cAMP estimula la transcripción al unirse al promotor. • La presencia de glucosa y su subsecuente degradacion determinan poco cAMP. El activador CAP no se une al promotor y hay una escasa transcripción del operón lac. 6 El operón lac: resumen • En ausencia de lactosa, el represor se une al operador con independencia de los niveles de cAMP: transcripción reprimida. • En presencia de lactosa y glucosa, el represor no se puede unir al operador. Sin embargo, sólo se produce una pequeña cantidad de mRNA: la degradación de glucosa hace descender el cAMP, y no se puede formar el complejo cAMP-CAP para estimular la transcripción. • En presencia de sólo lactosa, el represor no se puede unir al operador, los niveles de cAMP son altos, y el complejo cAMPCAP se une al promotor produciendo grandes cantidades de mRNA. El operón triptófano (trp) • • • • • • • • • Control negativo. 5 genes estructurales (trpA, trpB, trpC, trpD, trpE). promotor operador. Región líder (trpL), que incluye un sitio atenuador (att). Gen regulador trpR. Dos sistemas de control: Cualitativo (interacción represor-operador). Cuantitativo (atenuación. Actúa una vez iniciada la transcripción). 7 El operón trp: interacción operador-represor • El gen regulador trpR codifica una proteína represora inactiva (aporrepresor) que se activa al unirse con el triptófano (efector). • Ausencia de triptófano: – El aporrepresor no se une al operador: Transcripción del operón trp y síntesis de triptófano • Presencia de triptófano: – El aporrepresor se activa al combinarse con el triptófano, uniéndose al operador. Se bloquea la transcripción del operón trp reprimiendo la síntesis de triptófano El operón trp: atenuación • • • Atenuación: controla el número de transcritos completos del operón trp, haciéndolo inversamente proporcional a la cantidad de triptófano. La región líder codifica un pequeño polipéptido (con dos codones triptófano antes del STOP) y contiene 4 regiones complementarias que pueden formar bucles que constituyen señales de transcripción. Inmediatamente después del inicio de la transcripción, la región 1 y 2 forma un bucle: señal de breve pausa que permite la unión del ribosoma al RNA. La traducción ocurre inmediatamente después de la RNA polimerasa. 8 El operón trp: atenuación Casi ausencia de triptófano: • • • El ribosoma se para al llegar a los codones trp y cubre la región 1. Regiones 2 y 3 forman un bucle: señal de antiterminación. Se transcriben todos los genes. Presencia escasa de triptófano: • • • El ribosoma se desplaza hasta el codón STOP, cubriendo las regiones 1 y2. Regiones 3 y 4 forman un bucle: señal de terminación Se interrumpe la transcripción antes de llegar a los genes estructurales. Regulación de la expresión génica en eucariotas 9 Control de la expresión génica en eucariotas • Eucariotas carecen de operones • Regulación más compleja: – Respuesta rápida para adaptarse a cambios ambientales – Respuesta a largo plazo para regular los procesos de desarrollo y diferenciación celular • Varios niveles de control de la expresión: – – – – – Transcripción del mRNA Procesamiento del mRNA Trasporte del mRNA Degradación del mRNA Traducción y degradación proteica Control transcripcional • Control de la transcripción del mRNA: El más importante. Determina si un gen se transcribe, cómo se transcribe, y cuánto se transcribe. • Secuencias reguladoras: intentificadoras (activan la transcripción) y silenciadoras (reprimen la transcripción). Actúan en combinación con los factores de transcripción. • Control positivo y negativo 10 Control transcripcional: cambios cromosómicos • La regiones cromosómicas que son transcripcionalmente activas están menos condensadas que las regiones transcripcionalmente inactivas. • La zonas promotoras de los genes que se están transcribiendo también presentan un menor grado de empaquetamiento cromosómico • Las histonas de los nucleosomas pueden reprimir la transcripción de los genes al cubrir las regiones promotoras (particularmente la caja TATA), impidiendo la unión de las proteínas reguladoras y factores de transcripción necesarios para iniciar la transcripción. • Los genes podrían activarse mediante la unión de proteínas activadoras a secuencias intensificadoras, que desintegrarían el nucleosoma permitiendo así el acceso de las proteínas reguladoras. Control transcripcional: hormonas • Las hormonas son moléculas efectoras producidas por ciertos tipos celulares y que producen una respuesta fisiológica en otras células. • Cada tipo de hormona actúa sobre un tipo determinado de células, gracias a la unión con receptores específicos. • Constituyen un sistema de regulación de respuesta rápida. 11 Control transcripcional: hormonas • Proteínas inducidas por esteroides: A excepción de la hormona esteroide glucocorticode, los receptores de esteroides se localizan en un número reducido de tejidos. Control transcripcional: hormonas • Los genes regulados por una misma hormona esteroide poseen una secuencia común, llamada elemento de unión a la hormona esteroide (HRE), a la que se une el complejo formado por la unión de la hormona y su receptor. • Los elementos HRE se localizan en las secuencias intensificadoras, a menudo en copias múltiples. • La misma hormona esteroide puede activar distintos tipos de genes. • La expresión génica en plantas también está regulada en parte por hormonas 12 Control transcripcional: metilación del DNA • El DNA de muchos eucariotas es metilado después de la replicación en un cierto número de bases, la mayoría de las cuales son citosinas. El enzima DNA metilasa cataliza la transformación de citosina en 5metilcitosina (5mC). • El porcentaje de 5mC es muy variable en eucariotas, y aumenta con la complejidad de los organismos (3% en mamíferos, pero casi inexistente en Drosophila). • La distribución de 5mC no es aleatoria: La mayoría se encuentra en la secuencia CG (90% en mamíferos). • Los genes más metilados se expresan menos. La metilación podría reprimir la expresión génica, o ser el resultado de un bajo nivel de expresión. Control del procesamiento del mRNA calcitonina • Regula la producción de mRNAs maduros a partir de moléculas de premRNA: proteínas codificadas por el mismo gen pueden diferir en su estructura y función. • Poliadenilación alternativa: Distintas señales de poliadenilación determinan diferentes premRNAs • Splicing alternativo: Formas alternativas de eliminar intrones generan distintos mRNA maduros 13 Control del procesamiento del mRNA • El splicing alternativo desempeña un papel fundamental en la determinación del sexo en Drosophila • Modelo de regulación en cascada: El distinto procesamiento del pre-mRNA del gen sex-lethal en machos y hembras, determina la forma en la que se procesan los pre-mRNAs de los genes tra y dsx, y que finalmente provocan la diferenciación somática como machos o hembras. Control del transporte del mRNA • Muchos de los pre-mRNAs nunca abandonan el núcleo celular • Modelo de retención por el spliceosoma: – El pre-mRNA es retenido en el núcleo hasta que finaliza su procesamiento en los spliceosomas. – El mRNA maduro se libera del spliceosoma y atraviesa la membrana nuclear hacia el citoplasma. La presencia de la caperuza 5’ , y posiblemente de señales especícas, promueven la salida del mRNA del núcleo. 14 Control de la traducción del mRNA • Los ribosomas pueden seleccionar y traducir mRNAs específicos. • El mecanismo no es bien conocido. – Ciertas proteínas podrían asociarse a los mRNAs para evitar su degradación e impedir su traducción. – La cola poli(A) promueve la iniciación de la traducción y también podría jugar un papel importante, pues es más corta en los mRNAs inactivos que activos. Un secuencia específica en el extremo 3’ del mRNA podría estar implicada en el acortamiento de la cola poli(A). Control de la degradación del mRNA • Todos los RNAs son degradados una vez en el citoplasma. Su estabilidad es variable: – rRNA y tRNA son muy estables – mRNA: Algunos se degradan en minutos, mientras que otros duran meses • La degradación del mRNA es un mecanismo muy importante de regulación de la expresión génica en eucariotas. Según los casos, puede precisar o no del acortamiento de la cola poli(A) antes de la eliminación de la caperuza 5´, tras lo cual el mRNA es degradado por las exonucleasas 5’-3’. • La estabilidad de los mRNAs puede cambiar en respuesta a señales reguladoras. 15 Control de la degradación del mRNA Control de la degradación proteica • Determinan la vida media de una proteína. • En eucariotas, la degradación de proteínas precisa de la unión del cofactor proteico ubiquitina, que las marca para su proteolisis. • El aminoácido N-terminal determina la velocidad con la que se unen las moléculas de ubiquitina a la proteína y, por tanto, fija la vida media de la proteína. 16 Genética del desarrollo Genética del desarrollo • Los eucariotas complejos contienen múltiples tipos de células, tejidos y órganos, aunque todas las células de un mismo organismo contienen un mismo genoma. • Diferenciación: Formación de diferentes tipos de células, tejidos y órganos mediante la regulación de la expresión de los genes. • Desarrollo: Es un programa de actividades genéticas sucesivas en el que se integran diversos subprogramas de la diferenciación celular. • Los procesos de desarrollo y diferenciación son el resultado de un patrón programado de activación y represión de la expresión génica 17 Actividad genómica en eucariotas multicelulares • Un 20-40% del genoma de eucariotas es altamente repetitivo, mientras que el DNA restante es moderadamente repetitivo o de copia única. • Sólo una pequeña fracción del DNA se transcribe en un momento dado para producir proteínas (6% en erizos). • Se desconoce la función del DNA que nunca se transcribe. Algunos autores defienden que se trata de DNA basura sin función alguna, aunque también se ha propuesto que podría desempeñar un papel regulador aún por determinar. Constancia del DNA genómico durante el desarrollo • El genoma de cualquier célula animal o vegetal contiene el mismo DNA que el cigoto del cual deriva. • Distintos experimentos en plantas han permitido demostrar que células somáticas aisladas poseen la capacidad de generar un nuevo individuo, demostrando así que el DNA no cambia durante el desarrollo. 18 Constancia del DNA genómico durante el desarrollo • La clonación en 1997 de la oveja Dolly a partir del núcleo de una célula adulta confirmó que también en los animales cualquier célula es capaz de originar un nuevo individuo. • El hecho de que cualquier núcleo de una célula somática adulta contenga toda la información necesaria para dirigir la formación de un nuevo individuo desde el principio se conoce como totipotencia. Actividad génica diferencial entre tejidos durante el desarrollo • La hemoglobina humana esta formada por cuatro polipéptidos. Se producen distintos tipos de hemoglobinas a lo largo del desarrollo como resultado de la activación o inactivación de genes que codifican diferentes globinas. – Embrión: (2ζ + 2ε) – Feto: (2α + 2γ ) – Adultos: (2α + 2δ ) • Los genes están dispuestos en el cromosoma en el mismo orden en el que se expresan. 19 Bases genéticas de la diversidad de anticuerpos • Cada linfocito B sintetiza un único tipo de anticuerpo que es específico de cada antígeno. • La unión antígeno-anticuerpo determina la proliferación del linfocito B que lo sintetiza (selección clonal). • Anticuerpos: – Formados por dos cadenas pesadas idénticas (H) y dos cadenas ligeras idénticas (L) unidas por puentes disulfuro. – Cada cadena posee una parte constante (CH, CL) y otra variable (VH, VL) que es la que se une al antígeno. • Los mamíferos sintetizan 106-108 tipos de anticuerpos, que surgen por recombinación somática durante el desarrollo de los linfocitos B. Anticuerpos: recombinación de la cadena ligera • • • En las células germinales, los genes que determinan la cadena ligera están formados por pequeños segmentos dispuestos en tandem. Existen tres tipos de segmentos, que codifican las regiones variables (Vk), de unión (Jk) y constante (Ck). Se necesita un segmento de cada tipo para sintetizar una cadena ligera funcional. Durante el desarrollo del linfocito B, la recombinación somática determina que uno de los segmentos Vk se combine al azar con un segmento de la región Jx y el segmento Ck En el ratón: 350 x 4 x 1 =1400 cadenas ligeras posibles 20 Anticuerpos: recombinación de la cadena pesada • • • • Los genes que determinan la cadena pesada también están formados por segmentos pertenecientes a la región variable (VH), unión (Jx) y constante (CK). Poseen un región adicional (DK) que incrementa la diversidad. Durante el desarrollo, los segmentos de cada región se unen al azar por recombinación somática. En ratón: 500 x 12 x 4 = 24000 cadenas pesadas posibles En total: 1400 x 24000 = 33.600.000 moléculas posibles de anticuerpo. Regulación genética del desarrollo: Drosophila • Las estructuras corporales son resultado del establecimiento de gradientes moleculares a lo largo de los ejes antero-posterior y dorsoventral en el huevo, que subsecuentemente determinan la formación de compartimentos en el embrión que, a su vez, determinan los segmentos que forman el adulto. • Genes de efecto materno: determinan los gradientes en el huevo. Genes de segmentación: dividen el embrión en parasegmentos (no coinciden exactamente con los segmentos visibles en el adulto). Genes homeóticos: especifican la identidad de cada segmento en el adulto. • • 21 Regulación genética del desarrollo: Drosophila Genes homeóticos • • • • • • Especifican la identidad de cada segmento en el adulto. Complejo Antennapedia (ANT-C): formado por 5 genes que determinan la región anterior. Complejo Bithorax (BX-C): formado por 3 genes que determinan la región posterior. Todos estos genes comparten una secuencia muy similar de 180 pb, llamada secuencia homeobox. La región del polipéptido que codifican se llama homeodominio, y es la que se une al DNA de los genes que regulan. La secuencia homeobox se ha encontrado en muchos genes reguladores de Drosophila y otras especies. Forman la familia homeobox o Hox, compuesta por genes muy conservados en la evolución. Los genes homeóticos se encuentran en la mayoría de los grupos animales. Regulación genética del desarrollo: Drosophila Mutantes homeóticos Mutaciones Antennapedia • Mutaciones Bithorax Las mutaciones en genes homeóticos pueden determinar que se formen estructuras corporales distintas a las que se originarían normalmente. Muchas veces son letales. 22